Method Article
Cómo cuantificar la fracción de proteínas fluorescentes fotoactivado a granel y en células vivas
En este artículo
Resumen
Aquí, presentamos un protocolo que implica genéticamente acoplado espectralmente distinto photoactivatable y proteínas fluorescentes. Estas quimeras de la proteína fluorescente permiten la cuantificación de la fracción PA-FP que es fotoactivado a ser fluorescentes, es decir, la eficacia de la fotoactivación. El protocolo revela que diversos modos de fotoactivación rendimiento fotoactivación diferentes eficiencias.
Resumen
Photoactivatable y - proteínas fluorescentes convertibles (PA-FPs) se han utilizado en microscopía de fluorescencia células vivas para el análisis de la dinámica de las células y conjuntos de proteínas. Hasta ahora, ningún método ha estado disponible para cuantificar a granel y en vivo las células expresan cuántos de los PA-FPs son fotoactivado a fluorescencia.
Aquí, presentamos un protocolo de reglas internas, es decir, genéticamente acoplado espectralmente distintos (photoactivatable) proteínas fluorescentes, a ratiometrically cuantificar la fracción de FPs PA todas expresadas en una célula que se encienden al ser fluorescente. Mediante este protocolo, se muestra que diferentes modos de fotoactivación rindieron fotoactivación diferentes eficiencias. Fotoactivación de alta potencia corta con un láser confocal de barrido microscopio (CLSM) dio lugar a hasta cuatro veces inferior fotoactivación eficacia que cientos de exposiciones de bajo nivel aplicadas por CLSM o un pulso corto aplicado por iluminación de campo amplio. Mientras que el protocolo ha sido ejemplificado aquí para GFP (PA-) y (PA) cereza, puede en principio ser aplicado a cualquier par de proteína fluorescente espectralmente distintos photoactivatable o photoconvertible y cualquier montaje experimental.
Introducción
En 2002, la primera photoactivatable términos generales aplicable (PA-GFP1) y photoconvertible (Kaede2) proteínas fluorescentes fueron descritas. Estas proteínas fluorescentes ópticos resaltador cambian sus propiedades espectrales sobre la irradiación con luz UV, es decir, se convierten en brillantes (proteínas fluorescentes photoactivatable, es decir, PA-FPs), o cambiar su color (photoconvertible FPs). Hasta la fecha, varios reversibles e irreversibles photoactivatable y photoconvertible proteínas fluorescentes han sido desarrolladas3,4. En estudios de conjunto o a granel, marcadores ópticos se han utilizado para estudiar la dinámica de proteínas o células enteras y la conectividad de los compartimentos subcelulares. Además, marcadores ópticos conectados una sola molécula base superresolution técnicas como Palma5 y FPALM6de imagen.
Aunque la fotoquímica procesos durante la fotoactivación o - conversión se han descrito muchos marcadores ópticos y estructuras cristalográficas incluso antes y después de la fotoactivación / - conversión se hicieron disponibles7 , 8, el mecanismofísico subyacente de foto de fotoactivación y - la conversión no se entiende totalmente. Además, hasta ahora sólo crudas estimaciones existen de la eficacia de fotoactivación y - conversión, es decir, la fracción de proteínas fluorescentes expresada que es realmente photoconverted o fotoactivado a ser fluorescente. In vitro los estudios ensemble han divulgado cuantificar el cambio en los espectros de absorción y la cantidad de nativos y activa la proteína en un gel9,10,11.
Aquí, presentamos un protocolo de quimeras de la proteína fluorescente para evaluar la fracción de proteínas fluorescentes fotoactivado a granel y en células vivas. Cuando se trabaja con proteínas fluorescentes genéticamente codificadas, la cantidad absoluta de proteínas expresadas varía de célula a célula y se desconoce. Si una célula expresando una FP PA muestra una señal más brillante después de la fotoactivación de otra célula, no puede ser distinguido si esta señal más brillante se debe a la mayor expresión de la PA-FP o una fotoactivación más eficiente de la PA-FP. Para estandarizar el nivel de expresión en las células, se introducen a reglas internas de genéticamente acopladas espectralmente distintas proteínas fluorescentes. Por el acoplador de la información genética de una proteína fluorescente de photoactivatable a un espectral distinta de proteínas fluorescentes, reglas internas se crean todavía se expresará en una cantidad total desconocida pero en una cantidad relativa fija y conocida de 1: 1 esta estrategia permite la caracterización cuantitativa de los diferentes esquemas de luz UV fotoactivación, es decir, la evaluación de la cantidad relativa de PA-FPs que puede ser fotoactivado con diferentes modos de fotoactivación y permite así Definir esquemas de fotoactivación que son más efectivos que otros. Además, esta estrategia permite en principio la evaluación de la cuantificación absoluta de la fracción de fotoactivado PA-FP. Para ello, es importante darse cuenta de que los estudios presentados de conjunto basada en la intensidad que hace que el análisis más complejo de lo dispuesto en el presente Protocolo. Parámetros de determinación de la medida intensidad fluorescente, brillo molecular es decir, diferentes, absorbancia y espectros de emisión y efectos de traste, necesitan ser considerados al comparar intensidades de fluorescencia de diferentes proteínas fluorescentes.
La cuantificación de basada en intensidad radiométrica presentados de fotoactivación eficiencia se ejemplifica para PA-GFP y PA-cereza en células vivas, pero en principio ampliamente aplicable y puede ser utilizada para cualquier proteína fluorescente photoactivatable bajo cualquier condición experimental.
Protocolo
1. construcción de plásmido
- Generar puntas de prueba de fusión de dos colores. Utilizar un vector de expresión de células de mamífero (véase Tabla de materiales) en la que mCherry112 y13 de la PA-mCherry1 se han insertado con la restricción de sitios africanas y BsrGI.
- Orden personalizado oligo-nucleótidos para amplificar las variantes monoméricas de eGFP y que contiene la mutación de A206K, es decir, mEGFP y PA-mEGFP14 sin un codón de parada como un fragmento de BamHI SalI PA-eGFP. Utilice la cartilla del N-terminal 5'-AAT TAA CAG TCG ACG ATG GTG AGC AAG GGC GAG G 3' y el C-terminal primer 5'-AAT ATA TGG ATC CCG CTT GTA CAG CTC GTC gato GC 3' e inserte este fragmento de BamHI SalI en el sitio de clonación múltiple del vector de expresión. Esto creará al vinculador RNPPV de cinco aminoácidos entre la proteína fluorescente verde y rojo. Nos referiremos a las quimeras de fluoróforo como GFP, cereza, PA-GFP, cereza y GFP — PA-cereza para el resto de este artículo.
Nota: El acoplamiento de fluoróforos y creación de reglas internas para evaluar la eficacia de la fotoactivación se pueden hacer con cualquier par fluoróforo espectralmente distintos, por ejemplo superfolder GFP PA 15/ 16de PA-TagRFP, etc. Muchas de las proteínas fluorescentes de photoactivatable son derivados de GFP. Por lo tanto, la secuencia del primer señalada anteriormente puede utilizarse para muchas proteínas fluorescentes para construir una quimera fluoróforo en un vector de expresión de células de mamífero.
2. cultura y transfección de la célula
- Utilizar cualquier línea de célula estándar como HeLa, NRK, Cos-7 o una línea celular específica para experimentos específicos de fotoactivación. Uso de DMEM (suplementado con suero bovino fetal 10% y la glutamina 2 mM) y tripsina sin rojo de fenol (véase Tabla de materiales) para reducir la fluorescencia de fondo.
- Separar las células de un cultivo confluente con tripsina, contar el número de células en suspensión de células mediante una cámara de Neubauer y semilla de 5.000-10.000 células por pocillo. Alternativamente, use 1 gota de una pipeta de 2 mL de una suspensión 10 mL de un cultivo de células confluentes crecido en un frasco de cultivo celular de 25 T o 3 gotas de una suspensión 5 mL de un cultivo confluente en un frasco de cultivo celular de 12,5 T.
- Cultivar células en cámaras de 8 pozos con tapa #1.0 (véase Tabla de materiales) de cristal para microscopía de fluorescencia células vivas.
- Transfectar células 24 h después de la galjanoplastia utilizando reactivos comerciales (véase Tabla de materiales) según el protocolo del distribuidor con la GFP, cereza, PA-GFP, cereza y GFP — quimeras PA-Cherry.
- Imagen de células después de un total de 20 h post transfección para permitir la expresión de la proteína, el plegamiento y maduración.
3. la proyección de imagen y fotoactivación
- Imagen de células en una cámara ambiental humidificada y caliente a 37 grados centígrados. A los medios de comunicación de la célula del almacenador intermediario a pH fisiológico y dejarlo CO2-independiente, agregar 20 mM HEPES, o usar gas de CO2 a 5% del flujo.
- En primer lugar, las células que expresan la GFP de la imagen, construcción de cereza. Establecer los parámetros que definen la intensidad láser integrado de tiempo por píxel en una imagen confocal, es decir, tiempo de permanencia de pixel en microsegundos, transmisión de filtro sintonizable óptico acústico (AOTF) en por ciento y el zoom digital.
Nota: Utilizando un objetivo de 60 x y un zoom digital de x 3 permite la proyección de imagen de una célula en su totalidad mientras que proporciona suficiente aumento. Configurar tiempo de permanencia de pixel y 2-4 μs transmisión AOTF para el láser de 488 nm y 561 nm tal que las imágenes muestran una buena relación señal a ruido sin ningún blanqueo y no pixeles lo que indica saturación de intensidad de fluorescencia. - Imagen, utilizando la energía del laser set, transmisión AOTF, tiempo de permanencia de píxeles y zoom digital, 15-20 células expresando GFP, cereza.
- Entonces, la imagen con el mismo sistema láser potencia, tiempo de permanencia de pixel, transmisión AOTF y zoom digital células expresando GFP — PA-Cherry y PA-GFP, cereza. Búsqueda por expresar las células en el canal verde o canal rojo, respectivamente. Evitar la exposición prolongada de las células durante la búsqueda por expresar las células con el fin de no blanquear las proteínas fluorescentes.
- Establecer una serie de mini-temporal con una imagen de activación previa y tres imágenes de la activación. Las imágenes de activación posterior ayudará a identificar potenciales transitorios Estados oscuros debido a la exposición a los rayos UV.
Nota: En nuestras manos, cambios en la intensidad de la fluorescencia detectada debido a los transitorios Estados oscuros eran < 1% y podría ser descuidado, pero los Estados oscuros deben evaluarse para cada montaje experimental. - Para determinar la eficiencia de la fotoactivación, es decir, la fracción de PA-FPs que se enciende para ser fluorescente, en los experimentos de fotoactivación específicas ya establecidas, aplicar la misma configuración de fotoactivación a 15-20 células que expresan las reglas internas presentó aquí y proceder con el análisis de imagen (sección 4). Si a partir de establecer experimentos de fotoactivación, encontrar aquí unas cuantas opciones diferentes, basadas en nuestra experiencia experimental con PA-GFP y PA-cereza; modificar según sea necesario.
- Para fotoactivación instantáneo de PA-GFP y PA-cereza usando un láser confocal de barrido (CLSM) del microscopio, se aplican μW 90 de 405 nm láser luz en 3 o 5 repeticiones, respectivamente.
Nota: Con nuestra configuración microscópica, utilizando una transmisión de AOTM 38% y un tiempo de permanencia del pixel de 2 μs, medimos este poder láser de 405 nm en modo de línea de exploración en la lente del objetivo. Con estos ajustes, aproximadamente el 8% y 16% de los PA-GFP y PA-cereza expresada pueden ser fotoactivado a ser fluorescente. La serie de toda valoración utilizando CLSM por fotoactivación instantáneo ha sido había publicado previamente17. - Si una fracción mayor de fotoactivado PA-GFP y PA-Cherry fluoróforos es ventajoso por ejemplo, para lograr una mayor relación señal a ruido y fotoactivación no tiene que ser de inmediato, aplicar 40 μW de 405 nm láser de luz con 2 μs tiempo de permanencia de pixel y 6% Transmisión de la AOTF 450 iteraciones. Entonces, fotoactivación tendrá hasta 4 min en comparación con sólo 1-2 segundos, pero la eficacia de la fotoactivación para PA-GFP será 29% en vez del 8%, lo que permite una mayor relación señal a ruido.
- Para fotoactivación instantáneo de PA-GFP y PA-cereza usando un láser confocal de barrido (CLSM) del microscopio, se aplican μW 90 de 405 nm láser luz en 3 o 5 repeticiones, respectivamente.
- Si existe un sistema de iluminación digital de mosaico que contiene matrices de micro del espejo en un modulador de luz espacial, luz láser de 405 nm puede usarse para widefield-fotoactivación. Esto permite eficiente fotoactivación en milisegundos.
Nota: Con 1,6 mW láser potencia medida en el objetivo y un tiempo de exposición de 250 ms, 29% de los PA-GFP puede ser fotoactivado a ser fluorescente. - Imagen con los parámetros de fotoactivación conjunto 15-20 células expresando GFP PA — cereza y GFP — PA-cereza, respectivamente.
- Desde pH, especies reactivas de oxígeno y otros factores ambientales pueden influir en la eficacia de la fotoactivación, puede ser importante evaluar la eficiencia de fotoactivación de la PA-FPs en lo respectivo micromilieu subcelular donde la proteína de interés es situado. Por el fluoróforo quimeras de acoplamiento a la proteína de interés, como los autores lo han hecho con la proteína de membrana plasmática VSVG 18, fotoactivación eficacia puede ser evaluada en el compartimento subcelular específico de interés.
4. Análisis y algoritmo de la imagen para la cuantificación de basada en intensidad radiométrica de la eficacia de la fotoactivación
- Análisis de imágenes pueden hacerse con las plataformas ImageJ o Fiji de procesamiento de imágenes de código abierto. Determinar la intensidad de la fluorescencia de fondo en las células transfected no en el verde (B1) y el canal rojo (B2). Evitar perinuclear o cualquier área que muestra mayor auto-fluorescencia.
- Para determinar la intensidad de fluorescencia en una celda transfected, contornear el cuerpo de la célula con la herramienta Selección a mano alzada. Una vez más, evitar perinuclear u otras áreas mostrando auto fluorescencia.
- Restar el fondo de la intensidad de fluorescencia medidos en cada canal.
IG = IGreen_measured -B1
IR = IRed_measured -B2 - Utiliza la GFP: cereza construcción para calcular la proporción de rojo a verde (RtoGr) y corregir para amortiguamiento de donantes debido a la transferencia de energía de resonancia de fluorescencia (FRET). La eficacia de traste E fue determinada para ser 0.3 en experimentos anteriores para la GFP: cereza constructo usando el mismo aminoácido vinculador entre los dos fluoróforos18.
RtoGr = (IRed_measured -B2) / (IGreen_measured -B1)
RtoGrcorr = RtoGr * (1 – E)
Nota: En esta intensidad basado en enfoque, donantes que apaga para mEGFP y PA-mGFP pueden ser diferentes dado posibles propiedades espectrales distintas que no se han caracterizado. La tasa de traste (kET) y la distancia de Foerster (R0) dependen de la producción de quántum del donante que no ha sido determinada para mEGFP y PA-mGFP. - Utiliza la GFP: construcción de PA-Cherry para evaluar la fracción de fotoactivado PA-Cherry. Determinar la intensidad de fluorescencia esperado de PA-Cherry multiplicando la intensidad de la fluorescencia verde insaciable medido de la GFP, Cherry PA construir previa a la fotoactivación con la corregida rojo-verde-relación / (RtoGrcorr ).
IRed_expected (IGreen_measured -B1) = * RtoGrcorr
Nota: Como se indicó anteriormente, el molecular brillo de siempre en FP y el photoactivatable FP puede ser diferente. Vea la discusión para más información sobre cómo tener en cuenta estas diferencias. - Calcular la eficiencia de la fotoactivación de PA-Cherry como una fracción de la fluorescencia roja medida intensidad después de fotoactivación y la intensidad de fluorescencia esperada en el canal rojo.
(FPA-Cherry) = (IRed_measured -B2) / meRed_expected - Utilizar la PA-GFP, cereza constructo para evaluar la fracción de fotoactivado PA-GFP. Determinar la intensidad de fluorescencia esperado de PA-GFP dividiendo la intensidad de fluorescencia roja medida de la PA-GFP, cereza construir previa a la fotoactivación por el rojo-verde-relación / (RtoGr). Aquí, el RtoGr no necesita ser corregido de amortiguamiento, los donantes porque GFP y PA-GFP están sujetos a donantes que apaga a la misma cantidad.
Green_expected = (IRed_measured -B2) / RtoGr - Calcular la eficiencia de la fotoactivación de PA-GFP como una fracción de la fluorescencia verde medida intensidad después de fotoactivación y la intensidad de fluorescencia esperada en el canal verde.
(FPA-GFP) = (IGreen_measured -B1) / IGreen_expected
Resultados
El protocolo que presentamos muestra la cuantificación radiométrica de la fracción de proteínas fluorescentes que se fotoactiva que fluorescente (figura 1). Esta fracción varía dependiendo de la modalidad de fotoactivación.
Un resultado típico mediante fotoactivación de alta potencia de corto plazo con un láser confocal de barrido (CLSM) del microscopio se muestra en la figura 2C. Después de valorar la potencia del láser según lo medido en la lente del objetivo por pixel moradores tiempo y transmisión ATOF, la eficacia máxima de la fotoactivación para PA-GFP era cerca de 8% para PA-cereza alrededor de 16%. La eficiencia de la fotoactivación comparable bajo de PA-GFP y PA-Cherry puede explicarse por fotoactivación simultánea y - destrucción cuando se expone a una corriente determinista continua de fotones por CLSM. La corta vida de fluorescencia y los espectros de absorción diferentes, cambiado de puesto derecho del rojo PA-FPs puede contribuir a la eficacia más alta de la fotoactivación de la PA-cereza en comparación con el PA-GFP.
Uso de láser de baja potencia y cientos de iteraciones, se logra una mayor eficiencia de fotoactivación. 450 iteraciones de la luz UV por un CLSM entregado un total de 4 min produjeron una eficiencia de fotoactivación del 29% para PA-GFP (Figura 3C). La mayor eficiencia de fotoactivación con la exposición repetitiva a fotones de luz UV puede sugerir un proceso multi-step de fotoactivación. Por otra parte, la luz ultravioleta aplicada es suficiente para Fotoactivar pero no lo suficiente para photodestruct que lleva acumulada con el tiempo a una mayor fracción de proteínas fluorescentes fotoactivado.
Con iluminación de campo amplio, los fluoróforos son estocástico y repetitivamente expuestos a fotones de 405 nm. Aquí, exposición de sólo 250 ms rindió un 29% de eficiencia de fotoactivación para PA-GFP.

Figura 1: concepto de cómo determinar la eficiencia de la fotoactivación a granel y en células vivas. Por acoplamiento espectralmente distintas proteínas fluorescentes, se crean reglas internas que permiten la radiométrica intensidad-evaluación de la eficacia de la fotoactivación. Mide intensidades de Cherry PA y PA-GFP se relacionaron con intensidades esperadas. Intensidades previstas fueron derivados desde la determinación de la intensidad de fluorescencia de las siempre en proteínas fluorescentes GFP-Cherry, GFP-PA-cereza o quimeras PA-GFP-Cherry antes de fotoactivación. Figura modificada de Renz y Wunder 201717. Haga clic aquí para ver una versión más grande de esta figura.

Figura 2: a granel fotoactivación de la PA-GFP-cereza (a) y GFP-PA-Cherry (b) como instantáneamente y completamente como sea posible en un microscopio de escaneo láser confocal de células vivas. 8% de los PA-GFP expresado fue fotoactivado con un tiempo de permanencia de pixel de 2 μs y una transmisión AOTF de 38%, que resultan en energía del laser de 90 μW, medida en el objetivo y 3 iteraciones (c). Aumento de la potencia del láser de 405 nm no aumentó eficacia de fotoactivación. Figura modificada de Renz y Wunder 201717. Haga clic aquí para ver una versión más grande de esta figura.
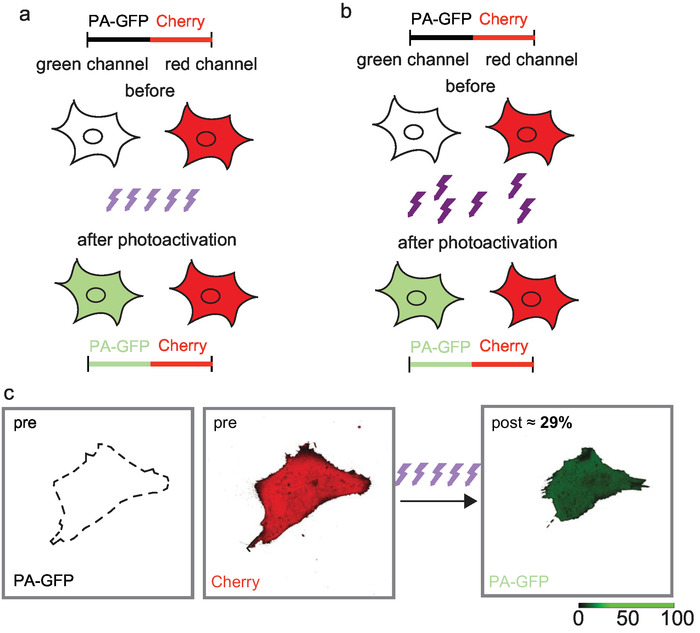
Figura 3: iterativo fotoactivación de baja potencia con un microscopio de escaneo láser confocal (a) y corta widefield alta potencia iluminación (b) rendimiento superior fotoactivación eficiencias. 29% de los PA-GFP fue fotoactivado con un tiempo de permanencia de pixel de 2 μs y una transmisión AOTF del 6%, que resultan en energía del laser de 40 μW, medida en el objetivo y 450 iteraciones (c). Figura modificada de Renz y Wunder 201717. Haga clic aquí para ver una versión más grande de esta figura.
Discusión
Hasta ahora, ningún método existió para determinar a granel la fracción de PA-FPs expresada en células vivas que se fotoactiva ser fluorescente. El protocolo presentado puede utilizarse para cualquier par de proteína fluorescente espectralmente distintos. Si bien ejemplificados aquí por el irreversible FPs PA PA-GFP y PA-cereza, este enfoque es en principio aplicable a las proteínas de la photoconvertible así. La proteína fluorescente espectralmente distinta, sin embargo, debe seleccionarse cuidadosamente para minimizar la superposición espectral dado que proteínas fluorescentes photoconvertible cambio su absorción y espectros de emisión, por ejemplo, de verde a rojo fluorescencia.
Como se indicó anteriormente, es importante señalar que el enfoque presentado es radiométrica y basada en la intensidad. Puede utilizarse para estandarizar el nivel de expresión desconocida en las células y definir diferencias relativas en la eficiencia de fotoactivación por diferentes modos de fotoactivación. El protocolo también puede utilizarse para evaluar la fracción absoluta de fotoactivado PA-FPs. Diferentes propiedades espectrales de diferentes FPs deben tenerse en cuenta.
El brillo molecular (MB) es el producto de rendimiento cuántico (QY), coeficiente de extinción (CE) y porcentaje absorbancia a la longitud de onda de excitación determinada en relación con el pico de absorbancia. Cereza12 y PA-Cherry13, se han publicado valores respectivos de QY y CE. La absorbancia por ciento a la longitud de onda de excitación determinada de 543 nm relativa al pico de absorbancia es de 0.5 y 0.7, respectivamente.
MBCherry = 0.22 * 72.000 * 0.5 = 7.920
MBPA-Cherry = 0,46 * 18.000 * 0.7 = 5.796
Así, el brillo molecular menor de PA-cereza comparado con cereza puede tenerse en cuenta dividiendoRed_expected por 1,37 (derivado de 7.920/5.796).
Sin embargo, se desconoce en qué condiciones de fotoactivación el brillo molecular publicado de PA-Cherry se ha determinado. Esto es importante, ya que aquí mostramos que el modo de fotoactivación cambia la fracción medida de fotoactivado PA-FPs. Además, para las versiones monoméricas comprende la mutación A206K. es decir, mEGFP y PA-mEGFP, no se ha publicado ningún brillo molecular.
En este enfoque basado en la intensidad radiométrica, el brillo molecular de la PA-FPs y las contrapartes FP siempre en una primera aproximación se han considerado idéntico. Nos decidimos por este enfoque, puesto que (i) para algunos FPs no se ha divulgado ningún brillo molecular, y (ii) es hasta ahora confuso en cómo ahora diferentes modos de fotoactivación pueden afectar el brillo molecular de la PA-FPs reportado en la literatura. Además, (iii) para un análisis comparativo de los conocimientos de la luminosidad molecular no es necesario; sólo es necesario para la determinación basada en la intensidad de la fracción absoluta de fotoactivado PA-FPs que puede calcularse como se muestra arriba.
Nuestro enfoque de quimeras de la proteína fluorescente como reglas internas muestra que la diferente exposición a la luz UV produce fotoactivación diferentes eficiencias. Por lo tanto, define opciones de cómo Fotoactivar un PA más grande-FP fracción y lograr una mejor relación señal a ruido. Por otra parte, abre oportunidades a diferencialmente Fotoactivar PA-FPs diferentes en la misma célula dada su respuesta diferencial a la luz UV o diferencialmente Fotoactivar el mismo PA-FP en diferentes compartimentos subcelulares, exponiéndolo manera diferente a los rayos UV. En Resumen, nuestro protocolo ayudará aún más la comprensión cuantitativa de los procesos celulares mediante PA-FPs en microscopía de células vivas.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Nos gustaría agradecer al laboratorio de Dorigo y el servicio de la proyección de imagen de Neurociencia en la escuela de medicina de la Universidad de Stanford para proporcionar espacio y equipo para este proyecto.
Materiales
| Name | Company | Catalog Number | Comments |
| pEGFP-N1 mammalian cell expression vector | Clontech | ||
| DMEM w/o phenol red | Thermo Fisher Scientific | 11054020 | |
| Trypsin w/o phenol red | Thermo Fisher Scientific | 15400054 | |
| L-Glutamine (200 mM) | Thermo Fisher Scientific | 25030081 | |
| HEPES | Thermo Fisher Scientific | 15630080 | |
| LabTek 8-well chambers #1.0 | Thermo Fisher Scientific | 12565470 | |
| Fugene 6 | Promega | E2691 |
Referencias
- Patterson, G. H., Lippincott-Schwartz, J. A photoactivatable GFP for selective photolabeling of proteins and cells. Science. 297 (5588), 1873-1877 (2002).
- Ando, R., Hama, H., Yamamoto-Hino, M., Mizuno, H., Miyawaki, A. An optical marker based on the UV-induced green-to-red photoconversion of a fluorescent protein. Proceedings of the National Academy of Sciences of the United States of America. 99 (20), 12651-12656 (2002).
- Renz, M., Lippincott-Schwartz, J. Ch. 9. The Fluorescent Protein Revolution Series in Cellular and Clinical Imaging. Day, R. N., Davidson, M. W. , CRC Press. 201-228 (2014).
- Shcherbakova, D. M., Sengupta, P., Lippincott-Schwartz, J., Verkhusha, V. V. Photocontrollable fluorescent proteins for superresolution imaging. Annual Review of Biophysics. , 303-329 (2014).
- Betzig, E., et al. Imaging intracellular fluorescent proteins at nanometer resolution. Science. 313 (5793), 1642-1645 (2006).
- Hess, S. T., Girirajan, T. P., Mason, M. D. Ultra-high resolution imaging by fluorescence photoactivation localization microscopy. Biophysical Journal. 91 (11), 4258-4272 (2006).
- Henderson, J. N., et al. Structure and mechanism of the photoactivatable green fluorescent protein. Journal of the American Chemical Society. 131 (12), 4176-4177 (2009).
- Subach, F. V., et al. Photoactivation mechanism of PAmCherry based on crystal structures of the protein in the dark and fluorescent states. Proceedings of the National Academy of Sciences of the United States of America. 106 (50), 21097-21102 (2009).
- Wiedenmann, J., et al. EosFP, a fluorescent marker protein with UV-inducible green-to-red fluorescence conversion. Proceedings of the National Academy of Sciences of the United States of America. 101 (45), 15905-15910 (2004).
- Habuchi, S., Tsutsui, H., Kochaniak, A. B., Miyawaki, A., van Oijen, A. M. mKikGR, a monomeric photoswitchable fluorescent protein. PLoS One. 3 (12), e3944(2008).
- McKinney, S. A., Murphy, C. S., Hazelwood, K. L., Davidson, M. W., Looger, L. L. A bright and photostable photoconvertible fluorescent protein. Nature Methods. 6 (2), 131-133 (2009).
- Shaner, N. C., et al. Improved monomeric red, orange and yellow fluorescent proteins derived from Discosoma sp. red fluorescent protein. Nature Biotechnology. 22 (12), 1567-1572 (2004).
- Subach, F. V., et al. Photoactivatable mCherry for high-resolution two-color fluorescence microscopy. Nature Methods. 6 (2), 153-159 (2009).
- Zacharias, D. A., Violin, J. D., Newton, A. C., Tsien, R. Y. Partitioning of lipid-modified monomeric GFPs into membrane microdomains of live cells. Science. 296 (5569), 913-916 (2002).
- Slocum, J. D., Webb, L. J. A Double Decarboxylation in Superfolder Green Fluorescent Protein Leads to High Contrast Photoactivation. Journal of Physical Chemistry Letters. 8 (13), 2862-2868 (2017).
- Subach, F. V., Patterson, G. H., Renz, M., Lippincott-Schwartz, J., Verkhusha, V. V. Bright monomeric photoactivatable red fluorescent protein for two-color super-resolution sptPALM of live cells. Journal of the American Chemical Society. 132 (18), 6481-6491 (2010).
- Renz, M., Wunder, C. Internal rulers to assess fluorescent protein photoactivation efficiency. Cytometry A. , (2017).
- Renz, M., Daniels, B. R., Vamosi, G., Arias, I. M., Lippincott-Schwartz, J. Plasticity of the asialoglycoprotein receptor deciphered by ensemble FRET imaging and single-molecule counting PALM imaging. Proceedings of the National Academy of Sciences of the United States of America. 109 (44), E2989-E2997 (2012).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados