Method Article
Mis à jour le rouleau Tube méthode d’imagerie Intermittent précisément localisé et répétitif au cours de la Culture à long terme des tranches de cerveau dans un système fermé
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
Présenté ici est la méthode du tube mis à jour le rouleau pour la culture et intermittent une imagerie du cerveau rongeur tranches sur plusieurs semaines avec un repositionnement précis sur couvre-objet en photodécoupe. Viabilité neuronale et la morphologie de la tranche sont bien entretenus. Sont prévues des adaptations de ce système entièrement fermé à l’aide de virus pour l’expression spécifique de type cellulaire.
Résumé
Coupes de cerveau de rongeurs cultivées sont utiles pour étudier le comportement moléculaire et cellulaire des neurones et des cellules gliales dans un environnement qui soutient plusieurs de leurs interactions normales en vivo . Tranches provenant d’une variété de lignées de souris transgéniques ou utilisation de vecteurs viraux pour l’expression des protéines fluorescent étiquetées ou reporters dans les coupes de cerveau de type sauvage permettent d’imagerie haute résolution en microscopie par fluorescence. Bien que plusieurs méthodes ont été développées pour l’imagerie des tranches de cerveau, alliant la culture tranche avec la capacité d’exécuter répétitives une imagerie de cellules spécifiques en tranches direct sur de longues périodes a posé des problèmes. Cela est particulièrement vrai lorsque les vecteurs viraux sont utilisées pour l’expression des protéines exogènes, puisque cela se fait mieux dans un système fermé pour protéger les utilisateurs et d’éviter toute contamination croisée. Nous rapportons des simples modifications apportées à la méthode de culture de rouleau tube cerveau tranche qui permettent pour l’imagerie haute résolution répétitive des tranches sur plusieurs semaines dans un système fermé. Cultivant les tranches sur des lamelles de photodécoupe permet l’utilisation de repères de rapidement et précisément repositionner la scène pour l’image du champ identique au fil du temps avant et après des traitements différents. Des exemples sont donnés pour l’utilisation de cette méthode combinée à une coloration neuronale spécifique et expression d’observer les changements dans l’architecture tranche hippocampe, expression neuronale induite par virale de protéines fluorescentes et le développement de la pathologie cofilin, qui a été précédemment observé dans l’hippocampe de la maladie d’Alzheimer (ma) en réponse au traitement de la tranche avec des oligomères de peptide amyloïde-β (Aβ).
Introduction
Des cultures primaires de neurones dissociés des régions du cerveau de rongeur sont un outil important utilisé par les chercheurs pour observer les réponses à pathologiquement impliqués des stimuli. Toutefois, ces études présentent l’inconvénient de regarder les neurones en 2D uniquement et sans leur système de soutien glial. En outre, sauf s’il est cultivé dans des conditions de très haute densité (640 neurones/mm2 ou environ 16 % de la surface) dans lequel il devient impossible de suivre l’excroissance au hasard d’une dendrite ou axone depuis plus d’une courte distance de son corps cellulaire, hippocampe viabilité neuronale pendant 4 semaines diminue de façon importante1, limitant l’utilisation de cultures dissociées des études étendues des pathologies liées au vieillissement. La mise en culture des tranches préparés à partir de cerveau de rongeur est une option intéressante qui permet de surmonter ces limitations en conservant une architecture cellulaire organisés et la viabilité des semaines ou des mois. Conditions pour le maintien de nombreuses régions du cerveau de rongeur dans la tranche de culture ont été décrits2.
Deux méthodes principales sont largement utilisés pour les cultures à long terme des tranches de cerveau : culture sur la membrane à l' interface air-liquide3 ou mise en culture sur les lamelles dans des tubes scellés a permis de tourner dans un incubateur à rouleau d’aération4. Tranches sur des membranes peuvent être photographiées directement avec la microscopie de fluorescence à haute résolution à l’aide d’un microscope droit et eau immersion objectifs5. Alternativement, tranches cultivés sur les membranes ont été transférés à la vaisselle en verre bas pour atteindre la bonne résolution des épines dendritiques, à l’aide d’un microscope inversé6. Toutefois, les deux méthodes d’imagerie tranches cultivés sur les membranes sont des systèmes ouverts qui nécessitent des modifications moyennes et utilisent souvent les antifongiques et/ou des antibiotiques pour prévenir ou réduire la contamination5,6. Tranches sur une membrane à l’interface air-médium maintiennent excellente morphologie et la survie, mais revenir à des emplacements précis en imagerie répétitive à fort grossissement est extrêmement difficile à moins que l’expérience est issue uniquement de petits groupes de cellules exprimant un marqueur fluorescent. Bien que les tranches cultivés sur les membranes ont été utilisés avec virale véhiculée par expression de transgènes5,6, protocoles de prévention des risques biotechnologiques peuvent nécessiter un système de culture clos être employées pour certains vecteurs viraux qui sont utilisés pour exprimant fluorescent étiquetée protéines et reporters de la physiologie cellulaire. En outre, les objectifs à immersion exigent décontamination entre les échantillons qui seront suivies dans la culture5. Une application importante des cultures interface membrane est de combiner une imagerie avec l’électrophysiologie de temps unique points7.
La méthode du tube à rouleaux avec lamelles couvre-objet à l’intérieur du tube en plastique ne permet pas de n’importe quel électrophysiologie ou l’imagerie haute résolution sans enlever le couvre-objet. Ainsi, cette méthode a été le plus souvent appliquée à des études à long terme dans lequel, après fixation observations ont été faites à8. Décrite ici est une méthode qui utilise la technique de culture de tube de rouleau, mais sur les tubes percés-out avec des tranches sur lamelles couvre-objet qui peut être photographiée de façon répétitive pour aussi longtemps que les cultures est maintenue. Le système clos ne requiert aucun changement médiatique pour l’imagerie et utilise des lamelles de photodécoupe pour fournir des repères qui permettent d’imagerie à fort grossissement, après quelques jours ou semaines, les domaines précis précédemment imagés.
Nous appliquons cette méthode pour analyser les changements dans l’hippocampe de rongeur, une région de majeur du cerveau impliquée dans la mémoire et l’apprentissage. L’hippocampe rongeur est souvent étudiée comme modèle pour des changements pathologiques ou liée à l’âge observée au cours du développement de troubles cognitifs9, telles que celles qui se produisent dans l’AD. Notre méthode est particulièrement bien adaptée à l’étude des changements pathologiques qui se développent au sein d’une seule tranche au fil du temps en réponse aux changements environnementaux, tels que l’augmentation des peptides bêta-amyloïdes, qui est caractéristique de l’AD8. Une pathologie associée du cerveau humain et les rongeur est la présence d’agrégats de cofilin-actine et tiges, les faisceaux contenant ce dernier des filaments dans laquelle cofilin et l’actine sont dans un rapport molaire 1:110,11, 12. tiges ont été observés dans les tranches fixes d’hippocampe de rat après un traitement de bêta-amyloïdes, ainsi que dans une tranche de cerveau de rongeurs vivants exprimant cofilin-GFP, soumis à l’hypoxie,8, et ils peuvent contribuer à la dysfonction synaptique chez AD et d’AVC. Ici, nous utilisons cette nouvelle méthode de culture pour observer l’évolution temporelle et la distribution dans les tranches des exogènes chimériques fluorescentes protéines exprimées introduites par différents virus. Ensuite, nous utilisons l’expression neuronale spécifique d’une construction de journaliste cofilin pour suivre l’évolution de la tige de cofilin et pathologie globale dans des tranches d’hippocampe en réponse au traitement par solubles oligomères bêta-amyloïdes (Aßo).
Protocole
L’utilisation des animaux suit élevage agréé et protocoles d’utilisation des animaux qui sont conformes à la protection des animaux et utiliser lignes directrices de la Colorado State University.
Remarque : Le protocole ci-dessous décrit la méthode de préparation et de la culture pour l’incubation à long terme et l’imagerie intermittent de tranches d’hippocampe. Une seule tranche hippocampe est attachée à une lamelle de photodécoupe spécialement préparé à l’aide d’un caillot de plasma, et puis les lamelles sont scellés sur le côté plat d’un tube de rouleau de Percé-out, qui est maintenu dans une étuve à rouleau. Les caillots plasmatiques sont dissous avec la plasmine avant l’infection virale pour l’expression de la protéine fluorescente et l’imagerie haute résolution. Un colorant vital neuronal fluorescent est utilisé pour les neurones dans les tranches d’image.
1. préparation du rouleau Tube Rack
- Utilisation du modèle illustré à la Figure 1A imprimé à la taille indiquée sur la barre d’échelle. Avec un clou, percer des trous petits (assez grands pour un marqueur à pointe fine) dans le modèle centré sur les trous.
- Définir le modèle sur le fond d’un plat de culture de tissu de 15 cm (diamètre nominal de 14 cm) et marquer la position des trous. Répétez cette procédure sur un deuxième plat.
- Avec des forets conçus pour être utilisé sur du plastique, percer six trous de diamètre de 1,5 cm sur chaque plat dans un arrangement hexagonal (4,8 cm Centre à centre) avec des centres de trou 2,5 cm du bord du plat. Percer trois trous (3 mm de diamètre) 12 mm du bord qui sont équidistantes placées entre deux des plus grands trous comme indiqué dans la Figure 1A.
- Avec le fond de chaque plat face à face, placer une vis de machine long 2.5 pouces (3/16 de pouce de diamètre) avec une rondelle plate à travers l’un des petits trous suivies d’une deuxième rondelle plate, une pièce de polyéthylène tube (entretoise, 4,7 cm), une autre rondelle plate , le deuxième plat de culture de tissu, une autre rondelle plate, une rondelle et un écrou.
- Répétez l’étape 1.4 sur les deux autres vis de machine et en serrant seulement vaguement jusqu'à ce que toutes les vis sont en place. Ensuite, serrez les écrous.
- Travailler les œillets (5/16 de pouce épais, diamètre du trou 5/8 po) dans les trous du plat inférieur pour obtenir le dernier tube carré (Figure 1B; illustré avec deux tubes en place). Une gommette sur chaque grille avec un numéro unique.
2. préparation du rouleau Tubes et lamelles couvre-objet
- Fabrication du gabarit pour le perçage du trou dans des tubes de rouleaux
- Percer un trou de 1,5 cm 8 cm de profondeur dans la partie centre de 2 x 4 x 5,5 po en bois blocs à un angle tel que le côté plat du rouleau tube volonté être presque parallèles avec le bloc lorsqu’il est inséré (Figure 2A).
- Agrandir le trou à l’aide d’une lime ronde bois à élargir et à cône le trou pour permettre l’insertion de tubes (tubes roulés sont légèrement plus grandes de diamètre près de l’extrémité du cap) (Figure 2B).
- Percer un trou vertical de 1,5 cm de diamètre, 5,5 cm du côté de l’édifice et centré sur le trou de côté (Figure 2C).
- Lorsque l’orifice du côté est conique assez, insérer un tube de rouleau qui est tracée à l’endroit désiré pour centrer le trou pour la tranche et positionner le tube pour l’endroit repéré est centré dans le trou vertical de 1,5 cm.
- Retirez le tube et mesurez la distance entre l’endroit à l’extrémité du tube. Marquer cette distance entre le centre du trou dans le gabarit et insérer un clou pour fournir un arrêt pour positionner correctement le tube de forage (flèche sur la Figure 2C).
- Utiliser une scie à métaux pour couper les ongles affleurer la surface de la cale en bois pour éviter les blessures.
- Ajouter les clips à ressort sur le fond de la sauteuse s’il y a une presse de foret avec machines à sous qui lui permettent d’être ancré (flècheFigure 2D noir). Sinon, utilisez C-pinces pour tenir le gabarit sur la perceuse.
- En utilisant le gabarit décrit ci-dessus pour tenir et positionner un tube de culture en plastique plat face 11 cm avec le côté plat vers le haut (Figure 2A), percer un trou de diamètre 6 mm avec le centre de 1,0 cm du bas et centré entre les parois du tube.
Remarque : Un foret conçu pour le plastique doit être utilisé. - Avec un outil d’ébavurage pivotant, lisser les bords du trou (Figure 2A) et faire 4 rainures à l’intérieur bord du trou (Figure 2E, encart) pour faciliter le drainage du trou pendant la rotation.
- Avec un trou de 12 mm, punch, couper des disques de diamètre de 12 mm de feuilles de caoutchouc non toxique double face adhésif silicone. À l’aide d’un poinçon de papier standard un trou (diamètre 6 mm), faire un trou au centre de chaque disque.
- Rincer les tubes percés avec l’éthanol à 70 %, air séchez-les dans une armoire, de la sécurité biologique et stériliser les tubes et les disques perforés adhésifs pendant 40 min sous la lampe UV (30 W à distance moyenne de 70 cm) dans l’armoire de sécurité biologique.
- Repositionner les tubes et les disques après 20 min afin que toutes les surfaces exposées sont stérilisés. Dans des conditions stériles, Décollez le blanc protecteur d’un disque adhésif et apposer le caoutchouc de silicone à l’extérieur d’un tube, en alignant les trous (Figure 2F).
ATTENTION : Pour éviter l’exposition aux UV, porter une protection oculaire et fermer le cabinet avant d’allumer la lampe UV. - Nettoyez 12 mm diamètre photodécoupe (100 Centre numéroté 1 mm carrés) lamelles de verre allemand. Maintenez les lamelles doucement avec une pince et tremper dans de l’éthanol absolu, suivi par l’eau, suivie d’éthanol absolu à nouveau et enfin tremper les lamelles dans une flamme pour brûler de l’éthanol. Permettre à lamelles couvre-objet à refroidir.
- Avec une pince en tenant les lamelles, tremper dans 2 % 3-aminopropyltriethoxysilane dans l’acétone pour 10 s. Rincer les lamelles couvre-objet avec de l’eau ultrapure et laisser sécher à l’air.
- Mettre les lamelles sur un papier filtre stérile à l’intérieur d’une biosécurité armoire et allumez la lampe à UV légère. Exposer chaque côté les lamelles pendant 20 min.
ATTENTION : Pour éviter l’exposition aux UV, porter une protection oculaire et fermer le cabinet avant d’allumer la lampe UV.
3. préparation de la tranche hippocampe
- Avant de débuter la dissection, préparer la moitié des lames de rasoir à double tranchant pour le chopper de tissu. Plier les lames longueur délicatement avec les doigts et casser en deux.
- Rincer les moitiés de la lame avec de l’acétone à l’aide d’un coton-tige pour nettoyer, rincer dans de l’éthanol absolu et séchage à l’air.
Remarque : Le protocole de culture de tranche de cerveau est indépendant de la souche de rat ou de souris ou de génotype. Plusieurs lignées de souris transgéniques avec différents fonds génétiques ont été utilisées.
- Avec une pince, tenir le côté postérieur de cerveau coupé vers le haut et la face ventrale contre le côté de la boîte de Pétri pour la stabilité. Doucement taquiner les méninges loin autour de la ligne sagittale médiane et enlever le tissu de cerveau moyen à l’aide d’une pince de Dumont #5 à pointe fine (Figure 3C, cercle en pointillés).
- Faire deux incisions le long du côté du cerveau pour l’écarter ouvert (Figure 3C, lignes pointillées). Une fois que le cerveau est placé face dorsale vers le bas et ouvrir de propagation, l’hippocampe fissure doit être visible (Figure 3D, flèche).
- Transférer le cerveau ouvert de propagation à un morceau de film plastique Polychlorotrifluoroéthylène et positionnez-le pour trancher sur la scène d’un découpeur de tissu. Humidifiez la lame GBSS/glucose, puis hacher l’hippocampe en tranches épaisses de ~ 300 µm.
- Avec une pipette de transfert, purger le cerveau en tranches sur le film plastique dans un plat frais 60 mm contenant GBSS/glucose (Figure 3E). Doucement, pinch off et taquiner, avec pinces à pointe fine, les méninges restantes et autres tissus non-hippocampique (Figure 3-F) des tranches (Figure 3G, H).
4. électrodéposition tranches
- Une fois les tranches ont été obtenus, placer 2 µL de plasma de poulet sur le centre du côté d’un lamelle couvre-objet préparé photodécoupe. Répandre le plasma légèrement pour atteindre une tache de diamètre de 3-4 mm.
Remarque : Le côté de photodécoupe est le dessus de la lamelle couvre-objet vu à travers un microscope à dissection, tels que les nombres sont correctement orientées. - Transférer 1 tranche de cerveau avec une spatule stérile de pointe étroite (Figure 4A) à l’endroit de plasma (Figure 4B). Utilisez la pince fermée pour garder la tranche à l’extrémité de la spatule en soulevant la tranche de la GBSS/glucose.
- Touchez la spatule pour le plasma spot sur la lamelle couvre-objet et avec une pince fermée, poussez la tranche sur la lamelle couvre-objet.
- Mix 2,5 µL de plasma avec 2,5 µL de la thrombine dans une éprouvette. Placer rapidement 2,5 µL de ce mélange sur et autour de la tranche et la pipette de haut en bas doucement pour mélanger (Figure 4B).
Remarque : Le plasma se coaguler dans 10-15 s, donc cela doit être fait rapidement. Si l’adhérence de la tranche est un problème, mélanger 5 plasma µL avec 5 thrombine µL et utiliser 4-5 µL sur la tranche, supprimant certains après avoir été mélangées afin que la tranche se trouve à plat sur la lamelle couvre-objet. - Retirez le plastique transparent couvrant du côté exposé de l’adhésif de caoutchouc de silicone déjà apposée sur un tube d’enroulement et la lamelle avec la tranche de cerveau sur l’adhésif en alignant la tranche dans le trou (Figure 4C).
- Pour assurer l’adhérence, comprime doux, même à la lamelle avec le pouce en appuyant uniformément sur la lamelle couvre-objet et en tenant pendant environ 1 min tout en transférant à l’armoire de sécurité biologique.
- Dans une armoire de sécurité biologique, ajouter 0,8 mL de milieu de culture complet Neurobasal A (Table des matières) dans chaque tube (Figure 4D).
- Couler un mélange d’air 5 % CO295 % grâce à une pipette Pasteur stérile coton-branché solidement maintenu par une pince. Rincer le tube avec le mélange de gaz et rapidement Boucher le tube qu’il soit retiré du autour de la pipette.
- Identifier les tubes avec le numéro de tranche et nombre des grilles. Insérer les tubes dans un rack de rouleau, veillant à ce qu’ils soient équilibrés géométriquement. S’il y a un nombre impair de tubes, ajouter des tubes pour équilibrer.
- Placez les paniers dans une étuve à 35 ° C à rouleaux avec rouleaux tournant le rack roller sur RPH 10-13 (Figure 4E). Pour garder le milieu en bas des tubes, inclinaison l’incubateur retour environ 5° en levant son front sur une planche.
- Entrez le numéro de tranche et le tube sur une feuille de calcul, qui sert à enregistrer toutes les informations de la tranche des traitements et des dates d’observation.
- Sur environ jour 6 dans la culture, ajouter 1 µL (0,002 U) de plasmine active dans chaque tube.
- Après la formation de caillots se dissoudre complètement (généralement en quelques heures), retirer le support et le remplacer par un milieu frais sans plasmine. Si nécessaire, tranches peuvent être incubées avec plasmine pendant la nuit et le milieu a changé le lendemain.
- Tranches sont généralement incubées pendant au moins 7-10 jours avant son utilisation dans des expériences. Aspirez le milieu et remplacez-le jour 3 ou 4, le 7e jour et tous les 7 jours.
5. préparation de vecteurs viraux pour l’Expression du transgène
NOTE : Expression de transgènes dans les neurones des cultures de la tranche est réalisée à l’aide de cerveaux de rongeurs génétiquement ou par l’introduction du transgène à l’infection par les déficients virus recombinant de la réplication.Adénovirus (AV), adeno-associated virus (AAV) et vecteurs recombinants lentivirus ont été utilisés dans nos cultures hippocampe tranche pour l’expression des chimères de différentes protéines fluorescentes dans des tranches de cerveau.
- Préparer la réplication AV déficient pour exprimer l’ARN d’intérêt selon les méthodes décrites ailleurs13,14. Titre les virus infectieux U/ml par la méthode de dilution en série à l’aide d’un anticorps dirigé contre une protéine virale exprimée comme décrit14. Pour observer les agrégats cofilin et formation de tige de cofilin-actine, utilisent le cofilin-R21Q-RDRF cDNA (plasmide 51279 #)16.
Remarque : Le 1 promoteur synapsin est un excellent choix pour l’expression spécifique neuronale15, alors que le promoteur de cytomégalovirus est utile pour la conduite des niveaux d’expression élevée dans beaucoup de types de cellule13. - Préparer AAV en co transfection de transfert plasmide contenant le gène d’intérêt et un plasmide rep/hab, avec ou sans un plasmide d’assistance, dans les cellules HEK293 emballage, qui alimentent le gène E1 viral, comme décrit précédemment17,18.
NOTE : AAV Recombinant est également pour une insertion ciblée dans l' hôte de génome cellule19. Pour le plasmide de transfert, nous utilisons cofilin humaine 1 avec une étiquette de protéine de fluorescence (plasmide #50856) mRFP1 de C-terminal clonée dans un synapsin contenant du promoteur AAV plasmide en aval du calcium capteur GCaMP5G20. Un morceau d’ADN codant pour la séquence peptidique Self clivage P2A est inséré par PCR entre GCaMP5G et cofilin-DP pendant la préparation du plasmide transfert de fournir l’expression de deux protéines d’un unique de transcription d’AAV21. - Préparer des vecteurs recombinants lentivirus par transfection Co du plasmide de transfert contenant le gène ou ADNc des signaux d’intérêt et de l’intégration, ainsi qu’une troisième génération de lentivirus emballage mélange qui divise la virale gag, pol, rev et gènes de vsv-g sur trois différentes plasmides22,23.
- Pour le plasmide de transfert, utilisez une seule étape de clonage système24 pour assembler le promoteur synapsin et cofilin-R21Q-RDRF cDNA (à partir de plasmide 51279 #) dans pLKO.1-GFP (plasmide #30323), avec le promoteur synapsin et cofilin-R21Q-RDRF remplaçant la hPGK promoteur et GFP cDNA, respectivement.
- Transfecter le plasmide final en cellules HEK293T de phosphate de calcium comme décrit précédemment23.
- Recueillir le moyen de quatre plats de 10 cm, se concentrer à 500 µL à l’aide de concentrateurs centrifuges 150K-coupure et stocker le lentivirus final à-80 ° C en petites parties aliquotes après rapide congélation dans l’azote liquide. Décongeler une aliquote qu’une seule fois pour infecter les cellules.
- Déterminer empiriquement que le volume de chaque type de virus disposés à parvenir au niveau de l’expression souhaitée en mettant en place un certain nombre de cultures différentes tranche de suivre l’expression des transgènes après l’infection avec des volumes différents de virus.
Remarque : En général, 1-10 µL du virus est utilisée par tranche.
6. traitements de la tranche
- Infectant les tranches avec le virus
- Travailler sous une hotte de sécurité biologique approuvé pour le travail de virus au niveau de sécurité biologique approprié pour le vecteur, mélanger une partie aliquote du virus (habituellement 1-10 µL) de 0,8 mL de milieu complet.
- Aspirez le milieu de la tranche à l’aide d’une pipette Pasteur stérile dans un piège de la collection contenant l’eau de Javel. Un piège secondaire est toujours utilisé entre le premier piège et la source de vide.
- Remplacer ce moyen de communication avec l’aliquote contenant le virus préparé ci-dessus, retourner les tubes de culture à une grille et placer dans un incubateur.
- Après 2 à 5 jours d’incubation des tranches avec le virus dans l’armoire de sécurité biologique, retirez le support contenant le virus avec une pipette stérile et placez-la dans un flacon contenant un agent antiviral approuvé pour tuer les virus.
- La coloration des neurones avec colorant vital
- Préparer et stocker les aliquotes de colorant vital neuronale fluorescent25 par rapide congélation 4 µL d’aliquotes dans l’azote liquide à une concentration de 100 µM et stocker ces à-20 ° C. Pas de gel/dégel la teinture plus d’une fois.
- Pour numéroter les neurones pour la visualisation en microscopie de fluorescence, décongeler une aliquote du colorant vital neuronale et diluer dans 4 mL de milieu Neurobasal complet (concentration de colorant final est 100 nM).
- Retirer le support de tranches par aspiration (ou avec une pipette de transfert si le milieu contient des virus) et remplacez-le par 0,8 mL de milieu contenant le colorant vital neuronale de 100 nM. Retourner les tranches à l’appareil de rouleaux dans l’incubateur.
- Après incubation des tranches pendant 2 h, aspirer le milieu contenant du colorant et remplacez-le par 0,8 mL de milieu complet frais sous une hotte de sécurité biologique.
Remarque : Marquage des neurones en tranches avec un colorant vital nécessite plusieurs heures d’incubation. Les premières images sont habituellement pris 24h après le traitement de teinture. Bien que les neurones sont spécifiquement étiquetés, il n’y a fluorescence de fond qui se décline en 2-3 jours pour donner mieux l’imagerie neuronale. Intensité du colorant vital diminue après 72 h. - Pour suivre les changements dans la morphologie de la tranche au fil du temps, réétiqueter les tranches tous les 7 jours.
7. Coupez l’imagerie
- Découvre les tranches sur un microscope inversé. Pour l’imagerie de fluorescence plus brillants, après 24 h avant l’imagerie, échanger le milieu de culture avec milieu Neurobasal A complet sans l’indicateur de pH de rouge de phénol.
Remarque : Pour les expériences rapportées ici, tranches sont lus sur un microscope à fluorescence confocale rotation inversée disque équipé d’un codage linéaire x, y scène avec contrôle z piezo et une caméra numérique haute résolution sensible. - Transférer le tube avec la culture de la tranche à être photographié de l’appareil de tube de rouleau pour le sur mesure porte-tube (Figure 5A), qui est placé dans l’adaptateur de la scène (Figure 5B), pour garder la lamelle perpendiculaire à l’objectif et de maintenir la tranche dans le même sens au cours de séances d’imagerie répétitives sur longs intervalles.
- Poussez le curseur sur la carte de l’étape serré contre le tube pour maintenir le tube en place (Figure 5B).
NOTE : Détails du porte-tube, scène adaptateur, et chauffage est accessible à : https://vpr.colostate.edu/min/custom-machining/roller-tube-holder/.
NOTE : Ces positions décalée de la marque de fiducial permettent de repérer cohérente de l’emplacement même de la lamelle couvre-objet lorsque la tranche est imagée, même si l’origine x, y de réglage de la marque de fiducial change lorsque l’adaptateur de tube ou de la scène sont retirés et remplacés.
NOTE : Les plans Image sont generalement à intervalles de 0,5 µm et 2 µm, selon la taille et la résolution souhaitée de l’image fonctionnalités. Construction d’une image 3D de qualité exige que les caractéristiques de l’image s’étendent sur plusieurs plans d’acquisition, et afin de visualiser les petits éléments, des intervalles plus petits sont donc requis entre les plans.
Remarque : La plupart des séances d’imagerie a rapporté ici étaient sous 18 min/tranche. Cependant, nous avons photographié quelques tranches de dix ou plusieurs fois et même en tant que tant que les 40 min en une seule séance, sans dommage apparent dans la survie à long terme de la tranche.
Résultats
Pour déterminer la précision avec laquelle les repères peuvent être utilisés pour reimage les mêmes cellules dans les mêmes champs au fil du temps, nous avons examiné les tranches cultivées sur des lamelles de photodécoupe (Figure 6A). Les neurones ont été visualisées par la souillure avec un colorant vital (100 nM pendant 2 h ; ne tache pas les cellules non neuronales), qui disparaît de neurones au fil du temps sans léser les cellules25. Nous avons identifié un fiducial marque dans une maille unique (Figure 6A, B), trouve une région de neurones de marqués au colorant vitales 24h après marquage, inscrite le x et y compenser les positions de la marque de fiducial (Figure 6B) et recueillis, en utilisant un objectif X 60, piles d’image confocale de cette région, en répétant l’imagerie sur 4 jours consécutifs. Les images de projection maximale d’une pile d’image de 30 µm prises au même endroit apparaissent (Figure 6A-C). Bien que certaines modifications morphologiques se produisent au sein de la région pendant 4 jours, les cellules identiques (dont plusieurs sont marqués) peuvent être suivis au fil du temps. L’intensité de la fluorescence du colorant vital a diminué au fil du temps, mais les neurones étaient toujours clairement identifiables 4 jours après marquage. Bien que la majeure partie de la fluorescence du colorant vital neuronale est diffus dans le cytoplasme, certains coloration ponctuée était toujours observée, qui est devenu plus perceptible comme arrière-plan fluorescence a diminué. En tranches malsains, une coloration ponctuée des cellules non neuronales est également observée en tranches s’est détériorées. Ces résultats démontrent que les cellules identifiées dans des tranches peuvent être répétitive photographiées à l’aide de repères pour les trouver.
Pour analyser les changements temporels organisation neuronale et la viabilité dans les tranches au cours de la culture à long terme, nous avons suivi les mêmes tranches pendant 5 semaines, marquage avec le colorant vital neuronale fluorescent une fois par semaine, 24 h avant l’imagerie. Ronds multiples de coloration avec ce colorant vital pendant plusieurs semaines une accumulation accrue des agrégats. Neurones dans les tranches fraîchement plaquées qui se trouvaient encore dans les caillots plasmatiques ont été chargés avec de la teinture et imagée à un jour in vitro (1 DIV). Les mêmes tranches ont été projetés encore une fois par semaine pendant 5 semaines. Images provenant d’une seule tranche avec un objectif 4 x apparaissent (Figure 7A-E). Les couches de cellules pyramidales du CA et DG sont brillamment marqués lorsqu’il est excité à 488 nm et fluorescence d’émission mesurée à > 620 nm. Sur une période de 5 semaines d’observation 19 tranches, trois tranches est sorti la lamelle, et deux autres perdu leur morphologie typique et est devenu opaques, une indication de leur mort. Ainsi, un taux de survie d’environ 70 % pour les expériences sont à considérer et tranches supplémentaires prêts à assurer un nombre suffisant d’analyse. Tranches ont été préparés à un réglage de l’épaisseur nominale sur le chopper de tissus de 300 µm. Après 5 semaines de culture, nous avons mesuré l’épaisseur de tranche par imagerie neuronales vitales colorant coloré des tranches de la lamelle vers le haut par l’intermédiaire de la tranche avec un objectif 40 huile sur un microscope confocal de disque de filature. Perte d’activité des neurones s’est produite à une moyenne de 257 nm (n = 5 tranches ayant plusieurs sites utilisés par tranche), ce qui démontre que très peu l’amincissement de la tranche avait eu lieu depuis l’époque de placage. Nous ne pourrions pas mesurer avec précision l’épaisseur de la tranche en microscopie par fluorescence lors de placage car le colorant vital pris au piège dans le caillot de plasma a donné une fluorescence diffuse rend difficile de mesurer avec précision la position à laquelle est survenue la perte d’orientation. Cependant, des images 3D des neurones dans les tranches sont facilement obtenues en tranches après avoir retiré le caillot de plasma. La tranche de DIV 21, montré à faible grossissement dans la Figure 7F, a été photographié avec un objectif X 60 sur un microscope confocal (étapes de 1 µm) 3 jours après le chargement avec le colorant vital neuronal. Une image 3D de 60 µm a été construite à partir des plans focaux (Figure 7G). Les neurones et leurs processus de neurites qui sont étiquetés avec le colorant vital peuvent être suivis en 3D. Morphologie et structure 3D des tranches ont été bien entretenus pendant au moins 3 mois, et les temps plus longs ont été utilisés dans la présente étude.
Des changements majeurs dans la morphologie d’une position déjà image a également eu lieu dans certaines tranches, ce qui suggère de ce mouvement de la tranche sur la lamelle peut avoir lieu. Certes, sur des intervalles plus longs entre l’imagerie, il devint plus difficile de savoir avec certitude que les cellules dans le domaine étant imagé étaient identiques à celles observées lors des sessions précédentes d’imagerie. Ainsi, images de projection maximale de piles confocal de tranches de marqués au colorant vitales acquis à l’emplacement identique avec l’objectif de 60 x à intervalles hebdomadaires s’étendant sur 4 semaines (Figure 8) montrent que la viabilité neuronale est bien entretenue, mais qu’il est difficile d’identifier un neurone spécifique au fil du temps, quand les images sont obtenues avec longs intervalles de temps entre les sessions. Vraisemblablement le modèle de cellules marquées avec plusieurs fluorophores serait plus facile à reconnaître, comme le sont les cellules en groupes localisés quand observée en faisant défiler une pile d’image.
Afin d’évaluer l’utilité des différents vecteurs viraux pour l’introduction d’un gène exogène dans les neurones des tranches d’hippocampe, nous avons comparé la tranche infectiosité utilisant AV et AAV recombinants vecteurs LENTIVIRAUX, chacun exprimant les différentes balises fluorescentes ou à l’aide promoteurs différents à l’expression de la voiture. AV (2 x 107 infectieuse U/tranche) exprimant cofilin-RDRF derrière un promoteur de CMV fort, cellules non spécifique a été utilisé pour infecter une tranche hippocampe de souris qui avait été cultivées 9 semaines sur les lamelles. Expression de cofilin-RDRF a été trouvée tout au long de la tranche à 5 jours après l’infection, avec une expression plus intense autour de la périphérie de tranche tel qu’observé avec un objectif de 4 x (Figure 9A). Les cellules dans la tranche exprimant cofilin-RDRF ont été également observés avec un objectif 20 X (Figure 9B), avec quelques taches ponctuée lumineux et diffusent également l’expression dans les neurones et les cellules non neuronales. Après 17 semaines de culture (infection après 8 semaines), tiges de cofilin spontanées avaient formé dans certaines cellules, vraisemblablement entraîné par la surexpression de la souche sauvage de26,cofilin-RDRF (Figure 9C)27.
Nous avons également démontré que AAV (1010 particules) pourraient être utilisées pour l’expression en tranches.Images de tranches infectés à 9 semaines de culture avec AAV, dans lequel un promoteur spécifique pour synapsin neuronal a conduit expression de GCaMP5-cofilin-RDRF avec une séquence de peptide P2A auto clivage dans l’éditeur de liens de la polyprotéine traduit21, ont été capturés 8 semaines après l’infection (17 semaines de culture). Dans les neurones exprimant les GCaMP5 et les cofilin-RDRF, quelques tiges de cofilin/agrégats formés (Figure 9,D-F). L’intensité de la fluorescence des tiges/agrégats était si forte que très peu de fluorescence d’un cofilin-RDRF diffuse pourrait être observée sans saturation complète et florissant de l’image de fluorescence cofilin des tiges. Cofilin spontanée tiges apparaissent dans les neurones dans lequel chimères de la protéine fluorescente cofilin sauvage ont été surexprimé26,27, ainsi que dans a souligné neurones10. Basé sur les titres d’adénovirus, qui sont déterminés sur la base d’infectiosité14 et le nombre de particules utilisé pour déterminer le titre d’AAV, environ 100 à 500 fois plus grand nombre de particules d’AAV est nécessaires pour obtenir approximativement le même pouvoir infectieux / expression en tranches par rapport à l’AV.
Pour suivre les recombinante expression induite par le lentivirus des protéines fluorescentes en tranches, tranches ont été infectés à 6 DIV avec 1, 3, 10 et 30 µL d’extraits d’un lentivirus recombinant pour expression spécifique neuronale (synapsin promoteur) de cofilin-R21Q-RDRF, développé comme un sonde pour cofilin-actine tige formation16d’imagerie de cellules vivantes. Tranches ont été marqués avec le colorant vital neuronale à 11 DIV et imagés dans des régions spécifiques pour la teinture et l’expression de cofilin-R21Q-RDRF sur DIV. 12 et 14 La tranche infectée par la quantité de 30 µL du virus n’ont pas survécu pour l’imagerie, mais les tranches en triple traitement avec les autres volumes de virus ont montré une expression dose-dépendante des RDRF. La figure 10 montre les images des tranches infectés par 1 µL et 10 µL de lentivirus à 6 et 8 jours après l’infection. Plusieurs régions de deux tranches différentes ont été quantifiées pour coloration conjointement des neurones avec le colorant vital et RDRF expression. Pour obtenir des tranches infectés par 1 µL de lentivirus, environ 28 % des neurones exprimé RDRF à 6 jours après l’infection, passant à 85 % par 8 jours après l’infection. Pour obtenir des tranches infectées avec 10 µL de lentivirus, environ 58 % des neurones exprimé RDRF à 6 jours après l’infection, augmentant à 86 % en 8 jours après l’infection. Ainsi, 1 µL de la lentivirus était suffisant pour fournir l’infectivité tranche généralisée et l’expression neuronale de 8 jours après l’infection.
Afin de démontrer que ce système de culture est utile dans le développement subséquent de la pathologie cofilin, tranches infectés par le lentivirus pour exprimer des cofilin-R21Q-RDRF dans les neurones ont été laissées non traitées ou traitées avec différentes concentrations (1 µM, 333 nM et 100 nM) de synthétique Aβ protéine humaine qui avait fait l’objet d’incubation au formulaire oligomères28. Résultats d’études antérieures ont démontré que synthétique Aβo induisent des tiges cofilin-actine chez 25 % des neurones de l’hippocampe dissociés29,30,31. Toutes les tranches de trois traités avec la concentration de 1 µM d’Aβ s’est détachés de la lamelle dans les 24 premières heures, alors que tous les véhicules traitement tranches (contrôle) et ceux traités avec le 333 nM et les 100 nM concentrations de bêta-amyloïdes a survécu pendant les deux semaines qu’ils ont été suivis. Les mêmes régions cellulaires (CA1, CA3 et DG) dans une tranche traités avec 100 nM Aβo ont été imagées (60 x, objectif) sur plusieurs jours. Tranches de contrôle qui ont été infectés sur 6 DIV avec lentivirus pour exprimer la synapsine conduit cofilinR21Q-RDRF avaient expression RDRF cellulaire diffuse par 15 DIV (Figure 11A). Tranches exposé à 100 nM Aβo à 14 DIV et imagé à DIV 15 ont montré que la distribution de cofilin-R21Q-RDRF est devenue ponctuée, apparaissant dans les deux tiges en forme de structures et agrégats (Figure 11B). Ces structures est devenu plus visible 6 jours après traitement de bêta-amyloïdes (Figure 11C, qui correspond au champ même de cellules comme Figure 11B). Dans de nombreux endroits riches en neurites où somas de cellules neuronales sont absents (Figure 11D), des baies ponctuées et bâtonnet de cofilinR21Q-RDRF développé (flèches dans la Figure 11C), similaire à la distribution de cofilin-actine tiges signalé précédemment dans les neurites des neurones Aβ imprégnées dans la culture29,30,31. Ainsi, cette nouvelle méthode pour la mise en culture et en observant des tranches d’hippocampe permettra aux utilisateurs de déterminer la viabilité à long terme des cellules sous forme de barres et de quel cofilin agrège et la réversibilité de la pathologie à divers stades de développement et facilement effectuer des mesures de dose-réponse sur les réactifs susceptibles de bloquer ou d’inverser la formation de la pathologie cofilin dans un plus en vivo-comme l’organisation cellulaire.

Figure 1 : Préparation de support rouleau. (A) modèle pour marquer l’emplacement des trous de forage sur le fond de plat de vitroplants de 15 cm. Si le chiffre est imprimé à la taille de la barre d’échelle illustrée, peut être découpé et utilisé pour marquer les positions sur une boîte de Petri de 15 cm pour percer les trous indiqués. (B) support rouleau muni de deux tubes insérés. Chaque grille est numéroté sur une étiquette bien visible sur le dessus de la grille. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.

Figure 2 : Préparation des tubes culture rouleau. Vue avant (A) sur le tube inséré dans un gabarit sur une perceuse pour percer le trou de 6 mm dans le tube. Ligne pointillée indique la position du tube face plate dans le gabarit. (B) fin vue le gabarit avec le tube inséré et foret aligné sur le trou. (C) vue de dessus du trou dans le gabarit pour le perçage des tubes roulés avec une mèche de 6 mm.Flèche blanche indique la position de l’ongle de coupure insérée comme une halte pour le positionnement des tubes et des lignes doubles noires sont pour l’alignement du foret. Tubes de printemps (D) clips (flèche noire), installées de façon sûre sur le fond de la sauteuse pour le maintenir en position lors du perçage. (E) après avoir percé le trou, les bords sont lissées avec un outil d’ébavurage et de rainures sont coupées sur le côté intérieur du trou (encart montre le trou vu à travers un microscope à dissection) pour améliorer le drainage moyen du trou pendant la rotation du tube. (F) Culture tube avec le trou aligné sur un trou dans l’adhésif de caoutchouc de silicone, auquel sera joint le couvre-objet. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
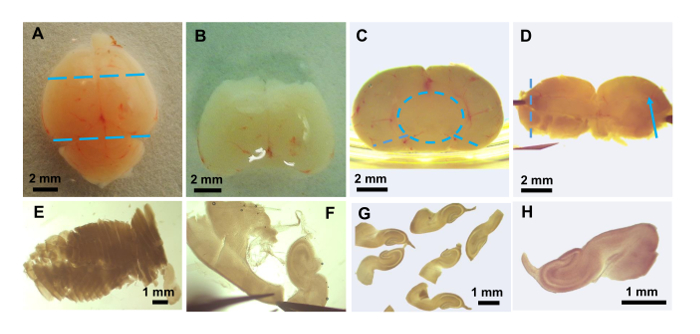
Figure 3 : Préparation de tranches de cerveau hippocampique. Photos prises avec un affichage de microscope de dissection : cerveau de souris intactes (A). Position des découpes pour retirer le cerveau et le cervelet sont montrés comme des lignes pointillées bleues. (B) après le retrait du cerveau et le cervelet. (C) morceau de cerveau de B est basculé à 90 ° avec la région occipitale (vers le cervelet) vers le haut. Positionnement de la pièce à côté du côté du plat aide à la suppression du mésencéphale (cercle en pointillés bleu) qui peut être taquiné loin de l’hippocampe, thalamus et hypothalamus restants. Deux coupes à l’aide de la pince (lignes pointillées bleues) permettent le morceau restant contenant l’hippocampe des deux hémisphères pourront s’étaler à plat. (D) le cerveau aplati pièce montrant le vaisseau sanguin le long de la fissure hippocampe (flèche bleue). Ce tissu est placé sur la feuille de plastique et transféré dans le hachoir de tissu à couper dans le sens de la ligne pointillée. (E) en tranches de tissu montrant un peu plus de la moitié de l’hippocampe après être retourné au GBSS/glucose. (F) finale dissection de l’hippocampe et le nettoyage des tranches pour enlever la matière non-hippocampique. (G) flottant plusieurs tranches après nettoyage final. (H) photo agrandie d’une seule tranche pour le transfert de la lamelle couvre-objet. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.

Figure 4 : Tranches d’électrodéposition et incubation. Tranche hippocampe de souris (A) A est supprimée de la boîte de Petri, sur la pointe d’une spatule à l’aide de la pointe de la pince pour contribuer à sortir il est exempt de la solution. (B), la tranche est placé à plat dans le centre d’une photodécoupe et traités 12 mm lamelle sur 2 µL de plasma de poulet et un autre 2,5 µL d’un mélange de 1:1 plasma/thrombine est ajoutée pour générer un caillot. (C) une fois que le caillot est défini (environ 1-2 min), le film est retire du cercle adhésif caoutchouc silicone sur un tube d’enroulement et la lamelle est positionnée avec le caillot centré dans le trou ; puis la lamelle est pressée en place avec un pouce et maintenue en position pendant environ 1 min. (D) ajouter 0,8 mL de milieu de culture complet. (E) rouleau tube titulaires à l’intérieur d’un incubateur de grand rouleau avec le front soulevé pour incliner 5 ° pour garder le milieu au bas des tubes. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.

Figure 5 : Support de tube de galet et stade plaque incubateur. (A) porte-Tube qui positionne le tube tel que la lamelle est maintenue en position pour l’imagerie. (B) porte-Tube monté sur une platine en adaptateur microscope. Curseurs sur le côté tenir le tube en toute sécurité pour l’imagerie. Étage (C) adaptateur avec les panneaux de porte et côté tube ajouté contenant bandes de chauffage reliés à un contrôleur de température thermoélectrique. Une fois que les tubes sont montés et positionnés, un massif à la boîte peut être ajouté pour aider à maintenir la température au cours de l’imagerie. Fil orange est le fil de thermocouple. Les plans pour la conception et la construction de l’adaptateur de l’étape et le chauffage sont disponibles à : https://vpr.colostate.edu/min/custom-machining/roller-tube-holder/. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.

Figure 6 : À l’aide de repères pour l’imagerie des mêmes cellules répétitive. (A) hippocampe tranche sur la lamelle (mailles de 1 mm) de photodécoupe avec subiculum déplié (queue) lus avec 4 x illumination champ objective et lumineux. Courbure du tube d’enroulement en plastique permet de créer un éclairage oblique qui améliore la visualisation de la grille. La boîte affiche la position et la taille d’un terrain de X 60. (B), une vue de la même tranche avec un objectif de 20 x pour trouver la marque de fiducial comme la pointe du bas de la 4 place 34. Les décalages x et de y sont présentés de façon reproductible localiser le centre de la zone souhaitée pour l’imagerie confocale grossissement plus élevé. (C–F) Une tranche marquée par neuronale agent vital que 13 DIV a été photographié à l’aide d’un objectif 60 x et ce qui rend une image de projection 30 µm sur 4 jours consécutifs (14-17 DIV). Neurones identiques ont été projetés chaque jour. La position du noyau dans chacune des trois neurones est indiquée par un symbole différent. Ils sont plus facilement identifiées en faisant défiler les piles image puisque leur change de position 3D légèrement. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
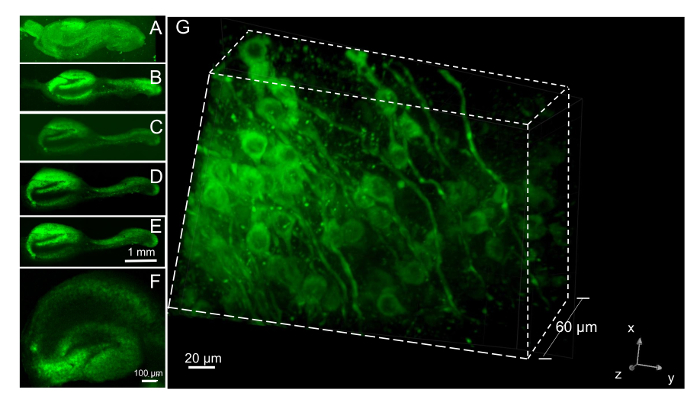
Figure 7 : Imagerie des neurones en tranches avec un colorant vital neuronal. (A) colorant vital neuronale teinté tranche en caillot de plasma prélevé 24h après ensemencement avec objectif 4 X. Colorant (100 nM) a été ajouté lors de la tranche a été d’abord placée dans le porte-tube de rouleau et délavée 2 h plus tard.Subiculum est enroulée autour de l’hippocampe dans le caillot. (B–E) Après dissolution du caillot par la plasmine ajouté à 6 DIV, la tranche a été rechargée 5 fois avec le colorant vital 24h avant l’imagerie à intervalles hebdomadaires. Les images ont été recueillies à 8, 21, 28 et 35 DIV (B–E, respectivement). (F) tranche Hippocampal cultivés pendant 3 semaines et teinté avec colorant vital neuronale 24h avant l’imagerie avec objectif 4 X. (G) Confocal pile d’images sur la même tranche que dans F montrant une vue 3D de 61 avions prises à intervalles de 1 µm. Neuronale colorant vital étiquettes clairement les neurites et corps cellulaire, mais il est exclu du noyau. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
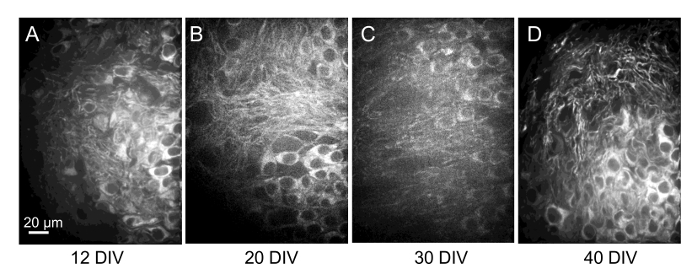
Figure 8 : Répétitives d’imagerie des neurones dans un seul endroit de la tranche pendant 4 semaines. Images de projection maximale de 30 piles d’image confocale µm du champ même de cellules (par positionnement) de tranches de marqués au colorant vitales prises à 12, 20, 30 et 40 DIV (A–D, respectivement). Il est difficile d’identifier répétitive des cellules individuelles dans des délais plus longs dans les images de projection. Cependant, même au cours de ces longues périodes, identification des cellules mêmes est souvent possible en faisant défiler les piles d’image ou créer des images 3D qui peut être tourné, comme illustré à la Figure 7G. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
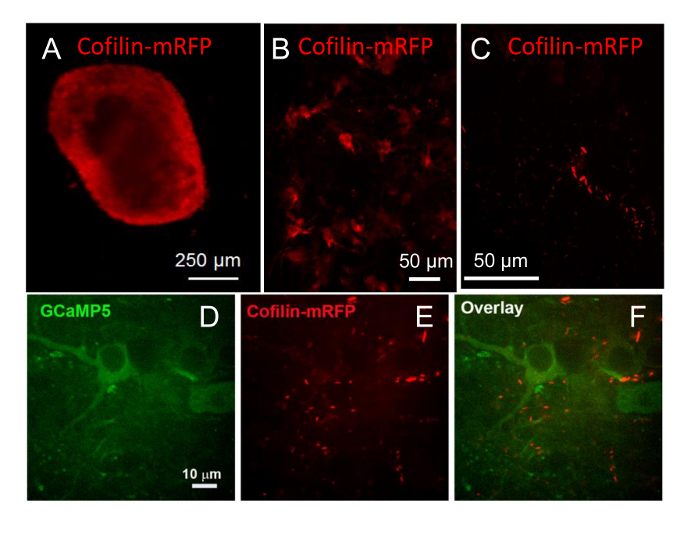
Figure 9 : Expression par l’intermédiaire d’origine virale et l’imagerie des protéines fluorescentes. (A) tranche Hippocampal cultivés pendant 9 semaines et infectées par l’adénovirus pour exprimer cofilin-RDRF derrière un promoteur de CMV. Expression a été trouvée tout au long de la tranche à 5 jours après l’infection, mais fluorescence a été plus brillant près de la périphérie de tranche. (B), à la même tranche a montré l’expression dans les cellules plus profondément dans la tranche vu avec objectif 20 X. Image est qu'une projection d’une pile de 20 images espacé dehors 2 µm. (C) la même tranche a été examiné après 17 semaines de culture (infection après 8 semaines) et RDRF cofilin a été observée dans les agrégats de la tige en forme comme le montre cette image de projection par une cheminée de 70 µm de 23 images, 3 µm en dehors, prise avec un objectif X 40. (D–F) Tranche hippocampe de souris infectée à 9 semaines de culture avec un AAV exprimant un GCaMP5-(P2A) - cofilin - RDRF derrière un promoteur synapsin. Fluorescence était visible dans les canaux rouges et verts après 10 jours. Une image du seul plan de la tranche montrant l’expression de (D) GCaMP5, une journaliste de calcium sensibles, (E) de nombreuses tiges contenant du cofilin et (F) une superposition d’image. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
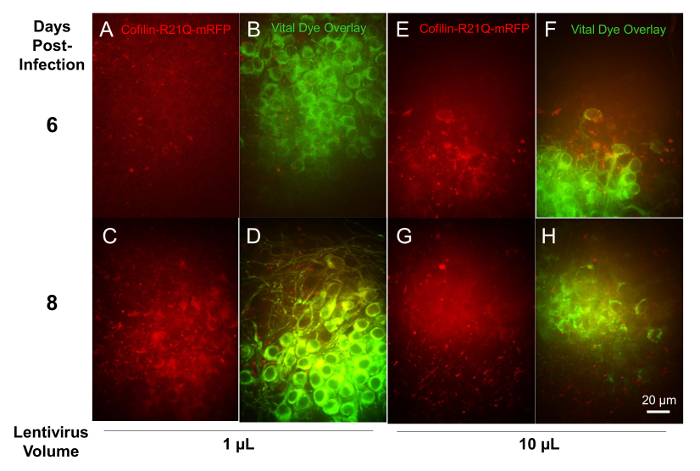
Figure 10 : Expression de cofilin-R21Q-RDRF pilotée par un promoteur synapsin dans les neurones infectés par des vecteurs LENTIVIRAUX recombinants. Détection d’un signal de fluorescence faible est observée par sur 3-4 jours après l’infection à l’aide de 10 µL de virus et peut être utilisable par 5 ou 6 jours, (E) comme on le voit dans ces images acquises avec un objectif 60 x. Même si cela prend plus de temps pour atteindre les mêmes niveaux d’expression avec 1 µL de virus, de 8 jours après l’infection, un fort pourcentage similaire des neurones (colorant vital étiqueté) exprimaient le cofilin-R21Q-RDRF. Seulement 27 % des neurones étaient positifs pour RDRF fluorescence à 6 jours après infection avec 1 µL (A, B), mais cela a augmenté de 85 % (C, D) de 8 jours. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
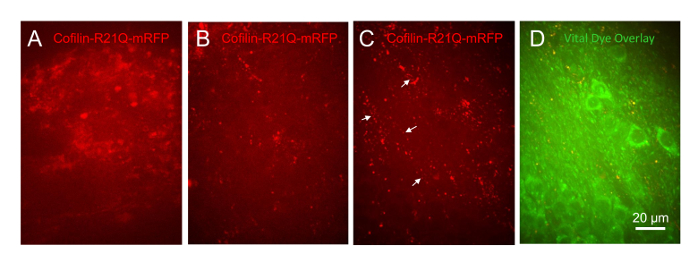
Figure 11 : Pathologie induite par l’oligomère cofilin Aβ dans des tranches d’hippocampe souris. Toutes les images prises comme 30 µm image empile avec un objectif de 60 x et sont présentés sous forme d’images de projection maximum. Tranches ont été infectés avec cofilin-R21Q-RDRF à 6 DIV. tranche (A) 15 DIV traités sur 14 DIV avec véhicule (DMSO/jambons F12 moyen utilisé pour générer l’Aβo). (B) tranche 15 DIV traités avec 100 nM Aβo. (C) même champ que dans B prise à 20 DIV et montré en (D) en surimpression avec étiquette neuronale colorant vital. Flèches indiquent les tableaux linéaires des agrégats de cofilin et de tiges dans la région de la tranche contenant des neurites, mais quelques corps cellulaires. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
Discussion
La méthode du tube rouleau décrite ici permet de culture à long terme et une imagerie direct du tissu cérébral en tranches. Un problème majeur avec la technique de tranche appliquée ici est dans le montage et l’entretien des tranches. Revêtements de lamelle qui prennent en charge la tranche adhérence, promouvoir l’amincissement de la tranche en améliorant l’excroissance des neurites et la migration des cellules hors la tranche ; ainsi, nous avons évité l’utilisation de ces substrats. L’insertion des groupes aminés sur le verre par un traitement avec 3-aminopropyltriethoxysilane améliore l’adhérence des tranches, mais trop ou trop peu plasma de poulet sur la lamelle peut également causer des problèmes d’adhérence conduisant à la perte de la tranche. Le volume du plasma nécessaire pour une bonne adhérence dépend de la taille de la tranche de cerveau cultivé et ainsi est plus grand pour les tranches d’hippocampe de rat, qui sont environ 4 fois plus grandes en superficie que les coupes de cerveau de souris. Si trop plasma caillots sous la tranche, adhésion cellulaire à la lamelle est altérée et traitement avec plasmine se desserre la tranche pour qu’il change de position ou se détache complètement. Cependant, trop peu de plasma dans le caillot peut conduire à la perte de tranche pendant les premiers jours de rotation dans l’incubateur. Dans une récente expérience impliquant des 39 tranches, trois ont été perdus, mais certaines de ces pertes ont pu tranche les dommages survenus pendant le processus de découpage en tranches. Néanmoins, nous nous préparons normalement environ 50 % des tranches plus que le nombre estimé nécessaire pour l’expérience. La deuxième cause principale des problèmes de la culture est la fuite du milieu autour du joint de la lamelle couvre-objet. Ce problème s’aggrave lorsque les lamelles ne sont pas fermement maintenus en position pendant au moins 1 min après leur apposer le sceau. Chaleur du pouce permet d’appliquer la pression probablement aide à terminer l’adhérence. Fuite qui se produit est souvent par le biais de canaux d’air minuscules sous la lamelle qui peuvent être observées avec un microscope à dissection. Ceux-ci disparaissent généralement sur l’utilisation de pression prolongée avec le pouce. Perte d’environ 2 % des cultures due à une fuite lente peut s’attendre, et donc il est conseillé d’attendre 10 jours après la mise en place des cultures avant d’effectuer les infections virales. Pression du pouce excessif, surtout si le produit inégalement dans l’ensemble de la lamelle couvre-objet, peut également causer la lamelle à craquer. Si la rupture est un problème, en appuyant sur les tubes à plat sur un tapis de souris en caoutchouc chauffé dans un incubateur pourrait aider à fournir une pression plus uniforme dans l’ensemble de la lamelle.
Décrit précédemment, méthodes de culture de tranche de cerveau sur la membrane à l’interface air-liquide (système ouvert) ou sur une lamelle de verre à l’intérieur d’un tube scellé en plastique (système fermé) sont très efficaces pour la survie à long terme de tranche, mais chaque méthode a ses points forts et faiblesses. Cultures de tranche sur la membrane à l’interface liquide air sont avantageuses pour la combinaison des études électrophysiologiques avec objectifs à immersion pour l' imagerie haute résolution7, mais présentent des inconvénients pour ce qui est de trouver le champ exact des cellules pour place dans le temps et risque de l’utilisateur et contamination objective lors de l’utilisation d’expression des gènes viraux. Utilisation de virus pour l’expression des transgènes est plus sûre et plus facile à réaliser dans un système fermé où la contamination des objectifs de microscope n’est pas un problème. Notre méthode de tube mis à jour le rouleau permet d’accéder de la tranche pour l’imagerie haute résolution, même si ce n’est pas favorable à des études électrophysiologiques.
Conditions de culture tranche ont été établies pour de nombreuses régions du cerveau rongeurs2, mais ici nous utilisons un hippocampe seulement parce que c’est l’une des régions cérébrales plus largement étudié et changements qui se produisent dans l’hippocampe sont d’un grand intérêt dans l’étude de troubles cognitifs. Les couches de cellules pyramidales du CA et DG maintiennent leur organisation au cours de plusieurs semaines de culture et peuvent être facilement observés morphologiquement. Nous avons utilisé une viabilité neuronale fluorescent nouvellement développés marqueur25, qui a des propriétés de fluorescence qui lui permettent d’être utilisé pour surveiller la viabilité neuronale et organisation dans des tranches d’hippocampe sur des périodes de jours au mois mais qui est aussi compatible avec l’utilisation de nombreuses autres protéines fluorescentes et les journalistes. Bien que pas optimal pour NeuO fluorescence25, nous pouvons exciter à NeuO à 488 emission nm et mesure à > 617 nm. Repères sur les lamelles de photodécoupe aidé à localiser les mêmes cellules répétitivement sur plusieurs jours de culture et nous a permis aux régions d’images identiques des tranches sur plusieurs semaines. Pratiquement aucun important amincissement des tranches a été constatée sur les lamelles de verre mis à jour le 5 semaines de culture, le point de temps plus longue pour laquelle nous avons obtenu des mesures d’épaisseur de tranche.
AV, AAV et vecteurs recombinants lentivirus fonctionnent bien pour exprimer des gènes exogènes en tranches. Lentivirus avec un promoteur spécifique neuronal est particulièrement utile pour l’obtention de l’expression dans un pourcentage très élevé (> 85 %) des neurones dans les 8 jours après l’infection. De plus, nous montrons que la pathologie de tige de cofilin-actine associée au développement des déficits cognitifs chez l’humain AD10,11 et Aβ surexprimant souris AD modèles32 peut être surveillée en tranche cultures traitées avec des concentrations relativement faibles (100 nM) de synthèse humaine Aβo. Nous envisageons que les futures applications de cette méthode inclura caractériser de nouvelles thérapies pour inverser la pathologie tige cofilin-actine et/ou anomalies épine dendritique correcte qui se produisent dans de nombreux troubles neurologiques33.
matériels
| Name | Company | Catalog Number | Comments |
| Bottoms from 15 cm culture dishes | VWR Scientific | 25384-326 | |
| Phillips Head Machine Screws (#10-32) | Ace Hardware | 2.5" long and 3/16" in diameter | |
| Flat Washers #10 | ACE Hardware | ||
| Machine Screw Nuts (#10-32) | ACE Hardware | ||
| Rubber Grommets | ACE Hardware | 5/16", thick; 5/8", hole diameter; 1.125", OD | |
| Polyethylene tubing (5/16"; OD, 3/16"; ID) | ACE Hardware | Cut to 1.8" length | |
| Lock Washer #10 | ACE Hardware | ||
| Drill Press, 5 speed | Ace Hardware | ProTech Model 1201 | |
| Nunclon Delta Flat-Sided Tubes | VWR | 62407-076 | |
| Drill bits, 3 mm, 6 mm and 15 mm | Ace Hardware | Diablo freud brand | Drill bits for cutting plastic. |
| Drill bits for wood, 1.5 cm and 1 mm | Ace Hardware | ||
| Wood file, 1/4" round | Ace Harware | ||
| Spring clips, 16 mm snap holder | Ace Hardware | ||
| Swivel Head Deburring Tool, 5" | Ace Hardware | 26307 | |
| Adhesive Silicone Sheet (Secure Seal) | Grace Bio-Labs | 666581 | 0.5 mm Thickness |
| 6 mm hole punch | Office Max | ||
| 12 mm hole punch | thepunchbunch.com | ||
| 70% Ethanol | |||
| Phototeched Coverslips, 12 mm diameter | Bellco Glass, Inc. | 1916-91012 | |
| Bunsen Burner | |||
| Absolute Ethanol | |||
| Nanopure Water | |||
| 3-aminopropyltriethoxylane | Sigma-Aldrich | A3648 | |
| Acetone | Sigma-Aldrich | 179124 | |
| #5 Dumont Forceps | Fine Science Tools | 11251-30 | |
| McIlwain Tissue Chopper | Ted Pella, Inc. | 10180 | |
| Double Edge Razor Blades | Ted Pella, Inc. | 121-6 | |
| Whatman Filter Paper | VWR | 28450-182 | Cut into 5.8 cm diameter circles |
| Poly-chloro-trifluoro-ethylene (Aclar) | Ted Pella, Inc. | 10501-10 | Cut into 5.8 cm diameter circles |
| #21 Surgical Blade | VWR Scientific | 25860-144 | |
| #5 Dumont Forceps | Fine Science Tools | 11251-30 | |
| Spatula, stainless with tapered end | VWR | 82027-518 | |
| Gey's Balanced Salt Solution | Sigma-Aldrich | G9779 | |
| Glucose | ThermoFisher Scientific | 15023-021 | 25% (w/v) Solution, 0.2 mm filter sterilized |
| Chicken Plasma | Cocalico Biologicals | 30-0300-5L | Rehydrate in sterile water, centrifuge at 2500 x g 30 min at 4 °C, quick freeze aliquots in liquid nitrogen and store at -80 °C. |
| Thrombin, Topical (Bovine) | Pfizer | Thrombin-JMI | Quick freeze aliquots in liquid nitrogen at 1,000 international units/mL in diluent provided and store at -80°C. Use at 250 units/mL. |
| Cell Roller System | Bellco Biotech | SciERA | |
| Roller Incubator | Forma | Model 3956 | |
| N21-MAX | ThermoFisher Scientific | AR008 | |
| Pen/Strep (100X) | ThermoFisher Scientific | 15140122 | |
| 200 mM Glutamine | ThermoFisher Scientific | 25030081 | |
| Glucose | ThermoFisher Scientific | 15023-021 | 25% (w/v) Solution, 0.2 mm filter sterilized |
| Neurobasal A | ThermoFisher Scientific | 10888-022 | Complete Medium: 48 mL Neurobasal A, 1 mL N21-MAX, 0.625 mL 200 mM Glutamine, 0.180 mL 25% Glucose, 0.250 mL 100x pen/strep. |
| Third generation lentivirus packaging | Life Technologies | K4975-00 | |
| 159 K cutoff centrifugal filters (Centricon) | EMD Millipore | ||
| Lentiviral cloning system (InFusion) | Clonetech | ||
| Plasmids 30323, 50856, 51279 | Addgene | ||
| Neuronal cell viability dye (NeuO) | Stemcell technologies | 1801 | Thaw once and quick freeze in 4 µL aliquots. Store at -20 °C |
| Inverted microscope | Olympus | IX83 | |
| Microscope objectives | Olympus | air: 4X, 20; oil: 40X, 60X, | |
| Spinning disc confocal system | Yokagawa | CSU22 | |
| Microscope EMCCD camera | Photometrics | Cascade II | |
| Linear encoded (x,y), piezo z flat top stage | ASI | ||
| Microscope lasers and integration | Intelligent Imaging Innovations | ||
| HEK293T cells | American Type Culture Collection | CRL-3216 | |
| Human Plasmin | Sigma Aldrich | P1867 | 0.002 U/mL in 0.1% bovine serum albumin (0.2 mm filter sterilized), quick freeze in liquid nitrogen and store at -80 °C. |
Références
- Brewer, G. J., Torricelli, J. R., Evage, E. K., Price, P. J. Optimized survival of hippocampal neurons in B27-supplemented Neurobasal, a new serum-free medium combination. J Neurosci Res. 35 (5), 567-576 (1993).
- Humpel, C. Organotypic brain slice cultures: a review. Neuroscience. 305, 86-98 (2015).
- Stoppini, L., Buchs, P. A., Muller, D. A simple method for organotypic cultures of nervous tissue. J Neurosci Methods. 37 (2), 173-182 (1991).
- Gähwiler, B. H. Organotypic monolayer cultures of nervous tissue. J Neurosci Methods. 4 (4), 329-342 (1981).
- Gogolla, N., Galimberti, I., DePaola, V., Caroni, P. Long-term imaging of neuronal circuits in organotypic hippocampal slice cultures. Nat Protoc. 1 (3), 1223-1226 (2006).
- Roo, M. D., Ribic, A. Analyzing structural plasticity of dendritic spines in organotypic slice culture. Methods Mol Biol. 1538, 277-289 (2017).
- Lee, K. F. H., Soares, C., Thivierge, J. -. P., Béīque, J. -. C. Correlated synaptic inputs drive dendritic calcium amplification and cooperative plasticity during clustered synapse development. Neuron. 89 (4), 784-799 (2016).
- Davis, R. C., Maloney, M. T., Minamide, L. S., Flynn, K. C., Stonebraker, M. A., Bamburg, J. R. Mapping cofilin-actin rods in stressed hippocampal slices and the role of cdc42 in amyloid-beta-induced rods. J Alzheimers Dis. 18 (1), 35-50 (2009).
- Clark, R. E., Squire, L. R. Similarity in form and function of the hippocampus in rodents, monkeys, and humans. Proc Natl Acad Sci U S A. 110, 10365-10370 (2013).
- Minamide, L. S., Striegl, A. M., Boyle, J. A., Meberg, P. J., Bamburg, J. R. Neurodegenerative stimuli induce persistent ADF/cofilin-actin rods that disrupt distal neurite function. Nature Cell Biol. 2 (9), 628-636 (2000).
- Rahman, T., et al. Cofilin rods and aggregates concur with tau pathology and the development of Alzheimer's disease. J Alzheimers Dis. 42 (4), 1443-1460 (2014).
- Bamburg, J. R., Bernstein, B. W. Actin dynamics and cofilin-actin rods in Alzheimer disease. Cytoskeleton(Hoboken). 73 (9), 477-497 (2016).
- He, T. C., Zhou, S., da Costa, L. T., Yu, J., Kinzler, K. W., Vogelstein, B. A simplified system for generating recombinant adenoviruses. Proc Natl Acad Sci U S A. 95 (5), 2509-2514 (1998).
- Minamide, L. S., et al. Production and use of replication-deficient adenovirus for transgene expression in neurons. Methods Cell Biol. 71, 387-416 (2003).
- Kügler, S., Kilic, E., Bähr, M. Human synapsin 1 gene promoter confers highly neuron-specific long-term transgene expression from an adenoviral vector in the adult rat brain depending on the transduced area. Gene Ther. 10 (4), 337-347 (2003).
- Mi, J., et al. A genetically encoded reporter for real-time imaging of cofilin-actin rods in living neurons. PLOS ONE. 8 (12), 83609 (2013).
- Wang, L., Blouin, V., Brument, N., Bello-Roufal, M., Francois, A. Production and purification of recombinant adeno-associated vectors. Methods Mol Biol. 807, 361-404 (2011).
- Matsushita, T., et al. Adeno-associated virus vectors can be efficiently produced without helper virus. Gene Therapy. 5 (7), 938-945 (1998).
- Ward, P., Walsh, C. E. Targeted integration of rAAV vector into the AAVS1 region. Virology. 433 (2), 356-366 (2012).
- Akerboom, J., et al. Optimization of a GCaMP calcium indicator for neural activity imaging. J Neurosci. 32 (40), 13819-13840 (2012).
- Kim, J. H., et al. High cleavage efficiency of a 2A peptide derived from porcine teschovirus-1 in human cell lines, zebrafish and mice. PLOS ONE. 6 (4), 18556 (2011).
- Benskey, M. J., Manfredsson, F. P. Lentivirus production and purification. Methods Mol Biol. 1382, 107-114 (2016).
- Huang, L., Chen, C. Autoprocessing of human immunodeficiency virus type 1 protease miniprecursor fusions in mammalian cells. AIDS Res Ther. 7, 27 (2010).
- Bordat, A., Houvenaghel, M. C., German-Retana, S. Gibson assembly: an easy way to clone polyviral full-length infectious cDNA clones expressing an ectopic VPg. Virol J. 12, 89 (2015).
- Er, J. C., et al. NeuO: a fluorescent chemical probe for live neuron labeling. Angew Chem Int Ed Engl. 54 (8), 2242-2246 (2015).
- Bernstein, B. W., Chen, H., Boyle, J. A., Bamburg, J. R. Formation of actin-ADF/cofilin rods transiently retards decline of mitochondrial potential and ATP in stressed neurons. Am J Physiol Cell Physiol. 291 (5), 828-839 (2006).
- Cichon, J., et al. Cofilin aggregation blocks intracellular trafficking and induces synaptic loss in hippocampal neurons. J Biol Chem. 287 (6), 3929-3939 (2012).
- Stine, W. B., Dahlgren, K. N., Krafft, G. A., LaDu, M. J. In vitro characterization of conditions for amyloid-beta peptide oligomerization and fibrillogenesis. J Biol Chem. 278 (13), 11612-11622 (2003).
- Maloney, M. T., Minamide, L. S., Kinley, A. W., Boyle, J. A., Bamburg, J. R. Beta-secretase-cleaved amyloid precursor protein accumulates at actin inclusions induced in neurons by stress or amyloid beta: a feedforward mechanisms for Alzheimer's disease. J Neurosci. 25 (49), 11313-11321 (2005).
- Davis, R. C., et al. Amyloid beta dimers/trimers potently induce cofilin-actin rods that are inhibited by maintaining cofilin phosphorylation. Mol Neurodegener. 6, 10 (2011).
- Walsh, K. P., et al. Amyloid-β and proinflammatory cytokines utilize a prion protein-dependent pathway to activate NADPH oxidase and induce cofilin-actin rods in hippocampal neurons. PLOS ONE. 9 (4), 95995 (2014).
- Woo, J. A., et al. RanBP9 at the intersection between cofilin and Aβ pathologies: rescue of neurodegenerative changes by RanBP9 reduction. Cell Death Disease. , 6 (2015).
- Shaw, A. E., Bamburg, J. R. Peptide regulation of cofilin activity in the CNS: a novel therapeutic approach for treatment of multiple neurological disorders. Pharmacol Ther. 175, 17-27 (2017).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon