Method Article
Imágenes de bioluminiscencia en tiempo real de la dinámica de señalización de muesca durante la neurogénesis de la murino
En este artículo
Resumen
Células neurales del tallo/progenitor exhiben varias dinámicas de expresión de los componentes de señalización de la muesca que conducen a diferentes resultados de los eventos celulares. Dicha expresión dinámica puede revelarse mediante monitoreo en tiempo real, no mediante análisis estáticos, utilizando un sistema de imágenes de bioluminiscencia altamente sensible que permite la visualización de cambios rápidos en expresiones génicas.
Resumen
La señalización de muesca regula el mantenimiento de las células madre/progenitoras neuronales mediante interacciones células celulares. Los componentes de la señalización Notch exhiben expresión dinámica. El efector de señalización de muesca Hes1 y el ligando de muesca Delta-like1 (Dll1) se expresan de manera oscilatoria en células neurales del tallo/progenitor. Debido a que el período de la expresión oscilatoria de estos genes es muy corto (2 h), es difícil monitorear su expresión cíclica. Para examinar estos cambios rápidos en la expresión génica o la dinámica de proteínas, se requieren reporteros de respuesta rápida. Debido a su rápida cinética de maduración y alta sensibilidad, el reportero de bioluminiscencia luciferasa es adecuado para monitorear los cambios rápidos en la expresión génica en las células vivas. Usamos un reportero luciferasa desestabilizado para monitorear la actividad promotora y un reportero fusionado con luciferasa para la visualización de la dinámica de proteínas a una sola célula. Estos reporteros de bioluminiscencia muestran una rápida rotación y generan señales muy débiles; por lo tanto, hemos desarrollado un sistema de imágenes de bioluminiscencia altamente sensible para detectar señales tan débiles. Estos métodos nos permiten monitorear varias dinámicas de expresión génica en células y tejidos vivos, que son información importante para ayudar a entender los estados celulares reales.
Introducción
El cerebro de los mamíferos se compone de un gran número de varios tipos de neuronas y células gliales. Todas las células se generan a partir de células neurales madre/progenitoras (NNP), que primero proliferan para expandir sus números, luego comienzan a diferenciarse en neuronas, y finalmente dan lugar a células gliales1,2,3,4,5. Una vez que las células se han diferenciado en neuronas, no pueden proliferar o aumentar su número, y, por lo tanto, el mantenimiento de los PNJ hasta etapas posteriores es importante. La señalización de muesca a través de interacciones célula-célula desempeña un papel importante en el mantenimiento de los PNJ6,7. Los ligandos de muesca interactúan con la proteína de membrana, Notch, en la superficie de las células vecinas y activa la proteína Notch. Después de la activación, se produce proteólisis de la proteína Notch, liberando así el dominio intracelular de Notch (NICD) de la membrana celular en el núcleo8,9,10. En el núcleo, NICD se une a las regiones promotoras de Hes1 y Hes5 (Hes1/5) y activa la expresión de estos genes. Hes1/5 reprime la expresión de los genes proneurales Ascl1 y Neurogenin1/2 (Neurog1/2)11,12,13,14. Debido a que los genes proneurales inducen la diferenciación neuronal, Hes1/5 desempeña un papel esencial en el mantenimiento de los PNJ. Además, como los genes proneurales pueden activar la expresión del ligando Delatal como 1 (Dll1), Hes1/5 también reprime la expresión de Dll1. Por lo tanto, la expresión de Dll1 conduce a que las celdas vecinas sean negativas para Dll1 a través de la señalización Notch. De esta manera, las células inhiben que las células adyacentes sigan su mismo destino, un fenómeno conocido como inhibición lateral8. En el cerebro en desarrollo, la inhibición lateral juega un papel en la generación de varios tipos de células diferentes.
La imagen en tiempo real a nivel de celda única revela expresiones dinámicas de los componentes de la señalización Notch en NPC15,16,17. La señalización de muesca activa la expresión de Hes1,pero la proteína Hes1 se une a su propio promotor y reprime su propia expresión. Además, Hes1 es una proteína extremadamente inestable, que se degrada por la vía ubiquitina-proteasoma; por lo tanto, la represión de su propio promotor es sólo de corta duración y luego la transcripción comienza de nuevo. De esta manera, la expresión de Hes1 oscila tanto a nivel de transcripción como traslacional en un ciclo de 2 h18. La expresión oscilatoria de Hes1, a su vez, induce la expresión oscilatoria de los genes diana aguas abajo, como Ascl1, Neurog2 y Dll1, a través de la represión periódica15,16,17,19. Mientras que los genes proneurales pueden inducir la diferenciación neuronal, su expresión oscilatoria no es suficiente para la diferenciación neuronal; más bien su expresión sostenida es esencial para la diferenciación neuronal. La expresión oscilatoria de genes proneurales es importante para mantener los PNJ en lugar de inducir la diferenciación neuronal14,15,16. La expresión de Dll1 oscila tanto a nivel de transcripción como traslacional durante diversas morfogénesis, como la neurogénesis y la somitogénesis. La expresión dinámica de Dll1 es importante para la morfogénesis normal y la expresión constante de Dll1 induce defectos en la neurogénesis y la somitogénesis17. Estos hallazgos demuestran la importante función que la dinámica de la expresión génica y la cinética proteica tienen en la regulación de diversos eventos de desarrollo (es decir, diferentes dinámicas de expresión producen diferentes resultados en los comportamientos celulares).
Para analizar la dinámica de la señalización de la muesca, el análisis estático de los tejidos y las células es insuficiente porque están cambiando constantemente. La toma de imágenes en tiempo real de células individuales es una poderosa herramienta para revelar la dinámica en la expresión génica. La expresión dinámica de las moléculas de señalización de Notch sufre respuestas cíclicas rápidas en el período de 2-3 h. Esta rápida expresión periódica presenta dos problemas difíciles para el monitoreo en tiempo real: (1) la expresión de las moléculas se suprime a niveles bajos, y (2) la rotación rápida requiere reporteros de respuesta rápida. Para superar estos problemas, previamente desarrollamos un método de imagen en tiempo real de bioluminiscencia20. Debido a que el reportero de bioluminiscencia tiene una mayor sensibilidad y un tiempo de maduración más corto que los reporteros fluorescentes, esta estrategia nos permite monitorear la dinámica rápida en las células vivas. Usando la visualización en tiempo real, encontramos que más genes mostraban una expresión dinámica de lo que habíamos pensado anteriormente. Además, ha aumentado el número de informes que muestran la expresión y la dinámica de proteínas en las células vivas y la importancia de estas dinámicas en diversos eventos biológicos, lo que sugiere un papel fundamental de la dinámica en las expresiones génicas21,22.
En este informe, se describe una manera de visualizar la expresión del ligando de la muesca Dll1 en los NNJ tanto en cultivos disociados como en cultivos de sectores corticales. Para monitorear la dinámica de la transcripción de Dll1 a niveles de una sola célula, generamos cultivos disociados de PNJ derivados del telencéfalo embrionario de ratones transgénicos que transportaban a un reportero pDll1-Ub-Fluc, un reportero de luciferasa desestabilizado impulsado por el promotor de Dll1. Para monitorear la dinámica de proteínas Dll1 in vivo, introdujimos el reportero de fusión Dll1-Fluc en los PNJ en la corteza y visualizamos la expresión del reportero en los PNJ en cultivos de rebanadas corticales. Las imágenes en tiempo real nos permitieron capturar las diversas características de la expresión génica y la dinámica de proteínas en células vivas a alta resolución temporal.
Protocolo
Todo el procedimiento, incluidos los sujetos de animales, ha sido aprobado por el Comité Institucional de Cuidado y Uso de Animales en el Instituto de Vida Fronteriza y Ciencias Médicas de la Universidad de Kioto.
1. Reporteros de bioluminiscencia
NOTA: El reportero de luciferasa es adecuado para medir la dinámica rápida de la actividad promotora mediante la fusión de la señal de degradación. Además, el reportero de fusión de luciferasa permite monitorear la dinámica proteica en una sola célula. Ambos tipos de reporteros están disponibles para el cultivo monocapa (cultivo de disociación) y el cultivo de tejidos (cultivo de rebanadas).
- Reportero de monitoreo de la actividad promotora de Dll117
NOTA: Para monitorear los rápidos cambios en la expresión génica, el reportero de respuesta rápida e inestable es esencial. La luciferasa de luciérnaga (Fluc) muestra una rápida maduración en comparación con los reporteros de fluoresceína. Debido a que la ubiquitina fusionó el reportero de luciferasa (Ub-Fluc) muestra una rápida degradación y un rápido volumen de negocios, es muy útil monitorear la expresión génica dinámica en las células vivas20.- Utilice el reportero deluciferase desestabilizado por el promotor Dll1, luciferasa ubiquitinada (pDll1-Ub-Fluc, Figura 1Un panel superior), para monitorear los cambios rápidos en la expresión de Dll1 a nivel de transcripción17.
- Generar una línea de ratón transgénica que lleve pDll1-Ub-Fluc para la expresión estable de este reportero.
- Reportero para el seguimiento de la dinámica de proteínas Dll117.
- Para la generación de ratones knock-in, inserte el ADNa de luciferasa en los 3 otermini de la región de codificación Dll1 para que se exprese la proteína de fusión Dll1-luciferasa (Reportero de Dll1-Fluc, Figura 1A,panel inferior)17.
- Supervise la dinámica de expresión de Dll1 a niveles de proteínas midiendo la actividad de la luciferasa.
- Para monitorear la dinámica de la proteína Dll1 a niveles de tejido, introduzca el reportero Dll1-Fluc en los PNJ en el telencéfalo embrionario por electroporación uterina.
2. Sistema de imágenes de bioluminiscencia
- Construya un sistema de imágenes en vivo de bioluminiscencia utilizando un microscopio invertido instalado con una cámara CCD refrigerada por agua de alta sensibilidad. Con el fin de reducir el ruido y obtener alta sensibilidad, enfríe la cámara con agua fría proporcionada por el circulador de agua a una temperatura optimizada de -90 oC.
- Para la toma de imágenes de células vivas/tejidos, instale el sistema de incubación, controlando la temperatura y el gas mezclado, en la etapa del microscopio.
- Para obtener imágenes de la expresión del reportero desestabilizado de luciferasa, utilice un ángulo numérico alto (NA: 1.30) lente objetivo de inmersión en aceite 40x. La distancia de trabajo de esta lente es de 0,2 mm, está disponible no sólo para el cultivo celular monocapa, sino también para el cultivo de rebanadas.
- Para la supervisión dual de la expresión del reportero de luciferasa y fluoresceína, instale el dispositivo de iluminación LED en la trayectoria de luz del microscopio.
- Controle las imágenes de lapso de tiempo con un software asociado con el microscopio (por ejemplo, el programa de adquisición multidimensional del software MetaMorph).
- Preparar todo el sistema en una habitación oscura, porque la señal del reportero luciferasa desestabilizado es muy débil, y para evitar la luz extraña evitar la adquisición.
3. Cultivos de disociación del tallo/progenitor neural (NPC)
- Preparación de medios de cultivo, platos y reactivos para la cultura de disociación del PNJ
- Preparar medios N2/B27 para el cultivo de células neurales del tallo/progenitor con una concentración final de 1x N2, 1x B27, 1 mM N-acetilcisteína, 10 ng/mL bFGF y 50 U/mL Penicilina/Streptomicina en medios DMEM/F12.
- Preparar la solución de papaína para la disociación que contiene 7 U/ml de papaína, 0.006% DNase y 1 mM N-acetilcisteína en medios EBSS.
- Prepare 100 mM de solución de luciferina sódica para la toma de imágenes de luminiscencia. Diluir 100 mM de solución de luciferina en medios N2/B27 a una concentración final de 1 mM.
NOTA: Luciferin muestra autofluoresceína sí mismo. En el caso de la luciferasa-fluorescencia de imágenes duales, especialmente EGFP, reducir la concentración de luciferina a 0,5 mM es mejor. - Recubrir un recipiente inferior de vidrio de 35 mm (diámetro de vidrio 27 mm) con 40 g/ml de solución de poli-L-lisina (PLL) durante 1 h a temperatura ambiente. Después de la incubación, lavar las placas 3x con PBS y dejar que se seque.
- Disección de embriones y disociación de PNJ
- Después de eutanasia restar un ratón transgénico pDll1-Ub-luc embarazada (día embrionario 12.5 (E12.5)) por asfixia deCO2 y luxación cervical, cortar a través del abdomen y sacar el útero.
NOTA: Los investigadores deben seguir las regulaciones y directrices del comité de investigación animal de su institución. Si se utilizan anestesia o analgésicos, úselos correctamente. - Transfiera el útero en una placa Petri de 10 cm que contenga 25 ml de PBS helado. Sacar los embriones del útero en PBS helado, usando microtijeras y fórceps finos.
- Cortar la cabeza de cada embrión usando tijeras y transferirla a platos Petri de 10 cm que contengan DMEM/F12 helado. Retire la epidermis y el cartílago que rodean el cerebro y transfiera el cerebro a una placa Petri de 35 mm que contenga hielo frío de 3 ml de medios N2/B27.
- Cortar el telencéfalo derecho e izquierdo del diencéfalo(Figura 1Ba-f, C,D). Retire las meninges que cubren la superficie del telencéfalo utilizando fórceps finos(Figura 1Bi). Usando fórceps finos de nuevo, saque la parte dorso-lateral de la corteza del telencéfalo(Figura 1Bg,h,j-l E).
- Transfiera los tejidos a nuevos tubos de 1,5 ml con una pipeta P1000 y retire cualquier medio adicional con una pipeta P200. Añadir 0,1 ml de solución de papaína por tejido cerebral (un par de corteza). Después de 15 min de incubación a 24oC, pipetee suavemente las muestras 10 veces con la pipeta P1000. Incubar las muestras de nuevo durante 15 min a 24 oC.
- Pipetear suavemente las muestras 10 veces con la pipeta P1000. Centrifugar las muestras durante 3 min a 400 x g y temperatura ambiente (RT). Descarta el sobrenadante.
- Añadir 1 ml de medios DMEM/F12 al pellet. Pipetear suavemente 10 veces con pipeta P1000. Centrifugar las muestras durante 3 min a 400 x g y RT. Deseche el sobrenadante. Repita esto al menos 2 veces más.
- Al pellet, añadir 0,5 ml de medios N2/B27 que contengan 1 mM de luciferina y mezclar bien. Sembrar las células (1 x 106 células) a un plato inferior de vidrio recubierto de PLL. Cultivo de las células en la incubadora de CO2 durante 1 h. Una vez que las células se adhieran al plato, agregue 2 mL de medios N2/B27, que contengan 1 mM de luciferina.
- Después de eutanasia restar un ratón transgénico pDll1-Ub-luc embarazada (día embrionario 12.5 (E12.5)) por asfixia deCO2 y luxación cervical, cortar a través del abdomen y sacar el útero.
- Visualización de la expresión del reportero luciferasa en la cultura de disociación del PNJ
- Inicie el sistema de imágenes en vivo de luminiscencia antes de realizar la disección. Para el cultivo de NPC, ajuste la temperatura de la incubadora de etapas a 37 oC y ajuste el ajuste de la mezcla de gas 20% O2, 5% CO2.
- Ponga el aceite de inmersión en la lente objetivo. Coloque la muestra en la etapa del microscopio. Vea el campo manualmente y elija la mejor posición y el foco de las celdas de interés. Haga clic en vivo para adquirir la imagen de prueba.
- Ejecute la adquisición de lapso de tiempo mediante adquisiciones de 2 dimensiones (luminiscencia y campo brillante) durante 24 horas con el programa de adquisición multidimensional. Seleccione El programa Adquisición multidimensional y establezca la configuración de adquisición de la siguiente manera (paso 3.3.6).
- Para la adquisición de imágenes luminiscencias, utilice los siguientes ajustes de cámara: velocidad de transferencia baja (50 kHz), 2 x 2/4 x 4 binning, tiempo de exposición de 10 min/5 min. Para la adquisición de imágenes de campo brillante, utilice los siguientes ajustes: velocidad de transferencia intermedia (1 MHz), 1 x 1 binning y tiempo de exposición de 100 ms.
4. En la electroporación utero
NOTA: Esto se realiza para la introducción del reportero Dll1-Fluc en las células progenitoras neuronales.
- Preparación de herramientas y reactivos para la electroporación
- Preparar micro capilares para la inyección de ADN en el ventrículo del telencéfalo embrionario. Estirar un capilar de hierba con calor y cortar y pulir la punta de la misma con la máquina de pulido.
- Preparar una mezcla de ADN con tinte (p. ej., azul Trypan). La concentración de cada ADN es de 1 g/l en PBS y añadir el tinte a una concentración final del 10%.
NOTA: Utilice la mezcla de ADN para la visualización de la dinámica de proteínas Dll1 de la siguiente manera: Dll1-Fluc (Reportero de proteínaDll1, Figura 2E superior)y pEF-EGFP (EF promotor impulsado por vector de expresión EGFP, Figura 2E inferior), monitoreando la ubicación y morfología de las células transinfectadas. - Preparar dos tipos de anestésicos: 3,88 mg/ml de pentobarbital y 0,12% de xilazina, en 1x PBS estéril.
- Utilice los instrumentos estériles y los guantes quirúrgicos durante el funcionamiento.
- Electroporación en el útero
- Anestetizar un ratón ICR embarazada en el tiempo (E12.5) por administración intraperitoneal de anestésicos con 500 ml de 3,88 mg/ml de pentobarbital y 500 ml de xilazina al 0,12%.
NOTA: Los investigadores deben seguir la regulación de los experimentos con animales. Si uno necesita usar anestesia o medicamentos para aliviar el dolor, úselos correctamente. - Corta el cabello y desinfecta la piel con exfoliación quirúrgica.
- Compruebe la falta de respuesta de pellizcar de dedo del dedo del dedo del dedo del tiempo. Una vez que no se observe el reflejo del pedal, haga un corte de 2-3 cm a través del abdomen a lo largo de la línea media. Coloque las gasas alrededor de la parte diseccionada y moje las gasas con PBS caliente para no secar la incisión. Saque suavemente el cuerno uterino derecho con fórceps en forma de anillo y cuente el número de embriones.
- Inyectar 1-2 l del ADN mezclado en el ventrículo del telencéfalo de embriones suavemente con micro capilar.
NOTA: Cuando la inyección es exitosa, se puede ver el color azul del tinte sobre la superficie del útero. - Humedezca el útero y el electrodo antes de proporcionar los pulsos de voltaje, y luego sostenga suavemente la cabeza de un embrión. Ponga el electrodo cargado positivamente en el lado del hemisferio, en el que se inyectó el ADN. Asegúrese de que el estado del pulso de electroporación es de 30-50 V para 50 ms, el intervalo de los pulsos es de 1 s (50 ms de carga y 950 ms sin carga). Proporcione 5 pulsos a un embrión y compruebe que las burbujas se generan a partir del electrodo cargado negativamente.
NOTA: Asegúrese de que la dirección del electrodo: el ADN se incorpora a las células en el lado del electrodo positivo. Durante este procedimiento, no mueva la posición del electrodo. - Repita los pasos 4.2.3-4.2.4 para otros embriones y devuelva el cuerno uterino a la cavidad abdominal. Realizar el mismo procedimiento a los embriones en el cuerno uterino izquierdo.
- Después de volver a colocar el lado izquierdo, sutura la incisión por una sutura de seda (4-0) con una aguja (17 mm). Antes de cerrar completamente la incisión, coloque PBS caliente en la cavidad abdominal
NOTA: El intervalo entre suturas es de alrededor de 2 mm. - Después de terminar la cirugía, coloque el ratón en una almohadilla de calentamiento para la recuperación después de la anestesia. Casa el ratón individualmente.
NOTA: Los investigadores deben seguir su regulación instiucional local de los experimentos con animales. Si uno necesita usar medicamentos para aliviar el dolor, úselos correctamente.
- Anestetizar un ratón ICR embarazada en el tiempo (E12.5) por administración intraperitoneal de anestésicos con 500 ml de 3,88 mg/ml de pentobarbital y 500 ml de xilazina al 0,12%.
5. Preparación de cultivos de rebanadas de la corteza en desarrollo y visualización de la expresión de reportero luciferasa en las rebanadas corticales
- Preparación de medios de cultivo y reactivos para cultivos de rebanadas de la corteza
- Preparar medios enriquecidos para el cultivo de rodajas corticales que contengan un 5% de suero bovino fetal y un 5% de suero de caballo en medios N2/B27.
- Burbuja 100% O2 gas a través de medios DMEM / F12 para la disección y la fabricación de rodajas corticales.
NOTA: Para utilizar el gas de oxígeno de forma segura, guarde el cilindro O2 en el gabinete del cilindro y supervise la concentración de oxígeno en el aire mediante un medidor de concentración de oxígeno. - Diluir 100 mM de solución de sodio luciferina en medios enriquecidos a una concentración final de 1 mM.
NOTA: Luciferin muestra autofluoresceína sí mismo. En el caso de la doble imagen luciferasa-fluorescencia, especialmente EGFP, menor concentración de luciferina (0,5 mM) es mejor. - Fije la incubadora multigás a 37 oC por 40% O2 y 5% CO2.
- Disección de embriones y examen de la expresión de reportero fluorescente
- Después de eutanasia del ratón embarazada al que los reporteros (E13.5) son introducidos por electroporación utero (paso 4.2), cortar a través del abdomen y sacar el útero.
NOTA: Los investigadores deben seguir la regulación de los experimentos con animales. Si usa anestesia o medicamentos para aliviar el dolor, úselos correctamente. - Transfiera el útero a una placa Petri de 10 cm que contenga PBS. Sacar los embriones del útero en PBS, usando microtijeras y fórceps finos. Cortar la cabeza de cada embrión y transferir a una placa Petri de 10 cm que contiene medios DMEM/F12 burbujeados con 100% O2 gas. Retire la epidermis y el cartílago que rodean el cerebro en los medios DMEM/F12.
- Ponga el cerebro en la tapa de la placa Petri con fórceps en forma de anillo y coloque la tapa en la etapa del microscopio estereoscópico de fluorescencia. Compruebe la región de la corteza que expresa la proteína fluorescente bajo la luz de excitación(Figura 2A,B).
- Transfiera el cerebro a una tabla de corte de goma de silicona llena de 30 ml de medios DMEM/F12 burbujeados con 100% O2 de gas. Retire las meninges que cubren la superficie del telencéfalo por fórceps finos.
- Cortar el borde entre la parte medial y lateral del telencéfalo dorsal y separarense en dos hemisferios utilizando un cuchillo microquirúrgico o fórceps finos. Usando un cuchillo microquirúrgico, corta la corteza como rayas y haz rodajas corticales(Figura 2C,D). Rebanadas de pipeta con medio usando pipeta y transfiéralas a medios enriquecidos en un plato de 35 mm.
- Coloque las rodajas en las inserciones de cultivo en los platos de fondo de vidrio con medios enriquecidos. Corrija la dirección de las rodajas con fórceps finos. Configure la superficie de corte de las rodajas en la superficie de la plaquita de referencia. Retire el medio adicional con una pipeta. Incubar las rodajas en la incubadora multi-gas establecida en 40% O2 y 5% CO2 a 37oC durante 30 min.
- Añadir 300 l de medios enriquecidos que contengan 1 mM de luciferina al exterior del inserto de cultivo en el plato inferior de vidrio.
- Después de eutanasia del ratón embarazada al que los reporteros (E13.5) son introducidos por electroporación utero (paso 4.2), cortar a través del abdomen y sacar el útero.
- Visualización de la expresión de reportero luciferasa en cultivos de sectores
- Inicie el sistema de imágenes en vivo de luminiscencia antes de iniciar la disección. Utilice la siguiente condición de cultivos de rodajas corticales: 40% O2, 5% CO2 y 37 oC.
- Ponga el aceite de inmersión en una lente objetivo 40x. Coloque el plato de muestra en la etapa del microscopio. Adquirir la imagen de prueba de fluorescente y establecer la posición y el plano de enfoque a la región de interés bajo la iluminación de la luz de excitación.
- Ejecutar la adquisición de lapso de tiempo por 3 adquisiciones dimensifiales (luminiscencia, fluorescencia y campo brillante) durante 24 h(Figura 2F-H). Para la adquisición de imágenes luminiscencias, utilice los siguientes ajustes de cámara: velocidad de transferencia baja (50 kHz), binning 2x2/4x4, tiempo de exposición de 10 min/5 min. Para la fluorescencia y la adquisición de imágenes de campo brillante, utilice los siguientes ajustes: velocidad de transferencia intermedia (1 MHz), binning 1x1 y tiempo de exposición de 100 ms.
6. Procesamiento y análisis de imágenes
- Conecte cada imagen y haga las imágenes de pila. Haga clic en Importar secuencia de imágenes en el menú Archivo (Archivo ? Importación de la lista de importaciones Secuencia de imágenes) y elija la carpeta donde se guardan las imágenes adquiridas. Ponga algunas palabras contenidas en el nombre de archivo en la columna de File name contains.
- Para eliminar el ruido de los rayos cósmicos en las imágenes de bioluminiscencia, aplique el plug-in SpikeNoise Filter de ImageJ/Fiji. Abra las imágenes de pila y haga clic en Filtro SpikeNoise.
- Aplique el plug-in Savitzky Golay Temporal Filter para obtener una dinámica clara de la expresión del reportero. Abra las imágenes de pila y haga clic en Filtro temporal Golay savitzky.
- Para medir la intensidad de la expresión del reportero, aplique el complemento Z Axis Profile Plus a cada celda. Seleccione las celdas y establezca los ROI, la región de interés y haga clic en Z Axis Profile Plus.
Resultados
Las expresiones de los genes Hes1/7 exhiben ciclo de oscilación de 2 h en varias líneas celulares y durante la somitogénesis. Además, el período de oscilación es muy corto y tanto sus ARNm como sus proteínas son extremadamente inestables con la vida media de alrededor de 20 min. Si se utiliza un reportero de respuesta lenta, no podemos rastrear una dinámica tan rápida, y si se utiliza un reportero estable, se acumula gradualmente mientras la expresión génica oscila. Por lo tanto, el reportero debe ser rápidamente degradado para monitorear la rápida rotación de estos genes expresados cíclicamente. Para superar estos problemas, utilizamos el reportero luciferasa para monitorear la expresión dinámica de los osciladores. Debido a que el reportero de bioluminiscencia tiene un corto tiempo de maduración y alta sensibilidad, nos permite monitorear la dinámica rápida de los osciladores ultradianos. Al igual que un reportero fluorescente, el reportero de luciferasa puede monitorear la dinámica de expresión de una proteína fusionándose con la secuencia de codificación genética(Figura 1A, Figura 2E y Figura 3D). Los productos genéticos fusionados con Luciferasa exhiben la misma expresión, rotación y cinética de translocación en las células que las proteínas endógenas. Además, para monitorear la actividad promotora del gen oscilante Dll1,utilizamos luciferasa ubiquitinada, un reportero luciferasa desestabilizado(Figura 1A y Figura 3A)23, cuya vida media es de aproximadamente 10 min20. Usando varios tipos de reporteros de luciferasa, generamos ratones transgénicos o ratones golpeados para obtener una expresión estable del reportero en NpCs durante la neurogénesis17. Para visualizar la expresión del reportero a niveles de una sola célula en el cultivo de tejidos, es preferible la introducción dispersa del reportero. Por lo tanto, utilizamos la transfección transitoria del gen reportero en NNP a través de electroporación utero(Figura 2F-H). El sistema de reportero de luciferasa se ha utilizado en el campo de los ritmos circadianos para monitorear las expresiones dinámicas de los genes del reloj durante un largo período (por ejemplo, 1 semana), lo que sugiere que la luciferina (D-luciferin), el sustrato de la enzima luciferasa luciérnaga, es muy estable y no tiene toxicidad para las células vivas24,25. Usualmente usamos luciferina en concentraciones de 1 mM en los medios de comunicación, lo que es suficiente para la toma de imágenes de células vivas durante la noche. Además, el sistema de imágenes basado en microscopios nos permite adquirir imágenes multidimensionales, imágenes de campo brillante, imágenes de fluorescencia e imágenes de quimioluminiscencia(Figura 2F-H y Figura 3E).
Utilizando estas condiciones, visualizamos las expresiones de varios genes de reloj ultradiano, incluyendo Hes1, Ascl1, Neurog2 y Dll1 (Figuras 2 y Figura 3). Los resultados representativos se muestran en la Figura 3. El reportero de la actividad promotora de Dll1 exhibió una expresión oscilatoria en PNJ derivada del telencéfalo de los ratones reportero de Dll1-Ub-Fluc. El reportero desestabilizado de luciferasa(Figura 3A) indicó una regulación aguda hacia arriba y hacia abajo de la expresión de la actividad promotora(Figura 3B,C). En este caso, las células progenitoras neuronales individuales mostraron un ciclo de oscilación de aproximadamente 2,5 h con varias amplitudes en el transcurso de 13 h(Figura 3C). El reportero de luciferasa de respuesta rápida nos permite capturar la dinámica de transmisión de la señalización de muesca entre dos células vivas(Figura 3D-F y Película Suplementaria S1). Preparamos dos tipos de mezclas de ADN: (1) Reportero promotor de Hes1 (pHes1-Ub-luc) y vector de expresión EGFP, y (2) Reportero de proteínaDll1 (Dll1-Luc) y vector de expresión mCherry y los transfectó a NpCs por separado. Luego recogimos los dos tipos de células y co-cultivamos para medir la expresión de los reporteros en células vivas. Los resultados representativos se muestran en la Figura 3E,F. Células positivas adyacentes de EGFP que transportan al reportero de Hes1 y a las células positivas de mCherry expresando a un reportero de proteínadlls Dll1 contactados entre sí durante la observación. La expresión del reportero Hes1 en una celda verde parecía comenzar unos 60 minutos después del contacto de dos celdas(Figura 3E,F). Esto sugirió que el retardo de tiempo para la transmisión de la señalización de la muesca entre las células adyacentes era de aproximadamente 1 h. Además, durante la transmisión de la señal, la expresión de proteína Dll1 mostró translocación dinámica en una célula roja(Figura 3E).

Figura 1: Disección del cerebro embrionario del ratón. (A) La estructura de los reporteros de luciferase, el reportero promotor de Dll1 y el reportero de proteínas Dll1. (B) Procedimiento de disección. (a) La vista lateral de un embrión de ratón. (b) Cortar la cabeza junto con la línea punteada. (c) Retire la epidermis y el cartílago de la brecha entre el telencéfalo y el cerebro medio. (d) Cortar el tejido circundante a lo largo de la línea media entre los hemisferios derecho e izquierdo. (e) Retire el tejido de la rotura central a ambos lados. (f) El telencéfalo y el cerebro medio después de la extracción del tejido circundante. (g) Separar el telencéfalo (tel), el cerebro medio (medio) y la bombilla olfativa (OB). (h) Sección transversal del telencéfalo, que se muestra en línea punteada en (g). (i) Retire las meninges que rodean la superficie del telencéfalo. (j) Separar el telencéfalo en dos partes: parte medial y lateral. (k) Cortar el borde de la corteza y la eminencia gangliónica (GE). (l) Parte dorso-lateral de la corteza se utiliza para el cultivo de disociación. (C) El cerebro en el día embrionario 14 (E14), que consiste en los hemisferios izquierdo(a ) y derecho (b) del telencéfalo y el cerebro medio (c). (D) Los hemisferios telencéfalos separados del cerebro medio. (E) Un hemisferio diseccionado del telencéfalo: la parte ventrolateral del telencéfalo, incluidas las eminencias gangliónicas (d), la parte dorsolateral del telencéfalo utilizada para cultivos disociados y cultivos de rodajas (e), la parte medial del telencéfalo (f) y las meninges que cubren la superficie del cerebro (g). Haga clic aquí para ver una versión más grande de esta figura.
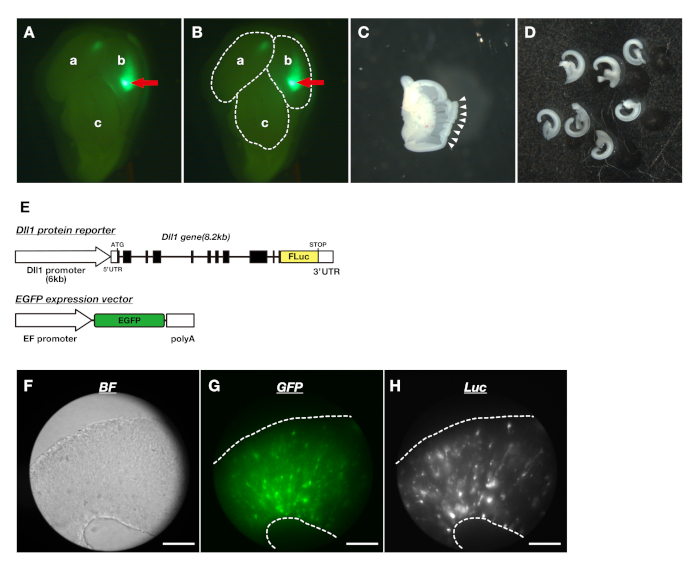
Figura 2: Hacer las rodajas corticales del telencéfalo embrionario y visualizar la dinámica de proteínas Dll1 en los cultivos de rodajas corticales. (A) Comprobación de la expresión de EGFP mediante microscopio estereoscópico de fluorescencia. La flecha roja muestra la región de la corteza que expresa EGFP. El hemisferio izquierdo (a), el hemisferio derecho (b) y el cerebro medio (c). (B) Las líneas de puntos indican los contornos de las regiones cerebrales que se muestran en (A). (C) Corteza diseccionada del telencéfalo dorsolateral. Las puntas de flecha blancas indican los bordes cortados. (D) Rebanadas corticales del telencéfalo dorso-lateral. (E) Estructuras genéticas de los reporteros para visualizar la dinámica de proteínas Dll1 en NpCs. El reportero de proteínas Dll1, Dll1-Fluc (superior) se introdujo en los PNJ con un vector de expresión EGFP (inferior) para monitorear la morfología y migración de las células en las rebanadas corticales. (F-H) Imágenes tridimensionales de campo brillante (F), expresión GFP (G) y bioluminiscencia (H) en la rebanada cortical. El sistema de imágenes de bioluminiscencia permitió rastrear simultáneamente las expresiones de luciferasa y reportero de fluorescencia. Barras de escala: 200 m. Por favor, haga clic aquí para ver una versión más grande de esta figura.
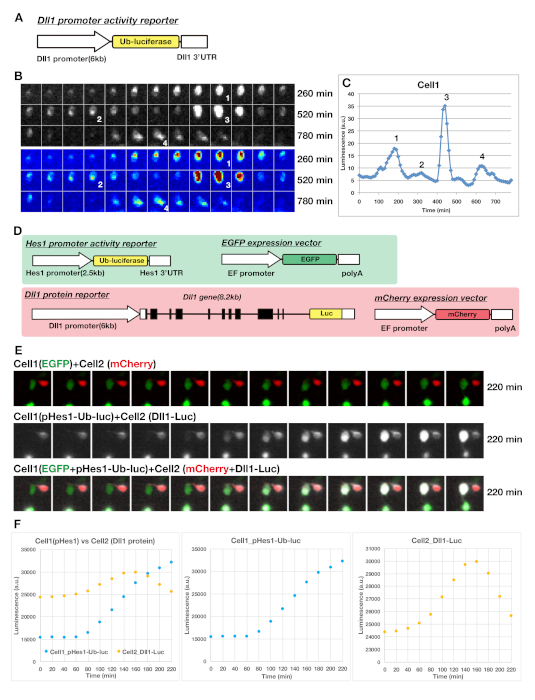
Figura 3: Datos representativos para la visualización y el análisis de la expresión dinámica del gen Dll1 en la cultura NPC. (A) El reportero construye para visualizar la expresión Dll1 a nivel transcripcional, utilizando un reportero luciferasa desestabilizado (Ub-luciferasa, luciferasa ubiquitizada). (B) Visualización de la expresión de Dll1 en un solo NPC con un reportero de bioluminiscencia. Los números del panel muestran los puntos pico de la expresión oscilatoria de Dll1 correspondientes a los números del panel C. (C) Una gráfica de curso temporal de la bioluminiscencia del NPC disociado mostrado en (B), que muestra la expresión dinámica de Dll1. (D-F) Visualización de la expresión del reportero promotor de Hes1 y reportero de proteínadlls Dll1 expresada en las células vecinas. (D) La estructura del reportero de actividad promotora de Hes1 (pHes1-Ub-luc) y del reportero de proteínas Dll1 (Dll1-Luc). (E y F) La célula positiva de EGFP que transportaba al reportero Hes1 (Cell1) y a la célula positiva mCherry que transportaba al reportero de proteínas Dll1 (Cell2) fueron co-cultivadas y se midió la luminiscencia de ambos tipos de reporteros. La expresión del reportero de Hes1 en la celda verde (celda1) parecía comenzar unos 60 minutos después de que las dos células entraran en contacto. Haga clic aquí para ver una versión más grande de esta figura.
Película suplementaria S1: Visualización de la expresión del reportero promotor de Hes1 y reportero de proteína Dll1 expresada en las células vecinas, relacionada con la Figura 3D-3F. Por favor, haga clic aquí para ver este video. (Haga clic con el botón derecho para descargar.)
Discusión
Los componentes de la señalización Notch muestran expresiones oscilatorias en sincronía durante la somitogénesis pero fuera de sincronía durante la neurogénesis, lo que lleva a las dificultades para capturar la dinámica de expresión mediante análisis estático en este último caso. Por lo tanto, se requiere monitoreo en tiempo real para revelar la dinámica de expresión de los componentes de señalización Notch, como Hes1 y Dll1. Debido a que los períodos de las expresiones de las oscilaciones Hes1 y Dll1 son extremadamente cortos, aproximadamente 2-3 h, se requiere una respuesta rápida y reporteros inestables para monitorear su dinámica de expresión. Para ello, hemos desarrollado el reportero de bioluminiscencia y el sistema de imágenes. El reportero de bioluminiscencia luciferasa muestra una rápida cinética de maduración y alta sensibilidad para rastrear la rápida rotación de tales expresiones genéticas cíclicas. La rápida rotación de los periodistas conduce a señales muy débiles generadas. Para detectar señales tan débiles producidas por los reporteros de bioluminiscencia, utilizamos un sistema de imágenes de bioluminiscencia optimizado, que incluye una cámara CCD refrigerada por agua de alta sensibilidad con una velocidad de lectura ultrabaja (50 kHz), que reduce el ruido al mínimo. Además, una lente objetivo de alta apertura numérica (N.A.) nos permite recoger al máximo la luz liberada del reportero luciferasa desestabilizado. Para obtener una sensibilidad más alta solemos utilizar binning más alto (por ejemplo, 2 x 2, 4 x 4, 8 x 8) y mantener el obturador abierto durante mucho tiempo (por ejemplo, 5-20 min). Debido a que la señal de los reporteros de luciferase es extremadamente débil, la interferencia de la luz del medio ambiente también presenta un problema en la detección de la señal débil: por lo tanto, la sala del microscopio debe estar completamente oscura. Este sistema nos permite medir las expresiones dinámicas de los genes Hes1 y Dll1 en los PNJ tanto en los niveles transcripcionales como proteicos(Figura 3). Además, podemos visualizar la dinámica proteica de la translocación intracelular con reporteros de proteínas fusionadas de luciferasa. Además, utilizando el reportero de luciferasa de respuesta rápida, podemos capturar la dinámica de transmisión de la señalización de muesca y medir el retardo de tiempo para la transmisión de la señal entre dos células vivas(Figura 3D-F y Película Suplementaria S1). Además, la combinación del reportero promotor (reportero de Ub-luciferase) y reportero de proteínas (fusión de luciferasa) de un solo gen nos permite medir el tiempo de retardo entre la transcripción y la traducción del gen. De esta manera, las múltiples imágenes de varios tipos de reporteros de luminiscencia y reporteros de fluorescencia están disponibles para medir el retardo de tiempo/ tasa para reacciones bioquímicas.
El monitoreo en tiempo real de las expresiones génicas a resolución de una sola célula ha revelado que hay variaciones en la dinámica de expresión del mismo gen. Algunas células expresan Dll1/Neurog2 de manera oscilatoria, pero otras muestran patrones sostenidos. Además, la dinámica de expresión diferente (oscilación versus estable) induce diferentes salidas en el estado de las células. Lo que sí parece estar claro es que diferentes dinámicas de expresión influyen en los comportamientos celulares de diferentes maneras, sugiriendo que la dinámica de expresión codifica más información21,22,26,27,28,29,30,31,32. Los análisis estáticos no pueden capturar la dinámica en la expresión génica, y los análisis en tiempo real son necesarios para entender los fenómenos biológicos para revelar la dinámica de expresión en eventos celulares. El enfoque que introducimos aquí puede monitorear la dinámica de los osciladores ultradianos durante el desarrollo neuronal a alta resolución temporal. Usando este método, encontramos que muchos más genes se expresan dinámicamente de lo que habíamos pensado anteriormente, y podemos rastrear no sólo la dinámica de la expresión de proteínas, sino también la dinámica en la localización de proteínas en la célula a alta resolución temporal. Estos patrones de expresión dinámica podrían tener significados biológicos que aún no se han aclarado.
Los reporteros de bioluminiscencia tienen una gran ventaja en resolución temporal y baja toxicidad para las células vivas en comparación con los reporteros de fluorescencia33. Sin embargo, a diferencia de las variaciones de color en los reporteros fluorescentes, sólo hay unos pocos colores en los reporteros de bioluminiscencia, imponiendo una limitación en el número de genes que se pueden monitorear simultáneamente. Sin embargo, cada vez más personas están siendo aisladas y clonadas de varias criaturas34,35,y utilizando tal variedad de luciferasas, podremos rastrear simultáneamente múltiples dinámicas de expresión génica en una sola célula a alta resolución temporal. El tamaño molecular de la luciferasa de la luciérnaga es mayor que los reporteros de fluorescencia, lo que presenta algunas dificultades en la construcción de proteínas fusionadas con reporteros para monitorear la dinámica de las proteínas, pero recientemente, una nueva, más pequeña y más brillante luciferasa ha sido clonada36,lo que nos permitiría visualizar la dinámica de proteínas más fácil que nunca. Un número creciente de informes han mostrado recientemente diferentes tipos de dinámicas en expresión génica y translocación de proteínas en diversos eventos biológicos21,22,26,27,28,29,30,31,32. Los análisis de tales dinámicas en la regulación espaciotemporal utilizando un sistema de monitoreo en tiempo real serían cada vez más importantes para capturar los estados reales de las células y revelar la regulación de los sistemas celulares.
Divulgaciones
Los autores no tienen ningún interés financiero conflictivo.
Agradecimientos
Agradecemos a Yumiko Iwamoto por apoyar la producción del vídeo. También estamos agradecidos a Akihiro Isomura por la discusión y los apoyos del análisis de imágenes, Hitoshi Miyachi por apoyos técnicos para la generación de animales transgénicos, Yuji Shinjo (Olympus Medical Science), Masatoshi Egawa (Olympus Medical Science), Takuya Ishizu ( Olympus Medical Science) y Ouin Kunitaki (Andor Japan) para el apoyo técnico y las discusiones del sistema de imágenes de bioluminiscencia. Este trabajo fue apoyado por Core Research for Evolutional Science and Technology (JPMJCR12W2) (R.K.), Grant-in-Aid for Scientific Research on Innovative Areas (MEXT 24116705 for H.S. y MEXT 16H06480 for R.K.), Grant-in-Aid for Scientific Research (C) (JSPS) (JSPS) (JSPS) (JSPS) (JSPS) (JSPS) (JSPS) (JSPS) (JSPS) (JSPS) (JSPS) (JSPS) (JSPS) (JSPS) (JSPS) (JSPS) (JSPS) (JSPS) (JSPS) (JSPS) (JSPS) (JSPS) (JSPS) (JSPS) (JSPS) (JSPS) (JSPS) (JSPS) (JSPS) (JSPS) (JSPS) (JSPS) (JSPS) (JSPS) (JSPS) (JSPS) (JSPS) (JSPS) (JSPS) (JSPS) (JSPS) (JSPS) (JSPS) (JSPS) (JSPS 18K06254) (H.S.), Fundación Takeda (R.K. y H.S.), y Plataforma para Enfoques Dinámicos al Sistema Vivo del Ministerio de Educación, Cultura, Deportes, Ciencia y Tecnología, Japón.
Materiales
| Name | Company | Catalog Number | Comments |
| Bioluminescence Imaging System | |||
| Chilled water circulator (chiller) | Julabo | Model: F12-ED | |
| Cooled CCD camera | Andor Technology | Model: iKon-M 934 | |
| Incubator system | TOKAI HIT | Model: INU-ONICS | |
| Inverted microscope | Olympus | Model: IX81 | |
| Inverted microscope | Olympus | Model: IX83 | |
| LED illumination device | CoolLED | Model: pE1 | |
| MetaMorph | MOLECULAR DEVICES | Model: 40000 | |
| Mix gas controller | Tokken | Model: TK-MIGM OLO2 | |
| Objective lens | Olympus | Model: UPLFLN 40X O | |
| Preparations for Dissection | |||
| Dissection microscope | Nikon | Model: SMZ-2B | |
| Fluorescence stereoscopic microscope | Leica | Model: MZ16FA | |
| Fine forceps | DUMONT | INOX No.5 | |
| Scissors, Micro scissors | |||
| Forceps | |||
| Ring-shaped forceps | |||
| 10-cm plastic petri dish | greiner | 664160-013 | |
| 35-mm plastic petri dish | greiner | 627160 | |
| PBS | Nacalai Tesque | 14249-24 | |
| DMEM/F12 | invitrogen | 11039-021 | |
| Reagents for NPC dissociation culture | |||
| B27 supplement | invitrogen | 12587-010 | |
| bFGF | invitrogen | 13256-029 | Stock solution: 1 μg/ml in 0.1% BSA/PBS |
| D-luciferin | Nacalai Tesque | 01493-85 | Stock solution: 100mM in 0.9% saline |
| DNase | Worthington Biochemical Corporation | LK003172 | Stock solution: 1000U/ml in EBSS |
| EBSS | Worthington Biochemical Corporation | LK003188 | |
| Glass bottom dish | IWAKI | 3910-035 | |
| N2 supplement (100x) | invitrogen | 17502-048 | |
| N-acetyl-cystein | Sigma | A-9165-25G | |
| Papain | Worthington Biochemical Corporation | LK003178 | Stock solution: 7U/ml in EBSS |
| Penicillin/Streptmycine | Nacalai Tesque | 09367-34 | |
| Poly-L-lysine | Sigma | P-6281 | 40 mg/ml in DW |
| Preparations for in utero electroporation | |||
| 50-ml syringe | TERUMO | 181228T | |
| Electrode | Neppagene | 7-mm | |
| Electroporator | Neppagene | CUY21 EDIT | |
| Forceps | |||
| Gauzes | Kawamoto co. | 7161 | |
| Micro capillary | Made in-house | ||
| PBS | Nacalai Tesque | 14249-24 | |
| Pentbarbital | Kyoritsuseiyaku | Somnopentyl | |
| Ring-shaped forceps | |||
| Scissors, Micro scissors | |||
| Suture needle | Akiyama MEDICAL MFG. CO | F17-40B2 | |
| Xylazine | Bayer | Seractal | |
| Preparations for Slice culture | |||
| 10-cm plastic petri dish | greiner | 664160-013 | |
| 35-mm plastic petri dish | greiner | 627160 | |
| Culture insert | Millipore | PICM01250 | |
| DMEM/F12 | invitrogen | 11039-021 | |
| Fetal Bovine Serum | Sigma | 172012-500ML | |
| Fine forceps | DUMONT | INOX No.5 | |
| Forceps | |||
| Horse Serum | Gibco | 16050-122 | |
| Micro surgical knife | Alcon | 19 Gauge V-Lance | |
| Multi-gas incubator | Panasonic | MCO-5MUV-PJ | |
| N2/B27 media | Made in-house | ref. NPC dissociatioin culture | |
| PBS | Nacalai Tesque | 14249-24 | |
| Ring-shaped forceps | |||
| Scissors, Micro scissors | |||
| Silicon rubber cutting board | Made in-house |
Referencias
- Ross, S. E., Greenberg, M. E., Stiles, C. D. Basic helix-loop-helix factors in cortical development. Neuron. 39, 13-25 (2003).
- Pontious, A., Kowalczyk, T., Englund, C., Hevner, R. F. Role of intermediate progenitor cells in cerebral cortex development. Developmental Neuroscience. 30, 24-32 (2007).
- Kriegstein, A., Alvarez-Buylla, A. The glial nature of embryonic and adult neural stem cells. Annual Review of Neuroscience. 32, 149-184 (2009).
- Paridaen, J. T., Huttner, W. B. Neurogenesis during development of the vertebrate central nervous system. EMBO Reports. 15, 351-364 (2014).
- Taverna, E., Götz, M., Huttner, W. B. The cell biology of neurogenesis: toward an understanding of the development and evolution of the neocortex. Annual Review of Cell and Developmental Biology. 30, 465-502 (2014).
- Louvi, A., Artavanis-Tsakonas, S. Notch signaling in vertebrate neural development. Nature Reviews Neuroscience. 7, 93-102 (2006).
- Pierfelice, T., Alberi, L., Gaiano, N. Notch in the Vertebrate Nervous System: An Old Dog with New Tricks. Neuron. 69, 840-855 (2011).
- Bray, S. Notch signalling: a simple pathway becomes complex. Nature Reviews Molecular Cell Biology. 7, 678-689 (2006).
- Bray, S. Notch signalling in context. Nature Reviews Molecular Cell Biology. 17, 722-735 (2016).
- Kopan, R., Ilagan, M. X. G. The Canonical Notch Signaling Pathway: Unfolding the Activation Mechanism. Cell. 137, 216-233 (2009).
- Ishibashi, M., et al. Persistent expression of helix-loop-helix factor HES-1 prevents mammalian neural differentiation in the central nervous system. The EMBO Journal. 13, 1799-1805 (1994).
- Ohtsuka, T., et al. Hes1 and Hes5 as notch effectors in mammalian neuronal differentiation. The EMBO Journal. 18, 2196-2207 (1999).
- Ohtsuka, T., Sakamoto, M., Guillemot, F., Kageyama, R. Roles of the Basic Helix-Loop-Helix Genes Hes1 and Hes5 in Expansion of Neural Stem Cells of the Developing Brain. Journal of Biological Chemistry. 276, 30467-30474 (2001).
- Kageyama, R., Ohtsuka, T., Kobayashi, T. The Hes gene family: repressors and oscillators that orchestrate embryogenesis. Development. 134, 1243-1251 (2007).
- Shimojo, H., Ohtsuka, T., Kageyama, R. Oscillations in Notch Signaling Regulate Maintenance of Neural Progenitors. Neuron. 58, 52-64 (2008).
- Imayoshi, I., et al. Oscillatory control of factors determining multipotency and fate in mouse neural progenitors. Science. 342, 1203-1208 (2013).
- Shimojo, H., et al. Oscillatory control of Delta-like1 in cell interactions regulates dynamic gene expression and tissue morphogenesis. Genes and Development. 30, 102-116 (2016).
- Hirata, H., et al. Oscillatory expression of the bHLH factor Hes1 regulated by a negative feedback loop. Science. 298, 840-843 (2002).
- Kageyama, R., Ohtsuka, T., Shimojo, H., Imayoshi, I. Dynamic Notch signaling in neural progenitor cells and a revised view of lateral inhibition. Nature Neuroscience. 11, 1247-1251 (2008).
- Masamizu, Y., et al. Real-time imaging of the somite segmentation clock: revelation of unstable oscillators in the individual presomitic mesoderm cells. Proceedings of the National Academy of Sciences of the United States of America. 103, 1313-1318 (2006).
- Levine, J. H., Lin, Y., Elowitz, M. B. Functional roles of pulsing in genetic circuits. Science. 342, 1193-1200 (2013).
- Purvis, J. E., Lahav, G. Encoding and decoding cellular information through signaling dynamics. Cell. 152, 945-956 (2013).
- Luker, G. D., Pica, C. M., Song, J., Luker, K. E., Piwnica-Worms, D. Imaging 26S proteasome activity and inhibition in living mice. Nature Medicine. 9, 969-973 (2003).
- Yamaguchi, S., et al. Synchronization of Cellular Clocks in the Suprachiasmatic Nucleus. Science. 302, 1408-1412 (2003).
- Kiyohara, Y. B., et al. The BMAL1 C terminus regulates the circadian transcription feedback loop. Proceedings of the National Academy of Sciences of the United States of America. 103, 10074-10079 (2006).
- Behar, M., Hoffmann, A. Understanding the temporal codes of intra-cellular signals. Current Opinion in Genetics and Development. 20, 684-693 (2010).
- Elowitz, M. B., Levine, A. J., Siggia, E. D., Swain, P. S. Stochastic Gene Expression in a Single Cell. Science. 297, 1183-1186 (2002).
- Nelson, D. E., et al. Oscillations in NF-kappaB signaling control the dynamics of gene expression. Science. 306, 704-708 (2004).
- Purvis, J. E., et al. p53 dynamics control cell fate. Science. 336, 1440-1444 (2012).
- Hansen, A. S., O'Shea, E. K. Promoter decoding of transcription factor dynamics involves a trade-off between noise and control of gene expression. Molecular Systems Biology. 9, 704 (2014).
- Hansen, A. S., O'Shea, E. K. cis Determinants of Promoter Threshold and Activation Timescale. Cell Reports. 12, 1226-1233 (2015).
- Johnson, H. E., Toettcher, J. E. Signaling Dynamics Control Cell Fate in the Early Drosophila Embryo. Developmental Cell. 48, 361-370 (2019).
- Badr, C. E., Tannous, B. A. Bioluminescence imaging: Progress and applications. Trends in Biotechnology. 29, 624-633 (2011).
- Nakajima, Y., Ohmiya, Y. Bioluminescence assays: multicolor luciferase assay, secreted luciferase assay and imaging luciferase assay. Expert Opinion on Drug Discovery. 5, 835-849 (2010).
- Nakajima, Y., et al. Enhanced beetle luciferase for high-resolution bioluminescence imaging. PLoS One. 5, (2010).
- Stacer, A. C., et al. NanoLuc reporter for dual luciferase imaging in living animals. Molecular Imaging. 12, (2013).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados