Konjugation: Eine Methode zur Übertragung der Ampicillinresistenz vom Spender auf den Empfänger E. coli
Überblick
Quelle: Alexander S. Gold1, Tonya M. Colpitts1
1 Department of Microbiology, Boston University School of Medicine, National Emerging Infections Diseases Laboratories, Boston, MA
Erstmals 1946 von Lederberg und Tatum entdeckt, ist die Konjugation eine Form des horizontalen Gentransfers zwischen Bakterien, die auf direkten physischen Kontakt zwischen zwei Bakterienzellen beruht (1). Im Gegensatz zu anderen Formen des Gentransfers, wie Transformation oder Transduktion, ist konjugation ein natürlich vorkommender Prozess, bei dem DNA unidirektional von einer Spenderzelle in eine Empfängerzelle abgesondert wird. Diese Richtung und die Fähigkeit für diesen Prozess, die genetische Vielfalt von Bakterien zu erhöhen, hat der Konjugation den Ruf als eine Form der bakteriellen "Paarung" verliehen, von der angenommen wird, dass sie wesentlich zum jüngsten Anstieg der antibiotikaresistenten Bakterien (2, 3). Durch den Einsatz selektiver Drücke, z. B. durch den Einsatz von Antibiotika, wurde die Konjugation für den Einsatz im Labor manipuliert, was sie zu einem leistungsfähigen Werkzeug für den horizontalen Gentransfer zwischen Bakterien und in einigen Fällen von Bakterien auf Hefe, Pflanze und Tier macht. Zellen (4). Neben Anwendungen im Labor ist der Bakterielle-Eukaryote-Gentransfer durch Konjugation ein spannender Weg des DNA-Transfers mit einer Vielzahl möglicher biotechnischer Anwendungen und natürlich vorkommender Implikationen (5).
Die Konjugation wird vermutlich durch einen "zweistufigen Mechanismus" (6) funktioniert. Erstens muss die Spenderzelle, bevor dna übertragen werden kann, direkten Zell-zu-Zell-Kontakt mit dem Empfänger herstellen. Dieser Prozess wurde am besten in gram-negativen Bakterien charakterisiert, von denen die am meisten untersuchten Escherichia coliist. Der Zell-zu-Zell-Kontakt wird durch das Vorhandensein eines komplexen Netzes extrazellulärer Filamente auf dem Spender hergestellt, der als Geschlechtspilus bekannt ist, ein konjugatives Element, das durch das übertragbare Gen kodiert wird, das als F-Faktor (Fruchtbarkeit) (7, 8) bezeichnet wird. Neben der Herstellung eines Kontakts zwischen Spender und Empfänger werden mehrere Proteine über den Geschlechtspilus zum Empfängerzytoplasma transportiert, wodurch ein Typ-IV-Sekretionssystem (T4SS) zwischen den beiden Zellen gebildet wird, eine notwendige Struktur für den zweiten Schritt der Konjugation, DNA-Transfer (6). Durch die Kombination dieser Funktion des Geschlechtspilus mit der Rollkreisreplikation der DNA ist die Spenderzelle in der Lage, DNA in Form eines transponierbaren Elements, wie eines Plasmids oder Transposons, durch ein "Shoot and Pump"-Modell (6) an den Empfänger zu übertragen. In diesem Fall ist das "Schießen" der Transport des Pilotproteins mit verknüpfter DNA durch das T4SS in die Empfängerzelle, und das "Pumpen" ist der aktive Transport von DNA zum Empfänger, ein Prozess, der vom T4SS abhängig ist und durch Kopplungsproteine katalysiert wird (6). Die in diesem Verfahren verwendete Maschinerie besteht aus einem Ursprung der Übertragungssequenz (oriT), die von der DNA in cis und trans-Genen bereitgestellt werden muss, die eine Relaxase, paarpaarbildung Komplex und Typ IV Kopplungsprotein kodieren, und kann in cis oder trans (9) vorhanden sein. Diese Relaxase spaltet die nic-Site innerhalb der OriT-Sequenz und heftet sich kovalent am 5' Ende des übertragenen Strangs an, um das Relaxosome zu produzieren, einen einsträngigen DNA-Relaxase-Komplex mit anderen Hilfsproteinen (9). Nach der Bildung verbindet sich das Relaxosom über das Kopplungsprotein Typ IV mit dem Paarungskomplex typ IV, das die Übertragung des ssDNA-Relaxase-Komplexes in Empfängerzellen durch den T4SS (10) ermöglicht. Einmal im Zytoplasma des Empfängers, kann sich die DNA in das Empfängergenom integrieren oder separat in Form eines Plasmids existieren, von denen beide die Expression ihrer Gene ermöglichen.
In diesem Experiment wurde der weit verbreitete Konjugationsspenderstamm E. coli WM3064 verwendet, um das Gen zur Ampicillinresistenz auf den Empfängerstamm E. coli J53 zu übertragen. Während beide Stämme der gramnegativen Bakterien gegen Tetracyclin resistent waren, hatte nur der Spenderstamm WM3064 das Gen für Ampicillinresistenz, für den im pWD2-oriT-Shuttlevektor kodiert, und war auxotroph bis Diaminopimelsäure (DAP) (11-13). Dieses Experiment bestand aus zwei Hauptschritten, der Vorbereitung von Spender- und Empfängerstämmen, gefolgt von der Übertragung des Ampicillinresistenzgens vom Spender zum Empfänger durch Konjugation (Abbildung 1).

Abbildung 1: Konjugationsschema. Dieser Schaltplan zeigt die erfolgreiche Übertragung eines Plasmids, nur ein Beispiel für ein transposierbares DNA-Element, von einer Spenderzelle zu einer Empfängerzelle mittels Konjugation. Bei Kontakt mit der Empfängerzelle durch die Spenderzelle über den Sexpilus repliziert sich das Plasmid durch Rollenkreisreplikation, bewegt sich durch den Multiproteinkomplex, der die beiden Zellen verbindet, und bildet ein neues vollwertiges Plasmid in der Empfängerzelle.
Durch die Inkubation einer Mischung aus Spender- und Empfängerzellen, die diese Zellen sukzessive in Gegenwart von Tetracyclin und DAP plattiert, ermöglichte dies die erfolgreiche Übertragung des Ampicillinresistenzgens. Nacheinander wurden Zellen, die aus dieser Mischung in Gegenwart von Tetracyclin und Ampicillin auswachsen, entfernt, alle Spenderzellen aufgrund des Mangels an DAP und alle Empfängerzellen entfernt, die möglicherweise nicht das Ampicillin-Resistenzgen gewonnen haben, was streng Empfänger-J53-Stamm ergibt. Bakterien, die ampicillinResistenz erworben haben (Abbildung 2). Nach der Durchführung wurde die erfolgreiche Übertragung des Ampicillinresistenzgens durch PCR bestätigt. Da die Konjugation erfolgreich war, enthielt der J53-Stamm von E. coli pWD2-oriT und war resistent gegen Ampicillin, und die Genkodierung für diese Resistenz ist durch PCR nachweisbar. Bei erfolglosem Erfolg hätte es jedoch keinen Nachweis des Ampicillinresistenzgens gegeben und Ampicillin würde immer noch als wirksames Antibiotikum gegen den J53-Stamm funktionieren.

Abbildung 2: Protokollschema. Dieser Schaltplan zeigt eine Übersicht über das vorgestellte Protokoll.
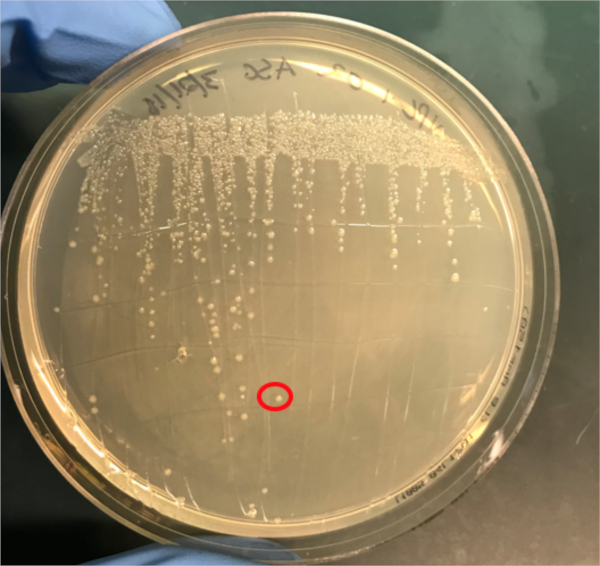
Abbildung 3A: Die Bestätigung einer erfolgreichen Konjugation durch PCR. A) Die Gefrierbestände der konjugierten und negativen Kontrollproben wurden auf Agarplatten ausgestreift und eine Kolonie (rot) für die DNA-Isolierung ausgewählt.
Verfahren
1. Einrichtung
- Autoklav ca. 1L von Luria-Bertani medium (LB). Diese sterile LB wird verwendet, um ca. 5 ml LB mit 0,3 ml Diaminopimelsäure (DAP) herzustellen.
- Sammeln Sie folgende Platten: LB Agarplatten mit 1X Tet und 0,3 mM DAP, LB Agarplatten nur mit 1X Tet und LB Agarplatten nur mit 1X Amp/Tet.
- Stellen Sie sicher, dass einige Glycerin und eine Box mit vorsterilisierten Kunststoff Pipettenspitzen in der Nähe sind.
- Vor Beginn der Arbeiten mit Mikroben, sterilisieren Sie den Arbeitsbereich mit 70% Ethanol. Tragen Sie immer die notwendige persönliche Schutzausrüstung, einschließlich Labormantel und Handschuhe.
- Nach der Fertigstellung alle Oberflächen und Handschuhe mit 70% Ethanol sterilisieren und die Hände waschen.
2. Vorbereitung der Spender- und Empfängerstämme
- 5 ml Bakterienkulturen der Spender- und Empfängerstämme vorbereiten und bei 37 °C mit Belüftung und Schütteln bei 220 Umdrehungen pro Minute über Nacht anbauen. Der Spenderstamm sollte in LB mit 0,3 mM DAP angebaut werden.
- Drehen Sie 1 ml beider Kulturen (ca. 3000 U/min für 5 Minuten) und waschen Sie die Zellen mit PBS.
- Setzen Sie die Zellen in 500 l Phosphatgepufferte Saline (PBS) wieder aus.
3. Konjugation
- Kombinieren Sie 50 l Empfängerzellen mit 50 l Spenderzellen in einem Mikrozentrifugenrohr und mischen Sie sie durch Pipettieren.
- Die Zellmischung 1 Stunde bei 37 °C inkubieren. Dies wird die Effizienz der Konjugation vor und während des nächsten Beschichtungsschritts verbessern.
- Pipetten Sie 100 l des Zellgemisches auf eine Agarplatte mit 1X Tet und 0,3 mM DAP. Nicht auf dem Teller verteilen.
- Pipetten Sie 100 l der Empfängerzellkultur auf eine Agarplatte mit 1X Tet und 0,3 mM DAP. Nicht auf dem Teller verteilen.
- Beide Platten über Nacht bei 37 °C inkubieren.
- Verschrotten Sie die Konjugationszellmischung und die Empfängerzellkultur mit einem sterilen Zellschaber. Übertragen Sie die Zellen in ein steriles Mikrozentrifugenrohr und setzen Sie die Zellen in 1 ml PBS wieder auf.
- Wirbeln Sie die Zellen und drehen Sie sie sanft nach unten (ca. 3000 U/min für 5 Minuten).
- Setzen Sie die Zellen in 1 ml PBS wieder aus.
- Die Konjugationsreaktionszellmischung auf einer LB-Agarplatte nur mit 1X Amp/Tet vereiteln. Auf dieser Platte werden nur die Empfängerbakterien, die das Amp-Resistenzgen durch Konjugation erfolgreich gewonnen haben, voraussichtlich wachsen.
- Die Empfängerzellen nur mit 1X Tet auf eine LB-Agarplatte geben. Auf dieser Platte werden nur nicht konjugierte Empfängerbakterien erwartet, die nicht über das Amp-Resistenzgen verfügen.
- Die Platten über Nacht bei 37 °C inkubieren.
- Wählen Sie einzelne Kolonien aus beiden Platten und verwenden Sie sie, um Übernachtkulturen in 5 ml Medien (37 °C mit Belüftung bei 220 Rpm) zu züchten.
4. DNA-Isolation
- Isolieren Sie DNA aus den zuvor hergestellten Kulturen mit 4,5 ml des gesamten Kulturvolumens durch DNA Miniprep.
- Um dies zu tun, elute die DNA mit 35 l Nuklease-freies Wasser.
- Reine DNA erzeugt ein Absorptionsverhältnis (A260/280) von etwa 1,8.
- Verwenden Sie die restlichen 0,5 ml jeder Kultur, um Glyzerinvorräte zuzubereiten, indem Sie eine 1:1-Mischung aus Bakterienkultur und 100% Glycerin herstellen.
- Lagern Sie die Gefriervorräte bei -80 °C.
5. Bestätigung der Plasmid-Transferkonjugation durch PCR
- Bereiten Sie zwei PCR-Master-Mixe vor, die jeweils einen anderen Satz von Vorwärts- und Rückwärtsprimern aufweisen, wobei einer auf ein 500 Base-Pair-Segment innerhalb des Ampicillin-Resistenz-Gens abzielt und der andere auf ein Segment innerhalb eines Housekeeping-Gens abzielt.
- Housekeeping-Genprimer wurden entwickelt, um ein DNA-Segment innerhalb des bakteriellen Gens zu verstärken, das für DNA-Gyrase B (14) kodiert.
- Die folgenden Reagenzienvolumina wurden verwendet, um 90 l jeder Mastermischung vorzubereiten:
7,5 l von 10 m Vorwärtsgrundierung
7,5 l von 10 m Reverse-Primer
75 l mit 2X PCR Master Mix
- Bereiten Sie die folgenden sechs PCR-Reaktionen mit 15 L-Master-Mix, 10 ng Vorlagen-DNA und nukleasefreiem Wasser bis zu einem Endvolumen von 25 l vor.
Konjugationsreaktion DNA und Ampicillin-Primer
Konjugationsreaktion DNA und Housekeeping Primer
Negative Kontroll-DNA und Ampicillin-Primer
Negativkontroll-DNA und Housekeeping-Primer
Keine DNA und Ampicillin-Primer
Keine DNA- und Housekeeping-Primer - Übertragen Sie diese Reaktionen auf eine PCR-Maschine mit vorgewärmten Block auf 98 C und beginnen Sie mit dem Thermocycling unter folgenden Bedingungen:
98 °C für 30 Sekunden
25-35 Zyklen von 98 °C für 5-10 Sekunden, 45-72 °C für 10 bis 30 Sekunden und 72 °C für 15-30 Sekunden pro kb
72 °C für 5-10 Minuten
Halten bei 4 °C - Laden Sie alle sechs PCR-Reaktionen auf ein 1% Agarose-Gel und laufen Sie bei 150V für ca. 20 Minuten.
- Visualisieren Sie das PC-Produkt mit einem UV-Beleuchtungsgerät.
Ergebnisse
Wenn die Konjugation erfolgreich war, wird ein 500 Basispaar-pcR-Produkt in dem Brunnen beobachtet, in dem PCR-Reaktion 1 geladen wurde (Gut #2 in Abbildung 3B), während keine Bänder in dem Brunnen beobachtet werden, in dem PCR-Reaktion 3 geladen wurde (Well #4 in Abbildung 3B). Das Vorhandensein dieses Bandes bestätigt die erfolgreiche Übertragung des Ampicillinresistenzgens und verleiht damit eine Ampicillinresistenz gegen den J53-Stamm von E. coli.
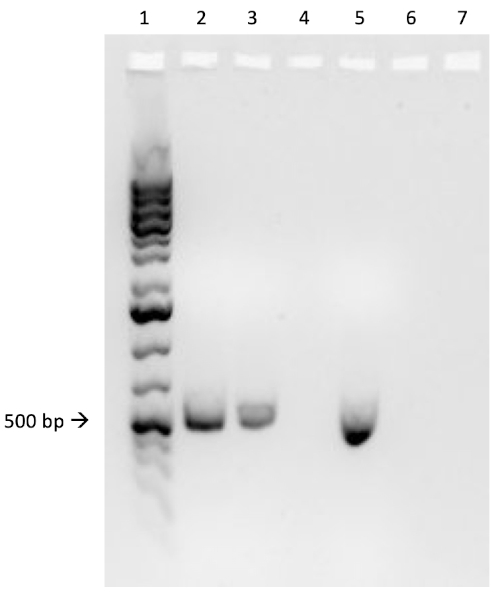
Abbildung 3B: Die Bestätigung einer erfolgreichen Konjugation durch PCR. B) Die PCR-Analyse wurde mit DNA durchgeführt, die aus der ausgewählten Kolonie isoliert wurde. Der Inhalt jedes Brunnens ist wie folgt: 1) DNA-Leiter, 2) Konjugations-DNA und Ampicillin-Primer, 3) Konjugations-DNA und Housekeeping-Primer, 4) Negative Kontroll-DNA und Ampicillin-Primer, 5) Negative Kontroll-DNA und Housekeeping-Primer, 6) Keine DNA und Ampicillin-Primer und 7) Keine DNA- und Negativkontrollgrundierungen. Das Vorhandensein eines PCR-Produkts mit einem Basispaar-PCR-Produkt von 500 ° Chr.-Band (gut 2) und das Fehlen dieses Produkts aus der PCR-Reaktion 3 (gut 4) bestätigen die erfolgreiche Konjugation.
Anwendung und Zusammenfassung
Konjugation ist ein natürlich vorkommender Prozess des horizontalen Gentransfers, der auf dem direkten Zell-zu-Zell-Kontakt einer Spenderzelle und einer Empfängerzelle beruht. Dieser Prozess wird unter allen Arten von Bakterien geteilt und war entscheidend für die bakterielle Evolution, vor allem Antibiotikaresistenz. Im Labor kann konjugation als effektive Methode des Gentransfers eingesetzt werden, die im Vergleich zu anderen Techniken viel weniger störend ist. Außerhalb des Labors bietet die Fähigkeit, DNA von Bakterien auf Eukaryoten über Konjugation zu übertragen, eine aufregende neue Möglichkeit der Gentherapie und das Verständnis der Implikationen dieser natürlich vorkommenden Gentransfers, z. B. die Beziehung zwischen bakterielle Infektion und Krebs, ist ein schnell aufkommendes Forschungsgebiet.
Referenzen
- Lederberg J, Tatum, E.L. Gene recombination in Escherichia coli Nature. 1946;158:558.
- Holmes R.K. J, M.G. Genetics: Exchange of Genetic Information. 4th Edition ed. Baron S, editor. Galveston, TX: University of Texas Medical Branch at Galveston; 1996.
- Cruz F, Davies, J. Horizontal gene transfer and the origin of species: lessons from bacteria. Trends in Microbiology. 2000;8:128-33.
- Llosa M, Cruz, F. Bacterial conjugation: a potential tool for genomic engineering. Ressearch in Microbiology. 2005;156:1-6.
- Lacroix B, Citovsky, V. Transfer of DNA from Bacteria to Eukaryotes. mBio. 2016;7(4):1-9.
- Llosa M, et al. Bacterial conjugation: a two-step mechanism for
- DNA transport. Molecular Microbiology. 2002;45:1-8.
- Grohmann E, Muth, G., Espinosa, M. Conjugative Plasmid Transfer in Gram-Positive Bacteria. Microbiology and Molecular Biology Reviews. 2003;67:277-301.
- Firth N, Ippen-Ihler, K, Skurray, RA. Structure and function of the F factor and mechanism of conjugation. Escherichia coli and salmonella: cellular and molecular biology. 1996;2:2377-401.
- Smillie C, Garcillan-Barcia MP, Francia MV, Rocha EPC, De La Cruz F. Mobility of Plasmids. Microbiology and Molecular Biology Reviews. 2010;74(3):434-52.
- Cascales E. Definition of a Bacterial Type IV Secretion Pathway for a DNA Substrate. 2004;304(5674):1170-3.
- Wang P, Yu Z, Li B, Cai X, Zeng Z, Chen X, et al. Development of an efficient conjugation-based genetic manipulation system for Pseudoalteromonas. Microbial Cell Factories. 2015;14(1):11.
- Yi H, Cho YJ, Yong D, Chun J. Genome Sequence of Escherichia coli J53, a Reference Strain for Genetic Studies. Journal of Bacteriology. 2012;194(14):3742-3.
- Baumann RLB, E. H.; Wiseman, J. S.; Vaal, M.; Nichols, J. S. Inhibition of Escherichia coli Growth and Diaminopimelic Acid Epimerase by 3-Chlorodiaminopimelic Acid. Antimicrobial Agents and Chemotherapy 1988;32:1119-23.
- Rocha D, Santos, CS, Pacheco LG. Bacterial reference genes for gene expression studies by RT-qPCR: survey and analysis. Antonie Van Leeuwenhoek. 2015;108:685-93.
pringen zu...
Videos aus dieser Sammlung:

Now Playing
Konjugation: Eine Methode zur Übertragung der Ampicillinresistenz vom Spender auf den Empfänger E. coli
Microbiology
38.1K Ansichten

Erstellen einer Winogradsky-Säule: Eine Methode zur Anreicherung der mikrobiellen Spezies in einer Sedimentprobe
Microbiology
128.7K Ansichten

Serielle Verdünnungen und Ausplattieren: Auszählung von Mikrobiom
Microbiology
314.8K Ansichten

Anreicherungskulturen: Kultivieren von aeroben und anaeroben Mikroben auf selektiven und differenziellen Medien
Microbiology
131.9K Ansichten

Reinkulturen und Streak-Plating: Isolierung einzelner Bakterienkolonien aus einer gemischten Probe
Microbiology
165.9K Ansichten

16S rRNA-Sequenzierung: Eine PCR-basierte Technik zur Identifizierung bakterieller Arten
Microbiology
188.2K Ansichten

Wachstumskurven: Erzeugen von Wachstumskurven mit Hilfe von koloniebildenden Einheiten und Messungen der optischen Dichte
Microbiology
294.1K Ansichten

Antibiotika-Empfindlichkeitstests: Epsilometertests zur Bestimmung der MHK-Werte von zwei Antibiotika und zur Bewertung der Antibiotika-Synergie
Microbiology
93.6K Ansichten

Mikroskopie und Färbungen: Gram-, Kapsel- und Endosporenfärbung
Microbiology
362.7K Ansichten

Plaque-Assay: Eine Methode zur Bestimmung des Virustiters als Plaque bildende Einheiten (PFU)
Microbiology
185.8K Ansichten

Transformation von E. coli-Zellen mit einem adaptierten Calciumchlorid-Verfahren
Microbiology
86.4K Ansichten

Phagen-Transduktion: Eine Methode zur Übertragung der Ampicillinresistenz von einem Spender auf den Empfänger E. coli
Microbiology
29.0K Ansichten
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten