Method Article
Eine einfache, schnelle und effektive Methode zur Analyse der Tumor-Xenotransplantation in transparenten Zebrafischembryonen
In diesem Artikel
Zusammenfassung
Wir beschreiben ein Protokoll für die Xenotransplantation in das Eigelb von transparenten Zebrafischembryonen, das durch eine einfache, schnelle Staging-Methode optimiert wird. Die Analysen nach der Injektion umfassen das Überleben und die Beurteilung der Krankheitslast von xenotransplantierten Zellen durch Durchflusszytometrie.
Zusammenfassung
In-vivo-Studien zum Tumorverhalten sind ein Grundpfeiler der Krebsforschung; Der Einsatz von Mäusen stellt jedoch erhebliche Herausforderungen in Bezug auf Kosten und Zeit dar. Hier stellen wir den larvalen Zebrafisch als Transplantationsmodell vor, das zahlreiche Vorteile gegenüber Mausmodellen hat, darunter einfache Handhabung, geringe Kosten und kurze Versuchsdauer. Darüber hinaus macht das Fehlen eines adaptiven Immunsystems während der Larvenstadien die Notwendigkeit überflüssig, immundefiziente Stämme zu erzeugen und zu verwenden. Während es etablierte Protokolle für die Xenotransplantation in Zebrafischembryonen gibt, stellen wir hier eine verbesserte Methode vor, die das Embryo-Staging für einen schnelleren Transfer, die Überlebensanalyse und den Einsatz von Durchflusszytometrie zur Beurteilung der Krankheitslast umfasst. Die Embryonen werden in Stadien gesetzt, um eine schnelle Zellinjektion in das Eigelb der Larven und eine Zellmarkierung zu ermöglichen, um die Konsistenz des injizierten Zellbolus zu überwachen. Nach der Injektion wird die Überlebensanalyse des Embryos bis zu 7 Tage nach der Injektion (dpi) bewertet. Schließlich wird auch die Krankheitslast durch Markierung übertragener Zellen mit einem fluoreszierenden Protein und Analyse mittels Durchflusszytometrie bewertet. Die Durchflusszytometrie wird durch eine standardisierte Methode zur Herstellung von Zellsuspensionen aus Zebrafischembryonen ermöglicht, die auch bei der Etablierung der Primärkultur von Zebrafischzellen verwendet werden könnte. Zusammenfassend lässt sich sagen, dass das hier beschriebene Verfahren eine schnellere Beurteilung des Verhaltens von Tumorzellen in vivo mit einer größeren Anzahl von Tieren pro Studienarm und auf kostengünstigere Weise ermöglicht.
Einleitung
Die Analyse des Verhaltens von Tumoren als Reaktion auf genetische Veränderungen oder medikamentöse Behandlung in vivo ist ein wesentliches Element der Krebsforschung 1,2,3,4. Solche Studien beinhalten meist die Verwendung von immungeschwächten Mausmodellen (Mus musculus)5; Xenotransplantationsstudien an Mäusen sind jedoch in vielerlei Hinsicht begrenzt, einschließlich begrenzter Kapazitäten, längerer Dauer, erheblicher Kosten und des Bedarfs an hochentwickelten Bildgebungsgeräten zur Überwachung des Fortschreitens interner Tumoren 6,7. Im Gegensatz dazu ermöglicht das Zebrafischmodell (Danio rerio) eine höhere Kapazität, eine kürzere Dauer, geringere Kosten und aufgrund seiner Transparenz eine einfache Überwachung des Krankheitsverlaufs 8,9.
Der Zebrafisch ist ein gut entwickeltes Wirbeltiermodellsystem mit Ex-utero-Entwicklung und hoher Fruchtbarkeit, wobei einzelne Weibchen mehr als 100 Embryonen produzieren10. Darüber hinaus sind Zebrafischembryonen transparent, was eine einfache Visualisierung von Entwicklungsprozessen mit Hilfe von Fluoreszenztechniken wie Reportern ermöglicht. Schließlich macht die Konservierung kritischer Entwicklungsprozesse sie zu einem idealen Modell für viele Arten von Studien, einschließlich des Verhaltens transplantierter maligner Zellen 11,12. Wildtyp-Zebrafischembryonen entwickeln Melanozyten, die sie im Alter von 2 Wochen optisch undurchsichtig machen, was jedoch durch die Erzeugung von Casper-Embryonen überwunden wurde (RoyA9; mitfaw2), die ein Leben lang transparent bleiben13. Aufgrund ihrer optischen Eigenschaften sind Casper-Zebrafische ideale Empfänger von transplantierten Tumorzellen 14,15,16. Die Xenotransplantation von Tumorzellen in Zebrafische hat in den letzten 2 Jahrzehnten an Bedeutung gewonnen 17,18,19,20,21. Zebrafischembryonen haben eine angeborene Immunität; Ihnen fehlt jedoch während ihres Larvenstadiums eine adaptive Immunität, wodurch sie funktionell immungeschwächt sind, was es ihnen ermöglicht, als effektive Wirte für transplantierte Tumor-Xenotransplantate zu dienen22.
Für die Tumortransplantation in Zebrafischembryonen sowie bei Erwachsenen wurden Protokolle entwickelt, die eine Reihe verschiedener Variablen berücksichtigen 23,24,25,26,27. Diese haben zahlreiche Stellen der Tumorablagerung im Zebrafisch untersucht, darunter Injektionen in das Dotter, den perivitellinischen Raum und das Herz und in verschiedenen Entwicklungsstadien 16,28. Die Umgebungstemperatur der Aquakultur für Zebrafisch-Xenotransplantate ist ebenfalls wichtig, da die Aufzucht von Zebrafischen in der Regel bei 28 °C erfolgt, während die Säugetierzellen bei 37 °C wachsen. Folglich muss eine Kompromisstemperatur gewählt werden, die von den Fischen toleriert wird, aber das Tumorwachstum unterstützt, und 34 °C scheinen beide Ziele zu erreichen29. Die Analyse des Verhaltens und der Progression von Tumoren nach Xenotransplantation ist ein weiterer wichtiger Schwerpunkt, der den Einsatz einer Vielzahl von bildgebenden Verfahren sowie die Überlebensanalyse umfasst30. Einer der Hauptvorteile des Zebrafischmodells ist die Verfügbarkeit einer großen Anzahl von Versuchstieren, die eine immense statistische Aussagekraft für In-vivo-Studien des Tumorverhaltens bieten. Bisherige Ansätze haben dieses Potenzial jedoch stark eingeschränkt, da langwierige Montageverfahren für Injektionen erforderlich sind.
Hier beheben wir diese Einschränkung durch die Entwicklung einer einfachen, schnellen Methode zur Stadierung von Embryonen, die einen hohen Durchsatz und eine Überwachung der Injektionsqualität mit der transparenten Casper-Zebrafischlinie ermöglicht. Dabei werden Xenotransplantate 2 Tage nach der Befruchtung (dpf) in den Dottersack der Casper-Zebrafischembryonen injiziert. Wir beobachten das Überleben von Embryonen nach Xenotransplantation im Rahmen der Tumorverhaltensanalyse. Des Weiteren zeigen wir die Abschätzung der Krankheitslast nach Xenotransplantation durch die Herstellung von Einzelzellsuspensionen und die Analyse mittels Durchflusszytometrie (Abbildung 1).
Protokoll
Die Haltung, Fütterung und Haltung von Zebrafischen erfolgte unter Standard-Aquakulturbedingungen bei 28,5 °C, wie beschrieben31. Alle Experimente mit Zebrafischen wurden bei dieser Temperatur durchgeführt; Nach der Xenotransplantation wurden die Tiere jedoch für die Dauer des Versuchs bei 34 °C kultiviert, gemäß den vom Institutional Animal Care and Use Committee (IACUC) genehmigten Verfahren.
1. Zucht (3 Tage vor der Injektion)
- Geben Sie Fischpaaren eine Woche vor der Zucht Trockenfutter (zusätzliches Futter; 5-6 Granulate pro Fisch), um die Tiergesundheit zu maximieren und die Anzahl der von Zuchtpaaren produzierten Embryonen zu erhöhen.
- Setzen Sie die Zuchttiere am Abend vor der Embryonenentnahme in Zuchtbecken mit einer Trennwand ein, die männliche und weibliche Fische trennt, wobei Sie Haremspaarungen von 2 Weibchen für jedes Männchen verwenden.
HINWEIS: Bei Versuchen mit 4 Armen mit 100 Tieren pro Arm sind 20 Zuchtpaare pro Versuch zu beschäftigen. Um die Anzahl der benötigten Zuchtpaare zu bestimmen, liegt eine gute Schätzung bei 50 Embryonen pro Casper-Zuchtpaar . Eine andere Möglichkeit besteht darin, einen robusteren, pigmentierten Zebrafischstamm zu verwenden und mit 1-Phenyl-2-Thioharnstoff (PTU) zu behandeln, um Pigmentflecken zu verhindern31. In der Praxis sollte das Experiment so skaliert werden, dass man genügend Embryonen hat, um 1 Tag nach der Injektion (dpi) die doppelte Anzahl der gewünschten Embryonen zu injizieren.
2. Embryonenentnahme (2 Tage vor der Injektion)
- Entfernen Sie am nächsten Morgen die Trennwände, damit sich die Fische vermehren können.
- Visualisieren Sie die Embryonen in den Tanks 20 Minuten (min) nach dem Entfernen der Trennwände.
- Sammeln Sie die Embryonen mit einem Sieb in einer 90-mm-Petrischale, die Embryowasser enthält, das wie im Zebrafischbuch31 beschrieben hergestellt wurde.
- Um Embryowasser herzustellen, fügen Sie 1,5 ml Stammsalze zu 1 l destilliertem Wasser und Methylenblau zu 0,1 % endgültig hinzu. Stellen Sie die Salzlösung her, indem Sie 40 g Meersalz (Tabelle der Materialien) in 1 l destilliertem Wasser auflösen. Die ionische Zusammensetzung des Embryonenwassers besteht aus K+ (0,68 mg/L), Cl− (31,86 mg/L), Na+ (17,77 mg/L), SO4− (4,47 mg/L), Mg2+ (2,14 mg/L) undCa2+ (0,68 mg/L).
- Lassen Sie den Zebrafisch eine zusätzliche Stunde lang brüten und sammeln Sie die daraus resultierenden Embryonen.
- Legen Sie die Embryonen für das Experiment zusammen.
- Entfernen Sie abends alle unbefruchteten oder toten Embryonen, die an ihrer abnormalen Morphologie erkennbar sind, und stellen Sie frisches Embryowasser zur Verfügung.
3. Pflege des Embryos und Vorbereitung des Werkzeugs für die Injektion (1 Tag vor der Injektion)
- Entfernen Sie am nächsten Morgen alle zusätzlichen toten Embryonen und stellen Sie frisches Embryowasser bereit.
- Bereiten Sie eine Agaroseplatte vor, indem Sie 1,5%ige Agarose in Embryowasser erhitzen und die erhitzte Mischung in eine 90 mm Petriplatte gießen. Eine 90-mm-Schale benötigt 30-35 mL der Mischung.
- Ziehen Sie nicht-filamentige Nadeln mit dem Nadelabzieher aus Glaskapillaren (Borosilikat). Die Nadeln werden (unter Hitzedruck) gezogen, wodurch ein geschlossenes Ende entsteht; Klemmen Sie sie mit einer Pinzette ab, um eine optimale Öffnung zu erzeugen. Die Eignung der Nadelöffnung ist zu beurteilen, indem das Volumen der pro Zeiteinheit verdrängten Flüssigkeit bestimmt wird (siehe Abschnitt 6.3 unten).
HINWEIS: Glaskapillaren können mit oder ohne zentrale Filamente erworben werden. Kapillaren ohne zentrale Filamente werden bevorzugt für Zellinjektionen verwendet. - Legen Sie die Nadeln in eine abgedeckte 90 mm Petriplatte in die Rillen aus Ton (Kindermodelliermasse), bis sie verwendet werden (Abbildung 2A).
4. Vorbereitung und Markierung von Leukämiezellen mit CM-Dil (Tag der Injektion)
- Halten Sie die zu transplantierenden Zellen unter optimalen Bedingungen für ihr Wachstum. Die hier verwendeten murinen Leukämiezellen waren entweder ausreichend (M82; Rpl22+/+) oder mangelhaft (M109; Rpl22-/-) für das ribosomale Protein Rpl22, das als Tumorsuppressorfungiert 32.
- Pelletzellen in einem konischen 50-ml-Röhrchen. Zählen, dann bei 300 x g bei Raumtemperatur (RT) 5 min zentrifugieren. Entsorgen Sie den Überstand.
HINWEIS: Die Anzahl der benötigten Zellen wird durch den Versuchsumfang und die Bedingungen bestimmt, aber 1 x 106 Zellen ist ein guter Ausgangspunkt. - CM-Dil-Färbung durchführen
HINWEIS: Die CM-Dil-Färbung ermöglicht die Überwachung des Injektionsbolus.- Stellen Sie eine Stammlösung von CM-Dil her, indem Sie eine 50 μg Durchstechflasche CM-Dil in 50 μl Dimethylsulfoxid (DMSO; 1 mg/ml oder ~1 mM endgültig) resuspendieren.
- Stellen Sie eine Arbeitslösung her, indem Sie die Brühe (4,8 μl Fleck/ml) in 1 % fötalem Rinderserum (FBS)/Hank's Balanced Salt Solution (HBSS) verdünnen, die alle unterstützenden Nahrungsergänzungsmittel enthält, die von den zu verwendenden Zellen benötigt werden.
- Die Zellen werden bei 1 x 106/100 μl in der Arbeitslösung des Farbstoffs resuspendiert.
- Bei 37 °C 10 min inkubieren.
HINWEIS: Die Färbebedingungen müssen für die verwendeten Zellen (Zeit usw.) optimiert werden. Die hier verwendeten Zellen erforderten zwei unterschiedliche 10-minütige Inkubationen bei unterschiedlichen Temperaturen, um eine optimale Färbung zu erreichen. - Mit 10 mL 1x HBSS bei Raumtemperatur (RT) waschen.
- Pelletzellen (Zentrifugation bei 300 x g für 5 min bei RT), den Überstand dekantieren, dann mit 10 mL HBSS resuspendieren und für eine zweite Wäsche wiederholen.
- Resuspendieren Sie die gefärbten Tumorzellen bei 40.000 Zellen/μl in 1 % FBS/PBS und allen erforderlichen unterstützenden Nahrungsergänzungsmitteln und halten Sie die Zellsuspension bis zur Injektion bei 34 °C.
HINWEIS: Für die Aufrechterhaltung der in dieser Studie verwendeten Zelllinien waren unterstützende Nahrungsergänzungsmittel erforderlich (z. B. 1 % FBS und Zytokine). Die Nahrungsergänzungsmittel und die Xenotransplantation müssen möglicherweise an die jeweiligen untersuchten Zelllinien angepasst werden. PBS wurde hier anstelle von Medien ausgewählt, um eine mögliche Toxizität im Eigelb zu vermeiden.
5. Dechorionation
- Dechorionieren Sie die Casper-Zebrafischembryonen manuell bei 2 dpf mit Insulininjektionsspritzen unter 2-facher Vergrößerung in einem Lichtmikroskop. Durchstechen Sie das Chorion mit einer Nadel, während Sie mit der anderen Nadel das Chorion ruhigstellen.
HINWEIS: Die Verwendung von Pronase zur Dechorionierung wird nicht empfohlen, da sie manchmal zu einer verminderten Gesundheit des Embryos führt. Die Dechorionisierung der Embryonen bei 2 dpf wird bevorzugt, da sie einfacher ist und die Embryonen weniger zerbrechlich sind als zu früheren Zeiten (1 dpf). - Achten Sie beim Dechorionieren darauf, die Embryonen nicht mit den Nadeln zu berühren. Das Berühren oder Beschädigen des Eigelbs von Embryonen durch versehentlichen Kontakt mit den Nadeln kann zum Tod führen.
- Entfernen Sie die abgestreiften Chorionen, indem Sie das Embryowasser wechseln.
6. Einrichten des Mikroinjektors und der Nadel
- Schalten Sie den Mikroinjektor und die Pumpe ein und richten Sie die Bedingungen ein, die für Mikroinjektionen von Zellen geeignet sind. Ein Einspritzdruck von 9-11 psi und eine Einspritzzeit von 0,5 Sekunden (s) sind ein guter Ausgangspunkt für das Abschneiden der Nadel und das Einstellen der Öffnung.
- Laden Sie die Suspension der Tumorzelllinie (~5 μl) vorsichtig in einem einzigen Durchgang in die Mikronadel, um die Bildung von Luftblasen zu vermeiden, die den Zellstrom in der Nadel stören.
- Schneiden Sie das Ende der Nadel mit einer Pinzette (Dumont-Zahl 5) ab, um eine Öffnung zu erzeugen, die den Auswurf von 10-15 Nanolitern (nL) Zellsuspension pro 0,2-0,3 s unterstützt.
HINWEIS: Die obige Berechnung des nL-Volumens erfolgt unter Verwendung von Kalibrierkapillaren, wobei 1 mm = 30 nL. Kurz gesagt, stellen Sie die Zeit auf 0,5 s ein, drücken Sie nach jedem Nadelclip auf Inject und sammeln Sie das Volumen in der Kalibrierkapillare. Dann wird die Länge des gesammelten Volumens mit der Skala unter einem Mikroskop gemessen, und das Clipping der Nadel wird gestoppt, wenn 30 nL in 0,5 s injiziert werden. Stellen Sie dann die Zeit der Injektionen auf 0,2-0,3 s ein (Injektion von ~10-15 nL Zellen)
7. Vorbereitung des Embryos für die Injektion
- Wählen Sie gesunde Embryonen unter dem Mikroskop aus und sortieren Sie alle mit Entwicklungsanomalien wie Herzödemen oder einem kurzen oder gekrümmten Rumpf aus.
- Betäuben Sie die Embryonen mit Tricain-Methansulfonat (MS-222; 0,16 g/l Embryowasser) für 1 min in einer 90 mm Petriplatte.
- Verwenden Sie eine Pasteurpipette aus Glas, um die Embryonen zu entnehmen. Ordnen Sie 10-15 Embryonen in einer seitlichen Position auf der 1,5%igen Agaroseplatte an (Abbildung 2B-D).
- Entfernen Sie überschüssiges Wasser mit der Pasteur-Pipette und lassen Sie die minimale Menge an Embryonenwasser übrig, die benötigt wird, um die Embryonen am Leben zu erhalten.
8. Ablauf der Injektion
- Überprüfen Sie unter dem Lichtmikroskop, ob sich die Zellen in der Nadelspitze angesammelt haben.
- Injizieren Sie die Embryonen mit der kalibrierten Nadel für 0,2-0,3 s (mit 10-15 nL entsprechend 400-600 Zellen) in das Eigelb der Embryonen.
- Wiederholen Sie die Injektion für alle Embryonen und sammeln Sie sie dann in frischem Embryowasser.
HINWEIS: Da sich Zellen an der Nadelspitze ansammeln, muss die Spitze alle 15-20 Injektionen leicht zurückgespült werden. Dies erfordert auch ein Zurücksetzen des Drucks und der Zeit bei jedem erneuten Clipping, um sicherzustellen, dass ein ähnliches Volumen injiziert wird. - Um sicherzustellen, dass der Vergleich des Verhaltens zweier unterschiedlicher Sätze übertragener Zellen gültig ist, überwachen Sie den Bolus der übertragenen Zellen. Tun Sie dies, indem Sie die Embryonen basierend auf der CM-Dil-Färbung (im RFP-Kanal) 1 Stunde nach der Injektion (hpi) sortieren und diejenigen mit optimaler Färbung ("guter Bolus") von denen mit minderwertiger Färbung ("minderer Bolus") trennen; Abbildung 3, gelber Pfeil).
- Entsorgen Sie die Embryonen mit einem minderwertigen Bolus oder verwenden Sie sie, um den Einfluss einer anderen Zelldosis auf das Fortschreiten der Krankheit zu beurteilen.
- Entfernen Sie alle toten Embryonen bis zum Ende des Tages, da ihr Tod eher mit einem Injektionstrauma als mit dem Tumorwachstum zusammenhängt. Embryonen aus der Analyse zu entfernen, die keine Zellen zurückhalten, da die Zellen wahrscheinlich aus der Injektionsstelle ausgetreten sind.
- Halten Sie die injizierten Embryonen für die Dauer des Experiments bei 34 °C in einer 90-mm-Schale mit ~60 Embryonen pro Platte.
HINWEIS: Nach 5 dpf wurde das Eigelb von den wachsenden Embryonen verzehrt, daher müssen die Embryonen für die Dauer des Experiments mit Pantoffeltierfutter versorgt werden. Um eine angemessene Ernährung zu gewährleisten, sollte den Embryonen täglich eine Lunge von 6 dpf (4 dpi) bis 9 dpf (7 dpi) verabreicht werden. Pantoffeltierchen werden durch Kultivierung in Kolben unter optimalen Ernährungs- und Temperaturbedingungen vermehrt, wie beschrieben31.
9. Überlebensanalyse
- Überwachen Sie die Embryonen für die nächsten 7 dpi und wechseln Sie das Embryowasser täglich. Der Wasserwechsel kann der Einfachheit halber auf abwechselnde Tage reduziert werden, wenn die Studie eine medikamentöse Behandlung umfasst.
- Überprüfen Sie die Gesundheit der Embryonen und bewerten Sie den Tod für die Dauer der Analyse.
HINWEIS: Die Versuchsdauer für dieses Experiment betrug 7 Tage, kann aber je nach Aggressivität des xenotransplantierten Tumors kürzer oder länger sein. - Verwenden Sie die CM-Dil-Fluoreszenz, um die Krankheitslast zu beurteilen (Abbildung 4A) und bestimmen Sie den Einfluss genetischer Veränderungen oder medikamentöser Behandlungen auf das Überleben mit Hilfe der Kaplan-Meier-Analyse und stellen Sie sie grafisch dar (z. B. mit GraphPad Prism; Abbildung 4B) 33. Urheberrecht
10. Einzelzellsuspension von Embryonen für die Durchflusszytometrie-Analyse
HINWEIS: Die Krankheitslast kann durch Durchflusszytometrie nach Xenotransplantation beurteilt werden; Dazu ist jedoch eine unauslöschliche Markierung der Tumorzellen erforderlich. Retroviral oder lentiviral verabreichtes rotfluoreszierendes Protein (RFP) oder mCherry ist wirksam, da es ein gutes Signal gegenüber der Autofluoreszenz von Zebrafischzellen liefert, die das Signal des grün fluoreszierenden Proteins verdeckt.
- Isolieren Sie Embryonen im dpi-Stadium Ihrer Wahl. Hier werden 5 dpi angezeigt (Abbildung 5).
- Sammeln Sie 30-40 Embryonen pro Zustand als Ausgangspunkt, aber die Anzahl der Embryonen kann je nach Stadium und Aggressivität der transplantierten Zellen variieren. Betäuben Sie die Embryonen wie oben beschrieben.
HINWEIS: Embryonen können als Replikate unterteilt werden, um eine statistische Signifikanz zu gewährleisten, wie hier gezeigt (Abbildung 5B). - Übertragen Sie die Embryonen in 1,5-ml-Zentrifugenröhrchen.
- Verwenden Sie 100 μl kalziumfreie Ringer-Lösung (Rezept31) pro Probe, um das Eigelb aufzulösen, da ein niedriger Kalziumspiegel das embryonale Gewebe weich macht und eine effektivere Gewebedissoziation ermöglicht.
- Pipettieren Sie 5 Minuten lang intermittierend auf und ab, um das Eigelb mit einer 200-μl-Pipettenspitze zu entfernen.
- 0,05 % Trypsin/PBS (ohne Phenolrot-Indikator) auf 29 °C vorwärmen und mit 27 μl Kollagenase IV (100 mg/ml) pro ml Trypsinlösung ergänzen. Für jede Embryonenprobe wird ein Volumen von 1 ml Lösung benötigt.
- Geben Sie 1 ml der Trypsin/Kollagenase-Lösung zu jeder Probe von deyotterten Embryonen und inkubieren Sie sie 30-35 Minuten lang bei 29 °C.
- Pipettieren Sie die Embryonen in dieser Lösung mit einer 1 ml-Pipettenspitze alle 5 Minuten auf und ab, bis die Struktur der Embryonen (Rückgrat) nicht mehr sichtbar ist.
- Stoppen Sie die Reaktion mit 200 μl FBS.
- Gut mischen und weitere 5 Minuten bei 29 °C inkubieren, um eine vollständige Inaktivierung des Trypsins zu gewährleisten.
HINWEIS: Für das Gewebedissoziationsprotokoll wird eine Temperatur von 29 °C verwendet, um den durch einen Hitzeschock verursachten Tod von Zebrafischzellen zu verhindern, der bei 37 °C auftritt. Wenn jedoch keine Konservierung der Zebrafischzellen erforderlich ist, kann der Aufschluss bei 37 °C durchgeführt werden. - Die Zellsuspension bei 300 x g für 5 min bei 4 °C pelletieren und den Überstand verwerfen.
- Resuspendieren Sie das Zellpellet in 4 °C PBS und pelletieren Sie es wie oben.
- Wiederholen Sie den Waschvorgang und seihen Sie die Zellen dann durch ein 70-μm-Zellsieb.
- Pelletieren und mit der Färbung für die Durchflusszytometrie-Analyse fortfahren.
HINWEIS: Wenn eine Kultivierung von primären Zebrafischzellen erforderlich ist, waschen Sie sie noch zweimal mit 4 °C PBS und resuspendieren Sie sie in L15-Medien (mit Antibiotika und 10 % FBS).
11. Fluoreszenzaktivierte Zellsortierung (FACS): Färbung und Sortierung von xenotransplantierten Zellen
- Die Zellsuspension in einem Färbemedium (HBSS mit 1% FBS) resuspendieren und 5 min bei 300 x g pelletieren.
- Resuspendieren Sie das Zellpellet im Färbemedium mit einem Antikörper, der mit den transplantierten Zellen reaktiv ist, um einen zweiten Marker (neben RFP oder mCherry) bereitzustellen, mit dem transplantierte Zellen von Zebrafischzellen unterschieden werden können. Hier wurden 50 μl Anti-Maus-CD45 (APC-CD45) pro Probe in einer Verdünnung von 1:50 verwendet (Abbildung 5).
- 20 min bei 4 °C inkubieren, bevor Sie wie oben beschrieben mit 1 mL des Färbemediums waschen, um den ungebundenen Antikörper zu entfernen.
- Nach dem Verwerfen des Überstands wird das Zellpellet in 200 μl Färbemedium resuspendiert, das den lebenswichtigen Farbstoff Helix NP Blue (1 μM) enthält, der eine Unterscheidung zwischen lebend und tot ermöglicht.
- Übertragen Sie die Zellmischung für die Durchflusszytometrie-Analyse in 5-ml-Polycarbonat-Röhrchen mit rundem Boden.
HINWEIS: Die parallele Färbung einer Tumorzellkontrolle schafft Klarheit beim Ziehen von Gates während der Durchflusszytometrie-Analyse.
12. Durchflusszytometrie
- Schalten Sie das verfügbare Durchflusszytometer mit einer niedrigen Durchflussrate (500 Ereignisse pro Sekunde oder weniger) ein, um die Parameter einzustellen.
- Verwenden Sie die Tumorzellkontrolle (die gleichen Zellen, die für die Xenotransplantation verwendet werden), um die Spannung für die Kanäle FSC (Vorwärtsstreuung), SSC (Seitenstreuung), BV421/CasB (Viabilität), CE-594 (mCherry) und APC (CD45.2) einzustellen.
HINWEIS: Einzeln gefärbte Proben sind erforderlich, um die Kompensationseinstellungen festzulegen, die eine Fluorochromüberlappung zwischen verschiedenen Flecken eliminieren. - Verwenden Sie die nicht injizierte Embryoprobe, um Einstellungen zu schaffen, die sowohl transplantierte als auch Zebrafischzellen aufnehmen.
- Erhöhen Sie die Flussrate auf 8000 Ereignisse pro Sekunde und zeichnen Sie 1 Million Ereignisse für jede Probe auf.
- Analysieren Sie die resultierenden Daten mit einer geeigneten Analysesoftware, indem Sie zunächst Einzellinge auswählen, indem Sie FSC-H v/s FSC-A (Höhe vs. Fläche) plotten, gefolgt von einem Diagramm zur Auswahl lebensfähiger Zellen. Die FlowJo-Software wird häufig für solche Analysen verwendet.
- Ziehen Sie mit der Tumorzellkontrolle ein Tor um die transplantierten Zellen durch FSC-A vs. SSC-A, dann verwenden Sie die Indikatorfärbungen, in diesem Fall CD45 und mCherry (Abbildung 5B).
HINWEIS: Das für den Tumor ausgewählte Größentor enthält auch Zebrafischzellen, was die Grundlage für die Normalisierung und Bestimmung der Krankheitslast bildet. - Analysieren Sie die Embryoproben mit den gleichen Gate-Einstellungen wie oben. Das endgültige Diagramm der Tumorfärbung und des Indikators für fluoreszierende Proteine wird ein Maß für die Krankheitslast liefern (Abbildung 5C).
HINWEIS: Hier wurde der Unterschied in der Krankheitslast für Embryonen aufgetragen, die einen guten Bolus aus injizierten Zellen erhielten, und für diejenigen, die einen minderwertigen Bolus erhielten. - Bei Bedarf sortieren Sie die Tumorzellen in den Embryonen mittels Durchflusszytometrie für die nachgelagerte molekulare Analyse.
Ergebnisse
Xenotransplantation
In Abbildung 1 ist ein umfassender Überblick über das gesamte Experiment und die Analyse dargestellt, die von der Embryonenproduktion bis zur Beurteilung des Krankheitsverlaufs durch Überlebens- und Krankheitslastanalyse mittels Durchflusszytometrie reicht. Dieser Ansatz bringt mehrere Verbesserungen mit sich, die die Reproduzierbarkeit und Skalierbarkeit der Xenotransplantation verbessern und eine neue Methode zur Bewertung der Krankheitslast bieten. Der Erfolg dieser Experimente hängt stark von der Gesundheit der transplantierten Zellen ab, da Zellen, die nicht gesund sind und sich in der logarithmischen Phase befinden, sich nach der Transplantation nicht vermehren. Die Dauer der Injektionssitzung ist ebenfalls ein kritischer Parameter. Nachdem die Tumorzellen vorbereitet wurden, ist es wichtig, die Injektion in den Zebrafisch innerhalb von 3-4 Stunden abzuschließen. Der in dieser Studie verwendete Ansatz ermöglicht es, eine größere Anzahl von Embryonen in diesem Zeitraum zu injizieren, indem sie einfach direkt auf der Seite auf einer Agaroseplatte platziert und in das Eigelb injiziert werden (Abbildung 2C,D). Darüber hinaus ist es unerlässlich, dass die optimale Nadelöffnung so gewählt wird, dass genügend Zellen injiziert werden (400-600 Zellen), aber die Öffnung nicht so groß ist, dass die Embryonen verletzt werden. Ein weiterer Aspekt ist der Einspritzdruck. Wir stellen fest, dass ein Druck von mehr als 12-13 psi das Eigelb der Embryonen stört und zum Tod führt. Eine weitere Variabilität, die diesem Verfahren inhärent ist, ist die Konsistenz der Injektion. Die zu injizierenden Zellen setzen sich am Ende der Injektionsnadel ab, was eine präzise Steuerung des Injektionsbolus erschwert. Wenn die Zellen xenotransplantiert werden, haben alle Embryonen das Potenzial, den gleichen Injektionsbolus zu erhalten, in der Praxis ist dies jedoch nicht der Fall (Abbildung 3). Die Anzahl der übertragenen Zellen kann je nach Verhalten der Tumorzellen (z. B. Verklumpung) und dem Kenntnisstand des Bedieners stark variieren. Wir haben diese Unsicherheit durch CM-Dil-Färbung/mCherry-Markierung angegangen, die eine Kategorisierung von Tieren nach der Injektion ermöglicht, die einen angemessenen und konsistenten Zellbolus erhalten haben, sowie von Tieren, die einen minderwertigen Bolus erhalten haben. Die CM-Dil-Färbung, die jedoch effektiver mit einem fluoreszierenden Protein markiert werden kann, hat den zusätzlichen Vorteil, dass sie die Überwachung des Krankheitsverlaufs entweder durch Mikroskopie oder durch Durchflusszytometrie erleichtert (Abbildung 4 und Abbildung 5).
Analyse des Tumorverhaltens
Die Tumorprogression kann mit einer einfachen Fluoreszenzmikroskopie, die sich auf RFP konzentriert, leicht überwacht werden (Abbildung 4A). Ebenso kann die traditionelle Überlebensüberwachung durch die Kaplan-Meier-Analyse (Log-Rank- und Wilcoxon-Test) durchgeführt werden (Abbildung 4B). Beeindruckend ist, dass es im Gegensatz zu mausbasierten Xenotransplantationsstudien, bei denen typischerweise 8-10 Tiere pro Studienarm vorhanden sind, bei der hier beschriebenen Zebrafischmethode nicht schwierig ist, Studienarme mit jeweils mehr als 60 Tieren zu erreichen (Abbildung 4B). Dies erhöht das Auflösungsvermögen von in vivo-Studien deutlich. Schließlich haben wir einen weiteren Ansatz für die Analyse der Krankheitslast mittels Durchflusszytometrie implementiert. Dies beinhaltet die Zerstörung einer entsprechenden Anzahl von Embryonen und die Analyse des Tumorzellgehalts der resultierenden Einzelzellsuspension mittels Durchflusszytometrie. Durch die Kombination eines tumorspezifischen Zelloberflächenmarkers mit dem fluoreszierenden Proteinindikator können die xenotransplantierten Mäuse/menschlichen Zellen mittels Durchflusszytometrie als Ansatz zur Beurteilung der Krankheitslast sicher identifiziert werden (Abbildung 5). Zu diesem Zweck sind rot fluoreszierende Proteine überlegen, da die grün fluoreszierenden Proteine kein Signal über die Autofluoreszenz der Zebrafischzellen des Wirts liefern konnten. Hier wurde mCherry zur Zellmarkierung und zum Monitoring im Rahmen der Xenotransplantation für die FACS-Analyse zusammen mit CD45 eingesetzt. Die doppelte Markierung ermöglichte es uns, Unterschiede in der Tumorlast zwischen guter und minderer Bolusimpfung zu messen (Abbildung 5B,C).
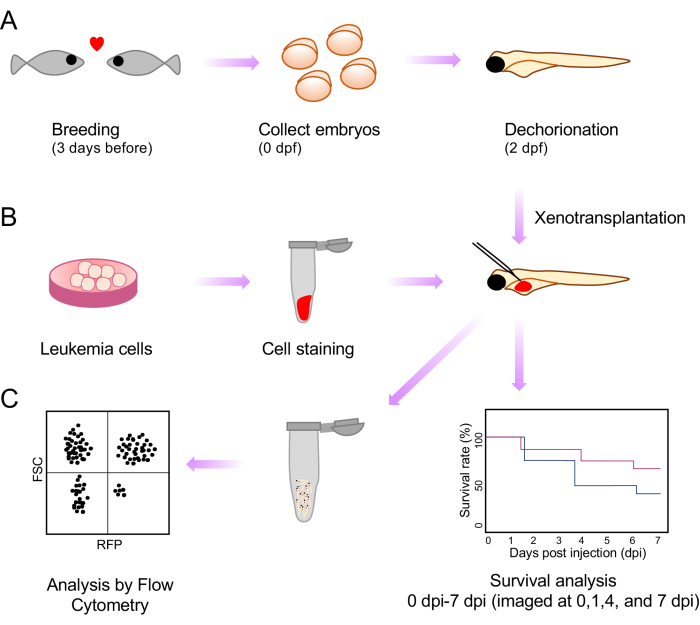
Abbildung 1: Schematische Darstellung des gesamten Verfahrens der Xenotransplantation und der Analyse nach der Injektion. (A) Der Aufbau der Zucht, die Embryonenentnahme und die Morphologie 2 Tage nach der Befruchtung (dpf) sind schematisiert. (B) Vorbereitung, Färbung und Injektion von Leukämiezellen für die Xenotransplantation in das Eigelb von Zebrafischembryonen. (C) Analysen nach Xenotransplantationen, einschließlich Überlebens- und Durchflusszytometrie. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
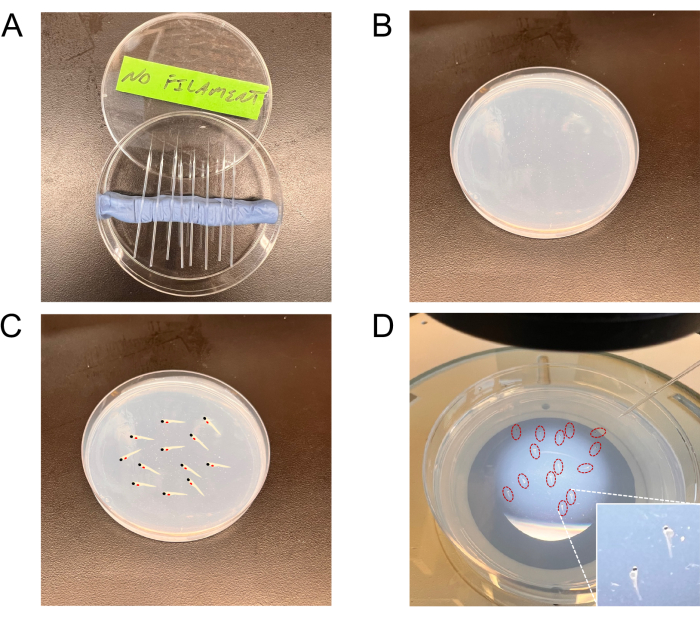
Abbildung 2: Repräsentative Bilder der Werkzeuge, die für die Injektionen verwendet wurden. (A) Gezogene Nadeln in einer Petriplatte. (B) Die Agaroseplatte für das Embryo-Staging. (C,D) Eine Tafel mit Embryonen, die für die Injektion auf der Embryoladeplatte bereitgestellt wurden (repräsentatives Diagramm in Feld C und echte Embryonen (rot eingekreist) in Feld D). Der Einsatz in der unteren rechten Ecke von Feld D zeigt eine Ansicht der inszenierten Embryonen mit höherer Vergrößerung. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
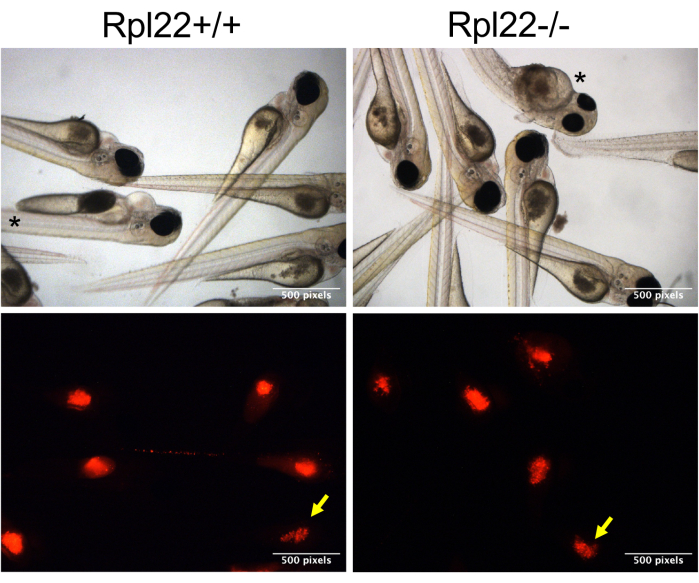
Abbildung 3: Repräsentative Bilder von xenotransplantierten Embryonen. Es werden Hellfeld- und Immunfluoreszenzbilder von CM-Dil-gefärbten (rot)-positiven Zellen im Eigelb der Casper-Embryonen bei 1 dpi gezeigt (Clutch-Bild). Embryonen mit einem minderwertigen Bolus sind mit einem gelben Pfeil gekennzeichnet, während Embryonen mit gestörter Morphologie mit einem Sternchen gekennzeichnet sind. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
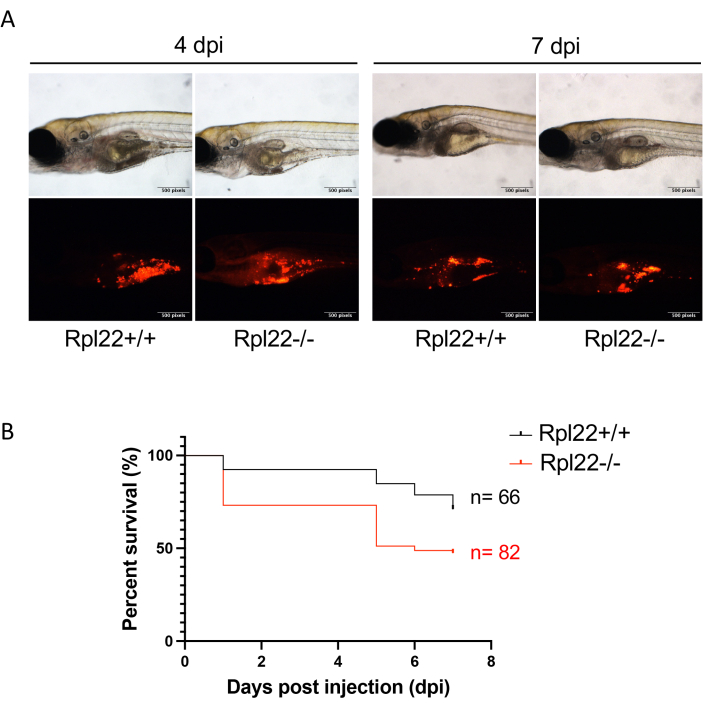
Abbildung 4: Beurteilung des Krankheitsverlaufs durch Fluoreszenzbildgebung und Überlebensanalyse. (A) Repräsentatives Bild von xenotransplantierten Embryonen bei 4 dpi und 7 dpi. (B) Das Kaplan-Meier-Diagramm, das die Überlebensanalyse von Embryonen mit zwei genetisch unterschiedlichen Leukämielinien zeigt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
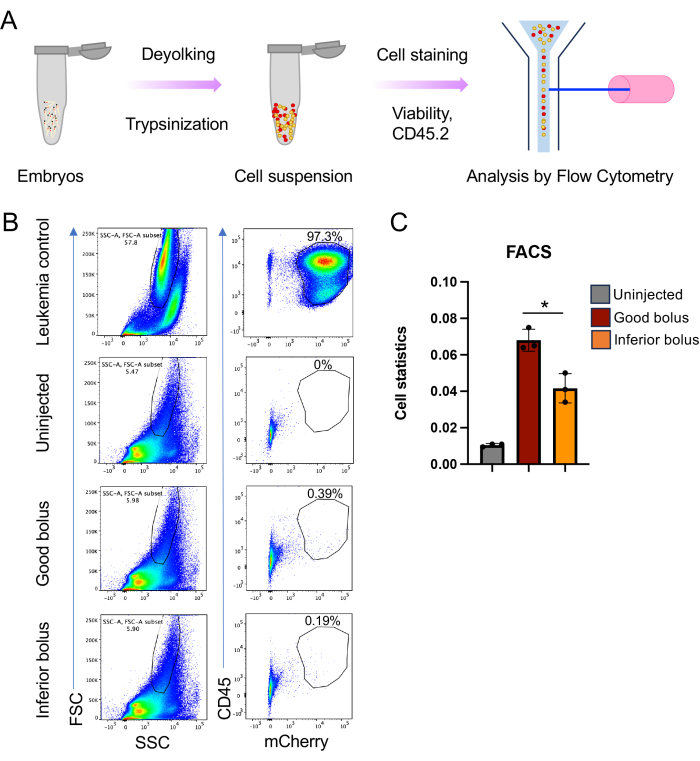
Abbildung 5: Durchflusszytometrische Analyse der Krankheitslast bei xenotransplantierten Zebrafischen. (A) Schematische Darstellung der Vorbereitung der Zellsuspension und der Durchflusszytometrie-Analyse. Kurz gesagt, Embryonen mit 4 dpi werden mit Trypsin und Kollagenase in Einzelzellsuspensionen zerlegt, gefolgt von einer Durchflusszytometrie. (B) Repräsentative Diagramme für die Durchflusszytometrie-Analyse, wobei das linke Bild in jedem Panel FSC-A vs. SSC-A-Diagramm und das rechte Bild CD45 v/s mCherry-Signale darstellt. (C) Balkendiagramm mit den Zellstatistiken für xenotransplantierte Zellen, wie sie aus dem CD45 v/s mCherry-Diagramm für nicht injizierte, gute und minderwertige Embryonen erhalten wurden (n = 45, 40 und 40 für jedes Replikat (n = 3); p-Wert * ≤ 0,05, berechnet unter Verwendung des ungepaarten t-Tests mit Welch-Korrektur in GraphPad Prism 9). Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Diskussion
Die Zebrafisch-Xenotransplantation hat sich als schnelle, robuste und kostengünstige Alternative zu Mausstudien erwiesen12. Obwohl über mehrere Ansätze zur Zebrafisch-Xenotransplantation berichtet wurde, hat unsere Anpassung zu signifikanten Verbesserungen geführt. Neben der Standardisierung der Parameter rund um das Verfahren konzentrieren sich diese Verbesserungen insbesondere auf die Beschleunigung der Geschwindigkeit, mit der Tumorinjektionen durchgeführt werden können, um so eine Erhöhung der Anzahl der Tiere pro Studienarm zu ermöglichen und die Tumormarkierung zur Überwachung der Qualität der Injektion und des Verhaltens nach der Injektion zu nutzen.
Während die hier beschriebenen Verbesserungen dieser Methode ein großes Potenzial haben, erfordert die erfolgreiche Umsetzung dieser Strategie einen erfahrenen Praktiker und eine Optimierung für die jeweilige Anwendung. Wir setzten Leukämiezellen ein. Folglich kann der Einsatz solider Krebsarten zusätzliche Herausforderungen mit sich bringen. Solche Tumoren können anfällig für Aggregation sein, was zu einer Variabilität bei der Abgabe des Zellbolus führen würde. aber auch unter solchen Umständen sollte die RFP-Kennzeichnung eine angemessene Qualitätskontrolle des Bolus nach der Injektion ermöglichen. Dies ist der GFP-Markierung oder grünen Farbstoffen überlegen, die durch Autofluoreszenz verdeckt werden. Schließlich minimiert die hier beschriebene Standardisierung der meisten Parameter, die den Erfolg beeinflussen (Gesundheit des Embryos, Aquakulturtemperatur, Nadelöffnung, Injektionsdruck usw.), die Variabilität dieses Prozesses.
Ein wichtiger Aspekt bei Xenotransplantationsexperimenten im Zebrafisch ist die Injektionsstelle. Hier haben wir gezeigt, dass die Injektionen in das Eigelb im Vergleich zu anderen technisch anspruchsvolleren Stellen, wie dem Perivitelinraum34, dem Cuviergang35 und der intrakardialen Injektion (Herzventrikel)36, recht einfach sind. Der Nachteil der Dotterinjektion besteht darin, dass es sich um ein lebenswichtiges Organ für die wachsenden Embryonen handelt, so dass darauf geachtet werden muss, dass der Nadeldurchmesser und der Druck sorgfältig kontrolliert werden, damit der Embryo nicht aufgrund eines Injektionstraumas stirbt. Der hier beschriebene Ansatz mildert diese Bedenken, indem er Verletzungen minimiert und offensichtliche Verletzungen oder Todesfälle um 1 dpi vernachlässigt, da diese Probleme nicht mit dem Tumorwachstum zusammenhängen. Die letzte Überlegung bezüglich der Injektionsstelle ist, dass unterschiedliche Mikroumgebungen eine mehr oder weniger große Fähigkeit haben können, die Vermehrung von xenotransplantierten Tumoren zu unterstützen. Folglich können vielleicht zuerst Dotterinjektionen durchgeführt werden, bevor zu anspruchsvolleren orthotopen Injektionen übergegangen wird. Der große Vorteil der Dotterinjektion besteht darin, dass sie kein präzises Embryonen-Staging erfordert und so eine schnellere Injektion einer größeren Anzahl von Embryonen ermöglicht, wodurch ihre Gesundheit besser erhalten und die statistische Aussagekraft zur Auflösung von Unterschieden im Verhalten transplantierter Tumorzellen erhöht wird.
Die Überwachung des Krankheitsverlaufs nach der Injektion wird in der Regel anhand der Auswirkungen auf das Überleben unter Verwendung der Kaplan-Meier-Analyse bewertet37; Tests zur Krankheitslast können jedoch auch sehr informativ sein. Bei transplantierten Zellen, die an der Injektionsstelle verbleiben, kann die Tumorlast mit verschiedenen Mikroskopiemethoden quantifiziert werden, sofern die Markierungsmethode für die Tumorzellen nicht durch Autofluoreszenz verdeckt wird29. Die CM-Dil-Färbung lässt sich leicht auflösen und wird von der Autofluoreszenz nicht beeinflusst, so dass sie sich gut zur Quantifizierung der Tumorlast lokalisierter Zellen eignet. Die Herausforderung tritt auf, wenn Tumorzellen nicht an der Injektionsstelle verbleiben und sich ausbreiten. In solchen Fällen ist die Durchflusszytometrie in Verbindung mit der unauslöschlichen genetischen Markierung mit rot fluoreszierenden Proteinen eine sehr effektive Methode zur Überwachung der Krankheitslast in standardisierten Gelege von Embryonen, da der markierte Tumor mit speziesspezifischen Färbungen analysiert werden kann, die sich von den Zebrafischzellen unterscheiden. Ein Manko von CM-Dil ist, dass es durch Zellteilungverdünnt wird 38. Dementsprechend bringt die Anpassung durch genetische Markierung der Tumoren mittels RFP oder mCherry erhebliche Vorteile mit sich. Die mCherry-Expression, gekoppelt mit einem tumorspezifischen Antikörper, ermöglicht die sichere Identifizierung transplantierter Zellen unter einem möglicherweise komplexen Muster von Hintergrundsignalen, die von den Zebrafischzellen des Wirts bereitgestellt werden.
Zusammengenommen stellen der optimierte Zebrafisch-Xenotransplantationsansatz und die Analysemethode, die in dieser Studie verwendet wurden, eine erhebliche Verbesserung einer bereits leistungsfähigen experimentellen Plattform dar.
Offenlegungen
Die Autoren erklären, dass keine konkurrierenden Interessen bestehen.
Danksagungen
Diese Arbeit wurde durch NIH-Zuschüsse R37AI110985 und P30CA006927, eine Zuwendung des Commonwealth of Pennsylvania, der Leukemia and Lymphoma Society und des Bishop Fund unterstützt. Diese Studie wurde auch von den Kerneinrichtungen von Fox Chase unterstützt, einschließlich der Zellkultur, der Durchflusszytometrie und der Labortiereinrichtung. Wir danken Dr. Jennifer Rhodes für die Instandhaltung der Zebrafisch- und Mikroinjektionsanlage am FCCC.
Materialien
| Name | Company | Catalog Number | Comments |
| 1-phenyl 2-thiourea (PTU) | Sigma | P7629 | |
| 70 micron cell strainer | Corning | CLS431751-50EA | |
| 90 mm Petri dish | Thermo Fisher Scientific | S43565 | |
| Agarose | Apex bioresearch | 20-102GP | |
| APC APC anti-mouse CD45.2 Antibody | Biolegend | 109814 | |
| BD FACSymphony A5 Cell Analyzer | BD Biosciences | BD FACSymphony A5 | |
| calibration capillaries | Sigma | P1424-1PAK | |
| Cell tracker CM-dil dye | Invitrogen | C7001 | |
| Collageanse IV | Gibco | 17104019 | |
| Dumont forceps number 55 | Fine science tools | 11255-20 | |
| FBS | Corning | 35-015-CV | |
| Fluorescence microscope | Nikon | model SMZ1500 | |
| Glass capillaries (Borosilicate) | World precision instruments | 1B100-4 | |
| HBSS | Corning | 21-023-CV | |
| Helix NP Blue | Biolegend | 425305 | |
| Instant Ocean Sea Salt | Instant ocean | SS15-10 | |
| Light microscope | Nikon | model SMZ1000 | |
| Methylene blue | Sigma | M9140-100G | |
| Microloader (long tips for laoding cells) | eppendorf | 930001007 | |
| P1000 micropipette puller | Sutter instruments | model P-97 | |
| PM 1000 cell microinjector | MicroData Instruments, Inc. (MDI) | PM1000 | |
| Tricaine methanesulphate (Ethyl 3- aminobenzoate methanesulphate) | Sigma | E10521-10G | |
| Trypsin-EDTA (0.5%), no phenol red | Gibco | 15400054 | |
| Zebrafish adult irradiated diet (dry feed) | Zeigler | 388763 |
Referenzen
- Sharma, G., Goyal, Y., Bhatia, S. Handbook of Animal Models and its Uses in Cancer Research. Preclinical Animal Models of Cancer: Applications and Limitations. , (2022).
- Singhal, S. S., et al. Recent advancement in breast cancer research: Insights from model organisms-Mouse models to zebrafish. Cancers. 15 (11), 2961 (2023).
- Liu, Y., et al. Patient-derived xenograft models in cancer therapy: technologies and applications. Signal Transduction and Targeted Therapy. 8 (1), 160 (2023).
- Fuochi, S., Galligioni, V. Disease Animal Models for Cancer Research. Cancer Cell Culture: Methods and Protocols. , (2023).
- Shaw, T. J., Senterman, M. K., Dawson, K., Crane, C. A., Vanderhyden, B. C. Characterization of intraperitoneal, orthotopic, and metastatic xenograft models of human ovarian cancer. Mol Ther. 10 (6), 1032-1042 (2004).
- Deroose, C. M., et al. Multimodality imaging of tumor xenografts and metastases in mice with combined small-animal PET, small-animal CT, and bioluminescence imaging. J Nucl Med. 48 (2), 295-303 (2007).
- Zeng, M., et al. Generation, evolution, interfering factors, applications, and challenges of patient-derived xenograft models in immunodeficient mice. Cancer Cell Int. 23 (1), 120 (2023).
- Adhish, M., Manjubala, I. Effectiveness of zebrafish models in understanding human diseases-A review of models. Heliyon. 9 (3), e14557 (2023).
- MacRae, C. A., Peterson, R. T. Zebrafish as a mainstream model for in vivo systems pharmacology and toxicology. Ann Rev Pharmacol Toxicol. 63, 43-64 (2023).
- Choe, S. -. K., Kim, C. -. H. Zebrafish: A powerful model for genetics and genomics. Int J Mol Sci. 24 (9), 8169 (2023).
- White, R., Rose, K., Zon, L. Zebrafish cancer: the state of the art and the path forward. Nat Rev Cancer. 13 (9), 624-636 (2013).
- Al-Hamaly, M. A., Turner, L. T., Rivera-Martinez, A., Rodriguez, A., Blackburn, J. S. Zebrafish cancer avatars: A translational platform for analyzing tumor heterogeneity and predicting patient outcomes. Int J Mol Sci. 24 (3), 2288 (2023).
- White, R. M., et al. Transparent adult zebrafish as a tool for in vivo transplantation analysis. Cell Stem Cell. 2 (2), 183-189 (2008).
- Hill, D., Chen, L., Snaar-Jagalska, E., Chaudhry, B. Embryonic zebrafish xenograft assay of human cancer metastasis. F1000Res. 7, 1682 (2018).
- Corkery, D. P., Dellaire, G., Berman, J. N. Leukaemia xenotransplantation in zebrafish--chemotherapy response assay in vivo. Br J Haematol. 153 (6), 786-789 (2011).
- Lin, J., et al. A clinically relevant in vivo zebrafish model of human multiple myeloma to study preclinical therapeutic efficacy. Blood. 128 (2), 249-252 (2016).
- Grissenberger, S., et al. High-content drug screening in zebrafish xenografts reveals high efficacy of dual MCL-1/BCL-XL inhibition against Ewing sarcoma. Cancer Lett. 554, 216028 (2023).
- Baxi, D. Zebrafish: A Versatile Animal Model to Study Tumorigenesis Process and Effective Preclinical Drug Screening for Human Cancer Research. Handbook of Animal Models and its Uses in Cancer Research. , (2022).
- Li, X., Li, M. The application of zebrafish patient-derived xenograft tumor models in the development of antitumor agents. Med Res Rev. 43 (1), 212-236 (2023).
- Yin, J., et al. Zebrafish patient-derived xenograft model as a preclinical platform for uveal melanoma drug discovery. Pharmaceuticals. 16 (4), 598 (2023).
- Nakayama, J., Makinoshima, H., Gong, Z. In vivo drug screening to identify anti-metastatic drugs in Twist1a-ER(T2) transgenic zebrafish. Bio Protoc. 13 (10), e4673-e4673 (2023).
- Lam, S., Chua, H., Gong, Z., Lam, T., Sin, Y. Development and maturation of the immune system in zebrafish, Danio rerio: a gene expression profiling, in situ hybridization and immunological study. Dev Comp Immunol. 28 (1), 9-28 (2004).
- Nicoli, S., Presta, M. The zebrafish/tumor xenograft angiogenesis assay. Nat Protoc. 2 (11), 2918-2923 (2007).
- Casey, M. J., et al. Transplantation of zebrafish pediatric brain tumors into immune-competent hosts for long-term study of tumor cell behavior and drug response. J Vis Exp. (123), e55712 (2017).
- Soh, G. H., Kögler, A. C., Müller, P. A simple and effective transplantation device for zebrafish embryos. J Vis Exp. (174), e62767 (2021).
- Martinez-Lopez, M., Póvoa, V., Fior, R. Generation of zebrafish larval xenografts and tumor behavior analysis. J Vis Exp. (172), e62373 (2021).
- Ren, J., Liu, S., Cui, C., Ten Dijke, P. Invasive behavior of human breast cancer cells in embryonic zebrafish. J Vis Exp. (122), e55459 (2017).
- Zhao, C., et al. A novel xenograft model in zebrafish for high-resolution investigating dynamics of neovascularization in tumors. PloS One. 6 (7), e21768 (2011).
- Cabezas-Sáinz, P., Pensado-López, A., Sáinz Jr, B., Sánchez, L. Modeling cancer using zebrafish xenografts: drawbacks for mimicking the human microenvironment. Cells. 9 (9), 1978 (2020).
- Haraoka, Y., Akieda, Y., Ishitani, T. Live-imaging analyses using small fish models reveal new mechanisms that regulate primary tumorigenesis. Yakugaku Zasshi. 139 (5), 733-741 (2019).
- Westerfield, M. . The Zebrafish Book. A Guide for the Laboratory Use of Zebrafish (Danio rerio). , (2000).
- Rao, S., et al. Inactivation of ribosomal protein L22 promotes transformation by induction of the stemness factor, Lin28B. Blood. 120 (18), 3764-3773 (2012).
- Goel, M. K., Khanna, P., Kishore, J. Understanding survival analysis: Kaplan-Meier estimate. Int J Ayurveda Res. 1 (4), 274-278 (2010).
- Usai, A., Di Franco, G., Gabellini, C., Morelli, L., Raffa, V. Establishment of zebrafish patient-derived xenografts from pancreatic cancer for chemosensitivity testing. J Vis Exp. (195), e63744 (2023).
- Murali Shankar, N., et al. Preclinical assessment of CAR-NK cell-mediated killing efficacy and pharmacokinetics in a rapid zebrafish xenograft model of metastatic breast cancer. Front Immunol. 14, 1254821 (2023).
- Takahi, M., et al. Xenograft of human pluripotent stem cell-derived cardiac lineage cells on zebrafish embryo heart. Biochem Biophys Res Commun. 674, 190-198 (2023).
- Rudner, L. A., et al. Shared acquired genomic changes in zebrafish and human T-ALL. Oncogene. 30 (41), 4289-4296 (2011).
- Regan, J. L., et al. RNA sequencing of long-term label-retaining colon cancer stem cells identifies novel regulators of quiescence. iScience. 24 (6), 102618 (2021).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten