Method Article
In Planta Genexpression und Gen-Editierung in Moso-Bambusblättern
In diesem Artikel
Zusammenfassung
In dieser Studie wurde eine neuartige Genexpressions- und Geneditierungsmethode, die durch Agrobacterium vermittelt wird, in Bambus entwickelt. Diese Methode verbesserte die Effizienz der Genfunktionsvalidierung bei Bambus erheblich, was erhebliche Auswirkungen auf die Beschleunigung des Prozesses der Bambuszüchtung hat.
Zusammenfassung
Für Bambus wurde eine neuartige In-Planta-Gentransformationsmethode entwickelt, die zeit- und arbeitsintensive Kallusinduktions- und Regenerationsprozesse vermeidet. Diese Methode beinhaltet die Agrobacterium-vermittelte Genexpression durch Verwundung und Vakuum für Bambussetzlinge. Es wurde erfolgreich die Expression von exogenen Genen wie dem RUBY-Reporter und dem Cas9-Gen in Bambusblättern nachgewiesen. Die höchste Transformationseffizienz für die Akkumulation von Betalain in RUBY-Sämlingen wurde mit dem Stamm GV3101 mit einem Prozentsatz von 85,2 % nach der Infektion erreicht. Obwohl sich die fremde DNA nicht in das Bambusgenom integrierte, war die Methode effizient bei der Expression der exogenen Gene. Darüber hinaus wurde mit dieser Methode auch ein Gen-Editing-System mit einem nativen Reporter entwickelt, aus dem eine durch das editierte Bambus-Violaxanthin-De-Epoxidase-Gen (PeVDE) erzeugte in Bambusblättern eine In-situ-Mutante mit einer Mutationsrate von 17,33 % erzeugte. Die Mutation von PeVDE führte zu verringerten nicht-photochemischen Quenching-Werten (NPQ) unter starkem Licht, die mit einem Fluorometer genau nachgewiesen werden können. Dies macht den editierten PeVDE zu einem potentiellen nativen Reporter sowohl für exogene als auch für endogene Gene in Bambus. Mit dem Reporter von PeVDE wurde ein Cinnamoyl-CoA-Reduktase-Gen mit einer Mutationsrate von 8,3% erfolgreich editiert. Durch diese Operation wird der Prozess der Gewebekultur oder der Kallusinduktion vermieden, was für die Expression exogener Gene und die endogene Geneditierung in Bambus schnell und effizient ist. Diese Methode kann die Effizienz der Überprüfung der Genfunktion verbessern und dazu beitragen, die molekularen Mechanismen wichtiger Stoffwechselwege in Bambus aufzudecken.
Einleitung
Die Untersuchung der Genfunktion in Bambus ist vielversprechend für das fortgeschrittene Verständnis von Bambus und die Erschließung seines Potenzials für genetische Modifikationen. Ein effektiver Weg hierfür kann durch den Prozess der Agrobacterium-vermittelten Infektion in Bambusblättern erreicht werden, bei dem das T-DNA-Fragment, das exogene Gene enthält, in die Zellen eingebracht wird, was anschließend zur Expression der Gene in den Blattzellen führt.
Bambus ist ein wertvoller und nachwachsender Rohstoff mit einem breiten Anwendungsspektrum in Fertigung, Kunst und Forschung. Bambus besitzt hervorragende Holzeigenschaften wie hohe mechanische Festigkeit, Zähigkeit, mäßige Steifigkeit und Flexibilität1, die heute in einer Vielzahl von Haushalts- und Industrieartikeln weit verbreitet sind, darunter Zahnbürsten, Strohhalme, Knöpfe, Einweggeschirr, unterirdische Rohrleitungen und Kühlturmfüller für die thermische Stromerzeugung. Daher spielt die Bambuszüchtung eine entscheidende Rolle bei der Gewinnung von Bambussorten mit hervorragenden Holzeigenschaften, um Kunststoffe zu ersetzen und den Kunststoffverbrauch zu reduzieren, die Umwelt zu schützen und den Klimawandel zu bekämpfen sowie einen erheblichen wirtschaftlichen Wert zu generieren.
Die traditionelle Bambuszüchtung steht jedoch aufgrund der langen vegetativen Wachstumsphase und der unsicheren Blütezeit vor Herausforderungen. Obwohl molekulare Züchtungstechniken entwickelt und auf die Bambuszüchtung angewendet wurden, ist der Prozess der Bambus-Gentransformation aufgrund der Kallusinduktions- und Regenerationsprozesse zeitaufwändig, arbeitsintensiv und kompliziert 2,3,4,5. Eine stabile genetische Transformation erfordert oft Agrobacterium-vermittelte Methoden, die Gewebekulturprozesse wie Kallusinduktion und Regeneration beinhalten. Bambus hat jedoch eine geringe Fähigkeit zur Kallusregeneration, was die Anwendung einer stabilen genetischen Transformation in Bambus stark einschränkt. Nachdem Agrobacterium Pflanzenzellen infiziert hat, dringt das T-DNA-Fragment in die Pflanzenzellen ein, wobei die Mehrheit der T-DNA-Fragmente nicht in die Zellen integriert bleibt, was zu einer vorübergehenden Expression führt. Nur ein kleiner Teil der T-DNA-Fragmente integriert sich zufällig in sein Chromosom, was zu einer stabilen Expression führt. Die transienten Expressionsniveaus zeigen eine Akkumulationskurve, die für jedes Gen, das aus einer von Agrobacterium gelieferten T-DNA exprimiert wird, variieren kann. In den meisten Fällen treten die höchsten Expressionsniveaus 3-4 Tage nach der Infiltration auf und nehmen nach 5-6 Tagen schnell ab 6,7. Frühere Studien haben gezeigt, dass mehr als 1/3 der Mutationen in geneditierten Pflanzen, die ohne Selektionsdruck für Resistenz erhalten wurden, auf die vorübergehende Expression von CRISPR/Cas9 zurückzuführen sind, während die restlichen weniger als 2/3 auf eine stabile Expression nach der DNA-Integration in das Genom zurückzuführensind 8. Dies deutet darauf hin, dass die Integration von T-DNA in das Pflanzengenom für die Genomierung nicht notwendig ist. Darüber hinaus hemmt der Selektionsdruck für Resistenzen das Wachstum nicht-transgener Zellen signifikant, was sich direkt auf den Regenerationsprozess infizierter Explantate auswirkt. Daher ist es möglich, durch die Verwendung einer transienten Expression ohne Selektionsdruck für Resistenz in Bambus eine nicht-integrierte Expression exogener Gene zu erreichen und die Genfunktion direkt in pflanzlichen Organen zu untersuchen. Somit kann eine einfache und zeitsparende Methode zur exogenen Genexpression und -editierung in Bambus9 entwickelt werden.
Die entwickelte exogene Genexpressions- und Geneditierungsmethode zeichnet sich durch ihre Einfachheit, Kosteneffizienz und den Verzicht auf teure Geräte oder komplexe Verfahren aus9. Bei dieser Methode wurde das endogene Violaxanthin-De-Epoxidase-Gen (PeVDE) aus Bambus als Reporter für die exogene Genexpression ohne Selektionsdruck verwendet. Dies liegt daran, dass das editierte PeVDE in Bambusblättern die Lichtschutzfähigkeit bei starkem Licht reduziert und eine Abnahme des nicht-photochemischen Quenching-Wertes (NPQ) zeigt, der durch Chlorophyll-Fluoreszenz-Bildgebung nachgewiesen werden kann. Um die Wirksamkeit dieser Methode zu demonstrieren, wurde ein weiteres endogenes Bambusgen, das Cinnamoyl-CoA-Reduktase-Gen (PeCCR5)9, mit diesem System ausgeschaltet und erfolgreich Mutanten dieses Gens erzeugt. Diese Technik kann für die funktionelle Charakterisierung von Genen verwendet werden, die Funktionen in Bambusblättern haben. Durch die vorübergehende Überexpression dieser Gene in Bambusblättern können ihre Expressionsniveaus erhöht werden, oder durch Gen-Editierung kann ihre Expression reduziert werden, was die Untersuchung von nachgeschalteten Genexpressionsniveaus, Blattphänotypen und Produktinhalten ermöglicht. Dies bietet einen effizienteren und praktikableren Ansatz für die Erforschung von Genfunktionen in Bambus. Diese Technik kann auf die funktionelle Charakterisierung von Genen angewendet werden, die in Bambusblättern funktionieren. Durch die vorübergehende Überexpression dieser Gene in Bambusblättern können ihre Expressionsniveaus erhöht werden, oder durch Gen-Editierung kann ihre Expression reduziert werden, was die Untersuchung von nachgeschalteten Genexpressionsniveaus, Blattphänotypen und Produktinhalten ermöglicht. Darüber hinaus ist es wichtig zu beachten, dass aufgrund der umfangreichen Polyploidisierung die Mehrheit der kommerziell wichtigen Gene in Bambusgenomen in mehreren Kopien vorhanden ist, was zu genetischer Redundanz führt. Dies stellt eine Herausforderung für die Durchführung der Multiplex-Genom-Editierung in Bambus dar. Vor der Anwendung stabiler genetischer Transformations- oder Geneditierungstechniken ist es entscheidend, Genfunktionen schnell zu validieren. Bei der Lösung des Problems der Mehrfachkopien besteht ein Ansatz darin, Transkriptomexpressionsprofile zu analysieren, um Gene zu identifizieren, die in bestimmten Stadien aktiv exprimiert werden. Darüber hinaus ermöglicht das Targeting der konservierten funktionellen Domänen dieser Genkopien das Design gemeinsamer Zielsequenzen oder den Einbau mehrerer Zielstellen in denselben CRISPR/Cas9-Vektor, was den gleichzeitigen Knockout dieser Gene ermöglicht. Dies bietet einen effizienteren und praktikableren Ansatz für die Erforschung von Genfunktionen in Bambus.
Protokoll
1. Vorbereitung von Bambussetzlingen
- Bereiten Sie Moso-Bambus-Sämlinge (Phyllostachys edulis) mit Samen vor, die in Guilin, Guangxi, China, geerntet wurden. Beginne damit, die Samen 2-3 Tage lang in Wasser einzuweichen, wobei du darauf achten solltest, das Wasser täglich zu wechseln. Als nächstes erstellst Du ein Substrat, indem Du Erde und Vermiculit in einem Verhältnis von 3:1 mischst.
- Säen Sie die eingeweichten Samen zur Keimung in das Substrat. Halten Sie die Sämlinge unter Laborbedingungen und halten Sie die Temperatur zwischen 18-25 °C. Sorgen Sie für eine 16 h helle/8 h dunkle Photoperiode mit einer Lichtintensität von 250-350 μmol/m2/s während der Lichtphase.
- Halten Sie eine relative Luftfeuchtigkeit von ca. 60 % aufrecht. Für eine Agrobacterium-Infektion verwenden Sie Sämlinge, die 15 Tage alt sind und eine Höhe von 2-10 cm haben, was das beste Stadium für die Transformation ist.
HINWEIS: Da die Bambusblüte unvorhersehbar ist, sind Samen nicht jedes Jahr verfügbar. Die Samen werden in der Regel 2-3 Jahre bei 4 °C in einer trockenen Umgebung gelagert und können noch eine Lebensfähigkeit von über 20% aufrechterhalten.
2. Herstellung von Plasmiden und Agrobacterium
- Plasmide: Um den transienten Expressionseffekt zu validieren, wird das pHDE-35S::RUBY-Konstrukt verwendet, das ein sichtbares Reportergen enthält, das vom CaMV 35S-Promotor 10 gesteuert wird. Für die Gen-Editierung wird das pCambia1300-Ubi::Cas9-Konstrukt verwendet, das das Cas9-Gen trägt, das vom Mais-Ubi-Promotor 11 gesteuert wird. Fügen Sie die sgRNA-Leitsequenzen von PeVDE und anderen Zielgenen zwischen den beiden AarI-Stellen in das pCambia1300-Ubi::Cas9-Konstrukt 9 ein.
- Fügen Sie die CRISPR/Cas9-Guide-RNA-Sequenzen zwischen die beiden AarI-Restriktionsendonuklease-Stellen des pCambia1300-Ubi::Cas9-Konstrukts ein, einschließlich der PeVDE - und PeCCR5-Zielgene .
- Fügen Sie GGCA zum 5'-Ende der 20-Nukleotid-Sequenz hinzu und synthetisieren Sie eine einzelsträngige DNA-Sequenz. Komplementieren Sie die 20-Nukleotid-Sequenz umgekehrt und fügen Sie AAAC zum 5'-Ende hinzu, dann synthetisieren Sie eine weitere einzelsträngige DNA-Sequenz.
- Beide einzelsträngigen DNA-Sequenzen werden in verdünntem Wasser auf eine Konzentration von 10 nM/L verdünnt, gründlich gemischt und bei 95 °C 5 min erhitzt. Lassen Sie die Mischung auf Raumtemperatur abkühlen, wodurch sich doppelsträngige Adapter bilden.
- Verbinden Sie die Adapter mit dem linearen pCambia1300-Ubi::Cas9-Fragment, das von der Aar I-Endonukleaseunter Verwendung der T4-DNA-Ligase verdaut wird, und sequenzieren Sie den konstruierten CRISPR/Cas9-Vektor, um das gewünschte Gen-Targeting-Konstrukt zu erhalten.
- Um Plasmide in Agrobacterium umzuwandeln, verwenden Sie die Gefrier-Auftau-Methode wie folgt: Mischen Sie 1 μL der Plasmide (Konzentration: 10 - 1.000 ng/μL) mit 100 μL Agrobacterium-kompetenten Zellen (Translationseffizienz: > 1 x 10 4 koloniebildende Einheiten/μg) und mischen Sie sie vorsichtig. Die Mischung für 5 Minuten auf Eis legen. Die Mischung für 5 Minuten in flüssigen Stickstoff überführen.
- Die Mischung im 37 °C warmen Wasserbad 5 min auftauen. Geben Sie 500 μl Luria-Bertani (LB)-Medium in die Mischung und inkubieren Sie es in einem Schüttelinkubator bei 28 °C bei 200 U/min für 2-3 h. Die pHDE-35S::RUBY-Plasmide werden einzeln in die AGL1-, GV3101-, LBA4044- und EHA105-Stämme von Agrobacterium tumefaciens (A. tumefaciens) eingebracht12. Die CRISPR/Cas9-Plasmide werden in den GV3101-Stamm von A. tumefaciens eingeführt.
- Agrobacterium wird in Hefeextrakt Pepton (YEP)-Medium (10 g Rindfleischextrakt, 10 g Hefeextrakt und 5 g NaCl pro L) mit den entsprechenden Antibiotika (Spectinomycin für 35S::RUBY und Kanamycin für CRISPR/Cas9) bei 28 °C gezüchtet. Die einzelnen Kolonien wurden 36-48 h nach der Kultivierung beobachtet.
- Pflücken Sie die Kolonien und geben Sie sie in flüssiges YEP-Medium (mit entsprechenden Antibiotika) für das weitere Wachstum um. Verwenden Sie nach 24-36 Stunden die Primer von RUBY-F und RUBY-R (Tabelle 1), um eine PCR durchzuführen, um den erfolgreichen Transfer von Plasmiden in Agrobacterium zu bestätigen.
- 1 ml des erfolgreich transformierten Agrobacteriums in 100 ml frisches flüssiges YEP-Medium (mit entsprechenden Antibiotika) überführen und dann über Nacht bei 28 °C auf einen OD600 von 0,8 wachsen lassen.
- Die Bakteriensuspension wird bei 4.000 x g für 5 min bei 4 °C zentrifugiert, das Bakterienpellet einmal mit dem Infiltrationsmedium der Suspensionen (10 mM MgCl2 und 10 mM MES-KOH [PH 5,6]) gespült und anschließend erneut zentrifugiert. Resuspendieren Sie das Bakterienpellet in einem Suspensionsinfiltrationsmedium auf einen OD600 von 0,6 für die Bambusumwandlung.
3. Agrobacterium-vermittelt im Planta-Transformationssystem
- Bereiten Sie sich auf die Transformation vor; Entferne die Sämlinge mit erdgebundenen Wurzeln vorsichtig aus dem Substrat und achte darauf, dass die Wurzeln intakt bleiben. Wickeln Sie die Sämlinge mit Alufolie ein, um die Feuchtigkeit zu erhalten und eine Ablösung der Erde zu verhindern (Abbildung 1A).
- Bringen Sie die eingewickelten Sämlinge für eine Dauer von 2 Stunden in eine Umgebung mit höherer Luftfeuchtigkeit (relative Luftfeuchtigkeit >90%) und geringerer Beleuchtung (Intensität weniger als 50 μmol/m2/s).
- Wickeln Sie mit einer scharfen Nadel aus einer Spritze den oberen Teil der gerollten, unreifen Blätter (ca. 1-2 cm von der Spitze) der Bambussämlinge ein- oder zweimal auf (wie durch die roten Dreiecke in Abbildung 1A angedeutet).
- Tauchen Sie anschließend den verwundeten oberen Teil der Sämlinge in Suspensionen von Agrobacterium. Führen Sie den gesamten Prozess, von der Wunde bis zum Eintauchen in Agrobacterium , schnell durch, da die frische Wunde die Impfeffizienz erhöht.
- Setzen Sie die Sämlinge sofort für 2 Minuten in eine Vakuumkammer mit einem Druck von 25-27 mmHg um (Abbildung 1B).
- Nach dem Vakuumieren packen Sie die Sämlinge vorsichtig aus und pflanzen sie wieder in das Substrat ein. Stellen Sie die Sämlinge 2 Tage lang in schwaches Licht oder Dunkelheit (<50 μmol/m2/s) mit hoher Luftfeuchtigkeit (RH>90%) bei Raumtemperatur (18-25 °C). Anschließend kultivieren Sie die Sämlinge unter normalen Wachstumsbedingungen und gießen Sie sie alle 5-7 Tage. Beobachte ihre Phänotypen, um Material für nachfolgende Experimente zu liefern.
4. Design von Single Guide RNAs (sgRNAs) für die Geneditierung
- Identifizierung einer Protospacer-Nachbarmotiv-Stelle (PAM), die sich in der Nähe einer spezifischen und konservierten Domäne der Zielgensequenz befindet. Stellen Sie sicher, dass die spezifische PAM-Sequenz in diesem CRISPR/Cas9-System NGG ist. Überprüfen Sie die On-Target-Spezifität der ausgewählten Sequenz, indem Sie eine BlastN-Suche in der Bambus-Genomdatenbank durchführen. Stellen Sie sicher, dass die sgRNA einzigartig ist, insbesondere im Upstream-Bereich und in der Nähe des PAM 9,11.
HINWEIS: Dieser Vergleich reduziert effektiv potenzielle Off-Target-Stellen im Genom, die von der Gen-Editierung betroffen sind. - Entwerfen Sie zwei sgRNAs (sgRNA-1 und sgRNA-2) auf dem ersten Exon des PeVDE-Gens 13. AgeI Restriktionsstellen stromaufwärts des PAM in sgRNA-1 und XbaI Restriktionsstellen stromaufwärts des PAM in sgRNA-2. Entwerfen Sie eine sgRNA auf dem vierten Exon des PeCCR5-Gens , das für das konservierte Motiv von KNWYCYGK kodiert. Dieses Motiv ist entscheidend für die Katalyse von CCRs14.
- Entwerfen Sie eine Spacer-Sequenz mit 20 Nukleotiden, die an die PAM-Stelle angrenzt. Diese Spacer-Sequenz wird das Cas9-Enzym an den Zielort für die DNA-Spaltung und die anschließende Gen-Editierung führen.
HINWEIS: Es ist ratsam, eine Zielregion vor der Endonuklease-Enzymspaltstelle innerhalb der PAM-Stelle zu wählen. Dies wird die Validierung der Effizienz der Geneditierung erleichtern.
5. Primer-Design und PCR
- Manuelles Entwerfen spezifischer Primer für die Amplifikation der PeVDE - und PeCCR5-Fragmente . Entwerfen Sie die vor- und nachgeschalteten Primer so, dass sie sich mindestens 100 bp außerhalb der Zielstelle befinden, mit einem Längenunterschied von über 100 bp, um eine deutliche Bandtrennung während der Elektrophorese zu ermöglichen. Entwerfen Sie Primer für die Amplifikation von RUBY innerhalb der ersten 500 bp des Gens. Eine Liste aller verwendeten Primer ist in Tabelle 1 enthalten.
- Verwenden Sie eine High-Fidelity-DNA-Polymerase für die PCR. In diesem Fall verwenden Sie die DNA-Polymerase mit hoher Genauigkeit und effizienter Amplifikation bei der Genklonierung.
- Bereiten Sie die PCR-Reaktionsmischung wie folgt vor: 5x Puffer (Mg2+ Plus): 4 μL; dNTP-Mischung (je 2,5 mM): 1,6 μl; Vorwärts- und Rückwärtsprimer (je 10 pmol): je 1 μl; Bambus-Genom-DNA (ca. 50 ng); DNA-Polymerase (2,5 U/μl): 0,2 μl; Verdünnen Sie das Wasser auf ein Gesamtvolumen von 20 μl.
- Befolgen Sie die PCR-Betriebsbedingungen: Anfängliche Denaturierung bei 98 °C für 5 min; Denaturierung bei 98 °C für 10 s; Glühen bei 56 °C für 5 s; Verlängerung bei 72 °C für 30 s; Wiederholen Sie die Denaturierung bis zur Verlängerung für 32 Zyklen; Enddehnung bei 72 °C für 5 min; Auf unbestimmte Zeit bei 4 °C halten.
HINWEIS: PCR-Bedingungen dienen als Beispiel und müssen möglicherweise für bestimmte Anwendungen oder Ziele optimiert werden.
6. DNA-Extraktion, Endonuklease-Enzymverdau und Sequenzierung
- Trennen Sie die Region mit niedrigeren NPQ-Werten von frischen Bambusblattblättern mit einer Schere, wie sie mit dem bildgebenden PAM-Fluorometer identifiziert wurde (siehe Schritt 7.4). Die Blattproben mit flüssigem Stickstoff einfrieren und die gefrorenen Blattproben in einen Mörser überführen. Mahlen Sie die Proben mit einem Stößel zu einem feinen Pulver und fügen Sie während des Mahlvorgangs genügend flüssigen Stickstoff hinzu. Dieser Schritt hilft bei der Freisetzung des zellulären Inhalts, einschließlich der DNA.
- Extrahieren Sie genomische DNA aus dem pulverisierten Blatt mit der Cetyltrimethylammonium-Ammoniumbromid-Methode (CTAB). 50 mg Blattpulverproben zu 800 μl einer 2%igen CTAB-Lösung geben. Die Probe wird gründlich gemischt und 30 Minuten lang bei 65 °C inkubiert, alle 5 Minuten leicht geschüttelt.
- Fügen Sie ein gleiches Volumen Chloroform/Isoamylalkohol (24:1, v/v) hinzu und schütteln Sie die Mischung kräftig. Nach der Zentrifugation bei 8.000 g für 8 min wird der Überstand in ein neues Röhrchen überführt.
- Man fügt ein gleiches Volumen Chloroform/Isoamylalkohol hinzu und wiederholt Schritt 6.3. Geben Sie ein gleiches Volumen eiskaltes Isopropanol hinzu und drehen Sie das Röhrchen mehrmals um, bevor Sie es 30 Minuten lang bei -20 °C platzieren. Nach Zentrifugation bei 8.000 x g für 5 min wird der Überstand verworfen.
- Waschen Sie das DNA-Pellet 2x mit 75%igem Ethanol bei 4 °C und lösen Sie es dann in 50 μl Wasser auf.
- Amplifizieren Sie die genomische DNA, die die Zielstelle der Zielgene enthält, sowohl aus Wildtyp- als auch aus Agrobacterium-infizierten Bambusblättern mit dem Protokoll in Schritt 5.4.
- Führen Sie den Enzymverdau der PCR-Produkte durch Endonuklease durch. Wählen Sie ein bestimmtes Enzym aus, das die gewünschten Restriktionsstellen innerhalb der amplifizierten DNA-Fragmente erkennt. Verwenden Sie AgeI und XbaI Endonukleasen für die Verdauung.
- Bereiten Sie eine Reaktionsmischung bestehend aus AgeI oder XbaI (20 Einheiten/μl)- 1 μl, 1 μg PCR-Produkte, 10x Puffer - 5 μl vor und fügen Sie Wasser zu einem Gesamtvolumen von 50 μl hinzu. 1 h bei 37 °C inkubieren.
- Analysieren Sie den Anteil der verdauten DNA-Fragmente mittels Gelelektrophorese. Vergleichen Sie die verdauten Fragmente aus Wildtyp- und Agrobacterium-infizierten Proben, um die Effizienz der Geneditierung zu beurteilen.
- Markieren Sie die Transposase-Adapter-Sequenzen TCGTCGGCAGCGTGTCAGATGTGTATAAGAGACAG und GTCTCGTGGGCTCGGAGATGTGTATAAGAGACAG am 5'-Ende der Vorwärts- bzw. Rückwärtsprimer für die Amplifikation von PeVDE- und PeCCR5-Fragmenten 9,15. Verwenden Sie das in Schritt 5.4 beschriebene Protokoll für die Verstärkung. Bereiten Sie die PCR-Produkte (vor der Verdauung) für die Tiefensequenzierung vor.
7. Messung der Chlorophyllfluoreszenz von NPQ-Werten in Blättern
- Setzen Sie die Bambussämlinge vor den Messungen 2 h lang hohen Lichtintensitäten von 1200 μmol/m2/s aus. Diese Belichtung erhöht die Menge des absorbierten Lichtquantums und aktiviert das Lichtschutzsystem in den Blättern.
- Verwenden Sie ein bildgebendes PAM-Fluorometer, um die in vivo PS II Chlorophyllfluoreszenz von Bambusblättern zu messen. Stellen Sie die aktinische Lichtintensität auf 800 μmol/m2/s für 6-minütige Fluoreszenzmessungen ein. Während dieser Zeit wird alle 30 s ein Sättigungsimpuls angewendet, um Chlorophyll-Fluoreszenzkurven zu erhalten. Die stabilen Werte der Kurven werden für Berechnungen13 verwendet.
- Berechnen Sie die nicht-photochemische Abschreckung (NPQ) mit der Formel:
NPQ = (F m - F m') / F m'
wobei F m die maximale Fluoreszenz im dunkeladaptierten Zustand und Fm' die maximale Fluoreszenz in jedem lichtadaptierten Zustand darstellt. - Überwachen Sie die NPQ-Werte über die visuelle Benutzeroberfläche der Bildgebungssoftware. Die Software ermöglicht die Echtzeitanalyse und -anzeige der Fluoreszenzdaten, einschließlich der NPQ-Werte.
Ergebnisse
Agrobacterium-vermittelt in der planta-Genexpression in Bambusblättern
Es hat sich gezeigt, dass das RUBY-Reportergen bei der Visualisierung der transienten Genexpression wirksam ist, da es in der Lage ist, leuchtend rotes Betalain aus Tyrosin10 zu produzieren. In dieser Studie wurde die Agrobacterium-vermittelte Transformation genutzt, um das exogene Ruby-Gen vorübergehend in Bambusblättern zu exprimieren (Abbildung 1). Am 3. Tag nach der Infektion wurde bei den unreifen gefalteten Blättern eine rote Färbung beobachtet, die am 5. Tag nach der Entfaltung der Blätter lebhafter wurde (blaues Dreieck, Abbildung 1C). Diese Ergebnisse zeigen, dass Agrobacterium erfolgreich die Expression des exogenen Ruby-Gens in Bambusblättern vermittelt hat und dass eine Betalainsynthese stattgefunden hat.
Darüber hinaus wurden vier Stämme von Agrobacterium (AGL1, LBA4404, EHA105 und GV3101) verglichen und es wurde festgestellt, dass der GV3101-Stamm die signifikanteste Betalain-Akkumulation in infizierten Bambusblättern verursachte, wobei der höchste Prozentsatz von 85,2 % der Keimlinge Betalain nach der Infektion akkumulierte, gefolgt von AGL1 (76,9 %) und dann EHA105 (49,1 %) und LBA4404 (31,3 %; Abbildung 1D). Dies deutet darauf hin, dass GV3101 die am besten geeignete Sorte für diesen Zweck ist. Es wurde eine High-Fidelity-PCR durchgeführt, um festzustellen, ob sich das Agrobacterium-vermittelte T-DNA-Fragment in das Bambuschromosom integriert hatte. Nach 40 PCR-Zyklen wurden keine Banden des Ruby-Gens nachgewiesen, was darauf hindeutet, dass das T-DNA-Fragment nicht oder nur in einer so geringen Anzahl integriert war, dass es nicht nachgewiesen werden konnte. Diese Ergebnisse schließen daraus, dass diese Genexpression vorübergehend ist.
Insgesamt zeigen diese Ergebnisse die Machbarkeit der Agrobacterium-vermittelten transienten Genexpression in planta in Bambus unter Verwendung des RUBY-Reportergens . Die rote Betalain-Farbe erwies sich jedoch als instabil und verschwand nach 3 Monaten der Infektion, was darauf hindeutet, dass das transiente Expressionssystem für die Langzeitbeobachtung nicht stabil ist.
In planta Gen-Editierung des Bambus-Violaxanthin-De-Epoxidase-Gens (PeVDE)
Die Agrobacterium-vermittelte Genexpression in planta ist eine transiente Methode der Genexpression in Bambus. Um zu untersuchen, ob ein transientes CRISPR/Cas9-System eine Gen-Editierung in Bambusblättern erreichen kann, wurde das Schlüsselenzym im Xanthophyll-Zyklus von Bambus, die Violaxanthin-De-Epoxidase (PeVDE), als Ziel für die Versuchs-Geneditierung ausgewählt. Single guide RNAs (sgRNAs) wurden auf dem ersten Exon des PeVDE-Gens (sgRNA-1) entworfen, das Restriktionsstellen des AltersI stromaufwärts des Protospacer-Nachbarmotivs (PAM) enthält, um die Validierung der Geneditierung zu erleichtern (Abbildung 2A).
Das CRISPR/Cas9-Konstrukt, das sgRNA-1 trägt, wurde in Agrobacterium transfiziert, um Bambusblätter zu transformieren. Nach 5-tägiger Infektion des Agrobakteriums, das die CRISPR/Cas9-Konstrukte enthält, die sgRNA-1 tragen, wurden die Bambuskeimlinge einer Hochlichtbehandlung unterzogen und anschließend ein Chlorophyll-Fluoreszenzparameter-Nachweis durchgeführt. Es wurden bestimmte Bereiche der Blattspreiten gefunden, die niedrigere nicht-photochemische Quenching-Werte (NPQ) aufwiesen (Abbildung 2B), was darauf hindeutet, dass die Lichtschutzfähigkeit dieser Bereiche unter intensivem Licht reduziert war. Da das PeVDE-Gen die Fähigkeit hat, überschüssige absorbierte Lichtenergie abzuleiten13, sind diese Bereiche mit niedrigeren NPQ-Werten wahrscheinlich die Regionen, in denen das PeVDE-Gen editiert wurde. Anschließend wurde eine Enzymverdauungs- und Sequenzierungsanalyse des PeVDE-Genfragments in diesen Bereichen der Blattspreiten durchgeführt (Abbildung 2C-D), und es wurde festgestellt, dass die Mutationsrate von sgRNA-1 17,33 % betrug, was darauf hindeutet, dass die Geneditierung in diesen Bereichen des PeVDE-Gens erfolgreich war.
Darüber hinaus wurde eine weitere sgRNA-Targeting-Stelle, sgRNA-2, die eine XbaI-Restriktionsstelle enthält, auf dem ersten Exon von PeVDE entworfen. Um die Möglichkeit einer langen Fragmentdeletion mit dualem sgRNA-Targeting zu untersuchen, wurde eine Geneditierung an beiden Zielstellen durchgeführt, was zu einer langen Fragmentdeletion führte (Abbildung 2E).
Editierte PeVDE-Mutante , die als Reporter im transienten Gen-Editing-System verwendet wird
Es wurde untersucht, ob die PeVDE-sgRNA als Reporter im transienten Geneditierungssystem dienen könnte. Das Cinnamoyl-CoA-Reduktase-Gen (PeCCR5) (Gen-ID: PH02Gene42984.t1) wurde zufällig ausgewählt, um den PeVDE-Reporter zu evaluieren . Ein sgRNA-Target für PeCCR5 wurde in seinem konservierten Motiv auf dem vierten Exon entworfen. Das CRISPR/Cas9-Konstrukt, das beide sgRNAs, PeVDE und PeCCR5, trägt, wurde in Bambusblätter umgewandelt (Abbildung 3A).
Nach einer 30-tägigen Agrobacterium-Infektion wurden die Sämlinge 20 Minuten lang mit hochintensivem Licht behandelt. Es wurde beobachtet, dass nur die Blattbereiche, die für das PeCCR5-Gen editiert wurden, keinen Einfluss auf die NPQ-Werte hatten, während die Blattbereiche, die mit sgRNAs von PeVDE und PeCCR5 transfiziert wurden, niedrigere NPQ-Werte aufwiesen (Abbildung 3B).
Anschließend wurde das PeCCR5-Fragment aus den Blattbereichen mit niedrigeren NPQ-Werten amplifiziert und sequenziert und fand mittels Tiefensequenzierung eine Mutationseffizienz von 8,3%. Daher diente der PeVDE-Reporter erfolgreich als transienter Gen-Editing-Reporter und kann zum Screening auf Gen-Editierung anderer endogener Bambus-Gene verwendet werden.
Insgesamt zeigen diese Ergebnisse die Machbarkeit der Bambus-Gen-Editierung mit CRISPR/Cas9 in Bambus.
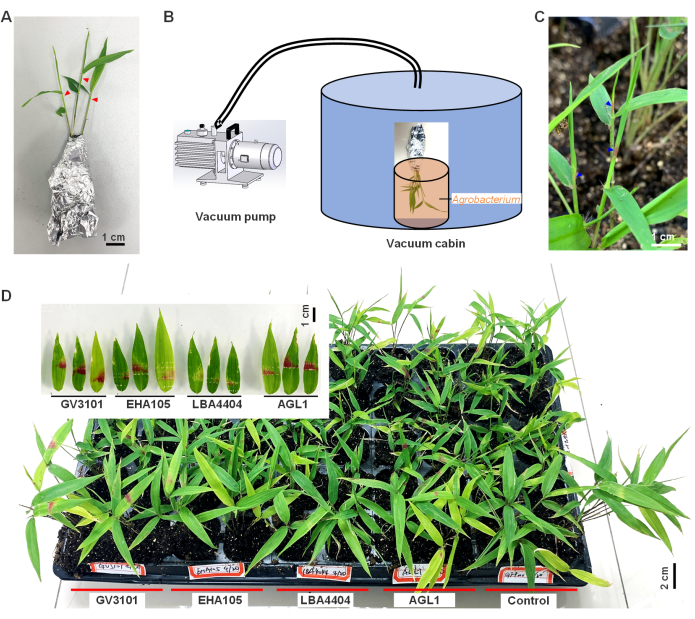
Abbildung 1: In-planta-Expression des Ruby-Gens und der Betalain-Akkumulation in Moso-Bambusblättern. (A) Moso-Bambus-Sämlinge, die in Alufolie eingewickelt und bereit für eine Agrobacterium-Infektion sind, mit roten Dreiecken, die Positionen anzeigen, die durch eine scharfe Nadel aus einer Spritze verletzt wurden. (B) Vakuum-Infiltrationsprozess von Bambussetzlingen. (C) Betalain-Akkumulation in Bambusblättern nach 3 Tagen der Infektion, beobachtet durch phänotypische Veränderungen. (D) Hier wurde mit vier Agrobacterium-Stämmen, AGL1, LBA4404, EHA105 und GV3101, eine RUBY-Gentransformation in Bambusblättern durchgeführt. GV3101 mit dem GFP-Konstrukt wurde als Negativkontrolle verwendet. Diese Zahl wurde von9 geändert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 2: In-planta-Expression und Gen-Editierung des PeVDE-Gens in Bambusblättern . (A) Orts- und Zielsequenzinformationen der sgRNA im PeVDE-Gen . Rote Dreiecke zeigen die Positionen der Vorwärts- und Rückwärtsprimer für die Fragmentverstärkung an. (B) NPQ und Rohbildgebung von Bambusblättern nach Infektion. Die Zahlen im NPQ-Bild stellen NPQ-Werte im Monitor der Bildverarbeitungssoftware dar. (C) Elektrophorese-Ergebnisse des PeVDE-Fragments vor und nach dem Age-I-Verdau. WT bezeichnet die nicht infizierten Wildtyp-Blätter, und + und - stehen für PeVDE-Fragmente mit bzw. ohne Age-I-Verdauung. (D) Tiefensequenzierungsergebnisse des PeVDE-Fragments in Blättern mit niedrigerem NPQ-Wert. Die roten, blauen und grauen Schriftarten in den Sequenzen stellen die Zielsites, PAM bzw. Einfügungen dar. Die roten Striche zeigen gelöschte Nukleotide an. (E) Sanger-Sequenzierungsergebnisse des PeVDE-Fragments nach Editierung mit sgRNA-1 und sgRNA-2. Diese Zahl wurde von9 geändert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 3: PeVDE-sgRNA als Reporter für das Screening der Gen-Editierung von PeCCR5. (A) Schematische Darstellung von CRISPR/Cas9-Konstrukten, die PeVDE- und PeCCR5-sgRNAs enthalten. (B) NPQ und Rohbilder von Bambusblättern nach Infektion mit den Konstrukten in (A). Weiße Dreiecke kennzeichnen Bereiche mit niedrigeren NPQ-Werten. Die Regenbogenfarbe stellt den Wert von NPQ/4 dar, wobei Rot dem Minimalwert und Violett 1 entspricht. (C) Die roten, blauen und grauen Schriftarten in den Sequenzen stellen die Zielseiten, PAM bzw. Einfügungen dar. Die roten Striche zeigen gelöschte Nukleotide an. Diese Zahl wurde von9 geändert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
| Name des Gens | Primer-Sequenz (5'-3') | Anwendung | ||
| RUBINROT | F: ATGGATCATGCGACCCTCG | Zur PCR-Amplifikation in infizierten Bambusblättern | ||
| R: GTACTCGTAGAGCTGCTGCAC | ||||
| PeVDE | F: TGTGGCTTCTAAAGCTCTGCAATCT | Für die Klonierung und Sequenzierung von Genen | ||
| R: TGTCAATGCTACAAGTCCTGGCA | ||||
| PeVDE-Target1 | F: GGCATAGCCCTCACGCAGCACCGG | Für die Entwicklung des PeVDE-sgRNA-1-Targets | ||
| R: AAACCCGGTGCTGCGTGAGGGCTA | ||||
| PeVDE-Target2 | F: GGCACTCCACGGTCCCAAATCTAG | Für die Entwicklung von PeVDE sgRNA-2-Targets | ||
| R: AAACCTAGATTTGGGACCGTGGAG | ||||
| PeCCR5-Target | F: GGCACTGGTACTGCTACGCTAAGA | Für die Entwicklung des PeCCR5 sgRNA-Targets | ||
| R: AAACTCTTAGCGTAGCAGTACCAG | ||||
Tabelle 1: Die Sequenzinformationen von Primern.
Diskussion
Diese Methode reduziert den Zeitaufwand im Vergleich zu herkömmlichen genetischen Transformationsmethoden, die in der Regel 1-2 Jahre dauern, erheblich und erreicht eine vorübergehende Expression exogener Gene und eine Genom-Editierung endogener Gene innerhalb von 5 Tagen. Diese Methode hat jedoch ihre Grenzen, da sie nur einen kleinen Teil der Zellen transformieren kann und die geneditierten Blätter chimärisch sind und nicht in der Lage sind, sich zu vollständigen Pflanzen zu regenerieren. Nichtsdestotrotz bietet diese in planta Genexpressions- und Gen-Editing-Technologie einen leistungsfähigen Ansatz für die funktionelle Verifizierung endogener Bambusgene.
Derzeit können die Genexpression und die Geneditierungstechnologie in planta nur in unreifen (gerollten) Blättern durchgeführt werden, nicht in reifen Blättern. Wenn sich die Blätter entfalten und vergrößern, steigt die Anzahl der geneditierten Zellen, die sich teilen, was eine Gen-Editierung in bestimmten Blattregionen ermöglicht. Die verwendete Agrobacterium-vermittelte Transformationsmethode führt jedoch nicht zu einer Insertion der exogenen T-DNA in das Bambuschromosom, was es schwierig macht, stabile Markergene in Bambuszu verwenden 6,9. Daher ist es schwierig, die genauen Standorte dieser Regionen zu bestimmen. Um dieses Problem anzugehen, wurde das PeVDE-Gen editiert, und der editierte Bereich zeigte eine verminderte Lichtschutzfähigkeit unter Starklichtbehandlung, was durch niedrigere NPQ-Werte angezeigt wird, die mit einem Chlorophyll-Fluorometer-Imaging-PAM leicht nachgewiesen werden können. So wurde PeVDE als Marker in Bambus entwickelt, um das Auftreten von exogener Genexpression und Geneditierung nachzuweisen. Aufgrund der hohen Konservierung dieses Gens über verschiedene Arten hinweg 13 kann es auch auf andere Pflanzen übertragen werden.
Durch die Ablagerung einer kutikulären Wachsschicht auf der Epidermis von Bambusblättern, gepaart mit der charakteristischen gekräuselten und eng umwickelten Morphologie unreifer Blätter, ist die Zugänglichkeit von Agrobacterium zu Blattzellen erheblich erschwert. Um die Wirksamkeit der Agrobacterium-Infektion zu verbessern, wurden physikalische Ansätze, einschließlich Wund- und Vakuuminfiltration, eingesetzt, um das Eindringen von Agrobacterium in die eingeschlossenen gekräuselten Bambusblätter zu fördern. Dieser Prozess ermöglicht eine unmittelbare Nähe zwischen Agrobacterium und den Blattzellen und erhöht so die Effizienz der genetischen Transformation. In der Zwischenzeit war dieses Gen-Editierungssystem bisher auf Bambusblätter beschränkt und kann nicht in Organen mit Fortpflanzungsfähigkeit wie Samen und Seitenknospen exprimiert werden, die an die nächste Generation vererbt werden können. Zukünftige Anwendungen der Technik werden optimiert, um eine in-planta-Genexpression und Gen-Editing-Technologie in reproduktionsfähigen Organen zu erreichen, mit dem Ziel, stabil vererbbare regenerierende Pflanzen zu erhalten.
Offenlegungen
Die Autoren erklären, dass sie keine konkurrierenden Interessen haben.
Danksagungen
Die Autoren danken dem National Key Research and Development Program of China (Grant No. 2021YFD2200502), der National Natural Science Foundation of China (Grant No. 31971736) für die finanzielle Unterstützung.
Materialien
| Name | Company | Catalog Number | Comments |
| 35S::RUBY | Addgene, United States | 160908 | Plamid construct |
| Agrobacterium competent cells of GV3101, EHA105,LBA4404, and AGL1 | Biomed, China | BC304-01, BC303-01, BC301-01, and BC302-01 | For Agrobacterium infection |
| CTAB | Sigma-Aldrich, United States | 57-09-0 | DNA extraction |
| Imaging-PAM fluorometer | Walz, Effeltrich, Germany | Detect chlorophyll fluorescence of bamboo leaves | |
| ImagingWin | Walz, Effeltrich, Germany | Software for Imaging-PAM fluorometer | |
| Paq CI or Aar I | NEB, United States | R0745S | Incorporate the target sequence onto the CRISPR/Cas9 vector. |
| PrimeSTAR Max DNA polymerase | Takara, Japan | R045Q | For gene cloning |
| T4 DNA ligase | NEB, United States | M0202V | Incorporate the target sequence onto the CRISPR/Cas9 vector. |
Referenzen
- Jiang, Z. H. World Bamboo and Rattan (in Chinese). , Liaoning Science and Technology Publishing House. Shenyang, China. (2002).
- Ye, S., et al. An efficient plant regeneration and transformation system of ma bamboo (Dendrocalamus latiflorus Munro) started from young shoot as explant. Frontiers in Plant Science. 8, 1298(2017).
- Ye, S., et al. Robust CRISPR/Cas9 mediated genome editing and its application in manipulating plant height in the first generation of hexaploid Ma bamboo (Dendrocalamus latiflorus Munro). Plant Biotechnology Journal. 18 (7), 1501-1503 (2020).
- Xiang, M., et al. Production of purple Ma bamboo (Dendrocalamus latiflorus Munro) with enhanced drought and cold stress tolerance by engineering anthocyanin biosynthesis. Planta. 254 (3), 50(2021).
- Huang, B., et al. An efficient genetic transformation and CRISPR/Cas9-based genome editing system for moso bamboo (Phyllostachys edulis). Frontiers in Plant Science. 13, 822022(2022).
- Lee, M. W., Yang, Y. Transient expression assay by agroinfiltration of leaves. Methods in Molecular Biology. 323, 225-229 (2006).
- Canto, T. Transient expression systems in plants: potentialities and constraints. Advances in Experimental Medicine and Biology. 896, 287-301 (2016).
- Chen, L., et al. A method for the production and expedient screening of CRISPR/Cas9-mediated non-transgenic mutant plants. Horticulture Research. 5, 13(2018).
- Sun, H., et al. A new biotechnology for in-planta gene editing and its application in promoting flavonoid biosynthesis in bamboo leaves. Plant Methods. 19 (1), 20(2023).
- He, Y., Zhang, T., Sun, H., Zhan, H., Zhao, Y. A reporter for noninvasively monitoring gene expression and plant transformation. Horticulture Research. 7 (1), 152(2020).
- Wang, C., Shen, L., Fu, Y., Yan, C., Wang, K. A simple CRISPR/Cas9 system for multiplex genome editing in rice. Journal of Genetics and Genomics. 42 (12), 703-706 (2015).
- McCormick, S., et al. Leaf disc transformation of cultivated tomato (L. esculentum) using Agrobacterium tumefaciens. Plant Cell Reports. 5 (2), 81-84 (1986).
- Lou, Y., et al. a violaxanthin de-epoxidase gene from moso bamboo, confers photoprotection ability in transgenic Arabidopsis under high light. Frontiers in Plant Science. 13, 927949(2022).
- Zhou, R., et al. Distinct cinnamoyl CoA reductases involved in parallel routes to lignin in Medicago truncatula. Proceedings of the National Academy of Sciences of the United States of America. 107 (41), 17803-17808 (2010).
- De Roeck, A., et al. Deleterious ABCA7 mutations and transcript rescue mechanisms in early onset Alzheimer's disease. Acta Neuropathologica. 134 (3), 475-487 (2017).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten