Method Article
Visualisierung von Pseudomonas aeruginosa im Sputum von Mukoviszidose-Patienten
In diesem Artikel
Zusammenfassung
Dieses Protokoll bietet Methoden zur Visualisierung von Bakterienzellen und Polysaccharid-Syntheselocus (Psl)-Polysacchariden im Sputum von Mukoviszidose-Patienten.
Zusammenfassung
Die frühzeitige Erkennung und Eradikation von Pseudomonas aeruginosa in der Lunge von Mukoviszidose-Patienten kann das Risiko einer chronischen Infektion verringern. Die Entwicklung chronischer P. aeruginosa-Infektionen ist mit einer Abnahme der Lungenfunktion und einer erhöhten Morbidität verbunden. Daher besteht ein großes Interesse an der Aufklärung der Gründe für das Scheitern der Eradikation von P. aeruginosa mit einer antibiotischen Therapie, die bei etwa 10-40% der pädiatrischen Patienten auftritt. Einer von vielen Faktoren, die die Wirtsclearance von P. aeruginosa und die Antibiotikaempfindlichkeit beeinflussen können, sind Variationen in der räumlichen Organisation (z. B. Aggregation oder Biofilmbildung) und der Polysaccharidproduktion. Daher waren wir daran interessiert, die in situ Charakteristika von P. aeruginosa im Sputum von Mukoviszidose-Patienten sichtbar zu machen. Eine Gewebereinigungstechnik wurde auf Sputumproben angewendet, nachdem die Proben in eine Hydrogelmatrix eingebettet wurden, um die 3D-Strukturen relativ zu den Wirtszellen zu erhalten. Nach der Gewebereinigung wurden fluoreszierende Markierungen und Farbstoffe hinzugefügt, um eine Visualisierung zu ermöglichen. Fluoreszenz-in-situ-Hybridisierung zur Visualisierung von Bakterienzellen, Bindung von fluoreszenzmarkierten Anti-Psl-Antikörpern zur Visualisierung des Exopolysaccharids und DAPI-Färbung zur Färbung von Wirtszellen, um strukturelle Einblicke zu erhalten. Diese Methoden ermöglichten die hochauflösende Bildgebung von P. aeruginosa im Sputum von Mukoviszidose-Patienten mittels konfokaler Laser-Scanning-Mikroskopie.
Einleitung
In dieser Studie wurden Experimente durchgeführt, um die in vivo Struktur von Pseudomonas aeruginosa im Sputum von pädiatrischen Mukoviszidose-Patienten (CF) sichtbar zu machen. P. aeruginosa-Infektionen werden bei 30-40% der pädiatrischen Mukoviszidose-Bevölkerung chronisch; Wenn sich chronische Infektionen erst einmal etabliert haben, sind sie fast unmöglich zu beseitigen1. P. aeruginosa-Isolate von Patienten mit früher Infektion sind im Allgemeinen anfälliger für antimikrobielle Mittel, daher werden diese mit anti-pseudomonalen Antibiotika behandelt, um die Etablierung einer chronischen Infektion zu verhindern2. Leider werden nicht alle P. aeruginosa-Isolate nach einer Antibiotikatherapie effektiv aus der Lunge entfernt. Die genauen Mechanismen, die mit dem Versagen von Antibiotika verbunden sind, sind noch nicht vollständig aufgeklärt. Frühere Studien haben gezeigt, dass Variationen in der Zelldichte, der Aggregation und der Polysaccharidproduktion die Wirksamkeit von Antibiotika beeinträchtigen können3. P. aeruginosa produziert drei extrazelluläre Polysaccharide: Pel, Psl und Alginat4. Die meisten Stämme von P. aeruginosa haben die genetische Fähigkeit, jedes der Exopolysaccharide zu exprimieren, obwohl oft ein Polysaccharidtyp überwiegend exprimiert wird5. Das Exopolysaccharid Alginat ist mit chronischen Infektionen in der Mukoviszidose-Lunge assoziiert, was zu einem mukoiden Phänotyp 6,7 führt. Die Polysaccharide Pel und Psl haben mehrere Funktionen, darunter die Unterstützung der anfänglichen Anheftung und der Aufrechterhaltung der Biofilmstruktur sowie die Verleihung von Antibiotikaresistenz8.
Methoden zur Visualisierung von In-vivo-Strukturen von Geweben wurden für eine Vielzahl von Probentypen entwickelt 9,10,11. In jüngerer Zeit wurden sie darauf zugeschnitten, mikrobielle Gemeinschaften in vivo im Sputum von Mukoviszidose-Patienten sichtbar zu machen12. Die Optimierung eines Gewebe-Clearing-Protokolls speziell für die Identifizierung von mikrobiellen Gemeinschaften innerhalb des Sputums wurde von DePas et al., 201612 entwickelt. Der Begriff MiPACT, der für Mikrobial identification after Passive CLARITY technique steht, wurde für die Klärung von Mukoviszidose-Sputumgeprägt 11,12. Bei Gewebereinigungstechniken werden die Proben zunächst fixiert und dann transparent gemacht, wobei ihre inhärente Architektur für die Färbung und mikroskopische Visualisierung intakt bleibt11. Die Fixierung und Klärung von Mukoviszidose-Sputumproben ermöglicht es den Forschern, Fragen im Zusammenhang mit der Biofilmstruktur, der bakteriellen Zelldichte, polymikrobiellen Assoziationen und Assoziationen zwischen Krankheitserregern und Wirtszellen zu beantworten. Der Vorteil der direkten Untersuchung von Bakterien, die im Sputum konserviert wurden, besteht darin, dass sie in einem wirtsspezifischen Kontext analysiert und visualisiert werden können. Obwohl die In-vitro-Züchtung klinischer Isolate im Labor für Experimente sehr aufschlussreich sein kann, sind solche Methoden nicht in der Lage, die Mukoviszidose-Lungenumgebung vollständig nachzubilden, was zu einer Diskrepanz zwischen Laborergebnissen und Patientenergebnissen führt.
Die hier vorgestellten Methoden können verwendet werden, um Sputum zu fixieren und zu reinigen, um Bakterien sichtbar zu machen, sei es von Mukoviszidose-Patienten oder Patienten mit anderen Atemwegsinfektionen. Die spezifische Art der Färbung und mikroskopischen Analyse, die hier beschrieben wird, ist die Fluoreszenz-in-situ-Hybridisierung (FISH), gefolgt von der Bindung von Anti-Psl-Antikörpern innerhalb des Hydrogels und der anschließenden Analyse mittels konfokaler Laser-Scanning-Mikroskopie (CLSM). Nach der Gewebereinigung können auch andere immunhistochemische und mikroskopische Methoden angewendet werden.
Protokoll
Für die Entnahme und Lagerung von Sputumproben von menschlichen Probanden ist die Genehmigung des Research Ethics Board (REB) erforderlich. Die hier vorgestellten Studien wurden vom Krankenhaus für kranke Kinder REB#1000058579 genehmigt.
1. Sputum-Entnahme
- Auswurf in einem sterilen Auffangbecher aufbewahren und sofort vor der Fixierung maximal 24 h bei 4 °C lagern.
HINWEIS: Wenn das Sputum zu lange bei 4 °C ohne Fixierung belassen wird, kann dies zu einem Zellabbau führen, insbesondere zum Abbau der weißen Blutkörperchen. Eine Fixierung so schnell wie möglich wird bevorzugt. - Überführen Sie die Sputumproben in ein steriles 15-ml-Röhrchen.
- Den Sputumproben wird ein gleiches Volumen von 4 % Paraformaldehyd (PFA) zugesetzt. Wenn die Sputumprobe beispielsweise 0,5 ml beträgt, fügen Sie 0,5 ml 4 % PFA hinzu. Durch sanfte Umkehrung mischen.
- Inkubieren Sie das Sputum über Nacht bei 4 °C.
- Waschen Sie die Probe, indem Sie 5 ml phosphatgepufferte Kochsalzlösung (PBS) zu je 2 ml fixiertem Sputum hinzufügen.
HINWEIS: Für diesen Waschschritt ist keine Zentrifugation erforderlich. - Entfernen Sie den Überstand vorsichtig mit einer Pipette.
HINWEIS: Vermeiden Sie es, den Auswurf mit der Pipette aufzusaugen, indem Sie die Spitze vom Auswurf wegzeigen und die Oberflächenflüssigkeit sehr langsam absaugen. Der Waschschritt beinhaltet keine Zentrifugation, um die strukturelle Integrität der Sputumpfropfen nicht zu stören. - Wiederholen Sie die Schritte 1.6-1.7 noch zwei weitere Male.
- Resuspendieren Sie das Pellet im 2-fachen Volumen von PBS mit 0,01 % (w/v) Natriumazid.
- Lagern Sie den Sputum bei 4 °C.
2. MiPACT (Tissue Clearing Technique) Verarbeitung von Sputum
- Entgasen Sie die Hydrogelkomponenten (30% 29:1 Acrylamid: Bis-Acrylamid, Härter und PBS) in einem verschlossenen Behälter mit einer anaeroben Packung vorher für 72 Stunden.
HINWEIS: Die anaerobe Packung und der versiegelte Behälter sind notwendig, da Sauerstoff die Acrylamidpolymerisation hemmt. Alternativ kann Sauerstoff entfernt werden, indem dieser Schritt in einer anaeroben Haube durchgeführt wird, indem ein Vakuum an einen versiegelten Behälter angelegt wird oder indemN2-Gas durch das Acrylamidgemisch sprudelt.- 2 ml einer 30%igen 29:1-Acrylamid:Bis-Acrylamid-Lösung in ein 15-ml-Röhrchen mit abgenommener Kappe im versiegelten anaeroben Behälter geben.
- Stellen Sie einen konzentrierten Härter mit 10 % (w/v) her, indem Sie 0,5 g Härter zu 5 ml PBS in einem 15-ml-Röhrchen hinzufügen. Lassen Sie die Kappe vom Röhrchen und bewahren Sie sie in dem verschlossenen anaeroben Behälter auf.
- Lassen Sie ein paar ml steriles PBS in einem Röhrchen mit abgenommener Kappe im anaeroben Behälter.
- 5 ml Hydrogellösung mit einer Endkonzentration von 0,2 % Härter und 4 % 29:1 Acrylamid:bis-acrylamid in PBS in einem 15 ml-Röhrchen herstellen. Durch Invertieren mischen und filtern Sie sie.
- Sputumproben aus dem Kühlschrank nehmen und dann unter sterilen Bedingungen mit einem Skalpell in kleine Abschnitte (ca. 5 mm Durchmesser) schneiden.
HINWEIS: Wenn der Auswurf recht flüssig ist, kann dieser Schritt trotzdem durchgeführt werden, es erfordert jedoch mehr Geduld und Übung, um den Auswurf mit einer Pinzette aus der Aufbewahrungslösung zu entfernen, bevor Sie das Skalpell verwenden, um die gewünschten Fraktionen zu trennen. - Legen Sie die geschnittenen Sputumproben in eine Vertiefung eines Objektträgers mit 8 Kammern.
- Geben Sie 300 μl filtersterilisierte Hydrogellösung aus Schritt 2.2 in jede der Vertiefungen, die Sputum enthalten.
- Legen Sie das 8-Kammer-Deckglas für 3 Stunden bei 37 °C in einen verschlossenen Behälter mit einer anaeroben Packung.
HINWEIS: Nach der Polymerisation sollte das Hydrogel mit eingebettetem Sputum die Konsistenz eines festen Gels haben. - Die erstarrten Hydrogel-Sputumproben werden in ein 15-ml-Kulturröhrchen mit 5 ml 8%igem Natriumdodecylsulfat (SDS) mit einem pH-Wert von 8 überführt und die Proben 3-14 Tage lang bei 37 °C (mit oder ohne Schütteln) klären lassen, bis das Sputum transparent wird.
HINWEIS: Die Reinigungszeiten hängen von der Zusammensetzung des Sputums ab und können im Allgemeinen durch Schütteln verkürzt werden. Achten Sie jedoch darauf, die Schüttelgeschwindigkeit nicht so weit zu erhöhen, dass die Integrität der Probe beeinträchtigt wird. DNA-reichere Proben brauchen im Vergleich zu schleimreichen Proben länger, um entfernt zu werden. - Die 8%ige SDS-Lösung in einen Abfallsammelbehälter umfüllen. Mit einer sterilen Pinzette wird jede in Hydrogel eingebettete Probe in ein steriles konisches 50-ml-Röhrchen übertragen. Geben Sie 10 ml PBS in jedes der konischen 50-ml-Röhrchen, um die Hydrogele zu waschen, und lassen Sie die Lösung vor dem Umfüllen 30 Minuten bis 1 Stunde einwirken. Wiederholen Sie dies noch 2x mehrmals.
HINWEIS: Dieser Waschschritt beinhaltet keine Zentrifugation. Überschüssige Flüssigkeit und Überstand werden vorsichtig mit einer Pipette entfernt. - Gewaschene Proben werden in PBS mit 0,01 % (w/v) Natriumazid und 1x RNase-Inhibitor bei 4 °C gelagert.
3. Protokoll der Hydrogel-Fluoreszenz-in-situ-Hybridisierung (FISH)
- Nehmen Sie die Hydrogel-Proben mit einer sterilen Pinzette aus ihrer Aufbewahrungslösung und legen Sie die Proben auf eine sterile Oberfläche (z. B. einen Objektträger oder eine Petrischale).
- Schneiden Sie die Hydrogele mit einem sterilen Skalpell in ~ 1 mm dicke Scheiben.
- Legen Sie die 1 mm dicken Abschnitte des Hydrogels in sterile 1,5-ml-Röhrchen.
- Bereiten Sie 1 ml den Hybridisierungspuffer (25 % Formamid, 0,9 M NaCl, 20 mM Tris-HCl [pH 7,6], 0,01 % SDS, gereinigtes und deionisiertesH2O) in einem 1,5-ml-Röhrchen vor.
- Die fluoreszenzmarkierte PseaerA-Sonde (150,7 nM12) wird in den Hybridisierungspuffer gegeben und durch Inversion gemischt.
HINWEIS: Formalinlösung sollte von einer offenen Flamme ferngehalten werden. Der Hybridisierungspuffer kann im Voraus hergestellt und in Aliquoten bei -20 °Cgelagert werden 13. - Geben Sie 200-500 μl Hybridisierungspuffer zu jedem ~1 mm Abschnitt des Hydrogels hinzu und stellen Sie sicher, dass die gesamte Hydrogelprobe untergetaucht ist.
- Lassen Sie die PseaerA-Sonde mit den Hydrogelproben hybridisieren, indem Sie sie ~ 18-24 h lang bei 46 °C im Dunkeln platzieren, ohne sie zu schütteln.
- Dekantieren Sie den Hybridisierungspuffer in einen Abfallsammelbehälter.
- Spülen Sie die Proben einmal mit filtersterilisiertem Waschpuffer (337,5 mM NaCl, 20 mM Tris-HCl, 5 mM EDTA (Ethylendiamintetraessigsäure) [pH 7,2], 0,01 % SDS und gereinigtem und deionisiertemH2O), indem Sie 1 ml Waschpuffer in jedes der 1,5-ml-Röhrchen geben und dann entfernen.
HINWEIS: Der Waschpuffer kann im Voraus hergestellt und bei Raumtemperatur (RT) gelagert werden. - Geben Sie 1 ml frischen Waschpuffer in die Röhrchen und inkubieren Sie die Proben dann 6 h lang im Dunkeln bei 48 °C, ohne zu schütteln.
4. Bindung von Hydrogel und Psl0096-Antikörpern
- Entfernen Sie den Waschpuffer steril mit einer 1-ml-Pipette.
- Spülen Sie die Proben mit einer 2%igen BSA (w/v) in PBS-Lösung, indem Sie 1 ml der 2%igen BSA-Lösung hinzufügen und entfernen.
- Geben Sie 500 μl der 2%igen BSA/PBS-Lösung zu den Hydrogelproben, um die unspezifische Proteinbindung zu blockieren. Inkubieren Sie die Proben dann über Nacht, im Dunkeln, bei RT, ohne zu schütteln.
- Steril entfernen Sie die Blockierungslösung mit einer 1-ml-Pipette.
- Bereiten Sie die Antikörperlösung Psl0096-Texas Red vor, indem Sie den Antikörper auf eine Endkonzentration von 0,112 μg/ml in 500 μl frischem 2%igem BSA/PBS verdünnen.
- Geben Sie die 500 μl Antikörperlösung zu den Hydrogelproben und inkubieren Sie sie bei RT für 6 h, lichtgeschützt, ohne zu schütteln.
5. DAPI (4′,6′-Diamidino-2-phenylindol)-Färbung
- Die Brechungsindexanpassungslösung (RIMS) wird hergestellt, indem 40 g nichtionisches Dichtegradientenmedium, 30 μl Tween20, 3 μg Natriumazid und 30 ml PBS in einen Kolben mit einem magnetischen Rührstab gegeben werden. Rühren Sie die Lösung 15 Minuten lang auf einem Magnetrührer oder bis sie sich vollständig aufgelöst hat.
- Der Filter sterilisiert die Lösung mit einer 10-ml-Spritze und einem sterilen 0,2-μm-Filter in ein konisches 50-ml-Röhrchen.
HINWEIS: Die Lösung kann bei 4 °C mehrere Monate gelagert werden. - Entfernen Sie die Antikörperlösung Psl0096-Texas Red mit einer sterilen 1-ml-Pipette.
- Spülen Sie die Hydrogelproben ab, indem Sie 1 ml PBS hinzufügen und dann entfernen.
- Inkubieren Sie die Hydrogelproben mit 250 μl RIMS-Lösung und 10 μg/ml DAPI bei RT mit leichtem Schütteln, im Dunkeln, über Nacht.
- Montieren Sie die Proben vor der konfokalen Bildgebung auf 0,9 mm oder 1,7 mm Perfusionskammern und versiegeln Sie sie mit einem Deckglas.
HINWEIS: Nach FISH und/oder Immunhistochemie und vor dem Eintauchen in RIMS können fluoreszierende Lektinfärbungen angewendet werden, wenn eine Visualisierung des Sputumschleims gewünscht wird12.
6. Bildgebung
- Führen Sie konfokale Laser-Scanning-Mikroskopie-Bildgebung mit Standardtechniken bei 25-facher, 40-facher, 63-facher oder 100-facher Vergrößerung durch.
Ergebnisse
Das Gesamtdesign des Experiments ist in Abbildung 1 und Abbildung 2 zusammengefasst. Abbildung 1 enthält eine Zusammenfassung der Protokolle für die Sputumverarbeitung und die Sputumreinigung. Die Sputumbearbeitung und -reinigung kann bis zu 17 Tage dauern. Das Protokoll kann jedoch gestoppt werden, und die Proben können nach der Fixierung mit PFA (Tag 2) oder nach der Gewebereinigung (Tage 5-17 je nach Reinigungszeit) gelagert werden. In Abbildung 2 sind die FISH- und Antikörperbindungsprotokolle zusammengefasst. Die FISH- und Antikörper-Färbeprotokolle dauern 4 Tage, sollten aber nach Beginn abgeschlossen und konfokale Bilder aufgenommen werden. Mit den oben genannten Protokollen können hochauflösende 3D-Bilder von P. aeruginosa-Zellen erhalten werden, wobei ihre in-situ-Struktur im Sputum visualisiert wird.
Die Reinigung von Sputum und die anschließende Anwendung von Fluoreszenzfärbungen, die in diesem Protokoll verwendet werden, ermöglicht eine detaillierte Visualisierung von P. aeruginosa in den Proben. Die in Abbildung 3 und Abbildung 4 gezeigten Sputumproben wurden von einem pädiatrischen CF-Patienten (17 Jahre alt) mit einer neu aufgetretenen P. aeruginosa-Infektion entnommen. In Abbildung 3A war ein Aggregat von Zellen in einer Sputumprobe zu sehen; Das Auftreten der gelblich-grünen Stäbchen war auf die Überlappung aller drei Fluorophore zurückzuführen. Obwohl wir keine Zellzahlen für diese Sputumprobe haben, fanden wir heraus, dass die Nachweisgrenze der Sonde bei 104 Zellen/ml liegt (siehe ergänzende Abbildung 1). In Abbildung 3B waren einzelne Stäbchenformen in Grün zu sehen, die von der speziesspezifischen Bindung der PseaerA-488-Sonde an P. aeruginosa-Zellen herrührten . Der rot dargestellte Antikörper Psl0096-Texas Red zeigt, wo sich das pseudomonale Exopolysaccharid im Sputum befand. in diesem Fall schien der Psl0096-Antikörper hauptsächlich mit den P. aeruginosa-Zellen zu überlappen (Abbildung 3C). Diese Methode ermöglichte auch die Visualisierung von pseudomonalen Zellen innerhalb des Sputums in Beziehung zu anderen Bakterienzellen und Wirtsstrukturen. In Abbildung 4 wurde ein Cluster von P. aeruginosa-Zellen in einer eukaryotischen Zelle phagozytiert und andere kleine Kokkenzellcluster in der Nähe beobachtet. Es wurde gezeigt, dass der Antikörper Psl0096 auch an Psl binden kann, das aus planktonischen P. aeruginosa produziert wird (siehe ergänzende Abbildung 2).
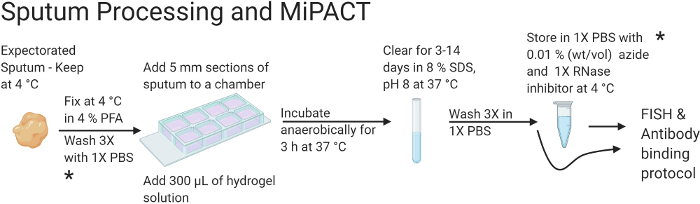
Abbildung 1: Flussdiagramm zur Veranschaulichung der Sputumverarbeitung und der MiPACT-Protokolle.
Zusammenfassend lässt sich sagen, dass Sputum fixiert wird, bevor es in eine Polyacrylamidlösung eingebettet wird. Nach der Klärung kann das Hydrogel entweder bei 4 °C gelagert oder mit dem FISH- und Antikörperbindungsprotokoll verwendet werden. Dieses Bild wurde mit BioRender.com erstellt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
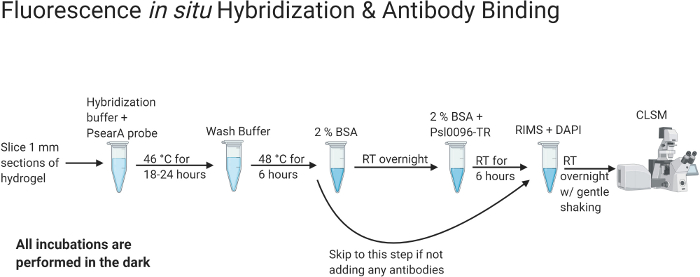
Abbildung 2: Flussdiagramm mit den Protokollen für die Fluoreszenz-in-situ-Hybridisierung und die Antikörperfärbung.
Sobald sich der Auswurf innerhalb der Hydrogelmatrix vollständig geklärt hat, können Färbemethoden angewendet werden. Dieses Bild wurde mit BioRender.com erstellt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 3: Immunfluoreszenzaufnahme einer Sputumprobe, die von einem Patienten mit einer frühen P. aeruginosa-Infektion entnommen wurde, eingebettet in eine Hydrogelmatrix.
Die Hydrogelprobe wurde mit einer PsearA-Alexa488-Sonde (grün), einem Psl0096-Texas Red-Antikörper (rot) und DAPI (blau) hybridisiert. (A) Sputumprobe unter allen 3 Kanälen, (B) Sputum nur unter dem grünen Kanal, der angibt, wo die PseaerA-488-Sonde gebunden ist, und (C) Sputum nur unter dem roten Kanal, in dem die rote Bindung von Psl0096-Texas aufgetreten ist. Die Bilder wurden mit 100-facher Vergrößerung aufgenommen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 4: Immunfluoreszenzaufnahme einer Sputumprobe, die von einem Patienten mit einer frühen P. aeruginosa-Infektion entnommen wurde, eingebettet in eine Hydrogelmatrix.
Die Hydrogelprobe wurde mit einer PsearA-Alexa488-Sonde (grün), einem Psl0096-Texas Red-Antikörper (rot) und DAPI (blau) hybridisiert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Ergänzende Abbildung 1: Fluoreszenz-in-situ-Hybridisierung einer planktonischen Kultur von PAO1 mit der PseaerA-488-Sonde. Bitte klicken Sie hier, um diese Abbildung herunterzuladen.
Ergänzende Abbildung 2: P. aeruginosa, gefärbt mit DAPI und dem Antikörper Psl0096-Texas Red. (A) Stamm PAO1-Δpsl und (B) Stamm PAO1. Bitte klicken Sie hier, um diese Abbildung herunterzuladen.
Diskussion
Der Zweck dieses Protokolls ist es, einen Einblick in die in-situ Organisation von P. aeruginosa-Zellen im Sputum von Mukoviszidose-Patienten zu ermöglichen. Sputumproben sollten bis zur Verarbeitung bei 4 °C gelagert werden, wenn sie nicht sofort fixiert werden können. Es wurde gezeigt, dass sich die Zellzahlen von P. aeruginosa im Sputum nicht signifikant ändern, wenn sie bei 1 h, 24 h oder 48 h verarbeitet werden, wenn sie bei 4 °C gelagert werden, obwohl die Bakterienzellzahl bei 25 °C für 24 oder 48 h als Folge des Bakterienwachstums signifikant ansteigt14. Für diese Studie wurden Sputumproben bei 4 °C bis maximal 24 h nach dem Auswurf gelagert. Es ist zu beachten, dass die Anzahl der Entzündungszellen nachweislich im Sputum abnimmt, wenn sie bei 4 °C belassen und mehr als 9 Stunden später verarbeitet werden15. Daher ist es wichtig, die spezifischen Zellen und Marker zu berücksichtigen, die man im Sputum visualisieren möchte, wenn man sich für eine Cut-off-Zeit für die Probenverarbeitung entscheidet.
Die Probenverarbeitung bei dieser Methode beginnt mit der Fixierung der Sputumproben in 4 % PFA. Paraformaldehyd vernetzt Bakterienzellen und ihre extrazelluläre Matrix und bewahrt ihre Strukturen für die mikroskopische Visualisierung und Analyse11,16. Wenn das Ziel darin besteht, die Gesamtzellzahl bestimmter Entzündungszellen im Sputum zu erhöhen, hat sich leider gezeigt, dass PFA die Anzahl dieser Zellen verringert, so dass andere Fixiermittel in Betracht gezogen werden sollten17. Eine weitere Einschränkung dieser Studie besteht darin, dass sie zeitaufwändig sein kann und über zwei Wochen dauern kann. Daher ist es möglicherweise nicht für die Entwicklung zu einer diagnostischen Methode geeignet, die zeitkritische Behandlungsentscheidungen erfordert. Darüber hinaus wäre diese Methode für das Verständnis der gesamten mikrobiellen Diversität in Sputumproben nicht geeignet, könnte aber mit anderen mikrobiellen Hochdurchsatz-Nachweismethoden wie einer qPCR kombiniert werden.
Die Zusammensetzung des Acrylamid-Hydrogels kann je nach Gewebetyp und Anwendung verändert werden11. Bei instabilen Proben wie CF-Sputum ist es notwendig, die Struktur mit einem 29:1-Acrylamid:Bis-Acrylamid-Gemisch (statt nur Acrylamid) zu unterstützen. Die Aufnahme von Paraformaldehyd in das Hydrogel kann die Struktur weiter stabilisieren, wobei längere Inkubationszeiten für die Diffusion von Sonden und Antikörpern erforderlich sind9. Die Zugabe von Formaldehyd zum Hydrogel kann auch verhindern, dass das Gewebe während des Reinigungsprozesses anschwillt, wenn dieser Effekt unerwünscht ist11.
Die derzeitige Methode zielt spezifisch auf P. aeruginosa-Zellen im Sputum von Mukoviszidose-Patienten ab. Alternative Methoden und Modifikationen dieses Protokolls können in Betracht gezogen werden, um die Visualisierung anderer Bakterien zu optimieren. Durch den Einsatz von spezies- und gattungsspezifischen FISH- und Hybridisierungskettenreaktionssonden (HCR) können andere Mukoviszidose-Erreger wie Staphylococcus aureus, Streptococcus sp. und Achromobacter xylosoxidans identifiziert werden12. In unserer Studie haben wir das pseudomonale Exopolysaccharid Psl. Andere Ziele, einschließlich Alginat oder Pel, können in zukünftigen Experimenten mit fluoreszierenden Antikörpern untersucht werden, die spezifisch für diese Exopolysaccharide sind. Die Anwendung der MiPACT-Methode zusammen mit der FISH- und Antikörperfärbung für CLSM dauert einige Wochen. Betrifft die Forschungsfrage nicht die 3-dimensionale räumliche Visualisierung des Sputums, gibt es schnellere Methoden, um vorhandene Bakterien sichtbar zu machen. Bisherige Methoden zur Visualisierung von Bakterien in Sputumproben verwenden Dünnschnitte oder Abstriche und umfassen: FISH18, Gram-Färbung und immunhistochemische Techniken, bei denen primäre Antikörper und Gegenfärbungen angewendet werden, um die Visualisierung von Biofilm-Exopolysacchariden und Bakterienzellen zu ermöglichen19,20.
Es gibt mehrere potenzielle zukünftige Anwendungen dieser Art von bildgebenden Verfahren. Die Fähigkeit, verschiedene bakterielle Organismen und ihre Interaktion mit Wirtszellen, wie z.B. Fresszellen, zu visualisieren, könnte unser Verständnis dafür verbessern, warum einige P. aeruginosa-Stämme effektiv aus den Atemwegen der Mukoviszidose entfernt werden, während andere Stämme dies nicht tun. Die Bildgebung von Bakterien in Atemwegsproben kann auch als Maß für die antimikrobielle Wirksamkeit und als Studienergebnis für neue Anti-Biofilm-Medikamente verwendet werden21. Darüber hinaus kann die Visualisierung der räumlichen Beziehung zwischen P. aeruginosa und anderen Organismen innerhalb des Mukoviszidose-Lungenmikrobioms, wie z. B. Staphylococcus aureus, dazu beitragen, die Rolle der Koinfektion/Kolonisierung in der Pathogenese pulmonaler Exazerbationen und deren Ansprechen auf eine Antibiotikabehandlung aufzuklären. Die In-vivo-Bildgebung von Bakterien kann auch auf andere Infektionen angewendet werden, einschließlich solcher mit beatmungsassoziierten Pneumonien oder chronischen Wundinfektionen22. Die gewonnenen Erkenntnisse können so für die zukünftige therapeutische Entwicklung genutzt werden.
Offenlegungen
Nichts.
Danksagungen
Die Autoren danken der Cystic Fibrosis Foundation, die diese Forschung finanziert hat, und MedImmune für die großzügige Spende von Anti-Psl0096-Antikörpern. Für diese Studie wurde die Bildgebung an der CAMiLoD-Bildgebungseinrichtung der University of Toronto durchgeführt.
Materialien
| Name | Company | Catalog Number | Comments |
| 29:1 acrylamide bisacrylamide, 30 % solution | BioRad | 161-0146 | |
| 8-Chambered Coverglass Nunc Lab-Tek | ThermoFischer Scientific | 155411 | |
| Anaerogen2.5L | Oxid Inc. | 35108 | |
| Coverwell perfusion chambers | Electron Microscopry Sciences | 70326 -12/-14 | |
| HistoDenz | Sigma | D2158 | |
| Protect RNA Rnase Inhibitor | Sigma | R7387 | |
| PseaerA - GGTAACCGTCCCCCTTGC | Eurofins | Order Details: Product: Modified DNA Oligo; Name: PseaerA; Sequence: [Alexa488]GGTAACCGTCCCCCTTGC; Synthesis: 50 nmol; Purification: HPLC; Ship state: Full yield (dry) | |
| Psl0096-Texas Red | Medimmune | The Psl0096-Texas red antibodies were a gift kindly provided by Medimmune and the company should be contacted for order inquiries. | |
| VA-044 Hardener | Wako | 27776-21-21 |
Referenzen
- Banerjee, D., Stableforth, D. The treatment of respiratory pseudomonas infection in cystic fibrosis: What drug and which way. Drugs. 60 (5), 1053-1064 (2000).
- Rosenfeld, M., Ramsey, B. W., Gibson, R. L. Pseudomonas acquisition in young patients with cystic fibrosis: Pathophysiology, diagnosis, and management. Current Opinion in Pulmonary Medicine. 9 (6), 492-497 (2003).
- Ciofu, O., Tolker-Nielsen, T. Tolerance and Resistance of Pseudomonas aeruginosa Biofilms to Antimicrobial Agents-How P. aeruginosa Can Escape Antibiotics. Frontiers in Microbiology. 10, 913 (2019).
- Billings, N., et al. The Extracellular Matrix Component Psl Provides Fast-Acting Antibiotic Defense in Pseudomonas aeruginosa Biofilms. PLoS Pathogens. 9 (8), 1003526 (2013).
- Franklin, M. J., Nivens, D. E., Weadge, J. T., Lynne Howell, P. Biosynthesis of the Pseudomonas aeruginosa extracellular polysaccharides, alginate, Pel, and Psl. Frontiers in Microbiology. 2, 167 (2011).
- Yang, L., et al. Polysaccharides serve as scaffold of biofilms formed by mucoid Pseudomonas aeruginosa. FEMS Immunology and Medical Microbiology. 65 (2), 366-376 (2012).
- Wozniak, D. J., et al. Alginate is not a significant component of the extracellular polysaccharide matrix of PA14 and PAO1 Pseudomonas aeruginosa biofilms. Proceedings of the National Academy of Sciences of the United States of America. 100 (13), 7907-7912 (2003).
- Baker, P., et al. Exopolysaccharide biosynthetic glycoside hydrolases can be utilized to disrupt and prevent Pseudomonas aeruginosa biofilms. Science Advances. 2 (5), 1-9 (2016).
- Yang, B., et al. Single-cell phenotyping within transparent intact tissue through whole-body clearing. Cell. 158 (4), 945-958 (2014).
- Chung, K., et al. Structural and molecular interrogation of intact biological systems. Nature. 497 (7449), 332-337 (2013).
- Treweek, J. B., et al. Whole-body tissue stabilization and selective extractions via tissue-hydrogel hybrids for high-resolution intact circuit mapping and phenotyping. Nature Protocols. 10 (11), 1860-1896 (2015).
- DePas, W. H., et al. Exposing the three-dimensional biogeography and metabolic states of pathogens in cystic fibrosis sputum via hydrogel embedding, clearing, and rRNA labeling. mBio. 7 (5), 1-11 (2016).
- Murray, M. P., Doherty, C. J., Govan, J. R. W., Hill, A. T. Do processing time and storage of sputum influence quantitative bacteriology in bronchiectasis. Journal of Medical Microbiology. 59 (7), 829-833 (2010).
- Efthimiadis, A., Jayaram, L., Weston, S., Carruthers, S., Hargreave, F. E. Induced sputum: Time from expectoration to processing. European Respiratory Journal. 19 (4), 706-708 (2002).
- Chao, Y., Zhang, T. Optimization of fixation methods for observation of bacterial cell morphology and surface ultrastructures by atomic force microscopy. Applied Microbiology and Biotechnology. 92 (2), 381-392 (2011).
- St-Laurent, J., Boulay, M. E., Prince, P., Bissonnette, E., Boulet, L. P. Comparison of cell fixation methods of induced sputum specimens: An immunocytochemical analysis. Journal of Immunological Methods. 308 (1-2), 36-42 (2006).
- Hogardt, M., et al. Specific and rapid detection by fluorescent situ hybridization of bacteria in clinical samples obtained from cystic fibrosis patients. Journal of Clinical Microbiology. 38 (2), 818-825 (2000).
- Hoffmann, N., et al. Erratum: Novel mouse model of chronic Pseudomonas aeruginosa lung infection mimicking cystic fibrosis. Infection and Immunity. 73 (8), 5290 (2005).
- Nair, B., et al. Utility of Gram staining for evaluation of the quality of cystic fibrosis sputum samples. Journal of Clinical Microbiology. 40 (8), 2791-2794 (2002).
- Howlin, R. P., et al. Low-Dose Nitric Oxide as Targeted Anti-biofilm Adjunctive Therapy to Treat Chronic Pseudomonas aeruginosa Infection in Cystic Fibrosis. Molecular Therapy. 25 (9), 2104-2116 (2017).
- Pestrak, M. J., et al. Treatment with the Pseudomonas aeruginosa glycoside hydrolase PslG combats wound infection by improving antibiotic efficacy and host innate immune activity. Antimicrobial Agents and Chemotherapy. 63 (6), 00234 (2019).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten