Method Article
Effiziente Erzeugung und Bearbeitung von Feeder-freie IPSCs aus menschlichen Zellen der Bauchspeicheldrüse mit dem CRISPR-Cas9-System
In diesem Artikel
Zusammenfassung
Dieses Protokoll beschreibt im Detail die Generation der Fußabdruck-freie induzierten pluripotenten Stammzellen (iPSCs) aus menschlichen Zellen der Bauchspeicheldrüse in den Feeder-freien Bedingungen, gefolgt von Bearbeitung mit CRISPR/Cas9 Ribonucleoproteins und Charakterisierung der geänderten einzellige Klone.
Zusammenfassung
Embryonalen und induzierte pluripotente Stammzellen können selbst zu erneuern und in mehreren Zelltypen des Körpers differenzieren. Die pluripotenten Zellen sind somit für die Forschung in der regenerativen Medizin begehrt und werden derzeit in klinischen Studien für Augenerkrankungen, Diabetes, Herzkrankheiten und anderen Erkrankungen. Das Potential, sich in spezialisierte Zelltypen, gekoppelt mit der jüngsten Fortschritte im Genom Bearbeitung Technologien einschließlich der CRISPR/Cas-System differenzieren haben zusätzliche Möglichkeiten für die Anpassung des Genoms des iPSC für die verschiedensten Anwendungen zur Verfügung gestellt einschließlich der Krankheit Modellierung, Gentherapie, und Vorspannen Wege der Differenzierung, um nur einige zu nennen. Unter den verfügbaren Bearbeitungen Technologien hat CRISPR/Cas9 von Streptococcus Pyogenes als ein Werkzeug der Wahl für die standortspezifische Bearbeitung des eukaryotischen Genoms entstanden. Das CRISPR sind leicht zugänglich, kostengünstig und höchst effizient in technische gezielte Bearbeitungen. Das System benötigt einen Cas9-Nuklease und ein Führer-Sequenz (20-Mer) spezifisch für die genomische Ziel anschlagenden ein 3-Nukleotid "NGG" Protospacer-neben-Motiv (PAM) für die Ausrichtung der Cas9 auf die gewünschte genomischen Locus, neben einem universellen Cas9 Bindung Tracer RNA ( zusammengerufen einzelne Führer RNS oder SgRNA). Hier präsentieren wir Ihnen eine Schritt-für-Schritt-Protokoll für die effiziente Erzeugung von Feeder-unabhängig und frei von Fußabdruck iPSC und beschreiben Methoden für die Genom-Bearbeitung der iPSC mit Cas9 Ribonucleoprotein (RNP) komplexe. Das Genom Bearbeitung Protokoll ist effektiv und kann leicht durch Pre-Komplexbildner SgRNAs für mehr als ein Ziel mit dem Cas9 Protein und gleichzeitig in die Zellen liefern gemultiplext werden. Schließlich beschreiben wir einen vereinfachten Ansatz zur Identifizierung und Charakterisierung von iPSCs mit gewünschten Bearbeitungen. Zusammengenommen dürften die skizzierten Strategien zur Erzeugung und Bearbeitung von iPSC für vielfältige Anwendungen zu optimieren.
Einleitung
Die Neuprogrammierung der menschlichen Körperzellen zu pluripotenten Zustand durch Überexpression der Neuprogrammierung Faktoren hat Stammzellenforschung mit Anwendungen in der Krankheit Modellierung, regenerative Medizin und Arzneimittelentwicklung revolutioniert. Mehrere nicht-viralen Neuprogrammierung Methoden sind lieferbar Umprogrammierung Faktoren und iPSCs zu generieren, aber der Prozess ist Arbeit intensiv und nicht sehr effizient1. Die virale Methoden sind zwar effizienter, mit Problemen der Virus Integration und Tumorigenität2,3,4. In diesem Manuskript berichten wir über die Verwendung von zytoplasmatischen Sendai-Virus für liefernde Umprogrammierung Faktoren und iPSC-Fußabdruck-freie Linien, die Integration von einer viralen Vektoren Sequenzen in ihre Genome5fehlt. Sendai-Virus ist ein RNA-Virus, wird aus der Zelle Zytoplasma ~ 10 Passagen nach Infektion verdünnt und produziert Neuprogrammierung Faktoren in Hülle und Fülle, führt zu schnellen und effizienten Umprogrammierung6,7. Die etablierten iPSCs können dann ohne weiteres Feeder-freies Medium zu vermeiden die Verwendung von Maus embryonalen Fibroblasten (MEFs) als Feeder Zellen8umgestellt werden.
In dieser Publikation neben umreißt das Sendai-Virus vermittelt Neuprogrammierung erläutern wir im folgenden eine verbesserte Protokoll zur Bearbeitung iPSCs, hat das Potenzial, unbegrenzte menschliche Zellen mit gewünschten genetischen Veränderungen für die Forschung zu liefern. Wir haben CRISPR/Cas9 Technologie zur Modifikation der iPSCs, verwendet, die nun für eine breite Palette von Anwendungen, einschließlich Knock-ins und Knockouts, groß angelegte genomische Deletionen, gepoolte Bibliothek screening für gen-Entdeckung, Gentechnik verwendet wird zahlreiche Modellorganismen, und Gen-Therapie9,10,11. Diese Technik beinhaltet die Bildung von komplexen von Streptococcus Pyogenes-abgeleitete Cas9 Nuklease und 20-Mer guide RNAs, die Zielerfassung über Basis-Paarung mit genomischen Zielsequenz angrenzend an 3' Nukleotid Protospacer angrenzenden erreichen Motiv (PAM) Sequenz. Die Cas9-Nuklease induziert eine gestrandete Doppelunterbrechung ~ 3 Nukleotide aus der PAM-Website, die vorwiegend durch nicht-homologe Ende verbinden (NHEJ) Weg Insertionen oder Deletionen in den offenen Leserahmen anschließend repariert und damit funktionsfähig ist Ko von Genen12.
Unsere verbesserte Protokoll enthält die Details für die Kultur der menschlichen Zellen der Bauchspeicheldrüse, deren Neuprogrammierung auf mitotically inaktiviert Maus embryonalen Fibroblasten (MEFs) höheren Wirkungsgrad der Neuprogrammierung, nachträgliche Anpassung an Feeder-freie Kultur auf Matrigel, Charakterisierung von etablierten iPSCs geführte CRISPR RNA Design und Vorbereitung, Lieferung in iPSCs als RNP-komplexe, einzelne Zelle Sortierung klonale Linien des bearbeiteten iPSCs generieren, einfach screening und Identifizierung von Bearbeitungen und Charakterisierung der einzelne Zelle klont. Genomische Deletionen wurden in dieser Studie effizient erzeugt, durch die Einführung von Cas9 Protein und zwei CRISPR SgRNA RNP komplexe induzieren doppelte gestrandete Pausen (DSB) und segmentieren Sie löschen die einzugreifen. Diese Methode nutzt die Verwendung von zwei Guides zur Erzeugung von Löschungen in den offenen Leserahmen, hohe Leistungsfähigkeit des NHEJ führt zu geringen Anzahl von Klone dieser Notwendigkeit charakterisiert werden und einfache Vorauswahl der Klone durch automatisierte Kapillar Elektrophorese-Einheit, Fragment Analyzer. Diese effektive Genom Bearbeitungsmethoden um menschliche stammzellbasierte Krankheitsmodelle generieren werden bald ein standard und Routine Ansatz in jedem Stammzell-Labor. Zu guter Letzt präzise Genom-Bearbeitung machen es möglich, über Stammzellen Krankheit Modellierung und potenziell könnte helfen, zellbasierte Therapien zu katalysieren.
Protokoll
1. Umprogrammierung Protokoll
- Generation von menschlichen iPSC aus primären menschlichen pankreatischen Zellen
- Mantel, ein 6-Well-Platte mit 1,5 mg/mL kalte Kollagen und lassen Sie es bei 37 ° C für 1 h Gel,
- Platte frühe Passage menschliche primäre Zellen der Bauchspeicheldrüse in Prigrow III Medium (~ 1-1,5 × 10 5 Zellen) auf einer Kollagen 6-Well-Platte am Tag-2 beschichtet bis etwa 2,5 × 10 5 Zellen oder mindestens 60 % zu erreichen confluency pro auch am Tag der Transduktion (Tag 0). Für die erste Studie Platte mindestens 2-3 Brunnen um einen Brunnen mit gewünschten Konfluenz am Tag 0 zu erhalten. Mindestens eine gut als ein Steuerelement verwenden, um die Zellen zählen.
- Am Tag der Transduktion (Tag 0) warm 1 mL Prigrow III Medium in einem Wasserbad für jedes gut, ausgestrahlt werden. Die Zellen von einem Steuerelement in 1 mL 10 % FBS Medium mit 1 mL Trypsin/EDTA für 5 min gut zu ernten oder bis die Zellen zu trennen, um eine Zelle Zählung durchführen. Damit 10 % FBS Medium hinzufügen DMEM, 10 % FBS, 1 % L-Glutamin, 1 % Natrium Pyruvat und 1 % nicht-essentiellen Aminosäuren.
- Zählen die Lebensfähigkeit der Zellen mit der Zelle-Zähler zu überprüfen und berechnen Sie das Volumen jedes Virus benötigt, um dem Ziel basiert auf der Zelle Nummer und Virus-Titer zu erreichen. Verwenden der Multiplizität der Infektion (MOI) von KOS = 5, Hc-Myc = 5 und hKlf4 = 3 für die Zellen der Bauchspeicheldrüsen.
- Tauwetter ein Satz von Sendai Vektor-Röhren-80 ° C eingelagert auf Eis und 1 mL Prigrow III Medium, sorgfältig die berechneten Mengen an jedes der drei Sendai Vektor Rohre hinzufügen vorgewärmt auf 37 ° C. Pipette vorsichtig um die Lösung zu mischen. Ein Brunnen in einer 6-Well-Platte reicht für transducing mit Sendai Virenvektoren reprogrammierte Zellen generieren.
- Prigrow III-Medium aus den Zellen abgesaugt und fügen Sie die Neuprogrammierung Virus-Mischung langsam in die Vertiefungen der Zellen. Inkubation der Zellen über Nacht in einem 37 ° C Inkubator mit einer befeuchteten Atmosphäre des 5 % CO 2.
- Sorgfältig verwerfen die Virus-Mischung auf Zellen und ersetzen Sie mit frischen Prigrow III Medium am nächsten Tag, 24 h nach Transduktion. Kulturzellen für 5D und das Medium wechseln täglich zu wechseln.
- Am 5. Tag bereiten MEFs auf 0,1 % Gelatine vorbeschichtet 10 cm Kultur Gerichte (1,3 x 10 6 Zellen/Gericht). MEFs in T175 Flaschen bis 4. Tag wachsen und dann am 5. Tag, setzen die Zellen auf 6.000 rads von γ-Strahlung Quelle vor der Aussaat den Zellen 13 oder die Nutzung der kommerziellen MEF Quelle.
- Am 6. Tag, verwenden Sie 1 mL der Zelle Ablösung Lösung für 10 min lösen transduced Zellen, Platte aller MEF Gerichte im Prigrow III Medium mit Rock-Inhibitor (5 mM Lager, letzte 10 µM) und inkubieren Sie über Nacht in einem 37 ° C Inkubator mit einem befeuchteten Atmosp Re 5 % CO 2.
- Das Prigrow III Medium verändern hESC Medium am nächsten Tag. Fügen Sie für die Herstellung von 500 mL hESC Medium, DMEM/F-12, 20 % (V/V) Ko-Serum Ersatz (KOSR), 5 ng/mL bFGF; 1 mM l-Glutamin; 100 µM nicht benötigten Aminosäuren und 100 µM 2-Mercaptoethanol. Das Medium jetzt jeden Tag sanft zu verändern.
- Beobachten die Platten regelmäßig für die Entstehung der Zelle Klumpen oder Kolonien bezeichnend für reprogrammierten Zellen. Die transformierten Zellen bilden klonalen Aggregate mit Kopfsteinpflaster Morphologie und großen Kern und Kernkörperchen. Markieren Sie die wahrscheinliche ' iPSC ' Kolonien und überprüfen sie regelmäßig für Wachstum.
- Fast vier Wochen nach Transduktion Kolonien sind bereit für die Kommissionierung oder Übertragung, bereiten 24-Well MEF Platten durch Ausplattieren 1,3 x 10 6 Zellen/Gelatine vorbeschichtete Platte wie zuvor für die Übertragung der einzelnen Kolonien.
- Vorbereiten-Matrix-Membran (z.B. Matrigel) durch Aliquotierung der Verdünnungsfaktor auf dem Zertifikat der Analyse zugrunde. Hinzu kommt eine aliquote 25 mL DMEM/F-12 zu beschichten vier 6-Well-Platten (1 mL/gut) und mindestens 1 h vor Gebrauch bei Raumtemperatur (RT) inkubieren. Wählen Sie manuell 12-24 Kolonien mit einem sterilen Pipettenspitze zu aspirieren sie und übertragen auf MEF-Platten in 500 µL hESC Medium.
- Aspirieren Sie die Medien in den Brunnen zu sanft zu stören oder zu die Kolonien auseinander zu brechen. Wählen Sie auch 24-48 Kolonien auf Matrix Membran beschichtet 24-Well-Platten. Verwenden Sie für die Membran beschichtete Platten mTeSR1 (ergänzt mit 10 µM Rock Inhibitor) für 24 h zu und ändern Sie sich täglich. Innerhalb von 6-10 d der Kommissionierung Kolonien, sie sind bereit, auf die neuen 24-Well und/oder 12-Well Membran-Platten für klonale Expansion übertragen werden.
- Bei Bedarf die Kolonien auf MEFs mit 1 mg/mL Kollagenase für 20 min lösen, waschen zweimal mit hESC/mTeSR1 und Transfer zum Membran beschichtet 12 oder 24-Well-Platten. Wenn es genug robusten Kolonien wachsen auf der Matrix-Membran aus Schritt 1.1.12 gibt, Einfrieren der Kolonien auf MEFs mit iPSC Einfrieren Medien gemäß den Hersteller ' s Leitlinien.
- Lösen die robuste Kolonien auf der Membran im Schritt 1.1.12 mit 500 µL Dispase für 20 Minuten überzogen und Platte wieder auf Matrix Membran beschichtet 12-Well Platten. Manuell abkratzen differenzierten Zellen oder kontaminierenden MEF Feeder für pluripotente auf der Membran-Klone zu bereichern.
- Erweitern die Klone nach 6-8 d durch den Anbau auf Membran Platten beschichtete durch Wehre mit Dispase Lösung als vor und Plattieren kleinzelligen Aggregate auf frischen Membran 6 oder 12-Well-Platte beschichtet. Änderung mTeSR1 Medium täglich. Charakterisieren Sie die umprogrammierten Klone durch alkalische Phosphatase Färbung, Immunostaining für Pluripotenz Marker (OCT3/4 und NANOG), FACS Analyse von pluripotenten Oberflächenmarker und Differenzierung Assays. Wachsen und Kultur die Klone auf Membran-beschichtete Platten für alle Assays einschließlich CRISPR Bearbeitung wie oben erklärt, es sei denn, andere Beschichtungslösung ausdrücklich erwähnt ist.
- Charakterisierung des menschlichen iPSCs
- alkalische Phosphatase Färbung
Hinweis: eine kommerzielle Kit wird hier verwendet. Siehe Tabelle der Materialien.- Vermeintliche menschliche iPSCs auf 24-Well-Platte und Kultur für 4-5-d-Platte mit täglich Medienwechsel.
- Starten die Färbung Protokoll, Aspirieren das Nährmedium und waschen Sie die Zellen mit 1 mL 1 X PBS mit 0,05 % Tween 20 (PBST).
- 0,5 mL der Lösung in das Kit auf die Zellen hinzufügen und 2 bis 5 min. Absaugen die Lösung bei RT inkubieren und dann die festen Zellen mit PBST waschen. Lassen Sie nicht die Brunnen trocken.
- Hinzufügen 0,5 mL frisch zubereitete AP Substratlösung pro Bohrloch durch das Mischen von Lösung A, B und C. Inkubation der Zellen im Dunkeln (verpackt mit Folie oder in einem dunklen Container) bei Raumtemperatur für 5 bis 15 min.
Hinweis: Eng überwachen die Farbe zu ändern und die Reaktion zu stoppen, wenn die Farbe hell, um unspezifische Färbung zu vermeiden wird. - Die Reaktion zu stoppen, indem die AP-Substratlösung Absaugen und waschen die Brunnen zweimal mit 2 mL 1 X PBS.
- Die Kolonien unter dem Mikroskop zu beobachten und die Bilder im 4 X oder 10 X Vergrößerung mit jeder Hellfeld-Mikroskop mit einer Kamera erfassen.
- Immunostaining
- menschlichen iPSCs 24-Well-Platte und Kultur für 4-5-d-Platte mit täglich Medienwechsel.
- Zellen einmal kurz mit PBS und Fix für 20 min bei 4 % Paraformaldehyd (PFA) in PBS waschen.
- Wash dreimal für 10 min (3 X, 10 min) mit PBS bei RT
- Sättigen unspezifische Websites mit 10 % normal Ziege oder Esel Serum (NS, wuchs je nach Tier Sekundärantikörper in) mit PBS-Puffer für 40 min bei RT Für nur intrazellulären Epitope sind 0,3 % TX-100 mit PBS-Puffer (TX/PBS), permeabilize.
- Waschen mit PBS bei RT 3 x 5 min.
- Verdünnen die OCT4, NANOG, TUJ1, NKX2-5 und SOX17 Antikörper in einer frischen Lösung 5 % NS mit PBS-Puffer und inkubieren Sie für 2 h bei RT oder über Nacht bei 4 ° c
- Waschen Sie 3 X 5 min. mit PBS bei RT
- Verdünnen geeignete sekundären Alexa Fluor 488 oder 568 Antikörper in 5 % NS/PBS und inkubieren Sie Zellen für 1 h bei RT
- Waschen Sie 3 X für 5 min bei RT.
- Mit DAPI mit PBS-Puffer 1 inkubieren0 min und waschen 2 X für 5 min bei RT mit einem Fluoreszenz-Mikroskop fotografieren.
- Fluoreszenz-aktivierte Zelle sortieren (FACS)
- Platte menschlichen iPSCs in einem 6-Well-Platte und Kultur bis die Kolonien werden 80 % Zusammenfluss (mindestens 10 5 Zellen /sample) mit täglichen mTeSR1 mittlere Veränderung.
- Am Tag des Experiments, die Zellen zu isolieren und zu einer einzigen Zelle Aussetzung zu distanzieren, wie oben beschrieben in das Protokoll. Waschen mit 10 % FBS Medium.
- Für Oberflächenmarker, Aufschwemmen in 0,5-1 mL 10 % FBS Medium und Keep on Ice
- Für intrazelluläre Marker Aufschwemmen in 1 mL 4 % PFA/PBS und 10 min bei Raumtemperatur inkubieren. Waschen Sie mit 10 % FBS Medium. Aufschwemmen in 0,1 % TX/PBS und inkubieren Sie für 15 min auf Eis. Mit 10 % FBS Medium wieder waschen. Aufschwemmen in 0,5-1 mL 10 % FBS Medium und Keep on Ice
- Hinzufügen 50 µL Zellsuspension zu 50 µL 10 % FBS, mittlere enthält empfohlene Menge von TRA-1-60, TRA-1-81 oder SSEA-4 Antikörper und inkubieren Sie für 30 min bis 1 h.
- Waschen zweimal mit 10 % FBS.
- In 100 µL 10 % FBS Medium mit fluoreszierenden Sekundärantikörper Aufschwemmen und inkubieren Sie für 30 min. Waschen zweimal mit 10 % FBS und Aufschwemmen im entsprechenden Volumen von 10 % FBS. Die lebenden Zellen unterscheiden sich von abgestorbenen Zellen durch Propidium Jodid Farbstoff hinzugefügt am < 1 µg/mL, Zellapoptose während des Prozesses zu schätzen.
- Tri-Linie Differenzierung
- Samen zwei 6-Well-Platten mit 1,5-2 x 10 6 iPSCs/Brunnen in mTeSR1 Medium mit 10 µM Rock Inhibitor.
- Erlauben die Zellen wachsen für 2-3-d mit täglich mTeSR1 mittlere Veränderung, bis der Brunnen 80-90 sind % konfluierende.
- Für Ektoderm Induktion hinzufügen neurale Induktion Medium (NIM) nach Angaben des Herstellers ' s Anweisungen in 2-3 Brunnen des 6-Well-Platten.
- Ändern Sie das Medium in RPMI mit Zuschlag von 2 % B27 minus Insulin und 12 µM CHIR99021 für Mesoderm Induktion in 2-3 Vertiefungen der Platten 14. Hinzufügen nur RPMI mit Zuschlag von 2 % B27 minus Insulin für die nächsten 24 Std. hinzufügen 5 µM IWP4 RPMI Medium mit 2 % B27 ergänzen minus Insulin für die nächsten 24 h. Nach 72 h, ersetzen Sie die Medien mit RPMI mit Zuschlag von 2 % B27 führt zu der Generation schlagen Herzzellen in 2-3-d
- Für Entoderm Induktion, das Medium in Vertiefungen der 6-Well-Platten MCDB 131 ergänzt mit 1,5 g/L Natrium Bicarbonat, 1 x Glutamax, 10 mM Glukose, 0,5 % BSA, 100 ng/mL GDF8 und 5µM CHIR99021 für 24 h Kultur im gleichen Medium ohne CHIR99021 verändern für 2-3-d 15.
- Die Immunostaining Protokoll ab Schritt 2 für alle drei Linien folgen und überprüfen Sie, ob der Ausdruck der entsprechenden Markierungen.
- alkalische Phosphatase Färbung
2. Bearbeitung des menschlichen Genoms iPSC verwenden CRISPR-Cas9
- Vorbereitung von Cas9 Protein- und SgRNA
- aliquoten Cas9 Protein in Mikrozentrifugenröhrchen unter sterilen Bedingungen und speichern die Aliquote durch Einfrieren der Rohre bei-80 ° c
- Design zwei targeting Guide RNAs pro gen der Software vom MIT (http://www.genome-engineering.org/crispr/) oder jede andere CRISPR Reiseführer Design Webtool anhand. Zielen Sie auf ersten oder zweiten Exon des Gens (basierend auf offenen Leserahmen), um Knockouts zu generieren. Wählen Sie die beiden führt, so dass sie dicht beieinander geschnitten (~ 30-100 bp auseinander) und wir können einfach visualisieren die Löschung mit den gleichen Satz von screening-Primer. Wählen Sie die Hilfslinien aus der Software-Ausgabe auf der Grundlage der höheren Qualitätsfaktor und geringere Anzahl von Weg-Ziel-Sites. Design-Screening Primer mit der Programm-Grundierung-Explosion (https://www.ncbi.nlm.nih.gov/tools/primer-blast/). Screening-Primer sollte mindestens 100 bp Weg von den Cas9 schneiden Sie Websites und die PCR-Produkt-Größe sollte vorzugsweise zwischen 400-700 bp für leichter Verstärkung durch PCR.
- Design der beiden komplementären SgRNA Oligo DNAs (19-22 Nukleotide in der Länge abhängig von der Reihenfolge der Anleitung) mit einer Bsa1 geschnitten Website an jedem Ende. Die Oligo DNAs kann kommerziell synthetisiert und geglüht um Doppel-Strang DNA bilden. Für Glühen, fügen Sie 1 µL 100 µM-Guides, 25 µL Puffer (10 mM Tris, pH7.5-8.0, 50 mM NaCl, 1 mM EDTA) Glühen und 23 µL Wasser. Brüten in einem Thermocycler so programmiert, dass bei 95 ° C für 2 min und dann nach und nach Abkühlen auf 25 ° C mehr als 45 min. starten
- Klon 2 µL der resultierende Fragment in einem Bsa1 Restriktionsenzym verdaut 1 µL des hauseigenen T7 Promotor pCR2.1 Vektor mit einer Cas9-Bindungsstelle mit T4 DNA-Ligase gemäß der Hersteller ' s Protokoll.
- Sequenz die geklonte DNA-Fragmente und Treue hergestellt, in-vitro- transkribieren die Klone mit einem T7 Kit, um einzelne Guide RNAs zu generieren. Reinigen der SgRNAs mit einem kommerziellen Kit und eluieren in RNase-freies Wasser. Die RNA-Konzentration zu überprüfen.
- Transfektion von HiPSCs
- HiPSCs in mTeSR1 Medium Kultur, wie oben beschrieben, bis die Zellen 40-50 % Zusammenfluss sind.
- Zwei Stunden vor Nucleofection, ersetzen Sie das Medium mit 2 mL vorgewärmte mTeSR1 Medium mit 10 µM Rock Inhibitor.
- Eine Stunde später, bereiten Ziel Brunnen für Nucleofected Zellen durch Absaugnadeln Membran, aus 12-Well-Platte wie oben beschrieben vorbereitet und durch vorgewärmte 1 mL mTeSR1 Medium mit 10 µM Rock Inhibitor zu ersetzen. Halten Sie bei 37 ° C für die Inkubation.
- Bereiten Nucleofection master-Mix (Skala entsprechend abhängig von den Proben) für jede Probe durch Hinzufügen von 16,4 µL P3 Primärzelle Ergänzung; 3,6 µL Ergänzung 1 aus dem Nucleofector Kit; 0,5 µg des Cas9-Proteins und 0,5 µg pro sgRNA in 22 µL pro Reaktionsvolumen. pMAX GFP Vektor war auch Nucleofected gemäß der Hersteller ' s Empfehlungen in Zellen, um die Effizienz der iPSC Transfektion grob schätzen.
- Waschen jeweils gut mit 2 mL PBS RT nach Rock-Hemmer aus der iPSC-Brunnen-haltigem Medium absaugen. Aspirieren Sie PBS, fügen Sie 1 mL der Zelle Ablösung Lösung und inkubieren Sie die Platte bei 37 ° C für 10 min.
- Zellen in 3 mL des mTeSR1 Mediums Aufschwemmen und pipette vorsichtig rauf und runter um eine einzelne Zelle Aussetzung zu generieren. Transfer dissoziiert Zellen zu einer Zentrifuge 15 mL tube mit 5 mL mTeSR1 Medium.
- Zellen mit Zelle Zähler zählen und rechnen für 0,5 x 10 6 Zellen/Transfektion erforderliche Gesamtvolumen. Gewünschte Menge an Zellen in 15 mL Zentrifugenröhrchen, Zentrifugieren bei 200 X g für 5 min bei RT und aspirieren überstand.
- Aufschwemmen jede Einheit von 0,5 x 10 6 Zellen in 22 µL der Transfektion master-Mix in Schritt 4 erstellt. Schnelle Übertragung von Zellen in die zentrale Kammer einen Brunnen aus einem Nucleocuvette Streifen. Legen Sie die Streifen in eine Nucleofector Gerät und Nucleofect Zellen mit Programm CB150.
- Nach Nucleofection, schnell hinzufügen 80 µL vorgewärmte mTESR1 Medium mit 10 µM-Rock-Inhibitor in jede Vertiefung der Nucleofected Zellen. Vorsichtig mischen, von oben und unten pipettieren.
- Zellen vom Strip entfernt sanft zu übertragen, in Vertiefungen der Membran vorbeschichtete 12-Well-Platte mit mTeSR1 Medium mit Rock-Inhibitor in Schritt 3 erstellt.
- Nach 1 d, ändern Sie in frischen mTeSR1 Medium ohne Rock-Inhibitor. Ernte Zellen 2-3 d nach Nucleofection für die Sortierung von einzelligen.
- Single-Cell Isolation der gezielten HiPSCs
- einen Tag vor der Sortierung, 96-Well Platten MEF durch impfen 2 x 10 6 Zellen/Gelatine-beschichtete Platte in 10 % FBS Medium vorzubereiten. Ca. 70-80 % der Klonkrieger Überleben nach Nucleofection und 2-3 MEF Platten können für jede Bearbeitung Experiment vorbereitet werden.
- Nach der Übernachtung Inkubation ändern Mittel-bis hESC Medium (wie zuvor beschrieben) ergänzt mit 100 ng /mL bFGF, 1 x SMC4 (Inhibitoren hinzugefügt, um die Medien um einzelne Zellviabilität zu verbessern), und 5 mg/mL Fibronektin Adhäsion zu fördern.
- Ersetzen Sie das Medium auf die HiPSCs in der 12-Well-Platte aus mTeSR1 zu mTeSR1 Medium ergänzt mit 1 x SMC4 für mindestens 2 h vor dem einzigen Zellsortierung.
- Aspirieren Sie das Medium aus HiPSCs und die Zellen sanft mit PBS waschen. Fügen Sie 500 µL Zelle Ablösung Lösung in jede Vertiefung und Inkubation bei 37 ° C für 10 min nach dem Absaugen der PBS. Die einzelligen Aussetzung durch Hinzufügen von 1 mL mTeSR1 (unsupplemented) in jede Vertiefung und Pipettieren sanft nach oben und unten mehrmals zu generieren.
- Legen Zellsuspension in eine 15 mL konische Rohr und Zentrifuge für 5 min bei 200 X g bei RT Aspirat überstand und Aufschwemmen der Zellen in 1 mL mTeSR1.
- Zellen in Einzelzellen mit 100 mm Düse unter sterilen Bedingungen mit einer Zelle in den einzelnen Brunnen der 96-Well-Platten, die in Schritt 1 erstellt eine Zelle Sorter sortiert.
- Vier Tage nach dem sortieren, Kolonie Bildung sollte; erkennbar an dieser Stelle ersetzen das Kulturmedium mit hESC Medium ergänzt mit 1 x SMC4.
- Acht Tage nach dem sortieren, ersetzen Medium mit hESC Medium und Kultur für 2 d.
- Lösen die Kolonien mit 1 mg/mL Kollagenase für 20 min und übertragen Sie sie auf 24-Well-Membran beschichtete Platten in mTeSR1 mit Rock-Inhibitor. Lassen Sie die einzelligen Klone wachsen und nach duplizieren oder erweitern sie ihre genomischen DNA zu extrahieren. Gen-spezifische Primer verwenden, um das Ziel DNA durch PCR verstärken.
- Einsatz Fragment Analyzer Kit für die erste Sichtung der PCR amplifizierten genomische Zielregion alle Klone von laufen sie durch das Gel/Farbstoff mischen gemäß der Hersteller ' Anweisungen. Schätzen Sie die daraus resultierenden Fragmentgröße in der Software PROSize durch den Vergleich mit den entsprechenden Leiter, die mit den Proben ausgeführt wird. Sequenz der erwarteten ' bearbeitet ' klont und analysieren Sequenzierungsdaten um die Bearbeitung zu bestätigen oder Löschungen.
- Für die Unterscheidung zwischen monoallelisches und biallelische Klone, verstärken die bearbeitete Sequenz mit PCR und Klon in pJET1.2 Vektor. Senden Sie mindestens 8-10 Klone für die Sequenzierung und überprüfen Sie die Sequenz um zu bestätigen, Bearbeitung in einem oder beiden Allele.
- Einmal erfolgreich gezielte iPSC Klone durch Genotypisierung identifiziert wurden, zu prüfen, um zu bestätigen, dass sie nicht Pluripotenz verloren haben oder Chromosomenanomalien durch den Prozess gewonnen.
Ergebnisse
In dieser Publikation haben wir eine einfache, aber effiziente Protokoll für die Generation der iPSC aus menschlichen Zellen der Bauchspeicheldrüse mit Integration oder Fußabdruck-kostenlose Sendai Virus Vektoren verfolgt. Abbildung 1A zeigt eine schematische Darstellung dieses Neuprogrammierung Protokolls. Die menschlichen Zellen der Bauchspeicheldrüsen waren kommerziell, erworben in Prigrow III Medium kultiviert und ausgestrahlt mit Sendai-Virus wie oben beschrieben. Ausgestrahlt Spindel förmigen Zellen der Bauchspeicheldrüse zeigte keine morphologischen Änderungen für ca. 5 Tage nach Sendai Virus Transduktion am Tag 0, aber dann mit größeren Kern und Kernkörperchen abgerundet werden. Einige frühe Kolonien wurden nach einer Woche Post-Transduktion gesehen aber sie wurden nicht aufgenommen, wie in unserer Erfahrung, diese Kolonien schnell distanzieren und sind in der Regel die "frühen teilweise umprogrammierten Zellen" feststellen, dass nicht robustere Kolonien. Ca. 10-15 Tage nach dem Transduktion, kleine helle Kolonien wurden beobachtet und nach Wachstum (Abbildung 1 b, oben rechts) abgeholt wurden. Menschlichen iPSC Kolonien sind kompakt, dicht gepackt mit klaren Kanten (gilt nicht als "voll reprogrammierten Zellen") (Abbildung 1 b, Tag 23 unten in der Mitte) und 'teilweise reprogrammierte Zellen' sind locker mit Lücken in der Kolonie (Abbildung 1 b, Tag 23 unten rechts). Die umprogrammierten Pankreas Zelle Kolonien wuchsen am Tag 18 (Abbildung 1 bLinks unten) und waren groß genug, um manuell vom Tag 23 (Abbildung 1 b, unten in der Mitte) abgeholt werden. Die Kolonien wurden auf Membran-beschichteten Platten vernickelt nach der Ernte zu Feeder-freien iPSC von Tag 30-40 (Abbildung 2A, links). Einige dieser "robust" Feeder-freie Kolonien wurden erweitert und für den Ausdruck der alkalischen Phosphatase, Pluripotenz und Oberflächenmarker im Feeder-freien Bedingungen gekennzeichnet. Alle Klone getestet wurden positiv für alkalische Phosphatase, da sie leuchtend rot nach Assay (Abb. 2A, rechts) gedreht. Die Pluripotenz Marker OCT4, NANOG, SSEA4, TRA-1-60 und TRA-1-81 wurden auch beobachtet, um hoch alle Klone von Immunostaining oder FACS-Analyse (Abb. 2 b und Abbildung 3A) zum Ausdruck gebracht werden.
Diese Ergebnisse zeigen, dass die menschlichen iPSC-Kolonien, die aus Zellen der Bauchspeicheldrüse erzeugt Pluripotenz Marker in den Feeder-freien Bedingungen ausgedrückt. Die Pluripotenz wurde auch durch gezielte Differenzierung der menschlichen iPSC, drei Mikrobeschichten beurteilt: Ektoderm, Mesoderm und Entoderm. Klone auf Membran differenziert potent TUJ1-positiven Neuronen (Ektoderm), NKX2-5-positiven schlagende Herzzellen (Mesoderm) und endodermische SOX17-positiven Zellen (Abb. 3 b).
Nach gründlichen Charakterisierung wurden 3-robust, Feeder-freie iPSC klonalen Linien für das Genom Bearbeitung Studien ausgewählt. Die beiden Führungen mit BsaI Schnitt Standorte an Enden für jedes Gen entwickelt wurden, nahe beieinander zu schneiden (z.B. n weniger als ~ 100 bp). Die schematische Darstellung des Protokolls ist in Abbildung 4dargestellt. Diese Löschung Strategie verwendet für die Zielgene erlaubt uns einen Teil der ersten oder zweiten Exon eines Gens problemlos löschen. Diese Strategie war sehr effizient und wir könnten bearbeitete Klone in jeden Versuch isolieren. Die Guides waren in BsaI verdaut Vektor- und in-vitro- transkribiert wie oben beschrieben geklont. Wir Nucleofected Führer (RNAs) und Cas9 Protein als RNP-komplexe für die schnelle und effektive Bearbeitung. Wir sortiert auch die Zellen als Einzelzellen auf MEFs, wie Wiederherstellung von Zellen höher im Vergleich zu den Zellen einzeln auf Membran-beschichteten Platten sortiert ist. Genomischer DNA wurde aus alle Klone isoliert, der Ziel-Teil war durch PCR verstärkt und Fragment Analyzer zum Bildschirm für Änderungen in den Größen von Ziel-Fragmente im Vergleich zu den Steuerelementen behoben. Die Fragmentgröße war im Vergleich zu der Leiter laufen mit den Proben zu "bearbeitete" Klone (Abbildung 4, unten) zu erkennen. Das Screening der Klone als wir, durch gehen konnte leicht gemacht > 90 Klone in einer Platte und die Ergebnisse wurden mit einer Genauigkeit von ± 3 bp erhalten. Die ausgewählten Klone wurden dann bestätigen Wildtyp und mutierte Klone sequenziert. Wildtyp-Klone hatte keine Löschung oder Zugabe von Basen mutierte Klone entweder hinzufügen oder löschen in der Sequenz im Vergleich zu den Wildtyp. Die Klone zeigten Streichung oder Ergänzung in der Sequenzierung erweitert und in geklonten pJET1.2 Vektor, monoallelisches und biallelische Klone durch Sequenzierung von mindestens 8-10 Kolonien pro Klonen zu identifizieren. Monoallelisches Klone hatten Basen in einem Allel hinzugefügt oder gelöscht und der andere war unverändert und ähnlich wie das Wildtyp-Allel. Biallelische Klone hatte Löschungen in beide Allele. Rund liefen drei hundert Klone für alle Zielgene, die etwa 9 monoallelisches Löschung Klone und 3 biallelische Löschung Klone in jedem Fall identifiziert. Dies entspricht in etwa zu einer Verringerung der 3-fold in der Frequenz der Generation biallelische Klone im Vergleich zu monoallelisches-Klone. Heterogenität innerhalb der Löschung Amplifikate reflektiert unvollkommen und inkonsistente NHEJ Reparatur. Die Expression des Gens wurde schließlich durch die Gewinnung von RNA von Zielzellen und die Durchführung der RT-PCR bestätigt.

Abbildung 1: Reprogrammierung von menschlichen Zellen der Bauchspeicheldrüse, Feeder-freie pluripotente Zellen (iPSC). (A) A schematische Protokoll für die Generation der iPSC aus menschlichen Zellen in der Bauchspeicheldrüse wird angezeigt. Die Zellen der Bauchspeicheldrüsen wurden auf Kollagen-beschichteten Platten angebaut und dann auf MEFs nach Transduktion mit menschlichen Sendai Virus Vektoren übertragen. Die komplett neu programmiert Kolonien wurden dann auf hESC qualifiziert Membran übertragen und gekennzeichnet. (B) Morphological ändert sich nach Sendai Virus Transduktion. Bevor Sie die Signalweiterleitung zeigen Zellen der Bauchspeicheldrüse typische Spindel-ähnliche Morphologie (oben links, Tag 0). Kleine, enge Kolonien erscheinen auf MEFs tagsüber 12, ähnlich wie menschliche pluripotente Zelle Kolonien. Normalerweise vollständig umprogrammierten menschlichen iPSC-Kolonien haben sehr klare Grenzen und können durch Tag 23-30 abgeholt werden. Eine dissoziierte Kolonie am 23. Tag wird unten rechts angezeigt. Alle Bilder wurden bei 100 X Vergrößerung (10 X-Objektiv und 10 X Okular) aufgenommen. Maßstabsleiste = 100 µm. Bitte klicken Sie hier für eine größere Version dieser Figur.
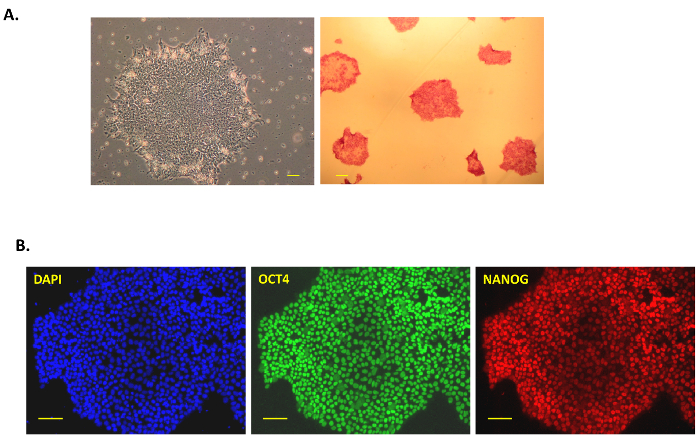
Abbildung 2: Charakterisierung der menschlichen iPSC. (A) A typische Phase Kontrast Bild einer iPSC-Kolonie nach Kommissionierung und Kultur in den Feeder-freien Bedingungen (40 X, 4 X Objektiv und 10 X Okular; links) nach Sendai Virus Neuprogrammierung des Pankreaszellen. Alkalischer Phosphatase gefärbt rot iPSC Kolonie ist auf der rechten Seite. Für die alkalische Phosphatase Färbung, die iPSC Kolonien wurden behoben und rot gefärbt, nach Zugabe von Substratlösung aus dem Kit. Die Bilder wurden bei 40 X aufgenommen. (B) Immunostaining für Pluripotenz Marker NANOG und OCT3/4 Zoll Feeder-freien menschlichen pankreatischen Zelle abgeleitet iPSCs. DAPI wurde verwendet, um den Kern als Steuerelement blau färben. Die Bilder wurden bei 100 X Vergrößerung. Maßstabsleiste = 100 µm in allen Bildern. Bitte klicken Sie hier für eine größere Version dieser Figur.
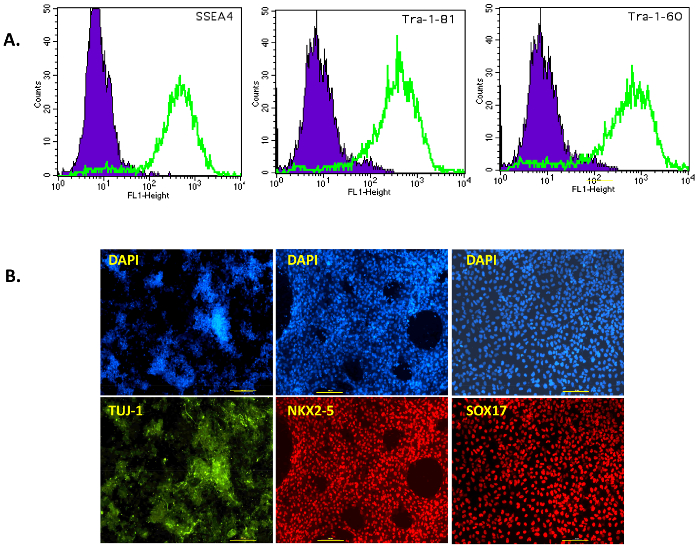
Abbildung 3: Charakterisierung der Pluripotenz und Differenzierung der iPSC. (A) FACS Analyse der Oberflächenmarker SSEA4, TRA-1-60 und TRA-1-81 im Feeder-freie iPSCs. Die Zellen wurden getrennt für einzelne Zellsuspension und gebeizt für Oberflächenmarker SSEA-4, TRA-1-60 und TRA-1-81. Die lila Spitze enthält iPSC negativ (Antikörper-Kontrolle) Zellen und Pankreas iPSC kann als grüne Gipfel visualisiert werden. (B) Immunofluorescent Bildgebung Germ Layer Markergene. Der Ausdruck der Marker TUJ1 Ektoderm (grün), NKX2-5 Mesoderm (rot) und SOX17 Entoderm (rot) in den Zellen nach gezielten Differenzierung der pankreatischen iPSC. Entsprechenden Kontrolle nuklearer Fleck DAPI ist blau. Die Bilder wurden bei 100 X Vergrößerung. Skalieren von Balken = 100 µm. Bitte klicken Sie hier für eine größere Version dieser Figur.

Abbildung 4: Schematische CRISPR/Cas9 Löschung Strategie. Ein SgRNA paar war gestaltete (rot markiert) mit BsaI geschnittenen Seiten Ende (dargestellt als F und R) vorzugsweise Ausrichtung der erste Exon geglüht, geklont in BsaI verdaut modifizierte Vektor (pCR2.1, BsaI Seite hervorgehoben grau), sequenziert und in-vitro- übersetzt. Die unterstrichenen Vektor zeigt des Teil nach BsaI Verdauung gelöscht. Die Position von screening-Primer wird in blau angezeigt. Cas9 und RNAs zu führen, waren Nucleofected im Feeder-freien iPSC als ein Komplex für genomische Deletionen. iPSCs wurden dann einzelne Zelle auf 96-Well-MEF-Platten, kultiviert, sortiert auf Membran übertragen, erweitert und mit Fragment Analyzer durchleuchtet. Für die Proben auf der Analyzer ausgeführt, wurden genomische DNA Cas9 Führer transfizierten Klone auf Membran extrahiert und durchlaufen die Gel/Farbstoff-Mix in den Analyzer für potenzielle "bearbeitete" Klone (Mitte unten) auswählen. Die zuständigen Leiter Marker, WT und potenzielle bearbeitete Klone sind gekennzeichnet. Die Expression des Gens wurde durch RNA extrahieren und mit Q-PCR Analyse bestätigt. Bitte klicken Sie hier für eine größere Version dieser Figur.
Diskussion
Umprogrammierung der menschlichen Körperzellen zu iPSCs hat einen wichtigen Impuls für die Felder der Grundlagenbiologie Forschung, personalisierte Medizin, Krankheit Modellierung, Arzneimittelentwicklung und regenerative Medizin16zur Verfügung gestellt. Viele aktuelle und am weitesten verbreitete Methoden der menschlichen iPSC Generation erfordern den Einsatz von Virus mit dem Risiko einer Integration in das Wirtsgenom oder episomal Vektoren mit geringer Effizienz Umprogrammierung. Hier präsentieren wir Ihnen eine effiziente Methode zur Erzeugung von Feeder-freie iPSCs aus menschlichen Zellen der Bauchspeicheldrüse mit Sendai-Virus, führt zu "Fußspur frei" und effiziente Umprogrammierung. Die daraus resultierenden menschlichen iPSCs sind frei von viralen transgene, Aufrechterhaltung der Pluripotenz und vermeiden Sie die Verwendung von unbekannten exogene Faktoren. Die Generation der iPSCs im Feeder-freien Bedingungen hat geringen Neuprogrammierung Effizienz, so dass wir die primäre Zellen der Bauchspeicheldrüsen auf Feeder-Zellen umprogrammiert und zog dann die Kolonien nach Feeder-freie Membran für Expansion, Kultur und Charakterisierung. Die Fusion dieser Techniken bietet stabile, verlässliche und effizienter iPSC-Programmierung.
Mit dieser Methode, was darauf hindeutet, dass Sendai-Virus verwendet werden, um die Vielzahl von Zellen neu zu programmieren wird auch iPSCs aus menschlichen Fibroblasten und andere primäre Zellen erzeugt. Wichtige Überlegungen sind: für optimale Konfluenz von Primärzellen müssen umprogrammiert werden, als zu wenige oder zu viele Zellen Auswirkungen Umprogrammierung Effizienz; eine frühere Passage von Primärzellen, wenn die Zellen noch optimal einteilen sind; sorgfältige Titration von der viralen Vektoren, dass die Ergebnisse bei minimalen Zellapoptose und einen hohen Wirkungsgrad zu einfach abholen der robuste Kolonien; überprüfen den Verlust von Sendai Vektor Transgene Ausdruck durch PCR an 10-15 Stellen um sicherzustellen, dass die iPSCs "Fußspur frei"; und streng sterilen Bedingungen für Kultur des iPSC auf Membran.
Wir verwendet eine komfortable und effiziente Methode der CRISPR Genom Änderung um das iPSC-Genom zu bearbeiten. Diese Technik kombiniert Cas9 Protein und SgRNA, RNP-komplexe zu erkennen und die komplementären DNA-Sequenzen zu Spalten. Ziel eine genomische Sequenz durch einfaches Ändern der 20 bp Guide RNA kann leicht angepasst. Unsere Methode bietet eine einfache Protokoll für effiziente und reproduzierbare Bearbeitung des menschlichen iPSCs sind arbeitsintensiver, schwer zu transduzieren und teuer im Unterhalt als andere Zellen. Wir entwerfen mindestens zwei benachbarte aber nicht überlappende Führer für jede Zielgen, sodass eine einheitliche Screening-Primer für das screening der bearbeiteten Klone verwendet werden kann. Verwendung von zwei Guides ~ 30-100 bp auseinander erzeugt darüber hinaus eine große Deletion, die leicht visualisiert werden, während der Vorauswahl und sorgt für Abbau des Proteins. Die Führungen können in HEK-293-Zellen zunächst auf Effizienz schätzen vor Beginn des Tests in iPSC getestet werden. Darüber hinaus ist es wichtig, die Parameter für die routinemäßige Transfektion von Reagenzien in iPSC zu etablieren. Mit pMAXGFP Plasmid, Transfektion Wirkungsgrade von > 80 % und Gene targeting/Störung Wirkungsgrade von 3-10 % iPSCs werden routinemäßig erreicht. Die direkte Zustellung der Cas9 RNP-komplexe in Zellen in unserem Protokoll sorgt für schnelles Handeln und schnelle Umsätze und hohe Rate der gezielten Veränderung unterhält. Unser Ansatz beschäftigt einzelne Zelle sortieren nach Nucleofection von Leitfäden und Cas9 Protein, Überwindung ein erhebliches Hindernis gen gezielt iPSC Bevölkerung vermischt und die Infektionsstaus der Zellen zu gewährleisten. Dieser "Roman" Gesamtkonzept ist unkompliziert, einfach, multiplex, dauert nur ein paar Wochen und erfordert keine antibiotische Auswahl. Wir waren nicht darauf bedacht den Verlust in Effizienz durch die Einführung von Löschungen von CRISPR sequentiell in eine Zelllinie oder iPSCs, obwohl Chromosomen Analyse muss in solchen Fällen um genomische Stabilität vorgenommen werden. Die Klone sollte auch für den Ausdruck der Pluripotenz Marker überprüft werden. Obwohl kein Schwerpunkt dieser Publikation, dieses Protokoll hinzugewählt genaue genetische Veränderung induzieren, indem unter anderem die Reparatur Vorlage in der Transfektion cocktail17,18sein kann.
Schließlich, basierend auf unserer Erfahrung, wichtige Überlegungen für iPSC bearbeiten Protokoll sind: rationales Design Guides zu minimieren oder ganz zu vermeiden aus Ziel Mutationen vor allem im Bereich"Samen" oder in der Nähe von PAM Motiv geändert wird; Optimierung der Effizienz der Nucleofection in die iPSCs um Lieferung von RNPs zu gewährleisten; Überprüfung der Qualität der vorbereiteten Guides; Titration der Führer und Cas9 Proteinkonzentrationen und Validierung ihrer Tätigkeit durch biochemische Spaltung in-vitro- Tests oder wie beschrieben in diesem Artikel im menschlichen Cellssuch als HEK Zellen, die sind einfacher zu Kultur und transfizieren; die Verwendung von frühen Durchgang iPSCs, die in der Log-Phase des Wachstums und der ~ 50 % Konfluenz zu erreichen sind; sorgfältiger Umgang mit Zellen mit sanften Medium ändert sich nach einzelnen Zelle sortieren um das Überleben der Zellen zu gewährleisten.
Wir glauben, dass die oben beschriebenen Protokoll abgegrenzt ausreichend detailliert die Leser mit einem Fahrplan zu erzeugen und Bearbeiten von iPSC in reproduzierbarer Weise ausstatten wird.
Offenlegungen
BT ist ein Gründungsmitglied der renovieren Biosciences Inc.
Danksagungen
Arbeit im Labor wurde unterstützt von Postdoc Fellowship-Stipendium an Dr. Anjali Nandal und explorative Grant aus Maryland Stem Cell Research Fund zu BT (TEDCO).
Materialien
| Name | Company | Catalog Number | Comments |
| Sendai viral vectors - CytoTune-iPS 2.0 Kit | Invitrogen | A16517 | Thaw on ice; S No: 1 |
| Trypsin EDTA | Gibco Life Tech | 25300-054 | 0.05%, 100 ml; S No: 2 |
| Rock inhibitor (Y-27632) | Milipore | SCM075 | Use 10 μM; S No: 3 |
| DMEM/F-12 medium | Invitrogen | 11330-032 | S No: 4 |
| Serum replacement (KSR) | Gibco | 10828028 | S No: 5 |
| DMEM | Invitrogen | 11960069 | 1X; S No: 6 |
| Fetal bovine serum | Thermo Scientific | SH30071.03 | Aliquot; S No: 7 |
| L-glutamine (Glutamax, 100X), liquid | Thermo Scientific | 35050061 | 1/100; S No: 8 |
| Non-Essential Amino Acids | Gibco | 11140-050 | 1/100; S No: 9 |
| 2-Mercaptoethanol | Gibco | 21985023 | 55 mM, 1/1,000; S No: 10 |
| Hausser Hemacytometers | Hausser Scientific | 02-671-54 | S No: 11 |
| 0.1% Gelatin Solution | STEMCELL Technologies | 7903 | Incubate at 37º C for 1 hour; S No: 12 |
| SSEA-4 antibody | Santacruz | sc-21704 | 1/100; S No: 13 |
| TRA-1-81 antibody | Cell Signaling | 4745S | 1/200; S No: 14 |
| OCT4 antibody | Santa Cruz | sc-5279 | 1/1,000; S No: 15 |
| Collagen I, Rat Tail | Life Technologies | A10483-01 | Keep cold; S No: 16 |
| Alexa Fluor fluorescent 488/ 568 (secondary antibodies) | Invitrogen | A21202/A10042 | 1/2,000; S No: 17 |
| DPBS | Hyclone | SH30028LS | 1X; S No: 18 |
| 100-mm tissue culture dish | Falcon | 353003 | S No: 20 |
| 96-well tissue culture plate | Falcon | 353078 | S No: 21 |
| 6-well tissue culture plate | Falcon | 353046 | S No: 22 |
| Dissecting scope | Nikon | SMZ745 | S No: 23 |
| Picking hood | NuAire | NU-301 | S No: 24 |
| 15 ml Centrifuge Tube | Greiner Bio-One | 188271 | S No: 25 |
| 50 ml Centrifuge Tube | Greiner Bio-One | 227261 | S No: 26 |
| Sodium pyruvate | Invitrogen | 11360 | S No: 28 |
| β-mercaptoethanol | Sigma | M7522 | S No: 29 |
| Prigrow III medium | ABM | TM003 | S No: 31 |
| Countess™ Cell Counter | Invitrogen | C10227 | S No: 32 |
| Faxitron X-ray system | Faxitron | CellRad | S No: 33 |
| Accutase | Innovative cell Technologies | AT-104 | S No: 34 |
| Collagenase | Life Technologies | 17104019 | 1mg/ml stock; S No: 35 |
| Dispase | STEMCELL Technologies | 7923 | S No: 36 |
| hESC qualified matrigel | BD Biosciences | 354277 | To dilute, use cold DMEM/F-12; S No: 37 |
| bFGF | R & D | 233-FB | Stock 10 ug/ml; S No: 38 |
| Paraformaldehyde | EMS | 15710 | 4% stock in PBS; S No: 39 |
| TRA-1-60 | Santa Cruz | sc-21705 | 1/100; S No: 40 |
| NANOG | ReproCELL | RCAB0004P-F | 1/100; S No: 41 |
| Tween 20 | Sigma | P9416-100ML | S No: 42 |
| Alkaline Phosphatase kit | Stemgent | 00-0055 | S No: 43 |
| Cas9 protein | PNA Bio | CP01-50 | Thaw and aliquot; S No: 44 |
| Goat or donkey serum | Sigma | D9663/G9023 | S No: 45 |
| Triton X-100 | Sigma | X100-100ML | S No: 46 |
| DAPI | Thermo Scientific | D1306 | S No: 47 |
| Tris | Sigma | 9285-100ML | S No: 48 |
| NaCL | Sigma | S7653-250G | S No: 49 |
| EDTA | Sigma | BP2482-500 | S No: 50 |
| T4 DNA ligase | NEB | M0202T | S No: 51 |
| Mega Shortscript T7 kit | Thermo Scientific | AM1354 | S No: 52 |
| Mega Clear kit | Thermo Scientific | AM1908 | S No: 53 |
| SMC4 | BD Biosciences | 354357 | S No: 54 |
| Fibronectin | STEMCELL Technologies | 7159 | S No: 55 |
| CloneJET cloning kit | Thermo Scientific | K1232 | S No: 56 |
| Fragment analyzerTM | Advanced Analytical | S No: 57 | |
| mTeSR1 medium kit | STEMCELL Technologies | 5850 | Warm to room temperature; S No: 58 |
| Freezing medium mFreSR™ | STEMCELL Technologies | 5855 | S No: 59 |
| Freezing medium CryoStor® | STEMCELL Technologies | 7930 | S No: 60 |
| MEFs | Globalstem | GSC-6301G | S No: 61 |
| L-glutamine | Invitrogen | 25030081 | S No: 62 |
| Human pancreatic cells | ABM | T0159 | S No: 63 |
| STEMdiff™ Neural Induction Medium | Stemcell Technologies | 5835 | S No: 64 |
| RPMI | Thermofisher | 11875-093 | S No: 65 |
| 2% B27-insulin | Thermofisher | A1895601 | S No: 66 |
| CHIR99021 | Stemcell Technologies | 72052 | S No: 67 |
| IWP4 | Stemcell Technologies | 72552 | S No: 68 |
| 2% B27 | Thermofisher | 17504044 | S No: 69 |
| MCDB 131 | Life Technologies | 10372019 | S No: 70 |
| Sodium bicarbonate | Sigma-Aldrich | S8761-100ML | S No: 71 |
| Glucose | Sigma-Aldrich | G8270-100G | S No: 72 |
| BSA | Proliant | 68700 | S No: 73 |
| GDF8 | Pepro-Tech | 120-00 | S No: 74 |
| TUJ1 antibody | EMD Milipore | AB9354 | S No: 75 |
| NKX2-5 antibody | Santa Cruz | Sc-14033 | S No: 76 |
| SOX17 antibody | R & D systems | AF1924 | S No: 77 |
| Propidium iodide | Thermo Scientific | P3566 | S No: 78 |
| Amaxa 4D-nucleofector™ | Lonza | AAF-1002 | S No: 79 |
| FACSAria II (cell sorter) | BD biosciences | SORP UV | S No: 80 |
Referenzen
- Malik, N., Rao, M. S. A review of the methods for human iPSC derivation. Methods Mol. Biol. 997, 23-33 (2013).
- Yu, J., et al. Induced pluripotent stem cell lines derived from human somatic cells. Science. 318, 1917-1920 (2007).
- Takahashi, K., Yamanaka, S. Induction of pluripotent stem cells from mouse embryonic and adult fibroblast cultures by defined factors. Cell. 126, 663-676 (2006).
- Sommer, C. A., et al. Induced pluripotent stem cell generation using a single lentiviral stem cell cassette. Stem cells. 27, 543-549 (2009).
- Macarthur, C. C., et al. Generation of human-induced pluripotent stem cells by a nonintegrating RNA Sendai virus vector in feeder-free or xeno-free conditions. Stem cells Int. , 564612(2012).
- Li, H. O., et al. A cytoplasmic RNA vector derived from nontransmissible Sendai virus with efficient gene transfer and expression. J Virol. 74, 6564-6569 (2000).
- Fusaki, N., Ban, H., Nishiyama, A., Saeki, K., Hasegawa, M. Efficient induction of transgene-free human pluripotent stem cells using a vector based on Sendai virus, an RNA virus that does not integrate into the host genome. Proc. Jpn. Acad. Ser. B, PhysBiol. Sci. 85, 348-362 (2009).
- Nakagawa, M., et al. A novel efficient feeder-free culture system for the derivation of human induced pluripotent stem cells. Sci. Rep. 4, 3594(2014).
- Hockemeyer, D., Jaenisch, R. Induced Pluripotent Stem Cells Meet Genome Editing. Cell stem cell. 18, 573-586 (2016).
- Komor, A. C., Badran, A. H., Liu, D. R. CRISPR-Based Technologies for the Manipulation of Eukaryotic Genomes. Cell. 169, 559(2017).
- Komor, A. C., Badran, A. H., Liu, D. R. CRISPR-Based Technologies for the Manipulation of Eukaryotic Genomes. Cell. 168, 20-36 (2017).
- Sander, J. D., Joung, J. K. CRISPR-Cas systems for editing, regulating and targeting genomes. Nat. Biotech. 32, 347-355 (2014).
- McElroy, S. L., Reijo Pera, R. A. Preparation of mouse embryonic fibroblast feeder cells for human embryonic stem cell culture. CSH Protoc. 2008, (2008).
- Lian, X., et al. Robust cardiomyocyte differentiation from human pluripotent stem cells via temporal modulation of canonical Wnt signaling. Proc. Natl. Acad. Sci. U.S.A. 109, E1848-E1857 (2012).
- Rezania, A., et al. Reversal of diabetes with insulin-producing cells derived in vitro from human pluripotent stem cells. Nat. Biotech. 32, 1121-1133 (2014).
- Shi, Y., Inoue, H., Wu, J. C., Yamanaka, S. Induced pluripotent stem cell technology: a decade of progress. Nat. Rev. Drug Discov. 16, 115-130 (2017).
- Wang, B., et al. Highly efficient CRISPR/HDR-mediated knock-in for mouse embryonic stem cells and zygotes. BioTechniques. 59, (2015).
- Liang, X., Potter, J., Kumar, S., Ravinder, N., Chesnut, J. D. Enhanced CRISPR/Cas9-mediated precise genome editing by improved design and delivery of gRNA, Cas9 nuclease, and donor DNA. J Biotech. 241, 136-146 (2017).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten