Method Article
Rekonstitution von Nukleosomen mit differentiell isotopenmarkierten Schwester Histone
In diesem Artikel
Zusammenfassung
Dieses Protokoll beschreibt die Rekonstitution von Nukleosomen unterschiedlich isotopenmarkierten Schwester Histone enthält. Zur gleichen Zeit, asymmetrisch posttranslational modifiziert Nukleosomen kann nach der Verwendung eines Histon vormodifiziert Kopie erzeugt werden. Diese Präparate können leicht zu untersuchen Modifikationsübersprechmechanismen verwendet werden, gleichzeitig auf beiden Schwester Histone, durch hochauflösende NMR-Spektroskopie.
Zusammenfassung
Asymmetrisch modifizierte Nukleosomen enthalten die beiden Kopien eines Histon (Schwester Histone) dekoriert mit unterschiedlichen Mengen von Post-translationale Modifikationen (PTM). Sie sind neu Spezies mit unbekannten Mitteln Niederlassungs- und funktionelle Auswirkungen identifiziert. Aktuelle analytische Methoden sind unzureichend, um die Kopie spezifische Auftreten von PTM auf die nukleosomalen Schwester Histone zu erkennen. Dieses Protokoll stellt ein biochemisches Verfahren zur in vitro Rekonstitution von Nukleosomen differentiell isotopenmarkierten Schwester Histone enthält. Der erzeugte Komplex kann auch asymmetrisch modifiziert werden, nachdem ein vormodifizierter Histon-Pool auch während der Histon Subkomplexe Neufaltung. Diese asymmetrischen Nukleosom Zubereitungen können leicht umgesetzt werden, mit Histon-modifizierende Enzyme Modifikation Übersprechens Mechanismen durch die asymmetrisch vorge eingebaut PTM unter Verwendung der kernmagnetischen Resonanz (NMR) -Spektroskopie auferlegt zu studieren. Insbesondere sind die Modifizierungsreaktionen inEchtzeit kann durch die Durchführung verschiedener Arten von NMR-Korrelationsexperimente, zugeschnitten für das jeweilige Isotop Typ unabhängig von den beiden Schwester Histone abgebildet werden. Diese Methode liefert die Mittel Übersprechmechanismen zu untersuchen, die zur Bildung und Ausbreitung von asymmetrischen PTM Muster auf nukleosomalen Komplexe beitragen.
Einleitung
Eukaryotische DNA ist eng in die Zellkerne in Chromatin verpackt. Der grundlegende Baustein des Chromatins ist die Nukleosomen Kernteilchen, die ~ 147 bp DNA um einen oktameren Komplex, bestehend aus zwei Kopien jeder der vier Core-Histone (H3, H4, H2A, H2B) gewickelt enthält. Histon-Proteine beherbergen eine Vielzahl von Post-translationale Modifikationen (PTM). Diese kovalente Substitutionen induzieren Veränderungen in der Chromatinstruktur, sowohl direkt durch die physikalische Chemie des Systems zu beeinflussen und indirekt durch die Einstellung von Chromatin-Remodeling - Aktivitäten 1, 2, 3. Durch diese Mittel steuern Histon PTMs Chromatin Zugänglichkeit und damit regeln alle DNA-basierten zellulären Funktionen 4.
PTM werden von Histon-modifizierenden Enzymsysteme vor allem auf die unstrukturierte N-terminalen Segmente (Schwänze) von Nukleosomen-Kern integriert histo installiertang. Aufgrund der vielen Modifikationsstellen auf der relativ kurzen Sequenz von Histon - tails beeinflussen PTMs einander durch Induzieren oder nachfolgenden Modifikationsreaktionen zu blockieren, eine Wirkung als Modifikationsübersprechens 5 bekannt. Aufgrund der insgesamt symmetrischen Architektur der Nukleosomen, Modifikationsreaktionen und Übersprechmechanismen wurden gedacht, um in ähnlicher Weise auf die zwei Kopien von jedem nukleosomalen Histon (Schwester Histone) auftreten. Dieses Konzept wurde vor kurzem in Frage gestellt und später widerlegt. Insbesondere zeigten in vitro enzymatische Tests auf freien Histon H3 Schwanz Peptide und auf Nukleosomen , dass eine Reihe von H3 - Kinasen 6 - Phosphorylierung in asymmetrischer Weise eingeführt. Zusätzlich Affinitätsreinigung basierten LC-MS / MS - Analyse zeigte das Vorhandensein von unsymmetrisch H3-methylierten Nukleosomen in verschiedene Arten von eukaryotischen Zellen 7. Somit bilden asymmetrisch modifizierten Nukleosomen neuartige Arten undWerkzeuge nötig sind, um die Mechanismen aufzudecken, die ihre Bildung steuern und die Übersprecheffekte zu analysieren, dass diese Asymmetrie ausüben könnten.
Üblicherweise wurden Western Blot (WB) und Massenspektrometrie (MS) -Analyse verwendet Histon PTMs zu detektieren. Trotz seiner einfachen Anwendung, leidet WB aus Spezifität / Kreuzreaktivität Probleme. Hinzu kommt, dass ist es nicht in der Lage 8 simultane Multi-PTM Analyse und direkte Quantifizierung der Modifikationsreaktionen durchzuführen. Auf der anderen Seite beschäftigt MS - Analyse ausgefeilte Instrumentierung , die Ausbildung auf hohem Niveau erfordert, bietet aber eine hohe Spezifität sowie gleichzeitige Kartierung und Quantifizierung von mehreren PTM 9. Jedoch sind beide Verfahren disruptive und die nukleosomalen Komplexe vor der Analyse dissoziiert, zu einer Mischung von Histonen und / oder Histon abgeleiteten Peptiden führt. Diese Manipulation entfernt die Möglichkeit, unabhängig zu unterscheidenModifikationsreaktionen, die auf jeder der beiden Schwester Histone auftreten und die Kopie spezifischen Modifikation Status der nukleosomalen Histone zu melden.
Kernspinresonanz (NMR) Spektroskopie als alternative Methode entwickelt PTM Reaktionen zuzuordnen. NMR ist eine unterbrechungsfreie und ermöglicht so die Kontrolle der PTM Ereignisse in einer Echtzeit - Weise in rekonstituierten Mischungen und sogar in intakten Zellen 10, 11. Die Entwicklung von Routinen zur schnellen Datenerfassung sowie für hochauflösende Abbildung basierend auf 2D heteronuklearen Korrelationsverfahren von isotopenmarkierten (15 N und / oder 13 C) Proben 12 erlaubt die gleichzeitige Abbildung von verschiedenen Arten von PTMs, beispiels als Serin / Threonin / Tyrosin - Phosphorylierung, Acetylierung Lysin / Methylierung und Arginin Methylierungs 13. In Abhängigkeit von der PTM untersuchten 15 N- oder 13 C-Kennzeichnung protocols kann das Protein funktionelle Gruppe zu markieren, die verwendet werden als eine Modifikation Reporter dient. Folglich kann PTM-Mapping durch Befolgen der charakteristischen chemischen Verschiebung Verschiebung der entsprechenden funktionellen Gruppe durchgeführt werden "Erfassen", um die Änderung in der chemischen Umgebung. In den meisten Fällen sowohl NH und CH chemischen Gruppen können die Entwicklung des PTM von Interesse zu berichten, verwendet werden.
Das aktuelle Protokoll beschreibt die Erzeugung von Nukleosomen unterschiedlich isotopenmarkierten Schwester Histone enthält. Es kombiniert die Flexibilität der NMR - Spektroskopie PTM zur Karte sowohl 1 H- 15 N und 1 H- 13 C - Korrelationsspektren mit der Nutzung von verschiedenen Protein - Affinität - Tags für die Reinigung der rekonstituierten ausgewählten Histon - Komplexen. Bemerkenswert ist, verwendet das Protokoll, das zwei verschiedene Pools eines bestimmten Histon für Nukleosomen Rekonstitution. Diese Pools sind unterschiedlich isotopenmarkierten (eins mit 15 N, die andere mit 13 C), und sie sind mit einem Polyhistidin fusionierte und einem Streptavidin Affinitätstag, respectively. Ein Tandem - Affinitätsreinigung Schema mit Ni-NTA und Streptavidin-basierte Chromatographie zunächst von Voigt et al. 7 verwendet wird , aus symmetrischen Pendants (1A) asymmetrische Spezies zu reinigen. Asymmetrische Histon Octamere werden anschließend zu rekonstituieren äquivalente nukleosomalen Komplexe (1B), unter Verwendung des Standardsalzdialyseverfahren 14 verwendet. Zusätzlich wird durch das gleiche Verfahren und durch eine der Histon-pools vormodifizierten aufweist, kann eine PTM asymmetrisch auf die resultierende Nukleosomen eingearbeitet werden. Die Umsetzung dieser Substrate mit Histon-modifizierende Enzyme und anschließender NMR-mapping Modifikations Ereignisse ermöglichen die Charakterisierung von Übersprechmechanismen sowohl in-cis (vormodifiziert Histon - copy) und in-trans (unmodified Histon - Kopie) (Abbildung 1C).
Protokoll
1. Rekonstitution von Nukleosomen mit differentiell isotopenmarkierten (und Asymmetrisch Modified) Schwester Histone
Hinweis: Das aktuelle Protokoll beschreibt die Rekonstitution von Nukleosomen mit unterschiedlich isotopenmarkierten Histon H3. Zu diesem Zweck wurden zwei Pools von Histon H3 verwendet wird; war 15 N-markierte und enthielt einen 6xHis-Tag am N-Terminus und die zweite war 13 C-markiertem und enthielt die Strep - Peptid (WSHPQFEK) am N-Terminus fusioniert ist . Beide Tags wurden von der nativen H3-Sequenz mit einer Tabakätzvirus (TEV) Protease-Erkennungsstelle getrennt. Zur Herstellung von Nukleosomen zusätzliche asymmetrisch geändert, einer der beiden H3-Pools in einem vormodifizierter Form enthalten. Premodification eines Histon - Pool kann durch die Umsetzung des isotopenmarkierten Histon von Interesse mit dem jeweiligen histonmodifizierende Enzym in Gegenwart der notwendigen für jede Art von Reaktions Cofaktoren / PTM Spender 1 durchgeführt werden ,6. Die effiziente Platzierung des jeweiligen PTM kann durch NMR-Spektroskopie oder Massenspektroskopie beurteilt werden. Die Mengen an Histone / DNA hier verwendet, sind grobe Empfehlungen eine Nukleosomen-Präparation mit einer ungefähren Konzentration von 10 uM zu erhalten (gemessen als DNA-Konzentration bei 260 nm) .Diese Endausbeute ist ausreichend, um gute Qualität NMR-Spektren mit relativ kurzen Erfassungszeiten aufzeichnen.
- Rekonstitution eines Histonoktamer mit unterschiedlich isotopenmarkierten (und asymmetrisch modifiziert) Histon H3
HINWEIS: Recombinant Histon - Proteine können in mg - Mengen in BL21 (DE3) pLysS - Zellen 14 hergestellt werden. An isotopenmarkierten Histonen, der gleiche Ausdruck / Reinigungsprotokoll wird gefolgt mit der Ausnahme , daß für das Bakterienwachstum einem Minimalmedium , das 15 NH 4 Cl und / oder 13 C-Glucose als Stickstoff- und Kohlenstoffquellen, beispiels 15 N- oder 13 erhalten C-Kennzeichnung respectively. Gereinigtes Histone sind gegen 5 mM β-Mercaptoethanol dialysiert und vor der Verwendung lyophilisiert gelagert.- Auflösen lyophilisierten Histon Aliquots, enthaltend ~ 5 mg jedes Histon in 1 ml Entfalten Puffer (7 M Guanidinium-HCl, 20 mM Tris pH 7,5, 10 mM DTT). Fügen Sie beide Arten von Histon H3. Mischen durch Pipettieren und nicht vortexen. Halten Sie die Röhrchen auf Eis für 30 min zu ermöglichen vollständige Entfaltung.
- Bestimmen die genaue Konzentration jedes Histon durch die Absorption bei 280 nm gemessen wurde gegen Puffer Entfalten und Verwenden Extinktionskoeffizienten berechnet mit dem ProtParam Werkzeug des ExPASy-Portal. 15
- Mischen Sie die Core-Histone zu äquimolaren Verhältnissen, unter Berücksichtigung von 50% jeder der beiden Histon H3-Pools gehören. Beginnen Sie mit den ganzen Inhalt der weniger konzentrierten Histon verwenden und dementsprechend alle anderen hinzuzufügen.
- Einstellen endgültige Proteinkonzentration auf ungefähr 1 mg / ml unter Verwendung von Puffer Entfalten.
- transfer die Mischung auf eine 6 kDa-Cutoff-Dialysemembran und dialysiert gegen 1-2 L gekühlter Rückfaltungspuffer (10 mM Tris pH 7,5, 2 M NaCl, 1 mM EDTA, 2 mM DTT) bei 4 ° C. Ändern des Puffers mindestens einmal nach der Dialyse für mindestens 3 h fortgeschritten ist. Führen Sie eine abschließende Dialyse O / N bei 4 ° C.
- Sammeln Sie dialysierte Material und entfernen Sie alle Niederschläge durch Zentrifugation in einer Mikrofuge für 5 min bei 4 ° C auf maximale Geschwindigkeit (in der Regel sollte kein Niederschlag beobachtet werden). Bestimmen Octamer Konzentration durch die Extinktion bei 280 nm gemessen wird. In den Berechnungen verwenden die zusätzlichen Extinktionskoeffizienten von Histonen, unter Berücksichtigung der doppelten jeden Wert.
- Konzentrat zu einem Endvolumen von ~ 2 ml einer 10 kDa-Cutoff-Zentrifugalfilter Einheit. Neu berechnen Oktamers Konzentration und Schätzung Verlust. An diesem Punkt wird ein Octamer Konzentration zwischen 50 bis 100 & mgr; M erhalten werden sollte.
- Schließen Sie das Gelfiltrationssäule (angegeben in der Tabelle der Materialien) zu einemFPLC-System und ins Gleichgewicht mit 2 Säulenvolumina (CV) von 0,22 um filtriert und entgast bei einer Fließgeschwindigkeit von 1 ml / min und einem Druck von 0,5 MPa Grenze Rückfaltungspuffer.
HINWEIS: Die Gelfiltrationslauf sollte im Kühlraum oder in einem kalten Schrank durchgeführt werden. - Spritzen Sie konzentrierte Material eine Durchflussrate von 1 ml / min und 1,0 sammeln - 1,5-ml-Fraktionen.
- Folgen Sie das chromatographische Profil und beobachten Histonoktamer Elution bei ~ 65-68 ml.
HINWEIS: Eine kleine Schulter des Elutionspeak und ein Peak bei ~ 80 ml weist auf die Existenz von freien H3-H4 - Tetramere und H2A-H2B Dimere sind. - Laufen 20 ul jeder gesammelten Fraktion in einer 18% SDS-PAGE-Gel.
HINWEIS: Verdünnen Sie die Proben mit einem Faktor von mindestens 2 mit Wasser , bevor sie auf das Gel laden die hohe Salzkonzentration und damit Vermeidung von Wettbewerbsverzerrungen zu verringern. Gleiches gilt für die anschließende SDS-PAGE Analysen von Proben in Puffer Neufaltung. - Pool relevant fractions mit reinem Octamere durch die gleichmäßige Verteilung der 4 Histone auf dem gefärbten Gel beurteilt und bestimmen Konzentration nach wie vor (Schritt 1.1.7).
- Verbinden einer 1 - ml - Ni-NTA - Säule (Table of Materials) mit einem FPLC - System und Äquilibrieren mit 10 CV von 0,22 um filtriert und entgast Puffer Neufaltung bei einer Fließgeschwindigkeit von 1 ml / min.
HINWEIS: Die Ni-NTA - Chromatographie sollte im Kühlraum oder in einem kalten Schrank durchgeführt werden. - Passieren gereinigter Octamer durch die Ni-NTA eine Strömungsgeschwindigkeit von 1 ml / min verwendet wird.
- Sammeln Sie Flow-Through und halten Proben für SDS-PAGE / WB-Analyse (20 & mgr; l und 4 & mgr; l, beziehungsweise). Anschließend waschen Säule mit 10 CV eines Basissignals erreicht wird, Puffer oder bis Neufaltung.
- Eluieren mit 10 CV von Rückfaltungspuffer, enthaltend 250 mM Imidazol eine Strömungsgeschwindigkeit von 1 ml / min verwendet wird. Halten Proben für SDS-PAGE / WB-Analyse (20 & mgr; l und 4 & mgr; l, jeweils).
- Äquilibrieren eine 1 ml-Affinitätssäule von acommercial Streptavidin (Table of Materials) mit 10 CV Umfaltungspufferkomponenten 250 mM Imidazol enthält eine Charge / Schwerkraftfluss Setup.
HINWEIS: Diese chromatographischen Schritt sollte im Kühlraum durchgeführt werden. - Pass Ni-NTA Elution durch die kommerzielle Streptavidin-Affinitätssäule.
- Sammeln Sie Fluss durch und halten Proben für SDS-PAGE / WB-Analyse (20 & mgr; l und 4 & mgr; l, beziehungsweise). Anschließend waschen mit 10 CV von Puffer Neufaltung.
- Eluieren mit 10 CV von Umfaltungspufferkomponenten 2,5 mM Desthiobiotin enthält. Halten Sie Proben für die SDS-PAGE / WB-Analyse (20 und 4 & mgr; l jeweils).
- Messen Sie Oktamers Konzentration, mit 10-mal weniger TEV-Protease und lassen Reaktion gehen über Nacht bei 4 ° C in einer Standard-Kunststoff-Zentrifugenröhrchen.
HINWEIS: Die notwendige Menge an TEV zu erhalten vollständigen Verdau (Entfernung der Affinitätstags) abhängig von der Herkunft (Konstrukt) und der Aktivität (frisch hergestellt wird rekombinantes TEV mehraktiv im Vergleich zu dem für längere Zeiträume bei gespeichert - 80 ° C). Versuchen Sie zunächst die Zugabe von 10-mal weniger TEV zum Oktamers verglichen (geschätzt als Proteinkonzentrationen) und entsprechend anpassen. - Check für den effizienten Verdau von 20 & mgr; l des Gemisches ausgeführt wird vor und nach der Zugabe TEV auf einem 18% SDS-PAGE-Gel. Wenn die Verdauung nicht vollständig ist, mehr TEV-Protease hinzufügen und weiter Inkubation für eine weitere 3-4 h.
- Dialysiere Probe gegen Puffer Neufaltung eine 50 kDa-Cutoff-Dialysemembran mit TEV-Protease zu entfernen und Pufferzusammensetzung anzupassen.
- Konzentriere das gereinigte asymmetrische Octamer eine 10 kDa-Cutoff Zentrifugalfiltereinheit verwendet (dieselbe Filtereinheit aus Schritt 1.1.7 können auch verwendet werden) auf ein Volumen von 1,0-2,0 ml. Messen Sie die Endkonzentration. Verwenden Sie entweder kurz nach der für Nukleosomen Rekonstitution oder anpassen, um 50% (v / v) Glycerin für einen längeren Zeitraum bei -20 ° C zu lagern. An dieser Stelle und mindestens 70% Verlust in den vorangegangenen Schritten erwartet, th50 uM - e Octamer-Konzentration im Bereich von 20 sein sollte.
- Nucleosome Rekonstitution
- Wenn Octamer in 50% Glycerin gelagert wurde, dialysiert über Nacht bei 4 ° C gegen Puffer Neufaltung bevor mit Nukleosomen Rekonstitution fortfahren.
- Passen DNA Salzkonzentration auf 2 M NaCl unter Verwendung einer 5 M NaCl-Stammlösung (eine Nukleosomen-Positionierung Sequenz enthält).
HINWEIS: Die gereinigte DNA entweder isoliert nach Reinigung eines Plasmids , das mehrere Kopien der gewünschten Sequenz synthetisiert oder durch PCR - Amplifizierung enthält konzentriert werden sollte speichernden ~ 50 & mgr; M in Wasser oder in einem Standard - Tris-EDTA - Puffer. - Mischungs Octamer und DNA unter Verwendung des optimalen Verhältnisses bestimmt von einem kleinen Test. Verwenden Rückfaltungspuffer das Endvolumen auf ~ 3,0 ml einzustellen. Fügen Sie immer die Oktamers letzte jede Möglichkeit zu verhindern, dass von der Octamer und die DNA bei <2 M Salzkonzentrationen zu mischen.
- Mit den ersten kleinen scDNA-Verhältnis, das zu einer optimalen Nukleosomen Ausbeuten führt: ale Rekonstitutionen die Oktamers zu bestimmen. Dazu montieren drei Reaktionen von 0,9: 1,0, 1,0: 1,0 und 1,1: 1,0 DNA: Oktamers Verhältnisse in einem Volumen zwischen 50 bis 100 & mgr; l. Typischerweise wird eine Konzentration von 5 & mgr; M DNA verwenden.
- Fahren Sie mit der Rekonstitution wie unten (Schritte 1.2.4-1.2.8) beschrieben und am Ende, schätzen die Menge an löslichen und gute Qualität wiederhergestellt Nukleosomen durch die DNA-Konzentration bei 260 nm gemessen und durch eine 6% nativer PAGE DNA Gel läuft , mit Ethidiumbromid gefärbt, bzw. (5A).
- Übertragen Sie die Mischung auf eine 6 kDa-Cutoff-Dialysemembran und dialysiert gegen 1 l Puffer über Nacht bei 4 ° C Neufaltung.
- Reduzieren die Salzkonzentration des Dialysepuffers in einem stufenweise von 2 auf 0,85, 0,64 und 0,2 M NaCl auf. Halten Probe in jedem Puffer für mindestens 3 h. Manchmal Niederschlag wird nach dem letzten Übergang gebildet. In diesem Fall continue Dialyse wie geplant und zu entfernen Präzipitat durch Zentrifugation Mikrofuge am Ende des nächsten Schritts.
- Transfer Dialysemembran in einem Becherglas mit 1 l Assaypuffer (25 mM NaH 2 PO 4 / Na 2 HPO 4 pH 6,8, 25 mM NaCl, 2 mM DTT) und Dialyse O / N bei 4 ° C fortgesetzt.
- Sammeln Probe, entfernen Sie alle Niederschlag bei Schritt 1.2.5 durch Zentrifugation in einer Mikrofuge für 5 min bei 4 ° C auf maximale Geschwindigkeit gebildet und konzentriert unter Verwendung eines 30 kDa-Cutoff-Zentrifugalfiltereinheit zu einem endgültigen Volumen von ~ 300 & mgr; l.
- Messen Sie DNA-Konzentration bei 260 nm, führen Sie eine 18% SDS-PAGE-Protein-Gel (4 ul der Probe) und einer 6% nativer PAGE DNA-Gel (0,2 ul der Probe) und speichern Probe bei 4 ° C für mehrere Wochen. Die Endkonzentration im Bereich von 10-20 & mgr; M sein sollte.
2. NMR-Analyse der Modifikationsreaktionen auf die beiden Schwester Histone
HINWEIS: Zuordnung2D - 1 H- 15 N - Korrelationsspektren von nukleosomalen Histonschwänze können Referenzen 16, 17, 18 zu finden. Zusätzlich Zuweisungen von CH - Gruppen von Lysine x, Serine und Threonine auf 2D - 1 H- 13 C - Korrelationsspektren können bei Referenz 16 zu finden.
- NMR - Einrichtung und Aufzeichnung von Referenzspektren
HINWEIS: Enzymatische Assays wurden durchgeführt unter Verwendung von 140 & mgr; l der Probe in Nucleosomen Assaypuffer in ein 3-mm - NMR - Röhrchen bei 300 K. Verschiedene NMR - Röhrchen mit anderen Probenvolumina verwendet werden können. Die oben erwähnte Temperatur ist angemessen guter Qualität zu erhalten 1 H- 15 N - Korrelationsspektren von nukleosomalen Histonschwänze und gute enzymatischen Aktivitäten. 2D-SOFAST-HMQC 1 H- 15 N und 2D-HSQC 1 H- 13 C - Spektren wurden jeweils mit einem Setup - tHut verleiht ähnliche Erfassungszeiten. Typische Aufnahmeparameter sind 128 Transienten mit 1,024 (1 H) x 128 (15 N) Komplexpunkten und 32 Transienten mit 1,024 (1 H) x 64 (13 C) komplexe Punkte für die beiden verschiedenen Typen von Korrelationsspektren. Für beide 2D-Spektren war die Akquisitionszeit ~ 30 min.- Auf 300 K als die Probentemperatur in dem Spektrometer. Fügen Sie 10% D 2 O (v / v) auf die Probe für das Spektrometer Frequenzverriegelung verwendet werden.
- Legen Sie die Probe in das Spektrometer und führen Sie die Grundoperationen (Verriegelung, Tuning, Shim). Darüber hinaus bestimmen die optimale Pulslängen und allgemeine Erfassungsparameter.
- Nehmen Sie 1D- und 2D - 1 H- 15 N und 1 H- 13 C - Referenzspektren.
- Prozessreferenzspektren und sicherzustellen, daß die Signale zum Abbilden der Modifizierungsreaktionen verwendet werden, werden gut aufgelöst und haben gute Intensitäten.
- Recover Probe und gibt it zu einem Mikrozentrifugenröhrchen.
- Aufbau der enzymatischen Reaktion, NMR - Kontrolle und quantitative Analyse
- Kopieren und vereinbaren Sie eine Reihe von verschachtelten 2D - 1 H- 15 N und 1 H- 13 C - Spektren. Ziel für eine Gesamterfassungszeit von mehreren Stunden und entsprechend anpassen, in einem neuen Durchlauf auf der Basis der enzymatischen Raten aus der ersten Reaktion erhalten.
- In der Probe , die notwendigen Cofaktoren für die jeweilige Art der Modifizierungsreaktion (1 mM ATP / 2 mM MgCl 2 für die Phosphorylierung, 1 mM Acetyl-CoA für die Acetylierung, 1 mM SAM für Methylierung, etc.).
- Fügen Sie das Enzym von Interesse, mischen durch Pipettieren, übertragen Sie die Probe wieder in das NMR-Röhrchen und anschließend in das Spektrometer (führen diese Operationen schnell).
HINWEIS: Wenn es keine Daten über die zu erwartende Enzymaktivität ist, passen die Substrat-zu-Enzym - Molverhältnis 10: 1. Führen Sie eine erste Probelauf und entsprechend anpassen, fürder eigentliche Lauf. - Führen Sie eine schnelle automatische Shim und den Rest von Parametern aus den Referenzspektren verwendet werden.
- Starten Sie die Serie der verschachtelten 2D - 1 H- 15 N und 1 H- 13 C - Spektren.
- Folgen Sie dem Verlauf der Reaktion in Echtzeit und der Erwerb stoppen, wenn die Beendigung der Reaktion oder eine gewünschte Niveau erreicht.
- Verfahren Spektren wie zuvor mit genau denselben Parametern.
- Wiederholen Sie den gesamten Lauf eine andere Erfassungsreihenfolge für die beiden Arten von Korrelationsspektren. Wenn im ersten Versuch ein 1 H- 15 N Korrelation erste in der Reihe war, beginnen jetzt mit einem 1 H- 13 C - Korrelation. Durch diese und für beide Spektren gleiche Erfassungszeit ist, ist es möglich, PTM Reaktionen auf beiden unterschiedlich isotopenmarkierten Histonen auf der gleichen Zeitskala zu planen und zu vergleichen. Darüber hinaus ist es möglich, Mittelwerte und Plot Fehlerbalken zu berechnen.
HINWEIS: Ein thorough Beschreibung der Methoden für die NMR - Signalanalyse und anschließende Quantifizierung der enzymatischen Reaktionen können hier 19 zu finden. - Wählen NMR Signale von jedem der Zeitverlauf-NMR-Spektrum, die das Fortschreiten der Reaktion berichten und berechnen ihre relativen Signalintensitäten eine Visualisierung und Analyse-Software für die NMR-Spektren verwendet wird. Tun Sie dies für Signale, die die unmodifizierte sowie den modifizierten Zustand melden. Extrahieren Sie Modifikationsstufen.
- Zeichnen Sie die berechneten Werte der Modifikationsstufen gegen die Reaktionszeit.
Ergebnisse
Richtig rückgefaltetem oktamere Arten werden nach einem Durchlauf der Rekonstitution Mischung durch eine Gelfiltrationssäule (Abbildung 2) isoliert. Die gebrauchsfertige oktameren Pool die drei verschiedenen Arten von Octamere enthält, wird auf dem Tandem-Affinitätsreinigung Schema unterworfen. Proben werden aus allen Schritten und durch SDS-PAGE und anschließend WB gesammelt. Überprüfung der korrekten Ausführung des Protokolls , das bei der Isolierung von asymmetrischen Spezies führt , wird durch Verfolgen des Vorhandenseins der beiden Affinitätsmarkierungen (His- und Strep-) in den verschiedenen Proben (Figur 3) erreicht. Anschließend werden die Affinitäts - Tags nach der TEV - Protease - Verdau (Figur 4) entfernt wird . Asymmetrische Octamere Affinitätsmarkierungen angeordnet sind, verwendet asymmetrische Nukleosomen zur Rekonstitution. Zunächst wird ein kleiner Testmaßstab Rekonstitution verwendet, um das optimale molare Verhältnis von DNA zu bestimmen: Octamer, dass in einem hohen ErgebnisseAusbeute Bildung eines monodispersen Komplexes. Anschließend wird die optimierte Mischungsverhältnis eingesetzt Nukleosomen in einem großen Maßstab zu rekonstituieren. Nukleosomen - Rekonstruktionen werden durch Protein / DNA - Gelen und NMR - Spektroskopie für die richtige Montage und die Anwesenheit der differentiell isotopenmarkierten Schwester H3 Histone bzw. (Abbildung 5) analysiert. Ein Anwendungsbeispiel ist in Abbildung 6 dargestellt. Insbesondere wird differentiell isotopenmarkierte phosphorylierte und asymmetrisch auf H3S10 Nukleosomen mit Gcn5 acetyltransferase umgesetzt und Acetylierung von H3K14 auf beiden H3 Kopien in Echtzeit durch NMR-Spektroskopie verfolgt. Die Analyse zeigt , dass H3S10 Phosphorylierung H3K14 Acetylierung von Gcn5 in cis im Vergleich zu in trans stimuliert.

Abbildung 1: Flussdiagramm für die Herstellung von differentiell Isotopen-labeled (und Asymmetrisch modifiziert) Nukleosomen. (A) Rekonstitution und Tandem - Affinitätsreinigung von differentiell isotopenmarkierten (und asymmetrisch modifiziert) Histon Octamere. (B) Nucleosome Rekonstitution durch die gereinigten asymmetrischen Octamere kombiniert und eine DNA - Nukleosomen-Positionierung Sequenz. (C) NMR - Überwachung von in-cis und in-trans - Modifikation Übersprechens nach der asymmetrisch verändert und unterschiedlich isotopenmarkierten Nukleosomen mit Histon-modifizierenden Enzyme mischen. Bitte klicken Sie hier , um eine größere Version dieser Figur zu sehen.
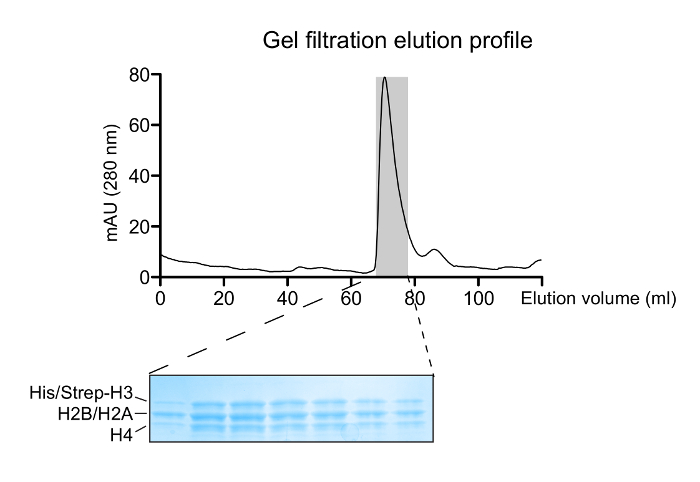
Abbildung 2: Rekonstitution von Histon Oktamere. Eine Gelfiltration Elutionsprofil der rückgefaltetem Histon Mischung. Der Hauptpeak bei ~ 70 mL entspricht den Histon Octamere. A 14 bis 20% Gradienten-SDS-PAGE-Gel (Coomassie-Blau-Färbung) der eluierten Fraktionen mit dem Octamer Spitze entspricht zeigt die korrekte Stöchiometrie Histon. Bitte klicken Sie hier , um eine größere Version dieser Figur zu sehen.

Abbildung 3: Reinigung von Asymmetric Histone Oktamere. 14-20% Gradienten-SDS-PAGE-Gel (Coomassiefärbung) und WB gegen die His- und Strep- Affinitätstags von Proben aus den verschiedenen Stufen des Tandemaffinitätsreinigung Schema. Bitte klicken Sie hier , um eine größere Version dieser Figur zu sehen.

Abbildung 4: Entfernen der Affinity - Tags. 18% SDS-PAGE-Gel (Coomassie-Färbung) gereinigtes Histon asymmetrisches Octamere vor und nach mit der TEV-Protease vermischt wird. Bitte klicken Sie hier , um eine größere Version dieser Figur zu sehen.

Abbildung 5: Biochemische und NMR - Charakterisierung von differentiell isotopenmarkierten Nukleosomen. (A) 6% nativer PAGE DNA - Gel (Ethidiumbromidfärbung) eines kleinen Test-Skala Nukleosomen Rekonstitution unterschiedliche Molverhältnisse von DNA unter Verwendung von : Oktamers (links). 6% nativer PAGE DNA-Gel (Ethidiumbromidfärbung) eines großen Nukleosomen Rekonstitution der optimierten DNA unter Verwendung von: Oktamers Molverhältnis (rechts). (B) 18% SDS-PAGE - Protein - Gel (Coomassiefärbung) der rekonstituierten Nukleosomen zeigt die korrekte Stöchiometrie der vier Core - Histone. (C) 1 H- 15 N SOFAST-HMQC - Spektrum der 15 N-markierten H3 Kopie unterschiedlich isotopenmarkierten Nukleosomen (nur H3 Schwanz Reste sind sichtbar - der Proteinkern aufgrund der langsamen Taumeln Rate unsichtbar ist). (D) 1 H- 13 C - HSQC - Spektrum der 13 C-markierten H3 Kopie differentiell isotopenmarkierten Nukleosomen. Bitte klicken Sie hier , um eine größere Version dieser Figur zu sehen.
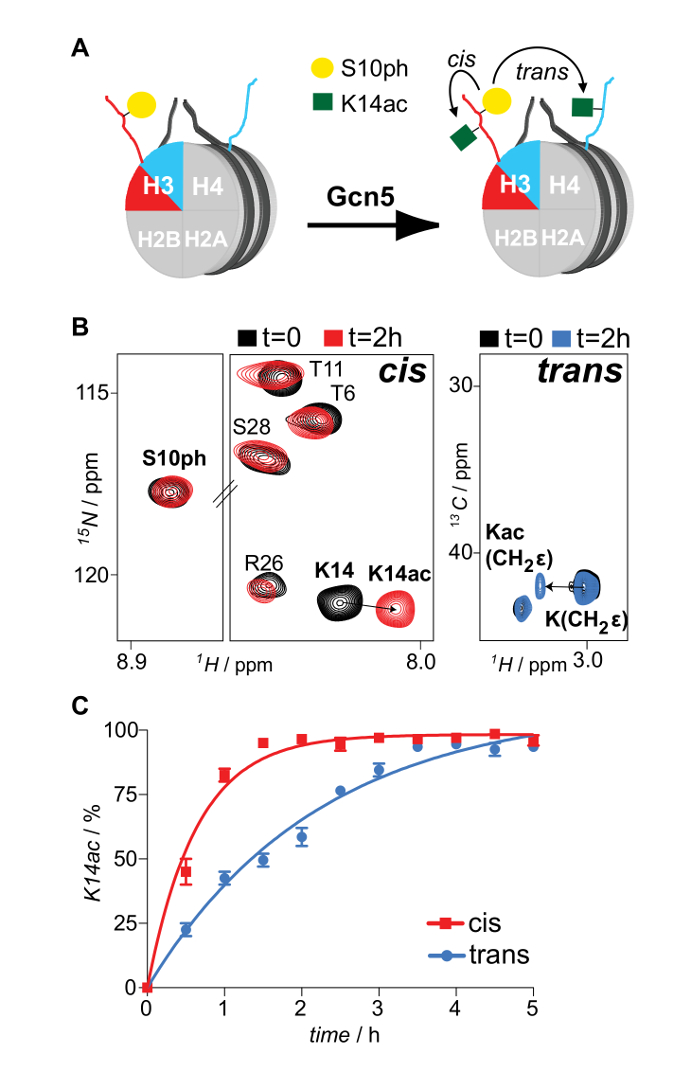
Abbildung 6: NMR Überwachung von In cis und trans Modifikation Übersprechen durch asymmetrische Phosphorylierung von H3S10 auf der Acetylierung Aktivität von Gcn5 Acetyltransferase H3K14 Imposed. (A) Schematische Darstellung von unterschiedlich isotopenmarkierten und asymmetrisch phosphoryliert auf H3S10 Nukleosomen reagierte mit Gcn5 Acetyltransferase und H3K14 Acetylierung Kartierung, in cis und trans. (B) 1 H- 15 N-SOFAST HMQC und 1 H- 13 C - HSQC - Spektren (ausgewählten Regionen) der asymmetrischen Nukleosomen vor und nach der Reaktion mit Gcn5. (C) Zeitaufgelöste NMR - Überwachung von H3K14 Acetylierung durch Gcn5 asymmetrisch auf H3S10 phosphoryliert Nukleosomen. Die Mittelwerte und Variations (Fehlerbalken) aus zwei unabhängigen Experimenten werden gezeigt. Mit Genehmigung 16. Bitte klicken Sie hier , um eine größere Version zu sehendiese Figur.
Diskussion
Für Nukleosomen Rekonstitution verwendet das aktuelle Protokoll , um eine 165 bp lange DNA - Vorlage , um die 601-Widom Nukleosomen Positioniersequenz 20, aber eine ähnliche Leistung wird erwartet , dass mit verschiedenen Längen von DNA - Matrizen enthalten. Das Protokoll wurde entworfen und verwendet unter Verwendung asymmetrischer Arten von Histon H3. Mit dem gleichen Prinzip kann das Verfahren auch für die anderen Core-Histone angewendet werden und zusätzlich verwendet werden können Komplexe, die zwei unterschiedliche Core-Histone mit unterschiedlichen Isotopenmarker zu rekonstituieren. Das Protokoll kann auch modifiziert werden, leicht zu anfangs Histon-Unterkomplexe Rekonstitution und nicht direkt Octamere Histon. Nach dieser Modifikation asymmetrische H3-H4 Tetramere können durch die Verwendung der gleichen Tandem Reinigungsschema wiederhergestellt werden. Letztere sind mit getrennt rekonstituiert H2A-H2B Dimere gemischt und DNA Nukleosomen 16 zu montieren. Die endgültigen Ausbeuten und die Gesamtleistung des modifizierten Protocol sind ähnlich wie die ursprüngliche.
Das Protokoll ist stark abhängig von der Fähigkeit der beiden Affinitätssäulen effizient die betreffenden Arten zu binden und so am Ende hochreine asymmetrische Komplexe zu ergeben. Es ist mühsam, direkter und konkreter Nachweis zu erbringen, dass die endgültige gereinigte Komplex ist in der Tat und nur asymmetrisch. Jedoch WB - Analyse von Proben aus allen Reinigungsschritten mit den entsprechenden Antikörpern liefert zuverlässige Indikation für den Erfolg des Tandem - Reinigungsschema (Abbildung 3). Die gleichen Ergebnisse und damit zusätzliche Sicherheit, wurden durch die Analyse der gleichen Proben auf das Vorhandensein der Affinitätstags durch NMR - Verfahren 16 erhalten. Dennoch, wenn WB-Analyse Kontamination asymmetrischer Art mit symmetrischen diejenigen anzeigt, kann eine kleine Variation in dem ersten Reinigungsschritt angewendet werden (Ni-NTA), die das Ergebnis zu verbessern. Insbesondere kann anstelle einer isokratischen Elution, ein Imidazol gradien t Elution (10-250 mM) eingesetzt und dann werden nur die ersten Elutionsfraktionen, die in den asymmetrischen Spezies hoch angereichert sind, werden vereinigt und führen Sie durch die Affinitätssäule. Dies kann durch WB-Analyse der Elutionsfraktionen auf die Anwesenheit des Strep Affinitäts-Tag bewertet.
Die Auswahl des Isotops Zeichens in Bezug auf das PTM von Interesse ist ziemlich flexibel , da für die meisten Fälle, NMR das gleiche PTM der Kartierung unter Verwendung von sowohl 1 H- 15 N und 1 H- 13 Ccorrelation Spektren der Lage ist. Jedoch ist für bestimmte Aminosäuren, wie Lysine, die chemische Verschiebung Dispersion für die Seitenkette 1 H- 13 C - Resonanzen ist ziemlich begrenzt. Zusätzlich wird, wenn die Zielstellen eines Lysin-modifizierenden Enzym nicht bekannt sind, ortsspezifische Auslesens von Lysin PTMs ist nicht einfach. In diesen Fällen beschäftigen alternative Methoden ortsselektive 13 C-Markierung, in Kombination mit halbsynthetischen Histon - Produktion"> 21 oder die Verwendung von neu NMR - Pulssequenzen aufgebaut , die spezifische Detektion von Modifikationszustände durch selektive Anregung ermöglicht Routinen . 22 NMR - Spektroskopie verleiht eher geringe Empfindlichkeit. In dieser Hinsicht relativ hohe Mengen an Nukleosomen (uM - Bereich) sind erforderlich , die auszuführen enzymatische Reaktionen. Dies verhindert die Entwicklung des Verfahrens auf ein Hochdurchsatzaufbau.
Gleichzeitig wurde ein neues chemisches Verfahren zur Synthese von chemisch reinen, asymmetrisch modifizierten Nukleosomen, mit hohen Ausbeuten eingeführt, definiert PTMs Durchführung 23. Dieser Ansatz ist verwendet worden, in Kombination mit Fluorographie, PTM Übersprechmechanismen zu untersuchen. Aufgrund der Niedrigauflösungsfähigkeit von fluorographischen Methoden in PTM Erfassung wurden Schlussfolgerungen abgeleitet indirekt auf eine Reihe von Nukleosomen tragen verschiedene Kombinationen von symmetrischen und asymmetrischen PTM Gesamtenzymaktivitäten zu vergleichen, eherals durch direkte Beobachtung von PTM Reaktionen auf die entsprechenden Stellen von jeder Schwester Histon. Jedoch Einbau von isotopenmarkierten Histonen und die Verwendung von NMR-Spektroskopie zur PTM Auslese könnte das vorgenannte Verfahren ein zusätzliches Werkzeug für die hochauflösende Analyse PTM asymmetrisch modifizierten Nukleosomen bilden.
Im Zusammenhang mit der Identifizierung von neuen Typen von asymmetrisch modifizierten Nukleosomen in Zukunft soll die aktuelle Methode zur Analyse von in cis ein hochauflösendes Werkzeug zu bilden und in trans PTM Übersprechens Reaktionen auf nukleosomalen Substraten. Insbesondere von hoher Bedeutung ist die Decodierung des Übersprechens Mechanismen, die zu bivalenten Nukleosomen 24 geben , die asymmetrisch enthalten, beide transcriptionsaktive und repressive PTM.
Offenlegungen
The author has nothing to disclose.
Danksagungen
Der Autor dankt Dr. Philipp Selenko (FMP-Berlin) für die Nasslaborfläche Bereitstellung von Infrastruktur und zur Durchführung Experimente und die Deutsche Forschungsgemeinschaft (DFG) für die Förderung der Arbeit durch ein Forschungsstipendium (LI 2402 / 2-1).
Materialien
| Name | Company | Catalog Number | Comments |
| Guanidinium HCl | Applichem | A14199 | Use high quality Gu-HCl |
| Tris | Roth | 4855.2 | |
| DTT | Applichem | A1101 | |
| NaCl | VWR chemicals | 27810.364 | |
| EDTA | Roth | 8043.2 | |
| Na2H2PO4 | Applichem | A1046 | |
| NaH2PO4 | Applichem | A3902 | |
| Imidazole | Applichem | A1073 | |
| d-Desthiobiotin | Sigma | D1411 | |
| Ni-NTA Superflow | Qiagen | 1034557 | |
| Strep-Tactin Superflow | IBA | 2-1207-001 | |
| His-probe antibody | Santa Cruz | sc-8036 | |
| Strep-tactin conjugated HRP | IBA | 2-1502-001 | |
| Hi-Load 16/600 Superdex 200 pg | GE Healthcare | 28-9893-35 | |
| 6 - 8 kDa dialysis membrane | Spectrumlabs | spectra/por 1, 132650 | |
| 50 kDa dialysis membrane | Spectrumlabs | spectra/por 7, 132129 | |
| 10 kDa centrigugal filter unit | Merck Millipore | UFC901024 | |
| 30 kDa centrigugal filter unit | Merck Millipore | UFC903024 | |
| Solution-state NMR spectrometer | at least 500 MHz operating frequency, equipped with a triple-resonance cryoprobe | ||
| Buffer name | Component | ||
| Unfolding Buffer | 7 M Guanidinium HCl | ||
| 20 mM Tris, pH 7.5 | |||
| 10 mM DTT | |||
| Refolding Buffer | 10 mM Tris, pH 7.5 | ||
| 2 M NaCl | |||
| 1 mM EDTA | |||
| 2 mM DTT | |||
| Assay Buffer | 25 mM Na2H2PO4, pH 6.8 | ||
| 25 mM NaCl | |||
| 2 mM DTT |
Referenzen
- Hansen, J. C. Conformational Dynamics of the Chromatin Fiber in Solution: Determinants, Mechanisms, and Functions. Annu. Rev. Biophys. Biomol. Struct. 31, 361-392 (2002).
- Clapier, C. R., Cairns, B. R. The Biology of Chromatin Remodeling Complexes. Annu. Rev. Biochem. 78, 273-304 (2009).
- Musselman, C. A., Lalonde, M., Cote, J., Kutateladze, T. G. Perceiving the epigenetic landscape through histone readers. Nat. Struct. Mol. Biol. 19 (12), 1218-1227 (2012).
- Suganuma, T., Workman, J. L. Signals and Combinatorial Functions of Histone Modifications. Annu. Rev. Biochem. 80, 473-499 (2011).
- Lee, J. S., Smith, E., Shilatifard, A. The Language of Histone Crosstalk. Cell. 142 (5), 682-685 (2010).
- Liokatis, S., et al. Phosphorylation of histone H3 Ser10 establishes a hierarchy for subsequent intramolecular modification events. Nat. Struct. Mol. Biol. 19 (8), 819-823 (2012).
- Voigt, P., et al. Asymmetrically Modified Nucleosomes. Cell. 151 (1), 181-193 (2012).
- Egelhofer, T. A., et al. An assessment of histone-modification antibody quality. Nat. Struct. Mol. Biol. 18 (1), 91-93 (2011).
- Huang, H., Lin, S., Garcia, B. A., Zhao, Y. Quantitative Proteomic Analysis of Histone Modifications. Chem. Rev. 115 (6), 2376-2418 (2015).
- Liokatis, S., Dose, A., Schwarzer, D., Selenko, P. Simultaneous Detection, of Protein Phosphorylation and Acetylation by High-Resolution NMR Spectroscopy. J. Am. Chem. Soc. 132 (42), 14704-14705 (2010).
- Binolfi, A., et al. Intracellular repair of oxidation-damaged α-synuclein fails to target C-terminal modification sites. Nat. Commun. 7, 10251(2016).
- Schanda, P., Kupce, E., Brutscher, B. SOFAST-HMQC experiments for recording two-dimensional heteronuclear correlation spectra of proteins within a few seconds. J. Biomol. NMR. 33 (4), 199-211 (2005).
- Theillet, F. X., et al. Cell signaling, post-translational protein modifications and NMR spectroscopy. J. Biomol. NMR. 54 (3), 217-236 (2012).
- Dyer, P. N., et al. Reconstitution of Nucleosome Core Particles from Recombinant Histones and DNA. Methods Enzymol. 375, 23-43 (2004).
- Artimo, P., et al. ExPASy :SIB bioinformatics resource portal. Nucleic Acids Res. 40 (W1), 597-603 (2012).
- Liokatis, S., klingberg, R., Tan, S., Schwarzer, D. Differentially Isotope-Labeled Nucleosomes to Study Asymmetric Histone Modification Crosstalk by Time-Resolved NMR Spectroscopy. Angew. Chem. Int. Ed. 55 (29), 8262-8265 (2016).
- Stützer, A., et al. Modulations of DNA Contacts by Linker Histones and Post-translational Modifications Determine the Mobility and Modifiability of Nucleosomal H3 Tails. Mol. Cell. 61 (2), 247-259 (2016).
- Zhou, B. R., et al. Histone H4 K16Q Mutation, an Acetylation Mimic, Causes Structural Disorder of Its N-terminal Basic Patch in the Nucleosome. J. Mol. Biol. 421 (1), 30-37 (2012).
- Theillet, F. X., et al. Site-specific NMR mapping and time-resolved monitoring of serine and threonine phosphorylation in reconstituted kinase reactions and mammalian cell extracts. Nat. Protoc. 8 (7), 1416-1432 (2013).
- Lowary, P. T., Widom, J. New DNA sequence rules for high affinity binding to histone octamer and sequence-directed nucleosome positioning. J. Mol. Biol. 276 (1), 19-42 (1998).
- Hackenberger, C. P., Schwarzer, D. Chemoselective ligation and modification strategies for peptides and proteins. Angew. Chem. Int. Ed. 47 (52), 10030-10074 (2008).
- Theillet, F. X., et al. Site-specific mapping and time-resolved monitoring of lysine methylation by high-resolution NMR spectroscopy. J. Am. Chem. Soc. 134 (18), 7616-7619 (2012).
- Lechner, C. C., Agashe, N. D., Fierz, B. Traceless Synthesis of Asymmetrically Modified Bivalent Nucleosomes. Angew. Chem. Int. Ed. 55 (8), 2903-2906 (2016).
- Bernstein, B. E., et al. A bivalent chromatin structure marks key developmental genes in embryonic stem cells. Cell. 125 (2), 315-326 (2006).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten