Method Article
核小体的重建与差异同位素标记的姐妹组蛋白
摘要
这个协议描述了含有差异同位素标记的妹妹组蛋白核小体重构。同时,可使用premodified组蛋白拷贝之后产生不对称翻译后修饰的核小体。这些制剂可以很容易地用于研究修改串扰机制,同时在两个姐姐组蛋白,通过使用高分辨率核磁共振光谱。
摘要
非对称修饰的核遏制饰有不同的集合翻译后后修饰的组蛋白的两个副本(妹妹组蛋白)。他们是新发现的物种,建立和功能影响未知的手段。目前的分析方法不足以检测的核小体组蛋白姐姐翻译后修饰的特定复制发生。这个协议提出了含差异同位素标记的妹妹组蛋白核小体在体外重构的生物化学方法。生成复杂的可也不对称修饰,组蛋白subcomplexes的折叠过程中包括组蛋白premodified池之后。这些非对称核制剂可以容易地与组蛋白修饰酶来研究使用核磁共振(NMR)谱由非对称预掺入PTM施加变形串扰机制反应。特别是,在修饰反应实时可以独立的两个姊妹组蛋白通过进行不同类型的核磁共振相关的实验中,对于各同位素类型量身定做进行映射。这种方法提供了手段来研究有助于不对称PTM方式对核小体复合物的形成和传播机制的串扰。
引言
真核生物DNA在细胞核内的紧密包装成染色质。染色质的基本构建块是一个包含DNA的周围由四个核心组蛋白的两个副本(H3,H4,H2A,H2B)的八聚体复合物包裹的〜147碱基对的核小体核心颗粒。组蛋白窝藏翻译后后修饰过多。这些共价的取代,都直接通过影响该系统的物理化学和间接通过招募染色质重塑活动1,2,3诱导染色质结构的改变。通过这些手段,组蛋白翻译后修饰控制染色质的无障碍,因此,调节所有基于DNA的细胞功能4。
翻译后修饰是由组蛋白修饰酶系统主要是对核小体纳入核心组织相容的非结构化N末端段(反面)安装网元。由于对组蛋白尾部的相对短的序列的许多修饰位点,翻译后修饰是通过诱导或阻断后续修饰反应,被称为修饰串扰5的效果相互影响。因为核小体的整体对称结构的,改性反应和串扰机制被认为类似地发生在每个核小体组蛋白(姐姐组蛋白)的两个副本。这个概念最近被质疑和反驳其后。特别是,关于自由组蛋白H3尾部的肽和在核小体的体外酶测定法表明,一组H3激酶以不对称的方式6中引入磷酸。此外,基于亲和纯化的LC-MS / MS分析表明在几种类型的真核细胞7的不对称H3甲基化的核小体的存在。因此,非对称改性的核构成新种,并需要工具来揭开该控制它们的形成和分析串扰效应,这种不对称可能发挥的机制。
通常,免疫印迹(WB)和质谱(MS)分析已被用来检测组蛋白翻译后修饰。尽管其应用方便,WB从特异性/交叉反应问题的困扰。最重要的是,它不能执行同步多PTM分析和修饰反应8的直接定量。另一方面,MS分析采用了精密的仪器,需要高水平的训练,但可提供高特异性以及同时映射和量化多个翻译后修饰9。 然而,这两种方法都是破坏性和核小体配合物分析之前被离解,从而产生的组蛋白和/或组蛋白衍生的肽的混合物。这种操作删除分辨独立的能力上每两个姐姐组蛋白的发生,并报告该核小体组蛋白的特定复制修改状态修饰反应。
演变作为一种替代方法,以映射的PTM反应核磁共振(NMR)谱。核磁共振是无中断的,因此允许PTM事件的实时方式中重构的混合物的监视,甚至在完整细胞10,11。例程快速数据采集,以及用于高分辨率映射基于二维发展同位素标记(15 N和/或13 C)的样品的异质核相关方法12允许不同类型的翻译后修饰的的同时映射,如如丝氨酸/苏氨酸/酪氨酸磷酸化,赖氨酸乙酰化/甲基化,和精氨酸甲基13。根据PTM正在调查中,15 N或13 C-PR标签otocols可以用来标记充当变形记者蛋白质的官能团。因此,PTM映射可以通过以下'感测'上的化学环境的改变相应的功能基团的特性的化学位移的位移来执行。在大多数情况下,无论是NH和CH的化学基团可以被用来报告感兴趣的PTM的演变。
目前的协议描述了含有差异同位素标记的妹妹组蛋白核的产生。它结合NMR光谱法的灵活性同时使用1 H 15 N和1 H 13 C相关光谱具有不同的蛋白的亲和标签的利用所选重组组蛋白复合物的纯化映射翻译后修饰。值得注意的是,该协议采用了核重建一个特定的组蛋白的两个不同的池。这些池是差异同位素标记(一个 15 N,其他与13℃),它们分别融合到多组氨酸和链霉亲和标签。以Ni-NTA和基于链霉亲和色谱串联亲和纯化方案最初使用Voigt 等 。 7被用来净化从对称的对应( 图1A)的不对称物种。非对称组蛋白八聚体随后用于重建等效核小体复合物( 图1B),使用标准的盐透析法14。此外,通过相同的方法,由具有组蛋白池之一预先改性,一PTM可以不对称结合到所得到的核小体。这些基板与组蛋白修饰酶和修改事件的后续核磁共振映射的反应使串扰机制表征无论是在顺式 (premodified组蛋白拷贝)和反式 (unmodifiED组蛋白复印件)( 图1C)。
研究方案
1.核小体的重建与差异同位素标记(和非对称修改)组蛋白姐姐
注:目前的协议描述了差异同位素标记的组蛋白H3核小体的重建。为此,组蛋白H3的两个池被使用;一个是15 N-标记的并含有在N-末端具有带6xHis标签,第二次是13 C标记,并含有在N-末端融合的链球菌肽(WSHPQFEK)。这两个变量从一个烟草蚀纹病毒(TEV)蛋白酶识别位点的本机H3序列分离。以制备额外不对称修饰的核,这两个H3池之一被包括在premodified形式。组蛋白池的修改前的可以由感兴趣同位素标记的组蛋白与相应的组蛋白修饰酶在必要的情况下为每种类型的反应的辅助因子/ PTM献血者1反应来进行6。各个PTM的有效放置可通过NMR光谱或质谱进行评估。组蛋白/ DNA的这里使用的量是粗糙的建议以获得具有10μm的近似浓度(如DNA浓度在260nm测得)。这最终产率是足以记录良好品质NMR谱与相对较短的采集时间核小体的制备。
- 与差异同位素标记(和不对称修改)组蛋白H3组蛋白八聚体的重组
注:重组组蛋白可以毫克的量来生产在BL21(DE3)plysS中的细胞14。以获得的同位素标记的组蛋白,相同的表达/纯化协议之后,使用对细菌生长的含有15 NH 4 Cl和/或13 C-葡萄糖作为氮源和碳源的基本培养基外,对15 N-或13 C-标签respectively。纯化的组蛋白被广泛地透析5mM的β巯基乙醇和使用之前储存冻干。- 溶解冻干组蛋白等分试样,含有〜5毫克每组蛋白在1mL去折叠缓冲液(7M胍盐酸,20毫摩尔Tris pH为7.5,10mM的DTT)。包括两种类型的组蛋白H3。吹打混匀,不要旋涡。保持冰管30分钟,使完全展开。
- 通过测量280nm处的吸光度对展开缓冲器和使用与ExPASy门户的protParam工具计算的消光系数确定每一个组蛋白的确切浓度。 15
- 混合核心组蛋白等摩尔比,牢记包括每两个组蛋白H3池的50%。通过使用较少的组蛋白集中的全部内容启动并相应地添加所有其他人。
- 调整最终蛋白质浓度至约1毫克/毫升用折叠缓冲液中。
- Transfer中混合物至6 kDa的截止透析膜和透析针对1-2大号冷却重折叠缓冲液(10mM的Tris pH为7.5,2M氯化钠,1mM EDTA中,2毫摩尔DTT)于4℃的。改变缓冲至少一次透析已经进行了最少3小时后。执行最终透析O / N在4℃。
- 收集透析材料,并在4℃下的最大速度在微量离心除去任何沉淀5分钟(通常无沉淀应观察)。通过测量280nm处的吸光度确定八聚体的浓度。在计算中,用组蛋白的添加消光系数,牢记双每个值。
- 浓缩,使用10 kDa的分界离心过滤单元〜2毫升的最终体积。重新计算八聚体的浓度和估计的损失。在这一点上,50之间的八聚体浓度 - 应取得100μM的。
- 凝胶过滤柱(在材料的表表示)连接到一个FPLC系统和平衡用的过滤微米并脱气以1mL /分钟的流速和0.5MPa的压力极限重折叠缓冲0.22 2柱体积(CV)。
注:凝胶过滤运行应在冷室中或在冷柜中进行。 - 使用1毫升/分钟的流速注入浓缩材料,并收集1.0 - 1.5毫升馏分。
- 按照色谱图,并在65〜观察组蛋白八聚体洗脱 - 68毫升。
注意:洗脱峰的小肩膀,并在〜80毫升的峰值分别代表自由H3-H4四聚体和H2A-H2B二聚体的存在。 - 在18%的SDS-PAGE凝胶上运行每个收集的级分的20微升。
注:加载它们在凝胶上,以减少高盐浓度,因此,避免扭曲之前用水以至少2倍稀释的样品。这同样适用于在重折叠缓冲液样品的随后的SDS-PAGE分析。 - 池相关FRA载有4组蛋白的凝胶染色的均匀分布判断,并确定浓度之前(步骤1.1.7)纯八聚体ctions。
- 1毫升Ni-NTA柱( 材料数据表 )连接到FPLC系统,用10 CV 0.22平衡微米过滤并脱气以1mL / min的流速重折叠缓冲液中。
注意:所述的Ni-NTA色谱应在冷室中或在冷柜中进行。 - 使用1毫升/分钟的流速通过在Ni-NTA通过纯化的八聚体。
- 收集流过,并保持样品SDS-PAGE / WB分析(分别为20μL和4μL)。接着,用10CV重折叠缓冲或直到达到基线信号的洗柱。
- 用10CV复性使用1毫升/分钟的流速含有250mM咪唑缓冲液中洗脱。为保持SDS-PAGE / WB分析的样品(分别为20μL和4μL)。
- 平衡的交流的1毫升亲和柱ommercial链霉( 材料表 )使用批处理/重力流设置含250 mM咪唑10 CV复性缓冲。
注:此色谱步骤应在冷室中进行。 - 通过商业链霉亲和柱通过的Ni-NTA洗脱。
- 通过收集流量和样本保持SDS-PAGE / WB分析(分别为20μL和4μL)。随后,用10CV复性缓冲。
- 用10CV折叠含有2.5mM脱硫生物素缓冲液中洗脱。为保持SDS-PAGE / WB分析的样品(20和4μL分别)。
- 测量八聚体浓度,添加量少10倍TEV蛋白酶和让反应在标准的塑料离心管中在4℃下继续过夜。
注:TEV的必需量,以获得完全消化(除去亲和标签的)依赖于原点(构建体)和活性(新鲜的,重组的TEV更活性相比在长时间储存所述一个 - 80℃)。尝试相比,八聚体(估计为蛋白质浓度)最初加入10倍的TEV少,并相应地调整。 - 通过运行前的混合物的20微升和后TEV除了在一个18%的SDS-PAGE凝胶上检查有效消化。如果消化不完全,增加更多的TEV蛋白酶和再持续3-4小时孵化。
- 透析针对使用50kDa的截止透析膜以除去TEV蛋白酶和调整缓冲组合物的重折叠缓冲液样品。
- 使用10 kDa的分界离心过滤单元(从步骤1.1.7相同的过滤器单元,也可使用),以1.0-2.0毫升体积浓缩纯化不对称八聚体。测量最终浓度。要么使用后不久为核小体重构或调整到50%(体积/体积)甘油在-20℃保存更长时间。在这一点上与前面的步骤中期望的至少70%的损失,第Ë八聚体浓度应在20〜 - 50微米。
- 核重建
- 如果八聚体被储存在50%甘油,在4℃下透析过夜针对与核小体重构继续之前重折叠缓冲液中。
- 调整的DNA通过使用5 M氯化钠原液(含有核小体定位序列)的盐浓度至2M NaCl的。
注:将纯化的DNA,它包含或所需序列的多拷贝的质粒的纯化后或者分离通过PCR扩增合成应储存浓缩至〜在水中的50微米或标准的Tris-EDTA缓冲液。 - 利用小规模试验确定最佳比例混合八聚体和DNA。使用重折叠缓冲液调整最终体积至〜3.0毫升。总是添加最后以防止混合的八聚体和在<2M的盐浓度该DNA的任何可能性的八聚体。
- 执行初始小-SC强麦reconstitutions确定八聚体:通向最佳产量核DNA的比例。对于这一点,装配的0.9三种反应:1.0,1.0:1.0和1.1:1.0 DNA:八聚体比率微升50-100之间的音量。通常情况下,使用5微米的DNA的浓度。
- 有如下(步骤1.2.4-1.2.8)中描述的重组进行,到了最后,通过测量260nm处的DNA浓度,并通过运行6%原生-PAGE DNA凝胶估计可溶性质量好的重组核小体量,用溴化乙锭,分别( 图5A)染色。
- 混合转移至6 kDa的截止透析膜和透析针对1升,4℃过夜重折叠缓冲液中。
- 从2减少透析缓冲液的盐浓度在一个逐步的方式,至0.85,0.64,和分别的0.2M NaCl洗涤。保持样品中的每个缓冲器至少3小时。有时沉淀在最后转换后形成的。在这种情况下Continue透析按计划和微量离心在下一步骤结束除去沉淀物。
- 转印透析膜在烧杯用1L测定缓冲液(25mM的NaH 2 PO 4 / 的 Na 2 HPO 4 pH 6.8的25 mM氯化钠,2mM的DTT)中,并继续透析O / N在4℃下的。
- 收集样品,除去任何沉淀物在最大速度步骤1.2.5通过离心在5分钟微量在4℃下形成,并使用30kDa的分界离心过滤单元来〜300微升的最终体积浓缩。
- 测量DNA浓度在260nm,运行了18%的SDS-PAGE蛋白质凝胶(4微升的样品)和6%的天然-PAGE DNA凝胶(0.2μL样品),并存储样本在4℃几周。终浓度应在10-20微米的范围内。
在两个姐妹组蛋白修饰反应2. NMR分析
注:转让核小体组蛋白尾巴的二维1 H- 15 N相关光谱可以参考16,17,18被发现。此外,CH的分配X在2D的赖氨酸,丝氨酸和苏氨酸组1 H- 13 C相关光谱可参考16找到。
- NMR设置和参考光谱的记录
注意:采用在测定缓冲液中的核小体样品的140微升酶测定法来进行到3毫米NMR管在与其他样品体积300K。不同的NMR管中,也可以使用。上述温度适宜,以获得高质量的核小体的组蛋白尾巴和良好的酶活性的1 H- 15 N相关光谱。 2D-梭华-Sofast-HMQC 1 H- 15 N和2D-HSQC 1 H- 13 C光谱带设置考勤分别录帽子赋予类似的采集时间。典型采集参数是128瞬变与1.024(1H)×128(15 N),复分和32瞬变与1.024(1H)×64(13℃)的两种不同类型的相关光谱的复杂分。对于二维光谱采集时间为〜30分钟。- 置300 K作为在光谱仪样品温度。加10%的D 2 O中(体积/体积),以将用于分光计频率锁定的样品。
- 放置在光谱仪样品和进行基本操作(锁定,调整垫片调整)。此外,确定最佳的脉冲长度和普通采集参数。
- 记录一维和二维1 H- 15 N和1 H- 13 C参考光谱。
- 过程参考光谱,并确保用于映射所述改性反应的信号被很好的解决,并具有良好的强度。
- 恢复样品和我转移吨至一个离心管。
- 酶促反应,NMR监测和定量分析的设置
- 复制并安排一系列交错的2D 1 H- 15 N和1 H- 13 C谱。瞄准的几个小时的总采集时间和基于来自该第一反应得到的酶促速率新的运行相应的调整。
- 添加到样品的必要辅因子用于各个类型的修饰反应(1毫摩尔ATP / 2mM的MgCl 2的磷酸化,1mM的乙酰-CoA乙酰化,1mM的SAM为甲基, 等 )。
- 加的目的酶,通过移液混合,在光谱仪(快速执行这些操作)将样品转移回到在NMR管,并随后。
注意:如果不存在关于期望的酶活性的数据,调整所述衬底对酶的摩尔比为10:1。执行第一次试运行,并相应地调整实际运行。 - 执行快速自动匀场,并使用参数的其余部分从参考光谱。
- 启动一系列交错二维1 H- 15 N和1 H- 13 C谱。
- 按照实时反应的进行,当反应达到完成,或者所需的水平停止采集。
- 流程光谱与完全相同的参数之前。
- 使用两种类型的相关光谱的不同习得顺序重复整个运行。如果在第一个实验中一个1 H 15 N个相关最早是在系列中,用1 H- 13 C相关现在开始。通过这一点,并具有相同的采集时间为两个光谱,能够绘制上,而在同一时间尺度两者差异同位素标记的组蛋白比较PTM反应。此外,有可能计算出平均值和情节误差棒。
注:thoroug该方法用于核磁共振信号分析和酶促反应的后续量化h的描述可以在这里找到19。 - 选择从每个时间进程NMR谱该报告反应的进行的NMR信号,并计算使用用于NMR光谱的可视化和分析软件它们的相对信号强度。为此该报告的未经修改以及修改状态的信号。提取修改级别。
- 绘制对反应时间修改级别的计算值。
结果
适当重折叠八聚体种类重构混合物的通过凝胶过滤柱运行( 图2)后分离。含有三种不同类型的八聚体的重组八聚池经受串联亲和纯化方案。样品从所有的步骤中收集并通过SDS-PAGE和随后WB分析。其导致非对称物种的隔离的协议的正确执行的验证是通过以下的不同样品中两个亲和标签(的His-和Strep-)的存在( 图3)来实现的。随后,将亲和标签被TEV蛋白酶消化( 图4)后除去。处置亲和标签的不对称八聚体被用于重建不对称的核。最初,小试规模的重组被用于确定DNA的最佳摩尔比:八聚体,在一个高的结果产量形成单分散复杂的。随后,最优化的混合比被用来重建核小体在大的规模。核reconstitutions通过蛋白质/ DNA凝胶和NMR谱的正确装配和差异同位素标记的妹妹H3组蛋白的存在下分别( 图5)分析。一个应用实例是图6中所描绘。具体地,差异同位素标记和上H3S10核不对称磷酸化与Gcn5和乙酰转移反应,和H3K14的两个H3份乙酰化接着在用NMR光谱的实时性。分析显示,H3S10磷酸化在独联体相比, 反式刺激H3K14乙酰化Gcn5和。

图1:流程图差异同位素-L的制备abeled(和非对称修改)核小体。 (A)差分同位素标记(和非对称修改)的组蛋白八聚体的重构与串联亲和纯化。 (B)通过组合纯化的非对称八聚体和DNA核小体定位序列核小体重构。 (℃)。NMR监测在顺 和反式的非对称地修改和差异同位素标记的核小体组蛋白修饰酶混合后修饰串扰。 请点击此处查看该图的放大版本。
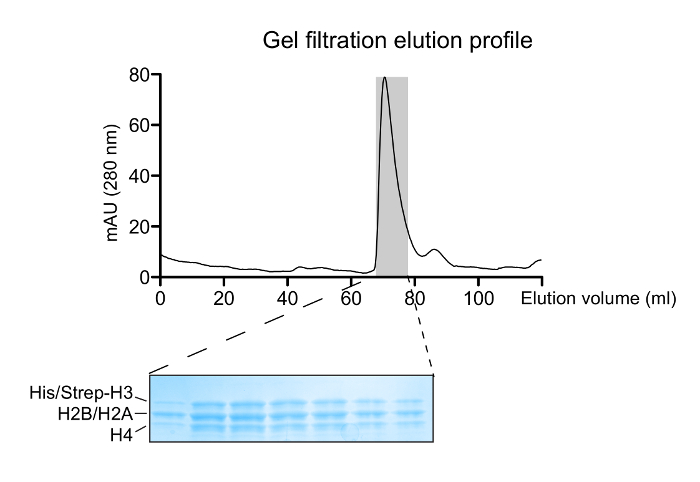
图2: 组蛋白八聚体的重构。复性蛋白混合物的凝胶过滤洗脱概况。在〜70毫升的主要峰对应于组蛋白八聚体。 A 14 - 对应于八聚体峰洗脱的级分的20%梯度SDS-PAGE凝胶(考马斯蓝染色)显示正确的组蛋白化学计量。 请点击此处查看该图的放大版本。

图3: 非对称组蛋白八聚体的分离纯化。 14-20%梯度SDS-PAGE凝胶(考马斯染色)和WB针对来自串联亲和纯化方案的不同阶段的样品的的His-和Strep-亲和标签。 请点击此处查看该图的放大版本。

图 4: 亲和标签的去除。 18%SDS-PAGE凝胶之前并与TEV蛋白酶混合后纯化的非对称组蛋白八聚体的(考马斯染色)。 请点击此处查看该图的放大版本。

图5: 差异同位素标记的核小体的生物化学和核磁共振表征。 (A)6%天然-PAGE DNA凝胶使用DNA的不同摩尔比小试规模的核小体重构的(溴化乙锭染色):八聚体(左)。使用优化的DNA大规模核小体重构的6%非变性-PAGE DNA凝胶(溴化乙锭染色):八聚体的摩尔比(右)。重建的核(B)的18%的SDS-PAGE蛋白质凝胶(考马斯染色)显示了四个核心组蛋白的正确化学计量。 (C)的差异同位素标记的核小体的15 N-标记的H3拷贝1 H 15 N梭华-Sofast-HMQC谱(仅H3尾部的残基是可见-蛋白质核心是不可见的由于缓慢翻滚速率)。 (D)的差异同位素标记的核小体的13 C标记的H3副本1 H- 13 C HSQC谱。 请点击此处查看该图的放大版本。
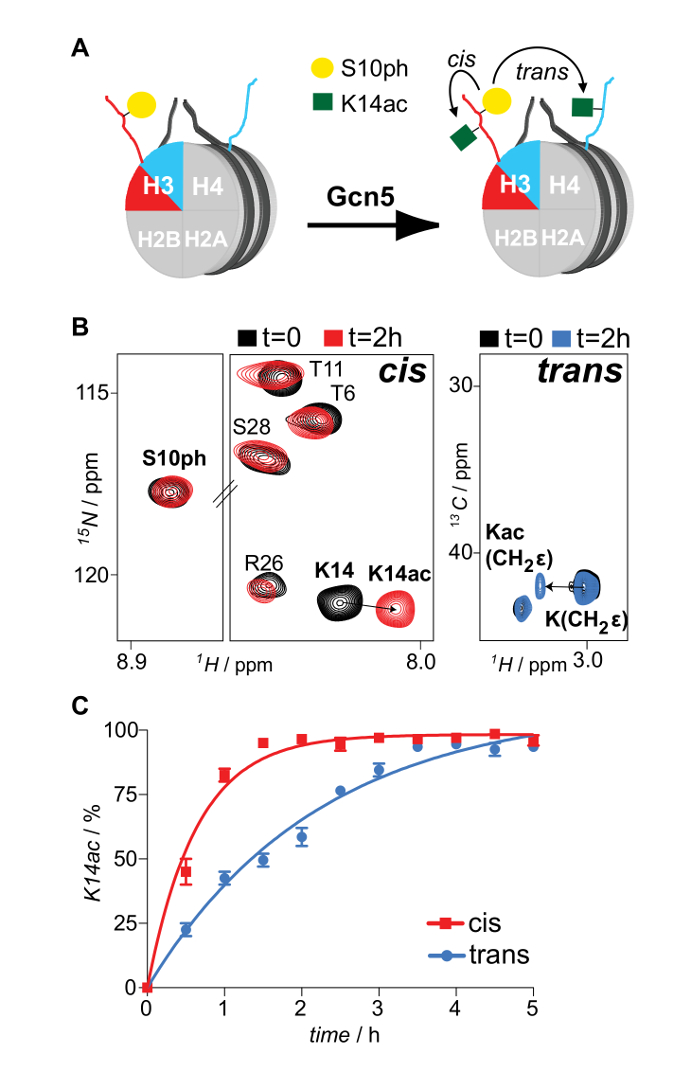
图6: 在顺式 和 反式 核磁共振监控 的修改串扰通过H3S10的不对称磷酸化Gcn5和乙酰转移酶的乙酰化H3K14活动强加的。 (A)的示意图差异同位素标记的和不对称的磷酸化的H3S10核与Gcn5和乙酰转移酶和乙酰化H3K14映射反应, 顺式和反式 。 (B)的1 H 15 N梭华-Sofast-HMQC和之前和Gcn5和反应后的非对称核的1 H 13 C HSQC光谱(选择的区域)。 (C)H3K14乙酰化对非对称H3S10磷酸化的核小体的时间分辨核磁共振监测由Gcn5和。从两个独立实验的平均值和变化(误差棒)被示出。转载许可16。 请点击此处查看大图这个数字。
讨论
对于核重建,目前协议利用含有601代表智慧核小体定位序列20 165 bp的DNA模板,但类似的性能是使用的模板DNA不同长度的预期。该协议的设计和采用使用非对称类型的组蛋白H3。用同样的原理,也可以应用于该方法的其他核心组蛋白和另外可用于重构携带具有不同同位素标记两个不同的核心组蛋白复合物。该协议可以稍微还修改了最初重组蛋白亚复合物,而不是直接组蛋白八聚体。以下这个变形例中,非对称H3-H4四聚体可通过采用同样的串联纯化方案进行复原。后者与分别重构H2A-H2B二聚体和DNA混合组装核16。最终产率和改性protoc的整体性能醇是类似于原始之一。
该协议是强烈地依赖于两个亲和柱,以有效地结合相关的物种,从而在最后的高纯度的不对称配合物,得到的能力。它是麻烦的,以提供直接和具体证明最终纯化复杂的确,只有非对称。然而,从与有关的抗体的所有纯化步骤样品的WB分析提供了用于串联纯化方案的成功( 图3)可靠指示。相同的结果,因此,额外的保证,通过分析相同样品通过NMR方法16存在下的亲和标签的获得。尽管如此,如果WB分析表明具有对称那些非对称物种的污染,一个小的变化,可以在第一纯化步骤(的Ni-NTA),可以提高疗效通过。特别是,而不是等度洗脱,咪唑gradien吨洗脱(10-250毫米)可以采用,然后,只有那些在不对称物种高度富集第一次洗脱级分合并,并通过亲和柱中运行。这可以通过在洗脱级分的链球菌亲和标签的存在的WB分析进行评估。
相对于感兴趣的PTM同位素标记的选择是相当灵活的,因为在大多数情况下,核磁共振能够同时使用1 H 15 N和1 H- 13 Ccorrelation谱映射相同的PTM的。然而,对于某些氨基酸,如赖氨酸,对于侧链1 H 13 C共振的化学位移分散是相当有限的。此外,当一个赖氨酸修饰酶的靶位点是未知的,赖氨酸翻译后修饰的位点特异性读出并不简单。在这些情况下,可替代的方法采用位点选择性的13 C-标记,结合半合成的组蛋白生产"> 21或使用新建立的NMR脉冲序列,使变形状态的特异性检测通过选择性的激励例程22。核磁共振光谱赋予相当低的灵敏度。在这方面,以执行需要较高量的核小体(μM范围)的酶促反应。这防止了方法以高通量设置的演变。
同时,一个新的化学方法已介绍了化学纯,不对称修饰核的合成中,以高产率,携带定义翻译后修饰23。这一方法已被使用,结合荧光自,研究PTM串扰机制。由于在检测翻译后修饰X光成像方法低分辨率能力,结论是通过在一组携带对称和非对称翻译后修饰的不同组合,而核的比较总体酶活性间接衍生比在每个姊妹组蛋白的相应位点的PTM反应直接观察。然而,同位素标记的组蛋白的掺入和用于PTM读出的使用NMR光谱法的可能构成上述方法为不对称改性的核小体的高分辨率PTM分析一个额外的工具。
在与新的类型在将来不对称修饰核小体的鉴定方面,当前的方法的目的是构成一个高分辨率的工具, 在顺式和在核小体衬底反 PTM串扰反应分析。特别是,具有高度重要性是,要包含不对称,无论是转录活性和压抑的翻译后修饰价核24产生的串扰机制的解码。
披露声明
The author has nothing to disclose.
致谢
作者感谢菲利普博士Selenko(FMP - 柏林)提供湿实验室空间和基础设施,通过研究补助经费的工作进行实验和德意志研究联合会(DFG)(LI 2402 / 2-1)。
材料
| Name | Company | Catalog Number | Comments |
| Guanidinium HCl | Applichem | A14199 | Use high quality Gu-HCl |
| Tris | Roth | 4855.2 | |
| DTT | Applichem | A1101 | |
| NaCl | VWR chemicals | 27810.364 | |
| EDTA | Roth | 8043.2 | |
| Na2H2PO4 | Applichem | A1046 | |
| NaH2PO4 | Applichem | A3902 | |
| Imidazole | Applichem | A1073 | |
| d-Desthiobiotin | Sigma | D1411 | |
| Ni-NTA Superflow | Qiagen | 1034557 | |
| Strep-Tactin Superflow | IBA | 2-1207-001 | |
| His-probe antibody | Santa Cruz | sc-8036 | |
| Strep-tactin conjugated HRP | IBA | 2-1502-001 | |
| Hi-Load 16/600 Superdex 200 pg | GE Healthcare | 28-9893-35 | |
| 6 - 8 kDa dialysis membrane | Spectrumlabs | spectra/por 1, 132650 | |
| 50 kDa dialysis membrane | Spectrumlabs | spectra/por 7, 132129 | |
| 10 kDa centrigugal filter unit | Merck Millipore | UFC901024 | |
| 30 kDa centrigugal filter unit | Merck Millipore | UFC903024 | |
| Solution-state NMR spectrometer | at least 500 MHz operating frequency, equipped with a triple-resonance cryoprobe | ||
| Buffer name | Component | ||
| Unfolding Buffer | 7 M Guanidinium HCl | ||
| 20 mM Tris, pH 7.5 | |||
| 10 mM DTT | |||
| Refolding Buffer | 10 mM Tris, pH 7.5 | ||
| 2 M NaCl | |||
| 1 mM EDTA | |||
| 2 mM DTT | |||
| Assay Buffer | 25 mM Na2H2PO4, pH 6.8 | ||
| 25 mM NaCl | |||
| 2 mM DTT |
参考文献
- Hansen, J. C. Conformational Dynamics of the Chromatin Fiber in Solution: Determinants, Mechanisms, and Functions. Annu. Rev. Biophys. Biomol. Struct. 31, 361-392 (2002).
- Clapier, C. R., Cairns, B. R. The Biology of Chromatin Remodeling Complexes. Annu. Rev. Biochem. 78, 273-304 (2009).
- Musselman, C. A., Lalonde, M., Cote, J., Kutateladze, T. G. Perceiving the epigenetic landscape through histone readers. Nat. Struct. Mol. Biol. 19 (12), 1218-1227 (2012).
- Suganuma, T., Workman, J. L. Signals and Combinatorial Functions of Histone Modifications. Annu. Rev. Biochem. 80, 473-499 (2011).
- Lee, J. S., Smith, E., Shilatifard, A. The Language of Histone Crosstalk. Cell. 142 (5), 682-685 (2010).
- Liokatis, S., et al. Phosphorylation of histone H3 Ser10 establishes a hierarchy for subsequent intramolecular modification events. Nat. Struct. Mol. Biol. 19 (8), 819-823 (2012).
- Voigt, P., et al. Asymmetrically Modified Nucleosomes. Cell. 151 (1), 181-193 (2012).
- Egelhofer, T. A., et al. An assessment of histone-modification antibody quality. Nat. Struct. Mol. Biol. 18 (1), 91-93 (2011).
- Huang, H., Lin, S., Garcia, B. A., Zhao, Y. Quantitative Proteomic Analysis of Histone Modifications. Chem. Rev. 115 (6), 2376-2418 (2015).
- Liokatis, S., Dose, A., Schwarzer, D., Selenko, P. Simultaneous Detection, of Protein Phosphorylation and Acetylation by High-Resolution NMR Spectroscopy. J. Am. Chem. Soc. 132 (42), 14704-14705 (2010).
- Binolfi, A., et al. Intracellular repair of oxidation-damaged α-synuclein fails to target C-terminal modification sites. Nat. Commun. 7, 10251(2016).
- Schanda, P., Kupce, E., Brutscher, B. SOFAST-HMQC experiments for recording two-dimensional heteronuclear correlation spectra of proteins within a few seconds. J. Biomol. NMR. 33 (4), 199-211 (2005).
- Theillet, F. X., et al. Cell signaling, post-translational protein modifications and NMR spectroscopy. J. Biomol. NMR. 54 (3), 217-236 (2012).
- Dyer, P. N., et al. Reconstitution of Nucleosome Core Particles from Recombinant Histones and DNA. Methods Enzymol. 375, 23-43 (2004).
- Artimo, P., et al. ExPASy :SIB bioinformatics resource portal. Nucleic Acids Res. 40 (W1), 597-603 (2012).
- Liokatis, S., klingberg, R., Tan, S., Schwarzer, D. Differentially Isotope-Labeled Nucleosomes to Study Asymmetric Histone Modification Crosstalk by Time-Resolved NMR Spectroscopy. Angew. Chem. Int. Ed. 55 (29), 8262-8265 (2016).
- Stützer, A., et al. Modulations of DNA Contacts by Linker Histones and Post-translational Modifications Determine the Mobility and Modifiability of Nucleosomal H3 Tails. Mol. Cell. 61 (2), 247-259 (2016).
- Zhou, B. R., et al. Histone H4 K16Q Mutation, an Acetylation Mimic, Causes Structural Disorder of Its N-terminal Basic Patch in the Nucleosome. J. Mol. Biol. 421 (1), 30-37 (2012).
- Theillet, F. X., et al. Site-specific NMR mapping and time-resolved monitoring of serine and threonine phosphorylation in reconstituted kinase reactions and mammalian cell extracts. Nat. Protoc. 8 (7), 1416-1432 (2013).
- Lowary, P. T., Widom, J. New DNA sequence rules for high affinity binding to histone octamer and sequence-directed nucleosome positioning. J. Mol. Biol. 276 (1), 19-42 (1998).
- Hackenberger, C. P., Schwarzer, D. Chemoselective ligation and modification strategies for peptides and proteins. Angew. Chem. Int. Ed. 47 (52), 10030-10074 (2008).
- Theillet, F. X., et al. Site-specific mapping and time-resolved monitoring of lysine methylation by high-resolution NMR spectroscopy. J. Am. Chem. Soc. 134 (18), 7616-7619 (2012).
- Lechner, C. C., Agashe, N. D., Fierz, B. Traceless Synthesis of Asymmetrically Modified Bivalent Nucleosomes. Angew. Chem. Int. Ed. 55 (8), 2903-2906 (2016).
- Bernstein, B. E., et al. A bivalent chromatin structure marks key developmental genes in embryonic stem cells. Cell. 125 (2), 315-326 (2006).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。