Method Article
Ein neuer Ansatz für die vergleichende Analyse von Multiproteinkomplexen Basierend auf
In diesem Artikel
Zusammenfassung
Die beschriebenen Vergleichs, quantitative Proteomik-Ansatz zielt auf die Erlangung Einblicke in die Zusammensetzung von Multiproteinkomplexen unter verschiedenen Bedingungen und wird durch den Vergleich genetisch verschiedenen Stämmen nachgewiesen. Für die quantitative Analyse gleichen Volumina verschiedener Fraktionen aus einem Saccharose-Dichtegradienten durch Massenspektrometrie vermischt und analysiert.
Zusammenfassung
Das eingeführte Protokoll bietet ein Tool für die Analyse von Multiproteinkomplexen in der Thylakoidmembran, durch aufschlussreiche Einblicke in die komplexe Zusammensetzung unter verschiedenen Bedingungen. In diesem Protokoll wird der Ansatz durch den Vergleich der Zusammensetzung des Proteins für den zyklischen Elektronenfluss (CEF) in Chlamydomonas reinhardtii, isoliert aus genetisch unterschiedlichen Stämmen Komplex verantwortlich demonstriert. Das Verfahren umfasst die Isolierung von Thylakoidmembranen, gefolgt von der Trennung in die Multiproteinkomplexen durch Saccharose-Dichtegradienten-Zentrifugation, SDS-PAGE, und Immunodetektion vergleichende quantitative Massenspektrometrie (MS) basierend auf Differenzial metabolische Markierung (14 N / 15 N) des Stämme analysiert. Waschmittel solubilisierten Thylakoidmembranen auf Saccharose-Dichtegradienten in gleichen Chlorophyllkonzentration geladen. Nach Ultrazentrifugation werden die Gradienten in Fraktionen, die durch Massen spectromet analysiert werden getrenntry zu gleichen Volumen. Dieser Ansatz ermöglicht die Untersuchung der Zusammensetzung innerhalb der Gradientenfraktionen und darüber hinaus, um das Migrationsverhalten verschiedener Proteine zu analysieren, vor allem die Konzentration auf ANR1, CAS und PGRL1. Außerdem ist dieses Verfahren durch die Bestätigung der Ergebnisse mit Immunoblotting und zusätzlich, indem sie die Ergebnisse aus früheren Studien (die Identifizierung und PSI-abhängigen Migration der Proteine, die zuvor beschrieben wurden, einen Teil der CEF-Superkomplex wie PGRL1 FNR sein und zeigte cyt f). Bemerkenswert ist, ist dieser Ansatz für eine breite Palette von Fragen, auf die dieses Protokoll verabschiedet werden kann und beispielsweise für vergleichende Analysen von Multi komplexe Zusammensetzung aus unterschiedlichen Umgebungsbedingungen eingesetzt isoliert anzugehen.
Einleitung
Photosyntheseprozesse Thylakoidmembran von Pflanzen und Algen in einer linearen und cyclischen Modus. Während der linearen Elektronenfluss (LEF) Photosystem I (PSI), Photosystem II (PSII) und Cytochrom b 6 / f letztlich Elektronen übertragen von Wasser zu NADP + 1, was zu der Erzeugung von NADPH und ATP 2. Im Gegensatz dazu zyklischen Elektronenfluss (CEF), die bekannt ist, um unter verschiedenen Umgebungsbedingungen, wie der Zustand 2 3 und 4 anaeroben Bedingungen, Ergebnisse in der Rückreduktion des oxidierten PSI durch Injizieren von Elektronen in die Elektronentransportkette induziert werden. Dieser Prozess kann entweder an der Stroma-Seite des Cytochrom b 6 / f-Komplex 1 oder am Pool Plastochinons 5 finden kann und erzeugt ATP, aber kein NADPH 2.
Das Ziel der vorliegenden Protokolls ist es, eine Massenspektrometrie (MS) basierend m demonstrierenethode für die vergleichende quantitative Analyse von Multiproteinkomplexen in der Thylakoidmembran von Chlamydomonas reinhardtii, um Einblick in die Zusammensetzung dieser Komplexe unter verschiedenen Bedingungen (durch Vergleich der genetisch verschiedene Stämme beispielhaft) zu gewinnen. Dieser Ansatz wurde in einer Veröffentlichung von Terashima et al angewendet. Im Jahr 2012, die eine Ca 2 +-abhängige Regulation der CEF in C. reinhardtii von einem Multiproteinkomplex einschließlich der Proteine CAS, ANR1 und PGRL1 6 vermittelt. Das Verfahren wird durch vergleichsweise Analyse der Zusammensetzung der CEF-Superkomplex in zwei genetisch verschiedene Stämme, wodurch der Vorteil der Markierung eines der beiden Stämme mit schweren Stickstoff (N 15) erläutert. Kurz gesagt, enthält das Protokoll die Vorbereitung der Thylakoidmembran, gefolgt von Waschmittel Solubilisierung und Fraktionierung der Photosynthesekomplexe in einem Saccharose-Dichtegradienten. Nach Fraktionierung des Gradienten, ausgewählt fracgen der beiden Stämme gemischt, basierend auf dem gleichen Volumen von SDS-PAGE, gefolgt von In-Gel-Verdauung und anschließende quantitative MS-Analyse getrennt.
Wie oben erwähnt, wird CEF unter verschiedenen Umweltbedingungen induziert und eine Veröffentlichung aus dem Jahr 2010 zeigt die Isolierung eines funktionellen CEF-Superkomplex vom Zustand 2 verriegelt Zellen von C. reinhardtii 7, die durch die Trennung solubilisierten Thylakoidmembranen auf einem Saccharose-Dichtegradienten-Ultrazentrifugation bei geführt wurde. Anders Iwai et al. 7, das vorgestellte Protokoll beschreibt die Isolierung des CEF-Superkomplex aus der anaeroben gewachsen C. reinhardtii Kulturen, indem Sie ein alternatives Verfahren. Dies umfasst Veränderungen in der Thylakoidmembran Isolierungsprotokoll als auch Unterschiede in der Lösungsschritt und der Trennung der Proteinkomplexe durch Ultrazentrifugation. In dem vorliegenden Protokoll Thylakoidmembranendurch Anwendung der von Chua und Bennoun 8 veröffentlichten Verfahren, die Puffer für thylakoid Herstellung von Iwai et al verwendet isoliert. enthielt 25 mM MES, 0,33 M Saccharose, 5 mM MgCl 2, 1,5 mM NaCl (pH 6,5), wie beschrieben, 9. Die Solubilisierung wurde mit 0,7-0,8% Detergens (n-Tridecyl-β-D-maltosid) für 30 min auf Eis im Fall von Iwai und Mitarbeitern durchgeführt, während das hier beschriebene Solubilisierung Verfahren beruht auf der Verwendung von 0,9% Detergenz (n -Dodecyl-β-D-maltosid (DM-β)) und nur für 20 Minuten auf Eis durchgeführt. Beide Gruppen verwendeten 0,8 mg Chlorophyll pro ml für die Solubilisierung mit dem jeweiligen Reinigungsmittel. Für die Trennung von Photosynthesekomplexen aus solubilisierten Thylakoidmembranen Iwai et al. Angewendet Saccharose-Konzentrationen zwischen 0,1 bis 1,3 M, während die Autoren dieses Protokoll Konzentrationen im Bereich von 0,4 bis 1,3 M. Der letzte Unterschied ist die Zentrifugation Geschwindigkeit, die geringer ist im Vergleich auf der earlier Veröffentlichung.
Solubilisierung von Thylakoidmembranen mit nichtionischen Detergenzien, gefolgt von Saccharose-Dichtegradienten-Fraktionierung wurde bereits in zahlreichen Studien, die von den 1980er Jahren bis heute 7, 9-14 und auch die Anwendung von metabolischer Markierung von Proteinen ist ein weit verbreitetes Verfahren in dem Gebiet der Proteomik angewendet. Die beschriebene Vorgehensweise gilt die 15 N metabolische Markierung für eine der beiden verglichenen Stämme durch Kultivieren in Gegenwart von Schwer Stickstoff als einzige Stickstoffquelle in Form von 15 N NH 4 Cl, die in den Aminosäuren, die zu einer Masse eingeschlossen ist Verschiebung in Abhängigkeit von der Aminosäuresequenz des Peptids. Bei der Analyse einer Mischung von 14 N und 15 N in einem MS ausführen kann diese Massenverschiebung verwendet werden, um die Probe für jede Ursprungs-Peptid und Peptid relativen Häufigkeiten berechnet werden kann, die relative Häufigkeit der entsprechenden bestimmening Protein 15.
Zahlreiche quantitative Proteomik-Studien an C. reinhardtii zur Verfügung, die eine definierte Menge an Protein zu vergleichen, um Veränderungen im Proteom zwischen experimentellen Bedingungen (zB Veränderungen im Proteom durch Nährstoff 16-19 oder Lichtstress 20,21) zu analysieren. Im Vergleich zu den Studien, in der aktuell vorgestellte Ansatz gleicher Volumina der Proben werden vereinigt und analysiert. Dieser Aufbau erlaubt es, das Migrationsverhalten der Proteine innerhalb der Gradient untersuchen und darüber hinaus zur Analyse der Zusammensetzung der verschiedenen Komplexe in Bezug auf die untersuchten Stämme.
Diese Methode wird vor allem durch die Konzentration auf drei Proteine erklärt werden: Der erste Kandidat ist der Chloroplasten lokalisierten Kalzium-Sensorprotein CAS, die gezeigt wurde, in Foto-Akklimatisierung in C beteiligt werden 22 reinhardtii. Calcium wird als ein wichtiges Signalten Ionen für Wege, die durch verschiedene biotische und abiotische Stressfaktoren aktiviert werden, schließlich zu Veränderungen in der Genexpression und Zellphysiologie 23 führt, und es wurde vorgeschlagen, dass Chloroplasten könnte die zelluläre Ca 2 + Signal beitragen über die CAS-Protein 22,24,25. Das zweite Protein ANR1 (anaerobe Reaktion 1 6), ein Protein, das gezeigt wurde, unter anoxischen Bedingungen wachsen in C induziert werden, 26 reinhardtii. Bemerkenswerterweise wurden CAS sowie ANR1 als Untereinheiten des CEF-Superkomplex und darüber hinaus durch Verwendung von Reverse genetische Ansätze, wurde gezeigt, dass beide Proteine beitragen funktionell CEF in vivo 6. Unterstützung ihrer Rolle als Funktionseinheiten dieses Proteinkomplexes identifiziert. Das dritte Protein ist die Thylakoidproteintrennung PGR5-Like 1 (PGRL1), die gezeigt wurde, in CEF in Chlamydomonas 4,27 sowie 5,28 in Arabidopsis beteiligt und war auch idin der Arbeit von Iwai et al entified. 7
Wildtyp (WT) gegen (vs) eine ΔPSI 29-Stamm und weisen eine Deletion des Gens PSAB, für einen wesentlichen Photosystem I Untereinheit, die ebenfalls Teil der Kodierung dieser Ansatz durch die die Ergebnisse von zwei verschiedenen Experimenten vorgestellt CEF-Superkomplex und WT vs pgrl1 ein Knock-out-Stamm 4. Für jeden dieser Versuche die quantitative Zusammensetzung der CEF-Superkomplex zwischen einem 15 N-und 14 N-markierten Stamm wurde verglichen.
Protokoll
1. Kultivierung von Chlamydomonas
- Das folgende C reinhardtii Stämme wurden in der vorliegenden Studie verwendet: WT cc124, WT CW15-Arg7 (zellwand und Arginin Auxotroph), ein ΔPSI mutierten Stamm 29 und ein pgrl1 Knock-out-Stamm 4.
- Alle Stämme wurden in Tris-Acetat-Phosphat-(TAP)-Medium 30, bei 25 ° C mit einem kontinuierlichen Lichtstärke von 20-50 &mgr; E / m 2 s und bei 120 Upm Schütteln gezüchtet. Die Kultur der ΔPSI sollten mit etwas Seidenpapier zur Belichtung von <5 uE / m 2 sec gewickelt werden.
- Die Kulturen der markierten Stämme (ΔPSI und WT cc124, respectively) enthielt 7,5 mM 15 N NH 4 Cl, während die Kulturen für die unmarkierten Stämme (WT CW15-Arg7 und pgrl1, respectively) enthielt 7,5 mM 14 N NH 4 Cl. Zellen für mindestens vier generiere wachsenIonen in voller Kennzeichnung und zu erreichen, müssen in der exponentiellen Wachstumsphase gehalten werden.
- Am Tag vor Beginn des Experiments zu zählen und zu verdünnen Zellen auf eine Dichte von 1 x 10 6 Zellen / ml in einem Volumen von mindestens 750 ml / Stamm.
Bitte beachten Sie, dass zwei Arten von diskontinuierlichen Saccharose-Dichtegradienten in dem folgenden Protokoll beschrieben. Die Photo Saccharose-Dichtegradienten nach Takahashi et al. 9 werden verwendet, um die verschiedenen photosynthetischen Proteinkomplexe aus isolierten und solubilisierten Thylakoide während über Nacht Zentrifugation trennen und haben den Tag vorbereitet werden, bevor (siehe Protokoll Nr. 2) und der thylakoid Saccharose-Dichtegradienten 8 sind in der thylakoid Isolierungsverfahren angewendet (siehe Protokoll Nr. 3).
2. Herstellung von Photo Sucrose Dichte Gradienten
- Zum Ausgießen der Gradienten drei Stammlösungen benötigt:
- 2 M Saccharose
- 10% β-DM in H 2 O
- 0,5 M Tricin, pH 8,0 (NaOH)
- Mit diesen Aktien, bereiten Sie die folgenden Lösungen:
Konzentration: 1,3 M 1,0 M 0,85 M 0,7 M 0,65 M 0,4 M 2 M Saccharose 13 ml 10 ml 8,5 ml 7 ml 6,5 ml 4 ml 10%46;-DM 100 ul 100 ul 100 ul 100 ul 100 ul 100 ul 0,5 M Tricin 200 ul 200 ul 200 ul 200 ul 200 ul 200 ul H 2 O 6,7 ml 9,7 ml 11,2 ml 12,7 ml 13,2 ml 15,7 ml - Verwenden Sie 14 mm x 89 mm Zentrifugenröhrchen und gießen die Gradienten sehr langsam, beginnend mit der höchsten Dichte Saccharose-Lösung niedriger Dichte Lösung.
- Pour nur jeweils 1 ml der 1,3 und 1,0 M Lösung und 2 ml für den Rest der Lösungen.
- Lassen Gradienten über Nacht im Kühlraum.
3. Anaerobe Induktion und Isolierung von Thylakoidmembranen 8
- Bevor mit der Isolierung von Thylakoidmembranen induzieren anaeroben Bedingungen unter Durchblasen von Argon 4 Stunden. Die Blasenbildung kann durch eine Glaspipette in die Kultivierung Kolben unter ständigem Mischen der Kultur mit einem Magnetrührer durchgeführt werden.
- Beginnen mit der Isolierung von Thylakoidmembranen durch Pelletieren der Zellen 5 min bei 2500 × g, resuspendieren in H1-Puffer (0,3 M Saccharose, 25 mM HEPES (pH 7,5), 5 mM MgCl 2).
(Bitte beachten Sie, dass beim Start mit der Isolierung von Thylakoidmembranen Proben should auf Eis gehalten werden, um den Proteinabbau zu vermeiden, werden alle Schritte der Zentrifugation bei 4 ° C durchgeführt und die Arbeit mit Handschuhen wird dringend empfohlen, Keratin-Kontamination zu vermeiden.) - Pellet-Zellen für 5 min bei 2.500 × g, Resuspendieren in Puffer H1.
- Brechen Sie die Zellen mit zwei Durchgängen durch einen Zerstäuber mit einem Stickstoffdruck von 1500 hPa (für die Stämme ohne Zellwand nicht nur ein Durchgang).
- Spin down Zellen für 7 min bei 2.500 x g.
(Nach diesem Schritt der Überstand sollte hellgrün sein, wenn nicht, war die Zellbruch nicht erfolgreich.) - Die Zellen in H2-Puffer (0,3 M Saccharose, 5 mM HEPES (pH 7,5), 10 mM EDTA (pH 8,0)) und Pellet-Zellen für 10 min bei 32.800 x g.
- Die Zellen in H3-Puffer (1,8 M Saccharose, 5 mM HEPES (pH 7,5), 10 mM EDTA (pH 8,0)) und homogenisieren Pellets mit einem Töpfer (keine grünen Partikel sollten am Ende dieser Arbeitsschritt bleiben).
- Bereiten thylakoid Saccharose-Dichtegradienten in WanneES (25 mm x 89 mm) von unten beginnend mit einer Schicht aus 12 ml H3-Puffer mit Zellen, gießen eine mittlere Schicht aus 12 ml H &sub4;-Puffer (1,3 M Saccharose, 5 mM HEPES (pH 7,5), 10 mM EDTA ( pH 8,0)) und auf 12 ml H5-Puffer (0,5 M Saccharose, 5 mM HEPES (pH 7,5), 10 mM EDTA (pH 8,0)). Seien Sie vorsichtig beim Gießen der Steigungen und vermeiden das Mischen der drei Schichten.
- Waage mit H5-Puffer und Zentrifuge thylakoid Saccharose-Dichtegradienten für 1 Stunde bei 70.700 x g.
- Entfernen thylakoid Bands aus den thylakoid Saccharose-Dichtegradienten und verdünnt sie mit einem geeigneten Volumen H6-Puffer (5 mM HEPES (pH 7,5), 10 mM EDTA (pH 8,0)).
- Spin down Zellen bei 37.900 g für 20 min. Wenn das Pellet ist nicht fest, ist mehr H6 Puffer für diesen Zentrifugationsschritt erforderlich.
- Resuspendieren Thylakoide in einem kleinen Volumen Puffer H6 und fahren mit der Bestimmung des Chlorophyllkonzentration.
4. Bestimmung des Chlorophyll-Konzentration 31
- Die Bestimmung der Chlorophyllmenge wird mit 80% Aceton durchgeführt.
- Mischen Sie 995 ul 80% Aceton und 5 ul der Thylakoide in H6-Puffer (Verdünnung 1:200).
- Vortex für mehrere Sekunden, bis keine grüne Partikel verbleiben und dann drehen sich mit 14,1 xg unten für 5 min.
- Nehmen Sie den Überstand und Messung der Extinktion bei 663,6 und 646,6 nm und 750 nm auf.
- Berechnen Sie die Chlorophyllmenge mit den folgenden Formeln:
- C Chl a [mg / ml] = (0,01225 * E 663,6-0,00255 * E 646,6) * Verdünnungsfaktor
- C Chl b [mg / ml] = (0,02031 * E 646,6-0,00491 * E 663,6) * Verdünnungsfaktor
- C Chl [mg / ml] = C Chl a + C Chl b
5. Laden von Photo Sucrose Dichte Gradienten
- Berechnen Sie die Beträge der Thylakoide, β-DM und H6-Puffer, die benötigt werdenfür jeden Farbverlauf:
- Jede Steigung sollte mit einem Gesamtvolumen von 700 ul mit 0,8 mg / ml Chlorophyll in 0,9% β-DM (auflösen β-DM in H 2 O) geladen werden, füllen Sie das Restvolumen mit H6-Puffer.
- Für Solubilisierung bereiten verschiedene Teilmengen mit Thylakoide, β-DM und H6 Puffer für jede Steigung.
- Lassen Proben für 20 min auf Eis mit regelmäßig Misch (Invertierung) alle paar Minuten (Solubilisierungsschritt).
- Zentrifuge bei maximaler Geschwindigkeit (14.000 × g) für 10 min bei 4 ° C.
(Nach der Zentrifugation sollte das Pellet klein und weißlich sein, während der Überstand ist dunkelgrün.) - Legen Sie Überstand an den Steigungen.
- Waage mit H6-Puffer und drehen mit Ultrazentrifuge bei 134.470 xg über Nacht (14 h) bei 4 ° C.
(Bitte beachten Sie, dass die erfolgreiche Trennung der Photosynthesekomplexe mit Photo Saccharose-Dichtegradienten wie in diesem Protokoll präsentiert wird nur erreicht, wenn using frisch isolierte Thylakoide, da eine Vorhersage für die Solubilisierung und komplexe Trennung ist schwierig, wenn die Arbeit mit Thylakoidmembranen, die vor eingefroren worden sind, zu machen.)
6. Fraktionierung von Photo Sucrose Dichte Gradienten
- Machen Sie Fotos von den Steigungen.
- Punktion ein Loch an der Unterseite des Rohres mit einer Nadel und fraktionieren Gradienten in 500 ul Röhrchen. Alternativ kann die Fraktionierung auch unter Verwendung einer Platte mit 96 Vertiefungen (Mikrotiterplatte) durchgeführt werden.
- Bei Verwendung einer Mikroplatte bestimmen Absorption der verschiedenen Fraktionen Gradienten bei 675 nm auf.
(Bei diesem Schritt können die Proben bei -80 ° C für einige Wochen gelagert werden.)
7. SDS-PAGE und Immun
(Bitte beachten Sie, dass nur für den Vergleich von WT vs ΔPSI ein Western-Blot-Analyse durchgeführt wurde, um die Fraktionen für die anschließende MS-Analyse auswählen. Für das Experiment WT vs pgrl1 frMaßnahmen wurden mittels der Absorption in den verschiedenen Fraktionen ausgewählt.)
- Separate 30 ul jeder Fraktion von WT und ΔPSI Steigung auf einem 13% SDS-Polyacrylamid-Gel-32.
- Führen Western-Blot-Analyse 33 mit den folgenden Antikörpern: ANR1 (TEF7) 26, 34 PGRL1, CAS 6, die PSI-Untereinheit PSAD 32 und die D1 PSII Kernuntereinheit. Sämtliche Antikörper mit einer Verdünnung von 1:1000 (für verstärkte Chemilumineszenz-Nachweistechnik), außer für den D1-Antikörper, der mit einer Verdünnung von 1:10000 eingesetzt werden sollte.
- Basierend auf den Ergebnissen der Immundetektion, nehmen Sie die Peak-Fraktionen von ANR1, PGRL1 und PSAD für WT und ΔPSI (hier den Fraktionen 6 und 13), mischen Sie 30 ul Fraktion 6 von WT und ΔPSI und tun das gleiche mit Fraktion 13 aus beide Präparate und trennen sie wieder auf einem 13% SDS-Polyacrylamid-Gel.
- Für den quantitativen Vergleich von WT vs pgrl1 nehmen Sie die CEF-Superkomplex Fraktionen durch Messung der Absorption in den verschiedenen Gradienten-Fraktionen bestimmt und mischen 30 ul beider Proben gefolgt von der Trennung auf einem 13% SDS-Polyacrylamidgel.
- Färben die Proteinbanden mit Coomassie-Lösung (85% phosphoriger Säure, 757 mM Ammoniumsulfat, 1,2% Coomassie Brilliant Blau, 20% Methanol) für 2 h bei Raumtemperatur oder über Nacht bei 4 ° C.
- Um unspezifische Färbung zu entfernen, waschen, mehrmals mit ddH 2 O.
- Schneiden Sie die Bahnen in 1 mm Gelstücke und fahren mit der In-Gel-Verdauung.
(Alternativ können die Proben ein paar Minuten in einer Vakuumzentrifuge getrocknet und für ein paar Wochen vor der weiteren in-Gel-Verdauung gespeichert.)
8. In-Gel-Verdauung (Geändert von Shevchenko et al. 35)
Bitte beachten Sie, alle Puffer und Lösungen kurz vor der Verwendung vorzubereiten und Glasflaschen für Puffer Acetonitril (ACN) enthalten.
ACHTUNG! Arbeiten mit ACN schädlich sein könnten, mehr Informationen können heruntergeladen werden unter: http://www.sciencelab.com/msds.php?msdsId=9927335 (2013).
- Für 30 Sekunden waschen Gel-Stücke mit> 10 Volumen ddH2O (~ 200 ul).
- Die Gelstücke mit 300 &mgr; l von 25 mM NH 4 HCO 3 für 15 min unter Schütteln inkubieren, Entfernen Flüssigkeit.
- Die Gelstücke mit 300 &mgr; l von 25 mM NH 4 HCO 3 in 50% ACN inkubieren (Herstellung durch Mischen ACN und 50 mM NH 4 HCO 3 in einem Verhältnis von 1:1) für 15 min unter Schütteln entfernen Flüssigkeit.
- Wenn die Gelstücke noch blau, wiederholen der NH 4 HCO 3 und NH 4 HCO 3 / ACN wäscht, bis die meisten der Coomassie entfernt.
(Obwohl der Großteil der Coomassie-Färbung entfernt werden sollte, ist es nicht notwendig, die Gelstücke vollständig entfärben.) - 100 l ACN, um die Gel-Stücke für 5 min zu entwässern. Die Gel-Stücke sollten einen Schrumpfnd schauen ganz weiß.
- Entfernen Sie so viel wie möglich ACN und fahren mit Trypsin Verdauung. Alternativ können die Proben bei -20 ° C für einige Wochen gelagert werden.
- In 10-20 ul pro Band von 20 ng / ul Trypsin in 10% ACN / 25 mM NH 4 HCO 3 (frisch verdünnt), halten Sie Proben auf Eis.
- Nach 30 Minuten prüfen, ob alle Lösung absorbiert wurde und noch mehr Trypsin-Puffer, falls erforderlich. Gel Stücke sollten vollständig mit Trypsin Puffer bedeckt sein. Halten Proben auf Eis.
- Lassen Sie Gel-Stücke für weitere 90 min auf Eis, um sie mit Trypsin zu sättigen.
(Critical Schritt: die Ausbeute an tryptischen Peptiden nimmt mit zunehmender Inkubationszeit auf Eis, wahrscheinlich aufgrund der langsamen Diffusion des Enzyms in der Polyacrylamid-Matrix ist es erforderlich, einen geringen Überschuß an Lösung zu haben für die Gelstücke zu vermeiden, dass die Stücke. fallen trocken während der Inkubation über Nacht.) - Digest bei 37 ° C für 4-6 Stunden. Für Experimente erfordernmaximale Peptidrückgewinnung, über Nacht zu verdauen.
- Nach der Verdauung Spin-Down verkürzte Puffer.
- Ultraschallbad Rohre für 5 min auf Peptide und Zentrifuge wieder eluieren.
- Überstand sammeln und übertragen sie in ein neues Röhrchen.
- Führen Peptid Extraktion mit 80 ul von 30% ACN / 1% Ameisensäure (FA) in einem Ultraschallbad für 15 min.
- Führen Extraktion mit 80 ul von 30% ACN / 1% FA in einem Ultraschallbad für 15 min.
- Führen Extraktion mit 80 ul von 70% ACN / 1% FA in einem Ultraschallbad für 15 min.
- Zentrifuge wieder und Pool-Überstand mit dem Eluat vor.
(FA dient Trypsin zu inaktivieren und erhöhen Peptidlöslichkeit.) - Chemische Peptid-Lösung vollständig in einer Vakuumzentrifuge.
(An dieser Stelle Proben können für ein paar Wochen in -20 ° C, bevor mit MS-Analyse zu speichern.) - Resuspendieren Peptide in 6 ul MS-Puffer (5% ACN, 0,1 v / v FA, Wasser) und beschallen für 2-5 min.
- Centrifuge Proben bei maximum Geschwindigkeit für 5 Minuten, um Partikelmaterial zu entfernen.
- Verwenden von 4 ul Überstand für die massenspektrometrische Analyse.
9. MS-Datenanalyse mit "Proteomatic"
Die Datenanalyse wurde mit dem Open-Source-Software "Proteomatic" (die bei http://www.proteomatic.org/ heruntergeladen werden kann), eine Plattform, die die Erzeugung und Beendigung von MS / MS-Daten Auswertung Rohrleitungen, durch die Verwendung erlaubt geführt freie und kommerzielle Software-36. Kurz gesagt, werden die nachfolgend beschriebenen Einstellungen und mehr Informationen finden Sie in 6,26 gefunden werden.
- Identifizierung und Quantifizierung der MS-Daten
Identifizierung und Quantifizierung von Proteinen aus der Experimente WT vs ΔPSI wurden mit OMSSA (Version 2.1.4 37) und qTrace bzw. 26 erfolgt, wie beschrieben, 6,26. Für die MS-Datenanalyse aus dem Experiment WT vs pgrl1 die folgende aktualisierte Pipeline verwendet:- Neben OMSSA 37, wurden die Proteine auch mit X identifiziert! Tandem (Version 2013.02.01 38). Beide Algorithmen wurden durch die Suche gegen einen durch die Kombination der JGI Chlamydomonas Gen Modell-Datenbank der Version 4.3 mit der AUGUSTUS Datenbank Version 10.2 sowie gegen eine beigetreten Datenbank der NCBI Datenbank angewendet erzeugt Datenbanken BK000554.2 und NC_001638.1.
- MS 2 Identifizierung von Peptiden wurde unter Verwendung einer ziel Köder Ansatz 39 durchgeführt. Ein Köder für jedes Protein wurde durch Mischen zufällig tryptischen Peptide unter Beibehaltung der Redundanz der nonproteotypic Peptiden geschaffen.
- Statistische Validierung der Peptide wurde mit der Software qvality (Version 0.3.3 40) mit einem hinteren Fehlerwahrscheinlichkeit (PEP) Schwellenwert von weniger als 0,01 und die Ergebnisse wurden mit einer Vorläufermasse Toleranz von 5 ppm filtriert geführt.
- Peptide aus OMSSA und X! Tandem-Läufe wurden zusammengefügt und mit qTrace quantifiziert 26 Anwendung der folgenden Filterstufen: 2 Identifizierung erfordern MS, fügen Protein-Namen / Gruppeninformationen und erfordern sowohl Schwester Peptiden.
Um zu untersuchen, ob Protein-Verhältnisse in den gemischten CEF-Superkomplex-Fraktionen von WT und pgrl1 signifikant unterschiedlich zueinander, wurde die statistische Analyse durchgeführt, die Anwendung der Software SPSS (Version 21). Die Untereinheiten des Cytochrom b 6 / f-Komplex wurden gegeneinander als auch die Proteine FNR FTSH2 HCF136 und gegen die PSI-Untereinheiten getestet. Die Peptid-Verhältnisse von jedem Scan Zahl pro Protein von allen vier Wiederholungen wurden auf Normalverteilung mit dem Shapiro-Wilks-Test geprüft. Da die Peptidpopulationen nicht normal verteilt (p <0,001) wurden nichtparametrische Statistik durchgeführt. Zunächst wurde der Kruskal-Wallis-Test angewendet Beurteilung einer erheblichen Unterschiede zwischen den Gruppen. Nur wenn dieser Test vorhergesagt signifikanten Unterschieds zwischen den Gruppen wurde eine weitere Analyse durchgeführt, um signifikante Unterschiede zwischen den beiden unabhängigen Gruppen mit dem Mann-Whitney-U-Test vorherzusagen.
Ergebnisse
Das eingeführte quantitative Proteomik Ansatz zielt darauf ab, die Zusammensetzung des Multiproteinkomplexen in Thylakoidmembranen durch die vergleichende Analyse der CEF-Superkomplex-Komponenten in genetisch verschiedenen C nachgewiesen charakterisieren reinhardtii Stämme. Das beschriebene Verfahren wurde erfolgreich von Terashima et al. 6 angelegt und umfasst die Isolierung der Thylakoidmembran von anaeroben Kulturen gezüchtet, gefolgt von Solubilisierung Waschmittel. Anschließend werden die Proben auf ein Saccharose-Dichtegradienten geladen auf der Grundlage gleicher Höhe und Chlorophyll-Komplexe werden durch Ultrazentrifugation fraktioniert. Zur quantitativen MS-Analyse gleiche Volumina von zwei Gradienten-Fraktionen aus verschiedenen Stämmen vermischt und vergleichend analysiert (Abbildung 1). Die vorgestellten Ergebnisse beinhalten den Vergleich der CEF-Superkomplex Fraktion zwischen WT CW15-Arg7 und ΔPSI sowie WT cc124 und pgrl1 sind.
Für den Vergleich von CEF-Superkomplex Komponenten zwischen WT und dem Stamm ΔPSI Fraktion 6, Beherbergung der CEF-Superkomplex in der WT und Fraktion 13, die Peak-Fraktion von ANR1 im ΔPSI Belastung, wie durch Immunoblot-Analyse ergab, (2A und 2B) wurden ausgewählt, 6. Abbildung 3 zeigt die relative Proteinverhältnisse als WT / ΔPSI (14 N / 15 N) für mehrere PSI-Kern und LHCI Proteine (Lichtsammelproteine der PSI auch als Lhca Proteine genannt) sowie für Proteine mit der CEF-Superkomplex zugeordnet. Wie erwartet, werden PSI-Kernproteine nur in WT-Proben von infinity Verhältnisse in beiden Fraktionen nachgewiesen gefunden. Lhca2 und Lhca9 sind in der WT angereichert, während Lhca4 -6 und -8 eine andere Regelung in beiden Fraktionen zu zeigen. Es ist wichtig zu beachten, dass LHCI Proteine synthetisiert und in der Regel 41 ΔPSI Mutante akkumuliert. Das Polypeptid Vergleich der PSI-LHCIKomplex aus C. reinhardtii WT mit dem LHCI Komplex der ΔPSI Mutante ergab, dass die Mehrheit der Lhca2, -3 und -9 scheint nur schwach gebunden LHCI und ferner, dass für eine stabile Bindung notwendig die Anwesenheit des PSI Kern. Im Gegensatz dazu Lhca1, -4, -6, -7, -8 und einen Komplex unabhängig von der Anordnung der PSI 14 zu bilden, die die Regulation der verschiedenen Lhca4, -6 und -8 in dem vorliegenden Experiment erklären könnte.
Für Proteine mit der CEF-Superkomplex zugeordnet werden, kann es zwischen denen, die in WT Fraktion 6 und Fraktion 13 des ΔPSI Mutante reichlicher unterschieden werden, welches eine starke PSI-abhängigen Migration durch Verhältnisse höher als eine für Fraktion 6 angezeigt und Verhältnisse niedriger als ein oder nicht in der Fraktion 13 erfasst (Lhcbm5, PGRL1, ANR1, CAS, PFD, HCF136 und cyt f) und diejenigen, die nicht so stark, aber dennoch ein PSI-abhängige Lokalisierung (FNR, FTSH1, FTSH2 und ATPC ).
Mit diesem Experiment Terashima et al. 6 zeigte eine PSI-abhängigen Migrationsverhalten für ANR1 und CAS (Fig. 2A-D und 3), wodurch sie als offenbaren neue Proteine der CEF-Superkomplex, der zuvor nicht identifiziert 7. Die Ergebnisse der MS-Analyse wurden durch Immun bestätigt und durch die Anwendung der reversen Genetik unterstützt, um eine funktionelle Rolle dieser Proteine in vivo CEF 6 bestätigen. Darüber hinaus diese Vergleichs MS-Ansatz unterstützt nachdrücklich die Ergebnisse aus früheren Studien, durch die Bestätigung Proteine, die bereits gezeigt haben, einen Teil des CEF-Superkomplex wie PGRL1, cyt f und FNR 7, sowie Lhcbm5 7,9 zu zeigen, eine PSI-abhängige Lokalisation im Saccharose-Dichtegradienten, die vergleichbar ANR1 und CAS ist.
Der Vergleich der Fraktion 6 und 13 ANR1 und ΔPSI Verwendung derselben approach (siehe Abbildung S8 6) zeigten eine ähnliche Zusammensetzung des CEF-Superkomplex in ANR1 und dem gleichen Trend für die Proteine ANR1, CAS und PGRL1 wie in dem Experiment WT vs ΔPSI enthüllt. Insbesondere, obwohl die Protein-Verhältnis von ANR1 in Fraktion 6 (ANR1 vs ΔPSI) ist vergleichbar mit dem Verhältnis der WT vs ΔPSI Experiment wurden die Mengen von CAS und PGRL1 scheinen im früheren Versuch, höher zu sein und könnte ein Ergebnis sein Kompensieren der Herunterregulierung der ANR1 6.
Außerdem ist die Anreicherung von ANR1 und CAS in der CEF-Superkomplex Fraktion könnte man auch ableiten, die PSI-abhängige Migration von mehreren anderen Proteinen aus diesem Experiment. Dies sind zwei ATP-abhängigen Metalloproteasen FTSH1 und FTSH2, die eine grundlegende Rolle bei der Verschlechterung des PSII Reaktionszentrum D1-Protein in den Chloroplasten spielen 42,43, ein Prefoldin-domain containing protein, das HCF136, die ein entscheidender Faktor für die Stabilität oder assembly von PSII 44 und der γ-Untereinheit der ATPase. Während für die später ein, die Verhältnisse zwischen Fraktion 6 und 13 keine so deutlichen Unterschied zeigen, müssen weitere Experimente durchgeführt, um zu überprüfen, ob die anderen Proteine können auch Kandidaten der CEF-Superkomplex sein werden.
Als Beweis für dieses Konzept Experiment vergleichenden Ansatz, analysierten wir die CEF-Superkomplex-Zusammensetzung in zwei Stämme, die beide PSI, so dass die Bildung dieses Komplexes. Die entsprechenden Fraktionen wurden auf der Grundlage der Absorptionsmessung der fraktionierten Gradienten gewählt. Fig. 4A und 4B zeigen die Ergebnisse der quantitativen Vergleich zwischen WT und einer pgrl1 Knockout-Stamm. Es sei darauf hingewiesen, dass ANR1 und CAS nicht merklich zwischen WT und pgrl1 geändert werden. Interessanterweise scheint HCF136 im WT angereichert werden, während FTSH2 scheint in pgrl1 angereichert werden. Eine statistisch significant Differenz für beide oben genannten Proteine auf alle PSI-Untereinheiten (einschließlich der LHCI Proteine) bestätigt werden.
Darüber hinaus cyt die Cytochrom b 6 / f-Komplex-Untereinheiten f und PETO gezeigt wurden signifikant von cyt b 6, cyt b 6 / f IV und PETC bzw. zu sein. Bemerkenswert ist, scheint cyt f zu sein in pgrl1 hochreguliert, während cyt b 6 und cyt b 6 / f IV sind etwas nach unten reguliert, obwohl die Regelung der cyt f Synthese beinhaltet wahrscheinlich cyt b 6 und cyt b 6 / f IV 45 . Die Atom-codierte PETC, die auch als Rieske Proteins bekannt ist zeigt eine ähnliche Regelung wie cyt b 6 und cyt b 6 / f IV (dh eine Down-Regulation in pgrl1). Dies wird als eine Verringerung oder die Abwesenheit des Proteins Rieske fällt noch zur Ansammlung des OTH führener cyt b 6 / f-Untereinheiten zu ~ 60% der WT-Ebene in C. 46 reinhardtii. Zusätzlich ist die Hochregulation von PETO in pgrl1 wichtig zu beachten, da diese locker gebundene Untereinheit wurde gezeigt, zu verringerten Niveaus in C akkumulieren reinhardtii Mutanten, denen entweder cyt b 6, cyt b 6 / f IV oder PETC 47, die in pgrl1 weniger häufig im Vergleich zu den WT im vorliegenden Experiment sind.
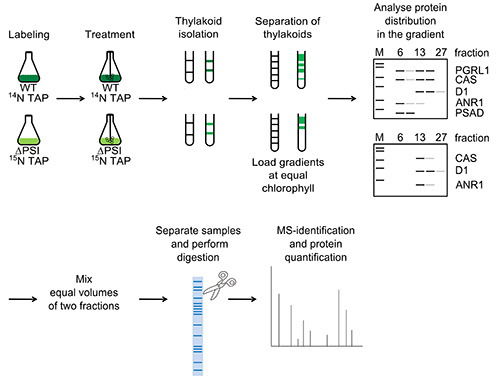
1. Versuchsablauf wie beim WT und ΔPSI dargestellt. Zellen metabolisch für mindestens vier Generationen markiert sind anaeroben Bedingungen durch Einleiten von Argon 4 Stunden lang induziert und Thylakoidmembranen werden durch Gradienten-Trennung isoliert. Die isolated Membranen mit β-DM gelöst, auf ein Saccharose-Dichte-Gradienten (für beide Stämme gleiche Mengen von Chlorophyll in diesem Schritt angewendet wird) geladen und über Nacht zentrifugiert. Die Gradienten-Fraktionen werden durch SDS-PAGE als auch die Immundetektion zur Verteilung von Proteinen innerhalb des Gradienten zu bestimmen, gesammelt und analysiert. Proteine mit einem PSI-abhängigen Migration gleicher Volumina der WT CEF-Superkomplex Fraktion (Fraktion Nr. 6) und dem entsprechenden 15 N-markierten Fraktion aus der ΔPSI Stamm identifizieren gemischt und vergleichend analysiert. Das gleiche wurde mit der Peakfraktion von ANR1 im ΔPSI Stamm (Fraktion Nr. 13) durchgeführt. Für die vergleichende quantitative MS-Ansatz wurden die gemischten Proteinproben durch SDS-PAGE getrennt, Proteinbanden wurden mit Coomassie-Blau-Lösung gefärbt, gefolgt von in-Gel-Verdauung vor MS-Analysen. Klicken Sie hier, um größere ima ansehenge.
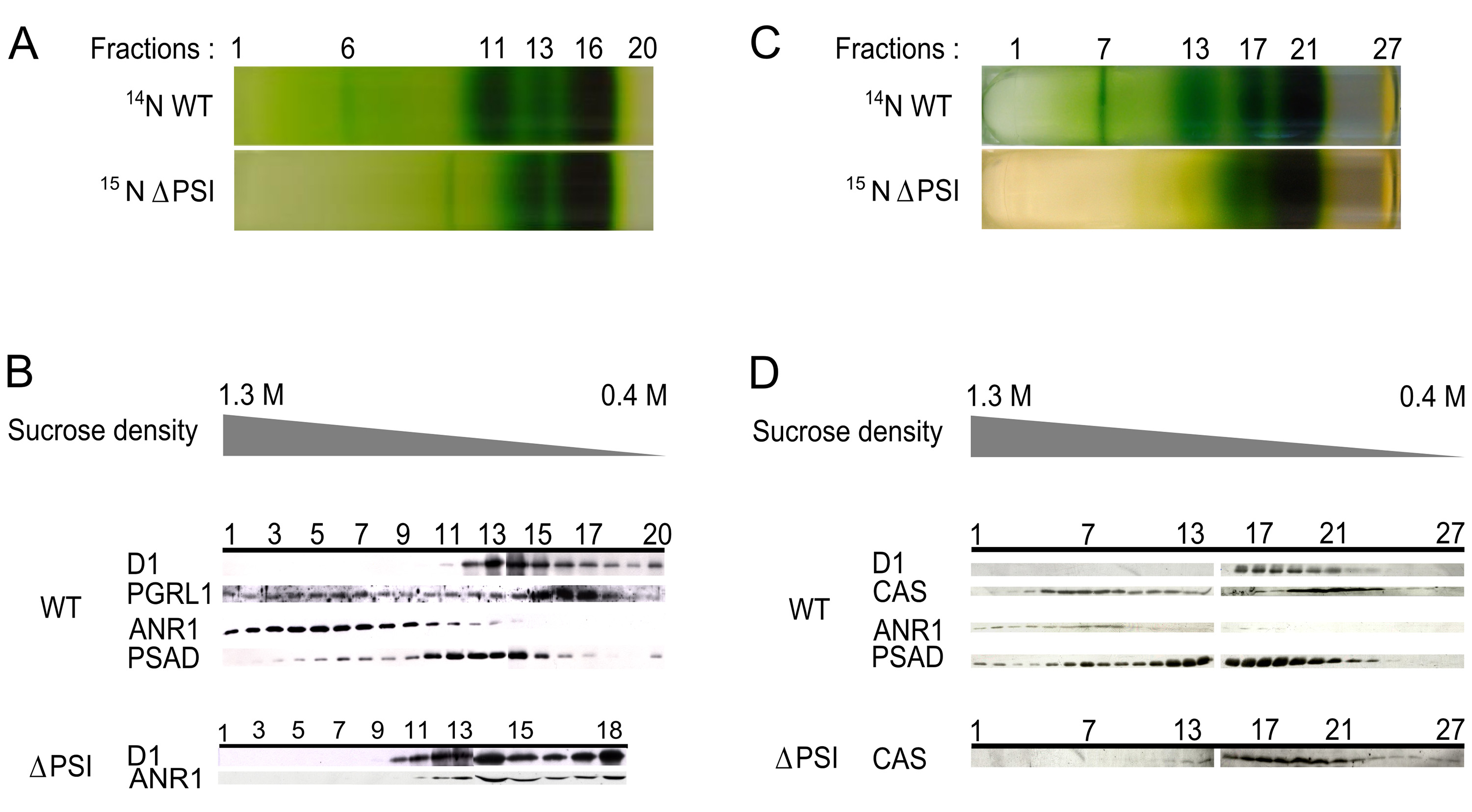
Abbildung 2. ANR1 CAS und wandern in die unteren Regionen in der Dichte ΔPSI Belastung. A:. Saccharose-Dichtegradienten von WT und ΔPSI Thylakoide aus anaeroben Bedingungen isoliert wurden in 20 Fraktionen getrennt B: Immundetektion von ANR1 in den 20 Fraktionen des Gradienten von WT und ΔPSI. Während dieses Protein der höheren Dichte in der Region lokalisierte WT (Fraktion 6) ist an der unteren Dichtebereiche in der ΔPSI Stamm (Fraktion 13) nach oben verschobenen C:. Sucrose-Dichtegradienten von WT und ΔPSI Thylakoide isoliert aus in den 27 Fraktionen des Gradienten von WT-und Immundetektion von ΔPSI CAS: anaeroben Bedingungen wurden in 27 Fraktionen getrennt D.. Während dieses Protein lokalisiert to die höhere Dichte im Bereich WT (Höhepunkt um Fraktion 7) ist an den unteren Dichtebereiche in der ΔPSI Stamm hochgeschaltet (Höhepunkt um Fraktion 19). (Diese Zahl hat sich von Terashima et al. 6 geändert wurde). Klicken Sie hier für eine größere Ansicht.

Abbildung 3. Relative Protein-Verhältnissen in Fraktion 6 bestimmt und 13 von WT und ΔPSI durch quantitative Massenspektrometrie. Fraktionen 6 und 13 von WT haben mit den jeweiligen Fraktionen, aus den 15 N-markierten ΔPSI Belastung verglichen. Die relativen Protein-Verhältnissen, die zwei biologischen Replikaten und drei MS-Läufen vertreten sind als WT / ΔPSI (14 dargestelltN / 15 N) sowohl analysierten Fraktionen mit Ausnahme von cyt f, die nur in Fraktion 6 identifiziert wurde. Diese vergleichende Quantifizierung zeigt, dass ANR1 und CAS zeigen eine PSI-abhängige Migration in der Saccharose-Dichtegradienten (diese Zahl hat sich von Terashima et al. 6 geändert wurde). Klicken Sie hier für eine größere Ansicht.

Abbildung 4. Relative Protein-Verhältnisse in der CEF-Superkomplex Fraktion von WT und pgrl1 durch quantitative Massenspektrometrie bestimmt. A: Saccharose-Dichtegradienten von WT und pgrl1 Thylakoide aus anaeroben Bedingungen isoliert. B: Relative Protein-Verhältnisse zwischen die CEF-Superkomplex Fraktion aus vier verschiedenen Wiederholungen ergeben dargestellt, als WT / pgrl 1 (14 N / 15 N). In diesem Experiment ANR1 und CAS sind nicht bemerkenswert verändert, während FTSH2 und HCF136 signifikante Unterschiede im Vergleich zu allen PSI-Untereinheiten (p ≤ 0,001 als durch *** rot gekennzeichnet; HCF136: 1900 b 6 / f-Komplex-Untereinheiten f und PETO gezeigt wurden signifikant von cyt b 6, cyt b 6 / f IV und PETC (p ≤ 0,001 ist, wie durch *** in blau angegeben; cyt f : 2684 Klicken Sie hier für eine größere Ansicht.
Diskussion
Verschiedene quantitative Proteomik-Studien unter Verwendung von stabilen Isotopenmarkierung sind in den letzten Jahren veröffentlicht worden. In diesen Versuchen in der Regel zwei verschiedenen Proben verglichen werden, von denen eine Probe mit einem stabilen Isotop markiert. Danach Proteine oder Peptide, die von den beiden Proben werden in einem gleichen Verhältnis vereinigt und gemeinsam weiter verarbeitet 48. Solche Studien wollen oft definiert isoliert zellulären Kompartimenten (zB Chloroplasten, Mitochondrien, oder Thylakoidmembranen), um unterschiedliche Stressbedingungen zu untersuchen, 26,34,49 ausgesetzt vergleichen Up-oder Down-Regulation von spezifischen Proteinen. Die beschriebenen Vergleichs quantitative Proteomik Ansatz zielt darauf ab, in der Thylakoidmembran Analyse der Zusammensetzung von Multiproteinkomplexen, und im Gegensatz zu den oben genannten Studien wird beim Vermischen des gleichen Volumens von Proben nach der Sucrose-Dichtetrennung von Thylakoidmembranen in gleichen Chlorophyllkonzentration vor geladen basierend ultracentrifugation. Dieses Verfahren wurde effektiv durch Terashima et al. 6, die vergleichend analysiert die Zusammensetzung der CEF-Superkomplex aus einem WT und einer ΔPSI Stamm Proteine migrieren PSI abhängig in einem Saccharose-Dichtegradienten isoliert identifizieren aufgebracht.
Der Erfolg dieser Strategie wird durch die Tatsache, dass die MS-Ergebnisse werden durch Immundetektion unterstützt bewährt. Zusätzlich Terashima et al. Konnten die Erkenntnisse aus früheren Studien bestätigen (dh die Identifikation und PSI-abhängige Migration von bereits beschrieben, einen Teil des CEF-Superkomplex, wie PGRL1, FNR und cyt f 7 sein, Proteine). Die Identifizierung von ANR1 und CAS als neuartige Komponenten des CEF-Maschinen 6 zeigt diesen Ansatz als ein sehr leistungsfähiges und effizientes Tool. Bemerkenswert ist, das isolierte CEF-Superkomplex in vitro-Aktivität zeigte, wodurch die erfolgreiche Reinigung einer Funktion zeigentional Multiproteinkomplex 6. Die Anwendbarkeit dieser Ansatz auch für zwei Stämme, die beide bilden eine CEF-Superkomplex wird durch den Vergleich zwischen WT und pgrl1 demonstriert.
Im Allgemeinen kann dieses Verfahren verwendet werden, um komplexe Zusammensetzungen in verschiedenen Stämmen oder Bedingungen vermessen werden, wie durch die Identifizierung von bisher nicht identifizierte Proteine, die mit dem CEF-Superkomplex Fraktion (dh die Anreicherung FTSH1, FTSH2, PFD und HCF136 in der gemeinsam gereinigten enthüllt CEF-Superkomplex Fraktion). Ob diese Proteine sind mögliche neue Kandidaten dieser komplexen und unser Verständnis für die unterschiedliche Regelung der FTHS2, HCF136 für die cyt b 6 / f-Untereinheiten zu verbessern, als auch durch den Vergleich von WT und pgrl1 ergab, sind weitere Experimente nötig.
Neben den beschriebenen Vorteile dieses Ansatzes gibt es several kritischen Schritte, die sorgfältig geprüft werden müssen, wenn die Arbeit mit diesem Protokoll: Das Brechen von Zellen mit dem Vernebler ist der erste wichtige Schritt. Wenn die Öffnung der Zellen nicht in der richtigen Weise durchgeführt wird, wird die Ausbeute der isolierten Thylakoide eher gering und könnte in kaum einem Nachweis des CEF-Superkomplex, nachdem über Nacht Zentrifugation führen. Die erfolgreiche Zellaufschluss aus der hellgrünen Farbe der Überstand nach dem Zentrifugieren entnommen werden. Im Gegensatz dazu, wenn der Druck während der Zellaufschluss zu hoch ist, Teile des Superkomplex möglicherweise zerfallen und wichtige Cofaktoren können verloren gehen. Ein weiterer Schritt beeinflussen die Ausbeute der Thylakoide ist die Aufwirbelung von Zellen mit einem Töpfer. Dies muss sorgfältig durchgeführt werden und am Ende dieses Schritts nur einige grüne Partikel bleiben sollte. Weiterhin können zur Herstellung der Saccharose-Dichtegradienten am Tag vor als auch für die Solubilisierung von Membrankomplexe, ist es sehr wichtig,dieses Protokoll genau folgen in Bezug auf die β-DM-Konzentration, denn dies ist entscheidend, um vollständig löslich Thylakoide 50 zu erhalten und damit auch für die Reinigung des CEF-Superkomplex. Der Lösungsschritt erfolgreich ausgeführt worden ist, wenn eine kleine weißliche Pellet wird nach dem anschließenden Zentrifugationsschritt beobachtet. Ein weiterer kritischer Punkt, der hier erwähnt werden sollte, ist die Zeit der über Nacht Ultrazentrifugation. Die Saccharose-Dichtegradienten sollten mindestens zwölf Stunden zentrifugiert, um eine vollständige Trennung der photoKomplexe erzielen.
Auch wenn das Protokoll unter Verwendung von C nachgewiesen reinhardtii für die vergleichende Analyse der CEF-Superkomplex-Zusammensetzung in zwei genetisch verschiedene Stämme, ist es leicht anpassbar auch für ein breites Spektrum von Fragen, wie die vergleichende Analysen von Multi komplexe Zusammensetzung aus unterschiedlichen Umwelt / experimental isoliertBedingungen oder durch Verwendung anderer Typen von Fraktionierung. Die einzigen Anforderungen dieses Ansatzes sind, dass die Modellorganismus in Zellkultur kultiviert werden, ist in der Lage, Ammonium als Stickstoffquelle und Proteinkomplexe können durch Saccharose-Dichtegradienten getrennt werden, was auf eine breite Anwendung dieses Verfahrens. Für die zukünftige Anwendung dieses Ansatzes SDS-PAGE-Auftrennung der Misch 14 N / 15 N-Proben und anschließende In-Gel-Verdauung konnte durch die Anwendung der Filtergestützten Probenvorbereitung (FASP)-Verfahren ersetzt werden. Dieses Verfahren schließt die Anwendung der starke Reinigungsmittel zur Solubilisierung mit dem Ziel, "aufzuräumen", bevor das Proteom die Verdauung und den gereinigten Peptide zu erhalten, verhindert, dass die Nachteile des In-Gel-Ansatz, der die Verdauung Peptid Erholung hemmen können, obwohl man, dass im Gel betrachten Verdauung beschrieben robust gegen Verschmutzungen, die bei der Verdauung 51 stören könnte sein.
Offenlegungen
Die Autoren erklären, keine konkurrierenden Finanz Interesse.
Danksagungen
MH dankt für die Unterstützung von der "Deutschen Forschungsgemeinschaft" (DFG). Autor Beiträge: MH entworfen Forschung; KT, JS und MT durchgeführt Forschung und analysiert die Daten, KT und MH schrieb die Zeitung.
Materialien
| Name | Company | Catalog Number | Comments |
| Chemicals | |||
| Acetic acid | AppliChem | A0662 | http://www.applichem.com/home/ |
| Acetone | AppliChem | A2300 | http://www.applichem.com/home/ |
| Acetonitrile Optigrade für LC-MS | Diagonal | 9340 | Harmful, work with gloves. See protocol text for further precautions. https://www.diagonal.de/ |
| Ammonium chloride 15N | Cambridge Isotope Laboratories | 39466-62-1 | http://www.isotope.com/cil/index.cfm |
| Ammonium chloride 14N | AppliChem | A0988 | http://www.applichem.com/home/ |
| Ammonium hydrogen phosphate | AppliChem | A3583 | http://www.applichem.com/home/ |
| Ammonium sulfate | AppliChem | A3598 | http://www.applichem.com/home/ |
| Coomassie brilliant blue R-250 | Fisher Scientific | 10041653 | http://www.de.fishersci.com/index.php/deindex |
| n-Dodecyl-β-D-maltoside | AppliChem | A0819 | http://www.applichem.com/home/ |
| EDTA | AppliChem | A2937 | http://www.applichem.com/home/ |
| Formic acid | AppliChem | A3858 | http://www.applichem.com/home/ |
| HEPES | AppliChem | A3724 | http://www.applichem.com/home/ |
| Magnesium chloride | AppliChem | A4425 | http://www.applichem.com/home/ |
| Methanol | AppliChem | A2954 | http://www.applichem.com/home/ |
| Phosphorous acid | AppliChem | A0989 | http://www.applichem.com/home/ |
| Dipotassium hydrogen phosphate | AppliChem | A1042 | http://www.applichem.com/home/ |
| Potassium dihydrogen phosphate | AppliChem | A1043 | http://www.applichem.com/home/ |
| Sodium hydroxide | AppliChem | A1551 | http://www.applichem.com/home/ |
| Sucrose | AppliChem | A1125 | http://www.applichem.com/home/ |
| Tricine | AppliChem | A3954 | http://www.applichem.com/home/ |
| Tris | AppliChem | A2264 | http://www.applichem.com/home/ |
| Trypsin (sequencing grade modified) and Trypsin buffer | Promega | V5111 | http://www.promega.de/ |
| Equipment | |||
| Nebulizer (BioNeb cell disruptor) | Glas-Col | http://www.glascol.com/product/subproduct/id/75 | |
| Centrifuge tubes (14 mm x 89 mm) | Beckman Coulter | 331372 | for preparation of Takahashi style gradients
http://www.beckmancoulter.de/ |
| Centrifuge tubes 25 mm x 89 mm | Beckman Coulter | 344058 | for preparation of thylakoid isolation gradients.
http://www.beckmancoulter.de/ |
| Coulter Avanti Centrifuge J-20 XP | Beckman Coulter | http://www.beckmancoulter.de/ | |
| Fuchs-Rosenthal cell couting chamber | Diagonal | 449/72 | https://www.diagonal.de/ |
| Homogenizer (Potter) 50 ml | Fisherbrand | 10618242 | http://www.de.fishersci.com/index.php/defisherbrand |
| Pistil for homogenizer | Fisherbrand | 105252220 | http://www.de.fishersci.com/index.php/defisherbrand |
| Ultracentrifuge (Optima XPN-80 Ultracentrifuge) | Beckman Coulter | website: http://www.beckmancoulter.de/ | |
| Other | |||
| Antibodies | Agrisera | http://www.agrisera.com/en/index.html |
Referenzen
- Joliot, P., Joliot, A. Cyclic electron flow in C3 plants. Biochim. Biophys. Acta. 1757 (5-6), 362-368 (2006).
- Shikanai, T. Cyclic electron transport around photosystem I: genetic approaches. Annu. Rev. Plant Biol. 58, 199-217 (2007).
- Finazzi, G., Furia, A., Barbagallo, R. P., Forti, G. State transitions, cyclic and linear electron transport and photophosphorylation in Chlamydomonas reinhardtii. Biochim. Biophys. Acta. 1413 (3), 117-129 (1999).
- Tolleter, D., et al. Control of hydrogen photoproduction by the proton gradient generated by cyclic electron flow in Chlamydomonas reinhardtii. Plant Cell. 23 (7), 2619-2630 (2011).
- Hertle, A. P., et al. PGRL1 Is the Elusive Ferredoxin-Plastoquinone Reductase in Photosynthetic Cyclic Electron Flow. Mol. Cell. 49 (3), 511-523 (2013).
- Terashima, M., et al. Calcium-dependent regulation of cyclic photosynthetic electron transfer by a CAS, ANR1, and PGRL1 complex. PNAS. 109 (43), 17717-17722 (2012).
- Iwai, M., Takizawa, K., Tokutsu, R., Okamuro, A., Takahashi, Y., Minagawa, J. Isolation of the elusive supercomplex that drives cyclic electron flow in photosynthesis. Nature. 464 (7292), 1210-1213 (2010).
- Chua, N. H., Bennoun, P. Thylakoid Membrane Polypeptides of Chlamydomonas reinhardtii: Wild-Type and Mutant Strains Deficient in Photosystem II Reaction Center. PNAS. 72 (6), 2175-2179 (1975).
- Takahashi, H., Iwai, M., Takahashi, Y., Minagawa, J. Identification of the mobile light-harvesting complex II polypeptides for state transitions in Chlamydomonas reinhardtii. PNAS. 103 (2), 477-482 (2006).
- Ikeuchi, M., Plumley, F. G., Inoue, Y., Schmidt, G. W. Phosphorylation of Photosystem II Components, CP43 Apoprotein, D1, D2, and 10 to 11 Kilodalton Protein in Chloroplast Thylakoids of Higher Plants. Plant Physiol. 85 (3), 638-642 (1987).
- Ruban, A. V., Lee, P. J., Wentworth, M., Young, A. J., Horton, P. Determination of the Stoichiometry and Strength of Binding of Xanthophylls to the Photosystem II Light Harvesting Complexes. J. Biol. Chem. 274 (15), 10458-10465 (1999).
- Barera, S., Pagliano, C., Pape, T., Saracco, G., Barber, J. Characterization of PSII-LHCII supercomplexes isolated from pea thylakoid membrane by one-step treatment with α- and β-dodecyl-D-maltoside. Phil. Trans. R. Soc. B. 367 (1608), 3389-3399 (2012).
- Kantzilakis, K., et al. A comparative approach towards thylakoid membrane proteome analysis of unicellular green alga Scenedesmus obliquus. Biochim. Biophys. Acta. 1768 (9), 2271-2279 (2007).
- Takahashi, Y., Yasui, T., Stauber, E. J., Hippler, M. Comparison of the subunit compositions of the PSI-LHCI supercomplex and the LHCI in the green alga Chlamydomonas reinhardtii. Biochemistry. 43 (24), 7816-7823 (2004).
- Thelen, J. J., Peck, S. C. Quantitative proteomics in plants: choices in abundance. Plant Cell. 19 (11), 3339-3346 (2007).
- Hsieh, S., et al. The Proteome of Copper, Iron, Zinc, and Manganese Micronutrient Deficiency in Chlamydomonas reinhardtii. Mol. Cell. Proteom. 12 (1), 65-86 (2013).
- Longworth, J., Noirel, J., Pandhal, J., Wright, P. C., Vaidyanathan, S. HILIC- and SCX-based quantitative proteomics of Chlamydomonas reinhardtii during nitrogen starvation induced lipid and carbohydrate accumulation. J. Proteome Res. 11 (12), 5959-5971 (2012).
- Malasarn, D., et al. Zinc deficiency impacts CO2 assimilation and disrupts copper homeostasis in Chlamydomonas reinhardtii. J. Biol. Chem. 288 (15), 10672-10683 (2013).
- Höhner, R., et al. The metabolic status drives acclimation of iron deficieny responses in Chlamydomonas reinhardtii as revealed by proteomics based hierarchical clustering and reverse genetics. Mol. Cell. , (2013).
- Mahong, B., Roytrakul, S., Phaonaklop, N., Wongratana, J., Yokthongwattana, K. Proteomic analysis of a model unicellular green alga, Chlamydomonas reinhardtii, during short-term exposure to irradiance stress reveals significant down regulation of several heat-shock proteins. Planta. 235 (3), 499-511 (2012).
- Förster, B., Mathesius, U., Pogson, B. J. Comparative proteomics of high light stress in the model alga Chlamydomonas reinhardtii. Proteomics. 6 (15), 4309-4320 (2006).
- Petroutsos, D., et al. The chloroplast calcium sensor CAS is required for photoacclimation in Chlamydomonas reinhardtii. Plant Cell. 23 (8), 2950-2963 (2011).
- Dodd, A. N., Kudla, J., Sanders, D. The language of calcium signaling. Annu. Rev. Plant Biol. 61, 593-620 (2010).
- Nomura, H., Komori, T., Kobori, M., Nakahira, Y., Shiina, T. Evidence for chloroplast control of external Ca2+-induced cytosolic Ca2+ transients and stomatal closure. Plant J. 53 (6), 988-998 (2008).
- Weinl, S., et al. A plastid protein crucial for Ca2+-regulated stomatal responses. New Phytol. 179 (3), 675-686 (2008).
- Terashima, M., Specht, M., Naumann, B., Hippler, M. Characterizing the anaerobic response of Chlamydomonas reinhardtii by quantitative proteomics. Mol. Cell. Proteomics. 9 (7), 1514-1532 (2010).
- Petroutsos, D., et al. PGRL1 participates in iron-induced remodeling of the photosynthetic apparatus and in energy metabolism in Chlamydomonas reinhardtii. J. Biol. Chem. 284 (47), 32770-32781 (2009).
- DalCorso, G., et al. A complex containing PGRL1 and PGR5 is involved in the switch between linear and cyclic electron flow in Arabidopsis. Cell. 132 (2), 273-285 (2008).
- Redding, K., et al. A systematic survey of conserved histidines in the core subunits of Photosystem I by site-directed mutagenesis reveals the likely axial ligands of P700. EMBO J. 17 (1), 50-60 (1998).
- Harris, E. H. . The Chlamydomonas Sourcebook. Introduction to Chlamydomonas and its laboratory use. , (2008).
- Porra, R. J., Thompson, W. A., Kriedemann, P. E. Determination of accurate extinction coefficients and simultaneous equations for assaying chlorophylls a and b extracted with four different solvents: verification of the concentration of chlorophyll standards by atomic absorption spectroscopy. Biochim. Biophys. Acta. 975 (3), 384-394 (1989).
- Naumann, B., Stauber, E. J., Busch, A., Sommer, F., Hippler, M. N-terminal processing of Lhca3 Is a key step in remodeling of the photosystem I-light-harvesting complex under iron deficiency in Chlamydomonas reinhardtii. J. Biol. Chem. 280 (21), 20431-20441 (2005).
- Hippler, M., Klein, J., Fink, a., Allinger, T., Hoerth, P. Towards functional proteomics of membrane protein complexes: analysis of thylakoid membranes from Chlamydomonas reinhardtii. Plant J. 28 (5), 595-606 (2001).
- Naumann, B., et al. Comparative quantitative proteomics to investigate the remodeling of bioenergetic pathways under iron deficiency in Chlamydomonas reinhardtii. Proteomics. 7 (21), 3964-3979 (2007).
- Shevchenko, A., Tomas, H., Havlis, J., Olsen, J. V., Mann, M. In-gel digestion for mass spectrometric characterization of proteins and proteomes. Nat. Prot. 1 (6), 2856-2860 (2006).
- Specht, M., Kuhlgert, S., Fufezan, C., Hippler, M. Proteomics to go: Proteomatic enables the user-friendly creation of versatile MS/MS data evaluation workflows. Bioinformatics. 27 (8), 1183-1184 (2011).
- Geer, L. Y., et al. Open mass spectrometry search algorithm. J. Proteome Res. 3 (5), 958-964 (2004).
- Craig, R., Beavis, R. C. TANDEM: matching proteins with tandem mass spectra. Bioinformatics. 20 (9), 1466-1467 (2004).
- Elias, J. E., Gygi, S. P. Target-decoy search strategy for increased confidence in large-scale protein identifications by mass spectrometry. Nat. Methods. 4 (3), 207-214 (2007).
- Käll, L., Storey, J. D., Noble, W. S. QVALITY: non-parametric estimation of q-values and posterior error probabilities. Bioinformatics. 25 (7), 964-966 (2009).
- Wollman, F. A., Bennoun, P. A new chlorophyll-protein complex related to photosystem I in Chlamydomonas reinhardtii. Biochim. Biophys. Acta. 680, 352-360 (1982).
- Bailey, S., et al. A critical role for the Var2 FtsH homologue of Arabidopsis thaliana in the photosystem II repair cycle in vivo. J. Biol. Chem. 277 (3), 2006-2011 (2002).
- Kato, Y., Sakamoto, W. Protein quality control in chloroplasts: a current model of D1 protein degradation in the photosystem II repair cycle. J. Biochem. 146 (4), 463-469 (2009).
- Meurer, J., Plücken, H., Kowallik, K. V., Westhoff, P. A nuclear-encoded protein of prokaryotic origin is essential for the stability of photosystem II in Arabidopsis thaliana. EMBO J. 17 (18), 5286-5297 (1998).
- Kuras, R., Wollman, F. A. The assembly of cytochrome b6/f complexes: an approach using genetic transformation of the green alga Chlamydomonas reinhardtii. EMBO J. 13 (5), 1019-1027 (1994).
- de Vitry, C., Finazzi, G., Baymann, F., Kallas, T. Analysis of the nucleus-encoded and chloroplast-targeted rieske protein by classic and site-directed mutagenesis of Chlamydomonas. Plant Cell. 11 (10), 2031-2044 (1999).
- Hamel, P., Olive, J., Pierre, Y., Wollman, F. A., de Vitry, C. A new subunit of cytochrome b6f complex undergoes reversible phosphorylation upon state transition. J. Biol. Chem. 275 (22), 17072-17079 (2000).
- Stanislas, T., et al. Quantitative proteomics reveals a dynamic association of proteins to detergent-resistant membranes upon elicitor signaling in tobacco. Mol. Cell. Proteomics. 8 (9), 2186-2198 (2009).
- Atteia, A., et al. A proteomic survey of Chlamydomonas reinhardtii mitochondria sheds new light on the metabolic plasticity of the organelle and on the nature of the alpha-proteobacterial mitochondrial ancestor. Mol. Biol. Evol. 26 (7), 1533-1548 (2009).
- Pagliano, C., Barera, S., Chimirri, F., Saracco, G., Barber, J. Comparison of the α and β isomeric forms of the detergent n-dodecyl-D-maltoside for solubilizing photosynthetic complexes from pea thylakoid membranes. Biochim. Biophys. Acta. 1817 (8), 1506-1515 (2012).
- Wisniewski, J. R., Zougman, A., Nagaraj, N., Mann, M. Universal sample preparation method for proteome analysis. Nat. Methods. 6 (5), 3-7 (2009).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten