Method Article
Metabolische Markierung und Membran Fraktionierung für Vergleichende Proteomanalyse von
In diesem Artikel
Zusammenfassung
Hier beschreiben wir ein robustes Verfahren für die Fraktionierung von pflanzlichen Plasmamembranen in Waschmittel-und Reinigungsmittel beständig löslichen Membranen auf der Basis einer Mischung von unmarkierten und in vivo vollständig 15 N markierten Arabidopsis thaliana Zellkulturen. Das Verfahren ist für die vergleichende Proteom-Studien angewendet, um Signalprozesse zu verstehen.
Zusammenfassung
Plasmamembran-Mikrodomänen sind Funktionen, basierend auf den physikalischen Eigenschaften der Lipid-Sterol-und Umwelt und haben bestimmte Rollen in Signalprozessen. Extrahieren Sterol angereicherte Membrandomänen von Pflanzenzellen zur Proteom-Analyse ist eine schwierige Aufgabe, vor allem aufgrund von mehreren Vorbereitungsschritte und Quellen für Verunreinigungen von anderen zellulären Kompartimenten. Die Plasmamembran bildet nur etwa 5-20% aller Membranen in einer Pflanzenzelle, und daher Trennung von hoch gereinigten Plasmamembranfraktion ist eine Herausforderung. Ein häufig verwendetes Verfahren beinhaltet wässrigen Zweiphasentrennung in Polyethylenglykol und Dextran, das Plasmamembran-Vesikel mit einer Reinheit von 95% 1 ergibt. Sterol-reichen Membranmikrodomänen in der Plasmamembran unlöslich sind bei der Behandlung mit kaltem nichtionische Detergentien bei alkalischen pH-Wert. Dieser Detergenz-resistenten Membranfraktion aus dem Großplasmamembran durch Ultrazentrifugation getrennt werden, wie inucrose Steigung 2. Anschließend kann Proteine aus der geringen Dichte Band des Saccharose-Gradienten von Methanol / Chloroform Fällung extrahiert werden. Extrahierte Protein wird dann Trypsin gespalten werden, entsalzt und schließlich durch LC-MS/MS analysiert. Unsere Extraktionsprotokoll für Sterol-reiche Mikrodomänen wird zur Herstellung von sauberen Detergenz-resistenten Membranfraktionen aus Arabidopsis thaliana Zellkulturen optimiert.
Wir verwenden vollständige metabolische Markierung von Arabidopsis thaliana Suspensionszellkulturen, die mit K 15 NO 3 als einzige Stickstoffquelle für die quantitative Proteomik vergleichende Studien nach der biologischen Behandlung von Zins 3. Durch Mischen gleicher Verhältnisse von markierten und unmarkierten Zellkulturen für die gemeinsame Proteinextraktion der Einfluss der Herstellungsschritte auf die endgültige quantitative Ergebnis wird auf einem Minimum gehalten. Auch Materialverlust während der Extraktion werden die beiden Kontroll-und Behandlungs Proben in der gleichen Weise einen Einflussd daher das Verhältnis von Licht und heben Peptid konstant. Bei dem vorgeschlagenen Verfahren entweder markiert oder unmarkiert Zellkultur erfährt eine biologische Behandlung, die andere dient als Steuer 4.
Einleitung
Im Jahr 1972, Jonathan Singer und Garth Nicolson schlug die Flüssig-Mosaik-Modell ein Strukturmodell von Zellmembranen, anstelle der Protein-Lipid-Protein-Sandwich-Modell, das in der Regel in den frühen 1960er Jahren angenommen wurde. Singer und Nicolson postuliert, dass die biologische Membran kann als eine zweidimensionale Flüssigkeit in dem alle Lipid-und Protein-Moleküle frei und leicht diffundieren 5 betrachtet werden. Seit dieser Zeit Strukturmodell der Plasmamembran und der Kenntnis der Membranzusammensetzung wurde noch komplexer. Insbesondere in der Plasmamembran-Strukturen, wie Protein-und Lipidkomplexe / Sterol basierend strukturell ungeordnete Mikrodomänen beobachtet werden. In künstlichen Modellmembranen 6,7 kann Sterole und Sphingolipide seitlich von anderen Lipidarten zu trennen, um Bereiche mit veränderten physikalischen Eigenschaften bilden. Diese Trennung innerhalb der Zellmembran wird hauptsächlich durch die Eigen Zuordnen Eigenschaften zwischen Sterole und einen hoch verursachtturated Kohlenwasserstoffketten phopsho-und Sphingolipide 8. Insbesondere die starren Sterin Ringe begünstigen Wechselwirkungen mit steifer und gerader gesättigten Lipidspezies und diese Wechselwirkungen zwingen benachbarten Kohlenwasserstoffketten in mehr verlängert Konformationen, die Erhöhung der Membrandicke und Härte.
Eine der häufigsten beobachteten Merkmale Terinmembrandomänen war ihrer Unlöslichkeit bei Behandlung mit nicht-ionischen Detergenzien, wie Triton X-100 und Brij 35. Diese Fraktionen wurden vermutlich identisch mit Membran-Mikrodomänen und wurden Reinigungsmittel resistente Membranen (DRM) genannt auf der Grundlage ihrer biochemischen Herstellungsverfahren 2. Die Verwendung von nichtionischen Reinigungsmitteln während der DRM-Extraktion erhielt einige Kritik als die biochemische DRM Zubereitung kann nicht direkt an spezifische Membranraum innerhalb der lebenden Zelle 9 entsprechen. Insbesondere scheint das Detergens zu Protein-Verhältnis von entscheidender Bedeutung in solchen Zubereitungen eins verschiedene Reinigungsmittel, sowie verschiedene Reinigungs Mengen können unterschiedliche Zusammensetzung der Reinigungsmittel beständig Membranfraktion 10 ergeben. Es gibt jedoch Hinweise darauf, dass bestimmte Proteinspezies spezifisch zu assoziieren mit diesen zellulären sterolreichen Membrandomänen, und daß diese Proteine auch in der biochemischen Präparaten von Detergenz-resistenten Membranfraktionen 11 angereichert. Der Kern von Proteinen, die in Pflanzen DRM-Fraktion gefunden wurden, und bei denen die Anwesenheit in DRM war Sterol abhängig waren besonders GPI-verankerte Proteine, wie Fasciclin artigen Arabinogalactan-Proteine (FLA) und Mitglieder der SKU-Proteinfamilie. Auch einige Signalproteine, wie Rezeptor-like Kinasen oder Phospholipasen gefunden 11. Diese Ergebnisse sind konsistent mit vielen proteomischen Untersuchungen an Säugermembrandomänen 12,13. Auch bei Pflanzen gibt es immer mehr Hinweise auf die Rolle von Membran-Mikrodomänen in Zusammenhang mit Stress-Reaktion 14 -16.
Das hier beschriebene Protokoll bietet ein robustes Verfahren zur Fraktionierung von Plasmamembran-Mikrodomänen und insbesondere ein Protein verwendet, um Reinigungsmittelkonzentration, die uns Stress induzierte Veränderungen des sterolreichen Membrankompartiment 4,11,14 darstellen kann.
Protokoll
VERFAHREN
Gemeinsame Reagenzien und Puffer in der Extraktionsprotokoll verwendet:
- JPL Medium für Arabidopsis thaliana Suspensionskulturen
- 3 &mgr; M H 3 BO 3
- 3 uM MnSO4 x H 2 O
- 1,1 &mgr; M ZnSO 4 x 7 H 2 O
- 0,15 uM KJ
- 0,03 &mgr; M Na 2 MoO 4 x 2 H 2 O
- 3 nM CoCl 2 x 6 H 2 O
- 3 nM CuSO 4 x 5 H 2 O
- 0,9 mM CaCl 2 x 2 H 2 O
- 0,5 mM MgSO 4 x 7 H 2 O
- 0,5 &mgr; M FeSO 4 x 7 H 2 O
- 0,5 &mgr; M Na 2 EDTA · 2 H 2 O
- 0,12 uM Thiamin HCl
- 0,16 uM Nicotinsäure
- 0,097 uM Pyridoxin-HCl
- 0,107 uM NAA
- 0,375 mM KH 2 PO 4
- 0,061 mM NaH2 PO 4 x 2 H 2 O
- 0,039 mM Na 2 HPO 4
- 0,027 mM Glycin
- 0,56 mM myo-Inosit
- 1,5% Saccharose
- 10 mM K 14 NO 3 oder 10 mM K 15 NO 3
HINWEIS: pH-Wert des Mediums sollte JPL auf 5,7 mit KOH eingestellt werden. Muß das Medium durch Filtration oder Autoklavieren vor der Verwendung sterilisiert werden.
- Buffer H
- 100 mM HEPES-KOH (pH 7,5)
- 250 mM Saccharose
- 10% (w / v) Glycerin
- 5 mM EDTA
- 5 mM Ascorbinsäure
- 0,6% (w / v) PVP K-25 oder K-30
- 5 mM DTT (Add frisch)
- 1 mM PMSF (Add frisch)
- Protease-und Phosphatase-Inhibitoren (Add frisch)
- 50 mM NaF
- 1 mM Na 3 VO 4
- 1 mM Benzamidin
- 0,3 uM mikrocystin
- 4 uM Leupeptin
- Protease-Inhibitor-Cocktail
- Buffer R
- 5 mM Kaliumphosphat
- 0,33 M Saccharose
- 3 mM KCl
- 0.1 mM EDTA
- 1 mM DTT (Add frisch)
- Buffer TNE
- 25 mM Tris-HCl, pH 7,5
- 150 mM NaCl
- 5 mM EDTA
- Zwei-Phasen-System (6 g-System ist geeignet für bis zu 15 g Probe Frischgewicht) Herstellungsverfahren
In 15 ml nachstehend aufgeführten und Falcon-Röhrchen-Mix Zutaten mischen:- 20% (w / w) Dextran T500 - 2,6 g
- 40% (w / w) Poly (ethylenglycol) (PEG 3350), - 1,3 g
- Saccharose - 0,678 g
- 0,2 M Kaliumphosphat, pH 7,8 bis 0,15 ml
- 2 M KCl - 0.014 ml
- Wasser hinzufügen, bis Gesamtmasse des Zweiphasensystems ist 6 g.
- Saccharoselösungen in TNE-Puffer
- 2,4 M, 1,6 M, 1,4 M, 0,15 M
- Reagenzien für die Trypsinverdauung
- UTU: 6 M Harnstoff, 2 M Thioharnstoff (pH 8,0 mit 10 mM Tris-HCl)
- Reduktionspuffer (1 &mgr; g / &mgr; l DTT in Wasser, 6,5 mM)
- Alkylierung Puffer (5 ug / ul iodoacetamide in Wasser, 27 mM)
- LysC Endopeptidase (0,5 ug / ul)
- Trypsin, modifiziert Sequenzierung Grad (0,4 ug / ul)
- Reagenzien für die Peptid Entsalzung über C 18
- Resuspension Lösung: 2% Trifluoressigsäure (TFA), 5% Acetonitril in Wasser
- Lösung A: 0,5% Essigsäure
- Lösung B: 80% Acetonitril, 0,5% Essigsäure
- Reagenzien für Phosphopeptidanreicherung
- Lösung A: 0,1% TFA, 5% Acetonitril
- Lösung B: 0,1% TFA, 80% Acetonitril
- TiO 2 10 um
- Ammoniak (Lager 25% ige Lösung)
- Piperidin (Aktien 100%)
PROTOCOLS
1. Metabolische Markierung von Arabidopsis thaliana Zellsuspensionskulturen
- Wachsen Arabidopsis Col-0-Zelleaus Blättern 17 in voller 14 N-JPL Medium abgeleitet Suspensionskulturen, und ein Satz von Kulturen (18 "Common Reagenzien und Puffer"-Bereich) in 15 N-JPL Medium in sterile Flasche bei konstanter Lichtverhältnissen bei 80 bis 100 mmol / m 2 s, 23 ° C unter konstantem Schütteln bei 120 UpM.
- Die Zellkulturen zu erhalten, anzuimpfen 400 ml frischem Medium mit JPL 40 ml von sieben Tage alten Zellkultur in 1 l-Kolben.
- Ernten von Zellkulturen erfolgt durch Vakuumansaugung durch die große Glastrichter mit einem Edelstahlgewebe. Zellen sammeln sich auf dem Netz Platte und in den Trichter und können leicht von dort abgeholt werden. Zellen werden empfohlen, um bei -80 ° C oder in flüssigem Stickstoff eingefroren werden vor dem Mahlen.
HINWEIS: 15 N metabolisch markierten Zellkulturen verwenden K 15 NO 3 als einziger Stickstoffquelle für mindestens zwei Durchgänge über zwei Wochen 19. In Erfahrungriments für vergleichende Proteomik, verwenden Sie eine 15 N markierte Zellkultur und pflegen eine Kultur, in unmarkierten Normalmedium. Biologische Behandlung wird dann entweder markiert oder unmarkiert Kultur angewendet werden, die andere dient als Kontrolle (Fig. 1). Für Proteinzubereitung werden beide Kulturen 20 kombiniert werden. Bei der Planung der Versuchsaufbau, empfehlen wir erwägen, eine wechselseitige Etikettierung Setup 4, in der die gleiche Behandlung ist, einmal zu den 15 N-markierten Zellen angewendet und einmal auf die nicht-markierten Zellen und die jeweiligen unmarkierten oder 15 N-markierten Zellen dienen als Kontrolle. In diesem Fall werden die doppelten Mengen an Zellkulturen benötigt.
2. Plasmamembran Reinigung
Hinweis: Alle weiteren Schritte werden in den Kühlraum und / oder auf Eis durchgeführt, es sei denn es ist anders angegeben.
- Homogenisieren Zellen von etwa 1 L von 7 Tagen alten Zellsuspensionskulturen in flüssigem Stickstoff.Das Material kann bei -80 ° C bis zur weiteren Verwendung gelagert werden.
- Mischen Sie die gleichen Mengen an markierten und unmarkierten gefrorene Zellkulturen basierend auf Frischgewicht in einem Becherglas und mit 2 Volumina Puffer H sofort. Im Falle von Membranmikrodomänen Herstellung wird vorgeschlagen, mindestens 7 g Frischgewicht der beiden markierten und unmarkierten Zellen verwenden.
- Becherglas auf einem Shaker und schütteln, bis das Material geschmolzen und die Lösung ist flüssig und ohne Eiskristallen.
- Filtern des Homogenats durch eine Schicht Miracloth in 50 ml Zentrifugenröhrchen.
- Bilanz Proben mit Puffer H und Zentrifuge bei 10.000 g für 8 min.
- Laden der Überstand in vorgekühlte Ultrazentrifugenröhrchen (zB Rotor Beckman Coulter SW31Ti), mit einem Volumen von etwa 32 ml
- Balance Proben mit Puffer H und Pellet die Mikrosomen durch Zentrifugation bei 100.000 × g bei 4 ° C für 30 min unter Verwendung von Beckman SW32Ti Rotors.
- Entfernen Sie den Überstand nach der Zentrifugation.
HINWEIS: Der Überstand enthält lösliche Proteine. Eine kleine Menge dieser Fraktion kann zur weiteren Proteinfällung und anschließender Analyse auch von löslichen Proteinen gespeichert.
- Das Membranpellet zu resuspendieren jeder Probe in entsprechenden Puffermenge R und Laden auf die Oberseite U1 Zweiphasensystem (Fig. 2). Wenn die 6 g Zweiphasensystem verwendet wird, geladen werden genau 2 g des resuspendierten Membranpellet.
Hinweis: Das Endvolumen des Puffers R hängt von der Größe des Zwei-Phasen-System, das geht zu verwenden (~ 1,6 ml Puffer R wird bei der Verwendung von 6 g System erforderlich). Es wird empfohlen, weniger Puffer für die Resuspendierung zu verwenden, so können reine Puffer auf Zwei-Phasen-System, anstatt zu viel Puffer zur Solubilisierung und nicht in der Lage, die gesamte Probe auf die Zweiphasen-System zu laden zugegeben werden, um das geforderte Gewicht zu erhalten.
- Laden Sie das gleiche Volumen an reinem Puffer wie für R th verwendete Probe Resuspension auf U2.
- Langsam mischen U1 und U2 Rohre durch Umdrehen ihnen 30x (ca. 1 Inversion / sec).
HINWEIS: Sie nicht zu stark geschüttelt werden die Zwei-Phasen-System. Es kann ein Fehlen von Phasentrennung in der Ultrazentrifugation verursachen.
- Zentrifugieren der Proben bei 1500 × g bei 4 ° C für 10 min.
- Entfernen Sie den Überstand nach der Zentrifugation.
- Oberphase aus U1 und U2 auf wiederholen Zentrifugation bei 1.500 × g bei 4 ° C für 10 min.
- Verschieben Sie den oberen Phase von U2 in die SW41Ti Ultrazentrifuge Rohren und verdünnen Sie es mit fünf Volumen Puffer R und gründlich mischen.
HINWEIS: Der letzte Oberphase kann in separate Ultrazentrifugenröhrchen aufgeteilt, bevor es fünf Mal verdünnt werden kann.
- Zentrifugieren der Proben bei 200.000 × g bei 4 ° C für eine Stunde mit dem Rotor SW41Ti.
- Membranpellet resuspendieren in small Höhe von TNE-Puffer (typischerweise 200 ul Puffer verwendet wird) für die Isolierung von Reinigungsmittel beständig Membranen.
ANMERKUNG: Eine kleine Menge dieser Fraktion kann zur Analyse von fraktionierten Plasmamembran gespeichert.
HINWEIS: Die Plasmamembranfraktion kann bei 4 ° C über Nacht vor der Fraktionierung in detergensbeständige Membranen und Reinigungsmittel lösliche Fraktionen gespeichert werden.
3. Reinigungsmittel beständig Membranherstellung
- Überprüfen Sie die Konzentration der Plasmamembranfraktion durch Bradford-Assay.
- Hinzufügen Triton X-100 zu der Plasmamembranfraktion. Die endgültige Konzentration des Reinigungsmittels sollte zwischen 0,5-1% bleiben. Die Reinigungsmittel, um Protein-Gewichtsverhältnis sollte zwischen 13-15 bleiben.
- Shake Proben bei etwa 100 UpM bei 4 ° C für 30 min.
- Hinzufügen drei Volumina von 2,4 M Saccharose in einem Volumen des behandelten Plasmamembran zu 1,8 M Endkonzentration von Saccharose zu erhalten.
- Vorbereiten des Saccharosegradienten in SW41Ti Ultrazentrifugenröhrchen durch Laden der entsprechenden Saccharoselösungen auf der Oberseite der 1,8 M Fraktion, wie in Fig. 3 dargestellt.
- Zentrifugieren der Proben bei 250.000 × g bei 4 ° C für 18 Stunden unter Verwendung des Beckman SW41Ti Rotors.
- Die Ultrazentrifugenröhrchen vorsichtig aus dem Rotor zu entfernen, um Brechender der Niedrigdichteband, das als eine milchig-Ring an der Grenzfläche von 0,15 M Saccharose M/1.4 sichtbar sein können, zu vermeiden. Manchmal kann nichts sehen, aber die Fraktion, die das Reinigungsmittel beständig Proteinfraktion enthält noch.
- Bruchteil von 0,75 ml Volumen von der Spitze des Gradienten entfernen. Die Fraktionen 2 und 3, die Volumen von rund 1,5 ml oberhalb der Inter Ring und 0,5 ml unterhalb decken stellen die Reinigungsmittel beständig Membranfraktion. Sammeln Sie diese Fraktionen in 15 ml Falcon-Röhrchen. Fraktionen 9 und 10 enthalten die Reinigungsmittel löslichen Membranfraktion und kann auch zum Vergleich gesammelt werden. Zur weiteren Analyse Pool fractIonen 2 und 3 sowie den Fraktionen 9 und 10.
4. Extraktion von Proteinen aus dem Waschmittel beständig Fraktion von Methanol / Chloroform
Hinweis: Alle Schritte werden bei Raumtemperatur durchgeführt.
- In 4 Bänden von Methanol zu den gesammelten Fraktionen und gründlich vortexen.
- 1 Volumen Chloroform, Vortex gründlich.
- In drei Wassermengen, Vortex gründlich.
- Zentrifugieren der Proben bei 2000 g für 5 min unter Verwendung einer Tischzentrifuge (z. B. Eppendorf 5417R).
- Entfernen Sie die wässrige Phase über der Proteinschicht in der Interphase.
- In 3 Volumina Methanol, Vortex gründlich.
- Zentrifugieren Sie die Proben bei 4000 g für 10 min.
- Überstand entfernen und trocknen Sie die Pellets. Das getrocknete Pellet ist bereit für in-Lösung Verdauung.
5. In-Lösung Trypsin Verdauung
HINWEIS: Bei diesem Verfahren werden alle Schritte getan zuRaumtemperatur, um unerwünschte Derivatisierung von Aminosäureseitenketten durch die Denaturierungsmittel reduzieren.
- Lösen Probe in kleinen Volumen von 6 M Harnstoff, 2 M thiurea, pH 8 (UTE). Verwendung nach geringem Volumen, wie es mit der Probe (üblicherweise etwa 40 ul) kompatibel.
- Spin Proben in UTU für 10 min, jede unlösliche Material zu pelletieren gelöst bei 5.000 xg in einer Tischzentrifuge (zB Eppendorf 5417R).
- Der pH-Wert der endgültigen Lösung sollte in der Nähe von 8,0 für optimale Trypsin Verdauung. Überprüfen Sie mit pH-Streifen.
- 1 ul Reduktionspuffer für jede 50 ug Probenprotein und inkubiere 30 Minuten bei Raumtemperatur.
HINWEIS: Nur erforderlich, eine grobe Schätzung der Proteingehalt ist. Wenn die Probenmenge begrenzt ist, ist es besser, um die Genauigkeit zu opfern Anstatt Probe auf einem Protein-Assay.
- 1 ul Alkylierung Puffer pro 50 ug Probenprotein und inkubiere 20 Minuten bei Raumtemperatur im Dunkeln.
- Mit 0,5 &mgr; l von lysC pro 50 ug Probenprotein und Inkubieren während drei Stunden bei Raumtemperatur unter konstantem Schütteln bei 700 Upm. Falls erforderlich, kann auch der Verdau über Nacht durchgeführt werden. Allerdings verlängert Zeit bei warmen Temperaturen nicht empfohlen, da es Peptidverlust durch Adsorption an Kunststoff-Rohrmaterial unter wässrigen pH 8 Bedingungen zu erhöhen.
- Verdünnte Probe mit vier Volumina 10 mM Tris-HCl, pH 8.
HINWEIS: Dieser Schritt ist absolut notwendig, um die Harnstoffkonzentration zu verdünnen, wie Trypsin ist sehr empfindlich auf hohe Salz.
- 1 &mgr; l 50 &mgr; g Trypsin pro Probenprotein und Inkubieren über Nacht bei Raumtemperatur unter konstantem Schütteln bei 700 Upm.
- Säure die Proben auf 0,2% TFA Endkonzentration auf pH 2 (sowie ungefähr 1/10 Volumen 2% Trifluoressigsäure) zu erreichen.
HINWEIS: Die Proben können bei - 20 ° C bis zur weiteren Verwendung, aber es ist besser, zu speichernsie auf StageTips bei 4 ° C, wenn es für eine kurze Zeit (eine Woche) oder speichern sie nach StageTips Entsalzung.
6. Herstellung von C 18-StageTips
- Legen Sie eine Empore Disk-C18 auf eine flache, saubere Oberfläche, wie ein Einweg-Kunststoff-Petrischale.
- Punch-Out eine kleine Festplatte mit einer stumpfen Injektionsnadel mit einem Durchmesser von 1,5 mm. Die Platte klebt in der Nadel und in eine Pipettenspitze übertragen werden.
- Schieben die Scheibe aus der Nadel befestigen und in die Verjüngung eine Pipettenspitze von einem Stück Quarzglas oder Schlaucharmatur in das Innere der Nadel.
HINWEIS: StageTips kann bei Raumtemperatur 21 trocken gelagert werden.
7. Die Verwendung von C 18-StageTips für Peptide Entsalzung und Konzentration
- Bedingen eine C 18-StageTip indem 50 ul Lösung B auf den vorbereiteten StageTip. Drehen Sie die Spitze in der Zentrifuge bei 2.000 rpm für 2 min in einer Tisch centrifuge (zB Eppendorf 5417R).
HINWEIS: Verwenden Sie Adapter StageTips in einer Eppendorf-Zentrifuge drehen, wird Flüssigkeit in einem 2 ml Reaktionsgefäß gesammelt werden. Für größere Vorbereitungen, Bühnen Tipps können auch in eine Spitze Rack mit 96 der 200 ul-Tipps platziert werden und Flüssigkeit kann in einer Mikrotiterplatte gesammelt werden.
HINWEIS: Verwenden Sie niemals höhere Geschwindigkeiten als 3.000 × g in einer Tischzentrifuge (zB Eppendorf 5417R) wegen der Gefahr von Spinn die Festplatte C 18 von der Spitze.
- Äquilibrieren Sie die StageTip mit 100 ul Lösung A Spin die Spitze in der Zentrifuge bei 5.000 xg in einer Tischzentrifuge (zB Eppendorf 5417R).
- Wiederholen Sie Schritt 2.
- Legen Sie die Probe auf die Platte durch vorsichtiges Pipettieren der Probe in die Pipettenspitze mit der Scheibe im Inneren.
HINWEIS: Eine Disc kann ca. binden. 100 &mgr; g Protein.
- Drehen Sie die Tipps in der centrifuge, bis das gesamte Volumen der Probe durch die C-18 Festplatte übergeben.
- Mit 100 ul Lösung A. Drehen Sie die Tipps in der Zentrifuge Waschen der StageTips zweimal.
HINWEIS: Die gewaschenen und geladen StageTips können bei 4 ° C für bis zu einer Woche gelagert werden.
- Eluiere Probe mit 40 ul der Lösung B. Sammeln des Eluats in ein frisches 1,5 ml Reaktionsgefäß.
- Konzentrieren sich die Probe in der Speed Vac. Unter idealen Bedingungen, stoppen Sie den Entwässerungsprozess, da es nur um 1 oder 2 ul Flüssigkeit verlassen.
HINWEIS: Getrocknete Proben in der -80 ° C über Jahre gelagert werden.
- Fügen Sie ein Gesamtvolumen von 9 ul Resuspensionslösung zur Probe und überträgt es auf der Mikrotiterplatte für die Massenanalyse spec.
Hinweis: Das Endvolumen des verwendeten Resuspensionspuffers ist abhängig von den Bedürfnissen der Experimentator und der Empfindlichkeit des Massenspektrometersverwendet.
8. Alternative Protokoll für Phosphopeptidanreicherung
HINWEIS: Phosphopeptidanreicherung, Proteinextraktion in Schritt 2 muss in Gegenwart von Phosphatase-Inhibitoren durchgeführt werden.
HINWEIS: Genaue Proteinkonzentration muss nicht für die folgenden Schritte festgelegt werden. Es ist genug, um eine grobe Schätzung der Proteingehalt haben, um unnötige Verluste zu vermeiden Probe
- Bereiten Sie die C 8-StageTips (nach dem gleichen Protokoll für die Herstellung von C 18-StageTips in Schritt 6).
- Übertragung 1 mg TiO 2-Perlen an die Spitze der vorbereiteten C 8-StageTips (Einsatz 1 mg TiO 2 pro 100 ug Protein).
- Äquilibrieren die TiO 2-Tipps mit 100 ul der Lösung C, Schleudern bei 2000 g für 5 min in einer Tischzentrifuge (zB Eppendorf 5417R).
- Mischungs 100 ug verdaut und entsalzt Peptide aus Schritt 7.7 (es sollte in 100 ul LösungB) mit 100 ul der Lösung A
- Legen Mischprobe auf die äquilibrierte TiO 2 Spalten; Schleudern bei 1000 × g, 5 min.
- Sammeln Sie die Durchfluss und laden Sie es auf der gleichen Spitze wieder (flow halten durch, nachdem zweiten Durchgang).
- Waschen Spitze mit 100 ul Lösung A; Spin bei 2.000 × g, 5 min in einer Tischzentrifuge (zB Eppendorf 5417R).
- Eluieren Probe mit 50 &mgr; l 5% igem Ammoniak.
- Eluieren Probe mit 50 &mgr; l 5% Piperidin (beide Eluaten kombiniert werden können).
- Unmittelbar säuern die Probe mit 50 ul 20% Phosphorsäure. pH-Wert sollte bei etwa 2 sein.
HINWEIS: Speichern Sie die TiO 2-Tipps und Abrufen der Pulver. Es kann mit Lösung B gewaschen und wieder für ein oder zwei weitere Runden verwendet werden.
- Wieder zu entsalzen angesäuerten Proben über 18 C-Tipps (siehe Schritt 8).
9. LC-MS/MS Analyse von Peptid-Mischungen
- Resuspendieren entsalzt phosphopeptides in Resuspensionspuffer.
- Last resuspendiert Probe auf LC-MS/MS System der Wahl erwerben und Spektren in Daten abhängigen Modus zu einem empfohlenen Auflösung von 60.000 vollen Halbwertsbreiten.
HINWEIS: Für eine optimale Fragmentierung, sollte entweder neutral-loss-Scan angewendet werden 22, oder falls vorhanden den mehrstufigen Aktivierungs 23. Wenn ETD verfügbar ist, wird Peptid-Rückgrat Fragmentierung ohne Verlust von Phosphorsäure 24 zu ermöglichen.
HINWEIS: Peaklisten von Rohdaten müssen extrahiert und zur Datenbank Identifikation sowie zur Quantifizierung vorgelegt. Hier beschreiben wir die Einstellungen für die Verwendung von Maskottchen und MSQuant, die Rohdaten aus LTQ, LTQ-Orbitrap und LTQ-FT Instrumente (Thermo Scientific) funktioniert.
- Auszug Peak-Liste mit DTAsupercharge innerhalb der "Helper" Menü in MSQuant. Verwenden Sie die Standardeinstellungen.
- Senden resultierende Peak-Liste-Datei in Mascot-Suchmaschine. Critical Einstellungen: Vorläuferionenmassentoleranz 10 ppm, MS / MS-Massentoleranz von 0,5 Da. Fest Änderungen: Carbamidomethylierung von Cysteinen, variable Modifikationen: Oxidation von Methionin, die Phosphorylierung von Serin, Threonin, Tyrosin. Die quantitative Methode: 15 N metabolische Markierung.
- Sparen Maskottchen Ergebnis als Web-Seite Datei.
- Legen Maskottchen Ergebnisdatei und RAW-Datei in MSQuant. Wählen Sie alle Proteine von Interesse und führen Sie "Automatische Quantifizierung". Das Programm wird Scan-Spektren aus der RAW-Datei lesen und Peak-Integration für die markierten und unmarkierten Partner jedes Peptids.
- Export quantitative Ergebnisse in ein Tabellenkalkulationsprogramm oder in eine Tab-getrennte Textdatei. Die Daten können dann zur weiteren statistischen Auswertung in Excel oder mit weiteren Softwarepaketen wie Statquant 25 oder 26 Cracker eingereicht werden.
Ergebnisse
Mit der vorgestellten Protokoll mit metabolisch markierten Arabidopsis-Zellkulturen ist es möglich, die Plasmamembranen aus Pflanzengewebe (Schritt 2, Fig. 2 und 4) zu isolieren und anzureichern Waschmittel beständig Membranfraktionen innerhalb der Plasmamembran (Schritt 3, Fig. 3 und 5). Anschließend kann das Protokoll Extraktion von Proteinen aus diesen Waschmittel beständig Membranfraktionen (Schritt 4) und die Verdauung des Proteins für die vergleichende Proteomik-Analyse (Schritt 5). Schließlich ist die optionale Anreicherung von Phosphopeptiden (Schritt 8) besonders interessant bei der Untersuchung zellulärer Signalprozesse. Der letzte Schritt ist dann die quantitative Datenanalyse (Schritt 9, Abbildung 6) von Protein Häufigkeitsverhältnisse in Membranfraktionen von markierten und unmarkierten Zellkultur.
Typische Ergebnisse nach dem Mischen der 2-Phasen-System (Schritt 2) und anschließender Zentrifugation sind eine klare gefärbte obere Phase und einegrün gefärbt untere Phase (Abbildung 2). Plasmamembranvesikel bevorzugt assoziieren mit der oberen PEG-Phase, während Vesikeln aus grünen Chloroplasten und andere Innen Membranen werden in den unteren Dextran-Phase eingearbeitet. Wenn die Farbtrennung nicht beobachtet werden kann, Schritt 2, wurde nicht korrekt durchgeführt, und die Plasmamembranen werden mit erheblichen Mengen von inneren Membranen kontaminiert werden. Anreicherung von Plasmamembran-Proteine durch proteomische Analyse von kleinen Aliquots der Plasmamembranfraktionen und Proteine in der unteren Phase des Zweiphasensystems angezeigt. Die untere Phase enthält die innere Membran der Zelle. Beispielsweise besonders typischen bekannten Plasmamembranproteinen, wie Rezeptor-Kinase-Proteine zeigten eine hohe Häufigkeit in der Plasmamembranfraktion (PM) und geringer Häufigkeit in der unteren Phase Fraktion (4A), die die innere Membran (IM). Im Gegenzug bekannten Proteinen des endoplasmatischen Retikulum zeigen hoheFülle in der inneren Membran-Fraktion und wurden nicht in die Plasmamembranfraktion (4B) beobachtet.
Die anschließende Anreicherung von detergensbeständige Membranfraktionen (Schritt 3) wird im Idealfall führen zur Bildung einer milchigen Ring von niedriger Dichte Membranvesikel bei etwa 1/5 der Rohrhöhe (Fig. 3). Wenn die Verstellung des Triton X-100-Konzentrationen oder die Waschmittel Protein-Verhältnis (Schritt 3.2) optimal für die Trennung in Waschmittel-und Reinigungsmittel beständig löslichen Fraktionen können zusätzliche Ringe in unteren Teilen des Gradienten beobachtet, und es wird auch Membran Klumpen sein in der geringen Dichte Band. In diesen Fällen ist die Reinigungsmittel auf Protein-Verhältnis oder gesamte Waschmittelmenge verschieden von der kritischen Mizellenkonzentration, die für reproduzierbare Trennung der beiden Membrandomäne Fraktionen. Mit der abschließenden Reinigungsmittel-Konzentration und Reinigungsmittel, um Protein-Verhältnis beschrieben hier, Enrichment von typischen Mikrodomänenproteine wie Remorin (AT3G61260) 27 könnte durch eine vergleichende massenspektrometrische Analyse von DRM-Fraktion, Reinigungsmittel lösliche Fraktion (Schritt 3.8) sowie fraktionierten Plasmamembran und der internen Membranen bestätigt werden. Höchste Fülle von Remorin-Protein wurde in Fraktionen gefunden DRM wie erwartet (Fig. 6A). Proteine, die nicht in den Membranmikrodomänen, wie zB die ABC-Transporter AT1G59870 (6B) vorhanden, nicht eine erhöhte Häufigkeit in der DRM-Fraktion zu zeigen. Auch typische Verunreinigungen, wie eine mitochondriale Protein (AT2G07707) von der ATPase-F0-Komplex (6C) und ribosomalen Protein (At1G001100) von der 60S Ribosomen (6D) nicht eine angereicherte Hülle und Fülle in der DRM-Fraktion zeigen, wenn auch minimale Mengen können noch identifiziert werden.
Nach der massenspektrometrischen Analyse-, Protein-Häufigkeiten Verhältnisse von Protein-Phosphorylierung im PlasmaMembranfraktionen von markierten und nicht markierten Zellkulturen quantitativ unterscheiden (Abbildung 6). Typische Ergebnisse aus der MSQuant ( msquant.alwaysdata.net ) Quantifizierung Fenster sollte eine Ionenintensitätsverhältnis von eins zu eins für die meisten der identifizierten Peptide (Fig. 6A) angezeigt. Diese Peptide mit Ionenintensitätsverhältnis erheblich von 1:1 abweichende gelten als Differenz zwischen dem markierten und unmarkierten Zellkultur geregelt und sind Kandidaten für die Anpassung an Proteinen, die durch die angelegte (6C)-Behandlung beeinflusst. Ein guter Qualitätskontrolle erfolgreich metabolische Markierung ist die Co-Elution von sowohl markiertem und unmarkiertem Peptid Versionen als einzelner Peak in der Nano-HPLC-Trennung (Schritt 9), wie in den 6B und D gezeigt.

Fig. 1 ist. Metabolische Markierung experimentelle Strategie. Schematische Darstellung von zwei Varianten der Versuchsplanung, wenn Sie voll Stoffwechsel markierten und unmarkierten A. thaliana Zellkulturen. Der Experimentator kann entweder entscheiden, 14 N-Zellen (unbenannt) als Steuerungs-und 15 N-Zellen (metabolisch markierten), die biologische Behandlung unterziehen oder umgekehrt zu verwenden. In einem experimentellen Design gegenseitige beide Varianten werden parallel durchgeführt.

2. Workflow zur Trennung von Pflanzen Membranvesikel einer wäßrigen Zweiphasensystem auf Basis von Polyethylenglykol (PEG) und Dextran. Plasmamembran-Vesikel in die obere Phase abgetrennt, während PEG Chloroplast und andere Innenmembranen mit dem zugehörigen Bottom Dextran-Phase nach dem Zentrifugieren. Mit zusätzlichen Waschen der oberen Phase besser Reinigung erreicht werden, aber auch Materialverlust erhöht.
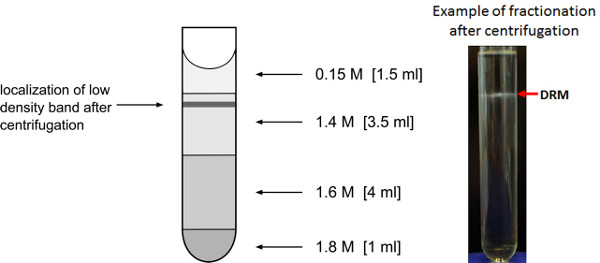
3. Anreicherung von Low-Density-Membranfraktionen. Darstellung der Saccharose-Gradienten für die Anreicherung von Low-Density-Reinigungsmittel beständig Membranfraktionen. Die zu erwartende Lage nach der Zentrifugation über Nacht der geringen Dichte Membranvesikels Band angezeigt.

4. Anreicherung von Plasmamembranproteinen nach der Reinigung in Zweiphasensystem. NormalizedProteinionenintensitäten für Proteine in der Plasmamembran (PM) und innere Membran (IM)-Fraktion gemessen. (A) Typische bekannte Bestandteile der Plasmamembran eine deutlich höhere Menge in PM Fraktion als IM-Fraktion. (B) Natur bekannten Komponenten der internen Membranen zeigt das entgegengesetzte Verhalten. Zur Normalisierung wurden Ionenintensität Summen für jedes Protein als ein Teil der Gesamtionenstärke pro Probe ausgedrückt und dann gemittelt zwischen Wiederholungen. Bruchteil der gesamten Ionenintensitäten wurden dann als relativ zum Mittelwert zwischen den zwei Behandlungen ausgedrückt. Fehlerbalken stellen die Standardabweichung der drei Präparate aus unabhängig voneinander gewachsen Zellkulturen. Klicken Sie hier, um eine größere Abbildung anzuzeigen .

5. . Fülle von Standard-Proteinmarker gemessen Fraktionen innerhalb Stücke stellen die Verteilung der normalisierten Proteinionenintensitäten ausgewählter Proteinmarker auf bestimmte subzellulärer Kompartimente (DRM - sterolreichen Mikrodomänen, PM - Plasmamembran, IM - interne Membranen, SP - lösliche Proteine). Fülle-Muster von (A) Remorin als Marker für sterolreichen Membranmikrodomänen, (B) ABC-Transporter-Familie Typ-Protein als Vertreter einer nicht Sterin-abhängige Protein, (C)-Untereinheit der mitochondrialen F0-Komplex, (D) 60S -Untereinheit der Ribosomen als typische Co-Reinigung von Verunreinigungen. Zur Normalisierung wurden Ionenintensität Summen für jedes Protein als ein Teil der Gesamtionenstärke pro Probe ausgedrückt und dann gemittelt zwischen Wiederholungen. Bruchteil der gesamten Ionenintensitäten wurden dann wie in bezug auf die mittlere zwischen den beiden trea ausgedrückttments. Fehlerbalken stellen die Standardabweichung der drei Präparate aus unabhängig voneinander gewachsen Zellkulturen. Klicken Sie hier, um eine größere Abbildung anzuzeigen .
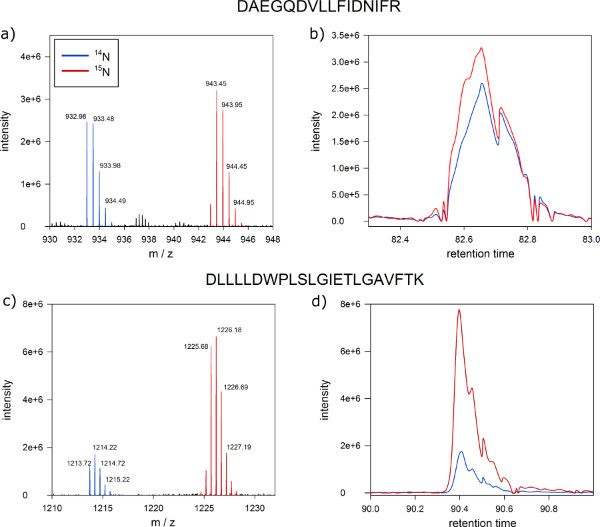
6. Erwartete Ergebnisse der quantitativen Protein-Massenspektrometrie. Screenshot von MSQuant der die erwartete 1.01 Ionenintensitätsverhältnis eines Peptids aus einer 1:1-Mischung von Proteinextrakten aus markierten und unmarkierten Arabidopsis-Zellen. (A) Full-Scan-Spektrum von Peptid DNNLLGK deutlich Abtrennen markierter (rechte Spitze) und unmarkierten (linker Peak) Version der Peptide auf der m / z-Achse. Die monoisotopischen Masse und das erste Isotop werden durch blaue Markierungen gekennzeichnet. (B) markierte (blau) eind unmarkierten (rot) Versionen des Peptids koeluieren als einzelner Peak bei Nano-HPLC-Chromatographie. (C) Full-Scan-Spektrum eines Peptids mit Ionenintensitätsverhältnis von 1:5 als ein Kandidat für ein differentiell regulierten Proteins. (D ) Auch Proteine mit unterschiedlichen Verhältnissen von 1:1 koeluieren in Reversed-Phase Chromatographie. Klicken Sie hier, um eine größere Abbildung anzuzeigen .
Diskussion
Die in diesem Papier Protokoll enthält viele Schritte und alle von ihnen entscheidend für reine und Vertreter Fraktionierung der Plasmamembran Anlage in Waschmittel beständig Membranen und Reinigungsmittel lösliche Fraktionen zu erhalten sind. Daher ist es wichtig, jeden Schritt zu folgen, wie angewiesen.
Behandlung der Plasmamembranfraktion mit nicht-ionischen Detergens (Schritt 3.2) den stärksten Einfluss auf die Qualität der Membran Mikrodomänen Fraktionierung. Um reproduzierbare Ergebnisse zwischen verschiedenen Präparationen zu erhalten, und um verschiedene Proben aus dem gleichen Experiment vergleichen zu können, ist es sehr wichtig, genau zu ermitteln, die Konzentration der Proteine in der Plasmamembran und das Reinigungsmittel immer in der gleichen Verhältnis in Bezug auf Proteingehalts und immer auf die gleiche Endkonzentration. Praktisch bedeutet dies, daß es manchmal notwendig ist, um Proben mit einem hohen Proteinkonzentration zu verdünnen oder unterschiedlicher Konzentration verwendet BeständeTriton X-100.
In biochemischen Untersuchungen, vor kurzem gab es eine Tendenz unter den Forschern, ein Reinigungsmittel-freie Methode zur Isolierung von Membranmikrodomänen 28-30 zu verwenden. Diese Verfahren beruhen auf einer mechanischen Störung der Plasmamembran durch Scherung durch einen sehr kleinen Nadel in Analogie zu der Druckzelle Französisch Maschine zu schaffen. Diese Verfahren wurden erfolgreich in Säugerzellkulturen, die reich an Mikrodomänen angewendet, aber Anwendung auf Zellwand enthaltenden Organismen (Hefe, Pflanzenzellen) wurde nicht berichtet. Verwendung der quantitativen Proteomik mit Sterol-störenden Mittel, wie Methyl-beta-cyclodextrin wurde gezeigt, dass die DRM-Fraktionen von Pflanzenplasmamembran hergestellt enthalten Proteine, die Marker für die Membran-Mikrodomänen 11.
Die endgültigen Ergebnisse der massenspektrometrischen Quantifizierung, die in Fig. 6 gezeigte typische Ergebnisse stellen die ideale Situation, indie die Ionenintensität für die Mehrzahl der markierten und unmarkierten Peptiden nahe identisch. Jedoch kann in einigen Fällen ein gewisses Maß an biologischer Unterschiede zwischen den Sets von markierten und unmarkierten Zellkulturen beobachtet werden. Somit werden vor der biologischen Behandlung von entweder markiert oder unmarkiert Zellkultur durchgeführt wird, die Analyse eines Gemisches von markierten und unmarkierten Kontrollzellen, sowohl ohne jegliche Behandlung, ermöglicht die Identifizierung von Peptiden mit einem divergierenden Verhältnissen von der idealen 0.59 Situationen. Diese Proteine mit divergierenden Verhältnisse gibt Hinweise für die biologische Variante. Um die Herausforderung der Scheidungsbehandlungseffekte aus biologischen Variation zu überwinden, deshalb schlagen wir vor, um die wechselseitige Etikettierung in gepaarten Experimenten 20 zu verwenden. Im reziproken Versuchsaufbau sind beide Varianten des Experiments durchgeführt, wie in Fig. 1 vorgeschlagen, und die Schritte des Protokolls sind, wie beschrieben, gefolgt. Dann Vergleichen der Ionenintensitätsverhältnis von proteins von beiden reziproken Experimenten kann man unterscheiden, ob die Unterschiede zwischen den Ionenintensitätsverhältnisse aufgrund biologischer Variation oder als eine Wirkung der Behandlung angewendet werden.
Ein weiteres Verfahren zur Lösung des Problems Differenzierung Behandlungswirkungen und biologischen Variation ist die Verwendung eines internen markierten Standards als Referenz für alle Behandlungen 31. In diesem Ansatz werden unmarkierte Kontrolle und unmarkierten Zellen, die biologische Behandlung zu unterziehen mit der gleichen Menge von Schwer markierten Kontroll-Zellen vermischt, die aus der gleichen Charge. Es ermöglicht, den Einfluß von biologischen Variation Peptidverhältnis, denn Proteine werden als signifikant angesehen, wenn Ionenintensitätsverhältnis von 14 N-Kontrolle / 15 N-Kontroll-und 14 N-Behandlung / 15 N-Kontrolle signifikant unterschiedlich sind. Dies ist möglich, da die markierten Standards ist genau das gleiche in allen Fällen.
Einer der drawbacks der hier beschriebenen DRM Anreicherungsverfahren sind die Co-Reinigung von Proteinen, die in der Waschmittelbeständigen Membranfraktion identifiziert werden können. In der Liste der Proteine aus dem Low-Density-Detergenz-resistenten Membranfraktion identifiziert, können wir beobachten, regelmäßig eine Vielzahl von ribosomalen Proteinen. Aufgrund der relativ geringen Dichte der ribosomalen Proteine wandern sie in der gleichen Fraktion als Sterol abhängige Membranproteine im Saccharosegradienten. Es war ohne Zweifel, dass die ribosomale Proteine sind eindeutig nicht Bestandteile der Membranmikrodomänen 11 gezeigt. Um diese und andere Co-Reinigung von Proteinen und nicht wirklich mit der detergensbeständige niedriger Dichte Membranfraktionen ausgeschlossen wird, schlagen wir vor, die Proteom-Analyse auch die Zusammensetzung der intrazellulären Membranen (IM), die von der unteren Phase aus dem Dextran-Zwei extrahiert werden kann, Phasensystem und die Häufigkeitsverhältnisse von mutmaßlichen Verunreinigungen zwischen der inneren Membran und der DRM vergleichenFraktion (siehe Beispiele in Abb. 5). DRM-co-Reinigung von Proteinen sollten ähnliche Häufigkeiten haben in beiden Fraktionen (IM-und DRM).
Fraktionierung von Zellextrakten und Membranmikrodomänen ist eine gemeinsame Strategie, um die Komplexität in einer Probe für Proteomanalyse 32 zu reduzieren. Daher ist die hier beschriebene Protokoll nützlich für alle Studien von Signalprozessen in der Plasmamembran von Pflanzenzellen. Biologische Anwendungen sind bei der Untersuchung Stressreaktionen sowie Pathogen-Infektionen, die alle zu einem großen Teil Veränderungen in der Plasmamembranzusammensetzung und Modifikation von Membranproteinen 14,15 induzieren. Neben dem Studium Stressreaktionen kann die Quantifizierung von Proteinhäufigkeiten in verschiedenen Membranfraktionen große Hilfe Annotation von noch unbekannten Proteinen 33 34,35.
Offenlegungen
Die Autoren erklären, dass sie keine finanziellen Interessen konkurrieren.
Der Autor, Witold Szymanski ist ein Mitarbeiter des Max-Planck-Institut für Molekulare Pflanzenphysiologie. Der Autor, ist Waltraud Schulze Mitarbeiter an der Universität Hohenheim, Deutschland.
Materialien
| Name | Company | Catalog Number | Comments |
| REAGENTS | |||
| Chemicals were ordered from Sigma-Aldrich unless noted otherwise | |||
| Ammonia (stock 25 % solution) | WAKO | 010-03166 | |
| TiO2 10 μm | GL-Science | 5020-75010 | |
| Empore Disk C18 | Varian | 12145004 | |
| Polyethylene glycol(PEG 3350) | Sigma | 88276 | |
| Dextran T500 | Roth | 9219.2 | |
| Trypsin | Promega | V5113 | |
| Protease inhibitor cocktail (PIC) | Sigma | P9599 | |
| K15NO3 | Cambridge Isotope Laboratories | NLM-765-PK | |
| EQUIPMENT | |||
| Optima L-80 XP Ultracentrifuge | Beckman | ||
| Plate reader | BioTek | ||
| EASY-nLC II nano-Liquid Chromatograph | Thermo Scientific | ||
| LTQ-Orbitrap mass spectrometer | Thermo Scientific | ||
| Centrifuge 5810R | Eppendorf | ||
| Centrifuge 5417R | Eppendorf | ||
| Thermomixer | Eppendorf | ||
| Speed Vac RVC 2-25 | Christ | ||
| Shaker Unimax 2010 | Heidolph | ||
Referenzen
- Alexandersson, E., Saalbach, G., Larsson, C., Kjellbom, P. Arabidopsis plasma membrane proteomics identifies components of transport, signal transduction and membrane trafficking. Plant Cell Physiol. 45, 1543-1556 (2004).
- Brown, D. A., Rose, J. K. Sorting of GPI-anchored proteins to glycolipid-enriched membrane subdomains during transport to the apical cell surface. Cell. 68, 533-544 (1992).
- Lanquar, V., et al. 15N-metabolic labeling for comparative plasma membrane proteomics in Arabidopsis cells. Proteomics. 7, 750-754 (2007).
- Kierszniowska, S., Walther, D., Schulze, W. X. Ratio-dependent significance thresholds in reciprocal 15N-labeling experiments as a robust tool in detection candidate proteins responding to biological treatment. Proteomics. 9, 1916-1924 (2009).
- Singer, S. J., Nicolson, G. L. The fluid mosaic model of the structure of cell membranes. Science. 175, 720-731 (1972).
- Simons, K., Ikonen, E. Functional rafts in cell membranes. Nature. 387, 569-5672 (1997).
- Baumgart, T., et al. Large-scale fluid/fluid phase separation of proteins and lipids in giant plasma membrane vesicles. Proc. Natl. Acad. Sci. USA. 104, 3165-3170 (2007).
- Silvius, J. R. Partitioning of membrane molecules between raft-and non raft-domains: insights from model membrane studies. Biochim. Biophys. Acta. 1746, 193-202 (2005).
- Tanner, W., Malinsky, J., Opekarova, M. In plant and animal cells, detergent-resistant membranes do not define functional membrane rafts. Plant Cell. , (2011).
- Lauwers, E., Andre, B. Association of yeast transporters with detergent-resistant membranes correlates with their cell-surface location. Traffic. 7, 1-15 (2006).
- Kierszniowska, S., Seiwert, B., Schulze, W. X. Definition of Arabidopsis sterol-rich membrane microdomains by differential treatment with methyl-ß-cyclodextrin and quantitative proteomics. Mol. Cell. Proteomics. 8, 612-623 (2009).
- Simons, K., Toomre, D. Lipid rafts and signal transduction. Nat. Rev. Mol. Cell Biol. 1, 31-39 (2000).
- Maeda, Y., Kinoshita, T. Structural remodeling, trafficking and functions of glycosylphosphatidylinositol-anchored proteins. Prog. Lipid Res. 50, (2011).
- Keinath, N. F., et al. PAMP-induced changes in plasma membrane compartmentalization reveal novel components of plant immunity. J. Biol. Chem. 285, 39140-39149 (2010).
- Stanislas, T., et al. Quantitative proteomics reveals a dynamic association of proteins to detergent resistant membranes upon elicitor signaling in tobacco. Mol. Cell. Proteomics. 8, 2186-2198 (2009).
- Minami, A., et al. Alterations in detergent-resistant plasma membrane microdomains in Arabidopsis thaliana during cold acclimation. Plant Cell Physiol. 50, 341-359 (2008).
- Pauly, M., Eberhard, S., Albersheim, P., Darvill, A., York, W. S. Effects of the mur1 mutation on xyloglucans produced by suspension-cultured Arabidopsis thaliana cells. Planta. 214, 67-74 (2001).
- Jouanneau, J. P., Peaud-Lenoel, C. Growth and synthesis of proteins in cell suspensions of a kinetin dependent tobacco. Physiol. Plant. 20, 834-850 (1967).
- Engelsberger, W. R., Erban, A., Kopka, J., Schulze, W. X. Metabolic labeling of plant cell cultures with K15NO3 as a tool for quantitative analysis of proteins and metabolites. Plant Methods. 2, 1-11 (2006).
- Arsova, B., Kierszniowska, S., Schulze, W. X. The use of heavy nitrogen in quantitative proteomics experiments in plants. Trends Plant Sci. 17, 102-112 (2012).
- Rappsilber, J., Ishihama, Y., Mann, M. Stop And Go Extraction tips for matrix-assisted laser desorption/ionization, nanoelectrospray, and LC/MS sample pretreatment in proteomics. Anal. Chem. 75, 663-670 (2003).
- Olsen, J. V., Macek, B. High accuracy mass spectrometry in large-scale analysis of protein phosphorylation. Methods Mol. Biol. 492, 131-142 (2009).
- Schroeder, M. J., Shabanowitz, J., Schwartz, J. C., Hunt, D. F., Coon, J. J. A neutral loss activation method for improved phosphopeptide sequence analysis by quadrupole ion trap mass spectrometry. Anal. Chem. 76, 3590-3598 (2004).
- Frese, C. K., et al. Improved peptide identification by targeted fragmentation using CID, HCD and ETD on an LTQ-Orbitrap Velos. Journal of Protrome Research. 10, 2377-2388 (2011).
- van Breukelen, B., vanden Toorn, H. W., Drugan, M. M., Heck, A. J. StatQuant: a post-quantification analysis toolbox for improving quantitative mass spectrometry. Bioinformatics. 25, 1472-1473 (2009).
- Zauber, H., Schulze, W. X. Proteomics wants cRacker: Automated standardized data analysis of LC/MS derived proteomic data. J. Proteome Res. 11, 5548-5555 (2012).
- Raffaele, S., Mongrand, S., Gamas, P., Niebel, A., Ott, T. Genome-wide annotation of remorins, a plant-specific protein family: Evolutionary and functional perspectives. Plant Physiol. 145, 593-600 (2007).
- Shah, M. B., Sehgal, P. B. Nondetergent isolation of rafts. Methods Mol. Biol. 398, 21-28 (2007).
- Persaud-Sawin, D. -. A., Lightcap, S., Harry, G. J. Isolation of rafts from mouse brain tissue by a detergent-free method. Journal of Lipid Research. 50, 759-767 (2009).
- Macdonald, L. J., Pike, L. J. A simplified method for the preparation of detergent-free lipid rafts. Journal of Lipid Research. 46, 1061-1067 (2005).
- Mühlhaus, T., Weiss, J., Hemme, D., Sommer, F., Schroda, M. Quantitative shotgun proteomics using a uniform 15N-labeled standard to monitor proteome dynamics in time course experiments reveals new insights into the heat stress response of Chlamydomonas reinhardtii. Mol. Cell. Proteomics. 10, M110.004739 (2011).
- Schulze, W., Usadel, B. Quantitation in mass-spectrometry-based proteomics. Annu. Rev. Plant Biol. 61, 491-516 (2010).
- Foster, L. J., de Hoog, C., Mann, M. Unbiased quantiative proteomics of lipid rafts reveales high specificity for signaling factors. Proc. Natl. Acad. Sci. USA. 100, 5813-5818 (2003).
- Lilley, K. S., Dupree, P. Plant organelle proteomics. Curr. Opin. Plant Biol. 10, 594-599 (2007).
- Sadowski, P. G., Groen, A. J., Dupree, P., Lilley, K. S. Sub-cellular localization of membrane proteins. Proteomics. 8, 3991-4011 (2008).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten