Method Article
Bitki Hücre Tiplerinin İzolasyonu ve Transkriptom Analizi
Bu Makalede
Özet
Yüksek verimli scRNA-seq yöntemlerinin fizibilitesi ve etkinliği, bitki araştırmalarında tek hücreli bir çağın habercisidir. Burada, spesifik Arabidopsis thaliana kök hücre tiplerinin izole edilmesi ve ardından transkriptom kütüphanesi yapımı ve analizi için sağlam ve eksiksiz bir prosedür sunulmaktadır.
Özet
Çok hücreli organizmalarda, gelişimsel programlama ve çevresel tepkiler, farklı hücre tiplerinde ve hatta hücresel heterojenite olarak bilinen hücreler içinde oldukça farklı olabilir. Son yıllarda, yeni nesil dizileme (NGS) teknikleriyle birleştirilen tek hücreli ve hücre tipi izolasyon, biyolojik süreçleri tek hücreli çözünürlükte incelemek için önemli araçlar haline gelmiştir. Bununla birlikte, bitki hücrelerinin izole edilmesi, bitkilerde tek hücreli yaklaşımların uygulanmasını sınırlayan bitki hücre duvarlarının varlığı nedeniyle nispeten daha zordur. Bu protokol, aşağı akış multi-omik analizi ve diğer çalışmalar için uygun olan, bitki hücreleri ile floresan ile aktive edilmiş hücre sıralama (FACS) tabanlı tek hücreli ve hücre tipi izolasyon için sağlam bir prosedürü açıklamaktadır. Arabidopsis kök floresan işaretleyici çizgilerini kullanarak, ksilem-kutup perisiklus hücreleri, lateral kök başlangıç hücreleri, lateral kök kapak hücreleri, korteks hücreleri ve endodermal hücreler gibi belirli hücre tiplerinin nasıl izole edildiğini gösteriyoruz. Ayrıca, Smart-seq2 kullanan etkili bir aşağı akış transkriptom analiz yöntemi de sağlanmaktadır. Hücre izolasyon yöntemi ve transkriptom analiz teknikleri, diğer hücre tiplerine ve bitki türlerine uyarlanabilir ve bitki biliminde geniş uygulama potansiyeline sahiptir.
Giriş
Hücreler tüm canlı organizmaların temel birimidir ve yapısal ve fizyolojik işlevleri yerine getirir. Çok hücreli organizmalardaki hücreler belirgin bir eşzamanlılık göstermesine rağmen, farklı tipteki hücreler ve bireysel hücreler, gelişim ve çevresel tepkiler sırasında transkriptomlarında farklılıklar gösterir. Yüksek verimli tek hücreli RNA dizilimi (scRNA-seq), hücresel heterojenliği anlamak için benzeri görülmemiş bir güç sağlar. ScRNA-seq'in bitki bilimlerinde uygulanması, bir bitki hücresi atlası1'in başarılı bir şekilde oluşturulmasına katkıda bulunmuş, bitki dokularındaki nadir hücresel taksonları tanımlamak için kullanılmıştır2, bitki dokularındaki hücre tiplerinin bileşimi hakkında fikir vermiş ve hücresel kimliği ve bitki gelişimi ve farklılaşması sırasında kullanılan önemli işlevleri tanımlamak için kullanılmıştır. Ek olarak, yeni belirteç genleri keşfetmek için bitki dokularındaki uzaysal zamansal gelişimsel yörüngeleri çıkarmak mümkündür 1,2,3 4 ve farklı bitkilerde aynı hücre tipinin evrimsel korunumunu ortaya çıkarmak için scRNA-seq kullanarak önemli transkripsiyon faktörlerinin işlevlerini incelemek5. Abiyotik stresler, bitki büyümesi ve gelişimi üzerindeki en önemli çevresel etkiler arasındadır. Tek hücreli transkriptom dizilimi yoluyla farklı tedavi koşulları altında bitki dokularındaki hücre tiplerinin bileşimindeki değişiklikleri araştırarak, abiyotik stres yanıt mekanizması6'yı da çözebilirsiniz.
ScRNA dizilimini kullanarak hücre tipleri arasındaki transkripsiyonel heterojenliği çözme potansiyeli, hücre izolasyon yöntemine ve dizileme platformuna bağlıdır. Floresan ile aktive edilmiş hücre sıralama (FACS), ışık saçılmasına ve hücrelerin floresan özelliklerine dayanan scRNA-seq için hücrelerin bir alt popülasyonunu izole etmek için yaygın olarak kullanılan bir tekniktir. Transgenik teknoloji ile floresan işaretleyici hatlarının geliştirilmesi, FACS7 ile hücre izolasyonunun verimliliğini büyük ölçüde artırmıştır. Smart-seq28 kullanarak scRNA-seq iletmek, hücresel heterojeniteyi disseke etme yeteneğini daha da geliştirir. Smart-seq2 yöntemi, gen tespiti için iyi bir duyarlılığa sahiptir ve düşük bir transkript girişi9 ile bile genleri tespit edebilir. Toplu hücre tipi toplamaya ek olarak, modern hücre sıralayıcıları, Smart-seq210 veya CEL-seq211 gibi diğer çoğullanmış RNA-seq yöntemlerini kullanarak tek hücreli çözünürlükte transkriptom analizine izin veren tek hücreli bir dizin sıralama formatı sağlar. Tek hücreli veya hücre tipi sıralama, paralel multi-omik çalışmalar12,13 gibi diğer birçok aşağı akış uygulaması için potansiyel olarak kullanılabilir. Burada, ksilem-kutup perisiklus hücreleri, lateral kök kapak hücreleri, lateral kök başlangıç hücreleri, korteks hücreleri ve endodermal hücreler gibi bitki hücre tiplerini FACS tarafından Arabidopsis thaliana marker hücre hatlarının köklerinden izole etmek için sağlam ve çok yönlü bir protokol sunulmaktadır. Protokol ayrıca aşağı akış transkriptom analizi için Smart-seq2 kütüphanesinin oluşturulmasını içerir.
Protokol
Aşağıdaki protokol, aşağıdaki kök hücre tipleri için floresan ve floresan işaretleyici çizgileri olmayan A. thaliana vahşi tip (WT) tohumlar için optimize edilmiştir: ksilem-kutuplu perisiklus hücreleri (J0121), lateral kök başlangıç hücreleri, lateral kök kapak hücreleri (J3411), endodermis ve korteks hücreleri (J0571) (Şekil 1A). Tüm işaretleyici çizgileri, daha önce yayınlanmış bir rapor14'ü takiben vahşi tip bir Arabidopsis tesisine GATA23 promotör güdümlü GFP yapısının sokulmasıyla oluşturulan lateral kök başlatma hücresi işaretleyici hattı hariç, ticari bir kaynaktan (Malzeme Tablosuna bakınız) elde edilmiştir.
1. Bitki materyalinin hazırlanması
- A. thaliana WT tohumlarını ve floresan işaretleyici hattı tohumlarını, tohumları oda sıcaklığında oda sıcaklığında 15 dakika boyunca dönen bir inkübatörde% 20 ağartıcıda inkübe ederek sterilize edin.
- Tohumları çift damıtılmış suda (ddH2O) üç ila beş kez durulayın. Bu adımı steril temiz bir tezgahta gerçekleştirin.
- WT ve muhabir hattı tohumlarını yarı güçlü Murashige ve Skoog (MS) ortamına %0,8 agar (w/v)15 ile kaplayın. 4 °C'de 2 gün boyunca tabakalaştıktan sonra bitkileri 5 gün boyunca dikey olarak büyütün (23 ° C'de 16 saat ışık).
2. Protoplasting
- Çözüm A ve Çözüm B olarak adlandırılan protopkalıcı çözeltiler 2,5'i hazırlayın (bkz.
- 400 mM mannitol, %0,05 BSA, 20 mM MES (pH 5,7), 10 mM CaCl2 ve 20 mM KCl içeren Çözelti A'yı hazırlayın (bkz. A Çözümünü −20 °C'de 1 aya kadar saklayın.
- Çözelti A'nın taze bir aliquotuna %1 (w/v) selülaz R10, %1 (w/v) selülaz RS, %1 (w/v) hemiselülaz, %0,5 (w/v) pektolyaz ve %1 (w/v) macerozyme R10 ekleyerek Çözelti B'yi hazırlayın.
- Deneye başlamadan önce A Çözeltisi ve B Çözeltisini buz üzerinde yavaşça çözün.
- Kökleri temiz bir bıçak veya makas kullanarak kesin ve kökleri ~ 0,5 cm'lik parçalara ayırın. Kökleri 1,5 mL B Çözeltisine batırın ve ardından oda sıcaklığında 1,5-2 saat boyunca hafifçe döndürün (yaklaşık 18 rpm'de).
- Kök protoplastlarını 40 μm süzgeç ağından süzün (bkz.
- Süzgeç ağını 1-2 mL Çözelti A ile durulayın.
- Adım 2.4 ve adım 2.5'teki sıvıları birleştirin ve 4 ° C'de 5 dakika boyunca 300 x g'de santrifüjleyin. Süpernatantı bir pipetle atın, hücre peletini 500-600 μL Çözelti A'da yeniden askıya alın ve ardından hemen buzun üzerine yerleştirin.
- Askıya alınmış hücre çözeltisini, hücre sıralama için yeni bir 5 mL test tüpüne aktarın.
3. Floresan Aktif Hücre Sıralama (FACS)
- Sıralayıcıdaki cihaz kurulum adımlarını açın ve tamamlayın (bkz. Floresan kanallarını seçin, otofloresan taban çizgisini belirlemek için kontrol olarak bir WT tesisi (floresan yok) kullanın ve floresan yoğunluğuna ve FSC/SSC teklilerine göre sıralama kapısını ayarlayın (Şekil 2).
- Hücrelerin zarar görmesini önlemek için 1,5 mL'lik bir toplama tüpüne 500 μL Çözelti A (adım 2.1.1) ekleyin. Tüp başına 2.000-3.000 hücre toplayın.
- Sıralamadan sonra, numuneleri hemen buzun üzerine yerleştirin, hücreleri içeren toplama tüpünü 300 x g'de 4 ° C'de 5 dakika boyunca santrifüj edin ve süpernatantı bir pipetle çıkarın.
- 2 μL sıralanmış hücre alın ve floresan mikroskobu kullanarak floresan olup olmadığını kontrol edin (bkz.
- Sıralanan hücreleri −80 °C'de saklayın veya kütüphane yapımı için hemen kullanın (adım 4).
- Tek hücreli dizin sıralama için, 96 delikli plakayı bağdaştırıcıya yerleştirin. Plakanın konumunu, damlacık plakanın orta deliğine düşecek şekilde kalibre edin. Sıralama yaparken tek hücreli sıralama modunu seçin, sıralanan hücrelerin hedef sayısını 1 olarak girin ve sıralamaya başlayın.
4. Smart-seq2 kütüphane hazırlığı
- Ultra düşük girdi miktarının bir sonucu olarak, kontaminasyonsuz bir ortamda tek hücreli tip RNA-seq kütüphanesi inşası gerçekleştirin. Deneye başlamadan önce, tezgahı bir yüzey dekontaminantı8 (bakınız Malzeme Tablosu) ve% 75 etanol ile temizleyin.
- 0.33 μL% 10 Triton X-100, 0.55 μL RNaz inhibitörü ve 0.22 μL 0.1 M DTT'yi birleştirerek lizis tamponunu (karışım A) (Tablo 1) hazırlayın (bkz.
- Sıralanan numuneye 1 μL karışım A ekleyin ve steril bir havaneyle öğütün. Tercih edilen numune hacmi ≤0,5 μL'dir; hacmi 14 μL'ye çıkarmak için RNaz içermeyen su kullanın. her bir hücre örneğini 0,2 mL ince duvarlı bir PCR tüpüne aktarın.
- 0.44 μL oligo-dT 30 VN ters transkripsiyon (RT) reaksiyon astarı (100 μM) ve 4.4 μL dNTP (10mM) içeren karışım B'yi hazırlayın (Tablo 2) (bkz.
- Her tüpteki 14 μL'lik numuneye 4,4 μL B karışımı ekleyin, numuneyi karıştırmak için yavaşça pipet yapın ve numuneyi 72 °C'de 3 dakika boyunca inkübe edin. Kuluçkadan sonra, oligo-dT'yi poli A kuyruğuna melezleştirmek için numuneleri hemen buz üzerine koyun.
- Ters transkripsiyon reaksiyonu karışımını (karışım C) hazırlayın (Tablo 3) (bakınız Malzeme Tablosu). Numuneleri içeren her tüpe 21,6 μL C karışımı ekleyin. RT programını ortak bir PCR cihazında açın (Tablo 4).
- Ön amplifikasyon reaksiyonunu buz üzerinde gerçekleştirin. 44 μL 2x PCR polimeraz karışımı ve 0.88 μL IS PCR primeri (10 μM) birleştirerek D karışımını hazırlayın (Tablo 5) (bkz . 40 μL RT reaksiyon ürününe 40,8 μL karışım D ekleyin ve ön amplifikasyon programını çalıştırın (Tablo 6).
- Ampure XP boncuklarını kullanarak ön amplifikasyon reaksiyonu ürünlerini saflaştırın (bkz. Adım 4.7'den itibaren her numuneye 48 μL boncuk (0,6:1 oranında) ekleyin ve pipetleme ile numuneleri nazikçe karıştırın.
- Numuneleri oda sıcaklığında 10 dakika boyunca inkübe edin. Numuneleri içeren 1,5 mL'lik tüpleri manyetik bir ayırma standına 5 dakika boyunca yerleştirin. Boncukları rahatsız etmeden süpernatantı numunelerden dikkatlice atın.
- Boncukları 200 μL% 80 etanol içinde yeniden askıya alarak yıkayın ve etanol içeren süpernatanı atmadan önce numuneleri manyetik ayırma standına ( Malzeme Tablosuna bakınız) 3 dakika daha yerleştirin.
- Numuneleri 10 dakika boyunca hava ile kurutun ve hava kurutması sırasında kontaminasyonu ve çapraz kontaminasyonu önlemek için tüpü örtün.
- Boncukları 20 μLddH2O içinde tekrar askıya alın, numuneleri oda sıcaklığında 5 dakika inkübe edin ve ardından manyetik ayırma standına 5 dakika boyunca yerleştirin.
- Her tüpten 18 μL süpernatantı pipetleyin ve numuneleri yeni 1,5 mL santrifüj tüplerine aktarın. Bir DNA niceleme kiti kullanarak cDNA'nın kalitesini değerlendirmek, bir parça analizörü kullanarak her bir ön kütüphanenin boyut dağılımını belirlemek için numunenin 1 μL'sini kullanın (bkz.
- Bir dizileme kütüphanesi hazırlama kiti kullanarak adım 4.13'ün kütüphane öncesi ürününden Illumina dizilimi16 için bir cDNA kütüphanesi 8 oluşturun (bkz.
- Ampure XP boncuklarını kullanarak kütüphaneleri saflaştırın (adım 4.14'ten itibaren), saflaştırılmış kütüphaneleri ölçün ve adım 4.13'ü takip eden her kütüphanenin boyut dağılımını belirleyin.
NOT: Her kütüphanenin eşit nanomollerini bir araya getirerek, hiçbirinin aynı Illumina indeksi kombinasyonuna sahip olmamasını sağlayın. Aksi takdirde, kütüphaneler istenen sıralama çıktısına dayalı bir oranda bir araya getirilebilir ve Illumina sıralayıcısının aynı şeridinde birlikte sıralanabilir. Genel olarak, her kütüphaneyi >10 milyon haritalanmış okuma sağlayan 4-6 GB derinliğe kadar sıralamak, Arabidopsis genomunun 20x-30x kapsama alanını sağlar. Daha düşük sıralama derinlikleri de kabul edilebilir, ancak diferansiyel ifade analizinin önemini etkileyebilir.
5. RNA-seq veri analizi
- Trim-Galore17 kullanarak ham okumaları kırpın, ardından hisat2 18 (daehwankimlab.github.io/hisat2) kullanarak referans genoma eşleyin ve Picard19 (broadinstitute.github.io/picard) kullanarak PCR çoğaltılmış parçaları çıkarın.
- Her numune için en az üç biyolojik replika kullanarak DESeq220 ile diferansiyel olarak eksprese edilen genlerin (DEG) ham sayım işlemesini ve müteakip analizini gerçekleştirin. Pheatmap paketi ile gen ekspresyonunun kümelenmesini gerçekleştirin ve bir ekspresyon ısı haritasında görselleştirin.
NOT: Genlerin ve TE'lerin (dönüştürülebilir elementler) RPKM (milyon eşlenmiş okuma başına kilobaz başına okuma) değerleri Stringtie18 (github.com/gpertea/stringtie) ile hesaplanmış ve bir genom tarayıcısında görselleştirilmiştir.
Sonuçlar
Protoplast izolasyonu
Bu protokol, floresan A. thaliana kök işaretleyici çizgilerinin protoplast sıralaması için etkilidir. Bu belirteç çizgileri, floresan proteinlerinin özellikle hedef hücre tiplerinde eksprese edilen genlerle füzyonu veya arttırıcı tuzak çizgileri kullanılarak geliştirilmiştir (Şekil 1). Çok sayıda doku ve organ, model bitkilerde ve mahsullerde spesifik floresan belirteçleri eksprese eden hücre tiplerine diseke edilmiştir.
FACS popülasyonu, sıralanmış hücreler ve kütüphane QC
Vahşi tip bir bitkiyi kontrol olarak kullanarak ve ileri saçılma (FSC) ve yan saçılma (SSC) gibi kapıları ayarlayarak, ilgilenilen hücrelerin büyük bir popülasyonunu ve otofloresan için bir taban çizgisini belirledik ve floresana özgü işaretli hücreleri başarıyla sıraladık (Şekil 2). Son sıralama kütüphanelerinin kalitesi (adım 4.15'ten itibaren) parça boyutu dağılım analizi ile belirlenmiştir. Yaklaşık 2.000 ksilem-kutuplu perisiklus hücresi, lateral kök primordia hücreleri, endodermis/korteks hücreleri ve lateral kök kapağı hücrelerinden oluşan RNA-seq kütüphanesinin temsili sonuçları Şekil 3A-D'de gösterilmiştir.
İfade kalıplarının analizi
Sıralama verilerinin kalitesi, sıralama derinliği, eşleme hızı ve fastQC raporları gibi çoklu analiz prosedürlerinden değerlendirilebilir. Dizileme verilerinin doğruluğu ve duyarlılığı, izole edilmiş hücre tipleri ile tüm doku arasındaki DEG analizinden tanımlanabilen bir dizi hücre tipi zenginleştirilmiş genin varlığı ile gösterilebilir. Bazı hücre tiplerinde önemli ölçüde daha yüksek ekspresyon seviyelerine sahip genler, hücre tipi zenginleştirilmiş genler olarak tanımlanabilir. Bu arada, her hücre tipinin genom-tarayıcı görünümleri, bilinen belirteç genlerinin ekspresyon seviyelerini göstermek ve işaretleyici genlerin ekspresyon modelinin hücre tipi ekspresyon verilerinde yeniden yapılandırılıp yapılandırılamayacağını test etmek için yan yana karşılaştırılabilir. Örnek olarak, dört kök hücre tipinden herhangi birinde zenginleştirilmiş genler kümelenmiş ve farklı hücre tipleri arasında gen ekspresyonunun özgüllüğünü gösteren ısı haritasında gösterilmiştir (Şekil 4). YUCCA3, MYB36, WOX5 ve PFA1 incelendi ve ifade kalıpları önceki raporlara göre beklendiği gibi 21,22,23,24 idi. Veri analizi işlem hatları ve temsili ham sıralama verileri genel bir depoda (github.com/gaolabsjtu/root_cell_types_RNAseq) kullanılabilir.
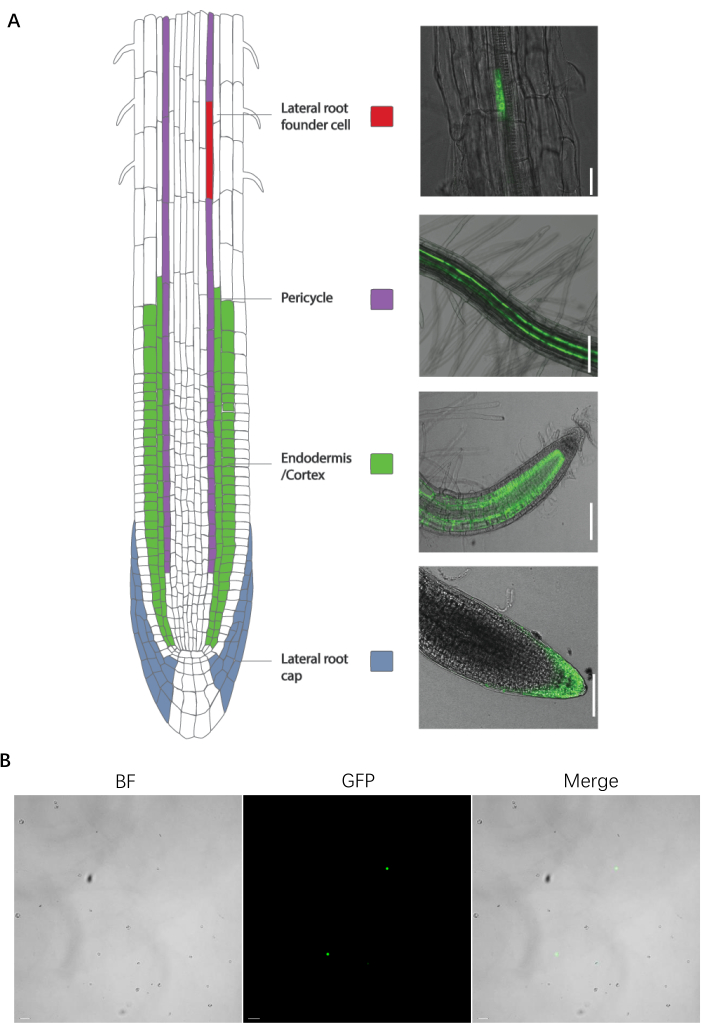
Şekil 1: Kök ve protoplast preparatının spesifik hücre tipi işaretleyici çizgisi . (A, sol) Geliştirici tuzak hattı tanıtımı ve görüntüleri. (A, sağda) Kökün spesifik hücre tipi işaretleyici çizgisi. (B) Sıralamadan önce protoplast preparatının bir görüntüsü. Yanal kök kurucu hücre, perisiklus, endodermis/korteks, lateral kök kapağı ve protoplastlar için ölçek çubukları sırasıyla 25 μm, 100 μm, 100 μm, 75 μm ve 20 μm'yi temsil eder. Bu şeklin daha büyük bir versiyonunu görmek için lütfen buraya tıklayın.

Şekil 2: Belirli kök hücre tipleri için FACS'ın kurulması. Bir FACS deneyi örneği: (A) SSC ve FSC kullanan ana popülasyon; (B) GFP-pozitif (+) ve GFP-negatif (-) kapılar; (C) sıralanmış hücrelerin parlak alan ve (D) floresan görüntüleri. Ölçek çubuğu 50 μm'yi temsil eder. Bu şeklin daha büyük bir versiyonunu görmek için lütfen buraya tıklayın.
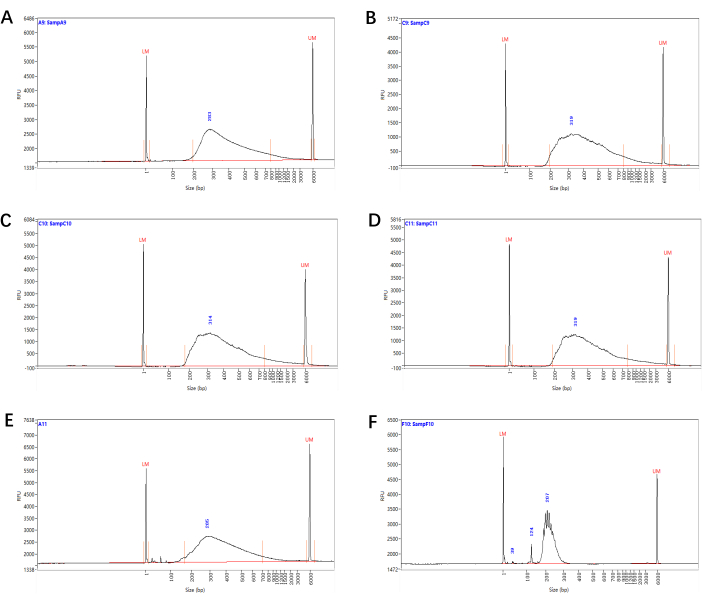
Şekil 3: Smart-seq2 sıralama kitaplıklarının boyut dağılımı. Ksilem-kutuplu perisiklus hücrelerinden (A), lateral kök primordia hücrelerinden (B), endodermis/korteks hücrelerinden (C) ve lateral kök kapak hücrelerinden (D) üretilen dizileme kütüphanelerinin temsili parça boyutu dağılımları (adım 4.15'ten itibaren). (E) Astar/adaptör dimer piklerine sahip, boyut seçiminden sonra da sıralanabilen küçük boyutlu bir kütüphane. (F) Başarısız kütüphane hazırlığına işaret eden, anormal boyutta dağılımı olan bir kütüphane. Bu şeklin daha büyük bir versiyonunu görmek için lütfen buraya tıklayın.

Şekil 4: Hücre tipi ile zenginleştirilmiş gen analizi. Her hücre tipi ile tüm kök arasında bir DEG analizi yapıldı; Her hücre tipinde dört kattan fazla upregülasyona sahip genler, hücre tipi ile zenginleştirilmiş genler olarak tanımlanmıştır. Bu genler birleştirildi, kümelendi ve ısı haritası (üst panel) grafiğinde görselleştirildi. Hücre tipi ile zenginleştirilmiş genler, bilinen birçok belirteç genini içeriyordu; YUCCA3, MYB36, WOX5 ve PFA1 gibi tipik belirteç genleri, genom tarayıcısında (alt panel) gösterilmek üzere seçildi. Kısaltmalar: LRP = lateral root primordia; Endo/Cor = endodermis/korteks; LRC = yanal kök kapağı. Bu şeklin daha büyük bir versiyonunu görmek için lütfen buraya tıklayın.
| Bileşen | Hacim (μL) |
| %10 Triton X-100 | 0.33 |
| RNaz inhibitörü | 0.55 |
| DTT (0,1 M) | 0.22 |
Tablo 1: Hücre lizis tamponunun hazırlanması için reaksiyon bileşenleri (karışım A).
| Bileşen | Hacim (μL) |
| Oligo-dT30VN (100 μM) | 0.44 |
| dNTP'ler (10 mM) | 4.4 |
Tablo 2: B karışımının hazırlanması için reaksiyon bileşenleri.
| Bileşen | Hacim (μL) |
| SuperScript IV arabelleği (5x) | 8.8 |
| Betain (5 m) | 8.8 |
| DTT (0,1 M) | 2.2 |
| MgCl2 (1 M) | 0.264 |
| TSO (100 μM) | 0.44 |
| SuperScript IV ters transkriptaz (200 U/μL) | 2.2 |
| RNaz inhibitörü | 1.1 |
Tablo 3: Ters transkripsiyon PCR karışımının (C karışımı) hazırlanması için reaksiyon bileşenleri.
| Devir | Sıcaklık (°C) | Saat |
| 1 | 50 | 90 dk |
| 2-11 | 55 | 2 dk |
| 50 | 2 dk | |
| 12 | 70 | 15 dk |
| 13 | 4 | ∞ |
Tablo 4: mRNA'dan cDNA sentezi için ters transkripsiyon (RT) PCR ayarları.
| Bileşen | Hacim (μL) |
| KAPA HiFi HotStart ReadyMix (2x) | 44 |
| IS PCR astarı (10 μM) | 0.88 |
Tablo 5: Ön amplifikasyon reaksiyonu karışımının (karışım D) hazırlanması için reaksiyon bileşenleri.
| Devir | Sıcaklık (°C) | Saat |
| 1 | 98 | 5 dk |
| 2-13 | 98 | 20 saniye |
| 67 | 30 saniye | |
| 72 | 3 dk | |
| 14 | 72 | 5 dk |
| 15 | 4 | ∞ |
Tablo 6: Ön amplifikasyon reaksiyonu için PCR program ayarları.
Tartışmalar
Smart-seq2 tabanlı protokol, yüzlerce hücreden güvenilir sıralama kitaplıkları oluşturabilir8. Başlangıç malzemesinin kalitesi, transkriptom analizinin doğruluğu için esastır. FACS, ilgilenilen hücreleri hazırlamak için güçlü bir araçtır, ancak bu prosedür, özellikle de protoplasting adım, bitki uygulamaları için optimize edilmelidir. Lazer yakalama mikrodiseksiyonu (LCM) veya manuel disseke hücreler de girdi25,26 olarak kullanılabilir, bu nedenle burada sağlanan protokol, bilinen belirteç genleri olan veya olmayan çeşitli bitki türlerinde ve hücre tiplerinde potansiyel olarak kullanılabilir.
Bitki materyalinin hazırlanması
Bitki gelişim biyolojisinde, FACS genellikle bir floresan belirteci eksprese eden zenginleştirilmiş hücre popülasyonlarını izole etmek için kullanılır. Böyle bir belirteci ifade eden bitkiler protoplastlanır ve protoplastlar sonunda hücre tipine özgü belirtecin ekspresyonuna dayanarak saf alt popülasyonlara ayrılır. Bu nedenle, floresan işaretleyicinin sinyali güçlü ve spesifik olmalıdır. Ek olarak, araştırmacı hangi belirteçlerin önceden sıralanabileceğini kontrol etmelidir. İhtiyaç duyulan tohum sayısı, ifade seviyesine, işaretleyicinin nerede ifade edildiğine, aşağı akış uygulamaları için kaç hücreye ihtiyaç duyulduğuna ve kaç kopyanın sıralandığına bağlıdır. İfade, bitki başına çok az hücreye sahip tek bir hücre tipindeyse, genellikle işaretleyicinin daha fazla sayıda hücrede eksprese edilmesinden daha fazla sayıda bitkiye ihtiyaç vardır. Bireysel işaretleyici çizgileri için gereken tohum sayısını ampirik olarak belirlemek en iyisidir. Sıralama için pencereyi optimize etmek ve sabit sayıda bitkiden kaç hücrenin ortaya çıkacağını bilmek için her bir işaretleyici çizgisiyle ön deneyler yapmak özellikle anlamlıdır. Deney materyali kökler ise, köklerin agar içine batmasını önlemek ve temiz köklerin kesilmesini kolaylaştırmak için, tohum kaplamasından önce orta içeren agar üzerine uygun büyüklükte ve otoklavlanmış bir ağ döşenebilir. Düşük sıcaklıktaki tabakalaşma, tohum çimlenmesine ve gelişmesine yardımcı olur, bu nedenle tohumları sterilize ettikten veya kapladıktan sonra 2 gün boyunca 4 ° C'de tabakalaştırmanızı ve ardından bitkileri dikey olarak büyütmenizi öneririz.
Protoplasting ve FACS
Tabakalaşmadan 5-6 gün sonra protoplasting ve sıralama iyi çalışır. Çözüm A ve Çözüm B, 0,22 μm süzgeç kullanılarak filtrelenmelidir. Ek olarak, B çözeltisinin -20 ° C'de daha uzun süre saklanması enzimlerin verimliliğini azaltır. Başlamadan önce, Çözelti A ve Çözelti B, yaklaşık 20 dakika süren buz üzerinde hafifçe çözülmelidir. Çözelti B çalkalanmamalıdır, çünkü sallanan Çözelti B enzimleri bozabilir ve aşırı kabarcık oluşumuna neden olabilir. Süzgeç ağını Çözelti A ile birden çok kez yıkamak, adım 2.5'te elde edilen hücre sayısını artırabilir. Adım 2.6'da açıklandığı gibi, protoplastlar peletin yakınında altta olduğundan ve görülemediğinden, tüm süpernatant aspire edilmemelidir. Sıralamadan önce, daha iyi sıralama verimliliği elde etmek için protoplastların konsantrasyonunun yaklaşık 105-10 6 hücre / mL olmasını sağlamak en iyisidir. Yeni bir deney kurarken, WT tesisinden aynı doku, sıralama kapısının kurulması için bir kontrol olarak gereklidir. Adım 3.1'de, önce floresan kanallarının (örneğin, PE ve FITC) seçilmesi ve SSC ve FSC'ye göre ana popülasyonu belirlemek için bir kontrol örneği kullanılması önerilir. Ek olarak, kontrol sinyalinin kapının solunda bulunduğundan emin olmak için floresan kanalının voltajını değiştirerek kapının konumunu ayarlamanız önerilir (yani, negatif grup 103'ün altında bulunur). Sıralama süresi, protoplasting veya sıralama nedeniyle ortaya çıkabilecek gen ekspresyonundaki değişiklikleri önlemek için 30 dakika veya 60 dakikadan az ile sınırlandırılmalıdır. Sıralamadan sonra, az sayıda sıralanmış hücre toplanmalı ve floresan açısından kontrol edilmelidir (adım 3.4). Aşağı akış kütüphane yapısı üzerinde herhangi bir etkiyi önlemek için süpernatanttan mümkün olduğunca fazla tampon çıkarılmalıdır (adım 3.3).
RNA-seq kütüphanesinin inşası
RNA-seq kütüphanesinin inşası için, RNA'nın bozulmasını azaltmak için sıralamadan hemen sonra hücrelerin kullanılmasını öneririz. Çok fazla hücreyle başlamanız önerilmez, çünkü bu yetersiz reaksiyonlar nedeniyle düşük kaliteli bir kütüphaneye neden olabilir. Adım 4.7 ve adım 4.14'teki PCR döngülerinin sayısı, RNA / cDNA'nın giriş miktarına bağlıdır. Daha az girdi olduğunda döngü sayısı artırılabilir veya daha fazla girdi olduğunda azaltılabilir. Bu nedenle, gerçek zamanlı PCR'yi aynı amplifikasyon prosedürüyle çalıştırmak için adım 4.7 ve adım 4.14'teki karışık örneklerden bazılarının alınması ve daha sonra ön kütüphanenin / kütüphanenin resmi amplifikasyonundan önce qPCR'nin sonuçlarına dayanarak nihai döngü sayısının belirlenmesi önerilir. Ek olarak, saflaştırma adımı 4.8'den önce, Ampure XP boncukları oda sıcaklığında en az 10 dakika dengelenmeli ve daha sonra iyice girdaplanmalıdır. Saflaştırma adımındaki boncukların hacmi 0.8: 1 oranının üzerine çıkarılmamalıdır. Aksi takdirde, bu astar dimerlerinin taşınmasını artıracaktır. Ek olarak, zor yeniden süspansiyonu önlemek için adım 4.11'de boncukların aşırı kurutulmasından kaçınılmalıdır. Nitelikli bir ön kütüphanenin ortalama büyüklüğü yaklaşık 1,5-2 kb ve az miktarda kısa (<500 bp) parçaya sahip olmalıdır. Kütüphanelerdeki anormal boyut dağılımları veya küçük boyutlu astar/adaptör dimer pikleri düşük kalitenin göstergeleridir ve bu numuneler atılmalı veya daha fazla boncuk saflaştırma turuna tabi tutulmalıdır (adım 4.15) (Şekil 3E-F).
Sınırlama
Bu protokol, hücreleri diğer bitki dokularından ve diğer bitki türlerinden izole etmek için uygulanabilir. Bununla birlikte, hücre izolasyon süreci büyük ölçüde protoplastların hazırlanmasına bağlıdır. Dokunun içinde bulunan ve / veya sayıca az olan vasküler hücreler ve dişi cinsiyet hücreleri gibi bazı hücre tiplerinin izole edilmesi zordur. Protoplastların hazırlanmasının zor olduğu hücreler için, floresanla aktive edilmiş çekirdekler sıralama (FANS)27 , kullanılabilecek isteğe bağlı bir yöntemdir. Bu arada, FACS ile spesifik hücre tiplerini elde etmek için bu protokolün kullanılması, floresan işaretleyici hatlarının mevcudiyetine bağlıdır. Bu tür işaretleyici çizgilerin eksikliği, bu yöntemlerin mahsullerde ve bahçecilik bitkilerinde kullanımını sınırlar. Bitkilerde yüksek verimli tek hücreli RNA-seq teknolojisinin uygulanması, hücre tipi belirteç hatları geliştirmek ve FACS tabanlı hücre tipi RNA-seq ve multi-omik çalışmaların uygulama kapasitesini genişletmek için daha fazla kullanılabilecek yeni hücre tipine özgü belirteç genlerini ortaya çıkaracaktır.
Açıklamalar
Yazarların açıklayacak hiçbir şeyi yok.
Teşekkürler
Bu protokolü Şangay Jiao Tong Üniversitesi Tarım ve Biyoloji Fakültesi'nin tek hücreli multi-omik tesisinde kurduk ve Çin Ulusal Doğa Bilimleri Vakfı (Hibe No. 32070608), Şangay Pujiang Programı (Hibe No. 20PJ1405800) ve Şangay Jiao Tong Üniversitesi (Hibe No. Agri-X20200202, 2019TPB05) tarafından desteklendik.
Malzemeler
| Name | Company | Catalog Number | Comments |
| 0.22 µm strainer | Sorfa | 622110 | |
| Agar | Yeasen | 70101ES76 | |
| Agilent fragment analyzer | Aglient | Aglient 5200 | |
| Agilent high-sensitivity DNA kit | Aglient | DNF-474-0500 | |
| Ampure XP beads | BECKMAN | A63881 | |
| Betaine | yuanye | S18046-100g | |
| Bleach | Mr Muscle | FnBn83BK | 20% (v/v) bleach |
| BSA | sigma | 9048-46-8 | |
| CaCl2 | yuanye | S24109-500g | |
| Cellulase R10 | Yakult (Japan) | 9012-54-8 | |
| Cellulase RS | Yakult (Japan) | 9012-54-8 | |
| Centrifuge tube (1.5 mL) | Eppendolf | 30121589 | |
| DNase, RNase, DNA and RNA Away Surface Decontaminants | Beyotime | R0127 | |
| dNTPs (10 mM) | NEB | N0447S | |
| DTT (0.1 M) | invitrogen | 18090050 | |
| Ethanol | Sinopharm Chemical Reagent Co., Ltd | 100092680 | |
| FACS | BD FACS Melody | BD-65745 | |
| FACS | Sony | SH800S | |
| Filter tip (1000 µL) | Thermo Scientific | TF112-1000-Q | |
| Filter tip (200 µL) | Thermo Scientific | TF140-200-Q | |
| Filter tip (10 µL) | Thermo Scientific | TF104-10-Q | |
| Filter tip (100 µL) | Thermo Scientific | TF113-100-Q | |
| Fluorescent microscope | Nikon | Eclipse Ni-E | |
| Four-Dimensional Rotating Mixer | Kylin -Bell | BE-1100 | |
| Hemicellulase | sigma | 9025-56-3 | |
| IS PCR primer | 5'-AAGCAGTGGTATCAACGCAGAG T-3' | ||
| KAPA HiFi HotStart ReadyMix(2X) | Roche | 7958935001 | |
| KCl | Sinopharm Chemical Reagent Co., Ltd | 7447-40-7 | |
| Macerozyme R10 | Yakult (Japan) | 9032-75-1 | |
| Magnetic separation stand | invitrogen | 12321D | |
| Mannitol | aladdin | 69-65-8 | |
| MES | aladdin | 145224948 | |
| MgCl2 | yuanye | R21455-500ml | |
| Microcentrifuges | Eppendorf | Centrifuge 5425 | |
| Micro-mini-centrifuge | Titan | Timi-10k | |
| MS | Phytotech | M519 | |
| Nextera XT DNA Library Preparation Kit | illumina | FC-131-1024 | |
| oligo-dT30VN primer | 5'-AAGCAGTGGTATCAACGCAGAG TACTTTTTTTTTTTTTTTTTTTTTTT TTTTTTTTTTVN-3' | ||
| PCR instrument | Thermal cycler | A24811 | |
| Pectolyase | Yakult (Japan) | 9033-35-6 | |
| Plant marker lines | Nottingham Arabidopsis Stock Centre (NASC) | ||
| Qubit 1x dsDNA HS Assay Kit | invitrogen | Q33231 | |
| Qubit 2.0 fluorometer | invitrogen | Q32866 | |
| RNase inhibitor | Thermo Scientific | EO0382 | |
| RNase-free water | invitrogen | 10977023 | |
| Solution A | 400 mM mannitol, 0.05 % BSA , 20 mM MES (pH5.7), 10 mM CaCl2, 20 mM KCl | ||
| Solution B | 1 % (w/v)cellulase R10, 1 % (w/v) cellulase RS, 1 % (w/v)hemicellulase, 0.5 % (w/v)pectolyase and 1 % (w/v) Macerozyme R10 in a fresh aliquot of solution A | ||
| Sterile pestle | BIOTREAT | 453463 | |
| Strainer (40 µm ) | Sorfa | 251100 | |
| SuperScript IV reverse transcriptase (200 U/µL) | invitrogen | 18090050 | |
| SuperScript IV buffer (5x) | invitrogen | 18090050 | |
| Test tube (5 mL) | BD Falcon | 352052 | |
| Thin-walled PCR tubes with caps (0.5 mL) | AXYGEN | PCR-05-C | |
| Triton X-100 | Sangon Biotech | A600198-0500 | |
| TSO primer | 5'-AAGCAGTGGTATCAACGCAGAG TACATrGrG+G-3' | ||
| Vortex | Titan | VM-T2 |
Referanslar
- Zhang, T. -. Q., Chen, Y., Liu, Y., Lin, W. -. H., Wang, J. -. W. Single-cell transcriptome atlas and chromatin accessibility landscape reveal differentiation trajectories in the rice root. Nature Communications. 12 (1), 2053 (2021).
- Denyer, T., et al. Spatiotemporal developmental trajectories in the Arabidopsis root revealed using high-throughput single-cell RNA sequencing. Developmental Cell. 48 (6), 840-852 (2019).
- Liu, Q., et al. Transcriptional landscape of rice roots at the single-cell resolution. Molecular Plant. 14 (3), 384-394 (2021).
- Liu, Z., et al. Global dynamic molecular profiling of stomatal lineage cell development by single-cell RNA sequencing. Molecular Plant. 13 (8), 1178-1193 (2020).
- Shahan, R., et al. A single-cell Arabidopsis root atlas reveals developmental trajectories in wild-type and cell identity mutants. Developmental Cell. 57 (4), 543-560 (2022).
- Wendrich, J. R., et al. Vascular transcription factors guide plant epidermal responses to limiting phosphate conditions. Science. 370 (6518), (2020).
- Carter, A. D., Bonyadi, R., Gifford, M. L. The use of fluorescence-activated cell sorting in studying plant development and environmental responses. The International Journal of Developmental Biology. 57 (6-8), 545-552 (2013).
- Picelli, S., et al. Full-length RNA-seq from single cells using Smart-seq2. Nature Protocols. 9 (1), 171-181 (2014).
- Wang, X., He, Y., Zhang, Q., Ren, X., Zhang, Z. Direct comparative analyses of 10X genomics chromium and Smart-seq2. Genomics Proteomics Bioinformatics. 19 (2), 253-266 (2021).
- Serrano-Ron, L., et al. Reconstruction of lateral root formation through single-cell RNAsequencing reveals order of tissue initiation. Molecular Plant. 14 (8), 1362-1378 (2021).
- Hashimshony, T., et al. CEL-Seq2: Sensitive highly-multiplexed single-cell RNA-Seq. Genome Biology. 17, 77 (2016).
- Macaulay, I. C., et al. G&T-seq: Parallel sequencing of single-cell genomes and transcriptomes. Nature Methods. 12 (6), 519-522 (2015).
- Angermueller, C., et al. Parallel single-cell sequencing links transcriptional and epigenetic heterogeneity. Nature Methods. 13 (3), 229-232 (2016).
- De Rybel, B., et al. A novel aux/IAA28 signaling cascade activates GATA23-dependent specification of lateral root founder cell identity. Current Biology. 20 (19), 1697-1706 (2010).
- Duncombe, S. G., Barnes, W. J., Anderson, C. T. Imaging the delivery and behavior of cellulose synthases in Arabidopsis thaliana using confocal microscopy. Methods in Cell Biology. 160, 201-213 (2020).
- Levy, S. E., Myers, R. M. Advancements in next-generation sequencing. Annual Review of Genomics and Human Genetics. 17 (1), 95-115 (2016).
- Ooi, C. C., et al. High-throughput full-length single-cell mRNA-seq of rare cells. PLoS One. 12 (11), e0188510 (2017).
- Pertea, M., Kim, D., Pertea, G. M., Leek, J. T., Salzberg, S. L. Transcript-level expression analysis of RNA-seq experiments with HISAT, StringTie and Ballgown. Nature Protocols. 11 (9), 1650-1667 (2016).
- Tsyganov, K., Perry, A., Archer, S., Powell, D. RNAsik: A Pipeline for complete and reproducible RNA-seq analysis that runs anywhere with speed and ease. Journal of Open Source Software. 3, 583 (2018).
- Love, M. I., Huber, W., Anders, S. Moderated estimation of fold change and dispersion for RNA-seq data with DESeq2. Genome Biology. 15 (12), 550 (2014).
- Kamiya, T., et al. The MYB36 transcription factor orchestrates Casparian strip formation. Proceedings of the National Academy of Sciences of the United States of America. 112 (33), 10533-10538 (2015).
- Zhang, Y., et al. Two types of bHLH transcription factor determine the competence of the pericycle for lateral root initiation. Nature Plants. 7 (5), 633-643 (2021).
- Haecker, A., et al. Expression dynamics of WOX genes mark cell fate decisions during early embryonic patterning in Arabidopsis thaliana. Development. 131 (3), 657-668 (2004).
- Chen, Q., et al. Auxin overproduction in shoots cannot rescue auxin deficiencies in Arabidopsis roots. Plant Cell Physiol. 55 (6), 1072-1079 (2014).
- Nichterwitz, S., et al. Laser capture microscopy coupled with Smart-seq2 for precise spatial transcriptomic profiling. Nature Communications. 7, 12139 (2016).
- Long, J., et al. Nurse cell--derived small RNAs define paternal epigenetic inheritance in Arabidopsis. Science. 373 (6550), (2021).
- Gutzat, R., et al. Arabidopsis shoot stem cells display dynamic transcription and DNA methylation patterns. EMBO Journal. 39 (20), e103667 (2020).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır