Method Article
植物細胞タイプの単離とトランスクリプトーム解析
要約
ハイスループットscRNA-seqメソッドの実現可能性と有効性は、植物研究におけるシングルセル時代の到来を告げるものです。ここでは、シ ロイヌナズ ナの根の特定の細胞タイプを単離し、その後のトランスクリプトームライブラリの構築と解析を行うための堅牢で完全な手順を紹介します。
要約
多細胞生物では、発生プログラミングと環境応答は、異なる細胞タイプまたは細胞内でさえ非常に異なる可能性があり、これは細胞の不均一性として知られています。近年、次世代シーケンシング(NGS)技術と組み合わせたシングルセルおよびセルタイプの分離は、シングルセル分解能で生物学的プロセスを研究するための重要なツールになっています。しかしながら、植物細胞壁が存在するため、植物細胞を単離することは比較的困難であり、それは植物における単一細胞アプローチの適用を制限する。このプロトコルは、植物細胞による蛍光活性化セルソーティング(FACS)ベースのシングルセルおよびセルタイプの分離のための堅牢な手順を説明しており、ダウンストリームのマルチオミクス解析やその他の研究に適しています。 シロイヌナ ズナの根蛍光マーカー株を用いて、木部極周回細胞、側根初期細胞、側根冠細胞、皮質細胞、内胚葉細胞などの特定の細胞種がどのように分離されるかを示します。さらに、Smart-seq2を用いた効果的なダウンストリームトランスクリプトーム解析法も提供します。細胞単離法とトランスクリプトーム解析技術は、他の細胞種や植物種にも適応でき、植物科学に幅広い応用の可能性を秘めています。
概要
細胞はすべての生物の基本単位であり、構造的および生理学的機能を果たします。多細胞生物の細胞は見かけのシンクロニシティを示しますが、異なる種類の細胞や個々の細胞は、発生中のトランスクリプトームと環境応答に違いを示します。ハイスループットシングルセルRNAシーケンシング(scRNA-seq)は、細胞の不均一性を理解するための前例のないパワーを提供します。scRNA-seqを植物科学に応用することは、植物細胞アトラスの構築に成功し1、植物組織2の希少な細胞分類群の同定に使用され、植物組織における細胞タイプの組成に関する洞察を提供し、細胞同一性と植物の発生および分化に使用される重要な機能を特定するために使用されてきました。さらに、植物組織1,2,3における時空間発生軌跡を推測して新しいマーカー遺伝子を発見し4、scRNA-seqを用いて重要な転写因子5の機能を研究することで、異なる植物における同じ細胞種の進化的保存を明らかにすることができます3。.非生物的ストレスは、植物の成長と発達に対する最も重要な環境影響の一つです。シングルセルトランスクリプトームシーケンシングを通じて、さまざまな処理条件下での植物組織の細胞タイプの組成の変化を調べることにより、非生物的ストレス応答メカニズムを解明することもできます6。
scRNAシーケンシングを使用して細胞タイプ間の転写不均一性を解決できる可能性は、細胞分離方法とシーケンシングプラットフォームによって異なります。蛍光活性化セルソーティング(FACS)は、光散乱と細胞の蛍光特性に基づいてscRNA-seqの細胞の亜集団を単離するために広く使用されている技術です。トランスジェニック技術による蛍光マーカー株の開発により、FACS7による細胞単離の効率が大幅に向上しました。Smart-seq28を用いてscRNA-seqを実施すると、細胞の不均一性を解剖する能力がさらに高まります。Smart-seq2法は遺伝子検出感度が高く、転写物入力量が少ない場合でも遺伝子を検出できます9。バルクセルタイプの収集に加えて、最新のセルソーターはシングルセルインデックスソーティングフォーマットを提供し、Smart-seq210またはCEL-seq211などの他のマルチプレックスRNA-seqメソッドを使用してシングルセル分解能でトランスクリプトーム解析を可能にします。シングルセルまたはセルタイプのソーティングは、並行マルチオミクス研究など、他の多くのダウンストリームアプリケーションに使用できる可能性があります12,13。ここでは、FACSによってシロイヌナズナマーカー細胞株の根から木部極周回細胞、側根冠細胞、側根初期細胞、皮質細胞、内胚葉細胞などの植物細胞タイプを単離するための堅牢で汎用性の高いプロトコルを紹介します。このプロトコルには、ダウンストリームトランスクリプトーム解析用のSmart-seq2ライブラリの構築もさらに含まれます。
プロトコル
以下のプロトコルは、次の根細胞タイプの蛍光および蛍光マーカーラインを持たないA. thaliana野生型(WT)種子用に最適化されています:木部極周環状細胞(J0121)、側根初期細胞、側根冠細胞(J3411)、内皮および皮質細胞(J0571)(図1A)。以前に発表された第14報告書に続いて、GATA23プロモーター駆動GFP構築物を野生型シロイヌナズナ植物に導入することによって生成された側根開始細胞マーカー株を除いて、すべてのマーカー株は市販の供給源(材料の表を参照)から入手した。
1.植物材料の調製
- A. thaliana WT種子および蛍光マーカー系統種子を、室温で15分間回転インキュベーター内で20%漂白剤中でインキュベートすることにより滅菌する。
- 種子を二重蒸留水(ddH2O)で3〜5回すすいでください。このステップは、滅菌済みのクリーンベンチで実行します。
- WTおよびレポーターラインシードを、0.8%寒天(w / v)15を含む半強度のムラシゲおよびスクーグ(MS)培地にプレートします。4°Cで2日間成層した後、植物を垂直に5日間(23°Cで16時間光)育てます。
2.プロトプラスト
- 溶液Aおよび溶液Bと呼ばれるプロトプラスト溶液2,5を調製する(材料表を参照)。
- 400 mM マンニトール、0.05% BSA、20 mM MES(pH 5.7)、10 mM CaCl2、および 20 mM KCl を含む溶液 A を調製 します (材料表を参照)。溶液Aを-20°Cで最大1ヶ月間保存します。
- 溶液Aの新しいアリコートに1%(w/v)セルラーゼR10、1%(w/v)セルラーゼRS、1%(w/v)ヘミセルラーゼ、0.5%(w/v)ペクトリアーゼ、および1%(w/v)マセロチームR10を加えて、溶液Bを調製します。
- 実験を開始する前に、溶液Aと溶液Bを氷上で静かに解凍します。
- きれいな刃やハサミで根を切り落とし、根を~0.5cmに切ります。根を1.5 mLの溶液Bに浸し、続いて室温で1.5〜2時間穏やかに回転(約18 rpm)します。
- ルートプロトプラストを40 μmのストレーナーメッシュでろ過します(材料表を参照)。
- ストレーナーメッシュを1〜2 mLの溶液Aですすぎます。
- ステップ2.4とステップ2.5の液体を混ぜ合わせ、300 x g で4°Cで5分間遠心分離します。 上清をピペットで廃棄し、細胞ペレットを500〜600 μLの溶液Aに再懸濁し、すぐに氷上に置きます。
- 再懸濁した細胞溶液を新しい5 mL試験管に移し、細胞を選別します。
3. 蛍光活性化セルソーティング(FACS)
- スイッチを入れて、ソーターで機器のセットアップ手順を完了します( 材料表を参照)。蛍光チャンネルを選択し、WTプラント(蛍光なし)をコントロールとして使用して自家蛍光のベースラインを決定し、蛍光強度とFSC/SSCシングレットに基づいてソーティングゲートを調整します(図2)。
- 500 μLの溶液A(ステップ2.1.1)を1.5 mLの収集チューブに加えて、細胞の損傷を防ぎます。チューブあたり2,000〜3,000個の細胞を収集します。
- 選別後、直ちにサンプルを氷上に置き、細胞を入れた回収チューブを300 x g で4°Cで5分間遠心分離し、ピペットで上清を除去した。
- 選別した細胞を2 μL採取し、蛍光顕微鏡を使用して蛍光を確認します( 材料表を参照)。
- ソートしたセルを-80°Cで保存するか、すぐにライブラリの構築に使用します(ステップ4)。
- シングルセルインデックスソーティングの場合は、96ウェルプレートをアダプターに入れます。液滴がプレートの中央の穴に落ちるようにプレートの位置を調整します。ソート時に単一セルソートモードを選択し、ソートされたセルの目標数を1として入力し、ソートを開始します。
4. スマートシーク2ライブラリの準備
- 超低入力の結果、汚染のない環境でシングルセル型RNA-seqライブラリ構築を行うことができます。実験を開始する前に、表面除染剤8 ( 材料表を参照)と75%エタノールでベンチを清掃してください。
- 0.33 μLの10%Triton X-100、0.55 μLのRNase阻害剤、および0.22 μLの0.1 M DTTを組み合わせて、溶解バッファー(混合物A)(表1)を調製します( 材料の表を参照)。
- 選別したサンプルに混合物Aを1 μL加え、滅菌乳棒で粉砕します。好ましいサンプル容量は≤0.5μLです。RNaseフリーの水を使用して、容量を14 μLに構成します。 各単一細胞サンプルを0.2 mLの薄肉PCRチューブに移します。
- 0.44 μLのオリゴdT 30 VN逆転写(RT)反応プライマー(100 μM)および4.4 μLのdNTP(10mM)を含む混合物Bを調製します(表2)( 材料の表を参照)。
- 各チューブ内の14 μLのサンプルに4.4 μLの混合物Bを加え、ピペットで穏やかにサンプルを混合し、サンプルを72°Cで3分間インキュベートします。インキュベーション後、直ちにサンプルを氷上に置いて、オリゴ-dTをポリAテールにハイブリダイズさせる。
- 逆転写反応混合物(混合物C)(表3)を準備する( 材料表参照)。サンプルを含む各チューブに21.6 μLの混合物Cを追加します。一般的なPCR機器でRTプログラムをオンにします(表4)。
- 氷上でプリアンプ反応を行う。44 μLの2x PCRポリメラーゼミックスと0.88 μLのIS PCRプライマー(10 μM)を組み合わせて混合物Dを調製します(表5)( 材料の表を参照)。40.8 μLの混合物Dを40 μLのRT反応生成物に加え、プリアンププログラムを実行します(表6)。
- Ampure XPビーズを使用してプリアンプ反応生成物を精製します( 材料の表を参照)。ステップ4.7の各サンプルに48 μLのビーズ(0.6:1の比率)を加え、ピペッティングでサンプルを穏やかに混合します。
- サンプルを室温で10分間インキュベートします。サンプルを含む1.5 mLチューブを磁気分離スタンドに5分間置きます。ビーズを乱すことなく、サンプルから上清を慎重に廃棄します。
- ビーズを200 μLの80%エタノールに再懸濁して洗浄し、エタノール含有上清を廃棄する前に、サンプルをさらに3分間磁気分離スタンド( 材料の表を参照)に置きます。
- サンプルを10分間風乾し、空気乾燥中の汚染や相互汚染を防ぐためにチューブを覆います。
- ビーズを20 μLのddH2Oに再懸濁し、サンプルを室温で5分間インキュベートした後、磁気分離スタンドに5分間置きます。
- 各チューブから18 μLの上清をピペットで取り出し、サンプルを新しい1.5 mL遠沈管に移します。1 μLのサンプルを使用して、DNA定量キットを使用してcDNAの品質を評価し、フラグメントアナライザー( 材料の表を参照)を使用して各プレライブラリのサイズ分布を決定し、残りのサンプルを-20°Cで保存します。
- シーケンシングライブラリ調製キットを使用して、ステップ4.13のプレライブラリ製品からIlluminaシーケンシング16用のcDNAライブラリ8を構築します(材料表を参照)。
- Ampure XPビーズを使用してライブラリを精製し(ステップ4.14から)、精製されたライブラリを定量し、ステップ4.13に従って各ライブラリのサイズ分布を決定します。
注:各ライブラリのナノモルを均等にプールし、イルミナインデックスの同じ組み合わせを持たないようにします。それ以外の場合は、ライブラリを目的のシーケンシング出力に基づく比率でプールし、イルミナシーケンサーの同じレーンで一緒にシーケンスすることができます。一般に、各ライブラリを4〜6 GBの深さまでシーケンシングすると、1,000万>のマップリードが得られ、 シロイヌナズナ ゲノムの20倍から30倍のカバレッジが得られます。より低いシーケンシング深度も許容されますが、発現差解析の有意性に影響を与える可能性があります。
5. RNA配列データ解析
- Trim-Galore17を使用して生のリードをトリミングし、続いてhisat2 18(daehwankimlab.github.io/hisat2)を使用して参照ゲノムにマッピングし、Picard19(broadinstitute.github.io/picard)を使用してPCR複製フラグメントを削除します。
- 各試料について少なくとも3つの生物学的反復を用いてDESeq220 を用いて差次発現遺伝子(DEG)の生計数処理およびその後の分析を行う。Pheatmapパッケージを使用して遺伝子発現のクラスタリングを実行し、発現ヒートマップで視覚化します。
注:遺伝子とTE(転移要素)のRPKM(100万マップリードあたりのキロ塩基あたりのリード)値は、Stringtie18 (github.com/gpertea/stringtie)で計算され、ゲノムブラウザで視覚化されました。
結果
プロトプラスト単離
このプロトコルは、蛍光 A.タリアナ 根マーカー株のプロトプラストソーティングに有効です。これらのマーカー株は、蛍光タンパク質と標的細胞型で特異的に発現する遺伝子との融合、またはエンハンサートラップ株の使用によって開発されました(図1)。多数の組織や臓器が、モデル植物や作物で特定の蛍光マーカーを発現する細胞型に解剖されています。
FACSポピュレーション、ソートされたセル、およびライブラリQC
野生植物を対照とし、前方散乱(FSC)や側方散乱(SSC)などのゲートを設定することで、目的の細胞の主要な集団と自家蛍光のベースラインを決定し、蛍光特異的なマーク細胞の選別に成功しました(図2)。最終的なシーケンシングライブラリの品質(ステップ4.15から)は、フラグメントサイズ分布分析によって決定されました。約2,000個の木部極周回細胞、側根原基細胞、内皮/皮質細胞、および側根冠細胞のRNA-seqライブラリの代表的な結果を図3A-Dに示します。
発現パターンの解析
シーケンシングデータの品質は、シーケンシングの深さ、マッピングレート、fastQCレポートなど、複数の解析手順から評価できます。シーケンシングデータの精度と感度は、単離された細胞型と組織全体との間のDEG分析から識別できる一連の細胞型濃縮遺伝子の存在によって実証できます。特定の細胞型において有意に高い発現レベルを有する遺伝子は、細胞型富化遺伝子として同定することができる。一方、各細胞型のゲノムブラウザビューを並べて比較し、既知のマーカー遺伝子の発現レベルを表示し、細胞型発現データでマーカー遺伝子の発現パターンを再構築できるかどうかをテストすることができます。一例として、4つの根細胞タイプのいずれかで富む遺伝子をクラスター化してヒートマップで示し、異なる細胞タイプ間の遺伝子発現の特異性を示しました(図4)。YUCCA3、MYB36、WOX5、およびPFA1を調べたところ、発現パターンは以前の報告21、22、23、24に従って予想通りでした。データ解析パイプラインと代表的な生のシーケンシングデータは、パブリックリポジトリ(github.com/gaolabsjtu/root_cell_types_RNAseq)で入手できます。
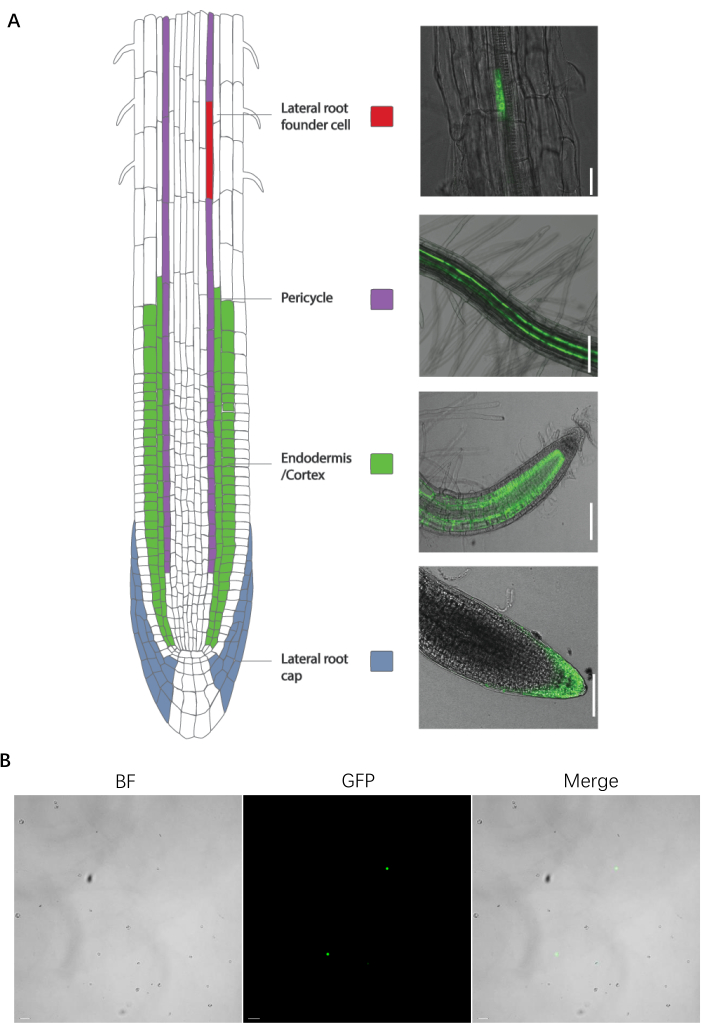
図1:根およびプロトプラスト調製物の特定の細胞型マーカー株 。 (A、左)エンハンサートラップラインの紹介と画像。(A右)根の特定の細胞型マーカーライン。(B)選別前のプロトプラスト調製物の画像。側根創始細胞、周皮、内皮/皮質、側根冠、およびプロトプラストのスケールバーは、それぞれ25 μm、100 μm、100 μm、75 μm、および20 μmを表します。 この図の拡大版を表示するには、ここをクリックしてください。

図2:特定の根細胞タイプに対するFACSの確立。 FACS実験の例:(A)SSCおよびFSCを使用する主要な集団;(b)GFP陽性(+)およびGFP陰性(−)ゲート;(C)明視野および(D)選別された細胞の蛍光画像。スケールバーは50μmを表します。 この図の拡大版を表示するには、ここをクリックしてください。
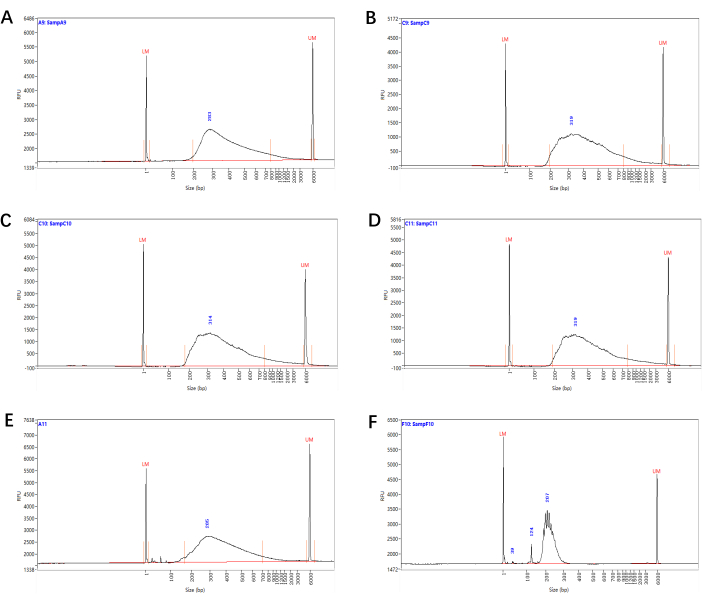
図3:Smart-seq2シーケンシングライブラリのサイズ分布。 木部極周回細胞(A)、側根原基細胞(B)、内皮/皮質細胞(C)および側根冠細胞(D)から生成されたシーケンシングライブラリの代表的なフラグメントサイズ分布(ステップ4.15から)。(E)プライマー/アダプターダイマーピークを持つ小型ライブラリで、サイズ選択後も配列決定できます。(F) ライブラリーの準備に失敗したことを示す異常なサイズ分布を持つライブラリー。 この図の拡大版を表示するには、ここをクリックしてください。

図4:細胞型濃縮遺伝子解析。 DEG分析は、各細胞型と根全体の間で実施されました。各細胞型で4倍以上のアップレギュレーションを有する遺伝子を細胞型富化遺伝子として同定した。これらの遺伝子を組み合わせ、クラスター化し、ヒートマップ(上のパネル)プロットで視覚化しました。細胞型が豊富な遺伝子には、多くの既知のマーカー遺伝子が含まれていました。 YUCCA3、 MYB36、 WOX5、 PFA1 などの典型的なマーカー遺伝子をゲノムブラウザに表示するように選択しました(下のパネル)。略語:LRP =側根原基;エンド/コー=内皮/皮質;LRC =ラテラルルートキャップ。 この図の拡大版を表示するには、ここをクリックしてください。
| コンポーネント | 容量(μL) |
| 10% トリトン X-100 | 0.33 |
| RNase阻害剤 | 0.55 |
| DTT (0.1 M) | 0.22 |
表1:細胞溶解緩衝液(混合物A)を調製するための反応成分。
| コンポーネント | 容量(μL) |
| オリゴdT30VN (100 μM) | 0.44 |
| dNTP (10 mM) | 4.4 |
表2:混合物Bを調製するための反応成分。
| コンポーネント | 容量(μL) |
| 上付き文字 IV バッファ (5x) | 8.8 |
| ベタイン (5 M) | 8.8 |
| DTT (0.1 M) | 2.2 |
| マグネシウムCl2 (1 M) | 0.264 |
| TSO (100 μM) | 0.44 |
| スーパースクリプトIV逆転写酵素(200 U/μL) | 2.2 |
| RNase阻害剤 | 1.1 |
表3:逆転写PCR混合物(混合物C)を調製するための反応成分。
| サイクル | 気温(°C) | 時間 |
| 1 | 50 | 90分 |
| 2-11 | 55 | 2 ミン |
| 50 | 2 ミン | |
| 12 | 70 | 15分 |
| 13 | 4 | ∞ |
表4:mRNAからcDNAを合成するための逆転写(RT)PCR設定。
| コンポーネント | 容量(μL) |
| KAPA HiFi HotStart ReadyMix (2x) | 44 |
| IS PCRプライマー (10 μM) | 0.88 |
表5:プレアンプ反応混合物(混合物D)を調製するための反応成分。
| サイクル | 気温(°C) | 時間 |
| 1 | 98 | 5 ミン |
| 2-13 | 98 | 20秒 |
| 67 | 30秒 | |
| 72 | 3 ミン | |
| 14 | 72 | 5 ミン |
| 15 | 4 | ∞ |
表6:増幅前反応のPCRプログラム設定。
ディスカッション
Smart-seq2ベースのプロトコルは、数百のセルから信頼性の高いシーケンシングライブラリを生成できます8。出発物質の品質は、トランスクリプトーム解析の精度に不可欠です。FACSは目的の細胞を調製するための強力なツールですが、この手順、特にプロトプラストステップは、植物用途に最適化する必要があります。レーザーキャプチャーマイクロダイセクション(LCM)または手動解剖細胞も入力として使用することができるため、ここで提供されるプロトコルは、既知のマーカー遺伝子の有無にかかわらず、さまざまな植物種および細胞タイプで使用できる可能性があります。
植物材料の調製
植物発生生物学において、FACSは通常、蛍光マーカーを発現する濃縮細胞集団を単離するために使用されます。このようなマーカーを発現する植物はプロトプラストされ、プロトプラストは最終的に細胞型特異的マーカーの発現に基づいて純粋な亜集団に分類されます。したがって、蛍光マーカーのシグナルは強く特異的でなければなりません。さらに、研究者はどのマーカーをソートできるかを事前に確認する必要があります。必要なシードの数は、発現レベル、マーカーが発現される場所、ダウンストリームアプリケーションに必要な細胞数、およびソートされる反復の数によって異なります。発現が植物当たりの細胞数が非常に少ない単一細胞型である場合、一般に、マーカーがより多くの細胞で発現される場合よりも多くの植物が必要である。個々のマーカーラインに必要なシードの数を経験的に決定するのが最善です。各マーカーラインで予備実験を実行して、ソートのウィンドウを最適化し、固定数の植物から生じる細胞の数を知ることは特に意味があります。実験材料が根である場合、根が寒天に沈むのを防ぎ、きれいな根の切断を容易にするために、シードプレーティングの前に、適切なサイズのオートクレーブメッシュを培地含有寒天に置くことができます。低温成層は種子の発芽と発育を助けるので、殺菌またはメッキした後、種子を4°Cで2日間層別化し、植物を垂直に成長させることをお勧めします。
プロトプラストとFACS
層別化の5〜6日後にプロトプラストと選別がうまくいきます。溶液Aと溶液Bは、0.22 μmのストレーナーを使用してろ過する必要があります。さらに、溶液Bを-20°Cで長期間保存すると、酵素の効率が低下します。開始する前に、溶液Aと溶液Bを氷上で穏やかに解凍する必要がありますが、これには約20分かかります。 溶液Bを振ると酵素が破壊され、過剰な気泡形成を引き起こす可能性があるため、溶液Bを振とうしないでください。ストレーナーメッシュを溶液Aで複数回洗浄すると、ステップ2.5で得られるセルの数を増やすことができます。ステップ2.6で説明したように、プロトプラストはペレットの近くの底部にあり、見ることができないため、上清全体を吸引しないでください。選別する前に、より良い選別効率を達成するために、プロトプラストの濃度が約10 5-10 6細胞/ mLであることを確認するのが最善です。新しい実験を設定する場合、WT植物からの同じ組織が、選別ゲートを設定するための対照として必要とされる。ステップ3.1では、最初に蛍光チャネル(PEやFITCなど)を選択し、コントロールサンプルを使用してSSCとFSCに従って主要な集団を決定することをお勧めします。さらに、制御信号がゲートの左側に位置することを確実にするために蛍光チャネルの電圧を変化させることによってゲートの位置を調整することが提案される(すなわち、負の基が103より下に位置する)。選別時間は、プロトプラストまたは選別によって起こり得る遺伝子発現の変化を防ぐために、30分または60分未満に制限されなければならない。選別後、少数の選別された細胞を収集し、蛍光をチェックする必要があります(ステップ3.4)。下流のライブラリ構築への影響を避けるために、上清からできるだけ多くのバッファーを除去する必要があります(ステップ3.3)。
RNA-seqライブラリの構築
RNA-seqライブラリの構築には、RNAの分解を抑えるために、ソーティング直後の細胞を使用することをお勧めします。あまりにも多くの細胞から始めることは、不十分な反応のために低品質のライブラリをもたらす可能性があるため、お勧めできません。ステップ4.7およびステップ4.14のPCRサイクル数は、RNA/cDNAの入力量によって異なります。サイクル数は、入力が少ない場合は増やし、入力が多い場合は下げることができます。したがって、ステップ4.7とステップ4.14の混合サンプルの一部を取得して、同じ増幅手順でリアルタイムPCRを実行し、プレライブラリ/ライブラリの正式な増幅の前にqPCRの結果に基づいて最終的なサイクル数を決定することをお勧めします。さらに、精製ステップ4.8の前に、Ampure XPビーズを室温で少なくとも10分間平衡化してから、よくボルテックスする必要があります。精製ステップでのビーズの量は、0.8:1の比率を超えて増やさないでください。そうしないと、プライマーダイマーのキャリーオーバーが増加します。さらに、困難な再懸濁を防ぐために、ステップ4.11でビーズを過度に乾燥させないようにする必要があります。修飾されたプレライブラリは、平均サイズが約1.5〜2 kbで、少量の短い(<500 bp)フラグメントが必要です。ライブラリ内の異常なサイズ分布または小さなサイズのプライマー/アダプターダイマーピークは低品質の指標であり、これらのサンプルは廃棄するか、ビーズ精製をさらにラウンドする必要があります(ステップ4.15)(図3E-F)。
制限
このプロトコルは、他の植物組織および他の植物種から細胞を単離するために適用することができる。しかしながら、細胞単離プロセスはプロトプラストの調製に大きく依存する。血管細胞や女性の性細胞など、組織の内部に位置していたり、数が少ない細胞型もあります。プロトプラストの調製が困難な細胞に対しては、蛍光活性化核選別(FANS)27 が任意の方法として使用できる。一方、このプロトコルを使用してFACSによって特定の細胞タイプを取得することは、蛍光マーカー株の利用可能性に依存します。そのようなマーカー線の欠如は、作物および園芸植物におけるこれらの方法の使用を制限する。ハイスループットシングルセルRNA-seq技術を作物に適用することで、新しい細胞型特異的マーカー遺伝子が明らかになり、細胞型マーカー株の開発にさらに使用でき、FACSベースの細胞型RNA-seqおよびマルチオミクス研究の適用能力が広がります。
開示事項
著者は開示するものは何もありません。
謝辞
このプロトコルは、上海交通大学農生物学部のシングルセルマルチオミクス施設で設定され、中国国家自然科学財団(助成金番号32070608)、上海浦江プログラム(助成金番号20PJ1405800)、上海交通大学(助成金番号Agri-X20200202、2019TPB05)の支援を受けました。
資料
| Name | Company | Catalog Number | Comments |
| 0.22 µm strainer | Sorfa | 622110 | |
| Agar | Yeasen | 70101ES76 | |
| Agilent fragment analyzer | Aglient | Aglient 5200 | |
| Agilent high-sensitivity DNA kit | Aglient | DNF-474-0500 | |
| Ampure XP beads | BECKMAN | A63881 | |
| Betaine | yuanye | S18046-100g | |
| Bleach | Mr Muscle | FnBn83BK | 20% (v/v) bleach |
| BSA | sigma | 9048-46-8 | |
| CaCl2 | yuanye | S24109-500g | |
| Cellulase R10 | Yakult (Japan) | 9012-54-8 | |
| Cellulase RS | Yakult (Japan) | 9012-54-8 | |
| Centrifuge tube (1.5 mL) | Eppendolf | 30121589 | |
| DNase, RNase, DNA and RNA Away Surface Decontaminants | Beyotime | R0127 | |
| dNTPs (10 mM) | NEB | N0447S | |
| DTT (0.1 M) | invitrogen | 18090050 | |
| Ethanol | Sinopharm Chemical Reagent Co., Ltd | 100092680 | |
| FACS | BD FACS Melody | BD-65745 | |
| FACS | Sony | SH800S | |
| Filter tip (1000 µL) | Thermo Scientific | TF112-1000-Q | |
| Filter tip (200 µL) | Thermo Scientific | TF140-200-Q | |
| Filter tip (10 µL) | Thermo Scientific | TF104-10-Q | |
| Filter tip (100 µL) | Thermo Scientific | TF113-100-Q | |
| Fluorescent microscope | Nikon | Eclipse Ni-E | |
| Four-Dimensional Rotating Mixer | Kylin -Bell | BE-1100 | |
| Hemicellulase | sigma | 9025-56-3 | |
| IS PCR primer | 5'-AAGCAGTGGTATCAACGCAGAG T-3' | ||
| KAPA HiFi HotStart ReadyMix(2X) | Roche | 7958935001 | |
| KCl | Sinopharm Chemical Reagent Co., Ltd | 7447-40-7 | |
| Macerozyme R10 | Yakult (Japan) | 9032-75-1 | |
| Magnetic separation stand | invitrogen | 12321D | |
| Mannitol | aladdin | 69-65-8 | |
| MES | aladdin | 145224948 | |
| MgCl2 | yuanye | R21455-500ml | |
| Microcentrifuges | Eppendorf | Centrifuge 5425 | |
| Micro-mini-centrifuge | Titan | Timi-10k | |
| MS | Phytotech | M519 | |
| Nextera XT DNA Library Preparation Kit | illumina | FC-131-1024 | |
| oligo-dT30VN primer | 5'-AAGCAGTGGTATCAACGCAGAG TACTTTTTTTTTTTTTTTTTTTTTTT TTTTTTTTTTVN-3' | ||
| PCR instrument | Thermal cycler | A24811 | |
| Pectolyase | Yakult (Japan) | 9033-35-6 | |
| Plant marker lines | Nottingham Arabidopsis Stock Centre (NASC) | ||
| Qubit 1x dsDNA HS Assay Kit | invitrogen | Q33231 | |
| Qubit 2.0 fluorometer | invitrogen | Q32866 | |
| RNase inhibitor | Thermo Scientific | EO0382 | |
| RNase-free water | invitrogen | 10977023 | |
| Solution A | 400 mM mannitol, 0.05 % BSA , 20 mM MES (pH5.7), 10 mM CaCl2, 20 mM KCl | ||
| Solution B | 1 % (w/v)cellulase R10, 1 % (w/v) cellulase RS, 1 % (w/v)hemicellulase, 0.5 % (w/v)pectolyase and 1 % (w/v) Macerozyme R10 in a fresh aliquot of solution A | ||
| Sterile pestle | BIOTREAT | 453463 | |
| Strainer (40 µm ) | Sorfa | 251100 | |
| SuperScript IV reverse transcriptase (200 U/µL) | invitrogen | 18090050 | |
| SuperScript IV buffer (5x) | invitrogen | 18090050 | |
| Test tube (5 mL) | BD Falcon | 352052 | |
| Thin-walled PCR tubes with caps (0.5 mL) | AXYGEN | PCR-05-C | |
| Triton X-100 | Sangon Biotech | A600198-0500 | |
| TSO primer | 5'-AAGCAGTGGTATCAACGCAGAG TACATrGrG+G-3' | ||
| Vortex | Titan | VM-T2 |
参考文献
- Zhang, T. -Q., Chen, Y., Liu, Y., Lin, W. -H., Wang, J. -W. Single-cell transcriptome atlas and chromatin accessibility landscape reveal differentiation trajectories in the rice root. Nature Communications. 12 (1), 2053(2021).
- Denyer, T., et al. Spatiotemporal developmental trajectories in the Arabidopsis root revealed using high-throughput single-cell RNA sequencing. Developmental Cell. 48 (6), 840-852 (2019).
- Liu, Q., et al. Transcriptional landscape of rice roots at the single-cell resolution. Molecular Plant. 14 (3), 384-394 (2021).
- Liu, Z., et al. Global dynamic molecular profiling of stomatal lineage cell development by single-cell RNA sequencing. Molecular Plant. 13 (8), 1178-1193 (2020).
- Shahan, R., et al. A single-cell Arabidopsis root atlas reveals developmental trajectories in wild-type and cell identity mutants. Developmental Cell. 57 (4), 543-560 (2022).
- Wendrich, J. R., et al. Vascular transcription factors guide plant epidermal responses to limiting phosphate conditions. Science. 370 (6518), (2020).
- Carter, A. D., Bonyadi, R., Gifford, M. L. The use of fluorescence-activated cell sorting in studying plant development and environmental responses. The International Journal of Developmental Biology. 57 (6-8), 545-552 (2013).
- Picelli, S., et al. Full-length RNA-seq from single cells using Smart-seq2. Nature Protocols. 9 (1), 171-181 (2014).
- Wang, X., He, Y., Zhang, Q., Ren, X., Zhang, Z. Direct comparative analyses of 10X genomics chromium and Smart-seq2. Genomics Proteomics Bioinformatics. 19 (2), 253-266 (2021).
- Serrano-Ron, L., et al. Reconstruction of lateral root formation through single-cell RNAsequencing reveals order of tissue initiation. Molecular Plant. 14 (8), 1362-1378 (2021).
- Hashimshony, T., et al. CEL-Seq2: Sensitive highly-multiplexed single-cell RNA-Seq. Genome Biology. 17, 77(2016).
- Macaulay, I. C., et al. G&T-seq: Parallel sequencing of single-cell genomes and transcriptomes. Nature Methods. 12 (6), 519-522 (2015).
- Angermueller, C., et al. Parallel single-cell sequencing links transcriptional and epigenetic heterogeneity. Nature Methods. 13 (3), 229-232 (2016).
- De Rybel, B., et al. A novel aux/IAA28 signaling cascade activates GATA23-dependent specification of lateral root founder cell identity. Current Biology. 20 (19), 1697-1706 (2010).
- Duncombe, S. G., Barnes, W. J., Anderson, C. T. Imaging the delivery and behavior of cellulose synthases in Arabidopsis thaliana using confocal microscopy. Methods in Cell Biology. 160, 201-213 (2020).
- Levy, S. E., Myers, R. M. Advancements in next-generation sequencing. Annual Review of Genomics and Human Genetics. 17 (1), 95-115 (2016).
- Ooi, C. C., et al. High-throughput full-length single-cell mRNA-seq of rare cells. PLoS One. 12 (11), e0188510(2017).
- Pertea, M., Kim, D., Pertea, G. M., Leek, J. T., Salzberg, S. L. Transcript-level expression analysis of RNA-seq experiments with HISAT, StringTie and Ballgown. Nature Protocols. 11 (9), 1650-1667 (2016).
- Tsyganov, K., Perry, A., Archer, S., Powell, D. RNAsik: A Pipeline for complete and reproducible RNA-seq analysis that runs anywhere with speed and ease. Journal of Open Source Software. 3, 583(2018).
- Love, M. I., Huber, W., Anders, S. Moderated estimation of fold change and dispersion for RNA-seq data with DESeq2. Genome Biology. 15 (12), 550(2014).
- Kamiya, T., et al. The MYB36 transcription factor orchestrates Casparian strip formation. Proceedings of the National Academy of Sciences of the United States of America. 112 (33), 10533-10538 (2015).
- Zhang, Y., et al. Two types of bHLH transcription factor determine the competence of the pericycle for lateral root initiation. Nature Plants. 7 (5), 633-643 (2021).
- Haecker, A., et al. Expression dynamics of WOX genes mark cell fate decisions during early embryonic patterning in Arabidopsis thaliana. Development. 131 (3), 657-668 (2004).
- Chen, Q., et al. Auxin overproduction in shoots cannot rescue auxin deficiencies in Arabidopsis roots. Plant Cell Physiol. 55 (6), 1072-1079 (2014).
- Nichterwitz, S., et al. Laser capture microscopy coupled with Smart-seq2 for precise spatial transcriptomic profiling. Nature Communications. 7, 12139(2016).
- Long, J., et al. Nurse cell--derived small RNAs define paternal epigenetic inheritance in Arabidopsis. Science. 373 (6550), (2021).
- Gutzat, R., et al. Arabidopsis shoot stem cells display dynamic transcription and DNA methylation patterns. EMBO Journal. 39 (20), e103667(2020).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved