Method Article
Выделение и транскриптомный анализ типов растительных клеток
В этой статье
Резюме
Осуществимость и эффективность высокопроизводительных методов секвенирования скРНК предвещают эру одноклеточных исследований растений. Здесь представлена надежная и полная процедура выделения конкретных типов клеток корня Arabidopsis thaliana и последующего построения и анализа библиотеки транскриптома.
Аннотация
В многоклеточных организмах программирование развития и реакции окружающей среды могут сильно различаться в разных типах клеток или даже внутри клеток, что известно как клеточная гетерогенность. В последние годы выделение отдельных клеток и клеток в сочетании с методами секвенирования следующего поколения (NGS) стали важными инструментами для изучения биологических процессов при разрешении в одной клетке. Однако выделение растительных клеток относительно сложнее из-за наличия клеточных стенок растений, что ограничивает применение одноклеточных подходов у растений. Этот протокол описывает надежную процедуру выделения одноклеточных и клеточных клеток на основе флуоресцентно-активированной сортировки (FACS) с растительными клетками, которая подходит для последующего мультиомиксного анализа и других исследований. Используя флуоресцентные маркерные линии корня арабидопсиса , мы демонстрируем, как выделяются определенные типы клеток, такие как клетки перицикла ксилемы, начальные клетки латерального корня, клетки боковой крышки корня, клетки коры и энтодермальные клетки. Кроме того, предусмотрен эффективный последующий метод анализа транскриптома с использованием Smart-seq2. Метод выделения клеток и методы анализа транскриптома могут быть адаптированы к другим типам клеток и видам растений и имеют широкий потенциал применения в науке о растениях.
Введение
Клетки являются основной единицей всех живых организмов и выполняют структурные и физиологические функции. Хотя клетки в многоклеточных организмах демонстрируют кажущуюся синхронность, клетки разных типов и отдельные клетки демонстрируют различия в своих транскриптомах во время развития и реакций окружающей среды. Высокопроизводительное секвенирование одноклеточной РНК (scRNA-seq) обеспечивает беспрецедентную мощность для понимания клеточной гетерогенности. Применение scRNA-seq в науках о растениях способствовало успешному построению атласарастительных клеток 1, использовалось для идентификации редких клеточных таксонов в тканях растений2, дало представление о составе типов клеток в тканях растений и использовалось для идентификации клеточной идентичности и важных функций, используемых во время развития и дифференцировки растений. Кроме того, можно вывести пространственно-временные траектории развития в тканях растений 1,2,3, открыть новые гены-маркеры 4 и изучить функции важных факторов транскрипции5 с помощью scRNA-seq, чтобы выявить эволюционное сохранение одного и того же типа клеток у разных растений 3. Абиотические стрессы являются одними из наиболее важных факторов воздействия окружающей среды на рост и развитие растений. Исследуя изменения в составе типов клеток в тканях растений в различных условиях лечения с помощью секвенирования одноклеточного транскриптома, можно также разрешить механизм абиотической реакциина стресс 6.
Потенциал разрешения транскрипционной гетерогенности между типами клеток с использованием секвенирования скРНК зависит от метода выделения клеток и платформы секвенирования. Сортировка клеток, активируемая флуоресценцией (FACS), является широко используемым методом выделения субпопуляции клеток для scRNA-seq на основе рассеяния света и флуоресцентных свойств клеток. Разработка флуоресцентных маркерных линий с помощью трансгенной технологии значительно повысила эффективность выделения клеток с помощью FACS7. Проведение секвенирования скРНК с использованием Smart-seq28 еще больше усиливает способность препарировать клеточную гетерогенность. Метод Smart-seq2 обладает хорошей чувствительностью для детекции генов и может обнаруживать гены даже при низком входе транскрипта9. В дополнение к массовому сбору типов клеток, современные сортировщики клеток предоставляют формат сортировки индекса одной ячейки, что позволяет проводить анализ транскриптома с разрешением одной клетки с использованием Smart-seq210 или других мультиплексных методов секвенирования РНК, таких как CEL-seq211. Сортировка по одной ячейке или по ячейке потенциально может быть использована для многих других последующих приложений, таких как параллельные мультиомические исследования12,13. Здесь представлен надежный и универсальный протокол для выделения типов растительных клеток, таких как клетки перицикла ксилемы, клетки боковой корневой крышки, начальные клетки бокового корня, клетки коры и энтодермальные клетки из корней маркерных клеточных линий Arabidopsis thaliana с помощью FACS. Кроме того, протокол включает в себя создание библиотеки Smart-seq2 для последующего анализа транскриптома.
протокол
Следующий протокол был оптимизирован для семян дикого типа (WT) A. thaliana без флуоресцентных и флуоресцентных маркерных линий для следующих типов корневых клеток: клетки перицикла ксилемы-полюса (J0121), начальные клетки бокового корня, клетки боковой корневой крышки (J3411), клетки энтодермы и коры (J0571) (рис. 1A). Все маркерные линии были получены из коммерческого источника (см. Таблицу материалов), за исключением маркерной линии клеток инициации бокового корня, которая была получена путем введения конструкции GFP, управляемой промотором GATA23, в растение Arabidopsis дикого типа в соответствии с ранее опубликованным отчетом14.
1. Подготовка растительного сырья
- Стерилизуют семена A. thaliana WT и семена флуоресцентной маркерной линии, инкубируя семена в 20% отбеливателе во вращающемся инкубаторе при комнатной температуре в течение 15 минут.
- Промойте семена в воде двойной дистилляции (ddH2O) от трех до пяти раз. Выполните этот шаг на стерильной чистой скамье.
- Нанесите семена WT и репортерной линии на среднюю средность Murashige и Skoog (MS) половинной крепости с 0,8% агара (мас./об.)15. Выращивайте растения вертикально в течение 5 дней (16 часов света при 23 ° C) после стратификации в течение 2 дней при 4 ° C.
2. Протопластинг
- Приготовьте протопластные растворы2,5, называемые раствором А и раствором В (см. Таблицу материалов).
- Приготовьте раствор А, содержащий 400 мМ маннита, 0,05% BSA, 20 мМ MES (рН 5,7), 10 мМ CaCl2 и 20 мМ KCl (см. Таблицу материалов). Храните раствор А при температуре −20 °C до 1 месяца.
- Приготовьте раствор B, добавив 1% (мас./об.) целлюлазы R10, 1% (мас./об.) целлюлазы RS, 1% (мас./об.) гемицеллюлазы, 0,5% (мас./об.) пектолиазы и 1% (мас./об.) мацероцима R10 в свежей аликвоте раствора А. Храните раствор B при -20 °C до 1 месяца.
- Аккуратно разморозьте раствор А и раствор Б на льду перед началом эксперимента.
- Срежьте корни чистым лезвием или ножницами и измельчите корни на кусочки ~0,5 см. Погрузите корни в 1,5 мл раствора B с последующим осторожным вращением (примерно при 18 об/мин) при комнатной температуре в течение 1,5-2 часов.
- Отфильтруйте корневые протопласты через сетчатый фильтр 40 мкм (см. Таблицу материалов).
- Промойте сетку сетчатого фильтра 1-2 мл раствора А.
- Смешайте жидкости на шагах 2.4 и 2.5 и центрифугируйте при 300 x g в течение 5 мин при 4 °C. Выбросьте надосадочную жидкость с помощью пипетки, ресуспендируйте клеточную гранулу в 500-600 мкл раствора А, а затем немедленно поместите ее на лед.
- Перенесите ресуспендированный клеточный раствор в новую пробирку объемом 5 мл для сортировки клеток.
3. Сортировка клеток, активируемая флуоресценцией (FACS)
- Включите и завершите шаги настройки прибора на сортировщике (см. Таблицу материалов). Выберите флуоресцентные каналы, используйте установку WT (без флуоресценции) в качестве элемента управления, чтобы определить базовую линию для автофлуоресценции, и отрегулируйте сортировочный затвор в зависимости от интенсивности флуоресценции и синглетов FSC/SSC (рис. 2).
- Добавьте 500 мкл раствора А (этап 2.1.1) в пробирку для сбора объемом 1,5 мл, чтобы предотвратить повреждение клеток. Соберите 2 000-3 000 клеток в пробирке.
- После сортировки немедленно поместите образцы на лед, центрифугируйте сборную пробирку, содержащую клетки при 300 x g , в течение 5 мин при 4 ° C и удалите надосадочную жидкость пипеткой.
- Возьмите 2 мкл отсортированных клеток и проверьте наличие флуоресценции с помощью флуоресцентного микроскопа (см. Таблицу материалов).
- Храните отсортированные ячейки при температуре −80 °C или сразу же используйте их для создания библиотеки (шаг 4).
- Для сортировки индекса по одной ячейке поместите пластину с 96 лунками в адаптер. Откалибруйте положение пластины так, чтобы капля попадала в центральное отверстие пластины. Выберите режим сортировки по одной ячейке при сортировке, введите целевое количество отсортированных ячеек равным 1 и начните сортировку.
4. Подготовка библиотеки Smart-seq2
- В результате сверхмалого количества входных данных выполните построение библиотеки РНК-секвенирования одноклеточного типа в среде, свободной от загрязнения. Перед началом эксперимента очистите стенд поверхностным обеззараживающим средством8 (см. Таблицу материалов) и 75% этанолом.
- Приготовьте буфер для лизиса (смесь А) (таблица 1), объединив 0,33 мкл 10% Triton X-100, 0,55 мкл ингибитора РНКазы и 0,22 мкл 0,1 М DTT (см. Таблицу материалов).
- Добавьте 1 мкл смеси А в отсортированный образец и измельчите стерильным пестиком. Предпочтительный объем образца составляет ≤0,5 мкл; используйте воду, не содержащую РНКазы, чтобы восполнить объем до 14 мкл. Перенесите каждый образец клетки в тонкостенную пробирку для ПЦР объемом 0,2 мл.
- Приготовьте смесь B, содержащую 0,44 мкл реакционного праймера с обратной транскрипцией (RT) oligo-dT 30 VN (100 мкМ) и 4,4 мкл dNTP (10мМ) (таблица 2) (см. Таблицу материалов).
- Добавьте 4,4 мкл смеси B к 14 мкл образца в каждой пробирке, аккуратно перемешайте образец пипеткой и инкубируйте образец при 72 ° C в течение 3 минут. После инкубации немедленно поместите образцы на лед, чтобы гибридизировать олиго-dT с поли-А-хвостом.
- Приготовьте реакционную смесь обратной транскрипции (смесь С) (табл. 3) (см. Таблицу материалов). Добавьте 21,6 мкл смеси С в каждую пробирку, содержащую образцы. Включите программу ЛТ на обычном ПЦР-приборе (табл. 4).
- Проведите реакцию предварительного усиления на льду. Приготовьте смесь D, соединив 44 мкл смеси 2x ПЦР-полимеразы и 0,88 мкл праймера IS PCR (10 мкМ) (таблица 5) (см. Таблицу материалов). Добавьте 40,8 мкл смеси D к 40 мкл продукта реакции RT и запустите программу предварительного усиления (таблица 6).
- Очистите продукты реакции предварительного усиления с помощью гранул Ampure XP (см. Таблицу материалов). Добавьте 48 мкл шариков (соотношение 0,6:1) в каждый образец с шага 4.7 и аккуратно перемешайте образцы с помощью пипетки.
- Инкубируют образцы при комнатной температуре в течение 10 мин. Поместите пробирки объемом 1,5 мл с образцами на стенд для магнитной сепарации на 5 минут. Аккуратно сбрасывайте надосадочную жидкость с образцов, не повреждая бусины.
- Промойте шарики, ресуспендировав их в 200 мкл 80% этанола, и поместите образцы на стенд для магнитной сепарации (см. Таблицу материалов) еще на 3 минуты, прежде чем выбросить надосадочную жидкость, содержащую этанол.
- Высушите образцы на воздухе в течение 10 минут и накройте пробирку, чтобы предотвратить загрязнение и перекрестное загрязнение во время сушки на воздухе.
- Ресуспендируют шарики в 20 мкл ddH2O, инкубируют образцы при комнатной температуре в течение 5 мин, а затем помещают их на стенд для магнитной сепарации на 5 мин.
- Пипеткой вынимают 18 мкл надосадочной жидкости из каждой пробирки и переносят образцы в новые центрифужные пробирки объемом 1,5 мл. Используйте 1 мкл образца для оценки качества кДНК с помощью набора для количественного определения ДНК, определите распределение по размерам каждой предварительной библиотеки с помощью анализатора фрагментов (см. Таблицу материалов) и храните оставшийся образец при -20 ° C.
- Создайте библиотекукДНК 8 для секвенированияIllumina 16 из предбиблиотечного продукта шага 4.13, используя набор для подготовки библиотеки секвенирования (см. Таблицу материалов).
- Очистите библиотеки (из шага 4.14) с помощью шариков Ampure XP, количественно определите очищенные библиотеки и определите распределение размеров каждой библиотеки после шага 4.13.
ПРИМЕЧАНИЕ: Объедините равные наномоли каждой библиотеки, убедившись, что ни один из них не имеет одинаковой комбинации индекса Illumina. В противном случае библиотеки могут быть объединены в соотношение, основанное на желаемом выходе секвенирования, и секвенированы вместе на одной полосе секвенсора Illumina. Как правило, секвенирование каждой библиотеки на глубину 4-6 ГБ, что дает >10 миллионов картированных чтений, обеспечивает 20-30-кратный охват генома Arabidopsis . Меньшая глубина секвенирования также приемлема, но может повлиять на значимость анализа дифференциального выражения.
5. Анализ данных RNA-seq
- Обрежьте необработанные чтения с помощью Trim-Galore17 с последующим сопоставлением с эталонным геномом с помощью hisat218 (daehwankimlab.github.io/hisat2) и удалите дублированные фрагменты ПЦР с помощью Picard19 (broadinstitute.github.io/picard).
- Проведите обработку необработанного подсчета и последующий анализ дифференциально экспрессируемых генов (DEG) с помощью DESeq220 , используя по крайней мере три биологические реплики для каждого образца. Выполните кластеризацию экспрессии генов с помощью пакета Pheatmap и визуализируйте тепловую карту экспрессии.
ПРИМЕЧАНИЕ: Значения RPKM (чтений на килобазу на миллион картированных чтений) генов и TE (переносимых элементов) были рассчитаны с помощью Stringtie18 (github.com/gpertea/stringtie) и визуализированы в браузере генома.
Результаты
Изоляция протопласта
Этот протокол эффективен для сортировки протопластов флуоресцентных линий корневых маркеров A. thaliana. Эти маркерные линии были разработаны путем слияния флуоресцентных белков с генами, экспрессируемыми специфически в типах клеток-мишеней, или с использованием линий-ловушек энхансеров (рис. 1). Многочисленные ткани и органы были расчленены на типы клеток, экспрессирующие специфические флуоресцентные маркеры в модельных растениях и сельскохозяйственных культурах.
Заполнение FACS, отсортированные ячейки и контроль качества библиотеки
Используя растение дикого типа в качестве контрольных и установочных ворот, таких как прямое рассеяние (FSC) и боковое рассеяние (SSC), мы определили основную популяцию интересующих клеток и базовый уровень для автофлуоресценции и успешно отсортировали флуоресцентно-специфические маркированные клетки (рис. 2). Качество библиотек окончательного секвенирования (начиная с шага 4.15) определяли методом анализа распределения фрагментов по размерам. Репрезентативные результаты библиотеки RNA-seq около 2,000 клеток перицикла ксилемы, клеток зачатков боковых корней, клеток эндодермы/коры головного мозга и клеток боковой крышки корня показаны на рисунке 3A-D.
Анализ паттернов экспрессии
Качество данных секвенирования можно оценить с помощью нескольких процедур анализа, таких как глубина секвенирования, скорость сопоставления и отчеты fastQC. Точность и чувствительность данных секвенирования может быть продемонстрирована наличием ряда генов, обогащенных клеточным типом, которые могут быть идентифицированы с помощью анализа DEG между изолированными типами клеток и всей тканью. Гены со значительно более высокими уровнями экспрессии в определенных типах клеток могут быть идентифицированы как гены, обогащенные клеточным типом. Между тем, представления браузера генома каждого типа клеток можно сравнивать бок о бок, чтобы показать уровни экспрессии известных маркерных генов и проверить, можно ли реконструировать паттерн экспрессии маркерных генов в данных экспрессии клеточного типа. Например, гены, обогащенные в любом из четырех типов корневых клеток, были сгруппированы и показаны на тепловой карте, которая показала специфичность экспрессии генов среди различных типов клеток (рис. 4). Были исследованы YUCCA3, MYB36, WOX5 и PFA1, и паттерны экспрессии соответствовали ожиданиям в соответствии с предыдущими отчетами21,22,23,24. Конвейеры анализа данных и репрезентативные необработанные данные секвенирования доступны в общедоступном репозитории (github.com/gaolabsjtu/root_cell_types_RNAseq).
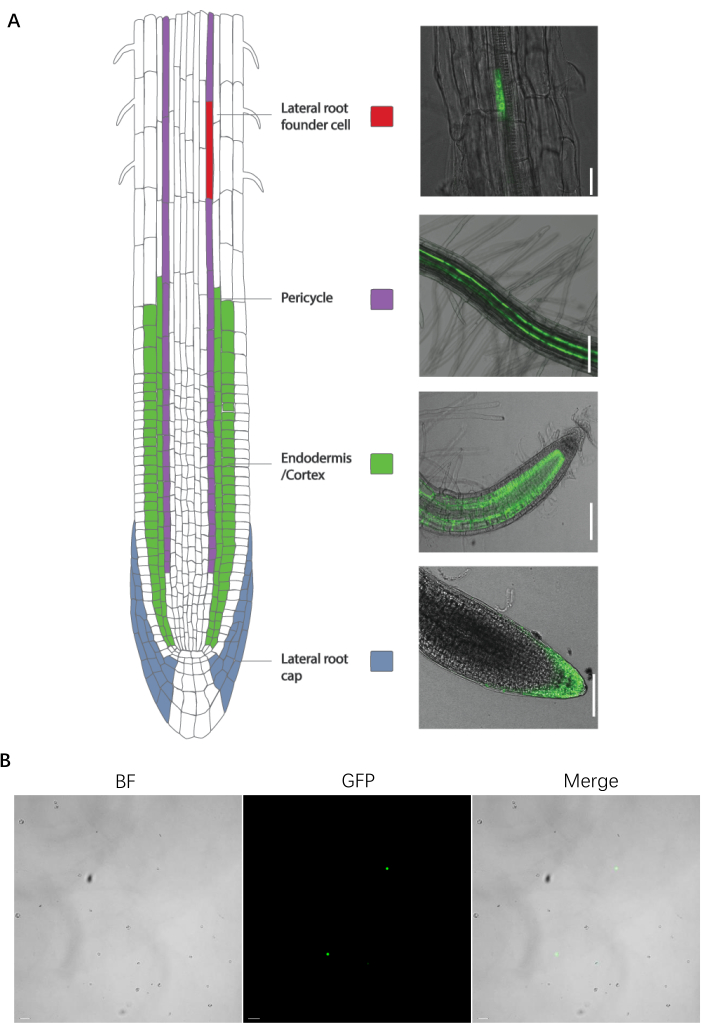
Рисунок 1: Маркерная линия конкретного типа клеток препарата корня и протопласта . (А, слева) Введение линии ловушки Enhancer и изображения. (А, справа) Маркерная линия конкретного типа ячейки корня. (B) Изображение препарата протопласта перед сортировкой. Масштабные линейки для клетки-основателя латерального корня, перицикла, эндодермы/коры, боковой крышки корня и протопластов представляют 25 мкм, 100 мкм, 100 мкм, 75 мкм и 20 мкм соответственно. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.

Рисунок 2: Создание FACS для конкретных типов корневых клеток. Пример эксперимента СУИМ: а) основная популяция, использующая КСК и КФС; (B) GFP-положительные (+) и GFP-отрицательные (-) вентили; (C) светлое поле и (D) флуоресцентные изображения отсортированных клеток. Масштабная линейка представляет 50 мкм. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
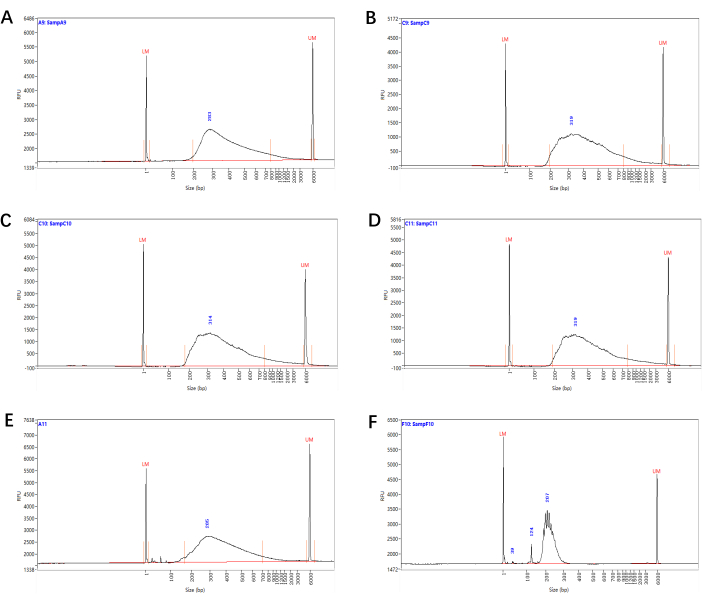
Рисунок 3: Распределение по размерам библиотек секвенирования Smart-seq2. Репрезентативные распределения фрагментов по размерам библиотек секвенирования (из шага 4.15), полученных из клеток перицикла ксилемного полюса (A), клеток зачатков боковых корней (B), клеток эндодермы/коры (C) и клеток боковой крышки корня (D). (E) Библиотека небольшого размера с пиками димера праймера/адаптера, которые все еще могут быть секвенированы после выбора размера. (F) библиотека с ненормальным распределением размеров, что свидетельствует о неудачной подготовке библиотеки. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.

Рисунок 4: Анализ генов, обогащенных клеточным типом. Анализ DEG проводился между каждым типом клеток и всем корнем; Гены с более чем четырехкратной активацией в каждом типе клеток были идентифицированы как гены, обогащенные клеточным типом. Эти гены были объединены, сгруппированы и визуализированы на графике тепловой карты (верхняя панель). Гены, обогащенные клеточным типом, включали многие известные гены-маркеры; типичные маркерные гены, такие как YUCCA3, MYB36, WOX5 и PFA1 , были выбраны для отображения в браузере генома (нижняя панель). Сокращения: LRP = боковые корневые зачатки; Эндо/Кор = эндодерма/кора; LRC = боковая корневая крышка. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
| Компоненты | Объем (мкл) |
| 10% Triton X-100 | 0.33 |
| Ингибитор РНКазы | 0.55 |
| DTT (0,1 М) | 0.22 |
Таблица 1: Компоненты реакции для приготовления буфера лизиса клеток (смесь А).
| Компоненты | Объем (мкл) |
| Oligo-dT30VN (100 мкМ) | 0.44 |
| dNTP (10 мМ) | 4.4 |
Таблица 2: Реакционные компоненты для приготовления смеси Б.
| Компоненты | Объем (мкл) |
| Буфер SuperScript IV (5x) | 8.8 |
| Бетаин (5 м) | 8.8 |
| DTT (0,1 М) | 2.2 |
| MgCl2 (1 м) | 0.264 |
| TSO (100 мкМ) | 0.44 |
| Обратная транскриптаза SuperScript IV (200 ЕД/мкл) | 2.2 |
| Ингибитор РНКазы | 1.1 |
Таблица 3: Компоненты реакции для приготовления ПЦР-смеси с обратной транскрипцией (смесь С).
| Цикл | Температура (°C) | Время |
| 1 | 50 | 90 мин |
| 2-11 | 55 | 2 мин |
| 50 | 2 мин | |
| 12 | 70 | 15 мин |
| 13 | 4 | ∞ |
Таблица 4: Настройки ПЦР с обратной транскрипцией (ОТ) для синтеза кДНК из мРНК.
| Компоненты | Объем (мкл) |
| KAPA HiFi HotStart ReadyMix (2x) | 44 |
| Праймер IS PCR (10 мкМ) | 0.88 |
Таблица 5: Реакционные компоненты для приготовления реакционной смеси предварительного усиления (смесь D).
| Цикл | Температура (°C) | Время |
| 1 | 98 | 5 мин |
| 2-13 | 98 | 20 с |
| 67 | 30 с | |
| 72 | 3 мин | |
| 14 | 72 | 5 мин |
| 15 | 4 | ∞ |
Таблица 6: Настройки программы ПЦР для реакции предварительной амплификации.
Обсуждение
Протокол на основе Smart-seq2 может генерировать надежные библиотеки секвенирования из нескольких сотен ячеек8. Качество исходного материала имеет важное значение для точности анализа транскриптома. FACS является мощным инструментом для подготовки клеток, представляющих интерес, но эта процедура, особенно этап протопластинга, должна быть оптимизирована для применения в растениях. Лазерная захватная микродиссекция (LCM) или ручная рассеченная клетка также может быть использована в качестве входныхданных 25,26, поэтому представленный здесь протокол потенциально может быть использован для различных видов растений и типов клеток с известными маркерными генами или без них.
Подготовка растительного сырья
В биологии развития растений FACS обычно используется для выделения обогащенных клеточных популяций, экспрессирующих флуоресцентный маркер. Растения, которые экспрессируют такой маркер, протопластируются, и протопласты в конечном итоге сортируются в чистые субпопуляции на основе экспрессии маркера, специфичного для клеточного типа. Поэтому сигнал флуоресцентного маркера должен быть сильным и специфическим. Кроме того, исследователь должен заранее проверить, какие маркеры можно отсортировать. Количество необходимых семян зависит от уровня экспрессии, где экспрессируется маркер, сколько ячеек необходимо для последующих применений и сколько реплик отсортировано. Если экспрессия находится в одном типе клеток с очень небольшим количеством клеток на растение, обычно требуется большее количество растений, чем если бы маркер экспрессировался в большем количестве клеток. Лучше всего опытным путем определить количество семян, необходимое для отдельных маркерных линий. Особенно важно проводить предварительные эксперименты с каждой маркерной линией, чтобы оптимизировать окно для сортировки и знать, сколько клеток получится из фиксированного количества растений. Если экспериментальным материалом являются корни, то для предотвращения погружения корней в агар и облегчения срезания чистых корней на содержащий среду агар перед посевом можно уложить автоклавную сетку подходящего размера и автоклавную сетку. Низкотемпературная стратификация способствует прорастанию и развитию семян, поэтому мы рекомендуем стратифицировать семена при температуре 4 °C в течение 2 дней после стерилизации или покрытия, а затем выращивать растения вертикально.
Протопластинг и FACS
Хорошо работают протопластинг и сортировка через 5-6 дней после стратификации. Раствор А и раствор Б необходимо отфильтровать с помощью сетчатого фильтра 0,22 мкм. Кроме того, хранение раствора B при температуре -20 °C в течение более длительных периодов времени снижает эффективность ферментов. Перед началом раствор А и раствор Б следует аккуратно разморозить на льду, что занимает около 20 мин. Раствор Б нельзя встряхивать, так как встряхивание раствора Б может нарушить работу ферментов и вызвать чрезмерное образование пузырьков. Многократная промывка сетчатого фильтра раствором А может увеличить количество ячеек, полученных на шаге 2.5. Как описано на шаге 2.6, всю надосадочную жидкость не следует отсасывать, так как протопласты находятся внизу рядом с гранулой и не видны. Перед сортировкой лучше всего убедиться, что концентрация протопластов составляет около 10 5-10 6 клеток/мл для достижения лучшей эффективности сортировки. При постановке нового эксперимента в качестве элемента управления для настройки сортировочного затвора требуется та же ткань из растения WT. На этапе 3.1 рекомендуется сначала выбрать флуоресцентные каналы (например, PE и FITC) и использовать контрольный образец для определения основной популяции в соответствии с SSC и FSC. Дополнительно предлагается отрегулировать положение затвора путем изменения напряжения флуоресцентного канала для обеспечения того, чтобы управляющий сигнал располагался слева от затвора (т.е. отрицательная группа расположена ниже 103). Время сортировки должно быть ограничено 30 минутами или менее 60 минутами, чтобы предотвратить изменения экспрессии генов, которые могут произойти из-за протопластинга или сортировки. После сортировки следует собрать небольшое количество отсортированных клеток и проверить их на флуоресценцию (этап 3.4). Из надосадочной жидкости должно быть удалено как можно больше буфера, чтобы избежать какого-либо воздействия на конструкцию нижестоящей библиотеки (этап 3.3).
Построение библиотеки RNA-seq
Для построения библиотеки RNA-seq мы рекомендуем использовать клетки сразу после сортировки, чтобы уменьшить деградацию РНК. Не рекомендуется начинать со слишком большого количества клеток, так как это может привести к некачественной библиотеке из-за неадекватных реакций. Количество циклов ПЦР на шагах 4.7 и 4.14 зависит от входного количества РНК/кДНК. Количество циклов может быть увеличено, когда вход меньше, или уменьшен, когда вход больше. Поэтому рекомендуется взять часть смешанных образцов из шага 4.7 и шага 4.14 для запуска ПЦР в реальном времени с той же процедурой амплификации, а затем определить окончательное количество циклов на основе результатов кПЦР перед формальной амплификацией пребиблиотеки/библиотеки. Кроме того, перед этапом очистки 4.8 шарики Ampure XP должны быть уравновешены при комнатной температуре в течение не менее 10 минут, а затем хорошо перемешаны. Объем шариков на этапе очистки не должен быть увеличен выше соотношения 0,8:1. В противном случае это увеличит унос грунтовочных димеров. Кроме того, следует избегать пересушивания шариков на шаге 4.11, чтобы предотвратить затрудненную ресуспендирование. Квалифицированная предварительная библиотека должна иметь средний размер около 1,5-2 кб и небольшое количество коротких (<500.н.) фрагментов. Аномальное распределение по размерам или небольшие пики праймера/адаптера димера в библиотеках являются показателями низкого качества, и эти образцы должны быть выброшены или подвергнуты дальнейшим циклам очистки шариков (этап 4.15) (рис. 3E-F).
Ограничения
Этот протокол может быть применен для выделения клеток из других тканей растений и других видов растений. Однако процесс выделения клеток сильно зависит от подготовки протопластов. Некоторые типы клеток трудно выделить, такие как сосудистые клетки и женские половые клетки, которые расположены внутри ткани и/или немногочисленны. Для клеток, для которых трудно получить протопласты, факультативным методом может быть использована сортировка ядер, активируемых флуоресценцией (FANS)27 . Между тем, использование этого протокола для получения конкретных типов клеток с помощью FACS зависит от наличия флуоресцентных маркерных линий. Отсутствие таких маркерных линий ограничивает использование этих методов в сельскохозяйственных культурах и садовых растениях. Применение высокопроизводительной технологии секвенирования одноклеточной РНК в сельскохозяйственных культурах позволит выявить новые гены-маркеры, специфичные для клеточного типа, которые могут быть в дальнейшем использованы для разработки маркерных линий клеточного типа и расширения возможностей применения исследований РНК-секвенирования клеточного типа на основе FACS и мультиомических исследований.
Раскрытие информации
Авторам раскрывать нечего.
Благодарности
Мы создали этот протокол в одноклеточном мультиомиксном центре Школы сельского хозяйства и биологии Шанхайского университета Цзяотун при поддержке Национального фонда естественных наук Китая (грант No 32070608), Шанхайской программы Пуцзян (грант No 20PJ1405800) и Шанхайского университета Цзяотун (грант No Agri-X20200202, 2019TPB05).
Материалы
| Name | Company | Catalog Number | Comments |
| 0.22 µm strainer | Sorfa | 622110 | |
| Agar | Yeasen | 70101ES76 | |
| Agilent fragment analyzer | Aglient | Aglient 5200 | |
| Agilent high-sensitivity DNA kit | Aglient | DNF-474-0500 | |
| Ampure XP beads | BECKMAN | A63881 | |
| Betaine | yuanye | S18046-100g | |
| Bleach | Mr Muscle | FnBn83BK | 20% (v/v) bleach |
| BSA | sigma | 9048-46-8 | |
| CaCl2 | yuanye | S24109-500g | |
| Cellulase R10 | Yakult (Japan) | 9012-54-8 | |
| Cellulase RS | Yakult (Japan) | 9012-54-8 | |
| Centrifuge tube (1.5 mL) | Eppendolf | 30121589 | |
| DNase, RNase, DNA and RNA Away Surface Decontaminants | Beyotime | R0127 | |
| dNTPs (10 mM) | NEB | N0447S | |
| DTT (0.1 M) | invitrogen | 18090050 | |
| Ethanol | Sinopharm Chemical Reagent Co., Ltd | 100092680 | |
| FACS | BD FACS Melody | BD-65745 | |
| FACS | Sony | SH800S | |
| Filter tip (1000 µL) | Thermo Scientific | TF112-1000-Q | |
| Filter tip (200 µL) | Thermo Scientific | TF140-200-Q | |
| Filter tip (10 µL) | Thermo Scientific | TF104-10-Q | |
| Filter tip (100 µL) | Thermo Scientific | TF113-100-Q | |
| Fluorescent microscope | Nikon | Eclipse Ni-E | |
| Four-Dimensional Rotating Mixer | Kylin -Bell | BE-1100 | |
| Hemicellulase | sigma | 9025-56-3 | |
| IS PCR primer | 5'-AAGCAGTGGTATCAACGCAGAG T-3' | ||
| KAPA HiFi HotStart ReadyMix(2X) | Roche | 7958935001 | |
| KCl | Sinopharm Chemical Reagent Co., Ltd | 7447-40-7 | |
| Macerozyme R10 | Yakult (Japan) | 9032-75-1 | |
| Magnetic separation stand | invitrogen | 12321D | |
| Mannitol | aladdin | 69-65-8 | |
| MES | aladdin | 145224948 | |
| MgCl2 | yuanye | R21455-500ml | |
| Microcentrifuges | Eppendorf | Centrifuge 5425 | |
| Micro-mini-centrifuge | Titan | Timi-10k | |
| MS | Phytotech | M519 | |
| Nextera XT DNA Library Preparation Kit | illumina | FC-131-1024 | |
| oligo-dT30VN primer | 5'-AAGCAGTGGTATCAACGCAGAG TACTTTTTTTTTTTTTTTTTTTTTTT TTTTTTTTTTVN-3' | ||
| PCR instrument | Thermal cycler | A24811 | |
| Pectolyase | Yakult (Japan) | 9033-35-6 | |
| Plant marker lines | Nottingham Arabidopsis Stock Centre (NASC) | ||
| Qubit 1x dsDNA HS Assay Kit | invitrogen | Q33231 | |
| Qubit 2.0 fluorometer | invitrogen | Q32866 | |
| RNase inhibitor | Thermo Scientific | EO0382 | |
| RNase-free water | invitrogen | 10977023 | |
| Solution A | 400 mM mannitol, 0.05 % BSA , 20 mM MES (pH5.7), 10 mM CaCl2, 20 mM KCl | ||
| Solution B | 1 % (w/v)cellulase R10, 1 % (w/v) cellulase RS, 1 % (w/v)hemicellulase, 0.5 % (w/v)pectolyase and 1 % (w/v) Macerozyme R10 in a fresh aliquot of solution A | ||
| Sterile pestle | BIOTREAT | 453463 | |
| Strainer (40 µm ) | Sorfa | 251100 | |
| SuperScript IV reverse transcriptase (200 U/µL) | invitrogen | 18090050 | |
| SuperScript IV buffer (5x) | invitrogen | 18090050 | |
| Test tube (5 mL) | BD Falcon | 352052 | |
| Thin-walled PCR tubes with caps (0.5 mL) | AXYGEN | PCR-05-C | |
| Triton X-100 | Sangon Biotech | A600198-0500 | |
| TSO primer | 5'-AAGCAGTGGTATCAACGCAGAG TACATrGrG+G-3' | ||
| Vortex | Titan | VM-T2 |
Ссылки
- Zhang, T. -. Q., Chen, Y., Liu, Y., Lin, W. -. H., Wang, J. -. W. Single-cell transcriptome atlas and chromatin accessibility landscape reveal differentiation trajectories in the rice root. Nature Communications. 12 (1), 2053 (2021).
- Denyer, T., et al. Spatiotemporal developmental trajectories in the Arabidopsis root revealed using high-throughput single-cell RNA sequencing. Developmental Cell. 48 (6), 840-852 (2019).
- Liu, Q., et al. Transcriptional landscape of rice roots at the single-cell resolution. Molecular Plant. 14 (3), 384-394 (2021).
- Liu, Z., et al. Global dynamic molecular profiling of stomatal lineage cell development by single-cell RNA sequencing. Molecular Plant. 13 (8), 1178-1193 (2020).
- Shahan, R., et al. A single-cell Arabidopsis root atlas reveals developmental trajectories in wild-type and cell identity mutants. Developmental Cell. 57 (4), 543-560 (2022).
- Wendrich, J. R., et al. Vascular transcription factors guide plant epidermal responses to limiting phosphate conditions. Science. 370 (6518), (2020).
- Carter, A. D., Bonyadi, R., Gifford, M. L. The use of fluorescence-activated cell sorting in studying plant development and environmental responses. The International Journal of Developmental Biology. 57 (6-8), 545-552 (2013).
- Picelli, S., et al. Full-length RNA-seq from single cells using Smart-seq2. Nature Protocols. 9 (1), 171-181 (2014).
- Wang, X., He, Y., Zhang, Q., Ren, X., Zhang, Z. Direct comparative analyses of 10X genomics chromium and Smart-seq2. Genomics Proteomics Bioinformatics. 19 (2), 253-266 (2021).
- Serrano-Ron, L., et al. Reconstruction of lateral root formation through single-cell RNAsequencing reveals order of tissue initiation. Molecular Plant. 14 (8), 1362-1378 (2021).
- Hashimshony, T., et al. CEL-Seq2: Sensitive highly-multiplexed single-cell RNA-Seq. Genome Biology. 17, 77 (2016).
- Macaulay, I. C., et al. G&T-seq: Parallel sequencing of single-cell genomes and transcriptomes. Nature Methods. 12 (6), 519-522 (2015).
- Angermueller, C., et al. Parallel single-cell sequencing links transcriptional and epigenetic heterogeneity. Nature Methods. 13 (3), 229-232 (2016).
- De Rybel, B., et al. A novel aux/IAA28 signaling cascade activates GATA23-dependent specification of lateral root founder cell identity. Current Biology. 20 (19), 1697-1706 (2010).
- Duncombe, S. G., Barnes, W. J., Anderson, C. T. Imaging the delivery and behavior of cellulose synthases in Arabidopsis thaliana using confocal microscopy. Methods in Cell Biology. 160, 201-213 (2020).
- Levy, S. E., Myers, R. M. Advancements in next-generation sequencing. Annual Review of Genomics and Human Genetics. 17 (1), 95-115 (2016).
- Ooi, C. C., et al. High-throughput full-length single-cell mRNA-seq of rare cells. PLoS One. 12 (11), e0188510 (2017).
- Pertea, M., Kim, D., Pertea, G. M., Leek, J. T., Salzberg, S. L. Transcript-level expression analysis of RNA-seq experiments with HISAT, StringTie and Ballgown. Nature Protocols. 11 (9), 1650-1667 (2016).
- Tsyganov, K., Perry, A., Archer, S., Powell, D. RNAsik: A Pipeline for complete and reproducible RNA-seq analysis that runs anywhere with speed and ease. Journal of Open Source Software. 3, 583 (2018).
- Love, M. I., Huber, W., Anders, S. Moderated estimation of fold change and dispersion for RNA-seq data with DESeq2. Genome Biology. 15 (12), 550 (2014).
- Kamiya, T., et al. The MYB36 transcription factor orchestrates Casparian strip formation. Proceedings of the National Academy of Sciences of the United States of America. 112 (33), 10533-10538 (2015).
- Zhang, Y., et al. Two types of bHLH transcription factor determine the competence of the pericycle for lateral root initiation. Nature Plants. 7 (5), 633-643 (2021).
- Haecker, A., et al. Expression dynamics of WOX genes mark cell fate decisions during early embryonic patterning in Arabidopsis thaliana. Development. 131 (3), 657-668 (2004).
- Chen, Q., et al. Auxin overproduction in shoots cannot rescue auxin deficiencies in Arabidopsis roots. Plant Cell Physiol. 55 (6), 1072-1079 (2014).
- Nichterwitz, S., et al. Laser capture microscopy coupled with Smart-seq2 for precise spatial transcriptomic profiling. Nature Communications. 7, 12139 (2016).
- Long, J., et al. Nurse cell--derived small RNAs define paternal epigenetic inheritance in Arabidopsis. Science. 373 (6550), (2021).
- Gutzat, R., et al. Arabidopsis shoot stem cells display dynamic transcription and DNA methylation patterns. EMBO Journal. 39 (20), e103667 (2020).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены