Method Article
Florojenik RNA Aptamers için In Vitro ve Hücresel Turn-on Kinetiğinin Belirlenmesi
* Bu yazarlar eşit katkıda bulunmuştur
Bu Makalede
Özet
Protokol, florojenik RNA aptamers Ispanak2 ve Brokolinin kinetiğini belirlemek için iki yöntem sunar. İlk yöntem, florojenik aptamer kinetiğinin in vitro olarak bir plaka okuyucu ile nasıl ölçüleceğini açıklarken, ikinci yöntem hücrelerdeki florojenik aptamer kinetiğinin akış sitometrisi ile ölçülmesini detaylandırır.
Özet
Florojenik RNA aptamerleri, RNA'ları etiketlemek ve görselleştirmek, gen ekspresyonu hakkında rapor vermek ve metabolitlerin ve sinyal moleküllerinin seviyelerini tespit eden floresan biyosensörleri aktive etmek için canlı hücrelere uygulanmıştır. Bu sistemlerin her birindeki dinamik değişiklikleri incelemek için, gerçek zamanlı ölçümlerin elde edilmesi arzu edilir, ancak ölçümlerin doğruluğu, florojenik reaksiyonun kinetiğinin örnekleme frekansından daha hızlı olmasına bağlıdır. Burada, sırasıyla bir numune enjektörü ve bir akış sitometresi ile donatılmış bir plaka okuyucu kullanarak florojenik RNA aptamers için in vitro ve hücresel açma kinetiğini belirleme yöntemlerini açıklıyoruz. Ispanak2 ve Brokoli aptamerlerinin floresan aktivasyonu için in vitro kinetiğin iki fazlı ilişki reaksiyonları olarak modellenebileceğini ve sırasıyla 0.56 s-1 ve 0.35 s-1'lik farklı hızlı faz hızı sabitlerine sahip olduğunu gösteriyoruz. Ek olarak, Escherichia coli'deki Ispanak2'nin floresan aktivasyonu için hücresel kinetiğin, Gram-negatif bakterilere boya difüzyonu ile daha da sınırlı olduğunu, dakika zaman ölçeğinde doğru örnekleme sıklığını sağlamak için hala yeterince hızlı olduğunu gösteriyoruz. Floresan aktivasyon kinetiğini analiz etmek için kullanılan bu yöntemler, geliştirilen diğer florojenik RNA aptamerlerine uygulanabilir.
Giriş
Florojenik reaksiyonlar, floresan sinyali üreten kimyasal reaksiyonlardır. Florojenik RNA aptamerler tipik olarak floresan kuantum verimini arttırmak için küçük bir molekül boyasını bağlayarak bu işlevi yerine getirir (Şekil 1A)1. Farklı florojenik RNA aptamer sistemleri geliştirilmiştir ve spesifik RNA aptamer dizilerinden ve buna karşılık gelen boya ligandlarından oluşur1. Florojenik RNA aptamerleri, mRNA'ların ve kodlamayan RNA'ların canlı hücre görüntülemesini sağlayan floresan etiketler olarak RNA transkriptlerine eklenmiştir 2,3,4. Ayrıca, raporlama fonksiyonu RNA seviyesi5,6 olması dışında, yeşil floresan proteininin (GFP) muhabir olarak kullanılmasına benzer şekilde, gen ekspresyonunun floresan raporlayıcıları olarak promotör dizilerinden sonra yerleştirilmişlerdir. Son olarak, florojenik RNA aptamerleri, belirli bir küçük moleküle yanıt olarak florojenik reaksiyonu tetiklemek için tasarlanmış RNA bazlı floresan biyosensörlere dahil edilmiştir. RNA bazlı floresan biyosensörler, çeşitli floresan olmayan metabolitlerin ve sinyal molekülleri 7,8,9,10,11'in canlı hücre görüntülemesi için geliştirilmiştir.
RNA lokalizasyonu, gen ekspresyonu ve küçük molekül sinyallerindeki dinamik değişiklikleri görselleştirmek için florojenik RNA aptamerlerinin geliştirilmesine artan ilgi vardır. Bu uygulamaların her biri için, gerçek zamanlı ölçümlerin elde edilmesi arzu edilir, ancak ölçümlerin doğruluğu, florojenik reaksiyonun kinetiğinin örnekleme frekansından daha hızlı olmasına bağlıdır. Burada, florojenik RNA aptamers Ispanak2 12 ve Brokoli13 için in vitro kinetiği bir örnek enjektör ile donatılmış bir plaka okuyucu kullanarak belirlemek ve bir akış sitometresi kullanarak Escherichia coli'de eksprese edilen Ispanak2 için hücresel açılma kinetiğini belirlemek için yöntemler açıklanmaktadır. Bu iki RNA aptamer seçildi çünkü RNA lokalizasyonu 2,3,4'ü incelemek için uygulandılar, muhabirler 5,6 ve biyosensörler 7,8,9,10,11'de kullanıldılar ve karşılık gelen boya ligandları (DFHBI veya DFHBI-1T) ticari olarak temin edilebilir. Literatürde belirlenen in vitro özelliklerinin bir özeti, protokol gelişimini (örneğin, kullanılan dalga boyları ve boya konsantrasyonları) bildiren Tablo 1 4,13,14'te verilmiştir. Bu sonuçlar, RNA aptamerlerinden etkilenen florojenik reaksiyonların hızlı olduğunu ve istenen hücre biyolojik uygulamaları için doğru ölçümleri engellememesi gerektiğini göstermektedir.
Protokol
1. In vitro kinetik deneyi
- DNA şablonlarının PCR ile hazırlanması
- PCR reaksiyonlarını ayarlayın: PCR reaksiyonlarını hazırlamak için, aşağıdaki reaktifleri ince duvarlı bir PCR tüpünde birleştirin:
33 μL çift damıtılmış su (ddH2O)
Yüksek doğrulukta DNA polimeraz için 10 μL 5x tampon
Her biri 2 mM'lik 5 μL deoksiribonükleozid trifosfat (dNTP)
0,5 μL 40 μM ileri astar
0,5 μL 40 μM ters astar
DNA şablonunun 0,5 μL (10-100 ng) (yalnızca Ispanak2 PCR için; Brokoli primerleri örtüşür)
0.5 μL yüksek doğrulukta DNA polimeraz (son ekle)
NOT: Sentetik oligonükleotidler genellikle kuru olarak sevk edilir. Stok çözeltileri hazırlamak için, bilinen bir ddH2O hacmi (100 μL önerilir) ekleyin ve tükenme katsayısının çevrimiçi olarak en yakın komşu kuralları tarafından hesaplanabileceği Bira yasasına göre konsantrasyonu belirlemek için bu çözeltinin A260'ını ölçün. Bu stok çözeltisi daha sonra seyreltmeleri PCR'de kullanıma uygun hale getirmek için kullanılabilir. - Termosikler protokolünü çalıştırın.
- Tam uzunlukta Ispanak2 ve Brokoli DNA şablonlarını yükseltmek için aşağıdaki termosikler protokolünü kullanın:
İlk denatürasyon:2 dk için 98 °C
35 döngü:
Denatürasyon:15 s için 98 °C denatürasyon
Tavlama:30 s için 72 °C
Uzantı:30 s için 72 °C
Son uzatma: 5 dakika boyunca 72 °C - Reaksiyondan sonra, istenen DNA ürününün varlığını doğrulamak için ürünün küçük bir alikotunu% 2 agaroz jeli ile düşük moleküler ağırlıklı bir merdivenle (boyut aralığı: 25-766 nükleotidler) analiz edin.
- Ticari olarak temin edilebilen jel ekstraksiyonu veya PCR temizleme kitleri ile ürünü saflaştırın ve ddH2O veya üretici tarafından sağlanan tamponla salın.
NOT: Bir PCR temizleme kiti seçerken, kolon moleküler ağırlık kesiminin T7-Brokoliyi (81 nükleotid) tutacak kadar düşük olduğundan emin olun, aksi takdirde PCR ürünü kaybolacaktır.
NOT: İsteğe bağlı duraklatma noktası: DNA'yı -20 °C'de saklayın.
- Tam uzunlukta Ispanak2 ve Brokoli DNA şablonlarını yükseltmek için aşağıdaki termosikler protokolünü kullanın:
- PCR reaksiyonlarını ayarlayın: PCR reaksiyonlarını hazırlamak için, aşağıdaki reaktifleri ince duvarlı bir PCR tüpünde birleştirin:
- Ispanak2 ve Brokoli RNA'nın in vitro transkripsiyon (IVT) ile hazırlanması
- Transkripsiyon reaksiyonlarını ayarlayın.
- 100 μL'lik bir transkripsiyon reaksiyonu hazırlamak için, aşağıdaki reaktifleri 1.5 mL'lik bir mikrosantrifüj tüpünde birleştirin: 10 μL 10x transkripsiyon tamponu + 20 μL 10 mM ribonükleozid trifosfatlar (rNTP'ler) + 1-64 μL DNA şablonu (toplam 1 μg) + 2 μL inorganik pirofosfataz + ddH 2 O ila 98 μL +2μL T7 RNA polimeraz (son ekle).
- Bu reaksiyonu 37 °C'de 4 saat boyunca inkübe edin. %20 sakaroz, %0,1 sodyum dodesil sülfat (SDS), 1x Tris-Borat-EDTA (1x TBE) tamponu ve ~18 M üreden oluşan 100 μL 2x üre jel yükleme tamponu (2x ULB) ekleyerek reaksiyonu söndürün.
NOT: İsteğe bağlı duraklatma noktası: Söndürülmüş reaksiyonu -20 °C'de saklayın.
- Transkripsiyon reaksiyonlarını ayarlayın.
- RNA'nın poliakrilamid jel elektroforezi (PAGE) saflaştırılması
- Ispanak2 ve Brokoli RNA'nın PAGE saflaştırılması
DİKKAT: Polimerize olmayan (sıvı veya toz halinde) akrilamid son derece toksiktir. Toz haline getirilmiş akrilamid tartılıyorsa, bunu bir duman davlumbazında yapın. Her zaman uygun koruyucu ekipman giyin ve ellerinizi iyice yıkayarak akrilamid tozu veya çözeltisi ile kirlenmiş eldivenleri derhal çıkarın. Akrilamid cilde doğrudan temas ederse, maruz kalan bölgeyi en az 15 dakika boyunca sabun ve suyla yıkayın. Akrilamid gözlerle doğrudan temas ederse, 15 dakika boyunca suyla yıkayın.- PAGE jelini hazırlayın: İstenmeyen abortif transkriptleri ve reaksiyona girmemiş rNTP'leri tam uzunluktaki üründen çıkarmak için,% 6'lık bir üre-poliakrilamid jeli hazırlayın. Genel olarak 8 delikli tarak ile 28 cm x 16,5 cm x 1,5 mm jeller kullanılabilir. Rezervuarları doldurmak için 1x TBE tampon kullanarak jel ve elektroforez ekipmanını kurun.
- RNA numunelerini PAGE jeline yükleyin: Jeli şerit başına bir adet söndürülmüş 200 μL reaksiyonla yükleyin. Ayrı bir şeritte, jelde sırasıyla 106 nükleotid ve 26 nükleotitte,15'te göç eden izleyici boyaları ksilen siyanol ve bromofenol mavisi ile 2x ULB yükleyin. Sonraki adımlarda olası kontaminasyonu önlemek için her numune arasında boş bir şerit bırakın.
- PAGE jelini çalıştırın: 95-nt Ispanak2 ve 49-nt Brokoliyi kendi kesilmiş ürünlerinden ayırmak için, jeli 25 W'ta 1.5-2 saat çalıştırın, bu noktada bromofenol mavi boyası jel uzunluğunun ~ 5 / 6'sını geçirmiş olacaktır.
- RNA örneklerini PAGE jelinde görselleştirin: Jelin etrafındaki cam plakaları sökün ve jeli her iki taraftaki plastik ambalajla örtün, sargı üzerindeki şeritleri etiketleyin. Karanlık bir odadaki RNA bantlarını, sarılmış jeli UV ışığı altında floresan bir TLC plakasına yerleştirerek UV gölgeleme ile görselleştirin. Ürüne karşılık gelen RNA bantlarının kenarını bir işaretleyici ile hızlı bir şekilde özetleyin ve UV ışınlarına maruz kalmaktan kaynaklanan hasarı en aza indirmek için UV lambasını kapatın.
- RNA numunelerini PAGE jelinden tüketin ve çıkarın: Her numune için taze bir tıraş bıçağı ile, istenen ürün bantlarını tüketin, ~ 1 mm küplere zar atın ve jel parçalarını, oda sıcaklığında (RT) 2 saat veya 4 ° C'de bir gece boyunca bir rotatörde RNA'yı çıkarmak için 500 μL ezme ıslatma tamponuna sahip 2 mL mikro santrifüj tüpüne ekleyin.
NOT: İsteğe bağlı duraklatma noktası: numune -20 °C'de saklanabilir. - RNA'yı çökeltin
- Jel parçalarını tamponda ekstrakte edilen RNA'dan ayırmak için, numuneyi 4 ° C'de 20 dakika boyunca 13.000 x g'de santrifüj edin ve ardından süpernatantı çıkarmak ve yeni bir 2 mL mikrosantrifüj tüpüne yüklemek için dar uçlu bir pipet kullanın.
- RNA'yı çökeltmek için, 1.5 mL buz gibi soğuk etanol ve 1 μL 20 mg / mL glikojen, vorteks ekleyin ve -20 ° C veya -80 ° C'de en az 1 saat saklayın.
NOT: İsteğe bağlı duraklama noktası: RNA birkaç ay boyunca -20 ° C'de saklanabilir.
- RNA çökeltinin toplanması: Çökeltilmiş RNA'yı 4 ° C'de 20 dakika boyunca 13.000 x g'de santrifüjleme ile pelet edin. Süpernatantı çıkarın ve peleti 30 μL ddH2O veya 1x TE tamponunda yeniden askıya almadan önce kalan etanolün açık hava altında (~ 1 saat) buharlaşmasına izin verin.
NOT: Bu işlem tipik olarak ~ 10 μM'lik bir son RNA konsantrasyonu ile sonuçlanır.
NOT: İsteğe bağlı duraklama noktası: RNA örneği birkaç ay boyunca -20 ° C'de saklanabilir.
- RNA stok konsantrasyonlarını belirleyin.
- Hidroliz reaksiyonu için bir RNA aliquot hazırlayın.
- Bu testi yapmak için, ilk olarak, stok RNA örneğinin A 260'ını belirlemek için bir UV / Vis nano spektrofotometresi kullanın ve numunenin ddH2O'da ~10 absorbans birimine (AU) seyreltilmiş bir aliquotunu yapın.
- 0.5 mL'lik bir PCR tüpünde aşağıdaki reaksiyonu hazırlayın: 16 μL ddH 2 O + 2 μL 10x Na 2 CO3 tamponu + ~ 10 AU'ya seyreltilmiş2μL RNA aliquot. Reaksiyonu 95 ° C'de 90 dakika boyunca inkübe edin ve ardından RT'ye soğumasını bekleyin.
- Nükleotid absorbanslarını kullanarak RNA konsantrasyonunu belirleyin: Bu numunenin A260'ını bir UV / Vis nano spektrofotometre ile ölçün ve aşağıdaki formülü kullanarak RNA konsantrasyonunu hesaplayın:

burada c, RNA'nın konsantrasyonudur, b yol uzunluğudur, i, spesifik nükleotiddir (A, C, G veya U), n i, RNA dizisindeki nükleotid i'nin frekansıdır ve ε i, nükleotid i'nin molar yok olma katsayısıdır. Orijinal stok RNA konsantrasyonunu belirlemek için, c'yi seyreltme faktörü ile çarpın.
NOT: İsteğe bağlı duraklatma noktası: RNA birkaç ay boyunca -20 ° C'de saklanabilir.
- Hidroliz reaksiyonu için bir RNA aliquot hazırlayın.
- Ispanak2 ve Brokoli RNA'nın PAGE saflaştırılması
- Bir in vitro plaka okuyucu kinetik tahlili
- RNA renatürasyon programını ayarlayın: Kaydet'e basmadan önce aşağıdaki adımların her birini eklemek için Yeni Program Oluştur > Yeni Faz Ekle > Yeni Adım Ekle'yi birden çok kez seçerek aşağıdaki termosikler protokolünü oluşturun:
3 dk için 70 °C
45 sn için 65 °C
45 sn için 60 °C
45 sn için 55 °C
45 sn için 50 °C
45 sn. için 45 °C
45 sn için 40 °C
45 sn için 35 °C
45 sn için 30 °C - RNA'yı yeniden doğandırın: Ispanak2 ve Brokoli RNA'larını yeniden denatüre etmek için, 0,5 mL ince duvarlı birPCR tüpünde ddH 2 O'da her RNA'nın 2 μM stoğunu hazırlayın ve ardından 1 μM RNA çözeltisi yapmak için eşit hacimde 2x renatürasyon tamponu (80 mM HEPES, pH 7.5 [KOH], 250 mM KCl, 6 mM MgCl2) ekleyin. Tüpleri termosiklusa ekleyin, kaydedilen renatürasyon programını açın ve Çalıştır'a basın.
NOT: Bir termosikler mevcut değilse, RNA'lar bunun yerine 70 ° C'lik bir ısı bloğunda 3 dakika boyunca inkübe edilebilir ve daha sonra tezgahta RT'ye yavaşça soğumaya bırakılabilir. - Bağlayıcı reaksiyon tamponunu hazırlayın: Tamponu bir aptamer-boya bağlama reaksiyonuna hazırlamak için, tampon bileşenleri içeren bir ana karışım yapın (69,5 μL ddH 2 O + 4 μL 1 M HEPES, pH 7,5 [KOH] + 6,2 μL 2 M KCl + 0,3 μL 1 M MgCl2). Kaç numune ve çoğaltmaya ihtiyaç duyulduğuna bağlı olarak, bu değerleri numune sayısı artı bir ile çarpın. Genel olarak, RNA örneği başına üç replika tatmin edicidir.
- Plaka okuyucuyu hazırlayın.
- Plaka okuyucu enjektör programını ayarlayın: Floresan plaka okuyucusunda, Sıcaklık'ı seçin ve istenen sıcaklığı 37 ° C'ye ayarlayın, kinetiği deneylere başlamadan önce sıcaklığın bu değere eşit olduğundan emin olun. Plaka okuyucu yazılımını açın, Ayarlar > Edinme Görünümü'nü seçin ve kinetik ölçümler için aşağıdaki programı girin:
Döngü: Her kuyu için
Temel ayar: 60 anahat okuması
SmartInject ayarları: 10 μL enjeksiyon (taban çizgisi okunduktan sonra gerçekleşir)
Floresan (veya FL) şunları okur:
Uyarma: 448 nm (bant genişliği: 9 nm)
Emisyon: 506 nm (bant genişliği: 15 nm)
Kartuş: MONO (s/n 3297)
Zamanlama:
Toplam okuma süresi: 10 dk
Okuma aralığı: 0,5 s
PMT ve Optik: Okuma başına 6 flaş
Döngü: Sonraki kuyu - Enjektörü hazırlayın
- Enjektörü yıkayın ve aspire edin: Plaka okuyucu enjektörünü hazırlamak için önce Plaka okuyucuya enjekte et'i seçin, yönlendirildiğinde plaka okuyucuya bir atık toplama plakası sağlayın ve ardından Yıka'yı seçin ve plaka okuyucudaki talimatları izleyerek enjeksiyon tüpünü 1 mL hacimli ddH2O, ddH2O'da %75 etanol ile temizleyin, ve sonra ddH2O. Ardından, enjektörün fazla sıvıyı çıkarmasına izin vermek için Aspirat'ı seçin.
- Enjektörü astarlayın: Önceki ekrana çıktıktan sonra, deneyler sırasında numunelere saf, konsantre ligand eklenmesini sağlamak için enjekte edilecek iki adet 260 μL hacim ligand ile enjektörü astarlamak için Prime'ı seçin - bu durumda, ddH2O'da 100 μM DFHBI ile astarlayın.
- Plaka okuyucu enjektör programını ayarlayın: Floresan plaka okuyucusunda, Sıcaklık'ı seçin ve istenen sıcaklığı 37 ° C'ye ayarlayın, kinetiği deneylere başlamadan önce sıcaklığın bu değere eşit olduğundan emin olun. Plaka okuyucu yazılımını açın, Ayarlar > Edinme Görünümü'nü seçin ve kinetik ölçümler için aşağıdaki programı girin:
- İn vitro kinetik deneyler gerçekleştirin : Bir kinetik deneyi gerçekleştirmek için, önce 96 kuyucuklu berrak alt plakanın bir kuyucuğuna 80 μL bağlayıcı tampon ana karışımı ekleyin, ardından 10 μL renatüre RNA. Bu plakanın ve enjektördeki DFHBI çözeltisinin, plaka okuyucuda 15 dakika boyunca 37 ° C'ye dengelenmesine izin verin.
- Okuma Alanı altındaki plaka okuyucu yazılımı Ayarları'nda, analiz edilecek kuyuyu seçin ve ardından daha önce açıklanan kinetik programını yürütmek için Giriş sekmesi altında Çalıştır'ı seçin. Tüm denemeler tamamlanana kadar bu işlemi tekrarlayın.
NOT: Kritik olarak, RNA kinetiğinin replikalar ve numuneler arasında aynı koşullar altında ölçülmesini sağlamak için kinetiği deneyleri her seferinde bir kuyu halinde yapılmalıdır. - Enjektörü yıkayın: Kalan DFHBI çözeltisini enjeksiyon tüpünden çıkarmak için, enjeksiyon tüpünü adım 1.4.4.2.1'de açıklandığı gibi 1 mL ddH 2 O hacimleri, ddH2O'da% 75 etanol ve ardındanddH2O ile yıkayın.
NOT: İsteğe bağlı duraklatma noktası.
- RNA renatürasyon programını ayarlayın: Kaydet'e basmadan önce aşağıdaki adımların her birini eklemek için Yeni Program Oluştur > Yeni Faz Ekle > Yeni Adım Ekle'yi birden çok kez seçerek aşağıdaki termosikler protokolünü oluşturun:
- Florojenik RNA aptamerlerinin in vitro kinetiğinin analizi.
- Verileri analiz yazılımına girin: Verileri işlenmek üzere kolayca kopyalamak için deneysel verileri elektronik tablo olarak dışa aktarın. Analiz yazılımında, XY formatında yeni bir veri tablosu oluşturun. X sütununa, her okuma zaman noktasını girin, t = 0 DFHBI enjeksiyonunun zamanıdır. Y sütununa, ilgili zaman noktasındaki replikalar arasındaki ortalama floresan değerlerini t = 0'dan başlayarak girin.
- Verileri normalleştirin ve grafiklendirin: Floresan değerlerini normalleştirmek için Normalleştir'> > Veri İşlemeyi Analiz Et'e ve ardından Tamam'a tıklayın. Normalleştirme, sonuçları kesirler halinde sunma ve Sonuçların Grafiğini Çizme için veri kümesinin en küçük ve en büyük değerlerini kullanmayı seçin ve ardından Tamam'a tıklayın. Zaman içinde normalleştirilmiş ortalama floresan grafiğini oluşturmak için [Veri Kümesi Adı] Normalleştir simgesine tıklayın ve Grafik Ailesi: Yalnızca Noktalarla XY'yi seçin.
NOT: Hata çubukları, az sayıda zaman noktasının grafiğinin çizildiği durumlarda görmek için yararlı olabilir. Bunlar istenirse, XY biçiminde bir veri tablosu seçerken, çoğaltma değerlerini yan yana sütunlara girmek için "Y" altındaki seçeneği belirleyin. Elde edilen tablonun grafiğini varsayılan seçenekler seçiliyken çizmek, hata çubukları olan bir grafik oluşturur. - Kinetik parametreleri elde etmek için eğri uydurma gerçekleştirin: Kinetik verilere bir eğri sığdırmak için Verileri Analiz Et > Analiz Et'e tıklayın ve XY Analizleri sekmesi altında Doğrusal Olmayan Regresyon (Eğriye Sığdırma) seçeneğini belirleyin. Model sekmesi altında, kinetik verileri aşağıdaki iki fazlı ilişkilendirme denklemiyle sığdırmak için Üstel > İki Fazlı İlişkilendirme'ye tıklayın:
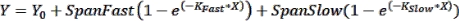
burada Y, X zamanındaki floresandır, Y 0, t = 0'daki floresandır, KHızlı ve K Yavaş, sırasıyla hızlı ve yavaş hız sabitleridir ve SpanFast ve SpanSlow, sırasıyla hızlı ve yavaş hızlar tarafından hesaplanan floresan açma aralıklarıdır (temsili sonuçlara bakınız, Şekil 1). Hız sabitlerini, t1/2 değerlerini ve PercentFast değerlerini görüntülemek için Nonlin Fit sekmesine tıklayın.
NOT: Tüm bu değerler için standart sapma elde etmek amacıyla, bireysel plaka okuyucu deney kopyaları yukarıda açıklandığı şekilde işlenebilir.
2. Hücresel kinetik deneyi
- E. coli suşlarının hazırlanması
- BL21 Star (DE3) E. coli hücrelerini, üreticinin protokolünü izleyerek ~ 100 ng pET31b tRNA-Ispanak2 yapısı ile dönüştürün.
NOT: Plazmid yapısı ticari olarak temin edilebilir (Plazmid #79783). - Karbenikilin (Karbonhidrat: 50 mg/mL) plakaları içeren LB agar üzerindeki hücreleri plakalayın ve 12-16 saat boyunca 37 °C'de inkübe edin. Plazmidler içeren hücreler plaka üzerinde koloniler halinde büyüyecektir.
NOT: İsteğe bağlı duraklama noktası: Plakalar üzerindeki dönüştürülmüş BL21 Yıldız hücreleri, 1 hafta boyunca parafilme sarılmış 4 °C'de saklanabilir.
- BL21 Star (DE3) E. coli hücrelerini, üreticinin protokolünü izleyerek ~ 100 ng pET31b tRNA-Ispanak2 yapısı ile dönüştürün.
- Büyüyen hücreler ve florojenik RNA aptamer ekspresyonunu indüklemek
- Karbenikilin (Karbonhidrat: 50 mg / mL) içeren 2 mL'lik bir indükleyici olmayan ortam (NI) kültürünü, dönüştürülmüş BL21 Yıldız hücrelerinin tek bir kolonisi ile aşılayın. Bunu en az üç biyolojik kopya için tekrarlayın. Kültürleri 37 ° C'de 22-24 saat boyunca 250 rpm'de bir inkübatörde / çalkalayıcıda inkübe edin.
NOT: İsteğe bağlı duraklama noktası: NI ortamında yetiştirilen hücreler plazmidi tutar ve 1 hafta boyunca 4 ° C'de saklanabilir. - NI ortamındaki büyümeden sonra, kültürü 100x, karbenikilin (Karbonhidrat: 50 mg / mL) içeren taze bir 3 mL ZYP-5052 otoindüksiyon ortamına (AI) seyreltin. İfadeyi indüklemek için hücreleri 37 ° C'de 250 rpm'de 250 rpm'de 16-18 saat boyunca büyütün.
NOT: Kültürler için tipik OD600 , 18 saatlik büyümeden sonra 2.0-3.3 arasında değişecektir. Optimum hücre yoğunluğu aralığı 2.5-3.0 arasındadır.
- Karbenikilin (Karbonhidrat: 50 mg / mL) içeren 2 mL'lik bir indükleyici olmayan ortam (NI) kültürünü, dönüştürülmüş BL21 Yıldız hücrelerinin tek bir kolonisi ile aşılayın. Bunu en az üç biyolojik kopya için tekrarlayın. Kültürleri 37 ° C'de 22-24 saat boyunca 250 rpm'de bir inkübatörde / çalkalayıcıda inkübe edin.
- Hücresel kinetik deneyinin gerçekleştirilmesi
- Akış sitometresini kurun.
- Akış sitometresini ve cihaza bağlı bilgisayarı açın. Akış sitometresi yazılımına giriş yaptıktan sonra, Cihaz sekmesinin altında, Başlangıç simgesine tıklayın. Doğru ölçümlü izleme başlatmayı sağlamak için yazılım ekranında belirtilen adımları izleyin.
NOT: Bazı akış sitometreleri cihaz başlatma dizisini Astarlama olarak adlandırır. Deney için kullanılacak akış sitometresi için üreticinin protokolünü izlediğinizden emin olun. - Bir performans testi çalıştırın (varsa). Ana Menü sekmesi altında, Performans Testi'ne tıklayın. Bir kültür tüpünde, üreticinin performans izleme boncuklarından üç damlayı 3 mL odaklama sıvısına ekleyin.
- Kültür tüpünü numune tüp kaldırıcıya yerleştirin ve kaldırıcıyı kaldırın. Performans Testini Çalıştır'ı tıklamadan önce, izleme boncuklarının tüpünün Lot numarasının Performans Testi Kurulumu ekranında belirtilenlerle aynı olduğundan emin olun. Testi çalıştırmak için Performans Testi'ni tıklatın.
- Bu deney için akış sitometresi yazılımını, tek hücreli floresan için aşağıdaki edinme parametreleriyle ayarlayın:
Uyarma Lazeri: 488 nm
Emisyon Kanalı: GFP (FITC olarak da adlandırılır)
Edinme Hacmi: 40 μL (toplam çekme hacmi 90 μL)
Akış Hızı: 200 μL/dk
Her ölçüm için hücre sayısı: 30.000
Cihaz Ayarları:
Gerilim:
FSC: 480 V
SSC: 400 V
BL1: 540 V
BL2: 392 V
BL3: 422 V
- Akış sitometresini ve cihaza bağlı bilgisayarı açın. Akış sitometresi yazılımına giriş yaptıktan sonra, Cihaz sekmesinin altında, Başlangıç simgesine tıklayın. Doğru ölçümlü izleme başlatmayı sağlamak için yazılım ekranında belirtilen adımları izleyin.
- Deneme dosyalarını ayarlayın.
- Akış sitometresi kullanıcı adını sağ tıklayarak Deneme Gezgini sekmesinde yeni bir deneme dosyası oluşturun. Açılır pencerede Yeni Deneme'yi seçin. Bilgisayar ekranında yeni bir pencere açıldığında Tamam'ı seçin.
- Yeni deneme dosyasında, "Grup" klasörüne sağ tıklayın ve Yeni Örnek Tüp Ekle'yi seçin. Her belirli zaman noktası için numune tüplerini etiketleyin ve Örnek'e sağ tıklayıp açılır menüden Yeniden Adlandır'ı seçerek çoğaltın. Çalışmanın amaçlanan zaman akışı için toplam çoğaltma sayısı ve zaman noktaları için bu adımı yineleyin.
- Seyreltik bir hücre çözeltisi hazırlayın: Yeni bir kültür tüpünde, 1,5 mL 1x PBS çözeltisi ekleyin. Ardından, seyreltilmiş bir hücre çözeltisi oluşturmak için AI ortamındaki 3 μL indüklenmiş hücreyi 1x PBS çözeltisine ekleyin. Farklı kültür tüplerindeki her biyolojik çoğaltma için bu adımı tekrarlayın.
- Hücrelerin arka plan floresansını ölçün: Boya eklemeden önce, 1x PBS çözeltisindeki hücreleri içeren her biyolojik replikasyon kültür tüpünün okumalarını alın. Bu, hücrelerin floresan arka planının, boya eklendikten sonra kıvrımın zamanla açıldığını gözlemlemek için ölçülmesidir.
- Bir okuma yapmak için, kültür tüpünü numune tüp kaldırıcıya yerleştirin ve kaldırıcıyı elle numune enjeksiyon iğnesine kaldırın. Koleksiyon Paneli sekmesinde uygun örnek dosyayı seçin ve Kaydet'i tıklatın.
- Çalıştırma tamamlandığında, numune tüp kaldırıcıyı kültür tüpü ile elle indirin. Bu, akışkan sistemi yıkayacak ve her biyolojik replikasyon numunesi arasındaki taşımayı en aza indirecek bir Durulama adımı başlatacaktır. Çalıştırma tamamlandıktan sonra veriler otomatik olarak bilgisayara kaydedilir.
- En az üç biyolojik replikasyon için hücresel floresan arka planını ölçmek üzere 2.3.4 içindeki adımları tekrarlayın. Sonraki örnek dosyaya geçmek için, Kayıt simgesinin altındaki örnek tüp adının yanındaki sağ ok simgesini tıklatarak bir sonraki örnek dosyayı seçin.
- Boya içeren hücreler için zaman noktalarında floresan ölçün.
- 50 μM DFBHI-1T'lik bir nihai konsantrasyon vermek için hücrelerle 1x PBS çözeltisine 1.4 μL konsantre bir boya stoğu (DMSO'da 50 mM DFHBI-1T) ekleyin. Ardından, kültür tüpü kapağını sabitleyin ve ardından ilk kez nokta okumayı yapmadan önce çözeltiyi eşit şekilde karıştırmak için kültür tüplerini 3x-5x ters çevirin.
NOT: E. coli için kültür tüpleri içindeki DMSO'nun toplam yüzdesi% 10'u geçmemelidir, çünkü bu hücre canlılığını etkileyebilir16. - Kapağı çıkarın ve kültür tüpünü numune tüp kaldırıcıya yerleştirin. Tutucuyu elle numune enjeksiyon iğnesine kaldırın ve uygun numune dosyasının altında Kaydet simgesini tıklatın. Ayrıca, deneme için Başlat'a basarak bir zamanlayıcı başlatın.
- Çalıştırma tamamlandıktan sonra tüp kaldırıcıyı elle indirin ve 2.3.5.1-2.3.5.2 arasındaki adımları tekrarlayın. (zamanlayıcı çalışırken) konsantre DFHBI-1T'nin 1.4 μL'sini ekleyerek, kültür tüplerini ters çevirerek ve kalan tüm biyolojik kopyalar için okumalar alarak. Bu ilk kayıtlar, tüm biyolojik kopyalar için 0 dakikada elde edilen okumalar olacaktır. Bunu her biyolojik replikasyon için birer birer yapın.
NOT: Kayıt akışı sitometrisi alımına basıldığı zamanı not edin. Deneme boyunca zaman noktaları için bu zaman dilimine bağlı kalın. - Numune tüp kaldırıcıdaki kültür tüplerini numune enjeksiyon iğnesine kaldırarak, uygun numune dosyasını seçerek, Kaydet'i tıklatarak ve her çalıştırma tamamlandıktan sonra kaldırıcıyı elle indirerek okumalar yapmaya devam edin. Test edilen tüm ek zaman noktaları ve biyolojik kopyalar için bunu yapın. Deneme tamamlanana kadar adımları tekrarlayın.
NOT: DFHBI-1T'nin çözelti içinde alüminyum folyo ile kaplanarak fotobeyazlatılmasını önlemek için numuneleri ışıktan uzak tutun.
- 50 μM DFBHI-1T'lik bir nihai konsantrasyon vermek için hücrelerle 1x PBS çözeltisine 1.4 μL konsantre bir boya stoğu (DMSO'da 50 mM DFHBI-1T) ekleyin. Ardından, kültür tüpü kapağını sabitleyin ve ardından ilk kez nokta okumayı yapmadan önce çözeltiyi eşit şekilde karıştırmak için kültür tüplerini 3x-5x ters çevirin.
- Boyasız hücreler için zaman noktalarında floresan ölçün (Kontrol).
- Üreticinin protokolünü takip eden negatif kontrollerle deneyi tekrar etmeden önce akış sitometresi için uygun temizleme protokollerini çalıştırın. Bu, önceki deneyden kontrol zaman noktası analizi deneyine geçişi en aza indirmek için yapılır. Aşağıda, deneyler arasındaki akış sitometresi için izlenen adımlar verilmiştir:
- Tüp kaldırıcıya boş bir kültür tüpünü elle yerleştirin, tüp tutucuyu kaldırın ve Cihaz sekmesindeki Kaydı Aç simgesine tıklayın. Bu, yapışkan numuneleri temizlemek için akışkan sistemde bir geri yıkama çalıştıracaktır. Tüp tutucuyu elle indirin ve Unclog dizisi tamamlandıktan sonra tüpü çıkarın.
- Yeni bir kültür tüpü ile,% 10'luk bir ağartıcı çözeltisinin 3 mL'sini ekleyin, kültür tüpünü tüp tutucuya yerleştirin ve tutucuyu elle numune enjeksiyon iğnesine kaldırın. Ek olarak, akış sitometresine uygunsa, otomatik numune alma cihazına temiz bir 96 delikli plaka yerleştirin.
- Akışkan sistemi boyunca %10 ağartıcı içeren bir temizleme dizisi çalıştırmak için SIP'i Sanitize Et /Otomatik Numune Alma Cihazı SIP'i Sanitize Et simgesine tıklayın. Temizleme sırasını tamamlamak için tüp tutucuyu indirin.
- Denetim zaman noktası çözümleme çalışması için örnek dosyaları adım 2.3.3'teki yönergeleri izleyerek ayarlayın.
- Yeni bir kültür tüpünde, 1.5 mL'lik 1x PBS çözeltisinde seyreltik bir hücre çözeltisi hazırlayın. Seyreltilmiş bir hücre çözeltisi oluşturmak için AI ortamındaki indüklenmiş hücrelerin 3 μL'sini 1x PBS çözeltisine ekleyin. Her biyolojik çoğaltma için bu adımı yineleyin.
- Kültür tüpüne hücrelerle 1x PBS çözeltisine birer birer 1,4 μL DMSO ekleyin ve aynı zaman noktalarını test edin. Kültür tüpü kapağını sabitleyin ve ardından ilk kez nokta okumayı yapmadan önce çözeltiyi eşit şekilde karıştırmak için kültür tüplerini 3x-5x ters çevirin. Bunu her biyolojik replikasyon için birer birer yapın.
- Kontrol hücrelerinin analizi için, 2.3.5.2-2.3.5.4 adımlarını kullanarak, boyalı hücrelerle aynı protokolü izleyin.
- Üreticinin protokolünü takip eden negatif kontrollerle deneyi tekrar etmeden önce akış sitometresi için uygun temizleme protokollerini çalıştırın. Bu, önceki deneyden kontrol zaman noktası analizi deneyine geçişi en aza indirmek için yapılır. Aşağıda, deneyler arasındaki akış sitometresi için izlenen adımlar verilmiştir:
- Akış sitometresini kapatın: Enstrümantasyonun düzgün bir şekilde kapatılması için üreticinin protokolünü izleyin. Akış sitometresi için, cihaz aşağıdaki şekilde kapatılmaya hazırlanır:
- 2.3.6.1.1-2.3.6.1.3 adımlarını izleyerek akış sitometresi için ilk temizleme protokolünü gerçekleştirin.
- Kültür tüpünü% 10 ağartıcı çözeltisi ile 3 mL odaklama sıvısı içeren bir kültür tüpü ile değiştirin. Tüp tutucuyu elle kaldırın ve Kapat simgesinin altında açılır menüye tıklayın ve Tamamlandı'yı seçin.
NOT: İsteğe bağlı duraklatma noktası.
- Akış sitometresini kurun.
- Akış sitometrisi verilerinin analizi
- Analiz için tüm FCS dosyalarını dışa aktarın. FCS dosyalarını bir akış sitometrisi analiz yazılımıyla açın.
- Yalnızca Hücre FCS dosyalarından birini kullanarak, ileri dağılım (FSC) ve yan dağılım (SSC) nokta grafiğinden (FSC-Alanı/SSC-Alanı) bir kapı oluşturun ve her iki günlük eksenini kullanarak herhangi bir sinyali enkazdan hariç tutun. Bu kapıyı oluşturmak için, akış sitometrisi analiz yazılımındaki AutoGate simgesine tıklayın ve Gate 1 olarak adlandırın. Aynı geçidi, veri işleme çalışma alanındaki Tüm Örnekler sekmesi altında test edilen tüm örneklere uygulayın. Bu, tüm FCS dosyalarının altındaki Kapı 1'in işlenmesine neden olur.
- Adım 2.4.2'de kullanılan Yalnızca hücre FCS dosyasını, çift tıklatarak yeni bir alt küme dosyası oluşturun. Eksen ayarlarını FSC-Alanı/FSC-Yüksekliği olarak değiştirin, her ikisi de günlük eksenlerini kullanır. Oval bir kapı oluşturmak için akış sitometrisi analiz yazılımındaki AutoGate simgesine tıklayın ve buna Kapı 2 adını verin. Bu kapıyı, test edilen tüm örneklere adım 2.4.2'de kapı kümesinin altında bir alt küme olarak uygulayın. Bu, tüm örneklerin her FCS dosyasıyla ilişkilendirilmiş Kapı 1 > Kapı 2'ye sahip olmasına neden olur.
- Adım 2.4.2 ve adım 2.4.3'te ayarlanan her iki geçidin de Kapı 2'yi çift tıklatarak uygulandığı başka bir alt küme dosyası oluşturun. Eksen ayarlarını FSC-Alanı/Histogram olarak değiştirin. Bu histogram geçidini test edilen tüm örneklere bir alt küme olarak uygulayın, böylece tüm örneklerde her FCS dosyasıyla ilişkilendirilmiş Kapı 1 > Kapı 2 > Kapı 2 bulunur.
NOT: Histogramlar, histogramların düzen penceresine taşınmasına yardımcı olmak ve veri işleme ile daha fazla organizasyon oluşturmak için Kapı 2'den Histogram'a yeniden adlandırılabilir. - Ortalama floresan yoğunluğu (MFI) değerlerini analiz etmek için düzen penceresini açın. Her zaman noktası için histogram geçitlerini tıklatıp mizanpaj penceresine sürükleyin.
- MFI sonuçlarını düzen penceresinde görüntülemek üzere test edilen her numune için "∑ Ortalaması: BL1-A" (GFP) için istatistiksel bir analiz gerçekleştirin.
- En az üç bağımsız biyolojik kopya için zaman noktası analizi başına MFI değerleri için standart sapmayı hesaplayın.
- Mizanpaj penceresini PDF dosyası olarak dışa aktararak her zaman noktası için histogram ve MFI değerlerini kaydedin.
NOT: İsteğe bağlı duraklatma noktası.
- Grafik akışı sitometri verileri
- Her zaman noktası için histogram ve MFI değerlerini içeren PDF dosyasını açın. MFI değerleri bir veri analiz yazılımına kopyalanır. Veri grafik yazılımında, XY biçiminde yeni bir veri tablosu oluşturun.
- Aşağıdakilerin seçili olduğu bir XY Tablosu oluşturmak için seçin:
Veri Tablosu: Yeni bir tabloya veri girme veya alma
Seçenekler:
X: Geçen Süreler
Y: Yan yana alt sütunlara (üç ila dört) çoğaltma değerleri girin - X ekseninde, deneme ve kontrol çalıştırmaları için tüm zaman noktalarını etiketleyin.
- Grup A'da, boya eklenmiş hücrelerin floresan analizi için tüm biyolojik replikalar için MFI değerlerini her zaman noktasına girin.
- Grup B'de, boya (DMSO) eklenmemiş hücrelerin analizi için tüm biyolojik replikaların MFI değerlerini her zaman noktasına girin.
- Sonuçları gözlemlemek için, Grafikler sekmesinin altındaki [Veri Kümesi Adı Ekle] öğesine tıklayın. Bu, veri noktalarını, her zaman noktasında standart sapmayı (s.d.) temsil eden hata çubuklarıyla birlikte araç olarak görüntüler. X ekseni geçen süreyi, Y ekseni ise MFI değerlerini temsil eder.
- Aşağıdakilerin seçili olduğu bir XY Tablosu oluşturmak için seçin:
- Her zaman noktası için histogram ve MFI değerlerini içeren PDF dosyasını açın. MFI değerleri bir veri analiz yazılımına kopyalanır. Veri grafik yazılımında, XY biçiminde yeni bir veri tablosu oluşturun.
Sonuçlar
In vitro kinetik
Sentetik oligonükleotidler olarak satın alınan DNA şablonlarının ve primerlerinin dizileri Tablo 2'de, reaktif tarifleri ise Ek Dosya 1'de gösterilmiştir. PCR amplifikasyonu, sonraki in vitro transkripsiyon (IVT) reaksiyonu için gerekli olan T7 promotörü ile DNA şablonunun miktarını artırmak için kullanılır. Ek olarak, PCR amplifikasyonu aynı reaksiyonda iki amaç için kullanılabilir: primer uzantısı ile tam uzunlukta Brokoli DNA şablonunu üretmek ve DNA şablonunu büyütmek.
RNA'yı sentezlemek için IVT reaksiyonundan sonra, PAGE saflaştırması, istenmeyen kesilmiş transkriptleri, bozulmuş ürünleri ve reaksiyona girmemiş rNTP'leri tam uzunluktaki RNA ürününden çıkaracaktır. Bu tür saflaştırma tercih edilir, çünkü kesilmiş veya bozulmuş RNA'lar RNA konsantrasyonlarının yanlış belirlenmesine neden olur. Nükleotid bazları UV ışığını emdiğinden, jel üzerindeki RNA bantları ve rNTP'ler UV altında floresan TLC plakasına karşı gölgeler olarak görselleştirilebilir. Böylece, doğru ürün boyutuna karşılık gelen bantlar seçici olarak çıkarılabilir.
En yakın komşu yöntemi, yok olma katsayılarını ve dolayısıyla yapılandırılmış RNA'ların konsantrasyonlarını abartır, çünkü baz eşleşmesi17 nedeniyle hipokromisiteyi hesaba katmaz. Bu nedenle, doğru RNA konsantrasyonlarını belirlemek için, RNA'yı bireysel NMP'lere hidrolize etmek için nötr pH termal hidroliz testleri yapılmıştır18. Doğru yok olma katsayısı, RNA dizisindeki NMP'lerin yok olma katsayılarının toplamı olarak hesaplandı.
DFHBI'nin Ispanak2 ve Brokoli'ye bağlanmasının kinetiği, bir plaka okuyucu testi kullanılarak belirlendi. RNA ilk önce boya bağlama için doğru konformasyonda olmasını sağlamak için yeniden denatüre edildi. Plaka okuyucu kinetik testi için reaksiyon koşulları 40 mM HEPES, pH 7.5 (KOH), 125 mM KCl, 3 mM MgCl2, 100 nM renatüre RNA ve 10 μM DFHBI'den oluşuyordu ve reaksiyon 37 ° C'de ölçüldü. MgCl 2'nin bu sıcaklığı ve konsantrasyonu, fizyolojik koşulları 19 taklit etmek için seçildi, ancak 28 ° C ve10 mM MgCl2 koşulları da gelişmiş aptamer katlama için kullanılabilir.
Hem florojenik aptamers Ispanak2 hem de Brokoli, DFHBI boyasına bağlanmak için iki fazlı ilişki kinetiği gösterir (Şekil 2). Kinetik veriler, her iki aptamer için bir fazlı ilişkiden ziyade iki fazlı ilişki ile daha iyi uyuyordu (Ek Şekil 1). Hızlı ve yavaş çağrışımlar için oran sabitleri ve t1/2 değerleri en uygun eğri ile belirlenmiştir (Tablo 3). Floresan açma büyüklüğünün yüzde kaçının daha hızlı DFHBI bağlayıcı RNA popülasyonu tarafından hesaplandığını açıklayan PercentFast da belirlendi.
Bağlanma yetkin durumdaki ıspanak2, Brokoli'den daha hızlı açılma gösterir (t 1/2 = 1.2 s'ye karşı2.0 s). Her iki aptamer için ikinci faz kinetiği benzerdir (t1/2 = 180 s) ve muhtemelen numunenin bir alt popülasyonu için ortak bir hız sınırlayıcı adıma karşılık gelir (PercentFast = Ispanak2 ve Brokoli için sırasıyla% 68 ve% 60). Genel olarak, bu sonuçlar, iyi katlanmış Ispanak2 ve Brokoli aptamerlerinin çok hızlı açılma kinetik sergilediğini ve ilk yarı-maksimum dönüşün boya ilavesinden 1-2 s içinde olduğunu göstermektedir.
Hücresel kinetik
pET31b plazmidine klonlanan DNA yapılarının dizileri Tablo 2'de gösterilmiştir ve reaktif tarifleri Ek Dosya 1'de gösterilmiştir. Florojenik RNA aptamerlerinin DNA yapıları tipik olarak hücresel deneyler için bir tRNA iskelesi içinde bulunur. BL21 Star (DE3) E. coli suşu, RNA stabilitesini artıran RNaz E'de mutasyona sahip bir ekspresyon suşudur.
Floresan zaman noktası ölçümleri ilk 45 dakika boyunca her 5 dakikada bir kaydedildi, ardından 1 saat, 1,5 saat ve 2 saatte bir okumalar yapıldı ve toplam 12 zaman noktası artı yalnızca hücre okuması verildi. En kısa zaman aralığının 5 dakika olması, her zaman noktasında birden fazla biyolojik replikanın ölçülmesine izin verdi ve 30 s ila 1 dakikalık replikasyon ölçümleri arasında düzenli boşluk bırakıldı. Zaman kursu deneyi için kullanılan 1x PBS çözeltisinde seyreltilen hücrelerin toplam hacmi 1.5 mL idi.
Hücreler, tek hücre popülasyonunun ortalama floresan yoğunluğunu (MFI) belirlemeden önce kapılıydı. Gateing, analiz edilecek hücre popülasyonunu belirlemek için dağılım grafiğinde bir alan seçer. Bu işlem, herhangi bir enkaz veya çarpma okumasının analize dahil edilmesini önler. Gösterilen akış sitometrisi analizi için, 30.000 olay kaydedildi ve bu da gating'den sonra analiz edilen 10.000-20.000 olayla sonuçlandı.
Hücresel floresan kinetiği, hem E. coli'ye boya difüzyonunun hem de hücresel ortamdaki RNA aptamerine boya bağlayıcı kinetiğin bir fonksiyonudur (Şekil 1B). Ispanak2-tRNA eksprese eden hücreler için, boya ilavesi ile numune analizi arasındaki kısa zaman gecikmesi (saniye cinsinden) nedeniyle ortalama floresan yoğunluğunun (MFI) "0" zaman noktasında hemen arttığı gözlenmiştir (Şekil 3A). Ayrıca, hücresel floresan, 5 dakikalık ilk zaman noktasında maksimum dengeli MFI değerine (40.441 ± 990) ulaşmıştır. Buna karşılık, kontrol hücreleri düşük arka plan floresansı (416) gösterir ve DMSO ilavesi ile MFI değerinde değişiklik olmaz (Şekil 3B). Boyalı hücreler ile boyasız hücreler arasındaki bir karşılaştırma, floresan aktivasyonunun hücrelerde 98 kat ± 2 olduğunu ortaya koymaktadır. Genel olarak, bu sonuçlar, E. coli hücrelerinde eksprese edilen Ispanak2-tRNA'nın, boya ilavesinden 5 dakikadan daha kısa bir süre içinde maksimum açılma ile hızlı açılma kinetik sergilediğini göstermektedir.
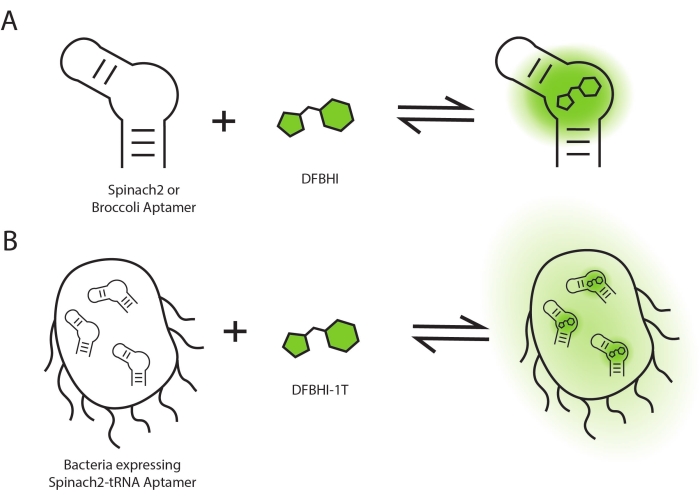
Şekil 1: Floresan aktivasyonunun şeması. Floresan aktivasyonu, hücrelerdeki boya moleküllerine (A) in vitro ve (B) bağlanan RNA aptamers üzerinde meydana gelir. Bu şeklin daha büyük bir versiyonunu görüntülemek için lütfen buraya tıklayın.

Resim 2: Florojenik aptamerlerin in vitro kinetiği. Florojenik aptamers (A, B) Ispanak2 veya (C, D) Brokolinin temsili in vitro kinetiği, iki fazlı ilişki ile modellenmiştir, t = 0 s, DFHBI ilavesinin zaman noktasıdır (son DFHBI konsantrasyonu: 10 μM). Deneyler üçlü olarak yapıldı. Tüm hata çubukları ortalamadan standart sapmaları temsil eder. Uyumdan, hem hızlı hem de yavaş ilişkilendirme reaksiyonu bileşenleri için t1/2 değerleri elde edildi. Bu şeklin daha büyük bir versiyonunu görüntülemek için lütfen buraya tıklayın.
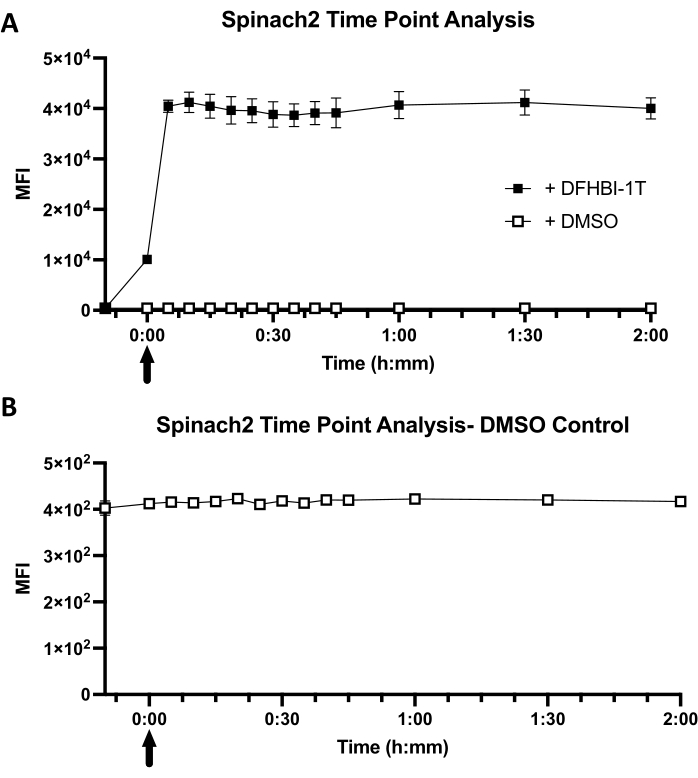
Şekil 3: Bir tRNA iskelesinde Ispanak2'nin temsili hücresel kinetiği . (A) 2 saat boyunca tRNA-Ispanak2 boya alımının zaman noktası analizi. DFHIB-1T veya DMSO'ya eklenmeden önce yalnızca hücre taban çizgisi alındı. Zaman noktaları ilk 45 dakika boyunca her 5 dakikada bir alındı, ardından 1 saat, 1,5 saat ve 2 saatte bir zaman noktası okuması yapıldı. Ok, DFHBI-1T veya DMSO'nun BL21 Yıldız hücrelerle 1x PBS çözümüne ne zaman eklendiğini temsil eder. Analiz için DFHBI-1T'nin son konsantrasyonu 50 μM'dir. DMSO kontrolü için, DMSO ilavesi, DFHBI-1T boya ilavesi için kullanılan eşit hacimde (1.4 μL) idi. (B) BL21 Star E. coli hücreleri ile DMSO kontrol zaman noktası analizinin yakın çekimi . Ortalama floresan yoğunluğu (MFI), boya veya DMSO ile BL21 Star hücrelerinin genel floresan okumasını gösterir. Veriler, üç biyolojik replikanın ortalama ± standart sapmasını temsil etmektedir. Bu şeklin daha büyük bir versiyonunu görüntülemek için lütfen buraya tıklayın.
| Aptamer-Boya Çifti | Uzunluk (nt) | Maksimum abs (nm) | Max em (nm) | Yok olma katsayısı (M-1· cm-1) | Kuantum verimi | Parlaklık | Kd (nm) | Tm (°C) | Referans |
| Ispanak2-DFHBI | 95 | 445 | 501 | 26100 | 0.7 | 63 | 1450 | 37 | 4 |
| Ispanak2-DFHBI-1T | 95 | 482 | 505 | 31000 | 0.94 | 100 | 560 | 37 | 13, 14 |
| Brokoli-DFHBI-1T | 49 | 472 | 507 | 29600 | 0.94 | 96 | 360 | 48 | 13 |
Tablo 1: Ispanak2-DFHBI4, Ispanak2-DFHBI-1T 13,14 ve Brokoli-DFHBI-1T 13'ün daha önce yayınlanmış fotofiziksel ve biyokimyasal özellikleri.
| Ispanak2 + T7 promotör | 5'-CGATCCCGCGAAATTAATACGACTCACTATAGGATGTAACTGAATGAAATGGTGAA GGACGGGTCCAGTAGGCTGCTTCGGCAGCCTACTTGTTGAGTAGAGTGTGAGCTCC GTAACTAGTTACATC-3' | ||
| Brokoli + T7 promotör | 5'-CGATCCCGCGAAATTAATACGACTCACTATAGgagacggtcgg gtccagatattcgtatctgtcgagtagagtgtgggctc-3' | ||
| tRNA-Ispanak2 yapısı (pET31b plazmidinde) | 5'-CGATCCCGCGAAATTAATACGACTCACTATAGGGGCCCGGATAGCTCAGTCGGT AGAGCAGCGGCCGGATGTAACTGAATGAAATGGTGAAGGACGGGTCCAGTAGGCT GCTTCGGCAGCCTACTTGTTGAGTAGAGTGAGCTCCGTAACTAGTTACATCcesaret CCGCGGGTCCAGGGTTCAAGTCCCTGTTCGGGCGCCA TAGCATAACCCCTTGGGGCCTCTAAACGGGTCTTGAGGGGTTTTTTG3' | ||
| Ispanak2 İleri Astar | 5'-CGATCCCGCGAAATTAATACGACTCACTATAG-3' | ||
| Ispanak2 Ters Astar | 5'-GATGTAACTAGTTACGGAGC-3' | ||
| Brokoli İleri Astar | 5'-CGATCCCGCGAAATTAATACGACTCACTATAGgagacggtcgggtccagatattcgtatctg-3' | ||
| Brokoli Ters Astar | 5'-gagcccacactctcgacagatacgaatctggacccgaccgtctc-3' | ||
Tablo 2: İn vitro ve hücresel kinetik çalışmalar için kullanılan DNA dizilerini ve primerleri içeren DNA dizi tablosu. Bold= T7 destekçisi; Altı çizili = tRNA iskelesi; Caps = Ispanak2; küçük harf = Brokoli; Kalın italik = T7 sonlandırıcı.
| Aptamer | Hızlı t1/2 (ler) | Yavaş t1/2 (ler) | KHızlı (s-1) | KYavaş (s-1) | Yüzde Hızlı |
| Ispanak2 | 1.2 ± 0.2 | 180 ± 10 | 0.56 ± 0.07 | 0.0039 ± 0.0002 | 68 ± 5 |
| Brokoli | 2.0 ± 0.2 | 180 ± 30 | 0.35 ± 0.05 | 0.0039 ± 0.0006 | 60 ± 3 |
Tablo 3: Takılan verilerden elde edilen Ispanak2 ve Brokoli aptamerlerinin in vitro kinetiği değerleri. Veriler, üç replikanın ortalama ± standart sapması olarak raporlanır.
Ek Şekil 1: Florojenik aptamerlerin in vitro kinetiğini temsil eder. Florojenik aptamerlerin (A,C) Ispanak2 veya (B,D) Brokolinin temsili in vitro kinetiği, DFHBI ilavesinin zaman noktasını gösteren oklarla (A,B) 600 s veya (C,D) 20 s ölçüm zamanlarında tek fazlı ilişki ile modellenmiştir (son DFHBI konsantrasyonu: 10 μM). Deneyler üçlü olarak yapıldı. Genel olarak, bu veriler, floresan sinyali daha uzun bir süre boyunca izlendiğinde iki fazlı bir ilişkilendirme modeline göre tek fazlı bir ilişkilendirme modeline göre daha az uygundur. Bu Dosyayı indirmek için lütfen tıklayınız.
Ek Dosya 1: In vitro kinetik deneyi için tarifler. Bu Dosyayı indirmek için lütfen tıklayınız.
Tartışmalar
In vitro kinetik deneyi için, aynı genel protokol, hem ligand bağlayıcı hem de florofor bağlayıcı alan8 içeren RNA tabanlı bir floresan biyosensörün in vitro kinetiğini ölçmek için değiştirilebilir. Bu durumda, RNA, ligand yanıt kinetiği elde etmek için ligand enjekte edildikten sonra ölçümlerden önce florofor ile inkübe edilmelidir. Replikalar arasında yüksek değişkenlik gözlenirse, her numunenin ölçümden önce 96 delikli plakada aynı süre boyunca dengelenmesine izin verilip verilmediğini kontrol ederek sorun giderilebilir. Her numune veya replikasyon, tüm numuneleri bir kerede hazırlamak yerine, bir kuyucukta ayrı ayrı hazırlanmalı ve 15 dakikalık dengeleme adımından hemen sonra ölçülmelidir.
Hücresel kinetik deneyi için, protokol daha kısa veya daha uzun süreli kurslar için değiştirilebilir, ancak biyolojik replikasyonların sayısını planlamak ve gerekli hücre çözeltisi hacmini ayarlamak çok önemlidir. Adımları dikkatli bir şekilde gerçekleştirmek için yeterli zamana sahip olmak için her biyolojik replikasyon okumasının 30 s ila 1 dakika arasında bir aralıkta bırakılması önerilir. Başka bir modifikasyon, RNA aptamerini alternatif bir negatif kontrol olarak bağlamayan farklı bir florojenik boyayı test etmektir, bu da arka plan üzerinde floresan aktivasyonu göstermemelidir. Tutarsız sonuçlar gözlenirse, bir önceki çalıştırmadan diğerine hücrelerin veya boyaların taşmasını önlemek için üreticinin farklı deneysel çalışmalar arasındaki protokolünü takiben akış sitometresinin uygun şekilde temizlendiğini kontrol ederek sorun giderilebilir.
Sunulan in vitro yöntem, florojenik aptamerler veya RNA bazlı florojenik biyosensörler arasındaki kinetiği karşılaştırmak için yararlı olsa da, elde edilen kinetik değerler sıcaklığa, magnezyum konsantrasyonuna veya kullanılan diğer tampon bileşenlerine bağlı olarak değişebilir. Ayrıca, bu yöntem daha önce farklı florojenik RNA sistemlerini karakterize etmek için kullanılan iyi tanımlanmış koşullar sağlarken, diğer biyolojik makromoleküllerin varlığı nedeniyle hücre içi ortam mükemmel bir şekilde temsil edilemez.
Programlanabilir bir enjektör ile donatılmış floresan plaka okuyucunun veri toplama için ölü zamanı yokken, akış sitometresi cihazı gözlemlenebilir ölü zaman nedeniyle sınırlı zamansal doğruluğa sahiptir. "Kaydet" düğmesine tıklandığında ve veri toplama başladığında ~ 5 sn gecikme vardır. Cihazın 30.000 olayı ölçmesi için ek bir ~ 5 s gecikme meydana gelir; Bu örnek edinme süresi, hücrelerin 1x PBS'de ne kadar seyreltik olduğuna bağlı olarak biraz değişecektir.
Hücresel deneylerdeki bir diğer potansiyel sınırlama, 1x PBS'deki hücre canlılığıdır. Uzatılmış zaman noktası analizleri için, ölü hücreleri boyamak için propidyum iyodür kullanılarak hücre canlılığı kontrol edilebilir20. Boya agregasyonu, akış sitometresi tarafından yapılan floresan ölçümlerinin doğruluğunu da sınırlayabilir. Sulu çözeltilerde çözünürlüğü çok sınırlı olan boyalar birikebilir ve akış sitometresinde hücre olarak sayılabilecek kadar büyük parçacıklar olarak görünebilir. Bu nedenle, kapılı bölgedeki agregaları kontrol etmek için yalnızca boya deneysel kontrolleri çalıştırmak önemlidir.
Daha önce, Brokolinin 1 mM Mg 2 + in vitro'da Ispanak2 ile karşılaştırılabilir parlaklığa sahip olduğu gösterilmiştir, ancak Brokoli-tRNA, Ispanak2-tRNA13'e kıyasla canlı E. coli'de ~ iki kat daha fazla floresan yoğunluğu sergiler. Bildiğimiz kadarıyla, Ispanak2 ve Brokoli florojenik aptamerler için boya bağlayıcı kinetikler daha önce karşılaştırılmamış ve iki fazlı bir ilişki ile modellenmemiştir. Her iki RNA aptamer için başlangıçtaki hızlı hız sabitleri, boya bağlama cebinin önceden katlandığını ve boyanın bağlanması için yapısal bir değişikliğe gerek olmadığını destekler, bu da X-ışını kristalografisi ve UV eritme deneyleri21,22 ile tutarlıdır. Daha yavaş bir oran sabitine sahip ikinci aşama daha önce bildirilmemiştir, çünkü durdurulmuş akış ve floresan ömrü ölçümleri gibi diğer deneyler Ispanak aptamerini daha kısa bir süre (20 s ve 300 s) 23,24 için analiz etmiştir. Çok daha yavaş olan ikinci faz, veriler 600 sn için analiz edildiğinde floresanda gözlenen biüstel bir artışla sonuçlanır. Bu yavaş adım, bağlanma yetersizliğinden bağlanma yetkin RNA durumuna kadar hız sınırlayıcı bir yeniden katlama adımına veya bağlı boyanın trans formlarından cis formlarına hız sınırlayıcı bir fotodönüşüm adımına bağlanabilir. İkinci mekanizma daha önce biüstel bir floresan profili24 vermek üzere modellenmişti ve ilgili aptamer Baby Spanach25 üzerindeki absorpsiyon ve uyarma spektrumları arasındaki farkın yakın tarihli bir analizi ile desteklenmiştir.
İn vitro bulguların genel önemi, florojenik RNA aptamer ile boya ilişkisinin gerçek zamanlı RNA lokalizasyonunu ve gen ekspresyon çalışmalarını sınırlamadığını göstermeleridir. Ispanak2 kullanan RNA tabanlı floresan biyosensörler için, ölçülen açma kinetiği, burada ölçülen ikinci faz kinetiğine benzerdir10 , çünkü biyosensörler yeniden katlanma adımı gerektirir ve bu nedenle, neredeyse gerçek zamanlı sinyalizasyon çalışmalarını mümkün kılmak için yeterince hızlı olmalıdır.
Hücresel kinetiğin, Ispanak2 için gözlenen in vitro kinetiğinden farklı olması bekleniyordu. Önemli bir fark, dış ve iç zarları geçmeyi içeren E. coli hücrelerine boya difüzyonunun ek bir adımının olmasıdır. Ek olarak, hücresel ortam, moleküler kalabalıklaşma, iyon bileşimi ve konsantrasyonlarının yanı sıra RNA ve boya konsantrasyonları açısından boya-aptamer ilişkisi için farklı koşullar ortaya koymaktadır.
Sonuçların genel önemi, hücresel floresanın 5 dakikadan daha kısa sürede maksimum sinyale ulaşması ve en az 2 saat boyunca sabit kalmasıdır, bu da bu zaman aralığında gerçek zamanlı RNA lokalizasyonu ve gen ekspresyon çalışmalarına olanak tanır. Ispanak2 kullanan RNA tabanlı bir biyosensör için, daha önce ligand ilavesinden sonraki 4-5 dakika içinde önemli bir floresan tepkisinin gözlenebileceğini, ancak maksimum sinyale ulaşmanın daha uzun sürdüğünü (15-30 dakika)8 göstermiştik. Birlikte ele alındığında, bu bulgular hücrelere boya difüzyonunun, RNA tabanlı biyosensörlerle yapılan in vivo deneyler için pratik hız sınırlayıcı adım olmadığını göstermektedir. Son olarak, bu deneysel protokol hücrelerdeki diğer florojenik RNA sistemlerini analiz etmek için uygulanabilir.
Burada sunulan deneysel protokoller diğer florojenik RNA sistemlerini analiz etmek için uygulanabilir. Bu çalışmada analiz edilen iki aptamer olan Ispanak2 ve Brokoli'nin ötesinde, farklı emisyon profilleri, gelişmiş fotostabilite, daha sıkı bağlanma afiniteleri ve floroforları değiştirme yeteneği sağlayan diğer florojenik RNA sistemleri geliştirilmiştir (yakın zamanda gözden geçirilmiştir1). Floresan özelliklerine ek olarak, bu sistemler için in vitro ve in cells için açma kinetiğinin karşılaştırılması, farklı hücre biyolojik uygulamalarına uygunluklarını değerlendirmek için önemlidir. Sonuçlar ayrıca aptamerin yapısal ön katlanmasını veya yeniden düzenlenmesini de destekleyebilir. Tartışıldığı gibi, bazı modifikasyonlarla, bu protokoller RNA tabanlı biyosensörleri analiz etmek için de uygulanmıştır8.
Açıklamalar
Yazarlar çıkar çatışması olmadığını beyan ederler.
Teşekkürler
Bu çalışma MCH'YE aşağıdaki hibelerle desteklenmiştir: NSF-BSF 1815508 ve NIH R01 GM124589. MRM, eğitim hibesi NIH T32 GM122740 tarafından kısmen desteklenmiştir.
Malzemeler
| Name | Company | Catalog Number | Comments |
| Agarose | Thermo Fischer Scientific | BP160500 | |
| Agarose gel electrophoresis equipment | Thermo Fischer Scientific | B1A-BP | |
| Alpha D-(+)-lactose monohydrate | Thermo Fischer Scientific | 18-600-440 | |
| Amber 1.5 mL microcentrifuge tubes | Thermo Fischer Scientific | 22431021 | |
| Ammonium persulfate (APS) | Sigma-Aldrich | A3678 | |
| Ammonium sulfate ((NH4)2SO4) | Sigma-Aldrich | A4418 | |
| Attune NxT Flow cytometer | Thermo Fischer Scientific | A24861 | |
| Attune 1x Focusing Fluid | Thermo Fischer Scientific | A24904 | |
| Attune Shutdown Solution | Thermo Fischer Scientific | A24975 | |
| Attune Performance Tracking Beads | Thermo Fischer Scientific | 4449754 | |
| Attune Wash Solution | Thermo Fischer Scientific | J24974 | |
| Boric acid | Sigma-Aldrich | B6768 | |
| Bromophenol blue | Sigma-Aldrich | B0126 | |
| Carbenicillin disodium salt | Sigma-Aldrich | C3416 | |
| Chlorine Bleach | Amazon | B07J6FJR8D | |
| Corning Costar 96-well plate | Daigger Scientific | EF86610A | |
| Culture Tubes, 12 mm x 75 mm, 5 mL with attached dual position cap | Globe Scientific | 05-402-31 | |
| DFHBI | Sigma-Aldrich | SML1627 | |
| DFHBI-1T | Sigma-Aldrich | SML2697 | |
| D-Glucose (anhydrous) | Acros Organics | AC410955000 | |
| Dimethyl sulfoxide (DMSO) | Sigma-Aldrich | D8418 | |
| Dithiothreitol (DTT) | Sigma-Aldrich | DTT-RO | |
| DNA loading dye | New England Biolabs | B7025S | |
| DNA LoBind Tubes (2.0 mL) | Eppendorf | 22431048 | |
| dNTPs: dATP, dCTP, dGTP, dTTP | New England Biolabs | N0446S | |
| EDTA, pH 8.0 | Gibco, Life Technologies | AM9260G | |
| Ethanol (EtOH) | Sigma-Aldrich | E7023 | |
| Filter-tip micropipettor tips | Thermo Fischer Scientific | AM12635, AM12648, AM12655, AM12665 | |
| FlowJo Software | BD Biosciences | N/A | FlowJo v10 Software |
| Fluorescent plate reader with heating control | VWR | 10014-924 | |
| Gel electrophoresis power supply | Thermo Fischer Scientific | EC3000XL2 | |
| Glycerol | Sigma-Aldrich | G5516 | |
| Glycogen AM95010 | Thermo Fischer Scientific | AM95010 | |
| GraphPad Prism | Dotmatics | N/A | Analysis software from Academic Group License |
| Heat block | Thomas Scientific | 1159Z11 | |
| HEPES | Sigma-Aldrich | H-4034 | |
| Inorganic pyrophosphatase | Sigma-Aldrich | I1643-500UN | |
| Low Molecular Weight DNA Ladder | New England Biolabs | N3233L | Supplied with free vial of Gel Loading Dye, Purple (6x), no SDS (NEB #B7025). |
| Magnesium chloride hexahydrate (MgCl2) | Sigma-Aldrich | M2670 | |
| Magnesium sulfate (MgSO4) | Fisher Scientific | MFCD00011110 | |
| Microcentrifuge tubes (1.5 mL) | Eppendorf | 22363204 | |
| Microcentrifuge with temperature control | Marshall Scientific | EP-5415R | |
| Micropipettors | Gilson | FA10001M, FA10003M, FA10005M, FA10006M | |
| Micropipettor tips | Sigma-Aldrich | Z369004, AXYT200CR, AXYT1000CR | |
| Millipore water filter with BioPak unit | Sigma-Aldrich | CDUFBI001, ZRQSVR3WW | |
| Narrow micropipettor pipette tips | DOT Scientific | RN005R-LRS | |
| PBS, 10x | Thermo Fischer Scientific | BP39920 | |
| PCR clean-up kit | Qiagen | 28181 | |
| PCR primers and templates | Integrated DNA technologies | ||
| PCR thermocycler for thin-walled PCR tubes | Bio-Rad | 1851148 | |
| PCR thermocycler for 0.5 mL tubes | Techne | 5PRIME/C | |
| pET31b-T7-Spinach2 Plasmid | Addgene | Plasmid #79783 | |
| Phusion High-Fidelity DNA polymerase | New England Biolabs | M0530L | Purchase of Phusion High-Fideldity Enzyme is supplied with 5x Phusion HF Buffer, 5x Phusion GC Buffer, and MgCl2 and DMSO solutions. |
| Polyacrylamide gel electrophoresis gel comb, C.B.S. Scientific | C.B.S. Scientific | VGC-1508 | |
| Polyacrylamide gel electrophoresis equipment | C.B.S. Scientific | ASG-250 | |
| Potassium chloride (KCl) | Sigma-Aldrich | P9333 | |
| Potassium phosphate monobasic | Sigma-Aldrich | P5655 | |
| Razor blades | Genesee Scientific | 38-101 | |
| rNTPs: ATP, CTP, GTP, UTP | New England Biolabs | N0450L | |
| SDS | Sigma-Aldrich | L3771 | |
| Short wave UV light source | Thermo Fischer Scientific | 11758221 | |
| Sodium carbonate (Na2CO3) | Sigma-Aldrich | S7795 | |
| Sodium chloride (NaCl) | Sigma-Aldrich | S7653 | |
| Sodium hydroxide (NaOH) | Sigma-Aldrich | S8045 | |
| Sodium phosphate dibasic, anhydrous | Thermo Fischer Scientific | S375-500 | |
| SoftMax Pro | Molecular Devices | N/A | SoftMax Pro 6.5.1 (platereader software) obtained through Academic Group License |
| Sterile filter units | Thermo Fischer Scientific | 09-741-88 | |
| Sucrose | Sigma-Aldrich | S0389 | |
| SYBR Safe DNA gel stain | Thermo Fischer Scientific | S33102 | |
| TAE buffer for agarose gel electrophoresis | Thermo Fischer Scientific | AM9869 | |
| Tetramethylethylenediamine (TEMED) | Sigma-Aldrich | T9281 | |
| Tris base | Sigma-Aldrich | TRIS-RO | |
| Tryptone (granulated) | Thermo Fischer Scientific | M0251S | |
| T7 RNA polymerase | New England Biolabs | M0251S | |
| Urea-PAGE Gel system | National Diagnostics | EC-833 | |
| UV fluorescent TLC plate | Sigma-Aldrich | 1.05789.0001 | |
| UV/Vis spectrophotometer | Thermo Fischer Scientific | ND-8000-GL | |
| Vortex mixer | Thermo Fischer Scientific | 2215415 | |
| Xylene cyanol | Sigma-Aldrich | X4126 | |
| Yeast Extract (Granulated) | Thermo Fischer Scientific | BP9727-2 |
Referanslar
- Su, Y., Hammond, M. C. RNA-based fluorescent biosensors for live cell imaging of small molecules and RNAs. Current Opinion in Biotechnology. 63, 157-166 (2020).
- Zhang, J., et al. Tandem spinach array for mRNA Imaging in living bacterial cells. Scientific Reports. 5, 17295(2015).
- Wang, Z., et al. In spatial complementation of aptamer-mediated recognition enables live-cell imaging of native RNA transcripts in real time. Angewandte Chemie. 57 (4), 972-976 (2018).
- Strack, R. L., Disney, M. D., Jaffrey, S. R. A superfolding Spinach2 reveals the dynamic nature of trinucleotide repeat-containing RNA. Nature Methods. 10 (12), 1219-1224 (2013).
- Thavarajah, W., et al. Point-of-use detection of environmental fluoride via a cell-free riboswitch-based biosensor. ACS Synthetic Biology. 9 (1), 10-18 (2020).
- You, M., Litke, J. L., Jaffrey, S. R. Imaging metabolite dynamics in living cells using a Spinach-based riboswitch. Proceedings of the National Academy of Sciences of the United States of America. 112 (21), 2756-2765 (2015).
- Kellenberger, C. A., Wilson, S. C., Sales-Lee, J., Hammond, M. C. RNA-based fluorescent biosensors for live cell imaging of second messengers cyclic di-GMP and cyclic AMP-GMP. Journal of the American Chemical Society. 135 (13), 4906-4909 (2013).
- Manna, S., Truong, J., Hammond, M. C. Guanidine biosensors enable comparison of cellular turn-on kinetics of riboswitch-based biosensor and reporter. ACS Synthetic Biology. 10 (3), 566-578 (2021).
- Bose, D., Su, Y., Marcus, A., Raulet, D. H., Hammond, M. C. An RNA-based fluorescent biosensor for high-throughput analysis of the cGAS-cGAMP-STING pathway. Cell Chemical Biology. 23 (12), 1539-1549 (2016).
- Wang, X. C., Wilson, S. C., Hammond, M. C. Next-generation RNA-based fluorescent biosensors enable anaerobic detection of cyclic di-GMP. Nucleic Acids Research. 44 (17), 139(2016).
- Paige, J. S., Thinh, N. -D., Wenjiao, S., Jaffrey, S. R. Fluorescence imaging of cellular metabolites with RNA. Science. 335 (6073), 1194(2012).
- Paige, J. S., Wu, K. Y., Jaffrey, S. R. RNA mimics of green fluorescent protein. Science. 333 (6042), 642-646 (2011).
- Filonov, G. S., Moon, J. D., Svensen, N., Jaffrey, S. R. Broccoli: Rapid selection of an RNA mimic of green fluorescent protein by fluorescence-based selection and directed evolution. Journal of the American Chemical Society. 136 (46), 16299-16308 (2014).
- Song, W., Strack, R. L., Svensen, N., Jaffrey, S. R. Plug-and-play fluorophores extend the spectral properties of spinach. Journal of the American Chemical Society. 136 (4), 1198-1201 (2014).
- Sambrook, J., Fritsch, E., Maniatis, T. Molecular Cloning: A Laboratory Manual. , Cold Spring Harbor Laboratory Press. Cold Spring Harbor, NY. (1989).
- Basch, H., Gadebusch, H. H. In vitro antimicrobial activity of dimethylsulfoxide. Applied Microbiology. 16 (12), 1953-1954 (1968).
- Kallansrud, G., Ward, B. A comparison of measured and calculated single- and double-stranded oligodeoxynucleotide extinction coefficients. Analytical Biochemistry. 236 (1), 134-138 (1996).
- Wilson, S. C., Cohen, D. T., Wang, X. C., Hammond, M. C. A neutral pH thermal hydrolysis method for quantification of structured RNAs. RNA. 20 (7), 1153-1160 (2014).
- Szatmári, D., et al. Intracellular ion concentrations and cation-dependent remodelling of bacterial MreB assemblies. Scientific Reports. 10, 12002(2020).
- Boulos, L., Prévost, M., Barbeau, B., Coallier, J., Desjardins, R. LIVE/DEAD® BacLightTM: Application of a new rapid staining method for direct enumeration of viable and total bacteria in drinking water. Journal of Microbiological Methods. 37 (1), 77-86 (1999).
- Huang, H., et al. A G-quadruplex-containing RNA activates fluorescence in a GFP-like fluorophore. Nature Chemical Biology. 10 (8), 686-691 (2014).
- Jeng, S. C. Y., Chan, H. H. Y., Booy, E. P., McKenna, S. A., Unrau, P. J. Fluorophore ligand binding and complex stabilization of the RNA Mango and RNA Spinach aptamers. RNA. 22 (12), 1884-1892 (2016).
- Han, K. Y., Leslie, B. J., Fei, J., Zhang, J., Ha, T. Understanding the photophysics of the Spinach-DFHBI RNA aptamer-fluorogen complex to improve live-cell RNA imaging. Journal of the American Chemical Society. 135 (50), 19033-19038 (2013).
- Wang, P., et al. Photochemical properties of Spinach and its use in selective imaging. Chemical Science. 4 (7), 2865-2873 (2013).
- Dao, N. T., et al. Photophysics of DFHBI bound to RNA aptamer Baby Spinach. Scientific Reports. 11, 7356(2021).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiDaha Fazla Makale Keşfet
This article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır