Method Article
קביעת קינטיקה במבחנה והפעלה תאית עבור אפטאמרים של RNA פלואורוגניים
In This Article
Summary
הפרוטוקול מציג שתי שיטות לקביעת הקינטיקה של הרנ"א הפלואורוגני אפטאמרים תרד2 וברוקולי. השיטה הראשונה מתארת כיצד למדוד קינטיקה פלואורוגנית של אפטמר במבחנה באמצעות קורא צלחת, ואילו השיטה השנייה מפרטת את מדידת הקינטיקה הפלואורוגנית של אפטמר בתאים על ידי ציטומטריה של זרימה.
Abstract
אפטאמרים פלואורוגניים של RNA יושמו בתאים חיים כדי לתייג ולהמחיש רנ"א, לדווח על ביטוי גנים ולהפעיל ביוסנסורים פלואורסצנטיים המזהים רמות של מטבוליטים ומולקולות איתות. על מנת לחקור את השינויים הדינמיים בכל אחת מהמערכות הללו, רצוי לקבל מדידות בזמן אמת, אך דיוק המדידות תלוי בקינטיקה של התגובה הפלואורוגנית שתהיה מהירה יותר מתדר הדגימה. במאמר זה נתאר שיטות לקביעת הקינטיקה החוץ-גופית והתאית עבור אפטאמרים פלואורוגניים של RNA באמצעות קורא צלחות המצויד במזרק דגימה וציטומטר זרימה, בהתאמה. אנו מראים כי ניתן למדל את הקינטיקה במבחנה עבור הפעלת הפלואורסצנציה של האפטמרים תרד2 וברוקולי כתגובות אסוציאציה דו-פאזיות ויש להם קבועי קצב פאזה מהירים שונים של 0.56 s−1 ו- 0.35 s−1, בהתאמה. בנוסף, אנו מראים כי הקינטיקה התאית להפעלת פלואורסצנטיות של תרד2 ב-Escherichia coli, המוגבלת עוד יותר על ידי דיפוזיית צבע לחיידקים גראם-שליליים, עדיין מהירה מספיק כדי לאפשר תדירות דגימה מדויקת על ציר הזמן הדק. שיטות אלה לניתוח קינטיקה של הפעלת פלואורסצנציה ישימות לאפטמרים פלואורוגניים אחרים של RNA שפותחו.
Introduction
תגובות פלואורוגניות הן תגובות כימיות היוצרות אות פלואורסצנטי. אפטאמרים פלואורוגניים של RNA בדרך כלל מבצעים את הפונקציה הזו על-ידי קשירת צבע מולקולה קטנה כדי לשפר את התפוקה הקוונטית הפלואורסצנטית שלה (איור 1A)1. פותחו מערכות אפטמר RNA פלואורוגניות שונות המורכבות מרצפי RNA אפטמר ספציפיים ומליגנדות הצבעהמתאימות 1. אפטאמרים של RNA פלואורוגניים צורפו לתעתיקי RNA כתגי פלואורסצנט המאפשרים הדמיה של תאים חיים של mRNA ו-RNAsשאינם מקודדים 2,3,4. הם גם הוצבו לאחר רצפי מקדמים כמדווחים פלואורסצנטיים של ביטוי גנים, בדומה לשימוש בחלבון פלואורסצנטי ירוק (GFP) כמדווח, אלא שפונקציית הדיווח היא ברמת RNA 5,6. לבסוף, אפטאמרים של RNA פלואורוגניים שולבו בביוסנסורים פלואורסצנטיים מבוססי RNA, שנועדו לעורר את התגובה הפלואורוגנית בתגובה למולקולה קטנה ספציפית. ביוסנסורים פלואורסצנטיים מבוססי RNA פותחו להדמיית תאים חיים של מטבוליטים שונים שאינם פלואורסצנטיים ומולקולות איתות 7,8,9,10,11.
יש עניין גובר בפיתוח אפטמרים של RNA פלואורוגניים כדי לדמיין שינויים דינמיים בלוקליזציה של RNA, ביטוי גנים ואותות של מולקולות קטנות. עבור כל אחד מהיישומים הללו, רצוי לקבל מדידות בזמן אמת, אך הדיוק של המדידות תלוי בקינטיקה של התגובה הפלואורוגנית להיות מהירה יותר מתדר הדגימה. במאמר זה נתאר שיטות לקביעת הקינטיקה במבחנה עבור רנ"א פלואורוגני אפטאמרים תרד2 12 וברוקולי13 באמצעות קורא צלחות המצויד במזרק דגימה ולקביעת הקינטיקה הדלקתית התאית עבורתרד2 המתבטאת ב-Escherichia coli באמצעות ציטומטר זרימה. שני אפטאמרים אלה של RNA נבחרו מכיוון שהם יושמו כדי לחקור לוקליזציה של RNA 2,3,4, הם שימשו בכתבים 5,6 וביוסנסורים 7,8,9,10,11, וליגנדות הצבע המתאימות (DFHBI או DFHBI-1T) זמינות מסחרית. סיכום של תכונותיהם במבחנה שנקבעו בספרות מובא בטבלה 1 4,13,14, אשר ביססה את התפתחות הפרוטוקול (למשל, אורכי הגל וריכוזי הצבעים שבהם נעשה שימוש). תוצאות אלה מראות כי התגובות הפלואורוגניות המושפעות מאפטאמרים של RNA הן מהירות ואינן אמורות לעכב מדידות מדויקות עבור היישומים הביולוגיים הרצויים של התא.
Protocol
1. ניסוי קינטיקה במבחנה
- הכנת תבניות DNA על ידי PCR
- הגדר תגובות PCR: כדי להכין תגובות PCR, שלב את הריאגנטים הבאים בצינור PCR בעל דופן דקה:
33 μL של מים מזוקקים כפולים (ddH2O)
10 μL של מאגר 5x עבור DNA פולימראז באיכות גבוהה
5 μL של 2 mM כל טריפוספט דאוקסיריבונוקלאוזיד (dNTP)
0.5 μL של פריימר קדמי של 40 μM
0.5 μL של פריימר הפוך 40 μM
0.5 μL (10-100 ננוגרם) של תבנית DNA (עבור תרד2 PCR בלבד; פריימרים ברוקולי חופפים)
0.5 μL של DNA פולימראז בעל נאמנות גבוהה (הוסף אחרון)
הערה: אוליגונוקלאוטידים סינתטיים נשלחים לעתים קרובות יבשים. כדי להכין פתרונות מלאי, הוסף נפח ידוע (מומלץ 100 μL) של ddH2O, ומדוד את A260 של תמיסה זו כדי לקבוע את הריכוז על ידי חוק באר, שבו מקדם ההכחדה יכול להיות מחושב על ידי הכללים של השכן הקרוב ביותר באינטרנט. לאחר מכן ניתן להשתמש בפתרון מלאי זה כדי לבצע דילולים המתאימים לשימוש ב- PCR. - הפעל את פרוטוקול התרמו-ציקלר.
- השתמש בפרוטוקול התרמוציקלר הבא כדי להגביר תבניות דנ"א באורך מלא של תרד2 וברוקולי:
דנטורציה ראשונית: 98 מעלות צלזיוס למשך 2 דקות
35 מחזורים:
דנטורציה: דנטורציה של 98 מעלות צלזיוס למשך 15 שניות
חישול: 72 מעלות צלזיוס למשך 30 שניות
הארכה:72 מעלות צלזיוס למשך 30 שניות
הארכה סופית: 72°C למשך 5 דקות. - לאחר התגובה, לנתח aliquot קטן של המוצר על ידי 2% ג'ל agarose יחד עם סולם משקל מולקולרי נמוך (טווח גודל: 25-766 נוקלאוטידים) כדי לאשר את נוכחותו של מוצר ה- DNA הרצוי.
- טהרו את המוצר על ידי מיצוי ג'ל מסחרי או ערכות ניקוי PCR והתחמקו עם ddH2O או החיץ המסופק על ידי היצרן.
הערה: ודא בעת בחירת ערכת ניקוי PCR כי חתך המשקל המולקולרי של העמודה נמוך מספיק כדי לשמור על T7-ברוקולי (81 נוקלאוטידים), אחרת מוצר ה-PCR יאבד.
הערה: נקודת השהיה אופציונלית: אחסן את הדנ"א ב- −20 °C.
- השתמש בפרוטוקול התרמוציקלר הבא כדי להגביר תבניות דנ"א באורך מלא של תרד2 וברוקולי:
- הגדר תגובות PCR: כדי להכין תגובות PCR, שלב את הריאגנטים הבאים בצינור PCR בעל דופן דקה:
- הכנת תרד2 וברוקולי RNA על ידי שעתוק חוץ גופי (IVT)
- הגדר את תגובות השעתוק.
- כדי להכין תגובת שעתוק של 100 μL, שלב את הריאגנטים הבאים בצינור מיקרוצנטריפוגה של 1.5 מ"ל: 10 μL של 10x מאגר שעתוק + 20 μL של 10 mM ריבונוקלאוזיד טריפוספטים (rNTPs) + 1-64 μL של תבנית DNA (סה"כ 1 מיקרוגרם) + 2 μL של pyrophosphatase אנאורגני + ddH 2 O עד 98 μL +2μL של T7 RNA פולימראז (להוסיף אחרון).
- לדגום תגובה זו במשך 4 שעות ב 37 מעלות צלזיוס. הרוות את התגובה על ידי הוספת 100 μL של 2x מאגר העמסת ג'ל אוריאה (2x ULB), המורכב מ-20% סוכרוז, 0.1% נתרן דודציל סולפט (SDS), חיץ טריס-בוראט-EDTA (1x TBE) ו-~18 M אוריאה.
הערה: נקודת השהיה אופציונלית: אחסן את התגובה המרווה ב- −20 °C.
- הגדר את תגובות השעתוק.
- אלקטרופורזה בג'ל פוליאקרילאמיד (PAGE) טיהור RNA
- טיהור דף של תרד2 וברוקולי RNA
אזהרה: אקרילאמיד שאינו פולימרי (נוזלי או אבקתי) הוא רעיל ביותר. אם אתם שוקלים אבקת אקרילאמיד, עשו זאת במכסה אדים. תמיד ללבוש ציוד מגן מתאים מיד להסיר כפפות מזוהמות עם אבקת אקרילאמיד או פתרון, לשטוף ידיים ביסודיות. אם אקרילאמיד בא במגע ישיר עם העור, יש לשטוף את האזור החשוף למשך 15 דקות לפחות במים וסבון. אם אקרילאמיד בא במגע ישיר עם העיניים, יש לשטוף אותן במים למשך 15 דקות.- הכן את ג'ל PAGE: כדי להסיר תעתיקים לא רצויים של הפלות ו- rNTPs לא מגיבים מהמוצר באורך מלא, הכן ג'ל אוריאה-פוליאקרילאמיד של 6%. באופן כללי, 28 ס"מ x 16.5 ס"מ x 1.5 מ"מ ג'לים ניתן להשתמש עם מסרק 8 באר. הגדר את ציוד הג'ל והאלקטרופורזה, באמצעות חיץ TBE 1x למילוי המאגרים.
- טען דגימות RNA בג'ל PAGE: טען את הג'ל עם תגובה אחת מרוותה של 200 μL לכל נתיב. בנתיב נפרד, טען 2x ULB עם צבעי גשש קסילן ציאנול וכחול ברומופנול, אשר נודדים בג'ל ב 106 נוקלאוטידים ו 26 נוקלאוטידים, בהתאמה15. השאירו נתיב ריק בין כל דגימה כדי למנוע זיהום פוטנציאלי בשלבים הבאים.
- הפעל את ג'ל PAGE: כדי להפריד בין 95-nt תרד 2 וברוקולי 49-nt מהמוצרים החתוכים שלהם בהתאמה, הפעל את הג'ל במשך 1.5-2 שעות ב 25 W, ואז הצבע הכחול ברומופנול יעבור ~ 5/6 מאורך הג'ל.
- דמיינו את דגימות ה-RNA בג'ל PAGE: פרקו את לוחות הזכוכית סביב הג'ל וכסו את הג'ל בניילון נצמד משני הצדדים, תוך סימון הנתיבים על העטיפה. דמיינו פסי RNA בחדר חשוך על ידי הצללת UV על ידי הנחת הג'ל העטוף על צלחת TLC פלואורסצנטית תחת אור UV. שרטטו במהירות את הקצה של רצועות ה-RNA המתאימות למוצר בעזרת סמן וכבו את מנורת ה-UV כדי למזער את הנזק מחשיפה לקרינת UV.
- הבלו וחלץ את דגימות ה-RNA מג'ל ה-PAGE: עם סכין גילוח טרי לכל דגימה, הוציאו את רצועות המוצר הרצויות, הכניסו קוביות לקוביות של ~1 מ"מ, והוסיפו את חתיכות הג'ל לצינור מיקרוצנטריפוגה של 2 מ"ל עם 500 μL של חיץ ספיגת קראש כדי לחלץ RNA על מסובב למשך שעתיים בטמפרטורת החדר (RT) או לילה בטמפרטורה של 4 מעלות צלזיוס.
הערה: נקודת השהיה אופציונלית: הדגימה עשויה להיות מאוחסנת ב- −20 °C. - לזרז את הרנ"א
- כדי להפריד את חתיכות הג'ל מהרנ"א שחולץ במאגר, צנטריפוגה של הדגימה ב 13,000 x g במשך 20 דקות ב 4 מעלות צלזיוס, ולאחר מכן להשתמש פיפטה צרת קצה כדי לחלץ את supernatant ולהעמיס אותו לתוך צינור microcentrifuge חדש 2 מ"ל.
- כדי לזרז את הרנ"א, הוסיפו 1.5 מ"ל של אתנול קר כקרח ו-1 μL של 20 מ"ג/מ"ל גליקוגן, מערבולת, ואחסנו לפחות שעה אחת ב-−20 מעלות צלזיוס או −80 מעלות צלזיוס.
הערה: נקודת השהיה אופציונלית: ניתן לאחסן את הרנ"א בטמפרטורה של −20 °C למשך מספר חודשים.
- אוסף של משקע RNA: גלולה את הרנ"א המזורז על ידי צנטריפוגה ב 13,000 x g במשך 20 דקות ב 4 מעלות צלזיוס. הסר את הסופר-נאטנט ואפשר לאתנול הנותר להתאדות באוויר הפתוח (~1 שעות) לפני החייאת הכדור ב-30 μL של ddH2O או 1x TE buffer.
הערה: תהליך זה מביא בדרך כלל לריכוז RNA סופי של ~10 μM.
הערה: נקודת השהיה אופציונלית: דגימת הרנ"א עשויה להיות מאוחסנת בטמפרטורה של −20 °C למשך מספר חודשים.
- קבע את ריכוזי מלאי ה- RNA.
- הכינו RNA aliquot לתגובת ההידרוליזה.
- כדי לבצע בדיקה זו, תחילה, השתמש בספקטרופוטומטר ננו UV/Vis כדי לקבוע את A260 של דגימת ה-RNA של המניה ולבצע אליקוט מדולל של הדגימה ל~10 יחידות ספיגה (AU) ב-ddH2O.
- הכן את התגובה הבאה בצינור PCR של 0.5 מ"ל: 16 μL של ddH 2 O + 2 μL של 10x Na 2 CO3 מאגר +2μL של RNA aliquot מדולל ל ~ 10 AU. לדגום את התגובה במשך 90 דקות ב 95 מעלות צלזיוס, ולאחר מכן לאפשר לו להתקרר RT.
- קבע את ריכוז ה-RNA באמצעות ספיגת נוקלאוטידים: מדוד את ה-A260 של דגימה זו באמצעות ספקטרופוטומטר ננו-UV/Vis וחשב את ריכוז ה-RNA באמצעות הנוסחה הבאה:

כאשר c הוא ריכוז הרנ"א, b הוא אורך הנתיב, i הוא הנוקלאוטיד הספציפי (A, C, G או U), n i הוא התדירות של נוקלאוטיד i ברצף הרנ"א, ו-ε i הוא מקדם ההכחדה הטוחנת של נוקלאוטיד i. כדי לקבוע את ריכוז ה-RNA המקורי של המניה, הכפל את c בגורם הדילול.
הערה: נקודת השהיה אופציונלית: ניתן לאחסן את הרנ"א בטמפרטורה של −20 °C למשך מספר חודשים.
- הכינו RNA aliquot לתגובת ההידרוליזה.
- טיהור דף של תרד2 וברוקולי RNA
- ביצוע in vitro מבחן קינטיקה של קורא צלחות
- הגדר את תוכנית רנטורציה של RNA: צור את פרוטוקול התרמו-ציקלר הבא על-ידי בחירה באפשרות צור תוכנית חדשה > הוסף שלב חדש > הוסף שלב חדש מספר פעמים כדי להוסיף כל אחד מהשלבים הבאים לפני לחיצה על שמור:
70 מעלות צלזיוס למשך 3 דקות
65 מעלות צלזיוס למשך 45 שניות
60 מעלות צלזיוס למשך 45 שניות
55 מעלות צלזיוס למשך 45 שניות
50 מעלות צלזיוס למשך 45 שניות
45 מעלות צלזיוס למשך 45 שניות
40 מעלות צלזיוס למשך 45 שניות
35 מעלות צלזיוס למשך 45 שניות
30 מעלות צלזיוס למשך 45 שניות - טבע מחדש של הרנ"א: כדי ליצור מחדש רנ"א של תרד2 וברוקולי, הכן 2 מאגרי מיקרומטר של כל רנ"א ב-ddH 2 O בצינור PCR דק של 0.5 מ"ל, ולאחר מכן הוסף נפח שווה של 2x חיץ רנטורציה (80 mM HEPES, pH 7.5 [KOH], 250 mM KCl, 6 mM MgCl2) כדי ליצור פתרונות RNA של 1 μM. הוסף את הצינורות ל- thermocycler, פתח את תוכנית הרנטורציה שנשמרה ולחץ על הפעל.
הערה: אם תרמוציקלר אינו זמין, ניתן לדגור את ה-RNAs במקום זאת על בלוק חום של 70 מעלות צלזיוס למשך 3 דקות ולאחר מכן לאפשר להם להתקרר באיטיות ל-RT על הספסל. - הכן את מאגר תגובת הקשירה: כדי להכין את המאגר לתגובת קשירה אחת של אפטאמר-צבע, צור תערובת אב המכילה רכיבי מאגר (69.5 μL של ddH 2 O +4 μL של 1 M HEPES, pH 7.5 [KOH] + 6.2 μL של 2 M KCl + 0.3 μL של 1 M MgCl2). בהתאם למספר הדגימות והשכפולים הדרושים, הכפל ערכים אלה במספר הדגימות בתוספת אחת. באופן כללי, שלושה שכפולים לכל דגימת RNA משביעי רצון.
- הכינו את קורא הצלחות.
- הגדר את תוכנית המזרקים של קורא הלוחות: בקורא הלוחות הפלואורסצנטיים, בחר Temp והגדר את הטמפרטורה הרצויה ל- 37 °C, וודא שהטמפרטורה התאזן לערך זה הרבה לפני תחילת ניסויי קינטיקה. פתח את תוכנת קורא הלוחות, בחר הגדרות > תצוגת רכישה והזן את התוכנית הבאה למדידות קינטיות:
לולאה: לכל באר
הגדרה בסיסית: 60 קריאות בסיסיות
הגדרות SmartInject: הזרקת 10 μL (שתתרחש לאחר קריאת הבסיס)
פלואורסצנציה (או FL) קוראת:
עירור: 448 ננומטר (רוחב פס: 9 ננומטר)
פליטת מזהמים: 506 ננומטר (רוחב פס: 15 ננומטר)
מחסנית: מונו (s/n 3297)
תזמון:
זמן קריאה כולל: 10 דקות
מרווח קריאה: 0.5 שניות
PMT ואופטיקה: 6 הבזקים לכל קריאה
לולאה: הבאר הבאה - הכן את המזרק
- לשטוף ולשאוף את המזרק: כדי להכין את מזרק קורא הצלחות, בחר תחילה הזרק על קורא הצלחות, ספק צלחת איסוף פסולת לקורא הצלחות כאשר אתה מכוון, ולאחר מכן בחר שטיפה, ונקה את צינור ההזרקה בהתאם להוראות בקורא הצלחת עם נפחים של 1 מ"ל של ddH 2 O, 75% אתנול ב- ddH2O, ולאחר מכן ddH2O. לאחר מכן, בחר שואף, ומאפשר למזרק להוציא נוזלים עודפים.
- הפריים את המזרק: לאחר היציאה למסך הקודם, בחר Prime כדי להכין את המזרק עם שני נפחי ליגנד של 260 μL שיש להזריק כדי להבטיח שליגנד טהור ומרוכז יתווסף לדגימות במהלך ניסויים - במקרה זה, פריים עם 100 μM DFHBI ב- ddH2O.
- הגדר את תוכנית המזרקים של קורא הלוחות: בקורא הלוחות הפלואורסצנטיים, בחר Temp והגדר את הטמפרטורה הרצויה ל- 37 °C, וודא שהטמפרטורה התאזן לערך זה הרבה לפני תחילת ניסויי קינטיקה. פתח את תוכנת קורא הלוחות, בחר הגדרות > תצוגת רכישה והזן את התוכנית הבאה למדידות קינטיות:
- ביצוע ניסויים בקינטיקה חוץ גופית : כדי לבצע ניסוי קינטיקה אחד, הוסף תחילה 80 μL של תערובת מאסטר חיץ מחייב שהוכנה בעבר לבאר אחת של צלחת תחתית ברורה היטב 96 היטב, ולאחר מכן 10 μL של RNA renatured. אפשרו לצלחת זו ולתמיסת DFHBI במזרק להתאזן ל-37 מעלות צלזיוס למשך 15 דקות בקורא הלוחות.
- בתוכנת קורא הצלחות הגדרות תחת אזור קריאה, בחר את הבאר לניתוח ולאחר מכן, תחת הכרטיסיה בית , בחר הפעל כדי להפעיל את תוכנית הקינטיקה שתוארה קודם לכן. חזור על תהליך זה עד להשלמת כל הניסויים.
הערה: באופן קריטי, ניסויי קינטיקה צריכים להתבצע היטב אחד בכל פעם כדי להבטיח שהקינטיקה של הרנ"א נמדדת בתנאים זהים בין שכפולים לדגימות. - לשטוף את המזרק: כדי להסיר את תמיסת DFHBI הנותרת מצינור ההזרקה, לשטוף את צינור ההזרקה כמתואר בשלב 1.4.4.2.1 עם 1 מ"ל נפחים של ddH 2 O, 75% אתנול ב ddH2 O, ולאחר מכן ddH2O.
הערה: נקודת השהיה אופציונלית.
- הגדר את תוכנית רנטורציה של RNA: צור את פרוטוקול התרמו-ציקלר הבא על-ידי בחירה באפשרות צור תוכנית חדשה > הוסף שלב חדש > הוסף שלב חדש מספר פעמים כדי להוסיף כל אחד מהשלבים הבאים לפני לחיצה על שמור:
- ניתוח של קינטיקה חוץ גופית של אפטאמרים RNA פלואורוגניים.
- הזן נתונים לתוכנת הניתוח: ייצא נתונים ניסיוניים כגיליון אלקטרוני כדי להעתיק בקלות את הנתונים לעיבוד. בתוכנת הניתוח, צור טבלת נתונים חדשה בתבנית XY. בעמודה X, הזן כל נקודת זמן לקריאה, כאשר t = 0 הוא הזמן של הזרקת DFHBI. בעמודה Y, הזן את ערכי הפלואורסצנציה הממוצעים בין משכפלים באותה נקודת זמן מתאימה, החל מ- t = 0.
- נרמל וגרף את הנתונים: כדי לנרמל את ערכי הפלואורסצנציה, לחץ על נתח > עיבוד נתונים > נרמל ולאחר מכן לחץ על אישור. בחר להשתמש בערכים הקטנים והגדולים ביותר של ערכת הנתונים לצורך נורמליזציה, כדי להציג תוצאות כשברים, כדי ליצור גרף של התוצאות ולאחר מכן לחץ על אישור. ליצירת גרף של הפלואורסצנציה הממוצעת המנורמלת לאורך זמן, לחצו על הסמל ' נרמול של [שם ערכת הנתונים] ' ובחרו 'משפחת תרשימים: XY עם נקודות בלבד'.
הערה: קווי שגיאה יכולים להיות שימושיים להצגה במקרים שבהם מספר קטן של נקודות זמן מוצגות בגרף. אם אלה רצויים, בעת בחירת טבלת נתונים בתבנית XY, בחר באפשרות תחת "Y" כדי להזין ערכים משוכפלים בעמודות זו לצד זו. יצירת גרפים של הטבלה המתקבלת עם אפשרויות ברירת המחדל שנבחרו תפיק תרשים עם קווי שגיאה. - בצע התאמת עקומה כדי לקבל פרמטרים קינטיים: כדי להתאים עקומה לנתונים הקינטיים, לחץ על נתח > ניתוח נתונים ובחר רגרסיה לא ליניארית (התאמת עקומה) תחת הכרטיסייה XY Analyses . תחת הכרטיסייה מודל , לחץ על שיוך מעריכי > דו-פאזי כדי להתאים את נתוני הקינטיקה למשוואת האסוציאציה הדו-פאזית הבאה:
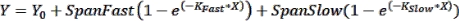
כאשר Y הוא הפלואורסצנציה בזמן X, Y 0 הוא הפלואורסצנציה ב- t = 0, K Fast ו- K Slow הם קבועי קצב מהיר ואיטי, בהתאמה, ו- SpanFast ו- SpanSlow הם הטווחים של הפעלת פלואורסצנציה המוסברים על ידי הקצבים המהירים והאיטיים, בהתאמה (ראה תוצאות מייצגות, איור 1). לחץ על הכרטיסיה התאמה ללא לין כדי להציג קבועי קצב, ערכיt 1/2 וערכי PercentFast.
הערה: כדי להשיג סטיית תקן עבור כל הערכים הללו, ניתן לעבד שכפולים של ניסויים בקורא לוחות בודדים באותו אופן כפי שתואר לעיל.
2. ניסוי קינטיקה תאית
- הכנת זני אי קולי
- להפוך BL21 כוכב (DE3) E . coli תאים עם ~ 100 ng של pET31b tRNA-תרד2 לבנות בהתאם לפרוטוקול של היצרן.
הערה: מבנה פלסמיד זמין מסחרית (פלסמיד #79783). - צלחת את התאים על אגר LB המכיל קרבניצ'ילין (פחמימות: 50 מ"ג/מ"ל) צלחת ודגירה ב 37 מעלות צלזיוס במשך 12-16 שעות. תאים המכילים פלסמידים יגדלו כמושבות על הצלחת.
הערה: נקודת השהיה אופציונלית: ניתן לאחסן תאי כוכב BL21 שעברו טרנספורמציה על צלחות בטמפרטורה של 4 מעלות צלזיוס כשהם עטופים בפרפילם למשך שבוע אחד.
- להפוך BL21 כוכב (DE3) E . coli תאים עם ~ 100 ng של pET31b tRNA-תרד2 לבנות בהתאם לפרוטוקול של היצרן.
- גידול תאים וגרימת ביטוי של RNA אפטמר פלואורוגני
- לחסן תרבית של 2 מ"ל של מדיה לא משררה (NI) המכילה קרבניצ'ילין (פחמימות: 50 מ"ג/מ"ל) עם מושבה אחת של תאי כוכב BL21 שעברו טרנספורמציה. חזור על כך עבור לפחות שלושה שכפולים ביולוגיים. דגירה של התרביות ב-37 מעלות צלזיוס באינקובטור/שייקר ב-250 סל"ד למשך 22-24 שעות.
הערה: נקודת השהיה אופציונלית: תאים הגדלים במדיית NI שומרים על הפלסמיד וניתן לאחסן אותם בטמפרטורה של 4 מעלות צלזיוס למשך שבוע אחד. - לאחר הצמיחה במדיית NI, יש לדלל את התרבית פי 100 ל-3 מ"ל טריים של מדיה אוטואינדוקציה (AI) של ZYP-5052 המכילה קרבניצ'ילין (פחמימות: 50 מ"ג/מ"ל). לגדל את התאים ב-37 מעלות צלזיוס באינקובטור/שייקר ב-250 סל"ד למשך 16-18 שעות כדי לגרום לביטוי.
הערה: OD600 טיפוסי לתרביות ינוע בין 2.0-3.3 לאחר 18 שעות של צמיחה. טווח צפיפות התאים האופטימלי הוא בין 2.5-3.0.
- לחסן תרבית של 2 מ"ל של מדיה לא משררה (NI) המכילה קרבניצ'ילין (פחמימות: 50 מ"ג/מ"ל) עם מושבה אחת של תאי כוכב BL21 שעברו טרנספורמציה. חזור על כך עבור לפחות שלושה שכפולים ביולוגיים. דגירה של התרביות ב-37 מעלות צלזיוס באינקובטור/שייקר ב-250 סל"ד למשך 22-24 שעות.
- ביצוע ניסוי הקינטיקה התאית
- הגדר את ציטומטר הזרימה.
- הפעל את ציטומטר הזרימה ואת המחשב המחובר למכשיר. לאחר הכניסה לתוכנה עבור ציטומטר הזרימה, תחת מכשיר הכרטיסייה, לחץ על סמל ההפעלה . בצע את השלבים המצוינים במסך התוכנה כדי להבטיח אתחול מכשור תקין.
הערה: ציטומטרים מסוימים של זרימה מכנים את רצף ההפעלה של המכשיר Priming. הקפידו לעקוב אחר פרוטוקול היצרן עבור ציטומטר הזרימה שישמש לניסוי. - הפעל בדיקת ביצועים (אם רלוונטי). תחת הכרטיסייה תפריט ראשי , לחץ על מבחן ביצועים. בצינור תרבית, הוסף שלוש טיפות של חרוזי מעקב הביצועים של היצרן לתוך 3 מ"ל של נוזל מיקוד.
- הנח את צינור התרבית לתוך מרים צינור הדגימה והרם את מרימי המשקולות. לפני לחיצה על הפעל בדיקת ביצועים, ודא שהלוט # של הצינור של חרוזי המעקב זהה למה שמצוין במסך הגדרת בדיקת ביצועים . לחץ על בדיקת ביצועים כדי להפעיל את הבדיקה.
- הגדר את תוכנת ציטומטר הזרימה לניסוי זה עם פרמטרי הרכישה הבאים עבור פלואורסצנציה חד-תאית:
לייזר עירור: 488 ננומטר
ערוץ פליטה: GFP (נקרא גם FITC)
נפח רכישה: 40 μL (עם נפח משיכה כולל של 90 μL)
קצב זרימה: 200 μL / min
ספירת תאים עבור כל מדידה: 30,000
הגדרות מכשיר:
מתח:
FSC: 480 V
SSC: 400 וולט
BL1: 540 וולט
BL2: 392 V
BL3: 422 V
- הפעל את ציטומטר הזרימה ואת המחשב המחובר למכשיר. לאחר הכניסה לתוכנה עבור ציטומטר הזרימה, תחת מכשיר הכרטיסייה, לחץ על סמל ההפעלה . בצע את השלבים המצוינים במסך התוכנה כדי להבטיח אתחול מכשור תקין.
- הגדר את קבצי הניסוי.
- צור קובץ ניסוי חדש בכרטיסיה סייר הניסויים על-ידי לחיצה באמצעות לחצן העכבר הימני על שם המשתמש של ציטומטר הזרימה. בחר ניסוי חדש בחלון הנפתח. כאשר חלון חדש במסך המחשב צץ, בחר אישור.
- בקובץ הניסוי החדש, לחץ באמצעות לחצן העכבר הימני על התיקייה "קבוצה" ובחר הוסף צינור דגימה חדש. תייג את צינורות הדגימה עבור כל נקודת זמן ספציפית ושכפל אותה על ידי לחיצה ימנית על דגימה ובחירה באפשרות שנה שם בתפריט הנפתח. חזור על שלב זה עבור המספר הכולל של עותקים משוכפלים ונקודות זמן עבור מהלך הזמן המיועד של המחקר.
- הכן תמיסת תאים מדוללים: בצינור תרבית חדש, הוסף 1.5 מ"ל של תמיסת PBS אחת. לאחר מכן, הוסף 3 μL של תאים מושרים במדיית AI לתמיסת PBS 1x כדי ליצור תמיסת תאים מדוללים. חזור על שלב זה עבור כל שכפול ביולוגי בצינורות תרבית שונים.
- מדוד את פלואורסצנטיות הרקע של התאים: לפני הוספת צבע, קח קריאות של כל צינור תרבית שכפול ביולוגי המכיל תאים בתמיסת PBS אחת. זאת כדי שהרקע הפלואורסצנטי של התאים יימדד כדי לראות את הקפל נדלק לאורך זמן לאחר הוספת הצבע.
- כדי לבצע קריאה, הנח את צינור התרבית לתוך מרים צינור הדגימה, והרם את המרים ביד אל מחט הזרקת הדגימה. בחרו בקובץ לדוגמה המתאים בכרטיסייה ' חלונית אוסף ' ולחצו על ' רשומה'.
- בסיום הריצה, הנמיכו את מרים צינור הדגימה בעזרת צינור התרבית ביד. פעולה זו תיזום שלב שטיפה שישטוף את המערכת הנוזלית וימזער את הנשיאה בין כל דגימת שכפול ביולוגית. הנתונים יישמרו באופן אוטומטי במחשב לאחר השלמת הריצה.
- חזור על השלבים בתוך 2.3.4 כדי למדוד את הרקע הפלואורסצנטי התאי עבור לפחות שלושה שכפולים ביולוגיים. כדי לעבור לקובץ לדוגמה הבא, בחר את הקובץ לדוגמה הבא על-ידי לחיצה על סמל החץ ימינה ליד שם צינור הדגימה מתחת לסמל הקלטה .
- מדידת פלואורסצנטיות בנקודות זמן עבור תאים עם צבע.
- הוסף 1.4 μL של מלאי צבע מרוכז (50 mM DFHBI-1T ב- DMSO) לתמיסת PBS אחת עם תאים כדי לתת ריכוז סופי של 50 μM DFBHI-1T. לאחר מכן, אבטח את מכסה צינור התרבית, ולאחר מכן הפוך את צינורות התרבית 3x-5x כדי לערבב את התמיסה באופן שווה לפני ביצוע הקריאה הנקודתית הראשונה.
הערה: האחוז הכולל של DMSO בתוך צינורות התרבית עבור E. coli לא יעלה על 10%, מכיוון שהדבר יכול להשפיע על כדאיות התא16. - הסר את המכסה והנח את צינור התרבית לתוך מרים צינור הדגימה. הרם את המחזיק ביד אל מחט הזרקת הדגימה, ותחת קובץ הדגימה המתאים, לחץ על תקליט סמל. בנוסף, התחל טיימר על-ידי לחיצה על התחל עבור הניסוי.
- הורד את מרים הצינורות ביד לאחר השלמת הריצה, וחזור על שלבים 2.3.5.1-2.3.5.2. (כאשר הטיימר פועל) על ידי הוספת 1.4 μL של DFHBI-1T המרוכז, היפוך צינורות התרבית, ולקיחת קריאות עבור כל השכפולים הביולוגיים הנותרים. ההקלטות הראשונות הללו יהיו הקריאות המתקבלות ב-0 דקות עבור כל השכפולים הביולוגיים. עשו זאת אחד בכל פעם עבור כל שכפול ביולוגי.
הערה: רשום לעצמך את הזמן שבו לוחצים על רכישת הציטומטריה של זרימת הרשומות. לדבוק בזמן הזה מזעזע עבור נקודות זמן במהלך הניסוי. - המשך לקרוא על ידי העלאת צינורות התרבית במרים צינור הדגימה למחט הזרקת הדגימה, בחירת קובץ הדגימה המתאים, לחיצה על הקלט והורדת המרים ביד לאחר השלמת כל ריצה. עשו זאת עבור כל נקודות הזמן הנוספות והשכפולים הביולוגיים הנבדקים. חזור על השלבים עד להשלמת הניסוי.
הערה: שמור את הדגימות הרחק מאור כדי למנוע הלבנת תמונות של DFHBI-1T בתמיסה על-ידי כיסוי הדגימות ברדיד אלומיניום.
- הוסף 1.4 μL של מלאי צבע מרוכז (50 mM DFHBI-1T ב- DMSO) לתמיסת PBS אחת עם תאים כדי לתת ריכוז סופי של 50 μM DFBHI-1T. לאחר מכן, אבטח את מכסה צינור התרבית, ולאחר מכן הפוך את צינורות התרבית 3x-5x כדי לערבב את התמיסה באופן שווה לפני ביצוע הקריאה הנקודתית הראשונה.
- מדידת פלואורסצנטיות בנקודות זמן עבור תאים ללא צבע (בקרה).
- הפעל את פרוטוקולי הניקוי המתאימים עבור ציטומטר הזרימה לפני שתחזור שוב על הניסוי עם בקרות שליליות בהתאם לפרוטוקול היצרן. זה נעשה כדי למזער כל העברה מהניסוי הקודם לניסוי ניתוח נקודת הזמן של הבקרה. להלן השלבים הבאים עבור ציטומטר הזרימה בין הניסויים:
- הנח שפופרת תרבות ריקה במרים הצינורות ביד, הרם את מחזיק הצינור ולחץ על סמל Unclog בכרטיסייה מכשיר . פעולה זו תפעיל backflush במערכת fluidics כדי לנקות דגימות דביקות. הורד את מחזיק הצינור ביד והסר את הצינור לאחר סיום רצף Unclog .
- עם צינור תרבית חדש, להוסיף 3 מ"ל של תמיסת אקונומיקה 10%, למקם את צינור התרבית לתוך מחזיק הצינור, ולהרים את המחזיק ביד אל מחט הזרקת הדגימה. בנוסף, יש להכניס לוחית נקייה של 96 בארות לתוך הדגימה האוטומטית, אם רלוונטי לציטומטר הזרימה.
- לחץ על סמל SIP של חיטוי SIP/חיטוי אוטומטי כדי להפעיל רצף ניקוי עם 10% אקונומיקה בכל מערכת הנוזלים. הנמיכו את מחזיק הצינור כדי להשלים את רצף הניקוי.
- הגדר את הקבצים לדוגמה עבור הפעלת ניתוח נקודת הזמן של הבקרה בהתאם להוראות בשלב 2.3.3.
- בצינור תרבית חדש, הכינו תמיסת תאים מדוללים ב-1.5 מ"ל של תמיסת PBS אחת. הוסף 3 μL מהתאים המושרים במדיית AI לתמיסת PBS 1x כדי ליצור תמיסת תאים מדוללים. חזור על שלב זה עבור כל שכפול ביולוגי.
- הוסף 1.4 μL של DMSO לתוך צינור התרבית אחד בכל פעם לתמיסת PBS 1x עם תאים ובדוק את אותן נקודות זמן. אבטח את מכסה צינור התרבית, ולאחר מכן הפוך את צינורות התרבית 3x-5x כדי לערבב את התמיסה באופן שווה לפני ביצוע הקריאה הנקודתית הראשונה. עשו זאת אחד בכל פעם עבור כל שכפול ביולוגי.
- בצע את אותו פרוטוקול לניתוח תאי בקרה כמו עבור תאים עם צבע, באמצעות שלבים 2.3.5.2-2.3.5.4.
- הפעל את פרוטוקולי הניקוי המתאימים עבור ציטומטר הזרימה לפני שתחזור שוב על הניסוי עם בקרות שליליות בהתאם לפרוטוקול היצרן. זה נעשה כדי למזער כל העברה מהניסוי הקודם לניסוי ניתוח נקודת הזמן של הבקרה. להלן השלבים הבאים עבור ציטומטר הזרימה בין הניסויים:
- כבה את ציטומטר הזרימה: עקוב אחר פרוטוקול היצרן לכיבוי תקין של המכשור. עבור ציטומטר הזרימה, המכשיר מוכן לכיבוי באופן הבא:
- בצע את פרוטוקול הניקוי הראשוני עבור ציטומטר הזרימה בשלבים 2.3.6.1.1-2.3.6.1.3.
- החלף את צינור התרבית בתמיסת אקונומיקה 10% בצינור תרבית עם 3 מ"ל של נוזל מיקוד. הרם את מחזיק הצינור ביד, ותחת סמל הכיבוי , לחץ על התפריט הנפתח ובחר יסודי.
הערה: נקודת השהיה אופציונלית.
- הגדר את ציטומטר הזרימה.
- ניתוח נתוני ציטומטריה של זרימה
- ייצא את כל קבצי FCS לניתוח. פתח את קבצי FCS באמצעות תוכנה לניתוח ציטומטריה של זרימה.
- באמצעות אחד מקבצי FCS של תאים בלבד, צור שער ממתווה הנקודה של הפיזור הקדמי (FSC) והפיזור הצדדי (SSC) (FSC-Area/SSC-Area), תוך שימוש בשני צירי יומן הרישום כדי לא לכלול אותות מפסולת. כדי ליצור שער זה, לחץ על סמל AutoGate בתוכנת ניתוח ציטומטריה של זרימה וקרא לו שער 1. החל שער זהה על כל הדגימות שנבדקו תחת הכרטיסיה כל הדוגמאות בסביבת העבודה של עיבוד הנתונים. התוצאה תהיה שער 1 מתחת לכל קבצי FCS המעובדים.
- צור קובץ ערכת משנה חדש עם קובץ FCS של תא בלבד המשמש בשלב 2.4.2 על-ידי לחיצה כפולה עליו. שנה את הגדרות הציר ל- FSC-Area/FSC-Height, כאשר שניהם משתמשים בצירי יומן. לחץ על סמל AutoGate בתוכנת ניתוח ציטומטריה של זרימה כדי ליצור שער סגלגל, וקרא לו שער 2. החל שער זה כתת-ערכה מתחת לערכת השערים בשלב 2.4.2 על כל הדוגמאות שנבדקו. התוצאה תהיה שלכל הדוגמאות שער 1 > שער 2 ישויכו לכל קובץ FCS.
- צור קובץ ערכת משנה נוסף כאשר שני השערים מוגדרים בשלב 2.4.2 ושלב 2.4.3 מוחלים על-ידי לחיצה כפולה על שער 2. שנה את הגדרות הציר ל- FSC-Area/היסטוגרמה. החל שער היסטוגרמה זה כתת-ערכה על כל הדגימות שנבדקו, וכתוצאה מכך כל הדגימות כוללות שער 1 > שער 2 > שער 2 המשויכים לכל קובץ FCS.
הערה: ניתן לשנות את שם ההיסטוגרמות משער 2 להיסטוגרמה כדי לסייע בהעברת ההיסטוגרמות לחלון הפריסה, וכן כדי ליצור ארגון רב יותר בעיבוד הנתונים. - כדי לנתח את ערכי עוצמת הפלואורסצנציה הממוצעת (MFI), פתח את חלון הפריסה. לחץ וגרור את שערי ההיסטוגרמה עבור כל נקודת זמן לחלון הפריסה.
- בצע ניתוח סטטיסטי עבור "ממוצע ∑: BL1-A" (GFP) עבור כל דגימה שנבדקה כדי להציג את תוצאות ה- MFI בחלון הפריסה.
- חשב את סטיית התקן עבור ערכי MFI לניתוח נקודת זמן עבור לפחות שלושה שכפולים ביולוגיים בלתי תלויים.
- שמרו את ההיסטוגרמות ואת ערכי MFI לכל נקודת זמן באמצעות ייצוא חלון הפריסה כקובץ PDF.
הערה: נקודת השהיה אופציונלית.
- נתוני ציטומטריה של זרימת גרף
- פתחו את קובץ PDF הכולל את ההיסטוגרמות ואת ערכי MFI לכל נקודת זמן. ערכי MFI יועתקו לתוכנת ניתוח נתונים. בתוכנת גרפים של נתונים, צור טבלת נתונים חדשה בתבנית XY.
- בחר כדי ליצור טבלת XY כשהאפשרות הבאה נבחרה:
טבלת נתונים: הזנה או ייבוא של נתונים לטבלה חדשה
אפשרויות:
X: זמנים שחלפו
Y: הזן (שלושה עד ארבעה) ערכים משוכפלים בעמודות משנה זו לצד זו - תייג על ציר X את כל נקודות הזמן לניסוי ולריצות הבקרה.
- בקבוצה A, הזן את ערכי ה-MFI עבור כל השכפולים הביולוגיים לתוך כל נקודת זמן לצורך ניתוח פלואורסצנטי של התאים עם תוספת הצבע.
- בקבוצה B, הזן את ערכי MFI עבור כל השכפולים הביולוגיים בכל נקודת זמן לצורך ניתוח התאים ללא תוספת צבע (DMSO).
- כדי לראות את התוצאות, לחץ על [הוסף שם ערכת נתונים] תחת הכרטיסיה תרשימים . פעולה זו תציג את נקודות הנתונים כאמצעי, עם קווי שגיאה המייצגים את סטיית התקן (s.d.) בכל נקודת זמן. ציר ה-X מייצג את הזמן שחלף, וציר ה-Y מייצג את ערכי ה-MFI.
- בחר כדי ליצור טבלת XY כשהאפשרות הבאה נבחרה:
- פתחו את קובץ PDF הכולל את ההיסטוגרמות ואת ערכי MFI לכל נקודת זמן. ערכי MFI יועתקו לתוכנת ניתוח נתונים. בתוכנת גרפים של נתונים, צור טבלת נתונים חדשה בתבנית XY.
תוצאות
קינטיקה במבחנה
הרצפים של תבניות הדנ"א והפריימרים, שנרכשו כאוליגונוקלאוטידים סינתטיים, מוצגים בטבלה 2, ומתכוני הריאגנטים מוצגים בקובץ משלים 1. הגברת PCR משמשת להגדלת כמות תבנית הדנ"א באמצעות מקדם T7, הנדרש לתגובת השעתוק במבחנה (IVT) הבאה. בנוסף, ניתן להשתמש בהגברת PCR לשתי מטרות באותה תגובה: ליצור את תבנית הדנ"א של ברוקולי באורך מלא על ידי הרחבת פריימר, כמו גם להגדיל את תבנית הדנ"א.
לאחר תגובת ה- IVT לסינתזה של RNA, טיהור PAGE יסיר כל תעתיקים חתוכים לא רצויים, מוצרים פגומים ו- rNTPs לא מגיבים ממוצר ה- RNA באורך מלא. סוג זה של טיהור עדיף מכיוון שרנ"א קטוע או מושפל יגרום לקביעה לא מדויקת של ריכוזי RNA. מאחר שבסיסי נוקלאוטידים בולעים אור UV, ניתן לדמיין רצועות RNA ו-rNTPs על הג'ל תחת UV כצללים על לוח TLC פלואורסצנטי. לפיכך, להקות המתאימות לגודל המוצר הנכון ניתן לחלץ באופן סלקטיבי.
שיטת השכן הקרוב ביותר מעריכה יתר על המידה את מקדמי ההכחדה, ולכן את ריכוזי הרנ"א המובנים, שכן היא אינה לוקחת בחשבון היפוכרומיות עקב זיווג בסיס17. לכן, כדי לקבוע ריכוזי RNA מדויקים, בוצעו מבחני הידרוליזה תרמית של pH ניטרלי כדי לבצע הידרוליזה של הרנ"א ל- NMPsבודדים 18. מקדם ההכחדה המדויק חושב כסכום מקדמי ההכחדה של NMPs ברצף הרנ"א.
הקינטיקה של קשירת DFHBI לתרד2 וברוקולי נקבעה באמצעות בדיקת קורא צלחת. רנ"א עבר חידוש ראשון כדי להבטיח שהוא יהיה בקונפורמציה הנכונה לקשירת צבע. תנאי התגובה לבדיקת הקינטיקה של קורא הצלחות כללו 40 mM HEPES, pH 7.5 (KOH), 125 mM KCl, 3 mM MgCl2, 100 nM RNA renatured ו-10 μM DFHBI, והתגובה נמדדה ב-37 מעלות צלזיוס. טמפרטורה וריכוז אלה של MgCl 2 נבחרו כדי לחקות תנאים פיזיולוגיים19, אם כי תנאים של 28 מעלות צלזיוס ו 10 mM MgCl2 עשויים לשמש גם לקיפול אפטמר משופר.
גם האפטמרים הפלואורוגניים, תרד2 וגם ברוקולי, מציגים קינטיקה דו-פאזית לקשירת צבע DFHBI (איור 2). נתוני הקינטיקה התאימו טוב יותר על ידי שיוך דו-פאזי מאשר על ידי שיוך חד-פאזי עבור שני האפטמרים (איור משלים 1). קבועי הקצב וערכיt 1/2 עבור האסוציאציות המהירות והאיטיות נקבעו על ידי עקומת ההתאמה הטובה ביותר (טבלה 3). PercentFast, המתאר איזה אחוז מגודל ההדלקה הפלואורסצנטית אחראי על ידי אוכלוסיית ה-RNA הקושרת DFHBI המהירה יותר, נקבע גם הוא.
תרד2 במצב מחייב מראה הפעלה מהירה יותר מאשר ברוקולי (t 1/2 = 1.2 שניות לעומת2.0 שניות). הקינטיקה של השלב השני עבור שני האפטמרים דומה (t1/2 = 180 שניות) וככל הנראה תואמת שלב מגביל קצב משותף עבור תת-אוכלוסייה של המדגם (PercentFast = 68% ו- 60% עבור תרד2 וברוקולי, בהתאמה). בסך הכל, התוצאות הללו מראות כי אפטאמרים מקופלים היטב של תרד 2 וברוקולי מפגינים קינטיקה מהירה מאוד, כאשר ההדלקה הראשונית החצי-מקסימלית נמצאת בטווח של 1-2 שניות מתוספת הצבע.
קינטיקה תאית
הרצפים של מבני הדנ"א, אשר משוכפלים לתוך פלסמיד pET31b, מוצגים בטבלה 2, ומתכוני ריאגנטים מוצגים בקובץ משלים 1. מבני הדנ"א של אפטאמרים של רנ"א פלואורוגניים כלולים בדרך כלל בתוך פיגום tRNA לניסויים תאיים. זן BL21 Star (DE3) E. coli הוא זן ביטוי עם מוטציה ב-RNase E המגבירה את יציבות ה-RNA.
מדידות נקודת הזמן הפלואורסצנטית נרשמו כל 5 דקות במשך 45 הדקות הראשונות, ולאחר מכן קריאות בשעה, 1.5 שעות ושעתיים, מה שנתן בסך הכל 12 נקודות זמן בתוספת הקריאה בתא בלבד. העובדה שמרווח הזמן הקצר ביותר הוא 5 דקות אפשרה למדוד מספר שכפולים ביולוגיים בכל נקודת זמן, עם ריווח קבוע בין מדידות המשוכפלות של 30 שניות עד דקה. הנפח הכולל של תאים מדוללים בתמיסת PBS אחת ששימשה לניסוי במהלך הזמן היה 1.5 מ"ל.
התאים היו מגודרים לפני קביעת עוצמת הפלואורסצנטיות הממוצעת (MFI) של אוכלוסיית התאים הבודדים. Gating בוחר אזור בחלקת הפיזור כדי לקבוע את אוכלוסיית התאים שתנותח. תהליך זה מונע כל פסולת או קריאות מרובות מלהיכלל בניתוח. לצורך ניתוח ציטומטריה של זרימה שהוצג, נרשמו 30,000 אירועים, מה שהביא לכך ש-10,000-20,000 אירועים נותחו לאחר ההטלה.
הקינטיקה הפלואורסצנטית התאית היא פונקציה של דיפוזיית צבע לתוך E. coli וקינטיקה קושרת צבע ל-RNA aptamer בתוך הסביבה התאית (איור 1B). עבור תאים המבטאים תרד2-tRNA, נצפה כי עוצמת הפלואורסצנציה הממוצעת (MFI) עולה באופן מיידי בנקודת הזמן "0", עקב פיגור הזמן הקצר (בשניות) בין הוספת צבע לניתוח דגימה (איור 3A). יתר על כן, פלואורסצנטיות תאית כבר הגיעה לערך MFI המקסימלי השווה שלה (40,441 ± 990) בנקודת הזמן הראשונה של 5 דקות. לעומת זאת, תאי בקרה מראים פלואורסצנטיות נמוכה ברקע (416) וללא שינוי בערך MFI עם תוספת DMSO (איור 3B). השוואה בין תאים עם צבע לבין תאים ללא צבע מגלה כי הפעלת פלואורסצנציה היא פי 98 ± 2 בתאים. בסך הכל, תוצאות אלה מראות כי תרד2-tRNA המתבטא בתאי E. coli מציג קינטיקה של הפעלה מהירה, עם הפעלה מקסימלית תוך פחות מ-5 דקות מתוספת הצבע.
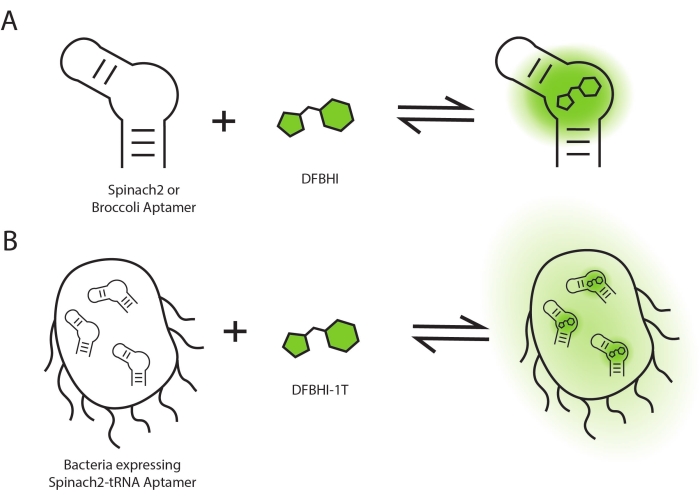
איור 1: סכמת הפעלה פלואורסצנטית. הפעלה פלואורסצנטית מתרחשת כאשר רנ"א אפטמרים נקשרים למולקולות צבע (A) במבחנה ו-(B) בתאים. אנא לחץ כאן כדי להציג גרסה גדולה יותר של נתון זה.

איור 2: קינטיקה חוץ-גופית של האפטמרים הפלואורוגניים. קינטיקה חוץ-גופית מייצגת של האפטאמרים הפלואורוגניים (A,B) תרד2 או (C,D) ברוקולי במודל של קשר דו-פאזי, כאשר t = 0 s היא נקודת הזמן של תוספת DFHBI (ריכוז DFHBI סופי: 10 μM). הניסויים בוצעו במשולש. כל קווי השגיאה מייצגים סטיות תקן מהממוצע. מההתאמה התקבלו ערכיt 1/2 עבור רכיבי התגובה של האסוציאציה המהירה והאיטית. אנא לחץ כאן כדי להציג גרסה גדולה יותר של נתון זה.
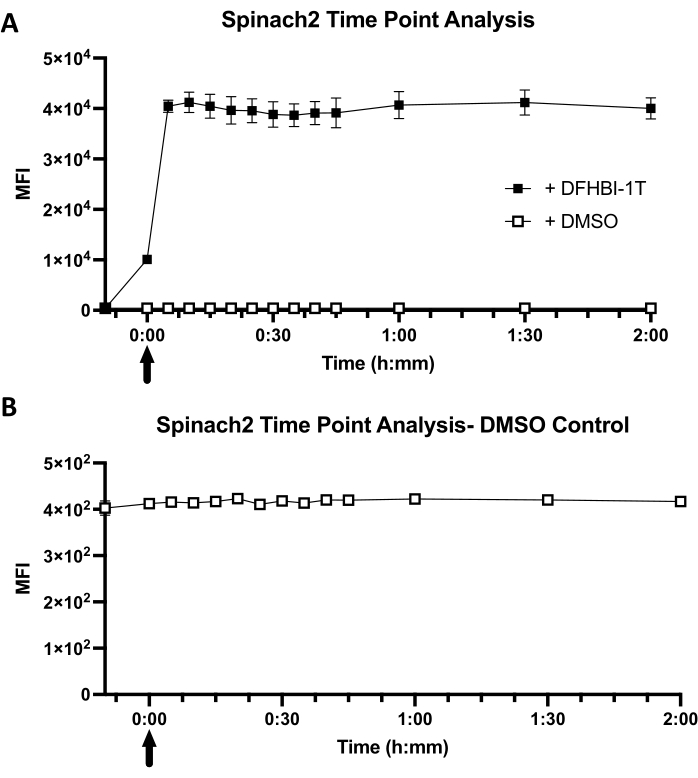
איור 3: קינטיקה תאית מייצגת של תרד2 בפיגום tRNA. (A) ניתוח נקודת זמן של ספיגת צבע tRNA-Spinach2 במהלך 2 שעות. קו בסיס לתאים בלבד נלקח לפני הוספת DFHIB-1T או DMSO. נקודות זמן נלקחו כל 5 דקות במשך 45 הדקות הראשונות, ולאחר מכן קריאת נקודת זמן בשעה, 1.5 שעות ושעתיים. החץ מייצג מתי DFHBI-1T או DMSO נוספו לתמיסת PBS אחת עם תאי כוכב BL21. הריכוז הסופי של DFHBI-1T לניתוח הוא 50 μM. עבור בקרת DMSO, התוספת של DMSO הייתה בנפח שווה (1.4 μL) ששימש לתוספת צבע DFHBI-1T. (B) תקריב של ניתוח נקודת הזמן של בקרת DMSO עם תאי BL21 Star E. coli. עוצמת פלואורסצנציה ממוצעת (MFI) מציינת את הקריאה הפלואורסצנטית הכוללת של תאי כוכב BL21 עם צבע או DMSO. הנתונים מייצגים את סטיית התקן הממוצעת ± של שלושה שכפולים ביולוגיים. אנא לחץ כאן כדי להציג גרסה גדולה יותר של נתון זה.
| זוג אפטאמר-צבע | אורך (nt) | מקסימום שרירי בטן (ננומטר) | מקס אם (ננומטר) | מקדם הכחדה (M-1· CM-1) | תפוקה קוונטית | בהירות | Kd (ננומטר) | ט"מ (°C) | הפניה |
| תרד2-DFHBI | 95 | 445 | 501 | 26100 | 0.7 | 63 | 1450 | 37 | 4 |
| תרד2-DFHBI-1T | 95 | 482 | 505 | 31000 | 0.94 | 100 | 560 | 37 | 13, 14 |
| ברוקולי-DFHBI-1T | 49 | 472 | 507 | 29600 | 0.94 | 96 | 360 | 48 | 13 |
טבלה 1: תכונות פוטופיזיות וביוכימיות שפורסמו בעבר של תרד2-DFHBI4, תרד2-DFHBI-1T 13,14 וברוקולי-DFHBI-1T 13.
| מקדם תרד2 + T7 | 5'-CGATCCCGCGAAATTAATACGACTCACTATAGGATGTAACTGAATGAAATGGTGAA GGACGGGTCCAGTAGGCTGCTTCGGCGCCTGTTGAGTAGAGTGTGAGCTCC GTAACTAGTTACATC-3' | ||
| ברוקולי + מקדם T7 | 5'-CGATCCCGCGAAATTAATACGACTCACTATAGgagacggtcgg gtccagatattcgtatctgtcgagtgtgggctc-3' | ||
| מבנה tRNA-Spinach2 (בפלסמיד pET31b) | 5'-CGATCCCGCGAAATTAATACGACTCACTATAGGGGCCCGGATAGCTCAGTCGGT AGAGCAGCGGCCGGATGTAACTGAATGAAATGGTGAAGGACGGGTCCAGTAGGCT GCTTCGGCAGCCTACTTGTTGAGTAGAGTGAGCTCCGTAACTAGTTACATCCGG CCGCGGGTCCAGGGTTCAAGTCCCTGTTCGGGGCCA TAGCATAACCCCTTGGGGCCTAACGGGTCTTGAGGGGTTTTG-3' | ||
| תרד2 פריימר קדמי | 5'-CGATCCCGCGAAATTAATACGACTCACTATAG-3' | ||
| תרד2 פריימר הפוך | 5'-GATGTAACTAGTTACGGAGC-3' | ||
| פריימר קדמי ברוקולי | 5'-CGATCCCGCGAAATTAATACGACTCACTATAGgagacggtcgggtccagatattcgtatctg-3' | ||
| ברוקולי פריימר הפוך | 5'-gagcccacactactcgacagatacgaatctggacccgaccgtctc-3' | ||
טבלה 2: טבלת רצפי דנ"א המכילה רצפי דנ"א ופריימרים המשמשים למחקרי קינטיקה חוץ גופית ותאית. נועז = מקדם T7; מסומן בקו תחתון = פיגום tRNA; כובעים = תרד2; אותיות קטנות = ברוקולי; נטוי מודגש = שליחות קטלנית T7.
| אפטמר | מהיר t1/2 (s) | Slow t1/2 (s) | Kמהיר (s-1) | Kאיטי (s-1) | אחוז מהיר |
| תרד2 | 1.2 ± 0.2 | 180 ± 10 | 0.56 ± 0.07 | 0.0039 ± 0.0002 | 68 ± 5 |
| ברוקולי | 2.0 ± 0.2 | 180 ± 30 | 0.35 ± 0.05 | 0.0039 ± 0.0006 | 60 ± 3 |
טבלה 3: ערכי קינטיקה במבחנה של אפטמרים תרד2 וברוקולי הנגזרים מנתונים מותאמים. הנתונים מדווחים כממוצע ± סטיית התקן של שלושה משוכפלים.
איור משלים 1: קינטיקה חוץ-גופית מייצגת של האפטמרים הפלואורוגניים. קינטיקה מייצגת במבחנה של האפטמרים הפלואורוגניים (A,C) תרד2 או (B,D) ברוקולי במודל של קשר חד-פאזי ב-(A,B) 600 שניות או (C,D) 20 שניות זמני מדידה, עם חצים המציינים את נקודת הזמן של תוספת DFHBI (ריכוז DFHBI סופי: 10 μM). הניסויים בוצעו במשולש. באופן כללי, נתונים אלה מתאימים פחות על ידי מודל שיוך חד-פאזי מאשר מודל שיוך דו-פאזי כאשר אות פלואורסצנטי מנוטר למשך זמן ארוך יותר. אנא לחץ כאן כדי להוריד קובץ זה.
קובץ משלים 1: מתכונים לניסוי קינטיקה חוץ גופית. אנא לחץ כאן כדי להוריד קובץ זה.
Discussion
עבור ניסוי הקינטיקה במבחנה, ניתן לשנות את אותו פרוטוקול כללי כדי למדוד את הקינטיקה במבחנה של ביוסנסור פלואורסצנטי מבוסס RNA המכיל גם תחום קושר ליגנד וגם תחום קושר פלואורופור 8. במקרה זה, יש לדגור את הרנ"א עם הפלואורופור לפני המדידות בעת הזרקת הליגנד על מנת לקבל קינטיקה של תגובת ליגנד. אם נצפתה שונות גבוהה בין המשכפלים, ניתן לפתור בעיות על ידי בדיקה שכל דגימה מותרת לשיווי משקל למשך אותה כמות זמן בלוח 96 הבאר לפני המדידה. כל דגימה או שכפול צריכים להיות מוכנים בנפרד בבאר ולמדוד מיד לאחר שלב שיווי המשקל של 15 דקות, במקום להכין את כל הדגימות בבת אחת.
עבור ניסוי הקינטיקה התאית, ניתן לשנות את הפרוטוקול למסלולי זמן קצרים או ארוכים יותר, אך חיוני לתכנן את מספר השכפולים הביולוגיים ולהתאים את נפח פתרון התא הדרוש. מומלץ לרווח כל קריאת שכפול ביולוגית בין 30 שניות לדקה אחת כדי שיהיה מספיק זמן לבצע את השלבים בזהירות. שינוי נוסף הוא לבדוק צבע פלואורוגני אחר שאינו קושר את ה-RNA aptamer כבקרה שלילית חלופית, שלא אמורה להראות הפעלת פלואורסצנטיות על הרקע. אם נצפות תוצאות לא עקביות, ניתן לפתור בעיות על-ידי בדיקה שציטומטר הזרימה מנוקה כראוי בהתאם לפרוטוקול היצרן בין ריצות ניסוי שונות כדי למנוע דימום של תאים או צבעים מהריצה הקודמת לאחרת.
בעוד ששיטת ה-in vitro המוצגת שימושית להשוואת הקינטיקה בין אפטמרים פלואורוגניים או ביוסנסורים פלואורוגניים מבוססי RNA, הערכים הקינטיים המתקבלים עשויים להשתנות בהתאם לטמפרטורה, לריכוז המגנזיום או לרכיבי חיץ אחרים שבהם נעשה שימוש. כמו כן, בעוד ששיטה זו מספקת תנאים מוגדרים היטב ששימשו בעבר לאפיון מערכות RNA פלואורוגניות שונות, הסביבה התוך-תאית אינה יכולה להיות מיוצגת באופן מושלם בשל נוכחותם של מקרומולקולות ביולוגיות אחרות.
בעוד שלקורא הלוחות הפלואורסצנטיים המצויד במזרק הניתן לתכנות אין זמן מת לאיסוף נתונים, למכשיר הציטומטר של הזרימה יש דיוק זמני מוגבל בשל הזמן המת הניתן לצפייה. יש פיגור של ~ 5 שניות בין לחיצה על כפתור "הקלט" לבין תחילת רכישת הנתונים. פיגור נוסף של ~5 שניות מתרחש כדי שהמכשיר ימדוד 30,000 אירועים; זמן רכישת דגימה זה ישתנה מעט בהתאם למידת דילול התאים ב- PBS אחד.
מגבלה פוטנציאלית נוספת לניסויים תאיים היא כדאיות התא ב-PBS אחד. לניתוח נקודות זמן ממושכות, ניתן לבדוק את כדאיות התאים באמצעות פרופידיום יודיד כדי להכתים תאים מתים20. צבירת צבע יכולה גם להגביל את הדיוק של מדידות פלואורסצנטיות המבוצעות על ידי ציטומטר הזרימה. צבעים עם מסיסות מוגבלת מאוד בתמיסות מימיות יכולים להצטבר ולהופיע כחלקיקים גדולים מספיק כדי להיספר כתאים בציטומטר הזרימה. לכן, חשוב להפעיל בקרות ניסיוניות של צבע בלבד כדי לבדוק אם יש אגרגטים באזור המגודר.
בעבר הוכח כי לברוקולי יש בהירות דומה לתרד2 ב-1 mM Mg2+in vitro, אך ברוקולי-tRNA מציג עוצמת פלואורסצנטיות גדולה פי שניים ב-E. coli חי בהשוואה לתרד2-tRNA13. למיטב ידיעתנו, הקינטיקה הקושרת צבע עבור תרד2 ואפטאמרים פלואורוגניים ברוקולי לא הושוו בעבר ועוצבו על ידי קשר דו-שלבי. קבועי הקצב המהיר ההתחלתיים של שני ה-RNA תומכים בכך שכיס קשירת הצבע מקופל מראש ואין צורך בשינויים מבניים כדי שהצבע ייקשר, מה שעולה בקנה אחד עם קריסטלוגרפיה של קרני רנטגן וניסויים בהתכת UV21,22. השלב השני עם קבוע קצב איטי יותר לא דווח בעבר מכיוון שניסויים אחרים כגון עצירת זרימה ומדידות אורך חיים פלואורסצנטיות ניתחו את אפטמר התרד למשך זמן קצר יותר (20 שניות ו-300 שניות)23,24. השלב השני האיטי בהרבה גורם לעלייה דו-אקספוננציאלית נצפתה בפלואורסצנציה כאשר הנתונים מנותחים במשך 600 שניות. ניתן לייחס את הצעד האיטי הזה לצעד מגביל קצב ממצב RNA מחייב למצב RNA בעל יכולת מחייבת, או לצעד פוטו-המרה מגביל קצב מצורות טרנס לציס של הצבע המאוגד. המנגנון האחרון עוצב בעבר כדי לתת פרופיל פלואורסצנטי דו-אקספוננציאלי24 ונתמך על ידי ניתוח עדכני של ההבדל בין ספקטרום הקליטה והעירור על האפטר הקשור, בייבי תרד25.
המשמעות הכוללת של הממצאים במבחנה היא שהם מראים כי קשר הצבע לאפטמר RNA פלואורוגני אינו מגביל מחקרי לוקליזציה של RNA וביטוי גנים בזמן אמת. עבור ביוסנסורים פלואורסצנטיים מבוססי RNA המשתמשים בתרד2, הקינטיקה המופעלת הנמדדת דומה לקינטיקה של השלב השני שנמדדה כאן10 מכיוון שהביוסנסורים דורשים צעד מתקפל מחדש, ולכן צריכים להיות מהירים מספיק כדי לאפשר מחקרי איתות כמעט בזמן אמת.
היה צפוי שהקינטיקה התאית תהיה שונה מהקינטיקה במבחנה שנצפתה עבור תרד2. הבדל מרכזי אחד הוא שיש שלב נוסף של דיפוזיית צבע לתוך תאי E. coli , הכולל חציית הממברנות החיצוניות והפנימיות. בנוסף, הסביבה התאית מציבה תנאים שונים לקשר צבע-אפטמר מבחינת צפיפות מולקולרית, הרכב יונים וריכוזים, כמו גם ריכוזי RNA וצבע.
המשמעות הכוללת של התוצאות היא שהפלואורסצנציה התאית מגיעה לאות המרבי תוך פחות מ-5 דקות ונשארת יציבה למשך שעתיים לפחות, מה שמאפשר לוקליזציה של RNA בזמן אמת ומחקרי ביטוי גנים בטווח זמן זה. עבור ביוסנסור מבוסס RNA המשתמש בתרד2, הראינו בעבר כי ניתן לראות תגובה פלואורסצנטית משמעותית תוך 4-5 דקות מתוספת הליגנד, אך ההגעה לאות המקסימלי אורכת זמן רב יותר (15-30 דקות)8. יחד, ממצאים אלה מצביעים על כך שדיפוזיה של צבע לתאים אינה הצעד המעשי להגבלת קצב בניסויי in vivo עם ביוסנסורים מבוססי RNA. לבסוף, פרוטוקול ניסיוני זה יכול להיות מיושם כדי לנתח מערכות RNA פלואורוגניות אחרות בתאים.
ניתן ליישם את הפרוטוקולים הניסיוניים המוצגים כאן לניתוח מערכות RNA פלואורוגניות אחרות. מעבר לשני האפטמרים שנותחו במחקר זה, תרד2 וברוקולי, פותחו מערכות RNA פלואורוגניות אחרות המספקות פרופילי פליטה שונים, יציבות פוטו-יציבה משופרת, זיקות קשירה הדוקות יותר ויכולת לשנות פלואורופורים (נסקרה לאחרונה 1). בנוסף לתכונות הפלואורסצנטיות שלהן, חשוב לבחון את הקינטיקה המפעילה של מערכות אלה במבחנה ובתאים כדי להעריך את התאמתן ליישומים ביולוגיים שונים של תאים. התוצאות עשויות גם לתמוך בקיפול מקדים מבני או בסידור מחדש של האפאמר. כפי שנדון, עם כמה שינויים, פרוטוקולים אלה יושמו גם כדי לנתח ביוסנסורים מבוססי RNA8.
Disclosures
המחברים מצהירים על היעדר ניגוד עניינים.
Acknowledgements
עבודה זו נתמכה על ידי המענקים הבאים ל- MCH: NSF-BSF 1815508 ו- NIH R01 GM124589. MRM נתמך חלקית על ידי מענק הכשרה NIH T32 GM122740.
Materials
| Name | Company | Catalog Number | Comments |
| Agarose | Thermo Fischer Scientific | BP160500 | |
| Agarose gel electrophoresis equipment | Thermo Fischer Scientific | B1A-BP | |
| Alpha D-(+)-lactose monohydrate | Thermo Fischer Scientific | 18-600-440 | |
| Amber 1.5 mL microcentrifuge tubes | Thermo Fischer Scientific | 22431021 | |
| Ammonium persulfate (APS) | Sigma-Aldrich | A3678 | |
| Ammonium sulfate ((NH4)2SO4) | Sigma-Aldrich | A4418 | |
| Attune NxT Flow cytometer | Thermo Fischer Scientific | A24861 | |
| Attune 1x Focusing Fluid | Thermo Fischer Scientific | A24904 | |
| Attune Shutdown Solution | Thermo Fischer Scientific | A24975 | |
| Attune Performance Tracking Beads | Thermo Fischer Scientific | 4449754 | |
| Attune Wash Solution | Thermo Fischer Scientific | J24974 | |
| Boric acid | Sigma-Aldrich | B6768 | |
| Bromophenol blue | Sigma-Aldrich | B0126 | |
| Carbenicillin disodium salt | Sigma-Aldrich | C3416 | |
| Chlorine Bleach | Amazon | B07J6FJR8D | |
| Corning Costar 96-well plate | Daigger Scientific | EF86610A | |
| Culture Tubes, 12 mm x 75 mm, 5 mL with attached dual position cap | Globe Scientific | 05-402-31 | |
| DFHBI | Sigma-Aldrich | SML1627 | |
| DFHBI-1T | Sigma-Aldrich | SML2697 | |
| D-Glucose (anhydrous) | Acros Organics | AC410955000 | |
| Dimethyl sulfoxide (DMSO) | Sigma-Aldrich | D8418 | |
| Dithiothreitol (DTT) | Sigma-Aldrich | DTT-RO | |
| DNA loading dye | New England Biolabs | B7025S | |
| DNA LoBind Tubes (2.0 mL) | Eppendorf | 22431048 | |
| dNTPs: dATP, dCTP, dGTP, dTTP | New England Biolabs | N0446S | |
| EDTA, pH 8.0 | Gibco, Life Technologies | AM9260G | |
| Ethanol (EtOH) | Sigma-Aldrich | E7023 | |
| Filter-tip micropipettor tips | Thermo Fischer Scientific | AM12635, AM12648, AM12655, AM12665 | |
| FlowJo Software | BD Biosciences | N/A | FlowJo v10 Software |
| Fluorescent plate reader with heating control | VWR | 10014-924 | |
| Gel electrophoresis power supply | Thermo Fischer Scientific | EC3000XL2 | |
| Glycerol | Sigma-Aldrich | G5516 | |
| Glycogen AM95010 | Thermo Fischer Scientific | AM95010 | |
| GraphPad Prism | Dotmatics | N/A | Analysis software from Academic Group License |
| Heat block | Thomas Scientific | 1159Z11 | |
| HEPES | Sigma-Aldrich | H-4034 | |
| Inorganic pyrophosphatase | Sigma-Aldrich | I1643-500UN | |
| Low Molecular Weight DNA Ladder | New England Biolabs | N3233L | Supplied with free vial of Gel Loading Dye, Purple (6x), no SDS (NEB #B7025). |
| Magnesium chloride hexahydrate (MgCl2) | Sigma-Aldrich | M2670 | |
| Magnesium sulfate (MgSO4) | Fisher Scientific | MFCD00011110 | |
| Microcentrifuge tubes (1.5 mL) | Eppendorf | 22363204 | |
| Microcentrifuge with temperature control | Marshall Scientific | EP-5415R | |
| Micropipettors | Gilson | FA10001M, FA10003M, FA10005M, FA10006M | |
| Micropipettor tips | Sigma-Aldrich | Z369004, AXYT200CR, AXYT1000CR | |
| Millipore water filter with BioPak unit | Sigma-Aldrich | CDUFBI001, ZRQSVR3WW | |
| Narrow micropipettor pipette tips | DOT Scientific | RN005R-LRS | |
| PBS, 10x | Thermo Fischer Scientific | BP39920 | |
| PCR clean-up kit | Qiagen | 28181 | |
| PCR primers and templates | Integrated DNA technologies | ||
| PCR thermocycler for thin-walled PCR tubes | Bio-Rad | 1851148 | |
| PCR thermocycler for 0.5 mL tubes | Techne | 5PRIME/C | |
| pET31b-T7-Spinach2 Plasmid | Addgene | Plasmid #79783 | |
| Phusion High-Fidelity DNA polymerase | New England Biolabs | M0530L | Purchase of Phusion High-Fideldity Enzyme is supplied with 5x Phusion HF Buffer, 5x Phusion GC Buffer, and MgCl2 and DMSO solutions. |
| Polyacrylamide gel electrophoresis gel comb, C.B.S. Scientific | C.B.S. Scientific | VGC-1508 | |
| Polyacrylamide gel electrophoresis equipment | C.B.S. Scientific | ASG-250 | |
| Potassium chloride (KCl) | Sigma-Aldrich | P9333 | |
| Potassium phosphate monobasic | Sigma-Aldrich | P5655 | |
| Razor blades | Genesee Scientific | 38-101 | |
| rNTPs: ATP, CTP, GTP, UTP | New England Biolabs | N0450L | |
| SDS | Sigma-Aldrich | L3771 | |
| Short wave UV light source | Thermo Fischer Scientific | 11758221 | |
| Sodium carbonate (Na2CO3) | Sigma-Aldrich | S7795 | |
| Sodium chloride (NaCl) | Sigma-Aldrich | S7653 | |
| Sodium hydroxide (NaOH) | Sigma-Aldrich | S8045 | |
| Sodium phosphate dibasic, anhydrous | Thermo Fischer Scientific | S375-500 | |
| SoftMax Pro | Molecular Devices | N/A | SoftMax Pro 6.5.1 (platereader software) obtained through Academic Group License |
| Sterile filter units | Thermo Fischer Scientific | 09-741-88 | |
| Sucrose | Sigma-Aldrich | S0389 | |
| SYBR Safe DNA gel stain | Thermo Fischer Scientific | S33102 | |
| TAE buffer for agarose gel electrophoresis | Thermo Fischer Scientific | AM9869 | |
| Tetramethylethylenediamine (TEMED) | Sigma-Aldrich | T9281 | |
| Tris base | Sigma-Aldrich | TRIS-RO | |
| Tryptone (granulated) | Thermo Fischer Scientific | M0251S | |
| T7 RNA polymerase | New England Biolabs | M0251S | |
| Urea-PAGE Gel system | National Diagnostics | EC-833 | |
| UV fluorescent TLC plate | Sigma-Aldrich | 1.05789.0001 | |
| UV/Vis spectrophotometer | Thermo Fischer Scientific | ND-8000-GL | |
| Vortex mixer | Thermo Fischer Scientific | 2215415 | |
| Xylene cyanol | Sigma-Aldrich | X4126 | |
| Yeast Extract (Granulated) | Thermo Fischer Scientific | BP9727-2 |
References
- Su, Y., Hammond, M. C. RNA-based fluorescent biosensors for live cell imaging of small molecules and RNAs. Current Opinion in Biotechnology. 63, 157-166 (2020).
- Zhang, J., et al. Tandem spinach array for mRNA Imaging in living bacterial cells. Scientific Reports. 5, 17295(2015).
- Wang, Z., et al. In spatial complementation of aptamer-mediated recognition enables live-cell imaging of native RNA transcripts in real time. Angewandte Chemie. 57 (4), 972-976 (2018).
- Strack, R. L., Disney, M. D., Jaffrey, S. R. A superfolding Spinach2 reveals the dynamic nature of trinucleotide repeat-containing RNA. Nature Methods. 10 (12), 1219-1224 (2013).
- Thavarajah, W., et al. Point-of-use detection of environmental fluoride via a cell-free riboswitch-based biosensor. ACS Synthetic Biology. 9 (1), 10-18 (2020).
- You, M., Litke, J. L., Jaffrey, S. R. Imaging metabolite dynamics in living cells using a Spinach-based riboswitch. Proceedings of the National Academy of Sciences of the United States of America. 112 (21), 2756-2765 (2015).
- Kellenberger, C. A., Wilson, S. C., Sales-Lee, J., Hammond, M. C. RNA-based fluorescent biosensors for live cell imaging of second messengers cyclic di-GMP and cyclic AMP-GMP. Journal of the American Chemical Society. 135 (13), 4906-4909 (2013).
- Manna, S., Truong, J., Hammond, M. C. Guanidine biosensors enable comparison of cellular turn-on kinetics of riboswitch-based biosensor and reporter. ACS Synthetic Biology. 10 (3), 566-578 (2021).
- Bose, D., Su, Y., Marcus, A., Raulet, D. H., Hammond, M. C. An RNA-based fluorescent biosensor for high-throughput analysis of the cGAS-cGAMP-STING pathway. Cell Chemical Biology. 23 (12), 1539-1549 (2016).
- Wang, X. C., Wilson, S. C., Hammond, M. C. Next-generation RNA-based fluorescent biosensors enable anaerobic detection of cyclic di-GMP. Nucleic Acids Research. 44 (17), 139(2016).
- Paige, J. S., Thinh, N. -D., Wenjiao, S., Jaffrey, S. R. Fluorescence imaging of cellular metabolites with RNA. Science. 335 (6073), 1194(2012).
- Paige, J. S., Wu, K. Y., Jaffrey, S. R. RNA mimics of green fluorescent protein. Science. 333 (6042), 642-646 (2011).
- Filonov, G. S., Moon, J. D., Svensen, N., Jaffrey, S. R. Broccoli: Rapid selection of an RNA mimic of green fluorescent protein by fluorescence-based selection and directed evolution. Journal of the American Chemical Society. 136 (46), 16299-16308 (2014).
- Song, W., Strack, R. L., Svensen, N., Jaffrey, S. R. Plug-and-play fluorophores extend the spectral properties of spinach. Journal of the American Chemical Society. 136 (4), 1198-1201 (2014).
- Sambrook, J., Fritsch, E., Maniatis, T. Molecular Cloning: A Laboratory Manual. , Cold Spring Harbor Laboratory Press. Cold Spring Harbor, NY. (1989).
- Basch, H., Gadebusch, H. H. In vitro antimicrobial activity of dimethylsulfoxide. Applied Microbiology. 16 (12), 1953-1954 (1968).
- Kallansrud, G., Ward, B. A comparison of measured and calculated single- and double-stranded oligodeoxynucleotide extinction coefficients. Analytical Biochemistry. 236 (1), 134-138 (1996).
- Wilson, S. C., Cohen, D. T., Wang, X. C., Hammond, M. C. A neutral pH thermal hydrolysis method for quantification of structured RNAs. RNA. 20 (7), 1153-1160 (2014).
- Szatmári, D., et al. Intracellular ion concentrations and cation-dependent remodelling of bacterial MreB assemblies. Scientific Reports. 10, 12002(2020).
- Boulos, L., Prévost, M., Barbeau, B., Coallier, J., Desjardins, R. LIVE/DEAD® BacLightTM: Application of a new rapid staining method for direct enumeration of viable and total bacteria in drinking water. Journal of Microbiological Methods. 37 (1), 77-86 (1999).
- Huang, H., et al. A G-quadruplex-containing RNA activates fluorescence in a GFP-like fluorophore. Nature Chemical Biology. 10 (8), 686-691 (2014).
- Jeng, S. C. Y., Chan, H. H. Y., Booy, E. P., McKenna, S. A., Unrau, P. J. Fluorophore ligand binding and complex stabilization of the RNA Mango and RNA Spinach aptamers. RNA. 22 (12), 1884-1892 (2016).
- Han, K. Y., Leslie, B. J., Fei, J., Zhang, J., Ha, T. Understanding the photophysics of the Spinach-DFHBI RNA aptamer-fluorogen complex to improve live-cell RNA imaging. Journal of the American Chemical Society. 135 (50), 19033-19038 (2013).
- Wang, P., et al. Photochemical properties of Spinach and its use in selective imaging. Chemical Science. 4 (7), 2865-2873 (2013).
- Dao, N. T., et al. Photophysics of DFHBI bound to RNA aptamer Baby Spinach. Scientific Reports. 11, 7356(2021).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved