Method Article
Determinación de cinética de encendido in vitro y celular para aptámeros de ARN fluorogénicos
* Estos autores han contribuido por igual
En este artículo
Resumen
El protocolo presenta dos métodos para determinar la cinética de los aptámeros de ARN fluorogénico Spinach2 y Brócoli. El primer método describe cómo medir la cinética del aptámero fluorogénico in vitro con un lector de placas, mientras que el segundo método detalla la medición de la cinética del aptámero fluorogénico en las células mediante citometría de flujo.
Resumen
Los aptámeros de ARN fluorogénico se han aplicado en células vivas para etiquetar y visualizar ARN, informar sobre la expresión génica y activar biosensores fluorescentes que detectan niveles de metabolitos y moléculas de señalización. Para estudiar los cambios dinámicos en cada uno de estos sistemas, es deseable obtener mediciones en tiempo real, pero la precisión de las mediciones depende de que la cinética de la reacción fluorogénica sea más rápida que la frecuencia de muestreo. Aquí, describimos métodos para determinar la cinética de encendido in vitro y celular para aptámeros de ARN fluorogénicos utilizando un lector de placas equipado con un inyector de muestras y un citómetro de flujo, respectivamente. Mostramos que la cinética in vitro para la activación de fluorescencia de los aptámeros de espinaca2 y brócoli se puede modelar como reacciones de asociación de dos fases y tienen diferentes constantes de velocidad de fase rápida de 0.56 s−1 y 0.35 s−1, respectivamente. Además, mostramos que la cinética celular para la activación de fluorescencia de la espinaca2 en Escherichia coli, que está aún más limitada por la difusión del colorante en las bacterias gramnegativas, sigue siendo lo suficientemente rápida como para permitir una frecuencia de muestreo precisa en la escala de tiempo diminuta. Estos métodos para analizar la cinética de activación de fluorescencia son aplicables a otros aptámeros de ARN fluorogénicos que se han desarrollado.
Introducción
Las reacciones fluorogénicas son reacciones químicas que generan una señal de fluorescencia. Los aptámeros de ARN fluorogénico suelen realizar esta función uniendo un colorante de molécula pequeña para mejorar su rendimiento cuántico de fluorescencia (Figura 1A)1. Se han desarrollado diferentes sistemas de aptámeros de ARN fluorogénicos que consisten en secuencias específicas de aptámeros de ARN y los correspondientes ligandos de colorante1. Los aptámeros de ARN fluorogénicos se han añadido a las transcripciones de ARN como etiquetas fluorescentes que permiten obtener imágenes de células vivas de ARNm y ARN no codificantes 2,3,4. También se han colocado después de secuencias promotoras como reporteros fluorescentes de expresión génica, similar al uso de proteína fluorescente verde (GFP) como reportero, excepto que la función de informe está en el nivel de ARN 5,6. Finalmente, los aptámeros de ARN fluorogénico se han incorporado a biosensores fluorescentes basados en ARN, que están diseñados para desencadenar la reacción fluorogénica en respuesta a una molécula pequeña específica. Se han desarrollado biosensores fluorescentes basados en ARN para obtener imágenes de células vivas de varios metabolitos no fluorescentes y moléculas de señalización 7,8,9,10,11.
Existe un creciente interés en el desarrollo de aptámeros de ARN fluorogénicos para visualizar cambios dinámicos en la localización del ARN, la expresión génica y las señales de moléculas pequeñas. Para cada una de estas aplicaciones, es deseable obtener mediciones en tiempo real, pero la precisión de las mediciones depende de que la cinética de la reacción fluorogénica sea más rápida que la frecuencia de muestreo. Aquí, describimos métodos para determinar la cinética in vitro para los aptámeros de ARN fluorogénicos Spinach212 y Brócoli13 utilizando un lector de placas equipado con un inyector de muestras y para determinar la cinética de activación celular para Spinach2 expresada en Escherichia coli utilizando un citómetro de flujo. Estos dos aptámeros de ARN fueron elegidos porque se han aplicado para estudiar la localización de ARN 2,3,4, se han utilizado en reporteros5,6 y biosensores 7,8,9,10,11, y los ligandos de colorante correspondientes (DFHBI o DFHBI-1T) están disponibles comercialmente. Un resumen de sus propiedades in vitro determinadas en la literatura se da en la Tabla 1 4,13,14, que informó el desarrollo del protocolo (por ejemplo, las longitudes de onda y las concentraciones de colorantes utilizadas). Estos resultados demuestran que las reacciones fluorogénicas afectadas por los aptámeros de ARN son rápidas y no deben impedir mediciones precisas para las aplicaciones biológicas celulares deseadas.
Protocolo
1. Experimento cinético in vitro
- Preparación de plantillas de ADN por PCR
- Configure la(s) reacción(es) de PCR: Para preparar reacciones de PCR, combine los siguientes reactivos en un tubo de PCR de pared delgada:
33 μL de agua de doble destilación (ddH2O)
10 μL de tampón 5x para ADN polimerasa de alta fidelidad
5 μL de 2 mM de cada desoxirribonucleósido trifosfato (dNTP)
0,5 μL de cebador directo de 40 μM
0,5 μL de cebador inverso de 40 μM
0,5 μL (10-100 ng) de plantilla de ADN (solo para PCR de espinacas2; Los cebadores de brócoli se superponen)
0,5 μL de ADN polimerasa de alta fidelidad (añadir el último)
NOTA: Los oligonucleótidos sintéticos a menudo se envían secos. Para preparar soluciones madre, agregue un volumen conocido (se recomiendan 100 μL) deddH2O, y mida la A260 de esa solución para determinar la concentración según la ley de Beer, donde el coeficiente de extinción se puede calcular mediante reglas de vecino más cercano en línea. Esta solución madre se puede utilizar para hacer diluciones apropiadas para su uso en PCR. - Ejecute el protocolo del termociclador.
- Utilice el siguiente protocolo de termociclador para amplificar las plantillas completas de ADN de espinaca2 y brócoli:
Desnaturalización inicial: 98 °C durante 2 min
35 ciclos:
Desnaturalización:98 °C desnaturalización durante 15 s
Recocido:72 °C durante 30 s
Extensión:72 °C durante 30 s
Extensión final: 72 °C durante 5 min. - Después de la reacción, analice una pequeña alícuota del producto en gel de agarosa al 2% junto con una escalera de bajo peso molecular (rango de tamaño: 25-766 nucleótidos) para confirmar la presencia del producto de ADN deseado.
- Purificar el producto mediante extracción en gel disponible comercialmente o kits de limpieza por PCR y eluya conddH2Oo el tampón proporcionado por el fabricante.
NOTA: Al elegir un kit de limpieza de PCR, asegúrese de que el límite de peso molecular de la columna sea lo suficientemente bajo como para retener T7-Brócoli (81 nucleótidos), o de lo contrario se perderá el producto de PCR.
NOTA: Punto de pausa opcional: almacenar el ADN a -20 °C.
- Utilice el siguiente protocolo de termociclador para amplificar las plantillas completas de ADN de espinaca2 y brócoli:
- Configure la(s) reacción(es) de PCR: Para preparar reacciones de PCR, combine los siguientes reactivos en un tubo de PCR de pared delgada:
- Preparación de ARN de Espinacas2 y Brócoli por transcripción in vitro (IVT)
- Configure la(s) reacción(es) de transcripción.
- Para preparar una reacción de transcripción de 100 μL, combine los siguientes reactivos en un tubo de microcentrífuga de 1,5 ml: 10 μL de tampón de transcripción 10x + 20 μL de trifosfatos de ribonucleósidos de 10 mM (rNTPs) + 1-64 μL de plantilla de ADN (total de 1 μg) + 2 μL de pirofosfatasa inorgánica +ddH2O a 98 μL + 2 μL de ARN polimerasa T7 (añadir último).
- Incubar esta reacción durante 4 h a 37 °C. Apague la reacción agregando 100 μL de 2x tampón de carga de gel de urea (2x ULB), compuesto de sacarosa al 20%, dodecil sulfato de sodio (SDS) al 0,1%, 1x tampón Tris-Borato-EDTA (1x TBE) y ~18 M de urea.
NOTA: Punto de pausa opcional: conservar la reacción de enfriamiento a -20 °C.
- Configure la(s) reacción(es) de transcripción.
- Electroforesis en gel de poliacrilamida (PAGE) purificación de ARN
- PÁGINA purificación de ARN de espinaca2 y brócoli
PRECAUCIÓN: La acrilamida no polimerizada (líquida o en polvo) es extremadamente tóxica. Si pesa acrilamida en polvo, hágalo en una campana extractora. Siempre use el equipo de protección adecuado y quítese inmediatamente los guantes contaminados con polvo o solución de acrilamida, lavándose bien las manos. Si la acrilamida entra en contacto directo con la piel, lave el área expuesta durante al menos 15 minutos con agua y jabón. Si la acrilamida entra en contacto directo con los ojos, enjuáguelos con agua durante 15 minutos.- Prepare el gel PAGE: Para eliminar las transcripciones abortivas no deseadas y los rNTP sin reaccionar del producto completo, prepare un gel de urea-poliacrilamida al 6%. En general, los geles de 28 cm x 16,5 cm x 1,5 mm se pueden usar con un peine de 8 pocillos. Configure el equipo de gel y electroforesis, utilizando 1x tampón TBE para llenar los depósitos.
- Cargue la(s) muestra(s) de ARN en el gel PAGE: Cargue el gel con una reacción apagada de 200 μL por carril. En un carril separado, cargue 2x ULB con colorantes rastreadores de cianol de xileno y azul de bromofenol, que migran en el gel a 106 nucleótidos y 26 nucleótidos, respectivamente15. Deje un carril vacío entre cada muestra para evitar una posible contaminación en los próximos pasos.
- Ejecute el gel PAGE: Para separar las espinacas de 95 nt2 y el brócoli de 49 nt de sus respectivos productos truncados, ejecute el gel durante 1.5-2 h a 25 W, momento en el cual el colorante azul de bromofenol habrá migrado ~ 5/6 de la longitud del gel.
- Visualice la(s) muestra(s) de ARN en el gel PAGE: Desmonte las placas de vidrio alrededor del gel y cubra el gel con una envoltura de plástico en ambos lados, etiquetando los carriles de la envoltura. Visualice las bandas de ARN en una habitación oscura mediante sombra UV colocando el gel envuelto en una placa TLC fluorescente bajo luz UV. Delinee rápidamente el borde de las bandas de ARN correspondientes al producto con un marcador y apague la lámpara UV para minimizar el daño causado por la exposición a los rayos UV.
- Extirpe y extraiga la(s) muestra(s) de ARN del gel PAGE: Con una cuchilla de afeitar nueva para cada muestra, extraiga las bandas de producto deseadas, corte en cubos de ~ 1 mm y agregue las piezas de gel a un tubo de microcentrífuga de 2 ml con 500 μL de tampón de remojo de trituración para extraer ARN en un rotador durante 2 h a temperatura ambiente (RT) o durante la noche a 4 °C.
NOTA: Punto de pausa opcional: la muestra puede almacenarse a -20 °C. - Precipitar el ARN
- Para separar las piezas de gel del ARN extraído en tampón, centrifugar la muestra a 13.000 x g durante 20 min a 4 °C, y luego utilizar una pipeta de punta estrecha para extraer el sobrenadante y cargarlo en un nuevo tubo de microcentrífuga de 2 ml.
- Para precipitar el ARN, agregue 1,5 ml de etanol helado y 1 μL de glucógeno de 20 mg / ml de vórtice y guárdelo durante al menos 1 h a -20 ° C o -80 ° C.
NOTA: Punto de pausa opcional: el ARN puede almacenarse a -20 °C durante unos meses.
- Recolección del precipitado de ARN: Granular el ARN precipitado por centrifugación a 13.000 x g durante 20 min a 4 °C. Retire el sobrenadante y permita que el etanol restante se evapore al aire libre (~ 1 h) antes de resuspender el pellet en 30 μL de ddH2O o 1x tampón TE.
NOTA: Este proceso generalmente resulta en una concentración final de ARN de ~ 10 μM.
NOTA: Punto de pausa opcional: la muestra de ARN puede almacenarse a -20 °C durante unos meses.
- Determinar las concentraciones de ARN.
- Preparar una alícuota de ARN para la reacción de hidrólisis.
- Para realizar este ensayo, primero, use un nano espectrofotómetro UV / Vis para determinar el A260 de la muestra de ARN madre y haga una alícuota diluida de la muestra a ~ 10 unidades de absorbancia (AU) en ddH2O.
- Preparar la siguiente reacción en un tubo de PCR de 0,5 ml: 16 μL de ddH2O + 2 μL de tampón 10x Na2CO 3 + 2μL de ARN alícuota diluido a ~10 UA. Incubar la reacción durante 90 min a 95 °C, y luego dejar que se enfríe a RT.
- Determine la concentración de ARN utilizando absorbancias de nucleótidos: Mida el A260 de esta muestra con un nano espectrofotómetro UV/Vis y calcule la concentración de ARN utilizando la siguiente fórmula:

donde c es la concentración de ARN, b es la longitud del camino, i es el nucleótido específico (A, C, G o U), n i es la frecuencia del nucleótido i en la secuencia de ARN y ε i es el coeficiente de extinción molar del nucleótido i. Para determinar la concentración original de ARN madre, multiplique c por el factor de dilución.
NOTA: Punto de pausa opcional: El ARN puede almacenarse a -20 °C durante unos meses.
- Preparar una alícuota de ARN para la reacción de hidrólisis.
- PÁGINA purificación de ARN de espinaca2 y brócoli
- Realización de un in vitro Ensayo cinético del lector de placas
- Configure el programa de renaturalización de ARN: Cree el siguiente protocolo de termociclador seleccionando Crear nuevo programa > Agregar nueva fase > Agregar nuevo paso varias veces para agregar cada uno de los siguientes pasos antes de presionar Guardar:
70 °C durante 3 min
65 °C durante 45 s
60 °C durante 45 s
55 °C durante 45 s
50 °C durante 45 s
45 °C durante 45 s
40 °C durante 45 s
35 °C durante 45 s
30 °C durante 45 s - Renaturalizar el ARN: Para renaturalizar los ARN de espinaca2 y brócoli, prepare 2 μM de cada ARN enddH2Oen un tubo de PCR de pared delgada de 0,5 ml, y luego agregue un volumen igual de tampón de renaturalización 2x (80 mM HEPES, pH 7.5 [KOH], 250 mM KCl, 6 mM MgCl2) para producir soluciones de ARN de 1 μM. Agregue los tubos al termociclador, abra el programa de renaturalización guardado y presione Ejecutar.
NOTA: Si un termociclador no está disponible, los ARN pueden incubarse en un bloque de calor de 70 ° C durante 3 minutos y luego dejarse enfriar lentamente a RT en el banco. - Prepare el tampón de reacción de unión: Para preparar el tampón para una reacción de unión aptámero-colorante, haga una mezcla maestra que contenga componentes tampón (69.5 μL deddH2O + 4 μL de 1 M HEPES, pH 7.5 [KOH] + 6.2 μL de 2 M KCl + 0.3 μL de 1 M MgCl2). Dependiendo de cuántas muestras y réplicas se necesiten, multiplique estos valores por el número de muestras más uno. En general, tres réplicas por muestra de ARN es satisfactorio.
- Prepare el lector de placas.
- Configure el programa del inyector del lector de placas: En el lector de placas de fluorescencia, seleccione Temperatura y ajuste la temperatura deseada a 37 °C, asegurándose de que la temperatura se haya equilibrado con este valor mucho antes de comenzar los experimentos cinéticos. Abra el software del lector de placas, seleccione Configuración > Vista de adquisición e introduzca el siguiente programa para mediciones cinéticas:
Bucle: Para cada pozo
Configuración de línea base: 60 lecturas de línea base
Ajustes de SmartInject: inyección de 10 μL (que ocurrirá después de las lecturas de referencia)
La fluorescencia (o FL) dice:
Excitación: 448 nm (ancho de banda: 9 nm)
Emisión: 506 nm (ancho de banda: 15 nm)
Cartucho: MONO (s/n 3297)
Cronometraje:
Tiempo total de lectura: 10 min
Intervalo de lectura: 0,5 s
PMT y óptica: 6 destellos por lectura
Bucle: Siguiente pozo - Preparar el inyector
- Lave y aspire el inyector: Para preparar el inyector lector de placas, primero seleccione Inyectar en el lector de placas, suministre una placa de recolección de residuos al lector de placas cuando se le indique, y luego seleccione Lavar y limpie el tubo de inyección siguiendo las instrucciones del lector de placas con volúmenes de 1 ml de ddH 2 O, etanol al 75% en ddH2O, y luego ddH2O. A continuación, seleccione Aspirado, permitiendo que el inyector expulse el exceso de líquido.
- Prepare el inyector: Después de salir a la pantalla anterior, seleccione Prime para preparar el inyector con dos volúmenes de ligando de 260 μL que se inyectarán para garantizar que se agregue un ligando puro y concentrado a las muestras durante los experimentos; en este caso, cebador con 100 μM DFHBI en ddH2O.
- Configure el programa del inyector del lector de placas: En el lector de placas de fluorescencia, seleccione Temperatura y ajuste la temperatura deseada a 37 °C, asegurándose de que la temperatura se haya equilibrado con este valor mucho antes de comenzar los experimentos cinéticos. Abra el software del lector de placas, seleccione Configuración > Vista de adquisición e introduzca el siguiente programa para mediciones cinéticas:
- Realizar experimentos cinéticos in vitro : Para realizar un experimento cinético, primero agregue 80 μL de mezcla maestra de tampón de unión previamente preparada a un pocillo de una placa de fondo transparente de 96 pocillos, seguido de 10 μL de ARN renaturalizado. Deje que esta placa y la solución DFHBI en el inyector se equilibren a 37 °C durante 15 minutos en el lector de placas.
- En la configuración del software del lector de placas en Área de lectura, seleccione el pozo que desea analizar y, a continuación, en la pestaña Inicio , seleccione Ejecutar para ejecutar el programa de cinética descrito anteriormente. Repita este proceso hasta que se completen todos los experimentos.
NOTA: Críticamente, los experimentos cinéticos deben realizarse uno bien a la vez para garantizar que la cinética de ARN se mida en condiciones idénticas entre réplicas y muestras. - Lave el inyector: Para extraer la solución DFHBI restante del tubo de inyección, lave el tubo de inyección como se describe en el paso 1.4.4.2.1 con volúmenes de 1 ml deddH2O, etanol al 75% enddH2Oy luego ddH2O.
NOTA: Punto de pausa opcional.
- Configure el programa de renaturalización de ARN: Cree el siguiente protocolo de termociclador seleccionando Crear nuevo programa > Agregar nueva fase > Agregar nuevo paso varias veces para agregar cada uno de los siguientes pasos antes de presionar Guardar:
- Análisis de la cinética in vitro de aptámeros de ARN fluorogénicos.
- Ingrese datos en el software de análisis: exporte datos experimentales como una hoja de cálculo para copiar fácilmente los datos para su procesamiento. En el software de análisis, cree una nueva tabla de datos en formato XY. En la columna X, introduzca cada punto de tiempo de lectura, siendo t = 0 la hora de la inyección DFHBI. En la columna Y, introduzca los valores de fluorescencia promedio entre réplicas en ese punto de tiempo respectivo comenzando en t = 0.
- Normalizar y graficar los datos: Para normalizar los valores de fluorescencia, haga clic en Analizar > procesamiento de datos > Normalizar y, a continuación, haga clic en Aceptar. Elija usar los valores más pequeños y más grandes del conjunto de datos para la normalización, para presentar los resultados como fracciones y para Graficar los resultados y, a continuación, haga clic en Aceptar. Para crear un gráfico de la fluorescencia promediada normalizada a lo largo del tiempo, haga clic en el icono Normalizar de [Nombre del conjunto de datos] y seleccione Familia de gráficos: XY con solo puntos.
NOTA: Las barras de error pueden ser útiles para ver en los casos en que se grafica un pequeño número de puntos de tiempo. Si lo desea, al elegir una tabla de datos de formato XY, seleccione la opción debajo de "Y" para ingresar valores replicados en columnas lado a lado. Al graficar la tabla resultante con las opciones predeterminadas seleccionadas, se producirá un gráfico con barras de error. - Realizar ajuste de curva para obtener parámetros cinéticos: Para ajustar una curva a los datos cinéticos, haga clic en Analizar > Analizar datos y seleccione Regresión no lineal (Ajuste de curva) en la pestaña Análisis XY . En la pestaña Modelo , haga clic en Asociación exponencial > de dos fases para ajustar los datos cinéticos con la siguiente ecuación de asociación de dos fases:
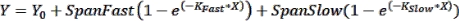
donde Y es la fluorescencia en el momento X, Y 0 es la fluorescencia en t = 0, K Rápido y KLento son constantes de velocidad rápida y lenta, respectivamente, y SpanFast y SpanSlow son los rangos de activación de fluorescencia explicados por las velocidades rápidas y lentas, respectivamente (ver resultados representativos, Figura 1). Haga clic en la pestaña Nonlin Fit para ver las constantes de velocidad, t1/2 valores y valores PercentFast.
NOTA: Para obtener una desviación estándar para todos estos valores, las réplicas individuales del experimento del lector de placas se pueden procesar de la misma manera que se describió anteriormente.
2. Experimento de cinética celular
- Preparación de cepas de E. coli
- Transformar células BL21 Star (DE3) de E. coli con ~100 ng de construcción pET31b tRNA-Spinach2 siguiendo el protocolo del fabricante.
NOTA: La construcción de plásmido está disponible comercialmente (Plásmido #79783). - Colocar en placa las células en placas de agar LB que contengan carbenicilina (Carb: 50 mg/mL) e incubar a 37 °C durante 12-16 h. Las células que contienen plásmidos crecerán como colonias en la placa.
NOTA: Punto de pausa opcional: Las celdas BL21 Star transformadas en placas se pueden almacenar a 4 °C envueltas en parafilm durante 1 semana.
- Transformar células BL21 Star (DE3) de E. coli con ~100 ng de construcción pET31b tRNA-Spinach2 siguiendo el protocolo del fabricante.
- Crecimiento de células e inducción de la expresión de aptámero de ARN fluorogénico
- Inocular un cultivo de 2 ml de medios no inductores (NI) que contengan carbenicilina (Carb: 50 mg/ml) con una sola colonia de las células BL21 Star transformadas. Repita esto para al menos tres réplicas biológicas. Incubar los cultivos a 37 °C en una incubadora/agitadora a 250 rpm durante 22-24 h.
NOTA: Punto de pausa opcional: Las células cultivadas en medios de NI retienen el plásmido y pueden almacenarse a 4 °C durante 1 semana. - Después del crecimiento en medios de NI, diluya el cultivo 100x en 3 ml frescos de medios de autoinducción (AI) ZYP-5052 que contengan carbenicilina (carbohidratos: 50 mg / ml). Cultivar las células a 37 °C en una incubadora/agitadora a 250 rpm durante 16-18 h para inducir la expresión.
NOTA: El OD600 típico para cultivos oscilará entre 2.0-3.3 después de 18 h de crecimiento. El rango óptimo de densidad celular está entre 2.5-3.0.
- Inocular un cultivo de 2 ml de medios no inductores (NI) que contengan carbenicilina (Carb: 50 mg/ml) con una sola colonia de las células BL21 Star transformadas. Repita esto para al menos tres réplicas biológicas. Incubar los cultivos a 37 °C en una incubadora/agitadora a 250 rpm durante 22-24 h.
- Realización del experimento de cinética celular
- Configure el citómetro de flujo.
- Encienda el citómetro de flujo y la computadora conectados al instrumento. Una vez que haya iniciado sesión en el software del citómetro de flujo, en la pestaña Instrumento , haga clic en el icono Inicio . Siga los pasos indicados en la pantalla del software para garantizar la inicialización correcta de la instrumentación.
NOTA: Algunos citómetros de flujo llaman a la secuencia de inicio del instrumento Priming. Asegúrese de seguir el protocolo del fabricante para el citómetro de flujo que se utilizará para el experimento. - Ejecute una prueba de rendimiento (si corresponde). En la pestaña Menú principal , haga clic en Prueba de rendimiento. En un tubo de cultivo, agregue tres gotas de las perlas de seguimiento del rendimiento del fabricante en 3 ml de fluido de enfoque.
- Coloque el tubo de cultivo en el elevador de tubos de muestra y levante el elevador. Antes de hacer clic en Ejecutar prueba de rendimiento, asegúrese de que el lote # del tubo de las cuentas de seguimiento sea el mismo que se indica en la pantalla Configuración de prueba de rendimiento . Haga clic en Prueba de rendimiento para ejecutar la prueba.
- Configure el software del citómetro de flujo para este experimento con los siguientes parámetros de adquisición para la fluorescencia de una sola célula:
Láser de excitación: 488 nm
Canal de emisión: GFP (también llamado FITC)
Volumen de adquisición: 40 μL (con un volumen total de extracción de 90 μL)
Caudal: 200 μL / min
Recuentos de células para cada medición: 30,000
Configuración del instrumento:
Voltaje:
FSC: 480 V
SSC: 400 V
BL1: 540 V
BL2: 392 V
BL3: 422 V
- Encienda el citómetro de flujo y la computadora conectados al instrumento. Una vez que haya iniciado sesión en el software del citómetro de flujo, en la pestaña Instrumento , haga clic en el icono Inicio . Siga los pasos indicados en la pantalla del software para garantizar la inicialización correcta de la instrumentación.
- Configure los archivos experimentales.
- Cree un nuevo archivo de experimento en la pestaña Explorador de experimentos haciendo clic con el botón derecho en el nombre de usuario del citómetro de flujo. Seleccione Nuevo experimento en la ventana desplegable. Cuando aparezca una nueva ventana en la pantalla de la computadora, seleccione Aceptar.
- En el nuevo archivo de experimento, haga clic derecho en la carpeta "Grupo" y seleccione Agregar nuevo tubo de muestra. Etiquete los tubos de muestra para cada punto de tiempo específico y replique haciendo clic derecho en Muestra y seleccionando Cambiar nombre en el menú desplegable. Repita este paso para el número total de réplicas y puntos de tiempo para el curso de tiempo previsto del estudio.
- Prepare una solución celular diluida: En un nuevo tubo de cultivo, agregue 1.5 ml de 1x solución de PBS. A continuación, agregue 3 μL de células inducidas en medios de IA en la solución 1x PBS para hacer una solución celular diluida. Repita este paso para cada réplica biológica en diferentes tubos de cultivo.
- Mida la fluorescencia de fondo de las células: Antes de agregar el tinte, tome lecturas de cada tubo de cultivo de replicación biológica que contenga células en una solución de PBS 1x. Esto es para que el fondo fluorescente de las células se mida para observar el pliegue que se enciende con el tiempo una vez que se agrega el tinte.
- Para tomar una lectura, coloque el tubo de cultivo en el elevador de tubos de muestra y levante el elevador con la mano hacia la aguja de inyección de muestra. Seleccione el archivo de muestra adecuado en la ficha Panel de recopilación y haga clic en Grabar.
- Cuando se complete la carrera, baje el elevador de tubos de muestra con el tubo de cultivo a mano. Esto iniciará un paso de enjuague que enjuagará el sistema fluídico y minimizará el arrastre entre cada muestra de réplica biológica. Los datos se guardarán automáticamente en el equipo una vez finalizada la ejecución.
- Repita los pasos dentro de 2.3.4 para medir el fondo fluorescente celular para al menos tres réplicas biológicas. Para pasar al siguiente archivo de ejemplo, seleccione el siguiente archivo de ejemplo haciendo clic en el icono de flecha derecha cerca del nombre del tubo de muestra debajo del icono Grabar.
- Mida la fluorescencia en puntos de tiempo para las células con tinte.
- Añadir 1,4 μL de un colorante concentrado (50 mM DFHBI-1T en DMSO) en 1x solución de PBS con células para obtener una concentración final de 50 μM DFBHI-1T. A continuación, asegure la tapa del tubo de cultivo y luego invierta los tubos de cultivo 3x-5x para mezclar la solución de manera uniforme antes de tomar la primera lectura del punto de tiempo.
NOTA: El porcentaje total de DMSO dentro de los tubos de cultivo para E. coli no debe exceder el 10%, ya que esto puede afectar la viabilidad celular16. - Retire la tapa y coloque el tubo de cultivo en el elevador de tubos de muestra. Levante el soporte con la mano hacia la aguja de inyección de muestra y, debajo del archivo de muestra adecuado, haga clic en el icono Grabar . Además, inicie un temporizador presionando Inicio para el experimento.
- Baje el elevador de tubos a mano una vez finalizada la carrera y repita los pasos 2.3.5.1-2.3.5.2. (con el temporizador funcionando) agregando 1.4 μL del DFHBI-1T concentrado, invirtiendo los tubos de cultivo y tomando lecturas para todas las réplicas biológicas restantes. Estas primeras grabaciones serán las lecturas obtenidas a 0 min para todas las réplicas biológicas. Haga esto uno a la vez para cada réplica biológica.
NOTA: Anote el momento en que se presiona la adquisición de citometría de flujo de registro. Adhiérase a este escalonamiento de tiempo para los puntos de tiempo en el transcurso del experimento. - Continúe tomando lecturas elevando los tubos de cultivo en el elevador de tubos de muestra a la aguja de inyección de muestra, seleccionando el archivo de muestra adecuado, haciendo clic en Grabar y bajando el elevador con la mano después de completar cada carrera. Haga esto para todos los puntos de tiempo adicionales y réplicas biológicas que se están probando. Repita los pasos hasta que se complete el experimento.
NOTA: Mantenga las muestras fuera de la luz para evitar el fotoblanqueo de DFHBI-1T en solución cubriendo las muestras con papel de aluminio.
- Añadir 1,4 μL de un colorante concentrado (50 mM DFHBI-1T en DMSO) en 1x solución de PBS con células para obtener una concentración final de 50 μM DFBHI-1T. A continuación, asegure la tapa del tubo de cultivo y luego invierta los tubos de cultivo 3x-5x para mezclar la solución de manera uniforme antes de tomar la primera lectura del punto de tiempo.
- Medir la fluorescencia en puntos de tiempo para células sin colorante (Control).
- Ejecute los protocolos de limpieza apropiados para el citómetro de flujo antes de repetir el experimento nuevamente con controles negativos siguiendo el protocolo del fabricante. Esto se hace para minimizar cualquier transferencia del experimento anterior al experimento de análisis de puntos de tiempo de control. A continuación se presentan los pasos seguidos para el citómetro de flujo entre experimentos:
- Coloque un tubo de cultivo vacío en el elevador de tubos con la mano, levante el soporte del tubo y haga clic en el icono Destapar en la pestaña Instrumento . Esto ejecutará un retrolavado en el sistema de fluidos para limpiar cualquier muestra pegajosa. Baje el soporte del tubo a mano y retire el tubo una vez que se haya realizado la secuencia de desobstrucción .
- Con un tubo de cultivo nuevo, agregue 3 ml de una solución de lejía al 10%, coloque el tubo de cultivo en el soporte del tubo y levante el soporte con la mano hacia la aguja de inyección de muestra. Además, coloque una placa limpia de 96 pocillos en el muestreador automático si corresponde al citómetro de flujo.
- Haga clic en el icono SIP Sanitize SIP/Sanitize Autosampler para ejecutar una secuencia de limpieza con un 10% de lejía en todo el sistema de fluidos. Baje el portatubos para completar la secuencia de limpieza.
- Establezca los archivos de ejemplo para la ejecución del análisis de puntos de tiempo de control siguiendo las instrucciones del paso 2.3.3.
- En un nuevo tubo de cultivo, prepare una solución celular diluida en 1,5 ml de 1x solución de PBS. Agregue 3 μL de las células inducidas en medios de IA en la solución 1x PBS para hacer una solución celular diluida. Repita este paso para cada réplica biológica.
- Agregue 1.4 μL de DMSO en el tubo de cultivo uno a la vez a la solución 1x PBS con células y pruebe los mismos puntos de tiempo. Asegure la tapa del tubo de cultivo y luego invierta los tubos de cultivo 3x-5x para mezclar la solución uniformemente antes de tomar la primera lectura del punto de tiempo. Haga esto uno a la vez para cada réplica biológica.
- Siga el mismo protocolo para el análisis de células de control que para células con colorante, utilizando los pasos 2.3.5.2-2.3.5.4.
- Ejecute los protocolos de limpieza apropiados para el citómetro de flujo antes de repetir el experimento nuevamente con controles negativos siguiendo el protocolo del fabricante. Esto se hace para minimizar cualquier transferencia del experimento anterior al experimento de análisis de puntos de tiempo de control. A continuación se presentan los pasos seguidos para el citómetro de flujo entre experimentos:
- Apague el citómetro de flujo: Siga el protocolo del fabricante para el apagado adecuado de la instrumentación. Para el citómetro de flujo, el instrumento está preparado para el apagado de la siguiente manera:
- Realice el protocolo de limpieza inicial para el citómetro de flujo siguiendo los pasos 2.3.6.1.1-2.3.6.1.3.
- Reemplace el tubo de cultivo con una solución de lejía al 10% con un tubo de cultivo con 3 ml de líquido de enfoque. Levante el portatubos con la mano y, debajo del icono Apagado , haga clic en el menú desplegable y seleccione Completo.
NOTA: Punto de pausa opcional.
- Configure el citómetro de flujo.
- Análisis de los datos de citometría de flujo
- Exporte todos los archivos FCS para su análisis. Abra los archivos FCS con un software de análisis de citometría de flujo.
- Usando uno de los archivos FCS solo de celda, genere una puerta a partir del diagrama de puntos de dispersión directa (FSC) y dispersión lateral (SSC) (FSC-Area/SSC-Area), utilizando ambos ejes de registro para excluir cualquier señal de los desechos. Para crear esta puerta, haga clic en el icono AutoGate en el software de análisis de citometría de flujo y asígnele el nombre Puerta 1. Aplique esta misma puerta a todas las muestras probadas en la ficha Todas las muestras del área de trabajo de procesamiento de datos. Esto dará como resultado que se procese la Puerta 1 debajo de todos los archivos FCS.
- Cree un nuevo archivo de subconjunto con el archivo FCS de solo celda utilizado en el paso 2.4.2 haciendo doble clic en él. Cambie la configuración del eje a FSC-Area/FSC-Height, y ambas utilizan ejes de registro. Haga clic en el icono AutoGate en el software de análisis de citometría de flujo para generar una puerta ovalada, llamándola Puerta 2. Aplique esta puerta como un subconjunto debajo del conjunto de puertas en el paso 2.4.2 a todas las muestras probadas. Esto dará como resultado que todas las muestras tengan Gate 1 > Gate 2 asociadas con cada archivo FCS.
- Cree otro archivo de subconjunto con ambas puertas establecidas en los pasos 2.4.2 y 2.4.3 aplicadas haciendo doble clic en Puerta 2. Cambie la configuración del eje a FSC-Area/Histogram. Aplique esta puerta de histograma como un subconjunto a todas las muestras probadas, lo que hace que todas las muestras tengan la Puerta 1 > la Puerta 2 > la Puerta 2 asociadas con cada archivo FCS.
NOTA: Los histogramas se pueden cambiar de Puerta 2 a Histograma para ayudar a mover los histogramas a la ventana de diseño, así como para crear más organización con el procesamiento de datos. - Para analizar los valores medios de intensidad de fluorescencia (MFI), abra la ventana de diseño. Haga clic y arrastre las puertas del histograma para cada punto de tiempo a la ventana de diseño.
- Realice un análisis estadístico para "∑ Media: BL1-A" (GFP) para cada muestra probada para mostrar los resultados de MFI en la ventana de diseño.
- Calcule la desviación estándar para los valores de MFI por análisis de punto de tiempo para al menos tres réplicas biológicas independientes.
- Guarde los histogramas y los valores de MFI para cada punto de tiempo exportando la ventana de diseño como un archivo PDF.
NOTA: Punto de pausa opcional.
- Graficar datos de citometría de flujo
- Abra el archivo PDF que contiene los histogramas y los valores de MFI para cada punto de tiempo. Los valores de MFI se copiarán en un software de análisis de datos. En el software de gráficos de datos, cree una nueva tabla de datos en formato XY.
- Seleccione esta opción para crear una tabla XY con lo siguiente seleccionado:
Tabla de datos: Introducir o importar datos en una tabla nueva
Opciones:
X: Tiempos transcurridos
Y: Introduzca (de tres a cuatro) valores replicados en subcolumnas en paralelo - Etiqueta en el eje X todos los puntos de tiempo para el experimento y las ejecuciones de control.
- En el Grupo A, ingrese los valores de MFI para todas las réplicas biológicas en cada punto de tiempo para el análisis de fluorescencia de las células con el colorante agregado.
- En el Grupo B, ingrese los valores de MFI para todas las réplicas biológicas en cada punto de tiempo para el análisis de las células sin colorante (DMSO) agregado.
- Para observar los resultados, haga clic en [Insertar nombre del conjunto de datos] en la pestaña Gráficos . Esto mostrará los puntos de datos como medias, con barras de error que representan la desviación estándar (s.d.) en cada punto de tiempo. El eje X representa el tiempo transcurrido y el eje Y representa los valores de MFI.
- Seleccione esta opción para crear una tabla XY con lo siguiente seleccionado:
- Abra el archivo PDF que contiene los histogramas y los valores de MFI para cada punto de tiempo. Los valores de MFI se copiarán en un software de análisis de datos. En el software de gráficos de datos, cree una nueva tabla de datos en formato XY.
Resultados
Cinética in vitro
Las secuencias de los moldes y cebadores de ADN, que se compran como oligonucleótidos sintéticos, se muestran en la Tabla 2, y las recetas de reactivos se muestran en el Archivo Suplementario 1. La amplificación por PCR se utiliza para aumentar la cantidad de plantilla de ADN con el promotor T7, que se requiere para la posterior reacción de transcripción in vitro (IVT). Además, la amplificación por PCR se puede utilizar para dos propósitos en la misma reacción: para generar la plantilla de ADN de brócoli de longitud completa mediante la extensión del cebador, así como para ampliar la plantilla de ADN.
Después de la reacción IVT para sintetizar ARN, la purificación de PAGE eliminará cualquier transcripción truncada no deseada, productos degradados y rNTP sin reaccionar del producto de ARN de longitud completa. Se prefiere este tipo de purificación porque los ARN truncados o degradados causarán la determinación inexacta de las concentraciones de ARN. Dado que las bases de nucleótidos absorben la luz UV, las bandas de ARN y los rNTP en el gel se pueden visualizar bajo UV como sombras contra una placa TLC fluorescente. Por lo tanto, las bandas correspondientes al tamaño correcto del producto se pueden extraer selectivamente.
El método del vecino más cercano sobrestima los coeficientes de extinción y, por lo tanto, las concentraciones de ARN estructurados, ya que no tiene en cuenta la hipocromicidad debida al apareamiento de bases17. Por lo tanto, para determinar las concentraciones precisas de ARN, se realizaron ensayos de hidrólisis térmica de pH neutro para hidrolizar el ARN a NMP individuales18. El coeficiente de extinción exacto se calculó como una suma de los coeficientes de extinción de los NMP en la secuencia de ARN.
La cinética de la unión de DFHBI a Spinach2 y Brócoli se determinó mediante un ensayo de lector de placas. El ARN se renaturalizó primero para garantizar que estuviera en la conformación correcta para la unión del tinte. Las condiciones de reacción para el ensayo cinético del lector de placas consistieron en 40 mM HEPES, pH 7.5 (KOH), 125 mM KCl, 3 mM MgCl2, 100 nM ARN renaturalizado y 10 μM DFHBI, y la reacción se midió a 37 °C. Esta temperatura y concentración de MgCl 2 fueron elegidas para imitar las condiciones fisiológicas 19, aunque las condiciones de 28 °C y10 mM MgCl2 también se pueden utilizar para mejorar el plegamiento del aptámero.
Tanto los aptámeros fluorogénicos Spinach2 como el Brócoli muestran una cinética de asociación de dos fases para unirse al colorante DFHBI (Figura 2). Los datos cinéticos se ajustaron mejor a la asociación de dos fases que a la asociación de una fase para ambos aptámeros (Figura complementaria 1). Las constantes de velocidad y los valores t1/2 para las asociaciones rápida y lenta fueron determinadas por la curva de mejor ajuste (Tabla 3). También se determinó PercentFast, que describe qué porcentaje de la magnitud de activación de fluorescencia se explica por la población de ARN de unión DFHBI más rápida.
La espinaca2 en el estado competente para la unión muestra una activación más rápida que el brócoli (t 1/2 = 1.2 s vs.2.0 s). La cinética de la segunda fase para ambos aptámeros es similar (t1/2 = 180 s) y probablemente corresponde a un paso común de limitación de velocidad para una subpoblación de la muestra (PercentFast = 68% y 60% para espinacas2 y brócoli, respectivamente). En general, estos resultados muestran que los aptámeros de espinaca2 y brócoli bien plegados exhiben una cinética de encendido muy rápida, con el encendido inicial medio máximo dentro de 1-2 s de la adición de colorante.
Cinética celular
Las secuencias de las construcciones de ADN, que se clonan en el plásmido pET31b, se muestran en la Tabla 2, y las recetas de reactivos se muestran en el Archivo Suplementario 1. Las construcciones de ADN de los aptámeros de ARN fluorogénico están típicamente contenidas dentro de un andamio de ARNt para experimentos celulares. La cepa BL21 Star (DE3) E. coli es una cepa de expresión con una mutación en la RNasa E que aumenta la estabilidad del ARN.
Las mediciones del punto de tiempo de fluorescencia se registraron cada 5 minutos durante los primeros 45 minutos, seguidas de lecturas a 1 h, 1,5 h y 2 h, dando un total de 12 puntos de tiempo más la lectura de solo celda. Tener el intervalo de tiempo más corto de 5 minutos permitió medir múltiples réplicas biológicas en cada punto de tiempo, con un espaciamiento regular entre las mediciones replicadas de 30 s a 1 min. El volumen total de células diluidas en 1x solución de PBS utilizada para el experimento de curso de tiempo fue de 1,5 ml.
Las células fueron cerradas antes de determinar la intensidad media de fluorescencia (MFI) de la población de células individuales. Gating selecciona un área en el diagrama de dispersión para determinar la población celular que se analizará. Este proceso evita que se incluyan en el análisis lecturas de residuos o multiplicadores. Para el análisis de citometría de flujo mostrado, se registraron 30,000 eventos, lo que resultó en 10,000-20,000 eventos analizados después del gating.
La cinética de fluorescencia celular es una función tanto de la difusión del colorante en E. coli como de la cinética de unión del colorante al aptámero de ARN dentro del entorno celular (Figura 1B). Para las células que expresan espinaca2-ARNt, se observó que la intensidad media de fluorescencia (MFI) aumenta inmediatamente en el punto de tiempo "0", debido al corto retraso (en segundos) entre la adición del colorante y el análisis de la muestra (Figura 3A). Además, la fluorescencia celular ya ha alcanzado su valor máximo de MFI equilibrado (40.441 ± 990) en el primer punto de tiempo de 5 min. En contraste, las células de control muestran baja fluorescencia de fondo (416) y ningún cambio en el valor de MFI con la adición de DMSO (Figura 3B). Una comparación entre células con colorante y células sin colorante revela que la activación de la fluorescencia es 98 veces mayor ± 2 en las células. En general, estos resultados muestran que el ARNt de espinaca2 expresado en las células de E. coli exhibe una cinética de encendido rápido, con un encendido máximo dentro de menos de 5 minutos de la adición de colorante.
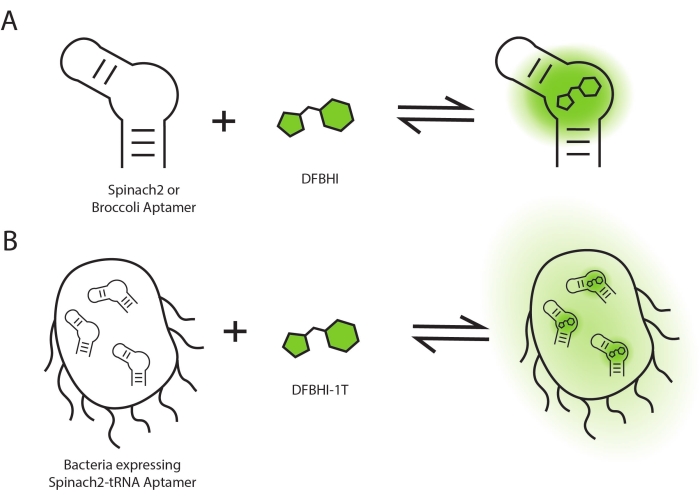
Figura 1: Esquema de activación de fluorescencia. La activación de la fluorescencia ocurre cuando los aptámeros de ARN se unen a las moléculas de colorante (A) in vitro y (B) en las células. Haga clic aquí para ver una versión más grande de esta figura.

Figura 2: Cinética in vitro de los aptámeros fluorogénicos. Cinética representativa in vitro de los aptámeros fluorogénicos (A,B) Spinach2 o (C,D) Brócoli modelados por asociación bifásica, siendo t = 0 s el punto de tiempo de adición de DFHBI (concentración final de DFHBI: 10 μM). Los experimentos se realizaron por triplicado. Todas las barras de error representan desviaciones estándar de la media. A partir del ajuste, se obtuvieron valores de t1/2 para los componentes de reacción de asociación rápida y lenta. Haga clic aquí para ver una versión más grande de esta figura.
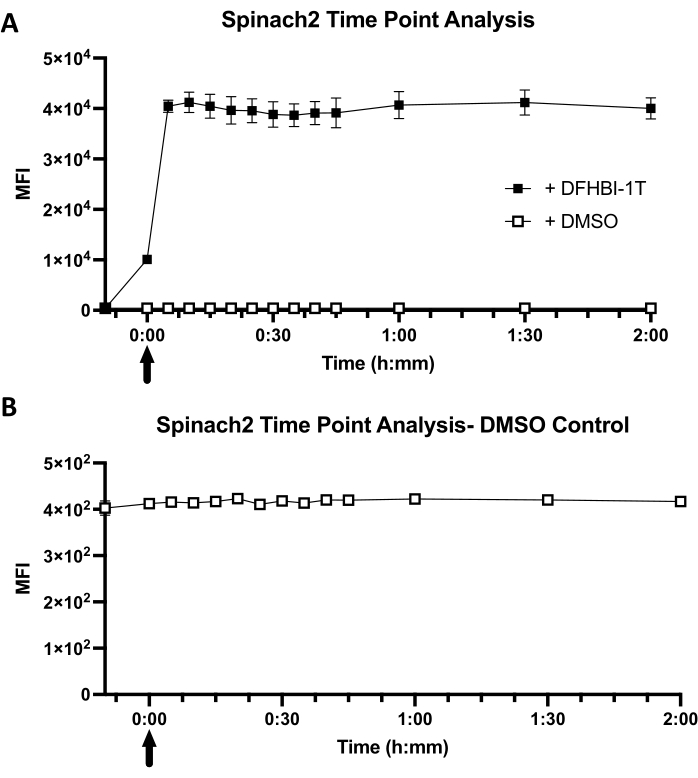
Figura 3: Cinética celular representativa de Spinach2 en un andamio de ARNt. (A) Análisis de puntos de tiempo de la captación de colorante ARNt-Espinaca2 en el transcurso de 2 h. Se tomó una línea de base solo de células antes de agregar DFHIB-1T o DMSO. Los puntos de tiempo se tomaron cada 5 minutos durante los primeros 45 minutos, seguidos de una lectura de puntos de tiempo a 1 h, 1.5 h y 2 h. La flecha representa cuándo se agregó el DFHBI-1T o DMSO en una solución PBS 1x con celdas BL21 Star. La concentración final de DFHBI-1T para el análisis es de 50 μM. Para el control DMSO, la adición de DMSO fue en un volumen igual (1,4 μL) utilizado para la adición de colorante DFHBI-1T. (B) Un primer plano del análisis del punto de tiempo de control DMSO con células BL21 Star E. coli. La intensidad media de fluorescencia (MFI) indica la lectura fluorescente general de las células estrella BL21 con colorante o DMSO. Los datos representan la media ± la desviación estándar de tres réplicas biológicas. Haga clic aquí para ver una versión más grande de esta figura.
| Par Aptamer-Dye | Longitud (nt) | Abdominales máximos (nm) | Max em (nm) | Coeficiente de extinción (M-1· cm-1) | Rendimiento cuántico | Brillo | Kd (nm) | Tm (°C) | Referencia |
| Espinaca2-DFHBI | 95 | 445 | 501 | 26100 | 0.7 | 63 | 1450 | 37 | 4 |
| Espinaca2-DFHBI-1T | 95 | 482 | 505 | 31000 | 0.94 | 100 | 560 | 37 | 13, 14 |
| Brócoli-DFHBI-1T | 49 | 472 | 507 | 29600 | 0.94 | 96 | 360 | 48 | 13 |
Tabla 1: Propiedades fotofísicas y bioquímicas publicadas previamente de Spinach2-DFHBI4, Spinach2-DFHBI-1T 13,14 y Broccoli-DFHBI-1T 13.
| Espinaca2 + T7 promotor | 5'-CGATCCCGGGAAATTAATACGACTCACTATAGGATGTAACTGAATGAAATGGTGAA GGACGGGTCCAGTAGGCTGCTTCGGCAGCCTACTTGTTGAGTAGAGTGTGAGCTCC GTAACTAGTTACATC-3' | ||
| Brócoli + promotor T7 | 5'-CGATCCCGGGAAATTAATACGACTCACTATAGgagacggtcgg GTCCAGATATTCGTATCTGTCGAGTAGAGTGTGGGCTC-3' | ||
| Construcción tRNA-Spinach2 (en plásmido pET31b) | 5'-CGATCCCGGGAAATTAATACGACTCACTATAGGGGCCCGGATAGCTCAGTCGGT AGAGCAGCGGCCGGATGTAACTGAATGAAATGGTGAAGGACGGGTCCAGTAGGCT GCTTCGGCAGCCTACTTGTTGAGTAGAGTGTGAGCTCCGTAACTAGTTACATCCGG CCGCGGGTCCAGGGTTCAAGTCCCTGTTCGGGCGCCA TAGCATAACCCCTTGGGGCCTCTAAACGGGTCTTGAGGGGTTTTTTG-3' | ||
| Espinaca2 Imprimación Directa | 5'-CGATCCCGGGAAATTAATACGACTCACTATAG-3' | ||
| Spinach2 Reverse Primer | 5'-GATGTAACTAGTTACGGAGC-3' | ||
| Imprimación directa de brócoli | 5'-CGATCCCGGGAAATTAATACGACTCACTATAGgagacggtcgggtccagatattcgtatctg-3' | ||
| Imprimación inversa de brócoli | 5'-gagcccacactctactcgacagatacgaatctggacccgaccgtctc-3' | ||
Tabla 2: Tabla de secuencias de ADN que contiene secuencias de ADN y cebadores utilizados para estudios cinéticos in vitro y celulares. Negrita= promotor T7; Subrayado = andamio de ARNt; Caps = Espinacas2; minúsculas = brócoli; Cursiva en negrita = terminador T7.
| Aptámero | Rápido t1/2 (s) | Lento t1/2 (s) | KRápido (s-1) | KLento (s-1) | Porcentaje rápido |
| Espinacas2 | 1.2 ± 0.2 | 180 ± 10 | 0,56 ± 0,07 | 0,0039 ± 0,0002 | 68 ± 5 |
| Brécol | 2.0 ± 0.2 | 180 ± 30 | 0,35 ± 0,05 | 0,0039 ± 0,0006 | 60 ± 3 |
Tabla 3: Valores cinéticos in vitro de los aptámeros de espinaca2 y brócoli derivados de los datos ajustados. Los datos se informan como la media ± desviación estándar de tres réplicas.
Figura complementaria 1: Cinética in vitro representativa de los aptámeros fluorogénicos. Cinética representativa in vitro de los aptámeros fluorogénicos (A,C) Spinach2 o (B,D) Brócoli modelados por asociación monofásica a (A,B) 600 s o (C,D) 20 s tiempos de medición, con flechas que indican el punto de tiempo de adición de DFHBI (concentración final de DFHBI: 10 μM). Los experimentos se realizaron por triplicado. En general, estos datos son menos adecuados por un modelo de asociación de una fase que un modelo de asociación de dos fases cuando la señal de fluorescencia se monitorea durante más tiempo. Haga clic aquí para descargar este archivo.
Archivo complementario 1: Recetas para el experimento cinético in vitro. Haga clic aquí para descargar este archivo.
Discusión
Para el experimento de cinética in vitro , se puede modificar el mismo protocolo general para medir la cinética in vitro de un biosensor fluorescente basado en ARN que contiene un dominio de unión a ligando y de unión a fluoróforos8. En este caso, el ARN debe incubarse con el fluoróforo antes de las mediciones al inyectar el ligando para obtener la cinética de respuesta del ligando. Si se observa una alta variabilidad entre las réplicas, se puede solucionar el problema verificando que se permita que cada muestra se equilibre durante la misma cantidad de tiempo en la placa de 96 pocillos antes de la medición. Cada muestra o réplica debe prepararse individualmente en un pozo y medirse inmediatamente después del paso de equilibrio de 15 minutos, en lugar de preparar todas las muestras a la vez.
Para el experimento de cinética celular, el protocolo puede modificarse para cursos de tiempo más cortos o más largos, pero es fundamental planificar el número de réplicas biológicas y ajustar el volumen de solución celular necesario. Se recomienda espaciar cada lectura de réplica biológica entre 30 s a 1 minuto para tener el tiempo adecuado para realizar los pasos cuidadosamente. Otra modificación es probar un colorante fluorogénico diferente que no se une al aptámero de ARN como un control negativo alternativo, que no debe mostrar activación de fluorescencia sobre el fondo. Si se observan resultados inconsistentes, se puede solucionar el problema verificando que el citómetro de flujo se limpie correctamente siguiendo el protocolo del fabricante entre diferentes ejecuciones experimentales para evitar cualquier sangrado de células o tintes de la ejecución anterior a la siguiente.
Si bien el método in vitro presentado es útil para comparar la cinética entre aptámeros fluorogénicos o biosensores fluorogénicos basados en ARN, los valores cinéticos obtenidos pueden cambiar dependiendo de la temperatura, la concentración de magnesio u otros componentes tampón utilizados. Además, si bien este método proporciona condiciones bien definidas que se han utilizado previamente para caracterizar diferentes sistemas de ARN fluorogénico, el entorno intracelular no puede representarse perfectamente debido a la presencia de otras macromoléculas biológicas.
Mientras que el lector de placas de fluorescencia equipado con un inyector programable no tiene tiempo muerto para la adquisición de datos, el instrumento del citómetro de flujo tiene una precisión temporal limitada debido al tiempo muerto observable. Hay un retraso de ~ 5 s entre el momento en que se hace clic en el botón "Grabar" y el momento en que comienza la adquisición de datos. Se produce un retraso adicional de ~5 s para que el instrumento mida 30,000 eventos; este tiempo de adquisición de la muestra variará ligeramente dependiendo de cuán diluidas estén las células en 1x PBS.
Otra limitación potencial para los experimentos celulares es la viabilidad celular en 1x PBS. Para análisis de puntos de tiempo prolongados, la viabilidad celular se puede verificar utilizando yoduro de propidio para teñir las células muertas20. La agregación de colorantes también puede limitar la precisión de las mediciones de fluorescencia realizadas por el citómetro de flujo. Los colorantes con solubilidad muy limitada en soluciones acuosas pueden agregarse y aparecer como partículas lo suficientemente grandes como para ser contadas como células en el citómetro de flujo. Por lo tanto, es importante ejecutar controles experimentales solo con colorante para verificar si hay agregados en la región cerrada.
Anteriormente, se demostró que el brócoli tiene un brillo comparable a la espinaca2 a 1 mM Mg2 + in vitro, pero el brócoli-ARNt exhibe ~ dos veces mayor intensidad de fluorescencia en E. coli viva en comparación con la espinaca2-ARNt13. Hasta donde sabemos, la cinética de unión al colorante para los aptámeros fluorogénicos de espinaca2 y brócoli no se ha comparado antes y se ha modelado mediante una asociación de dos fases. Las constantes iniciales de velocidad rápida para ambos aptámeros de ARN apoyan que el bolsillo de unión del colorante esté plegado previamente y no se necesiten cambios estructurales para que el tinte se una, lo que es consistente con la cristalografía de rayos X y los experimentos de fusión UV21,22. La segunda fase con una constante de velocidad más lenta no ha sido reportada previamente porque otros experimentos como el flujo detenido y las mediciones de vida útil de fluorescencia analizaron el aptámero de espinacas para una duración más corta (20 s y 300 s)23,24. La segunda fase, mucho más lenta, da como resultado un aumento biexponencial observado en la fluorescencia cuando se analizan los datos durante 600 s. Este paso lento puede atribuirse a un paso de repliegue limitante de la velocidad de un estado de ARN incompetente para la unión a un estado de ARN competente para la unión o un paso de fotoconversión limitante de la velocidad de las formas trans a cis del colorante unido. Este último mecanismo fue modelado previamente para dar un perfil de fluorescencia biexponencial24 y está respaldado por un análisis reciente de la diferencia entre los espectros de absorción y excitación en el aptámero relacionado, Baby Spinach25.
La importancia general de los hallazgos in vitro es que muestran que la asociación del colorante al aptámero de ARN fluorogénico no limita la localización del ARN en tiempo real y los estudios de expresión génica. Para los biosensores fluorescentes basados en ARN que emplean Spinach2, la cinética de encendido medida es similar a la cinética de segunda fase medida aquí10 porque los biosensores requieren un paso de repliegue y, por lo tanto, deben ser lo suficientemente rápidos como para permitir estudios de señalización casi en tiempo real.
Se esperaba que la cinética celular fuera diferente de la cinética in vitro observada para la espinaca2. Una diferencia clave es que hay un paso adicional de difusión de tinte en las células de E. coli , que implica cruzar las membranas externas e internas. Además, el entorno celular plantea diferentes condiciones para la asociación colorante-aptámero en términos de apiñamiento molecular, composición de iones y concentraciones, así como concentraciones de ARN y colorante.
La importancia general de los resultados es que la fluorescencia celular alcanza la señal máxima en menos de 5 minutos y permanece estable durante al menos 2 h, lo que permite la localización de ARN en tiempo real y estudios de expresión génica en este rango de tiempo. Para un biosensor basado en ARN que emplea Spinach2, previamente demostramos que se podía observar una respuesta de fluorescencia significativa dentro de los 4-5 minutos de la adición del ligando, pero que alcanzar la señal máxima lleva más tiempo (15-30 min)8. Tomados en conjunto, estos hallazgos indican que la difusión de colorantes en las células no es el paso práctico de limitación de velocidad para experimentos in vivo con biosensores basados en ARN. Finalmente, este protocolo experimental se puede aplicar para analizar otros sistemas de ARN fluorogénico en células.
Los protocolos experimentales presentados aquí se pueden aplicar para analizar otros sistemas de ARN fluorogénico. Más allá de los dos aptámeros analizados en este estudio, Spinach2 y Brócoli, se han desarrollado otros sistemas de ARN fluorogénico que proporcionan diferentes perfiles de emisión, fotoestabilidad mejorada, afinidades de unión más estrechas y la capacidad de cambiar fluoróforos (recientemente revisado1). Además de sus propiedades de fluorescencia, la evaluación comparativa de la cinética de encendido para estos sistemas in vitro y en células es importante para evaluar su idoneidad para diferentes aplicaciones biológicas celulares. Los resultados también pueden apoyar el plegado previo estructural o la reorganización del aptámero. Como se discutió, con algunas modificaciones, estos protocolos también se han aplicado para analizar biosensores basados en ARN8.
Divulgaciones
Los autores declaran no tener conflictos de intereses.
Agradecimientos
Este trabajo fue apoyado por las siguientes subvenciones a MCH: NSF-BSF 1815508 y NIH R01 GM124589. MRM fue parcialmente apoyado por la subvención de capacitación NIH T32 GM122740.
Materiales
| Name | Company | Catalog Number | Comments |
| Agarose | Thermo Fischer Scientific | BP160500 | |
| Agarose gel electrophoresis equipment | Thermo Fischer Scientific | B1A-BP | |
| Alpha D-(+)-lactose monohydrate | Thermo Fischer Scientific | 18-600-440 | |
| Amber 1.5 mL microcentrifuge tubes | Thermo Fischer Scientific | 22431021 | |
| Ammonium persulfate (APS) | Sigma-Aldrich | A3678 | |
| Ammonium sulfate ((NH4)2SO4) | Sigma-Aldrich | A4418 | |
| Attune NxT Flow cytometer | Thermo Fischer Scientific | A24861 | |
| Attune 1x Focusing Fluid | Thermo Fischer Scientific | A24904 | |
| Attune Shutdown Solution | Thermo Fischer Scientific | A24975 | |
| Attune Performance Tracking Beads | Thermo Fischer Scientific | 4449754 | |
| Attune Wash Solution | Thermo Fischer Scientific | J24974 | |
| Boric acid | Sigma-Aldrich | B6768 | |
| Bromophenol blue | Sigma-Aldrich | B0126 | |
| Carbenicillin disodium salt | Sigma-Aldrich | C3416 | |
| Chlorine Bleach | Amazon | B07J6FJR8D | |
| Corning Costar 96-well plate | Daigger Scientific | EF86610A | |
| Culture Tubes, 12 mm x 75 mm, 5 mL with attached dual position cap | Globe Scientific | 05-402-31 | |
| DFHBI | Sigma-Aldrich | SML1627 | |
| DFHBI-1T | Sigma-Aldrich | SML2697 | |
| D-Glucose (anhydrous) | Acros Organics | AC410955000 | |
| Dimethyl sulfoxide (DMSO) | Sigma-Aldrich | D8418 | |
| Dithiothreitol (DTT) | Sigma-Aldrich | DTT-RO | |
| DNA loading dye | New England Biolabs | B7025S | |
| DNA LoBind Tubes (2.0 mL) | Eppendorf | 22431048 | |
| dNTPs: dATP, dCTP, dGTP, dTTP | New England Biolabs | N0446S | |
| EDTA, pH 8.0 | Gibco, Life Technologies | AM9260G | |
| Ethanol (EtOH) | Sigma-Aldrich | E7023 | |
| Filter-tip micropipettor tips | Thermo Fischer Scientific | AM12635, AM12648, AM12655, AM12665 | |
| FlowJo Software | BD Biosciences | N/A | FlowJo v10 Software |
| Fluorescent plate reader with heating control | VWR | 10014-924 | |
| Gel electrophoresis power supply | Thermo Fischer Scientific | EC3000XL2 | |
| Glycerol | Sigma-Aldrich | G5516 | |
| Glycogen AM95010 | Thermo Fischer Scientific | AM95010 | |
| GraphPad Prism | Dotmatics | N/A | Analysis software from Academic Group License |
| Heat block | Thomas Scientific | 1159Z11 | |
| HEPES | Sigma-Aldrich | H-4034 | |
| Inorganic pyrophosphatase | Sigma-Aldrich | I1643-500UN | |
| Low Molecular Weight DNA Ladder | New England Biolabs | N3233L | Supplied with free vial of Gel Loading Dye, Purple (6x), no SDS (NEB #B7025). |
| Magnesium chloride hexahydrate (MgCl2) | Sigma-Aldrich | M2670 | |
| Magnesium sulfate (MgSO4) | Fisher Scientific | MFCD00011110 | |
| Microcentrifuge tubes (1.5 mL) | Eppendorf | 22363204 | |
| Microcentrifuge with temperature control | Marshall Scientific | EP-5415R | |
| Micropipettors | Gilson | FA10001M, FA10003M, FA10005M, FA10006M | |
| Micropipettor tips | Sigma-Aldrich | Z369004, AXYT200CR, AXYT1000CR | |
| Millipore water filter with BioPak unit | Sigma-Aldrich | CDUFBI001, ZRQSVR3WW | |
| Narrow micropipettor pipette tips | DOT Scientific | RN005R-LRS | |
| PBS, 10x | Thermo Fischer Scientific | BP39920 | |
| PCR clean-up kit | Qiagen | 28181 | |
| PCR primers and templates | Integrated DNA technologies | ||
| PCR thermocycler for thin-walled PCR tubes | Bio-Rad | 1851148 | |
| PCR thermocycler for 0.5 mL tubes | Techne | 5PRIME/C | |
| pET31b-T7-Spinach2 Plasmid | Addgene | Plasmid #79783 | |
| Phusion High-Fidelity DNA polymerase | New England Biolabs | M0530L | Purchase of Phusion High-Fideldity Enzyme is supplied with 5x Phusion HF Buffer, 5x Phusion GC Buffer, and MgCl2 and DMSO solutions. |
| Polyacrylamide gel electrophoresis gel comb, C.B.S. Scientific | C.B.S. Scientific | VGC-1508 | |
| Polyacrylamide gel electrophoresis equipment | C.B.S. Scientific | ASG-250 | |
| Potassium chloride (KCl) | Sigma-Aldrich | P9333 | |
| Potassium phosphate monobasic | Sigma-Aldrich | P5655 | |
| Razor blades | Genesee Scientific | 38-101 | |
| rNTPs: ATP, CTP, GTP, UTP | New England Biolabs | N0450L | |
| SDS | Sigma-Aldrich | L3771 | |
| Short wave UV light source | Thermo Fischer Scientific | 11758221 | |
| Sodium carbonate (Na2CO3) | Sigma-Aldrich | S7795 | |
| Sodium chloride (NaCl) | Sigma-Aldrich | S7653 | |
| Sodium hydroxide (NaOH) | Sigma-Aldrich | S8045 | |
| Sodium phosphate dibasic, anhydrous | Thermo Fischer Scientific | S375-500 | |
| SoftMax Pro | Molecular Devices | N/A | SoftMax Pro 6.5.1 (platereader software) obtained through Academic Group License |
| Sterile filter units | Thermo Fischer Scientific | 09-741-88 | |
| Sucrose | Sigma-Aldrich | S0389 | |
| SYBR Safe DNA gel stain | Thermo Fischer Scientific | S33102 | |
| TAE buffer for agarose gel electrophoresis | Thermo Fischer Scientific | AM9869 | |
| Tetramethylethylenediamine (TEMED) | Sigma-Aldrich | T9281 | |
| Tris base | Sigma-Aldrich | TRIS-RO | |
| Tryptone (granulated) | Thermo Fischer Scientific | M0251S | |
| T7 RNA polymerase | New England Biolabs | M0251S | |
| Urea-PAGE Gel system | National Diagnostics | EC-833 | |
| UV fluorescent TLC plate | Sigma-Aldrich | 1.05789.0001 | |
| UV/Vis spectrophotometer | Thermo Fischer Scientific | ND-8000-GL | |
| Vortex mixer | Thermo Fischer Scientific | 2215415 | |
| Xylene cyanol | Sigma-Aldrich | X4126 | |
| Yeast Extract (Granulated) | Thermo Fischer Scientific | BP9727-2 |
Referencias
- Su, Y., Hammond, M. C. RNA-based fluorescent biosensors for live cell imaging of small molecules and RNAs. Current Opinion in Biotechnology. 63, 157-166 (2020).
- Zhang, J., et al. Tandem spinach array for mRNA Imaging in living bacterial cells. Scientific Reports. 5, 17295(2015).
- Wang, Z., et al. In spatial complementation of aptamer-mediated recognition enables live-cell imaging of native RNA transcripts in real time. Angewandte Chemie. 57 (4), 972-976 (2018).
- Strack, R. L., Disney, M. D., Jaffrey, S. R. A superfolding Spinach2 reveals the dynamic nature of trinucleotide repeat-containing RNA. Nature Methods. 10 (12), 1219-1224 (2013).
- Thavarajah, W., et al. Point-of-use detection of environmental fluoride via a cell-free riboswitch-based biosensor. ACS Synthetic Biology. 9 (1), 10-18 (2020).
- You, M., Litke, J. L., Jaffrey, S. R. Imaging metabolite dynamics in living cells using a Spinach-based riboswitch. Proceedings of the National Academy of Sciences of the United States of America. 112 (21), 2756-2765 (2015).
- Kellenberger, C. A., Wilson, S. C., Sales-Lee, J., Hammond, M. C. RNA-based fluorescent biosensors for live cell imaging of second messengers cyclic di-GMP and cyclic AMP-GMP. Journal of the American Chemical Society. 135 (13), 4906-4909 (2013).
- Manna, S., Truong, J., Hammond, M. C. Guanidine biosensors enable comparison of cellular turn-on kinetics of riboswitch-based biosensor and reporter. ACS Synthetic Biology. 10 (3), 566-578 (2021).
- Bose, D., Su, Y., Marcus, A., Raulet, D. H., Hammond, M. C. An RNA-based fluorescent biosensor for high-throughput analysis of the cGAS-cGAMP-STING pathway. Cell Chemical Biology. 23 (12), 1539-1549 (2016).
- Wang, X. C., Wilson, S. C., Hammond, M. C. Next-generation RNA-based fluorescent biosensors enable anaerobic detection of cyclic di-GMP. Nucleic Acids Research. 44 (17), 139(2016).
- Paige, J. S., Thinh, N. -D., Wenjiao, S., Jaffrey, S. R. Fluorescence imaging of cellular metabolites with RNA. Science. 335 (6073), 1194(2012).
- Paige, J. S., Wu, K. Y., Jaffrey, S. R. RNA mimics of green fluorescent protein. Science. 333 (6042), 642-646 (2011).
- Filonov, G. S., Moon, J. D., Svensen, N., Jaffrey, S. R. Broccoli: Rapid selection of an RNA mimic of green fluorescent protein by fluorescence-based selection and directed evolution. Journal of the American Chemical Society. 136 (46), 16299-16308 (2014).
- Song, W., Strack, R. L., Svensen, N., Jaffrey, S. R. Plug-and-play fluorophores extend the spectral properties of spinach. Journal of the American Chemical Society. 136 (4), 1198-1201 (2014).
- Sambrook, J., Fritsch, E., Maniatis, T. Molecular Cloning: A Laboratory Manual. , Cold Spring Harbor Laboratory Press. Cold Spring Harbor, NY. (1989).
- Basch, H., Gadebusch, H. H. In vitro antimicrobial activity of dimethylsulfoxide. Applied Microbiology. 16 (12), 1953-1954 (1968).
- Kallansrud, G., Ward, B. A comparison of measured and calculated single- and double-stranded oligodeoxynucleotide extinction coefficients. Analytical Biochemistry. 236 (1), 134-138 (1996).
- Wilson, S. C., Cohen, D. T., Wang, X. C., Hammond, M. C. A neutral pH thermal hydrolysis method for quantification of structured RNAs. RNA. 20 (7), 1153-1160 (2014).
- Szatmári, D., et al. Intracellular ion concentrations and cation-dependent remodelling of bacterial MreB assemblies. Scientific Reports. 10, 12002(2020).
- Boulos, L., Prévost, M., Barbeau, B., Coallier, J., Desjardins, R. LIVE/DEAD® BacLightTM: Application of a new rapid staining method for direct enumeration of viable and total bacteria in drinking water. Journal of Microbiological Methods. 37 (1), 77-86 (1999).
- Huang, H., et al. A G-quadruplex-containing RNA activates fluorescence in a GFP-like fluorophore. Nature Chemical Biology. 10 (8), 686-691 (2014).
- Jeng, S. C. Y., Chan, H. H. Y., Booy, E. P., McKenna, S. A., Unrau, P. J. Fluorophore ligand binding and complex stabilization of the RNA Mango and RNA Spinach aptamers. RNA. 22 (12), 1884-1892 (2016).
- Han, K. Y., Leslie, B. J., Fei, J., Zhang, J., Ha, T. Understanding the photophysics of the Spinach-DFHBI RNA aptamer-fluorogen complex to improve live-cell RNA imaging. Journal of the American Chemical Society. 135 (50), 19033-19038 (2013).
- Wang, P., et al. Photochemical properties of Spinach and its use in selective imaging. Chemical Science. 4 (7), 2865-2873 (2013).
- Dao, N. T., et al. Photophysics of DFHBI bound to RNA aptamer Baby Spinach. Scientific Reports. 11, 7356(2021).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados