Method Article
Ağırlıklı Korelasyon Ağlarına Dayalı Farklı Habitatlarda Kök Mikrobiyotasının Ayrışması
* Bu yazarlar eşit katkıda bulunmuştur
Bu Makalede
Özet
Toprak, su ve rizosfer gibi çeşitli ekolojik mikrobiyal toplulukların birlikteliğini değerlendirmek için ağ analizi uygulandı. Burada sunulan, farklı ekolojik ortamlar nedeniyle mikrobiyal topluluklarda oluşabilecek farklı birlikte oluşum ağlarını analiz etmek için WGCNA algoritmasının nasıl kullanılacağına ilişkin bir protokoldür.
Özet
Kök mikrobiyomu bitki büyümesinde ve çevresel adaptasyonda önemli bir rol oynar. Ağ analizi, farklı ortamlarda farklı mikrobiyal türlerin etkileşim ilişkisini veya birlikte oluşum modelini etkili bir şekilde keşfedebilen toplulukları incelemek için önemli bir araçtır. Bu makalenin amacı, farklı ekolojik ortamlar nedeniyle mikrobiyal topluluklarda oluşabilecek farklı birlikte oluşum ağlarını analiz etmek için ağırlıklı korelasyon ağı algoritmasının nasıl kullanılacağı hakkında ayrıntılı bilgi sağlamaktır. Deneyin tüm analizi WGCNA paketinde gerçekleştirilir. WGCNA, ağırlıklı korelasyon ağı analizi için bir R paketidir. Bu yöntemleri göstermek için kullanılan deneysel veriler, pirinç (Oryza sativa) kök sisteminin üç nişi için NCBI (Ulusal Biyoteknoloji Bilgi Merkezi) veritabanından mikrobiyal topluluk verileriydi. Üç nişin her birinde mikrobiyal topluluğun ortak bolluk ağlarını oluşturmak için ağırlıklı korelasyon ağı algoritmasını kullandık. Daha sonra endosfer, rhizoplane ve rizosfer toprağı arasında diferansiyel co-bolluk ağları tespit edildi. Buna ek olarak, ağdaki çekirdek cins, ağ işlevlerinde önemli bir düzenlenmiş rol oynayan "WGCNA" paketi ile elde edilmiştir. Bu yöntemler, araştırmacıların mikrobiyal ağın çevresel bozulmaya tepkisini analiz etmelerini ve farklı mikrobiyal ekolojik yanıt teorilerini doğrulamalarını sağlar. Bu yöntemlerin sonuçları, pirincin endosfer, rizosplan ve rizosfer toprağında tanımlanan önemli diferansiyel mikrobiyal ağların.
Giriş
Mikrobiyom araştırmasının ekosistem süreçlerini anlamak ve manipüle etmek için önemli etkileri vardır1,2. Mikrobiyal popülasyonlar, özellikleri mikroorganizmaların çevresel değişikliklere tepkisini etkileyebilecek ekolojik ağlarla birbirine bağlanır3,4. Ayrıca, bu ağların özellikleri mikrobiyal toplulukların stabilitesini etkiler ve toprak fonksiyonu ile yakından ilişkilidir5. Ağırlıklı gen korelasyon ağı analizi artık genler ve mikrobiyal topluluklar arasındaki ilişki üzerine araştırma için yaygın olarak uygulanmaktadır6. Önceki çalışmalar esas olarak farklı genlerin veya popülasyonların ağları ile dış dünya arasındaki ilişkilere odaklanmıştır7. Bununla birlikte, mikrobiyal popülasyonların farklı çevresel koşullar altında oluşturduğu korelasyon ağlarındaki farklılıklar çok az araştırılmıştır. Bu makalede sunulan araştırmanın amacı, farklı çevresel koşullar altında toplanan mikrobiyom örneklerinden oluşan bir ortak oluşum ağı oluşturmak için WGCNA algoritmasının hızlı bir şekilde uygulanması hakkında içgörüler ve ayrıntılar sağlamaktır. Analiz sonuçlarına dayanarak, popülasyonun bileşimini ve farklılıklarını değerlendirdik ve farklı mikrobiyal popülasyonlar arasındaki ilişkiyi daha da tartıştık. Aşağıdaki temel ağ algoritması8 ağırlıklı korelasyon ağı algoritması uygulandı. İlk olarak, Operasyonel Taksonomik Birimler (OTU) ifade profilleri arasındaki Pearson korelasyon katsayısı hesaplanarak bir benzerlik matrisinin oluşturulması gerekiyordu. Daha sonra, bitişiklik işlevlerinin parametreleri (güç veya sigmoid bitişiklik işlevleri) ölçeksiz bir topoloji kriteri ile benimsendi, benzerlik matrisi bir bitişiklik matrisine dönüştürüldü ve her birlikte oluşum ağı bir bitişiklik matrisine karşılık geldi. Tutarlı ifade profillerine sahip OPU'ları modüller halinde gruplandırmak için TOM tabanlı benzerlik ile birlikte ortalama bağlantı hiyerarşik kümeleme kullandık. Ayrıca, muhafazakar istatistikler ve ilgili parametre analiz modülleri arasındaki ilişkiyi hesapladık ve sonunda modüldeki hub OTU'yu tanımladık. Bu yöntemler özellikle farklı çevresel koşullar altında çeşitli mikrobiyal popülasyonlar arasındaki ağ yapılarındaki farklılıkların analizi için uygundur. Bu yazıda, ortak ifade ağı geliştirme yöntemini, modüller arasındaki benzerliklerin analizini ayrıntılı olarak anlattık ve farklı modül ağlarındaki çekirdek türleri elde etmek için uygulanan prosedürdeki adımlara kısa bir genel bakış sağladık.
Protokol
1. Veri İndirme
- NCBI veritabanını oluşturan PRJNA386367 katılım verilerini indirin. PRJNA386367 katılım verilerinden, 2014 yılında Arbuckle, Kaliforniya'daki batık bir pirinç tarlasında 14 hafta boyunca yetiştirilen pirinç bitkilerinden rizosfer, rhizoplane ve endosfer mikrobiyom verilerini seçin.
NOT: Rizosfer, rhizoplane ve endosfer mikrobiyom verileri PRJNA386367 katılımda OPU'lar tablosu tarafından sunulmuştur.
2. Optimum güç değeri belirleme
NOT: WGCNA paketi aşağıdaki işlev parametrelerinin tümlerini içerir. WGCNA, ağırlıklı korelasyon ağı analizi için bir R paketidir. Anahtar komut satırları Ek S1'e başvurur.
- R dil ortamında, Rstudio yazılımını açın ve WGCNA paketini yükleyin.
- Verileri yükleyin ve verilerin doğruluğunu kontrol etmek için goodSamplesGenes işlevini kullanın. Komut satırlarını yürütün:
"gsg = goodSamplesGenes(datExpr0, ayrıntılı = 3)
gsg$allOK "
Çalıştır 'ıtıklatın. - Aykırı değerleri kontrol edin ve gereksinimleri karşılayan örnekleri saklayın. Denetim sonucu DOĞRU olduğunda, sonraki adıma geçin. Sonucu kaydedin.
- Farklı güç değerleri altında verinin iki grubunun ölçeksiz dizini R2'yi hesaplamak için PickSoftThreshold işlevini kullanın. Komut satırını yürütün:
"sft = pickSoftThreshold(datExpr0, powerVector = güçler, ayrıntılı = 5)"
Çalıştır 'ıtıklatın. - Sonuçları görselleştirin (Şekil 1). Komut satırını yürütün:
"plot(sft$fitIndices[,1], -sign(sft$fitIndices[,3])*sft$fitIndices[,2],
xlab="Yumuşak Eşik (güç)",ylab="Ölçeksiz Topoloji Modeli Sığdır,R^2 imzalı",type="n",
main = yapıştırma("ES_Scale bağımsızlığı"));
metin(sft$fitIndices[,1], -sign(sft$fitIndices[,3])*sft$fitIndices[,2],
etiketler=güçler,cex=cex1,col="kırmızı");
abline(h=0,9,col="kırmızı")
plot(sft$fitIndices[,1], sft$fitIndices[,5],
xlab="Yumuşak Eşik (güç)",ylab="Ortalama Bağlantı", type="n",
main = yapıştır("ES_Mean bağlantı"))
metin(sft$fitIndices[,1], sft$fitIndices[,5], labels=powers, cex=cex1,col="red")"
Çalıştır 'ıtıklatın.
NOT: Ağırlıklı korelasyon ağı algoritmasının öncülü, kurulan ortak ifade ağ yapısının ölçeksiz topoloji kriterinin standartlarına uygun olması ve sağlamlığını artırmasıdır. 1'e yakın ölçeksiz dizin, ölçeksiz ağa daha yakın bir ağ yapısını gösterir. - Ölçek içermeyen R 2 endeksi0,9'dan büyük olduğunda güç değerini seçin ve bir sonraki analiz adımına geçin.
NOT: Ölçeksiz dizin 1'e yakın olduğunda, ağ yapısı ölçeksiz ağa daha yakındır. İki veya daha fazla ağı analiz ederken, birlikte ifade edilen ağlar arasındaki karşılaştırılmayı karşılamak için her ağı ölçeksiz ağın güç değerine yakın hale getirmek gerekir.
3. Ortak ifade ağının ve modül tanımlamasının inşası
NOT: Yukarıdaki hesaplanan güç değerine bağlı olarak, birlikte oluşum ağı oluşturulur. Anahtar komut satırları Ek S2'ye başvurur.
- Sembolik bir birlikte oluşum ağının inşası için imzalı parametreler eklemek üzere WGCNA paketindeki bitişiklik işlevini kullanın. Komut satırını yürütün:
"bitişiklik = bitişiklik(datExpr0, güç = softPower)"
Çalıştır 'ıtıklatın. - Üst düzey çakışan bir ağ geliştirmek ve benzerlik ağını hesaplamak için TOM benzerliği işlevini uygulayın. Komut satırını yürütün:
"TOM = TOMsimilarity(bitişiklik);
dissTOM = 1-TOM"
Çalıştır 'ıtıklatın.
NOT: Topoloji çakışma ağ türünü ayarlamak için imzalı parametre eklendi. - Hiyerarşik kümeleme için ortalama bağlantı hiyerarşik kümeleme yöntemini seçmek için hclust işlevini kullanın. Komut satırını yürütün:
"geneTree = hclust(as.dist(dissTOM), yöntem = "ortalama");"
Çalıştır 'ıtıklatın. - Dinamik dal kesme gerçekleştirmek için cutreeDynamic işlevini kullanın ve minClusterSize parametresini 30 olarak ayarlayın. Modül tanıma sonucunu alın. Komut satırını yürütün:
"dynamicMods = cutreeDynamic(dendro = geneTree, distM = dissTOM, deepSplit = 2, pamRespectsDendro = FALSE, minClusterSize = minModuleSize);"
Çalıştır 'ıtıklatın.
NOT: En küçük modül boyutu 30'dan düşük olamaz. - Her OTU modülünün modül eigenes modülEigenes işlevi tarafından hesaplayın. Komut satırını yürütün:
"MEList = moduleEigengenes(datExpr0, renkler = dynamicColors)
MEs = MEList$eigengenes"
Çalıştır 'ıtıklatın.
NOT: Eigen modülü, modüldeki genel OTU ifade düzeyini temsil etti. Belirli bir OTU değil, tekil ağ değeri ayrışmasıyla elde edilen her kümenin ilk ana bileşeniydi. - Eigen modülünün korelasyon katsayısına göre küme işlevini gerçekleştirin. Modülleri 0,25'ten düşük bir değerle birleştirmek için mergeCloseModules işlevini kullanın. Komut satırını yürütün:
"merge = mergeCloseModules(datExpr0, dynamicColors, cutHeight = MEDissThres, ayrıntılı = 3)"
Çalıştır 'ıtıklatın. - Son olarak, her ortak ifade ağının modül atama görüntüleme diyagramını elde etmek için görselleştirme için plotDendroAndColors işlevini kullanın (Şekil 2). Modül atama tablosunun her OTin'ine karşılık gelen modül ilişkilendirmesini ayıklamak için tablo işlevini kullanın. Komut satırını yürütün:
"plotDendroAndColors(geneTree, mergedColors, "Birleştirilmiş dinamik",dendroLabels = YANLIŞ,
askıda kalma = 0,03,addGuide = DOĞRU, guideHang = 0,05,
main = "ES_Gene dendrogram ve modül renkleri")"
Çalıştır 'ıtıklatın.
NOT: Birlikte ifade eden ağın modül atama diyagramında, farklı renkler farklı modülleri, gri ise herhangi bir modüle sınıflandırılamayan OTU'ları temsil eder. Gri modüldeki daha fazla sayıda OTU, ifade matrisinin erken aşama ön işleme kalitesinin düşük olduğunu gösterir.
4. Modül karşılaştırması
NOT: Bu yöntem, iki ekolojik mikrobiyal topluluğun ağ modüllerini karşılaştırmak için kullanılabilir. Bu makalede, mikrobiyal ağ modüllerinin endosfer ve rhizoplane, endosfer ve rizosfer, rizosfer ve rhizosplane arasındaki farklarını karşılaştırın.
- Koruma testi
- Önceki adımlarda kaydedilen iki veri kümesinin parametrelerini ve sonuçlarını yükleyin.
- Bir grup mikrobiyal verinin ağ modülü atama sonucunu başvuru grubu olarak, diğer grubu ise test grubu olarak ayarlayın.
- Z_summary ve medianRank muhafazakarlık istatistiksel parametrelerinin değerlerini hesaplamak için modulePreservation işlevini kullanın. Komut satırını yürütün:
"system.time({mp=modulePreservation(multiExpr,
multiColor,referenceNetworks=1,
nPermutation=100, randomSeed=1,quickCor=0,verbose=3)})"
Çalıştır 'ıtıklatın.
NOT: Bu sonuç modüller arasındaki muhafazakarlığı ölçebilir. Z_summary>10, iki modülün yüksek oranda korunduğunu gösterirken, Z_summary<2 korunmayan modülleri gösterir. medianRank, sıralama ile değerlendirilen modülün göreli korunmasını ifade eder. Daha yüksek medianRank değerleri korunmayan modülleri gösterir. (Anahtar komut satırları Ek S3'ebaşvurur.) - Sonuçları görselleştirmek için çizim işlevini kullanın (Şekil 3). parametreleri alın Z_summary ve medianRank (Tablo 1).
NOT: Hem 2'den küçük Z_summary değerini hem de en üstteki ortanca Rank değerini karşılayan ağ modülleri, iki ekolojik mikrobiyal topluluktaki en yüksek oranda korunmayan modüldür. - İki ağın en yüksek oranda korunmayan modülüne sahip modülü tanımlamak için yukarıda belirtilen iki istatistiksel parametrenin sonuçlarına dayanmaktadır.
- Modül üyeliğinin korelasyon analizi
- İki ağın modül atama sonuçlarının sırasıyla başvuru ve test grubu olarak ayarlandığını ayarlayın.
NOT: Ayarların Koruma testiyle aynı olması gerekir. - Birkaç aday modüldeki her OTU'nun kME (modül üyeliği) değerini ayıklamak için corPvalueStudent işlevini kullanın.
Komut satırını yürütün:
"Pvalue = as.data.frame(corPvalueStudent(as.matrix
(ModuleMembership), Örnekler))""
Çalıştır 'ıtıklatın.
NOT: kME modül üyeliği derecesini temsil ediyor. ME, modüldeki OTU ifadesinin genel düzeyini temsil eden eigen modül anlamına gelir. kME, her OTU ve ME arasındaki korelasyon katsayısıdır. OTU'nun ağdaki önemini OTU'nun kME değerine göre ölçün. (Anahtar komut satırları Ek S4'e başvurur.) - Ardından, iki ağdaki karşılık gelen OTU'ların kME değerinin korelasyon katsayısını hesaplamak ve korelasyon analiz diyagramını çizmek için verboseScatterplot işlevini kullanın (Şekil 4).
Komut satırını yürütün:
"verboseScatterplot(abs(TModuleMembership
[TmoduleGenes, Tcolumn]),
abs(NModuleMembership[NmoduleGenes, Ncolumn]),
xlab = yapıştır("kME in", "ES"),
ylab = yapıştır("kME in", "RP"),
main = yapıştır("lightyellow"),
cex.main = 1.7, cex.lab = 1.6, cex.axis = 1.6, col = modulecolor)"
Çalıştır 'ıtıklatın. - İki ağın OTU'sunun kME değerinin en küçük korelasyon katsayısına sahip modülü seçin. Bu modülün iki ağın en büyük farkına sahip olduğunu düşünün.
- İki ağın modül atama sonuçlarının sırasıyla başvuru ve test grubu olarak ayarlandığını ayarlayın.
5. Mikrobiyal diferansiyel ağ modülünün analizi
- Modülün OTU sıra kümesinin istatistiksel analizi ile baskın bakteri phyla'nın verilerini en büyük farkla elde edin.
NOT: Modülün en büyük farka sahip OTU sıra kümesi phyla taksonomisi ile toplanır. Baskın bakteri phyla% 10'dan fazlasını oluşturdu. - Ardından, en büyük fark modülünde OTU'nun etkileşim ilişkisi bilgilerini içeren dosyayı elde etmek için exportNetworkToCytoscape işlevini kullanın.
Komut satırını yürütün:
"cyt = exportNetworkToCytoscape(modTOM,
edgeFile = yapıştırma("NEW-ES_CytoscapeInput-edges-", modüller , ".txt", sep=""),
nodeFile = yapıştırma("NEW-ES_CytoscapeInput-nodes-", modüller, ".txt", sep=""),
ağırlıklı = DOĞRU,eşik = 0,5, nodeNames = modProbes,
altNodeNames = modGenes, nodeAttr = moduleColors[inModule])"
Çalıştır 'ıtıklatın. - Dosyayı Cytoscape'e aktarın. Eşiği 0,5 olarak ayarlayın ve diğer parametreleri gerektiği gibi ayarlayın.
- Diferansiyel mikroorganizmalardan oluşan bir ortak oluşum ağı oluşturun (Şekil 5).
- Ağdaki en önemli düzenleyici role sahip çekirdek cinsin bilgilerini elde etti.
NOT: OUT'un kME değerine göre çekirdek cins tanımlanabilir. - Son olarak, çekirdek cinsin işlevleri değerlendirildi ve tüm fark ağı üzerindeki etkisi analiz edildi.
Sonuçlar
Bu makaledeki temsili sonuçlar NCBI veritabanındaki 2014 Kaliforniya Abaker pirinç kökü mikrobiyom verilerinden indirildi (PRJNA386367)9. Veriler, su altında kalan bir pirinç tarlasında 14 hafta boyunca yetiştirilen pirinç bitkilerinden alınan rizosfer, rhizoplane ve endosfer mikrobiyom örneklerini içerir. WGCNA algoritmasını, ölçeksiz ağa yakın olan üç ağı karşılayan güç değerini seçmek için kullandık (Şekil 1) ve üç ortak ifade ağı geliştirdik (Şekil 2). Endosfer, rhizoplane ve rizosfer toprak mikrobiyal ortak ifade ağında sırasıyla 23, 22 ve 21 modül tanımlanmıştır. Bu sonuçlar, üç nişteki mikrobiyal etkileşim ağlarının sayısının temelde eşit olduğunu göstermektedir.
Endosfer, rhizoplane ve rizosfer toprağı içindeki mikrobiyal ağ modüllerindeki farklılıkları daha da karşılaştırdık. Üç niş grubun modüllerinin aşağıdaki koruma test sonuçları elde edilmiştir. Rizosfer toprağı ile rizosplan arasında son derece korunmuş olmayan üç modül mevcuttu (Şekil 3a, Tablo 1). Ayrıca, rizosfer toprağı ve endosferi (Şekil 3b, Tablo 2) ve rhizoplane ve endosfer arasında son derece korunmuş olmayan altı modül arasında dokuz son derece korunmuş modül mevcuttu ( Şekil3c, Tablo 3). Ayrıca, üç niş arasında son derece korunmayan modüller bulundu ve bu da üç niş arasında mikroorganizmaların bileşiminde büyük farklılıklar olduğunu gösterdi. Elde edilen konservatif olmayan modüllerin modül üyeliği korelasyon analizinin sonuçları Şekil 4'tegösterilmiştir. Şekilden, her iki niş arasında en az korelasyona sahip önemli ölçüde farklı bir modül, aralarındaki en önemli farkı temsil eden üç niş arasında görülebilir.
Rizosfer-rizoplan fark ağında (Şekil 5a), baskın fitülar Proteobakteri (%72.97) idi. Rizosfer-endosfer fark ağında(Şekil 5b),baskın phyla Proteobacteria (%66.36), Actinobacteria (%10.1) ve Bakterioidetler (%10.9) idi. Rhizoplane-endosfer fark ağında (Şekil 5c), baskın phyla Proteobacteria (%41.41), Bakterioidetler (%10.10), Firmicutes (%12.12) ve Verrucomicrobia idi.
Rhodobacter ve Novosphingobiumdahil olmak üzere üç çekirdek cins (Şekil 5a), Blvii28 ve Dechloromonasdahil altı çekirdek cins ( Şekil 5b ) ve Cellvibrio ve Geobacterdahil olmak üzere beş çekirdek cins (Şekil 5c), üç diferansiyel birlikte oluşum ağını önemli düzenleyici işlevler uygulamıştır. Dechloromonashariç tüm çekirdek cinsler, mevcut kök mikrobiyal toplulukların bolluğunu ve çeşitliliğini kritik derecede etkileyen pirinç köklerinin üç nişi arasında mikrobiyal popülasyonların ve türlerin göreceli bolluğunda önemli farklılıkların varlığını gösteren sadece bir ağ üzerinde etkili olmuştur.
Pirinç rhizosfer-endosfer fark ağında bulunan çekirdek cins Azospirillum,azot fiksasyonuna katıldı ve bitki büyümesini teşvik etti10. Ek olarak, rhizoplane-endosfer fark ağında önemli ölçüde zenginleştirilmiş olan Geobacter cinsi, birçok toprak ve çökeltide çözünmeyen Fe ve Mn oksitlerin azaltılmasını teşvik eden ana faktör olabilir11. Bu mikroorganizmalar kök nişlerdeki bir dizi mikrobiyal toplulukla etkileşime girer ve pirinç köklerinin büyümesi ve gelişmesi için kritik öneme sahip olabilecek mikrobiyal ağların düzenlenmesine aktif olarak katılırlar.
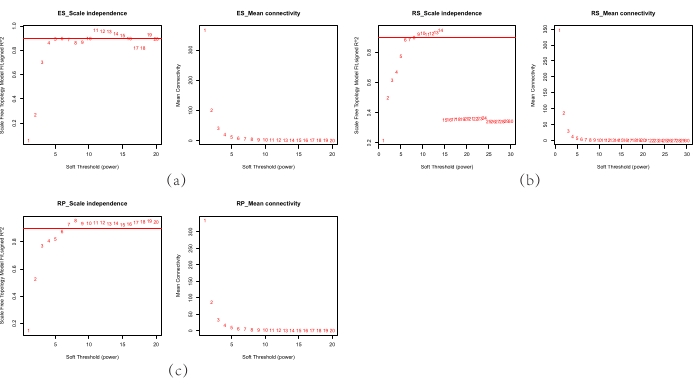
Şekil 1. Veri kümelerinde powerβ'nin değerlendirilmesi. (a) ES veri kümesindeki güç β değerlendirilmesi; (b) RS veri kümesindeki güç β değerlendirilmesi; (c) RP veri kümesindeki güç β değerlendirilmesi, ölçeksiz R2 endeksinin dağılımı (solda), ortalama bağlantının dağılımı (sağda) farklı yumuşak güç endeksleri boyunca. En iyi gücün değeri, R2 doygunluğa eğilimli olduğunda ve 0.8'den düşük olmadığında elde edildi. Karşılaştırılılabilirliklerini sağlamak için üç ortak ifade ağının aynı güç değerine ayarlı olması gerekiyordu. (RS: rizosfer toprağı, RP: rhizoplane ve ES: endosfer). Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.

Şekil 2. Ortalama bağlantı hiyerarşik kümeleme ile elde edilen OTU dendrogramı. (a)ES ağından OTU dendrogramı; (b) RS ağından OTU dendrogramı; (c) RP ağından OTU dendrogramı. Dendrogramın altındaki renk satırı, Dinamik Ağaç Kesme algoritması tarafından belirlenen modül atamasını gösterir. (RS: rizosfer toprağı, RP: rhizoplane ve ES: endosfer) Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.

Şekil 3. Koruma testinin sonuçları. (a) Analiz sonuçları, referans grubu olarak RS ortak ifade ağ modülü ayırma ve test grubu olarak RP ortak ifade ağ modülü ayırma sonuçlarına dayanır; (b) Analiz sonuçları, referans grubu olarak ES ortak ifade ağ modülü ayırmasına ve test grubu olarak RS ortak ifade ağ modülü ayırma sonuçlarına dayanır; (c) Analiz sonuçları, referans grubu olarak ES ortak ifade ağ modülü ayırmasını ve test grubu olarak RP ortak ifade ağ modülü ayırma sonuçlarını temel alır. Z_summary > 10, iki modülün yüksek oranda korunduğunu gösterirken, Z_summary < 2 korunmayan modülleri gösterir. medianRank, sıralama ile değerlendirilen modülün göreli korunmasını ifade eder. Daha yüksek medianRank değerleri korunmayan modülleri gösterir. (RS: rizosfer toprağı, RP: rhizoplane ve ES: endosfer) Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
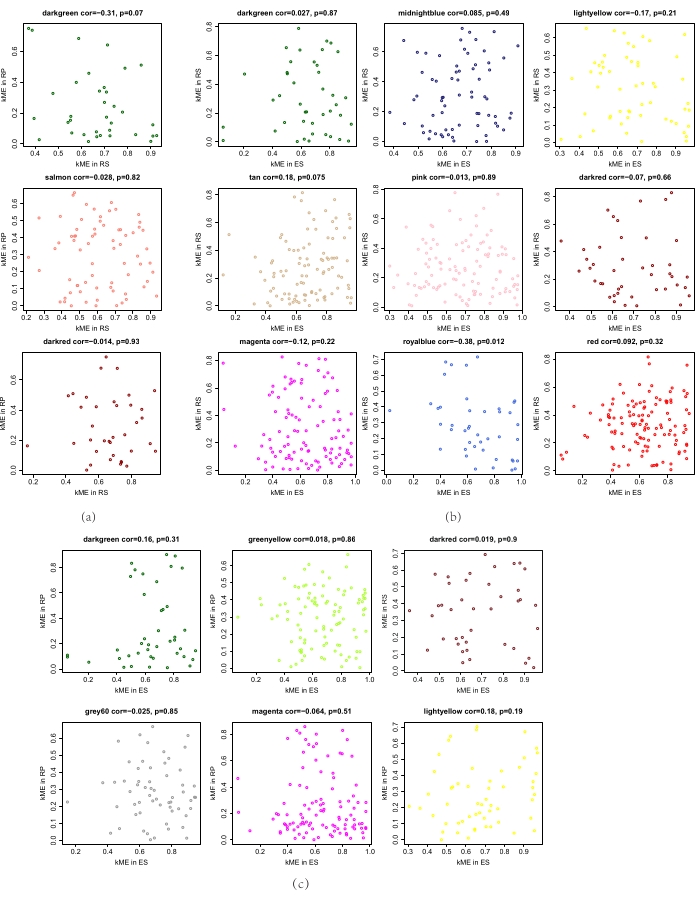
Şekil 4. Modül üyeliklerinin korelasyon analizi. (a) RS ve RP ağı arasındaki kME değerinin modül korelasyonu; (b) RS ve RP ağı arasındaki kME değerinin modül korelasyonu; (c) KME değerinin RP ve ES ağı arasındaki modül korelasyonu (RS: rhizosfer toprağı, RP: rhizoplane, ES: endosfer ve KME: modül üyeliği). Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
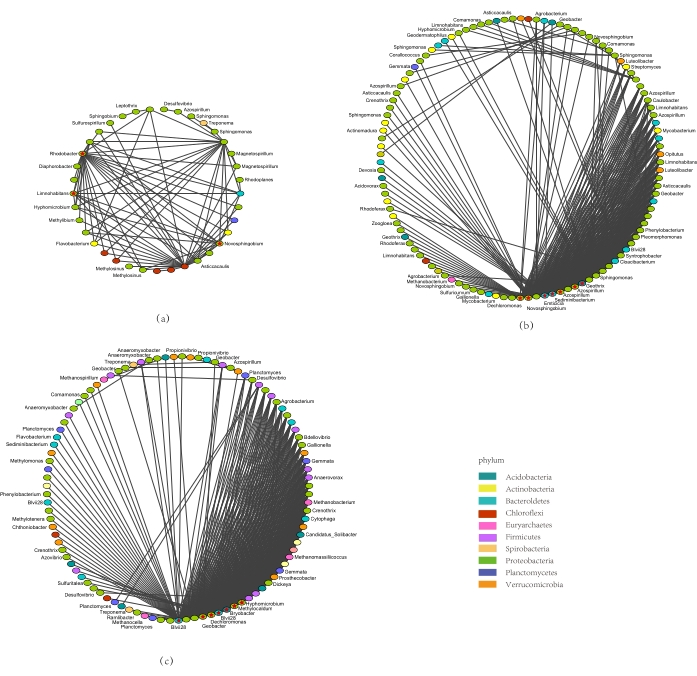
Şekil 5. Pirinç kökünde diferansiyel mikrobialpopülasyonun birlikte oluşum ağı. (a)RS-RP'deki diferansiyel mikrobiyal popülasyonların birlikte oluşum ağı; (b) ES-RS'de diferansiyel mikrobiyal popülasyonların birlikte oluşum ağı; (c) ES-RP'de diferansiyel mikrobiyal popülasyonların birlikte oluşum ağı. Analiz Cytoscape yazılımı kullanılarak gerçekleştirildi. Farklı renkler, şekilde farklı kapıları temsil eder. (RS: Rhizosphere toprağı, RP: rhizoplane ve ES: endosfer). Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
| modül | medianRank | Zsummary | modül | medianRank | Zsummary |
| greenyellow | 2 | 14 | sarı | 13 | 5.2 |
| pembe | 2 | 17 | gri60 | 14 | 3.1 |
| geceyarısı | 3 | 10 | royalblue | 15 | 2.1 |
| kahverengi | 4 | 18 | siyah | 16 | 2.7 |
| lightcyan | 6 | 8.5 | taba rengi | 17 | 3.2 |
| mor | 7 | 10 | somon | 18 | 1.3 |
| mavi | 7 | 22 | galibarda | 18 | 2.2 |
| yeşil | 8 | 12 | koyu renkli | 20 | -0.24 |
| Camgöbeği | 10 | 4.8 | altın | 20 | 14 |
| açık yeşil | 11 | 5.1 | darkgreen | 22 | -1.1 |
| lightyellow | 12 | 5.3 | gri | 22 | 0.21 |
| kırmızı | 12 | 6.1 |
Tablo 1. Rizzosfer toprağı ile rizosplan arasındaki Zsummary ve medianRank'ın sonucu.
| modül | medianRank | Zsummary | modül | medianRank | Zsummary |
| somon | 1 | 19 | lightcyan | 13 | 1.1 |
| siyah | 3 | 5.3 | kırmızı | 15 | 0.95 |
| sarı | 4 | 5.8 | geceyarısı | 15 | -0.0016 |
| açık yeşil | 5 | 0.27 | royalblue | 16 | 0.83 |
| greenyellow | 7 | 3 | galibarda | 16 | 0.52 |
| darkturquoise | 7 | 1.2 | darkgreen | 17 | 0.16 |
| gri60 | 9 | 1.1 | taba rengi | 18 | 0.64 |
| mavi | 10 | 3.9 | lightyellow | 19 | 0.52 |
| mor | 10 | 2.3 | koyu renkli | 19 | -0.18 |
| kahverengi | 12 | 2.3 | pembe | 19 | -0.71 |
| Camgöbeği | 12 | 0.78 | altın | 21 | 11 |
| yeşil | 13 | 1.7 |
Tablo 2. Rizosfer toprağı ve endosfer arasındaki Zsummary ve medianRank'ın sonucu.
| modül | medianRank | Zsummary | modül | medianRank | Zsummary |
| siyah | 1 | 15 | darkturquoise | 13 | 1.7 |
| somon | 2 | 27 | geceyarısı | 13 | 1.6 |
| sarı | 3 | 13 | açık yeşil | 13 | 0.64 |
| Camgöbeği | 4 | 5.4 | darkgreen | 14 | 1.5 |
| mavi | 8 | 3.9 | koyu renkli | 16 | 1.5 |
| lightcyan | 9 | 2.6 | mor | 17 | 2.3 |
| pembe | 10 | 3.6 | greenyellow | 18 | 0.8 |
| royalblue | 10 | 1.5 | lightyellow | 18 | 0.42 |
| kahverengi | 12 | 2.9 | galibarda | 19 | 0.2 |
| yeşil | 12 | 1.9 | altın | 21 | 18 |
| kırmızı | 12 | 1.9 | gri60 | 21 | -0.21 |
| taba rengi | 13 | 2.5 |
Tablo 3. Rhizoplane ve endosfer arasındaki Zsummary ve medianRank sonucu.
Ek S1: Bu Dosyayı indirmek için lütfen buraya tıklayın.
Ek S2: Bu Dosyayı indirmek için lütfen buraya tıklayın.
Tartışmalar
Korelasyon ağları biyoinformatik uygulamalarında giderek daha fazla kullanılmaktadır. WGCNA, biyolojik sistemin çeşitli unsurları arasındaki ilişkilerin açıklayıcı analizi için bir sistem biyolojisi yöntemidir12. R yazılım paketi WGCNA13 , 14,15üzerinde daha önceki çalışmalarda kullanılmıştır. Paket, ağ oluşturma, modül algılama, topolojik özelliklerin hesaplanması, veri simülasyonu, görselleştirme ve harici yazılımlarla etkileşim yeteneği için işlevler içerir. WGCNA, beyin kanseri 16 , maya hücre döngüsü17, fare genetiği18 , 19, primat beyin dokusu20,21,diyabet22vebitkilerdengen ifade verilerini analiz etmek için kapsamlı bir şekilde almıştır. Ağı oluşturmak için ağırlıklı gen korelasyon ağı analizini kullanın en az 8 örnek içermelidir. Bu yazıda, farklı ortamlarda mikrobiyal popülasyonlar arasındaki etkileşimleri açıklayan gen birlikte ifade ağlarına odaklandık. Farklı ortamlarda mikrobiyal popülasyonlar arasında diferansiyel ağlar elde ettik ve her ağdaki temel türleri belirledik. Temel türlerin toplum için önemli olduğu fikri, gıda ağı araştırmalarında yaygın olarak24. Karmaşık bir mikrobiyal topluluktaki bazı türler, bağırsak florasındaki Bakterioidler gibi topluluğun istikrarını ve işlevselliğini korumak için gerekli olabilir25. Mikrobiyal toplulukların analizi, belirli potansiyel öneme sahip türlerin hedef alınmasıyla önemli ölçüde basitleştirilebilir.
Temsili sonuçlarımız, yukarıda açıklanan yöntem kullanılarak tanımlanabilen mikrobiyal topluluklardaki farklılıkları vurgulamaktadır. Burada pirinç kök sisteminin farklı nişlerindeki mikroorganizmalar WGCNA'ya tabi tutuldu. Üç niş arasındaki fark, konservatif ve modül üyelik analizleri kullanılarak belirlendi. Fark modüllerinde anahtar türleri belirledik ve üç nişteki mikrobiyal toplulukların bileşimindeki farklılıklar hakkında bilgi edindik. Bu arada, birlikte oluşum ağı, pirincin köklerinde değişen mikroorganizmalar arasında önemli bir etkileşimin varlığını ortaya koydu. Bulgularımız, WGCNA'nın çeşitli ortamlardaki mikrobiyal topluluk farklılıklarının değerlendirilmesinde önemi ve fizibilitesi için doğrudan kanıt sunun.
Açıklamalar
Yazarların açıklayacak bir şeyi yok.
Teşekkürler
Bu makalenin geliştirilmesi, Çin Ulusal Doğa Bilimleri Vakfı-Guizhou İl Halk Hükümeti Karst Bilim Araştırma Merkezi Projesi (U1812401), Guizhou Normal Üniversitesi Doktora Araştırma Projesi (GZNUD[201) tarafından desteklenmiştir.7]1), Guizhou Eyaleti Bilim ve Teknoloji Destek Projesi (QKHZC[2021]YB459) ve Guiyang Bilim ve Teknoloji Projesi([2019]2-8).
Yazarlar Edwards J.A ve arkadaşlarına kamuya açık veritabanlarında pirinç mikrobiyom verileri sağladıkları ve topedit'ten (www.topeditsci.com) bu makalenin hazırlanması sırasındaki dilsel yardımı için destek sağladıkları için teşekkür etmek istiyor.
Malzemeler
| Name | Company | Catalog Number | Comments |
| R | The University of Auckland | version 4.0.2 | R is a free software environment for statistical computing and graphics. It compiles and runs on a wide variety of UNIX platforms, Windows and MacOS. |
| RStdio | JJ Allaire | version 1.4.1103 | The RStudio IDE is a set of integrated tools designed to help you be more productive with R and Python. |
| Cytoscape | version 3.7.1 | Cytoscape is an open source software platform for visualizing complex networks and integrating these with any type of attribute data. | |
| NCBI database | The National Center for Biotechnology Information advances science and health by providing access to biomedical and genomic information. |
Referanslar
- Philippot, L., Raaijmakers, J. M., Lemanceau, P., vander Putten, W. H. Going back to the roots: the microbial ecology of the rhizosphere. Nature Reviews Microbiology. 11, 789-799 (2013).
- Fierer, N. Embracing the unknown: disentangling the complexities of the soil microbiome. Nature Review Microbiology. 15 (10), 579-590 (2017).
- Jin, J., Wang, G. H., Liu, X. B., Liu, J. D., Chen, X. L., Herbert, S. J. Temporal and spatial dynamics of bacterial community in the rhizosphere of soybean genotypes grown in a black soil. Pedosphere. 19 (6), 808-816 (2009).
- Ma, B., et al. Genetic correlation network prediction of forest soil microbial functional organization. ISME J. 12 (10), 2492-2505 (2018).
- de Vries, F. T., et al. Soil bacterial networks are less stable under drought than fungal networks. Nature Communications. 9 (1), 3033 (2018).
- Colin, C., et al. Correlating transcriptional networks to breast cancer survival: a large-scale coexpression analysis. Carcinogenesis. (10), 2300-2308 (2013).
- Ma, B., Zhao, K., Lv, X., et al. Genetic correlation network prediction of forest soil microbial functional organization. ISME J. 12, 2492-2505 (2018).
- Zhang, B., Horvath, S. A general framework for weighted gene co-expression network analysis. Statistical applications in genetics and molecular biology. 4 (1), (2005).
- Edwards, J. A., et al. Compositional shifts in root-associated bacterial and archaeal microbiota track the plant life cycle in field-grown rice. PLoS Biology. 16 (2), 2003862 (2018).
- Bashan, Y., De-Bashan, L. E. How the Plant Growth-Promoting Bacterium Azospirillum Promotes Plant Growth-A Critical Assessment. Advances in Agronomy. 108, 77-136 (2010).
- Lovley, D. R., et al. Geobacter: The Microbe Electric's Physiology, Ecology, and Practical Applications. Advances in Microbial Physiology. 59, 1 (2011).
- Langfelder, P., Horvath, S. WGCNA: an R package for weighted correlation network analysis. BMC Bioinformatics. 9, 559 (2008).
- Zhang, B., Horvath, S. A General Framework for Weighted Gene Co-expression Network Analysis. Statistical Applications in Genetics and Molecular Biology. 4 (1), 17 (2005).
- Horvath, S., Dong, J. Geometric interpretation of Gene Co-expression Network Analysis. PLoS Computational Biology. 4 (8), 1000117 (2008).
- Langfelder, P., Horvath, S. Eigengene networks for studying the relationships between co-expression modules. BMC Systems Biology. 1, 54 (2007).
- Horvath, S., et al. Analysis of Oncogenic Signaling Networks in Glioblastoma Identifies ASPM as a Novel Molecular Target. Proceedings of the National Academy of Sciences of the United States of America. 103 (46), 17402-17407 (2006).
- Carlson, M. R., et al. and Sequence Conservation: Predictions from Modular Yeast Co-expression Networks. BMC Genomics. 7 (1), 40 (2006).
- Fuller, T., et al. Weighted Gene Co-expression Network Analysis Strategies Applied to Mouse Weight. Mammalian Genome. 6 (18), 463-472 (2007).
- Yin, L., Wang, Y., Lin, Y., et al. Explorative analysis of the gene expression profile during liver regeneration of mouse: a microarray-based study[J]. Artificial Cells Nanomedicine & Biotechnology. 47 (1), 1113-1121 (2019).
- Oldham, M., Horvath, S., Geschwind, D. Conservation and Evolution of Gene Co-expression Networks in Human and Chimpanzee Brains. Proceedings of the National Academy of Sciences of the United States of America. 103 (47), 17973-17978 (2006).
- Oldham, M. C., et al. Functional organization of the transcriptome in human brain. Nature Neuroscience. 11 (11), 1271-1282 (2008).
- Keller, M. P., et al. A gene expression network model of type 2 diabetes links cell cycle regulation in islets with diabetes susceptibility. Genome Research. 18 (5), 706-716 (2008).
- Weston, D., Gunter, L., Rogers, A., Wullschleger, S. Connecting genes, coexpression modules, and molecular signatures to environmental stress phenotypes in plants. BMC Systems Biology. 2 (1), 16 (2008).
- Jorda´n, F. Keystone species and food webs. Biological Sciences. 364, 1733-1741 (2009).
- Backhed, F., Ley, R. E., Sonnenburg, J. L., Peterson, D. A., Gordon, J. I. Host-Bacterial Mutualism in the Human Intestine. Science. 307, 1915-1920 (2009).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiDaha Fazla Makale Keşfet
This article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır