Method Article
Divergenz von Wurzelmikrobiota in verschiedenen Lebensräumen basierend auf gewichteten Korrelationsnetzwerken
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Die Netzwerkanalyse wurde angewendet, um die Assoziation verschiedener ökologischer mikrobieller Gemeinschaften wie Boden, Wasser und Rhizosphäre zu bewerten. Hier wird ein Protokoll vorgestellt, wie der WGCNA-Algorithmus verwendet werden kann, um verschiedene Co-Occurrence-Netzwerke zu analysieren, die in den mikrobiellen Gemeinschaften aufgrund unterschiedlicher ökologischer Umgebungen auftreten können.
Zusammenfassung
Das Wurzelmikrobiom spielt eine wichtige Rolle beim Pflanzenwachstum und der Anpassung an die Umwelt. Die Netzwerkanalyse ist ein wichtiges Werkzeug für die Untersuchung von Gemeinschaften, die die Interaktionsbeziehung oder das Co-Occurrence-Modell verschiedener mikrobieller Arten in verschiedenen Umgebungen effektiv untersuchen können. Der Zweck dieses Manuskripts ist es, Details darüber zu liefern, wie der gewichtete Korrelationsnetzwerkalgorithmus verwendet werden kann, um verschiedene Co-Occurrence-Netzwerke zu analysieren, die in mikrobiellen Gemeinschaften aufgrund unterschiedlicher ökologischer Umgebungen auftreten können. Alle Analysen des Experiments werden im WGCNA-Paket durchgeführt. WGCNA ist ein R-Paket für die gewichtete Korrelationsnetzwerkanalyse. Die experimentellen Daten, die zur Demonstration dieser Methoden verwendet wurden, waren mikrobielle Gemeinschaftsdaten aus der NCBI-Datenbank (National Center for Biotechnology Information) für drei Nischen des Reiswurzelsystems (Oryza sativa). Wir haben den algorithmus für gewichtete Korrelationsnetzwerke verwendet, um Co-Abundance-Netzwerke mikrobieller Gemeinschaften in jeder der drei Nischen zu konstruieren. Dann wurden differentielle Ko-Abundanz-Netzwerke zwischen Endosphäre, Rhizoplan und Rhizosphärenboden identifiziert. Darüber hinaus wurden die Kerngattungen im Netzwerk durch das "WGCNA"-Paket erhalten, das eine wichtige regulierte Rolle bei Netzwerkfunktionen spielt. Diese Methoden ermöglichen es den Forschern, die Reaktion mikrobieller Netzwerke auf Umweltstörungen zu analysieren und verschiedene mikrobielle ökologische Reaktionstheorien zu überprüfen. Die Ergebnisse dieser Methoden zeigen, dass die signifikanten differentiellen mikrobiellen Netzwerke in der Endosphäre, Rhizoplane und Rhizosphäre Boden von Reis identifiziert werden.
Einleitung
Mikrobiomforschung hat wichtige Implikationen für das Verständnis und die Manipulation von Ökosystemprozessen1,2. Mikrobielle Populationen sind durch interagierende ökologische Netzwerke miteinander verbunden, deren Eigenschaften die Reaktion von Mikroorganismen auf Umweltveränderungen beeinflussen können3,4. Darüber hinaus beeinflussen die Eigenschaften dieser Netzwerke die Stabilität mikrobieller Gemeinschaften und sind eng mit der Bodenfunktion verbunden5. Die gewichtete Genkorrelationsnetzwerkanalyse wurde nun häufig für die Erforschung der Beziehung zwischen Genen und mikrobiellen Gemeinschafteneingesetzt 6. Frühere Studien haben sich hauptsächlich auf die Assoziationen zwischen Netzwerken verschiedener Gene oder Populationen und der Außenwelt konzentriert7. Die Unterschiede in den Korrelationsnetzwerken, die mikrobielle Populationen unter verschiedenen Umweltbedingungen bilden, sind jedoch kaum untersucht. Der Zweck der in diesem Artikel vorgestellten Forschung besteht darin, Einblicke und Details zur schnellen Implementierung des WGCNA-Algorithmus zu liefern, um ein Netzwerk von Mikrobiomproben zu konstruieren, die unter verschiedenen Umweltbedingungen gesammelt wurden. Basierend auf den Analyseergebnissen bewerteten wir die Zusammensetzung und Unterschiede der Population und diskutierten die Beziehung zwischen verschiedenen mikrobiellen Populationen. Der folgende grundlegende Fluss des gewichteten Korrelationsnetzwerkalgorithmus8 wurde angewendet. Zunächst musste eine Ähnlichkeitsmatrix erstellt werden, indem der Pearson-Korrelationskoeffizient zwischen den OTU-Ausdrucksprofilen (Operational Taxonomic Units) berechnet wurde. Dann wurden die Parameter der Adjazenzfunktionen (die Power- oder die Sigmoid-Adjazenzfunktionen) mit einem skalenfreien Topologiekriterium übernommen, die Ähnlichkeitsmatrix wurde in eine Adjazenzmatrix umgewandelt und jedes Co-Occurrence-Netzwerk entsprach einer Adjazenzmatrix. Wir verwendeten hierarchisches Clustering mit durchschnittlicher Verknüpfung in Verbindung mit der TOM-basierten Unähnlichkeit, um OTUs mit kohärenten Ausdrucksprofilen in Module zu gruppieren. Darüber hinaus berechneten wir die Beziehung zwischen konservativer Statistik und den zugehörigen Parameteranalysemodulen und identifizierten schließlich die Hub-OTU im Modul. Diese Methoden eignen sich besonders für die Analyse der Unterschiede in den Netzwerkstrukturen zwischen verschiedenen mikrobiellen Populationen unter divergierenden Umweltbedingungen. In diesem Manuskript haben wir die Methode der Co-Expression Network-Entwicklung, die Analyse der Unterschiede zwischen den Modulen detailliert beschrieben und einen kurzen Überblick über die Schritte im Verfahren gegeben, um die Kernspezies in verschiedenen Modulnetzwerken zu erhalten.
Protokoll
1. Daten-Download
- Laden Sie die Daten des Beitritts PRJNA386367 aus der NCBI-Datenbank herunter. Wählen Sie aus den Daten des Beitritts PRJNA386367 die Rhizosphären-, Rhizoplan- und Endosphären-Mikrobiomdaten von Reispflanzen aus, die 2014 14 Wochen lang in einem untergetauchten Reisfeld in Arbuckle, Kalifornien, angebaut wurden.
HINWEIS: Die Rhizosphären-, Rhizoplan- und Endosphärenmikrobiomdaten wurden von der OTUs-Tabelle in Der Beitrittstabelle PRJNA386367 dargestellt.
2. Optimale Leistungswertermittlung
HINWEIS: Das WGCNA-Paket enthält alle folgenden Funktionsparameter. WGCNA ist ein R-Paket für die gewichtete Korrelationsnetzwerkanalyse. Die Tastenbefehlszeilen beziehen sich auf die Ergänzung S1.
- Öffnen Sie in der R-Sprachumgebung die Rstudio-Software und installieren Sie das WGCNA-Paket.
- Laden Sie die Daten und verwenden Sie die goodSamplesGenes-Funktion, um die Richtigkeit der Daten zu überprüfen. Führen Sie die Befehlszeilen aus:
"gsg = goodSamplesGenes(datExpr0, verbose = 3)
gsg$allOK "
Klicken Sie auf Ausführen. - Suchen Sie nach Ausreißern und lagern Sie Proben, die den Anforderungen entsprechen. Wenn das Prüfergebnis TRUE ist, fahren Sie mit dem nächsten Schritt fort. Speichern Sie das Ergebnis.
- Verwenden Sie die Funktion PickSoftThreshold, um den skalenfreien Index R 2 derbeiden Gruppen der Daten unter unterschiedlichen Leistungswerten zu berechnen. Führen Sie die Befehlszeile aus:
"sft = pickSoftThreshold(datExpr0, powerVector = powers, verbose = 5)"
Klicken Sie auf Ausführen. - Visualisieren Sie die Ergebnisse (Abbildung 1). Führen Sie die Befehlszeile aus:
"plot(sft$fitIndices[,1], -sign(sft$fitIndices[,3])*sft$fitIndices[,2],
xlab="Soft Threshold (power)",ylab="Scale Free Topology Model Fit,signed R^2",type="n",
main = paste("ES_Scale independence"));
text(sft$fitIndices[,1], -sign(sft$fitIndices[,3])*sft$fitIndices[,2],
labels=powers,cex=cex1,col="red");
abline(h=0,9;col="rot")
plot(sft$fitIndices[,1], sft$fitIndices[,5],
xlab="Soft Threshold (power)",ylab="Mean Connectivity", type="n",
main = paste("ES_Mean connectivity"))
text(sft$fitIndices[,1], sft$fitIndices[,5], labels=powers, cex=cex1,col="red")"
Klicken Sie auf Ausführen.
HINWEIS: Die Prämisse des gewichteten Korrelationsnetzwerkalgorithmus ist, dass die etablierte Co-Expression-Netzwerkstruktur den Standards des kriteriums der skalenfreien Topologie entspricht, wodurch ihre Robustheit erhöht wird. Ein skalenfreier Index näher an 1 gibt eine Netzwerkstruktur an, die näher am skalenfreien Netzwerk liegt. - Wählen Sie den Leistungswert aus, wenn der skalenfreie Index R2 größer als 0,9 ist, und fahren Sie mit dem nächsten Analyseschritt fort.
HINWEIS: Wenn der skalenfreie Index nahe bei 1 liegt, ist die Netzwerkstruktur näher am skalenfreien Netzwerk. Bei der Analyse von zwei oder mehr Netzwerken ist es notwendig, jedes Netzwerk in die Nähe des Leistungswerts des skalenfreien Netzwerks zu bringen, um die Vergleichbarkeit zwischen den co-ausgedrückten Netzwerken zu erfüllen.
3. Aufbau eines Co-Expression-Netzwerks und Modulidentifikation
HINWEIS: Basierend auf dem oben berechneten Leistungswert wird das Co-Occurrence-Netzwerk aufgebaut. Die Tastenbefehlszeilen beziehen sich auf die Ergänzung S2.
- Verwenden Sie die Adjazenzfunktion im WGCNA-Paket, um signierte Parameter für den Aufbau eines symbolischen Netzwerks für das gleichzeitige Auftreten hinzuzufügen. Führen Sie die Befehlszeile aus:
"adjacency = adjacency(datExpr0, power = softPower)"
Klicken Sie auf Ausführen. - Wenden Sie die TOM-Ähnlichkeitsfunktion an, um ein topologisches überlappendes Netzwerk zu entwickeln und das Unähnlichkeitsnetzwerk zu berechnen. Führen Sie die Befehlszeile aus:
"TOM = TOMsimilarity(adjacency);
dissTOM = 1-TOM"
Klicken Sie auf Ausführen.
HINWEIS: Der signierte Parameter wurde hinzugefügt, um den Netzwerktyp für die Topologieüberlappung festzulegen. - Verwenden Sie die hclust-Funktion, um die durchschnittliche hierarchische Clustering-Methode für die Verknüpfung für hierarchisches Clustering auszuwählen. Führen Sie die Befehlszeile aus:
"geneTree = hclust(as.dist(dissTOM), method = "average");"
Klicken Sie auf Ausführen. - Verwenden Sie die cutreeDynamic-Funktion, um dynamisches Verzweigungsschneiden durchzuführen, und legen Sie den Parameter minClusterSize auf 30 fest. Rufen Sie das Ergebnis der Modulerkennung ab. Führen Sie die Befehlszeile aus:
"dynamicMods = cutreeDynamic(dendro = geneTree, distM = dissTOM, deepSplit = 2, pamRespectsDendro = FALSE, minClusterSize = minModuleSize);"
Klicken Sie auf Ausführen.
HINWEIS: Die minimale Modulgröße darf nicht kleiner als 30 sein. - Berechnen Sie die Moduleigenie jedes OTUs-Moduls durch die ModuleEigengenes-Funktion. Führen Sie die Befehlszeile aus:
"MEList = moduleEigengenes(datExpr0, colors = dynamicColors)
MEs = MEList$eigengenes"
Klicken Sie auf Ausführen.
HINWEIS: Das Moduleigen stellte die gesamte OTU-Ausdrucksebene im Modul dar. Es war keine spezifische OTU, sondern die erste Hauptkomponente jedes Clusters, die durch singuläre Netzwerkwertzerlegung erhalten wurde. - Führen Sie die Clusterfunktion basierend auf dem Korrelationskoeffizienten des Moduleigens aus. Verwenden Sie die funktion mergeCloseModules, um die Module mit einem Wert kleiner als 0,25 zusammenzuführen. Führen Sie die Befehlszeile aus:
"merge = mergeCloseModules(datExpr0, dynamicColors, cutHeight = MEDissThres, verbose = 3)"
Klicken Sie auf Ausführen. - Verwenden Sie schließlich die plotDendroAndColors-Funktion zur Visualisierung, um das Modulzuweisungsanzeigediagramm jedes Co-Expression-Netzwerks abzustellen (Abbildung 2). Verwenden Sie die Tabellenfunktion, um die Modulzuordnung zu extrahieren, die jeder OTin der Modulzuordnungstabelle entspricht. Führen Sie die Befehlszeile aus:
"plotDendroAndColors(geneTree, mergedColors, "Merged dynamic",dendroLabels = FALSE,
hang = 0,03,addGuide = TRUE, guideHang = 0,05,
main = "ES_Gene Dendrogramm und Modulfarben")"
Klicken Sie auf Ausführen.
HINWEIS: Im Modulzuordnungsdiagramm des Co-Expressing-Netzwerks repräsentieren verschiedene Farben verschiedene Module und Grau OTUs, die nicht in ein Modul eingeteilt werden können. Eine größere Anzahl von OTUs im Graumodul weist darauf hin, dass die Vorverarbeitungsqualität der Ausdrucksmatrix im Frühstadium schlecht ist.
4. Modulvergleich
HINWEIS: Mit dieser Methode können die Netzwerkmodule zweier ökologischer mikrobieller Gemeinschaften verglichen werden. Vergleichen Sie in diesem Artikel die Unterschiede mikrobieller Netzwerkmodule zwischen Endosphäre und Rhizoplan, Endosphäre und Rhizosphäre, Rhizosphäre und Rhizoplane.
- Konservierungstest
- Laden Sie die Parameter und Ergebnisse der beiden Datensätze, die in den vorherigen Schritten gespeichert wurden.
- Legen Sie das Ergebnis der Netzwerkmodulzuweisung einer Gruppe mikrobieller Daten als Referenzgruppe fest, während die andere Gruppe als Testgruppe.
- Verwenden Sie die Funktion modulePreservation, um die Werte der statistischen Parameter für die Konservativität Z_summary und medianRank zu berechnen. Führen Sie die Befehlszeile aus:
"system.time({mp=modulePreservation(multiExpr,
multiColor,referenceNetworks=1,
nPermutation=100, randomSeed=1,quickCor=0,verbose=3)})"
Klicken Sie auf Ausführen.
HINWEIS: Dieses Ergebnis kann die Konservativität zwischen den Modulen quantifizieren. Z_summary>10 gibt an, dass zwei Module stark erhalten sind, während Z_summary<2 nicht erhaltene Module bezeichnet. medianRank drückt die relative Erhaltung des durch Ranking bewerteten Moduls aus. Höhere MedianRank-Werte bezeichnen nicht erhaltene Module. (Die Tastenbefehlszeilen beziehen sich auf die Ergänzung S3.) - Verwenden Sie die Plotfunktion, um die Ergebnisse zu visualisieren (Abbildung 3). Holen Sie sich die Parameter Z_summary und medianRank (Tabelle 1).
HINWEIS: Die Netzwerkmodule, die sowohl den Z_summary wert kleiner als 2 als auch den mittleren Rangwert an der Spitze erfüllen, sind das am stärksten nicht erhaltene Modul in den beiden ökologischen mikrobiellen Gemeinschaften. - Basierend auf den Ergebnissen der oben genannten zwei statistischen Parameter, um das Modul mit dem am stärksten nicht erhaltenen Modul der beiden Netzwerke zu identifizieren.
- Korrelationsanalyse der Modulzugehörigkeit
- Legen Sie die Modulzuordnungsergebnisse der beiden Netzwerke als Referenz bzw. Testgruppe fest.
HINWEIS: Die Einstellungen müssen mit den Einstellungen des Erhaltungstests identisch sein. - Verwenden Sie die funktion corPvalueStudent, um den kME-Wert (Modulmitgliedschaft) jeder OTU in mehreren Kandidatenmodulen zu extrahieren.
Führen Sie die Befehlszeile aus:
"Pvalue = as.data.frame(corPvalueStudent(as.matrix
(ModulMitgliedschaft), Beispiele))"
Klicken Sie auf Ausführen.
HINWEIS: kME steht für den Grad der Modulzugehörigkeit. ME steht für Module eigen, das die Gesamtebene des OTU-Ausdrucks im Modul darstellt. kME ist der Korrelationskoeffizient zwischen jeder OTU und dem ME. Quantifizieren Sie die Bedeutung von OTU im Netzwerk durch den kME-Wert von OTU. (Die Tastenbefehlszeilen beziehen sich auf die Ergänzung S4.) - Verwenden Sie dann die VerboseScatterplot-Funktion, um den Korrelationskoeffizienten des kME-Werts der entsprechenden OTUs in den beiden Netzwerken zu berechnen und das Korrelationsanalysediagramm zu zeichnen (Abbildung 4).
Führen Sie die Befehlszeile aus:
"verboseScatterplot(abs(TModuleMembership)
[TmoduleGenes, Tcolumn]),
abs(NModuleMembership[NmoduleGenes, Ncolumn]),
xlab = paste("kME in", "ES"),
ylab = paste("kME in", "RP"),
main = Paste("hellgelb"),
cex.main = 1,7, cex.lab = 1,6, cex.axis = 1,6, col = modulecolor)"
Klicken Sie auf Ausführen. - Wählen Sie das Modul mit dem kleinsten Korrelationskoeffizienten des kME-Wertes der OTU der beiden Netzwerke. Betrachten Sie dieses Modul als den größten Unterschied der beiden Netzwerke.
- Legen Sie die Modulzuordnungsergebnisse der beiden Netzwerke als Referenz bzw. Testgruppe fest.
5. Analyse des mikrobiellen Differentialnetzwerkmoduls
- Erhalten Sie Daten der dominanten Bakterienphyla durch statistische Analyse des OTU-Sequenzsatzes des Moduls mit dem größten Unterschied.
HINWEIS: Der OTU-Sequenzsatz des Moduls mit der größten Differenz wird durch die Taxonomie von Phyla summiert. Die dominante Bakterienphyla machte mehr als 10% aus. - Verwenden Sie dann die ExportNetworkToCytoscape-Funktion, um die Datei abzustellen, die die Interaktionsbeziehungsinformationen der OTU im größten Differenzmodul enthält.
Führen Sie die Befehlszeile aus:
"cyt = exportNetworkToCytoscape(modTOM,
edgeFile = paste("NEW-ES_CytoscapeInput-edges-", modules , ".txt", sep=""),
nodeFile = paste("NEW-ES_CytoscapeInput-nodes-", modules, ".txt", sep=""),
weighted = TRUE,threshold = 0,5, nodeNames = modProbes,
altNodeNames = modGenes, nodeAttr = moduleColors[inModule])"
Klicken Sie auf Ausführen. - Importieren Sie die Datei in Cytoscape. Stellen Sie den Schwellenwert auf 0,5 ein und passen Sie andere Parameter nach Bedarf an.
- Konstruieren Sie ein Co-Occurrence-Netzwerk differentieller Mikroorganismen (Abbildung 5).
- Erhalten Sie die Informationen der Kerngattung, die die wichtigste regulatorische Rolle im Netzwerk hat.
HINWEIS: Entsprechend dem kME-Wert von OUT kann die Kerngattung definiert werden. - Abschließend wurden die Funktionen der Kerngattung bewertet und ihr Einfluss auf das gesamte Differenzennetzwerk analysiert.
Ergebnisse
Die repräsentativen Ergebnisse in diesem Artikel wurden aus den Daten des kalifornischen Abaker-Reiswurzelmikrobioms von 2014 in der NCBI-Datenbank (PRJNA386367)9heruntergeladen. Die Daten umfassen die Rhizosphären-, Rhizoplan- und Endosphären-Mikrobiomproben von Reispflanzen, die 14 Wochen lang in einem untergetauchten Reisfeld angebaut wurden. Wir verwendeten den WGCNA-Algorithmus, um den Leistungswert auszuwählen, der die drei Netzwerke zufriedenstellte, die sich in der Nähe des skalenfreien Netzwerks befanden (Abbildung 1) und drei Co-Expression-Netzwerke entwickelten (Abbildung 2). In der Endosphäre, Rhizoplane und Rhizosphäre Boden mikrobielle Co-Expressionsnetzwerk wurden 23, 22 und 21 Module identifiziert. Diese Ergebnisse deuten darauf hin, dass die Anzahl der mikrobiellen Interaktionsnetzwerke in den drei Nischen grundsätzlich gleich war.
Wir verglichen weiter die Unterschiede in den mikrobiellen Netzwerkmodulen innerhalb der Endosphäre, Rhizoplane und Rhizosphäre Boden. Die folgenden Konservierungstestergebnisse der Module der drei Nischengruppen wurden erhalten. Zwischen dem Rhizosphärenboden und dem Rhizoplane existierten drei extrem nicht erhaltene Module (Abbildung 3a, Tabelle 1). Zusätzlich waren neun extrem nicht erhaltene Module zwischen dem Rhizosphärenboden und der Endosphäre vorhanden (Abbildung 3b, Tabelle 2) und sechs extrem nicht erhaltene Module zwischen Rhizoplane und Endosphäre (Abbildung 3c, Tabelle 3). Darüber hinaus wurden extrem nicht konservierte Module unter den drei Nischen gefunden, was auf das Vorhandensein großer Unterschiede in der Zusammensetzung der Mikroorganismen zwischen den drei Nischen hinweist. Die Ergebnisse der Korrelationsanalyse der Modulmitgliedschaft der erhaltenen nichtkonservativen Module sind in Abbildung 4 dargestellt. Aus der Abbildung ist unter den drei Nischen ein deutlich anderes Modul mit der geringsten Korrelation zwischen den beiden Nischen sichtbar, was den signifikantesten Unterschied zwischen ihnen darstellt.
Im Rhizosphären-Rhizoplan-Differenznetzwerk (Abbildung 5a) waren Proteobakterien (72,97%) der dominante Stamm. Im Rhizosphären-Endosphären-Differenznetzwerk (Abbildung 5b) waren die dominanten Phyla Proteobakterien (66,36%), Actinobakterien (10,1%) und Bacteroidetes (10,9%). Im Rhizoplan-Endosphären-Differenznetzwerk (Abbildung 5c) waren die dominanten Phyla Proteobakterien (41,41%), Bacteroidetes (10,10%), Firmicutes (12,12%) und Verrucomicrobia.
Drei Kerngattungen (Abbildung 5a), darunter Rhodobacter und Novosphingob, sechs Kerngattungen (Abbildung 5b), einschließlich Blvii28 und Dechloromonas, und fünf Kerngattungen (Abbildung 5c), einschließlich Cellvibrio und Geobacter, übten wichtige regulatorische Funktionen in den drei differentiellen Ko-Vorkommensnetzwerken aus. Alle Kerngattungen, mit Ausnahme von Dechloromonas,hatten Einfluss auf nur ein Netzwerk, was auf die Verfügbarkeit erheblicher Unterschiede in der relativen Häufigkeit mikrobieller Populationen und Arten zwischen den drei Nischen der Reiswurzeln hindeutet, die die Häufigkeit und Vielfalt der bestehenden mikrobiellen Wurzelgemeinschaften entscheidend beeinflussten.
Die Kerngattung Azospirillum, die im Rhizosphären-Endosphären-Differenznetzwerk von Reis vorhanden ist, war an der Stickstofffixierung beteiligt und förderte das Pflanzenwachstum10. Darüber hinaus kann die Gattung Geobacter, die im Rhizoplane-Endosphären-Differenznetzwerk signifikant angereichert wurde, der Hauptfaktor sein, der die Verringerung unlöslicher Fe- und Mn-Oxide in vielen Böden und Sedimenten induziert11. Diese Mikroorganismen interagieren mit einer Reihe von mikrobiellen Gemeinschaften in den Wurzelnischen und beteiligen sich aktiv an der Regulierung mikrobieller Netzwerke, die für das Wachstum und die Entwicklung von Reiswurzeln von entscheidender Bedeutung sein könnten.
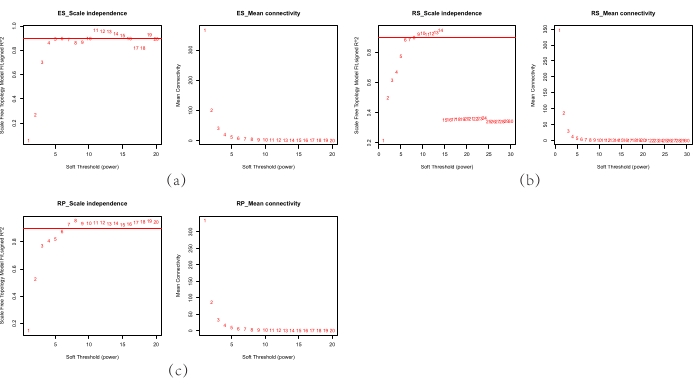
Abbildung 1. Auswertung von powerβ in den Datensätzen. a) Bewertung der im ES-Datensatz β; b) Bewertung der im RS-Datensatz β; (c) Auswertung der β im RP-Datensatz, Verteilung des skalenfreien Index R2 (links), Verteilung der mittleren Konnektivität (rechts) entlang verschiedener Soft-Power-Indizes. Der Wert der besten Leistung wurde erreicht, wenn R2 zur Sättigung neigte und nicht unter 0,8 lag. Die drei Co-Expression-Netzwerke mussten auf den gleichen Leistungswert eingestellt werden, um ihre Vergleichbarkeit zu gewährleisten. (RS: Rhizosphärenboden, RP: Rhizoplan und ES: Endosphäre). Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 2. OTU-Dendrogramm, das durch hierarchisches Clustering der durchschnittlichen Verknüpfung erhalten wird. a)OTU-Dendrogramm aus dem ES-Netz; b) OTU-Dendrogramm aus dem RS-Netz; c) OTU-Dendrogramm aus dem RP-Netzwerk. Die Farbzeile unter dem Dendrogramm gibt die Modulzuweisung an, die durch den Dynamic Tree Cut-Algorithmus bestimmt wird. (RS: Rhizosphärenboden, RP: Rhizoplan und ES: Endosphäre) Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 3. Ergebnisse des Konservierungstests. a) Die Analyseergebnisse basieren auf der Zuweisung des RS-Co-Expressionsnetzwerks als Referenzgruppe und den Rp-Coexpressionsnetzwerkmodulzuordnungsergebnissen als Testgruppe. b) Die Analyseergebnisse basieren auf der Zuweisung des ES-Co-Expression-Netzwerkmoduls als Referenzgruppe und den Ergebnissen der RS-Co-Expression-Netzwerk-Modulzuordnung als Testgruppe; (c) Die Analyseergebnisse basieren auf der ES-Co-Expression-Netzwerk-Modulzuordnung als Referenzgruppe und den RP-Co-Expression-Netzwerk-Modulzuordnungsergebnissen als Testgruppe. Z_summary > 10 zeigt an, dass zwei Module stark erhalten sind, während Z_summary < 2 nicht erhaltene Module bezeichnet. medianRank drückt die relative Erhaltung des durch Ranking bewerteten Moduls aus. Höhere MedianRank-Werte bezeichnen nicht erhaltene Module. (RS: Rhizosphärenboden, RP: Rhizoplan und ES: Endosphäre) Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
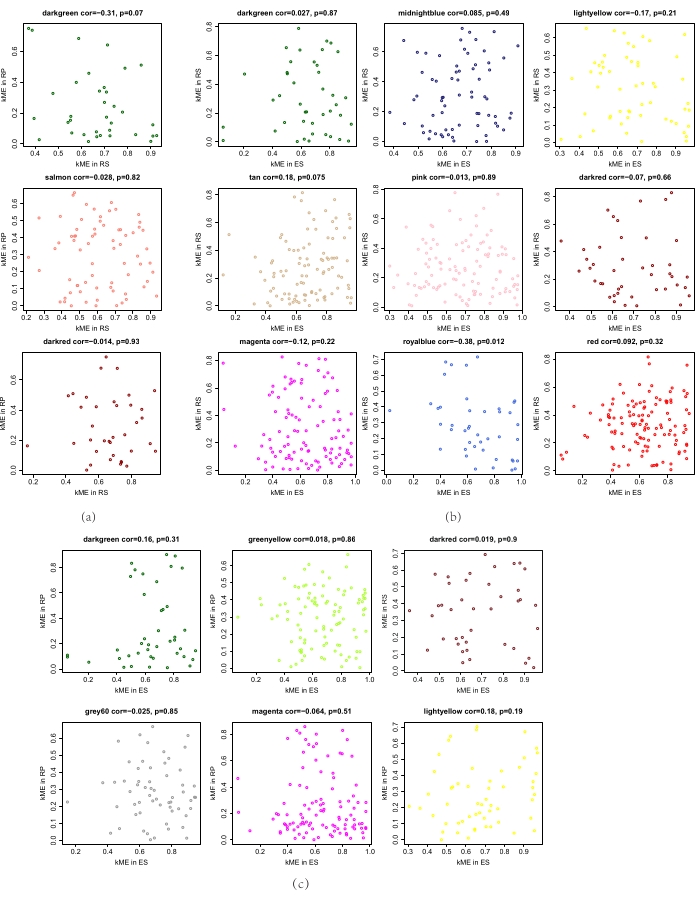
Abbildung 4. Korrelationsanalyse der Modulmitgliedschaften. a) Modulkorrelation des kME-Wertes zwischen dem RS- und RP-Netz; b) Modulkorrelation des kME-Wertes zwischen RS- und RP-Netz; (c) Modulkorrelation des kME-Wertes zwischen RP und ES-Netzwerk (RS: Rhizosphärenboden, RP: Rhizoplane, ES: Endosphäre und KME: Modulmitgliedschaft). Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
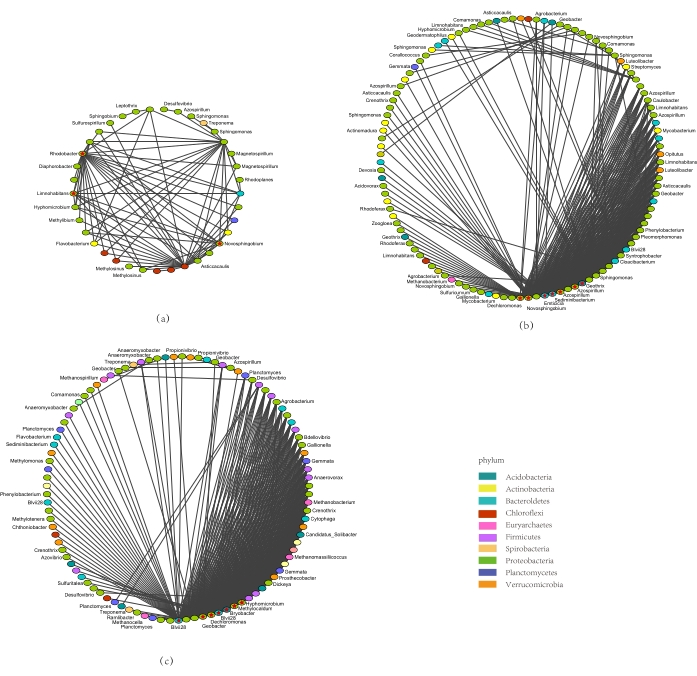
Abbildung 5. Co-Vorkommen Netzwerk der differentiellen Mikrobialitätpopulation in der Reiswurzel. a)Netzwerk des gleichzeitigen Auftretens differentieller mikrobieller Populationen in RS-RP; b) Netzwerk des gemeinsamen Auftretens differentieller mikrobieller Populationen in ES-RS; c) Koexetretensnetzwerk differentieller mikrobieller Populationen in ES-RP. Die Analyse wurde mit der Cytoscape-Software durchgeführt. Verschiedene Farben stellen verschiedene Tore in der Figur dar. (RS: Rhizosphärenboden, RP: Rhizoplane und ES: Endosphäre). Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
| Modul | medianRank | Zsummary | Modul | medianRank | Zsummary |
| grüngelb | 2 | 14 | gelb | 13 | 5.2 |
| rosa | 2 | 17 | grau60 | 14 | 3.1 |
| mitternachtblau | 3 | 10 | royalblue | 15 | 2.1 |
| braun | 4 | 18 | schwarz | 16 | 2.7 |
| Lightcyan | 6 | 8.5 | gerben | 17 | 3.2 |
| lila | 7 | 10 | Lachs | 18 | 1.3 |
| blau | 7 | 22 | Magenta | 18 | 2.2 |
| grün | 8 | 12 | dunkel | 20 | -0.24 |
| zyan | 10 | 4.8 | Gold | 20 | 14 |
| hellgrün | 11 | 5.1 | dunkelgrün | 22 | -1.1 |
| hellgelb | 12 | 5.3 | grau | 22 | 0.21 |
| rot | 12 | 6.1 |
Tabelle 1. Ergebnis von Zsummary und MedianRank zwischen dem Rhizosphärenboden und dem Rhizoplane.
| Modul | medianRank | Zsummary | Modul | medianRank | Zsummary |
| Lachs | 1 | 19 | Lightcyan | 13 | 1.1 |
| schwarz | 3 | 5.3 | rot | 15 | 0.95 |
| gelb | 4 | 5.8 | mitternachtblau | 15 | -0.0016 |
| hellgrün | 5 | 0.27 | royalblue | 16 | 0.83 |
| grüngelb | 7 | 3 | Magenta | 16 | 0.52 |
| dunkeltürkis | 7 | 1.2 | dunkelgrün | 17 | 0.16 |
| grau60 | 9 | 1.1 | gerben | 18 | 0.64 |
| blau | 10 | 3.9 | hellgelb | 19 | 0.52 |
| lila | 10 | 2.3 | dunkel | 19 | -0.18 |
| braun | 12 | 2.3 | rosa | 19 | -0.71 |
| zyan | 12 | 0.78 | Gold | 21 | 11 |
| grün | 13 | 1.7 |
Tabelle 2. Ergebnis von Zsummary und medianRank zwischen Rhizosphärenboden und Endosphäre.
| Modul | medianRank | Zsummary | Modul | medianRank | Zsummary |
| schwarz | 1 | 15 | dunkeltürkis | 13 | 1.7 |
| Lachs | 2 | 27 | mitternachtblau | 13 | 1.6 |
| gelb | 3 | 13 | hellgrün | 13 | 0.64 |
| zyan | 4 | 5.4 | dunkelgrün | 14 | 1.5 |
| blau | 8 | 3.9 | dunkel | 16 | 1.5 |
| Lightcyan | 9 | 2.6 | lila | 17 | 2.3 |
| rosa | 10 | 3.6 | grüngelb | 18 | 0.8 |
| royalblue | 10 | 1.5 | hellgelb | 18 | 0.42 |
| braun | 12 | 2.9 | Magenta | 19 | 0.2 |
| grün | 12 | 1.9 | Gold | 21 | 18 |
| rot | 12 | 1.9 | grau60 | 21 | -0.21 |
| gerben | 13 | 2.5 |
Tabelle 3. Ergebnis von Zsummary und medianRank zwischen Rhizoplane und Endosphäre.
Ergänzung S1: Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzung S2: Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzung S3: Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzung S4: Bitte klicken Sie hier, um diese Datei herunterzuladen.
Diskussion
Korrelationsnetzwerke werden zunehmend in bioinformatischen Anwendungen eingesetzt. WGCNA ist eine systembiologische Methode zur deskriptiven Analyse der Beziehungen zwischen verschiedenen Elementen eines biologischen Systems12. R-Softwarepaket wurde in früheren Arbeiten an WGCNA13,14,15verwendet. Das Paket enthält Funktionen für den Netzwerkaufbau, Modulerkennung, Berechnung topologischer Eigenschaften, Datensimulation, Visualisierung und Die Möglichkeit zur Anbindung an externe Software. WGCNA wurde ausgiebig eingesetzt, um Genexpressionsdaten von Hirntumoren16, Hefezellzyklus17, Mausgenetik18,19, Primatenhirngewebe20,21, Diabetes22und Pflanzen23zu analysieren . Verwenden Sie die gewichtete Genkorrelationsnetzwerkanalyse, um das Netzwerk zu konstruieren, das mindestens 8 Proben enthalten muss. In diesem Artikel haben wir uns auf Gen-Co-Expressionsnetzwerke konzentriert, die die Interaktionen zwischen mikrobiellen Populationen in verschiedenen Umgebungen beschreiben. Wir erhielten differentielle Netzwerke zwischen mikrobiellen Populationen in divergierenden Umgebungen und identifizierten die Schlüsselarten in jedem Netzwerk. Die Vorstellung, dass Schlüsselarten für die Gemeinschaft wichtig sind, wurde in der Nahrungsnetzforschung weit verbreitet24. Einige der Arten in einer komplexen mikrobiellen Gemeinschaft können wesentlich sein, um die Stabilität und Funktionalität der Gemeinschaft zu erhalten, wie Bacteroides in der Darmflora25. Die Analyse mikrobieller Gemeinschaften kann erheblich vereinfacht werden, indem auf bestimmte Arten von potenzieller Bedeutung abzielt.
Unsere repräsentativen Ergebnisse verdeutlichen die Unterschiede in mikrobiellen Gemeinschaften, die mit der oben beschriebenen Methode identifiziert werden können. Hier wurden Mikroorganismen in verschiedenen Nischen des Reiswurzelsystems WGCNA unterzogen. Die Differenz zwischen den drei Nischen wurde mittels konservativer und Modulzugehörigkeitsanalysen identifiziert. Wir haben die Schlüsselarten in den Differenzmodulen bestimmt und Informationen über die Unterschiede in der Zusammensetzung der mikrobiellen Gemeinschaften in den drei Nischen erhalten. In der Zwischenzeit zeigte das Co-Occurrence-Netzwerk das Vorhandensein einer signifikanten Wechselwirkung zwischen den sich verändernden Mikroorganismen in den Wurzeln von Reis. Unsere Ergebnisse liefern direkte Beweise für die Bedeutung und Machbarkeit von WGCNA bei der Bewertung mikrobieller Gemeinschaftsunterschiede in verschiedenen Umgebungen.
Offenlegungen
Die Autoren haben nichts preiszugeben.
Danksagungen
Die Entwicklung dieses Manuskripts wurde durch Mittel der National Natural Science Foundation of China-Guizhou Provincial People's Government Karst Science Research Center Project (U1812401), des Doctoral Research Project der Guizhou Normal University (GZNUD[2017]1), des Science and Technology Support Project der Provinz Guizhou (QKHZC[2021]YB459) und des Science and Technology Project des Guiyang([2019]2-8) unterstützt.
Die Autoren danken Edwards J.A et al. für die Bereitstellung von Reismikrobiomdaten in öffentlichen Datenbanken und die Unterstützung von TopEdit (www.topeditsci.com) für die sprachliche Unterstützung bei der Erstellung dieses Manuskripts.
Materialien
| Name | Company | Catalog Number | Comments |
| R | The University of Auckland | version 4.0.2 | R is a free software environment for statistical computing and graphics. It compiles and runs on a wide variety of UNIX platforms, Windows and MacOS. |
| RStdio | JJ Allaire | version 1.4.1103 | The RStudio IDE is a set of integrated tools designed to help you be more productive with R and Python. |
| Cytoscape | version 3.7.1 | Cytoscape is an open source software platform for visualizing complex networks and integrating these with any type of attribute data. | |
| NCBI database | The National Center for Biotechnology Information advances science and health by providing access to biomedical and genomic information. |
Referenzen
- Philippot, L., Raaijmakers, J. M., Lemanceau, P., vander Putten, W. H. Going back to the roots: the microbial ecology of the rhizosphere. Nature Reviews Microbiology. 11, 789-799 (2013).
- Fierer, N. Embracing the unknown: disentangling the complexities of the soil microbiome. Nature Review Microbiology. 15 (10), 579-590 (2017).
- Jin, J., Wang, G. H., Liu, X. B., Liu, J. D., Chen, X. L., Herbert, S. J. Temporal and spatial dynamics of bacterial community in the rhizosphere of soybean genotypes grown in a black soil. Pedosphere. 19 (6), 808-816 (2009).
- Ma, B., et al. Genetic correlation network prediction of forest soil microbial functional organization. ISME J. 12 (10), 2492-2505 (2018).
- de Vries, F. T., et al. Soil bacterial networks are less stable under drought than fungal networks. Nature Communications. 9 (1), 3033(2018).
- Colin, C., et al. Correlating transcriptional networks to breast cancer survival: a large-scale coexpression analysis. Carcinogenesis. (10), 2300-2308 (2013).
- Ma, B., Zhao, K., Lv, X., et al. Genetic correlation network prediction of forest soil microbial functional organization. ISME J. 12, 2492-2505 (2018).
- Zhang, B., Horvath, S. A general framework for weighted gene co-expression network analysis. Statistical applications in genetics and molecular biology. 4 (1), (2005).
- Edwards, J. A., et al. Compositional shifts in root-associated bacterial and archaeal microbiota track the plant life cycle in field-grown rice. PLoS Biology. 16 (2), 2003862(2018).
- Bashan, Y., De-Bashan, L. E. How the Plant Growth-Promoting Bacterium Azospirillum Promotes Plant Growth-A Critical Assessment. Advances in Agronomy. 108, 77-136 (2010).
- Lovley, D. R., et al. Geobacter: The Microbe Electric's Physiology, Ecology, and Practical Applications. Advances in Microbial Physiology. 59, 1(2011).
- Langfelder, P., Horvath, S. WGCNA: an R package for weighted correlation network analysis. BMC Bioinformatics. 9, 559(2008).
- Zhang, B., Horvath, S. A General Framework for Weighted Gene Co-expression Network Analysis. Statistical Applications in Genetics and Molecular Biology. 4 (1), 17(2005).
- Horvath, S., Dong, J. Geometric interpretation of Gene Co-expression Network Analysis. PLoS Computational Biology. 4 (8), 1000117(2008).
- Langfelder, P., Horvath, S. Eigengene networks for studying the relationships between co-expression modules. BMC Systems Biology. 1, 54(2007).
- Horvath, S., et al. Analysis of Oncogenic Signaling Networks in Glioblastoma Identifies ASPM as a Novel Molecular Target. Proceedings of the National Academy of Sciences of the United States of America. 103 (46), 17402-17407 (2006).
- Carlson, M. R., et al. and Sequence Conservation: Predictions from Modular Yeast Co-expression Networks. BMC Genomics. 7 (1), 40(2006).
- Fuller, T., et al. Weighted Gene Co-expression Network Analysis Strategies Applied to Mouse Weight. Mammalian Genome. 6 (18), 463-472 (2007).
- Yin, L., Wang, Y., Lin, Y., et al. Explorative analysis of the gene expression profile during liver regeneration of mouse: a microarray-based study[J]. Artificial Cells Nanomedicine & Biotechnology. 47 (1), 1113-1121 (2019).
- Oldham, M., Horvath, S., Geschwind, D. Conservation and Evolution of Gene Co-expression Networks in Human and Chimpanzee Brains. Proceedings of the National Academy of Sciences of the United States of America. 103 (47), 17973-17978 (2006).
- Oldham, M. C., et al. Functional organization of the transcriptome in human brain. Nature Neuroscience. 11 (11), 1271-1282 (2008).
- Keller, M. P., et al. A gene expression network model of type 2 diabetes links cell cycle regulation in islets with diabetes susceptibility. Genome Research. 18 (5), 706-716 (2008).
- Weston, D., Gunter, L., Rogers, A., Wullschleger, S. Connecting genes, coexpression modules, and molecular signatures to environmental stress phenotypes in plants. BMC Systems Biology. 2 (1), 16(2008).
- Jorda´n, F. Keystone species and food webs. Biological Sciences. 364, 1733-1741 (2009).
- Backhed, F., Ley, R. E., Sonnenburg, J. L., Peterson, D. A., Gordon, J. I. Host-Bacterial Mutualism in the Human Intestine. Science. 307, 1915-1920 (2009).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten