Method Article
Divergenza del microbiota radicale in diversi habitat in base a reti di correlazione ponderate
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
L'analisi della rete è stata applicata per valutare l'associazione di varie comunità microbiche ecologiche, come suolo, acqua e rizosfera. Qui è presentato un protocollo su come utilizzare l'algoritmo WGCNA per analizzare diverse reti di co-occorrenza che possono verificarsi nelle comunità microbiche a causa di diversi ambienti ecologici.
Abstract
Il microbioma radicale svolge un ruolo importante nella crescita delle piante e nell'adattamento ambientale. L'analisi di rete è uno strumento importante per lo studio delle comunità, che possono esplorare efficacemente la relazione di interazione o il modello di co-occorrenza di diverse specie microbiche in ambienti diversi. Lo scopo di questo manoscritto è quello di fornire dettagli su come utilizzare l'algoritmo della rete di correlazione ponderata per analizzare diverse reti di co-occorrenza che possono verificarsi nelle comunità microbiche a causa di diversi ambienti ecologici. Tutte le analisi dell'esperimento vengono eseguite nel pacchetto WGCNA. WGCNA è un pacchetto R per l'analisi ponderata della rete di correlazione. I dati sperimentali utilizzati per dimostrare questi metodi erano dati della comunità microbica dal database NCBI (National Center for Biotechnology Information) per tre nicchie del sistema radicale del riso (Oryza sativa). Abbiamo usato l'algoritmo della rete di correlazione ponderata per costruire reti di co-abbondanza di comunità microbica in ciascuna delle tre nicchie. Quindi, sono state identificate reti di co-abbondanza differenziale tra endosfera, rizopiano e suolo rizosfera. Inoltre, i generi principali in rete sono stati ottenuti dal pacchetto "WGCNA", che svolge un importante ruolo regolato nelle funzioni di rete. Questi metodi consentono ai ricercatori di analizzare la risposta della rete microbica ai disturbi ambientali e verificare diverse teorie di risposta ecologica microbica. I risultati di questi metodi mostrano che le significative reti microbiche differenziali identificate nel suolo dell'endosfera, del rizopiano e della rizosfera del riso.
Introduzione
La ricerca sul microbioma ha importanti implicazioni per la comprensione e la manipolazione dei processiecosistemici 1,2. Le popolazioni microbiche sono interconnesse da reti ecologiche interagenti, le cui caratteristiche possono influenzare la risposta dei microrganismi ai cambiamenti ambientali3,4. Inoltre, le proprietà di queste reti influenzano la stabilità delle comunità microbiche e sono strettamente associate alla funzione delsuolo 5. L'analisi ponderata della rete di correlazione genica è stata ora ampiamente applicata per la ricerca sulla relazione tra geni e comunità microbiche6. Studi precedenti si sono concentrati principalmente sulle associazioni tra reti di geni o popolazioni diverse e il mondo esterno7. Tuttavia, le differenze nelle reti di correlazione formate da popolazioni microbiche in diverse condizioni ambientali sono state scarsamente studiate. Lo scopo della ricerca presentata in questo documento è quello di fornire approfondimenti e dettagli sulla rapida implementazione dell'algoritmo WGCNA per costruire una rete di co-occorrenza di campioni di microbioma raccolti in diverse condizioni ambientali. Sulla base dei risultati dell'analisi, abbiamo valutato la composizione e le differenze della popolazione e discusso ulteriormente la relazione tra le diverse popolazioni microbiche. È stato applicato il seguente flusso di base dell'algoritmo di rete di correlazione ponderata8. In primo luogo, era necessario costruire una matrice di somiglianza calcolando il coefficiente di correlazione di Pearson tra i profili di espressione OTU (Operational Taxonomic Units). Quindi, i parametri delle funzioni di adiacenza (le funzioni di adiacenza di potenza o sigmoide) sono stati adottati con un criterio di topologia senza scala, la matrice di somiglianza è stata trasformata in una matrice di adiacenza e ogni rete di co-occorrenza corrispondeva a una matrice di adiacenza. Abbiamo utilizzato il clustering gerarchico di collegamento medio abbinato alla dissomiglianza basata su TOM per raggruppare le OTA con profili di espressione coerenti in moduli. Inoltre, abbiamo calcolato la relazione tra le statistiche conservative e i relativi moduli di analisi dei parametri, identificando infine l'OTU dell'hub nel modulo. Questi metodi sono particolarmente adatti per l'analisi delle differenze nelle strutture di rete tra varie popolazioni microbiche in condizioni ambientali divergenti. In questo manoscritto, abbiamo descritto in dettaglio il metodo di sviluppo della rete di co-espressione, l'analisi delle differenze tra i moduli e abbiamo fornito una breve panoramica dei passaggi della procedura applicata per ottenere le specie principali in diverse reti di moduli.
Protocollo
1. Download dei dati
- Scarica i dati dell'adesione PRJNA386367 dalla banca dati NCBI. Dai dati dell'adesione PRJNA386367, selezionare i dati del microbioma della rizosfera, del rizopiano e dell'endosfera dalle piante di riso coltivate per 14 settimane in un campo di riso sommerso ad Arbuckle, in California, nel 2014.
NOTA: I dati sul microbioma della rizosfera, del rizopiano e dell'endosfera sono stati presentati dalla tabella delle OTU nell'adesione PRJNA386367.
2. Determinazione ottimale del valore di potenza
NOTA: il pacchetto WGCNA contiene tutti i seguenti parametri funzionali. WGCNA è un pacchetto R per l'analisi ponderata della rete di correlazione. Le righe di comando chiave si riferiscono al Supplemento S1.
- Nell'ambiente in linguaggio R, aprire il software Rstudio e installare il pacchetto WGCNA.
- Caricare i dati e utilizzare la funzione goodSamplesGenes per verificare la correttezza dei dati. Eseguire le righe di comando:
"gsg = goodSamplesGenes(datExpr0, verbose = 3)
gsg$allOK "
Fare clic su Esegui. - Verificare la disponibilità di valori anomali e archiviare campioni che soddisfino i requisiti. Quando il risultato del controllo è TRUE, passare al passaggio successivo. Salvare il risultato.
- Utilizzare la funzione PickSoftThreshold per calcolare l'indice senza scala R2 dei due gruppi di dati con valori di potenza diversi. Eseguire la riga di comando:
"sft = pickSoftThreshold(datExpr0, powerVector = potenze, verbose = 5)"
Fare clic su Esegui. - Visualizzare i risultati (Figura 1). Eseguire la riga di comando:
"plot(sft$fitIndices[,1], -sign(sft$fitIndices[,3])*sft$fitIndices[,2],
xlab="Soft Threshold (power)",ylab="Scale Free Topology Model Fit,signed R^2",type="n",
main = paste("indipendenza ES_Scale"));
text(sft$fitIndices[,1], -sign(sft$fitIndices[,3])*sft$fitIndices[,2],
labels=powers,cex=cex1,col="red");
abline(h=0.9;col="rosso")
plot(sft$fitIndices[,1], sft$fitIndices[,5],
xlab="Soft Threshold (power)",ylab="Mean Connectivity", type="n",
main = incolla("connettività ES_Mean"))
text(sft$fitIndices[,1], sft$fitIndices[,5], labels=powers, cex=cex1,col="red")"
Fare clic su Esegui.
NOTA: la premessa dell'algoritmo di rete di correlazione ponderata è che la struttura di rete di coespressione stabilita è conforme agli standard del criterio della topologia senza scala, aumentandone la robustezza. Un indice senza scalabilità più vicino a 1 indica una struttura di rete più vicina alla rete senza scalabilità. - Selezionare il valore di potenza quando l'indice senza scala R2 al quadrato è maggiore di 0,9 e procedere alla fase successiva dell'analisi.
NOTA: quando l'indice senza scalabilità è vicino a 1, la struttura di rete è più vicina alla rete senza scalabilità. Quando si analizzano due o più reti, è necessario scegliere di rendere ogni rete vicina al valore di potenza della rete senza scala per soddisfare la comparabilità tra le reti co-espresse.
3. Costruzione di una rete di co-espressione e identificazione del modulo
NOTA: in base al valore di potenza calcolato sopra, viene costruita la rete di co-occorrenza. Le righe di comando chiave si riferiscono al Supplemento S2.
- Utilizzare la funzione di adiacenza nel pacchetto WGCNA per aggiungere parametri firmati per la costruzione di una rete di co-occorrenza simbolica. Eseguire la riga di comando:
"adiacenza = adiacenza(datExpr0, potenza = softPower)"
Fare clic su Esegui. - Applicare la funzione di somiglianza TOM per sviluppare una rete di sovrapposizione topologica e calcolare la rete di dissomiglianza. Eseguire la riga di comando:
"TOM = TOMsimilarity(adijacency);
dissTOM = 1-TOM"
Fare clic su Esegui.
Nota : il parametro signed è stato aggiunto per impostare il tipo di rete di sovrapposizione della topologia. - Utilizzare la funzione hclust per selezionare il metodo di clustering gerarchico di collegamento medio per il clustering gerarchico. Eseguire la riga di comando:
"geneTree = hclust(as.dist(dissTOM), metodo = "media");"
Fare clic su Esegui. - Utilizzate la funzione cutreeDynamic per eseguire il taglio dinamico dei rami e impostate il parametro minClusterSize su 30. Ottenere il risultato del riconoscimento del modulo. Eseguire la riga di comando:
"dynamicMods = cutreeDynamic(dendro = geneTree, distM = dissTOM, deepSplit = 2, pamRespectsDendro = FALSE, minClusterSize = minModuleSize);"
Fare clic su Esegui.
NOTA: la dimensione minima del modulo non può essere inferiore a 30. - Calcolare l'auto del modulo di ciascun modulo OTA mediante la funzione moduleEigenes. Eseguire la riga di comando:
"MEList = moduleEigengenes(datExpr0, colors = dynamicColors)
MEs = MEList$eigenes"
Fare clic su Esegui.
NOTA: l'auto del modulo rappresentava il livello di espressione OTU complessivo nel modulo. Non era una OTU specifica, ma il primo componente principale di ogni cluster ottenuto dalla scomposizione del valore di rete singolare. - Eseguire la funzione cluster in base al coefficiente di correlazione del modulo eigen. Utilizzare la funzione mergeCloseModules per unire i moduli con un valore inferiore a 0,25. Eseguire la riga di comando:
"merge = mergeCloseModules(datExpr0, dynamicColors, cutHeight = MEDissThres, verbose = 3)"
Fare clic su Esegui. - Infine, utilizzare la funzione plotDendroAndColors per la visualizzazione per ottenere il diagramma di visualizzazione dell'assegnazione del modulo di ogni rete di coespressione (Figura 2). Utilizzare la funzione table per estrarre l'attribuzione del modulo corrispondente di ogni OTin la tabella di assegnazione del modulo. Eseguire la riga di comando:
"plotDendroAndColors(geneTree, mergedColors, "Merged dynamic",dendroLabels = FALSE,
hang = 0,03,addGuide = TRUE, guideHang = 0,05,
main = "ES_Gene dendrogramma e colori del modulo")"
Fare clic su Esegui.
NOTA: nel diagramma di assegnazione dei moduli della rete co-espressiva, colori diversi rappresentano moduli diversi e il grigio rappresenta le OTA che non possono essere classificate in alcun modulo. Un numero maggiore di OTA nel modulo grigio indica che la qualità di pre-elaborazione in fase iniziale della matrice di espressione è scarsa.
4. Confronto dei moduli
NOTA: Questo metodo può essere utilizzato per confrontare i moduli di rete di due comunità microbiche ecologiche. In questo articolo, confronta le differenze dei moduli di rete microbica tra endosfera e rizopiano, endosfera e rizosfera, rizosfera e rizopiano.
- Test di conservazione
- Caricare i parametri e i risultati dei due set di dati salvati nei passaggi precedenti.
- Impostare il risultato dell'assegnazione del modulo di rete di un gruppo di dati microbici come gruppo di riferimento, mentre l'altro gruppo come gruppo di test.
- Utilizzare la funzione modulePreservation per calcolare i valori dei parametri statistici di conservativezza Z_summary e medianRank. Eseguire la riga di comando:
"system.time({mp=modulePreservation(multiExpr,
multiColor,referenceNetworks=1,
nPermutation=100, randomSeed=1,quickCor=0,verbose=3)})"
Fare clic su Esegui.
NOTA: questo risultato può quantificare la conservatività tra i moduli. Z_summary>10 indica che due moduli sono altamente conservati, mentre Z_summary<2 indica moduli non conservati. medianRank esprime la conservazione relativa del modulo valutato per classifica. Valori medianRank più elevati indicano moduli non conservati. (Le righe di comando dei tasti si riferiscono al supplemento S3.) - Utilizzare la funzione plot per visualizzare i risultati (Figura 3). Ottenere i parametri Z_summary e medianRank (Tabella 1).
NOTA: I moduli di rete che soddisfano sia il valore Z_summary inferiore a 2 che il valore Rank mediano in alto, è il modulo più altamente non conservato nelle due comunità microbiche ecologiche. - Sulla base dei risultati dei suddetti due parametri statistici per identificare il modulo con il modulo più altamente non conservato delle due reti.
- Analisi di correlazione dell'appartenenza al modulo
- Impostare i risultati di assegnazione del modulo delle due reti sono stati impostati rispettivamente come riferimento e gruppo di test.
NOTA: le impostazioni devono essere uguali a test di conservazione. - Utilizzare la funzione corPvalueStudent per estrarre il valore kME (appartenenza al modulo) di ogni OTU in diversi moduli candidati.
Eseguire la riga di comando:
"Pvalue = as.data.frame(corPvalueStudent(as.matrix
(ModuleMembership), Samples))"
Fare clic su Esegui.
NOTA: kME sta per il grado di appartenenza al modulo. ME sta per modulo eigen, che rappresenta il livello complessivo di espressione OTU nel modulo. kME è il coefficiente di correlazione tra ogni OTU e il ME. Quantificare l'importanza di OTU nella rete in modo dal valore kME di OTU. (Le righe di comando dei tasti si riferiscono al supplemento S4.) - Quindi, utilizzare la funzione verboseScatterplot per calcolare il coefficiente di correlazione del valore kME delle OTA corrispondenti nelle due reti e disegnare il diagramma di analisi di correlazione (Figura 4).
Eseguire la riga di comando:
"verboseScatterplot(abs(TModuleMembership
[TmoduleGenes, Tcolumn]),
abs(NModuleMembership[NmoduleGenes, Ncolumn]),
xlab = paste("kME in", "ES"),
ylab = paste("kME in", "RP"),
main = pasta("lightyellow"),
cex.main = 1.7, cex.lab = 1.6, cex.axis = 1.6, col = modulecolor)"
Fare clic su Esegui. - Selezionare il modulo con il più piccolo coefficiente di correlazione del valore kME dell'OTU delle due reti. Considera questo modulo per avere la più grande differenza delle due reti.
- Impostare i risultati di assegnazione del modulo delle due reti sono stati impostati rispettivamente come riferimento e gruppo di test.
5. Analisi del modulo di rete differenziale microbica
- Ottenere i dati dei phyla batterici dominanti attraverso l'analisi statistica del set di sequenze OTU del modulo con la più grande differenza.
NOTA: l'insieme di sequenze OTU del modulo con la differenza maggiore è sommato dalla tassonomia dei phyla. I batteri dominanti phyla rappresentavano oltre il 10%. - Quindi, utilizzare la funzione exportNetworkToCytoscape per ottenere il file contenente le informazioni sulla relazione di interazione dell'OTU nel modulo differenziale più grande.
Eseguire la riga di comando:
"cyt = exportNetworkToCytoscape(modTOM,
edgeFile = paste("NEW-ES_CytoscapeInput-edges-", modules , ".txt", sep=""),
nodeFile = paste("NEW-ES_CytoscapeInput-nodes-", modules, ".txt", sep=""),
ponderato = TRUE,soglia = 0,5, nodeNames = modProbes,
altNodeNames = modGenes, nodeAttr = moduleColors[inModule])"
Fare clic su Esegui. - Importare il file in Cytoscape. Impostare la soglia su 0,5 e regolare altri parametri in base alle esigenze.
- Costruire una rete di co-occorrenza di microrganismi differenziali (Figura 5).
- Ottenute le informazioni del genere core che ha il ruolo normativo più importante nella rete.
NOTA: In base al valore kME di OUT, è possibile definire il genere principale. - Infine, sono state valutate le funzioni del genere principale ed è stata analizzata la sua influenza sull'intera rete di differenze.
Risultati
I risultati rappresentativi in questo articolo sono stati scaricati dai dati del microbioma della radice di riso California Abaker del 2014 nel database NCBI (PRJNA386367)9. I dati includono i campioni di microbioma rizosfera, rizoplano ed endosfera da piante di riso coltivate per 14 settimane in un campo di riso sommerso. Abbiamo utilizzato l'algoritmo WGCNA per selezionare il valore di potenza che soddisfaceva le tre reti che erano vicine alla rete senza scala (Figura 1) e sviluppato tre reti di co-espressione (Figura 2). Nella rete di co-espressione microbica del suolo dell'endosfera, del rizopiano e della rizosfera, sono stati identificati rispettivamente 23, 22 e 21 moduli. Questi risultati indicano che il numero di reti di interazione microbica nelle tre nicchie era fondamentalmente uguale.
Abbiamo ulteriormente confrontato le differenze nei moduli della rete microbica all'interno dell'endosfera, del rizopiano e del suolo della rizosfera. Sono stati ottenuti i seguenti risultati dei test di conservazione dei moduli dei tre gruppi di nicchie. Tre moduli estremamente non conservati esistevano tra il suolo della rizosfera e il rizopiano (Figura 3a, Tabella 1). Inoltre, nove moduli estremamente non conservati erano presenti tra il suolo e l'endosfera della rizosfera (Figura 3b, Tabella 2) e sei moduli estremamente non conservati tra il rizopiano e l'endosfera (Figura 3c, Tabella 3). Inoltre, sono stati trovati moduli estremamente non conservati tra le tre nicchie, indicando la presenza di grandi differenze nella composizione dei microrganismi tra le tre nicchie. I risultati dell'analisi di correlazione dell'appartenenza al modulo dei moduli non conservativi ottenuti sono illustrati nella Figura 4. Dalla figura, un modulo significativamente diverso con la minima correlazione tra ciascuna delle due nicchie è visibile tra le tre nicchie, che rappresenta la differenza più significativa tra loro.
Nella rete di differenza rizosfera-rizopiano (Figura 5a), il phylum dominante era Proteobacteria (72,97%). Nella rete di differenza rizosfera-endosfera (Figura 5b), i phyla dominanti erano Proteobacteria (66,36%), Actinobacteria (10,1%) e Bacteroidetes (10,9%). Nella rete di differenza rizoplano-endosfera (Figura 5c), i phyla dominanti erano Proteobacteria (41,41%), Bacteroidetes (10,10%), Firmicutes (12,12%) e Verrucomicrobia.
Tre generi principali (Figura 5a), tra cui Rhodobacter e Novosphingobium, sei generi principali (Figura 5b), tra cui Blvii28 e Dechloromonas, e cinque generi principali (Figura 5c), tra cui Cellvibrio e Geobacter, hanno esercitato importanti funzioni regolatorie nelle tre reti di co-occorrenza differenziale. Tutti i generi principali, ad eccezione di Dechloromonas,hanno avuto influenza su una sola rete, indicando la disponibilità di notevoli differenze nell'abbondanza relativa di popolazioni microbiche e specie tra le tre nicchie di radici di riso, che hanno influenzato in modo critico l'abbondanza e la diversità delle comunità microbiche di radice esistenti.
Il genere principale Azospirillum,presente nella rete di differenza rizosfera-endosfera del riso, ha partecipato alla fissazione dell'azoto e ha promosso la crescita delle piante10. Inoltre, il genere Geobacter, che è stato significativamente arricchito nella rete di differenza rizoplano-endosfera, può essere il principale fattore che induce la riduzione degli ossidi di Fe e Mn insolubili in molti terreni e sedimenti11. Questi microrganismi interagiscono con una serie di comunità microbiche nelle nicchie delle radici e partecipano attivamente alla regolazione delle reti microbiche, che potrebbero essere di fondamentale importanza per la crescita e lo sviluppo delle radici di riso.
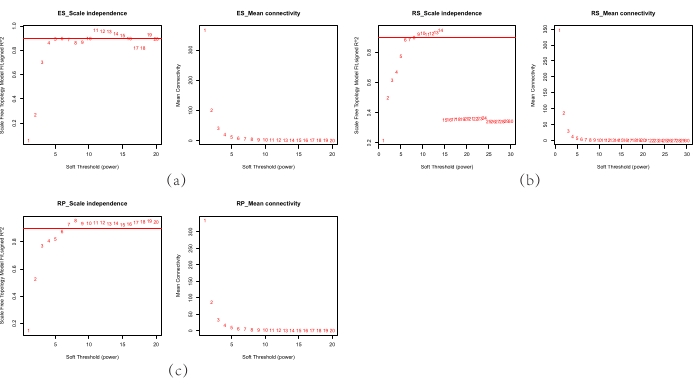
Figura 1. Valutazione di powerβ nei set di dati. a) valutazione dei β di potenza nell'set di dati ES; b) Valutazione dei β di potenza nel set di dati RS; (c) Valutazione della potenza β nel set di dati RP, distribuzione dell'indice privo di scala R2 (a sinistra), distribuzione della connettività media (a destra) lungo diversi indici di soft power. Il valore della migliore potenza è stato raggiunto quando R2 tendeva alla saturazione e non era inferiore a 0,8. Le tre reti di co-espressione dovevano essere impostate sullo stesso valore di potenza per garantirne la comparabilità. (RS: suolo rizosfera, RP: rizopiano ed ES: endosfera). Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 2. Dendrogramma OTU ottenuto mediante clustering gerarchico di collegamento medio. a)dendrogramma OTU dalla rete ES; b) dendrogramma OTU dalla rete RS; c) Dendrogramma OTU dalla rete RP. La riga di colore sotto il dendrogramma indica l'assegnazione del modulo determinata dall'algoritmo Dynamic Tree Cut. (RS: suolo rizosfera, RP: rizopiano ed ES: endosfera) Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 3. Risultati del test di conservazione. a) i risultati dell'analisi si basano sull'allocazione del modulo di rete di coespressione RS come gruppo di riferimento e sui risultati dell'allocazione del modulo di rete di coespressione RP come gruppo di prova; b) i risultati dell'analisi si basano sull'assegnazione del modulo di rete di coespressione ES come gruppo di riferimento e sui risultati dell'allocazione del modulo di rete di coespressione RS come gruppo di prova; c) I risultati dell'analisi si basano sull'allocazione del modulo di rete di coespressione ES come gruppo di riferimento e sui risultati dell'allocazione del modulo di rete di coespressione RP come gruppo di prova. Z_summary > 10 indica che due moduli sono altamente conservati, mentre Z_summary < 2 indica moduli non conservati. medianRank esprime la conservazione relativa del modulo valutato per classifica. Valori medianRank più elevati indicano moduli non conservati. (RS: suolo rizosfera, RP: rizopiano ed ES: endosfera) Fare clic qui per visualizzare una versione più grande di questa figura.
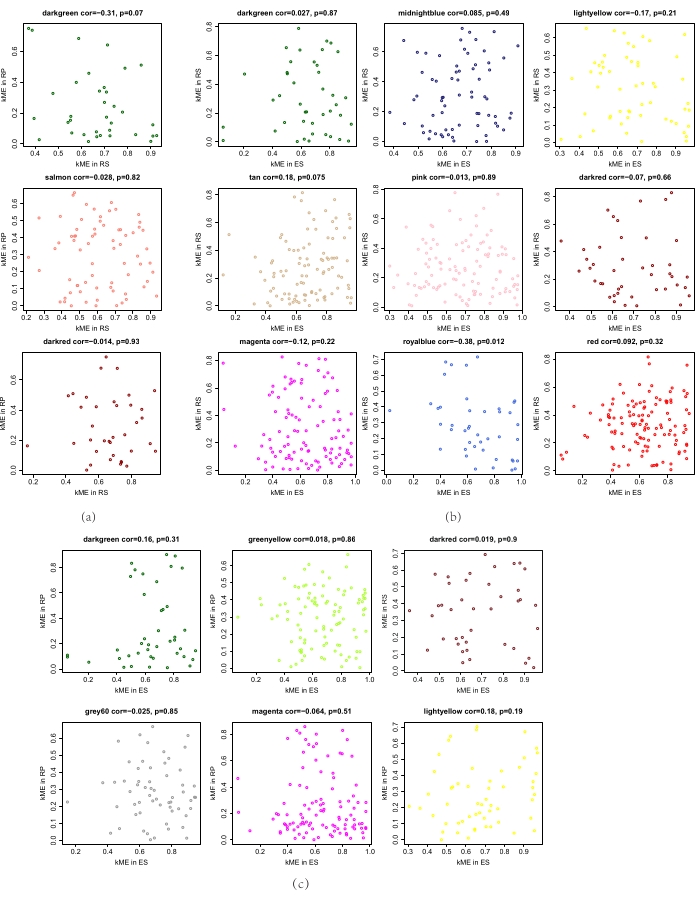
Figura 4. Analisi di correlazione delle appartenenze al modulo. a) correlazione del modulo del valore kME tra la rete RS e RP; b) correlazione del modulo del valore kME tra la rete RS e RP; (c) Correlazione del modulo del valore kME tra la rete RP ed ES (RS: suolo rizosfera, RP: rizopiano, ES: endosfera e KME: appartenenza al modulo). Fare clic qui per visualizzare una versione più grande di questa figura.
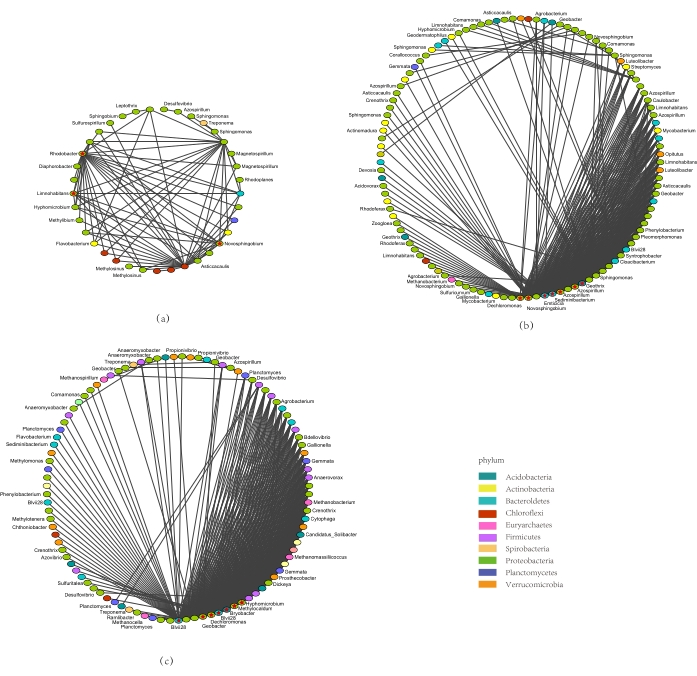
Figura 5. Rete di co-occorrenza di popolazione microbica differenziale nella radice del riso. a)rete di co-occorrenza di popolazioni microbiche differenziali in RS-RP; b) rete di co-occorrenza di popolazioni microbiche differenziali in ES-RS; c) Rete di co-occorrenza di popolazioni microbiche differenziali in ES-RP. L'analisi è stata eseguita utilizzando il software Cytoscape. Colori diversi rappresentano porte diverse nella figura. (RS: Suolo della rizosfera, RP: rizopiano ed ES: endosfera). Fare clic qui per visualizzare una versione più grande di questa figura.
| modulo | medianRank | Zsummary · | modulo | medianRank | Zsummary · |
| greenyellow | 2 | 14 | giallo | 13 | 5.2 |
| rosa | 2 | 17 | grigio60 | 14 | 3.1 |
| midnightblue | 3 | 10 | royalblue | 15 | 2.1 |
| marrone | 4 | 18 | nero | 16 | 2.7 |
| lightcyan | 6 | 8.5 | abbronzatura | 17 | 3.2 |
| porpora | 7 | 10 | salmone | 18 | 1.3 |
| blu | 7 | 22 | magenta | 18 | 2.2 |
| verde | 8 | 12 | darkred | 20 | -0.24 |
| ciano | 10 | 4.8 | oro | 20 | 14 |
| verde chiaro | 11 | 5.1 | verde scuro | 22 | -1.1 |
| lightyellow | 12 | 5.3 | grigio | 22 | 0.21 |
| rosso | 12 | 6.1 |
Tabella 1. Risultato di Zsummary e medianRank tra il suolo della rizosfera e il rizopiano.
| modulo | medianRank | Zsummary · | modulo | medianRank | Zsummary · |
| salmone | 1 | 19 | lightcyan | 13 | 1.1 |
| nero | 3 | 5.3 | rosso | 15 | 0.95 |
| giallo | 4 | 5.8 | midnightblue | 15 | -0.0016 |
| verde chiaro | 5 | 0.27 | royalblue | 16 | 0.83 |
| greenyellow | 7 | 3 | magenta | 16 | 0.52 |
| darkturquoise | 7 | 1.2 | verde scuro | 17 | 0.16 |
| grigio60 | 9 | 1.1 | abbronzatura | 18 | 0.64 |
| blu | 10 | 3.9 | lightyellow | 19 | 0.52 |
| porpora | 10 | 2.3 | darkred | 19 | -0.18 |
| marrone | 12 | 2.3 | rosa | 19 | -0.71 |
| ciano | 12 | 0.78 | oro | 21 | 11 |
| verde | 13 | 1.7 |
Tabella 2. Risultato di Zsummary e medianRank tra il suolo della rizosfera e l'endosfera.
| modulo | medianRank | Zsummary · | modulo | medianRank | Zsummary · |
| nero | 1 | 15 | darkturquoise | 13 | 1.7 |
| salmone | 2 | 27 | midnightblue | 13 | 1.6 |
| giallo | 3 | 13 | verde chiaro | 13 | 0.64 |
| ciano | 4 | 5.4 | verde scuro | 14 | 1.5 |
| blu | 8 | 3.9 | darkred | 16 | 1.5 |
| lightcyan | 9 | 2.6 | porpora | 17 | 2.3 |
| rosa | 10 | 3.6 | greenyellow | 18 | 0.8 |
| royalblue | 10 | 1.5 | lightyellow | 18 | 0.42 |
| marrone | 12 | 2.9 | magenta | 19 | 0.2 |
| verde | 12 | 1.9 | oro | 21 | 18 |
| rosso | 12 | 1.9 | grigio60 | 21 | -0.21 |
| abbronzatura | 13 | 2.5 |
Tabella 3. Risultato di Zsummary e medianRank tra il rizopiano e l'endosfera.
Supplemento S1: Fare clic qui per scaricare questo file.
Supplemento S2: Fare clic qui per scaricare questo file.
Supplemento S3: Fare clic qui per scaricare questo file.
Supplemento S4: Fare clic qui per scaricare questo file.
Discussione
Le reti di correlazione sono state sempre più utilizzate nelle applicazioni bioinformatiche. WGCNA è un metodo di biologia dei sistemi per l'analisi descrittiva delle relazioni tra vari elementi di un sistema biologico12. Il pacchetto software R è stato utilizzato in lavori precedenti su WGCNA13,14,15. Il pacchetto include funzioni per la costruzione di reti, il rilevamento di moduli, calcoli di proprietà topologiche, simulazione dei dati, visualizzazione e capacità di interfacciamento con software esterno. WGCNA è stato ampiamente impiegato per analizzare i dati di espressione genica dal cancro al cervello16,ciclo cellulare del lievito17,genetica del topo18,19,tessuto cerebrale dei primati20,21,diabete22e piante23. Utilizzare l'analisi ponderata della rete di correlazione genica per costruire la rete deve includere almeno 8 campioni. In questo articolo, ci siamo concentrati sulle reti di co-espressione genica che descrivono le interazioni tra popolazioni microbiche in ambienti diversi. Abbiamo ottenuto reti differenziali tra popolazioni microbiche in ambienti divergenti e identificato le specie chiave in ciascuna rete. L'idea che le specie chiave siano importanti per la comunità è stata ampiamente utilizzata nella ricerca sul food-web24. Alcune delle specie in una comunità microbica complessa possono essere essenziali per mantenere la stabilità e la funzionalità della comunità, come Bacteroides nella flora intestinale25. L'analisi delle comunità microbiche può essere sostanzialmente semplificata prendendo di mira specie specifiche di potenziale importanza.
I nostri risultati rappresentativi evidenziano le differenze nelle comunità microbiche, che possono essere identificate utilizzando il metodo sopra descritto. Qui, i microrganismi in diverse nicchie del sistema delle radici di riso sono stati sottoposti a WGCNA. Il differenziale tra le tre nicchie è stato identificato utilizzando analisi conservative e di appartenenza al modulo. Abbiamo determinato le specie chiave nei moduli di differenza e ottenuto informazioni sulle differenze nella composizione delle comunità microbiche nelle tre nicchie. Nel frattempo, la rete di co-occorrenza ha rivelato la presenza di un'interazione significativa tra i microrganismi che cambiano nelle radici del riso. I nostri risultati forniscono prove dirette del significato e della fattibilità del WGCNA nella valutazione delle differenze della comunità microbica in vari ambienti.
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Lo sviluppo di questo manoscritto è stato supportato da fondi della National Natural Science Foundation of China-Guizhou Provincial People's Government Karst Science Research Center Project (U1812401), del Doctoral Research Project della Guizhou Normal University (GZNUD[2017]1), del Science and Technology Support Project della provincia di Guizhou (QKHZC[2021]YB459) e del Science and Technology Project of the Guiyang([2019]2-8).
Gli autori desiderano ringraziare Edwards J.A et al per aver fornito dati sul microbioma del riso in database pubblici e il supporto di TopEdit (www.topeditsci.com) per la sua assistenza linguistica durante la preparazione di questo manoscritto.
Materiali
| Name | Company | Catalog Number | Comments |
| R | The University of Auckland | version 4.0.2 | R is a free software environment for statistical computing and graphics. It compiles and runs on a wide variety of UNIX platforms, Windows and MacOS. |
| RStdio | JJ Allaire | version 1.4.1103 | The RStudio IDE is a set of integrated tools designed to help you be more productive with R and Python. |
| Cytoscape | version 3.7.1 | Cytoscape is an open source software platform for visualizing complex networks and integrating these with any type of attribute data. | |
| NCBI database | The National Center for Biotechnology Information advances science and health by providing access to biomedical and genomic information. |
Riferimenti
- Philippot, L., Raaijmakers, J. M., Lemanceau, P., vander Putten, W. H. Going back to the roots: the microbial ecology of the rhizosphere. Nature Reviews Microbiology. 11, 789-799 (2013).
- Fierer, N. Embracing the unknown: disentangling the complexities of the soil microbiome. Nature Review Microbiology. 15 (10), 579-590 (2017).
- Jin, J., Wang, G. H., Liu, X. B., Liu, J. D., Chen, X. L., Herbert, S. J. Temporal and spatial dynamics of bacterial community in the rhizosphere of soybean genotypes grown in a black soil. Pedosphere. 19 (6), 808-816 (2009).
- Ma, B., et al. Genetic correlation network prediction of forest soil microbial functional organization. ISME J. 12 (10), 2492-2505 (2018).
- de Vries, F. T., et al. Soil bacterial networks are less stable under drought than fungal networks. Nature Communications. 9 (1), 3033 (2018).
- Colin, C., et al. Correlating transcriptional networks to breast cancer survival: a large-scale coexpression analysis. Carcinogenesis. (10), 2300-2308 (2013).
- Ma, B., Zhao, K., Lv, X., et al. Genetic correlation network prediction of forest soil microbial functional organization. ISME J. 12, 2492-2505 (2018).
- Zhang, B., Horvath, S. A general framework for weighted gene co-expression network analysis. Statistical applications in genetics and molecular biology. 4 (1), (2005).
- Edwards, J. A., et al. Compositional shifts in root-associated bacterial and archaeal microbiota track the plant life cycle in field-grown rice. PLoS Biology. 16 (2), 2003862 (2018).
- Bashan, Y., De-Bashan, L. E. How the Plant Growth-Promoting Bacterium Azospirillum Promotes Plant Growth-A Critical Assessment. Advances in Agronomy. 108, 77-136 (2010).
- Lovley, D. R., et al. Geobacter: The Microbe Electric's Physiology, Ecology, and Practical Applications. Advances in Microbial Physiology. 59, 1 (2011).
- Langfelder, P., Horvath, S. WGCNA: an R package for weighted correlation network analysis. BMC Bioinformatics. 9, 559 (2008).
- Zhang, B., Horvath, S. A General Framework for Weighted Gene Co-expression Network Analysis. Statistical Applications in Genetics and Molecular Biology. 4 (1), 17 (2005).
- Horvath, S., Dong, J. Geometric interpretation of Gene Co-expression Network Analysis. PLoS Computational Biology. 4 (8), 1000117 (2008).
- Langfelder, P., Horvath, S. Eigengene networks for studying the relationships between co-expression modules. BMC Systems Biology. 1, 54 (2007).
- Horvath, S., et al. Analysis of Oncogenic Signaling Networks in Glioblastoma Identifies ASPM as a Novel Molecular Target. Proceedings of the National Academy of Sciences of the United States of America. 103 (46), 17402-17407 (2006).
- Carlson, M. R., et al. and Sequence Conservation: Predictions from Modular Yeast Co-expression Networks. BMC Genomics. 7 (1), 40 (2006).
- Fuller, T., et al. Weighted Gene Co-expression Network Analysis Strategies Applied to Mouse Weight. Mammalian Genome. 6 (18), 463-472 (2007).
- Yin, L., Wang, Y., Lin, Y., et al. Explorative analysis of the gene expression profile during liver regeneration of mouse: a microarray-based study[J]. Artificial Cells Nanomedicine & Biotechnology. 47 (1), 1113-1121 (2019).
- Oldham, M., Horvath, S., Geschwind, D. Conservation and Evolution of Gene Co-expression Networks in Human and Chimpanzee Brains. Proceedings of the National Academy of Sciences of the United States of America. 103 (47), 17973-17978 (2006).
- Oldham, M. C., et al. Functional organization of the transcriptome in human brain. Nature Neuroscience. 11 (11), 1271-1282 (2008).
- Keller, M. P., et al. A gene expression network model of type 2 diabetes links cell cycle regulation in islets with diabetes susceptibility. Genome Research. 18 (5), 706-716 (2008).
- Weston, D., Gunter, L., Rogers, A., Wullschleger, S. Connecting genes, coexpression modules, and molecular signatures to environmental stress phenotypes in plants. BMC Systems Biology. 2 (1), 16 (2008).
- Jorda´n, F. Keystone species and food webs. Biological Sciences. 364, 1733-1741 (2009).
- Backhed, F., Ley, R. E., Sonnenburg, J. L., Peterson, D. A., Gordon, J. I. Host-Bacterial Mutualism in the Human Intestine. Science. 307, 1915-1920 (2009).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon