Method Article
基于加权相关网络的不同栖息地根微生物群的差异
摘要
应用网络分析来评估各种生态微生物群落(如土壤、水和岩石圈)的关联。这里介绍的是如何使用WGCNA算法来分析由于不同的生态环境而可能发生在微生物群落中的不同共生网络的协议。
摘要
根微生物群在植物生长和环境适应中起着重要作用。网络分析是研究社区的重要工具,可以有效探索不同环境中不同微生物物种的相互作用关系或共发生模式。本文稿的目的是提供如何使用加权相关网络算法分析因不同生态环境而可能发生在微生物群落中的不同共生网络的详细信息。实验的所有分析均在 WGCNA 包中执行。WGCNA 是用于加权相关网络分析的 R 包。用于证明这些方法的实验数据是来自NCBI(国家生物技术信息中心)数据库的微生物群落数据,用于水稻(Oryza水稻)根系的三个利基。我们使用加权相关网络算法在三个利基中每个利基中构建微生物群落的共同丰度网络。然后,确定了内层、地层和日光圈土壤之间的微分共丰度网络。此外,网络的核心属是通过"WGCNA"一揽子计划获得的,它在网络功能中起着重要的调节作用。这些方法使研究人员能够分析微生物网络对环境扰动的反应,并验证不同的微生物生态反应理论。这些方法的结果表明,水稻内层、地层和稻谷圈土壤中所识别的显著微分微生物网络。
引言
微生物群研究对理解和操纵生态系统过程具有重要的意义。微生物种群通过相互作用的生态网络相互连接,其特征会影响微生物对环境变化的反应。此外,这些网络的特性影响微生物群落的稳定,并与土壤功能5密切相关。加权基因相关性网络分析现已被广泛应用于基因与微生物群落关系的研究。先前的研究主要集中于不同基因或种群网络与外界之间的关联。然而,微生物群在不同环境条件下形成的相关网络差异几乎没有被研究过。本文所介绍的研究旨在提供有关快速实施WGCNA算法的见解和细节,以构建在不同环境条件下收集的微生物群样本的共发生网络。根据分析结果,我们评估了种群的组成和差异,并进一步讨论了不同微生物种群之间的关系。应用了加权相关网络算法8的以下基本流。首先,需要通过计算运营分类单位 (OTU) 表达配置文件之间的 Pearson 相关系数来构建相似性矩阵。然后,采用了具有无尺度拓扑标准的邻接函数参数(功率或 sigmoid 邻接函数),将相似性矩阵转换为邻接矩阵,并且每个共发生网络对应于邻接矩阵。我们使用平均链接分层聚类,以及基于 TOM 的差异,将具有连贯表达配置文件的 OTUs 组合成模块。此外,我们计算了保守统计与相关参数分析模块之间的关系,最终确定了模块中的集线器 OTU。这些方法特别适合分析不同环境条件下不同微生物种群之间网络结构的差异。在这份手稿中,我们详细描述了共同表达网络开发的方法,分析了模块之间的差异,并简要概述了在不同模块网络中获取核心物种的程序中应用的步骤。
研究方案
1. 数据下载
- 从 NCBI 数据库下载加入 PRJNA386367 的数据。从加入PRJNA386367的数据中,从2014年加利福尼亚州阿巴克尔的水下稻田中生长了14周的水稻植物中选择日光圈、日生行星和内层微生物群数据。
注:在加入PRJNA386367的O图表中,介绍了日星圈、日光和内层微生物群数据。
2. 最佳功率值确定
注:WGCNA 软件包包含以下所有功能参数。WGCNA 是用于加权相关网络分析的 R 包。关键命令线指补充S1。
- 在 R 语言环境中,打开 Rstudio 软件并安装 WGCNA 软件包。
- 加载数据并使用良好的采样生成功能来检查数据的正确性。执行命令行:
"gsg = 好样子生成 (datExpr0, 冗长 = 3)
格斯格 $阿洛克 "
单击 "运行"。 - 检查符合要求的离群值和存储样品。当检查结果为真实时,请继续下一步。保存结果。
- 使用 PickSoftThreshold 功能计算不同功率值下两组数据的无比例索引 R2。 执行命令线:
"sft = 选择软胸 (datExpr0, 电源矢量 + 权力, 冗长 = 5)"
单击 "运行"。 - 可视化结果(图1)。执行命令线:
"图(sft$拟合印度[,1],-符号(sft$拟合印度[,3])*sft$拟合印度[,2],
xlab= "软阈值 (功率)", ylab = "扩展免费拓扑模型适合, 签名 R+2", 类型 =n,
主 = 粘贴("ES_Scale独立"):
文本 (sft$拟合印度 [, 1], - 签名 (sft$拟合印度 [, 3]) * sft $适合印度 [, 2],
标签=权力,cex=cex1,科尔="红色");
阿伯林 (h=0.9,可可"红色")
情节 (sft$拟合印度 [, 1], sft $适合印度 [, 5],
xlab= "软阈值 (电源)", ylab = "平均连接", 类型 =n",
主 + 粘贴("ES_Mean连接")
文本 (sft$拟合印度 [, 1], sft$适合印度 [, 5], 标签 = 权力, cex = cex1, 科尔 [红色])
单击 "运行"。
注:加权相关网络算法的前提是,已建立的共同表达网络结构符合无尺度拓扑标准的标准,增强其稳健性。接近 1 的无比例指数表示更接近无规模网络的网络结构。 - 当无比例指数 R2 大于 0.9 时选择功率值,然后继续下一步分析。
注:当无规模指数接近1时,网络结构更接近无规模网络。在分析两个或两个以上的网络时,必须选择使每个网络接近无规模网络的功率值,以满足共同表达网络之间的可比性。
3. 构建共同表达网络和模块识别
注:根据上述计算的功率值,构建了共发生网络。关键命令线指补充S2。
- 使用 WGCNA 包中的邻存函数添加用于构建象征性共发生网络的签名参数。执行命令线:
"邻从 = 邻从 (datExpr0, 电源 = 软实力)"
单击 "运行"。 - 应用 TOM 相似性函数来开发拓扑重叠网络并计算异构网络。执行命令线:
"TOM = 托姆相似性(辅助性);
迪斯托姆 = 1 - 汤姆"
单击 "运行"。
注:已添加签名参数以设置拓扑重叠网络类型。 - 使用集束函数选择分级聚类的平均链接分层聚类方法。执行命令线:
"基因树 = hclust (阿斯. dist) 、 方法 = "平均");
单击 "运行"。 - 使用 cutree 动态功能执行动态分支切割,并将最小光谱参数设置为 30。获取模块识别结果。执行命令线:
"动态模式 = 树动力学 (登德罗 + 基因树, distm = 迪斯托姆, 深溅 = 2, 帕姆尊重登德罗 + 假, 小光谱大小 = 最小模块化);"
单击 "运行"。
注:最小模块大小不能小于 30。 - 通过模块"基因"功能计算每个 OTU 模块的模块 eigen。执行命令线:
"ME 列表 = 模块基因(datExpr0,颜色 = 动态颜色)
梅斯 = 梅利斯 $ 基因"
单击 "运行"。
注:模块 eigen 表示模块中的整体 OTU 表达水平。它不是一个特定的 OTU,而是通过单一网络值分解获得的每个聚类的第一个主要组件。 - 根据模块 eigen 的相关系数执行聚类函数。使用合并的关闭模块功能合并值低于 0.25 的模块。执行命令线:
"合并 = 合并关闭模式 (datExpr0, 动态颜色, 切高 = 梅迪斯特雷斯, 冗长 = 3)"
单击 "运行"。 - 最后,使用图文并存的可视化功能获取每个共同表达网络的模块分配显示图(图 2)。使用表功能提取每个 OTin 模块分配表对应的模块归因。执行命令线:
"绘图登德罗和彩色 (基因树, 合并颜色, "合并动态", 登德罗拉贝尔斯 = 错误,
挂 = 0.03, 添加指南 = 真, 指南行 = 0.05,
主 = "ES_Gene登德罗格拉姆和模块颜色")"
单击 "运行"。
注:在共同表达网络的模块分配图中,不同的颜色表示不同的模块,灰色表示不能归入任何模块的 OTUs。灰色模块中的大量 OTUs 表明表达矩阵的早期预处理质量较差。
4. 模块比较
注:此方法可用于比较两个生态微生物群落的网络模块。本文比较了内层和日光、内层和日光圈、日光圈和日光行星之间微生物网络模块的差异。
- 保存测试
- 加载前几步中保存的两个数据集的参数和结果。
- 将一组微生物数据的网络模块分配结果设置为参考组,而将另一组设置为测试组。
- 使用模块保留函数计算保守度统计参数Z_summary值和中位数值。执行命令线:
"系统.时间([mp]模块保存(多扩展,
多色,参考网络=1,
n 排列=100,随机种子=1,快速科尔=0,冗长=3)])
单击 "运行"。
注:此结果可以量化模块之间的保守性。Z_summary>10 表示两个模块保存高度,而 Z_summary<2 表示未保留模块。中位数表示按排名评估的模块的相对保存。较高的中位数值表示未保留的模块。(关键命令线指 补充S3。) - 使用绘图功能可视化结果(图3)。获取参数Z_summary和中位数(表1)。
注:网络模块满足Z_summary值小于2和中位数排名值在顶部,是两个生态微生物群落中保存最高度的模块。 - 根据上述两个统计参数的结果,确定该模块具有两个网络中保存程度最高的模块。
- 模块成员的相关性分析
- 将两个网络的模块分配结果分别设置为参考组和测试组。
注:设置必须与保存测试相同。 - 使用科普价值学生函数在多个候选模块中提取每个 OTU 的 kME(模块成员)值。
执行命令线:
"价值 = 数据. 框架 (科普价值学生 (as. 矩阵)
(模块成员),样本))
单击 "运行"。
注:kME 代表模块成员的程度。ME 代表模块 eigen,它表示模块中 OTU 表达的整体水平。kME 是每个 OTU 和 ME 之间的相关系数。通过 OTU 的 kME 值来量化 OTU 在网络中的重要性。(关键命令线指补充S4。) - 然后,使用冗长散射图函数计算两个网络中相应O图的 kME 值的相关系数,并绘制相关分析图(图 4)。
执行命令线:
"冗长散射图(abs(模态成员)
[特模块基因, 特科伦]),
腹肌 (N 模块成员 [N 模块基因, 恩科伦]),
xlab = 粘贴 ("kME 在","ES"),
ylab = 粘贴 ("kME 在"," RP"),
主 + 糊("淡黄色"),
cex. main = 1.7, cex. 实验室 = 1.6, cex. 轴 = 1.6, col = 模块颜色)"
单击 "运行"。 - 选择两个网络 OTU 的 KME 值相关系数最小的模块。认为此模块具有两个网络的最大差异。
- 将两个网络的模块分配结果分别设置为参考组和测试组。
5. 微生物微分网络模块分析
- 通过对模块 OTU 序列集的统计分析,获取差异最大的主导细菌植物的数据。
注:差异最大的模块的 OTU 序列集由植物分类法进行总结。占主导菌菌的占10%以上。 - 然后,使用导入网络ToCytoscape功能,在最大的差分模块中获取包含 OTU 交互关系信息的文件。
执行命令线:
"细胞 = 出口网络到西托斯景观 (模组,
边缘文件 = 粘贴 ("新ES_CytoscapeInput边缘 -", 模块, ".txt", sep=),
节点文件 = 粘贴("新ES_CytoscapeInput节点-",模块,".txt",sep=),
加权 = 真实, 阈值 = 0.5, 节点名 = 模组,
阿尔特诺德纳米斯 = 模组, 节点 + 模块颜色 [在模态])"
单击 "运行"。 - 将文件导入细胞景观。将阈值设置为 0.5,并根据需要调整其他参数。
- 构建微分微生物共发生网络(图5)。
- 获取了在网络中具有最重要监管作用的核心属的信息。
注:根据 OUT 的 kME 值,可以定义核心属。 - 最后,评估了核心属的功能,分析了其对整个差异网络的影响。
结果
本文的代表性结果从 NCBI 数据库 (PRJNA386367)9中的 2014 年加州阿巴克水稻根微生物群数据中下载。数据包括在水下稻田中生长了14周的稻田中的稻田、日光和内层微生物群样本。我们使用WGCNA算法来选择满足三个网络的功率值,这三个网络接近无规模网络(图1),并开发了三个共同表达网络(图2)。在内圈、日光和日光圈土壤微生物共表达网络中,分别确定了23个、22个和21个模块。这些结果表明,这三个利基的微生物相互作用网络的数量基本相等。
我们进一步比较了内层、日光和生球土壤中微生物网络模块的差异。获得了三个利基组模块的以下保存测试结果。在日光圈土壤和日光飞机之间存在三个极不保存的模块(图3a,表1)。此外,在日光圈土壤和内层(图3b,表2)和6个极不保存的模块之间存在rhizosphere土壤和内层(图3c,表3)之间的9个极不保存的模块。此外,在三个利基中发现了极不保存的模块,表明三个利基中的微生物成分存在巨大差异。图4说明了获得的非保守模块的模块成员关系分析结果。从数字上看,在三个利基市场中,每个利基的相关性最小,显著不同的模块可见一斑,这代表了它们之间最显著的差异。
在日志球-rhizoe平面差异网络(图5a)中,主要的植物是 蛋白细菌 (72.97%)。在日志球-内层差异网络(图5b)中,主要的植物是 蛋白菌 (66.36%)、 活性细菌 (10.1%)和 细菌 (10.9%)。在日志行星-内层差异网络(图5c)中,主要的植物是 蛋白菌 (41.41%)、 细菌 (10.10%)、 菲米丘特 (12.12%)和 维鲁科米罗比亚。
三个核心属(图5a),包括 罗多菌 和 诺沃斯芬戈比姆,六个核心属(图5b),包括 Blvii28 和 德氯莫纳斯,以及五个核心属(图5c),包括 细胞病毒 和 地球细菌,在三个微分共发生网络中发挥着重要的调控作用。除 德氯莫纳斯外,所有核心属只对一个网络产生影响,表明稻根三个利基区微生物种群和物种的相对丰度存在相当大的差异,严重影响了现有根微生物群落的丰度和多样性。
核心属 阿佐斯皮里姆,存在于水稻的日志圈内层差异网络,参与氮固定和促进植物生长10。此外,在地层-内层分界网中显著富集的 地菌 属可能是导致许多土壤和沉积物中不溶性费和Mn氧化物减少的主要因素。这些微生物与根基的一系列微生物群落相互作用,并积极参与微生物网络的调节,这对水稻根的生长和发展可能至关重要。
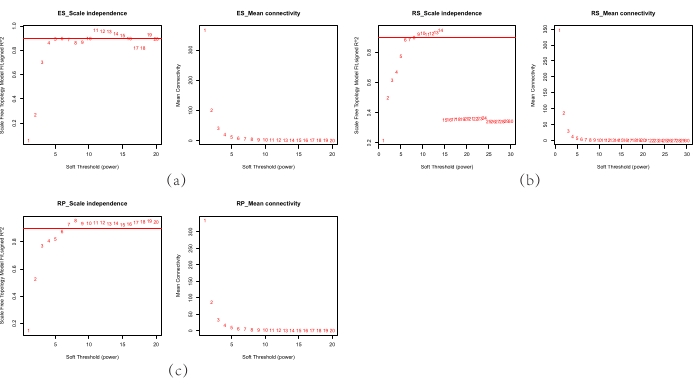
图1。数据集中电源的评估。 (a) ES 数据集中的功率β评估;(b) RS 数据集中的功率β评估;(c) RP 数据集中的功率β评估、无比例指数 R2(左)的分布、沿不同软实力指数的平均连接分布(右)。当R2趋于饱和且不低于0.8时,就实现了最佳功率的价值。必须将三个共同表达网络设置为相同的功率值,以确保其可比性。(RS: 日光圈土壤, RP: 日光飞机, 和 ES: 内层) 。 请单击此处查看此图的较大版本。

图2。通过平均链接分层聚类获得的 OTU 登德罗格。(a)来自ES网络的 OTU 登德罗格:(b) 来自RS网络的 OTU 登德罗格:(c) 来自RP网络的 OTU 登德罗格拉姆。登德罗格拉姆下方的颜色行表示由动态剪树算法确定的模块分配。(RS: 日光圈土壤, RP: 日光飞机, 和 ES: 内层) 请单击此处查看此图的较大版本。

图3。保存测试结果。 (a) 分析结果以RS共同表达网络模块分配为参考组,RP共同表达网络模块分配结果为测试组:(b) 分析结果以ES共同表达网络模块分配为参考组,RS共同表达网络模块分配结果为测试组:(c) 分析结果以ES共表达网络模块分配为参考组,RP共同表达网络模块分配结果为测试组。Z_summary >10 表示两个模块保存高度,而Z_summary < 2 表示未保留模块。中位数表示按排名评估的模块的相对保存。较高的中位数值表示未保留的模块。(RS: 日光圈土壤, RP: 日光飞机, 和 ES: 内层) 请单击此处查看此图的较大版本。
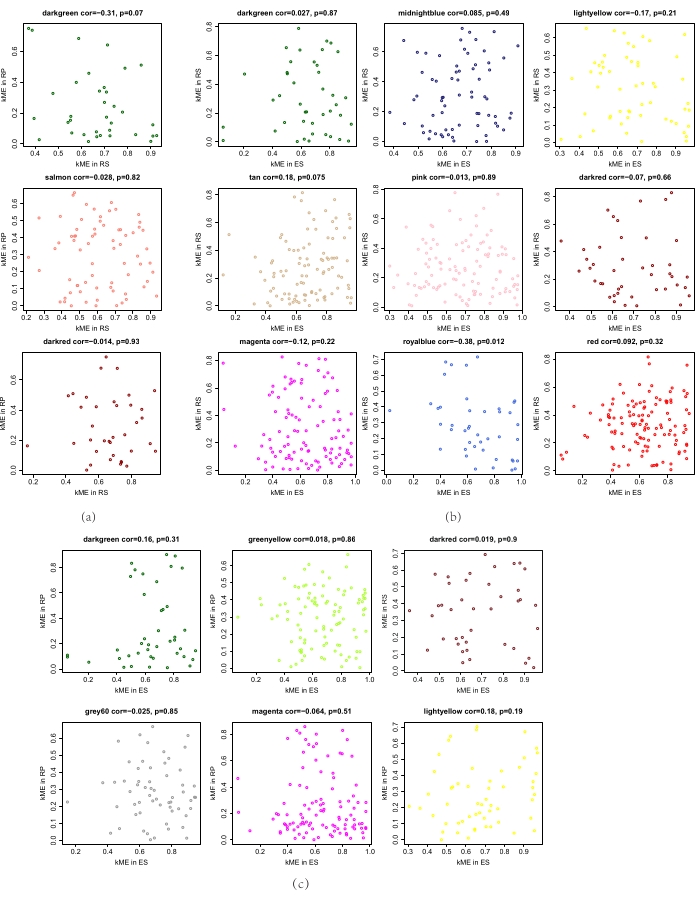
图4。模块成员的相关性分析。 (a) RS 和 RP 网络之间 kME 值的模块相关性;(b) RS 和 RP 网络之间 kME 值的模块相关性;(c) RP 和 ES 网络之间的 kME 值的模块相关性 (RS: 岩石圈土壤、RP: rhizosplane、ES:内层和 KME: 模块成员)。 请单击此处查看此图的较大版本。
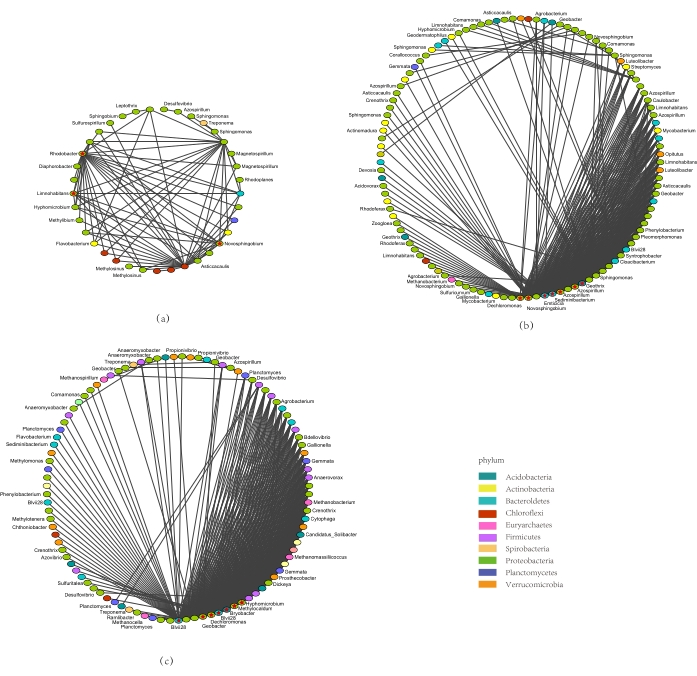
图5。水稻根部微分微生物的共发生网络。(a)RS-RP中微分微生物种群的共发生网络:(b) ES-RS中微分微生物种群的共发生网络:(c) ES-RP中微分微生物种群的共发生网络。分析是使用Cytoscape软件进行的。不同的颜色表示图中的不同门。(RS: 日光圈土壤, RP: 日光飞机, 和 ES: 内层) 。 请单击此处查看此图的较大版本。
| 模块 | 中位数 | 祖马里 | 模块 | 中位数 | 祖马里 |
| 绿黄色 | 2 | 14 | 黄色 | 13 | 5.2 |
| 粉红色 | 2 | 17 | 灰色60 | 14 | 3.1 |
| 午夜蓝 | 3 | 10 | 皇家蓝 | 15 | 2.1 |
| 棕色 | 4 | 18 | 黑 | 16 | 2.7 |
| 光青 | 6 | 8.5 | 鞣 | 17 | 3.2 |
| 紫色 | 7 | 10 | 鲑鱼 | 18 | 1.3 |
| 蓝 | 7 | 22 | 品红 | 18 | 2.2 |
| 绿 | 8 | 12 | 变暗 | 20 | -0.24 |
| 青色 | 10 | 4.8 | 金 | 20 | 14 |
| 浅绿色 | 11 | 5.1 | 黑绿色 | 22 | -1.1 |
| 淡黄色 | 12 | 5.3 | 灰色 | 22 | 0.21 |
| 红 | 12 | 6.1 |
表1。日志球土壤和日志飞机之间的中位兰克的结果。
| 模块 | 中位数 | 祖马里 | 模块 | 中位数 | 祖马里 |
| 鲑鱼 | 1 | 19 | 光青 | 13 | 1.1 |
| 黑 | 3 | 5.3 | 红 | 15 | 0.95 |
| 黄色 | 4 | 5.8 | 午夜蓝 | 15 | -0.0016 |
| 浅绿色 | 5 | 0.27 | 皇家蓝 | 16 | 0.83 |
| 绿黄色 | 7 | 3 | 品红 | 16 | 0.52 |
| 黑绿松石 | 7 | 1.2 | 黑绿色 | 17 | 0.16 |
| 灰色60 | 9 | 1.1 | 鞣 | 18 | 0.64 |
| 蓝 | 10 | 3.9 | 淡黄色 | 19 | 0.52 |
| 紫色 | 10 | 2.3 | 变暗 | 19 | -0.18 |
| 棕色 | 12 | 2.3 | 粉红色 | 19 | -0.71 |
| 青色 | 12 | 0.78 | 金 | 21 | 11 |
| 绿 | 13 | 1.7 |
表2。在日光圈土壤和内层之间产生中位兰克的结果。
| 模块 | 中位数 | 祖马里 | 模块 | 中位数 | 祖马里 |
| 黑 | 1 | 15 | 黑绿松石 | 13 | 1.7 |
| 鲑鱼 | 2 | 27 | 午夜蓝 | 13 | 1.6 |
| 黄色 | 3 | 13 | 浅绿色 | 13 | 0.64 |
| 青色 | 4 | 5.4 | 黑绿色 | 14 | 1.5 |
| 蓝 | 8 | 3.9 | 变暗 | 16 | 1.5 |
| 光青 | 9 | 2.6 | 紫色 | 17 | 2.3 |
| 粉红色 | 10 | 3.6 | 绿黄色 | 18 | 0.8 |
| 皇家蓝 | 10 | 1.5 | 淡黄色 | 18 | 0.42 |
| 棕色 | 12 | 2.9 | 品红 | 19 | 0.2 |
| 绿 | 12 | 1.9 | 金 | 21 | 18 |
| 红 | 12 | 1.9 | 灰色60 | 21 | -0.21 |
| 鞣 | 13 | 2.5 |
表3。在日志飞机和内层之间产生齐马利和中位兰克的结果。
补充 S1: 请点击这里下载此文件.
补充 S2: 请点击这里下载此文件.
补充 S3: 请点击这里下载此文件.
补充 S4: 请点击这里下载此文件.
讨论
相关网络越来越多地用于生物信息学应用。WGCNA是一种系统生物学方法,用于描述分析生物系统12的各种元素之间的关系。R软件包用于WGCNA13、14、15的早期工作。该软件包包括网络构建、模块检测、拓扑特性计算、数据模拟、可视化和与外部软件交互的能力等功能。WGCNA被广泛应用于分析来自脑癌16、酵母细胞周期17、小鼠遗传学18、19、灵长类脑组织20、21、糖尿病22和植物23的基因表达数据。使用加权基因相关网络分析构建网络必须包括至少8个样本。在本文中,我们重点介绍了描述不同环境中微生物群之间相互作用的基因共同表达网络。我们获得了不同环境中微生物种群之间的差分网络,并确定了每个网络中的关键物种。关键物种对社区很重要的观念在食品网研究中得到了广泛的应用。在复杂的微生物群落中的一些物种对于维持社区的稳定性和功能至关重要,例如肠道植物群落中的细菌25。通过针对具有潜在重要性的特定物种,可以大大简化微生物群落的分析。
我们的代表性结果突出了微生物群落的差异,可以使用上述方法进行识别。在这里,稻根系统不同利基的微生物受到WGCNA的控制。通过保守和模块成员分析,确定了这三个利基市场的差异。我们确定了差异模块中的关键物种,并获得了关于三个利基区微生物群落组成差异的信息。同时,共发生网络揭示了水稻根部微生物变化之间的显著相互作用。我们的发现为WGCNA在评估不同环境中微生物群落差异方面的重要性和可行性提供了直接证据。
披露声明
作者没有什么可透露的。
致谢
该手稿的研制得到了中国贵州省人民政府喀斯特科学研究中心项目(U1812401)、贵州师范大学博士研究项目(GZNUD[201])的资助 7[1),贵州省科技支撑项目(QKHZC[2021]YB459)和贵阳科技项目([2019]2-8)。
作者感谢Ededs J.A等人在公共数据库中提供水稻微生物群数据,并感谢TopEdit(www.topeditsci.com)在编写这份手稿期间提供的语言援助。
材料
| Name | Company | Catalog Number | Comments |
| R | The University of Auckland | version 4.0.2 | R is a free software environment for statistical computing and graphics. It compiles and runs on a wide variety of UNIX platforms, Windows and MacOS. |
| RStdio | JJ Allaire | version 1.4.1103 | The RStudio IDE is a set of integrated tools designed to help you be more productive with R and Python. |
| Cytoscape | version 3.7.1 | Cytoscape is an open source software platform for visualizing complex networks and integrating these with any type of attribute data. | |
| NCBI database | The National Center for Biotechnology Information advances science and health by providing access to biomedical and genomic information. |
参考文献
- Philippot, L., Raaijmakers, J. M., Lemanceau, P., vander Putten, W. H. Going back to the roots: the microbial ecology of the rhizosphere. Nature Reviews Microbiology. 11, 789-799 (2013).
- Fierer, N. Embracing the unknown: disentangling the complexities of the soil microbiome. Nature Review Microbiology. 15 (10), 579-590 (2017).
- Jin, J., Wang, G. H., Liu, X. B., Liu, J. D., Chen, X. L., Herbert, S. J. Temporal and spatial dynamics of bacterial community in the rhizosphere of soybean genotypes grown in a black soil. Pedosphere. 19 (6), 808-816 (2009).
- Ma, B., et al. Genetic correlation network prediction of forest soil microbial functional organization. ISME J. 12 (10), 2492-2505 (2018).
- de Vries, F. T., et al. Soil bacterial networks are less stable under drought than fungal networks. Nature Communications. 9 (1), 3033 (2018).
- Colin, C., et al. Correlating transcriptional networks to breast cancer survival: a large-scale coexpression analysis. Carcinogenesis. (10), 2300-2308 (2013).
- Ma, B., Zhao, K., Lv, X., et al. Genetic correlation network prediction of forest soil microbial functional organization. ISME J. 12, 2492-2505 (2018).
- Zhang, B., Horvath, S. A general framework for weighted gene co-expression network analysis. Statistical applications in genetics and molecular biology. 4 (1), (2005).
- Edwards, J. A., et al. Compositional shifts in root-associated bacterial and archaeal microbiota track the plant life cycle in field-grown rice. PLoS Biology. 16 (2), 2003862 (2018).
- Bashan, Y., De-Bashan, L. E. How the Plant Growth-Promoting Bacterium Azospirillum Promotes Plant Growth-A Critical Assessment. Advances in Agronomy. 108, 77-136 (2010).
- Lovley, D. R., et al. Geobacter: The Microbe Electric's Physiology, Ecology, and Practical Applications. Advances in Microbial Physiology. 59, 1 (2011).
- Langfelder, P., Horvath, S. WGCNA: an R package for weighted correlation network analysis. BMC Bioinformatics. 9, 559 (2008).
- Zhang, B., Horvath, S. A General Framework for Weighted Gene Co-expression Network Analysis. Statistical Applications in Genetics and Molecular Biology. 4 (1), 17 (2005).
- Horvath, S., Dong, J. Geometric interpretation of Gene Co-expression Network Analysis. PLoS Computational Biology. 4 (8), 1000117 (2008).
- Langfelder, P., Horvath, S. Eigengene networks for studying the relationships between co-expression modules. BMC Systems Biology. 1, 54 (2007).
- Horvath, S., et al. Analysis of Oncogenic Signaling Networks in Glioblastoma Identifies ASPM as a Novel Molecular Target. Proceedings of the National Academy of Sciences of the United States of America. 103 (46), 17402-17407 (2006).
- Carlson, M. R., et al. and Sequence Conservation: Predictions from Modular Yeast Co-expression Networks. BMC Genomics. 7 (1), 40 (2006).
- Fuller, T., et al. Weighted Gene Co-expression Network Analysis Strategies Applied to Mouse Weight. Mammalian Genome. 6 (18), 463-472 (2007).
- Yin, L., Wang, Y., Lin, Y., et al. Explorative analysis of the gene expression profile during liver regeneration of mouse: a microarray-based study[J]. Artificial Cells Nanomedicine & Biotechnology. 47 (1), 1113-1121 (2019).
- Oldham, M., Horvath, S., Geschwind, D. Conservation and Evolution of Gene Co-expression Networks in Human and Chimpanzee Brains. Proceedings of the National Academy of Sciences of the United States of America. 103 (47), 17973-17978 (2006).
- Oldham, M. C., et al. Functional organization of the transcriptome in human brain. Nature Neuroscience. 11 (11), 1271-1282 (2008).
- Keller, M. P., et al. A gene expression network model of type 2 diabetes links cell cycle regulation in islets with diabetes susceptibility. Genome Research. 18 (5), 706-716 (2008).
- Weston, D., Gunter, L., Rogers, A., Wullschleger, S. Connecting genes, coexpression modules, and molecular signatures to environmental stress phenotypes in plants. BMC Systems Biology. 2 (1), 16 (2008).
- Jorda´n, F. Keystone species and food webs. Biological Sciences. 364, 1733-1741 (2009).
- Backhed, F., Ley, R. E., Sonnenburg, J. L., Peterson, D. A., Gordon, J. I. Host-Bacterial Mutualism in the Human Intestine. Science. 307, 1915-1920 (2009).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。