Method Article
Создание трансгенных растений с одной копии вставки с помощью бинарный вектор BIBAC-GW
В этой статье
Резюме
С помощью бинарный вектор pBIBAC-GW делает создания трансгенных растений с нетронутыми одной копии вставки, легкий процесс. Здесь ряд протоколов представлены Путеводитель читателя через процесс создания трансгенных растениях Arabidopsis и тестирования растений для целостности и копировать количество вставок.
Аннотация
При генерации трансгенных растений, обычно цель заключается в том, чтобы иметь стабильные выражение трансген. Это требует единого, нетронутыми интеграции трансген, интеграции нескольких копирования часто подвергаются сайленсинга генов. Шлюз совместимых бинарный вектор, основанные на бактериальных искусственных хромосом (pBIBAC-GW), как и другие производные pBIBAC, позволяет вставки одной копии трансгенов с высокой эффективностью. Как улучшение в оригинальной pBIBAC шлюз кассету был клонирован в pBIBAC-GW, так что последовательности интерес может теперь быть легко включены в вектор передачи ДНК (T-ДНК) путем клонирования шлюза. Обычно, преобразования с pBIBAC-GW приводит к эффективности 0,2 – 0,5%, при котором половина мутация нести нетронутыми поштучными интеграции T-ДНК. PBIBAC-GW векторов доступны с устойчивость к глюфосинату аммония или DsRed флуоресценции в пальто семян для отбора в растениях и устойчивость к канамицину как выделение бактерий. Здесь, что руководство читателя через процесс создания трансгенных растений, с помощью pBIBAC-GW представлены серии протоколов: начиная от комбинирование последовательности интерес в pBIBAC-GW вектора выбора, к заводе преобразования с Agrobacterium, выбор мутация и тестирования растений для целостности и копировать количество вставок, используя ДНК blotting. Внимание уделяется разработке стратегии переноса ДНК признать одним и несколькими копиями внедрений в одной системе и нескольких локусов.
Введение
При генерации трансгенных растений, обычно цель заключается в том, чтобы иметь комплексный transgene(s) стабильно выразил. Это достигается за счет нетронутыми Поэкземплярная интеграция трансген. Несколько интеграции может привести к увеличению выражение трансген, но и для подавления экспрессии гена. Глушителей трансгенов является более вероятным, если вставленный последовательности расположены в тандеме или перевернутый повторяется1,2,3,4. Бинарных векторов используются как Трансфер в Agrobacterium-опосредованной преобразования экспериментов, чтобы доставить последовательности интерес в геномах растений. Количество внедрений в геном растения зависит количество копию бинарный вектор. Agrobacterium tumefaciens5,6 Многие часто используемые бинарных векторов высокой копии векторы и поэтому дают высокий средний трансген копии номер: 3.3 до 4,9 копий в проростках Arabidopsis5.
Количество внедрений T-ДНК может быть снижен с помощью бинарных векторов, которые имеют низкий копия число в A. tumefaciens, например BIBAC7, или путем запуска T-ДНК от A. tumefaciens хромосомы5. Среднее количество внедрений трансген в таких случаях является ниже 25,8,9,10. Из-за одной копии в A. tumefaciens, а также кишечная палочка, BIBAC-производные может поддерживать и доставить конструкции, как большой, как11150 КБ.
GW-совместимых BIBAC векторов10,12 позволяют легко введение в vector с использованием шлюза клонирования генов интерес. Использование шлюза технологии значительно упрощает процесс клонирования, но также преодолевает общие проблемы, связанные с большими низким копии число векторов13,14, таких как низкая доходность ДНК и ограниченный выбор уникальных ограничений сайты, доступные для клонирования7,11. PBIBAC-GW производные доступны либо сопротивление к глюфосинату аммония (pBIBAC бар-GW) или DsRed флуоресценции в пальто (pBIBAC ЗП-GW) семян для выбора растений (рис. 1)10,12. Для обоих векторов канамицин сопротивления ген используется в качестве маркера выделения бактерий.
PBIBAC-GW векторов объединить: (1) легкий дизайн и генетических манипуляций в E. coliи (2) нетронутыми поштучными внедрений в planta с высокой эффективностью. PBIBAC-GW векторов урожайность в среднем 1,7 внедрений в проростках Arabidopsis с примерно половина трансгенных растений, перевозящих один интегрированный10T-ДНК.
Стабильные выражение трансгенов является требованием для большинства мутация генерируется. Стабильная трансген выражение может быть достиган нетронутыми, одной копии интеграций. Работа с трансгенных растений, перевозящих нетронутыми, одной копии интеграции является, однако, еще более важно, если например, целью является изучение эффективности процессов, основанных на хроматина, например мутагенеза, рекомбинации, или ремонт и зависимость от этих процессы на геномной расположение и структура хроматина в месте вставки. Для наших интересов изучать зависимость олигонуклеотида Направленный мутагенез (ODM) в контексте местных геномной, набор репортер линий с неповрежденными, одной копии интеграций мутагенеза Репортер ген был сгенерированный (рис. 2)10. С помощью этого набора строк, было показано, что эффективность ODM колеблется от трансгенных локусов, интегрированный в разных местах генома, несмотря на довольно аналогичные уровни выражения трансген.
протокол
1. Вставка последовательности интерес в бинарный вектор
-
Подготовка записи шлюза и бинарных векторов.
- Изолируйте запись шлюза вектор, содержащий фрагмент ДНК или гена интереса, используя набор мини-prep согласно предложения поставщика.
Примечание: BIBAC-GW векторов требуют использования канамицин (км) для выделения бактерий, поэтому, используйте вектора входа с другой маркер сопротивления вместо канамицин. Например pENTR-gm вектор, перевозящих ген сопротивления гентамицин, является хорошим выбором12. - Распространять и изолировать BIBAC-GW вектор интересов. Использование штамма кишечной палочки , устойчив к токсичности ccdB гена присутствует в шлюз кассеты. Изоляция BIBAC-векторов с использованием протоколов или комплекты, специально предназначенные для крупных плазмид согласно предложения поставщика.
- Изолируйте запись шлюза вектор, содержащий фрагмент ДНК или гена интереса, используя набор мини-prep согласно предложения поставщика.
-
Осуществляют шлюза реакции.
- Подготовьте реакции рекомбинации LR согласно предложения поставщика. Смешайте следующие компоненты в пробки microcentrifuge 1,5 мл при комнатной температуре (RT): 100-300 нг записи клон (supercoiled), 300 нг BIBAC-GW вектора, LR Clonase реакции буфера (конечная концентрация: 1 x). Отрегулируйте громкость смеси 16 мкл с TE (10 мм трис, 1 ЭДТА, рН 8,0). Наконец добавьте 4 мкл LR Clonase фермент смесь и перемешать, vortexing. Инкубируйте смесь при 25 ° C в течение 1 ч.
- Прекратить LR реакции, добавив 2 мкл раствора протеиназы K (2 мкг/мкл) в смеси, подготовленный в шаг 1.2.1. Смешать и инкубировать при 37 ° C за 10 мин.
-
Преобразование E. coli с шлюза реакционную смесь электропорации.
- Опреснения LR реакционной смеси перед электропорации.
Примечание: Этот шаг очень важен для успешной электропорации. Ниже диализ метод описан, но другие методы, такие как высыпания с ацетат натрия и этанола могут также использоваться.- Подготовка установки для диализа фильтр LR реакции. 20 мл сверхчистого деионизованной воды наливайте в стерильных Петри. Поместите диск мембранный фильтр (размер пор = 0,025 мкм) на поверхности воды.
- Пипетка весь LR реакции тщательно поверх мембраны и позволяют смесь dialyze на RT 1 h.
- Добавьте 5 мкл обессоленной LR микс электро компетентных клеток DH10B в кювет электропорации (0,1 см). Electroporate клетки (1,5 V/см, сопротивление 200 Ω, емкость 25 МКФ) и сразу же добавить 1 мл подогретым супер оптимального Catabolite репрессий (SOC) среднего к клеткам, следуют инкубации при 37 ° C за 45 мин, 180 об/мин. (Средний SOC, экстракт 1 L: 20 г Bacto Триптон, 5 г дрожжей, 0,5 г NaCl, 2,5 мл 1 м KCl, 20 мл 1 м фильтр стерилизации глюкозы).
Примечание: DH10B клетки можно заменить для других клеток кишечной палочки , которые стабильно поддерживать большие плазмид.
Примечание: Условия оптимального электропорации зависит от используемого устройства электропорации. - Пелле бактерий на максимальной скорости, для 30 s с помощью microcentrifuge, удалить избыток SOC и Ресуспензируйте гранулы в приблизительно 50-100 мкл Бертани Лурия (LB) среды. Распространение бактерий на пластины канамицин LB (км-фунтов) (концентрация км, 40 мкг/мл) и инкубировать пластины при 37 ° C на ночь. (Средний LB, 1 L: 10 g Триптон Bacto, 5 г дрожжей экстракт, 10 г NaCl. Для твердой питательной, добавьте агар, 15 г/Л).
- Опреснения LR реакционной смеси перед электропорации.
-
Идентифицировать рекомбинированные производные BIBAC-GW и изолировать плазмида ДНК.
- Чтобы иметь возможность расти на тарелках км-ЛБ, E. coli клетки должен содержать рекомбинированные плазмида BIBAC-GW, в котором с требуемой вставки подставляется ccdB последовательность. Для проверки, если колонии бактерий на табличке содержат правильные плазмид, имея BIBAC-GW позвоночника и вставка интерес используйте колонии полимеразной цепной реакции (ПЦР)15 .
- Чтобы определить основу pBIBAC бар-GW, выполните15 реакции PCR праймеры DM1969 5'-GCGACGAGCCAGGGATAG-3 'и DM1970 5'-ATCAGTGCGCAAGACGTGAC-3'. Этот грунт набор усиливает 563 bp фрагмента гена бар .
- Чтобы проверить наличие BIBAC-ЗП-GW позвоночника, выполните15 реакции PCR, используя праймеры M737 5'-CGTGTAAAAAGCTTAGACTG-3 'и M892 5'-AACAGATGGTGGCGTCCC-3'. Эта грунтовка комбинация усиливает 791 bp фрагмент перекрытия cruciferin promotor и ППП последовательности.
- Выполните15 реакций PCR с использованием гена специфические праймеры для проверки на наличие Вставка интерес.
- Прививать один положительный колонии в 2 – 5 мл LB носитель, содержащий канамицин (40 мкг/мл) для изоляции ДНК16. Инкубируйте при 37 ° C на орбитальный шейкер на 180 об/мин, на ночь.
- Изолировать плазмидной ДНК (см. шаг 1.1.2).
- Чтобы иметь возможность расти на тарелках км-ЛБ, E. coli клетки должен содержать рекомбинированные плазмида BIBAC-GW, в котором с требуемой вставки подставляется ccdB последовательность. Для проверки, если колонии бактерий на табличке содержат правильные плазмид, имея BIBAC-GW позвоночника и вставка интерес используйте колонии полимеразной цепной реакции (ПЦР)15 .
2. Подготовка A. tumefaciens цветочные погружения Arabidopsis
-
Преобразование BIBAC-GW производные A. tumefaciens.
- Подготовьте электро компетентных клеток A. tumefaciens штамм C58C1 проведение pCH32 вспомогательный плазмида7. Рост бактерий в присутствии Тетрациклин (5 мкг/мл) и рифампицин (100 мкг/мл), чтобы выбрать для pCH32 и обеспечить рост только Agrobacterium клеток.
- Добавить 0,25 – 0,5 мкг ДНК pBIBAC-GW производной, предварительно растворяют в 10 – 20 мкл стерильные сверхчистого деионизированной воды, до 20 мкл компетентных Agrobacterium клеток в электропорации кюветы (0,1 см). Держите клетки на льду.
- Electroporate клетки (1,5 V/см, сопротивление 400 Ω, емкость 25 МКФ). Сразу же после электропорация добавить средне SOC подогретым (28 ° C) 1 мл на бактерии и инкубировать клетки на 28 ° C в течение 60-90 мин.
- Распространение 100 мкл и остальная часть бактерии на отдельных плит LB, содержащих рифампицин (100 мкг/мл), тетрациклином (5 мкг/мл) и канамицин (40 мкг/мл) и инкубировать в темноте на 28 ° C в течение 1-2 дней.
-
Подготовьте Agrobacterium подвеска.
- Проверить методом ПЦР пару A. tumefaciens колоний от плиты, подготовленную на этапе 2.1.4, наличие правильного вектора (см. шаг 1.4.1).
- Полоска единую колонию подтвердил содержит бинарный вектор с соответствующим вставки на табличке LB, содержащие антибиотики (см. шаг 2.1.4). Растут на 28 ° C на ночь.
- Повторите, полос с одной колонии, полученного на шаге 2.2.2.
- Прививать единую колонию в 2,5 мл среды LC с антибиотиками (см. шаг 2.1.4) для preculture. Инкубируйте на 28 ° C в течение 8 часов или на ночь, на 180 об/мин. (LC средний, 1 L: 10 g Триптон Bacto, 5 г дрожжей экстракт, 0,5 г NaCl, 2,5 г MgSO4 · 7 H2O, 2 g мальтоза).
- Добавьте preculture из шага 2.2.4. 250 мл LC с антибиотиками (см. шаг 2.1.4) и расти на 28 ° C, на 180 об/мин, на ночь.
- Пелле культуры спиннинг на 5500 g x 12 мин вновь приостановить гранулы в 100 мл раствора, содержащего 5%-ая сахароза, 0,05% Silwet L-77, 0.5 x г-жа налить подвеска в стерильный контейнер для цветочные погружения растений.
3. Arabidopsis преобразования
-
Подготовьте растения арабидопсис для преобразования.
- Растут растения арабидопсис в теплице или климат контролируемый рост палата до тех пор, пока они цветут (12 горшки с 9 растений на погружение).
- Клип первый болты, чтобы позволить более вторичных болты выйти. Растения будут готовы для погружения 4 – 6 дней после отсечения, когда растения имеют много незрелых цветок головы и не много оплодотворенной siliques.
-
Цветочные погружения
- Окуните соцветия на 5 – 10 s в Agrobacterium подвеска, подготовленную на этапе 2.2.6. Используйте нежное агитации.
- Оберните надземной части растения в фильме цепляться, чтобы сохранить влажность высокая и охватывают горшки с коробку держать растения в темноте. Инкубируйте 2 дней в камере парниковых/роста растения.
- Удалите поле и цепляться пленки и расти растения к зрелости в камере парниковых/рост.
Примечание: Для повышения эффективности преобразования, же растения могут быть повторно опущенные 7 дней после первого погружения. - Урожай семян. Бассейн и анализировать (T1) семена растений, преобразована с этой же конструкции, как один набор.
-
Экран для трансгенных растений.
- Для блокировки для трансгенных растений превращается с pBIBAC ЗП-GW производной, анализировать семена, с помощью микроскопии флуоресцирования. Для того чтобы обнаружить DsRed выражение в семя пальто, изображение семена на возбуждение 560 Нм и выбросов 600-650 Нм. Отделите флуоресцентные семена от не люминесцентные коллегами с помощью щипцов.
- Для блокировки для трансгенных растений превращается с pBIBAC бар-GW производной, сеять семена в поддоны заполнены с почвой (~ 2500 семена/0.1 м2). Для обеспечения даже распространение семян за поддоны, приостановить семена в 0,1% агар в 0,5 x фотосинтетическую Скуг среднего (МС) и распространение семян с помощью пипетки 1 мл.
Примечание: Чтобы стимулировать семена прорастают в синхронном режиме, Проинкубируйте семена по крайней мере 2 дня при 4 ° C. Это можно сделать до или после посева семян.- Spray саженцы с 0,5% раствором глюфосинату аммония 2 недели и 3 недели после посева в лотки. Использование 500 мл раствора глюфосинату аммония на 1 м2.
- Передавать Баранчики живых саженцы. Типичный образ поднос с саженцев до и после лечения (второй) глюфосинату аммония показаны на рисунке 3.
- Анализировать глюфосинату аммония устойчивостью растений методом ПЦР на наличие конструкции интерес (см. шаг 1.4.1. для грунтовки).
- Изолировать ДНК генома растений для ПЦР с помощью метода, описанного в Edwards et al. 17
4. Характеризуя мутация число и целостности T-ДНК внедрений
- Стратегия ограничения пищеварения
Примечание: Определите количество внедрений T-ДНК и их целостность ДНК промокательной с помощью энзимов ограничения. Этот метод позволяет определить единый, но также повторил внедрений на одном или разных локусов генома.- Использование ряда пищеварения ограничения для выявления возможных различных интеграционных моделей:
- Выберите фермент, который режет раз в середине T-ДНК, чтобы иметь возможность самостоятельно исследовать последовательности вверх и вниз по течению от места ограничения (рис. 4A и Рисунок 7а C). Смотрите Рисунок 4Aи фигура легенда для ожидаемых результатов и интерпретации.
- Выберите фермента или комбинация ферментов, вырезая всю последовательность интерес одновременно (рис. 4B и Рисунок 7а-D). Любое отклонение от известных длина показывает усечение комплексной кассеты.
Примечание: позаботьтесь, чтобы использовать только энзимов ограничения, которые не чувствительны к метилирование цитозина.
- Использование ряда пищеварения ограничения для выявления возможных различных интеграционных моделей:
- Подготовьте образцы геномной ДНК.
- Изолируйте геномной ДНК из растений, перевозящих конструкции интерес. CTAB ДНК алкалический метод может использоваться для изоляции ДНК18. Для ДНК блот анализ ДНК Arabidopsis , необходима 2 – 2,5 мкг геномной ДНК. Растворите ДНК в 50 мкл TE.
- Проверьте целостность ДНК электрофорез геля16. Нетронутыми геномной ДНК переносит как одна дискретных полоса в верхней части геля. Деградации ДНК могут быть признаны в качестве присутствие мазок. Чтобы избежать повреждения ДНК из-за повторного замораживания оттаивания, геномной ДНК образцов хранятся при 4 ° C.
- Дайджест геномной ДНК (2-2,5 мкг в случае Arabidopsis геномной ДНК) в общем объеме 50 мкл, на ночь, используя буфер условия, предложенные поставщиком фермента.
- В пробирке Смешайте 2 – 2,5 мкг Arabidopsis геномной ДНК, 5 мкл 10 x ограничение буфера и 5 U энзима ограничения в общей сложности 50 мкл сверхчистого обессоленной воды.
- Добавьте краситель погрузки (1 x конечная концентрация) ограничение образцы перед загрузкой на геле. Для хорошего визуального отслеживания, используйте один из следующих: comigrating с небольших фрагментов (бромфеноловый синий, 350 – 400 bp), или comigrating с более крупных фрагментов (Cylene cyanol, 3 – 4 kbp).
- Побегите гель ДНК.
- Подготовьте длинный (20 см) 0,5 x геля агарозы КЭ19. % Агарозном геле зависит от размеров фрагмента ожидается. 0,8 – 1% оптимально отделяет фрагменты > 1 kb в размер. Использовать 1 – 1,5% агарозы для фрагментов < 1 КБ. Не добавляйте бромид Ethidium геля. (5 x TBE, 1 L: 54 g Trizma базы, 27,5 г борной кислоты, 3.75 г ЭДТА).
Примечание: Для предотвращения загрязнения ДНК, использования геля лотков, которые не используются для фракционировать плазмиды и ПЦР усиленный ДНК. - Загрузите образцы гель19.
- Добавьте маркеры ДНК в гель, который находятся в диапазоне размеров от ожидаемого фрагментов. Загрузка приблизительно 1 мкг маркера (50-250 нг различных размеров фрагмента) на геле разрешить визуализации УФ светом.
- Размер фракционировать ДНК при низком напряжении (40 – 50 V/500 mA) на ночь.
- Подготовьте длинный (20 см) 0,5 x геля агарозы КЭ19. % Агарозном геле зависит от размеров фрагмента ожидается. 0,8 – 1% оптимально отделяет фрагменты > 1 kb в размер. Использовать 1 – 1,5% агарозы для фрагментов < 1 КБ. Не добавляйте бромид Ethidium геля. (5 x TBE, 1 L: 54 g Trizma базы, 27,5 г борной кислоты, 3.75 г ЭДТА).
- Подготовка к передаче ДНК из геля агарозы мембраны нейлона.
- Перевести гель в отдельный лоток и пятно на 20 – 25 минут в 0,5 x TBE содержащий бромид Ethidium (5 мкг/мл), повернув на 40 об/мин на орбитальный шейкер.
- Визуализируйте гель на УФ transilluminator. Проверьте размер разделение геномной ДНК, включая видимость дискретных спутниковой полосы (рис. 5A). Мазок к ниже, молекулярным весом размеров указывает деградации ДНК.
- УФ transilluminator заложить прозрачность геля и отметьте положение слотов и маркер полосы с маркером (Рисунок 5B). Это облегчит определение размера гибридизированные фрагментов позднее, в случае, если маркер последовательности не гибридизируйте специально с зонд ДНК. Указав маркер фрагментов на этот шаг, можно отслеживать размер гибридизировать фрагментов.
- Поместите гель обратно в лоток, промыть сверхчистого деионизированной воды и погружаться в 0,25 М HCl за 15 мин до раздробляет ДНК в гель. Промыть сверхчистого деионизированной водой. Использование достаточно HCl и воды для покрытия гель в лоток и поверните поднос с погруженной геля на 40 об/мин на орбитальный шейкер.
- Проинкубируйте гель в буфере денатурации 30 мин мыть сверхчистого деионизированной водой. Использование достаточно буфера и воды для покрытия гель в лоток и поверните поднос с погруженной геля на 40 об/мин на орбитальный шейкер. (Денатурация буфера: NaOH 0.5 М, 1,5 М NaCl).
- Проинкубируйте гель в буфере нейтрализации 30 мин мыть сверхчистого деионизированной водой. Используйте достаточно буфера и воды для покрытия гель в лоток, поверните поднос с погруженной геля на 40 об/мин на орбитальный шейкер. (Нейтрализация буфера: 0,5 М трис, 1,5 М NaCl, 220 мм HCl, рН 7,6).
- Передать ДНК мембраны нейлона.
- Подготовка установки для капиллярной передачи геномной ДНК. Поместите лоток заполнен с 20 x солевой раствор натрия цитрата (SSC) пластиковые пластины (примерно размер геля или больше). Сложите кусок толстой фильтровальной бумаги над лоток, так что оба из ее концов висят в ССК. Вырежьте кусок гель размера мембрана положительно заряженный нейлон Hybond N + и 2 куски толщиной фильтровальной бумаги. (20 x SSC: 3 M NaCl, цитрат натрия 0,3 М).
- Подготовьте blotting установки (Рисунок 6), поместив гель, слоты вниз на верхней части фильтра бумаги на пластиковой пластине. Место Hybond N+ мембраны на вершине, следуют 2 слоя фильтровальной бумаги. Предварительно мокрой каждый слой в 20 x SSC перед их добавлением в Ассамблее. Убедитесь в том удалить все пузырьки воздуха между слоями, как они препятствуют передаче ДНК.
- Обложка к Ассамблее с толстым слоем папиросной бумаги. Место пластиковую пластину с весом, таких как маленькую бутылку, на вершине. Убедитесь, что давление поровну через гель. Это обеспечит надлежащую передачу ДНК.
Примечание: Вес должен быть около 200 – 300 г; тяжестей препятствовать передаче ДНК. - Обложка области вокруг Ассамблеи, включая подвергаются фильтровальной бумаги, с цепляться пленки (рис. 6), чтобы избежать испарения 20 x SSC буфер и капиллярных сил к мембраны нейлона. Пятно на ночь.
- Отметьте положение слотов, имя и дата на вершине мембраны с карандаш и удалить мембрану из сборки. Обратите внимание, что снизу, которая соприкасалась с гель, несет в себе ДНК.
- Немедленно исправьте ДНК мембраны, УФ-облучения (2400 µJ/м2) с помощью УФ сшивателя.
Примечание: Сшивки условия зависят от типа используемых мембраны.
Примечание: на данный момент мембраны может храниться при-20 ° C и использоваться для гибридизации с зондом позже. Промойте мембрану из сшитого в 2 x SSC и печать, в его предварительно сложить полипропиленовую полиэтиленовые трубки перед его размещением в-20 ° C.
- Подготовьте зонд для гибридизации.
- Усилить последовательность для использования в качестве зонда для ДНК промокательной ПЦР20. 50-100 нг фрагмента ПЦР 250 kbp bp-2 используется в качестве датчика. Две отдельные датчики, один гибридизировать к правой границе проксимальной региона и другой левой границе проксимальной региона T-ДНК, может использоваться для оценки наличия всего T-ДНК (рис. 4A и рис. 7).
- Развести 50-100 нг ПЦР продукта в 24 мкл сверхчистого обессоленной воды в пробирке.
- Денатурируйте разбавленного продукта ПЦР при кипячении в течение 5 мин в стакан воды или тепла блока, а затем охладить прямо на льду.
- Оттепель готовые GCT-микс на льду. (GCT-микс: dGTP, дЦТФ, dTTP (все 0,5 мм), случайные hexamers 43,2 нг/мкл, Acetylated BSA 1.33 мг/мл, 33 мм β-меркаптоэтанол, 0,67 М Hepes, 0,17 мм трис рН 6,8, 17 мм MgCl).
- Добавьте 21 мкл GCT-микс и 2 U фрагменты фрагмент продукт PCR.
- 2 мкл [32P] АТФ в смеси и инкубировать при 37 ° C в течение 1 ч.
Предупреждение: Все шаги с участием СПС [32P] должны осуществляться в обстановке для радиоактивных работы при использовании соответствующей защиты.
Примечание: Убедитесь, АТФ [32P] свежие (не более 1 половине времени прошло). - Подготовьте торфов G-50 (крупный или средний) столбец21 для очистки помечены зонд из неинкорпорированных (радиоактивных) нуклеотидов. Возьмите шприц 2 мл и охватывают розетки с небольшой круг толстый фильтровальной бумаги. Добавить 2 мл G-50 торфов растворенного в TE в шприц и удалите все жидкости из столбца, спиннинг.
- Поместить столбец в 15 мл пластиковую трубку, загрузить помеченные зонд на столбце и спина при комнатной температуре (центрифуги на 750 x g, позволяют rpm для увеличения до 750 x g, затем остановить центрифуги и дайте вращение снизиться до 0 x g) для элюировать зонд. Добавить 200 мкл TE столбцов и спин элюировать оставшихся зонд; Повторите один раз. В этих условиях обозначенных фрагментов ДНК исключаются из торфов матрицы и элюировать, в то время как бесплатные нуклеотидов остаются в столбце.
- Используйте 300 мкл помечены зонда в гибридизации трубки. Держите оставшиеся помечены зонд при-20 ° C для последующего использования. Однако имейте тайм [32P] АТФ в виду.
- Гибридизируйте блот ДНК.
- Забег 2 x SSC и гибридизации буфера (15 мл на гибридизации трубки, максимум 2 помарки в метро) до 65 ° C. (Буфер гибридизации: декстран сульфата 10%, 1% SDS, 1 M NaCl, 50 мм трис рН 7,5, растворяют при 65 ° C, держать аликвоты при-20 ° C).
- Подогрейте печь гибридизации до 65 ° C.
- Место нейлоновая сетка в поднос с немного с подогревом (65 ° C) 2 x SSC для покрытия лоток. Место ДНК блот поверх сетки, с ДНК стороной вверх. Roll пятно вместе с сеткой и вставьте рулон в трубу гибридизации. Слить избыток 2 x SSC.
- В пробки microcentrifuge, кипения 150 мкл ДНК спермы лосося (концентрации 10 мг/мл) в гибридизации трубка для 5 мин (см. также шаг 4.6.3). Cool сразу на льду и добавлять подогретую буфер гибридизации.
- Добавьте решение спермы буфера лосось гибридизации в трубку с пятном. Предварительно гибридизируйте при 65 ° C для по крайней мере 1 час в вращающееся колесо, при 12 об/мин.
- При предварительной инкубации в шаге 4.7.5. почти закончена, кипения 300 мкл помечены зонда для 5 мин (см. также шаг 4.6.3) и сразу же добавить в блот после инкубации.
- Гибридизируйте на ночь в вращающееся колесо на 63 ° C, 12 об/мин. Не Пипетка зонда непосредственно на пятно, но в решение спермы буфера лосось гибридизации.
- Промойте пятно.
- Разогрейте моющих растворов (1 x ГНПП, 0.1% SDS и 0,1 x SSPE, 0.1% SDS) до 65 ° C. (20 x SSPE: 3 M NaCl, 230 мм NaH2PO4, 20 мм ЭДТА, рН 7,0).
- Распоряжаться гибридизации раствор и добавить около 100 – 150 мл 1 x SSPE, 0,1% раствор SDS для гибридизации трубки, закройте трубку и вращать вручную. Залейте гибридизации и моющего раствора в соответствующие жидких радиоактивных отходов.
- Добавить около 100 – 150 мл 1 x SSPE, 0,1% раствор SDS к трубе, закройте и инкубировать трубки для 15 мин на 63 ° C на вращающееся колесо, 12 об/мин. Надлежащим образом утилизируйте моющий раствор.
- Добавить около 100 – 150 мл 0,1 x SSPE, 0,1% раствор SDS для гибридизации трубки, закрыть и поверните трубку для 5 мин на 63 ° C, 12 об/мин. Надлежащим образом утилизируйте моющий раствор.
- Возьмите пятно из трубки и поместите его в лоток, содержащий достаточное разогретой 0.1 x ГНПП, 0.1% SDS и трясти за 3 мин на водяной бане тряски на 65 ° C. Тем временем промойте сетку в поддон с водой.
- Вынуть помаркой, поместить его между предварительно сложенном пластика (полиэтиленовые трубки), тщательно вытрите лишнюю жидкость и пусть пятно сухой кратко. Обратите внимание, что жидкость разрушит phosphorimager экрана.
- Уплотнение пятно в пластик с трех сторон. Удалите все лишнюю жидкость, окружающих блот и закрыть пластиковой трубки, уплотнения с четвертой стороны. Срезают излишки пластика. Убедитесь, что запечатанном пятно не протекает и что пластик сухой снаружи.
- Подвергайте экран phosphorimager.
- Место запечатанном пятно в phosphorimager кассету, с phosphorimager экрана с ДНК стороны пятно. Закройте кассету и оставить для ± 2 – 4 дня, в зависимости от прочности радиоактивных маркировки и чувствительность phosphorimager.
- Проверьте phosphorimager экрана с помощью phosphorimager. Позаботьтесь, чтобы разоблачить на экране как можно меньше света перед сканированием. Сохраните изображение. Стереть экран от сигнала, подвергая его яркий свет.
- Анализ помаркой.
- Анализ зависит от стратегии ограничения на шаге 4.1. При анализе помаркой, подготовленный согласно этой стратегии, показанный в шаге 4.1.1.1 (рис. 4A), граф количество обнаруженных фрагментов. В этой стратегии количество гибридизированные фрагментов относится к числу внедрений T-ДНК.
- Сравните количество фрагментов, обнаружены с зондом для левой (рис. 7B) и справа (рис. 7 c) T-ДНК.
Примечание: Если обнаружен, то либо i) несколько копий T-ДНК (либо в прямой или инвертированный ориентации) или ii) неполно интегрированный различное количество гибридизировать фрагментов T-ННО присутствуют. - Оценить размеры гибридизированные фрагментов на пятно на основе размера маркера полос и сравните размеры гибридизированные фрагментов с фрагмента ожидаемых размеров рассчитаны на основе стратегии ограничения тандем вставок (рис. 4A) Определите возможные тандем T-ННО. На рисунке 4A можно используйте в качестве руководства для вычисления размера ожидаемых фрагментов.
Примечание: Если размер гибридизированные фрагментов не согласны с расчетными, то наиболее вероятновероятн один настоящий интеграции не полным.
- Сравните количество фрагментов, обнаружены с зондом для левой (рис. 7B) и справа (рис. 7 c) T-ДНК.
- При анализе помаркой, подготовленный согласно стратегии, показанный в шаге 4.1.1.2 (рис. 4B), оценить размер фрагмента гибридизировать на пятно, на основе размера маркера полос и сравнить его с ожидаемого размера. Нетронутыми вставки создает единый фрагмент с определенной длины. Любое отклонение ожидаемых длины указывает неполной интеграции (рис. 7 d).
- Анализ зависит от стратегии ограничения на шаге 4.1. При анализе помаркой, подготовленный согласно этой стратегии, показанный в шаге 4.1.1.1 (рис. 4A), граф количество обнаруженных фрагментов. В этой стратегии количество гибридизированные фрагментов относится к числу внедрений T-ДНК.
- Полоса помарку для повторного гибридизации (необязательно).
Примечание: Же блот может гибридизированных последовательно с различными зондами. Прежде чем продолжить с новым датчиком, Стрип предыдущих гибридизированные зонд из пятно.- Чтобы удалить зонд из помаркой, место пятно в лоток с его ДНК-лицевой стороной вниз. Вылейте излишек 0,5% SDS в лоток. Отварите мембрана для 2-5 мин. Продолжительность лечения зависит от размера и GC-содержание зонда используется. Длиннее и GC-богаче зонды требуется более длительное лечение.
- После зачистки, гибридизируйте блот с другой зонд, или печать и хранить при температуре от-20 ° C.
Результаты
С помощью системы BIBAC-GW, репортер конструкции для изучения ODM в растениях были созданные10. Конструкции были разработаны в pENTR-gm вектора входа шлюза12 и вставляется в pBIBAC бар-GW (рис. 1) с использованием реакции рекомбинации шлюза LR.
Арабидопсис были преобразованы с pDM19, BIBAC-бар-GW плазмида с mTurquoise-eYFP репортер, перевозящих кодоном трансляционная стоп рамка eYFP чтения в позиции 120 (mTurquoise2-eYFP * 40) (Рисунок 2)10. В общей сложности 126 растения арабидопсис были превращается (9 растения в горшок, 14 горшки). Семена этих растений были объединены, посеяли на поддоны с почвой и позволено расти за две недели до начала лечения раствором глюфосинату аммония. Только саженцы, выражая бар Джин (присутствует в BIBAC-бар-GW) выжить глюфосинату аммония лечения (рис. 3). В общей сложности были определены 11 мутация преобразована с pDM19, соответствующее преобразование эффективности 0,02% семян проанализированы.
Для 11 мутация изолированные ДНК промокательной был использован для определения количества Т-ДНК интеграций. Для этой цели геномной ДНК был сокращен с BglII или Scaя (стратегия разработана на рисунке 4A). Оба этих энзимов ограничения только один раз разрезать на T-ДНК последовательности (рис. 7A). Гибридизации с зондами, признавая бар и eYFP, кодирующие области допускается определение числа соответствующих фрагментов ДНК.
Количество отдельных ДНК фрагментов на помарки, допускается для оценки количества вставок T-ДНК в линии репортер (Таблица 1). Один гибридизировать фрагментов с бара и eYFP зонда указано наличие единого интеграции T-ДНК. От 11 мутация проанализированы шесть осуществляется одной интеграций. Среднее количество внедрений был 1.2.
Для 6 линий, перевозящих один интеграции T-ДНК целостность конструкции вставленной репортер был испытан с использованием ДНК промокательной (стратегия разработана на рисунке 4В). Геномная ДНК был сокращен с BglII и Pciя выпустить 5,5 КБ фрагмент, содержащий Джин фьюжн бар и mTurquoise-eYFP (рис. 7A). Зонд против eYFP был использован для определения ожидаемого фрагмент. Все заводы испытания перевозимых фрагмент нетронутыми. Обратите внимание, что фрагмент изучены исключает слева и право T-ДНК границы и поэтому не проверяет целостность всего T-ДНК, но только часть, содержащая трансгенов интерес.
Экспрессии гена флуоресцентные репортер был определен в независимых поштучными трансгенных линий только отличающихся геномной расположение T-ДНК. Относительное стенограммы уровни репортер инициативе промоутер mTurquoise-eYFP CaMV 35S были измерены по RT-ПЦР в четыре линии репортер DM19, перевозящих нетронутыми, одной копии интеграций, из которых геномные позиция была определены10. Изменения в уровнях выражения гена репортера между линиями был незначительным: максимальная разница в уровнях РНК mTurquoise-eYFP был в 2 раза (рис. 8A).
Далее в эти линии репортер была проведена ODM. Три из четырех линий независимым репортером показали довольно аналогичные ODM эффективности (Рисунок 8B). Однако, одна линия, DM19 [4] 1, принесли весьма низкая эффективность ODM, по сравнению с другими линиями. Эти результаты показывают, что ODM зависит от местных геномной контекста. Каким образом местные геномной контексте интеграции T-ДНК в DM19 [4] 1 отличается от других линий предстоит определить. Анализ доступных наборов знаков активных и неактивных хроматина в геномной местах интеграции T-ДНК в не трансгенных растений не представила ответ10.

Рисунок 1: функциональные карты pBIBAC-GW векторов. pBIBAC-GW производные доступны либо сопротивление к глюфосинату (бар) или DsRed флуоресценции в семя пальто (DsRed) как маркер выделения в растениях. Для обоих векторов канамицин сопротивления ген представляет маркер выделения бактерий. Шлюз, ccdB кассеты отображается между зеленых стрелок, представляющие рекомбинации сайтов attR1 и attR2. Пожалуйста, нажмите здесь, чтобы посмотреть большую версию этой фигуры.

Рисунок 2: конструкция репортер мутагенеза. MTurquoise-eYFP репортер гены движет promotor 35S CaMV. MTurquoise кодирования региона сливается с eYFP, кодирование региона перевозящих C-A мутации в нуклеотидной позиции 120, что приводит к преждевременной остановки трансляционная кодон TAA и досрочное прекращение перевода синтез белка. 3′ Nopaline синтетазы (3' nos) сплайсингу сигнал используется для завершения транскрипции конструкции22. Сигнал ядерной локализации (NLS) используется для переведенных белков в ядро. Пожалуйста, нажмите здесь, чтобы посмотреть большую версию этой фигуры.

Рисунок 3: лоток заполнен Arabidopsis саженцев до и после лечения глюфосинату аммония. Саженцы, не выражая бар ген, который присутствует в pBIBAC бар-GW T-ДНК умирают после опрыскивают раствором глюфосинату аммония. Фотографии показывают же лоток для рассады (A) перед распылением с глюфосинату аммония, через 14 дней после посева и (B) 10 дней спустя, после распыляется дважды. Пожалуйста, нажмите здесь, чтобы посмотреть большую версию этой фигуры.
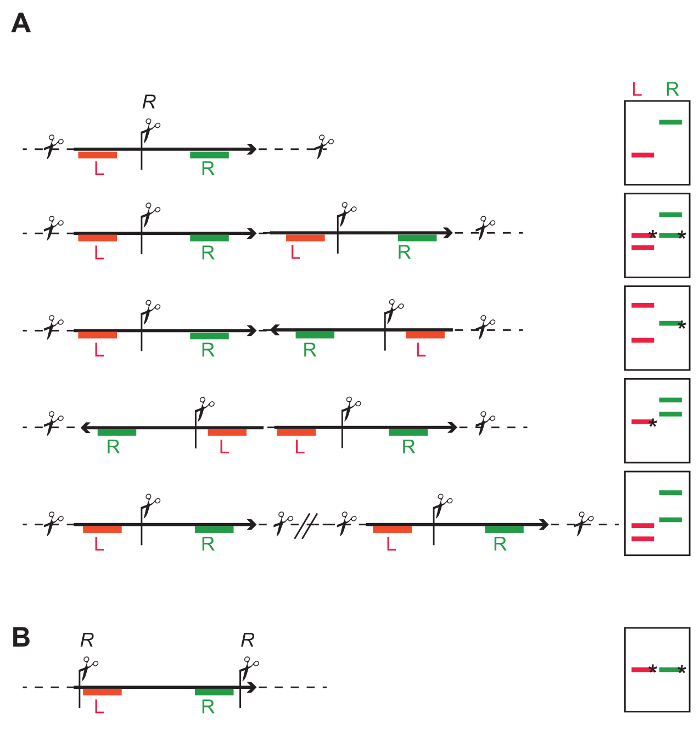
Рисунок 4: стратегии ограничения общего ДНК для определения числа и целостности из вставлены T-УНУ. (A) одно ограничение сайта (R) в середине T-ДНК позволяет независимым зондирующего слева (красный Л) и правой частью T-ДНК (зеленый R). Мультфильмы о праве показывают, что в зависимости от интеграции с одно - или multi - копировать T-ДНК, различных диапазонов узоры получаются с промокательной ДНК. Полос, отмеченные * имеет определенной длины, в то время как продолжительность других групп зависит от ближайшего сайта ограничение фланкируя геномной ДНК. Одной вставки: L и R зонд оба дают один независимый фрагмент. Размер ожидаемых среднем фрагментов может рассчитываться на основе на частоте ограничение сайта в геноме. Минимальный размер — это расстояние от места ограничения на левой границе (LB) или правой границе (РБ), в зависимости от чего интеграции является быть исследован, и если Т-ДНК нетронутыми. Тандеме повторить: Зонды для L и R дают оба двух фрагментов; для каждого датчика, один из фрагментов включает фланговые геномной ДНК второй фрагмент имеет ожидаемый размер и идентифицируется как зонды. Инвертированный повторить: В зависимости от направленности комплексной кассету один L и два R фрагментов, или два L и один R могут быть определены. Отдельных одного вставки: Результатом является число независимых фрагментов, и количество фрагментов соответствует числу внедрений. (B) ограничения сайтов на концах T-ДНК позволяет определить целостность фрагмента между места ограничения. Пожалуйста, нажмите здесь, чтобы посмотреть большую версию этой фигуры.
Рисунок 5: агарозном геле с ограничением шаблона и соответствия прозрачности. (A) на геле агарозы дна genomic переваривается с EcoRя показано. Правильное пищеварение ДНК свидетельствует наличие дискретных спутниковой полосы. (B) Маркировка позиции слотов и маркер полос на прозрачности делает возможным позже легко вычислить размер гибридизировать фрагментов. Здесь используются маркеры MRC Голландии (синий и красный), обозначается м. пожалуйста, нажмите здесь, чтобы посмотреть большую версию этой фигуры.
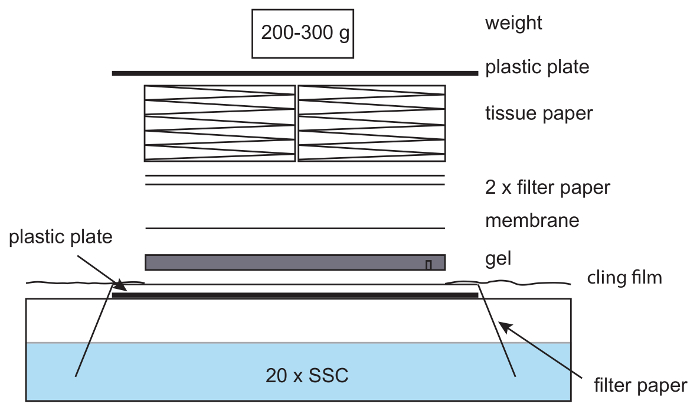
Рисунок 6: Настройка для капиллярной blotting. В капилляр промокательной установки фильтровальная бумага помещается на пластиковой пластине с конца бумаги, висит в 20 x SSC буфера. Бумага увлажненный с 20 x SSC и геля агарозы, размещен на вершине, следуют мембраны нейлона, фильтровальная бумага и стек тканей. Легкий вес находится на вершине. Осторожность для удаления пузырьков воздуха между гель, бумага и мембраны. Цепляться пленки используется во избежание высыхания установки. Пожалуйста, нажмите здесь, чтобы посмотреть большую версию этой фигуры.
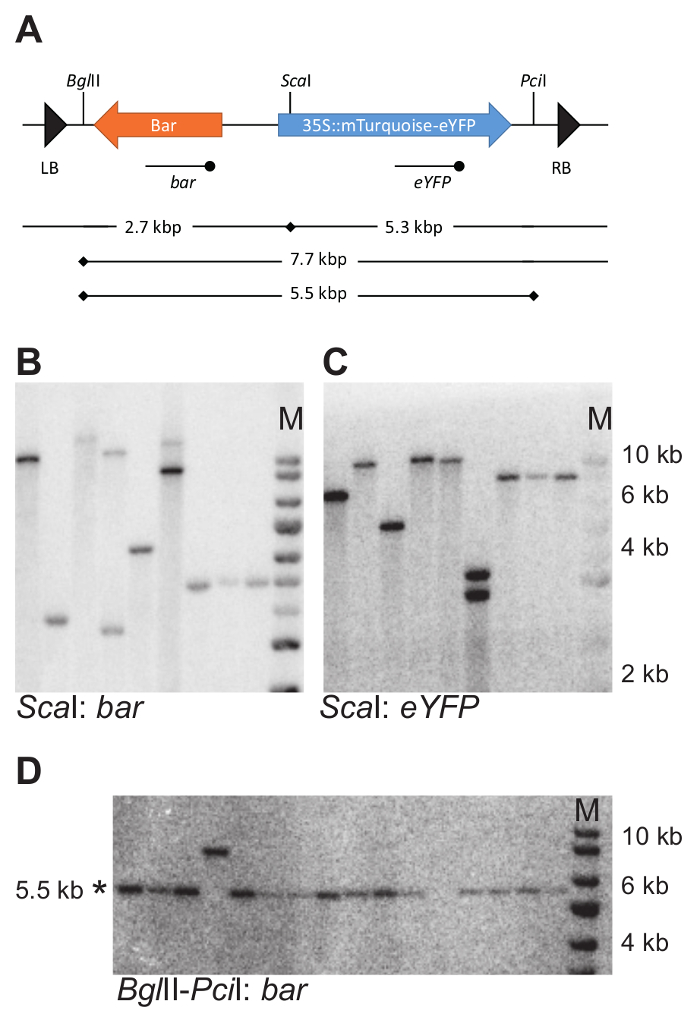
Рисунок 7: пример ДНК промокательной стратегии и экспериментальных результатов. (A) ДНК промокательной стратегии для определения числа и целостности T-ДНК интеграций. Резка местоположения выбранного энзимов ограничения в T-ДНК указаны с вертикальной чертой. EYFP и бар датчики, используемые для гибридизации с переваривается геномной ДНК, указаны с помощью строки с терминала точкой ниже T-ДНК. (DB–) Пример ДНК помарок. Геномная ДНК был сокращен с Scaя и пятно был исследован с бар и eYFP зонда (B и C). Геномная ДНК был вырезать с BglII и Pciя и исследован с зондом бар . Нетронутыми фрагменты являются 5,5 kbp в размер (D). Обратите внимание, что набор образцов в D отличается от указанных в B и C. * указывает размер ожидаемого фрагментов; М, маркер. В B, C и D используется же размер маркера. Пожалуйста, нажмите здесь, чтобы посмотреть большую версию этой фигуры.
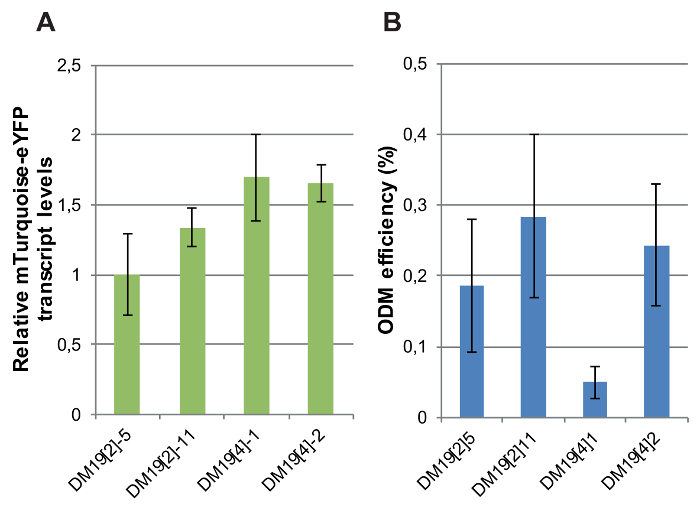
Рисунок 8: уровни выражения mTurquoise-eYFP и ODM эффективности линии репортер независимых mTurquoise-eYFP. (A) относительная mTurquoise-eYFP Стенограмма уровнях измеряется RT-ПЦР в DM19 репортер линий. Для нормализации Стенограмма уровни актина были использованы. (B) ODM эффективность измеряется в линиях репортер DM19. Для A и B, бары указывают в среднем по крайней мере пять биологических реплицирует. Планки погрешностей указывают SEM. пожалуйста, нажмите здесь, чтобы посмотреть большую версию этой фигуры.
| Тип T-ДНК Локус | Количество внедрений T-ДНК | Репортер линия | Количество обнаруженных фрагментов | Целостность | |||
| SCA Я | BGL II | BGL II/PciI | |||||
| Бар | eYFP | Бар | eYFP | eYFP | |||
| Интеграция в одном локусе | 1 | 19 [2] -2 | 1 | 1 | 1 | 1 | + |
| 1 | 19 [2] -5 | 1 | 1 | 1 | 1 | + | |
| 1 | 19 [2] -9 | 1 | 1 | 1 | 1 | + | |
| 1 | 19 [2] -11 | 1 | 1 | 1 | 1 | + | |
| 1 | 19 [4] -1 | 1 | 1 | 1 | 1 | + | |
| 1 | 19 [4] -2 | 1 | 1 | 1 | 1 | + | |
| 2, Перевернутый повторить | 19 [2] -10 | 2 | 1 | 1 | 1 | + | |
| 2, неполной интеграции | 19 [2] -3 | 1 | 2 | 1 | 1 | ND | |
| Несколько Локус интеграций | 2 | 19 [2] -6 | 2 | 2 | 2 | ND | |
| 2 | 19 [2] -7 | 2 | 2 | 2 | 2 | ND | |
| 3/4 | 19 [2] -1 | 4 | 3 | 3 | 3 | ND | |
| ND — не определен. | |||||||
Таблица 1: Резюме промокательной данных для изоляции после преобразования с pDM19 мутация ДНК.
Обсуждение
Решающее значение для генерации мутация с одной, нетронутыми интеграций трансген – это выбор бинарный вектор, используемый. BIBAC семьи векторов были использованы для доставки последовательности интересов многих растений видов23,24,25,26,27,28. Векторные BIBAC, включая BIBAC-GW, доходность поштучными интеграций с высокой эффективностью: среднее количество вставок в строке составляет 1,5-2, по сравнению с 3 или выше для наиболее часто используемых бинарных векторов5,9, 29. как значительное улучшение по сравнению с другими BIBAC векторов, с BIBAC-GW векторов, последовательности интерес может быть легко вставлен с помощью шлюза рекомбинации сайты12. Изменение вектора преодолеть общие проблемы BIBAC векторов при использовании обычных клонирования стратегии: i очень ограниченное количество уникальных ограничений сайтов и ii) ДНК низкой урожайности. Шлюз рекомбинации сайтов сделать BIBAC-GW векторов привлекательной альтернативой для других бинарных векторов для создания трансгенных растений.
Здесь описан ряд протоколов, от генерации BIBAC-GW производные, содержащими последовательности, интерес, к заводе преобразования и ДНК блот анализ числа и целостности трансгенных последовательностей. Несколько протоколов сообщили в этом документе, шлюз клонирования, электропорация бактерий, и трансформации растений, являются распространенной практикой во многих лабораториях и может также осуществляться с незначительными изменениями. Важно знать, что BIBAC-GW — это вектор одной копии в E. coli и A. tumefaciens. Таким образом при изоляции ДНК, урожайность низкая; рекомендуется активизировать процесс изоляции.
Мутация, перевозящих несколько интеграции T-ДНК введено трансгенов часто подвергаются1,2,4,30экспрессию гена и поэтому для большинства приложений следует избегать. Для выявления трансгенных растений с одной, нетронутыми интеграций, рекомендуется использовать ДНК анализ помаркой. Хотя методы, отличные от ДНК промокательной может использоваться для определения числа копии T-ДНК и целостность T-ННО в трансгенных линий (сегрегации анализ, хвост-PCR, Количественная ПЦР (ПЦР) и цифровой капелька ПЦР), хотя трудовой, интенсивный, ДНК промокательной часто является метод выбора. Сегрегации анализ не способен различать между несколькими и интеграции T-ДНК в одной локусов. ХВОСТ-PCR часто недостаточной оценки копия номер, особенно если более чем один Т-ДНК интеграция является настоящей31, и ПЦР необходимо разработать оптимизации для надежные результаты31,32. Цифровые капелька ПЦР является довольно точный метод для обнаружения копии номер если требуемое оборудование доступны31. Дополнительным преимуществом промокательной ДНК является поверхностным обнаружения усеченного T-ННО, которая легко пропустили всех методов на основе ПЦР.
С ДНК анализ помаркой гибридизировать фрагментов на пятно нужно быть хорошо идентифицируемой в сигнал и размер. Известно, что несколько факторов влияет на результат промокательной ДНК. Помимо надлежащего подбора энзимов ограничения (стратегия указано на рис. 4) и размер маркеров, достаточно ДНК хорошего качества не требуется. Менее 2 мкг геномной Arabidopsis ДНК не даст четко выраженные фрагментов. Когда имеешь дело с больших геномов, требуется больше ДНК. Для получения достаточного количества Arabidopsis ДНК, можно использовать цветочные ткани или 1 - неделя старый саженцев. Пакет сеянцев, выращенных на одной чашке Петри дает 2-8 мкг ДНК. Чтобы избежать деградации ДНК при изоляции, следует позаботиться обработать растительный материал быстро. Кроме того, геномной ДНК должны быть высокомобильна в трис-ЭДТА для уменьшения ее деградации, nucleases и хранить при 4 ° C вместо 20 ° C до предотвращения ДНК уменьшение поперечного сечения из-за повторил циклов замораживания оттаивания. Если вы не уверены, что все образцы ДНК полностью усваиваются, предлагается rehybridize ДНК пятно с зондом, признавая эндогенные, уникальные геномной региона. При выборе последовательности зонда для определения последовательности трансгенных или эндогенных, важно выбрать только уникальных последовательностей. Чтобы иметь возможность точно определить размер гибридизированные фрагментов, позиции ДНК гель слотов и ДНК маркер полосы должны быть помечены на прозрачности (Рисунок 5B) при визуализации бромид Ethidium-окрашенных гель (Рисунок 5A) на УФ transilluminator. В случае, если маркер последовательности не гибридизируйте с зонд ДНК, или гибридизации частичной, это единственный способ, позволяющий отслеживать вниз размер гибридизировать фрагментов.
После того, как осторожность для достижения хорошей гибридизации сигнал и оценить размер фрагмента, интерпретация результатов blotting проста. При использовании только один энзима ограничения и гибридизировать с различными зонды обнаружения либо в левой или правой части Т-ДНК, количество обнаруженных фрагментов отражает количество вставок T-ДНК. Например, Рисунок 7B, C показывает же ДНК помаркой, гибридизированных с различными зондами, бар (рис. 7B) и расширенной желтый флуоресцентный белок (eYFP) (рис. 7 c), используя стратегию, показано на рис. 7A. Все полосы, за исключением 4, показывают одинаковое количество фрагментов на обоих помарки: два фрагмента для линии 6 и единый фрагмент для всех других линий. Это количество обнаруженных фрагментов — количество вставок T-ДНК.
Когда количество фрагментов обнаружены с зондами привязки либо левой или правой частью T-ДНК отличается (что касается Рисунок 7BC, строка 4), либо неполной вставки присутствуют, или T-ННО вставили в тандем. Тандем вставки отображения длина-случайный фрагмент одного из фрагментов T-ДНК (рис. 4A, правая панель) и можно определить путем сравнения размер гибридизированные фрагментов с того, что ожидается, основанный на стратегии ограничения. Дополнительного слоя ватмановской стратегия может быть необходимо подтвердить тандем T-ДНК вставок. В образце показано на линии 4 (рис. 7B, C) двух вставок расположены в Перевернутый повторить ориентации.
При оценке целостности T-ДНК, или часть его, длина фрагмента гибридизировать может быть рассчитываются на основе стратегии ограничения. Любое отклонение от ожидаемого размера указывает на наличие неполной вставки. К примеру в Рисунок 7 d, в переулок 4, фрагмент гибридизировать мигрирует на 8, kbp (вместо ожидаемого 5.5 kbp) указанием размера увеличение фрагмента из-за отсутствия одного из места ограничения.
BIBAC-GW векторы являются отличными инструментами для создания одной копии нетронутыми внедрений в ряде видов растений. Протокол, сообщили здесь обеспечивает надежные процедуры для выявления растений с одной, нетронутыми интеграций трансген интерес.
Раскрытие информации
Авторы заявляют, что без финансовых интересов или другие конфликты интересов.
Благодарности
Это исследование поддерживается голландской технологии фонд STW (12385), которая является частью Нидерландской организации научных исследований (НВО), и которая частично финансируется министерства экономических дел (Грант 12385 OTP для MS). Мы благодарим Кэрол м. Гамильтон (Корнельский университет, Соединенные Штаты) за предоставление pCH20, костяк BIBAC-GW векторов.
Материалы
| Name | Company | Catalog Number | Comments |
| Kanamycin sulphate monohydrate | Duchefa | K0126 | |
| Gentamycin sulphate | Duchefa | G0124 | |
| Rifampicin | Duchefa | R0146 | |
| Tetracycline hydrochloride | Sigma | T-3383 | |
| DB3.1 competent cells | Thermo Scientific - Invitrogen | 11782-018 | One Shot ccdB Survival 2 T1R Competent Cells (A10460) by Invitrogen or any other ccdB resistant E. coli strain can be used instead |
| DH10B competent cells | Thermo Scientific - Invitrogen | 18290-015 | |
| Gateway LR clonase enzyme mix | Thermo Scientific - Invitrogen | 11791-019 | |
| tri-Sodium citrate dihydrate | Merck | 106432 | |
| Trizma base | Sigma-Aldrich | T1503 | |
| EDTA disodium dihydrate | Duchefa | E0511 | |
| Proteinase K | Thermo Scientific | EO0491 | |
| Bacto tryptone | BD | 211705 | |
| Yeast extract | BD | 212750 | |
| Sodium chloride | Honeywell Fluka | 13423 | |
| Potassium chloride | Merck | 104936 | |
| D(+)-Glucose monohydrate | Merck | 108346 | |
| Electroporation Cuvettes, 0.1 cm gap | Biorad | 1652089 | |
| Electroporator Gene Pulser | BioRad | ||
| Magnesium sulfate heptahydrate | Calbiochem | 442613 | |
| D(+)-Maltose monohydrate 90% | Acros Organics | 32991 | |
| Sucrose | Sigma-Aldrich | 84100 | |
| Silwet L-77 | Fisher Scientific | NC0138454 | |
| Murashige Skoog medium | Duchefa | M0221 | |
| Agar | BD | 214010 | |
| Glufosinate-ammonium (Basta) | Bayer | 79391781 | |
| Restriction enzymes | NEB | ||
| Ethidium Bromide | Bio-Rad | 1610433 | |
| Electrophoresis system | Bio-Rad | ||
| Sodium hydroxide | Merck | 106498 | |
| Hydrochloric acid | Merck | 100316 | |
| Blotting nylon membrane Hybond N+ | Sigma Aldrich | 15358 | or GE Healthcare Life Sciences (RPN203B) |
| Whatman 3MM Chr blotting paper | GE Healthcare Life Sciences | 3030-931 | |
| dNTP | Thermo Fisher | R0181 | |
| Acetylated BSA | Sigma-Aldrich | B2518 | |
| HEPES | Sigma-Aldrich | H4034 | |
| 2-Mercaptoethanol | Merck | 805740 | |
| Sephadex G-50 Coarse | GE Healthcare Life Sciences | 17004401 | or Sephadex G-50 Medium (17004301) |
| Dextran sulfate sodium salt | Sigma-Aldrich | D8906 | |
| Sodium Dodecyl Sulfate | US Biological | S5010 | |
| Salmon Sperm DNA | Sigma-Aldrich | D7656 | |
| Sodium dihydrogen phosphate monohydrate | Merck | 106346 | |
| Storage Phosphor screen and casette | GE Healthcare Life Sciences | 28-9564-74 | |
| Phosphor imager | GE Healthcare Life Sciences | Typhoon FLA 7000 | |
| UV Crosslinker | Stratagene | Stratalinker 1800 | |
| cling film (Saran wrap) | Omnilabo | 1090681 | |
| Agarose | Thermo Scientific - Invitrogen | 16500 | |
| Boric acid | Merck | 100165 | |
| DNA marker ‘Blauw’; DNA ladder. | MRC Holland | MCT8070 | |
| DNA marker ‘Rood’; DNA ladder | MRC Holland | MCT8080 | |
| Hexanucleotide Mix | Roche | 11277081001 | |
| Large-Construct Kit | Qiagen | 12462 | |
| Heat-sealable polyethylene tubing, clear | various providers | the width of the tubing should be wider than that of blotting membrane | |
| Heat sealer | |||
| Membrane filter disk | Merck | VSWP02500 | |
| Magnesium chloride | Merck | 105833 | |
| Hybridization mesh | GE Healthcare Life Sciences | RPN2519 |
Ссылки
- Jorgensen, R. A., Cluster, P. D., English, J., Que, Q., Napoli, C. A. Chalcone synthase cosuppression phenotypes in petunia flowers: comparison of sense vs. antisense constructs and single-copy vs. complex T-DNA sequences. Plant Mol Biol. 31 (5), 957-973 (1996).
- Stam, M., et al. Post-transcriptional silencing of chalcone synthase in Petunia by inverted transgene repeats. Plant J. 12, 63-82 (1997).
- Stam, M., Viterbo, A., Mol, J. N., Kooter, J. M. Position-dependent methylation and transcriptional silencing of transgenes in inverted T-DNA repeats: implications for posttranscriptional silencing of homologous host genes in plants. Mol Cell Biol. 18 (11), 6165-6177 (1998).
- Jin, Y., Guo, H. S. Transgene-induced gene silencing in plants. Methods Mol Biol. 1287, 105-117 (2015).
- Oltmanns, H., et al. Generation of Backbone-Free, Low Transgene Copy Plants by Launching T-DNA from the Agrobacterium Chromosome 1[W][OA]. Plant Physiol. , (2010).
- Ye, X., et al. Enhanced production of single copy backbone-free transgenic plants in multiple crop species using binary vectors with a pRi replication origin in Agrobacterium tumefaciens. Transgenic Res. , (2011).
- Hamilton, C. M. A binary-BAC system for plant transformation with high-molecular-weight DNA. Gene. 200 (1-2), 107-116 (1997).
- Frary, A., Hamilton, C. M. Efficiency and stability of high molecular weight DNA transformation: an analysis in tomato. Transgenic Res. 10 (2), 121-132 (2001).
- Vega, J. M., et al. Agrobacterium-mediated transformation of maize (Zea mays) with Cre-lox site specific recombination cassettes in BIBAC vectors. Plant Mol Biol. 66 (6), 587-598 (2008).
- Anggoro, D. T., Tark-Dame, M., Walmsley, A., Oka, R., de Sain, M., Stam, M. BIBAC-GW-based vectors for generating reporter lines for site-specific genome editing in planta. Plasmid. 89, 27-36 (2017).
- Hamilton, C. M., Frary, A., Lewis, C., Tanksley, S. D. Stable transfer of intact high molecular weight DNA into plant chromosomes. Proc Natl Acad Sci U S A. 93 (18), 9975-9979 (1996).
- Belele, C. L., Sidorenko, L., Stam, M., Bader, R., Arteaga-Vazquez, M. A., Chandler, V. L. Specific tandem repeats are sufficient for paramutation-induced trans-generational silencing. PLoS Genet. 9 (10), e1003773 (2013).
- Shizuya, H., et al. Cloning and stable maintenance of 300-kilobase-pair fragments of human DNA in Escherichia coli using an F-factor-based vector. Proc Natl Acad Sci U S A. 89 (18), 8794-8797 (1992).
- Shi, X., Zeng, H., Xue, Y., Luo, M. A pair of new BAC and BIBAC vectors that facilitate BAC/BIBAC library construction and intact large genomic DNA insert exchange. Plant Methods. 7, 33 (2011).
- Woodman, M. E., et al. Direct PCR of Intact Bacteria (Colony PCR). Curr Protoc Microbiol. , A.3D.1-A.3D.7 (2016).
- Ausubel, F. M., et al. Mol Biol. Current Protocols in Molecular Biology. 1, (2003).
- Edwards, K., Johnstone, C., Thompson, C. A simple and rapid method for the preparation of plant genomic DNA for PCR analysis. Nucleic Acids Res. 19 (6), 1349 (1991).
- Clarke, J. D. Cetyltrimethyl ammonium bromide (CTAB) DNA miniprep for plant DNA isolation. Cold Spring Harb Protoc. (3), (2009).
- Lee, P. Y., Costumbrado, J., Hsu, C. Y., Kim, Y. H. Agarose Gel Electrophoresis for the Separation of DNA Fragments. J Vis Exp. (62), e3923 (2012).
- Green, M. R., Sambrook, J. . Molecular Cloning. , (2012).
- Sosa, J. M. Chromatography with Sephadex Gels. Anal Chem. 52 (6), 910-912 (1980).
- Depicker, A., Stachel, S., Dhaese, P., Zambryski, P., Goodman, H. M. Nopaline synthase: transcript mapping and DNA sequence. J Mol Appl Genet. 1 (6), 561-573 (1982).
- Feng, J., Vick, B. A., Lee, M. K., Zhang, H. B., Jan, C. C. Construction of BAC and BIBAC libraries from sunflower and identification of linkage group-specific clones by overgo hybridization. Theor Appl Genet. 113 (1), 23-32 (2006).
- Lee, M. K., et al. Construction of a plant-transformation-competent BIBAC library and genome sequence analysis of polyploid Upland cotton (Gossypium hirsutum L). BMC Genomics. 14, 208 (2013).
- Wang, W., et al. A large insert Thellungiella halophila BIBAC library for genomics and identification of stress tolerance genes. Plant Mol Biol. 72 (1-2), 91-99 (2010).
- Wu, C., et al. A BAC- and BIBAC-based physical map of the soybean genome. Genome Res. 14 (2), 319-326 (2004).
- Xu, Z., et al. Genome physical mapping from large-insert clones by fingerprint analysis with capillary electrophoresis: a robust physical map of Penicillium chrysogenum. Nucleic Acids Res. 33 (5), e50 (2005).
- Zhang, M., et al. Genome physical mapping of polyploids: a BIBAC physical map of cultivated tetraploid cotton, Gossypium hirsutum L. PLoS One. 7 (3), e33644 (2012).
- Frary, A., Hamilton, C. M. Efficiency and stability of high molecular weight DNA transformation: An analysis in tomato. Transgenic Res. , (2001).
- Stam, M., Viterbo, A., Mol, J. N. M., Kooter, J. M. Position-Dependent Methylation and Transcriptional Silencing of Transgenes in Inverted T-DNA Repeats: Implications for Posttranscriptional Silencing of Homologous Host Genes in Plants. Cell Biol. 18 (11), 6165-6177 (1998).
- Głowacka, K., Kromdijk, J., Leonelli, L., Niyogi, K. K., Clemente, T. E., Long, S. P. An evaluation of new and established methods to determine T-DNA copy number and homozygosity in transgenic plants. Plant Cell Environ. 39 (4), 908-917 (2016).
- Stefano, B., Patrizia, B., Matteo, C., Massimo, G. Inverse PCR and Quantitative PCR as Alternative Methods to Southern Blotting Analysis to Assess Transgene Copy Number and Characterize the Integration Site in Transgenic Woody Plants. Biochem Genet. 54 (3), 291-305 (2016).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеThis article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены