Method Article
Generierung von transgenen Pflanzen mit Single-Copy-Einschübe mit binären Vektor BIBAC-GW
In diesem Artikel
Zusammenfassung
Mit einem pBIBAC-GW binären Vektor macht Erzeugung transgene Pflanzen mit intakten Single Copy Einfügungen, ein einfacher Vorgang. Hier präsentiert eine Reihe von Protokollen, die führen des Lesers durch den Prozess der Generierung von transgenen Pflanzen in Arabidopsis und Versuchsanlagen für Unversehrtheit und Anzahl der Einsätze zu kopieren.
Zusammenfassung
Beim Generieren von transgenen Pflanzen ist in der Regel das Ziel, stabile Expression ein Transgen zu haben. Dies erfordert eine einheitliche, intakte Integration des Transgens, Multi-Kopie-Integrationen häufig Gen-silencing ausgesetzt sind. Der Gateway-kompatiblen binären Vektor basierend auf bakterielle künstliche Chromosomen (pBIBAC-GW), wie andere pBIBAC-Derivate, ermöglicht das Einfügen von Single Copy transgene mit hohem Wirkungsgrad. Als eine Verbesserung der ursprünglichen pBIBAC hat eine Gateway-Kassette in pBIBAC-GW, geklont worden, so dass die Sequenzen von Interesse jetzt leicht in den Vektor Transfer DNA (T-DNA) durch Klonen Gateway integriert werden können. Die Transformation mit pBIBAC-GW wird häufig, einen Wirkungsgrad von 0,2 – 0,5 %, wobei die Hälfte der gentechnisch veränderten Pflanzen eine intakte Single Copy-Integration der T-DNA tragen. Die pBIBAC-GW-Vektoren sind mit Resistenz gegenüber Glufosinat-Ammonium oder DsRed Fluoreszenz in Samenhüllen in Pflanzen zur Auswahl und mit einer Resistenz gegen Kanamycin als Auswahl in Bakterien. Hier ist eine Reihe von Protokollen, die der Leser durch den Prozess der Erzeugung transgener Pflanzen mit pBIBAC-GW präsentiert: ausgehend von Rekombination die Sequenzen von Interesse in der pBIBAC-GW-Vektor der Wahl, um die Transformation mit Pflanzen Agrobacterium, Auswahl der gentechnisch veränderten Pflanzen und Versuchsanlagen für Unversehrtheit und Kopie Zahl der Einsätze mit DNA zu beflecken. Aufmerksamkeit gilt beim Entwerfen einer DNA-befleckenden Strategie, Single und copy Integrationen auf einzelne und mehrere Loci zu erkennen.
Einleitung
Beim Generieren von transgenen Pflanzen ist in der Regel das Ziel, die integrierte transgene stabil ausgedrückt zu haben. Dies kann durch intakte Einzelkopie Integrationen von ein Transgen. Mehrfachintegrationen können zu erhöhten Ausdruck ein Transgen, sondern auch Gen-silencing führen. Der transgene Silencing ist wahrscheinlicher, wenn die eingefügte Sequenzen in Tandem- oder invertierte Wiederholungen1,2,3,4angeordnet sind. Binäre Vektoren dienen als Shuttles in Agrobakterium-vermittelten Umwandlung Experimente durchführen, um die Sequenzen von Interesse in Pflanzengenomen liefern. Die Zahl der Integrationen in ein pflanzliches Genom ist abhängig von der Kopienzahl des binären Vektor in Agrobacterium Tumefaciens5,6. Viele häufig verwendete binäre Vektoren sind hohe Kopie Vektoren, und daher ergeben eine hohe durchschnittliche Transgen Exemplarzahl: 3,3 bis 4,9 Kopien in Arabidopsis5.
Die Anzahl der T-DNA Integrationen kann mithilfe von binäre Vektoren, bei denen eine niedrige Kopienzahl in A. Tumefaciens, z. B. BIBAC7oder durch die Einführung einer T-DNS von A. Tumefaciens Chromosom5gesenkt werden. Die durchschnittliche Anzahl der Transgen-Integrationen in solchen Fällen liegt unter 25,8,9,10. Durch die Single Copy in A. Tumefaciens, und auch in Escherichia coli, BIBAC-Derivate erhalten und Konstrukte, die so groß wie 150 kb11liefern können.
GW-kompatible BIBAC Vektoren10,12 erlauben einfache Einführung von Genen von Interesse in den Vektor durch Gateway zu klonen. Die Verwendung von Gateway-Technologie vereinfacht das Klonen Verfahren, aber auch häufige Probleme im Zusammenhang mit großen niedrige Kopienzahl Vektoren13,14, wie eine geringe DNA-Ausbeute und eine begrenzte Auswahl an einzigartigen Beschränkung überwindet Standorte für das Klonen7,11. Die pBIBAC-GW-Derivate sind entweder Widerstand zu Glufosinat-Ammonium (pBIBAC-BAR-GW) oder DsRed Fluoreszenz in Samenhüllen (pBIBAC-RFP-GW) in Pflanzen (Abbildung 1)10,12zur Auswahl zur Verfügung. Für beide Vektoren ist eine Kanamycin-Resistenz-Gen als Selektionsmarker in Bakterien verwendet.
Kombinieren Sie die pBIBAC-GW-Vektoren: (1) einfache Design und Genmanipulation in E. Coli, und (2) intakt Single Copy Integrationen in Planta bei hohem Wirkungsgrad. Die pBIBAC-GW Vektoren Rendite durchschnittlich 1,7 Integrationen in Arabidopsis mit etwa der Hälfte der transgenen Pflanzen mit einer einzigen integrierten T-DNA10.
Stabile Expression von transgenen ist eine Voraussetzung für die meisten gentechnisch veränderten Pflanzen erzeugt. Stabile Transgene Ausdruck kann durch intakte, Single Copy Integrationen erreicht werden. Arbeiten mit transgenen Pflanzen tragen intakte, Single Copy Integrationen ist jedoch noch wichtiger, wenn zum Beispiel das Ziel, die Effizienz der Chromatin-basierte Prozesse wie Mutagenese, Rekombination, oder Reparatur und die Abhängigkeit von diesen zu studieren Prozesse der Chromatinstruktur an der Insertionsstelle und genomische Position. Für unser Interesse um die Abhängigkeit des Oligonukleotids gerichtet Mutagenese (ODM) auf den lokalen genomische Kontext zu untersuchen war eine Reihe von Reporter Linien mit intakten, Single Copy Integrationen von ein Reportergen Mutagenese erzeugte (Abbildung 2)10. Mit diesem Satz von Linien, zeigte sich, dass die ODM-Effizienz variiert zwischen transgenen Loci integriert genomische Standorten trotz der Transgene Ausdruck ziemlich ähnlich.
Protokoll
1. Einfügen von Sequenzen von Interesse in binären Vektor
-
Bereiten Sie den Gateway-Eintrag und binäre Vektoren.
- Isolieren Sie den Gateway Eintrag Vektor mit einer DNA-Fragment oder gen von Interesse, die mit einem Mini-Prep Kit entsprechend den Vorschlägen des Lieferanten.
Hinweis: BIBAC-GW Vektoren erfordern den Einsatz von Kanamycin (Km) zur Auswahl in Bakterien, daher, einen Eintrag Vektor mit einen weiteren Widerstand Marker anstelle von Kanamycin verwenden. Zum Beispiel ist die pENTR-gm-Vektor, Durchführung einer Gentamicin-Resistenz-Gen, eine gute Wahl-12. - Vermehren und isolieren des BIBAC-GW-Vektors von Interesse. Verwenden Sie eine E. Coli -Stamm, die resistent gegen die Toxizität des CcdB -Gens in der Gateway-Kassette. Isolieren BIBAC-Vektoren mit Protokolle oder Bausätze speziell für große Plasmide gemäss den Vorschlägen des Lieferanten.
- Isolieren Sie den Gateway Eintrag Vektor mit einer DNA-Fragment oder gen von Interesse, die mit einem Mini-Prep Kit entsprechend den Vorschlägen des Lieferanten.
-
Führen Sie eine Gateway-Reaktion.
- Bereiten Sie die LR Rekombination Reaktion nach den Vorschlägen des Lieferanten. Mischen Sie die folgenden Komponenten in einem 1,5 mL Microcentrifuge Schlauch bei Raumtemperatur (RT): 100 – 300 ng Eintrag Klon (supercoiled), 300 ng BIBAC-GW Vektors, LR Clonase Reaktion Puffer (Endkonzentration: 1 X). Stellen Sie die Lautstärke der Mischung zu 16 µL mit TE (10 mM Tris, 1 mM EDTA, pH 8,0). Schließlich fügen Sie 4 µL der LR Clonase Enzym-Mix und Mix durch aufschütteln. Inkubieren Sie die Mischung bei 25 ° C für 1 h.
- Beenden Sie die LR-Reaktion durch Zugabe von 2 µL Proteinase K-Lösung (2 µg/µL) auf die Mischung, die in Schritt 1.2.1 vorbereitet. Mischen und bei 37 ° C für 10 min inkubieren.
-
E. Coli mit dem Gateway Reaktionsgemisch durch Elektroporation zu verwandeln.
- Entsalzen Sie das LR Reaktionsgemisch vor Elektroporation.
Hinweis: Dieser Schritt ist entscheidend für erfolgreiche Elektroporation. Unter einer Dialyse Methode ist beschrieben, aber auch andere Methoden wie z. B. Niederschlag mit Natriumacetat und Vinylalkohol kann auch benutzt werden.- Bereiten Sie das Setup für Filter Dialyse der LR-Reaktion. Gießen Sie 20 mL entionisiertem Reinstwasser in eine sterile Petrischale. Legen Sie eine Membran-Filterscheibe (Porengröße = 0,025 µm) auf der Wasseroberfläche.
- Die gesamte LR Reaktion sorgfältig auf die Membran Pipette und lassen Sie die Mischung für 1 h bei RT Dialyse.
- Elektro-kompetente DH10B Zellen in eine Küvette Elektroporation (0,1 cm) 5 µL des entsalzten LR-Mix hinzufügen. Electroporate der Zellen (1,5 V/cm, Widerstand 200 Ω, Kapazität 25 µF), und fügen Sie 1 mL vorgewärmten Super optimale Catabolite Repression (SOC) Medium sofort zu den Zellen, gefolgt von Inkubation bei 37 ° C für 45 min, 180 u/min. (SOC-Medium, 1 L: 20 g Bacto Tryptone, 5 g Hefe-Extrakt, 0,5 g NaCl, 2,5 mL 1M KCl, 20 mL 1 M Filter-sterilisiert Glukose).
Hinweis: DH10B Zellen können anderen E. Coli -Zellen ersetzt werden, die große Plasmide stabil zu halten.
Hinweis: Die optimale Elektroporation Bedingungen sind abhängig von der Elektroporation Gerät verwendet. - Pellet-Bakterien bei maximaler Geschwindigkeit für 30 s mit einem Microcentrifuge entfernen überschüssige SOC und Aufschwemmen das Pellet in ca. 50-100 µL Medium Luria-Bertani (LB). Die Bakterien auf Kanamycin-LB (Km-LB) Platten (Km Konzentration, 40 µg/mL) zu verbreiten, und über Nacht die Platten bei 37 ° C inkubieren. (LB-Medium, 1 L: 10 g Bacto Tryptone, 5 g Hefe-Extrakt, 10 g NaCl. Fügen Sie für festen Medium Agar, 15 g/L) hinzu.
- Entsalzen Sie das LR Reaktionsgemisch vor Elektroporation.
-
Identifizieren Sie der rekombinierten BIBAC-GW-Derivate und isolieren Sie Plasmid DNA zu.
- Um auf Km-LB-Platten wachsen zu können, sollte die E. Coli -Zellen die neukombinierte Plasmid BIBAC-GW enthalten, in dem die CcdB -Sequenz mit dem gewünschten Einsatz ersetzt wird. Verwenden Sie Kolonie Polymerase-Kettenreaktion (PCR)15 zu überprüfen, ob die Bakterien-Kolonien auf der Platte die richtigen Plasmide enthalten, mit der BIBAC-GW-Backbone und den Einsatz von Interesse.
- Ermittlung der pBIBAC-BAR-GW-Rückgrat, durchführen eine PCR15 Reaktion mit dem Primer DM1969 5'-GCGACGAGCCAGGGATAG-3 'und DM1970 5'-ATCAGTGCGCAAGACGTGAC-3'. Diese Grundierung Satz verstärkt 563 bp Fragment des Bar -Gens.
- Um auf das Vorhandensein des BIBAC-RFP-GW-Backbones zu überprüfen, führen Sie eine PCR15 Reaktion, unter Verwendung der Primer M737 5'-CGTGTAAAAAGCTTAGACTG-3 'und M892 5'-AACAGATGGTGGCGTCCC-3'. Diese Grundierung Kombination verstärkt 791 bp Fragment Überlappung der Cruciferin-Promotor und Rfp -Sequenz.
- Führen Sie PCR15 Reaktionen durch Gen-spezifische Primer für das Vorhandensein des Einsatzes von Interesse zu überprüfen.
- Eine einzige positive Kolonie in 2 – 5 mL LB-Medium mit Kanamycin (40 µg/mL) für DNA-Isolierung16zu impfen. Inkubation bei 37 ° C auf einem Orbitalschüttler mit 180 u/min, über Nacht.
- Das Plasmid DNA zu isolieren (siehe Schritt 1.1.2).
- Um auf Km-LB-Platten wachsen zu können, sollte die E. Coli -Zellen die neukombinierte Plasmid BIBAC-GW enthalten, in dem die CcdB -Sequenz mit dem gewünschten Einsatz ersetzt wird. Verwenden Sie Kolonie Polymerase-Kettenreaktion (PCR)15 zu überprüfen, ob die Bakterien-Kolonien auf der Platte die richtigen Plasmide enthalten, mit der BIBAC-GW-Backbone und den Einsatz von Interesse.
2. Vorbereitung von A. Tumefaciens zum floralen Dippen von Arabidopsis
-
BIBAC-GW-Derivate, A. Tumefacienszu verwandeln.
- Bereiten Sie Elektro-kompetente Zellen von A. Tumefaciens Stamm C58C1 tragen die pCH32 Helfer Plasmid-7. Wachsen Sie Bakterien in Gegenwart von Tetracyclin (5 µg/mL) und Rifampicin (100 µg/mL), wählen für pCH32 und Wachstum von Agrobacterium -Zellen zu gewährleisten.
- Hinzufügen von 0,25-0,5 µg DNA eines pBIBAC-GW-Derivats, vorab in 10 – 20 µL steriler ultrapure deionisiertes Wasser, zu 20 µL kompetente Agrobacterium -Zellen in Elektroporation Küvetten (0,1 cm) aufgelöst. Halten Sie Zellen auf Eis.
- Electroporate Zellen (1,5 V/cm, Widerstand 400 Ω, Kapazität 25 µF). Unmittelbar nach der Elektroporation die Bakterien 1 mL vorgewärmten (28 ° C) SOC Medium hinzu und inkubieren Sie die Zellen bei 28 ° C für 60-90 min.
- Verbreiten Sie, 100 µL und der Rest der Bakterien auf separaten LB-Platten mit Rifampicin (100 µg/mL), Tetracyclin (5 µg/mL) und Kanamycin (40 µg/mL) und 1-2 Tage im Dunkeln bei 28 ° C inkubieren.
-
Bereiten Sie die Agrobacterium Suspension.
- Überprüfen Sie mittels PCR ein paar A. Tumefaciens Kolonien von der Platte in Schritt 2.1.4, auf das Vorhandensein des richtigen Vektors vorbereitet (siehe Schritt 1.4.1).
- Streifen eine einzige Kolonie bestätigte gegenüber den binären Vektor mit dem entsprechenden Einsatz auf eine LB-Platte mit Antibiotika (siehe Schritt 2.1.4) enthalten. Bei 28 ° C über Nacht wachsen.
- Wiederholen Sie die Streifen mit einer einzigen Kolonie in Schritt 2.2.2 erhalten.
- Eine einzige Kolonie in 2,5 mL LC Medium ergänzt mit Antibiotika (siehe Schritt 2.1.4) für Vorkultur zu impfen. Bei 28 ° C für mindestens 8 Stunden oder über Nacht, mit 180 u/min inkubieren. (LC Medium 1 L: 10 g Bacto Tryptone, 5 g Hefe-Extrakt, 0,5 g NaCl, 2,5 g MgSO4 · 7 H2O, 2 g Maltose).
- Fügen Sie die Vorkultur aus Schritt 2.2.4. 250 ml LC mit Antibiotika (siehe Schritt 2.1.4) ergänzt und bei 28 ° C bei 180 u/min, über Nacht wachsen.
- Pellets der Kultur von Spinnen bei 5.500 x g für 12 min. wieder auszusetzen das Pellet in 100 mL Lösung mit 5 % Saccharose, 0,05 % Silwet L-77, 0,5 x ms gießen die Aussetzung in einen sterilen Behälter für Blumen Eintauchen der Pflanzen.
(3) Arabidopsis Transformation
-
Umwandlung der Arabidopsis -Pflanzen vorbereiten.
- Wachsen Sie Arabidopsis -Pflanzen in einem Gewächshaus oder Klima kontrolliertes Wachstum, bis sie blühen (12 Töpfe mit 9 Pflanzen pro eintauchen).
- Befestigen Sie die ersten Schrauben um weitere sekundäre Bolzen entstehen zu lassen. Pflanzen sind bereit zum Dippen 4 – 6 Tage nach abschneiden, wenn die Pflanzen viele unreife Blütenköpfchen und nicht viele Siliques befruchtet.
-
Floral eintauchen
- Tauchen Sie die Blütenstände für 5 – 10 s Agrobacterium Suspension in Schritt 2.2.6 vorbereitet. Verwenden Sie sanfte Erregung.
- Wickeln Sie die oberirdischen Teile der Pflanzen in Frischhaltefolie, die Luftfeuchtigkeit hoch zu halten, und decken Sie die Blumentöpfe mit einer Box, die Pflanzen im Dunkeln zu halten. 2 Tage inkubieren Sie die Pflanzen in einem Gewächshaus/Wachstum Kammer.
- Entfernen Sie Feld und die Klarsichtfolie und wachsen Sie die Pflanzen bis zur Fälligkeit in einem Gewächshaus/Wachstum.
Hinweis: Um die Effizienz der Umwandlung kann die gleichen Pflanzen wieder eingetauchte 7 Tage nach dem ersten eintauchen. - Die Samen zu ernten. Pool und die Samen (T1) von Pflanzen, verwandelt sich das gleiche Konstrukt, als einen einzigen Satz zu analysieren.
-
Bildschirm für transgene Pflanzen.
- Auf dem Bildschirm für transgene Pflanzen mit einem pBIBAC-RFP-GW-Derivat umgewandelt, analysieren Sie die Samen mit Fluoreszenz-Mikroskopie. Um DsRed Ausdruck in Samenhüllen erkennen, Bild die Samen bei einer Anregung von 560 nm und Emission von 600 – 650 nm. Trennen Sie die fluoreszierenden Samen von nicht-fluoreszierende Pendants mit Pinzette.
- Auf dem Bildschirm für transgene Pflanzen mit einem pBIBAC-BAR-GW-Derivat umgewandelt, die Saat in Schalen gefüllt mit Erde (~ 2.500 Samen/0,1 m2). Um sicherzustellen, eine sogar Ausbreitung von Samen über Schalen, Samen in 0,1 % Agar in 0,5 x Murashige Skoog Medium (MS), und verteilt die Samen mit Hilfe einer 1 mL-Pipette.
Hinweis: Um die Samen zu keimen in einer synchronen Weise zu fördern, inkubieren Sie die Samen für mindestens 2 Tage bei 4 ° c Dies kann vor oder nach der Aussaat der Samen erfolgen.- Sprühen Sie die Sämlinge mit Glufosinat-Ammonium-Lösung 0,5 % 2 Wochen, 3 Wochen nach der Aussaat in Schalen. 500 mL Glufosinat-Ammonium-Lösung pro 1 m2zu verwenden.
- Übertragen Sie Überlebenden Sämlinge auf einzelne Töpfe. Ein typisches Bild eines Tabletts mit Setzlingen vor und nach der (zweiten) Glufosinat-Ammonium-Behandlung sind in Abbildung 3dargestellt.
- Analysieren Sie die Glufosinat-Ammonium-resistenten Pflanzen mittels PCR auf das Vorhandensein des Konstrukts von Interesse (siehe Schritt 1.4.1. für Primer).
- Isolieren der genomischen Pflanzen-DNA für die PCR mit der Methode von Edwards Et Al. beschrieben 17
4. Charakterisierung von gentechnisch veränderten Pflanzen für die Anzahl und die Integrität der T-DNA-Integrationen
- Strategie der Einschränkung Verdauung
Hinweis: Bestimmen Sie die Anzahl der T-DNA-Integrationen und ihre Integrität von DNA mit Restriktionsenzymen beflecken. Diese Methode ermöglicht das identifizieren einzelner, aber auch wiederholte Integrationen auf dem gleichen oder einem anderen Loci im Genom.- Verwenden Sie eine Reihe von Einschränkungen Verdauung der verschiedenen Integration möglich Muster zu erkennen:
- Wählen Sie ein Enzym, das einmal in der Mitte der T-DNA in der Lage sein, selbständig Sequenzen vor- und nachgelagerten des Standortes Einschränkung (Abbildung 4A und Abbildung 7A-C) Sonde schneidet. Siehe Abbildung 4Aund die Figur-Legende für die erwarteten Ergebnisse und Interpretation.
- Wählen Sie ein Enzym oder eine Kombination von Enzymen, die die gesamte Sequenz des Interesses auf einmal (Abbildung 4 b und Abbildung 7A-D) ausschneiden. Jede Abweichung von den bekannten Länge zeigt Abschneiden der integrierte Kassette.
Hinweis: darauf achten Sie, nur Restriktionsenzyme verwenden, die nicht empfindlich auf Cytosin Methylierung sind.
- Verwenden Sie eine Reihe von Einschränkungen Verdauung der verschiedenen Integration möglich Muster zu erkennen:
- Bereiten Sie die genomischen DNA-Proben.
- Isolieren Sie die genomische DNA aus Pflanzen tragen das Konstrukt von Interesse. Eine CTAB DNA Miniprep-Methode kann für DNA-Isolierung18verwendet werden. Für die DNA-Analyse von Arabidopsis DNA beflecken, 2 – 2,5 µg genomische DNA erforderlich ist. Lösen Sie die DNA in 50 µL TE.
- Überprüfen Sie die Integrität der DNA durch Gel-Elektrophorese16. Intakter genomischer DNA wandert als eine diskrete Band an der Spitze des Gels. DNA-Abbau ist das Vorhandensein von einem Abstrich erkennbar. Zur Vermeidung von DNA-Schäden durch wiederholtes Einfrieren Auftauen sind genomische DNA-Proben bei 4 ° c aufbewahrt.
- Die genomische DNA (2 – 2,5 µg bei Arabidopsis genomischer DNA) in einem Gesamtvolumen von 50 µL, über Nacht, mit Pufferbedingungen vorgeschlagen durch den Enzym-Lieferanten zu verdauen.
- Mischen Sie in einem Reagenzglas 2 – 2,5 µg von Arabidopsis genomischer DNA, 5 µL 10-fach Einschränkung Puffer und 5 U Restriktionsenzym in insgesamt 50 µL ultrapure deionisiertes Wasser.
- Die Einschränkung Proben vor dem Verladen auf einem Gel laden Farbstoff (1 x Endkonzentration) hinzugefügt werden. Für gute visuelle Verfolgung, verwenden Sie eine der folgenden Schritte aus: comigrating mit kleinen Fragmenten (Bromophenol Blue, 350 – 400 bp), oder comigrating mit größeren Fragmente (Cylene Cyanol, 3 – 4 Kbp).
- Führen Sie die DNA-Gel.
- Bereiten Sie eine lange (20 cm) 0,5 x TBE-Agarose-Gel-19. Prozent der Agarose im Gel richtet sich nach den Fragment-Größen erwartet. 0,8-1 % trennt optimal Fragmente > 1 kb groß. Verwenden Sie 1 bis 1,5 % Agarose für Fragmente < 1 kb. Das Gel keine Interkalation Bromid hinzu. (5 X TBE, 1 L: 54 g Trizma Basis, 27,5 g Borsäure, 3,75 g EDTA).
Hinweis: Um DNA-Kontamination zu verhindern, verstärkt Einsatz Gel Trays, die nicht verwendet werden, um fraktionieren Plasmid und PCR DNA. - Laden Sie die Proben auf Gel19.
- Das Gel, das in der Größenordnung der erwarteten Fragmente sind fügen Sie DNA-Markern hinzu. Laden Sie ca. 1 µg Marker (50-250 ng der anderen Größe Fragment) auf das Gel um Visualisierung durch UV-Licht zu ermöglichen.
- Größe-fraktionieren der DNS mit einer niedrigen Spannung (40 – 50 V/500 mA) über Nacht.
- Bereiten Sie eine lange (20 cm) 0,5 x TBE-Agarose-Gel-19. Prozent der Agarose im Gel richtet sich nach den Fragment-Größen erwartet. 0,8-1 % trennt optimal Fragmente > 1 kb groß. Verwenden Sie 1 bis 1,5 % Agarose für Fragmente < 1 kb. Das Gel keine Interkalation Bromid hinzu. (5 X TBE, 1 L: 54 g Trizma Basis, 27,5 g Borsäure, 3,75 g EDTA).
- Bereiten Sie für die Übertragung der DNA aus dem Agarosegel auf die Nylon-Membran.
- Übertragen Sie das Gel auf ein separates Fach, und färben Sie es für 20-25 min in 0,5 X TBE mit Interkalation Bromid (5 µg/mL) durch Drehen am 40 u/min auf einem Orbitalschüttler.
- Visualisieren Sie das Gel auf einem UV-Transilluminator. Überprüfen Sie die Größe Trennung von genomischer DNA, einschließlich der Sichtbarkeit der diskrete Sat-Bands (Abb. 5A). Ein Abstrich auf geringere Molekulargewicht Größen zeigt DNA-Abbau.
- Legen Sie auf die UV-Transilluminator eine Transparenz über das Gel, und markieren Sie die Schlitze und die Marker-Bands mit einem Marker (Abb. 5 b). Dies erleichtert die Bestimmung der Größe der hybridisierten Fragmente später für den Fall, dass die Marker Sequenzen nicht speziell mit der DNA-Sonde hybridisieren. Durch die Angabe der Marker Fragmente bei diesem Schritt, ist es möglich, die Größe der hybridisieren Fragmente zu verfolgen.
- Legen Sie das Gel zurück in die Schublade, mit hochreinen entionisiertem Wasser spülen Sie, und Tauchen Sie es in 0,25 M HCl für 15 min auf die DNA in das Gel fragmentize. Mit hochreinen deionisiertes Wasser waschen. Benutzen Sie genügend HCl und Wasser, um das Gel in die Schale zu decken, und drehen Sie das Fach mit dem untergetauchten Gel 40 u/min auf einem Orbitalschüttler.
- Inkubieren Sie das Gel in Denaturierung Puffer für 30 min. Waschen mit entionisiertem Reinstwasser. Benutzen Sie genügend Puffer und Wasser, um das Gel in die Schale zu decken, und drehen Sie das Fach mit dem untergetauchten Gel 40 u/min auf einem Orbitalschüttler. (Denaturierung Puffer: 0,5 M NaOH, 1,5 M NaCl).
- Inkubieren Sie das Gel in Neutralisation Puffer für 30 min. Waschen mit entionisiertem Reinstwasser. Verwenden Sie ausreichend Wasser und Puffer zu decken das Gel in die Schale, drehen Sie das Tablett mit getauchten Gel 40 u/min auf einem Orbitalschüttler. (Neutralisation Puffer: 0,5 M Tris, 1,5 M NaCl, 220 mM HCl, pH 7.6).
- Übertragen Sie die DNA auf einer Nylon-Membran.
- Bereiten Sie das Setup für Kapillare Transfer der genomischen DNA. Setzen Sie eine Kunststoffplatte (ungefähr die Größe des Gels oder größer) auf ein Tablett mit 20 x Kochsalzlösung Natriumcitrat (SSC) gefüllt. Tablett, umklappen Sie ein Stück dickes Filtrierpapier so dass beide Enden in der SSC hängen. Schneiden Sie eine Gel-großes Stück der positiv geladenen Nylon Membrane Hybond N + und 2 Stück dicken Filterpapier. (20 X SSC: 3 M NaCl, Natriumcitrat 0,3 M).
- Bereiten Sie das befleckende Setup (Abbildung 6), indem man das Gel Schlitze unten oben auf dem Filterpapier auf der Kunststoffplatte. Legen Sie eine Hybond n + Membran an der Spitze, gefolgt von 2 Lagen Filterpapier. Jede Schicht in 20 Vornässen X SSC bevor Sie sie auf die Assembly hinzufügen. Achten Sie darauf, keine Luftblasen zwischen Schichten zu entfernen, da diese behindern den DNA-Transfer.
- Decken Sie die Assembly mit einer dicken Schicht von Tissue-Papier. Setzen Sie eine Kunststoffplatte mit einem Gewicht, wie eine kleine Flasche, an der Spitze. Stellen Sie sicher, dass der Druck über das Gel gleichmäßig verteilt ist. Dies gewährleistet korrekte Transfer der DNA.
Hinweis: Das Gewicht sollte ca. 200-300 g; schwere Gewichte behindern den DNA-Transfer. - Decken Sie den Bereich rund um die Montage, einschließlich der exponierten Filterpapier mit Frischhaltefolie (Abbildung 6) zur Vermeidung von Verdampfung von 20 X SSC Puffer und Ziel die Kapillare Kräfte gegen die Nylon-Membran. Fleck über Nacht.
- Markieren Sie die Position der Steckplätze, Namen und/oder Datum oben auf die Membran mit Bleistift, und entfernen Sie die Membran aus der Assembly. Beachten Sie, dass die Unterseite, die in Kontakt mit dem Gel, wurde die DNA trägt.
- Die DNA auf der Membran durch UV-Bestrahlung (2.400 µJ/m2) mit einem UV-Vernetzer sofort zu beheben.
Hinweis: Vernetzung Bedingungen hängen von der Art der Membran verwendet.
Hinweis: an dieser Stelle die Membran kann bei-20 ° C gelagert und später für die Hybridisierung mit einer Sonde verwendet. Spülen Sie die vernetzte Membran in 2 x SSC und Dichtung es heißsiegelfähigen Polyethylen-Schlauch vor der Platzierung bei-20 ° c in vorgefaltet
- Bereiten Sie die Sonde für die Hybridisierung.
- Verstärken Sie die Sequenz, als die Sonde für das Beflecken DNA durch PCR20verwendet werden. 50-100 ng eines 250 bp-2 Kbp-PCR-Fragments wird als Sonde verwendet. Zwei separate Sonden, einer Hybridisierung der rechten proximalen Grenzregion und andererseits in die linken proximalen Grenzregion der T-DNA, können verwendet werden, zu beurteilen, die Anwesenheit der gesamten T-DNA (Abbildung 4A und Abbildung 7).
- 50-100 ng PCR-Produktes in 24 µL des hochreinen deionisiertes Wasser im Reagenzglas zu verdünnen.
- Denaturieren Sie die verdünnte PCR-Produkt durch Kochen es für 5 min in einen Becher Wasser oder Hitze Block, dann direkt auf Eis abkühlen.
- Vorgefertigten GCT-Mix auf dem Eis auftauen. (GCT-Mix: dGTP, dCTP, dTTP (alle 0,5 mM), zufällige Hexamers 43,2 ng/µL, Acetylated BSA 1,33 mg/mL, 33 mM β-Mercaptoethanol, 0,67 M Hepes 0,17 mM Tris, pH 6,8, 17 mM MgCl).
- Das PCR-Produkt 21 µL der GCT-Mix und 2 U Klenow Fragment hinzufügen.
- Die Mischung 2 µL [32P] ATP hinzu und Inkubation bei 37 ° C für 1 h.
Achtung: Alle Schritte mit [32P] ATP in einer Umgebung, die vorgesehenen radioaktiven Arbeit während der Verwendung des entsprechenden Schutzes durchgeführt werden müssen.
Hinweis: Sicherstellen, dass die [32P] ATP ist frisch (nicht mehr als 1 Halbzeit vergangen). - Vorbereiten einer Sephadex G50 (grobe und mittlere) Spalte21 die markierte Sonde von unincorporated Nukleotide (radioaktive) zu reinigen. Nehmen Sie eine 2 mL Spritze, und bedecken Sie die Steckdose mit einem kleinen Kreis von dicken Filterpapier. Fügen Sie in die Spritze 2 mL Sephadex G50 TE aufgelöst, und entfernen Sie alle Flüssigkeit aus der Spalte von Spinnen.
- Setzen Sie die Säule in eine 15 mL Plastikrohr, laden die markierte Sonde auf die Spalte und spin bei Raumtemperatur (750 X g die Zentrifuge festgesetzt, erlauben die Drehzahl erhöhen, bis 750 X g erreicht ist, dann halt der Zentrifuge und erlauben die Drehung auf 0 X g zurückgehen) zu eluieren die Sonde. Hinzufügen der Spalte und Spin, die restlichen Sonde eluieren 200 µL TE; einmal wiederholen. Unter diesen Bedingungen beschrifteten DNA-Fragmente sind ausgeschlossen von der Sephadex-Matrix und eluieren, während freie Nukleotide in der Spalte bleiben.
- Verwenden Sie die markierte Sonde pro Hybridisierung Rohr 300 µL. Halten Sie die restlichen markierte Sonde bei-20 ° C zur späteren Verwendung. Aber, behalten Sie die Hälfte der Zeit [32P] ATP im Hinterkopf.
- Hybridisieren Sie den DNA-Blot.
- Heat 2 X SSC und Hybridisierung Puffer (15 mL pro Hybridisierung Röhre, maximal 2 Flecken pro Röhre) bis 65 ° C. (Hybridisierung Puffer: 10 % Dextran Sulfat, 1 % SDS, 1 M NaCl, 50 mM Tris pH 7.5, lösen sich bei 65 ° C, halten Aliquote bei-20 ° C).
- Hybridisierung Backofen auf 65 ° c vorheizen
- Platzieren Sie Nylon-Netzgewebe in einer Schale mit ein wenig beheizten (65 ° C) 2 X SSC um das Fach zu decken. Legen Sie die DNA-Fleck auf der Oberseite des Netzes mit der DNA-Seite nach oben. Rollen Sie den Blot zusammen mit dem Netz und legen Sie die Rolle in einer Hybridisierung-Röhre. Gießen Sie die überschüssige 2 X SSC.
- Kochen in einem Microcentrifuge Schlauch 150 µL Lachs Spermien-DNA (Konzentration 10 mg/mL) pro Hybridisierung Röhre für 5 min (siehe auch Punkt 4.6.3). Kühlen Sie sofort auf Eis und auf den vorgewärmten Hybridisierung Puffer hinzufügen.
- Das Rohr mit der Blot die Hybridisierung Puffer-Lachs-Sperma-Lösung hinzufügen. Bereits bei 65 ° C für mindestens 1 h in einem rotierenden Rad, 12 u/min hybridisieren.
- Wenn die Vorinkubation im Schritt 4.7.5. ist fast fertig, die markierte Sonde für 5 min kochen 300 µL (siehe auch Punkt 4.6.3) und der Fleck nach der Inkubation sofort hinzufügen.
- Über Nacht in das drehende Rad bei 63 ° C, 12 u/min hybridisieren. Die Sonde direkt auf den Fleck, sondern in der Hybridisierung Puffer-Lachs Sperma Lösung nicht pipette.
- Waschen Sie den Fleck.
- Heizen Sie die Waschlösungen (1 x SSPE, 0,1 % SDS und 0,1 X SSPE, 0,1 % SDS) bis 65 ° C. (20 X SSPE: 3 M NaCl, 230 mM NaH2PO4, 20 mM EDTA, pH 7,0).
- Die Hybridisierung Lösung zu entsorgen und fügen Sie ca. 100 – 150 mL 1 X SSPE, 0,1 % SDS-Lösung zur Hybridisierung Röhre, in der Nähe der u-Bahn und von hand drehen. Gießen Sie die Hybridisierung und Waschlösung in den entsprechenden flüssigen radioaktiven Abfällen.
- Fügen Sie etwa 100 – 150 mL 1 X SSPE, 0,1 % SDS-Lösung mit dem Rohr schließen und das Rohr für 15 min bei 63 ° C in das drehende Rad, 12 u/min inkubieren. Entsorgen Sie die Waschlösung entsprechend.
- Fügen Sie etwa 100 – 150 mL 0,1 X SSPE, 0,1 % SDS-Lösung zur Hybridisierung Röhre, schließen, und das Rohr für 5 min bei 63 ° C, 12 u/min drehen. Entsorgen Sie die Waschlösung entsprechend.
- Nehmen Sie den Fleck aus dem Rohr und legen Sie sie in eine Schale mit ausreichend vorgeheizten 0,1 x SSPE, 0,1 % SDS und schütteln für 3 min schütteln Wasserbad bei 65 ° C. Inzwischen, spülen Sie das Netz in eine Schale mit Wasser gefüllt.
- Herausnehmen Sie den Blot, legen Sie es zwischen vorgefalteten Kunststoff (Polyethylen-Schlauch) sorgfältig wischen Sie überschüssige Flüssigkeit ab und lassen Sie den Fleck kurz trocknen. Beachten Sie, dass Flüssigkeit Phosphorimager Bildschirm ruinieren wird.
- Versiegeln Sie den Blot in Kunststoff auf drei Seiten. Entfernen Sie alle überschüssige Flüssigkeit rund um den Fleck zu, und schließen Sie das Plastikrohr durch Versiegelung der vierten Seite. Schneiden Sie den Überschuss aus Kunststoff. Stellen Sie sicher der versiegelte Blot nicht undicht ist und, dass der Kunststoff außen trocken ist.
- Setzen Sie die Phosphorimager Bildschirm.
- Platzieren Sie den versiegelten Blot in einer Kassette Phosphorimager mit dem Phosphorimager Bildschirm nach der DNA-Seite der Fleck. Schließen der Kassette und lassen für ± 2 – 4 Tage, je nach der Stärke der radioaktiven Beschriftung und Empfindlichkeit der Phosphorimager.
- Scannen Sie den Phosphorimager-Bildschirm mit einer Phosphorimager. Achten Sie darauf, um den Bildschirm möglichst wenig Licht vor dem Scannen zu entlarven. Speichern Sie das Bild. Löschen Sie den Bildschirm von Signal von hellem Licht aussetzen.
- Analysieren Sie den Fleck.
- Die Analyse richtet sich nach der Strategie in Schritt 4.1 verwendet. Bei der Analyse des Blot zubereitet nach der Strategie im Schritt 4.1.1.1 (Abb. 4A) gezeigt, die Anzahl der Fragmente entdeckt. Bei dieser Strategie bezieht sich die Anzahl der hybridisierten Fragmente auf die Anzahl der T-DNA-Integrationen.
- Vergleichen Sie die Anzahl der Fragmente mit einer Sonde für den linken (Abb. 7 b) und rechten (Abbildung 7) Teil der T-DNA nachgewiesen.
Hinweis: Wenn eine unterschiedliche Anzahl von hybridisieren Fragmente entdeckt, dann entweder (i) mehrere T-DNA-Kopien (entweder im umgekehrten oder direkte Ausrichtung) oder (Ii) nicht vollständig integriert sind T-DNAs vorhanden. - Schätzen Sie die Größen der hybridisierten Fragmente auf der Blot basierend auf der Größe der Marker-Bands, und vergleichen Sie die Größen der hybridisierten Fragmente mit dem erwarteten Fragment, die Größen auf die Strategie von Tandem Insertionen (Abb. 4A) berechnet identifizieren Sie mögliche Tandemanordnung der T-DNA. Verwenden Sie die Abbildung 4A als Leitfaden zur Berechnung der Größe der erwarteten Fragmente.
Hinweis: Wenn die Größe der hybridisierten Fragmente mit berechneten nicht einverstanden ist, ist dann wohl einer der derzeitigen Integrationen nicht abgeschlossen.
- Vergleichen Sie die Anzahl der Fragmente mit einer Sonde für den linken (Abb. 7 b) und rechten (Abbildung 7) Teil der T-DNA nachgewiesen.
- Bei der Analyse des Blot zubereitet nach Strategie gezeigt im Schritt 4.1.1.2 (Abbildung 4 b) schätzen Sie die Größe des hybridisieren Fragments auf den Fleck, basierend auf der Größe der Markierung Bands und vergleichen Sie es mit der erwarteten Größe. Eine intakte Einfügung ergibt ein einzelnes Fragment mit einer definierten Länge. Jede Abweichung von der erwarteten Länge zeigt unvollständige Integration (Abbildung 7).
- Die Analyse richtet sich nach der Strategie in Schritt 4.1 verwendet. Bei der Analyse des Blot zubereitet nach der Strategie im Schritt 4.1.1.1 (Abb. 4A) gezeigt, die Anzahl der Fragmente entdeckt. Bei dieser Strategie bezieht sich die Anzahl der hybridisierten Fragmente auf die Anzahl der T-DNA-Integrationen.
- Streifen der Fleck für die erneute Hybridisierung (optional).
Hinweis: Der gleiche Fleck kann nacheinander mit verschiedenen Sonden hybridisiert. Bevor Sie mit einer neuen Sonde Streifen einer vorherigen hybridisierten Sonde vom Fleck.- Um die Sonde vom Fleck zu entfernen, legen Sie den Blot in ein Fach mit seiner DNA-Seite nach unten. Gießen Sie einen Überschuss von 0,5 % SDS in die Schale. Kochen Sie die Membran für 2 – 5 min. Die Dauer der Behandlung richtet sich nach der Größe und der GC-Gehalt der Sonde verwendet. Länger und GC-reicher Sonden benötigen eine längere Behandlung.
- Nach dem Ausschalen, den Fleck mit einer anderen Sonde hybridisieren oder Dichtung und Lagerung bei-20 ° C.
Ergebnisse
Mit dem BIBAC-GW-System waren Reporter Konstrukte für ein Studium ODM in Pflanzen erzeugten10. Konstrukte wurden in der Gateway-Eintrag Vektor pENTR-gm12 entworfen und in pBIBAC-BAR-GW (Abbildung 1) mit dem Gateway LR Rekombination Reaktion eingefügt.
Arabidopsis verwandelten sich mit pDM19, ein BIBAC-BAR-GW-Plasmid mit einer mTurquoise-eYFP Reporter tragen ein translational Stopcodon in den eYFP Leserahmen an Position 120 (mTurquoise2-eYFP * 40) (Abbildung 2)10. Insgesamt wurden 126 Arabidopsis -Pflanzen transformierten (9 Pflanzen pro Topf, 14 Töpfe). Samen dieser Pflanzen wurden gebündelt, auf Tabletts mit Erde gesät und für zwei Wochen vor der Behandlung mit Glufosinat-Ammonium Lösung wachsen durfte. Nur Sämlinge mit dem Ausdruck des Bar -Gens (vorhanden in BIBAC-BAR-GW) überleben Glufosinat-Ammonium-Behandlung (Abbildung 3). Insgesamt wurden 11 transgene verwandelt mit pDM19 identifiziert, entsprechend einem Transformationseffizienz von 0,02 % der Samen analysiert.
Für die 11 gentechnisch veränderten Pflanzen isoliert war Blot-DNA verwendet, um die Anzahl der T-DNA Integrationen festzustellen. Zu diesem Zweck wurde genomischer DNA geschnitten mit BglII oder Scaich (Strategie als ausgeklügelte auf Abbildung 4A). Beide diese Restriktionsenzyme schneiden nur einmal in der T-DNA-Sequenz (Abb. 7A). Hybridisierung mit Sonden in Anerkennung der Bar und eYFP Codierung Regionen erlaubt Erkennung der Anzahl der jeweiligen DNA-Fragmente.
Die Anzahl der einzelnen DNA-Fragmente auf die Flecken dürfen für die Schätzung der Zahl der T-DNA Einfügungen in den Reporter-Linien (Tabelle 1). Einzelnen hybridisieren Fragmente mit der Bar und eYFP Sonde angezeigt die Anwesenheit einer einzigen T-DNA-Integration. Von den 11 transgene analysiert durchgeführt sechs einzelnen Integrationen. Die durchschnittliche Zahl der Integrationen betrug 1,2.
6 Zeilen mit einer einzigen Integration der T-DNA wurde die Integrität des eingefügten Reporter Konstrukts getestet mit DNA beflecken (Strategie als ausgeklügelte auf Abbildung 4 b). Genomischer DNA wurde mit BglII und Pcischneide ich ein 5,5 kb-Fragment enthält die Bar und das mTurquoise-eYFP Fusionsgen (Abb. 7A) freizugeben. Eine Sonde gegen eYFP wurde verwendet, um die erwarteten Fragment zu erkennen. Alle Pflanzen getestet trug eine intakte Fragment. Das Fragment untersucht schließt der linken und der rechten T-DNA-Grenze, und deshalb prüft nicht die Integrität der gesamten T-DNA, aber nur der Teil mit der transgene von Interesse.
Die Expression des Reportergens fluoreszierende wurde in unabhängigen Single Copy transgenen Linien unterscheiden sich nur durch die genomische Position der T-DNA bestimmt. Relative Abschrift waren des CaMV-35 s-Promoter-gesteuerte mTurquoise-eYFP-Reporter gemessen durch RT-qPCR in DM19 Reporter vierzeilig mit intakten, Single Copy Integrationen von denen die genomische Position bestimmt10war. Die Variation der Reporter-gen Ausdruck Ebenen zwischen den Zeilen war gering: die maximale Differenz in den mTurquoise-eYFP RNA Levels war 2-fold (Abb. 8A).
Als nächstes wurde die ODM in diesen Zeilen Reporter durchgeführt. Drei von den vier unabhängige Reporter Linien zeigte ziemlich ähnlich ODM Wirkungsgrade (Abbildung 8 b). Jedoch eine Zeile, DM19 [4] 1, ergab eine sehr geringe ODM-Effizienz im Vergleich zu den anderen Linien. Diese Ergebnisse zeigen, dass die lokalen genomische Kontext der ODM betroffen ist. In welcher Weise die lokalen genomische Kontext der T-DNA Integration in DM19 [4] 1 unterscheidet sich von den anderen Linien Überreste identifiziert werden. Analyse der verfügbaren Datensätzen auf aktive und inaktive Chromatin Markierungen an den genomischen T-DNA-Integration-Standorten in nicht-transgenen Pflanzen bieten keine Antwort10.

Abbildung 1: funktionale Karten von pBIBAC GW Vektoren. pBIBAC-GW-Derivate sind mit entweder Resistenz gegenüber Glufosinat (Bar) oder DsRed Fluoreszenz im Samenkorn-Mäntel (DsRed) als Selektionsmarker in Pflanzen. Für beide Vektoren ist eine Kanamycin-Resistenz-Gen die Auswahlmarkierung in Bakterien. Das Gateway CcdB Kassette zeigt sich zwischen grünen Pfeilspitzen vertreten Rekombination Websites AttR1 und AttR2. Bitte klicken Sie hier für eine größere Version dieser Figur.

Abbildung 2: Mutagenese Reporter Konstrukt. Die mTurquoise-eYFP Reporter Gene orientieren sich an der CaMV-35 s-Promotor. Die mTurquoise Region Kodierung ist mit einer Region, die eine C-A-Mutation an Nukleotid Position 120, was zu einem vorzeitigen translationale Stopcodon TAA und vorzeitige Beendigung der Übersetzung des Fusionsproteins Codierung eYFP verschmolzen. 3′ Nopaline Synthase (3' Nos) Polyadenylation Signal wird verwendet, um die Transkription der Konstrukt22zu kündigen. Nukleare Lokalisierung Signal (NLS) wird verwendet, um die übersetzten Proteine in den Zellkern zu richten. Bitte klicken Sie hier für eine größere Version dieser Figur.

Abbildung 3: Tablett voller Arabidopsis Sämlinge vor und nach der Behandlung von Glufosinat-Ammonium. Sämlinge, die nicht mit dem Ausdruck des Bar -Gens, das in der pBIBAC-BAR-GW-T vorhanden-DNA-Matrize nach besprüht mit Glufosinat-Ammonium-Lösung. Die Fotos zeigen das gleiche Fach der Keimlinge (A) vor dem Sprühen mit Glufosinat-Ammonium, 14 Tage nach der Aussaat, und (B) 10 Tage später, nach zweimal besprüht. Bitte klicken Sie hier für eine größere Version dieser Figur.
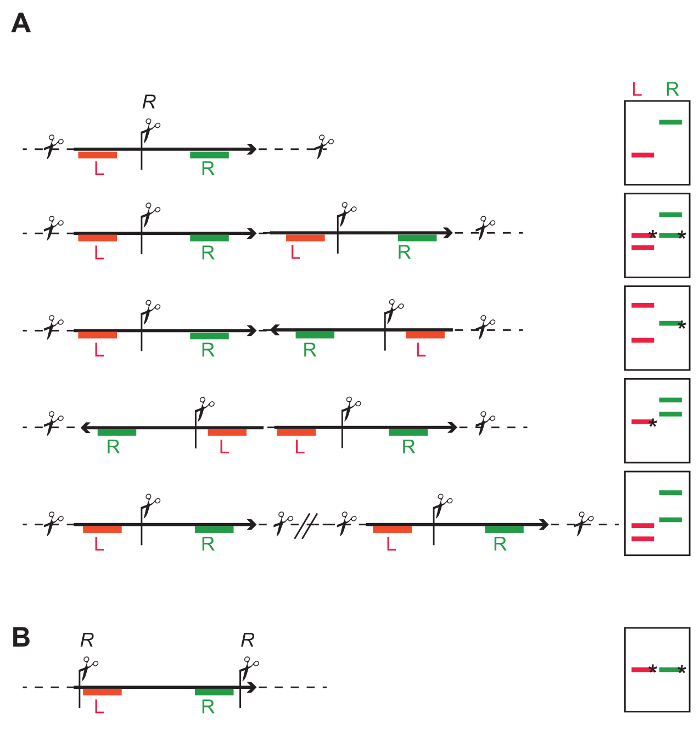
Abbildung 4: allgemeine DNA Beschränkung Strategie zu identifizieren, die Anzahl und die Unversehrtheit des eingefügt T-DNAs. (A) eine Beschränkung Site (R) in der Mitte der T-DNA ermöglicht unabhängige Sondierung der linken (roten L) und rechten Teil der T-DNA (grüne R). Die Cartoons auf der rechten Seite zeigen, dass je nach Einzel- oder multi - copy T-DNA Integrationen, Streifenbildung Mustern mit Blot-DNA gewonnen werden. Banden markiert mit einem * haben eine definierte Länge, während die Länge der anderen Bands hängt von der nächste Einschränkung-Standort in der flankierenden genomische DNA. Einzelne Insert: Die L und R Sonde beide geben ein unabhängiges Fragment. Die erwartete durchschnittliche Fragmentgröße kann basierend auf der Frequenz des Standortes Einschränkung im Genom berechnet. Die Mindestgröße ist der Abstand von der Einschränkung-Website an den linken Rand (LB) oder rechten Rand (RB), je nachdem, welches Ende der Integration sondiert werden, und wenn die T-DNA intakt ist. Tandem Repeat: Die Sonden für L und R geben beide zwei Fragmente; für jede Sonde eines der Fragmente enthält flankierender genomischen DNA, das zweite Fragment hat eine erwartete Größe und wird durch beide Sonden identifiziert. Inverted Repeat: Abhängig von der Richtwirkung der integrierte Kassette können entweder ein L und zwei R Fragmente oder zwei L und eine R identifiziert werden. Einzelnen einzelnen Einfügungen: Das Ergebnis ist eine Reihe von unabhängigen Fragmente, und die Anzahl der Fragmente entspricht die Zahl der Integrationen. (B) Beschränkung Websites an den Extremitäten der T-DNA bestimmen die Integrität des Fragments zwischen die Restriktionsschnittstellen können. Bitte klicken Sie hier für eine größere Version dieser Figur.
Abbildung 5: ein Agarosegel mit Einschränkung Muster und die passenden Transparenz. (A) auf das Agarosegel, verdaut genomischer DNA mit EcoRich wird angezeigt. Die richtige Verdauung der DNA wird durch die Anwesenheit von diskreten Sat-Bands veranschaulicht. (B) markieren die Position der Steckplätze und Marker Bands auf eine Transparenz zu einem späteren Zeitpunkt problemlos ermöglicht berechnen Sie die Größe der Fragmente Hybridisierung. MRC Holland Marker (blau und rot) kommen hier zum Einsatz, angezeigt durch M. Klicken Sie bitte hier, um eine größere Version dieser Figur.
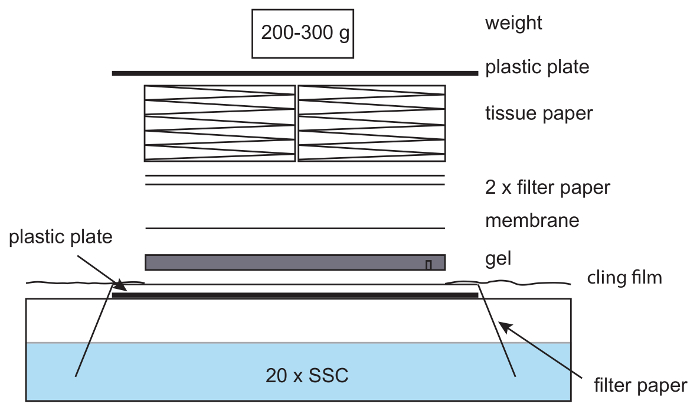
Abbildung 6: Setup für das Kapillare beflecken. In einer Kapillare Blot-Setup Filterpapier eine Kunststoffplatte mit den Enden des Papiers hängen in 20 X SSC Puffer im Vordergrund. Das Papier ist benetzt mit 20 X SSC und ein Agarosegel platziert an der Spitze, gefolgt von einer Nylon-Membran, Filterpapier und einen Stapel von Gewebe. An der Spitze befindet sich ein geringes Gewicht. Darauf wird geachtet, Luftblasen zwischen Gel, Papier und Membran zu entfernen. Klarsichtfolie wird verwendet, um zu vermeiden, trocknen aus dem Setup. Bitte klicken Sie hier für eine größere Version dieser Figur.
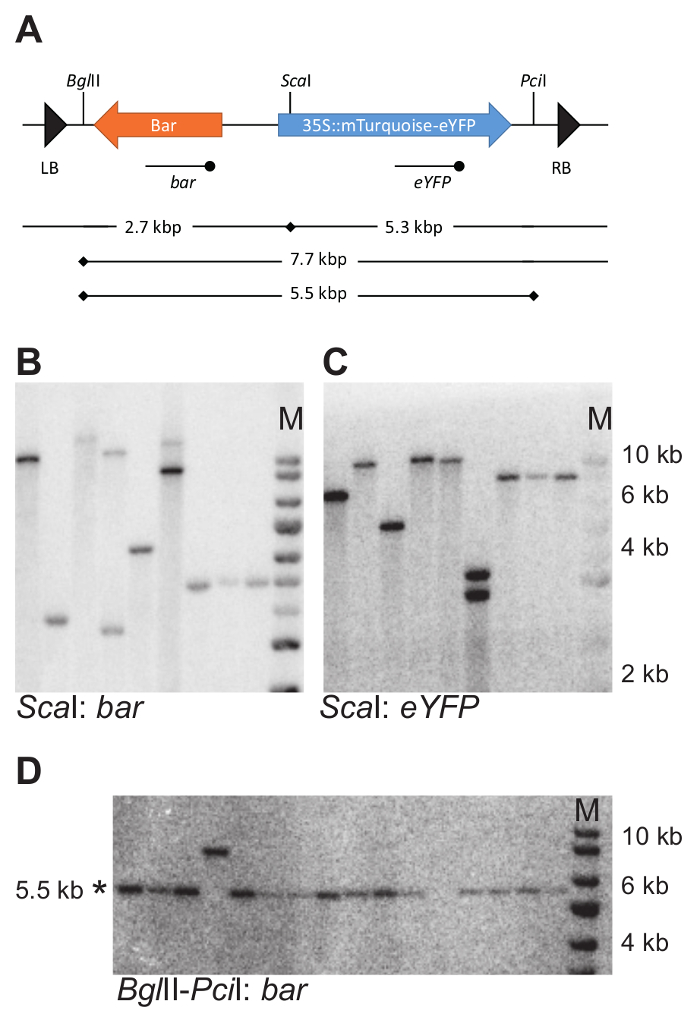
Abbildung 7: Beispiel für DNA beflecken, Strategie und experimentelle Ergebnisse. (A) DNA Blot-Strategie zu bestimmen, die Anzahl und die Unversehrtheit der T-DNA-Integrationen. Schneiden-Standorte der ausgewählten Restriktionsenzyme innerhalb der T-DNA sind mit vertikalen Balken gekennzeichnet. Die eYFP und Bar Sonden verwendet für die Hybridisierung mit der verdauten genomischen DNA sind gekennzeichnet mit einer Linie mit dem terminal Punkt unterhalb der T-DNA. (B–D) Beispiel DNA blots. Genomischer DNA wurde geschnitten, mit Scaich und der Fleck war mit einer Bar und eYFP Sonde (B und C) sondiert. Genomischer DNA wurde mit BglII und Pciich geschnitten und sondiert mit einer Bar -Sonde. Intakte Fragmente sind 5,5 Kbp in Größe (D). Beachten Sie, dass der Satz von Proben in D unterscheidet sich von den dargestellten in B und C. * zeigt die erwarteten Fragmentgröße; M, Markierung. In B, C und D wird die gleiche Größenmarkierung verwendet. Bitte klicken Sie hier für eine größere Version dieser Figur.
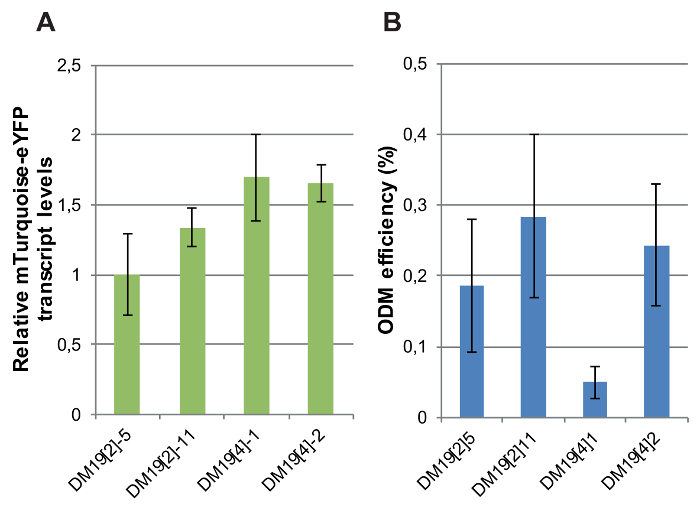
Abbildung 8: mTurquoise-eYFP Ausdruck Niveaus und ODM Effizienz bei unabhängigen mTurquoise-eYFP Reporter Linien. (A) Relative mTurquoise-eYFP Transkript Ebenen von RT-qPCR in DM19 Reporter Linien gemessen. Für die Normalisierung wurden Transkript Aktin verwendet. (B) ODM Effizienz in die DM19 Reporter Linien gemessen. Für A und B, bars geben den Durchschnitt von mindestens fünf biologischen repliziert. Fehlerbalken zeigen SEM Klicken Sie bitte hier, um eine größere Version dieser Figur.
| Typ T-DNA locus | Anzahl der T-DNA-Integrationen | Reporter-Linie | Anzahl der Fragmente erkannt | Integrität | |||
| SCA Ich | BGL II | BGL II/PciI | |||||
| Bar | eYFP | Bar | eYFP | eYFP | |||
| Einzigen Locus Integrationen | 1 | 19 [2]-2 | 1 | 1 | 1 | 1 | + |
| 1 | 19 [2]-5 | 1 | 1 | 1 | 1 | + | |
| 1 | 19 [2]-9 | 1 | 1 | 1 | 1 | + | |
| 1 | 19 [2]-11 | 1 | 1 | 1 | 1 | + | |
| 1 | 19 [4]-1 | 1 | 1 | 1 | 1 | + | |
| 1 | 19 [4]-2 | 1 | 1 | 1 | 1 | + | |
| 2, invertiert wiederholen | 19 [2]-10 | 2 | 1 | 1 | 1 | + | |
| 2, unvollständige integration | 19 [2]-3 | 1 | 2 | 1 | 1 | ND | |
| Locus Mehrfachintegrationen | 2 | 19 [2]-6 | 2 | 2 | 2 | ND | |
| 2 | 19 [2]-7 | 2 | 2 | 2 | 2 | ND | |
| 3/4 | 19 [2]-1 | 4 | 3 | 3 | 3 | ND | |
| ND – nicht bestimmt. | |||||||
Tabelle 1: Zusammenfassung der DNA Blot-Daten für gentechnisch veränderten Pflanzen isoliert nach der Transformation mit pDM19.
Diskussion
Entscheidend für die Erzeugung von gentechnisch veränderten Pflanzen mit einzelnen, intakte Integrationen von ein Transgen ist die Wahl der binären Vektor verwendet. BIBAC Familie Vektoren wurden zur Sequenzen von Interessen, viele Pflanzen Arten23,24,25,26,27,28zu liefern. BIBAC Vektoren, einschließlich BIBAC-GW, Ausbeute Single Copy Integrationen mit hohem Wirkungsgrad: die durchschnittliche Anzahl der Einschübe pro Zeile beträgt 1,5 bis 2, im Vergleich zu 3 oder höher, für die am häufigsten verwendete binäre Vektoren5,9, 29. als wesentliche Verbesserung im Vergleich zu anderen BIBAC Vektoren, mit der BIBAC-GW-Vektoren, die Sequenzen von Interesse können leicht eingefügt werden mit Gateway Rekombination Seiten12. Die modifizierte Vektoren zu überwinden die allgemeinen Probleme der BIBAC Vektoren als in konventionellen Klonen Strategien verwendet: i) eine sehr begrenzte Anzahl von einzigartigen Restriktionsschnittstellen und (Ii) eine niedrige DNA ergeben. Die Gateway-Rekombination-Sites machen BIBAC-GW Vektoren eine attraktive Alternative zu anderen binären Vektoren zur Erzeugung transgener Pflanzen.
Hier wird eine Reihe von Protokollen, von der Generierung BIBAC-GW-Derivate mit Sequenzen von Interesse, um Pflanzen Transformation und DNA-Analyse für Anzahl und Unversehrtheit der transgenen Sequenzen Fleck beschrieben. Einige der Protokolle berichtet in diesem Papier, Gateway Klonen, Elektroporation von Bakterien und Pflanzentransformation, sind gängige Praxis in vielen Laboratorien und kann auch mit leichten Modifikationen durchgeführt werden. Es ist wichtig zu wissen, dass BIBAC-GW ein Single Copy Vektor in E. Coli und A. Tumefaciensist. Deshalb wenn DNA zu isolieren, ist die Ausbeute gering; Es wird empfohlen, scale-up der Isolierung-Verfahren.
In gentechnisch veränderten Pflanzen tragen mehrere T-DNA-Integrationen eingeführte transgene unterliegen oft Gen-silencing-1,2,4,30, und sollte daher für die meisten Anwendungen vermieden werden. Um transgene Pflanzen mit einzelnen, intakte Integrationen zu ermitteln, empfiehlt es sich, DNA-Blot Analyse verwenden. Während andere Methoden als DNA beflecken kann zur Bestimmung der T-DNA Kopienzahl und Integrität der T-DNA in transgenen Linien verwendet werden (Segregationsanalyse, TAIL-PCR, quantitative PCR (qPCR) und Digital Tröpfchen PCR), obwohl Labor intensive, Blot-DNA ist oft die Methode der Wahl. Segregationsanalyse ist nicht in der Lage, zwischen mehreren unterscheiden und einzelne T-DNA Integrationen an einzelnen Loci. TAIL-PCR oft unter Schätzungen die Kopie Nummer, vor allem wenn mehrere T-DNA-Integration vorhanden31, und qPCR benötigt aufwendige Optimierung für zuverlässige Ergebnisse31,32. Digitale Tropfen PCR ist eine recht genaue Methode für die Kopie Nummer Erkennung, wenn die erforderliche Ausrüstung zur Verfügung31ist. Der Vorteil von DNA zu beflecken ist facile Erkennung von abgeschnittenen T-DNA, die in allen PCR-basierten Techniken leicht übersehen wird.
Mit DNA-Blot Analyse müssen die hybridisieren Fragmente auf den Fleck im Signal und Größe gut erkennbar sein. Mehrere Faktoren sind dafür bekannt, das Ergebnis der DNA-beflecken beeinflussen. Neben der Wahl eines geeigneten Restriktionsenzymen (angegeben in Abbildung 4-Strategie) und Größe Marker, ausreichend DNA von guter Qualität erforderlich ist. Weniger als 2 µg genomische Arabidopsis DNA wird nicht gut erkennbare Fragmente ergeben. Beim Umgang mit größeren Genome ist mehr DNA erforderlich. Um ausreichende Mengen an Arabidopsis DNA zu erhalten, können Blumen Gewebe oder 1 Woche alt Sämlinge verwendet werden. Eine Charge von Sämlingen gewachsen auf einer Petrischale ergibt 2 – 8 µg DNA. Zur Vermeidung von DNA-Abbau während Isolierung sollte darauf geachtet werden, um das Pflanzenmaterial schnell zu verarbeiten. Darüber hinaus genomischer DNA Nukleinsäuretablette in Tris-EDTA, dessen Abbau zu reduzieren durch Nukleasen und bei 4 ° C gelagert werden sollte, anstatt-20 ° C, um zu verhindern, dass DNA nicking aufgrund wiederholt Einfrieren Auftauen Zyklen. Wenn Sie unsicher sind, dass alle DNA-Proben vollständig verdaut werden, wird es vorgeschlagen, den DNA-Fleck mit einer Sonde erkennt eine endogene, einzigartige genomische Region rehybridize. Bei der Auswahl der Sonde Sequenzen zur Identifizierung von transgenen oder endogener Sequenzen ist es wichtig, nur einzigartige Sequenzen auszuwählen. Um die Größe der hybridisierten Fragmente genau bestimmen zu können, die Positionen der DNA gel Schlitze und DNA-Marker Bänder geprägt sein sollten auf eine Transparenz (Abb. 5 b) wenn eine Interkalation Bromid-gefärbten Gel (Abb. 5A) Visualisierung auf einem UV- Transilluminator. Für den Fall, dass die Marker Sequenzen nicht hybridisieren mit den Sonden-DNA oder Hybridisierung ist teilweise, ist es die einzige Möglichkeit, die Größe der Fragmente Hybridisierung aufzuspüren.
Nachdem darauf geachtet wird, gute Hybridisierung Signal und Schätzung der die Fragmentgröße zu erreichen, ist die Interpretation der befleckenden Ergebnisse einfach. Bei Verwendung nur ein Restriktionsenzym und Hybridisierung mit verschiedenen erkennen entweder den linken oder rechten Teil der T-DNA Sonden, spiegelt die Anzahl der gefundenen Fragmente die Anzahl der T-DNA Einfügungen. Zum Beispiel Abbildung 7 b, C zeigt die gleichen DNA-Blot, hybridisiert mit verschiedenen Sonden, Bar (Abb. 7 b) und verbesserte gelb fluoreszierenden Proteins (eYFP) (Abbildung 7), mit der Strategie, dargestellt in Abbildung 7A. Alle Spuren außer 4, zeigen eine gleiche Anzahl von Fragmenten auf beide Blots: zwei Fragmente für die Linie 6 und ein einziges Fragment für alle anderen Zeilen. Diese Anzahl an gefundenen Fragmente ist die Anzahl der T-DNA Einfügungen.
Wenn die Anzahl der Fragmente erkannt mit Sonden binden entweder links oder rechts Teil der T-DNA unterscheidet sich (wie Abbildung 7 bC, Linie 4), entweder unvollständige Einfügungen sind vorhanden, oder der T-DNA in Tandemanordnung eingefügt haben. Die Tandem-Einfügungen anzeigen nicht-zufällige Fragmentlänge für eines der T-DNA-Fragmente (Abbildung 4A, rechts) und durch den Vergleich der hybridisierten Fragmentgröße mit was erwartet wird, auf der Grundlage der Einschränkung Strategie identifiziert werden können. Eine zusätzliche befleckende Strategie kann erforderlich sein, um die Tandemanordnung der T-DNA Einfügungen zu bestätigen. In der Probe auf Linie 4 (Abb. 7 b, C) sind zwei Einfügungen in eine umgekehrte Wiederholung Orientierung angeordnet.
Bei der Schätzung der Unversehrtheit der T-DNA, bzw. eines Teils davon, kann die Länge des Fragments hybridisieren basierend auf der Strategie berechnet werden. Jede Abweichung von der erwarteten Größe zeigt das Vorhandensein einer unvollständigen Einfügung. Zum Beispiel wandert in Abbildung 7, in Bahn 4, hybridisieren Fragment bei 8 Kbp (statt der erwarteten 5,5 Kbp), eine erhöhte Fragmentgröße aufgrund des Fehlens eines die Restriktionsschnittstellen angibt.
BIBAC-GW-Vektoren sind hervorragende Werkzeuge für die Erzeugung von Single Copy intakt Integrationen in einer Reihe von Pflanzenarten. Das Protokoll hier berichtet bietet ein zuverlässiges Verfahren zur Identifizierung von Pflanzen mit einzelnen, intakte Integrationen von Transgen von Interesse.
Offenlegungen
Die Autoren erklären keine finanziellen Interessenkonflikte oder andere Interessenkonflikte.
Danksagungen
Diese Forschung wird unterstützt durch die niederländische Technologie Stiftung STW (12385), gehört die niederländische Organisation für wissenschaftliche Forschung (NWO), und die ist teilweise finanziert durch das Ministry of Economic Affairs (OTP Grant 12385, MS). Wir danken für die Bereitstellung von pCH20, das Rückgrat der BIBAC-GW Vektoren Carol M. Hamilton (Cornell University, USA).
Materialien
| Name | Company | Catalog Number | Comments |
| Kanamycin sulphate monohydrate | Duchefa | K0126 | |
| Gentamycin sulphate | Duchefa | G0124 | |
| Rifampicin | Duchefa | R0146 | |
| Tetracycline hydrochloride | Sigma | T-3383 | |
| DB3.1 competent cells | Thermo Scientific - Invitrogen | 11782-018 | One Shot ccdB Survival 2 T1R Competent Cells (A10460) by Invitrogen or any other ccdB resistant E. coli strain can be used instead |
| DH10B competent cells | Thermo Scientific - Invitrogen | 18290-015 | |
| Gateway LR clonase enzyme mix | Thermo Scientific - Invitrogen | 11791-019 | |
| tri-Sodium citrate dihydrate | Merck | 106432 | |
| Trizma base | Sigma-Aldrich | T1503 | |
| EDTA disodium dihydrate | Duchefa | E0511 | |
| Proteinase K | Thermo Scientific | EO0491 | |
| Bacto tryptone | BD | 211705 | |
| Yeast extract | BD | 212750 | |
| Sodium chloride | Honeywell Fluka | 13423 | |
| Potassium chloride | Merck | 104936 | |
| D(+)-Glucose monohydrate | Merck | 108346 | |
| Electroporation Cuvettes, 0.1 cm gap | Biorad | 1652089 | |
| Electroporator Gene Pulser | BioRad | ||
| Magnesium sulfate heptahydrate | Calbiochem | 442613 | |
| D(+)-Maltose monohydrate 90% | Acros Organics | 32991 | |
| Sucrose | Sigma-Aldrich | 84100 | |
| Silwet L-77 | Fisher Scientific | NC0138454 | |
| Murashige Skoog medium | Duchefa | M0221 | |
| Agar | BD | 214010 | |
| Glufosinate-ammonium (Basta) | Bayer | 79391781 | |
| Restriction enzymes | NEB | ||
| Ethidium Bromide | Bio-Rad | 1610433 | |
| Electrophoresis system | Bio-Rad | ||
| Sodium hydroxide | Merck | 106498 | |
| Hydrochloric acid | Merck | 100316 | |
| Blotting nylon membrane Hybond N+ | Sigma Aldrich | 15358 | or GE Healthcare Life Sciences (RPN203B) |
| Whatman 3MM Chr blotting paper | GE Healthcare Life Sciences | 3030-931 | |
| dNTP | Thermo Fisher | R0181 | |
| Acetylated BSA | Sigma-Aldrich | B2518 | |
| HEPES | Sigma-Aldrich | H4034 | |
| 2-Mercaptoethanol | Merck | 805740 | |
| Sephadex G-50 Coarse | GE Healthcare Life Sciences | 17004401 | or Sephadex G-50 Medium (17004301) |
| Dextran sulfate sodium salt | Sigma-Aldrich | D8906 | |
| Sodium Dodecyl Sulfate | US Biological | S5010 | |
| Salmon Sperm DNA | Sigma-Aldrich | D7656 | |
| Sodium dihydrogen phosphate monohydrate | Merck | 106346 | |
| Storage Phosphor screen and casette | GE Healthcare Life Sciences | 28-9564-74 | |
| Phosphor imager | GE Healthcare Life Sciences | Typhoon FLA 7000 | |
| UV Crosslinker | Stratagene | Stratalinker 1800 | |
| cling film (Saran wrap) | Omnilabo | 1090681 | |
| Agarose | Thermo Scientific - Invitrogen | 16500 | |
| Boric acid | Merck | 100165 | |
| DNA marker ‘Blauw’; DNA ladder. | MRC Holland | MCT8070 | |
| DNA marker ‘Rood’; DNA ladder | MRC Holland | MCT8080 | |
| Hexanucleotide Mix | Roche | 11277081001 | |
| Large-Construct Kit | Qiagen | 12462 | |
| Heat-sealable polyethylene tubing, clear | various providers | the width of the tubing should be wider than that of blotting membrane | |
| Heat sealer | |||
| Membrane filter disk | Merck | VSWP02500 | |
| Magnesium chloride | Merck | 105833 | |
| Hybridization mesh | GE Healthcare Life Sciences | RPN2519 |
Referenzen
- Jorgensen, R. A., Cluster, P. D., English, J., Que, Q., Napoli, C. A. Chalcone synthase cosuppression phenotypes in petunia flowers: comparison of sense vs. antisense constructs and single-copy vs. complex T-DNA sequences. Plant Mol Biol. 31 (5), 957-973 (1996).
- Stam, M., et al. Post-transcriptional silencing of chalcone synthase in Petunia by inverted transgene repeats. Plant J. 12, 63-82 (1997).
- Stam, M., Viterbo, A., Mol, J. N., Kooter, J. M. Position-dependent methylation and transcriptional silencing of transgenes in inverted T-DNA repeats: implications for posttranscriptional silencing of homologous host genes in plants. Mol Cell Biol. 18 (11), 6165-6177 (1998).
- Jin, Y., Guo, H. S. Transgene-induced gene silencing in plants. Methods Mol Biol. 1287, 105-117 (2015).
- Oltmanns, H., et al. Generation of Backbone-Free, Low Transgene Copy Plants by Launching T-DNA from the Agrobacterium Chromosome 1[W][OA]. Plant Physiol. , (2010).
- Ye, X., et al. Enhanced production of single copy backbone-free transgenic plants in multiple crop species using binary vectors with a pRi replication origin in Agrobacterium tumefaciens. Transgenic Res. , (2011).
- Hamilton, C. M. A binary-BAC system for plant transformation with high-molecular-weight DNA. Gene. 200 (1-2), 107-116 (1997).
- Frary, A., Hamilton, C. M. Efficiency and stability of high molecular weight DNA transformation: an analysis in tomato. Transgenic Res. 10 (2), 121-132 (2001).
- Vega, J. M., et al. Agrobacterium-mediated transformation of maize (Zea mays) with Cre-lox site specific recombination cassettes in BIBAC vectors. Plant Mol Biol. 66 (6), 587-598 (2008).
- Anggoro, D. T., Tark-Dame, M., Walmsley, A., Oka, R., de Sain, M., Stam, M. BIBAC-GW-based vectors for generating reporter lines for site-specific genome editing in planta. Plasmid. 89, 27-36 (2017).
- Hamilton, C. M., Frary, A., Lewis, C., Tanksley, S. D. Stable transfer of intact high molecular weight DNA into plant chromosomes. Proc Natl Acad Sci U S A. 93 (18), 9975-9979 (1996).
- Belele, C. L., Sidorenko, L., Stam, M., Bader, R., Arteaga-Vazquez, M. A., Chandler, V. L. Specific tandem repeats are sufficient for paramutation-induced trans-generational silencing. PLoS Genet. 9 (10), e1003773 (2013).
- Shizuya, H., et al. Cloning and stable maintenance of 300-kilobase-pair fragments of human DNA in Escherichia coli using an F-factor-based vector. Proc Natl Acad Sci U S A. 89 (18), 8794-8797 (1992).
- Shi, X., Zeng, H., Xue, Y., Luo, M. A pair of new BAC and BIBAC vectors that facilitate BAC/BIBAC library construction and intact large genomic DNA insert exchange. Plant Methods. 7, 33 (2011).
- Woodman, M. E., et al. Direct PCR of Intact Bacteria (Colony PCR). Curr Protoc Microbiol. , A.3D.1-A.3D.7 (2016).
- Ausubel, F. M., et al. Mol Biol. Current Protocols in Molecular Biology. 1, (2003).
- Edwards, K., Johnstone, C., Thompson, C. A simple and rapid method for the preparation of plant genomic DNA for PCR analysis. Nucleic Acids Res. 19 (6), 1349 (1991).
- Clarke, J. D. Cetyltrimethyl ammonium bromide (CTAB) DNA miniprep for plant DNA isolation. Cold Spring Harb Protoc. (3), (2009).
- Lee, P. Y., Costumbrado, J., Hsu, C. Y., Kim, Y. H. Agarose Gel Electrophoresis for the Separation of DNA Fragments. J Vis Exp. (62), e3923 (2012).
- Green, M. R., Sambrook, J. . Molecular Cloning. , (2012).
- Sosa, J. M. Chromatography with Sephadex Gels. Anal Chem. 52 (6), 910-912 (1980).
- Depicker, A., Stachel, S., Dhaese, P., Zambryski, P., Goodman, H. M. Nopaline synthase: transcript mapping and DNA sequence. J Mol Appl Genet. 1 (6), 561-573 (1982).
- Feng, J., Vick, B. A., Lee, M. K., Zhang, H. B., Jan, C. C. Construction of BAC and BIBAC libraries from sunflower and identification of linkage group-specific clones by overgo hybridization. Theor Appl Genet. 113 (1), 23-32 (2006).
- Lee, M. K., et al. Construction of a plant-transformation-competent BIBAC library and genome sequence analysis of polyploid Upland cotton (Gossypium hirsutum L). BMC Genomics. 14, 208 (2013).
- Wang, W., et al. A large insert Thellungiella halophila BIBAC library for genomics and identification of stress tolerance genes. Plant Mol Biol. 72 (1-2), 91-99 (2010).
- Wu, C., et al. A BAC- and BIBAC-based physical map of the soybean genome. Genome Res. 14 (2), 319-326 (2004).
- Xu, Z., et al. Genome physical mapping from large-insert clones by fingerprint analysis with capillary electrophoresis: a robust physical map of Penicillium chrysogenum. Nucleic Acids Res. 33 (5), e50 (2005).
- Zhang, M., et al. Genome physical mapping of polyploids: a BIBAC physical map of cultivated tetraploid cotton, Gossypium hirsutum L. PLoS One. 7 (3), e33644 (2012).
- Frary, A., Hamilton, C. M. Efficiency and stability of high molecular weight DNA transformation: An analysis in tomato. Transgenic Res. , (2001).
- Stam, M., Viterbo, A., Mol, J. N. M., Kooter, J. M. Position-Dependent Methylation and Transcriptional Silencing of Transgenes in Inverted T-DNA Repeats: Implications for Posttranscriptional Silencing of Homologous Host Genes in Plants. Cell Biol. 18 (11), 6165-6177 (1998).
- Głowacka, K., Kromdijk, J., Leonelli, L., Niyogi, K. K., Clemente, T. E., Long, S. P. An evaluation of new and established methods to determine T-DNA copy number and homozygosity in transgenic plants. Plant Cell Environ. 39 (4), 908-917 (2016).
- Stefano, B., Patrizia, B., Matteo, C., Massimo, G. Inverse PCR and Quantitative PCR as Alternative Methods to Southern Blotting Analysis to Assess Transgene Copy Number and Characterize the Integration Site in Transgenic Woody Plants. Biochem Genet. 54 (3), 291-305 (2016).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten