Method Article
BIBAC GW バイナリ ベクトルを使用してシングル コピー挿入と遺伝子組換え植物を生成します。
要約
PBIBAC GW バイナリ ベクトルを使用そのままシングル コピー挿入、簡単なプロセスを生成する遺伝子組換え植物になります。ここでは、一連のプロトコルは、形質転換シロイヌナズナ植物を生成し、イヌイットのための植物をテストのプロセスを介してリーダーを指導し、挿入の数がコピーされます。
要約
遺伝子組換え植物を生成、するとき一般に目的は transgene を安定に発現をしています。複数コピーの統合遺伝子サイレンシングにさらされている多くの場合と、遺伝子の 1 つ、そのまま統合が必要です。他の pBIBAC の誘導体のような細菌人工染色体 (pBIBAC GW)、に基づいてゲートウェイ互換性がある二進ベクトルは、高効率で単一コピーの遺伝子の挿入をことができます。、元の pBIBAC に改善は興味のシーケンス組み込むことが今簡単にベクトル転送 DNA (T-DNA) ゲートウェイを複製して、ゲートウェイ カセットは pBIBAC GW にクローン化されています。一般的 pBIBAC GW と変換結果 0.2 – 0.5 の効率 %、T DNA のままのシングル コピー統合を運ぶ、遺伝子組み換えの半分という。グルホシネート アンモニウム抵抗または種皮の植物では、選択した DsRed 蛍光と細菌の選択項目としてカナマイシン抵抗 pBIBAC GW ベクトルがあります。ここでは、一連のプロトコルが表示 pBIBAC GW を用いたトランスジェニック植物を生成するプロセスを介してリーダーを指導: 選択するの変換を植物の pBIBAC GW ベクトルに興味のシーケンスを再結合から始まってアグロバクテリウム、各種、遺伝子組み換え、DNA にしみが付くことを使用して挿入のイヌイットとコピーの数のための植物をテストします。注目は、単一および複数の座位でシングルとマルチ コピー統合を認識する DNA あぶらとり戦略の設計に与えられます。
概要
遺伝子組換え植物を生成、するとき通常目的は安定を表明した統合された transgene(s) を持つことです。これは、そのままシングル コピー transgene の統合によって達成できます。複数の統合は、遺伝子サイレンシングにも、遺伝子の発現増加につながることができます。遺伝子のサイレンシングは挿入されたシーケンスはタンデムまたは反転繰り返し1,2,3,4に配置された場合に生じやすい。バイナリのベクトル、アグロバクテリウムのシャトルとして使用されます-植物ゲノムに興味のシーケンスを提供する変換実験を介する。植物のゲノムに統合の数はアグロバクテリウム5,6.にバイナリのベクトルのコピー数に依存多くの一般的に使用されるバイナリ ベクトル高いコピー ベクトル、したがって高の平均遺伝子コピー数をもたらす:シロイヌナズナ53.3 4.9 にコピーします。
T DNA の統合の数は、BIBAC7などまたはA. 根頭がんしゅ病菌染色体5T DNA を起動することにより、 A. 根頭がんしゅ病菌で低コピー数を持つバイナリのベクトルを使用して下げることが。このような場合の transgene の統合の平均数は 25,8,9,10以下です。あることが原因シングル コピー A. 根頭がんしゅ病菌で、大腸菌、BIBAC 誘導体が維持し、150 kb11と同じ大きさの構造を提供します。
GW 互換 BIBAC ベクトル10、12は、ゲートウェイのクローン作成を使用してベクターに興味の遺伝子の簡単な紹介を許可します。ゲートウェイ技術を使用するクローンの手順を大幅に簡略化、大規模な低コピー数ベクトル13,14, 低 DNA 収量などユニークな制限の限られた選択に関連付けられている一般的な問題の解決にも7,11のクローンを作成できるサイト。グルホシネート アンモニウム (pBIBAC-バー-GW) にどちらかの抵抗性または種皮 (pBIBAC RFP GW) 植物 (図 1)10,12で選択した DsRed 蛍光 pBIBAC GW 誘導体があります。両方のベクトルのカナマイシン耐性遺伝子は、細菌の選択マーカーとして使用されます。
PBIBAC GW ベクトルを組み合わせる: (1) 簡単なデザインと大腸菌、および (2) そのままシングル コピー統合planta で高効率で遺伝子操作。単一を運ぶ遺伝子組換え植物のおよそ半分とシロイヌナズナにおける平均 1.7 インテグレーションの pBIBAC GW ベクトル収量は T-DNA10を統合されています。
遺伝子を安定に発現は、ほとんど遺伝子組み換え生成のための要件です。遺伝子発現の安定は、そのまま、単一コピーの統合によって実現できます。そのまま、単一コピーの統合を運ぶ遺伝子組換え植物の使用は、しかし、さらに重要な場合たとえば、目的これらの依存性や修復、組み換え、変異などのクロマチンによるプロセスの効率化を研究することですゲノムの位置と挿入部位のクロマチン構造上のプロセス。我々 の関心ローカル ゲノム コンテキストに関する監督オリゴヌクレオチド変異 (ODM) の依存性を研究する突然変異遺伝子のままに、単一コピーの統合記者行のセットだった生成された (図 2)10。この行セットを使用して、むしろ似ている遺伝子発現レベルにもかかわらず、ゲノムの異なる場所で統合された形質転換における遺伝子座間の ODM 効率が異なることが示されました。
プロトコル
1. バイナリのベクトルに興味のシーケンスを挿入します。
-
ゲートウェイのエントリとバイナリのベクトルを準備します。
- DNA のフラグメントまたはサプライヤーの提案によると小型準備キットを使用して興味の遺伝子を含むゲートウェイ エントリ ベクトルを分離します。
注: BIBAC GW ベクトルしたがって細菌の選択にカナマイシン (Km) を使用する必要、カナマイシンの代わりに別の抵抗のマーカーでエントリ ベクトルを使用します。例えば、ゲンタマイシン耐性遺伝子を運ぶ力がある pENTR gm ベクトルは良い選択12です。 - 反映し、興味の BIBAC GW ベクトルを分離します。ゲートウェイ カセット内に存在のccdB遺伝子毒性に耐性のある大腸菌の菌株を使用します。分離 BIBAC ベクトルを使用してプロトコルまたはサプライヤーの提案によると大規模なプラスミッドのために特別に設計されたキットします。
- DNA のフラグメントまたはサプライヤーの提案によると小型準備キットを使用して興味の遺伝子を含むゲートウェイ エントリ ベクトルを分離します。
-
ゲートウェイの反応を行います。
- サプライヤーの提案によると LR 再結合反応を準備します。常温 (RT) 1.5 mL 遠心チューブに次のコンポーネントをミックス: 100-300 ng エントリ クローン (スーパー)、300 BIBAC GW ベクトル、LR Clonase 反作用バッファーの ng (最終濃度: 1 x)。TE (10 mM Tris, EDTA、pH 8.0 の 1 mM) の 16 μ L を混合物の量を調整します。最後に、ボルテックスで LR Clonase 酵素ミックス、およびミックスの 4 μ L を追加します。25 ° C 1 時間で混合物を孵化させなさい。
- 1.2.1 準備した混合物にプロテイナーゼ K 溶液 (2 μ g/μ L) の 2 μ L を追加することで LR 反応を停止させます。混合し、37 ° c 10 分間加温します。
-
ゲートウェイの反応混合物を電気穿孔法による大腸菌を変換します。
- エレクトロポレーションの前に LR 反応混合物の塩分を除きます。
注: この手順は、エレクトロポレーションの成功にとって重要です。透析下メソッドは酢酸ナトリウムの析出など記載したが、他のメソッドとエタノールを使用もできます。- LR 反応フィルター透析のセットアップを準備します。超純水の脱イオン水 20 mL を滅菌シャーレに注ぎ。膜フィルター ディスク (細孔サイズ = 0.025 μ m) 水面上。
- ピペットの膜の上に注意深く全体の LR 反応と RT で 1 h の dialyze に混合物を許可します。
- エレクトロポレーション キュベット (0.1 cm) の電気主務 DH10B セルに脱塩の LR ミックスの 5 μ L を追加します。Electroporate 電池 (1.5 V/cm、抵抗 200 Ω、容量 25 μ F) 45 分、180 rpm の 37 ° C で培養後、細胞を 1 mL に予め温めておいたスーパー最適カタボライト抑制 (SOC) 媒体をすぐに追加。(SOC 培地 1 l: 20 g バクト トリプトン、酵母の 5 g の抽出、, 塩化ナトリウム 0.5 g 1 M の KCl の 2.5 mL、20 mL 1 M のフィルター滅菌グルコース)。
注: DH10B セルは、安定の大型プラスミドを維持他の大腸菌の細胞の代わりに使用できます。
注: 最適なエレクトロポレーション条件使用エレクトロポレーションのデバイスに依存しています。 - ペレット 30 最大速度で細菌、遠心機を使用して余分な SOC を削除し、ルリア ベルターニカミラ (LB) 培地の約 50-100 μ L でペレットを再懸濁します。カナマイシン LB (Km LB) 板 (Km 濃度、40 μ g/mL) に細菌を広げるし、37 ° C でプレートを一晩インキュベートします。(LB 培地 1 l: 10 g バクト トリプトン、酵母の 5 g のエキス、塩化ナトリウム 10 g。固体媒体の追加寒天 15 g/L)。
- エレクトロポレーションの前に LR 反応混合物の塩分を除きます。
-
られた BIBAC GW 誘導体を識別し、プラスミド DNA を隔離します。
- Km ポンドのプレート上に成長することができるには、大腸菌の細胞は、 ccdBシーケンスがされます希望挿入られた BIBAC GW プラスミドを含める必要があります。コロニーのポリメラーゼの連鎖反応 (PCR) の15を使用して、プレート上の細菌のコロニーが興味の挿入 BIBAC GW のバックボーンを持ち、正しいプラスミドを持つかどうかをチェックします。
- PBIBAC-バー-GW のバックボーンを識別する実行してプライマー DM1969 5'-GCGACGAGCCAGGGATAG-3 で PCR15反応 'と DM1970 5'-ATCAGTGCGCAAGACGTGAC-3'。このプライマー セットバー遺伝子の 563 の bp のフラグメントを増幅します。
- BIBAC RFP GW バックボーンの存在を確認するプライマー M737 5'-CGTGTAAAAAGCTTAGACTG-3 を使用して PCR15反応に従います 'と M892 5'-AACAGATGGTGGCGTCCC-3'。このプライマーの組み合わせは、cruciferin のプロモーターとrfpのシーケンスを重複 791 の bp のフラグメントを増幅します。
- 興味の挿入の有無をチェックする遺伝子特異的プライマーを用いた PCR15反応を実行します。
- 2-5 ml の DNA 分離16カナマイシン (40 μ g/mL) を含む LB 培地の単一肯定的なコロニーを接種します。180 rpm で軌道シェーカーで 37 ° C で一晩インキュベートします。
- プラスミド DNA を分離 (1.1.2 の手順を参照してください)。
- Km ポンドのプレート上に成長することができるには、大腸菌の細胞は、 ccdBシーケンスがされます希望挿入られた BIBAC GW プラスミドを含める必要があります。コロニーのポリメラーゼの連鎖反応 (PCR) の15を使用して、プレート上の細菌のコロニーが興味の挿入 BIBAC GW のバックボーンを持ち、正しいプラスミドを持つかどうかをチェックします。
2. A. 根頭がんしゅ病菌のシロイヌナズナの花浸漬のための準備
-
A. 根頭がんしゅ病菌BIBAC GW 誘導体を変換します。
- プラスミド7 A. 根頭がんしゅ病菌系統 C58C1、pCH32 を運ぶヘルパーの電子有能なセルの準備します。テトラサイクリン (5 μ g/mL) および pCH32 を選択し、アグロバクテリウムセルだけの成長を確保するリファンピシン (100 μ g/mL) の存在下で細菌を成長します。
- 追加 0.25 – 0.5 μ g pBIBAC GW 誘導体の DNA は前 10-20 μ L 滅菌超純水脱イオン水、エレクトロポレーション キュベット (0.1 cm) に 20 μ L 有能なアグロバクテリウムセルに分解します。氷の上の細胞を保ちます。
- Electroporate 電池 (1.5 V/cm の抵抗 400 Ω、容量 25 μ F)。エレクトロポレーション、直後に細菌に 1 mL に予め温めておいた (28 ° C) SOC 培地を追加し、28 ° C, 60-90 分で細胞をインキュベートします。
- 100 μ L とリファンピシン (100 μ g/mL)、テトラサイクリン (5 μ G/ml) とカナマイシン (40 μ G/ml) を含む別の LB の版上の細菌の残りの部分を広げるし、1-2 日間の 28 ° C で暗闇の中で孵化させなさい。
-
アグロバクテリウムの懸濁液を準備します。
- PCR プレート ステップ 2.1.4, 正しいベクトルの存在のために準備からA. 根頭がんしゅ病菌のコロニー数を確認 (手順 1.4.1 参照)。
- ストリーク (手順 2.1.4 参照) 抗生物質を含む LB プレート上での適切な挿入とバイナリのベクトルを含む単一コロニーを確認しました。28 ° C で一晩成長します。
- 2.2.2 の手順で得られた単一のコロニーでストリー キングを繰り返します。
- 清酒の抗生物質 (手順 2.1.4 参照) で LC 培の 2.5 ml 単一コロニーを接種します。28 ° C 少なくとも 8 時間、または一晩、180 rpm で孵化させなさい。(LC 媒体バクト トリプトン、酵母の 5 グラムの 1 l: 10 g 抽出, 塩化ナトリウム 0.5 g、2.5 g MgSO4 · 7 H2O、マルトースの 2 g)。
- 2.2.4 のステップから、清酒を追加します。250 mL LC 抗生物質 (手順 2.1.4 参照) を添加したし、一晩 180 rpm で 28 ° C で成長します。
- ペレット 5,500 x g で 12 分間で回して文化再 0.05 5% ショ糖を含む 100 mL の溶液内のペレットを中断植物の Silwet L-77、x さんを注ぐ花浸漬滅菌コンテナーに懸濁液 0.5%。
3. Arabidopsis の変形
-
変換のシロイヌナズナ植物を準備します。
- 花盛り (浸漬あたり 9 植物 12 鍋) までは、温室や気候制御成長室シロイヌナズナ植物を育てます。
- 出現する以上のセカンダリ ボルトを許可する最初のボルトをクリップします。植物が植物は多くの未熟な頭花と多くはないが、クリッピング後の 4-6 日を浸漬のため準備ができて siliques を受精します。
-
花を浸漬
- 2.2.6 準備したアグロバクテリウム懸濁液中の 5-10 s の花序をつけます。穏やかな攪拌を使用します。
- サランラップ、高湿度を保つ植物の地上部分をラップし、暗闇の中での植物を保つためにボックスで植木鉢をカバーします。温室/成長チャンバ内 2 日は植物を孵化させなさい。
- ボックスとしがみつくフィルムを取り外して温室/成長チャンバ内に成熟植物を育てます。
注: 変換の効率を高めるためには、最初に浸漬した後再浸漬 7 日同じ植物を指定ことができます。 - 種を収穫します。プールし、同じ構成要素、1 つのセットで、植物の種子 (T1) を分析します。
-
遺伝子組換え植物の画面。
- PBIBAC RFP GW 誘導体に変換された遺伝子組換え植物の画面、蛍光顕微鏡を利用した種子を分析します。種皮にした DsRed 発現を検出するために 560 nm の励起と 600-650 nm の発光で種子をイメージします。鉗子を使用して対応する非蛍光灯から蛍光種を分けます。
- 遺伝子組換え植物 pBIBAC-バー-GW 誘導体変換の画面に土 (~ 2,500 種/0.1 m2) を詰めたトレイに種をまきます。トレイ上の種子も広がりを 0.1% 寒天培地培 (MS) × 0.5 で種子を中断し、1 mL ピペットを使用して種を広めるため。
注: 同期的な方法で発芽する種子を刺激するには、4 ° C で、少なくとも 2 日間種をインキュベートします。これは、種子を播種前後に行うことができます。- 0.5% グルホシネート アンモニウム溶液 2 週間とトレーに播種後 3 週間で苗をスプレーします。グルホシネート アンモニウム溶液 1 m2あたり 500 mL を使用します。
- 個々 のポットに残っている苗を転送します。(2 番目) のグルホシネート アンモニウム治療前後の苗をトレイの典型的なイメージは、図 3のとおりです。
- 興味の構造の存在を PCR によってグルホシネート アンモニウム耐性植物を分析 (1.4.1 の手順を参照してください。 プライマーのため)。
- エドワーズらで説明したメソッドを使用して PCR のため植物ゲノム DNA を隔離します。17
4. 番号および T DNA の統合性の遺伝子組み換えを特徴付ける
- 制限の消化力の戦略
注: は、DNA は制限の酵素を使用してしみが付くことによって T DNA の統合との整合性の数を決定します。このメソッドは、シングル、識別することができますが、またゲノムの同じまたは異なる座位で統合を繰り返した。- 可能な限りのさまざまな統合パターンを識別する制限の消化力のシリーズを使用します。
- T DNA シーケンス (図 4 aおよび図 7 a C) 制限サイトの下流・上流のプローブを独立してできるように途中で一度切断する酵素を選択します。図 4 aと予想される結果の解釈図凡例を参照してください。
- 酵素または酵素 (図 4 b 図 7A-D) すぐに興味のシーケンス全体を切り取るの組み合わせを選択します。既知の長さから任意の偏差は、統合されたカセットの切り捨てを示します。
注: のみシトシンのメチル化に敏感ではない制限酵素を使用する注意してください。
- 可能な限りのさまざまな統合パターンを識別する制限の消化力のシリーズを使用します。
- ゲノムの DNA のサンプルを準備します。
- 興味の構造を運ぶ植物由来の DNA を分離します。DNA 分離18CTAB DNA の miniprep メソッドを使用できます。DNA のしみシロイヌナズナDNA の解析、ゲノム DNA の 2-2.5 μ g が必要です。TE の 50 μ L の DNA を溶解します。
- ゲルの電気泳動16DNA の整合性をチェックします。そのままのゲノム DNA をゲルの上部に 1 つの離散的バンドとして移行します。DNA 分解は、塗抹標本の存在として認識することができます。繰り返しの凍結融解による DNA 損傷を避けるためには、ゲノムの DNA のサンプルが 4 ° C で維持されます。
- ゲノム DNA (シロイヌナズナのゲノム DNA の場合 2-2.5 μ g) 総量が一晩、50 μ L の酵素サプライヤーによって提案されたバッファー状態を消化します。
- テスト チューブに 50 μ L の純水脱イオンの合計で 5 U 制限の酵素、5 μ L の 10 倍の制限バッファー、シロイヌナズナのゲノム DNA の 2-2.5 μ g を混ぜます。
- ゲルにロードする前に制限サンプルにローディングの染料 (最終濃度 x 1) を追加します。良い視覚追跡のため、次のいずれかを使用して、: comigrating の小さな断片 (ブロモフェノールの青、350-400 bp)、または大きい断片 (サイリーン cyanol、3-4 kbp) と comigrating。
- DNA ゲルを実行します。
- 長い (20 cm) 0.5 TBE agarose ゲル19× を準備します。ゲルの agarose の割合は、期待されるフラグメントのサイズに依存します。0.8-1% が最適のフラグメントを分ける > 1 kb のサイズです。1-1.5% の agarose を使用して、フラグメントの < 1 kb。エチジウム ブロマイドをゲルに追加しないでください。(5 x TBE、1 l: 54 g Trizma のベース、ホウ酸、EDTA の 3.75 g の 27.5 g)。
注: DNA の汚染を防ぐためには、プラスミド、PCR を分別に慣れていない使用ゲル トレイは DNA を増幅しました。 - ゲル19のサンプルを読み込みます。
- 予想されるフラグメントのサイズの範囲は、ゲルに DNA マーカーを追加します。紫外線によって可視化できるようにゲル上のマーカー (サイズの異なるフラグメントの 50-250 ng) の約 1 μ g をロードします。
- サイズ - 一晩分別低電圧 (40-50 V/500 mA) DNA。
- 長い (20 cm) 0.5 TBE agarose ゲル19× を準備します。ゲルの agarose の割合は、期待されるフラグメントのサイズに依存します。0.8-1% が最適のフラグメントを分ける > 1 kb のサイズです。1-1.5% の agarose を使用して、フラグメントの < 1 kb。エチジウム ブロマイドをゲルに追加しないでください。(5 x TBE、1 l: 54 g Trizma のベース、ホウ酸、EDTA の 3.75 g の 27.5 g)。
- ナイロン膜に agarose のゲルから DNA を転送する準備します。
- 別のトレイにゲルを転送し、0.5 で 20-25 分間染色エチジウム ブロマイドを含む x TBE (5 μ G/ml) 軌道シェーカーで 40 rpm で回転。
- UV transilluminator のゲルを視覚化します。離散衛星バンド (図 5 a) の可視性を含むゲノム DNA のサイズ分離を確認します。低分子量サイズへの中傷は、DNA 分解を示します。
- UV transilluminator、ゲル上の透明度を置くし、マーカーペン (図 5 b) とスロットとマーカー バンドの位置をマークします。マーカー シーケンスを行うプローブ DNA と特異的交配しない場合に後で交配させられたフラグメントのサイズを決定する簡単になります。この段階でマーカーのフラグメントを示すことで交雑のフラグメントのサイズを追跡することが可能です。
- ジェルをトレーに、超高純度の脱イオン水ですすいでください、ゲル内の DNA を fragmentize 15 分 0.25 M HCl で水没します。超純水の脱イオン水で洗ってください。トレイにジェルをカバーする十分な塩酸と水を使用して、軌道のシェーカーで 40 rpm で冠水したゲルとトレイ。
- 超純水の脱イオン水で 30 分洗浄用変性バッファーのゲルを孵化させなさい。トレイにジェルをカバーする十分なバッファーと水を使用して、軌道のシェーカーで 40 rpm で冠水したゲルとトレイ。(変性バッファー: 0.5 M NaOH、1.5 M の NaCl)。
- 超純水の脱イオン水で 30 分洗浄用中和バッファーのゲルを孵化させなさい。トレイにジェルをカバー、軌道シェーカーで 40 rpm で冠水したゲルとトレイを回転させるのに十分なバッファーと水を使用します。(中和バッファー: 0.5 M 1.5 M の NaCl、HCl、220 mM Tris pH 7.6)。
- DNA をナイロン膜に転送します。
- ゲノムの DNA の毛管転送のセットアップを準備します。プラスチック板 (約サイズのゲルのまたはより大きい) を生理食塩水ナトリウム クエン酸 (SSC) x 20 で満たされたトレイにかぶせます。両端が SSC に掛かっているので、トレイ、上厚いフィルター ペーパーの部分を折る。ピースは正荷電のナイロン膜 Hybond N +、ゲルの大きさと厚いフィルター ペーパーの 2 個をカットします。(20 x SSC: 3 M 塩化ナトリウム、クエン酸ナトリウム 0.3 M)。
- ゲルのプラスチック板にフィルター紙の上に下のスロットを配置することによって (図 6) のしみのセットアップを準備します。上では、Hybond N+ 膜を配置フィルター紙の 2 層が続きます。前 20 の各レイヤーを濡れているそれらをアセンブリに追加する前に x SSC。これらは DNA の伝達を阻害層の間に空気の泡を削除することを確認します。
- ティッシュ ペーパーの厚い層を持つアセンブリをカバーします。上の小瓶など、重量をプラスチックの板を配置します。圧力はゲルを均等に分けられていることを確認します。これは、DNA の適切な転送が保証されます。
注: 重量は約 200-300 g; する必要があります。重い重量を妨げる DNA 伝達。 - 20 x SSC バッファーとナイロン膜に向かって強制的にキャピラリー ターゲットの蒸発を避けるために寝かせます (図 6) の露出のフィルター ペーパーを含むアセンブリを囲む領域をカバーします。一晩のしみ。
- 鉛筆で、スロット、名前、および/または膜の上に日付の位置をマークし、膜をアセンブリから削除します。ゲルと接してきた下部側が DNA を運ぶことに注意してください。
- UV 架橋剤を使用して紫外線照射 (2,400 μ J/m2) による膜への DNA がすぐに解決します。
メモ: 架橋条件は膜の種類に依存します。
注: この時点で膜-20 ° C で保存でき後でプローブとの交配のため使用します。SSC とそれはで中古-20 ° C でそれを置く前に heat-sealable ポリエチレン チューブを畳んだシール x 2 の架橋膜を洗浄します。
- 交配のプローブを準備します。
- DNA は PCR20でしみが付くことのためのプローブとして使用するシーケンスを増幅します。50 100 250 bp 2 kbp PCR のフラグメントの ng は、プローブとして使用されます。2 つの独立したプローブ、右枠線の近位部と他の T-DNA の左枠線の近位部に交配させることの 1 つは、全体 T-DNA (図 4 aおよび図 7) の存在を評価するために使用できます。
- 試験管に純水脱イオン水の 24 μ L の PCR の製品の 50-100 ng を希釈します。
- 希薄化後の PCR の製品を水または熱ブロックのビーカーに 5 分間沸騰させることによって変性し、氷で直接冷やします。
- 氷の上の既成 GCT ミックスを解凍します。(GCT ミックス: dGTP dCTP、dTTP (すべて 0.5 mM)、ランダムな hexamers 43.2 ng/μ L、アセチル化 BSA 1.33 mg/mL、β-メルカプトエタノール、0.67 M Hepes 0.17 mM Tris pH 6.8, 17 mM MgCl の 33 mM)。
- GCT ミックス 21 μ L と 2 U の Klenow のフラグメントを PCR の製品に追加します。
- ミックスに [32P] ATP の 2 μ L を追加し、37 ° C 1 時間インキュベートします。
注意: [32P] ATP に関連するすべての手順は、適切な保護を使用している間放射性作業用に指定された環境で実施する必要があります。
注: [32P] ATP が新鮮なことを確認 (以上 1 時間の半分が経過)。 - (放射性) 非法人のヌクレオチドから分類されたプローブの浄化のセファデックス G-50 (粗・中) 列21を準備します。2 mL 注射器を取るし、厚いフィルター ペーパーの小さな円でコンセントをカバーします。セファデックス G-50 の 2 mL は、注射器に TE に溶解を追加し、回転によって列からすべての液体を削除します。
- 15 mL プラスチック チューブに列を配置、列に分類されたプローブの負荷および室温 (750 × g で遠心分離機を設定、750 x g に達するまで増加し回転を許可する、遠心分離機を停止し、0xg に低下する回転を許可する) でスピンの溶出がプローブ。列との溶出が残りプローブ; スピンに TE の 200 μ L を追加します。もう一度繰り返します。これらの条件でラベル付けされた DNA のフラグメントは、セファデックス マトリックスから除外され、溶出、遊離ヌクレオチドの列のまま。
- 分類されたプローブの交配管あたりの 300 μ L を使用します。後で使用するための-20 ° C で残りの分類されたプローブを維持します。ただし、[32P] ATP の半分の時間を覚えておいて。
- DNA のしみを交配させます。
- 熱 2 x SSC と交配バッファー (ハイブリダイゼーション チューブ、チューブごと 2 しみの最大あたり 15 mL) 65 ° c(交配バッファー: 10% デキストラン硫酸、1 %sds、1 M NaCl、50 mM Tris pH 7.5、65 ° C の溶解、-20 ° C で因数を保つ)。
- 65 ° c. にハイブリダイゼーション オーブンを予熱します。
- ほんの少し加熱 (65 ° C) 2 のトレイにナイロン メッシュを配置、トレイをカバーする x SSC。DNA 側を使用して、メッシュの上に DNA のしみを上に置きます。しみを一緒にメッシュ ロール、ロールをハイブリダイゼーション チューブに挿入します。余分な 2 を注ぐ x SSC。
- 微量遠心チューブにサケ精子 DNA の 150 μ L を沸騰 (濃度 10 mg/mL) あたり 5 分間交配管 (また見なさいステップ 4.6.3)。すぐに氷で冷却し、予熱した交配バッファーに追加します。
- しみとチューブに交配バッファー サケ精子ソリューションを追加します。少なくとも 1 h 12 rpm で、ホイールの回転で 65 ° C で交配させる前。
- とき 4.7.5 の手順で中古のインキュベーション。ほぼ完成 5 分標識プローブの沸騰 300 μ L (また見なさいステップ 4.6.3) し、孵化後すぐにしみへを追加。
- 63 ° C、12 rpm で回転している車輪に一晩を交配させます。直接、ブロットが交配バッファー サケ精子ソリューションにプローブをピペットないです。
- しみを洗います。
- 洗濯ソリューションを予熱 (0.1、0.1% SDS SSPE x 1 0.1% SDS x SSPE) 65 ° c(20 x SSPE: 3 M の NaCl、230 mM NaH2PO4、20 ミリメートルの EDTA、pH 7.0)。
- 交配ソリューションを破棄し、1 の約 100-150 mL を追加 x SSPE、ハイブリダイゼーション チューブに 0.1 %sds の溶液、チューブを閉じ、手で回転させます。交配および洗浄ソリューションの適切な液体放射性廃棄物を注ぐ。
- 1 の約 100-150 mL を追加 x SSPE、チューブに 0.1 %sds の溶液を閉じて、ホイールの回転、12 rpm で 63 ° C で 15 分間チューブを孵化させなさい。洗濯ソリューションを適切に破棄します。
- 約 100-150 mL の 0.1 を追加 x SSPE、ハイブリダイゼーション チューブに 0.1 %sds の溶液を閉じて、63 ° C、12 rpm で 5 分間チューブを回転させます。洗濯ソリューションを適切に破棄します。
- チューブからしみを取るし、十分な予熱した 0.1 x SSPE、0.1 %sds および 65 ° C で揺れ水お風呂で 3 分間振るトレイ一方、水で満たされたトレイにメッシュをすすいでください。
- しみを取り出す、折り曲げ済みプラスチック (ポリエチレン チューブ) の間に、慎重に余分な液体を拭き取り、簡潔に乾燥しみができます。液体が phosphorimager 画面を台無しにすることに注意してください。
- 3 つの側面のプラスチックでしみをシールします。しみを取り巻くすべての余分な液体を除去し、第 4 側面をシールでプラスチック製のチューブを閉じます。プラスチックの剰余金を切った。シールのしみが漏れていないプラスチックが外側に乾いていることを確認します。
- Phosphorimager 画面を公開します。
- Phosphorimager カセットにしみの DNA 側に直面している phosphorimager 画面とシールのしみを配置します。カセットを閉じるし、2-4 日間、放射化ラベリングの強さと、phosphorimager の感度に応じて ± おきます。
- Phosphorimager を使用して、phosphorimager 画面をスキャンします。スキャンする前に光を可能な限り少しの画面を公開する注意してください。画像を保存します。明るい光にさらすことによって信号から画面を消去します。
- しみを分析します。
- 分析は、4.1 のステップで使用される制限戦略によって異なります。4.1.1.1 (図 4 a) の手順で示した戦略にしたがって調製されたしみを分析する場合は、検出されたフラグメントの数します。この戦略では交配させられたフラグメントの数は T DNA の統合の数を指します。
- T DNA の右側 (図 7) と左 (図 7 b) のプローブで検出されたフラグメントの数を比較します。
注: 交雑フラグメント数が異なるが検出されると、[いずれか i) 複数 T DNA コピー (反転または直接向きのどちらか) または ii) 不完全統合 T Dna が存在します。 - しみの交配させられたフラグメントのサイズ マーカー バンドのサイズに基づいて、予想されるフラグメント サイズ タンデム挿入 (図 4 a) の制限戦略を基に算出して交配させられたフラグメントのサイズの比較を推定します。T DNAs の可能なタンデム配置を特定します。ガイドとして図 4 aを使用して、予想されるフラグメントのサイズを計算します。
注記: 交配させられたフラグメントのサイズが計算されるものと一致しません、する場合、現在の統合の最も可能性の高い 1 つは完了。
- T DNA の右側 (図 7) と左 (図 7 b) のプローブで検出されたフラグメントの数を比較します。
- 4.1.1.2 (図 4 b) の手順で示した戦略にしたがって調製されたしみを分析する際のマーカー バンドのサイズに基づいてブロットに交雑のフラグメントのサイズを見積もるし、予想されるサイズと比較します。そのまま挿入には、定義済みの長さを持つ 1 つのフラグメントが得られます。予定の長さの任意の偏差は、不完全な統合 (図 7) を示します。
- 分析は、4.1 のステップで使用される制限戦略によって異なります。4.1.1.1 (図 4 a) の手順で示した戦略にしたがって調製されたしみを分析する場合は、検出されたフラグメントの数します。この戦略では交配させられたフラグメントの数は T DNA の統合の数を指します。
- 再交配 (省略可能) のためのしみを除去します。
注: 同じしみを連続で交配して異なるプローブを持つことができます。新しいプローブを持つ前に、しみから以前ハイブリッド プローブを削除します。- しみからプローブを削除するには、とその DNA 側を下向きにトレイにしみを配置します。0.5 %sds の余剰をトレイに注ぐ。2-5 分の膜を沸騰させます。治療期間は、サイズと使用されるプローブの GC 含量に依存します。長く、GC 豊富なプローブには、長い治療が必要があります。
- ストリッピング後、別のプローブとしみを交配またはシール、-20 ° C で保存
結果
生成された10BIBAC GW システムを使用して、植物の ODM を勉強するため記者構造だった構造は、ゲートウェイ エントリ ベクトル力がある pENTR gm12に設計され、pBIBAC-バー-GW (図 1) ゲートウェイ LR 再結合反応を利用した挿入します。
シロイヌナズナの pDM19、位置 120 eYFP 読み枠で翻訳終止コドンを運ぶ mTurquoise eYFP 記者と BIBAC-バー-GW プラスミドで形質転換した (mTurquoise2 eYFP * 40) (図 2)10。合計で126 シロイヌナズナは変換 (14 ポット ポット当たり 9 植物).これらの植物の種子がプール、土とトレイに播種し、二週間前のグルホシネート アンモニウム溶液による治療を成長すること。(BIBAC-バー-GW の現在) にあるバーを発現苗のみ生き残るグルホシネート アンモニウム治療 (図 3)。合計では、分析種の 0.02% の変換効率に対応する 11 遺伝子組み換え pDM19 と変形は識別されました。
分離された 11 の遺伝子組み換え、DNA にしみが付くことだった T DNA の統合の数を決定する使用されます。そのため、ゲノム DNA で切られたBglII またはSca私 (図 4 aの工夫として戦略)。これらの制限の酵素の両方は T DNA シーケンス (図 7 a) に一度だけカットします。バーと eYFP コード領域を認識するプローブとの交配は、それぞれの DNA 断片の数の検出を許可しました。
記者行 (表 1) に T-DNA 挿入の数を推定できるしみにフラグメント個々 の DNA の数。バーと eYFP プローブを用いた単一交雑フラグメントには、単一 DNA の統合の存在が示されました。分析 11 遺伝子組み換えから六つは単一の統合を実施しました。統合の平均数は 1.2 でした。
6 行を単一 DNA の統合を実施、挿入されたレポーターの構造の整合性は、(図 4 bの工夫として戦略) に DNA のしみが付くことを使用してテストされました。ゲノム DNA は、バーと mTurquoise eYFP 融合遺伝子 (図 7 a) を含む 5.5 kb フラグメントをリリースするBglII とPciで切られました。EYFP に対してプローブは、予想されるフラグメントを検出する使用されました。テストしたすべての植物は、そのままフラグメントを運んだ。フラグメントを検討メモは左と右 T-DNA 境界線を除外し、しかし興味の遺伝子を含む部分のみ全体 T - dna の整合性を検査しません。
蛍光レポーター遺伝子の発現は、T-DNA のゲノムの位置のみが異なる独立した単一コピー トランスジェニックは行で決定されました。CaMV 35S プロモーターに駆動される mTurquoise eYFP 記者の相対的な成績レベルは、ゲノムの位置は断固としたな10そのまま、単一コピーの統合を運ぶ 4 DM19 記者行 Rt-qpcr によって測定されました。行の間レポーター遺伝子発現レベルの変動はマイナーだった: mTurquoise eYFP RNA レベルでの最大の違いは 2 倍 (図 8 a)。
次に、ODM は、これらの記者行を行った。独立したレポーターの 4 行のうち 3 人は幾分同じような ODM 効率 (図 8 b) を示した。ただし、1 行、DM19 [4] 1、他の線と比較して非常に低い ODM 効率が得られました。ODM はローカルのゲノムのコンテキストで影響を受けることが示唆されました。どのような方法で DM19 で T DNA の統合のローカル ゲノム コンテキスト [4] 1 とは異なる他の行を識別する残っています。非形質転換植物におけるゲノム DNA の統合サイトでアクティブおよび非アクティブのクロマチン マークで使用可能なデータセットの解析は、答え10を提供しませんでした。

図 1: pBIBAC GW のベクトルの機能マップします。グルホシネート (バー) にどちらかの抵抗性または植物の選択マーカーとして種皮 (下流) にした DsRed 蛍光 pBIBAC GW 誘導体があります。両方のベクトル、カナマイシン耐性遺伝子は細菌の選択マーカーです。AttR1 およびattR2 サイトのccdBカセットは再結合を表す緑の矢印の間にゲートウェイ。この図の拡大版を表示するのにはここをクリックしてください。

図 2: 突然変異誘発記者コンストラクト。MTurquoise eYFP レポーター遺伝子は、CaMV 35S プロモーターによって駆動されます。MTurquoise の地域のコーディングは、コード領域の塩基位置 120 C の突然変異を運ぶ、時期尚早の並進停止コドン TAA、および融合タンパク質の翻訳の早期終了に終って、eYFP が融合されます。3 ' Nopaline 合成酵素 (nos 3') 起こる信号を使用して、構築22の転写を終了します。核局在化信号 (NLS) は、核に翻訳されたタンパク質をターゲットに使用されます。この図の拡大版を表示するのにはここをクリックしてください。

図 3: グルホシネート アンモニウム治療前後シロイヌナズナ実生でトレイがいっぱい。PBIBAC-バー-GW T にあるバーの遺伝子を発現していない苗-DNA グルホシネート アンモニウム溶液を噴霧されて死ぬ。写真は、2 回散布されて後、播種後 14 日目、グルホシネート アンモニウムと (B) 10 日後に散布する前に苗 (A) の同じトレイを表示します。この図の拡大版を表示するのにはここをクリックしてください。
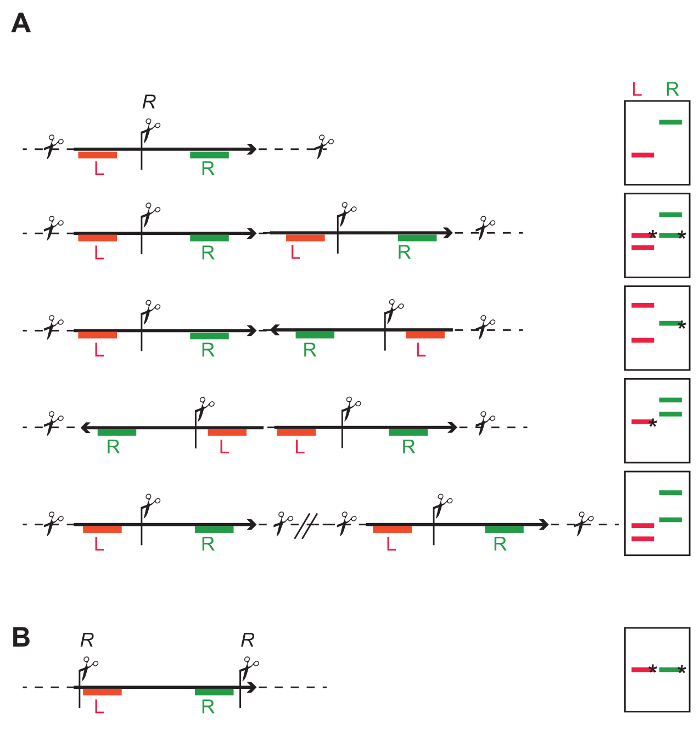
図 4: 数との厳守を識別する一般的な DNA 制限戦略挿入 T DNAs 。T DNA の真ん中に 1 つ (A) 制限サイト (R) により、左 (赤 L) と T DNA (緑 R) の右側の部分の独立した調査。右の漫画は別のバンド パターンは DNA にしみが付くことで得られたシングル- または多-コピー T DNA の統合によってことを示します。バンドが付いて、* 他のバンドの長さによって異なります並ぶゲノム DNA に最も近い制限サイト定義された長さがあります。単一挿入:L と R 両方を与える 1 つの独立したフラグメントをプローブします。予想される平均フラグメントのサイズは、ゲノムの制限サイトの周波数に基づいて計算できます。最小サイズは、統合の終わりは調査して T DNA はそのままかどうかに応じて制限サイトから左の境界線 (LB) または右の境界線 (RB) までの距離です。タンデム繰り返し:L と R のプローブを与える両方の 2 つのフラグメント。各プローブは並ぶゲノム DNA のフラグメントの 1 つが含まれています、2 番目のフラグメントが予想されるサイズと両方のプローブによって識別されます。反転繰り返し:統合カセットの方向によって、1 L と 2 つの R フラグメントや 2 つの L など 1 R のいずれかを識別できます。個々 の単一挿入:結果は多数の独立したフラグメント、フラグメントの数は、統合の数に対応します。(B) 制限サイト T DNA の四肢に許可制限のサイト間フラグメントの整合性を判断します。この図の拡大版を表示するのにはここをクリックしてください。
図 5: 制限のパターンと一致するの透明性とアガロースゲル。(A) DNA 消化EcoRagarose のゲルに私が表示されます。離散衛星バンドの存在によって DNA の適切な消化力を示します。スロットと透明度のマーカー バンドの位置が可能になり後で簡単に (B) のマーキングは、交配のサイズを計算します。ここでは、MRC オランダ マーカー (青と赤) が使用されますが、m. によって示されるこの図の拡大版を表示するのにはここをクリックしてください。
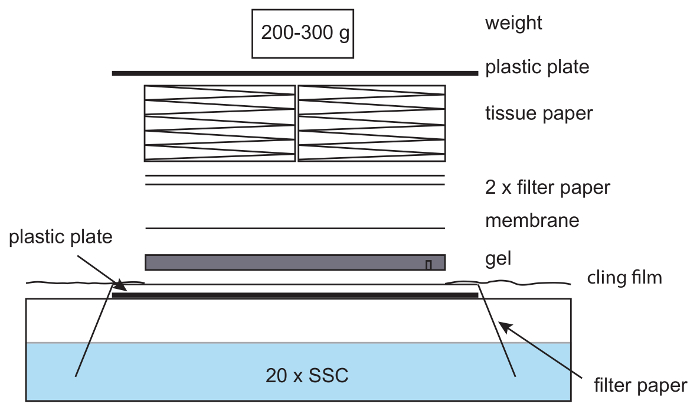
図 6: キャピラリーにしみが付くことのためのセットアップ。セットアップをしみ毛細血管、フィルター ペーパーはプラスチック板に 20 x SSC バッファーにぶら下がっている紙の先端を配置します。紙は 20 と接液 x SSC と、上部に配置 agarose のゲルに続いてナイロン膜、フィルター ペーパーおよび組織のスタック。軽量化は、上部に配置されます。ゲル、紙と膜の間の空気の泡を削除に注意です。サランラップは、セットアップの乾燥を避けるために使用されます。この図の拡大版を表示するのにはここをクリックしてください。
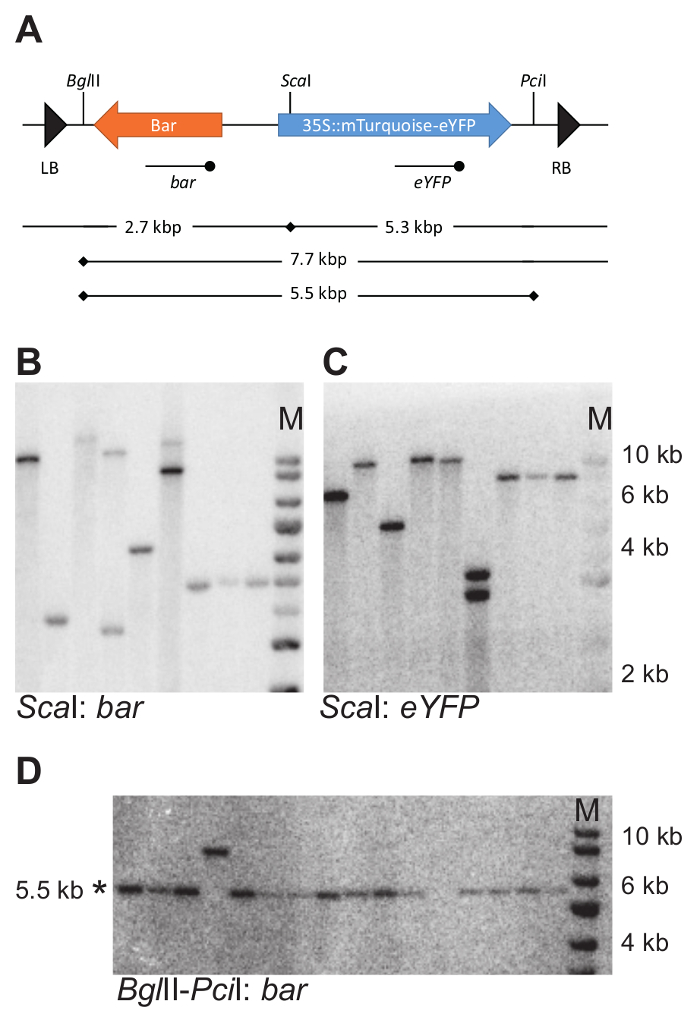
図 7: DNA の戦略と実験結果しみが付くことの例です。(A) DNA は、数と T DNA の統合の厳守を決定する戦略をしみが付きます。T DNA 内で選択した制限酵素の切断位置は、垂直バーで示されます。消化されたゲノム DNA の交配のためのeYFPとバーのプローブは、T-DNA の端末点と線を使用して示されます。(B-D)例の DNA のしみ。ゲノム DNA は、 Sca私としみだった (BとC) は、バーとeYFPの両方のプローブとプローブで切られました。ゲノム DNA は、 BglII およびPciカット私は、バープローブとプローブします。そのままフラグメントは、5.5 kbp の範囲のサイズ (D) です。D のサンプルのセット B と C に示したものとは異なることに注意してください *; 予想されるフラグメントのサイズを示しますM のマーカー。B、C、および D に同じサイズのマーカーが使用されます。この図の拡大版を表示するのにはここをクリックしてください。
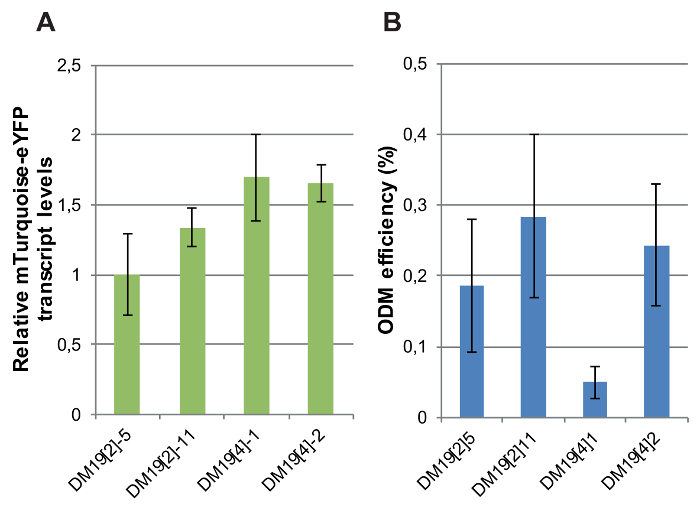
図 8: mTurquoise eYFP 発現レベルと独立した mTurquoise eYFP 記者行で ODM 効率。(A) 相対 mTurquoise eYFP トラン スクリプト レベル DM19 記者ラインで RT qPCR による測定します。正規化のアクチンの転写レベルが使用されました。(B) ODM 効率 DM19 記者ラインで測定します。A と B、バーの少なくとも 5 つの生物学的複製の平均を示します。誤差範囲を示す SEM.この図の拡大版を表示するのにはここをクリックしてください。
| T-DNA 遺伝子座の種類 | T DNA の統合の nr | 記者行 | 検出されたフラグメント数 | 整合性 | |||
| Sca私 | BglII | BglII/PciI | |||||
| バー | eYFP | バー | eYFP | eYFP | |||
| 単一軌跡統合 | 1 | [2] 19-2 | 1 | 1 | 1 | 1 | + |
| 1 | [2] 19-5 | 1 | 1 | 1 | 1 | + | |
| 1 | [2] 19-9 | 1 | 1 | 1 | 1 | + | |
| 1 | [2] 19-11 | 1 | 1 | 1 | 1 | + | |
| 1 | 19 [4]-1 | 1 | 1 | 1 | 1 | + | |
| 1 | 19 [4]-2 | 1 | 1 | 1 | 1 | + | |
| 2、反転を繰り返す | [2] 19-10 | 2 | 1 | 1 | 1 | + | |
| 2、不完全な統合 | [2] 19-3 | 1 | 2 | 1 | 1 | ND | |
| 複数の軌跡の統合 | 2 | [2] 19-6 | 2 | 2 | 2 | ND | |
| 2 | [2] 19-7 | 2 | 2 | 2 | 2 | ND | |
| 3/4 | [2] 19-1 | 4 | 3 | 3 | 3 | ND | |
| ND-未定します。 | |||||||
表 1:DNA pDM19 の変換後分離された遺伝子組み換えのデータをしみが付くことの要約です。
ディスカッション
生成する重要な遺伝子組み換え transgene の 1 つ、そのまま統合は使用バイナリ ベクトルの選択です。BIBAC 家族のベクトルは、種植物多く23,24,25,26,27,28に興味のシーケンスを提供する使用されています。BIBAC ベクトル、BIBAC-GW を含む高効率単一コピーの統合をもたらす: 1 行挿入の平均数は 1.5 に 2、最も一般的に使用されるバイナリ ベクトル5,9,の 3 以上を比較29. BIBAC GW ベクトル他 BIBAC ベクトルと比較して主要な改善として興味のシーケンス容易にゲートウェイ再結合サイト12を使用して挿入することができます。変更されたベクトル ベクトル BIBAC 従来のクローン作成戦略で使用する場合の一般的な問題を克服する: i) 固有の制限のサイトと ii) 低 DNA の非常に限られた数をもたらします。ゲートウェイ再結合サイトは、遺伝子組換え植物を生成するための他のバイナリ ベクトルは魅力的な代替 BIBAC GW のベクトルを作る。
ここ一連の BIBAC GW 誘導体の興味のシーケンスを含んでいる、植物情報の変換、および DNA 数と正常遺伝子のシーケンス解析をしみの生成からのプロトコルを説明しています。プロトコルのいくつかは本稿で報告したゲートウェイのクローニング、細菌と植物形質転換、エレクトロポレーション多くの実験室で一般的な方法、わずかな変更も行えます。A. 根頭がんしゅ病菌のエシェリヒア属大腸菌のベクトルのシングル コピーである BIBAC GW を知ることが重要です。したがって、DNA を隔離するときは、収量が低いです。分離手順をスケール アップすることをお勧めします。
遺伝子組み換え DNA に複数の統合を運ぶで導入遺伝子は、ジーンサイレンシング1,の2,の4、30の影響を受ける多くの場合、したがって、ほとんどのアプリケーション避けるべき。そのまま、単一の統合と遺伝子組換え植物を識別するためには、DNA のしみの分析を使用することをお勧めします。DNA 以外の方法ながらしみが付く使用できます T DNA コピー数および形質転換線トン Dna の完全性を決定する (分離分析、テール PCR、定量的 PCR (qPCR) およびデジタル液滴 PCR) 労働集中、DNA にしみが付くことが多いが、選択の方法。分離分析は複数の間で区別することは、単一の単一遺伝子 T DNA 統合。TAIL-PCR しばしば過小見積もりコピー数は、1 つ以上の DNA の統合は現在31qPCR は、信頼性の高い結果31,32の精巧な最適化を必要がある場合に特に。必要な機器が利用可能な31の場合、デジタル液滴 PCR、コピー数検出のためではなく正確な方法です。DNA にしみが付くことの加えられた利点はすべて PCR ベース テクニックで逃したが簡単に切り捨てられた T Dna の安易な検出。
Dna のしみとしみ断章の交雑が信号とサイズでも識別できる必要があります。いくつかの要因は、DNA にしみが付くことの結果に影響を与える知られています。(図 4に示されている戦略) の制限の酵素の適切な選択だけでなく、サイズ マーカー、質の良い十分な DNA が必要。シロイヌナズナのゲノムは、DNA の未満 2 μ g はよく識別の断片を得られません。大きいゲノムを扱う場合より多くの DNA が必要です。シロイヌナズナDNA の十分な量を得るためには、花の組織や 1 週間の古い苗を使用できます。1 つのペトリ皿上に成長した苗のバッチは、2-8 μ g の DNA を得られます。分離中に DNA の分解を避けるためには、高速の植物材料の処理に注意をすべき。さらに、ゲノム DNA を核酸、によるその劣化を抑えるトリス EDTA で再停止され、4 ° C で保存する必要がありますではなく DNA に刻み目をつける人を防ぐために-20 ° C 凍結融解のサイクルを繰り返します。よくわからない全ての DNA サンプルが完全に消化される場合は、内因性、ユニークなゲノム領域を認識プローブ DNA のしみを rehybridize することをお勧めします。Transgenic または内因性シーケンスを識別するためのプローブの順序を選択すると、のみ一意のシーケンスを選択することが重要です。交配させられたフラグメントのサイズを正確に決定することができるには、DNA の位置ゲル スロットおよび DNA マーカー バンド マークする必要が透明性 (図 5 b) にエチジウム ブロマイド染色ゲル (図 5 a) を視覚化するとき、uvtransilluminator。場合にマーカー シーケンスを行うプローブ DNA、ない交配または交配は部分的、断片を交配させることのサイズを追跡する唯一の方法です。
注意は良いシグナルとフラグメントのサイズの推定値を達成するために、しみの結果の解釈は簡単です。のみ 1 つの制限の酵素を使用して、異なる交配させることを調査するときどちらかを検出 T DNA の左または右の部分、検出されたフラグメントの数は T-DNA 挿入の数を反映します。例えば、バー (図 7 b) と強化された黄色い蛍光蛋白質 (eYFP) (図 7)、図 7 aに示すように戦略を使用して別のプローブとハイブリダイズ図 7 bCが同じ DNA しみを示しています。4 を除く、すべての車線両方のしみにフラグメントの数と同じ数を表示: 6 行と他のすべての行に対して 1 つのフラグメントのための 2 つのフラグメント。この検出されたフラグメントの数は、T-DNA 挿入数です。
左プローブのバインドのいずれかで検出されたフラグメントの数や、(図 7 b、C、4 線) については T DNA の右側の部分が異なる場合、不完全な挿入が存在、または T Dna が直列に挿入されています。タンデム挿入 (図 4 a、右側のパネル)、T DNA フラグメントの 1 つの非ランダムなフラグメント長を表示および制限戦略に基づいて期待されるハイブリッド フラグメントのサイズを比較することで識別することができます。追加のしみが付く戦略は、T-DNA 挿入のタンデム配置を確認する必要ことがあります。4 (図 7 bC) の行に示されているサンプルでは、2 つの挿入が反転繰り返し方向に並べられます。
T-DNA、またはその一部の厳守を見積もるとき交雑のフラグメントの長さ計算できますに基づいて制限戦略。予想サイズから任意の偏差は、不完全な挿入の存在を示します。たとえば、図 7の第 4、レーンで交雑フラグメント移行 8 kbp (予想される 5.5 kbp) ではなく制限のサイトのいずれかの不足のための高められたフラグメント サイズを示すにします。
BIBAC GW ベクトルは、植物種数の単一コピーのままの統合を生成するための優れたツールです。ここで報告されるプロトコルは、興味の遺伝子の 1 つ、そのままインテグレーション環境で植物を識別するために信頼性の高い手順を提供します。
開示事項
著者はない競合する金銭的な利益やその他の利害の関係を宣言します。
謝辞
この研究は、オランダ技術財団 STW (12385) オランダ組織科学的研究 (NWO) の一部であるし、するが部分的で、経済情勢の大臣 (MS に OTP グラント 12385) 資金を供給によってサポートされます。 我々 は pCH20、BIBAC GW ベクトルのバックボーンを提供するキャロル ・ マクゴーギー ・ ハミルトン (コーネル大学、米国) をありがちましょう。
資料
| Name | Company | Catalog Number | Comments |
| Kanamycin sulphate monohydrate | Duchefa | K0126 | |
| Gentamycin sulphate | Duchefa | G0124 | |
| Rifampicin | Duchefa | R0146 | |
| Tetracycline hydrochloride | Sigma | T-3383 | |
| DB3.1 competent cells | Thermo Scientific - Invitrogen | 11782-018 | One Shot ccdB Survival 2 T1R Competent Cells (A10460) by Invitrogen or any other ccdB resistant E. coli strain can be used instead |
| DH10B competent cells | Thermo Scientific - Invitrogen | 18290-015 | |
| Gateway LR clonase enzyme mix | Thermo Scientific - Invitrogen | 11791-019 | |
| tri-Sodium citrate dihydrate | Merck | 106432 | |
| Trizma base | Sigma-Aldrich | T1503 | |
| EDTA disodium dihydrate | Duchefa | E0511 | |
| Proteinase K | Thermo Scientific | EO0491 | |
| Bacto tryptone | BD | 211705 | |
| Yeast extract | BD | 212750 | |
| Sodium chloride | Honeywell Fluka | 13423 | |
| Potassium chloride | Merck | 104936 | |
| D(+)-Glucose monohydrate | Merck | 108346 | |
| Electroporation Cuvettes, 0.1 cm gap | Biorad | 1652089 | |
| Electroporator Gene Pulser | BioRad | ||
| Magnesium sulfate heptahydrate | Calbiochem | 442613 | |
| D(+)-Maltose monohydrate 90% | Acros Organics | 32991 | |
| Sucrose | Sigma-Aldrich | 84100 | |
| Silwet L-77 | Fisher Scientific | NC0138454 | |
| Murashige Skoog medium | Duchefa | M0221 | |
| Agar | BD | 214010 | |
| Glufosinate-ammonium (Basta) | Bayer | 79391781 | |
| Restriction enzymes | NEB | ||
| Ethidium Bromide | Bio-Rad | 1610433 | |
| Electrophoresis system | Bio-Rad | ||
| Sodium hydroxide | Merck | 106498 | |
| Hydrochloric acid | Merck | 100316 | |
| Blotting nylon membrane Hybond N+ | Sigma Aldrich | 15358 | or GE Healthcare Life Sciences (RPN203B) |
| Whatman 3MM Chr blotting paper | GE Healthcare Life Sciences | 3030-931 | |
| dNTP | Thermo Fisher | R0181 | |
| Acetylated BSA | Sigma-Aldrich | B2518 | |
| HEPES | Sigma-Aldrich | H4034 | |
| 2-Mercaptoethanol | Merck | 805740 | |
| Sephadex G-50 Coarse | GE Healthcare Life Sciences | 17004401 | or Sephadex G-50 Medium (17004301) |
| Dextran sulfate sodium salt | Sigma-Aldrich | D8906 | |
| Sodium Dodecyl Sulfate | US Biological | S5010 | |
| Salmon Sperm DNA | Sigma-Aldrich | D7656 | |
| Sodium dihydrogen phosphate monohydrate | Merck | 106346 | |
| Storage Phosphor screen and casette | GE Healthcare Life Sciences | 28-9564-74 | |
| Phosphor imager | GE Healthcare Life Sciences | Typhoon FLA 7000 | |
| UV Crosslinker | Stratagene | Stratalinker 1800 | |
| cling film (Saran wrap) | Omnilabo | 1090681 | |
| Agarose | Thermo Scientific - Invitrogen | 16500 | |
| Boric acid | Merck | 100165 | |
| DNA marker ‘Blauw’; DNA ladder. | MRC Holland | MCT8070 | |
| DNA marker ‘Rood’; DNA ladder | MRC Holland | MCT8080 | |
| Hexanucleotide Mix | Roche | 11277081001 | |
| Large-Construct Kit | Qiagen | 12462 | |
| Heat-sealable polyethylene tubing, clear | various providers | the width of the tubing should be wider than that of blotting membrane | |
| Heat sealer | |||
| Membrane filter disk | Merck | VSWP02500 | |
| Magnesium chloride | Merck | 105833 | |
| Hybridization mesh | GE Healthcare Life Sciences | RPN2519 |
参考文献
- Jorgensen, R. A., Cluster, P. D., English, J., Que, Q., Napoli, C. A. Chalcone synthase cosuppression phenotypes in petunia flowers: comparison of sense vs. antisense constructs and single-copy vs. complex T-DNA sequences. Plant Mol Biol. 31 (5), 957-973 (1996).
- Stam, M., et al. Post-transcriptional silencing of chalcone synthase in Petunia by inverted transgene repeats. Plant J. 12, 63-82 (1997).
- Stam, M., Viterbo, A., Mol, J. N., Kooter, J. M. Position-dependent methylation and transcriptional silencing of transgenes in inverted T-DNA repeats: implications for posttranscriptional silencing of homologous host genes in plants. Mol Cell Biol. 18 (11), 6165-6177 (1998).
- Jin, Y., Guo, H. S. Transgene-induced gene silencing in plants. Methods Mol Biol. 1287, 105-117 (2015).
- Oltmanns, H., et al. Generation of Backbone-Free, Low Transgene Copy Plants by Launching T-DNA from the Agrobacterium Chromosome 1[W][OA]. Plant Physiol. , (2010).
- Ye, X., et al. Enhanced production of single copy backbone-free transgenic plants in multiple crop species using binary vectors with a pRi replication origin in Agrobacterium tumefaciens. Transgenic Res. , (2011).
- Hamilton, C. M. A binary-BAC system for plant transformation with high-molecular-weight DNA. Gene. 200 (1-2), 107-116 (1997).
- Frary, A., Hamilton, C. M. Efficiency and stability of high molecular weight DNA transformation: an analysis in tomato. Transgenic Res. 10 (2), 121-132 (2001).
- Vega, J. M., et al. Agrobacterium-mediated transformation of maize (Zea mays) with Cre-lox site specific recombination cassettes in BIBAC vectors. Plant Mol Biol. 66 (6), 587-598 (2008).
- Anggoro, D. T., Tark-Dame, M., Walmsley, A., Oka, R., de Sain, M., Stam, M. BIBAC-GW-based vectors for generating reporter lines for site-specific genome editing in planta. Plasmid. 89, 27-36 (2017).
- Hamilton, C. M., Frary, A., Lewis, C., Tanksley, S. D. Stable transfer of intact high molecular weight DNA into plant chromosomes. Proc Natl Acad Sci U S A. 93 (18), 9975-9979 (1996).
- Belele, C. L., Sidorenko, L., Stam, M., Bader, R., Arteaga-Vazquez, M. A., Chandler, V. L. Specific tandem repeats are sufficient for paramutation-induced trans-generational silencing. PLoS Genet. 9 (10), e1003773 (2013).
- Shizuya, H., et al. Cloning and stable maintenance of 300-kilobase-pair fragments of human DNA in Escherichia coli using an F-factor-based vector. Proc Natl Acad Sci U S A. 89 (18), 8794-8797 (1992).
- Shi, X., Zeng, H., Xue, Y., Luo, M. A pair of new BAC and BIBAC vectors that facilitate BAC/BIBAC library construction and intact large genomic DNA insert exchange. Plant Methods. 7, 33 (2011).
- Woodman, M. E., et al. Direct PCR of Intact Bacteria (Colony PCR). Curr Protoc Microbiol. , A.3D.1-A.3D.7 (2016).
- Ausubel, F. M., et al. Mol Biol. Current Protocols in Molecular Biology. 1, (2003).
- Edwards, K., Johnstone, C., Thompson, C. A simple and rapid method for the preparation of plant genomic DNA for PCR analysis. Nucleic Acids Res. 19 (6), 1349 (1991).
- Clarke, J. D. Cetyltrimethyl ammonium bromide (CTAB) DNA miniprep for plant DNA isolation. Cold Spring Harb Protoc. (3), (2009).
- Lee, P. Y., Costumbrado, J., Hsu, C. Y., Kim, Y. H. Agarose Gel Electrophoresis for the Separation of DNA Fragments. J Vis Exp. (62), e3923 (2012).
- Green, M. R., Sambrook, J. . Molecular Cloning. , (2012).
- Sosa, J. M. Chromatography with Sephadex Gels. Anal Chem. 52 (6), 910-912 (1980).
- Depicker, A., Stachel, S., Dhaese, P., Zambryski, P., Goodman, H. M. Nopaline synthase: transcript mapping and DNA sequence. J Mol Appl Genet. 1 (6), 561-573 (1982).
- Feng, J., Vick, B. A., Lee, M. K., Zhang, H. B., Jan, C. C. Construction of BAC and BIBAC libraries from sunflower and identification of linkage group-specific clones by overgo hybridization. Theor Appl Genet. 113 (1), 23-32 (2006).
- Lee, M. K., et al. Construction of a plant-transformation-competent BIBAC library and genome sequence analysis of polyploid Upland cotton (Gossypium hirsutum L). BMC Genomics. 14, 208 (2013).
- Wang, W., et al. A large insert Thellungiella halophila BIBAC library for genomics and identification of stress tolerance genes. Plant Mol Biol. 72 (1-2), 91-99 (2010).
- Wu, C., et al. A BAC- and BIBAC-based physical map of the soybean genome. Genome Res. 14 (2), 319-326 (2004).
- Xu, Z., et al. Genome physical mapping from large-insert clones by fingerprint analysis with capillary electrophoresis: a robust physical map of Penicillium chrysogenum. Nucleic Acids Res. 33 (5), e50 (2005).
- Zhang, M., et al. Genome physical mapping of polyploids: a BIBAC physical map of cultivated tetraploid cotton, Gossypium hirsutum L. PLoS One. 7 (3), e33644 (2012).
- Frary, A., Hamilton, C. M. Efficiency and stability of high molecular weight DNA transformation: An analysis in tomato. Transgenic Res. , (2001).
- Stam, M., Viterbo, A., Mol, J. N. M., Kooter, J. M. Position-Dependent Methylation and Transcriptional Silencing of Transgenes in Inverted T-DNA Repeats: Implications for Posttranscriptional Silencing of Homologous Host Genes in Plants. Cell Biol. 18 (11), 6165-6177 (1998).
- Głowacka, K., Kromdijk, J., Leonelli, L., Niyogi, K. K., Clemente, T. E., Long, S. P. An evaluation of new and established methods to determine T-DNA copy number and homozygosity in transgenic plants. Plant Cell Environ. 39 (4), 908-917 (2016).
- Stefano, B., Patrizia, B., Matteo, C., Massimo, G. Inverse PCR and Quantitative PCR as Alternative Methods to Southern Blotting Analysis to Assess Transgene Copy Number and Characterize the Integration Site in Transgenic Woody Plants. Biochem Genet. 54 (3), 291-305 (2016).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
ISSN 2578-6326
Copyright © 2023 MyJoVE Corporation. All rights reserved
当社はcookieを使用しています。
「続行」をクリックすることで、当社のcookieへの同意となります。