Method Article
Geração de plantas transgênicas com inserções de cópia única usando vetor binário BIBAC-GW
Neste Artigo
Resumo
Usando um vetor binário pBIBAC-GW faz gerar plantas transgénicas com inserções de cópia única intactas, um processo fácil. Aqui, uma série de protocolos é apresentada que guia o leitor através do processo de geração de plantas transgênicas de Arabidopsis e teste as plantas para integridade e copiar o número de inserções.
Resumo
Ao gerar plantas transgênicas, em geral, o objetivo é ter expressão estável de um transgene. Isto requer única intacta de uma integração do transgene, como cópia multi integrações são frequentemente sujeitas a silenciamento de genes. O vetor binário compatível com o Gateway com base em cromossomos artificiais bacterianos (pBIBAC-GW), como outros derivados de pBIBAC, permite a inserção dos transgenes cópia única com alta eficiência. Como uma melhoria para o pBIBAC original, uma gaveta de Gateway foi clonada em pBIBAC-GW, para que as sequências de interesse podem agora ser facilmente incorporadas a transferência de vector DNA (T-DNA) clonando Gateway. Comumente, a transformação com pBIBAC-GW resulta em uma eficiência de 0,2-0,5%, segundo o qual metade dos transgênicos a transportar uma integração de cópia única intacta do T-DNA. Os vetores de pBIBAC-GW estão disponíveis com resistência ao glufosinato-amônio ou DsRed fluorescência em revestimentos de semente para seleção em plantas e com resistência à canamicina como uma seleção em bactérias. Aqui, uma série de protocolos é apresentada que guia o leitor através do processo de geração de plantas transgênicas usando pBIBAC-GW: a partir de recombinar as sequências de interesse em vetor de escolha, para plantar a transformação com pBIBAC-GW Agrobacterium, seleção dos transgênicos e teste as plantas para integridade e cópia número de inserções usando a DNA mancha. Atenção é dada a concepção de uma estratégia de mancha de DNA para reconhecer integrações de single e multi cópias em único e múltiplos loci.
Introdução
Ao gerar plantas transgênicas, geralmente, o objetivo é ter o transgene integrado estàvel expressa. Isto pode ser conseguido por integrações intacta cópia única de um transgene. Múltiplas integrações podem levar ao aumento da expressão de um transgene, mas também para o silenciamento de genes. Silenciamento de transgenes é mais provável se sequências inseridas são dispostas em tandem ou repetições invertidas1,2,3,4. Vetores binários são usadas como naves em Agrobacterium-mediada experimentos de transformação para entregar as sequências de interesse em genomas de planta. O número de integrações no genoma planta é dependente do número de cópia do vetor binário em Agrobacterium tumefaciens5,6. Muitos vetores binários comumente usados são vetores de alta cópia e, portanto, produzir um número de cópia do transgene média alta: cópias de 3.3 para 4,9 em Arabidopsis5.
O número de integrações de T-DNA pode ser diminuído usando vetores binários que têm um número baixo-cópia em a. tumefaciens, tais como BIBAC7, ou através do lançamento de um T-DNA da . tumefaciens cromossoma5. O número médio de integrações do transgene em tais casos é inferior a 25,8,9,10. Devido a ser cópia única na . tumefaciens e também em Escherichia coli, BIBAC-derivados podem manter e entregar as construções tão grandes quanto 150 kb,11.
Compatível com o GW BIBAC vetores10,12 permitir fácil introdução de genes de interesse para o vetor usando Gateway clonagem. O uso da tecnologia de Gateway simplifica o procedimento de clonagem, mas também supera os problemas comuns associados com13,grandes vetores de baixo-cópia-número14, como um baixo rendimento de DNA e uma seleção limitada de restrição exclusiva sites disponíveis para clonagem de7,11. Os derivados de pBIBAC-GW estão disponíveis com qualquer resistência ao glufosinato-amônio (pBIBAC-BAR-GW) ou DsRed fluorescência em revestimentos de semente (pBIBAC-RFP-GW) para seleção em plantas (Figura 1)10,12. Para os dois vetores, um gene de resistência canamicina é usado como marcador de seleção em bactérias.
Combinam os vetores de pBIBAC-GW: design (1) fácil e manipulação genética em e. colie integrações de cópia única (2) intacto em planta com alta eficiência. O rendimento de vetores de pBIBAC-GW em integrações de média 1,7 em Arabidopsis com aproximadamente metade das plantas transgênicas carregando um único integrado T-DNA10.
Expressao de transgenes é um requisito para a maioria dos transgênicos gerados. Expressão do transgene estável pode ser conseguida integrações intactas, cópia única. Trabalhar com plantas transgênicas carregando integrações intactas, cópia única é, no entanto, ainda mais importante, se por exemplo, o objetivo é estudar a eficiência de processos baseados em cromatina, tais como recombinação, mutagênese ou reparação e a dependência destes processos no genoma e a estrutura da cromatina no local da inserção. Para nosso interesse, para estudar a dependência de mutagênese do oligonucleotide dirigido (ODM) no contexto genômico do local, um conjunto de linhas de repórter com integrações intactos, cópia única de um gene repórter de mutagénese foi gerado (Figura 2)10. Usando este conjunto de linhas, foi demonstrado que a eficiência ODM varia entre loci transgénicos integrado em diferentes localizações genômicas, apesar dos níveis de expressão do transgene ser bastante semelhante.
Protocolo
1. Inserir sequências de interesse em vetor binário
-
Prepare a entrada de Gateway e vetores binários.
- Isole o vetor de Gateway de entrada que contém um fragmento de DNA ou o gene de interesse usando um kit de mini-preparação de acordo com as sugestões do fornecedor.
Nota: BIBAC-GW vetores requerem o uso de canamicina (Km) para a seleção de bactérias, portanto, usa um vetor de entrada com outro marcador de resistência ao invés de canamicina. Por exemplo, o vetor pENTR-gm, carregando um gene de resistência de gentamicina, é uma boa escolha de12. - Propagar e isolar o vetor BIBAC-GW de interesse. Use uma cepa de Escherichia coli que é resistente à toxicidade do gene ccdB presente dentro da gaveta de Gateway. BIBAC-vetores isolados usando protocolos ou kits projetados especificamente para grandes plasmídeos de acordo com as sugestões do fornecedor.
- Isole o vetor de Gateway de entrada que contém um fragmento de DNA ou o gene de interesse usando um kit de mini-preparação de acordo com as sugestões do fornecedor.
-
Realize uma reação de Gateway.
- Prepare a reação de recombinação LR de acordo com as sugestões do fornecedor. Misturar os componentes a seguir em um tubo de microcentrifugadora de 1,5 mL à temperatura ambiente (RT): 100 – 300 ng clone de entrada (supercoiled), 300 ng do vetor BIBAC-GW, amortecedor da reação Clonase LR (concentração final: 1 x). Ajuste o volume da mistura para 16 µ l com TE (10 mM Tris, 1 mM de EDTA, pH 8.0). Finalmente, adicione 4 µ l do mix de enzima Clonase LR e mistura num Vortex. Incube a mistura a 25 ° C, durante 1 h.
- Finalizar a reação de LR adicionando 2 µ l de solução de Proteinase K (2 µ g / µ l) para a mistura preparada na etapa 1.2.1. Misturar e incubar a 37 ° C por 10 min.
-
Transforme e. coli com a mistura de reação de Gateway por eletroporação.
- Do desalt mistura reacional LR antes da eletroporação.
Nota: Este passo é essencial para sucesso eletroporação. Abaixo uma diálise método é descritos, mas outros métodos tais como precipitação com acetato de sódio e etanol também pode ser usado.- Prepare a instalação para diálise de filtro da reação de LR. 20 mL de água desionizada ultrapura despeje uma placa de Petri estéril. Coloque um disco de filtro de membrana (tamanho de poros = 0,025 µm) na superfície da água.
- Pipetar a reação inteira de LR cuidadosamente na parte superior da membrana e permita que a mistura Dialize em RT por 1h.
- Adicione 5 µ l do mix LR demolhado para electro-competente DH10B células em uma cubeta de eletroporação (0,1 cm). Electroporate as células (1.5 V/cm, resistência 200 Ω, capacitância 25 µF) e imediatamente, adicionar 1 mL de meio de repressão (SOC) previamente aquecido Super Catabolite ideal para as células, seguidas de incubação a 37 ° C por 45 min, 180 rpm. (Meio SOC, extrato de 1 l: 20 g de Bacto triptona, 5 g de fermento, 0,5 g de NaCl, 2,5 mL de 1M de KCl, 20 mL de 1 M filtro esterilizado glicose).
Nota: DH10B células podem ser substituídas por outras células de Escherichia coli que estàvel mantêm grandes plasmídeos.
Nota: As condições de eletroporação ideal são dependentes do dispositivo electroporation usado. - Pelota de bactérias em velocidade maxima por 30 s usando microcentrifuga, remover excesso SOC e resuspenda o pellet em aproximadamente 50-100 µ l de meio Luria-Bertani (LB). Espalhar as bactérias em placas de canamicina-LB (Km-LB) (concentração Km, 40 µ g/mL) e incubar as placas a 37 ° C durante a noite. (Meio LB, 1 l: 10 g de Bacto triptona, 5 g de fermento extrato, 10 g de NaCl. Para o meio sólido adicione o agar, 15 g/L).
- Do desalt mistura reacional LR antes da eletroporação.
-
Identificar os derivados de BIBAC-GW recombinados e isolar o Plasmídeo.
- Para ser capaz de crescer em placas de Km-LB, as células de Escherichia coli devem conter o plasmídeo BIBAC-GW recombinado, no qual a sequência de ccdB é substituída com a inserção desejada. Use colônia reação em cadeia da polimerase (PCR)15 para verificar se as colônias de bactérias na placa de contenham os plasmideos corretos, tendo a espinha dorsal BIBAC-GW e a inserção de interesse.
- Para identificar a espinha dorsal pBIBAC-BAR-GW, realizar uma reação de15 PCR com os iniciadores DM1969 5'-GCGACGAGCCAGGGATAG-3 'e DM1970 5'-ATCAGTGCGCAAGACGTGAC-3'. Este conjunto de cartilha amplifica um fragmento de bp 563 do gene bar .
- Para verificar a presença da espinha dorsal BIBAC-RFP-GW, realizar uma reação de15 PCR, utilizando primers M737 5'-CGTGTAAAAAGCTTAGACTG-3 'e M892 5'-AACAGATGGTGGCGTCCC-3'. Esta combinação de cartilha amplifica um fragmento de bp 791 sobrepostas a sequência de promotor e rfp de cruciferin.
- Realize reações de15 PCR utilizando primers específicos do gene para verificar a presença do inserto de interesse.
- Inocule uma única colônia positiva em 2 – 5 mL de meio LB contendo canamicina (40 µ g/mL) para isolamento de DNA16. Incube a 37 ° C em um agitador orbital a 180 rpm, durante a noite.
- Isolar o plasmídeo (consulte a etapa 1.1.2).
- Para ser capaz de crescer em placas de Km-LB, as células de Escherichia coli devem conter o plasmídeo BIBAC-GW recombinado, no qual a sequência de ccdB é substituída com a inserção desejada. Use colônia reação em cadeia da polimerase (PCR)15 para verificar se as colônias de bactérias na placa de contenham os plasmideos corretos, tendo a espinha dorsal BIBAC-GW e a inserção de interesse.
2. preparação da . tumefaciens para mergulhar Floral de Arabidopsis
-
Transforme BIBAC-GW derivados da . tumefaciens.
- Prepare células electro-competente de auxiliar de estirpe C58C1 carregando o pCH32 da . tumefaciens plasmídeo7. Cresce bactérias na presença de tetraciclina (5 µ g/mL) e rifampicina (100 µ g/mL) para selecionar para pCH32 e garantir o crescimento de células de Agrobacterium apenas.
- Adicionar 0,25 – 0,5 µ g de DNA de um derivado pBIBAC-GW, previamente dissolvido em 10 – 20 µ l estéril ultrapura água desionizada, para 20 µ l Agrobacterium pilhas competentes em cubetas de eletroporação (0,1 cm). Manter as células no gelo.
- Electroporate as células (1.5 V/cm, resistência 400 Ω, capacitância 25 µF). Imediatamente após a eletroporação, adicionar 1 mL de meio de SOC pré-aquecido (28 ° C) para as bactérias e incube as celulas a 28 ° C, durante 60 a 90 min.
- Espalhou-se 100 µ l e o resto das bactérias em chapas separadas de LB contendo rifampicina (100 µ g/mL), tetraciclina (5 µ g/mL) e canamicina (40 µ g/mL) e incubar no escuro a 28 ° C durante 1-2 dias.
-
Prepare a suspensão de Agrobacterium .
- PCR para verificar algumas das colônias a. tumefaciens do prato preparado no passo 2.1.4, para a presença do vetor correto (consulte a etapa 1.4.1).
- Raia uma única colônia confirmada para conter o vetor binário com o apropriado inserir uma placa LB contendo antibióticos (consulte a etapa 2.1.4). Cresce a 28 ° C durante a noite.
- Repita as estrias com uma única colônia obtida na etapa 2.2.2.
- Inocule uma colônia única em 2,5 mL de meio de LC suplementado com antibióticos (consulte a etapa 2.1.4) para preculture. Incube a 28 ° C, durante pelo menos 8 h ou durante a noite, a 180 rpm. (Média de LC, 10 l: 1G de Bacto triptona, 5 g de fermento extrato, 0,5 g de NaCl, 2,5 g de MgSO4 · 7 H2O, 2 g de Maltose).
- Adicione o preculture da etapa 2.2.4. até 250 mL LC suplementadas com antibióticos (consulte a etapa 2.1.4) e crescer a 28 ° C, a 180 rpm, durante a noite.
- Pelota a cultura por fiação a 5.500 x g, durante 12 min. Ressuspender o pellet em 100 mL de solução contendo 5% de sacarose, 0,05% Silwet L-77, 0.5 x MS. Pour a suspensão em um recipiente estéril para mergulhar floral das plantas.
3. transformação de Arabidopsis
-
Prepare as plantas de Arabidopsis para transformação.
- Cultivar plantas de Arabidopsis em uma estufa ou clima câmara de crescimento controlado até que eles são de floração (12 potes com 9 plantas por imersão).
- Prenda os parafusos primeiros para permitir mais parafusos secundários a emergir. As plantas estão prontas para mergulhar 4-6 dias após o recorte, quando as plantas têm muitas cabeças de flor imaturas e muitos não fertilizado siliques.
-
Mergulhando floral
- Mergulhe inflorescências para 5 a 10 s em Agrobacterium suspensão preparada na etapa 2.2.6. Use agitação suave.
- Embrulhar as partes acima do solo, das plantas em película aderente para manter a umidade alta e cobrir os potenciômetros da planta com uma caixa para manter as plantas no escuro. Incube as plantas 2 dias em uma câmara de estufa/crescimento.
- Remova a caixa e a película aderente e crescer as plantas para a maturidade em uma câmara de estufa/crescimento.
Nota: Para aumentar a eficiência de transformação, as mesmas plantas podem ser re-mergulhados 7 dias após o primeiro mergulho. - Colha as sementes. Piscina e analisar as sementes (T1) de plantas, transformadas com a construção da mesma, como um único conjunto.
-
Tela para plantas transgênicas.
- Para tela de plantas transgénicas transformadas com um derivado de pBIBAC-RFP-GW, analise as sementes usando microscopia de fluorescência. A fim de detectar DsRed expressão em revestimentos de semente, imagem as sementes em uma excitação de 560 nm e emissão de 600-650 nm. Separe as sementes fluorescentes fluorescente não homólogos usando fórceps.
- Para tela de plantas transgénicas transformadas com um derivado de pBIBAC-BAR-GW, semear as sementes em bandejas, enchidas com o solo (~ 2.500 sementes/0.1 m2). Para garantir um mesmo espalhando sementes bandejas, suspender as sementes em ágar de 0,1% em 0.5 x médio Murashige Skoog (MS) e espalhar as sementes usando uma pipeta de 1 mL.
Nota: Para estimular as sementes que germinam de forma síncrona, incubar as sementes pelo menos 2 dias a 4 ° C. Isso pode ser feito antes ou após a semeadura as sementes.- Pulverize as mudas com 0,5% solução de glufosinato-amônio 2 semanas e 3 semanas após a semeadura em bandejas. Use 500 mL da solução de glufosinato-amônio por 1 m2.
- Transferência de mudas para potes individuais de sobreviver. Uma imagem típica de uma bandeja com mudas antes e após o tratamento de glufosinato-amônio (segundo) são mostrados na Figura 3.
- Analisar as plantas resistentes ao glufosinato-amônio por PCR para a presença da construção de interesse (consulte a etapa 1.4.1. para primeiras demão).
- Isolar o DNA genômico de planta para o PCR usando o método descrito por Edwards et al . 17
4. caracterizando transgênicos para o número e a integridade das integrações de T-DNA
- Estratégia de digestões de restrição
Nota: Determine o número de integrações de T-DNA e sua integridade pela DNA mancha usando enzimas de restrição. Este método permite identificar único, mas também repetiu integrações no mesmo ou diferentes loci no genoma.- Use uma série de digestões de restrição para identificar os padrões de integração diferentes possíveis:
- Selecione uma enzima que corta uma vez no meio do T-DNA, para poderes independentemente sonda sequências de montante e a jusante do site restrição (Figura 4A e Figura 7A-C). Ver Figura 4Ae a lenda de figura de resultados esperados e interpretação.
- Selecione uma enzima ou combinação de enzimas, cortando toda a sequência de interesse de uma só vez (Figura 4B e Figura 7A-D). Qualquer desvio do comprimento conhecido indica truncamento da fita integrado.
Nota: tenha cuidado para usar somente as enzimas de restrição que não são sensíveis a metilação de citosina.
- Use uma série de digestões de restrição para identificar os padrões de integração diferentes possíveis:
- Prepare as amostras de DNA genômicas.
- Isole o DNA genômico de plantas carregando o construto de interesse. Um método de miniprep CTAB DNA pode ser usado para isolamento de DNA18. DNA análise de Arabidopsis DNA do borrão, 2 – 2,5 µ g de DNA genômico é necessária. Dissolva o DNA em 50 µ l de TE.
- Verificar a integridade do DNA por eletroforese de gel de16. DNA genômico intacto migra como uma banda discreta na parte superior do gel. Degradação do DNA pode ser reconhecida como a presença de uma mancha. Para evitar danos ao DNA devido ao congelamento-descongelamento repetido, amostras de DNA genômicas são mantidas a 4 ° C.
- Digeri o DNA genômico (2-2,5 µ g no caso de DNA genômico de Arabidopsis ) em um volume total de 50 µ l, durante a noite, usando condições de reserva, sugeridas pelo fornecedor da enzima.
- Em um tubo de ensaio, misture 2 – 2,5 µ g de DNA genômico de Arabidopsis , 5 µ l 10x buffer de restrição e 5 U da enzima de restrição num total de 50 µ l ultrapura deionizada.
- Adicione corante de carregamento (1 x concentração final) para as amostras de restrição antes de carregar em um gel. Para o bom acompanhamento visual, use um dos seguintes: comigrating com pequenos fragmentos (azul de bromofenol, 350 – 400 bp), ou comigrating com fragmentos maiores (Cylene cianol, 3 – 4 kbp).
- Funcione o gel de DNA.
- Prepare um longo (20 cm) 0.5 x TBE de gel de agarose19. A porcentagem de agarose no gel varia de acordo com os tamanhos de fragmento esperados. 0,8 – 1% separa otimamente fragmentos > 1 kb em tamanho. Use 1-1,5% de agarose para fragmentos < 1 kb. Não adicione o brometo de Ethidium ao gel. (5 x TBE, 54 l: 1G de Trizma base, 27,5 g de ácido bórico, 3,75 g de EDTA).
Nota: Para evitar a contaminação de DNA, bandejas de gel de uso que não são usadas para fracionar o plasmídeo e PCR ampliaram DNA. - Carrega as amostras no gel de19.
- Adicione marcadores de DNA para o gel que estão na faixa de tamanho dos fragmentos esperados. Carga de aproximadamente 1 µ g de marcador (50-250 ng do fragmento de tamanho diferente) sobre o gel para permitir a visualização pela luz UV.
- Tamanho-fractionate o DNA em uma tensão baixa (40 – 50 V/500 mA) durante a noite.
- Prepare um longo (20 cm) 0.5 x TBE de gel de agarose19. A porcentagem de agarose no gel varia de acordo com os tamanhos de fragmento esperados. 0,8 – 1% separa otimamente fragmentos > 1 kb em tamanho. Use 1-1,5% de agarose para fragmentos < 1 kb. Não adicione o brometo de Ethidium ao gel. (5 x TBE, 54 l: 1G de Trizma base, 27,5 g de ácido bórico, 3,75 g de EDTA).
- Prepare-se para a transferência do DNA do gel do agarose para a membrana de nylon.
- Transferir o gel para uma bandeja separada e manchá-la por 20 – 25 min em 0,5 x TBE contendo brometo de etídio (5 µ g/mL), rodando a 40 rpm em um agitador orbital.
- Visualize o gel em um transiluminador UV. Verifique se a separação de tamanho do DNA genômico, incluindo visibilidade das bandas satélite discreta (Figura 5A). Um esfregaço para tamanhos de peso molecular inferiores indica degradação do DNA.
- O transiluminador de UV, colocar uma transparência sobre o gel e marque a posição dos slots e as bandas dos marcadores com uma caneta de marcador (Figura 5B). Isto irá facilitar a determinação do tamanho dos fragmentos hibridizados mais tarde caso as sequências de marcador não cruzar especificamente com a sonda de DNA. Indicando os fragmentos de marcador para esta etapa, é possível controlar o tamanho dos fragmentos hybridizing.
- Coloque o gel de volta na bandeja, enxagúe com água desionizada ultrapura e submirja em 0,25 M HCl por 15 min para fragmentar o DNA dentro do gel. Lavar com água desionizada ultrapura. Usar HCl e água suficiente para cobrir o gel na bandeja e gire a moldeira com o gel submerso a 40 rpm em um agitador orbital.
- Incube o gel no buffer de desnaturação por 30 min. lavagem com água deionizada ultrapura. Usar o tampão e água suficiente para cobrir o gel na bandeja e gire a moldeira com o gel submerso a 40 rpm em um agitador orbital. (Buffer de desnaturação: 0,5 M de NaOH, 1,5 M NaCl).
- Incube o gel no buffer de neutralização por 30 min. lavagem com água deionizada ultrapura. Use o tampão e água suficiente para cobrir o gel na bandeja, gire a bandeja com gel submerso a 40 rpm em um agitador orbital. (Buffer de neutralização: 0,5 M Tris, 1,5 M NaCl, HCl, de 220 mM pH 7,6).
- Transferência do DNA para uma membrana de nylon.
- Prepare a instalação para transferência capilar do DNA genômico. Coloque uma placa de plástico (aproximadamente o tamanho do gel ou maior) sobre uma bandeja cheia de 20 x solução salina-citrato de sódio (SSC). Dobre um pedaço de papel de filtro grosso sobre a bandeja, para que ambas suas extremidades estão pendurados no SSC. Corte um pedaço de gel de tamanho de membrana de nylon positivamente carregado Hybond N + e 2 pedaços de papel de filtro grosso. (20x SSC: 3 M NaCl, citrato de sódio 0,3 M).
- Prepare a instalação mancha (Figura 6), colocando o gel, vagas para baixo na parte superior do papel de filtro sobre a placa de plástico. Coloque uma membrana Hybond N+ na parte superior, seguido por 2 camadas de papel de filtro. Pre-molhado cada camada em 20 x SSC antes de adicioná-los à assembleia. Certifique-se de remover quaisquer bolhas de ar entre as camadas, pois estas impedem a transferência de DNA.
- Cubra a montagem com uma camada espessa de papel de seda. Coloque uma placa de plástico com peso, tais como uma pequena garrafa, na parte superior. Certifique-se que a pressão está igualmente dividida sobre o gel. Isto irá assegurar a transferência adequada de DNA.
Nota: O peso deve ser cerca de 200-300 g; pesos pesados dificultam a transferência de DNA. - Cobrir a área que circunda o assembly, incluindo papel de filtro exposta, com película aderente (Figura 6) para evitar a evaporação do buffer de x SSC 20 e alvo que capilar forças em direção a membrana de nylon. Borre durante a noite.
- Marque a posição dos slots, nome, e/ou data na parte superior da membrana com lápis e remover a membrana do assembly. Observe que o lado inferior que tem estado em contacto com o gel, carrega o DNA.
- Corrigi imediatamente o DNA para a membrana por irradiação UV (2.400 µ j/m2) usando um agente reticulante UV.
Nota: Condições de reticulação dependem do tipo de membrana utilizada.
Nota: neste ponto a membrana pode ser armazenada a-20 ° C e usada para a hibridação com uma sonda mais tarde. Lavar a membrana de quitosana em 2x SSC e selo-pre-dobrado em tubos de polietileno Heat antes de colocá-lo a-20 ° C.
- Prepare a sonda para a hibridação.
- Amplifica a sequência a ser usada como a sonda para a mancha de DNA por PCR20. 50-100 ng de um fragmento PCR 250 kbp bp-2 é usado como uma sonda. Duas sondas separadas, um cruzamento para a região proximal da borda direita e outro para a região proximal da borda esquerda do T-DNA, podem ser usadas para avaliar a presença do T-DNA inteiro (Figura 4A e Figura 7).
- Dilua 50-100 ng do produto PCR em 24 µ l de água deionizada ultrapura num tubo de ensaio.
- Desnaturar o produto do PCR diluído fervendo por 5 min num copo de água ou calor do bloco e, em seguida, fixe diretamente no gelo.
- Degelo do premade GCT-mistura no gelo. (GCT-mistura: dGTP, dCTP, dTTP (todos 0,5 mM), aleatório hexâmeros 43,2 ng / µ l, Acetylated BSA 1,33 mg/mL, 33mm de β-Mercaptoetanol, 0,67 M Hepes, 0,17 mM pH do Tris 6,8, 17 mM MgCl).
- Adicione 21 µ l do GCT-mix e 2 U de Klenow fragmento para o produto do PCR.
- Adicionar 2 µ l de ATP [32P] à mistura e incubar a 37 ° C, durante 1 h.
Atenção: Todas as etapas que envolvem a ATP [32P] precisam ser realizados em um ambiente designado pelo trabalho radioativo enquanto estiver usando a proteção apropriada.
Nota: Certifique-se que o ATP [32P] é fresco (não mais de 1 meia-hora já passou). - Prepare um G-50 de Sephadex coluna (grosso ou médio)21 para purificar as sondas marcadas de nucleotídeos (radioativos) sem personalidade jurídica. Pegue uma seringa de 2 mL e cobrir a saída com um pequeno círculo de papel de filtro grosso. Adicionar 2 mL de Sephadex G-50 dissolvido em TE para a seringa e retire todo o líquido da coluna pela fiação.
- Coloque a coluna em um tubo de plástico de 15 mL, carregar as sondas marcadas na coluna e girar à temperatura ambiente (definir o centrifugador a x 750 g, permitem que o rpm aumentar até 750 g x é atingido, então pare o centrifugador e permitir a rotação que recusar a 0 x g) para eluir a sonda. Adicionar 200 µ l de TE para a coluna e rotação para eluir a sonda restante; repeti uma vez. Nestas condições, rotulada de fragmentos de DNA são excluídos da matriz Sephadex em eluir, enquanto livre nucleotídeos permanecem na coluna.
- Use 300 µ l das sondas marcadas por tubo de hibridização. Manter a restante sondas marcadas a-20 ° C para uso posterior. No entanto, ter a metade do tempo de ATP [32P] em mente.
- Cruzar o borrão de DNA.
- Calor 2 x SSC e tampão de hibridização (15 mL por tubo de hibridização, máximo de 2 blots por tubo) para 65 ° C. (Tampão de hibridização: 10% de sulfato de dextrano, SDS 1%, 1 M NaCl, pH de Tris 50mm 7.5, dissolver a 65 ° C, manter alíquotas a-20 ° C).
- Pré-aqueça o forno de hibridização a 65 ° C.
- Coloque o engranzamento de nylon em uma bandeja com um pouco de aquecida (65 ° C) 2 x SSC para cobrir a bandeja. Coloque o borrão de DNA em cima da malha, com o lado de DNA. Rolar o blot juntamente com a malha e inserir o rolo em um tubo de hibridização. Deite fora o excesso 2 x SSC.
- Em um tubo de microcentrifugadora, ferver 150 µ l de DNA de esperma de salmão (concentração de 10 mg/mL) por tubo de hibridização por 5 min (Veja também passo 4.6.3). Arrefecer imediatamente no gelo e adicionar para o tampão de hibridização pré-aquecido.
- Adicione a solução de esperma de salmão-tampão hibridização para o tubo com o Borrão. Pre-cruzar a 65 ° C, durante pelo menos 1 h em uma roda giratória, em 12 rpm.
- Quando a pré-incubação na etapa 4.7.5. está quase terminado, ferva 300 µ l das sondas marcadas por 5 min (Veja também passo 4.6.3) e adicione imediatamente para o Borrão após a incubação.
- Cruzar a noite na roda rotativa a 63 ° C, 12 rpm. Não pipete a sonda diretamente sobre a mancha, mas para a solução de esperma de salmão-tampão hibridização.
- Lave o Borrão.
- Pré-aqueça as soluções de lavagem (1 x SEPI, 0,1% SDS e 0.1 x SSPE, 0,1% SDS) para 65 ° C. (20 x SSPE: 3 M NaCl, 230mm NaH2PO4, 20mm EDTA, pH 7,0).
- Descartar a solução de hibridização e adicionar cerca de 100-150 mL de 1 x SSPE, solução de SDS 0,1% para o tubo de hibridização, fechar o tubo e gire com a mão. Despeje a hibridação e lavagem com solução em resíduos radioactivos líquidos apropriados.
- Adicionar cerca de 100-150 mL de 1 x SSPE, solução de SDS 0,1% para o tubo, fechar e incubar o tubo por 15 min a 63 ° C, na roda giratória, 12 rpm. Descarte a solução de lavagem adequada.
- Adicionar cerca de 100-150 mL de 0.1 x SSPE, solução de SDS 0,1% para o tubo de hibridização, fechar e gire o tubo por 5 min a 63 ° C, 12 rpm. Descarte a solução de lavagem adequada.
- Pegue o borrão do tubo e colocá-lo em uma bandeja contendo suficientes pré-aquecido 0.1 x SEPI, 0,1% SDS e agitar durante 3 min em um tremendo banho de água a 65 ° C. Enquanto isso, lave a malha em uma bandeja cheia de água.
- Tirar a mancha, coloque-a entre as de plástico pre-dobrado (tubulação de polietileno), cuidadosamente Limpe o excesso de líquido e deixe a mancha secar brevemente. Note-se que o líquido vai estragar a tela phosphorimager.
- Sele o borrão em plástico nos três lados. Retire todo excesso de líquido em torno da mancha e feche o tubo plástico selando o quarto lado. Corte o excesso de plástico. Certifique-se de blot selado não está vazando, e que o plástico é seco do lado de fora.
- Expor a tela phosphorimager.
- Coloque o blot selado em uma gaveta de phosphorimager, com a tela de phosphorimager, voltada para o lado de DNA do blot. Feche a gaveta e deixar por ± 2-4 dias, dependendo a força do etiquetar radioativo e sensibilidade do phosphorimager.
- Varredura da tela de phosphorimager usando um phosphorimager. Cuide-se para expor a tela o menos possível à luz antes da digitalização. Salve a imagem. Apaga a tela do sinal por expô-lo à luz brilhante.
- Analise o Borrão.
- A análise depende da estratégia de restrição utilizada na etapa 4.1. Ao analisar o blot preparado de acordo com a estratégia mostrada na etapa 4.1.1.1 (Figura 4A), conte o número dos fragmentos detectados. Nesta estratégia, o número de fragmentos hibridizados refere-se ao número de integrações de T-DNA.
- Compare o número de fragmentos detectados com uma sonda para a esquerda (Figura 7B) e parte do T-DNA (Figura 7).
Nota: Se um número diferente de fragmentos hybridizing é detectado, então qualquer i) várias cópias T-DNA (seja na orientação invertida ou direta) ou integrado ii) incompleta T-DNAs estão presentes. - Estimar o tamanho dos fragmentos hibridizados em blot baseado no tamanho das bandas marcador e compara os tamanhos dos fragmentos hibridizados com o fragmento esperado os tamanhos calculados com base na estratégia de restrição do inserções em tandem (Figura 4A) Identifica o arranjo possível em tandem dos T-DNAs. Use a Figura 4A como um guia para calcular o tamanho dos fragmentos esperados.
Nota: Se o tamanho dos fragmentos hibridizados não concorda com riscos calculados, então mais provável um dos presentes as integrações não está completo.
- Compare o número de fragmentos detectados com uma sonda para a esquerda (Figura 7B) e parte do T-DNA (Figura 7).
- Ao analisar o blot preparado de acordo com a estratégia mostrada na etapa 4.1.1.2 (Figura 4B), estimar o tamanho do fragmento hybridizing sobre o blot baseado no tamanho das bandas do marcador e compará-lo com o tamanho esperado. Uma inserção intacta produz um único fragmento com um comprimento definido. Qualquer desvio do comprimento esperado indica integração incompleta (Figura 7).
- A análise depende da estratégia de restrição utilizada na etapa 4.1. Ao analisar o blot preparado de acordo com a estratégia mostrada na etapa 4.1.1.1 (Figura 4A), conte o número dos fragmentos detectados. Nesta estratégia, o número de fragmentos hibridizados refere-se ao número de integrações de T-DNA.
- Tira o Borrão para re-hibridização (opcional).
Nota: O mesmo borrão pode ser hibridizado consecutivamente com diferentes sondas. Antes de continuar com uma nova sonda, tira uma sonda anterior hibridizada de blot.- Para remover a sonda do blot, coloque o borrão em uma bandeja com seu DNA-lado virado para baixo. Despeje um excedente de 0,5% SDS a bandeja. Ferva a membrana por 2 – 5 min. A duração do tratamento depende do tamanho e GC-conteúdo da sonda usada. Sondas de GC-mais rico e mais precisam de um tratamento mais longo.
- Após o descascamento, cruzar o blot com outra sonda, ou selo e armazenar a-20 ° C.
Resultados
Usando o sistema de BIBAC-GW, construções de repórter para estudar ODM em plantas foram gerados10. Construções foram projetadas no Gateway de entrada vetor pENTR-gm12 e inseridas no pBIBAC-BAR-GW (Figura 1), usando a reação de recombinação Gateway LR.
Arabidopsis foram transformados com pDM19, um BIBAC-BAR-GW plasmídeo com um repórter de mTurquoise-eYFP carregando um códon de parada translacional no quadro de leitura de eYFP na posição 120 (mTurquoise2-eYFP * 40) (Figura 2)10. No total, 126 Arabidopsis plantas foram transformadas (9 plantas por vaso, 14 potes). As sementes dessas plantas foram em pool, semeadas em bandejas com solo e permitidas crescer durante duas semanas antes do tratamento com solução de glufosinato-amônio. Só mudas que expressa o gene bar (presente no BIBAC-BAR-GW) sobrevivem tratamento de glufosinato-amônio (Figura 3). No total, foram identificados 11 transgenics transformadas com pDM19, correspondente a uma eficiência de transformação de 0,02% das sementes analisadas.
Para os 11 transgênicos isolados, mancha de DNA foi usado para determinar o número de integrações de T-DNA. Para esse efeito, DNA genômico foi cortada com BglII ou Scaeu (estratégia como idealizadas na Figura 4A). Ambos destas enzimas de restrição cortar apenas uma vez na sequência T-DNA (Figura 7A). Hibridização com sondas reconhecendo o bar e eYFP regiões codificantes permitiu a deteção do número dos respectivos fragmentos de DNA.
O número de DNA individual fragmentos sobre os borrões permitidos para estimar o número de inserções de T-DNA nas linhas de repórter (tabela 1). Fragmentos de hybridizing único com sonda a barra e o eYFP indicaram a presença de uma única integração do T-DNA. Desde o 11 transgenics analisado, seis realizado única integrações. O número médio de integrações foi 1.2.
Para 6 linhas carregando uma única integração do T-DNA, a integridade da construção de repórter inserido foi testada usando a DNA mancha (estratégia como idealizadas na Figura 4B). DNA genômico foi cortada com BglII e Pcieu liberar um fragmento de 5,5 kb, contendo o gene de fusão a barra e mTurquoise-eYFP (Figura 7A). Uma sonda contra eYFP foi usada para detectar o fragmento esperado. Todas as plantas testadas carregava um fragmento intacto. Observe que o fragmento examinado exclui a esquerda e a borda direita T-DNA e, portanto, não se examina a integridade do T-DNA inteiro, mas apenas a parte que contém os transgenes de interesse.
A expressão do gene repórter fluorescente foi determinada em transgênicas-linhas de cópia única independentes, diferindo apenas pela localização genômica do T-DNA. Níveis de transcrição relativa do repórter CaMV 35S promotor-driven mTurquoise-eYFP foram medidos por RT-qPCR em quatro linhas de repórter DM19 carregando integrações intactas, cópia única de que a posição de genômica foi determinado10. A variação nos níveis de expressão de gene repórter nas entrelinhas foi menor: a diferença máxima de mTurquoise-eYFP níveis de RNA foi 2 vezes (Figura 8A).
Em seguida, o ODM foi realizado nestas linhas de repórter. Três das quatro linhas independentes repórter mostraram bastante semelhantes eficiências ODM (Figura 8B). No entanto, uma linha, DM19 [4] 1, rendeu uma eficiência muito baixa de ODM em comparação com as outras linhas. Estes resultados indicam que o ODM é afetado pelo contexto genômico local. De que maneira o contexto local genômico da integração T-DNA em DM19 [4] 1 difere do que nas outras linhas continua a ser identificado. Análise de conjuntos de dados disponíveis em marcas de cromatina ativos e inativos dos locais de integração de T-DNA genômico em plantas não-transgênicas não forneceu uma resposta10.

Figura 1: mapas funcionais dos vetores de pBIBAC-GW. pBIBAC-GW derivados estão disponíveis com qualquer resistência ao glufosinato (bar) ou fluorescência DsRed em revestimentos de semente (DsRed) como um marcador de seleção em plantas. Para os dois vetores, um gene de resistência canamicina é o marcador de seleção em bactérias. Gateway gaveta ccdB é mostrada entre pontas de seta verdes representando a recombinação sites attR1 e attR2. Clique aqui para ver uma versão maior desta figura.

Figura 2: construção de repórter do Mutagenesis. Os genes de repórter de mTurquoise-eYFP são movidos pelo CaMV 35S promotor. A mTurquoise região de codificação é fundido a uma eYFP codificação região carregando uma mutação C-A no nucleotídeo posição 120, resultando em um códon de parada translacional prematuro TAA e encerramento prematuro da tradução da proteína de fusão. 3 o ' sinal de poliadenilação Nopaline Synthase (nos 3') é usado para finalizar a transcrição do constructo22. Sinal de localização nuclear (NLS) é usado para direcionar as proteínas traduzidas para o núcleo. Clique aqui para ver uma versão maior desta figura.

Figura 3: bandeja cheia de plântulas de Arabidopsis antes e após o tratamento de glufosinato-amônio. Mudas não expressando o gene bar que está presente na T pBIBAC-BAR-GW-morrer de DNA após ser pulverizado com solução de glufosinato-amônio. As fotos mostram a mesma bandeja de mudas (A) antes de pulverizar com glufosinato-amônio, 14 dias após a semeadura e (B) 10 dias mais tarde, depois de ser pulverizado duas vezes. Clique aqui para ver uma versão maior desta figura.
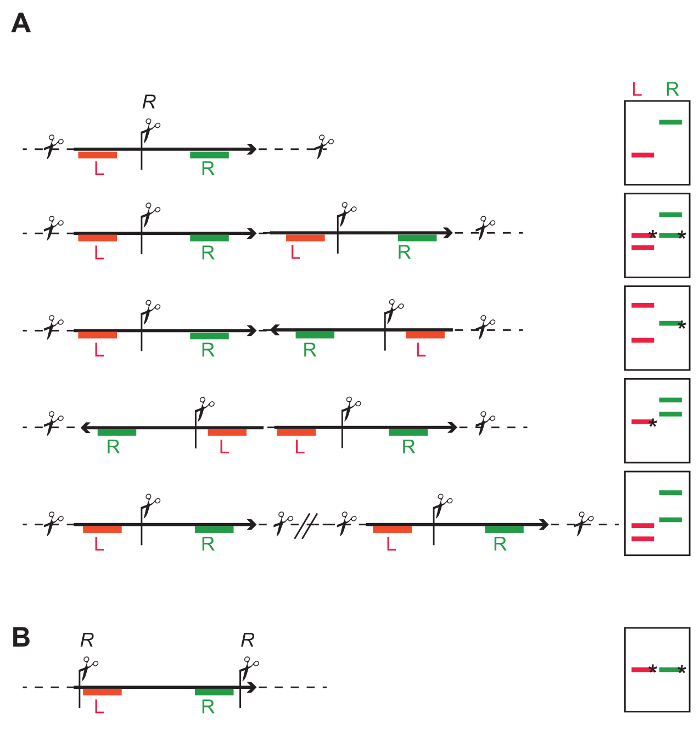
Figura 4: estratégia de restrição geral de DNA para identificar o número e a integridade de inserido DNAs-T. (A), um sítio de restrição (R) no meio do T-DNA permite sondagem independente de esquerda (L vermelho) e a parte direita do T-DNA (R verde). Os desenhos da direita mostram que dependendo de integrações de single - ou multi - cópias T-DNA, diferentes padrões de bandas de tempestade são obtidos com a mancha de DNA. Bandas marcados com um * têm um comprimento definido, enquanto o comprimento das outras bandas varia de acordo com o site mais próximo de restrição no DNA genômico flanqueando. Inserção única: A L e R sonda ambos dar um fragmento independente. O tamanho do fragmento média esperada pode ser calculado com base na frequência do site restrição no genoma. O tamanho mínimo é a distância entre o local de restrição para a borda esquerda (LB) ou borda direita (RB), dependendo de qual lado da integração está sendo sondado, e se o T-DNA está intacto. Repetição em tandem: As sondas para L e R dar ambos os dois fragmentos; para cada um dos fragmentos inclui flanqueando o DNA genômico de teste, o segundo fragmento tem um tamanho esperado e é identificado por ambas as sondas. Repetição invertida: Dependendo a direção da fita integrada, um "L" e dois fragmentos de R, ou dois L e um R podem ser identificado. Inserções única individuais: O resultado é um número de fragmentos independentes, e o número de fragmentos corresponde ao número de integrações. (B) restrição sites as extremidades do T-DNA permitem determinar a integridade do fragmento entre os sítios de restrição. Clique aqui para ver uma versão maior desta figura.
Figura 5: um gel de agarose com padrão de restrição e a transparência correspondente. (A) sobre o gel de agarose, o DNA genômico digerido com EcoRé mostrado. A boa digestão do DNA é ilustrada pela presença das bandas satélite discretos. (B) marcação a posição dos slots e bandas de marcador em uma transparência torna possível para depois facilmente calcular o tamanho de fragmentos de cruzamento. Aqui, marcadores de MRC-Holland (azul e vermelho) são usados, indicado por M. , por favor clique aqui para ver uma versão maior desta figura.
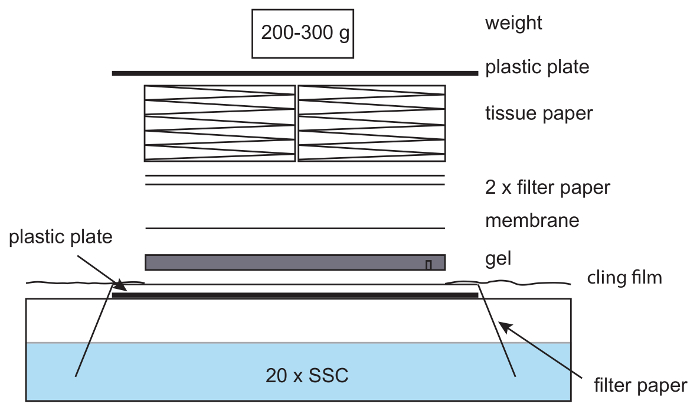
Figura 6: configuração para a mancha capilar. Em um capilar mancha instalação, papel de filtro é colocado sobre uma placa de plástico com as extremidades do papel pendurado em 20 de x SSC buffer. O papel é molhado com 20 x SSC e um gel de agarose, colocado no topo, seguiram por uma membrana de nylon, filtro de papel e uma pilha de tecidos. Um peso leve é colocado na parte superior. Cuidado é tomado para remover bolhas de ar entre o gel, o papel e a membrana. Película aderente é usada para evitar a secagem fora da instalação. Clique aqui para ver uma versão maior desta figura.
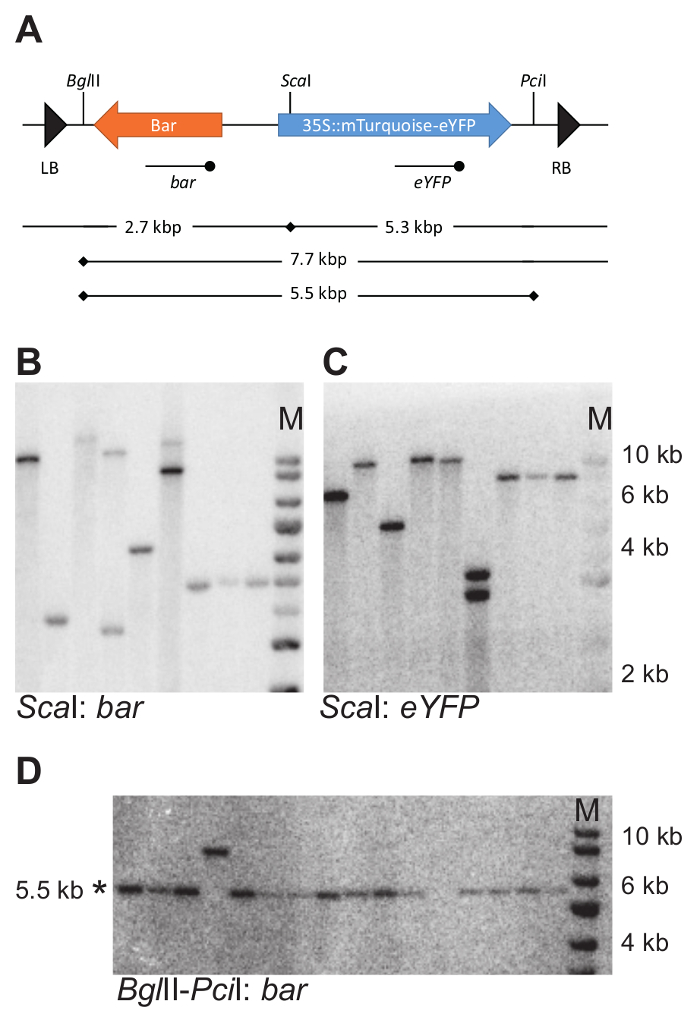
Figura 7: exemplo de DNA mancha a estratégia e os resultados experimentais. (A) DNA mancha estratégia para determinar o número e a integridade das integrações de T-DNA. Locais de corte das enzimas restrição selecionadas dentro do T-DNA são indicadas com barras verticais. As sondas eYFP e bar usadas para hibridização com DNA genômico digerido são indicadas usando uma linha com o ponto terminal abaixo do T-DNA. (B–D) DNA exemplo borrões. DNA genômico foi cortado com Sca, eu e o Borrão foi sondada com sonda de um bar e o eYFP (B e C). DNA genômico foi cortado com BglII e Pcieu e analisado com uma sonda de bar . Fragmentos intactos são 5,5 kbp em tamanho (D). Nota que o conjunto de amostras em D difere as mostradas em B e C. * indica o tamanho do fragmento esperado; M, marcador. Em B, C e D, é usado o mesmo marcador de tamanho. Clique aqui para ver uma versão maior desta figura.
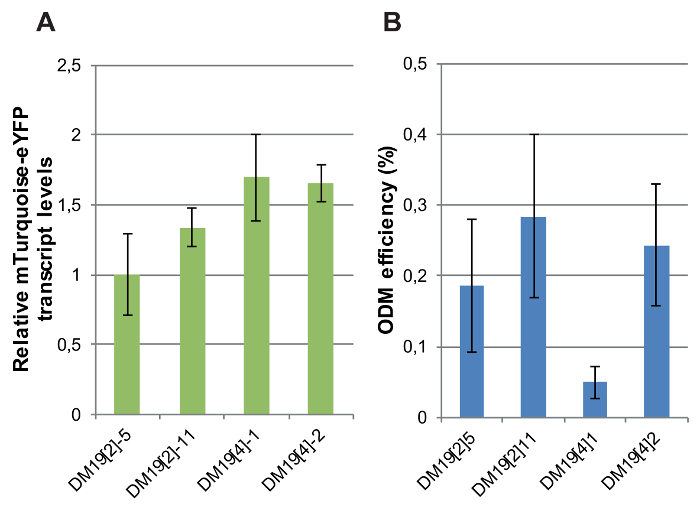
Figura 8: níveis de expressão de mTurquoise-eYFP e eficiências ODM em linhas de repórter independente mTurquoise-eYFP. Níveis de transcrição relativa mTurquoise-eYFP (A) medidos por RT-qPCR em linhas de repórter DM19. Para normalização, utilizaram-se os níveis de transcrição de actina. (B) ODM eficiência medido nas linhas DM19 repórter. Para A e B, barras indicam a média de pelo menos cinco réplicas biológicas. Barras de erro indicam SEM. clique aqui para ver uma versão maior desta figura.
| Tipo de locus de T-DNA | NR de integrações de T-DNA | Linha de repórter | Número de fragmentos detectados | Integridade | |||
| SCA Eu | BGL II | BGL II/PciI | |||||
| bar | eYFP | bar | eYFP | eYFP | |||
| Integrações de único locus | 1 | 19 [2] -2 | 1 | 1 | 1 | 1 | + |
| 1 | 19 [2] -5 | 1 | 1 | 1 | 1 | + | |
| 1 | 19 [2] -9 | 1 | 1 | 1 | 1 | + | |
| 1 | 19 [2] -11 | 1 | 1 | 1 | 1 | + | |
| 1 | 19 [4] -1 | 1 | 1 | 1 | 1 | + | |
| 1 | 19 [4] -2 | 1 | 1 | 1 | 1 | + | |
| Repita 2, invertido | 19 [2] -10 | 2 | 1 | 1 | 1 | + | |
| 2 integração incompleta | 19 [2] -3 | 1 | 2 | 1 | 1 | ND | |
| Várias integrações de locus | 2 | 19 [2] -6 | 2 | 2 | 2 | ND | |
| 2 | 19 [2] -7 | 2 | 2 | 2 | 2 | ND | |
| 3/4 | 19 [2] -1 | 4 | 3 | 3 | 3 | ND | |
| ND-não determinado. | |||||||
Tabela 1: Resumo do DNA mancha dados para transgenics isolado após transformação com pDM19.
Discussão
Crítico para a geração de transgênicos com integrações único, intactos de um transgene é a escolha do vetor binário usado. Vetores de família BIBAC têm sido utilizados para entregar muitos planta espécie23,24,25,26,,27,28-sequências de interesses. Vetores BIBAC, incluindo BIBAC-GW, rendem cópia única integrações com eficiência elevada: o número médio de inserções por linha é de 1,5 a 2, em comparação a 3 ou superior para binário mais comumente usado vetores5,9, 29. como uma grande melhoria em comparação com outros vetores BIBAC, com os vetores de BIBAC-GW, as sequências de interesse podem ser facilmente inseridas usando Gateway recombinação sites12. Os vetores modificados superar os problemas gerais de vetores BIBAC quando usado em estratégias de clonagem convencionais: i) um número muito limitado de sítios de restrição exclusiva e ii) um DNA de baixo rendimento. Os sites de recombinação de Gateway fazem BIBAC-GW vetores uma alternativa atraente para outros vetores binários para gerar plantas transgênicas.
Aqui uma série de protocolos, de geração de BIBAC-GW derivados contendo sequências de interesse, para plantar a transformação e DNA análise borrão para número e integridade das sequências transgénicas é descrita. Vários dos protocolos relataram neste trabalho, Gateway de clonagem, eletroporação de bactérias e a transformação da planta, é prática comum em muitos laboratórios e também pode ser realizada com pequenas modificações. É importante saber que o BIBAC-GW é um vetor de cópia única, em e. coli e na . tumefaciens. Portanto, quando isolar o DNA, o rendimento é baixo; recomenda-se intensificar o processo de isolamento.
Em transgenics carregando várias integrações de T-DNA, transgenes introduzidos são frequentemente sujeitas a1,2,4,30de silenciamento do gene e deve, portanto, a maioria dos aplicativos ser evitados. Para identificar plantas transgénicas com integrações única, intactas, recomenda-se usar a análise de DNA do borrão. Enquanto métodos diferentes de DNA mancha pode ser usada para determinar o número de cópia de T-DNA e integridade dos T-DNAs em linhas transgénicas (análise de segregação, TAIL-PCR, PCR (qPCR) quantitativos e digital gota PCR), apesar do trabalho intensivo, DNA mancha é muitas vezes o método de escolha. Análise de segregação não é capaz de diferenciar entre múltiplo e único integrações de T-DNA em loci único. TAIL-PCR frequentemente menores estimativas a cópia número, especialmente se mais de uma integração de T-DNA é presente31e qPCR precisa de otimização elaborada para resultados fiáveis31,32. Digital gota PCR é um método bastante preciso para a deteção de número de cópia se o equipamento necessário está disponível31. O benefício adicionado de DNA mancha é fácil detecção de T-DNAs truncados, que é facilmente perdida em todas as técnicas de PCR-baseados.
Com análise de DNA do borrão, hybridizing fragmentos sobre o blot precisam ser bem identificáveis no sinal e tamanho. Vários fatores são conhecidos por afetar o resultado do DNA mancha. Além de uma seleção adequada de enzimas de restrição (estratégia indicada na Figura 4) e marcadores de tamanho, suficiente DNA de boa qualidade é necessário. Menos de 2 µ g do genoma de Arabidopsis DNA não renderá fragmentos bem identificáveis. Quando se lida com genomas maiores, mais DNA é necessária. Para obter quantidades suficientes de Arabidopsis DNA, podem ser usadas tecidos florais ou mudas 1 - semana de idade. Um lote de mudas cultivadas em uma placa de Petri rende 2 – 8 µ g de DNA. A fim de evitar a degradação do DNA durante o isolamento, deve ter-se cuidado para processar o material de planta rápido. Além disso, DNA genômico deve ser resuspended em Tris-EDTA para reduzir a sua degradação por nucleases e armazenado a 4 ° C em vez de-20 ° C para evitar DNA roubando devido a repetidos ciclos de congelação-degelo. Se não tiver certeza que todas as amostras de DNA são digeridas completamente, é aconselhável rehybridize o borrão de DNA com sonda de reconhecer uma região genômica endógena, original. Ao selecionar sequências de sonda para a identificação de sequências transgénicas ou endógenas, é crucial selecionar apenas sequências únicas. Para poder determinar com precisão o tamanho dos fragmentos hibridizados, as posições de DNA gel vagas e faixas do marcador de ADN devem ser marcadas em uma transparência (Figura 5B) ao visualizar um gel manchado de brometo de etídio (Figura 5A) sobre um UV transiluminador. No caso das sequências marcador não cruzar com a sonda de DNA ou hibridização é parcial, é a única maneira de rastrear o tamanho dos fragmentos de cruzamento.
Uma vez que é tomado para conseguir sinal de hibridação bom e estimativa do tamanho do fragmento, a interpretação dos resultados mancha é simples. Quando usando apenas uma enzima de restrição e hybridizing com diferentes sondas de detecção também a parte esquerda ou direita do T-DNA, o número de fragmentos detectados reflete o número de inserções de T-DNA. Por exemplo, a figura 7B, C mostra o mesmo borrão de DNA, hibridizadas com diferentes sondas, bar (Figura 7B) e reforçada proteína fluorescente amarela (eYFP) (Figura 7), usando a estratégia mostrada na Figura 7A. Todas as faixas, exceto 4, mostram um número igual de fragmentos no ambos os borrões: dois fragmentos para linha 6 e um fragmento único para todas as outras linhas. Este número de fragmentos detectados é o número de inserções de T-DNA.
Quando o número de fragmentos detectados com sondas que vincule a esquerda ou direita parte do T-DNA difere (quanto a Figura 7BC, linha 4), ou incompletas inserções estão presentes, ou os T-DNAs inseriu-se no arranjo em tandem. As inserções em tandem exibir comprimento do fragmento não-aleatório para um dos fragmentos do T-DNA (Figura 4A, painel direito) e podem ser identificadas, comparando o tamanho do fragmento hibridizadas com o esperado com base na estratégia de restrição. Uma estratégia de mancha adicional pode ser necessários para confirmar a disposição em tandem de inserções do T-DNA. No exemplo mostrado na linha 4 (Figura 7B, C), duas inserções são arranjadas em uma orientação de repetição invertida.
Ao estimar a integridade de um T-DNA, ou parte dele, o comprimento do fragmento hybridizing pode ser calculado com base na estratégia de restrição. Qualquer desvio o tamanho esperado indica a presença de uma inserção incompleta. Por exemplo, na Figura 7, na faixa 4, um fragmento hybridizing migra no kbp 8, (em vez do esperado 5.5 kbp) indicando um fragmento maior tamanho devido à falta de um dos sites de restrição.
BIBAC-GW vetores são excelentes ferramentas para a geração de cópia única intactas integrações em um número de espécies de plantas. O protocolo relatado aqui fornece um procedimento confiável para identificar plantas com integrações única, intactas de um transgene de interesse.
Divulgações
Os autores declaram não concorrentes interesses financeiros ou outros conflitos de interesse.
Agradecimentos
Esta pesquisa é suportada pelo holandês tecnologia Foundation STW (12385), que faz parte da organização de países baixos para a pesquisa científica (NWO), e que é parcialmente financiado pelo Ministério dos assuntos económicos (OTP Grant 12385-MS). Agradecemos a Carol M. Hamilton (Universidade de Cornell, Estados Unidos) para a prestação de pCH20, a espinha dorsal dos vetores BIBAC-GW.
Materiais
| Name | Company | Catalog Number | Comments |
| Kanamycin sulphate monohydrate | Duchefa | K0126 | |
| Gentamycin sulphate | Duchefa | G0124 | |
| Rifampicin | Duchefa | R0146 | |
| Tetracycline hydrochloride | Sigma | T-3383 | |
| DB3.1 competent cells | Thermo Scientific - Invitrogen | 11782-018 | One Shot ccdB Survival 2 T1R Competent Cells (A10460) by Invitrogen or any other ccdB resistant E. coli strain can be used instead |
| DH10B competent cells | Thermo Scientific - Invitrogen | 18290-015 | |
| Gateway LR clonase enzyme mix | Thermo Scientific - Invitrogen | 11791-019 | |
| tri-Sodium citrate dihydrate | Merck | 106432 | |
| Trizma base | Sigma-Aldrich | T1503 | |
| EDTA disodium dihydrate | Duchefa | E0511 | |
| Proteinase K | Thermo Scientific | EO0491 | |
| Bacto tryptone | BD | 211705 | |
| Yeast extract | BD | 212750 | |
| Sodium chloride | Honeywell Fluka | 13423 | |
| Potassium chloride | Merck | 104936 | |
| D(+)-Glucose monohydrate | Merck | 108346 | |
| Electroporation Cuvettes, 0.1 cm gap | Biorad | 1652089 | |
| Electroporator Gene Pulser | BioRad | ||
| Magnesium sulfate heptahydrate | Calbiochem | 442613 | |
| D(+)-Maltose monohydrate 90% | Acros Organics | 32991 | |
| Sucrose | Sigma-Aldrich | 84100 | |
| Silwet L-77 | Fisher Scientific | NC0138454 | |
| Murashige Skoog medium | Duchefa | M0221 | |
| Agar | BD | 214010 | |
| Glufosinate-ammonium (Basta) | Bayer | 79391781 | |
| Restriction enzymes | NEB | ||
| Ethidium Bromide | Bio-Rad | 1610433 | |
| Electrophoresis system | Bio-Rad | ||
| Sodium hydroxide | Merck | 106498 | |
| Hydrochloric acid | Merck | 100316 | |
| Blotting nylon membrane Hybond N+ | Sigma Aldrich | 15358 | or GE Healthcare Life Sciences (RPN203B) |
| Whatman 3MM Chr blotting paper | GE Healthcare Life Sciences | 3030-931 | |
| dNTP | Thermo Fisher | R0181 | |
| Acetylated BSA | Sigma-Aldrich | B2518 | |
| HEPES | Sigma-Aldrich | H4034 | |
| 2-Mercaptoethanol | Merck | 805740 | |
| Sephadex G-50 Coarse | GE Healthcare Life Sciences | 17004401 | or Sephadex G-50 Medium (17004301) |
| Dextran sulfate sodium salt | Sigma-Aldrich | D8906 | |
| Sodium Dodecyl Sulfate | US Biological | S5010 | |
| Salmon Sperm DNA | Sigma-Aldrich | D7656 | |
| Sodium dihydrogen phosphate monohydrate | Merck | 106346 | |
| Storage Phosphor screen and casette | GE Healthcare Life Sciences | 28-9564-74 | |
| Phosphor imager | GE Healthcare Life Sciences | Typhoon FLA 7000 | |
| UV Crosslinker | Stratagene | Stratalinker 1800 | |
| cling film (Saran wrap) | Omnilabo | 1090681 | |
| Agarose | Thermo Scientific - Invitrogen | 16500 | |
| Boric acid | Merck | 100165 | |
| DNA marker ‘Blauw’; DNA ladder. | MRC Holland | MCT8070 | |
| DNA marker ‘Rood’; DNA ladder | MRC Holland | MCT8080 | |
| Hexanucleotide Mix | Roche | 11277081001 | |
| Large-Construct Kit | Qiagen | 12462 | |
| Heat-sealable polyethylene tubing, clear | various providers | the width of the tubing should be wider than that of blotting membrane | |
| Heat sealer | |||
| Membrane filter disk | Merck | VSWP02500 | |
| Magnesium chloride | Merck | 105833 | |
| Hybridization mesh | GE Healthcare Life Sciences | RPN2519 |
Referências
- Jorgensen, R. A., Cluster, P. D., English, J., Que, Q., Napoli, C. A. Chalcone synthase cosuppression phenotypes in petunia flowers: comparison of sense vs. antisense constructs and single-copy vs. complex T-DNA sequences. Plant Mol Biol. 31 (5), 957-973 (1996).
- Stam, M., et al. Post-transcriptional silencing of chalcone synthase in Petunia by inverted transgene repeats. Plant J. 12, 63-82 (1997).
- Stam, M., Viterbo, A., Mol, J. N., Kooter, J. M. Position-dependent methylation and transcriptional silencing of transgenes in inverted T-DNA repeats: implications for posttranscriptional silencing of homologous host genes in plants. Mol Cell Biol. 18 (11), 6165-6177 (1998).
- Jin, Y., Guo, H. S. Transgene-induced gene silencing in plants. Methods Mol Biol. 1287, 105-117 (2015).
- Oltmanns, H., et al. Generation of Backbone-Free, Low Transgene Copy Plants by Launching T-DNA from the Agrobacterium Chromosome 1[W][OA]. Plant Physiol. , (2010).
- Ye, X., et al. Enhanced production of single copy backbone-free transgenic plants in multiple crop species using binary vectors with a pRi replication origin in Agrobacterium tumefaciens. Transgenic Res. , (2011).
- Hamilton, C. M. A binary-BAC system for plant transformation with high-molecular-weight DNA. Gene. 200 (1-2), 107-116 (1997).
- Frary, A., Hamilton, C. M. Efficiency and stability of high molecular weight DNA transformation: an analysis in tomato. Transgenic Res. 10 (2), 121-132 (2001).
- Vega, J. M., et al. Agrobacterium-mediated transformation of maize (Zea mays) with Cre-lox site specific recombination cassettes in BIBAC vectors. Plant Mol Biol. 66 (6), 587-598 (2008).
- Anggoro, D. T., Tark-Dame, M., Walmsley, A., Oka, R., de Sain, M., Stam, M. BIBAC-GW-based vectors for generating reporter lines for site-specific genome editing in planta. Plasmid. 89, 27-36 (2017).
- Hamilton, C. M., Frary, A., Lewis, C., Tanksley, S. D. Stable transfer of intact high molecular weight DNA into plant chromosomes. Proc Natl Acad Sci U S A. 93 (18), 9975-9979 (1996).
- Belele, C. L., Sidorenko, L., Stam, M., Bader, R., Arteaga-Vazquez, M. A., Chandler, V. L. Specific tandem repeats are sufficient for paramutation-induced trans-generational silencing. PLoS Genet. 9 (10), e1003773 (2013).
- Shizuya, H., et al. Cloning and stable maintenance of 300-kilobase-pair fragments of human DNA in Escherichia coli using an F-factor-based vector. Proc Natl Acad Sci U S A. 89 (18), 8794-8797 (1992).
- Shi, X., Zeng, H., Xue, Y., Luo, M. A pair of new BAC and BIBAC vectors that facilitate BAC/BIBAC library construction and intact large genomic DNA insert exchange. Plant Methods. 7, 33 (2011).
- Woodman, M. E., et al. Direct PCR of Intact Bacteria (Colony PCR). Curr Protoc Microbiol. , A.3D.1-A.3D.7 (2016).
- Ausubel, F. M., et al. Mol Biol. Current Protocols in Molecular Biology. 1, (2003).
- Edwards, K., Johnstone, C., Thompson, C. A simple and rapid method for the preparation of plant genomic DNA for PCR analysis. Nucleic Acids Res. 19 (6), 1349 (1991).
- Clarke, J. D. Cetyltrimethyl ammonium bromide (CTAB) DNA miniprep for plant DNA isolation. Cold Spring Harb Protoc. (3), (2009).
- Lee, P. Y., Costumbrado, J., Hsu, C. Y., Kim, Y. H. Agarose Gel Electrophoresis for the Separation of DNA Fragments. J Vis Exp. (62), e3923 (2012).
- Green, M. R., Sambrook, J. . Molecular Cloning. , (2012).
- Sosa, J. M. Chromatography with Sephadex Gels. Anal Chem. 52 (6), 910-912 (1980).
- Depicker, A., Stachel, S., Dhaese, P., Zambryski, P., Goodman, H. M. Nopaline synthase: transcript mapping and DNA sequence. J Mol Appl Genet. 1 (6), 561-573 (1982).
- Feng, J., Vick, B. A., Lee, M. K., Zhang, H. B., Jan, C. C. Construction of BAC and BIBAC libraries from sunflower and identification of linkage group-specific clones by overgo hybridization. Theor Appl Genet. 113 (1), 23-32 (2006).
- Lee, M. K., et al. Construction of a plant-transformation-competent BIBAC library and genome sequence analysis of polyploid Upland cotton (Gossypium hirsutum L). BMC Genomics. 14, 208 (2013).
- Wang, W., et al. A large insert Thellungiella halophila BIBAC library for genomics and identification of stress tolerance genes. Plant Mol Biol. 72 (1-2), 91-99 (2010).
- Wu, C., et al. A BAC- and BIBAC-based physical map of the soybean genome. Genome Res. 14 (2), 319-326 (2004).
- Xu, Z., et al. Genome physical mapping from large-insert clones by fingerprint analysis with capillary electrophoresis: a robust physical map of Penicillium chrysogenum. Nucleic Acids Res. 33 (5), e50 (2005).
- Zhang, M., et al. Genome physical mapping of polyploids: a BIBAC physical map of cultivated tetraploid cotton, Gossypium hirsutum L. PLoS One. 7 (3), e33644 (2012).
- Frary, A., Hamilton, C. M. Efficiency and stability of high molecular weight DNA transformation: An analysis in tomato. Transgenic Res. , (2001).
- Stam, M., Viterbo, A., Mol, J. N. M., Kooter, J. M. Position-Dependent Methylation and Transcriptional Silencing of Transgenes in Inverted T-DNA Repeats: Implications for Posttranscriptional Silencing of Homologous Host Genes in Plants. Cell Biol. 18 (11), 6165-6177 (1998).
- Głowacka, K., Kromdijk, J., Leonelli, L., Niyogi, K. K., Clemente, T. E., Long, S. P. An evaluation of new and established methods to determine T-DNA copy number and homozygosity in transgenic plants. Plant Cell Environ. 39 (4), 908-917 (2016).
- Stefano, B., Patrizia, B., Matteo, C., Massimo, G. Inverse PCR and Quantitative PCR as Alternative Methods to Southern Blotting Analysis to Assess Transgene Copy Number and Characterize the Integration Site in Transgenic Woody Plants. Biochem Genet. 54 (3), 291-305 (2016).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados