Method Article
Análise de genes diferenciais e validação de dissulfideptose em modelo de lesão de isquemia-reperfusão miocárdica de rato
Neste Artigo
Resumo
O protocolo descreve a identificação de vários novos genes diferenciais relacionados à dissulfideptose associados à lesão de isquemia-reperfusão miocárdica por análise de bioinformática e validação experimental.
Resumo
A lesão de isquemia-reperfusão miocárdica (MIRI) é uma lesão adicional que ocorre durante o processo de restauração do fluxo sanguíneo do tecido cardíaco após lesão induzida por isquemia. O MIRI afeta seriamente a eficácia e o prognóstico de curto e longo prazo da reperfusão após infarto do miocárdio. No momento, o mecanismo do MIRI não está totalmente claro. A dissulfideptose é um novo modo de morte celular, e a relação entre o MIRI e a expressão de genes relacionados à dissulfideptose (DRGs) ainda não está clara. Em primeiro lugar, este estudo explora os genes diferencialmente expressos associados à dissulfideptose no MIRI por meio de análise de bioinformática. Em segundo lugar, ao construir um modelo de rato de MIRI, os DRGs foram detectados. Este estudo identificou 12 genes relacionados, incluindo Myh9, SLC7A11, SLC3A2, Myh7b, ACTB, FLNB, Actn1, Actn4, Flnc, Dbn1 e Pdlim1. O tecido miocárdico de ratos com IRMI mostra danos patológicos óbvios e eventos de apoptose. Os resultados da imuno-histoquímica indicaram que a estimulação do MIRI aumentou a expressão da proteína GLUT1 no tecido miocárdico, mas restringiu a expressão da proteína F-actina. Além disso, diferenças significativas na expressão de três proteínas foram validadas usando conjuntos de dados externos e modelos de ratos MIRI. Este estudo demonstrou que os DRGs tinham valor preditivo significativo no MIRI, fornecendo novas perspectivas para explorar biomarcadores e potenciais alvos terapêuticos do MIRI.
Introdução
O infarto agudo do miocárdio (IAM) é uma condição cardiovascular grave e continua sendo uma das principais causas globais de mortalidade. A intervenção coronária percutânea reduziu significativamente as taxas de mortalidade em pacientes com IAM1. No entanto, a terapia de reperfusão com o objetivo de restaurar o suprimento sanguíneo miocárdico é acompanhada por uma série de respostas patológicas e fisiológicas adversas. Esses processos podem resultar em aumento do tamanho do infarto, morte celular miocárdica, arritmias ventriculares sustentadas e mortesúbita 2. A lesão miocárdica de isquemia-reperfusão (IRMI) é uma condição cardiovascular complexa influenciada por fatores como citocinas, quimiocinas, fatores de crescimento, estresse oxidativo e sobrecarga de cálcio3. Mitigar o MIRI continua sendo um desafio significativo.
Recentemente, a dissulfideptose surgiu como uma nova forma de morte celular caracterizada pelo rápido colapso da rede de actina do citoesqueleto devido ao acúmulo excessivo de dissulfetos, incluindo cisteína, dentro das células, resultante da depleção de NADPH+. O acúmulo excessivo de dissulfeto interrompe as ligações dissulfeto entre as proteínas do citoesqueleto, levando à migração de actina e morte celular 4,5. Ao contrário das formas relatadas anteriormente de morte celular, como apoptose, necrose, piroptose e ferroptose, a dissulfideptose é iniciada pela agregação excessiva de dissulfetos intracelulares e não é antagonizada por inibidores específicos de outras vias de morte celular. Como uma morte celular distinta, as evidências indicaram que a superexpressão SLC7A11 evocada por deficiência de glicose celular pode desencadear dissulfidptose4. Após o MIRI, a secreção insuficiente de insulina pode induzir hipoglicemia e subsequente dissulfidptose, o que pode causar mais danos às células do miocárdio e pode ser um dos novos mecanismos do MIRI5.
Neste estudo, o objetivo principal foi utilizar bancos de dados abrangentes de expressão gênica, como o Gene Expression Omnibus (GEO), para analisar a expressão gênica diferencial entre amostras normais e MIRI. Realizamos uma análise de referência cruzada entre genes diferencialmente expressos e genes associados à dissulfideptose, com o objetivo de identificar genes relacionados à dissulfideptose (DRGs) que exibem expressão diferencial no MIRI. Algoritmos de aprendizado de máquina foram empregados para identificar genes-chave e validamos os padrões de expressão dos genes diferenciais de interesse selecionados usando um modelo animal. Essa abordagem fornece uma nova perspectiva para obter uma compreensão mais profunda dos mecanismos potenciais subjacentes ao início e progressão do MIRI. O foco principal desta pesquisa foi investigar um novo mecanismo de morte celular dentro do MIRI, descobrir genes diferenciais associados e conduzir experimentos preliminares para validar esses achados. O objetivo final era identificar novos alvos terapêuticos para mitigar o MIRI com base nesse mecanismo emergente de morte celular.
Protocolo
Para este estudo, nove ratos machos da raça Sprague-Dawley (SD), com idades entre 6 e 8 semanas e pesando 180-220 g, foram selecionados do Centro de Pesquisa de Animais Experimentais de Hubei [SCXK (Hubei) 20200018]. Os ratos foram mantidos em biotérios livres de patógenos específicos para aclimatação por 1 semana, com ciclo claro e escuro de 12 h/12 h, bebendo e comendo livremente. O presente estudo foi conduzido com a aprovação do Comitê de Ética Animal do Terceiro Hospital Afiliado da Universidade Médica de Zunyi (número de aprovação: (2016)-1-56). Todos os procedimentos foram realizados de acordo com as recomendações descritas no Guia para o Cuidado e Uso de Animais de Laboratório publicado pelos Institutos Nacionais de Saúde dos EUA. Medidas rigorosas foram implementadas para minimizar o número de animais utilizados e mitigar seu sofrimento.
1. Análise da expressão diferencial de genes relacionados ao MIRI e dissulfideptose
NOTA: Os dados de sequenciamento de RNA do MIRI denominado GSE214122 foram selecionados para rastrear genes associados à dissulfideptose de acordo com o relatório da literatura6.
- Abra o banco de dados GEO, digite GSE214122 na caixa Pesquisar e clique em Enter. Baixe o(s) arquivo(s) de matriz de série gerado(s) no formato TXT na seção Download.
NOTA: Antes de realizar a análise de dados, normalmente é necessário pré-processar os dados baixados. Isso inclui verificar se há valores ausentes, remover amostras e genes de baixa qualidade e realizar normalização ou padronização nos dados. O conjunto de dados de GSE214122 recuperado continha amostras dos grupos sham-operated e MIRI. - Realize a análise diferencial da expressão gênica das amostras usando o pacote DESeq27. Considere genes com |log2FC|>1 (mudança de dobra maior que 2) e FDR<0,05 como estatisticamente significativos, resultando na identificação de genes diferencialmente expressos associados ao MIRI. Gere um mapa de calor da expressão gênica diferencial na etapa 1.2 usando o pacote de mapa de mapa de calor7 do R.
- Construa um diagrama de Venn usando o pacoteVennDiagram 7 do R (consulte a Tabela de Materiais). Gere gráficos de vulcão para visualizar genes relacionados à dissulfideptose usando um pacote EnhancedVolcano em R7.
- Construa uma rede PPI conectando os genes diferencialmente expressos do MIRI com genes de dissulfideptose usando o banco de dados STRING7 (ver Tabela de Materiais).
2. Construção do modelo MIRI de rato
- Jejue os ratos durante a noite antes de realizar a anestesia. Anestesiar ratos por injeção intraperitoneal de pentobarbital sódico a 2% (40 mg/kg) e prender os membros a uma tábua de ratos em decúbito dorsal. Confirme a profundidade da anestesia sem resposta ao beliscão do dedo do pé, perda do reflexo de endireitamento e reflexo de piscadela. Use pomada veterinária para evitar o ressecamento durante a anestesia.
- Desinfete a pele do pescoço e do tórax com iodóforo e desiodado com álcool 75%. Raspe o pelo do pescoço e do peito esquerdo com um barbeador elétrico e faça uma incisão mediana no pescoço de aproximadamente 2,5 cm de comprimento com um bisturi (consulte a Tabela de Materiais).
- Separe a traqueia com uma pinça e realize a intubação endotraqueal com uma conexão a um ventilador de pequenos animais3 (ver Tabela de Materiais).
- Corte 4 cm longitudinalmente ao longo do lado esquerdo do esterno com uma tesoura oftálmica e separe sem corte os músculos subcutâneos com uma pinça para expor a pleura entre a terceira e a quarta costelas.
- Faça uma incisão de 4-5 cm do processo xifóide até o meio do segundo espaço intercostal esquerdo, segurando o bisturi em um ângulo de 45°. Separe suave e lentamente os músculos peitoral maior e serrátil anterior usando uma pinça oftálmica para acessar o espaço intercostal.
- Faça uma incisão transversal de 1,5 cm entre a terceira e quarta costelas esquerdas usando uma tesoura oftálmica. Após dissecar o pericárdio com pinça oftálmica, eleve o apêndice atrial esquerdo com uma pinça para identificar o óstio coronário na raiz da aorta.
- Ligadura a artéria coronária descendente anterior entre o cone pulmonar e o apêndice atrial esquerdo usando uma sutura 6-0 (ver Tabela de Materiais).
- Após 30 min, afrouxar a sutura para permitir a reperfusão por 120 min, seguido de fechamento das camadas muscular e cutânea com sutura 6-0. Aplique penicilina (ver Tabela de Materiais) injetando intraperitonealmente com 80.000-100.000 unidades.
- No grupo simulado, execute todos os procedimentos como feitos para o grupo modelo, mas apenas rosqueie a artéria coronária descendente anterior sem ligadura ou reperfusão.
- Assim que os ratos recuperarem a consciência, remova os tubos endotraqueais. Devolva os ratos ao biotério para recuperação, onde terão livre acesso a comida e água. Alojar o rato submetido à cirurgia em uma única gaiola com sistema de ventilação independente em laboratório estéril.
- Durante 30 min de isquemia e 120 min de reperfusão, realizar ecocardiografia. A modelagem foi considerada bem-sucedida se a fração de ejeção do ventrículo esquerdo fosse menor que 50%1,6.
3. Colha tecido cardíaco3
- Após 120 min de reperfusão, colocar os ratos no recipiente de eutanásia para implementar a eutanásia por inalação de dióxido de carbono. Colete os tecidos cardíacos do rato imediatamente.
- Remova os pelos abdominais dos ratos com um dispositivo de barbear e esterilize a área anterior do tórax com iodo e álcool 75%. Disseque a cavidade abdominal do rato ao longo da linha média do abdômen com um bisturi.
- Corte a pele do processo xifóide para cima até o espaço submandibular com uma tesoura cirúrgica e retire sem corte a pele e o tecido subcutâneo com uma pinça em ambos os lados para expor a caixa torácica e a camada muscular cervical superficial (ver Tabela de Materiais).
- Abra o tórax e a pleura ao longo da linha média com uma tesoura cirúrgica para expor o coração, prenda a aorta com um clipe vascular e corte a artéria com uma tesoura oftálmica. Retire o coração e disseque em bloco de tecido 1 mm3 em solução salina fria em banho de gelo e fixe com paraformaldeído a 4% por 48 h (ver Tabela de Materiais).
4. Coloração de hematoxilina-eosina (H & E)
- Desidratação e incorporação: Desidrate o bloco de tecido cardíaco mergulhando em sequência em álcool 75%, álcool 85%, álcool 95% (I), álcool 95% (II), álcool 100% (I) e álcool 100% (II) por 1 h em cada solução. Transparenteize com xileno (I) e xileno (II) mergulhando por 15 min em cada solução. Coloque o bloco de tecido cardíaco em parafina líquida (I), parafina (II) e parafina (III) por 20-30 min em sequência. Deixe o bloco de parafina esfriar naturalmente até a solidificação.
- Corte o bloco de tecido cardíaco embebido em parafina em seções de 4 μm usando uma máquina de fatiar parafina (consulte a Tabela de Materiais). Colocar as secções planas com uma escova macia num banho-maria a 45 °C e, em seguida, recolher as secções totalmente expandidas com uma lâmina.
- Desparafinação: Asse as fatias no forno por 30-60 min a 55 °C e desparafine com xileno (I) por 5-10 min e xileno (II) por 5-10 min. Em seguida, lave o xileno com etanol absoluto por 1-5 min, etanol 95% por 1-5 min e etanol 75% por 1-5 min e enxágue com água da torneira por 1-5 min.
- Coloração: Manchar com uma solução de hematoxilina por 5-20 min, enxaguar com água da torneira por 5-10 s, manchar com uma solução de eosina por 15-30 s e lavar com etanol 75%-85% por 30 s.
- Desidratação, transparente e vedação: Desidratar com etanol 95% (I) por 0,5-2 min e etanol 95% (II) por 2-5 min, seguido de etanol absoluto (I) por 2-5 min e etanol absoluto (II) por 2-5 min. Transparenteize com xileno (I) por 1 min e xileno (II) por 1 min e, em seguida, sele a fatia adicionando duas gotas de resina neutra na fatia e coloque uma lamínula. Pressione levemente a lamínula para encher a fatia com resina neutra (consulte a Tabela de Materiais).
- Observe cada fatia sob um microscópio óptico com ampliação de 200x de maneira duplo-cega por dois técnicos independentes.
- Selecione três campos visuais aleatórios para avaliar a congestão miocárdica, hemorragia, fibrose, necrose e degeneração. Use os seguintes critérios de pontuação: 0 indicou nenhuma lesão; 0-1 indicaram lesões menores que 1/4 da área designada; 1-2 lesões indicadas variaram de aproximadamente 1/4 a 1/2 da área designada; 2-3 lesões indicadas variaram de aproximadamente 1/2 a 3/4 da área designada; e 3-4 lesões indicadas eram maiores que 3/4 da área designada.
5. Ensaio TUNEL
- Diluir a proteinase K com solução tampão fosfato (PBS) a 40 μg/ml e adicionar 100 μl de proteinase K a 4 μm de secções preparadas no passo 4.3 para cobrir toda a área da amostra e incubar durante 10 min à temperatura ambiente.
NOTA: A proteinase K está incluída no kit de detecção de apoptose TUNEL (consulte a Tabela de Materiais). - Lave a seção 2x com PBS por 5 min cada, remova o excesso de líquido por sucção com papel de filtro e mantenha a seção úmida em uma caixa molhada.
NOTA: A proteinase K deve ser lavada para não interferir nas reações de rotulagem subsequentes. - Adicionar uniformemente 50 μl de solução de reacção TUNEL contendo 2 μl de enzima TdT e 48 μl de tampão de reacção TUNEL e colocar a secção numa caixa húmida para incubar a 37 °C no escuro durante 2 h.
NOTA: A enzima TdT e o tampão de reação TUNEL estão incluídos no kit de detecção de apoptose TUNEL. - Descarte a solução de reação TUNEL e enxágue 2x com PBS por 5 min cada. Lave a seção 3x por 5 min cada com PBS contendo 0,1% de Triton X-100 e 5 mg / mL de BSA.
- Adicione uniformemente 50 μL de solução de coloração DAPI e incube por 10 min em temperatura ambiente no escuro. Descarte a solução de coloração DAPI e mergulhe a seção em PBS, 3x, 5 min de cada vez.
- Obter sinais vermelhos e azuis DAPI TUNEL nos comprimentos de onda de excitação e emissão de 593/614 nm e 364/454 nm, respectivamente. Capture imagens sob um microscópio de fluorescência com ampliação de 200x (consulte a Tabela de Materiais).
6. Coloração imuno-histoquímica
- Adicione 200 μL de tampão bloqueador de peroxidase endógena aprimorada à seção na etapa 4.3 e incube por 10 min em temperatura ambiente no escuro. Lave a seção 2x com água destilada por 5 min cada.
- Adicione 1 mL de solução melhorada de recuperação de antígeno citrato e 49 mL de água bidestilada à caixa de incubação de anticorpos e misture suavemente. Mergulhe a seção na solução de recuperação de antígeno e aqueça a 95 ° C por 20 min antes de esfriar à temperatura ambiente (consulte a Tabela de Materiais).
- Lave a seção 2x com PBS por 5 min cada. Coloque a seção na caixa de incubação de anticorpos para incubar com 100 μL de anticorpos primários contra F-actina (1:100), GLUT1 (1:200), Myh9 (1:200), SLC7A11 (1:200) e SLC3A2 (1:200) durante a noite a 4 °C.
- Lave a seção 2x com PBS por 5 min cada. Adicione 100 μL de anticorpo secundário à seção e incube por 20 min em temperatura ambiente.
- Lave a seção 2x com PBS por 5 min cada. Manchar com 100 μL de solução de trabalho DAB por 5 min em temperatura ambiente e interromper a reação de cor lavando com água purificada.
NOTA: A solução de trabalho DAB foi preparada misturando a solução cromogênica DAB A e a solução cromogênica DAB B na proporção de 1:1, que está incluída no kit de desenvolvimento de cor da peroxidase de rábano DAB (consulte a Tabela de Materiais). - Pinte a seção com 100 μL de solução de coloração de hematoxilina por 3 min e lave com água corrente por 3 min (consulte a Tabela de Materiais).
NOTA: O kit de coloração de hematoxilina e eosina inclui uma solução de coloração de hematoxilina. - Mergulhe a seção em etanol 75%, etanol 95% e etanol 100% por 2 min em cada.
- Sele a seção com resina neutra, cubra com uma lamínula e examine a seção sob um microscópio óptico com ampliação de 200x (consulte a Tabela de Materiais). Tecidos exibindo uma cor marrom são reações positivas. Calcule a taxa de expressão positiva como a razão entre a área corada e a área total do campo de visão. Realizar análises quantitativas utilizando o software Image J.
7. Detecção de Western blot
NOTA: O tampão de lise e o inibidor de protease foram incluídos em um kit de quantificação de proteína BCA (ver Tabela de Materiais).
- Pesar 0,1 g de amostras miocárdicas frescas da etapa 3.5 e misturar com 1 mL de tampão de lise e 10 μL de inibidor de protease, seguido de trituração em um recipiente de moagem de vidro e centrifugação a 10.304 x g por 10 min a 4 ° C.
- Coletar o sobrenadante e realizar a quantificação de proteínas usando o método BCA de acordo com as instruções do fabricante8. Ferva o sobrenadante a 95 °C durante 5 min.
- Separe proteínas de diferentes pesos moleculares usando gel separador a 10% carregando 10 μL de amostra para eletroforese de proteínas a 80/120 V.
- Transfira as proteínas do gel para uma membrana de PVDF9. Em seguida, bloqueie a membrana com leite em pó desnatado a 5% por 1 h e incube durante a noite a 4 ° C com anticorpos primários Myh9, SLC7A11, SLC3A2 e β-tubulina em uma diluição de 1: 1000 (ver Tabela de Materiais).
- Após a lavagem com PBS 3x por 5 min cada, incube a membrana com anticorpo secundário (1:5000; ver Tabela de Materiais) à temperatura ambiente por 2 h, seguida de lavagem com PBS 3x por 5 min cada antes da visualização com um instrumento de visualização de proteínas (ver Tabela de Materiais).
- Calcule a proporção da escala de cinza das bandas para β-tubulina usando o software Image J 8,9.
8. Análise estatística
- Expresse os dados como média ± desvio padrão. Analise os dados usando a análise de variância unidirecional seguida pelo teste post hoc de diferença menos significativa. Realize a análise estatística usando uma ferramenta de análise estatística comercial (consulte a Tabela de Materiais) e considere p < 0,05 como estatisticamente significativo.
Resultados
Triagem de DRGs no MIRI
O conjunto de dados GSE214122 do Gene Expression Omnibus incluiu três dados de amostras simuladas e três MIRI. Usando o pacote DESeq2 em R, 1233 genes diferencialmente expressos (DEGs) foram identificados entre MIRI e amostras simuladas. Com base em |log2FC|>2 e FDR<0,05, 417 genes significativamente diferentes foram selecionados usando o pacote de mapa de trigo de R (Figura 1A). Em seguida, 15 genes de interseção entre esses 1233 DEGs e 106 DRGs foram apresentados na Figura 1B. Os 15 DRGs diferencialmente expressos foram anotados por um gráfico de vulcões, com 12 DRGs sendo rotulados no mapa do vulcão, conforme ilustrado na Figura 1C. Uma rede PPI para os 15 DRGs foi então construída usando STRING com três genes na rede não sendo conectados e, consequentemente, removidos. A rede PPI final compreendia 12 genes, conforme ilustrado na Figura 1D.
MIRI promove dano às células miocárdicas de rato
Os resultados da coloração HE demonstraram que nos grupos controle (sem procedimentos cirúrgicos) e simulado (com os mesmos procedimentos cirúrgicos do grupo modelo, mas sem procedimentos de isquemia-reperfusão), as células miocárdicas exibiram morfologia regular, fibras miocárdicas bem organizadas e sem sinais evidentes de edema ou infiltração de células inflamatórias. Em contraste, o grupo modelo exibiu arranjo de células miocárdicas desorganizado, inchaço, contagem de células reduzida, fragmentação das fibras miocárdicas e infiltração perceptível de células espumosas e um grande número de células inflamatórias (Figura 2A).
Os resultados da coloração TUNEL revelaram que, em comparação com os grupos controle e simulado, o grupo modelo exibiu um aumento significativo nas células apoptóticas marcadas com fluorescência vermelha nos tecidos miocárdicos de ratos (Figura 2B). A taxa de apoptose foi acentuadamente elevada (Figura 2C, p < 0,01) no grupo modelo, sem diferenças estatisticamente significativas observadas entre os grupos controle e simulado.
Expressão de F-actina e GLUT1 em tecido miocárdico de rato de MIRI
Em comparação com os grupos controle e simulado, o grupo modelo exibiu um aumento significativo na expressão de GLUT1 (Figura 3A, B, p < 0,01) e uma diminuição significativa na expressão de F-actina (Figura 3C, D, p < 0,01). Não foram observadas diferenças significativas entre os grupos controle e sham para a expressão das proteínas GLUT1 e F-actina.
Expressão de Myh9, SLC7A11 e SLC3A2 em tecido miocárdico de rato de MIRI
A análise imuno-histoquímica foi empregada pela primeira vez para avaliar os níveis de expressão de DRGs no modelo MIRI de ratos. Os resultados mostraram que, em comparação com os grupos controle e simulado, o grupo modelo apresentou níveis de expressão significativamente aumentados de Myh9 (Figura 4A, B, p < 0,01) e SLC3A2 (Figura 4E, F, p < 0,01). Em contraste, a expressão de SLC7A11 foi significativamente reduzida (Figura 4C,D, p < 0,01). Da mesma forma, os resultados do western blotting mostraram que, em comparação com os grupos controle e simulado, o grupo modelo aumentou significativamente os níveis de expressão de Myh9 (Figura 5A, B, p < 0,01) e SLC3A2 (Figura 5A, D, p < 0,01), enquanto a expressão de SLC7A11 (Figura 5A, C, p < 0,01) foi significativamente reduzida.

Figura 1: Triagem de genes relacionados à dissulfideptose no MIRI. (A) Mapa de calor de 1233 genes para análise de expressão diferencial em MIRI e amostras simuladas. (B) Diagrama de Venn de DEGs e DRGs. Havia 15 genes comuns entre os 1233 DEGs e 99 DRGs, conforme mostrado na caixa. (C) Gráficos de vulcões de DEGs no conjunto de dados GSE214122. Pontos vermelhos e verdes no gráfico, respectivamente, indicam genes regulados positivamente e diminuídos. (D) A rede PPI dos 12 DRGs do hub foi construída usando o banco de dados STRING. DEGs, genes diferencialmente expressos; DRGs, genes relacionados à dissulfideptose. Clique aqui para ver uma versão maior desta figura.

Figura 2: MIRI promove dano às células miocárdicas. (A) Imagens representativas das lesões miocárdicas avaliadas pela coloração H&E (barra de escala = 50 μm). (B) Apoptose de cardiomiócitos detectada pelo ensaio TUNEL. O grupo modelo exibiu um aumento significativo nas células apoptóticas marcadas com fluorescência vermelha nos tecidos miocárdicos de ratos (barra de escala = 50 μm). (C) Os resultados estatísticos da taxa de apoptose analisando a intensidade do sinal de fluorescência vermelha usando o software Image J. **p < 0,01, o grupo modelo vs. o grupo simulado, n=3. Os dados são expressos como a média ± desvio padrão. Clique aqui para ver uma versão maior desta figura.
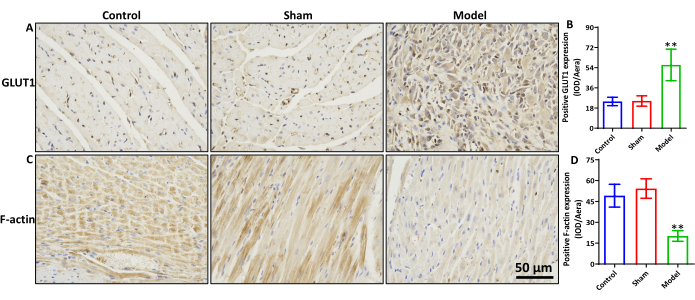
Figura 3: Expressão de F-actina e GLUT1 no tecido miocárdico de MIRI de ratos. (A) coloração IHC de GLUT1 e (B) resultados estatísticos de sua expressão proteica. (C) coloração IHC de F-actina e (D) resultados estatísticos de sua expressão proteica. barra de escala = 50 μm. **p < 0,01, o grupo modelo vs. o grupo simulado, n=6. Os dados são expressos como a média ± desvio padrão. Clique aqui para ver uma versão maior desta figura.

Figura 4: Análise da coloração IHC da expressão das proteínas relacionadas à dissulfideptose Myh9, SLC7A11 e SLC3A2 no tecido miocárdico de MIRI de ratos. (A) Coloração IHC de Myh9 e (B) resultados estatísticos de sua expressão proteica. (C) coloração IHC de SLC7A11 e (D) resultados estatísticos de sua expressão proteica. (E) coloração IHC de SLC3A2 e (F) resultados estatísticos de sua expressão proteica. barra de escala = 50 μm. **p < 0,01, o grupo modelo vs. o grupo simulado, n=3. Os dados são expressos como a média ± desvio padrão. Clique aqui para ver uma versão maior desta figura.

Figura 5: Análise de Western blot da expressão das proteínas relacionadas à dissulfideptose Myh9, SLC7A11 e SLC3A2 no tecido miocárdico de MIRI de ratos. (A) Bandas de proteínas representativas. Resultados estatísticos da expressão da proteína (B) Myh9, (C) SLC7A11 e (D) SLC3A2. **p < 0,01, o grupo modelo vs. o grupo simulado, n=3. Os valores em tons de cinza das proteínas foram analisados usando o software Image J. Os dados são expressos como a média ± desvio padrão. Clique aqui para ver uma versão maior desta figura.
Discussão
A dissulfideptose está intimamente associada ao citoesqueleto de actina, uma estrutura celular crítica essencial para manter a forma e a viabilidade celular. Composto por filamentos de actina, o citoesqueleto de actina confere forma e estrutura celular geral. A F-actina serve como um marcador para o citoesqueleto celular e, sob condições de falta de glicose, as ligações dissulfeto aumentam significativamente, levando à regulação negativa da F-actina. Esse fenômeno afeta principalmente processos e vias relacionadas ao citoesqueleto de actina e à adesão celular10,11.
No contexto do metabolismo da glicose, os transportadores de glicose (GLUTs) desempenham um papel vital na manutenção do equilíbrio dinâmico da glicose. O GLUT1, um dos primeiros membros descobertos da família GLUT, é uma proteína relacionada à captação de glicose com ampla distribuição tecidual. É expresso principalmente emhemácias12 e células endoteliais da barreira hematoencefálica13, onde participa do transporte transmembrana de glicose. O GLUT1 pode estar intimamente relacionado ao desenvolvimento de doenças associadas ao metabolismo anormal da glicose. Em modelos de falta de glicose, a expressão de GLUT1 é regulada positivamente, promovendo a morte de células tumorais. No modelo MIRI, a regulação positiva da expressão de GLUT1 piora o MIRI, mas melhorias são observadas quando o GLUT1 é regulado negativamente, tornando-o um alvo chave sob falta de glicose14. A imuno-histoquímica revelou um aumento intracelular significativo na proteína transportadora de glicose GLUT1 no modelo MIRI em comparação com os grupos controle e simulado. Ao mesmo tempo, a F-actina foi significativamente diminuída intracelularmente em comparação com os grupos controle e simulado. Isso sugere que o colapso do citoesqueleto de actina em condições de falta de glicose pode contribuir para o agravamento do MIRI, tornando o colapso do citoesqueleto de actina o mecanismo primário da dissulfideptose. Portanto, especulamos que a regulação positiva de GLUT1 no modelo MIRI induza dissulfideptose, levando a lesão de isquemia-reperfusão agravada.
O gene Myh9 codifica a cadeia pesada da miosina não muscular IIA, uma miosina citoplasmática amplamente expressa envolvida em vários processos que requerem a geração de forças mecânicas intracelulares e o reposicionamento do citoesqueleto de actina15. Pacientes com trombocitopenia congênita devido a mutações no gene Myh9 são propensos à formação de trombos e infarto agudo do miocárdio16. Portanto, Myh9 pode ser um fator preditivo para IAM ou MIRI. SLC7A11 e SLC3A2 são proteínas que auxiliam as células na obtenção de cisteína e glutationa, essenciais para manter a saúde e o equilíbrio celular. SLC7A11 pode mitigar a peroxidação lipídica e o estresse oxidativo para desempenhar um papel protetor no MIRI17. Nosso estudo explorou as potenciais funções biológicas e o significado terapêutico dos genes relacionados à dissulfideptose por meio do banco de dados GEO. Encontramos uma sobreposição limitada entre MIRI e genes relacionados à dissulfideptose, com um total de 12 genes sobrepostos. Por meio da análise de valor P e Log2Fold Change, identificamos quatro genes que estavam mais intimamente relacionados no modelo MIRI. Os resultados indicaram uma regulação positiva significativa da expressão de Myh9 e uma regulação negativa significativa da expressão de SLC7A11 no grupo MIRI. No entanto, não foi observada diferença estatisticamente significativa na expressão de ACTC. Os resultados experimentais confirmaram que Myh9 e SLC7A11 estão envolvidos no MIRI, e seus mecanismos podem estar intimamente relacionados à dissulfideptose mediada pela depleção de NADPH. Esses achados fornecem pistas importantes para identificar novos fatores preditivos e estratégias de tratamento personalizadas para MIRI (Figura 5).
Em resumo, a identificação e caracterização dos mecanismos de morte celular não apenas promovem nossa compreensão fundamental da homeostase celular, mas também destacam o papel significativo desempenhado pela dissulfideptose no MIRI. A deficiência deste método é a falta de experimentos críticos de validação para dissulfideptose. Atualmente, é possível detectar e identificar dissulfetoptose por meio da sonda fluorescente DCP-Bio1, que se liga a ligações dissulfeto, marcando dissulfetos intracelulares e extracelulares e facilitando a observação de dissulfetos em células ou tecidos18. Essas novas sondas podem ser empregadas para estudar a dinâmica espaço-temporal entre dissulfideptose e MIRI.
Divulgações
Os autores não têm nada a divulgar.
Agradecimentos
Esta pesquisa foi apoiada pelo Departamento Provincial de Ciência e Tecnologia de Guizhou (Qiankehe [2022]-583) e pela Administração Provincial de Medicina Tradicional Chinesa de Guizhou (QZYY-2016-019).
Materiais
| Name | Company | Catalog Number | Comments |
| 75% alcohol | Hunan Tongruijian Pharmaceutical Co. Ltd., Hunan, China | 85026 | |
| 6-0 nylon suture | Shanghai Pudong Jinhuan Medical Supplies Co. Ltd., Shanghai, China | CS002 | |
| BCA protein quantification kit | Beyotime Biotechnology, Shanghai, China | P0011 | |
| Bull serum albumin | Beyotime Biotechnology, Shanghai, China | ST2254 | |
| Centrifuge | Hunan Kaida Scientific Instrument Co. Ltd., Hunan, China | KH19A | |
| DAB horseradish peroxidase color development kit | Beyotime Biotechnology, Shanghai, China | P0203 | |
| DAPI staining solution | Beyotime Biotechnology, Shanghai, China | C1006 | |
| DESeq2 package | Version 4.1 | ||
| Electric razor | Kelmerpp | 235376 | |
| Enhanced endogenous peroxidase blocking buffer | Beyotime Biotechnology, Shanghai, China | P0100B | |
| Filter paper | Nanjing Keruicai Equipment Co., Ltd., Nanjing, China | 1.00049E+11 | |
| Fluorescence microscope | Nikon | ECLIPSE Ci | |
| GEO database | https://www.ncbi.nlm.nih.gov/geo/ | ||
| Glass grinder | Shanghai Leigu Instrument Co. Ltd., Shanghai, China | B-013002 | |
| GraphPad Prism | GraphPad Software | V8.0 | |
| Hematoxylin and eosin staining kit | Beyotime Biotechnology, Shanghai, China | C0105M | |
| Image J software | National Institutes of Health, Bethesda, USA | v1.8.0 | |
| Improved citrate antigen retrieval solution (50X) | Beyotime Biotechnology, Shanghai, China | P0083 | |
| Iodophor | Folca, Shenzhen, China | 1.00077E+11 | |
| Optical microscope | Nikon | ECLIPSE Ci | |
| Phosphate buffer solution | Beyotime Biotechnology, Shanghai, China | C0221A | |
| Primary antibodies against GLUT1 | Proteintech Group, Inc, Wuhan, China | 21829-1-AP | |
| Primary antibodies against MYH9 | Proteintech Group, Inc, Wuhan, China | 11128-1-AP | |
| Primary antibodies against SLC3A2 | Wuhan Lingsi Biotechnology Co., Ltd., Wuhan, China | LJS-D-7468 | |
| Primary antibodies against SLC7A11 | Proteintech Group, Inc, Wuhan, China | 26864-1-AP | |
| Protein visualization instrument | Thermo Fisher Scientific Inc. | iBright CL750 | |
| Rat board | Zhengzhou Haopai Biotechnology Co. Ltd., Zhengzhou, China | JPB-E | |
| Scalpel | Shanghai Lianhui Medical Supplies Co., Ltd., Shanghai, China | 1.00471E+13 | |
| Secondary antibody | Wuhan Boster Biological Technology, Ltd., Wuhan, China | BA1054 | |
| STRING database | https://cn.string-db.org/ | Version 12.0 | |
| Triton X-100 | Beyotime Biotechnology, Shanghai, China | ST1722 | |
| TUNEL apoptosis detection kit | Proteintech Group, Inc., Wuhan, China | PF00009 |
Referências
- Lu, Z., et al. Tripartite motif 38 attenuates cardiac fibrosis after myocardial infarction by suppressing TAK1 activation via TAB2/3 degradation. iScience. 25 (8), 104780 (2022).
- Tu, Q., et al. EGCG decreases myocardial infarction in both I/R and MIRI rats through reducing intracellular Ca2+ and increasing TnT levels in cardiomyocytes. Adv Clin Exp Med. 30 (6), 607-616 (2021).
- Li, Q., et al. Histone deacetylase HDAC4 participates in the pathological process of myocardial ischemia-reperfusion injury via MEKK1/JNK pathway by binding to miR-206. Cell Death Dis. 7 (1), 240 (2021).
- Liu, X., et al. Actin cytoskeleton vulnerability to disulfide stress mediates disulfidptosis. Nat Cell Biol. 25 (3), 404-414 (2023).
- Yang, L., et al. Analysis of risk genes associated with disulfidptosis-related myocardial ischemia-reperfusion injury. J Chongqing Med Univ. 49 (4), 401-408 (2024).
- Xiao, G., et al. CXCR1 and its downstream NF-κB inflammation signaling pathway as a key target of Guanxinning injection for myocardial ischemia/reperfusion injury. Front Immunol. 13, 1007341 (2022).
- Zhu, E. Y., et al. Screening of immune-related secretory proteins linking chronic kidney disease with calcific aortic valve disease based on comprehensive bioinformatics analysis and machine learning. J Transl Med. 21, 359 (2023).
- Shi, D. C., et al. Potential of Dendrobium officinale oligosaccharides to alleviate chronic colitis by modulating inflammation and gut microbiota. FoodMedHomo. 2, 9420077 (2025).
- Ning, E. J., et al. Artemisia argyi polysaccharide alleviates intestinal inflammation and intestinal flora dysbiosis in lipopolysaccharide-treated mice. FoodMedHomo. 1 (1), 9420008 (2024).
- Zhou, H., Wang, J., Zhu, P., Hu, S., Ren, J. Ripk3 regulates cardiac microvascular reperfusion injury: The role of IP3R-dependent calcium overload, XO-mediated oxidative stress and F-action/filopodia based cellular migration. Cell Signal. 45, 12-22 (2018).
- Antonipillai, J., Rigby, S., Bassler, N., Peter, K., Bernard, O. Pharmacological inhibition of LIM kinase pathway impairs platelets functionality and facilitates thrombolysis. Exp Cell Res. 382 (2), 111458 (2019).
- Kaumeyer, B. A., et al. GLUT1 immunohistochemistry is a highly sensitive and relatively specific marker for erythroid lineage in benign and malignant hematopoietic tissues. Am J Clin Pathol. 158 (2), 228-234 (2022).
- Patching, S. G. Glucose transporters at the blood-brain barrier: Function, regulation and gateways for drug delivery. Mol Neurobiol. 54 (2), 1046-1077 (2017).
- Zhou, H., et al. 3-iodothyronamine inhibits apoptosis induced by myocardial ischemia reperfusion via the Akt/FoxO1 signaling pathway. Ann Trans Med. 10 (4), 168 (2022).
- Pecci, A., et al. MYH9: Structure, functions and role of non-muscle myosin IIA in human disease. Gene. 664, 152-167 (2018).
- Girolami, A., et al. Occurrence of thrombosis in congenital thrombocytopenic disorders: A critical annotation of the literature. Blood Coagulat Fibrino. 24 (1), 18-22 (2013).
- Li, P., et al. SLC7A11-associated ferroptosis in acute injury diseases: Mechanisms and strategies. Eur Rev Med Pharmaco Sci. 27 (10), 4386-4398 (2023).
- Wu, X., et al. Dihydrotanshinone I preconditions myocardium against ischemic injury via PKM2 glutathionylation sensitive to ROS. Acta Pharma Sinica B. 13 (1), 113-127 (2023).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados