Method Article
Analisi dei geni differenziali e convalida della disulfideptosi nel modello di lesione da ischemia-riperfusione miocardica di ratto
In questo articolo
Riepilogo
Il protocollo descrive l'identificazione di diversi nuovi geni differenziali correlati alla disulfideptosi associati al danno da ischemia-riperfusione miocardica mediante analisi bioinformatica e validazione sperimentale.
Abstract
Il danno da ischemia-riperfusione miocardica (MIRI) è una lesione aggiuntiva che si verifica durante il processo di ripristino del flusso sanguigno del tessuto cardiaco dopo un danno indotto dall'ischemia. La MIRI influisce seriamente sull'efficacia e sulla prognosi a breve e lungo termine della riperfusione dopo infarto del miocardio. Al momento, il meccanismo di MIRI non è del tutto chiaro. La disulfideptosi è una nuova modalità di morte cellulare e la relazione tra MIRI ed espressione dei geni correlati alla disulfideptosi (DRG) non è ancora chiara. In primo luogo, questo studio esplora i geni differenzialmente espressi associati alla disulfideptosi nella MIRI attraverso l'analisi bioinformatica. In secondo luogo, costruendo un modello di ratto di MIRI, i DRG sono stati ulteriormente rilevati. Questo studio ha identificato 12 geni correlati, tra cui Myh9, SLC7A11, SLC3A2, Myh7b, ACTB, FLNB, Actn1, Actn4, Flnc, Dbn1 e Pdlim1. Il tessuto miocardico dei ratti con MIRI mostra evidenti danni patologici ed eventi di apoptosi. I risultati dell'immunoistochimica hanno indicato che la stimolazione MIRI aumenta l'espressione della proteina GLUT1 nel tessuto miocardico, ma limita l'espressione della proteina F-actina. Inoltre, sono state convalidate differenze significative nell'espressione di tre proteine utilizzando set di dati esterni e modelli di ratto MIRI. Questo studio ha dimostrato che i DRG avevano un valore predittivo significativo nella MIRI, fornendo nuove prospettive per l'esplorazione di biomarcatori e potenziali bersagli terapeutici della MIRI.
Introduzione
L'infarto miocardico acuto (IMA) è una grave condizione cardiovascolare e rimane una delle principali cause globali di mortalità. L'intervento coronarico percutaneo ha ridotto significativamente i tassi di mortalità nei pazienti con infarto miocardicomiocardico 1. Tuttavia, la terapia di riperfusione volta a ripristinare l'afflusso di sangue al miocardio è accompagnata da una serie di risposte patologiche e fisiologiche avverse. Questi processi possono provocare un aumento delle dimensioni dell'infarto, la morte delle cellule miocardiche, aritmie ventricolari sostenute e morte improvvisa2. Il danno da ischemia-riperfusione miocardica (MIRI) è una condizione cardiovascolare complessa influenzata da fattori quali citochine, chemochine, fattori di crescita, stress ossidativo e sovraccarico di calcio3. La mitigazione della MIRI rimane una sfida significativa.
Recentemente, la disulfideptosi è emersa come una nuova forma di morte cellulare caratterizzata da un rapido collasso della rete di actina citoscheletrica a causa di un eccessivo accumulo di disolfuri, inclusa la cisteina, all'interno delle cellule, derivante dalla deplezione di NADPH+. L'eccessivo accumulo di disolfuro interrompe i legami disolfuro tra le proteine del citoscheletro, portando alla migrazione dell'actina e alla morte cellulare 4,5. A differenza delle forme di morte cellulare precedentemente riportate come l'apoptosi, la necrosi, la piroptosi e la ferroptosi, la disulfideptosi è iniziata dall'eccessiva aggregazione di disolfuri intracellulari e non è antagonizzata da inibitori specifici di altre vie di morte cellulare. Come morte cellulare distintiva, l'evidenza ha indicato che l'eccessiva espressione di SLC7A11 evocata dalla carenza di glucosio cellulare può innescare la disulfidptosi4. Dopo la MIRI, un'insufficiente secrezione di insulina può indurre ipoglicemia e successiva disulfidptosi, che può causare ulteriori danni alle cellule miocardiche e può essere uno dei nuovi meccanismi della MIRI5.
In questo studio, l'obiettivo principale era quello di utilizzare database completi di espressione genica, come il Gene Expression Omnibus (GEO), per analizzare l'espressione genica differenziale tra campioni normali e MIRI. Abbiamo condotto un'analisi cross-referenziale tra geni differenzialmente espressi e geni associati a disulfideptosi, con l'obiettivo di identificare geni correlati alla disulfideptosi (DRGs) che mostrano espressione differenziale in MIRI. Sono stati impiegati algoritmi di apprendimento automatico per identificare i geni chiave e abbiamo convalidato i modelli di espressione dei geni differenziali selezionati di interesse utilizzando un modello animale. Questo approccio fornisce una nuova prospettiva per ottenere una comprensione più profonda dei potenziali meccanismi alla base dell'insorgenza e della progressione della MIRI. L'obiettivo principale di questa ricerca è stato quello di studiare un nuovo meccanismo di morte cellulare all'interno di MIRI, scoprire geni differenziali associati e condurre esperimenti preliminari per convalidare questi risultati. L'obiettivo finale era quello di identificare nuovi bersagli terapeutici per mitigare la MIRI sulla base di questo meccanismo emergente di morte cellulare.
Protocollo
Per questo studio, nove ratti maschi di Sprague-Dawley (SD), di età compresa tra 6 e 8 settimane e del peso di 180-220 g, sono stati selezionati dall'Hubei Experimental Animal Research Center [SCXK (Hubei) 20200018]. I ratti sono stati tenuti in stalle per animali privi di agenti patogeni specifici per acclimatarsi per 1 settimana, con un ciclo di luce e buio di 12 ore/12 ore, bevendo e mangiando liberamente. L'attuale studio è stato condotto con l'approvazione del Comitato Etico Animale del Terzo Ospedale Affiliato dell'Università di Medicina di Zunyi (numero di approvazione: (2016)-1-56). Tutte le procedure sono state eseguite in conformità con le raccomandazioni delineate nella Guida per la cura e l'uso degli animali da laboratorio pubblicata dal National Institutes of Health degli Stati Uniti. Sono state implementate misure rigorose per ridurre al minimo il numero di animali utilizzati e per mitigare le loro sofferenze.
1. Analisi dell'espressione differenziale di geni correlati a MIRI e disulfideptosi
NOTA: I dati di sequenziamento dell'RNA di MIRI denominati GSE214122 sono stati selezionati per lo screening dei geni associati alla disulfideptosi secondo il rapportodi letteratura 6.
- Apri il database GEO, digita GSE214122 nella casella di ricerca e fai clic su Invio. Scarica i file di matrice di serie generati in formato TXT nella sezione Download.
NOTA: prima di eseguire l'analisi dei dati, è in genere necessario pre-elaborare i dati scaricati. Ciò include il controllo dei valori mancanti, la rimozione di campioni e geni di bassa qualità e l'esecuzione della normalizzazione o standardizzazione dei dati. Il set di dati GSE214122 recuperato conteneva campioni provenienti dai gruppi gestiti da sham e MIRI. - Eseguire l'analisi dell'espressione genica differenziale dei campioni utilizzando il pacchetto DESeq27. Considerare i geni con |log2FC|>1 (variazione di piega maggiore di 2) e FDR<0,05 come statisticamente significativi, con conseguente identificazione di geni differenzialmente espressi associati a MIRI. Genera una mappa termica dell'espressione genica differenziale nel passaggio 1.2 utilizzando il pacchetto pheatmap7 di R.
- Costruisci un diagramma di Venn utilizzando il pacchetto VennDiagram7 di R (vedi Tabella dei materiali). Genera grafici Volcano per visualizzare i geni correlati alla disulfideptosi utilizzando un pacchetto EnhancedVolcano in R7.
- Costruire una rete PPI che colleghi i geni MIRI differenzialmente espressi con i geni della disulfideptosi utilizzando il database STRING7 (vedi Tabella dei materiali).
2. Costruzione del modello MIRI del ratto
- Digiunare i ratti durante la notte prima di eseguire l'anestesia. Anestetizzare i ratti mediante iniezione intraperitoneale di pentobarbital di sodio al 2% (40 mg/kg) e fissare gli arti a una tavola per ratti in posizione supina. Confermare la profondità dell'anestesia in assenza di risposta al pizzicamento delle dita dei piedi, alla perdita del riflesso di raddrizzamento e al riflesso dell'occhiolino. Utilizzare un unguento veterinario per prevenire la secchezza durante l'anestesia.
- Disinfettare la pelle del collo e del torace con iodoforo e deiodinato con alcol al 75%. Radere il pelo del collo e del torace sinistro con un rasoio elettrico e praticare un'incisione mediana del collo di circa 2,5 cm di lunghezza con un bisturi (vedi Tabella dei materiali).
- Separare la trachea con una pinza ed eseguire l'intubazione endotracheale con un collegamento a un ventilatore per piccoli animali3 (vedere la Tabella dei materiali).
- Tagliare longitudinalmente 4 cm lungo il lato sinistro dello sterno con le forbici oftalmiche e separare senza mezzi termini i muscoli sottocutanei con una pinza per esporre la pleura tra la terza e la quarta costola.
- Praticare un'incisione di 4-5 cm dal processo xifoideo al centro del secondo spazio intercostale sinistro tenendo il bisturi a un angolo di 45°. Separare delicatamente e lentamente i muscoli pettorale maggiore e dentato anteriore utilizzando una pinza oftalmica per accedere allo spazio intercostale.
- Praticare un'incisione trasversale di 1,5 cm tra la terza e la quarta costola sinistra utilizzando le forbici oftalmiche. Dopo aver sezionato il pericardio con una pinza oftalmica, sollevare l'appendice atriale sinistra con una pinza per identificare l'ostio coronarico alla radice aortica.
- Legare l'arteria coronaria discendente anteriore sinistra tra il cono polmonare e l'appendice atriale sinistra utilizzando una sutura 6-0 (vedi Tabella dei materiali).
- Dopo 30 minuti, allentare la sutura per consentire la riperfusione per 120 minuti, seguita dalla chiusura degli strati muscolari e cutanei utilizzando una sutura 6-0. Applicare la penicillina (vedi Tabella dei materiali) iniettando per via intraperitoneale 80.000-100.000 unità.
- Nel gruppo fittizio, eseguire tutte le procedure come fatto per il gruppo modello, ma infilare solo l'arteria coronaria discendente anteriore sinistra senza legatura o riperfusione.
- Una volta che i ratti riprendono conoscenza, rimuovere i tubi endotracheali. Riporta i ratti alla struttura per animali per il recupero, dove avranno libero accesso a cibo e acqua. Alloggiare il ratto sottoposto a intervento chirurgico in una gabbia singola con sistema di ventilazione indipendente in un laboratorio sterile.
- Durante 30 minuti di ischemia e 120 minuti di riperfusione, eseguire l'ecocardiografia. La modellazione è stata considerata di successo se la frazione di eiezione ventricolare sinistra misurava meno del 50%1,6.
3. Raccogliere il tessuto cardiaco3
- Dopo 120 minuti di riperfusione, mettere i ratti nel contenitore per l'eutanasia per attuare l'eutanasia mediante inalazione di anidride carbonica. Raccogli immediatamente i tessuti cardiaci del ratto.
- Rimuovere i peli addominali dei ratti con un dispositivo di rasatura e sterilizzare la zona anteriore del torace con iodio e alcol al 75%. Sezionare la cavità addominale del ratto lungo la linea mediana dell'addome con un bisturi.
- Tagliare la pelle dal processo xifoideo verso l'alto fino allo spazio sottomandibolare con le forbici chirurgiche e staccare bruscamente la pelle e il tessuto sottocutaneo con una pinza su entrambi i lati per esporre la gabbia toracica e lo strato muscolare cervicale superficiale (vedi Tabella dei materiali).
- Apri il torace e la pleura lungo la linea mediana con una forbice chirurgica per esporre il cuore, blocca l'aorta con una clip vascolare e taglia l'arteria con le forbici oftalmiche. Estrarre il cuore e sezionare in un blocco di tessutoda 1 mm 3 in soluzione salina fredda su bagno di ghiaccio e fissare con paraformaldeide al 4% per 48 ore (vedi Tabella dei Materiali).
4. Colorazione con ematossilina-eosina (H&E)
- Disidratazione e inclusione: disidratare il blocco di tessuto cardiaco immergendolo in sequenza in alcol al 75%, alcol all'85%, alcol al 95% (I), alcol al 95% (II), alcol al 100% (I) e alcol al 100% (II) per 1 ora in ciascuna soluzione. Rendere trasparenti con xilene (I) e xilene (II) immergendo per 15 minuti in ciascuna soluzione. Posizionare il blocco di tessuto cardiaco in paraffina liquida (I), paraffina (II) e paraffina (III) per 20-30 minuti in sequenza. Lasciare raffreddare naturalmente il blocco di paraffina fino alla solidificazione.
- Tagliare il blocco di tessuto cardiaco incluso in paraffina in sezioni di 4 μm utilizzando una macchina affettatrice per paraffina (vedi Tabella dei materiali). Stendere le sezioni in piano con una spazzola morbida in un bagno d'acqua a 45 °C e quindi raccogliere le sezioni completamente espanse con un vetrino.
- Deceratura: Cuocere le fette in forno per 30-60 minuti a 55 °C e decerare con xilene (I) per 5-10 minuti e xilene (II) per 5-10 minuti. Quindi, lavare lo xilene con etanolo assoluto per 1-5 minuti, etanolo al 95% per 1-5 minuti ed etanolo al 75% per 1-5 minuti e risciacquare con acqua di rubinetto per 1-5 minuti.
- Colorazione: Macchiare con una soluzione di ematossilina per 5-20 minuti, risciacquare con acqua di rubinetto per 5-10 secondi, colorare con una soluzione di eosina per 15-30 secondi e lavare con etanolo al 75%-85% per 30 secondi.
- Disidratante, trasparente e sigillante: disidratare con etanolo (I) al 95% per 0,5-2 minuti ed etanolo al 95% (II) per 2-5 minuti, seguito da etanolo assoluto (I) per 2-5 minuti ed etanolo assoluto (II) per 2-5 minuti. Trasparentizzare con xilene (I) per 1 min e xilene (II) per 1 min, quindi sigillare la fetta aggiungendo due gocce di resina neutra sulla fetta e posizionare un bicchiere di copertura. Premere leggermente il vetro di copertura per riempire la fetta con resina neutra (vedi Tabella dei materiali).
- Osserva ogni fetta al microscopio ottico con un ingrandimento di 200x in doppio cieco da due tecnici indipendenti.
- Seleziona tre campi visivi casuali per valutare la congestione miocardica, l'emorragia, la fibrosi, la necrosi e la degenerazione. Utilizzare i seguenti criteri di punteggio: 0 indicava nessuna lesione; 0-1 le lesioni indicate erano inferiori a 1/4 dell'area designata; 1-2 lesioni indicate variavano da circa 1/4-1/2 dell'area designata; 2-3 lesioni indicate variavano da circa 1/2-3/4 dell'area designata; e 3-4 lesioni indicate erano maggiori di 3/4 dell'area designata.
5. Saggio TUNEL
- Diluire la proteinasi K con una soluzione tampone fosfato (PBS) a 40 μg/mL e aggiungere 100 μL di proteinasi K alle sezioni di 4 μm preparate al punto 4.3 per coprire l'intera area del campione e incubare per 10 minuti a temperatura ambiente.
NOTA: La proteinasi K è inclusa nel kit di rilevamento dell'apoptosi TUNEL (vedere la tabella dei materiali). - Lavare la sezione 2 volte con PBS per 5 minuti ciascuna, rimuovere il liquido in eccesso aspirando con carta da filtro e mantenere la sezione umida in una scatola bagnata.
NOTA: La proteinasi K deve essere lavata in modo pulito per non interferire con le successive reazioni di marcatura. - Aggiungere uniformemente 50 μL di soluzione di reazione TUNEL contenente 2 μL di enzima TdT e 48 μL di tampone di reazione TUNEL e porre la sezione in una scatola bagnata per incubare a 37 °C al buio per 2 ore.
NOTA: L'enzima TdT e il tampone di reazione TUNEL sono inclusi nel kit di rilevamento dell'apoptosi TUNEL. - Eliminare la soluzione di reazione TUNEL e risciacquare 2 volte con PBS per 5 minuti ciascuno. Lavare la sezione 3 volte per 5 minuti ciascuna con PBS contenente lo 0,1% di Triton X-100 e 5 mg/mL di BSA.
- Aggiungere uniformemente 50 μl di soluzione colorante DAPI e incubare per 10 minuti a temperatura ambiente al buio. Eliminare la soluzione colorante DAPI e immergere la ciocca in PBS, 3 volte, 5 minuti ogni volta.
- Ottieni i segnali TUNEL red e DAPI blue a lunghezze d'onda di eccitazione ed emissione rispettivamente di 593/614 nm e 364/454 nm. Acquisisci immagini con un microscopio a fluorescenza con un ingrandimento di 200x (vedi Tabella dei materiali).
6. Colorazione immunoistochimica
- Aggiungere 200 μl di tampone bloccante la perossidasi endogena potenziato alla sezione al passaggio 4.3 e incubare per 10 minuti a temperatura ambiente al buio. Lavare la sezione 2 volte con acqua distillata per 5 minuti ciascuna.
- Aggiungere 1 mL di soluzione migliorata per il recupero dell'antigene del citrato e 49 mL di acqua a doppia distillazione nella scatola di incubazione degli anticorpi e mescolare delicatamente. Immergere la sezione nella soluzione di recupero dell'antigene e riscaldare a 95 °C per 20 minuti prima di raffreddarla a temperatura ambiente (vedere la Tabella dei materiali).
- Lavare la sezione 2 volte con PBS per 5 minuti ciascuna. Posizionare la sezione nella scatola di incubazione degli anticorpi per incubare con 100 μL di anticorpi primari contro F-actina (1:100), GLUT1 (1:200), Myh9 (1:200), SLC7A11 (1:200) e SLC3A2 (1:200) per una notte a 4 °C.
- Lavare la sezione 2 volte con PBS per 5 minuti ciascuna. Aggiungere 100 μl di anticorpo secondario alla sezione e incubare per 20 minuti a temperatura ambiente.
- Lavare la sezione 2 volte con PBS per 5 minuti ciascuna. Colorare con 100 μL di soluzione di lavoro DAB per 5 minuti a temperatura ambiente e arrestare la reazione del colore risciacquando con acqua purificata.
NOTA: La soluzione di lavoro DAB è stata preparata mescolando la soluzione cromogenica DAB A e la soluzione cromogenica DAB B in un rapporto 1:1, che è incluso nel kit di sviluppo del colore della perossidasi di rafano DAB (vedi Tabella dei materiali). - Colorare la sezione con 100 μL di soluzione colorante di ematossilina per 3 minuti e lavare con acqua corrente per 3 minuti (vedere la Tabella dei materiali).
NOTA: Il kit di colorazione ematossilina ed eosina include una soluzione di colorazione ematossilina. - Immergere la sezione in etanolo al 75%, etanolo al 95% ed etanolo al 100% per 2 minuti ciascuna.
- Sigillare la sezione con resina neutra, coprire con un vetrino coprioggetto ed esaminare la sezione al microscopio ottico con un ingrandimento di 200x (vedi Tabella dei materiali). I tessuti che mostrano un colore marrone sono reazioni positive. Calcola il tasso di espressione positiva come il rapporto tra l'area colorata e l'area totale del campo visivo. Esegui analisi quantitative utilizzando il software Image J.
7. Rilevamento di Western blot
NOTA: Il tampone di lisi e l'inibitore della proteasi sono stati inclusi in un kit di quantificazione della proteina BCA (vedi Tabella dei materiali).
- Pesare 0,1 g di campioni miocardici freschi della fase 3.5 e miscelare con 1 mL di tampone di lisi e 10 μL di inibitore della proteasi, quindi macinare in un contenitore di vetro e centrifugare a 10,304 x g per 10 minuti a 4 °C.
- Raccogliere il surnatante ed eseguire la quantificazione delle proteine utilizzando il metodo BCA secondo le istruzioni del produttore8. Far bollire il surnatante a 95 °C per 5 minuti.
- Separare proteine di diverso peso molecolare utilizzando il gel di separazione al 10% caricando un campione da 10 μL per l'elettroforesi delle proteine a 80/120 V.
- Trasferire le proteine sul gel su una membrana in PVDF9. Successivamente, bloccare la membrana con latte scremato in polvere al 5% per 1 ora e incubare per una notte a 4 °C con anticorpi primari Myh9, SLC7A11, SLC3A2 e β-tubulina a una diluizione di 1:1000 (vedere la Tabella dei materiali).
- Dopo il lavaggio con PBS 3 volte per 5 minuti ciascuno, incubare la membrana con l'anticorpo secondario (1:5000; vedere la Tabella dei materiali) a temperatura ambiente per 2 ore, quindi lavare con PBS 3 volte per 5 minuti ciascuno prima della visualizzazione con uno strumento di visualizzazione delle proteine (vedere la Tabella dei materiali).
- Calcola il rapporto della scala di grigi delle bande con la β-tubulina utilizzando il software Image J 8,9.
8. Analisi statistica
- Esprimere i dati come media ± deviazione standard. Analizza i dati utilizzando l'analisi unidirezionale della varianza seguita dal test post hoc della differenza meno significativa. Eseguire analisi statistiche utilizzando uno strumento di analisi statistica commerciale (vedi Tabella dei materiali) e considerare p < 0,05 come statisticamente significativo.
Risultati
Screening dei DRG in MIRI
Il set di dati GSE214122 di Gene Expression Omnibus includeva tre campioni fittizi e tre dati MIRI. Utilizzando il pacchetto DESeq2 in R, sono stati identificati 1233 geni differenzialmente espressi (DEG) tra MIRI e campioni fittizi. Sulla base di |log2FC|>2 e FDR<0.05, 417 geni significativamente diversi sono stati ulteriormente selezionati utilizzando il pacchetto pheatmap di R (Figura 1A). Quindi, nella Figura 1B sono stati presentati 15 geni di intersezione tra questi 1233 DEG e 106 DRG. I 15 DRG differenzialmente espressi sono stati ulteriormente annotati da un grafico del vulcano, con 12 DRG etichettati sulla mappa del vulcano, come illustrato nella Figura 1C. Una rete PPI per i 15 DRG è stata quindi costruita utilizzando STRING con tre geni nella rete non collegati e di conseguenza rimossi. La rete PPI finale comprendeva 12 geni, come illustrato nella Figura 1D.
MIRI promuove il danno alle cellule miocardiche di ratto
I risultati della colorazione HE hanno dimostrato che nei gruppi di controllo (nessuna procedura chirurgica) e fittizi (con le stesse procedure chirurgiche del gruppo modello ma nessuna procedura di ischemia-riperfusione), le cellule miocardiche mostravano una morfologia regolare, fibre miocardiche ben organizzate e nessun segno evidente di edema o infiltrazione di cellule infiammatorie. Al contrario, il gruppo modello ha mostrato una disposizione disorganizzata delle cellule miocardiche, gonfiore, riduzione della conta cellulare, frammentazione delle fibre miocardiche e notevole infiltrazione di cellule schiumose e un gran numero di cellule infiammatorie (Figura 2A).
I risultati della colorazione TUNEL hanno rivelato che, rispetto ai gruppi di controllo e sham, il gruppo modello ha mostrato un aumento significativo delle cellule apoptotiche marcate con fluorescenza rossa all'interno dei tessuti miocardici di ratto (Figura 2B). Il tasso di apoptosi era marcatamente elevato (Figura 2C, p < 0,01) nel gruppo modello, senza differenze statisticamente significative osservate tra il gruppo di controllo e quello fittizio.
Espressione di F-actina e GLUT1 nel tessuto miocardico di ratto di MIRI
Rispetto ai gruppi di controllo e sham, il gruppo modello ha mostrato un aumento significativo dell'espressione di GLUT1 (Figura 3A, B, p < 0,01) e una diminuzione significativa dell'espressione di F-actina (Figura 3C, D, p < 0,01). Non sono state osservate differenze significative tra i gruppi di controllo e sham per l'espressione della proteina GLUT1 e F-actina.
Espressione di Myh9, SLC7A11 e SLC3A2 nel tessuto miocardico di ratto di MIRI
L'analisi immunoistochimica è stata impiegata per la prima volta per valutare i livelli di espressione dei DRG nel modello MIRI di ratto. I risultati hanno mostrato che, rispetto ai gruppi di controllo e sham, il gruppo modello aveva livelli di espressione significativamente aumentati di Myh9 (Figura 4A, B, p < 0,01) e SLC3A2 (Figura 4E, F, p < 0,01). Al contrario, l'espressione di SLC7A11 è risultata significativamente ridotta (Figura 4C, D, p < 0,01). Di conseguenza, i risultati del western blotting hanno mostrato che, rispetto ai gruppi di controllo e sham, il gruppo modello aveva livelli di espressione significativamente aumentati di Myh9 (Figura 5A, B, p < 0,01) e SLC3A2 (Figura 5A, D, p < 0,01) mentre l'espressione di SLC7A11 (Figura 5A, C, p < 0,01) era significativamente ridotta.

Figura 1: Screening di geni correlati alla disulfideptosi in MIRI. (A) Heatmap di 1233 geni per l'analisi dell'espressione differenziale su campioni MIRI e sham. (B) Diagramma di Venn di DEG e DRG. C'erano 15 geni comuni tra i 1233 DEG e i 99 DRG, come mostrato nella scatola. (C) Grafici vulcanici dei DEG nel set di dati GSE214122. I punti rossi e verdi sul grafico, rispettivamente, indicano geni sovraregolati e sottoregolati. (D) La rete PPI dei 12 DRG hub è stata costruita utilizzando il database STRING. DEG, geni differenzialmente espressi; DRGs, geni correlati alla disulfideptosi. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 2: MIRI promuove il danno alle cellule miocardiche. (A) Immagini rappresentative delle lesioni miocardiche valutate mediante colorazione H&E (barra della scala = 50 μm). (B) Apoptosi dei cardiomiociti rilevata dal test TUNEL. Il gruppo modello ha mostrato un aumento significativo delle cellule apoptotiche marcate con fluorescenza rossa all'interno dei tessuti miocardici di ratto (barra della scala = 50 μm). (C) I risultati statistici del tasso di apoptosi analizzando l'intensità del segnale di fluorescenza rossa utilizzando il software Image J. **p < 0,01, il gruppo modello vs. il gruppo sham, n=3. I dati sono espressi come media ± deviazione standard. Clicca qui per visualizzare una versione più grande di questa figura.
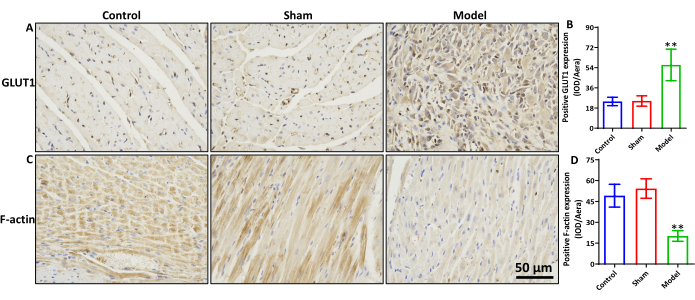
Figura 3: Espressione di F-actina e GLUT1 nel tessuto miocardico di MIRI di ratto. (A) colorazione IHC di GLUT1 e (B) risultati statistici della sua espressione proteica. (C) colorazione IHC della F-actina e (D) risultati statistici della sua espressione proteica. Barra della scala = 50 μm. **P < 0,01, il gruppo del modello contro il gruppo fittizio, n=6. I dati sono espressi come media ± deviazione standard. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 4: Analisi della colorazione IHC dell'espressione delle proteine correlate alla disulfideptosi Myh9, SLC7A11 e SLC3A2 nel tessuto miocardico di MIRI di ratto. (A) Colorazione IHC di Myh9 e (B) risultati statistici della sua espressione proteica. (C) colorazione IHC di SLC7A11 e (D) risultati statistici della sua espressione proteica. (E) colorazione IHC di SLC3A2 e (F) risultati statistici della sua espressione proteica. Barra della scala = 50 μm. **P < 0,01, il gruppo del modello rispetto al gruppo fittizio, n=3. I dati sono espressi come media ± deviazione standard. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 5: Analisi Western blot dell'espressione delle proteine correlate alla disulfideptosi Myh9, SLC7A11 e SLC3A2 nel tessuto miocardico di MIRI di ratto. (A) Bande proteiche rappresentative. Risultati statistici dell'espressione delle proteine (B) Myh9, (C) SLC7A11 e (D) SLC3A2. **p < 0,01, il gruppo modello vs. il gruppo sham, n=3. I valori della scala di grigi delle proteine sono stati analizzati utilizzando il software Image J. I dati sono espressi come media ± deviazione standard. Clicca qui per visualizzare una versione più grande di questa figura.
Discussione
La disulfideptosi è strettamente associata al citoscheletro di actina, una struttura cellulare critica essenziale per mantenere la forma e la vitalità delle cellule. Composto da filamenti di actina, il citoscheletro di actina conferisce forma e struttura cellulare complessive. La F-actina funge da marcatore per il citoscheletro cellulare e, in condizioni di carenza di glucosio, i legami disolfuro aumentano in modo significativo, portando alla sottoregolazione della F-actina. Questo fenomeno colpisce principalmente i processi e le vie legate al citoscheletro di actina e all'adesione cellulare10,11.
Nel contesto del metabolismo del glucosio, i trasportatori del glucosio (GLUT) svolgono un ruolo fondamentale nel mantenimento dell'equilibrio dinamico del glucosio. GLUT1, uno dei primi membri scoperti della famiglia GLUT, è una proteina correlata all'assorbimento del glucosio con un'ampia distribuzione tissutale. È espresso principalmente nei globuli rossi12 e nelle cellule endoteliali della barriera emato-encefalica13, dove partecipa al trasporto transmembrana del glucosio. GLUT1 può essere strettamente correlato allo sviluppo di malattie associate a un metabolismo anomalo del glucosio. Nei modelli di carenza di glucosio, l'espressione di GLUT1 è sovraregolata, promuovendo la morte delle cellule tumorali. Nel modello MIRI, l'upregolazione dell'espressione di GLUT1 peggiora il MIRI, ma si osservano miglioramenti quando GLUT1 è sottoregolato, rendendolo un bersaglio chiave in caso di carenza di glucosio14. L'immunoistochimica ha rivelato un significativo aumento intracellulare della proteina trasportatrice del glucosio GLUT1 nel modello MIRI rispetto ai gruppi di controllo e sham. Allo stesso tempo, la F-actina è risultata significativamente ridotta a livello intracellulare rispetto ai gruppi di controllo e sham. Ciò suggerisce che il collasso del citoscheletro di actina in condizioni di carenza di glucosio può contribuire al peggioramento della MIRI, rendendo il collasso del citoscheletro di actina il meccanismo primario della disulfideptosi. Pertanto, ipotizziamo che l'upregolazione di GLUT1 nel modello MIRI induca disulfideptosi, portando a un danno da ischemia-riperfusione aggravato.
Il gene Myh9 codifica per la catena pesante della miosina IIA non muscolare, una miosina citoplasmatica ampiamente espressa coinvolta in vari processi che richiedono la generazione di forze meccaniche intracellulari e il riposizionamento citoscheletrico dell'actina15. I pazienti con trombocitopenia congenita dovuta a mutazioni del gene Myh9 sono inclini alla formazione di trombi e all'infarto miocardico acuto16. Pertanto, Myh9 può essere un fattore predittivo per l'AMI o la MIRI. SLC7A11 e SLC3A2 sono proteine che aiutano le cellule a ottenere cisteina e glutatione, essenziali per mantenere la salute e l'equilibrio cellulare. SLC7A11 può mitigare la perossidazione lipidica e lo stress ossidativo per svolgere un ruolo protettivo in MIRI17. Il nostro studio ha esplorato le potenziali funzioni biologiche e il significato terapeutico dei geni correlati alla disulfideptosi attraverso il database GEO. Abbiamo trovato una sovrapposizione limitata tra i geni correlati alla MIRI e alla disulfideptosi, con un totale di 12 geni sovrapposti. Attraverso l'analisi del valore P e del Log2Fold Change, abbiamo identificato quattro geni che erano più strettamente correlati nel modello MIRI. I risultati hanno indicato una significativa sovraregolazione dell'espressione di Myh9 e una significativa sottoregolazione dell'espressione di SLC7A11 nel gruppo MIRI. Tuttavia, non è stata osservata alcuna differenza statisticamente significativa nell'espressione di ACTB. I risultati sperimentali hanno confermato che Myh9 e SLC7A11 sono coinvolti nella MIRI e i loro meccanismi possono essere strettamente correlati alla disulfideptosi mediata dalla deplezione di NADPH. Questi risultati forniscono indizi importanti per identificare nuovi fattori predittivi e strategie di trattamento personalizzate per la MIRI (Figura 5).
In sintesi, l'identificazione e la caratterizzazione dei meccanismi di morte cellulare non solo promuovono la nostra comprensione fondamentale dell'omeostasi cellulare, ma evidenziano anche il ruolo significativo svolto dalla disulfideptosi nella MIRI. Il difetto di questo metodo è la mancanza di esperimenti critici di validazione per la disulfideptosi. Attualmente, è possibile rilevare e identificare la disulfideptosi utilizzando la sonda fluorescente DCP-Bio1, che si lega ai legami disolfuro, marcando i disolfuri intracellulari ed extracellulari e facilitando l'osservazione dei disolfuri nelle cellule o nei tessuti18. Tali nuove sonde possono essere impiegate per studiare le dinamiche spazio-temporali tra disulfideptosi e MIRI.
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Questa ricerca è stata sostenuta dall'Ufficio provinciale di scienza e tecnologia del Guizhou (Qiankehe [2022]-583) e dall'Amministrazione provinciale di medicina tradizionale cinese del Guizhou (QZYY-2016-019).
Materiali
| Name | Company | Catalog Number | Comments |
| 75% alcohol | Hunan Tongruijian Pharmaceutical Co. Ltd., Hunan, China | 85026 | |
| 6-0 nylon suture | Shanghai Pudong Jinhuan Medical Supplies Co. Ltd., Shanghai, China | CS002 | |
| BCA protein quantification kit | Beyotime Biotechnology, Shanghai, China | P0011 | |
| Bull serum albumin | Beyotime Biotechnology, Shanghai, China | ST2254 | |
| Centrifuge | Hunan Kaida Scientific Instrument Co. Ltd., Hunan, China | KH19A | |
| DAB horseradish peroxidase color development kit | Beyotime Biotechnology, Shanghai, China | P0203 | |
| DAPI staining solution | Beyotime Biotechnology, Shanghai, China | C1006 | |
| DESeq2 package | Version 4.1 | ||
| Electric razor | Kelmerpp | 235376 | |
| Enhanced endogenous peroxidase blocking buffer | Beyotime Biotechnology, Shanghai, China | P0100B | |
| Filter paper | Nanjing Keruicai Equipment Co., Ltd., Nanjing, China | 1.00049E+11 | |
| Fluorescence microscope | Nikon | ECLIPSE Ci | |
| GEO database | https://www.ncbi.nlm.nih.gov/geo/ | ||
| Glass grinder | Shanghai Leigu Instrument Co. Ltd., Shanghai, China | B-013002 | |
| GraphPad Prism | GraphPad Software | V8.0 | |
| Hematoxylin and eosin staining kit | Beyotime Biotechnology, Shanghai, China | C0105M | |
| Image J software | National Institutes of Health, Bethesda, USA | v1.8.0 | |
| Improved citrate antigen retrieval solution (50X) | Beyotime Biotechnology, Shanghai, China | P0083 | |
| Iodophor | Folca, Shenzhen, China | 1.00077E+11 | |
| Optical microscope | Nikon | ECLIPSE Ci | |
| Phosphate buffer solution | Beyotime Biotechnology, Shanghai, China | C0221A | |
| Primary antibodies against GLUT1 | Proteintech Group, Inc, Wuhan, China | 21829-1-AP | |
| Primary antibodies against MYH9 | Proteintech Group, Inc, Wuhan, China | 11128-1-AP | |
| Primary antibodies against SLC3A2 | Wuhan Lingsi Biotechnology Co., Ltd., Wuhan, China | LJS-D-7468 | |
| Primary antibodies against SLC7A11 | Proteintech Group, Inc, Wuhan, China | 26864-1-AP | |
| Protein visualization instrument | Thermo Fisher Scientific Inc. | iBright CL750 | |
| Rat board | Zhengzhou Haopai Biotechnology Co. Ltd., Zhengzhou, China | JPB-E | |
| Scalpel | Shanghai Lianhui Medical Supplies Co., Ltd., Shanghai, China | 1.00471E+13 | |
| Secondary antibody | Wuhan Boster Biological Technology, Ltd., Wuhan, China | BA1054 | |
| STRING database | https://cn.string-db.org/ | Version 12.0 | |
| Triton X-100 | Beyotime Biotechnology, Shanghai, China | ST1722 | |
| TUNEL apoptosis detection kit | Proteintech Group, Inc., Wuhan, China | PF00009 |
Riferimenti
- Lu, Z., et al. Tripartite motif 38 attenuates cardiac fibrosis after myocardial infarction by suppressing TAK1 activation via TAB2/3 degradation. iScience. 25 (8), 104780 (2022).
- Tu, Q., et al. EGCG decreases myocardial infarction in both I/R and MIRI rats through reducing intracellular Ca2+ and increasing TnT levels in cardiomyocytes. Adv Clin Exp Med. 30 (6), 607-616 (2021).
- Li, Q., et al. Histone deacetylase HDAC4 participates in the pathological process of myocardial ischemia-reperfusion injury via MEKK1/JNK pathway by binding to miR-206. Cell Death Dis. 7 (1), 240 (2021).
- Liu, X., et al. Actin cytoskeleton vulnerability to disulfide stress mediates disulfidptosis. Nat Cell Biol. 25 (3), 404-414 (2023).
- Yang, L., et al. Analysis of risk genes associated with disulfidptosis-related myocardial ischemia-reperfusion injury. J Chongqing Med Univ. 49 (4), 401-408 (2024).
- Xiao, G., et al. CXCR1 and its downstream NF-κB inflammation signaling pathway as a key target of Guanxinning injection for myocardial ischemia/reperfusion injury. Front Immunol. 13, 1007341 (2022).
- Zhu, E. Y., et al. Screening of immune-related secretory proteins linking chronic kidney disease with calcific aortic valve disease based on comprehensive bioinformatics analysis and machine learning. J Transl Med. 21, 359 (2023).
- Shi, D. C., et al. Potential of Dendrobium officinale oligosaccharides to alleviate chronic colitis by modulating inflammation and gut microbiota. FoodMedHomo. 2, 9420077 (2025).
- Ning, E. J., et al. Artemisia argyi polysaccharide alleviates intestinal inflammation and intestinal flora dysbiosis in lipopolysaccharide-treated mice. FoodMedHomo. 1 (1), 9420008 (2024).
- Zhou, H., Wang, J., Zhu, P., Hu, S., Ren, J. Ripk3 regulates cardiac microvascular reperfusion injury: The role of IP3R-dependent calcium overload, XO-mediated oxidative stress and F-action/filopodia based cellular migration. Cell Signal. 45, 12-22 (2018).
- Antonipillai, J., Rigby, S., Bassler, N., Peter, K., Bernard, O. Pharmacological inhibition of LIM kinase pathway impairs platelets functionality and facilitates thrombolysis. Exp Cell Res. 382 (2), 111458 (2019).
- Kaumeyer, B. A., et al. GLUT1 immunohistochemistry is a highly sensitive and relatively specific marker for erythroid lineage in benign and malignant hematopoietic tissues. Am J Clin Pathol. 158 (2), 228-234 (2022).
- Patching, S. G. Glucose transporters at the blood-brain barrier: Function, regulation and gateways for drug delivery. Mol Neurobiol. 54 (2), 1046-1077 (2017).
- Zhou, H., et al. 3-iodothyronamine inhibits apoptosis induced by myocardial ischemia reperfusion via the Akt/FoxO1 signaling pathway. Ann Trans Med. 10 (4), 168 (2022).
- Pecci, A., et al. MYH9: Structure, functions and role of non-muscle myosin IIA in human disease. Gene. 664, 152-167 (2018).
- Girolami, A., et al. Occurrence of thrombosis in congenital thrombocytopenic disorders: A critical annotation of the literature. Blood Coagulat Fibrino. 24 (1), 18-22 (2013).
- Li, P., et al. SLC7A11-associated ferroptosis in acute injury diseases: Mechanisms and strategies. Eur Rev Med Pharmaco Sci. 27 (10), 4386-4398 (2023).
- Wu, X., et al. Dihydrotanshinone I preconditions myocardium against ischemic injury via PKM2 glutathionylation sensitive to ROS. Acta Pharma Sinica B. 13 (1), 113-127 (2023).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon