Method Article
Plug-in Micro-Manager de código aberto para imagens de visualização ao vivo de dipolos fluorescentes
Neste Artigo
Resumo
Desenvolvemos um plug-in Micro-Manager de código aberto, que permite a observação ao vivo de dipolos fluorescentes em um microscópio de iluminação estruturada. O plug-in suporta a observação da orientação do dipolo 2D e 3D.
Resumo
A microscopia de polarização de fluorescência (FPM) pode obter imagens da posição e orientação do dipolo dos fluoróforos. Apesar das conquistas da microscopia de polarização de fluorescência de super-resolução, sua dependência da pós-aquisição dificulta a observação em tempo real. A microscopia de iluminação estruturada polarizada (pSIM) oferece imagens de super-resolução de dipolos fluorescentes com velocidade de imagem rápida e é adequada para aplicações de células vivas. Desenvolvemos uma implementação de código aberto para reconstrução em tempo real de imagens de polarização e exibição dos dipolos fluorescentes. Além disso, estendemos o método para obter o mapeamento de orientação 3D (3DOM), ampliando sua utilidade para estudos biológicos complexos. Além disso, apresentamos uma introdução completa à extensão de um microscópio SIM existente em imagens de polarização e fornecemos um guia de configuração detalhado do Micro-Manager 2.0 para controlar o microscópio, permitindo a visualização em tempo real da imagem polarizada. Além disso, fornecemos o código MATLAB para reconstrução completa, abrangendo pSIM e 3DOM. Este guia abrangente tem como objetivo ajudar os iniciantes a dominar rapidamente e iniciar facilmente as operações.
Introdução
A microscopia de polarização de fluorescência (FPM) surgiu como uma técnica poderosa para obter imagens simultâneas da posição e da orientação do dipolo dos fluoróforos, oferecendo insights profundos sobre imagens biológicas 1,2. Ao facilitar a observação da direção das orientações das biomoléculas, o FPM revela o intrincado arranjo de macromoléculas como actina 3,4,5, microtúbulo5, septina6, filamento de DNA 7-9, complexo de poros nucleares10 e proteínas de membrana11. Seus recursos de alta velocidade, não invasivos e compatíveis com células vivas permitem o rastreamento da dinâmica de rotação molecular com alta resolução temporal11,12. Quando integrado com sondas de bioforça, o FPM não apenas mapeia as magnitudes de força na resolução subcelular, mas também mede as direções das forças, avançando assim nossa compreensão dos processos biomecânicos, revelando a direção das forças13.
Nas últimas décadas, a microscopia de polarização de fluorescência de super-resolução passou por uma rápida evolução. Um avanço notável neste campo é a microscopia de localização de orientação de molécula única, também denominada SMOLM, que pode localizar a posição e a orientação dos fluoróforos, permitindo assim a localização multidimensional. A medição de polarização no SMOLM pode ser realizada usando modulação de excitação de polarização7, detecção polarizada multicanal3 ou função de propagação de ponto sensível à polarização projetada (PSF) 14 . Apesar do SMOLM atingir resolução espacial da ordem de dezenas de nanômetros e medir a polarização de moléculas individuais, ele sofre com o tempo de imagem prolongado. Isso se deve aos ciclos repetitivos de piscar e localização de fluoróforos, que representam um desafio para imagens de taxa de vídeo e aplicações de células vivas.
Em contraste, a microscopia de iluminação estruturada polarizada (pSIM) oferece uma resolução espacial de aproximadamente até 100 nm, juntamente com a aquisição de informações de polarização com o mesmo conjunto de dados SIM. Notavelmente, o pSIM pode atingir velocidades de imagem de taxa de vídeo e é altamente compatível com imagens de células vivas, sem requisitos rigorosos em moléculas fluorescentes. Recentemente, o pSIM revelou com sucesso a estrutura do anel de actina no esqueleto periódico associado à membrana (MPS) 5 e permitiu o mapeamento de super-resolução de forças biológicas13 .
No entanto, o pSIM requer reconstrução de imagem pós-aquisição, o que impede a visualização em tempo real dos resultados da polarização. Esse atraso dificulta a observação imediata do fenômeno biológico, impedindo que os pesquisadores capturem rapidamente os fenômenos biológicos de interesse e façam ajustes em tempo real nas amostras e nas condições de imagem. Para resolver essa limitação, desenvolvemos uma implementação de código aberto que facilita a aquisição de imagens e a reconstrução e exibição em tempo real dos resultados de polarização, com base na plataforma ImageJ e Micro-Manager (https://github.com/KarlZhanghao/live-pol-imaging).
Além disso, embora o pSIM tenha se limitado a fornecer informações de polarização 2D no plano, recentemente estendemos seus recursos para obter mapeamento de orientação 3D usando quase o mesmo equipamento15, denominado mapeamento de orientação 3D (3DOM). Este software de código aberto também fornece o controle, reconstrução e visualização do 3DOM. Os módulos de reconstrução e visualização também são compatíveis com o aplicativo de rastreamento de orientação de molécula única. Todas essas funcionalidades aumentam a utilidade da imagem de polarização em estudos biológicos complexos.
Protocolo
1. Extensão de um microscópio de iluminação estruturada existente para imagens de polarização
- Prepare um microscópio SIM.
NOTA: Assumimos que os leitores têm uma certa base na configuração do microscópio e já possuem um microscópio de luz estruturado. Se você não tiver experiência relevante, consulte este artigo, que fornece uma descrição detalhada de como construir um microscópio SIM16. A unidade de controle de polarização de "HWP+WQP+LCVR" pode ser substituída por uma placa de onda de vórtice de pizza em nosso trabalho pSIM5 ou uma placa de meia onda de pizza17. - Meça as taxas de extinção de polarização de três direções carregando diferentes padrões no modulador de luz espacial (SLM). Coloque e gire um polarizador logo após a objetiva e use um medidor de potência para medir a potência do laser. Para pSIM, gire a placa de onda para manter a polarização s de três direções com taxa de extinção > 10.
- Para 3DOM, gire o HWP2 para manter a polarização p de seis direções. Substitua a máscara espacial do SIM pela máscara espacial 3DOM para permitir a passagem de feixes de 1 ordem de seis direções (consulte a Figura 1B).
NOTA: a polarização s é necessária no pSIM enquanto a polarização p é necessária no 3DOM. A configuração detalhada está incluída em outro lugar para pSIM5 e 3DOM15.
2. Configuração do Micro-Manager
- Baixe a versão Micro-Manager 2.0 do site oficial, instale o software seguindo as instruções.
- Prepare os drivers de dispositivo e o software correspondentes para se conectar com êxito ao sistema Micro-Manager. Neste sistema de microscópio, use o Micro-Manager para controlar a câmera, o estágio de translação, o laser, a placa DAQ e o microscópio.
NOTA: Os instrumentos suportados estão listados em https://micro-manager.org/Device_Support. Consulte a Tabela de Materiais para obter detalhes sobre os instrumentos a serem conectados. Instale drivers ou software oficial/de suporte e conecte o instrumento ao computador usando um cabo USB. - Adicione os instrumentos ao Micro-Manager usando o Assistente de Configuração de Hardware.
- Abra o software e selecione Nenhum na lista suspensa inicial.
- Selecionar dispositivos | Assistente de configuração de hardware... | Crie uma nova configuração e clique em Avançar para entrar na página de configuração.
- Localize o plug-in Câmera/Laser/Platina/DAQ/microscópio na lista suspensa de hardware e clique em Adicionar | ESTÁ BEM | A seguir.
- Vá para Selecionar dispositivos padrão e escolha a configuração do obturador automático. Defina a câmera padrão, o obturador padrão e o estágio de foco padrão. Clique em Avançar até que Salvar configuração e sair seja alcançado e salve o nome do arquivo de configuração. Clique em Concluir.
- Configurações comuns de modelo
- Salve um modelo para o modo ao vivo e o modo de snap do SIM.
- Defina o tempo de exposição e o modo de disparo da seguinte forma: escolha Grupo '+' em Definições de configuração, nomeie o nome do grupo como 'Modo', selecione Modo de exposição e disparo, clique em OK.
- Escolha Predefinição '+' para adicionar predefinições diferentes para o grupo 'Modo'. Nomeie uma predefinição 'ao vivo' para configurações de exposição do modo ao vivo e outra 'SIM' para o modo SIM. Por exemplo, no modo 'ao vivo', defina o tempo de exposição para 15 ms, enquanto para o modo de snap do SIM, defina 10 ms para o tempo de exposição e o modo de disparo é Padrão (Sobreposição).
NOTA: Um arquivo de configuração (MMConfig_psim_demo.cfg) está incluído em nosso repositório de código, que pode ser carregado diretamente pelo Micro-Manager 2.0.
3. Calibração do sistema com esferas fluorescentes
- Mergulhe as lamínulas em etanol a 75%, lave as lamínulas 3x com água deionizada (ddH2O) e seque-as.
- Vortex as esferas fluorescentes de 100 nm (diluídas a 1:1.000 com solução salina tamponada com fosfato [PBS]) e pipetar a suspensão diretamente nas lamínulas. Incube por 5-10 min em um ambiente escuro e, em seguida, lave suavemente com PBS.
- Adicione 20 μL de meio de montagem na lâmina do microscópio e posicione a lamínula sobre ela, certificando-se de evitar bolhas. Mantenha a temperatura a 4 °C e mantenha a lâmina no escuro, para que a lâmina com esferas fluorescentes de 100 nm esteja preparada para a calibração do sistema.
- Para pSIM, adquira três imagens das três fases diferentes de esferas fluorescentes. Um código MATLAB personalizado (Bead_calib.m) pega as imagens como entrada e gera duas imagens de calibração (calib1.tif, calib2.tif) para análise posterior.
NOTA: Conforme demonstrado anteriormente5, o experimento pSIM pode ser realizado na maioria dos sistemas comerciais. Em microscópios SIM construídos em casa ou comerciais, a calibração do sistema é necessária para eliminar o erro de medição de dipolos fluorescentes, causado pela iluminação não uniforme durante três direções de padrão. O experimento de calibração assume a polarização isotrópica de esferas fluorescentes e requer apenas as imagens brutas durante a aquisição do SIM. O procedimento de calibração detalhado está incluído em nosso trabalho original5. A saída de duas imagens de calibração calib1.tif e calib2.tif é necessária para a reconstrução do pSIM, que indica a energia de iluminação pixel a pixel da direção 2 do padrão; 3 refere-se à direção do padrão 1.
4. Preparação da amostra: actina em células fixas
- Mergulhe as lamínulas em etanol a 75%, lave as lamínulas 3x com ddH2O. Coloque as lamínulas em uma placa de cultura de células de seis poços.
- Cultura de células U2OS (linhagem celular ATCC HTB-96) em Meio de Eagle Modificado de Dulbecco (DMEM) suplementado com 10% (v/v) de Soro Fetal Bovino (FBS) a 37 °C e 5% de CO2 em lamínulas até atingirem aproximadamente 75% de confluência.
- Lave suavemente as células 3x com PBS. Para fixação celular, aplique paraformaldeído a 4% por 15 min em temperatura ambiente. Após a fixação, lave as células 3x novamente com PBS.
- Para aumentar a permeabilidade da membrana, trate as células com 0,1% de Triton X-100 em PBS por 5 min e, em seguida, lave as células 3x com PBS.
- Dissolva a faloidina fluorescente com 150 μL de DMSO para solução estoque (66μM). Prepare a solução de trabalho do corante de faloidina diluindo 5 μL da solução estoque em 995 μL de PBS. Incube as células no escuro com a solução de trabalho do corante Faloidina à temperatura ambiente por ~ 1 h. Lave as células por 3 x 3 min com PBS.
- Adicione 20 μL de meio de montagem na lâmina do microscópio. Coloque cuidadosamente a lamínula na lâmina sem bolhas. Guarde a lâmina a 4 °C e guarde-a em local escuro.
5. Preparação da amostra: λ-DNA in vitro
- Dissolva 0,5 g de Polimetilmetacrilato em 10 mL de materiais de base de prótese. Após a dissolução completa, aplique uniformemente a solução na superfície da lâmina e espere que ela evapore para formar um filme.
- Diluir 0,3 μL de λ-DNA e 32 μL de 1.000x SYTOX Orange Nucleic Acid Stain em 968 μL de PBS. Adicione 1 μL da solução preparada à lâmina e deixe secar.
- Sele a lamínula na lâmina com o suporte para proteger e preservar a amostra. Armazene a lâmina a 4 °C em um ambiente escuro.
6. Exames de imagem
- Coloque a lamínula no suporte de amostra e ajuste ao plano focal no modo ao vivo. Selecione um ROI de 512 x 512 pixels e ajuste o tempo de exposição e a intensidade do laser.
- Abra o software de controle independente do modulador de luz espacial e escolha o caminho de sequência de padrão de iluminação apropriado para pSIM / 3DOM ou modo ao vivo.
NOTA: Para pSIM, o padrão de iluminação inclui três ângulos de polarização diferentes e três fases diferentes. Para 3DOM, o padrão de iluminação inclui seis ângulos de polarização diferentes. - Selecione o modo de captura do SIM , selecione Multi-D Acq., escolha Pontos de tempo e defina a contagem (9 ou 6 imagens para pSIM ou 3DOM) e o intervalo (0 ms) na janela aberta. Clique em Adquirir!
- Clique em TriggerSLM.bsh (um arquivo de configuração está incluído em nosso repositório de código), o cartão de aquisição MyDaq ativa o modulador de luz espacial para tirar as fotos.
7. Plug-in de visualização ao vivo de pSIM e 3DOM
- Configure a FakeCamera e o caminho de acesso à imagem: carregue o script (MMConfig_FakeCam_demo.cfg) clicando em Dispositivos | Carregar configuração de hardware.... Configurar o diretório local de imagens em Dispositivos | Navegador de propriedades de dispositivos... | Máscara de caminho da câmera (Figura 2A-C).
NOTA: A parte de aquisição do script depende da configuração do hardware, por isso fornecemos uma versão (MMConfig_FakeCam_demo.cfg) usando o dispositivo FakeCamera para ler imagens locais no disco rígido para demonstração. Os usuários podem fazer alterações em nossa versão. Lembre-se de alterar o caminho de leitura da imagem (máscara Camera-Path). - Defina o Painel de Acesso Rápido clicando em Ferramentas | Painel de Acesso Rápido | Crie um novo painel. Clique no ícone Configurações no canto inferior esquerdo, droga 'Run Script' para a parte superior, que pode carregar o 'psim.bsh' e '3DOM.bsh' (Figura 2D) .
NOTA: O sistema Quick Access Panel permite que os usuários criem janelas personalizadas e acessem facilmente os controles no Micro-Manager, que são necessários com mais frequência. 'Ao vivo', 'Snap' ou outros canais também podem ser adicionados, conforme mostrado na Figura 2D. - Tomando o pSIM como exemplo, execute a reconstrução da imagem de polarização em tempo real clicando em 'psim.bsh'; observe as imagens reconstruídas em tempo real com resultados de polarização (Figura 2E). Lembre-se de alterar o caminho das imagens de calibração no arquivo 'psim.bsh' (calibPath = " caminho para o arquivo de imagens de calibração").
NOTA: O plug-in live-view é um script beanshell (psim.bsh e 3DOM.bsh), que pode ser executado no Painel de Scripts. O plug-in adquire nove imagens para pSIM ou seis imagens para 3DOM e calcula os resultados do mapeamento de orientação com base nas imagens de campo amplo. As imagens de mapeamento de orientação são exibidas para fornecer informações de polarização em tempo real, o que facilita a inspeção imediata, apesar de sua baixa resolução.
8. Análise de dados com reconstrução de super-resolução
- Instale o MATLAB R2019b a partir do website (consulte a Tabela de Materiais).
- Abra o software; para a reconstrução do pSIM, obtenha-se nove imagens, consistindo em três fases diferentes para cada um dos três ângulos de orientação. Certifique-se de que os nomes das nove imagens sejam de '1.tif' a '9.tif', coloque-os no arquivo chamado 'Input' e execute o programa chamado 'PSIM.m'. Observe a imagem de campo amplo reconstruída, a imagem SIM e a imagem pSIM com a roda de cores.
NOTA: A reconstrução do pSIM precisa de duas etapas: uma etapa do SIM e uma etapa de PM. Os dados são analisados por um programa MATLAB personalizado para facilitar a depuração. Obtenha o código de reconstrução da pasta 'reconsr' no GitHub. - Para 3DOM, capture seis figuras diferentes de direções de polarização. Mescle as seis imagens, nomeie o arquivo resultante como 'Raw_data.tif' e execute o programa chamado 'Recon_3DOM.m'. Observe a imagem de campo amplo reconstruída e a imagem 3DOM com resultados azimutais e os resultados do ângulo polar.
NOTA: Durante o processo de reconstrução, pode ocorrer um erro'--'função ou variável 'bfopen' não é reconhecida'. 'bfopen' é uma função na biblioteca de bio-formatos e é comumente usada para abrir e ler arquivos de imagem. Insira addpath ('path_to_bioformats_toolbox') na janela de comando do MATLAB. Substitua 'path_to_bioformats_toolbox' pelo caminho real da pasta da biblioteca. Também adicionamos a dependência Bio-Formats ao repositório GitHub.
Resultados
O método pSIM pode ser realizado em microscópios SIM com base na interferência usando feixes de laser polarizados s. A interferência de polarização s é o tipo de SIM mais amplamente utilizado e gera faixas de iluminação de alto contraste. O protótipo acadêmico de uma configuração de microscópio está incluído no trabalho original do pSIM5. Brevemente introduzido na Figura 1, um modulador de luz espacial (SLM) gera a ordem ±1 dos feixes difrativos e uma placa de meia onda de pizza mantém a polarização s de três direções. Enquanto outras alternativas de SIM podem usar grades para gerar feixes difrativos ou usar outros métodos de modulação de polarização para manter a polarização s, esses microscópios também podem ser estendidos para imagens de polarização.
Os microscópios 3DOM utilizam um sistema de imagem óptica semelhante (Figura 1A) com duas diferenças. Primeiro, o 3DOM não requer interferência de dois feixes de laser. Em vez disso, ele usa apenas os feixes +1 para controlar a polarização de excitação. Conforme demonstrado no trabalho original15, o 3DOM requer um mínimo de seis direções para calcular a orientação do dipolo 3D. Portanto, a máscara espacial 3DOM contém seis direções, cada uma com um orifício (consulte a Figura 1B). Outra diferença é que o 3DOM usa feixes de laser polarizados p, que podem ser alcançados girando o HWP2. O SLM gera luz difratada em vários ângulos oblíquos para alterar a porção da polarização p.
No sistema pSIM, a luz de saída é modulada em luz polarizada s, girando o ângulo das placas de meia onda. A máscara do pSIM é simétrica, o que permite que ±1 luz de ordem passe simultaneamente. Por outro lado, no 3DOM, ajustando o ângulo da placa de meia onda, o laser é transformado em luz polarizada p. A máscara do 3DOM não é assimétrica e só permite a passagem de +1 luz de ordem. Ao ajustar a frequência da faixa do SLM, ele altera o ângulo de difração dos feixes de laser. Assim, a porção de polarização fora do plano é modulada.
Ao analisar dois dipolos fluorescentes adjacentes que compartilham o mesmo ângulo azimutal, mas ângulos polares diferentes, tanto o pSIM quanto o 3DOM podem distinguir dipolos próximos dentro do limite de difração. Além disso, o 3DOM pode efetivamente distinguir diferentes dipolos com orientação 3D no domínio da polarização (Figura 1C,D). Isso ocorre porque a tecnologia 3DOM pode obter mais informações espaciais alterando o estado de polarização da luz excitada para obter uma medição precisa da orientação do dipolo no espaço 3D. Em contraste, a tecnologia pSIM depende principalmente de informações de polarização 2D e não pode aproveitar ao máximo as características de orientação 3D; portanto, não fornece informações sobre a orientação dos dipolos na direção vertical.
Como os resultados do mapeamento orientacional requerem pós-processamento, desenvolvemos e apresentamos aqui o 'plug-in Live-view' ('psim.bsh' e '3DOM.bsh') para o Micro-Manager 2.0. Para permitir que os usuários de computador sem uma câmera real conectada aprendam a usar o plug-in, primeiro configuramos o Micro-Manager com FakeCamera.In Figura 2, detalhamos o processo operacional do pSIM para obter efeitos de polarização em tempo real e as etapas operacionais do 3DOM são semelhantes. Na Figura 2A, a interface de software do Micro-Manager 2.0 é exibida, juntamente com as etapas de configuração necessárias para configurar nosso plug-in para operação. Na Figura 2B, mostramos a configuração de hardware usando dispositivos de demonstração de FakeCamera e DStage. A configuração de hardware do FakeCamera é capaz de simular vários comportamentos de uma câmera real, como exposição, foco e balanço de branco, permitindo que os desenvolvedores realizem o desenvolvimento e teste de software sem a necessidade de uma câmera real. Os usuários devem alterar o caminho de leitura da imagem com base nas circunstâncias reais, conforme mostrado na Figura 2C. A Figura 2D exibe um Painel de Acesso Rápido, que pode conter atalhos para uma série de funções ou configurações comumente usadas, permitindo que os usuários acessem e usem essas funções mais rapidamente. Os plug-ins de visualização em tempo real, 'psim.bsh' e '3DOM.bsh', podem ser carregados a partir deste painel. As etapas operacionais específicas podem ser encontradas em detalhes na seção 7 do protocolo. Os scripts 'psim.bsh' e '3DOM.bsh' são usados para gerar instantaneamente as imagens de mapeamento de orientação. Este plugin 'psim.bsh' permite a visualização imediata dos efeitos de polarização durante o estágio de captura de imagem pSIM. A Figura 2E ilustra um resultado típico da imagem de polarização do pSIM. A roda de cores indica a relação entre a orientação do dipolo e a pseudocor, o que é útil para os pesquisadores tomarem decisões imediatas sobre quais imagens capturar com base nos dados de polarização exibidos.
As células U2OS foram marcadas com faloidina e o processo de reconstrução da imagem do pSIM foi realizado usando MATLAB. Nove fotos foram capturadas de três estados de polarização diferentes e três fases diferentes. As setas duplas verdes marcam a direção da luz polarizada na Figura 3A. A partir da intensidade clara e escura da imagem, a orientação de polarização dos dipolos fluorescentes pode ser determinada.
Na Figura 3B, a figura da esquerda apresenta os resultados da super-resolução do SIM, enquanto a figura da direita mostra os resultados do pSIM. Notavelmente, o pSIM atinge a mesma capacidade de resolução espacial que o SIM e oferece o dobro da resolução espacial em comparação com a microscopia de campo amplo. Ao mapear cores específicas para direções distintas do filamento de actina, a roda de cores fornece uma representação visual que nos permite interpretar o alinhamento dos dipolos fluorescentes dentro dos filamentos de actina da célula. A cor do filamento de actina mostra que os dipolos estão aproximadamente alinhados com a orientação do filamento.
A Figura 4A mostra a distribuição da intensidade de emissão sob modulação de excitação de polarização; 3DOM pode produzir seis modulações de polarização diferentes. Nesta configuração, a luz excitada da molécula fluorescente é modulada alterando o estado de polarização, afetando assim a intensidade de emissão da molécula. Isso permite que os pesquisadores infiram a orientação das moléculas medindo a intensidade da fluorescência em diferentes estados de polarização. A Figura 4B mostra a intensidade de fluorescência do λ-DNA sob diferentes estados de polarização na Figura 4A. As moléculas de λ-DNA exibem diferentes intensidades de fluorescência em diferentes estados de polarização, o que indica que a orientação do dipolo de fluorescência e do ambiente tem uma influência significativa na intensidade do sinal de fluorescência. A Figura 4C exibe imagens de super-resolução do λ-DNA obtidas pelo 3DOM, mostrando resultados azimutais e resultados de ângulo polar. Nossas observações revelam que as moléculas do corante estavam alinhadas quase perpendicularmente às fitas de DNA. Como o eixo do DNA se dobra, a orientação média das moléculas do corante girará de forma correspondente. Os resultados de nossas medições de orientação validam a inserção de fluoróforos STOYOX Orange entre bases de DNA adjacentes, e o alinhamento do momento de dipolo de absorção é perpendicular ao eixo do DNA.
No 3DOM, uma luz de excitação inclinada é, portanto, necessária para obter as informações do eixo z da molécula alvo. Em nosso sistema, o SLM é usado para realizar o ajuste da luz de iluminação. Combinado com as características do SLM, se o SLM for carregado com um padrão listrado, a luz que sai do SLM será composta por 0 e ±1 ordens de luz (e as ordens +1 e -1 de luz são simétricas). Portanto, usamos a luz de ordem +1 como luz de excitação e utilizamos a máscara assimétrica para filtrar a luz de ordens 0 e -1. Além disso, a luz de excitação oblíqua pode ser realizada não apenas usando SLM, mas também usando um galvanômetro de varredura ou movendo a lente na frente da lente objetiva lateralmente.
O uso de seis estados de polarização de excitação diferentes no 3DOM é ditado por seu princípio15. Ajustando o modelo de imagem direta, o modelo obtido consiste em seis partes. Portanto, não é possível usar menos de seis ajustes dos estados de polarização, e um excesso de seis resultaria em redundância de informações. O princípio específico do 3DOM é mostrado abaixo.
Em coordenadas esféricas, caracterizamos a molécula fluorescente usando o anjo azimutal ρ e o ângulo polar η da seguinte forma:

ρ é o ângulo no sentido horário entre a projeção da molécula no plano x-y e o eixo x, e η é o ângulo entre a molécula e o eixo z.
A luz de excitação p-polarizada é caracterizada como:

Onde α e β representam o ângulo azimutal e o ângulo de incidência oblíquo da luz de excitação, respectivamente. A eficiência de absorção de fótons é  , e assumindo que a emissão de w dipolos no plano focal é capturada dentro do enésimo pixel, então o fóton fluorescente emitido pelo dipolo é denotado como:
, e assumindo que a emissão de w dipolos no plano focal é capturada dentro do enésimo pixel, então o fóton fluorescente emitido pelo dipolo é denotado como:


Onde C representa a irradiância da amostra e  representa o estado de polarização da luz de excitação conforme descrito a seguir:
representa o estado de polarização da luz de excitação conforme descrito a seguir:

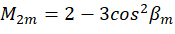

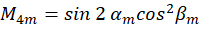
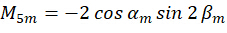
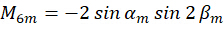
Ao combinar o coeficiente 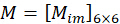 , a orientação
, a orientação 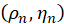 do enésimo pixel pode ser resolvida tomando as imagens
do enésimo pixel pode ser resolvida tomando as imagens  adquiridas como entrada da seguinte forma:
adquiridas como entrada da seguinte forma:
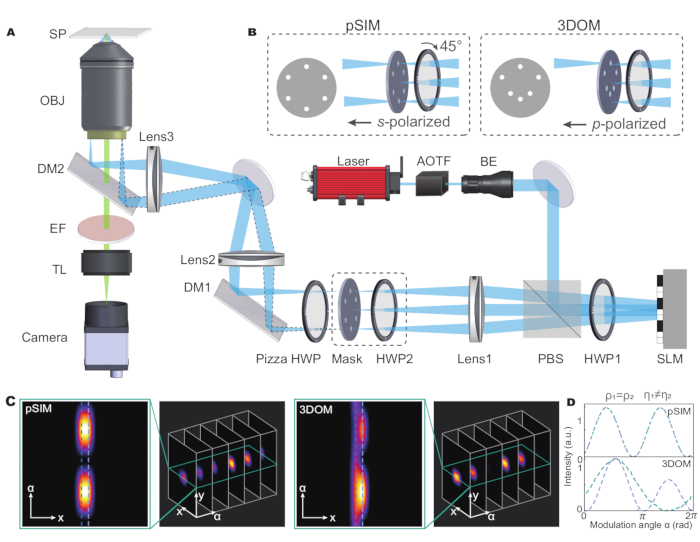
Figura 1: Ilustração esquemática dos microscópios pSIM e 3DOM. (A) Configuração experimental para pSIM e 3DOM. (B)As principais diferenças entre os sistemas pSIM e 3DOM são a máscara e o HWP2. A máscara do pSIM garante que a luz de ordem ±1 passe simultaneamente e a luz de saída seja polarizada em s girando o HWP2. A máscara do 3DOM passa apenas pela luz de ordem +1 e a luz de saída é polarizada em p. (C, D) Comparação das variações da intensidade de fluorescência de emissão entre pSIM e 3DOM. Abreviaturas: pSIM = microscopia de iluminação estruturada polarizada; 3DOM = mapeamento de orientação 3D; AOTF = filtro sintonizável acústico-óptico; BE = expansor de feixe; PBS = divisor de feixe de polarização; HWP = placa de meia onda; SLM = modulador de luz espacial; DM = espelho dicróico; OBJ = objetivo; SP = plano de amostra; EF = filtro de emissão; TL = lente de tubo. Esse valor foi modificado de Zhanghao et al.5 e Zhong et al.15. Clique aqui para ver uma versão maior desta figura.

Figura 2: Captura de tela do software e resultados representativos do pSIM. (A) A interface do Micro-Manager 2.0. Algumas opções podem ser usadas para configurar arquivos. (B) Configuração do plug-in de visualização ao vivo com FakeCamera e DStage. (C)Interface de seleção de caminho de leitura de imagem (máscara de caminho). (D) Configurando o Painel de Acesso Rápido e carregando 'Executar Script' com 'psim.bsh' e '3DOM.bsh'. (E) A reconstrução da imagem em tempo real demonstra os efeitos de polarização. As orientações do dipolo são pseudocoloridas. A roda de cores indica a relação entre a orientação do dipolo e a pseudocor. Abreviaturas: pSIM = microscopia de iluminação estruturada polarizada; 3DOM = mapeamento de orientação 3D. Clique aqui para ver uma versão maior desta figura.
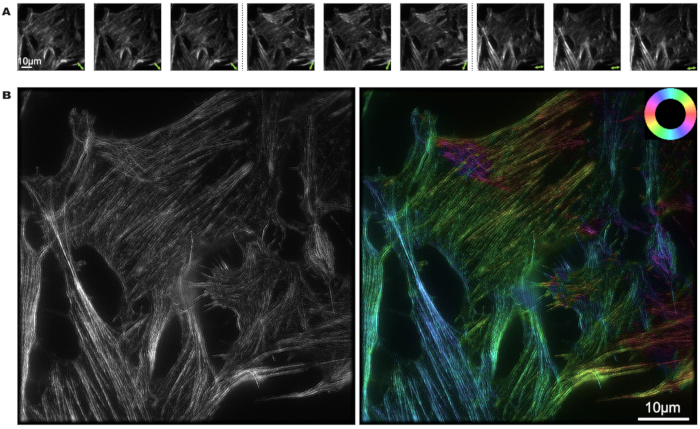
Figura 3: Imagem pSIM de filamentos de actina em célula U2OS marcada com faloidina. (A) Modulação do estado de polarização da luz de excitação 3x em pSIM, capturando três fases distintas para cada estado de polarização. As setas duplas indicam a direção da luz polarizada. (B) Imagem SIM e pSIM de um filamento de actina em A, onde a imagem da esquerda mostra os resultados do SIM e a imagem da direita mostra os resultados do pSIM com ângulo de polarização. A roda de cores posicionada no canto superior direito ilustra a relação entre a pseudocor e a orientação do dipolo. Barras de escala = 10 μm. Abreviatura: pSIM = microscopia de iluminação estruturada polarizada. Clique aqui para ver uma versão maior desta figura.

Figura 4: SYTOX Laranja-marcado λ-DNA Laranja-marcado imageado por 3DOM. (A) Modulação do estado de polarização da luz de excitação 6x em 3DOM. (B) A distribuição da intensidade de emissão sob modulação de excitação de polarização de A. (C) Imagem 3DOM de uma fita λ-DNA, onde a imagem da esquerda mostra resultados azimutais e a imagem da direita mostra resultados de ângulo polar. Barras de escala = 10 μm. Abreviação: 3DOM = mapeamento de orientação 3D. Clique aqui para ver uma versão maior desta figura.
Discussão
Em nosso estudo, desenvolvemos um plugin que permite a visualização em tempo real de duas técnicas de imagem de polarização, pSIM e 3DOM. Ambas as tecnologias podem ser executadas em um sistema SIM existente com pequenas modificações. Fornecemos as etapas detalhadas para instalar o microscópio pSIM e 3DOM e configurar o Micro-Manager para controlar o microscópio e demonstrar como obter os resultados de polarização de visualização ao vivo. Os resultados experimentais incluem o filamento de actina fotografado por pSIM e λ-DNA fotografado por 3DOM. A orientação dos dipolos de faloidina é aproximadamente paralela à direção dos filamentos de actina, enquanto a orientação dos fluoróforos STOYOX Orange é perpendicular ao eixo do DNA. A microscopia de iluminação estruturada também pode obter imagens de alta resolução espaço-temporal do aparelho de Golgi, rede de microtúbulos18 e retículo endoplasmático19.
Desenvolvemos plugins personalizados para pSIM e 3DOM; Os usuários podem escolher o programa de visualização adequado para o modo de disparo. Durante o processo de execução do plug-in de visualização em tempo real, se o plug-in não iniciar corretamente, os usuários devem primeiro verificar a configuração do caminho no arquivo de configuração em relação à leitura de imagens de câmeras virtuais, garantindo que o caminho tenha sido modificado com precisão para o local real onde as imagens estão armazenadas. Posteriormente, é essencial garantir que o caminho para ler as imagens de calibração no plug-in de visualização em tempo real também tenha sido atualizado de acordo. Diferentes versões do Micro-Manager podem introduzir novos recursos, mas também podem resultar em problemas de compatibilidade. Portanto, operações específicas precisam ser ajustadas de acordo com a versão real em uso.
A limitação da tecnologia reside no fato de que, embora os plug-ins de visualização em tempo real atuais nos permitam observar a direção da polarização de fluorescência dos dipolos em ambos os pSIM e 3ODM, a resolução das imagens ainda é inadequada, sendo apenas suficiente para observações aproximadas. No entanto, imagens de alta resolução são particularmente cruciais durante o processo de análise e tomada de decisão. Para obter detalhes mais finos, ainda precisamos contar com a plataforma MATLAB e empregar algoritmos avançados de processamento de imagem, como técnicas de reconstrução de super-resolução para processamento aprofundado. A falta de reconstrução completa com nosso plugin se deve ao baixo desempenho do script beanshell usado. A reconstrução completa usando o beanshell levará muito mais tempo do que a aquisição da imagem. O trabalho futuro inclui a implementação da reconstrução completa com Java e computação paralela e a inclusão do recurso na visualização ao vivo.
Esperamos que este plugin melhore a eficiência do experimento observando o estado de polarização em tempo real. A observação instantânea permite que os pesquisadores observem imediatamente as orientações do dipolo nas amostras para que possam localizar imediatamente o alvo de interesse ou um evento raro durante o rastreamento em tempo real. O protocolo também fornece a configuração de hardware e controle de software, o que torna o pSIM e o 3DOM mais fáceis de usar.
Divulgações
Os autores declaram não haver conflitos de interesse.
Agradecimentos
Este trabalho foi apoiado pelo Programa Nacional de Pesquisa e Desenvolvimento da China (2022YFC3401100).
Materiais
| Name | Company | Catalog Number | Comments |
| 100 nm Fluorescent beads | Invitrogen | F8801 | |
| 4% Formaldehyde solution | Invitrogen | R37814 | |
| Camera | Tucsen | Dhyana 400BSI V3 | https://www.tucsen.com/download-software/ |
| Denture base materials (Type I Thermally setting type, liquid) | New Century Dental | N/A | |
| Dulbecco’s Modified Eagle’s Medium | Gibco | C11995500BT | |
| Eclipse TE2000 Inverted Microscope | Nikon | TE2000 E | |
| Fetal Bovine Serum | Gibco | 10099141C | |
| MATLAB R2019b | MathWorks | Version R2019b | https://ww2.mathworks.cn/downloads/ |
| MetroCon V4.0 | Kopin | Version 4.0 | Software of Spatial light modulator |
| Micro-Manager 2.0 | μΜanager | Version 2.0 | Download Micro-Manager Latest Release |
| MS-2000 XYZ Automated Stage | Applied Scientific Instrumentation | MIM3 | https://www.asiimaging.com/support/downloads/usb-support-on-ms-2000-wk-controllers/ |
| myDAQ | National Instruments | 781325-01 | Software and Driver Downloads - NI |
| OBIS 561 nm LS 20 mW Laser | Coherent | 1325777 | |
| Phalloidin-AF568 | Invitrogen | A12380 | |
| Phosphate buffered saline | Corning | 21-040-CV | |
| Poly Methyl Methacrylate | Solarbio | M9810 | |
| ProLong Diamond | Invitrogen | P36980 | |
| Spatial light modulator | Kopin | SXGA-12 | |
| SYTOX orange nucleic acid stain | Invitrogen | S11368 | |
| Triton X-100 | Invitrogen | HFH10 | |
| Trypsin | Gibco | 25200056 | |
| λ-DNA | Invitrogen | S11368 |
Referências
- Zhanghao, K., Gao, J., Jin, D., Zhang, X., Xi, P. Super-resolution fluorescence polarization microscopy. J Innov Opt Health Sci. 11 (01), 1730002 (2018).
- Alonso, M. A., Brasselet, S. Polarization microscopy: from ensemble structural imaging to single-molecule 3D orientation and localization microscopy. Optica. 10 (11), 1486-1510 (2023).
- Valades Cruz, C. A., et al. Quantitative nanoscale imaging of orientational order in biological filaments by polarized superresolution microscopy. Pro Natl Acad Sci USA. 113 (7), E820-E828 (2016).
- Zhanghao, K., et al. Super-resolution dipole orientation mapping via polarization demodulation. Light Sci Appli. 5 (10), e16166 (2016).
- Zhanghao, K., et al. Super-resolution imaging of fluorescent dipoles via polarized structured illumination microscopy. Nat Commun. 10 (1), 4694 (2019).
- Vrabioiu, A. M., Mitchison, T. J. Structural insights into yeast septin organization from polarized fluorescence microscopy. Nature. 443 (7110), 466-469 (2006).
- Backer, A. S., Lee, M. Y., Moerner, W. E. Enhanced DNA imaging using super-resolution microscopy and simultaneous single-molecule orientation measurements. Optica. 3 (6), 659-666 (2016).
- Backer, A. S., et al. Single-molecule polarization microscopy of DNA intercalators sheds light on the structure of S-DNA. Sci Adv. 5 (3), eaav1083 (2019).
- Hulleman, C. N., et al. Simultaneous orientation and 3D localization microscopy with a Vortex point spread function. Nat Commun. 12 (1), 5934 (2021).
- Kampmann, M., Atkinson, C. E., Mattheyses, A. L., Simon, S. M. Mapping the orientation of nuclear pore proteins in living cells with polarized fluorescence microscopy. Nat Struct Mol Biol. 18 (6), 643-649 (2011).
- Lazar, J., Bondar, A., Timr, S., Firestein, S. J. Two-photon polarization microscopy reveals protein structure and function. Nat Methods. 8 (8), 684-690 (2011).
- Dong, B., et al. Parallel Three-Dimensional Tracking of Quantum Rods Using Polarization-Sensitive Spectroscopic Photon Localization Microscopy. ACS Photonics. 4 (7), 1747-1752 (2017).
- Blanchard, A., et al. Turn-key mapping of cell receptor force orientation and magnitude using a commercial structured illumination microscope. Nat Commun. 12 (1), 4693 (2021).
- Toprak, E., et al. Defocused orientation and position imaging (DOPI) of myosin V. Proc Natl Acad Sci USA. 103 (17), 6495-6499 (2006).
- Zhong, S., et al. Three-dimensional dipole orientation mapping with high temporal-spatial resolution using polarization modulation. PhotoniX. 5 (1), 12 (2024).
- Young, L. J., Ströhl, F., Kaminski, C. F. A Guide to Structured Illumination TIRF Microscopy at High Speed with Multiple Colors. J Vis Exp. (111), e53988 (2016).
- Huang, X., et al. long-term, super-resolution imaging with Hessian structured illumination microscopy. Nat Biotechnol. 36 (5), 451-459 (2018).
- Ando, R., et al. StayGold variants for molecular fusion and membrane-targeting applications. Nat Methods. 21 (4), 648-656 (2024).
- Hirano, M., et al. A highly photostable and bright green fluorescent protein. Nat Biotechnol. 40 (7), 1132-1142 (2022).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados