Method Article
Open-Source-Micro-Manager-Plugin für die Live-View-Bildgebung von fluoreszierenden Dipolen
In diesem Artikel
Zusammenfassung
Wir haben ein Open-Source-Micro-Manager-Plugin entwickelt, das die Live-View-Beobachtung von fluoreszierenden Dipolen auf einem Mikroskop mit strukturierter Beleuchtung ermöglicht. Das Plugin unterstützt die Beobachtung sowohl der 2D- als auch der 3D-Dipolausrichtung.
Zusammenfassung
Mit der Fluoreszenzpolarisationsmikroskopie (FPM) können die Position und Dipolorientierung von Fluorophoren abgebildet werden. Trotz der Errungenschaften der superhochauflösenden Fluoreszenzpolarisationsmikroskopie behindert ihre Abhängigkeit von der Nacherfassung die Echtzeitbeobachtung. Die polarisierte strukturierte Beleuchtungsmikroskopie (pSIM) bietet eine hochauflösende Abbildung von fluoreszierenden Dipolen mit hoher Bildgebungsgeschwindigkeit und eignet sich gut für Anwendungen mit lebenden Zellen. Wir haben eine Open-Source-Implementierung für die Echtzeit-Rekonstruktion von Polarisationsbildern und die Darstellung der fluoreszierenden Dipole entwickelt. Darüber hinaus haben wir die Methode erweitert, um ein 3D-Orientierungsmapping (3DOM) zu erreichen, wodurch ihr Nutzen für komplexe biologische Studien erweitert wurde. Darüber hinaus haben wir eine ausführliche Einführung in die Erweiterung eines bestehenden SIM-Mikroskops für die Polarisationsbildgebung gegeben und eine detaillierte Konfigurationsanleitung des Micro-Manager 2.0 zur Steuerung des Mikroskops bereitgestellt, die eine Echtzeitvorschau der polarisierten Bildgebung ermöglicht. Darüber hinaus haben wir den MATLAB-Code für die vollständige Rekonstruktion bereitgestellt, der sowohl pSIM als auch 3DOM umfasst. Dieser umfassende Leitfaden soll Anfängern helfen, die Operationen schnell zu beherrschen und einfach mit ihnen zu beginnen.
Einleitung
Die Fluoreszenzpolarisationsmikroskopie (FPM) hat sich zu einer leistungsfähigen Technik entwickelt, mit der sowohl die Position als auch die Dipolorientierung von Fluorophoren gleichzeitig abgebildet werden können, und bietet tiefgreifende Einblicke in die biologische Bildgebung 1,2. Durch die Erleichterung der Richtungsbeobachtung der Orientierungen von Biomolekülen enthüllt FPM die komplizierte Anordnung von Makromolekülen wie Aktin 3,4,5, Mikrotubuli5, Septin6, DNA-Filament 7-9, Kernporenkomplex10 und Membranproteine11. Seine schnellen, nicht-invasiven und mit lebenden Zellen kompatiblen Fähigkeiten ermöglichen die Verfolgung der molekularen Rotationsdynamik mit hoher zeitlicher Auflösung11,12. In Verbindung mit Bioforce-Sonden kartiert FPM nicht nur Kraftgrößen in subzellulärer Auflösung, sondern misst auch Kraftrichtungen, wodurch unser Verständnis biomechanischer Prozesse durch die Aufdeckung der Richtung von Kräften verbessertwird 13.
In den letzten Jahrzehnten hat sich die superauflösende Fluoreszenzpolarisationsmikroskopie rasant weiterentwickelt. Ein bemerkenswerter Fortschritt auf diesem Gebiet ist die Einzelmolekül-Orientierungslokalisierungsmikroskopie, auch SMOLM genannt, die sowohl die Position als auch die Orientierung von Fluorophoren lokalisieren und so eine mehrdimensionale Lokalisierung ermöglichen kann. Die Polarisationsmessung in SMOLM kann mit Hilfe der Polarisationsanregungsmodulation7, der mehrkanaligen polarisierten Detektion3 oder der speziell entwickelten polarisationsempfindlichen Punktspreizfunktion (PSF)14 durchgeführt werden. Obwohl SMOLM eine räumliche Auflösung in der Größenordnung von Dutzenden von Nanometern erreicht und die Polarisation einzelner Moleküle misst, leidet es unter einer verlängerten Bildgebungszeit. Dies ist auf die sich wiederholenden Zyklen des Blinkens und der Lokalisierung von Fluorophoren zurückzuführen, die eine Herausforderung für die Videobildgebung und Lebendzellanwendungen darstellen.
Im Gegensatz dazu bietet die polarisierte strukturierte Beleuchtungsmikroskopie (pSIM) eine räumliche Auflösung von etwa 100 nm, gekoppelt mit der Erfassung von Polarisationsinformationen mit demselben SIM-Datensatz. Insbesondere kann pSIM Bildgebungsgeschwindigkeiten mit Videorate erreichen und ist hochgradig kompatibel mit der Bildgebung lebender Zellen, ohne strenge Anforderungen an fluoreszierende Moleküle. Kürzlich ist es pSIM gelungen, die Aktinringstruktur im membranassoziierten periodischen Skelett (MPS)5 aufzudecken und eine superauflösende Kartierung biologischer Kräfte zu ermöglichen13.
pSIM erfordert jedoch eine Bildrekonstruktion nach der Aufnahme, was eine Echtzeit-Visualisierung der Polarisationsergebnisse verhindert. Diese Verzögerung behindert die unmittelbare Beobachtung biologischer Phänomene und hindert die Forscher daran, biologische Phänomene von Interesse schnell zu erfassen und Echtzeitanpassungen an Proben und Bildgebungsbedingungen vorzunehmen. Um diese Einschränkung zu beheben, haben wir eine Open-Source-Implementierung entwickelt, die die Bildaufnahme und die Echtzeit-Rekonstruktion und Anzeige von Polarisationsergebnissen auf Basis der ImageJ- und Micro-Manager-Plattform (https://github.com/KarlZhanghao/live-pol-imaging) erleichtert.
Während pSIM bisher auf die Bereitstellung von 2D-Polarisationsinformationen in der Ebene beschränkt war, haben wir kürzlich seine Fähigkeiten erweitert, um eine 3D-Orientierungskartierung mit fast der gleichen Ausrüstung15 zu erreichen, die als 3D-Orientierungskartierung (3DOM) bezeichnet wird. Diese Open-Source-Software bietet auch die Steuerung, Rekonstruktion und Visualisierung von 3DOM. Die Rekonstruktions- und Visualisierungsmodule sind auch mit der Anwendung zur Verfolgung der Einzelmolekülorientierung kompatibel. All diese Funktionalitäten erhöhen den Nutzen der Polarisationsbildgebung in komplexen biologischen Studien.
Protokoll
1. Erweiterung eines bestehenden Mikroskops mit strukturierter Beleuchtung für die Polarisationsbildgebung
- Bereiten Sie ein SIM-Mikroskop vor.
HINWEIS: Wir gehen davon aus, dass die Leser über eine gewisse Grundlage im Mikroskopaufbau verfügen und bereits über ein Strukturiertlichtmikroskop verfügen. Wenn Sie nicht über einschlägige Erfahrung verfügen, lesen Sie dieses Dokument, das eine detaillierte Beschreibung des Baus eines SIM-Mikroskops16 enthält. Das Polarisationssteuergerät von "HWP+WQP+LCVR" kann bei unserer pSIM work5 durch eine Pizzawirbel-Wellenplatte oder eine Pizza-Halbwellenplatte17 ersetzt werden. - Messen Sie Polarisationsextinktionsverhältnisse von drei Streifenrichtungen, indem Sie verschiedene Muster auf den räumlichen Lichtmodulator (SLM) laden. Platzieren und drehen Sie einen Polarisator direkt hinter dem Objektiv und verwenden Sie ein Leistungsmesser, um die Laserleistung zu messen. Drehen Sie für pSIM die Wellenplatte, um die s-Polarisation in drei Richtungen mit einem Extinktionsverhältnis > 10 beizubehalten.
- Drehen Sie für 3DOM den HWP2, um die p-Polarisation in sechs Richtungen beizubehalten. Ersetzen Sie die SIM-Raummaske durch die 3DOM-Raummaske, um Strahlen 1-Ordnung mit sechs Richtungen passieren zu lassen (siehe Abbildung 1B).
HINWEIS: In pSIM ist eine s-Polarisation erforderlich, während in 3DOM eine p-Polarisation erforderlich ist. Eine detaillierte Einrichtung ist an anderer Stelle für pSIM5 und 3DOM15 enthalten.
2. Einrichtung des Mikro-Managers
- Laden Sie die Version Micro-Manager 2.0 von der offiziellen Website herunter, installieren Sie die Software, indem Sie den Anweisungen folgen.
- Bereiten Sie die entsprechenden Gerätetreiber und Software vor, um eine erfolgreiche Verbindung mit dem Micro-Manager-System herzustellen. Verwenden Sie in diesem Mikroskopsystem den Micro-Manager, um die Kamera, den Translationstisch, den Laser, die Datenerfassungskarte und das Mikroskop zu steuern.
HINWEIS: Die unterstützten Instrumente sind in https://micro-manager.org/Device_Support aufgeführt. In der Materialtabelle finden Sie Details zu den anzuschließenden Instrumenten. Installieren Sie Treiber oder offizielle/Support-Software und verbinden Sie das Gerät über ein USB-Kabel mit dem Computer. - Fügen Sie die Instrumente mithilfe des Hardwarekonfigurationsassistenten zu Micro-Manager hinzu.
- Öffnen Sie die Software und wählen Sie Keine aus der anfänglichen Dropdown-Liste aus.
- Wählen Sie Geräte | Hardware-Konfigurations-Assistent... | Erstellen Sie eine neue Konfiguration und klicken Sie auf Weiter, um die Konfigurationsseite aufzurufen.
- Suchen Sie das Plug-In Kamera / Laser / Tisch / Datenerfassung / Mikroskop in der Dropdown-Hardwareliste und klicken Sie auf Hinzufügen | OK | Weiter.
- Gehen Sie zu Standardgeräte auswählen und wählen Sie die Einstellung für den automatischen Verschluss. Legen Sie die Standardkamera, den Standardauslöser und die Standardfokusstufe fest. Klicken Sie auf Weiter , bis Konfiguration und Beenden speichern erreicht ist, und speichern Sie den Namen der Konfigurationsdatei. Klicken Sie auf Fertig stellen.
- Allgemeine Vorlageneinstellungen
- Speichern Sie eine Vorlage für den Live-Modus und den SIM-Snap-Modus.
- Stellen Sie die Belichtungszeit und den Auslösemodus wie folgt ein: Wählen Sie in den Konfigurationseinstellungen Gruppe ' +', benennen Sie den Gruppennamen als 'Modus', wählen Sie Belichtungs - und Auslösemodus und klicken Sie auf OK.
- Wählen Sie Voreinstellung '+' , um verschiedene Voreinstellungen für die Gruppe 'Modus' hinzuzufügen. Nennen Sie eine Voreinstellung "live" für die Belichtungseinstellungen im Live-Modus und eine andere "SIM" für den SIM-Modus. Stellen Sie beispielsweise im Live-Modus die Belichtungszeit auf 15 ms ein, während Sie im SIM-Snap-Modus 10 ms auf die Belichtungszeit einstellen und der Auslösemodus Standard (Überlappung) ist.
HINWEIS: In unserem Code-Repository ist eine Konfigurationsdatei (MMConfig_psim_demo.cfg) enthalten, die direkt von Micro-Manager 2.0 geladen werden kann.
3. Systemkalibrierung mit fluoreszierenden Kügelchen
- Tauchen Sie die Deckgläser in 75%iges Ethanol, spülen Sie die Deckgläser 3x mit entionisiertem Wasser (ddH2O) ab und trocknen Sie sie.
- Die 100 nm fluoreszierenden Kügelchen (verdünnt auf 1:1.000 mit phosphatgepufferter Kochsalzlösung [PBS]) vortexen und die Suspension direkt auf die Deckgläser pipettieren. 5-10 Minuten in dunkler Umgebung inkubieren und dann vorsichtig mit PBS waschen.
- Geben Sie 20 μl Eindeckmedium auf den Objektträger und positionieren Sie das Deckglas darüber, wobei Sie darauf achten, dass keine Blasen entstehen. Halten Sie die Temperatur bei 4 °C und halten Sie den Objektträger im Dunkeln, damit der Objektträger mit 100 nm fluoreszierenden Kügelchen für die Systemkalibrierung vorbereitet ist.
- Nehmen Sie für pSIM drei Bilder der drei verschiedenen Phasen von fluoreszierenden Kügelchen auf. Ein benutzerdefinierter MATLAB-Code (Bead_calib.m) nimmt die Bilder als Eingabe und gibt zwei Kalibrierungsbilder (calib1.tif, calib2.tif) zur weiteren Analyse aus.
HINWEIS: Wie bereits in5 gezeigt, kann das pSIM-Experiment auf den meisten kommerziellen Systemen durchgeführt werden. Bei selbstgebauten oder kommerziellen SIM-Mikroskopen ist eine Systemkalibrierung erforderlich, um den Messfehler von Fluoreszenzdipolen zu eliminieren, der durch die ungleichmäßige Beleuchtung in drei Musterrichtungen verursacht wird. Das Kalibrierungsexperiment geht von der isotropen Polarisation fluoreszierender Kügelchen aus und benötigt nur die Rohbilder während der SIM-Aufnahme. Ein detailliertes Kalibrierungsverfahren ist in unserer Originalarbeitenthalten 5. Die Ausgabe von zwei Kalibrierungsbildern calib1.tif und calib2.tif ist für die pSIM-Rekonstruktion erforderlich, die die pixelweise Beleuchtungsenergie der Musterrichtung 2 angibt; 3 bezieht sich auf Musterrichtung 1.
4. Probenvorbereitung: Aktin in fixierten Zellen
- Tauchen Sie die Deckgläser in 75%iges Ethanol, spülen Sie die Deckgläser 3x mit ddH2O aus. Legen Sie die Deckgläser in eine Sechs-Well-Zellkulturplatte.
- Kultur von U2OS-Zellen (ATCC HTB-96-Zelllinie) in Dulbecco's Modified Eagle's Medium (DMEM), ergänzt mit 10 % (v/v) fötalem Rinderserum (FBS) bei 37 °C und 5 % CO2 auf Deckgläsern, bis sie eine Konfluenz von etwa 75 % erreichen.
- Waschen Sie die Zellen vorsichtig 3x mit PBS. Zur Zellfixierung 4% Paraformaldehyd 15 min lang bei Raumtemperatur auftragen. Nach der Fixierung die Zellen noch einmal 3x mit PBS waschen.
- Um die Membranpermeabilität zu verbessern, behandeln Sie die Zellen 5 Minuten lang mit 0,1 % Triton X-100 in PBS und waschen Sie die Zellen dann 3x mit PBS.
- Fluoreszierendes Phalloidin mit 150 μl DMSO für Stammlösung (66 μM) auflösen. Bereiten Sie die Phalloidin-Farbstoff-Arbeitslösung vor, indem Sie 5 μl der Stammlösung in 995 μl PBS verdünnen. Inkubieren Sie die Zellen im Dunkeln mit Phalloidin-Farbstoff-Arbeitslösung bei Raumtemperatur für ~1 h. Waschen Sie die Zellen 3 x 3 min mit PBS.
- Geben Sie 20 μl Eindeckmedium auf den Objektträger. Legen Sie das Deckglas vorsichtig und blasenfrei auf den Objektträger. Lagern Sie den Objektträger bei 4 °C und bewahren Sie ihn an einem dunklen Ort auf.
5. Probenvorbereitung: in-vitro λ-DNA
- Lösen Sie 0,5 g Polymethylmethacrylat in 10 ml Prothesenbasismaterialien. Tragen Sie nach der gründlichen Auflösung die Lösung gleichmäßig auf die Oberfläche des Objektträgers auf und warten Sie dann, bis sie verdunstet ist, um einen Film zu bilden.
- 0,3 μl λ-DNA und 32 μl 1.000x SYTOX Orange Nukleinsäure-Färbung in 968 μl PBS verdünnen. Geben Sie 1 μl der vorbereiteten Lösung auf den Objektträger und lassen Sie ihn trocknen.
- Versiegeln Sie das Deckglas mit dem Eindeckmittel auf dem Objektträger, um die Probe zu schützen und zu konservieren. Lagern Sie den Objektträger bei 4 °C in einer dunklen Umgebung.
6. Bildgebung
- Setzen Sie das Deckglas auf den Probenhalter und stellen Sie es im Live-Modus auf die Fokusebene ein. Wählen Sie einen ROI von 512 x 512 Pixeln und passen Sie die Belichtungszeit und die Laserintensität an.
- Öffnen Sie die unabhängige Steuerungssoftware des Raumlichtmodulators und wählen Sie den geeigneten Sequenzpfad für das Beleuchtungsmuster für pSIM / 3DOM oder den Live-Modus.
HINWEIS: Bei pSIM umfasst das Beleuchtungsmuster drei verschiedene Polarisationswinkel und drei verschiedene Phasen. Bei 3DOM umfasst das Beleuchtungsmuster sechs verschiedene Polarisationswinkel. - Wählen Sie den SIM-Aufnahmemodus , wählen Sie Multi-D Acq., wählen Sie Time Points und stellen Sie die Anzahl (9 oder 6 Bilder für pSIM oder 3DOM) und das Intervall (0 ms) im geöffneten Fenster ein. Klicken Sie auf Erwerben!
- Klicken Sie auf TriggerSLM.bsh (eine Konfigurationsdatei ist in unserem Code-Repository enthalten)), die MyDaq-Erfassungskarte aktiviert den räumlichen Lichtmodulator, um die Bilder aufzunehmen.
7. Live-View-Plugin von pSIM und 3DOM
- Konfigurieren Sie die FakeCamera und den Bildzugriffspfad: Laden Sie das Skript (MMConfig_FakeCam_demo.cfg), indem Sie auf Geräte | Hardwarekonfiguration laden.... Konfigurieren Sie das lokale Verzeichnis der Bilder unter Geräte | Eigenschaftenbrowser "Geräte"... | Kamerapfadmaske (Abbildung 2A-C).
HINWEIS: Der Erfassungsteil des Skripts hängt von der Hardware-Konfiguration ab, daher haben wir eine Version (MMConfig_FakeCam_demo.cfg) bereitgestellt, die das FakeCamera-Gerät verwendet, um lokale Bilder auf der Festplatte zu Demonstrationszwecken zu lesen. Benutzer können Änderungen in unserer Version vornehmen. Denken Sie daran, den Bildlesepfad (Kamera-Pfad-Maske) zu ändern. - Stellen Sie das Schnellzugriffsfeld ein, indem Sie auf Extras | Schnellzugriff Paneel | Neues Panel erstellen. Klicken Sie auf das Symbol "Einstellungen " in der unteren linken Ecke und aktivieren Sie "Run Script" im oberen Teil, wodurch die Dateien "psim.bsh" und "3DOM.bsh" geladen werden können (Abbildung 2D).
HINWEIS: Das Schnellzugriffspanel-System ermöglicht es Benutzern, benutzerdefinierte Fenster zu erstellen und einfach auf die Steuerelemente im Micro-Manager zuzugreifen, was am häufigsten benötigt wird. "Live", "Snap" oder andere Kanäle können ebenfalls hinzugefügt werden, wie in Abbildung 2D dargestellt. - Führen Sie am Beispiel von pSIM eine Echtzeit-Polarisationsbildrekonstruktion durch, indem Sie auf "psim.bsh" klicken. Beobachten Sie die in Echtzeit rekonstruierten Bilder mit Polarisationsergebnissen (Abbildung 2E). Denken Sie daran, den Pfad der Kalibrierungsbilder in der Datei 'psim.bsh' zu ändern (calibPath = " Pfad zur Datei mit den Kalibrierungsbildern").
HINWEIS: Das Live-View-Plugin ist ein Beanshell-Skript (psim.bsh und 3DOM.bsh), das im Skript-Panel ausgeführt werden kann. Das Plugin erfasst neun Bilder für pSIM oder sechs Bilder für 3DOM und berechnet die Ergebnisse des Orientation-Mappings basierend auf den Weitfeldbildern. Die Orientation-Mapping-Bilder werden so angezeigt, dass sie Echtzeit-Polarisationsinformationen liefern, die trotz der geringen Auflösung eine sofortige Inspektion ermöglichen.
8. Datenanalyse mit superauflösender Rekonstruktion
- Installieren Sie MATLAB R2019b von der Website (siehe Materialtabelle).
- Öffnen Sie die Software. Für die pSIM-Rekonstruktion erhalten Sie neun Bilder, die aus drei verschiedenen Phasen für jeden der drei Orientierungswinkel bestehen. Stellen Sie sicher, dass die Namen der neun Bilder zwischen '1.tif' und '9.tif' liegen, fügen Sie sie in die Datei mit dem Namen 'Input' ein und führen Sie das Programm mit dem Namen 'PSIM.m' aus. Beobachten Sie das rekonstruierte Weitfeldbild, das SIM-Bild und das pSIM-Bild mit dem Farbrad.
HINWEIS: Die Rekonstruktion für pSIM erfordert zwei Schritte: einen SIM-Schritt und einen PM-Schritt. Die Daten werden von einem speziell geschriebenen MATLAB-Programm analysiert, um das Debuggen zu erleichtern. Rufen Sie den Rekonstruktionscode aus dem Ordner "reconsr" in GitHub ab. - Erfassen Sie für 3DOM sechs verschiedene Polarisationsrichtungen. Führen Sie die sechs Bilder zusammen, nennen Sie die resultierende Datei "Raw_data.tif" und führen Sie das Programm mit dem Namen "Recon_3DOM.m" aus. Betrachten Sie das rekonstruierte Weitfeldbild und das 3DOM-Bild mit azimutalen Ergebnissen und den Ergebnissen des Polarwinkels.
HINWEIS: Während des Rekonstruktionsprozesses kann ein Fehler auftreten'--'Funktion oder Variable 'bfopen' wird nicht erkannt'. 'bfopen' ist eine Funktion in der Bioformate-Bibliothek und wird häufig zum Öffnen und Lesen von Bilddateien verwendet. Geben Sie addpath ('path_to_bioformats_toolbox') in das MATLAB-Befehlsfenster ein. Ersetzen Sie "path_to_bioformats_toolbox" durch den tatsächlichen Pfad des Bibliotheksordners. Außerdem haben wir dem GitHub-Repository die Abhängigkeit Bio-Formats hinzugefügt.
Ergebnisse
Die pSIM-Methode kann an SIM-Mikroskopen durchgeführt werden, basierend auf der Interferenz mit s-polarisierten Laserstrahlen. s-Polarisationsinterferenz ist der am weitesten verbreitete SIM-Typ und erzeugt kontrastreiche Beleuchtungsstreifen. Der akademische Prototyp eines Mikroskopaufbaus ist in der Originalarbeit von pSIM5 enthalten. Ein räumlicher Lichtmodulator (SLM), der in Abbildung 1 kurz vorgestellt wurde, erzeugt die ±1-Ordnung diffraktiver Strahlen, und eine Halbwellenplatte aus Pizza hält die s-Polarisation in drei Richtungen aufrecht. Während andere SIM-Alternativen Gitter zur Erzeugung diffraktiver Strahlen oder andere Polarisationsmodulationsmethoden zur Aufrechterhaltung der s-Polarisation verwenden können, können diese Mikroskope auch für die Polarisationsbildgebung erweitert werden.
Die 3DOM-Mikroskope verwenden ein ähnliches optisches Bildgebungssystem (Abbildung 1A) mit zwei Unterschieden. Erstens benötigt das 3DOM keine Interferenz von zwei Laserstrahlen. Stattdessen verwendet es nur die +1-Strahlen, um die Anregungspolarisation zu steuern. Wie in der ursprünglichen Arbeit15 gezeigt, benötigt 3DOM mindestens sechs Richtungen, um die 3D-Dipolorientierung zu berechnen. Daher enthält die 3DOM-Raummaske sechs Richtungen mit jeweils einer Lochblende (siehe Abbildung 1B). Ein weiterer Unterschied besteht darin, dass der 3DOM p-polarisierte Laserstrahlen verwendet, was durch Drehen des HWP2 erreicht werden kann. Das SLM erzeugt gebeugtes Licht in verschiedenen schrägen Winkeln, um den Anteil der p-Polarisation zu ändern.
Beim pSIM-System wird das austretende Licht durch Drehen des Winkels von Halbwellenplatten in s-polarisiertes Licht moduliert. Die Maske der pSIM ist symmetrisch, wodurch Licht ±1 Ordnung gleichzeitig durchgelassen werden kann. Umgekehrt wird bei 3DOM der Laser durch Einstellen des Winkels der Halbwellenplatte in p-polarisiertes Licht umgewandelt. Die Maske von 3DOM ist nicht asymmetrisch und lässt nur Licht von +1 Ordnung durch. Durch die Einstellung der Streifenfrequenz des SLM verändert es den Beugungswinkel von Laserstrahlen. Somit wird der Anteil der Polarisation außerhalb der Ebene moduliert.
Durch die Analyse von zwei benachbarten fluoreszierenden Dipolen, die den gleichen Azimutalwinkel, aber unterschiedliche Polarwinkel haben, können sowohl pSIM als auch 3DOM Dipole in der Nähe innerhalb der Beugungsgrenze unterscheiden. Darüber hinaus kann 3DOM verschiedene Dipole mit 3D-Orientierung im Polarisationsbereich effektiv unterscheiden (Abbildung 1C,D). Dies liegt daran, dass die 3DOM-Technologie mehr räumliche Informationen erhalten kann, indem sie den Polarisationszustand des angeregten Lichts ändert, um eine genaue Messung der Dipolausrichtung im 3D-Raum zu erreichen. Im Gegensatz dazu stützt sich die pSIM-Technologie hauptsächlich auf 2D-Polarisationsinformationen und kann die Vorteile der 3D-Orientierungseigenschaften nicht voll ausschöpfen. Es gibt daher keine Auskunft über die Ausrichtung der Dipole in vertikaler Richtung.
Da die Ergebnisse des Orientational Mapping eine Nachbearbeitung erfordern, haben wir das 'Live-view plugin' ('psim.bsh' und '3DOM.bsh') für Micro-Manager 2.0 entwickelt und vorgestellt. Um Computerbenutzern ohne angeschlossene reale Kamera die Verwendung des Plugins zu ermöglichen, konfigurieren wir zunächst den Micro-Manager mit FakeCamera.In Abbildung 2, wir haben den Betriebsprozess für pSIM detailliert beschrieben, um Echtzeit-Polarisationseffekte zu erzielen, und die Betriebsschritte für 3DOM sind ähnlich. In Abbildung 2A ist die Softwareoberfläche von Micro-Manager 2.0 dargestellt, zusammen mit den notwendigen Einrichtungsschritten, die erforderlich sind, um unser Plugin für den Betrieb zu konfigurieren. In Abbildung 2B zeigen wir die Hardwarekonfiguration mit Demonstrationsgeräten von FakeCamera und DStage. Die Hardwarekonfiguration von FakeCamera ist in der Lage, verschiedene Verhaltensweisen einer echten Kamera zu simulieren, wie z. B. Belichtung, Fokussierung und Weißabgleich, wodurch Entwickler Softwareentwicklung und -tests durchführen können, ohne dass eine tatsächliche Kamera erforderlich ist. Benutzer sollten den Bildlesepfad basierend auf den tatsächlichen Umständen ändern, wie in Abbildung 2C dargestellt. Abbildung 2D zeigt einen Schnellzugriffsbereich, der Verknüpfungen zu einer Reihe häufig verwendeter Funktionen oder Einstellungen enthalten kann, sodass Benutzer schneller auf diese Funktionen zugreifen und diese verwenden können. Die Echtzeit-Vorschau-Plugins 'psim.bsh' und '3DOM.bsh' können von diesem Panel geladen werden. Die konkreten Betriebsschritte finden Sie im Detail in Protokollabschnitt 7. Die Skripte "psim.bsh" und "3DOM.bsh" werden verwendet, um sofort die Ausrichtungs-Mapping-Bilder zu generieren. Dieses Plugin 'psim.bsh' ermöglicht die sofortige Visualisierung von Polarisationseffekten während der pSIM-Bildaufnahmephase. Abbildung 2E zeigt ein typisches Ergebnis der Polarisationsbildgebung von pSIM. Das Farbrad zeigt die Beziehung zwischen der Dipolorientierung und der Pseudofarbe an, was für Forscher hilfreich ist, um auf der Grundlage der angezeigten Polarisationsdaten sofort zu entscheiden, welche Bilder aufgenommen werden sollen.
U2OS-Zellen wurden mit Phalloidin markiert, und der Bildrekonstruktionsprozess von pSIM wurde mit MATLAB durchgeführt. Es wurden neun Fotos aus drei verschiedenen Polarisationszuständen und drei verschiedenen Phasen aufgenommen. Die grünen Doppelpfeile markieren die Richtung des polarisierten Lichts in Abbildung 3A. Aus der Hell- und Dunkelintensität des Bildes kann die Polarisationsorientierung der Fluoreszenzdipole bestimmt werden.
In Abbildung 3B zeigt die linke Abbildung die Superauflösungsergebnisse der SIM-Karte, während die rechte Abbildung die pSIM-Ergebnisse zeigt. Bemerkenswert ist, dass pSIM die gleiche räumliche Auflösung wie SIM erreicht und im Vergleich zur Weitfeldmikroskopie eine doppelt so hohe räumliche Auflösung bietet. Durch die Zuordnung bestimmter Farben zu unterschiedlichen Richtungen der Aktinfilamente bietet das Farbrad eine visuelle Darstellung, die es uns ermöglicht, die Ausrichtung der fluoreszierenden Dipole innerhalb der Aktinfilamente der Zelle zu interpretieren. Die Farbe des Aktinfilaments zeigt, dass die Dipole ungefähr mit der Filamentorientierung ausgerichtet sind.
Abbildung 4A zeigt die Verteilung der Emissionsintensität unter Polarisationsanregungsmodulation; 3DOM kann sechs verschiedene Polarisationsmodulationen erzeugen. In dieser Konfiguration wird das angeregte Licht des fluoreszierenden Moleküls durch Änderung des Polarisationszustands moduliert, wodurch die Emissionsintensität des Moleküls beeinflusst wird. Dies ermöglicht es den Forschern, die Orientierung von Molekülen abzuleiten, indem sie die Intensität der Fluoreszenz in verschiedenen Polarisationszuständen messen. Abbildung 4B zeigt die Fluoreszenzintensität von λ-DNA unter verschiedenen Polarisationszuständen in Abbildung 4A. λ-DNA-Moleküle weisen in verschiedenen Polarisationszuständen unterschiedliche Fluoreszenzintensitäten auf, was darauf hindeutet, dass die Ausrichtung des Fluoreszenzdipels und der Umgebung einen signifikanten Einfluss auf die Intensität des Fluoreszenzsignals hat. Abbildung 4C zeigt eine superhochauflösende Bildgebung der λ-DNA, die durch 3DOM erzielt wurde, und zeigt sowohl azimutale Ergebnisse als auch Ergebnisse des Polarwinkels. Unsere Beobachtungen zeigen, dass die Farbstoffmoleküle fast senkrecht zu den DNA-Strängen ausgerichtet waren. Da sich die DNA-Achse biegt, dreht sich die durchschnittliche Ausrichtung der Farbstoffmoleküle entsprechend. Die Ergebnisse unserer Orientierungsmessungen bestätigen die Insertion von STOYOX Orange Fluorophoren zwischen benachbarten DNA-Basen, und die Ausrichtung des Absorptionsdipolmoments ist senkrecht zur DNA-Achse.
In 3DOM ist daher ein gekipptes Anregungslicht erforderlich, um die z-Achsen-Information des Zielmoleküls zu erhalten. In unserem System wird SLM verwendet, um die Einstellung des Beleuchtungslichts zu realisieren. In Kombination mit den Eigenschaften von SLM besteht das aus dem SLM austretende Licht aus Lichtordnungen von 0 und ±1 (und die Lichtordnungen +1 und -1 sind symmetrisch), wenn das SLM mit einem Streifenmuster belastet ist (und die Lichtordnungen +1 und -1 symmetrisch sind). Daher verwenden wir das Licht der +1 Ordnung als Anregungslicht und die asymmetrische Maske, um das Licht der 0 und -1 Ordnung herauszufiltern. Darüber hinaus kann das schräge Anregungslicht nicht nur durch den Einsatz von SLM, sondern auch durch den Einsatz eines scannenden Galvanometers oder durch seitliches Bewegen der Linse vor der Objektivlinse realisiert werden.
Die Verwendung von sechs verschiedenen Anregungspolarisationszuständen in 3DOM wird durch sein Prinzip15 vorgegeben. Durch Anpassen des Forward-Imaging-Modells besteht das erhaltene Modell aus sechs Teilen. Daher ist es nicht möglich, weniger als sechs Anpassungen der Polarisationszustände zu verwenden, und ein Überschuss von sechs würde zu einer Redundanz der Information führen. Das spezifische Prinzip des 3DOM ist im Folgenden dargestellt.
In sphärischen Koordinaten charakterisieren wir das fluoreszierende Molekül mit Hilfe des azimutalen Winkels ρ und des Polarwinkels η wie folgt:

ρ ist der Winkel im Uhrzeigersinn zwischen der Projektion des Moleküls auf der x-y-Ebene und der x-Achse, und η ist der Winkel zwischen dem Molekül und der z-Achse.
Das p-polarisierte Anregungslicht zeichnet sich aus als:

Wobei α und β den azimutalen Winkel bzw. den schrägen Einfallswinkel des Anregungslichts darstellen. Die Photonenabsorptionseffizienz ist ,  und unter der Annahme, dass die Emission von w Dipolen in der Fokusebene innerhalb des n-ten Pixels erfasst wird, dann wird das vom Dipol emittierte Fluoreszenzphoton wie folgt bezeichnet:
und unter der Annahme, dass die Emission von w Dipolen in der Fokusebene innerhalb des n-ten Pixels erfasst wird, dann wird das vom Dipol emittierte Fluoreszenzphoton wie folgt bezeichnet:


Dabei steht C für die Bestrahlungsstärke der Probe und  stellt den Polarisationszustand des Anregungslichts dar, wie wie folgt beschrieben:
stellt den Polarisationszustand des Anregungslichts dar, wie wie folgt beschrieben:

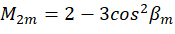

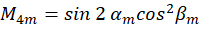
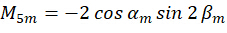
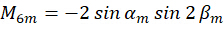
Durch die Kombination des Koeffizienten 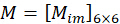 kann die Ausrichtung
kann die Ausrichtung 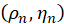 des n-ten Pixels gelöst werden, indem die erfassten Bilder
des n-ten Pixels gelöst werden, indem die erfassten Bilder  wie folgt als Eingabe verwendet werden:
wie folgt als Eingabe verwendet werden:
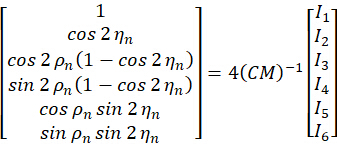
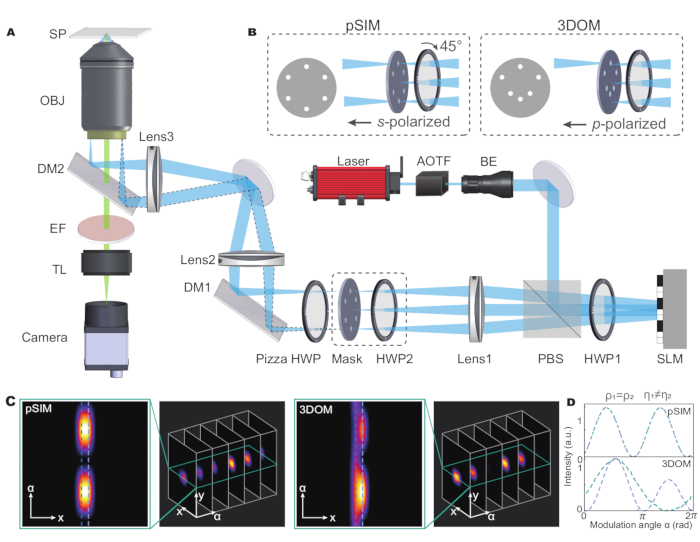
Abbildung 1: Schematische Darstellung der pSIM- und 3DOM-Mikroskope. (A) Versuchsaufbau für pSIM und 3DOM. (B)Die Hauptunterschiede zwischen dem pSIM- und dem 3DOM-System sind die Maske und HWP2. Die Maske der pSIM sorgt dafür, dass Licht ±1 Ordnung gleichzeitig durchgelassen wird und das ausgehende Licht durch Drehen des HWP2 s-polarisiert wird. Die Maske von 3DOM dringt nur durch Licht +1 Ordnung, und das ausgehende Licht ist p-polarisiert. (C, D) Vergleich der Schwankungen der Emissionsfluoreszenzintensität zwischen pSIM und 3DOM. Abkürzungen: pSIM = polarisierte strukturierte Beleuchtungsmikroskopie; 3DOM = 3D-Orientierungs-Mapping; AOTF = akusto-optischer abstimmbarer Filter; BE = Strahlaufweiterer; PBS = Polarisations-Strahlteiler; HWP = Halbwellenplatte; SLM = Raumlichtmodulator; DM = dichroitischer Spiegel; OBJ = Ziel; SP = Abtastebene; EF = Emissionsfilter; TL = Tubus-Linse. Diese Abbildung wurde von Zhanghao et al.5 und Zhong et al.15 modifiziert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 2: Software-Screenshot und repräsentative Ergebnisse von pSIM. (A)Die Benutzeroberfläche von Micro-Manager 2.0. Einige Optionen können zum Konfigurieren von Dateien verwendet werden. (B) Konfiguration des Live-View-Plugins mit FakeCamera und DStage. (C)Auswahlschnittstelle für den Bildlesepfad (Pfadmaske). (D) Einrichten des Schnellzugriffsfensters und Laden von 'Skript ausführen' mit 'psim.bsh' und '3DOM.bsh'. (E) Die Echtzeit-Bildrekonstruktion zeigt die Polarisationseffekte. Die Dipolorientierungen sind pseudofarbig. Das Farbrad zeigt die Beziehung zwischen der Dipolorientierung und der Pseudofarbe an. Abkürzungen: pSIM = polarisierte strukturierte Beleuchtungsmikroskopie; 3DOM = 3D-Orientierungs-Mapping. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
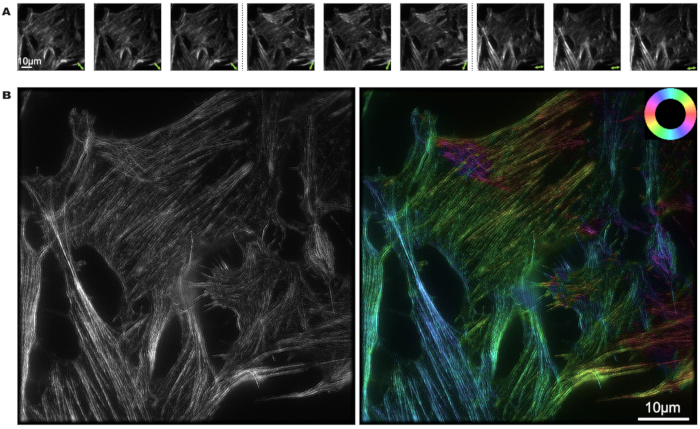
Abbildung 3: pSIM-Bildgebung von Aktinfilamenten in Phalloidin-markierten U2OS-Küvetten. (A) Modulation des Polarisationszustands des Anregungslichts um 3x in pSIM, wobei drei verschiedene Phasen für jeden Polarisationszustand erfasst werden. Die Doppelpfeile zeigen die Richtung des polarisierten Lichts an. (B) SIM- und pSIM-Bild eines Aktinfilaments in A, wobei das linke Bild die SIM-Ergebnisse und das rechte Bild die pSIM-Ergebnisse mit dem Polarisationswinkel zeigt. Das Farbrad oben rechts veranschaulicht die Beziehung zwischen der Pseudofarbe und der Dipolorientierung. Maßstabsleisten = 10 μm. Abkürzung: pSIM = polarisierte strukturierte Beleuchtungsmikroskopie. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 4: SYTOX Orange-markierte Orange-markierte λ-DNA, abgebildet von 3DOM. (A) Modulation des Polarisationszustands des Anregungslichts um 6x in 3DOM. (B) Die Emissionsintensitätsverteilung unter Polarisationsanregungsmodulation von A. (C) 3DOM-Bild eines λ-DNA-Strangs, wobei das linke Bild azimutale Ergebnisse und das rechte Bild Polarwinkelergebnisse zeigt. Maßstabsleisten = 10 μm. Abkürzung: 3DOM = 3D-Orientierungsmapping. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Diskussion
In unserer Studie haben wir ein Plugin entwickelt, das eine Echtzeitvorschau von zwei Polarisationsbildgebungsverfahren, pSIM und 3DOM, ermöglicht. Beide Technologien können mit leichten Modifikationen in einem bestehenden SIM-System durchgeführt werden. Wir haben die detaillierten Schritte zur Installation des pSIM- und 3DOM-Mikroskops bereitgestellt und den Micro-Manager eingerichtet, um das Mikroskop zu steuern und zu demonstrieren, wie die Polarisationsergebnisse in der Live-Ansicht erhalten werden. Zu den experimentellen Ergebnissen gehören das Aktinfilament, das mit pSIM abgebildet wurde, und die λ-DNA, die mit 3DOM abgebildet wurde. Die Orientierung der Phalloidin-Dipole ist ungefähr parallel zur Richtung der Aktinfilamente, während die Orientierung der STOYOX Orange Fluorophore senkrecht zur DNA-Achse verläuft. Mit der strukturierten Beleuchtungsmikroskopie kann auch eine räumlich-zeitlich aufgelöste Abbildung des Golgi-Apparats, des Mikrotubuli-Netzwerks18 und des endoplasmatischen Retikulums19 erzielt werden.
Wir haben maßgeschneiderte Plugins sowohl für pSIM als auch für 3DOM entwickelt. Benutzer können das richtige Vorschauprogramm für ihren Aufnahmemodus auswählen. Wenn das Plug-in während des Ausführens des Echtzeitvorschau-Plug-ins nicht ordnungsgemäß gestartet werden kann, müssen Benutzer zunächst die Pfadeinstellung in der Konfigurationsdatei für das Lesen von Bildern der virtuellen Kamera überprüfen und sicherstellen, dass der Pfad genau an den tatsächlichen Speicherort geändert wurde, an dem die Bilder gespeichert sind. Anschließend ist unbedingt darauf zu achten, dass auch der Pfad zum Auslesen von Kalibrierungsbildern im Echtzeit-Vorschau-Plugin entsprechend aktualisiert wurde. Verschiedene Versionen des Micro-Managers können neue Funktionen einführen, aber auch zu Kompatibilitätsproblemen führen. Daher müssen bestimmte Vorgänge entsprechend der tatsächlich verwendeten Version angepasst werden.
Die Einschränkung der Technologie liegt in der Tatsache, dass wir mit aktuellen Echtzeit-Vorschau-Plugins zwar die Fluoreszenzpolarisationsrichtung von Dipolen sowohl in pSIM als auch in 3ODM beobachten können, die Auflösung der Bilder jedoch immer noch unzureichend ist und nur für grobe Beobachtungen ausreicht. Hochauflösende Bilder sind jedoch besonders wichtig für den Prozess der Analyse und Entscheidungsfindung. Um feinere Details zu erhalten, müssen wir uns immer noch auf die MATLAB-Plattform verlassen und fortschrittliche Bildverarbeitungsalgorithmen wie hochauflösende Rekonstruktionstechniken für eine tiefgreifende Verarbeitung einsetzen. Das Fehlen einer vollständigen Rekonstruktion mit unserem Plugin ist auf die geringe Leistung des verwendeten Beanshell-Skripts zurückzuführen. Die vollständige Rekonstruktion mit Beanshell dauert viel länger als die Bildaufnahme. Zu den zukünftigen Arbeiten gehört die Implementierung des vollständigen Umbaus mit Java und parallelem Computing sowie die Aufnahme des Features in die Live-Vorschau.
Wir hoffen, dass dieses Plugin die Effizienz des Experiments verbessert, indem es den Polarisationszustand in Echtzeit beobachtet. Die sofortige Beobachtung ermöglicht es den Forschern, die Dipolorientierung in Proben sofort zu beobachten, so dass sie das interessierende Ziel oder ein seltenes Ereignis während der Live-Zeit-Verfolgung sofort lokalisieren können. Das Protokoll bietet auch die Hardware-Einrichtung und die Softwaresteuerung, was pSIM und 3DOM benutzerfreundlicher macht.
Offenlegungen
Die Autoren erklären, dass keine Interessenkonflikte bestehen.
Danksagungen
Diese Arbeit wurde durch das National Key Research and Development Program of China (2022YFC3401100) unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| 100 nm Fluorescent beads | Invitrogen | F8801 | |
| 4% Formaldehyde solution | Invitrogen | R37814 | |
| Camera | Tucsen | Dhyana 400BSI V3 | https://www.tucsen.com/download-software/ |
| Denture base materials (Type I Thermally setting type, liquid) | New Century Dental | N/A | |
| Dulbecco’s Modified Eagle’s Medium | Gibco | C11995500BT | |
| Eclipse TE2000 Inverted Microscope | Nikon | TE2000 E | |
| Fetal Bovine Serum | Gibco | 10099141C | |
| MATLAB R2019b | MathWorks | Version R2019b | https://ww2.mathworks.cn/downloads/ |
| MetroCon V4.0 | Kopin | Version 4.0 | Software of Spatial light modulator |
| Micro-Manager 2.0 | μΜanager | Version 2.0 | Download Micro-Manager Latest Release |
| MS-2000 XYZ Automated Stage | Applied Scientific Instrumentation | MIM3 | https://www.asiimaging.com/support/downloads/usb-support-on-ms-2000-wk-controllers/ |
| myDAQ | National Instruments | 781325-01 | Software and Driver Downloads - NI |
| OBIS 561 nm LS 20 mW Laser | Coherent | 1325777 | |
| Phalloidin-AF568 | Invitrogen | A12380 | |
| Phosphate buffered saline | Corning | 21-040-CV | |
| Poly Methyl Methacrylate | Solarbio | M9810 | |
| ProLong Diamond | Invitrogen | P36980 | |
| Spatial light modulator | Kopin | SXGA-12 | |
| SYTOX orange nucleic acid stain | Invitrogen | S11368 | |
| Triton X-100 | Invitrogen | HFH10 | |
| Trypsin | Gibco | 25200056 | |
| λ-DNA | Invitrogen | S11368 |
Referenzen
- Zhanghao, K., Gao, J., Jin, D., Zhang, X., Xi, P. Super-resolution fluorescence polarization microscopy. J Innov Opt Health Sci. 11 (01), 1730002 (2018).
- Alonso, M. A., Brasselet, S. Polarization microscopy: from ensemble structural imaging to single-molecule 3D orientation and localization microscopy. Optica. 10 (11), 1486-1510 (2023).
- Valades Cruz, C. A., et al. Quantitative nanoscale imaging of orientational order in biological filaments by polarized superresolution microscopy. Pro Natl Acad Sci USA. 113 (7), E820-E828 (2016).
- Zhanghao, K., et al. Super-resolution dipole orientation mapping via polarization demodulation. Light Sci Appli. 5 (10), e16166 (2016).
- Zhanghao, K., et al. Super-resolution imaging of fluorescent dipoles via polarized structured illumination microscopy. Nat Commun. 10 (1), 4694 (2019).
- Vrabioiu, A. M., Mitchison, T. J. Structural insights into yeast septin organization from polarized fluorescence microscopy. Nature. 443 (7110), 466-469 (2006).
- Backer, A. S., Lee, M. Y., Moerner, W. E. Enhanced DNA imaging using super-resolution microscopy and simultaneous single-molecule orientation measurements. Optica. 3 (6), 659-666 (2016).
- Backer, A. S., et al. Single-molecule polarization microscopy of DNA intercalators sheds light on the structure of S-DNA. Sci Adv. 5 (3), eaav1083 (2019).
- Hulleman, C. N., et al. Simultaneous orientation and 3D localization microscopy with a Vortex point spread function. Nat Commun. 12 (1), 5934 (2021).
- Kampmann, M., Atkinson, C. E., Mattheyses, A. L., Simon, S. M. Mapping the orientation of nuclear pore proteins in living cells with polarized fluorescence microscopy. Nat Struct Mol Biol. 18 (6), 643-649 (2011).
- Lazar, J., Bondar, A., Timr, S., Firestein, S. J. Two-photon polarization microscopy reveals protein structure and function. Nat Methods. 8 (8), 684-690 (2011).
- Dong, B., et al. Parallel Three-Dimensional Tracking of Quantum Rods Using Polarization-Sensitive Spectroscopic Photon Localization Microscopy. ACS Photonics. 4 (7), 1747-1752 (2017).
- Blanchard, A., et al. Turn-key mapping of cell receptor force orientation and magnitude using a commercial structured illumination microscope. Nat Commun. 12 (1), 4693 (2021).
- Toprak, E., et al. Defocused orientation and position imaging (DOPI) of myosin V. Proc Natl Acad Sci USA. 103 (17), 6495-6499 (2006).
- Zhong, S., et al. Three-dimensional dipole orientation mapping with high temporal-spatial resolution using polarization modulation. PhotoniX. 5 (1), 12 (2024).
- Young, L. J., Ströhl, F., Kaminski, C. F. A Guide to Structured Illumination TIRF Microscopy at High Speed with Multiple Colors. J Vis Exp. (111), e53988 (2016).
- Huang, X., et al. long-term, super-resolution imaging with Hessian structured illumination microscopy. Nat Biotechnol. 36 (5), 451-459 (2018).
- Ando, R., et al. StayGold variants for molecular fusion and membrane-targeting applications. Nat Methods. 21 (4), 648-656 (2024).
- Hirano, M., et al. A highly photostable and bright green fluorescent protein. Nat Biotechnol. 40 (7), 1132-1142 (2022).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten