Method Article
Complemento Micro-Manager de código abierto para imágenes de visualización en vivo de dipolos fluorescentes
En este artículo
Resumen
Desarrollamos un complemento Micro-Manager de código abierto, que permite la observación en vivo de dipolos fluorescentes en un microscopio de iluminación estructurada. El plugin admite la observación de la orientación del dipolo 2D y 3D.
Resumen
La microscopía de polarización de fluorescencia (FPM) puede obtener imágenes de la posición y la orientación del dipolo de los fluoróforos. A pesar de los logros de la microscopía de polarización de fluorescencia de superresolución, su dependencia de la posterior adquisición dificulta la observación en tiempo real. La microscopía de iluminación estructurada polarizada (pSIM) ofrece imágenes de súper resolución de dipolos fluorescentes con una velocidad de imagen rápida y es adecuada para aplicaciones de células vivas. Desarrollamos una implementación de código abierto para la reconstrucción en tiempo real de imágenes de polarización y visualización de los dipolos fluorescentes. Además, ampliamos el método para lograr el mapeo de orientación 3D (3DOM), ampliando su utilidad para estudios biológicos complejos. Además, hemos presentado una introducción completa a la extensión de un microscopio SIM existente en imágenes de polarización y hemos proporcionado una guía de configuración detallada de Micro-Manager 2.0 para controlar el microscopio, lo que permite una vista previa en tiempo real de las imágenes polarizadas. Además, hemos proporcionado el código de MATLAB para la reconstrucción completa que abarca tanto pSIM como 3DOM. Esta guía completa tiene como objetivo ayudar a los principiantes a dominar rápidamente y comenzar fácilmente con las operaciones.
Introducción
La microscopía de polarización de fluorescencia (FPM) se ha convertido en una técnica potente para obtener imágenes simultáneas de la posición y la orientación dipolar de los fluoróforos, ofreciendo una visión profunda de las imágenes biológicas 1,2. Al facilitar la observación de la dirección de las orientaciones de las biomoléculas, FPM revela la intrincada disposición de macromoléculas como la actina 3,4,5, el microtúbulo5, la septina6, el filamentode ADN 7-9, el complejo de poros nucleares10 y las proteínas de membrana11. Sus capacidades de alta velocidad, no invasivas y compatibles con células vivas permiten el seguimiento de la dinámica de rotación molecular con alta resolución temporal11,12. Cuando se integra con sondas de biofuerza, FPM no solo mapea magnitudes de fuerza a resolución subcelular, sino que también mide direcciones de fuerza, avanzando así en nuestra comprensión de los procesos biomecánicos al revelar la dirección de las fuerzas13.
En las últimas décadas, la microscopía de polarización de fluorescencia de superresolución ha experimentado una rápida evolución. Un avance notable en este campo es la microscopía de localización de orientación de una sola molécula, también denominada SMOLM, que puede localizar tanto la posición como la orientación de los fluoróforos, permitiendo así la localización multidimensional. La medición de la polarización en SMOLM se puede realizar utilizando la modulación de excitación de polarización7, la detección polarizada multicanal3 o la función de dispersión de puntos sensible a la polarización (PSF) diseñada14. A pesar de que SMOLM logra una resolución espacial del orden de decenas de nanómetros y mide la polarización de moléculas individuales, sufre de un tiempo de imagen prolongado. Esto se debe a los ciclos repetitivos de parpadeo y localización de fluoróforos, que plantean un desafío para las imágenes de velocidad de video y las aplicaciones de células vivas.
Por el contrario, la microscopía de iluminación estructurada polarizada (pSIM) ofrece una resolución espacial de aproximadamente hasta 100 nm, junto con la adquisición de información de polarización con el mismo conjunto de datos SIM. En particular, pSIM puede alcanzar velocidades de imagen de velocidad de video y es altamente compatible con imágenes de células vivas, sin requisitos estrictos para las moléculas fluorescentes. Recientemente, pSIM ha revelado con éxito la estructura del anillo de actina en el esqueleto periódico asociado a la membrana (MPS)5 y ha permitido el mapeo de superresolución de las fuerzas biológicas13.
Sin embargo, pSIM requiere la reconstrucción de la imagen posterior a la adquisición, lo que impide la visualización en tiempo real de los resultados de polarización. Este retraso dificulta la observación inmediata de los fenómenos biológicos, lo que impide a los investigadores capturar rápidamente los fenómenos biológicos de interés y realizar ajustes en tiempo real a las muestras y las condiciones de imagen. Para abordar esta limitación, hemos desarrollado una implementación de código abierto que facilita la adquisición de imágenes y la reconstrucción en tiempo real y la visualización de los resultados de polarización, basada en la plataforma ImageJ y Micro-Manager (https://github.com/KarlZhanghao/live-pol-imaging).
Además, mientras que pSIM se ha limitado a proporcionar información de polarización 2D en el plano, recientemente hemos ampliado sus capacidades para lograr el mapeo de orientación 3D utilizando casi el mismo equipo15, denominado mapeo de orientación 3D (3DOM). Este software de código abierto también proporciona el control, la reconstrucción y la visualización de 3DOM. Los módulos de reconstrucción y visualización también son compatibles con la aplicación de seguimiento de orientación de molécula única. Todas estas funcionalidades mejoran la utilidad de las imágenes de polarización en estudios biológicos complejos.
Protocolo
1. Ampliación de un microscopio de iluminación estructurada existente para la obtención de imágenes de polarización
- Prepara un microscopio SIM.
NOTA: Suponemos que los lectores tienen una cierta base en la configuración del microscopio y ya poseen un microscopio de luz estructurada. Si carece de experiencia relevante, consulte este documento, que proporciona una descripción detallada de cómo construir un microscopio SIM16. La unidad de control de polarización de "HWP+WQP+LCVR" puede ser sustituida por una placa de onda de vórtice de pizza en nuestro pSIM work5 o una placa de media onda de pizza17. - Mida las relaciones de extinción de polarización de tres direcciones de rayas cargando diferentes patrones en el modulador de luz espacial (SLM). Coloque y gire un polarizador justo después del objetivo y use un medidor de potencia para medir la potencia del láser. Para pSIM, gire la placa de onda para mantener la polarización s de tres direcciones con una relación de extinción > 10.
- Para 3DOM, gire el HWP2 para mantener la polarización p de seis direcciones. Reemplace la máscara espacial SIM por la máscara espacial 3DOM para permitir el paso de haces de 1 orden de seis direcciones (consulte la Figura 1B).
NOTA: se requiere polarización s en pSIM, mientras que la polarización p se requiere en 3DOM. La configuración detallada se incluye en otra parte para pSIM5 y 3DOM15.
2. Configuración de Micro-Manager
- Descargue la versión Micro-Manager 2.0 desde el sitio web oficial, instale el software siguiendo las instrucciones.
- Prepare los controladores de dispositivo y el software correspondientes para conectarse correctamente al sistema Micro-Manager. En este sistema de microscopio, utilice Micro-Manager para controlar la cámara, la etapa de traslación, el láser, la placa DAQ y el microscopio.
NOTA: Los instrumentos compatibles se enumeran en https://micro-manager.org/Device_Support. Consulte la Tabla de materiales para obtener detalles sobre los instrumentos que se van a conectar. Instale los controladores o el software oficial/de soporte y conecte el instrumento a la computadora mediante un cable USB. - Añada los instrumentos a Micro-Manager mediante el Asistente de configuración de hardware.
- Abra el software y seleccione Ninguno en la lista desplegable inicial.
- Seleccionar dispositivos | Asistente de configuración de hardware... | Cree una nueva configuración y haga clic en Siguiente para ingresar a la página de configuración.
- Localice el complemento Camera / Laser / Stage / DAQ / Microscope en la lista desplegable de hardware y haga clic en Agregar | DE ACUERDO | Siguiente.
- Vaya a Seleccionar dispositivos predeterminados y elija la configuración del obturador automático. Establezca la cámara predeterminada, el obturador predeterminado y la etapa de enfoque predeterminada. Haga clic en Siguiente hasta que se alcance Guardar configuración y salida , y guarde el nombre del archivo de configuración. Haga clic en Finalizar.
- Configuración común de plantillas
- Guarde una plantilla para el modo en vivo y el modo de ajuste de SIM.
- Configure el tiempo de exposición y el modo de disparo de la siguiente manera: elija Grupo ' +' en Ajustes de configuración, asigne un nombre al grupo como 'Modo', seleccione Modo de exposición y disparo, haga clic en Aceptar.
- Elija Preset '+' para agregar diferentes presets para el grupo 'Mode'. Asigne un nombre preestablecido "live" para los ajustes de exposición del modo en vivo y otro "SIM" para el modo SIM. Por ejemplo, en el modo 'en vivo', establezca el tiempo de exposición en 15 ms, mientras que para el modo de ajuste de SIM, establezca 10 ms para el tiempo de exposición y el modo de disparo es Estándar (Superposición).
NOTA: Se incluye un archivo de configuración (MMConfig_psim_demo.cfg) en nuestro repositorio de código, que puede ser cargado directamente por Micro-Manager 2.0.
3. Calibración del sistema con perlas fluorescentes
- Sumerja los cubreobjetos en etanol al 75%, enjuague los cubreobjetos 3 veces con agua desionizada (ddH2O) y séquelos.
- Agite las perlas fluorescentes de 100 nm (diluidas a 1:1.000 con solución salina tamponada con fosfato [PBS]) y pipetee la suspensión directamente sobre los cubreobjetos. Incubar durante 5-10 minutos en un ambiente oscuro, luego lavar suavemente con PBS.
- Añada 20 μl de medio de montaje en el portaobjetos del microscopio y coloque el cubreobjetos sobre él, asegurándose de evitar cualquier burbuja. Mantenga la temperatura a 4 °C y mantenga el portaobjetos en la oscuridad, de modo que el portaobjetos con perlas fluorescentes de 100 nm esté preparado para la calibración del sistema.
- Para pSIM, adquiera tres imágenes de las tres fases diferentes de las perlas fluorescentes. Un código de MATLAB personalizado (Bead_calib.m) toma las imágenes como entrada y genera dos imágenes de calibración (calib1.tif, calib2.tif) para su posterior análisis.
NOTA: Como se demostró anteriormente5, el experimento pSIM se puede realizar en la mayoría de los sistemas comerciales. En los microscopios SIM caseros o comerciales, se requiere una calibración del sistema para eliminar el error de medición de los dipolos fluorescentes, causado por la iluminación no uniforme durante tres direcciones de patrón. El experimento de calibración asume la polarización isotrópica de las perlas fluorescentes y solo requiere las imágenes en bruto durante la adquisición de SIM. El procedimiento de calibración detallado está incluido en nuestro trabajo original5. La salida de dos imágenes de calibración calib1.tif y calib2.tif es necesaria para la reconstrucción de pSIM, que indica la energía de iluminación píxel a píxel de la dirección del patrón 2; 3 se refiere a la dirección del patrón 1.
4. Preparación de la muestra: actina en células fijas
- Sumerja los cubreobjetos en etanol al 75%, enjuague los cubreobjetos 3 veces con ddH2O. Coloque los cubreobjetos en una placa de cultivo celular de seis pocillos.
- Cultivo de células U2OS (línea celular ATCC HTB-96) en Medio de Águila Modificado de Dulbecco (DMEM) suplementado con 10% (v/v) de Suero Fetal Bovino (FBS) a 37 °C y 5% de CO2 en cubreobjetos hasta alcanzar aproximadamente el 75% de confluencia.
- Lave suavemente las celdas 3 veces con PBS. Para la fijación celular, aplique paraformaldehído al 4% durante 15 min a temperatura ambiente. Después de la fijación, lave las células 3 veces nuevamente con PBS.
- Para mejorar la permeabilidad de la membrana, trate las células con Triton X-100 al 0,1% en PBS durante 5 minutos, luego lave las células 3 veces con PBS.
- Disuelva la faloidina fluorescente con 150 μL de DMSO para la solución madre (66 μM). Prepare la solución de trabajo del colorante Phalloidin diluyendo 5 μL de la solución madre en 995 μL de PBS. Incubar las células en la oscuridad con una solución de trabajo de colorante de faloidina a temperatura ambiente durante ~ 1 h. Lave las celdas durante 3 x 3 minutos con PBS.
- Añada 20 μL de medio de montaje en el portaobjetos del microscopio. Coloque con cuidado el cubreobjetos en la corredera sin que queden burbujas. Guarde el portaobjetos a 4 °C y manténgalo en un lugar oscuro.
5. Preparación de la muestra: λ-DNA in vitro
- Disuelva 0,5 g de polimetilmetacrilato en 10 mL de materiales base para dentaduras postizas. Después de una disolución completa, aplique uniformemente la solución sobre la superficie del portaobjetos y luego espere a que se evapore para formar una película.
- Diluir 0,3 μL de λ-ADN y 32 μL de 1.000x Tinción de ácido nucleico SYTOX Orange en 968 μL de PBS. Agregue 1 μL de la solución preparada al portaobjetos y deje que se seque.
- Selle el cubreobjetos en el portaobjetos con el soporte para proteger y preservar la muestra. Guarde el portaobjetos a 4 °C en un ambiente oscuro.
6. Imágenes
- Coloque el cubreobjetos en el portamuestras y ajústelo al plano focal en el modo en vivo. Selecciona un ROI de 512 x 512 píxeles y ajusta el tiempo de exposición y la intensidad del láser.
- Abra el software de control independiente del modulador de luz espacial y elija la ruta de secuencia del patrón de iluminación adecuada para pSIM / 3DOM o modo en vivo.
NOTA: Para pSIM, el patrón de iluminación incluye tres ángulos de polarización diferentes y tres fases diferentes. Para 3DOM, el patrón de iluminación incluye seis ángulos de polarización diferentes. - Seleccione el modo de captura de SIM , seleccione Multi-D Acq., elija Puntos de tiempo y configure el Recuento (9 o 6 imágenes para pSIM o 3DOM) y el Intervalo (0 ms) en la ventana abierta. ¡Haz clic en Adquirir!
- Haga clic en TriggerSLM.bsh (se incluye un archivo de configuración en nuestro repositorio de código), la tarjeta de adquisición MyDaq activa el modulador de luz espacial para tomar las imágenes.
7. Plugin de visualización en vivo de pSIM y 3DOM
- Configure la FakeCamera y la ruta de acceso a la imagen: cargue el scrip (MMConfig_FakeCam_demo.cfg) haciendo clic en Dispositivos | Cargar configuración de hardware.... Configurar el directorio local de imágenes en Dispositivos | Explorador de propiedades de dispositivos... | Máscara de trayectoria de la cámara (Figura 2A-C).
NOTA: La parte de adquisición del script depende de la configuración del hardware, por lo que hemos proporcionado una versión (MMConfig_FakeCam_demo.cfg) utilizando el dispositivo FakeCamera para leer imágenes locales en el disco duro para su demostración. Los usuarios pueden realizar cambios en nuestra versión. Recuerde cambiar la ruta de lectura de la imagen (máscara Camera-Path). - Configure el Panel de acceso rápido haciendo clic en Herramientas | Panel de acceso rápido | Crear nuevo panel. Haga clic en el icono de Configuración en la esquina inferior izquierda, fármaco 'Ejecutar script' en la parte superior, que puede cargar el 'psim.bsh' y '3DOM.bsh' (Figura 2D) .
NOTA: El sistema de panel de acceso rápido permite a los usuarios crear ventanas personalizadas y acceder fácilmente a los controles de Micro-Manager, que es lo que se necesita con mayor frecuencia. También se pueden agregar canales 'Live', 'Snap' u otros, como se muestra en la Figura 2D. - Tomando pSIM como ejemplo, realice la reconstrucción de la imagen de polarización en tiempo real haciendo clic en 'psim.bsh'; observe las imágenes reconstruidas en tiempo real con resultados de polarización (Figura 2E). Recuerde cambiar la ruta de las imágenes de calibración en el archivo 'psim.bsh' (calibPath = " ruta al archivo de imágenes de calibración").
NOTA: El complemento de visualización en vivo es un script de infierno (psim.bsh y 3DOM.bsh), que se puede ejecutar en el Panel de script. El plugin adquiere nueve imágenes para pSIM o seis imágenes para 3DOM y calcula los resultados del mapeo de orientación en función de las imágenes de campo amplio. Las imágenes de mapeo de orientación se muestran para proporcionar información de polarización en tiempo real, lo que facilita la inspección inmediata, a pesar de su baja resolución.
8. Análisis de datos con reconstrucción de superresolución
- Instale MATLAB R2019b desde el sitio web (consulte la Tabla de materiales).
- Abra el software; para la reconstrucción de pSIM, obtenga nueve imágenes, que constan de tres fases diferentes para cada uno de los tres ángulos de orientación. Asegúrese de que los nombres de las nueve imágenes estén de '1.tif' a '9.tif', colóquelos en el archivo llamado 'Input' y ejecute el programa llamado 'PSIM.m'. Observe la imagen de campo amplio, la imagen SIM y la imagen pSIM reconstruidas con la rueda de colores.
NOTA: La reconstrucción de pSIM necesita dos pasos: un paso de SIM y un paso PM. Los datos se analizan mediante un programa de MATLAB escrito a medida para facilitar la depuración. Obtenga el código de reconstrucción de la carpeta 'reconsr' en GitHub. - Para 3DOM, capture seis figuras de direcciones de polarización diferentes. Combine las seis imágenes, asigne al archivo resultante el nombre 'Raw_data.tif' y ejecute el programa llamado 'Recon_3DOM.m'. Observe la imagen de campo amplio reconstruida y la imagen 3DOM con resultados acimutales y los resultados de ángulo polar.
NOTA: Durante el proceso de reconstrucción, puede ocurrir un error: no se reconoce la función o la variable 'bfopen'. 'bfopen' es una función de la biblioteca de bioformatos y se utiliza habitualmente para abrir y leer archivos de imagen. Escriba addpath ('path_to_bioformats_toolbox') en la ventana de comandos de MATLAB. Reemplace 'path_to_bioformats_toolbox' por la ruta de la carpeta de la biblioteca real. También agregamos la dependencia Bio-Formats al repositorio de GitHub.
Resultados
El método pSIM se puede realizar en microscopios SIM en función de la interferencia utilizando rayos láser polarizados s. La interferencia de polarización s es el tipo de SIM más utilizado y genera franjas de iluminación de alto contraste. El prototipo académico de una configuración de microscopio está incluido en el trabajo original de pSIM5. Brevemente presentado en la Figura 1, un modulador de luz espacial (SLM) genera el orden ±1 de haces difractivos y una placa de media onda mantiene la polarización s de tres direcciones. Mientras que otras alternativas a SIM pueden usar rejillas para generar haces difractivos o usar otros métodos de modulación de polarización para mantener la polarización s, estos microscopios también se pueden extender para imágenes de polarización.
Los microscopios 3DOM utilizan un sistema de imágenes ópticas similar (Figura 1A) con dos diferencias. En primer lugar, el 3DOM no requiere la interferencia de dos rayos láser. En cambio, solo usa los haces +1 para controlar la polarización de excitación. Como se demostró en el trabajo original15, 3DOM requiere un mínimo de seis direcciones para calcular la orientación del dipolo 3D. Por lo tanto, la máscara espacial 3DOM contiene seis direcciones, cada una con un agujero de alfiler (consulte la Figura 1B). Otra diferencia es que el 3DOM utiliza rayos láser polarizados p, que se pueden lograr girando el HWP2. El SLM genera luz difractada en varios ángulos oblicuos para cambiar la porción de polarización p.
En el sistema pSIM, la luz saliente se modula en luz polarizada s girando el ángulo de las placas de media onda. La máscara de pSIM es simétrica, lo que permite el paso simultáneo de ±1 orden de luz. Por el contrario, en 3DOM, al ajustar el ángulo de la placa de media onda, el láser se transforma en luz polarizada p. La máscara de 3DOM no es asimétrica y solo deja pasar la luz de orden +1. Al ajustar la frecuencia de raya del SLM, cambia el ángulo de difracción de los rayos láser. Por lo tanto, se modula la porción de polarización fuera del plano.
Al analizar dos dipolos fluorescentes adyacentes que comparten el mismo ángulo acimutal pero diferentes ángulos polares, tanto pSIM como 3DOM pueden distinguir dipolos cercanos dentro del límite de difracción. Además, 3DOM puede distinguir eficazmente diferentes dipolos con orientación 3D en el dominio de polarización (Figura 1C, D). Esto se debe a que la tecnología 3DOM puede obtener más información espacial cambiando el estado de polarización de la luz excitada para lograr una medición precisa de la orientación del dipolo en el espacio 3D. Por el contrario, la tecnología pSIM se basa principalmente en la información de polarización 2D y no puede aprovechar al máximo las características de orientación 3D; Por lo tanto, no proporciona información sobre la orientación de los dipolos en la dirección vertical.
Dado que los resultados del mapeo orientacional requieren posprocesamiento, desarrollamos y presentamos aquí el 'Complemento de visualización en vivo' ('psim.bsh' y '3DOM.bsh') para Micro-Manager 2.0. Para permitir que los usuarios de computadoras sin una cámara real conectada aprendan a usar el complemento, primero configuramos Micro-Manager con FakeCamera.In Figura 2, hemos detallado el proceso operativo para que pSIM logre efectos de polarización en tiempo real y los pasos operativos para 3DOM son similares. En la Figura 2A, se muestra la interfaz de software de Micro-Manager 2.0, junto con los pasos de configuración necesarios para configurar nuestro complemento para su funcionamiento. En la Figura 2B, se muestra la configuración de hardware utilizando dispositivos de demostración de FakeCamera y DStage. La configuración de hardware de FakeCamera es capaz de simular varios comportamientos de una cámara real, como la exposición, el enfoque y el balance de blancos, lo que permite a los desarrolladores realizar desarrollos y pruebas de software sin la necesidad de una cámara real. Los usuarios deben cambiar la ruta de lectura de la imagen en función de las circunstancias reales, como se muestra en la Figura 2C. La Figura 2D muestra un panel de acceso rápido, que puede contener accesos directos a una serie de funciones o configuraciones de uso común, lo que permite a los usuarios acceder y usar estas funciones más rápidamente. Los plugins de vista previa en tiempo real, 'psim.bsh' y '3DOM.bsh', se pueden cargar desde este panel. Los pasos operativos específicos se pueden encontrar en detalle en la sección 7 del protocolo. Los scripts 'psim.bsh' y '3DOM.bsh' se utilizan para generar instantáneamente las imágenes de mapeo de orientación. Este plugin 'psim.bsh' permite la visualización inmediata de los efectos de polarización durante la etapa de captura de imágenes pSIM. La Figura 2E ilustra un resultado típico de las imágenes de polarización de pSIM. La rueda de colores indica la relación entre la orientación del dipolo y el pseudocolor, lo que es útil para que los investigadores tomen decisiones inmediatas sobre qué imágenes capturar en función de los datos de polarización mostrados.
Las células U2OS se marcaron con faloidina y el proceso de reconstrucción de la imagen de pSIM se llevó a cabo con MATLAB. Se capturaron nueve fotos de tres estados de polarización diferentes y tres fases diferentes. Las flechas dobles verdes marcan la dirección de la luz polarizada en la Figura 3A. A partir de la intensidad de luz y oscuridad de la imagen, se puede determinar la orientación de polarización de los dipolos fluorescentes.
En la Figura 3B, la figura de la izquierda presenta los resultados de superresolución de la SIM, mientras que la figura de la derecha muestra los resultados de la pSIM. En particular, pSIM logra la misma capacidad de resolución espacial que SIM y ofrece el doble de resolución espacial en comparación con la microscopía de campo amplio. Al mapear colores específicos a distintas direcciones de filamento de actina, la rueda de colores proporciona una representación visual que nos permite interpretar la alineación de los dipolos fluorescentes dentro de los filamentos de actina de la célula. El color del filamento de actina muestra que los dipolos están aproximadamente alineados con la orientación del filamento.
La Fig. 4A muestra la distribución de la intensidad de la emisión bajo modulación de excitación por polarización; 3DOM puede producir seis modulaciones de polarización diferentes. En esta configuración, la luz excitada de la molécula fluorescente se modula cambiando el estado de polarización, lo que afecta la intensidad de emisión de la molécula. Esto permite a los investigadores inferir la orientación de las moléculas midiendo la intensidad de la fluorescencia en diferentes estados de polarización. La Figura 4B muestra la intensidad de fluorescencia del ADN-λ bajo diferentes estados de polarización en la Figura 4A. Las moléculas de λ-ADN exhiben diferentes intensidades de fluorescencia en diferentes estados de polarización, lo que indica que la orientación del dipolo de fluorescencia y el entorno tienen una influencia significativa en la intensidad de la señal de fluorescencia. La Figura 4C muestra imágenes de superresolución de λ-ADN logradas por 3DOM, que muestran tanto los resultados acimutales como los resultados del ángulo polar. Nuestras observaciones revelan que las moléculas de tinte estaban alineadas casi perpendicularmente a las hebras de ADN. Dado que el eje del ADN se dobla, la orientación promedio de las moléculas de tinte rotará en consecuencia. Los resultados de nuestras mediciones de orientación validan la inserción de fluoróforos STOYOX Orange entre bases de ADN adyacentes, y la alineación del momento dipolar de absorción es perpendicular al eje del ADN.
Por lo tanto, en 3DOM, se requiere una luz de excitación inclinada para obtener la información del eje z de la molécula objetivo. En nuestro sistema, SLM se utiliza para realizar el ajuste de la luz de iluminación. Combinado con las características del SLM, si el SLM está cargado con un patrón de rayas, la luz que sale del SLM se compondrá de 0 y ±1 órdenes de luz (y los órdenes de luz +1 y -1 son simétricos). Por lo tanto, usamos la luz de orden +1 como luz de excitación y utilizamos la máscara asimétrica para filtrar la luz de orden 0 y -1. Además, la luz de excitación oblicua se puede realizar no solo mediante el uso de SLM, sino también mediante el uso de un galvanómetro de barrido o moviendo la lente frente a la lente del objetivo lateralmente.
El uso de seis estados de polarización de excitación diferentes en 3DOM está dictado por su principio15. Al ajustar el modelo de imagen frontal, el modelo obtenido consta de seis partes. Por lo tanto, no es posible utilizar menos de seis ajustes de los estados de polarización, y un exceso de seis resultaría en redundancia de la información. El principio específico del 3DOM se muestra a continuación.
En coordenadas esféricas, caracterizamos la molécula fluorescente utilizando el ángel acimutal ρ y el ángulo polar η de la siguiente manera:

ρ es el ángulo en el sentido de las agujas del reloj entre la proyección de la molécula en el plano x-y y el eje x, y η es el ángulo entre la molécula y el eje z.
La luz de excitación polarizada p se caracteriza por:

Donde α y β representan el ángulo acimutal y el ángulo de incidencia oblicuo de la luz de excitación, respectivamente. La eficiencia de absorción de fotones es  , y suponiendo que la emisión de w dipolos en el plano focal se captura dentro del enésimo píxel, entonces el fotón fluorescente emitido por el dipolo se denota como:
, y suponiendo que la emisión de w dipolos en el plano focal se captura dentro del enésimo píxel, entonces el fotón fluorescente emitido por el dipolo se denota como:


Donde C representa la irradiancia de la muestra y  representa el estado de polarización de la luz de excitación como se describe a continuación:
representa el estado de polarización de la luz de excitación como se describe a continuación:

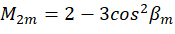

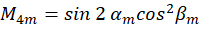
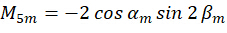
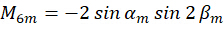
Combinando el coeficiente 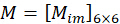 , la orientación
, la orientación 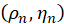 del enésimo píxel se puede resolver tomando las imágenes adquiridas
del enésimo píxel se puede resolver tomando las imágenes adquiridas  como entrada de la siguiente manera:
como entrada de la siguiente manera:
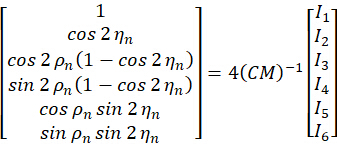
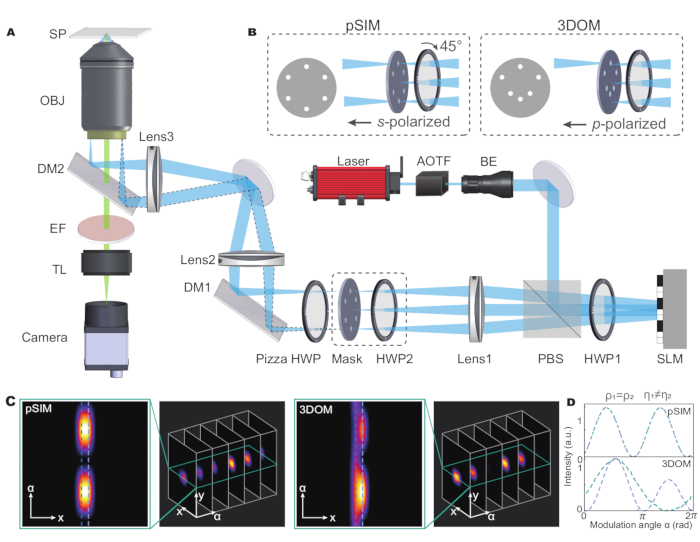
Figura 1: Ilustración esquemática de los microscopios pSIM y 3DOM. (A) Configuración experimental para pSIM y 3DOM. (B)Las principales diferencias entre los sistemas pSIM y 3DOM son la máscara y el HWP2. La máscara de la pSIM asegura que la luz de orden ±1 pase simultáneamente y la luz saliente se polariza en S girando el HWP2. La máscara de 3DOM solo pasa a través de la luz de orden +1 y la luz saliente está polarizada p. (C, D) Comparación de las variaciones de la intensidad de fluorescencia de emisión entre pSIM y 3DOM. Abreviaturas: pSIM = microscopía de iluminación estructurada polarizada; 3DOM = mapeo de orientación 3D; AOTF = filtro sintonizable acústico-óptico; BE = expansor de haz; PBS = divisor de haz de polarización; HWP = placa de media onda; SLM = modulador de luz espacial; DM = espejo dicroico; OBJ = objetivo; SP = plano de muestra; EF = filtro de emisiones; TL = lente de tubo. Esta figura fue modificada a partir de Zhanghao et al.5 y Zhong et al.15. Haga clic aquí para ver una versión más grande de esta figura.

Figura 2: Captura de pantalla del software y resultados representativos de pSIM. (A)La interfaz de Micro-Manager 2.0. Algunas opciones se pueden utilizar para configurar archivos. (B) Configuración del plugin live-view con FakeCamera y DStage. (C)Interfaz de selección de ruta de lectura de imágenes (máscara de ruta). (D) Configurar el Panel de Acceso Rápido y cargar 'Run Script' con 'psim.bsh' y '3DOM.bsh'. (E) La reconstrucción de la imagen en tiempo real demuestra los efectos de polarización. Las orientaciones de los dipolos son pseudocoloreadas. La rueda de colores indica la relación entre la orientación del dipolo y el pseudocolor. Abreviaturas: pSIM = microscopía de iluminación estructurada polarizada; 3DOM = mapeo de orientación 3D. Haga clic aquí para ver una versión más grande de esta figura.
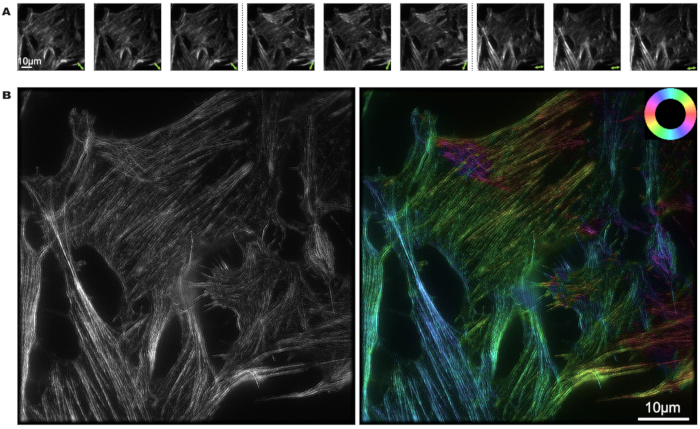
Figura 3: Imágenes pSIM de filamentos de actina en una célula U2OS marcada con faloidina. (A) Modulación del estado de polarización de la luz de excitación 3x en pSIM, capturando tres fases distintas para cada estado de polarización. Las flechas dobles indican la dirección de la luz polarizada. (B) Imagen SIM y pSIM de un filamento de actina en A, donde la imagen de la izquierda muestra los resultados de la SIM y la imagen de la derecha muestra los resultados de la pSIM con ángulo de polarización. La rueda de colores situada en la parte superior derecha ilustra la relación entre el pseudocolor y la orientación del dipolo. Barras de escala = 10 μm. Abreviatura: pSIM = microscopía de iluminación estructurada polarizada. Haga clic aquí para ver una versión más grande de esta figura.

Figura 4: SYTOX Naranja marcado con λ-DNA marcado con naranja imageizado por 3DOM. (A) Modulación del estado de polarización de la luz de excitación 6x en 3DOM. (B) La distribución de la intensidad de la emisión bajo la modulación de excitación por polarización de A. (C) Imagen 3DOM de una hebra de λ-ADN, donde la imagen de la izquierda muestra los resultados acimutales y la imagen de la derecha muestra los resultados del ángulo polar. Barras de escala = 10 μm. Abreviatura: 3DOM = mapeo de orientación 3D. Haga clic aquí para ver una versión más grande de esta figura.
Discusión
En nuestro estudio, desarrollamos un complemento que permite la vista previa en tiempo real de dos técnicas de imagen de polarización, pSIM y 3DOM. Ambas tecnologías se pueden realizar en un sistema SIM existente con ligeras modificaciones. Hemos proporcionado los pasos detallados para instalar el microscopio pSIM y 3DOM y configurar Micro-Manager para controlar el microscopio y demostrar cómo obtener los resultados de polarización de vista en vivo. Los resultados experimentales incluyen el filamento de actina fotografiado por pSIM y el λ-ADN fotografiado por 3DOM. La orientación de los dipolos de faloidina es aproximadamente paralela a la dirección de los filamentos de actina, mientras que la orientación de los fluoróforos naranjas STOYOX es perpendicular al eje del ADN. La microscopía de iluminación estructurada también puede lograr imágenes de alta resolución espacio-temporal del aparato de Golgi, la red de microtúbulos18 y el retículo endoplásmico19.
Hemos desarrollado plugins personalizados tanto para pSIM como para 3DOM; Los usuarios pueden elegir el programa de vista previa adecuado para su modo de disparo. Durante el proceso de ejecución del complemento de vista previa en tiempo real, si el complemento no se inicia correctamente, los usuarios primero deben verificar la configuración de la ruta en el archivo de configuración con respecto a la lectura de imágenes de cámaras virtuales, asegurándose de que la ruta se haya modificado con precisión a la ubicación real donde se almacenan las imágenes. Posteriormente, es esencial asegurarse de que la ruta para leer las imágenes de calibración en el complemento de vista previa en tiempo real también se haya actualizado en consecuencia. Las diferentes versiones de Micro-Manager pueden introducir nuevas funciones, pero también pueden dar lugar a problemas de compatibilidad. Por lo tanto, las operaciones específicas deben ajustarse de acuerdo con la versión real en uso.
La limitación de la tecnología radica en el hecho de que, aunque los actuales plugins de previsualización en tiempo real nos permiten observar la dirección de polarización de fluorescencia de los dipolos tanto en pSIM como en 3ODM, la resolución de las imágenes sigue siendo inadecuada, siendo simplemente suficiente para observaciones aproximadas. Sin embargo, las imágenes de alta resolución son particularmente cruciales durante el proceso de análisis y toma de decisiones. Para obtener detalles más finos, todavía necesitamos confiar en la plataforma MATLAB y emplear algoritmos avanzados de procesamiento de imágenes, como técnicas de reconstrucción de superresolución para el procesamiento en profundidad. La falta de reconstrucción completa con nuestro plugin se debe al bajo rendimiento del script beanshell utilizado. La reconstrucción completa con frijoles llevará mucho más tiempo que la adquisición de imágenes. El trabajo futuro incluye la implementación de la reconstrucción completa con Java y computación paralela y la inclusión de la característica en la vista previa en vivo.
Esperamos que este complemento mejore la eficiencia del experimento al observar el estado de polarización en tiempo real. La observación instantánea permite a los investigadores observar inmediatamente las orientaciones de los dipolos en los especímenes para que puedan localizar inmediatamente el objetivo de interés o un evento raro durante el seguimiento en tiempo real. El protocolo también proporciona la configuración de hardware y el control de software, lo que hace que pSIM y 3DOM sean más fáciles de usar.
Divulgaciones
Los autores declaran no tener conflictos de intereses.
Agradecimientos
Este trabajo contó con el apoyo del Programa Nacional de Investigación y Desarrollo Clave de China (2022YFC3401100).
Materiales
| Name | Company | Catalog Number | Comments |
| 100 nm Fluorescent beads | Invitrogen | F8801 | |
| 4% Formaldehyde solution | Invitrogen | R37814 | |
| Camera | Tucsen | Dhyana 400BSI V3 | https://www.tucsen.com/download-software/ |
| Denture base materials (Type I Thermally setting type, liquid) | New Century Dental | N/A | |
| Dulbecco’s Modified Eagle’s Medium | Gibco | C11995500BT | |
| Eclipse TE2000 Inverted Microscope | Nikon | TE2000 E | |
| Fetal Bovine Serum | Gibco | 10099141C | |
| MATLAB R2019b | MathWorks | Version R2019b | https://ww2.mathworks.cn/downloads/ |
| MetroCon V4.0 | Kopin | Version 4.0 | Software of Spatial light modulator |
| Micro-Manager 2.0 | μΜanager | Version 2.0 | Download Micro-Manager Latest Release |
| MS-2000 XYZ Automated Stage | Applied Scientific Instrumentation | MIM3 | https://www.asiimaging.com/support/downloads/usb-support-on-ms-2000-wk-controllers/ |
| myDAQ | National Instruments | 781325-01 | Software and Driver Downloads - NI |
| OBIS 561 nm LS 20 mW Laser | Coherent | 1325777 | |
| Phalloidin-AF568 | Invitrogen | A12380 | |
| Phosphate buffered saline | Corning | 21-040-CV | |
| Poly Methyl Methacrylate | Solarbio | M9810 | |
| ProLong Diamond | Invitrogen | P36980 | |
| Spatial light modulator | Kopin | SXGA-12 | |
| SYTOX orange nucleic acid stain | Invitrogen | S11368 | |
| Triton X-100 | Invitrogen | HFH10 | |
| Trypsin | Gibco | 25200056 | |
| λ-DNA | Invitrogen | S11368 |
Referencias
- Zhanghao, K., Gao, J., Jin, D., Zhang, X., Xi, P. Super-resolution fluorescence polarization microscopy. J Innov Opt Health Sci. 11 (01), 1730002(2018).
- Alonso, M. A., Brasselet, S. Polarization microscopy: from ensemble structural imaging to single-molecule 3D orientation and localization microscopy. Optica. 10 (11), 1486-1510 (2023).
- Valades Cruz, C. A., et al. Quantitative nanoscale imaging of orientational order in biological filaments by polarized superresolution microscopy. Pro Natl Acad Sci USA. 113 (7), E820-E828 (2016).
- Zhanghao, K., et al. Super-resolution dipole orientation mapping via polarization demodulation. Light Sci Appli. 5 (10), e16166(2016).
- Zhanghao, K., et al. Super-resolution imaging of fluorescent dipoles via polarized structured illumination microscopy. Nat Commun. 10 (1), 4694(2019).
- Vrabioiu, A. M., Mitchison, T. J. Structural insights into yeast septin organization from polarized fluorescence microscopy. Nature. 443 (7110), 466-469 (2006).
- Backer, A. S., Lee, M. Y., Moerner, W. E. Enhanced DNA imaging using super-resolution microscopy and simultaneous single-molecule orientation measurements. Optica. 3 (6), 659-666 (2016).
- Backer, A. S., et al. Single-molecule polarization microscopy of DNA intercalators sheds light on the structure of S-DNA. Sci Adv. 5 (3), eaav1083(2019).
- Hulleman, C. N., et al. Simultaneous orientation and 3D localization microscopy with a Vortex point spread function. Nat Commun. 12 (1), 5934(2021).
- Kampmann, M., Atkinson, C. E., Mattheyses, A. L., Simon, S. M. Mapping the orientation of nuclear pore proteins in living cells with polarized fluorescence microscopy. Nat Struct Mol Biol. 18 (6), 643-649 (2011).
- Lazar, J., Bondar, A., Timr, S., Firestein, S. J. Two-photon polarization microscopy reveals protein structure and function. Nat Methods. 8 (8), 684-690 (2011).
- Dong, B., et al. Parallel Three-Dimensional Tracking of Quantum Rods Using Polarization-Sensitive Spectroscopic Photon Localization Microscopy. ACS Photonics. 4 (7), 1747-1752 (2017).
- Blanchard, A., et al. Turn-key mapping of cell receptor force orientation and magnitude using a commercial structured illumination microscope. Nat Commun. 12 (1), 4693(2021).
- Toprak, E., et al. Defocused orientation and position imaging (DOPI) of myosin V. Proc Natl Acad Sci USA. 103 (17), 6495-6499 (2006).
- Zhong, S., et al. Three-dimensional dipole orientation mapping with high temporal-spatial resolution using polarization modulation. PhotoniX. 5 (1), 12(2024).
- Young, L. J., Ströhl, F., Kaminski, C. F. A Guide to Structured Illumination TIRF Microscopy at High Speed with Multiple Colors. J Vis Exp. (111), e53988(2016).
- Huang, X., et al. long-term, super-resolution imaging with Hessian structured illumination microscopy. Nat Biotechnol. 36 (5), 451-459 (2018).
- Ando, R., et al. StayGold variants for molecular fusion and membrane-targeting applications. Nat Methods. 21 (4), 648-656 (2024).
- Hirano, M., et al. A highly photostable and bright green fluorescent protein. Nat Biotechnol. 40 (7), 1132-1142 (2022).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados