Method Article
תוסף Micro-Manager בקוד פתוח להדמיה בתצוגה חיה של דיפולים פלואורסצנטיים
In This Article
Summary
פיתחנו תוסף Micro-Manager בקוד פתוח, המאפשר תצפית בתצוגה חיה של דיפולים פלואורסצנטיים במיקרוסקופ תאורה מובנה. התוסף תומך בהתבוננות בכיוון דיפול דו-ממדי ותלת-ממדי כאחד.
Abstract
מיקרוסקופ קיטוב פלואורסצנטי (FPM) יכול לדמות את המיקום והכיוון הדיפול של פלואורופורים. למרות ההישגים של מיקרוסקופ קיטוב פלואורסצנטי ברזולוציה גבוהה, ההסתמכות שלהם על לאחר הרכישה מעכבת תצפית בזמן אמת. מיקרוסקופ תאורה מובנה מקוטב (pSIM) מציע הדמיה ברזולוציה גבוהה של דיפולים פלואורסצנטיים עם מהירות הדמיה גבוהה ומתאים היטב ליישומי תאים חיים. פיתחנו יישום קוד פתוח לשחזור בזמן אמת של תמונות קיטוב והצגת הדיפולים הפלואורסצנטיים. בנוסף, הרחבנו את השיטה להשגת מיפוי אוריינטציה תלת מימדי (3DOM), והרחבנו את התועלת שלה למחקרים ביולוגיים מורכבים. יתר על כן, הצגנו מבוא יסודי להרחבת מיקרוסקופ SIM קיים על הדמיית קיטוב וסיפקנו מדריך תצורה מפורט של Micro-Manager 2.0 לשליטה במיקרוסקופ, המאפשר תצוגה מקדימה בזמן אמת של הדמיה מקוטבת. בנוסף, סיפקנו את קוד MATLAB לשחזור מלא המקיף הן את pSIM והן את 3DOM. מדריך מקיף זה נועד לסייע למתחילים לשלוט במהירות ולהתחיל בקלות בפעולות.
Introduction
מיקרוסקופ קיטוב פלואורסצנטי (FPM) התגלה כטכניקה רבת עוצמה להדמיה בו זמנית הן של המיקום והן של כיוון הדיפול של פלואורופורים, ומציעה תובנות עמוקות לגבי הדמיה ביולוגית 1,2. על ידי הקלה על תצפית כיוון על כיווני הביו-מולקולות, FPM חושף את הסידור המורכב של מקרו-מולקולות כגון אקטין 3,4,5, מיקרו-צינורית5, ספטין6, חוט DNA 7-9, קומפלקס נקבוביות גרעיניות10 וחלבוני ממברנה11. היכולות המהירות הגבוהה, הלא פולשניות והתואמות לתאים חיים מאפשרות מעקב אחר דינמיקת סיבוב מולקולרית ברזולוציה זמנית גבוהה11,12. בשילוב עם בדיקות ביו-כוח, FPM לא רק ממפה את גדלי הכוח ברזולוציה תת-תאית אלא גם מודד כיווני כוח, ובכך מקדם את הבנתנו את התהליכים הביומכניים על ידי חשיפת כיוון הכוחות13.
בעשורים האחרונים, מיקרוסקופ קיטוב פלואורסצנטי ברזולוציה גבוהה עבר אבולוציה מהירה. התקדמות בולטת בתחום זה היא מיקרוסקופ לוקליזציה של אוריינטציה של מולקולה אחת, המכונה גם SMOLM, שיכול למקם הן את המיקום והן את הכיוון של פלואורופורים, ובכך לאפשר לוקליזציה רב-ממדית. ניתן לבצע מדידת קיטוב ב-SMOLM באמצעות אפנון עירור קיטוב7, זיהוי מקוטב רב-ערוצי3, או פונקציית התפשטות נקודה רגישה לקיטוב (PSF) 14. למרות ש-SMOLM משיגה רזולוציה מרחבית בסדר גודל של עשרות ננומטר ומודדת את הקיטוב של מולקולות בודדות, היא סובלת מזמן הדמיה ממושך. זאת בשל המחזורים החוזרים ונשנים של מצמוץ פלואורופור ולוקליזציה, המהווים אתגר להדמיה בקצב וידאו ויישומי תאים חיים.
לעומת זאת, מיקרוסקופ תאורה מובנה מקוטב (pSIM) מציע רזולוציה מרחבית של כ-100 ננומטר, יחד עם רכישת מידע קיטוב עם אותו מערך נתונים של SIM. יש לציין כי pSIM יכול להשיג מהירויות הדמיה בקצב וידאו והוא תואם מאוד להדמיית תאים חיים, ללא דרישות מחמירות על מולקולות פלואורסצנטיות. לאחרונה, pSIM חשף בהצלחה את מבנה טבעת האקטין בשלד המחזורי הקשור לממברנה (MPS)5 ואיפשר מיפוי ברזולוציה גבוהה של כוחות ביולוגיים13.
עם זאת, pSIM דורש שחזור תמונה לאחר הרכישה, המונע הדמיה בזמן אמת של תוצאות הקיטוב. עיכוב זה מעכב את התצפית המיידית של תופעות ביולוגיות, ומונע מחוקרים ללכוד במהירות תופעות ביולוגיות מעניינות ולבצע התאמות בזמן אמת לדגימות ולתנאי הדמיה. כדי להתמודד עם מגבלה זו, פיתחנו יישום קוד פתוח המאפשר רכישת תמונות ושחזור והצגה בזמן אמת של תוצאות קיטוב, המבוססות על פלטפורמת ImageJ ו-Micro-Manager (https://github.com/KarlZhanghao/live-pol-imaging).
יתר על כן, בעוד ש-pSIM הוגבל לספק מידע על קיטוב דו-ממדי במישור, הרחבנו לאחרונה את יכולותיו להשיג מיפוי כיוון תלת-ממדי באמצעות כמעט אותו ציוד15, המכונה מיפוי כיוון תלת-ממדי (3DOM). תוכנת קוד פתוח זו מספקת גם שליטה, שחזור והדמיה של 3DOM. מודולי השחזור וההדמיה תואמים גם ליישום מעקב אחר כיוון מולקולה בודדת. כל הפונקציות הללו משפרות את התועלת של הדמיית קיטוב במחקרים ביולוגיים מורכבים.
Protocol
1. הרחבת מיקרוסקופ תאורה מובנה קיים להדמיית קיטוב
- הכן מיקרוסקופ SIM.
הערה: אנו מניחים שלקוראים יש בסיס מסוים בהגדרת מיקרוסקופ וכבר יש להם מיקרוסקופ אור מובנה. אם חסר לך ניסיון רלוונטי, עיין במאמר זה, המספק תיאור מפורט כיצד לבנות מיקרוסקופ SIM16. ניתן להחליף את יחידת בקרת הקיטוב של "HWP+WQP+LCVR" בצלחת גל מערבולת פיצה בעבודת pSIM5 שלנו או בצלחת חצי גל פיצה17. - מדוד יחסי הכחדת קיטוב של שלושה כיווני פסים על ידי טעינת דפוסים שונים על מאפנן אור מרחבי (SLM). הנח וסובב מקטב מיד לאחר המטרה והשתמש במד כוח כדי למדוד את עוצמת הלייזר. עבור pSIM, סובב את לוחית הגל כדי לשמור על קיטוב s של שלושה כיוונים עם יחס הכחדה > 10.
- עבור 3DOM, סובב את ה-HWP2 כדי לשמור על קיטוב p של שישה כיוונים. החלף את המסכה המרחבית של ה-SIM במסכה המרחבית 3DOM כדי לאפשר לאלומות מסדר אחד של שישה כיוונים לעבור דרכן (ראה איור 1B).
הערה: קיטוב s נדרש ב-pSIM ואילו קיטוב p נדרש ב-3DOM. הגדרה מפורטת כלולה במקום אחר עבור pSIM5 ו-3DOM15.
2. הגדרת מיקרו-מנהל
- הורד את גרסת Micro-Manager 2.0 מהאתר הרשמי, התקן את התוכנה על ידי ביצוע ההוראות.
- הכן את מנהלי ההתקנים והתוכנה המתאימים כדי להתחבר בהצלחה למערכת Micro-Manager. במערכת מיקרוסקופ זו, השתמש במיקרו-מנהל כדי לשלוט במצלמה, שלב התרגום, הלייזר, לוח ה-DAQ והמיקרוסקופ.
הערה: המכשירים הנתמכים מפורטים ב-https://micro-manager.org/Device_Support. עיין בטבלת החומרים לפרטים על המכשירים שיש לחבר. התקן מנהלי התקנים או תוכנה רשמית/תומכת וחבר את המכשיר למחשב באמצעות כבל USB. - הוסף את המכשירים ל-Micro-Manager באמצעות אשף קביעת תצורת החומרה.
- פתח את התוכנה ובחר ללא מהרשימה הנפתחת הראשונית.
- בחר מכשירים | אשף קביעת תצורת החומרה... | צור תצורה חדשה ולחץ על הבא כדי להיכנס לדף התצורה.
- אתר את התוסף מצלמה / לייזר / שלב / DAQ / מיקרוסקופ ברשימת החומרה הנפתחת ולחץ על הוסף | אישור | הבא.
- עבור אל בחר התקני ברירת מחדל ובחר הגדרת תריס אוטומטי. הגדר את מצלמת ברירת המחדל, תריס ברירת המחדל ושלב המיקוד המוגדר כברירת מחדל. לחץ על הבא עד שתגיע לשמירת תצורה ויציאה , ושמור את שם קובץ התצורה. לחץ על סיום.
- הגדרות תבנית נפוצות
- שמור תבנית עבור מצב חי ומצב הצמדה של SIM.
- הגדר את זמן החשיפה ומצב ההפעלה באופן הבא: בחר קבוצה '+' בהגדרות התצורה, תן שם לשם הקבוצה כ'מצב', בחר מצב חשיפה וטריגר, לחץ על אישור.
- בחר Preset '+' כדי להוסיף קביעות מוגדרות מראש שונות עבור הקבוצה 'Mode'. תן שם להגדרה מוגדרת מראש אחת 'חי' עבור הגדרות חשיפה במצב חי, ו-'SIM' אחר למצב SIM. לדוגמה, במצב 'חי', הגדר את זמן החשיפה ל-15 אלפיות השנייה, ואילו עבור מצב הצמדת SIM, הגדר 10 אלפיות השנייה לזמן החשיפה ומצב ההדק הוא סטנדרטי (חפיפה).
הערה: קובץ תצורה (MMConfig_psim_demo.cfg) כלול במאגר הקוד שלנו, אותו ניתן לטעון ישירות על ידי Micro-Manager 2.0.
3. כיול מערכת עם חרוזים פלואורסצנטיים
- טבלו את הכיסויים באתנול 75%, שטפו את הכיסויים פי 3 במים נטולי יונים (ddH2O) וייבשו אותם.
- מערבולת את חרוזי הפלואורסצנט של 100 ננומטר (מדולל ל-1:1,000 עם מי מלח חוצצים פוספט [PBS]) ופיפטה את המתלה ישירות על החלקות הכיסוי. יש לדגור במשך 5-10 דקות בסביבה חשוכה, ולאחר מכן לשטוף בעדינות עם PBS.
- הוסף 20 מיקרוליטר של אמצעי הרכבה על שקופית המיקרוסקופ, ומקם את הכיסוי מעליו, הקפד להימנע מבועות. שמור על הטמפרטורה על 4 מעלות צלזיוס ושמור על המגלשה בחושך, כך שהמגלשה עם חרוזי פלורסנט של 100 ננומטר מוכנה לכיול המערכת.
- עבור pSIM, רכשו שלוש תמונות של שלושת השלבים השונים של חרוזים פלואורסצנטיים. קוד MATLAB מותאם אישית (Bead_calib.m) לוקח את התמונות כקלט ומוציא שתי תמונות כיול (calib1.tif, calib2.tif) להמשך ניתוח.
הערה: כפי שהודגם קודם לכן5, ניתן לבצע את ניסוי ה-pSIM ברוב המערכות המסחריות. במיקרוסקופי SIM ביתיים או מסחריים, נדרש כיול מערכת כדי למנוע את שגיאת המדידה של דיפולים פלואורסצנטיים, הנגרמת על ידי תאורה לא אחידה במהלך שלושה כיווני תבנית. ניסוי הכיול מניח את הקיטוב האיזוטרופי של חרוזים פלואורסצנטיים ודורש רק את התמונות הגולמיות במהלך רכישת ה-SIM. נוהל כיול מפורט כלול בעבודה המקורית שלנו5. הפלט של שתי תמונות כיול calib1.tif ו-calib2.tif נדרש לשחזור pSIM, המציין את אנרגיית התאורה הפיקסלית של כיוון התבנית 2; 3 מתייחס לכיוון התבנית 1.
4. הכנת דגימה: אקטין בתאים קבועים
- טבלו את הכיסויים ב-75% אתנול, שטפו את הכיסויים פי 3 עם ddH2O. הניחו את הכיסויים בצלחת תרבית תאים עם שש בארות.
- תרבית תאי U2OS (קו תאים ATCC HTB-96) במדיום הנשר המותאם (DMEM) של Dulbecco בתוספת 10% (v/v) סרום בקר עוברי (FBS) ב-37 מעלות צלזיוס ו-5% CO2 על תלושי כיסוי עד שהם מגיעים לכ-75% מפגש.
- שטפו בעדינות את התאים פי 3 עם PBS. לקיבוע תאים, יש למרוח 4% פרפורמלדהיד למשך 15 דקות בטמפרטורת החדר. לאחר הקיבוע יש לשטוף את התאים שוב פי 3 עם PBS.
- כדי לשפר את חדירות הממברנה, טפל בתאים עם 0.1% Triton X-100 ב-PBS למשך 5 דקות, ולאחר מכן שטוף את התאים פי 3 עם PBS.
- ממיסים פלואורסצנטי פלואורסצנטי עם 150 מיקרוליטר של DMSO לתמיסת מלאי (66 מיקרומטר). הכן את תמיסת העבודה של צבע הפאלודין על ידי דילול 5 מיקרוליטר מתמיסת המלאי ב-995 מיקרוליטר של PBS. דגרו על התאים בחושך עם תמיסת עבודה של צבע פלואידין בטמפרטורת החדר למשך ~1 שעה. שוטפים את התאים למשך 3 x 3 דקות עם PBS.
- הוסף 20 מיקרוליטר של מדיום הרכבה על שקופית המיקרוסקופ. הנח בזהירות את הכיסוי על המגלשה ללא בועות. אחסן את השקופית בטמפרטורה של 4 מעלות צלזיוס ושמור אותה במקום חשוך.
5. הכנת דגימה: λ-DNA במבחנה
- ממיסים 0.5 גרם פולי מתיל מתאקרילט ב-10 מ"ל של חומרי בסיס תותבות. לאחר פירוק יסודי, יש למרוח את התמיסה באופן שווה על פני השקופית, ולאחר מכן להמתין עד שהיא תתאדה ליצירת סרט.
- לדלל 0.3 מיקרוליטר של λ-DNA ו-32 מיקרוליטר של 1,000x SYTOX כתם חומצת גרעין כתום ב-968 מיקרוליטר של PBS. הוסף 1 מיקרוליטר מהתמיסה המוכנה לשקופית והניח לה להתייבש.
- אטום את הכיסוי על המגלשה עם התושבת כדי להגן ולשמור על הדגימה. אחסן את השקופית בטמפרטורה של 4 מעלות צלזיוס בסביבה חשוכה.
6. הדמיה
- הנח את הכיסוי על הדגימה מחזיק והתאם למישור המוקד במצב חי. בחר החזר ROI של 512 x 512 פיקסלים והתאם את זמן החשיפה ועוצמת הלייזר.
- פתח את תוכנת הבקרה העצמאית של מאפנן האור המרחבי, ובחר את נתיב רצף דפוס התאורה המתאים עבור pSIM / 3DOM או מצב חי.
הערה: עבור pSIM, דפוס התאורה כולל שלוש זוויות קיטוב שונות ושלושה שלבים שונים. עבור 3DOM, דפוס התאורה כולל שש זוויות קיטוב שונות. - בחר את מצב לכידת ה-SIM , בחר Multi-D Acq., בחר נקודות זמן והגדר את הספירה (9 או 6 תמונות עבור pSIM או 3DOM) ואת המרווח (0 אלפיות השנייה) בחלון הפתוח. לחץ על Acquire!
- לחץ על TriggerSLM.bsh (קובץ תצורה כלול במאגר הקוד שלנו), כרטיס הרכישה של MyDaq מפעיל את אפנן האור המרחבי כדי לצלם את התמונות.
7. תוסף תצוגה חיה של pSIM ו-3DOM
- הגדר את נתיב הגישה ל-FakeCamera ולתמונה: טען את ה-scrip (MMConfig_FakeCam_demo.cfg) על ידי לחיצה על התקנים | טען תצורת חומרה.... הגדר את הספרייה המקומית של תמונות בהתקנים | דפדפן נכסי מכשירים... | מסיכת נתיב מצלמה (איור 2A-C).
הערה: חלק הרכישה של הסקריפט תלוי בהגדרת החומרה, לכן סיפקנו גרסה (MMConfig_FakeCam_demo.cfg) המשתמשת בהתקן FakeCamera כדי לקרוא תמונות מקומיות בדיסק הקשיח לצורך הדגמה. משתמשים יכולים לבצע שינויים בגרסה שלנו. זכור לשנות את נתיב קריאת התמונה (מסיכת נתיב מצלמה). - הגדר את לוח הגישה המהירה על ידי לחיצה על כלים | פנל לגישה מהירה | צור חלונית חדשה. לחץ על סמל ההגדרות בפינה השמאלית התחתונה, תרופה 'הפעל סקריפט' לחלק העליון, שיכול לטעון את 'psim.bsh' ו-'3DOM.bsh' (איור 2D).
הערה: מערכת לוח הגישה המהירה מאפשרת למשתמשים ליצור חלונות מותאמים אישית ולגשת בקלות לפקדים ב-Micro-Manager, הדרושים בתדירות הגבוהה ביותר. ניתן להוסיף גם 'חי', 'הצמד' או ערוצים אחרים , כפי שמוצג באיור 2D. - אם ניקח את pSIM כדוגמה, בצע שחזור תמונת קיטוב בזמן אמת על ידי לחיצה על 'psim.bsh'; התבונן בתמונות המשוחזרות בזמן אמת עם תוצאות קיטוב (איור 2E). זכור לשנות את נתיב תמונות הכיול בקובץ 'psim.bsh' (calibPath = " נתיב לקובץ תמונות כיול").
הערה: תוסף התצוגה החיה הוא סקריפט beanshell (psim.bsh ו-3DOM.bsh), שניתן להריץ בלוח הסקריפטים. התוסף רוכש תשע תמונות עבור pSIM או שש תמונות עבור 3DOM ומחשב את תוצאות מיפוי הכיוון על סמך תמונות השדה הרחב. תמונות מיפוי הכיוון מוצגות כדי לספק מידע קיטוב בזמן אמת, מה שמקל על בדיקה מיידית, למרות הרזולוציה הנמוכה שלו.
8. ניתוח נתונים עם שחזור ברזולוציה גבוהה
- התקן את MATLAB R2019b מהאתר (ראה טבלת החומרים).
- פתח את התוכנה; לשחזור pSIM, השג תשע תמונות, המורכבות משלושה שלבים שונים עבור כל אחת משלוש זוויות הכיוון. ודא ששמות תשע התמונות הם מ-'1.tif' עד '9.tif', הכנס אותן לקובץ בשם 'קלט' והפעל את התוכנית בשם 'PSIM.m'. התבונן בתמונת השדה הרחב, תמונת ה-SIM ותמונת ה-pSIM המשוחזרת באמצעות גלגל הצבעים.
הערה: השחזור של pSIM דורש שני שלבים: שלב SIM וצעד PM. הנתונים מנותחים על ידי תוכנת MATLAB הכתובה בהתאמה אישית לאיתור באגים קל יותר. קבל את קוד השחזור מתיקיית 'reconsr' ב- GitHub. - עבור 3DOM, צלמו שישה דמויות שונות של כיווני קיטוב. מיזגו את שש התמונות יחד, תנו לקובץ המתקבל את השם 'Raw_data.tif', והריצו את התוכנית בשם 'Recon_3DOM.m'. התבונן בתמונת השדה הרחב המשוחזרת ובתמונת 3DOM עם תוצאות אזימוטליות ותוצאות הזווית הקוטבית.
הערה: במהלך תהליך השחזור, עלולה להתרחש שגיאה' --'פונקציה או משתנה 'bfopen' אינם מזוהים. 'bfopen' היא פונקציה בספריית הפורמטים הביולוגיים והיא משמשת בדרך כלל לפתיחה וקריאה של קבצי תמונה. הזן addpath ('path_to_bioformats_toolbox') בחלון הפקודה של MATLAB. החלף את 'path_to_bioformats_toolbox' בנתיב תיקיית הספרייה בפועל. הוספנו גם את התלות ב-Bio-Formats למאגר GitHub.
תוצאות
ניתן לבצע את שיטת ה-pSIM במיקרוסקופי SIM המבוססים על ההפרעה באמצעות קרני לייזר מקוטבות s. הפרעות קיטוב s הן סוג ה- SIM הנפוץ ביותר ויוצרות פסי תאורה בעלי ניגודיות גבוהה. אב הטיפוס האקדמי של מערך מיקרוסקופ נכלל בעבודה המקורית של pSIM5. הוצג בקצרה באיור 1, מודולטור אור מרחבי (SLM) מייצר את הסדר ±1 של אלומות עקיפות וצלחת חצי גל פיצה שומרת על קיטוב s של שלושה כיוונים. בעוד שחלופות SIM אחרות עשויות להשתמש בסורגים כדי ליצור אלומות עקיפות או להשתמש בשיטות אפנון קיטוב אחרות כדי לשמור על קיטוב s, ניתן להרחיב את המיקרוסקופים הללו גם להדמיית קיטוב.
מיקרוסקופי 3DOM משתמשים במערכת הדמיה אופטית דומה (איור 1A) עם שני הבדלים. ראשית, ה-3DOM אינו דורש הפרעה של שתי קרני לייזר. במקום זאת, הוא משתמש רק בקורות +1 כדי לשלוט בקיטוב העירור. כפי שהודגם בעבודה המקורית15, 3DOM דורש מינימום של שישה כיוונים כדי לחשב את כיוון הדיפול התלת-ממדי. לכן, המסכה המרחבית 3DOM מכילה שישה כיוונים, כל אחד עם חור סיכה (ראו איור 1B). הבדל נוסף הוא שה-3DOM משתמש בקרני לייזר מקוטבות p, שניתן להשיג על ידי סיבוב ה-HWP2. ה-SLM מייצר אור עקיפה בזוויות אלכסוניות שונות כדי לשנות את החלק של קיטוב p.
במערכת pSIM, האור היוצא מווסת לאור מקוטב s על ידי סיבוב הזווית של לוחות חצי גל. המסכה של pSIM היא סימטרית, המאפשרת לאור מסדר ±1 לעבור בו זמנית. לעומת זאת, ב-3DOM, על ידי התאמת הזווית של לוחית חצי הגל, הלייזר הופך לאור מקוטב p. המסכה של 3DOM אינה א-סימטרית ומאפשרת רק ל-+1 לעבור אור. על ידי התאמת תדר הפסים של ה-SLM, הוא משנה את זווית העקיפה של קרני הלייזר. לפיכך, החלק של הקיטוב מחוץ למישור מווסת.
על ידי ניתוח שני דיפולים פלואורסצנטיים סמוכים החולקים את אותה זווית אזימוטלית אך זוויות קוטביות שונות, גם pSIM וגם 3DOM יכולים להבחין בדיפולים בקרבת מקום בתוך גבול העקיפה. יתר על כן, 3DOM יכול להבחין ביעילות בין דיפולים שונים עם אוריינטציה תלת-ממדית בתחום הקיטוב (איור 1C,D). הסיבה לכך היא שטכנולוגיית 3DOM יכולה להשיג מידע מרחבי רב יותר על ידי שינוי מצב הקיטוב של האור הנרגש כדי להשיג מדידה מדויקת של כיוון הדיפול במרחב תלת מימדי. לעומת זאת, טכנולוגיית pSIM מסתמכת בעיקר על מידע קיטוב דו-ממדי ואינה יכולה לנצל את מלוא היתרונות של מאפייני אוריינטציה תלת-ממדית; לכן הוא לא מצליח לספק מידע על כיוון הדיפולים בכיוון האנכי.
מכיוון שתוצאות המיפוי האוריינטלי דורשות עיבוד לאחר מכן, פיתחנו והצגנו כאן את 'תוסף Live-view' ('psim.bsh' ו-'3DOM.bsh') עבור Micro-Manager 2.0. כדי לאפשר למשתמשי מחשב ללא מצלמה אמיתית מחוברת ללמוד כיצד להשתמש בתוסף, אנו מגדירים תחילה את Micro-Manager עם FakeCamera.In איור 2, פירטנו את התהליך התפעולי עבור pSIM להשגת אפקטי קיטוב בזמן אמת, והשלבים התפעוליים עבור 3DOM דומים. באיור 2A, ממשק התוכנה של Micro-Manager 2.0 מוצג, יחד עם שלבי ההתקנה הדרושים הנדרשים כדי להגדיר את התוסף שלנו לפעולה. באיור 2B, אנו מציגים את תצורת החומרה באמצעות התקני הדגמה של FakeCamera ו-DStage. תצורת החומרה של FakeCamera מסוגלת לדמות התנהגויות שונות של מצלמה אמיתית, כגון חשיפה, מיקוד ואיזון לבן, ובכך לאפשר למפתחים לבצע פיתוח ובדיקות תוכנה ללא צורך במצלמה אמיתית. על המשתמשים לשנות את נתיב קריאת התמונה בהתבסס על הנסיבות בפועל כפי שמוצג באיור 2C. איור 2D מציג לוח גישה מהירה, שעשוי להכיל קיצורי דרך לסדרה של פונקציות או הגדרות נפוצות, המאפשר למשתמשים לגשת ולהשתמש בפונקציות אלה במהירות רבה יותר. ניתן לטעון את תוספי התצוגה המקדימה בזמן אמת, 'psim.bsh' ו-'3DOM.bsh', מלוח זה. ניתן למצוא את השלבים התפעוליים הספציפיים בפירוט בסעיף פרוטוקול 7. הסקריפטים 'psim.bsh' ו-'3DOM.bsh' משמשים ליצירה מיידית של תמונות מיפוי הכיוון. תוסף זה 'psim.bsh' מאפשר הדמיה מיידית של אפקטי הקיטוב במהלך שלב לכידת תמונת pSIM. איור 2E ממחיש תוצאה אופיינית של הדמיית קיטוב של pSIM. גלגל הצבעים מציין את הקשר בין כיוון הדיפול לצבע הפסאודו, מה שעוזר לחוקרים לקבל החלטות מיידיות לגבי אילו תמונות לצלם על סמך נתוני הקיטוב המוצגים.
תאי U2OS סומנו בפאלואידין, ותהליך שחזור התמונה של pSIM בוצע באמצעות MATLAB. תשע תמונות צולמו משלושה מצבי קיטוב שונים ושלושה שלבים שונים. החצים הכפולים הירוקים מסמנים את כיוון האור המקוטב באיור 3A. מעוצמת האור והחושך של התמונה, ניתן לקבוע את כיוון הקיטוב של הדיפולים הפלואורסצנטיים.
באיור 3B, האיור השמאלי מציג את תוצאות רזולוציית העל מ-SIM, בעוד שהאיור הימני מציג את תוצאות ה-pSIM. יש לציין כי pSIM משיג את אותה יכולת רזולוציה מרחבית כמו SIM ומציע רזולוציה מרחבית כפולה בהשוואה למיקרוסקופיה רחבה. על ידי מיפוי צבעים ספציפיים לכיווני חוטי אקטין מובחנים, גלגל הצבעים מספק ייצוג חזותי המאפשר לנו לפרש את היישור של דיפולים פלואורסצנטיים בתוך חוטי האקטין של התא. צבע חוט האקטין מראה כי דיפולים מיושרים בערך עם כיוון הנימה.
איור 4A מציג את התפלגות עוצמת הפליטה תחת אפנון עירור קיטוב; 3DOM יכול לייצר שישה אפנון קיטוב שונים. בתצורה זו, האור הנרגש של המולקולה הפלואורסצנטית מווסת על ידי שינוי מצב הקיטוב, ובכך משפיע על עוצמת הפליטה של המולקולה. זה מאפשר לחוקרים להסיק את הכיוון של מולקולות על ידי מדידת עוצמת הקרינה במצבי קיטוב שונים. איור 4B מראה את עוצמת הקרינה של λ-DNA במצבי קיטוב שונים באיור 4A. מולקולות λ-DNA מציגות עוצמות פלואורסצנטיות שונות במצבי קיטוב שונים, מה שמעיד על כך שלכיוון דיפול הקרינה והסביבה יש השפעה משמעותית על עוצמת אות הקרינה. איור 4C מציג הדמיה ברזולוציה גבוהה של λ-DNA שהושגה על ידי 3DOM, ומציג הן תוצאות אזימוטליות והן תוצאות זווית קוטבית. התצפיות שלנו מגלות שמולקולות הצבע היו מיושרות כמעט בניצב לגדילי ה-DNA. מכיוון שציר ה-DNA מתכופף, הכיוון הממוצע של מולקולות הצבע יסתובב בהתאם. תוצאות מדידות האוריינטציה שלנו מאמתות את החדרת הפלואורופורים הכתומים של STOYOX בין בסיסי ה-DNA הסמוכים, והיישור של מומנט דיפול הבליעה מאונך לציר ה-DNA.
ב-3DOM, לכן נדרש אור עירור מוטה כדי להשיג את מידע ציר ה-z של מולקולת המטרה. במערכת שלנו, SLM משמש למימוש התאמת אור התאורה. בשילוב עם המאפיינים של SLM, אם ה-SLM עמוס בתבנית פסים, האור היוצא מה-SLM יהיה מורכב מ-0 ו-±1 סדרי אור (וסדרי האור +1 ו-1 הם סימטריים). לכן, אנו משתמשים באור מסדר +1 כאור העירור ומשתמשים במסכה הא-סימטרית כדי לסנן את האור בסדר 0 ו-1. יתרה מכך, ניתן לממש את אור העירור האלכסוני לא רק על ידי שימוש ב-SLM, אלא גם על ידי שימוש בגלוונומטר סורק או על ידי הזזת העדשה מול עדשת האובייקט לרוחב.
שימוש בשישה מצבי קיטוב עירור שונים ב-3DOM מוכתב על ידי העיקרון שלו15. על ידי התאמת מודל ההדמיה קדימה, המודל המתקבל מורכב משישה חלקים. לכן, לא ניתן להשתמש בפחות משש התאמות של מצבי הקיטוב, ועודף של שש יגרום ליתירות מידע. העיקרון הספציפי של ה-3DOM מוצג להלן.
בקואורדינטות כדוריות, אנו מאפיינים את המולקולה הפלואורסצנטית באמצעות המלאך האזימוטלי ρ והזווית הקוטבית η כדלקמן:

ρ היא הזווית בכיוון השעון בין הקרנת המולקולה במישור ה-xy לבין ציר ה-x, ו-η היא הזווית בין המולקולה לציר ה-z.
אור העירור המקוטב p מאופיין כ:

כאשר α ו-β מייצגים את הזווית האזימוטלית ואת זווית ההתרחשות האלכסונית של אור העירור, בהתאמה. יעילות ספיגת הפוטון היא  , ובהנחה שפליטת דיפולים w במישור המוקד נלכדת בתוך הפיקסל ה-n, אז הפוטון הפלואורסצנטי הנפלט מהדיפול מסומן כ:
, ובהנחה שפליטת דיפולים w במישור המוקד נלכדת בתוך הפיקסל ה-n, אז הפוטון הפלואורסצנטי הנפלט מהדיפול מסומן כ:


כאשר C מייצג את קרינת הדגימה, ומייצג  את מצב הקיטוב של אור העירור כמתואר כדלקמן:
את מצב הקיטוב של אור העירור כמתואר כדלקמן:

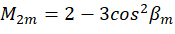

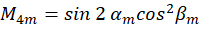
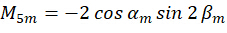
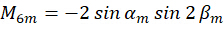
על ידי שילוב המקדם 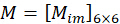 , ניתן לפתור את הכיוון
, ניתן לפתור את הכיוון 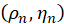 של הפיקסל ה-n על ידי לקיחת התמונות
של הפיקסל ה-n על ידי לקיחת התמונות  שנרכשו כקלט באופן הבא:
שנרכשו כקלט באופן הבא:
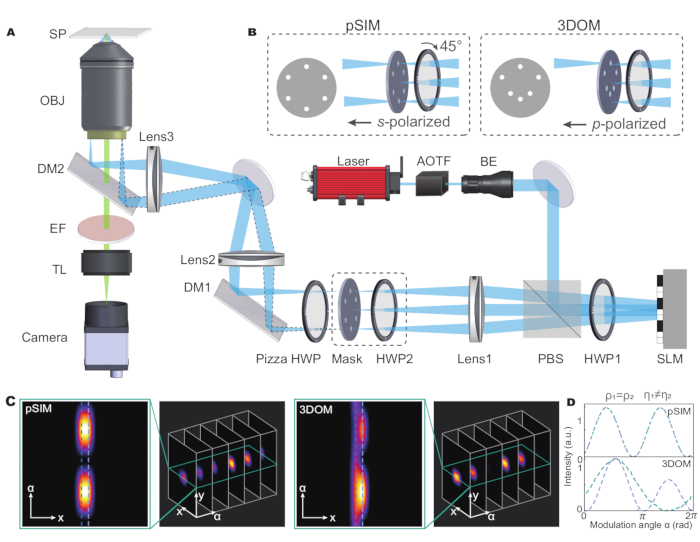
איור 1: איור סכמטי של מיקרוסקופי pSIM ו- 3DOM. (A) מערך ניסיוני עבור pSIM ו- 3DOM. (ב)ההבדלים העיקריים בין מערכות pSIM ו-3DOM הם המסכה ו-HWP2. המסכה של ה-pSIM מבטיחה שאור מסדר ±1 יעבור בו זמנית, והאור היוצא מקוטב ב-s על ידי סיבוב ה-HWP2. המסכה של 3DOM עוברת רק דרך אור מסדר +1, והאור היוצא מקוטב p. (ג, ד) השוואה בין וריאציות עוצמת הקרינה של הפליטה בין pSIM ל-3DOM. קיצורים: pSIM = מיקרוסקופ תאורה מובנה מקוטב; 3DOM = מיפוי כיוון תלת מימדי; AOTF = מסנן מתכוונן אקוסטו-אופטי; BE = מרחיב קרן; PBS = מפצל קרן קיטוב; HWP = צלחת חצי גל; SLM = מודולטור אור מרחבי; DM = מראה דיכרואית; OBJ = אובייקטיבי; SP = מישור מדגם; EF = מסנן פליטה; TL = עדשת צינור. נתון זה שונה מ-Zhanghao et al.5 ו-Zhong et al.15. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.

איור 2: צילום מסך תוכנה ותוצאות מייצגות של pSIM. (A) הממשק של Micro-Manager 2.0. אפשרויות מסוימות עשויות לשמש לקביעת תצורה של קבצים. (ב) תצורה של תוסף תצוגה חיה עם FakeCamera ו-DStage. (ג)ממשק הבחירה של נתיב קריאת תמונה (מסיכת נתיב). (ד) הגדרת לוח הגישה המהירה וטעינת 'Run Script' עם 'psim.bsh' ו-'3DOM.bsh'. (E) שחזור התמונה בזמן אמת מדגים את השפעות הקיטוב. כיווני הדיפול הם פסאודו-צבעוניים. גלגל הצבעים מציין את הקשר בין כיוון הדיפול לפסאודו-צבע. קיצורים: pSIM = מיקרוסקופ תאורה מובנה מקוטב; 3DOM = מיפוי כיוון תלת מימדי. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.
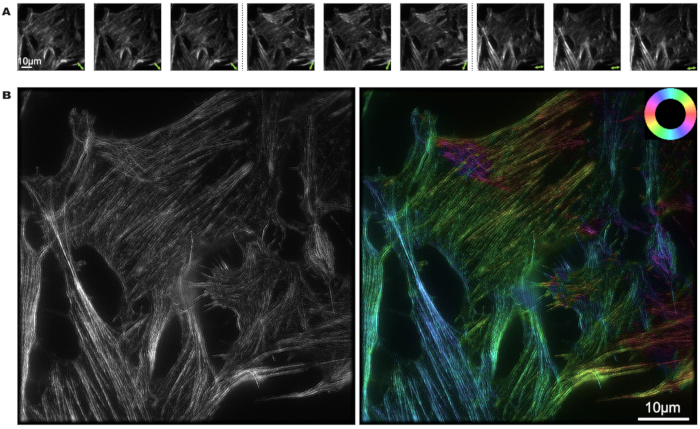
איור 3: הדמיית pSIM של חוטי אקטין בתא U2OS עם תווית פלואידין. (A) אפנון של מצב קיטוב אור העירור 3x ב-pSIM, לכידת שלושה פאזות נפרדות עבור כל מצב קיטוב. החצים הכפולים מציינים את כיוון האור המקוטב. (B) תמונת SIM ו-pSIM של חוטי אקטין ב-A, כאשר התמונה השמאלית מציגה תוצאות SIM והתמונה הימנית מציגה תוצאות pSIM עם זווית קיטוב. גלגל הצבעים הממוקם בפינה הימנית העליונה ממחיש את הקשר בין כיוון הפסאודו-צבע לכיוון הדיפול. פסי קנה מידה = 10 מיקרומטר. קיצור: pSIM = מיקרוסקופ תאורה מובנה מקוטב. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.

איור 4: SYTOX כתום עם תווית כתומה λ-DNA שצולמה על ידי 3DOM. (A) אפנון של מצב קיטוב אור העירור 6x ב-3DOM. (B) התפלגות עוצמת הפליטה תחת אפנון עירור קיטוב של A. (C) תמונת 3DOM של גדיל λ-DNA, כאשר התמונה השמאלית מציגה תוצאות אזימוטליות והתמונה הימנית מציגה תוצאות זווית קוטבית. פסי קנה מידה = 10 מיקרומטר. קיצור: 3DOM = מיפוי כיוון תלת מימדי. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.
Discussion
במחקר שלנו, פיתחנו תוסף המאפשר תצוגה מקדימה בזמן אמת של שתי טכניקות הדמיית קיטוב, pSIM ו-3DOM. ניתן לבצע את שתי הטכנולוגיות במערכת SIM קיימת עם שינוי קל. סיפקנו את השלבים המפורטים להתקנת מיקרוסקופ pSIM ו-3DOM והקמנו את Micro-Manager כדי לשלוט במיקרוסקופ ולהדגים כיצד להשיג את תוצאות הקיטוב בתצוגה חיה. תוצאות הניסוי כוללות את חוט האקטין שצולם על ידי pSIM ו-λ-DNA שצולם על ידי 3DOM. הכיוון של דיפולים הפאלואידין מקביל בערך לכיוון חוטי האקטין, בעוד שהכיוון של STOYOX Orange fluorophores מאונך לציר ה-DNA. מיקרוסקופ תאורה מובנה יכול גם להשיג הדמיה ברזולוציה מרחבית-זמנית גבוהה של מנגנון גולגי, רשת המיקרו-צינורות18 והרטיקולום האנדופלזמי19.
פיתחנו תוספים מותאמים אישית הן עבור pSIM והן עבור 3DOM; משתמשים יכולים לבחור את תוכנית התצוגה המקדימה המתאימה למצב הצילום שלהם. במהלך תהליך הפעלת תוסף התצוגה המקדימה בזמן אמת, אם התוסף לא מצליח להפעיל כראוי, על המשתמשים לבדוק תחילה את הגדרת הנתיב בקובץ התצורה לגבי קריאת תמונות מצלמה וירטואלית, ולוודא שהנתיב שונה במדויק למיקום בפועל שבו מאוחסנות התמונות. לאחר מכן, חיוני לוודא שגם הנתיב לקריאת תמונות כיול בתוסף התצוגה המקדימה בזמן אמת עודכן בהתאם. גרסאות שונות של Micro-Manager עשויות להציג תכונות חדשות, אך הן עלולות גם לגרום לבעיות תאימות. לכן, יש להתאים פעולות ספציפיות בהתאם לגרסה בפועל בשימוש.
מגבלת הטכנולוגיה נעוצה בעובדה שלמרות שתוספי התצוגה המקדימה הנוכחיים בזמן אמת מאפשרים לנו לצפות בכיוון הקיטוב הקרינה של דיפולים הן ב-pSIM והן ב-3ODM, הרזולוציה של התמונות עדיין אינה מספקת, והיא מספיקה רק לתצפיות גסות. עם זאת, תמונות ברזולוציה גבוהה הן חיוניות במיוחד במהלך תהליך הניתוח וקבלת ההחלטות. כדי לקבל פרטים עדינים יותר, אנו עדיין צריכים להסתמך על פלטפורמת MATLAB ולהשתמש באלגוריתמים מתקדמים לעיבוד תמונה כגון טכניקות שחזור ברזולוציה גבוהה לעיבוד מעמיק. היעדר שחזור מלא עם התוסף שלנו נובע מהביצועים הנמוכים של סקריפט השעועית בו נעשה שימוש. השחזור המלא באמצעות שעועית ייקח הרבה יותר זמן מרכישת תמונה. העבודה העתידית כוללת יישום השחזור המלא עם Java ומחשוב מקבילי והכללת התכונה בתצוגה המקדימה החיה.
אנו מקווים שתוסף זה ישפר את יעילות הניסוי על ידי התבוננות במצב הקיטוב בזמן אמת. התצפית המיידית מאפשרת לחוקרים לצפות באופן מיידי בכיווני דיפול בדגימות כך שיוכלו לאתר מיד את יעד העניין או אירוע נדיר במהלך מעקב בזמן חי. הפרוטוקול מספק גם את הגדרת החומרה ובקרת התוכנה, מה שהופך את pSIM ו-3DOM לידידותיים יותר למשתמש.
Disclosures
המחברים מצהירים שאין ניגודי אינטרסים.
Acknowledgements
עבודה זו נתמכה על ידי תוכנית המחקר והפיתוח הלאומית של סין (2022YFC3401100).
Materials
| Name | Company | Catalog Number | Comments |
| 100 nm Fluorescent beads | Invitrogen | F8801 | |
| 4% Formaldehyde solution | Invitrogen | R37814 | |
| Camera | Tucsen | Dhyana 400BSI V3 | https://www.tucsen.com/download-software/ |
| Denture base materials (Type I Thermally setting type, liquid) | New Century Dental | N/A | |
| Dulbecco’s Modified Eagle’s Medium | Gibco | C11995500BT | |
| Eclipse TE2000 Inverted Microscope | Nikon | TE2000 E | |
| Fetal Bovine Serum | Gibco | 10099141C | |
| MATLAB R2019b | MathWorks | Version R2019b | https://ww2.mathworks.cn/downloads/ |
| MetroCon V4.0 | Kopin | Version 4.0 | Software of Spatial light modulator |
| Micro-Manager 2.0 | μΜanager | Version 2.0 | Download Micro-Manager Latest Release |
| MS-2000 XYZ Automated Stage | Applied Scientific Instrumentation | MIM3 | https://www.asiimaging.com/support/downloads/usb-support-on-ms-2000-wk-controllers/ |
| myDAQ | National Instruments | 781325-01 | Software and Driver Downloads - NI |
| OBIS 561 nm LS 20 mW Laser | Coherent | 1325777 | |
| Phalloidin-AF568 | Invitrogen | A12380 | |
| Phosphate buffered saline | Corning | 21-040-CV | |
| Poly Methyl Methacrylate | Solarbio | M9810 | |
| ProLong Diamond | Invitrogen | P36980 | |
| Spatial light modulator | Kopin | SXGA-12 | |
| SYTOX orange nucleic acid stain | Invitrogen | S11368 | |
| Triton X-100 | Invitrogen | HFH10 | |
| Trypsin | Gibco | 25200056 | |
| λ-DNA | Invitrogen | S11368 |
References
- Zhanghao, K., Gao, J., Jin, D., Zhang, X., Xi, P. Super-resolution fluorescence polarization microscopy. J Innov Opt Health Sci. 11 (01), 1730002 (2018).
- Alonso, M. A., Brasselet, S. Polarization microscopy: from ensemble structural imaging to single-molecule 3D orientation and localization microscopy. Optica. 10 (11), 1486-1510 (2023).
- Valades Cruz, C. A., et al. Quantitative nanoscale imaging of orientational order in biological filaments by polarized superresolution microscopy. Pro Natl Acad Sci USA. 113 (7), E820-E828 (2016).
- Zhanghao, K., et al. Super-resolution dipole orientation mapping via polarization demodulation. Light Sci Appli. 5 (10), e16166 (2016).
- Zhanghao, K., et al. Super-resolution imaging of fluorescent dipoles via polarized structured illumination microscopy. Nat Commun. 10 (1), 4694 (2019).
- Vrabioiu, A. M., Mitchison, T. J. Structural insights into yeast septin organization from polarized fluorescence microscopy. Nature. 443 (7110), 466-469 (2006).
- Backer, A. S., Lee, M. Y., Moerner, W. E. Enhanced DNA imaging using super-resolution microscopy and simultaneous single-molecule orientation measurements. Optica. 3 (6), 659-666 (2016).
- Backer, A. S., et al. Single-molecule polarization microscopy of DNA intercalators sheds light on the structure of S-DNA. Sci Adv. 5 (3), eaav1083 (2019).
- Hulleman, C. N., et al. Simultaneous orientation and 3D localization microscopy with a Vortex point spread function. Nat Commun. 12 (1), 5934 (2021).
- Kampmann, M., Atkinson, C. E., Mattheyses, A. L., Simon, S. M. Mapping the orientation of nuclear pore proteins in living cells with polarized fluorescence microscopy. Nat Struct Mol Biol. 18 (6), 643-649 (2011).
- Lazar, J., Bondar, A., Timr, S., Firestein, S. J. Two-photon polarization microscopy reveals protein structure and function. Nat Methods. 8 (8), 684-690 (2011).
- Dong, B., et al. Parallel Three-Dimensional Tracking of Quantum Rods Using Polarization-Sensitive Spectroscopic Photon Localization Microscopy. ACS Photonics. 4 (7), 1747-1752 (2017).
- Blanchard, A., et al. Turn-key mapping of cell receptor force orientation and magnitude using a commercial structured illumination microscope. Nat Commun. 12 (1), 4693 (2021).
- Toprak, E., et al. Defocused orientation and position imaging (DOPI) of myosin V. Proc Natl Acad Sci USA. 103 (17), 6495-6499 (2006).
- Zhong, S., et al. Three-dimensional dipole orientation mapping with high temporal-spatial resolution using polarization modulation. PhotoniX. 5 (1), 12 (2024).
- Young, L. J., Ströhl, F., Kaminski, C. F. A Guide to Structured Illumination TIRF Microscopy at High Speed with Multiple Colors. J Vis Exp. (111), e53988 (2016).
- Huang, X., et al. long-term, super-resolution imaging with Hessian structured illumination microscopy. Nat Biotechnol. 36 (5), 451-459 (2018).
- Ando, R., et al. StayGold variants for molecular fusion and membrane-targeting applications. Nat Methods. 21 (4), 648-656 (2024).
- Hirano, M., et al. A highly photostable and bright green fluorescent protein. Nat Biotechnol. 40 (7), 1132-1142 (2022).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved