Method Article
蛍光双極子のライブビューイメージングのためのオープンソースのMicro-Managerプラグイン
要約
オープンソースのMicro-Managerプラグインを開発し、構造化照明顕微鏡で蛍光双極子のライブビュー観察を可能にしました。このプラグインは、2Dと3Dの両方の双極子向きの観測をサポートしています。
要約
蛍光偏光顕微鏡(FPM)は、蛍光色素の位置と双極子配向を画像化できます。超解像蛍光偏光顕微鏡の成果にもかかわらず、取得後への依存がリアルタイム観察を妨げています。偏光構造化照明顕微鏡(pSIM)は、蛍光双極子の超解像イメージングを高速に行え、生細胞アプリケーションに最適です。私たちは、偏光画像のリアルタイム再構成と蛍光双極子の表示のためのオープンソースの実装を開発しました。さらに、3D方位マッピング(3DOM)を達成するために手法を拡張し、複雑な生物学的研究への有用性を広げました。さらに、既存のSIMマイクロスコープを偏光イメージングに拡張する方法を徹底的に紹介し、マイクロスコープを制御するためのMicro-Manager 2.0の詳細な構成ガイドを提供し、偏光イメージングのリアルタイムプレビューを可能にしました。さらに、pSIMと3DOMの両方を網羅する完全な再構成のためのMATLABコードを提供しています。この包括的なガイドは、初心者が操作をすばやく習得し、簡単に開始できるように支援することを目的としています。
概要
蛍光偏光顕微鏡(FPM)は、蛍光色素の位置と双極子の配向の両方を同時にイメージングするための強力な技術として登場し、生物学的イメージング1,2に深い洞察を提供しています。FPMは、生体分子の配向の方向観察を容易にすることにより、アクチン3,4,5、微小管5、セプチン6、DNAフィラメント7-9、核孔複合体10、膜タンパク質11などの高分子の複雑な配列を明らかにします.その高速、非侵襲的、および生細胞互換の機能により、高い時間分解能11,12で分子回転ダイナミクスの追跡が可能になります。バイオフォースプローブと統合すると、FPMは細胞内分解能で力の大きさをマッピングするだけでなく、力の方向も測定し、力の方向を明らかにすることで生体力学的プロセスの理解を進める13。
過去数十年で、超解像蛍光偏光顕微鏡法は急速な進化を遂げました。この分野での注目すべき進歩は、SMOLMとも呼ばれる単一分子配向局在化顕微鏡法であり、蛍光色素の位置と配向の両方を局在化できるため、多次元の局在化が可能になります。SMOLMでの偏光測定は、偏光励起変調7、マルチチャネル偏光検出3、または工学的偏光感受性点像分布関数(PSF)14を使用して実行できます。SMOLMは数十ナノメートルオーダーの空間分解能を達成し、単一分子の分極を測定するにもかかわらず、イメージング時間が長くなるという課題があります。これは、蛍光色素の点滅と局在化の繰り返しサイクルによるもので、ビデオレートイメージングや生細胞アプリケーションには課題となっています。
対照的に、偏光構造化照明顕微鏡(pSIM)は、最大約100nmの空間分解能を提供し、同じSIMデータセットで偏光情報を取得することができます。特に、pSIMはビデオレートのイメージング速度を達成でき、蛍光分子に厳しい要件を課すことなく、生細胞イメージングと高い互換性があります。最近、pSIMは膜関連周期骨格(MPS)5 のアクチン環構造を明らかにすることに成功し、生体力の超解像マッピングを可能にしました13。
しかし、pSIMは取得後の画像再構成を必要とするため、偏光結果をリアルタイムで可視化することができません。この遅延は、生命現象の即時観察を妨げ、研究者が関心のある生物学的現象を迅速に捉え、サンプルやイメージング条件をリアルタイムで調整することを妨げます。この制限に対処するために、ImageJおよびMicro-Managerプラットフォーム(https://github.com/KarlZhanghao/live-pol-imaging)に基づいて、画像取得と偏光結果のリアルタイム再構成および表示を容易にするオープンソースの実装を開発しました。
また、pSIMはこれまで2次元面内偏波情報の提供に限られていましたが、最近では、3DOM(3D Orientation Mapping)と呼ばれるほぼ同じ機器15を用いて3D方位マッピングを実現する機能を拡張しました。このオープンソースソフトウェアは、3DOMの制御、再構築、および視覚化も提供します。再構成モジュールと可視化モジュールは、単一分子配向追跡アプリケーションとも互換性があります。これらすべての機能により、複雑な生物学的研究における偏光イメージングの有用性が向上します。
プロトコル
1. 既存の構造化照明顕微鏡を偏光イメージング用に拡張
- SIMマイクロスコープを準備します。
注:読者は顕微鏡のセットアップに一定の基礎があり、すでに構造化された光学顕微鏡を所有していることを前提としています。関連する経験がない場合は、SIMマイクロスコープの構築方法16について詳しく説明したこのホワイトペーパーを参照してください。「HWP+WQP+LCVR」の偏波制御ユニットは、当社のpSIMワーク5 ではピザ渦波板、またはピザ半波板17に置き換えることができる。 - 空間光変調器(SLM)に異なるパターンをロードすることにより、3つのストライプ方向の偏光消光比を測定します。対物レンズの直後に偏光子を配置して回転させ、パワーメーターを使用してレーザー出力を測定します。pSIMの場合、波長板を回転させて、消光比が10>3方向のs偏光を維持します。
- 3DOM の場合、HWP2 を回転させて 6 方向の p 偏光を維持します。SIM 空間マスクを 3DOM 空間マスクに置き換えて、6 方向の 1 次ビームが通過できるようにします ( 図 1B を参照)。
注:pSIMではS偏波が必要であり、3DOMではP偏波が必要です。pSIM5 と 3DOM15 の詳細なセットアップは、他の場所に含まれています。
2. Micro-Managerのセットアップ
- 公式WebサイトからMicro-Manager 2.0バージョンをダウンロードし、指示に従ってソフトウェアをインストールします。
- Micro-Managerシステムに正常に接続するために、対応するデバイスドライバーとソフトウェアを準備します。この顕微鏡システムでは、Micro-Managerを使用して、カメラ、並進ステージ、レーザー、DAQボード、顕微鏡を制御します。
注意: サポートされている機器は https://micro-manager.org/Device_Support に記載されています。接続する機器の詳細については、材料表 を参照してください。ドライバーまたは公式/サポートソフトウェアをインストールし、USBケーブルを使用して機器をコンピューターに接続します。 - ハードウェア構成ウィザードを使用して、装置をMicro-Managerに追加します。
- ソフトウェアを開き、最初のドロップダウンリストから [なし ]を選択します。
- デバイスの選択 |ハードウェア構成ウィザード... |新しい設定を作成し、[次へ] をクリックして設定ページに入ります。
- ドロップダウンハードウェアリストでカメラ/レーザー/ステージ/ DAQ/顕微鏡プラグインを見つけて、[ 追加]をクリックします|わかりました |次へ。
- [デフォルトのデバイスを選択]に移動し、自動シャッター設定を選択します。デフォルトのカメラ、デフォルトのシャッター、デフォルトのフォーカスステージを設定します。[Save configuration and exit] に到達するまで [Next] をクリックし、設定ファイルの名前を保存します。「終了」をクリックします。
- 一般的なテンプレート設定
- ライブモードとSIMスナップモードのテンプレートを保存します。
- 露光時間とトリガーモードを次のように設定します:構成設定でグループ「+」を選択し、グループ名に「モード」という名前を付けて、露出とトリガーモードを選択して、[OK]をクリックします。
- プリセット「+」を選択して、「モード」グループに異なるプリセットを追加します。ライブモードの露出設定には1つのプリセットに「live」、SIMモードにはもう1つのプリセットに「SIM」という名前を付けます。たとえば、「ライブ」モードでは露光時間を15ミリ秒に設定し、SIMスナップモードの場合は露光時間を10ミリ秒に設定し、トリガーモードは標準(オーバーラップ)に設定します。
注:設定ファイル(MMConfig_psim_demo.cfg)はコードリポジトリに含まれており、Micro-Manager 2.0で直接ロードできます。
3. 蛍光ビーズによるシステムキャリブレーション
- カバーガラスを75%エタノールに浸し、脱イオン水(ddH2O)でカバーガラスを3回すすぎ、乾燥させます。
- 100 nmの蛍光ビーズ(リン酸緩衝生理食塩水[PBS]で1:1,000に希釈)をボルテックスし、懸濁液をカバーガラスに直接ピペットで移します。暗所で5〜10分間インキュベートした後、PBSでやさしく洗浄します。
- 顕微鏡スライドに20 μLの封入剤を加え、気泡を避けながらカバースリップをその上に置きます。温度を4°Cに保ち、スライドを暗所に置いておくと、100 nmの蛍光ビーズが入ったスライドがシステムキャリブレーション用に準備されます。
- pSIMの場合、蛍光ビーズの3つの異なるフェーズの3つの画像を取得します。カスタム MATLAB コード (Bead_calib.m) は、イメージを入力として受け取り、さらに解析するために 2 つのキャリブレーション イメージ (calib1.tif、calib2.tif) を出力します。
注:前述の5で示したように、pSIMの実験はほとんどの商用システムで実行できます。自作または市販のSIM顕微鏡では、3パターン方向の不均一な照明によって引き起こされる蛍光双極子の測定誤差を排除するために、システムキャリブレーションが必要です。キャリブレーション実験では、蛍光ビーズの等方性偏光を前提としており、SIM取得中には生の画像のみが必要です。詳細なキャリブレーション手順は、当社のオリジナル作品に含まれています 5.pSIMの再構成には、calib1.tifとcalib2.tifの2つのキャリブレーション画像の出力が必要であり、これはパターン方向2のピクセル単位の照明エネルギーを示します。3はパターン方向1を指す。
4.サンプル調製:固定細胞内のアクチン
- カバースリップを75%エタノールに浸し、カバースリップをddH2Oで3回すすぎ、カバースリップを6ウェル細胞培養プレートに入れる。
- U2OS細胞(ATCC HTB-96細胞株)をダルベッコ改変イーグルス培地(DMEM)で培養し、37°Cで10%(v/v)ウシ胎児血清(FBS)を添加し、カバースリップ上で5%CO2 を約75%のコンフルエントに達するまで培養します。
- 細胞をPBSで3回優しく洗浄します。細胞固定には、4%パラホルムアルデヒドを室温で15分間適用します。固定後、PBSで細胞を再度3回洗浄します。
- 膜透過性を高めるには、0.1% Triton X-100をPBS溶液で5分間処理し、その後、PBSで細胞を3回洗浄します。
- 蛍光ファロイジンを150μLのDMSO溶液(66μM)に溶解します。Phalloidin色素ワーキング溶液を調製するには、ストック溶液5 μLをPBS995 μLで希釈します。暗所で細胞をファロイジン色素ワーキング溶液と室温で~1時間インキュベートします。細胞をPBSで3 x 3分間洗浄します。
- 20 μLの封入剤を顕微鏡スライドに加えます。カバースリップを気泡が入らないようにスライドに慎重に置きます。スライドは4°Cで保管し、暗い場所に保管してください。
5. サンプル調製:in-vitro λ-DNA
- 0.5 gのポリメチルメタクリレートを10 mLの義歯ベース材料に溶解します。完全に溶解した後、溶液をスライドの表面に均一に塗布し、蒸発して膜を形成するのを待ちます。
- 0.3 μL の λ-DNA と 32 μL の 1,000x SYTOX Orange Nucleic Acid Stain を 968 μL の PBS で希釈します。調製した溶液1μLをスライドに加え、乾燥させます。
- カバーガラスを封入剤でスライドに密封し、サンプルを保護および保存します。スライドは暗所で4°Cで保管してください。
6. イメージング
- カバースリップをサンプルホルダーに置き、ライブモードで焦点面に調整します。512 x 512ピクセルのROIを選択し、露光時間とレーザー強度を調整します。
- 空間光変調器の独立した制御ソフトウェアを開き、pSIM / 3DOMまたはライブモードの適切な照明パターンシーケンスパスを選択します。
注:pSIMの場合、照明パターンには3つの異なる偏光角度と3つの異なる位相が含まれます。3DOMの場合、照明パターンには6つの異なる偏光角度が含まれています。 - SIMキャプチャモードを選択し、Multi-D Acq.を選択し、Time Pointsを選択し、開いているウィンドウでカウント(pSIMまたは3DOMの場合は9または6枚の画像)と間隔(0ミリ秒)を設定します。[取得] をクリックします。
- TriggerSLM.bsh(設定ファイルはコードリポジトリに含まれています)をクリックすると、MyDaq取得カードが空間光変調器をアクティブにして写真を撮ります。
7. pSIMと3DOMのライブビュープラグイン
- FakeCameraと画像のアクセスパスを設定しますMMConfig_FakeCam_demo:デバイス|ハードウェア構成の読み込み....デバイスのイメージのローカルディレクトリを構成します|Devices プロパティ ブラウザ |カメラ パス マスク (図 2A-C)。
注:スクリプトの取得部分はハードウェアのセットアップに依存するため、 FakeCamera デバイスを使用してハードディスク上のローカル画像を読み取るバージョン(MMConfig_FakeCam_demo.cfg)を提供しました。ユーザーはバージョンに変更を加えることができます。画像の読み取りパス(カメラパスマスク)を変更することを忘れないでください。 - クイックアクセスパネルを設定するには、[ツール] |クイックアクセスパネル |新しいパネルを作成します。左下隅にある[設定]アイコンをクリックし、[スクリプトの実行]を上部に配置して、「psim.bsh」と「3DOM.bsh」をロードできます(図2D)。
注意: クイックアクセスパネル システムを使用すると、ユーザーはカスタマイズされたウィンドウを作成し、最も頻繁に必要とされるMicro-Managerのコントロールに簡単にアクセスできます。「ライブ」、「スナップ」、またはその他のチャネルも追加できます 、 図2Dに示すように。 - pSIMを例にとると、「psim.bsh」をクリックしてリアルタイムの偏波画像の再構築を実行します。偏光結果とともにリアルタイムで再構成された画像を観察します(図2E)。'psim.bsh' ファイルのキャリブレーション画像のパスを変更することを忘れないでください (calibPath = " キャリブレーション画像ファイルへのパス)。
注: ライブビュープラグインは、 スクリプトパネルで実行できる Bean シェルスクリプト (psim.bsh および 3DOM.bsh) です。プラグインは、pSIM用に9つの画像、または3DOM用に6つの画像を取得し、広視野画像に基づいて向きマッピング結果を計算します。方位マッピング画像が表示され、リアルタイムの偏光情報が得られるため、解像度が低いにもかかわらず、すぐに検査できます。
8. 超解像再構成によるデータ解析
- MATLAB R2019b を Web サイトからインストール します (資料の表を参照)。
- ソフトウェアを開きます。pSIM再構成では、3つの方位角のそれぞれについて3つの異なる位相からなる9つの画像を取得します。9 つのイメージの名前が '1.tif' から '9.tif' であることを確認し、それらを 'Input' という名前のファイルに入れて、'PSIM.m' という名前のプログラムを実行します。再構成された広視野画像、SIM画像、pSIM画像をカラーホイールで観察します。
注: pSIM の再構築には、SIM ステップと PM ステップの 2 つのステップが必要です。データは、デバッグを容易にするために、カスタムメイドのMATLABプログラムによって解析されます。GitHub の 'reconsr' フォルダから再構築コードを取得します。 - 3DOM の場合、6 つの異なる偏光方向の図形をキャプチャします。6つの画像を結合し、結果のファイルに「Raw_data.tif」という名前を付けて、「Recon_3DOM.m」という名前のプログラムを実行します。再構成された広視野画像と3DOM画像、方位角の結果、極角の結果を観察します。
注:再構築プロセス中に、「関数または変数「bfopen」が認識されません」というエラーが発生する可能性があります。「bfopen」はBio-formatsライブラリの関数で、画像ファイルを開いたり読み込んだりするために一般的に使用されます。MATLAB コマンド ウィンドウに addpath ('path_to_bioformats_toolbox') を入力します。'path_to_bioformats_toolbox' を実際のライブラリフォルダパスに置き換えます。また、Bio-Formats の依存関係を GitHub リポジトリに追加しました。
結果
pSIM法は、s偏光レーザービームを使用した干渉に基づいてSIM顕微鏡で実行できます。S偏光干渉は、最も広く使用されているタイプのSIMであり、高コントラストの照明ストライプを生成します。顕微鏡セットアップのアカデミックプロトタイプは、pSIM5のオリジナル作品に含まれています。 図1で簡単に紹介すると、空間光変調器(SLM)は±1オーダーの回折ビームを生成し、ピザ半波長板は3方向のS偏光を維持します。他のSIMの代替品では、回折ビームを生成するために格子を使用したり、s偏光を維持するために他の偏光変調法を使用したりすることがありますが、これらの顕微鏡は偏光イメージングのために拡張することもできます。
3DOM顕微鏡は、2つの違いがあるにもかかわらず、同様の光学イメージングシステム(図1A)を利用しています。まず、3DOMは2つのレーザービームの干渉を必要としません。代わりに、+1ビームのみを使用して励起偏光を制御します。元の作品15で示されているように、3DOMは、3Dダイポールの向きを計算するために最低6つの方向を必要とします。したがって、3DOM 空間マスクには 6 つの方向が含まれ、それぞれにピンホールがあります ( 図 1B を参照)。もう一つの違いは、3DOMはp偏光レーザービームを使用しており、これはHWP2を回転させることで実現できることです。SLMは、さまざまな斜め角度で回折光を生成して、p偏光の部分を変更します。
pSIMシステムでは、出射光を半波長板の角度を回すことでS偏光に変調します。pSIMのマスクは対称的であるため、±1次の光を同時に通過させることができます。逆に3DOMでは、半波長板の角度を調整することで、レーザーをp偏光に変換します。3DOMのマスクは非対称ではなく、+1次の光のみを通過させます。SLMのストライプ周波数を調整することで、レーザー光の回折角度を変化させます。したがって、面外偏光の部分は変調されます。
方位角が同じで極角が異なる2つの隣接する蛍光双極子を解析することにより、pSIMと3DOMはどちらも回折限界内で近くの双極子を区別できます。さらに、3DOMは、偏光領域で3D配向を持つ異なる双極子を効果的に区別することができます(図1C、D)。これは、3DOM技術が励起光の偏光状態を変えることで、より多くの空間情報を取得できるため、3次元空間での双極子方位を正確に測定できるためです。一方、pSIM技術は主に2次元の偏波情報に依存しており、3次元の配向特性を十分に活用することはできません。したがって、垂直方向の双極子の向きに関する情報を提供できません。
オリエンテーションマッピングの結果には後処理が必要なため、Micro-Manager 2.0用の「Live-viewプラグイン」(「psim.bsh」および「3DOM.bsh」)を開発し、ここで紹介しました。実際のカメラが接続されていないコンピューターユーザーがプラグインの使用方法を学べるように、最初に 図2 FakeCamera.In を使用してMicro-Managerを構成し、pSIMがリアルタイムの偏波効果を実現するための操作プロセスを詳しく説明し、3DOMの操作手順も同様です。 図2Aには、Micro-Manager 2.0のソフトウェアインターフェイスと、プラグインを動作用に構成するために必要なセットアップ手順が表示されています。 図2Bは、FakeCameraとDStageのデモデバイスを使用したハードウェア構成を示しています。FakeCameraのハードウェア構成は、露出、フォーカシング、ホワイトバランスなど、実際のカメラのさまざまな動作をシミュレートできるため、開発者は実際のカメラを必要とせずにソフトウェア開発とテストを行うことができます。ユーザーは、 図 2C に示すように、実際の状況に基づいて画像の読み取りパスを変更する必要があります。 Figure 2D は、一般的に使用される一連の機能または設定へのショートカットを含む クイック アクセス パネルを表示し、ユーザーがこれらの機能にすばやくアクセスして使用できるようにします。リアルタイムプレビュープラグイン「psim.bsh」と「3DOM.bsh」は、このパネルからロードできます。具体的な運用手順については、プロトコルセクション7で詳しく説明されています。「psim.bsh」および「3DOM.bsh」スクリプトは、向きマッピング画像を即座に生成するために使用されます。このプラグイン「psim.bsh」は、pSIMの画像取得段階で偏光効果を即座に可視化することができます。 図2E は、pSIMの偏光イメージングの典型的な結果を示しています。カラーホイールは、双極子方位と疑似カラーの関係を示しており、研究者が表示された偏光データに基づいてどの画像をキャプチャするかをすぐに決定するのに役立ちます。
U2OS細胞をファロイジンで標識し、pSIMの画像再構成プロセスをMATLABを用いて行いました。3つの異なる偏光状態と3つの異なる位相から9枚の写真が撮影されました。緑色の二重矢印は、 図3Aの偏光の方向を示しています。画像の明暗強度から、蛍光双極子の偏光方向を決定できます。
図3Bでは、左の図がSIMからの超解像結果を示し、右の図がpSIMの結果を示しています。特に、pSIMはSIMと同じ空間分解能を実現し、広視野顕微鏡と比較して2倍の空間分解能を提供します。特定の色をアクチンフィラメントの異なる方向にマッピングすることにより、カラーホイールは、細胞のアクチンフィラメント内の蛍光双極子の配列を解釈することを可能にする視覚的表現を提供します。アクチンフィラメントの色は、双極子がフィラメントの向きとほぼ整列していることを示しています。
図4A は、偏光励起変調下での放射強度分布を示しています。3DOMは、6つの異なる偏光変調を生成できます。この構成では、蛍光分子の励起光は偏光状態を変化させることで変調され、分子の発光強度に影響を与える。これにより、研究者は、さまざまな分極状態での蛍光強度を測定することにより、分子の配向を推測できます。 図4B は、 図4Aの異なる分極状態におけるλ-DNAの蛍光強度を示しています。λ-DNA分子は、異なる分極状態で異なる蛍光強度を示すため、蛍光双極子と環境の配向が蛍光シグナルの強度に大きく影響することを示しています。 図4C は、3DOMによって達成されたλ-DNAの超解像イメージングを示しており、方位角の結果と極角の結果の両方を示しています。私たちの観察により、色素分子はDNA鎖に対してほぼ垂直に整列していたことが明らかになりました。DNA軸が曲がるため、色素分子の平均配向はそれに応じて回転します。当社の配向測定の結果は、隣接するDNA塩基間にSTOYOX Orange蛍光色素が挿入され、吸収双極子モーメントの整列がDNA軸に対して垂直であることを検証しています。
そのため、3DOMでは、標的分子のz軸情報を得るために傾斜した励起光が必要です。当社のシステムでは、SLMを用いて照明光の調整を実現しています。SLM の特性と組み合わせると、SLM にストライプ パターンがロードされている場合、SLM から出る光は 0 と ±1 桁の光で構成されます (+1 と -1 の光は対称です)。そこで、励起光として+1次光を使用し、0次数と-1次光をフィルタで除去するために非対称マスクを利用します。また、斜め励起光は、SLMだけでなく、走査型検流計を使用したり、対物レンズの前にあるレンズを横方向に動かしたりすることでも実現できます。
3DOMで6つの異なる励起偏光状態を使用することは、その原理15によって決定されます。フォワードイメージングモデルを調整することにより、得られるモデルは6つの部分で構成されます。したがって、偏光状態の調整を6つ未満にすることはできず、6つを超えると情報の冗長性が発生します。3DOMの具体的な原理を以下に示します。
球面座標では、方位角の天使ρ と極角ηを使用して、次のように蛍光分子を特徴付けます。

ρ は x-y 平面上の分子の投影と x 軸との間の時計回りの角度であり、η は分子と z 軸との間の角度です。
p偏光励光は次のように特徴付けられます。

ここで、αとβは、それぞれ励起光の方位角と斜め入射角を表します。光子吸収効率は  であり、焦点面内のw双極子の放出がn番目のピクセル内に捕捉されると仮定すると、双極子によって放出される蛍光光子は次のように表されます。
であり、焦点面内のw双極子の放出がn番目のピクセル内に捕捉されると仮定すると、双極子によって放出される蛍光光子は次のように表されます。


ここで、 C はサンプルの放射照度を表し、 は次のように励起光の偏光状態を表します。
は次のように励起光の偏光状態を表します。

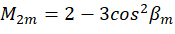

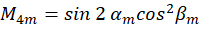
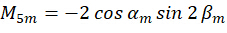
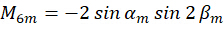
係数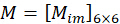 を組み合わせることにより、次のように取得した画像を入力として
を組み合わせることにより、次のように取得した画像を入力として とすることで、n番目のピクセルの向き
とすることで、n番目のピクセルの向き を解くことができます。
を解くことができます。
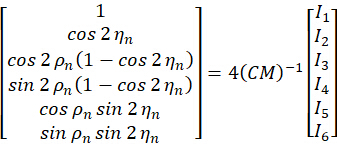
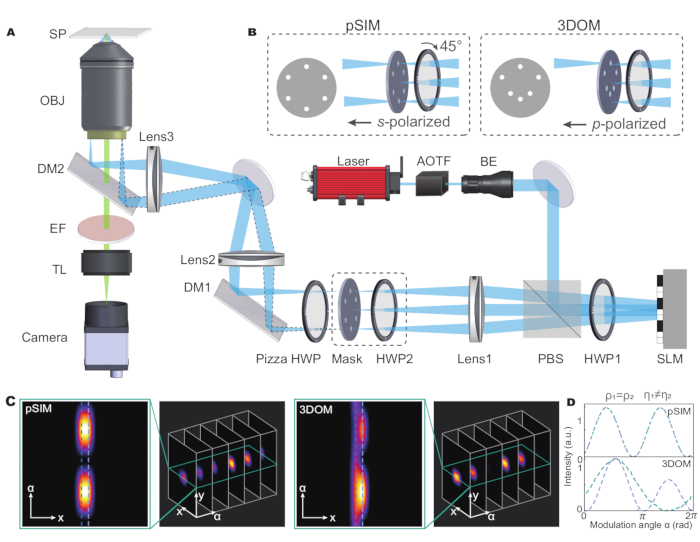
図1:pSIMと3DOM顕微鏡の概略図(A)pSIMと3DOMの実験セットアップ。(イ)pSIMシステムと3DOMシステムの主な違いは、マスクとHWP2です。pSIMのマスクにより、±1次数の光が同時に通過し、HWP2を回転させることで出射光がS偏光されます。3DOM のマスクは +1 次光のみを通過し、出射光は p 偏光されます。(C、D)pSIMと3DOMの発光蛍光強度の変動の比較。略語:pSIM = 偏光構造化照明顕微鏡;3DOM = 3Dオリエンテーションマッピング。AOTF = 音響光学チューナブルフィルタ;BE = ビームエキスパンダー;PBS =偏光ビームスプリッター;HWP = 半波長板;SLM = 空間光変調器;DM = ダイクロイックミラー;OBJ = 客観的;SP = サンプル平面;EF = 発光フィルタ;TL = 結像レンズ。この図は、Zhanghao et al.5 と Zhong et al.15 から修正されました。この図の拡大版を表示するには、ここをクリックしてください。

図2:ソフトウェアのスクリーンショットとpSIMの代表的な結果 (A)Micro-Manager 2.0のインターフェース。一部のオプションは、ファイルの設定に使用できます。(B) FakeCameraとDStageによるライブビュープラグインの設定。(ハ)画像読み取りパス(パスマスク)選択インターフェース。(D) クイックアクセスパネルを設定し、「スクリプトの実行」を「psim.bsh」と「3DOM.bsh」と一緒にロードします。(E)リアルタイムの画像再構成は、偏光効果を示しています。双極子の向きは疑似色です。カラーホイールは、双極子向きと擬似色との関係を示します。略語:pSIM = 偏光構造化照明顕微鏡;3DOM = 3D オリエンテーションマッピング。 この図の拡大版を表示するには、ここをクリックしてください。
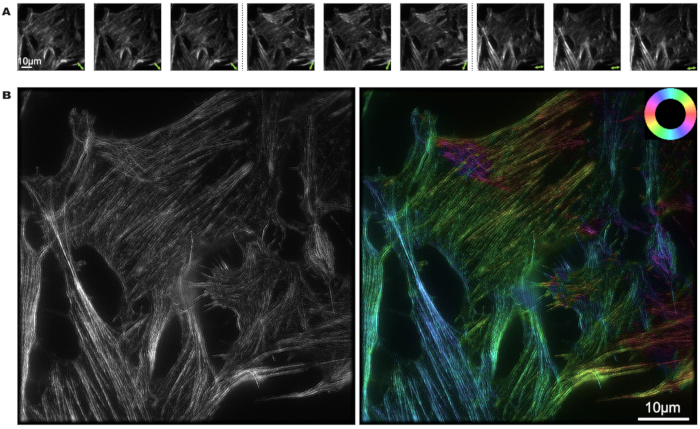
図3:ファロイジン標識U2OS細胞におけるアクチンフィラメントのpSIMイメージング (A)pSIMにおける励起光偏光状態3倍の変調、各偏光状態の3つの異なる位相を捉える。二重矢印は偏光の方向を示しています。 (B) AのアクチンフィラメントのSIMおよびpSIM画像、左の画像はSIMの結果、右の画像は偏光角でのpSIMの結果を示しています。右上に配置されたカラーホイールは、疑似色と双極子向きの関係を示しています。スケールバー = 10 μm。略語:pSIM = 偏光構造化照明顕微鏡。 この図の拡大版を表示するには、ここをクリックしてください。

図4:SYTOX Orange標識 3DOMで画像化されたOrange標識λ-DNA. (A) 3DOMにおける励起光偏光状態6xの変調。 (B) A. の偏光励起変調下での発光強度分布 (C)λ-DNA鎖の3DOM画像(左の画像は方位角の結果、右の画像は極角の結果を示しています。スケールバー = 10 μm。略語: 3DOM = 3D オリエンテーション マッピング。この図の拡大版を表示するには、ここをクリックしてください。
ディスカッション
私たちの研究では、pSIMと3DOMという2つの偏光イメージング技術をリアルタイムでプレビューできるプラグインを開発しました。どちらの技術も、わずかな変更を加えるだけで既存のSIMシステムで実行できます。pSIMと3DOMマイクロスコープのインストール手順と、マイクロスコープを制御するためのMicro-Managerのセットアップ、ライブビュー偏光結果の取得方法の詳細な手順を提供しました。実験結果には、pSIMでイメージングしたアクチンフィラメントと3DOMでイメージングしたλ-DNAが含まれています。ファロイジン双極子の配向はアクチンフィラメントの方向とほぼ平行ですが、STOYOX Orange蛍光色素の配向はDNA軸に対して垂直です。構造化照明顕微鏡は、ゴルジ装置、微小管ネットワーク18、および小胞体19の高時空間分解能イメージングも達成できる。
pSIMと3DOMの両方用にカスタマイズされたプラグインを開発しました。ユーザーは、撮影モードに適したプレビュープログラムを選択できます。リアルタイムプレビュープラグインの実行中に、プラグインが正常に起動しない場合、ユーザーはまず、仮想カメラ画像の読み取りに関する構成ファイルのパス設定を確認し、パスが画像が保存されている実際の場所に正確に変更されていることを確認する必要があります。その後、リアルタイムプレビュープラグインでキャリブレーション画像を読み取るためのパスもそれに応じて更新されていることを確認することが不可欠です。Micro-Managerのバージョンが異なれば、新機能が導入される場合がありますが、互換性の問題も発生する可能性があります。したがって、特定の操作は、実際に使用するバージョンに応じて調整する必要があります。
この技術の限界は、現在のリアルタイムプレビュープラグインでは、pSIMと3ODMの両方で双極子の蛍光偏光方向を観察できるにもかかわらず、画像の解像度はまだ不十分であり、大まかな観察には十分ではないという事実にあります。ただし、高解像度の画像は、分析と意思決定のプロセスにおいて特に重要です。より詳細に取得するには、MATLABプラットフォームに依存し、超解像再構成技術などの高度な画像処理アルゴリズムを採用して詳細な処理を行う必要があります。私たちのプラグインで完全な再構築が行われないのは、使用されるBeanshellスクリプトのパフォーマンスが低いためです。Beanshellを使用した完全な再構築には、画像取得よりもはるかに長い時間がかかります。今後の作業には、Javaと並列コンピューティングによる完全な再構築の実装と、ライブプレビューへの機能の組み込みが含まれます。
このプラグインが偏光状態をリアルタイムで観察することで、実験の効率を向上させることを願っています。この即時観測により、研究者は標本の双極子方位を即座に観察できるため、ライブタイムトラッキング中に関心のあるターゲットや珍しいイベントをすぐに見つけることができます。また、このプロトコルはハードウェアのセットアップとソフトウェア制御も提供し、pSIMと3DOMをより使いやすくします。
開示事項
著者は、利益相反を宣言しません。
謝辞
この研究は、中国の国家重点研究開発プログラム(2022YFC3401100)の支援を受けました。
資料
| Name | Company | Catalog Number | Comments |
| 100 nm Fluorescent beads | Invitrogen | F8801 | |
| 4% Formaldehyde solution | Invitrogen | R37814 | |
| Camera | Tucsen | Dhyana 400BSI V3 | https://www.tucsen.com/download-software/ |
| Denture base materials (Type I Thermally setting type, liquid) | New Century Dental | N/A | |
| Dulbecco’s Modified Eagle’s Medium | Gibco | C11995500BT | |
| Eclipse TE2000 Inverted Microscope | Nikon | TE2000 E | |
| Fetal Bovine Serum | Gibco | 10099141C | |
| MATLAB R2019b | MathWorks | Version R2019b | https://ww2.mathworks.cn/downloads/ |
| MetroCon V4.0 | Kopin | Version 4.0 | Software of Spatial light modulator |
| Micro-Manager 2.0 | μΜanager | Version 2.0 | Download Micro-Manager Latest Release |
| MS-2000 XYZ Automated Stage | Applied Scientific Instrumentation | MIM3 | https://www.asiimaging.com/support/downloads/usb-support-on-ms-2000-wk-controllers/ |
| myDAQ | National Instruments | 781325-01 | Software and Driver Downloads - NI |
| OBIS 561 nm LS 20 mW Laser | Coherent | 1325777 | |
| Phalloidin-AF568 | Invitrogen | A12380 | |
| Phosphate buffered saline | Corning | 21-040-CV | |
| Poly Methyl Methacrylate | Solarbio | M9810 | |
| ProLong Diamond | Invitrogen | P36980 | |
| Spatial light modulator | Kopin | SXGA-12 | |
| SYTOX orange nucleic acid stain | Invitrogen | S11368 | |
| Triton X-100 | Invitrogen | HFH10 | |
| Trypsin | Gibco | 25200056 | |
| λ-DNA | Invitrogen | S11368 |
参考文献
- Zhanghao, K., Gao, J., Jin, D., Zhang, X., Xi, P. Super-resolution fluorescence polarization microscopy. J Innov Opt Health Sci. 11 (01), 1730002 (2018).
- Alonso, M. A., Brasselet, S. Polarization microscopy: from ensemble structural imaging to single-molecule 3D orientation and localization microscopy. Optica. 10 (11), 1486-1510 (2023).
- Valades Cruz, C. A., et al. Quantitative nanoscale imaging of orientational order in biological filaments by polarized superresolution microscopy. Pro Natl Acad Sci USA. 113 (7), E820-E828 (2016).
- Zhanghao, K., et al. Super-resolution dipole orientation mapping via polarization demodulation. Light Sci Appli. 5 (10), e16166 (2016).
- Zhanghao, K., et al. Super-resolution imaging of fluorescent dipoles via polarized structured illumination microscopy. Nat Commun. 10 (1), 4694 (2019).
- Vrabioiu, A. M., Mitchison, T. J. Structural insights into yeast septin organization from polarized fluorescence microscopy. Nature. 443 (7110), 466-469 (2006).
- Backer, A. S., Lee, M. Y., Moerner, W. E. Enhanced DNA imaging using super-resolution microscopy and simultaneous single-molecule orientation measurements. Optica. 3 (6), 659-666 (2016).
- Backer, A. S., et al. Single-molecule polarization microscopy of DNA intercalators sheds light on the structure of S-DNA. Sci Adv. 5 (3), eaav1083 (2019).
- Hulleman, C. N., et al. Simultaneous orientation and 3D localization microscopy with a Vortex point spread function. Nat Commun. 12 (1), 5934 (2021).
- Kampmann, M., Atkinson, C. E., Mattheyses, A. L., Simon, S. M. Mapping the orientation of nuclear pore proteins in living cells with polarized fluorescence microscopy. Nat Struct Mol Biol. 18 (6), 643-649 (2011).
- Lazar, J., Bondar, A., Timr, S., Firestein, S. J. Two-photon polarization microscopy reveals protein structure and function. Nat Methods. 8 (8), 684-690 (2011).
- Dong, B., et al. Parallel Three-Dimensional Tracking of Quantum Rods Using Polarization-Sensitive Spectroscopic Photon Localization Microscopy. ACS Photonics. 4 (7), 1747-1752 (2017).
- Blanchard, A., et al. Turn-key mapping of cell receptor force orientation and magnitude using a commercial structured illumination microscope. Nat Commun. 12 (1), 4693 (2021).
- Toprak, E., et al. Defocused orientation and position imaging (DOPI) of myosin V. Proc Natl Acad Sci USA. 103 (17), 6495-6499 (2006).
- Zhong, S., et al. Three-dimensional dipole orientation mapping with high temporal-spatial resolution using polarization modulation. PhotoniX. 5 (1), 12 (2024).
- Young, L. J., Ströhl, F., Kaminski, C. F. A Guide to Structured Illumination TIRF Microscopy at High Speed with Multiple Colors. J Vis Exp. (111), e53988 (2016).
- Huang, X., et al. long-term, super-resolution imaging with Hessian structured illumination microscopy. Nat Biotechnol. 36 (5), 451-459 (2018).
- Ando, R., et al. StayGold variants for molecular fusion and membrane-targeting applications. Nat Methods. 21 (4), 648-656 (2024).
- Hirano, M., et al. A highly photostable and bright green fluorescent protein. Nat Biotechnol. 40 (7), 1132-1142 (2022).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved