Method Article
Ensaio de remontagem de luciferase dividida para medir contatos de retículo endoplasmático-mitocôndrias em células vivas
Neste Artigo
Resumo
Estabelecemos um ensaio de remontagem de luciferase dividida para monitorar os contatos retículo-mitocôndria endoplasmática em células vivas. Usando este ensaio, descrevemos um protocolo para medir quantitativamente o nível desses acoplamentos inter-organelas em HEK293T células, sob a condição de tratamento químico.
Resumo
Os locais de contato do retículo endoplasmático (ER)-mitocôndrias desempenham um papel crítico na saúde e homeostase celular, como a regulação do Ca2+ e da homeostase lipídica, dinâmica mitocondrial, biogênese de autofagossomos e mitofagossomos e apoptose. A falha em manter o acoplamento mitocondrial ER normal está implicada em muitas doenças neurodegenerativas, como doença de Alzheimer, doença de Parkinson, esclerose lateral amiotrófica e paraplegia espástica hereditária. É de considerável importância explorar como a desregulação dos contatos ER-mitocondriais pode levar à morte celular e se reparar esses contatos ao nível normal pode melhorar as condições neurodegenerativas. Assim, ensaios aprimorados que medem o nível desses contatos podem ajudar a esclarecer os mecanismos patogênicos dessas doenças. Em última análise, o estabelecimento de ensaios simples e confiáveis facilitará o desenvolvimento de novas estratégias terapêuticas. Aqui descrevemos um ensaio de luciferase dividida para medir quantitativamente o nível de contatos ER-mitocôndrias em células vivas. Este ensaio pode ser usado para estudar o papel fisiopatológico desses contatos, bem como para identificar seus moduladores na triagem de alto rendimento.
Introdução
As interações entre o RE e as mitocôndrias são vitais para a homeostase celular e sobrevivência 1,2,3,4. Evidências anteriores indicam que qualquer tipo de interrupção ou desregulação nos locais de contato ER-mitocôndrias pode contribuir para várias doenças neurodegenerativas, metabólicas e cardiovasculares, bem como câncer 5,6,7,8,9,10. Por exemplo, um aumento anormal da captação de Ca2+ nas mitocôndrias pode levar à morte celular pela abertura dos poros de transição de permeabilidade das mitocôndrias, que são comumente vistos em alguns modelos da doença de Alzheimer 5,11. Da mesma forma, a redução dos contatos ER-mitocôndrias pode resultar em diminuição da produção de ATP e comprometimento da ingestão de Ca2+, como visto em modelos de esclerose lateral amiotrófica 5,11,12. À medida que mais estudos estão sendo conduzidos no domínio dos contatos ER-mitocôndrias, proteínas e genes adicionais relacionados à doença que podem afetar esses contatos estão sendo descobertos. Apesar do conhecimento atual e das evidências que mostram o papel dos locais de contato ER-mitocôndrias, ainda é necessário muito trabalho para elucidar como esses contatos podem levar à perda da função celular e, finalmente, à morte celular.
Vários métodos foram desenvolvidos para avaliar a proximidade das duas membranas, a morfologia estrutural e a distância entre os dois locais de contato com as organelas 3,4,13. As abordagens para monitorar o acoplamento ER-mitocôndria incluem imagens baseadas em marcadores de fluorescência14,15, imagens baseadas em repórteres FRET16 e imagens baseadas em sonda de fluorescência dividida17,18, que usam epifluorescência e microscopia confocal. A microscopia de super-resolução e de resolução atômica também são ferramentas poderosas para visualizar com precisão os contatos entre organelas, embora sua utilização na análise do local de contato ainda seja limitada, pois requerem microscópios altamente dedicados e conhecimento técnico19. Além disso, microscopia eletrônica de transmissão (MET), microscopia eletrônica de varredura (MEV) e outras técnicas de EM, como tomografia eletrônica (ET) e microscopia crioeletrônica, são comumente usadas, pois fornecem imagens ultraestruturais de alta resolução dos locais de contato, que muitas vezes são impossíveis de explorar usando outras abordagens experimentais 20,21,22 . No entanto, esses métodos baseados em EM são uma técnica de rendimento muito baixo que também pode ser afetada por procedimentos de fixação química. Mais recentemente, métodos baseados em marcação de proximidade têm sido usados para detectar locais de contato, bem como para identificar novas proteínas do local de contato. Por exemplo, o ensaio de ligação de proximidade (PLA) foi usado para quantificar as proximidades de organelas23 , 24 , enquanto uma versão revisada do ensaio de ascorbato peroxidase (APEX) foi utilizada na identificação de novas proteínas do local de contato25 , 26 . É importante reconhecer que todos esses métodos descritos acima têm pontos fortes e limitações intrínsecas na detecção dos contatos entre as organelas. Assim, o emparelhamento de diferentes técnicas é necessário para obter uma interpretação completa dos locais de contato com as organelas.
Anteriormente, estabelecemos o ensaio de remontagem split-Renilla luciferase 8 (ensaio split-Rluc) para monitorar o nível de contatos da membrana ER-mitocôndrias (Figura 1A) 24 , 26 , 27 . Resumidamente, cada metade dividida da luciferase de Renilla é conjugada com uma sequência de direcionamento de ER ou mitocôndria. Quando transfectada em conjunto, cada metade dividida da enzima é expressa no RE ou na membrana mitocondrial. Quando o ER e as mitocôndrias são posicionados próximos um do outro, as metades divididas se juntam e reconstituem toda a enzima com atividade de luciferase. Para a construção split-Rluc, usamos Renilla luciferase 8 (Rluc8) em pBAD/Myc-His27 para o modelo inicial. O sítio de divisão (entre os aminoácidos 91 e 92) foi determinado com base em relatórios anteriores27. Para a metade N-terminal do Rluc8, as sequências de DNA para os aminoácidos 1-91 do Rluc8 foram fundidas à extremidade 3 'da marca FLAG e a sequência de direcionamento mitocondrial AKAP1 de camundongo no vetor pcDNA3.1 TOPO por PCR27. Para a metade C-terminal direcionada ao RE, as sequências de DNA que codificam os aminoácidos 92-311 foram fundidas à extremidade 5 'da etiqueta myc e à sequência de localização UBC6 ER da levedura. Aqui, atualizamos a construção do plasmídeo split-Rluc de modo que as metades divididas da luciferase Renilla sejam expressas em um único vetor (pCAG) sob o mesmo promotor e subsequentemente clivadas em dois fragmentos à medida que T2A, uma sequência de peptídeo 2A autoclivada do vírus Thosea asigna, é inserida entre as duas metades divididas ( Figura 1B ). O mapa e as sequências de DNA do plasmídeo são fornecidos no Arquivo Suplementar 1 e na Figura Suplementar S1. Usando este sistema, medimos os efeitos de três produtos químicos (inibindo GTPases envolvidas na polimerização de actina) nos contatos ER-mitocôndrias. Este ensaio split-Rluc é um sistema de ensaio simples, mas robusto, para triagem de alto rendimento para moduladores de contato entre organelas24.
Protocolo
1. Manutenção e semeadura celular (Dia 1)
- Manter HEK293T células em meios de cultura de células contendo meio de Eagle modificado de Dulbecco (DMEM) com 10% de soro fetal bovino (FBS) (em placas de cultura de 100 mm) em uma incubadora umidificada a 37 ° C com 5% de CO2.
- Antes de começar, verifique a confluência da placa visualizando ao microscópio. Quando as células atingirem aproximadamente 80-90% de confluência, prepare-se para semear as células em uma placa de cultura de 6 poços, removendo o meio e lavando com 10 mL de solução salina tamponada com fosfato de Dulbecco (DPBS).
- Remova o DPBS da placa de cultura e trate as células com 1 mL de vermelho de fenol tripsina-EDTA a 0,05%. Incubar por 3 min, depois retirar a placa da incubadora e adicionar 9 mL de meio de cultura (DMEM com 10% de FBS) para interromper a reação de tripsina. Pipete lentamente para cima e para baixo para garantir que todas as células ainda presas à placa sejam desalojadas. Transfira a suspensão celular para um tubo de 50 mL.
- Centrifugue a 300 × g por 5 min em temperatura ambiente. Remova o meio e ressuspenda os grânulos celulares com 1 mL de meio fresco (DMEM + 10% FBS).
- Conte o número de células usando um hemocitômetro.
- Garanta uma distribuição uniforme das células girando suavemente o tubo. Retire imediatamente 10 μL da suspensão celular e coloque em um tubo de microcentrífuga de 1,7 mL. Adicione 90 μL de mídia ao mesmo tubo.
- Depois de misturar as células e o meio suavemente, pegue a suspensão celular e aplique 10 μL em cada uma das câmaras do hemocitômetro, pipetando suavemente sob a lamínula.
- Usando uma objetiva de 10x, focalize as linhas de grade do hemocitômetro sob um microscópio. Uma vez focado, conte o número de células em um conjunto de 16 quadrados (4 x 4 quadrados) usando um contador de contagem manual. Repita até que todos os quatro conjuntos de 16 quadrados sejam contados.
- Calcule o número total de células/mL tomando a contagem média de células (de cada um dos 16 quadrados de canto) e multiplicando por 105.
NOTA: Em vez de usar um hemocitômetro para contar células, um contador de células automatizado pode ser usado de acordo com as instruções do fabricante.
- Assim que a contagem de células estiver concluída, prepare-se para plaquear as células em uma placa de 6 poços a 7,5 × 105 células /densidade de poço com 2 mL de meio/poço. Em um tubo de 50 mL, adicione o volume necessário de células junto com 13 mL de meio. Dispense 2 mL da suspensão de células diluídas em cada um dos 6 poços.
- Antes do plaqueamento, calcule o número de células necessárias para 7,5 × 105 células/poço usando C1V1 = C2V2 onde C1 é a contagem inicial de células (número de células/mL), V1 é o volume necessário da suspensão inicial de células, C2 é a densidade de células-alvo desejada (número de células/mL), e V2 é o volume final necessário para a semeadura celular.
- Após a distribuição, bata suavemente na placa em todos os lados para espalhar as células e, em seguida, coloque-a em uma incubadora umidificada a 37 ° C com 5% de CO2 durante a noite.
2. Transfecção celular mediada por polietilenoimina (PEI) e semeadura celular pós-transfecção (Dia 2)
- Remova a placa de 6 poços da incubadora e aspire os meios de cultura existentes de cada poço. Adicione 2 mL de meio fresco por poço.
- Transfecte as células com DNA de plasmídeo split-Rluc (pCAG-MitoRluc N-T2A-R lucCER-IRES-mCherry) dissolvido em tampão TE (pH 8,0).
- Para cada poço, misture o DNA (750 ng) e PEI (proporção de DNA / PEI = 1: 3) em 200 μL de DMEM em um tubo de microcentrífuga de 1,7 mL. Rapidamente vórtice o DNA / PEI no nível 8 por aproximadamente 2 s para garantir que eles se misturem bem para formar complexos. Depois disso, gire-o brevemente em uma mini centrífuga (<3 s).
- Deixe a mistura de DNA / PEI incubar em temperatura ambiente por 15 min e, em seguida, adicione a mistura (soltando-a) à superfície do meio de cultura na placa de 6 poços contendo células. Bata suavemente na placa (para distribuir uniformemente a mistura) e incube-a em uma incubadora umidificada a 37 ° C com 5% de CO2 por 6 h.
- Durante a incubação, prepare uma placa de 96 poços revestida com Poli-D-Lisina (PDL) adicionando primeiro 70 μL de PDL (50 μg/mL em DPBS) a cada poço e incubando em uma incubadora umidificada a 37 °C com 5% de CO2 por pelo menos 1 h. Remova a solução de PDL e lave cada poço 2x com 100 μL de DPBS. No final da segunda lavagem, certifique-se de que todos os DPBS restantes sejam removidos.
- Às 6 h após a transfecção, prepare-se para semear as células transfectadas em uma placa de 96 poços revestida com PDL.
- Retire a placa de 6 poços contendo células transfectadas da incubadora, aspire o meio e lave cada poço com 2 mL de DPBS. Depois de remover o DPBS, adicione 350 μL de vermelho de fenol tripsina-EDTA a 0,05% e incube por 1-2 min. Para interromper a reação da tripsina, adicione 1,7 mL de meio a cada poço.
- Depois de transferir as células junto com o meio para um tubo de 50 mL, gire o tubo a 300 × g por 5 min em uma centrífuga de mesa. Aspire o meio e ressuspenda o pellet celular com 1 mL de meio fresco. Conte o número de células usando um hemocitômetro (conforme descrito na etapa 1.5).
- Semeie as células em uma placa de 96 poços revestida com PDL a uma densidade de 4 × 104 células/poço em 100 μL de meio de cultura. Para isso, em um tubo de 50 mL, adicione o volume necessário de células (veja abaixo para cálculo) junto com o volume apropriado de meio (volume total de 12 mL). Usando uma pipeta multicanal, dispense 100 μL da suspensão de células diluídas em cada um dos 96 poços. Incubar as células durante 18 h numa incubadora humidificada a 37 °C com 5% de CO2.
- Calcule o número de células necessárias para 4 × 104 células/poço usando C1V1 = C2V2 onde C1 é a contagem inicial de células (número de células/mL), V1 é o volume necessário da suspensão inicial de células, C2 é a densidade de células-alvo desejada (número de células/mL) e V2 é o volume final necessário para a semeadura de células.
3. Tratamento químico e ensaio de luciferase em células vivas (Dia 3)
- Em um tubo de microcentrífuga de 1,7 mL, prepare as três soluções frescas de 50 μM de rosina (50 mM de estoque em DMSO), 25 μM de Ehop-016 (25 mM de estoque em DMSO) e 50 μM de ZCL278 (50 mM de estoque em DMSO) diluindo cada solução estoque (50 mM de Rhosina, 25 mM de Ehop-016 e 50 mL de ZCL278; todos em DMSO) 1:1.000 na quantidade necessária (50 μL/poço × número de poços) de meios de cultura. Para cada solução, adicione substrato de células vivas (estoque de 50 mM em DMSO) para Renilla luciferase diluída 1:2.000 até uma concentração final de 25 μM. Para fazer soluções de ZCL278 de 0,5, 5 e 50 μM, execute diluições seriais usando o ZCL278 inicial de 50 μM em meios de cultura.
NOTA: Se o tempo de incubação química for desejado por menos de 1-1,5 h, as células podem ser pré-incubadas com substrato de células vivas (para Renilla luciferase) por 1-1,5 h antes de serem tratadas com cada produto químico. Adicionamos produtos químicos e substrato de células vivas simultaneamente para otimizar o protocolo para triagem de medicamentos de alto rendimento. - Remova o meio de cada poço da placa de 96 poços contendo células transfectadas e adicione 50 μL de mistura de meios químicos e substratos a cada poço.
- Depois de incubar as células por 1 h, 2 h ou 5 h, retire a placa de cultura da incubadora, carregue-a no leitor de placas de luminescência e meça a luminescência.
- Antes de começar, certifique-se de que o leitor de placas esteja ligado. Acesse o software leitor de microplacas e crie um novo file clicando em Nova sessão. Defina a temperatura do leitor de placas para 37 °C clicando em Incubadora, marcando a opção de temperatura e digitando 37 °C. Em Layout da placa, escolha a opção Desconhecido e especifique os poços a serem medidos clicando e arrastando.
- Em Protocolo, clique em Luminescência e mantenha as configurações padrão. Quando terminar a configuração, carregue a placa com a tampa no leitor de placas e escolha Run Plate In. Clique em Iniciar e a janela de salvamento do arquivo aparecerá. Salve o arquivo em sua área de trabalho renomeando-o e clicando em Salvar.
NOTA: A luminescência continuará a ser produzida por >24 h após a adição do substrato da célula viva.
- Quando a leitura estiver concluída, remova a placa do leitor de placas e clique em Run Plate In para colocar o leitor de volta. Depois de concluído, coloque a placa de volta na incubadora umidificada (37 °C, 5% CO2) até a próxima leitura.
- Quando terminar, transfira os dados de luminescência para um programa gráfico para plotar a unidade de luminescência relativa (RLU) (eixo y) para cada variável (por exemplo, produtos químicos ou concentrações) (eixo x) e analise os dados.
4. Validação do ensaio split-Rluc com outros métodos.
- Realize outros ensaios, como ensaio de ligação de proximidade (PLA), microscopia eletrônica de transmissão e monitoramento da captação de cálcio mitocondrial para validar os resultados obtidos do ensaio split-Rluc, conforme descrito anteriormente24.
Resultados
Usamos o protocolo descrito acima para medir o nível de contatos ER-mitocôndrias após a adição de três compostos conhecidos por inibir GTPases específicas. CDC42, RHO e RAC são GTPases que promovem a polimerização da actina28 quando ativadas e são inibidas por ZCL278, Rhosin e Ehop-016, respectivamente24. HEK293T células transfectadas com split-Rluc foram tratadas com DMSO (controle), ZCL278 (50 μM), Rhosin (50 μM) ou Ehop-016 (25 μM) e incubadas por 1 h, 2 h e 5 h. Usando um leitor de placas, medimos a atividade de luc split-Rem pontos de tempo definidos (Figura 2).
As células tratadas com rhosina (inibidor de RHO GTPase) não mostraram alterações significativas na atividade da luciferase em comparação com as células de controle tratadas com DMSO em 1 h, 2 h e 5 h do tratamento (p > 0,9999, p = 0,6956, p > 0,9999) (Figura 2). Esses resultados demonstram que a Rhosina não afeta os contatos ER-mitocôndrias, sugerindo que não há envolvimento da atividade da GTPase RHO na regulação dos contatos entre as duas organelas.
Em contraste, o inibidor da RAC GTPase Ehop-016 mostrou atividade de luciferase significativamente menor do que o DMSO em 1 h, 2 h e 5 h de tratamento (p = 0,0106, p = 0,0009, p = 0,0024) (Figura 2). Esses dados indicam que a atividade da RAC GTPase é necessária para manter a interação normal ER-mitocôndrias na célula.
Finalmente, o inibidor de CDC42 ZCL278 mostrou as mudanças mais drásticas na atividade da luciferase (Figura 2). As células tratadas com ZCL278 mostraram uma diminuição significativa na atividade da luciferase em comparação com o DMSO controle e tiveram os menores valores de p em todos os três pontos de tempo, mantendo um p < 0,0001 por toda parte. Nossos dados demonstram uma forte inibição do acoplamento ER-mitocôndria quando a atividade da GTPase CDC42 é bloqueada por ZCL278, consistente com nosso relatório anterior usando o ensaio original split-Rluc realizado em células co-transfectadas com dois vetores que codificam cada metade da split-luciferase separadamente24.
Dados os resultados impressionantes com o ZCL278, especialmente quando comparado aos outros inibidores da GTPase, a atividade do ZCL278 foi testada em várias concentrações [0,5 μM, 5 μM, 50 μM] contra um controle DMSO. As atividades da luciferase medidas em 1 h, 2 h e 5 h após o tratamento mostraram uma resposta dose-dependente; 0,5 μM de ZCL278 resultou no efeito mais fraco na atividade da luciferase, enquanto 5 μM mostrou mais forte e 50 μM mostrou a maior redução na atividade enzimática reconstituída (Figura 3). Juntos, nossos resultados mostram que o ZCL278 regula negativamente a atividade do split-Rluc de maneira dose-dependente e, com o passar do tempo, concentrações mais altas de ZCL278 (inibidor de CDC42) ainda mostram uma mudança significativa em relação ao DMSO controle em comparação com concentrações mais baixas.
Como forma de validar nosso ensaio split-Rluc, ensaios independentes bem estabelecidos, como ensaio de ligadura de proximidade (PLA), microscopia eletrônica de transmissão (TEM) e monitoramento da captação de cálcio mitocondrial, podem ser realizados (Figura 4 e dados não mostrados)24. O isoproterenol, um agonista do receptor β2 adrenérgico, que é um dos compostos identificados em nossa triagem para promotores de contato ER-mitocôndrias ( Figura 4A ), foi usado para confirmar que a atividade da luciferase do split-Rluc após o tratamento com isoproterenol foi aumentada devido ao aumento dos contatos ER-mitocôndrias, conforme demonstrado por mudanças na intensidade do sinal de PLA, bem como na captação de cálcio mitocondrial ( Figura 4B-E ).
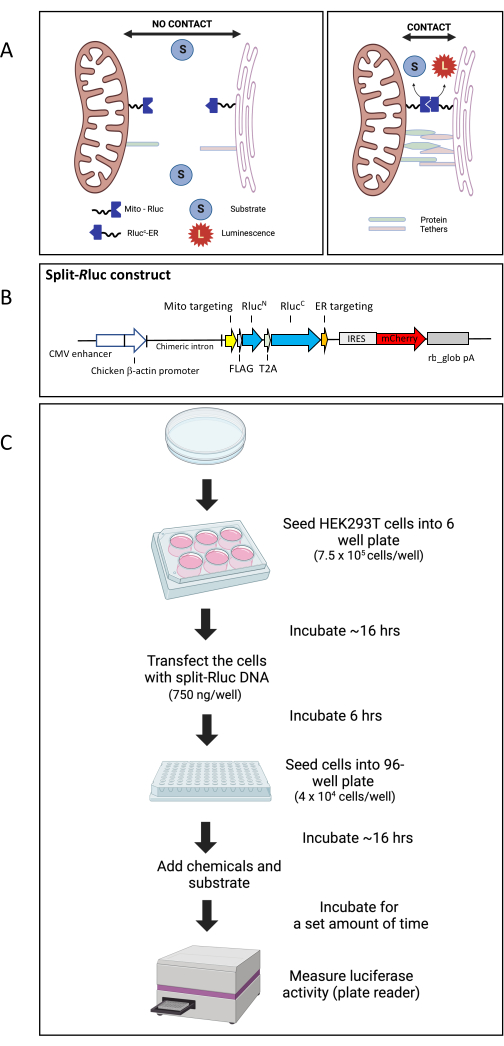
Figura 1: O ensaio de remontagem de Luc split-R. (A) Representação esquemática do ensaio Luc Split-R. Onde não há contatos de membrana entre as mitocôndrias e o retículo endoplasmático, o Mito-RlucN e o RlucC-ER não interagem e, portanto, não há atividade de Rluc8. No entanto, a interação entre o Mito-RlucN e o RlucC-ER, onde o ER e as membranas mitocondriais entram em contato, resulta em atividade enzimática completa, que é detectada pela conversão do substrato em luminescência. (B) Mapa esquemático da construção de DNA split-Rluc (pCAG-MitoRluc N-T2A-R lucCER-IRES-mCherry). (C) Diagrama do fluxo de trabalho para ensaio de luc split-Rem células HEK293T. Abreviaturas: Mito = mitocôndrias; ER = retículo endoplasmático; IRES, local de entrada ribossômico interno; rb_glob pA, sinal de poli adenilação da beta-globina de coelho; T2A, uma sequência do peptídeo 2A de autoclivagem do vírus Thosea asigna. Clique aqui para ver uma versão maior desta figura.

Figura 2: As atividades de luc split-Rcorrespondentes aos níveis de contatos ER-Mito nas células tratadas com inibidores da GTPase. (AC) Atividades de luciferase reconstituídas em (A) 1 h, (B) 2 h ou (C) 5 h após DMSO (controle), Rhosin [50 μM], Ehop-016 [25 μM] ou ZCL278 [50 μM] tratamento (n = 12 para cada grupo). Todos os dados foram analisados por meio de ANOVA one-way com comparações múltiplas. Os valores de p são relatados como p > 0,05 (ns), p ≤ 0,05 (*), p ≤ 0,01 (**), p ≤ 0,001 (***), p ≤ 0,0001 (****). As barras de erro indicam o erro médio ± padrão de medição. Abreviaturas: Mito = mitocôndrias; ER = retículo endoplasmático; RLU = unidade de luz relativa. Clique aqui para ver uma versão maior desta figura.

Figura 3: As atividades split-Rluc medindo os níveis de contatos ER-Mito nas células tratadas com ZCL278. (AC) Atividades de luciferase reconstituída em (A) 1 h, (B) 2 h ou (C) 5 h após DMSO (controle) ou ZCL278 na concentração [0,5 μM], [5 μM] ou [50 μM] (n = 12 para cada grupo). Todos os dados foram analisados por meio de ANOVA one-way com comparações múltiplas. Os valores de p são relatados como p > 0,05 (ns), p ≤ 0,05 (*), p ≤ 0,01 (**), p ≤ 0,001 (***), p ≤ 0,0001 (****). As barras de erro indicam o erro médio ± padrão de medição. Abreviaturas: Mito = mitocôndrias; ER = retículo endoplasmático; RLU = unidade de luz relativa. Clique aqui para ver uma versão maior desta figura.

Figura 4: Validação do ensaio de luc split-R. (A) Um exemplo de quantificação da atividade de Split-Rluc após o tratamento de células HEK293T com 11 drogas, incluindo isoproterenol, identificadas em uma triagem composta para promotores de contato ER-mitocôndrias. (B) Um exemplo de ensaio de ligação de proximidade em células HeLa que foram tratadas com DMSO ou isoproterenol (1 μM). O sinal PLA (vermelho) indica um contato próximo entre ER e Mito. Sec61-GFP (verde): para rotular ER; DAPI (azul): núcleo. (C) Quantificação do sinal de PLA em A (n = 4 experimentos com 77-97 células cada; Teste de Mann-Whitney). (D) Captação de cálcio mitocondrial em resposta à histamina (100 μM). A mudança de intensidade fluorescente Mito-R-GECO1 (a média foi obtida em várias células HeLa) plotada a cada 2,5 s em células tratadas com DMSO ou isoproterenol. A seta indica adição de histamina. (E) Esquerda: Pico máximo de captação de cálcio mitocondrial em A. Teste t bicaudal não pareado (n = 10, DMOS; 14, Isoproterenol). Direita: Taxa de captação de cálcio mitocondrial (como em A; 7,5 a 12,5 s). Teste de Mann-Whitney (n = 12, DMSO; 11, Isoproterenol). Essa figura é modificada de Lim et al.24. Abreviaturas: RLU = unidade de luz relativa; PLA = ensaio de ligadura de proximidade. Clique aqui para ver uma versão maior desta figura.
Tabela 1: Resumo das técnicas atuais para contatos ER-mitocôndrias. Lista de vantagens e limitações para cada método. Clique aqui para baixar esta tabela.
Arquivo Suplementar 1: Arquivo zip contendo sequência de DNA (formatos PDF e GBK) de pCAG-MitoRluc N-T2A-R lucCER-IRES-mCherry. Clique aqui para baixar este arquivo.
Figura Suplementar S1: Mapa de pCAG-MitoRluc N-T2A-R lucCER-IRES-mCherry. Clique aqui para baixar este arquivo.
Discussão
Usamos um ensaio de remontagem split-Renilla luciferase 8 (ensaio split-Rluc) para quantificar o nível de acoplamentos ER-mitocôndrias. Neste estudo, modificamos a construção original split-Rluc24 gerando um único vetor, pCAG-MitoRluc N-T2A-R lucCER-IRES-mCherry, codificando cada componente split-Rluc (MitoRlucN e RlucCER) e uma sequência de peptídeo 2A autoclivado do vírus Thosea asigna para garantir a expressão da mesma quantidade de cada metade dividida da luciferase. Como substrato para a luciferase Renilla, em vez de celenterazina, um substrato de células vivas modificado (derivado protegido da celenterazina; consulte a Tabela de Materiais) foi usado pelas seguintes razões. Primeiro, ao contrário da celenterazina, que gera autoluminescência em um ambiente aquoso (especialmente pior no soro), esse derivado protegido gera autoluminescência muito baixa (muitas vezes não detectável) em meio contendo 10% de soro, portanto, sua relação sinal-fundo é maior do que a gerada pela celenterazina. Em segundo lugar, diferentemente da celenterazina, onde a luminescência é mais forte logo após a adição do substrato, mas começa a diminuir muito rapidamente, a luminescência produzida pelo substrato vivo modificado atinge seu ponto mais alto atingível aproximadamente 1,5 h após a adição do substrato e permanece contínua por mais de 24 h. Essas mudanças na cinética da reação facilitam a medição da luminescência em uma placa de vários poços, o que é muito útil para triagem de alto rendimento. Além disso, a luminescência produzida pelo substrato protegido permanecendo constante a longo prazo permite a medição de um único conjunto de amostras em vários pontos de tempo, em oposição à medição de vários conjuntos de amostras usadas em vários pontos de tempo (ponto único por amostra).
Usando esta versão atualizada da construção split-Rluc, testamos se as GTPases RHO, RAC e CDC42 participam da formação de contatos ER-Mito. Adicionamos inibidores de GTPase, Rhosin (inibidor de RHO), Ehop-016 (inibidor de RAC) e ZCL278 (inibidor de CDC42), para HEK293T células que expressam componentes de luc split-R. Os resultados do nosso ensaio indicam que o ZCL278 é o inibidor mais potente da formação de contatos ER-mitocôndrias e o Ehop-016 é o próximo inibidor significativo, enquanto a Rhosin não afetou seus contatos. Esses dados sugerem que a polimerização de actina mediada por CDC42 (assim como a mediada por RAC em menor grau) é necessária para manter os contatos ER-mitocôndrias na célula, enquanto a polimerização de actina mediada por RHO provavelmente não é importante.
Para garantir a máxima eficiência, há muitas etapas críticas que devem ser consideradas para este protocolo. Em primeiro lugar, a quantidade de construção transfectada deve ser testada, pois muito DNA pode ter um efeito tóxico nas células. Em segundo lugar, como o número de células pode ter um impacto significativo nos níveis de luciferase medidos, manter a densidade das células tão próxima quanto o que foi declarado no protocolo é crucial para garantir a confluência ideal e consistente de 80% das células no momento do ensaio. Pode haver uma redução na luminescência se as células atingirem a confluência durante o tempo de exposição química, pois as células confluentes produzem menos proteína em geral. Terceiro, para garantir que as células não se desprendam durante a adição de produtos químicos e substrato, usamos uma placa de 96 poços pré-revestida com Poli-D-Lisina. Em quarto lugar, para os compostos (incluindo o substrato) usados neste protocolo, é importante mantê-los a -20 °C ou menos (-70 °C) em alíquotas e adicioná-los às células imediatamente descongeladas. Por fim, devido a possíveis flutuações de temperatura que podem afetar a atividade da luciferase, pode ser melhor optar por usar os poços mais internos da placa e ignorar os poços que revestem a parte externa da placa.
O ensaio split-Rluc8 oferece um método fácil, mas robusto, para determinar os contatos ER-mitocôndrias, ideal para triagem de alto rendimento; no entanto, existem algumas limitações que precisam ser observadas. Para a fisiologia celular, há a questão das mudanças no comportamento celular devido à constante superexpressão do construto DNA. Atualmente, estamos trabalhando em uma construção com um promotor induzível para combater as possíveis mudanças na fisiologia celular devido à superexpressão constante. Além disso, quando este ensaio é usado para fins de triagem, deve-se notar que resultados falso-positivos podem surgir de qualquer efeito indireto dos medicamentos em i) níveis de expressão, ii) eficiências direcionadas a organelas dos referidos fragmentos de proteínas, ou iii) atividades enzimáticas das luciferases reconstituídas. Devido a essas limitações, este ensaio deve ser testado em conjunto com outros métodos para validar ainda mais os resultados. A Tabela 1 refere-se a vários métodos de teste desses locais de contato, juntamente com suas vantagens e desvantagens. Aproveitamos outros métodos, incluindo, mas não se limitando a, ensaios de ligação de proximidade (PLA), que detectam proximidade entre proteínas, e microscopia eletrônica de transmissão (TEM), que permite uma visualização clara dos contatos de organelas, em conjunto com o ensaio split-Rluc para validar que as alterações resultantes na RLU são de fato devidas a mudanças nos locais de contato ER-mitocôndrias (Figura 4 e Lim et al.24). Também mostramos uma correlação entre alteração nos contatos ER-mitocôndrias e parâmetros funcionais, como transferência de cálcio e fissão mitocondrial (Figura 4 e Lim et al.24). Para garantir que o efeito dos medicamentos na atividade split-Rluc não seja devido ao aumento da expressão do gene split-Rluc, um inibidor de transcrição ou tradução pode ser adicionado junto com os medicamentos, como fizemos anteriormente24.
Para a aplicação do sistema split-Rluc em neurônios diferenciados em cultura onde a taxa de transfecção é baixa, a infecção viral (com lentivírus expressando componentes split-Rluc) seria uma abordagem alternativa. Seu uso pode ser estendido para criar um sistema de triagem onde as células (por exemplo, células-tronco pluripotentes induzidas por humanos) são projetadas para expressar de forma estável cada componente do split-Rluc, MitoRlucN e RlucCER, seja por infecção viral ou por edição de genes mediada por CRSPR. No entanto, para um sistema de expressão estável, deve-se estar ciente de que pode haver uma pressão de seleção para células que toleram melhor o sistema split-Rluc, o que pode não ser representativo do estado fisiológico normal. Essa adaptação ou seleção pode introduzir viés nos resultados experimentais.
Em resumo, apesar de algumas das limitações discutidas acima, o split-Rluc é um ensaio rápido, direto e poderoso para triagem de alto rendimento para pequenas moléculas ou genes que regulam os contatos inter-organelas na célula, como foi demonstrado em nosso estudo anterior de triagem de drogas24.
Divulgações
Os autores declaram não haver conflitos de interesse.
Agradecimentos
Os autores agradecem ao Dr. Jeffrey Golden (Cedars-Sinai Medical Center) pela revisão crítica do manuscrito. Este trabalho foi financiado em parte pelo Instituto Nacional de Doenças Neurológicas e Derrame (NINDS, R01NS113516).
Materiais
| Name | Company | Catalog Number | Comments |
| 1.7 mL SafeSeal Microcentrifuge Tube | Sorenson | 16070 | |
| 6-well plate TC Treated | USA Scientific | CC7682-7506 | |
| 10 mL Pipette Tips OneTip | USA Scientific | 1110-3700 | |
| 10 μL pipette tips OneTip | USA Scientific | 1110-3700 | |
| 20-200 μL Beveled tips OneTip | USA Scientific | 1111-1210 | |
| 50 mL Polypropylene Conical Tube | Falcon | 352070 | |
| 96-Well Flipper Microtube Racks | ThermoFisher Scientific | 8770-11 | |
| 96-well plate TC Treated | USA Scientific | CC7682-7596 | |
| 100 mm x 20 mm TC Treated Dish | USA Scientific | CC7682-3394 | |
| 1250 μL Tips OneTip | USA Scientific | 1112-1720 | |
| Centrifuge 5910 Ri - Refrigerated Centrifuge | Eppendorf | 5943000131 | |
| Dimethyl sulfoxide, anhydrous, ≥99.9% | Sigma-Aldrich | 276855-100ML | |
| DMEM, high glucose | ThermoFisher Scientific | 11965092 | |
| DPBS, no calcium, no magnesium | ThermoFisher Scientific | 14190144 | |
| EHop 016 | Bio-Techne Tocris | 6248 | Dissolve in DMSO; store at -70 °C |
| EnduRen Live Cell Substrate | Promega | E6481 | Store aliquots at -70 °C |
| Eppendorf 2-20 μL pipette | Eppendorf | 3123000039 | |
| Eppendorf Research plus 100-1000 μL pipette | Eppendorf | 3123000063 | |
| Eppendorf Research Plus 1-10 µL pipette | Eppendorf | 3123000020 | |
| Eppendorf Research plus 12-channel | Eppendorf | 3125000028 | |
| Eppendorf Research plus 200 μL pipette | Eppendorf | 3123000055 | |
| Fetal Bovine Serum, qualified, USDA-approved regions | ThermoFisher Scientific | 10437028 | |
| Forma Steri-Cycle CO2 Incubator, 184 L, Polished Stainless Steel | ThermoFisher Scientific | 381 | |
| Hand tally counter | Sigma-Aldrich | HS6594 | |
| HEK 293T Cells | ATCC | CRL-3216 | |
| Hemacytometer - Neubauer Bright Line, Double-Counting Chamber | LW Scientific | CTL-HEMM-GLDR | |
| Invitrogen TE Buffer | ThermoFisher Scientific | 8019005 | |
| Microscope | Zeiss | Axiovert 25 CFL | |
| Mini centrifuge | Benchmark Scientific | C1012 | |
| Multi Tube Rack For 50ml Conical, 15ml Conical, And Microcentrifuge Tubes | Boekel Scientific | 120008 | |
| PEI MAX - Transfection Grade Linear Polyethylenimine Hydrochloride (MW 40,000) | Polysciences | 24765-100MG | |
| Pipet-Aid XP | USA Scientific | 4440-0101 | |
| Poly-D-lysine hydrobromide | Sigma-Aldrich | P6407-5MG | |
| Rhosin hydrochloride | Bio-Techne Tocris | 5003 | Dissolve in DMSO; store at -70 °C |
| Trypsin-EDTA (0.05%), phenol red | ThermoFisher Scientific | 25300054 | |
| Varioskan LUX multimode microplate reader | ThermoFisher Scientific | VL0000D0 | |
| Vortex | ThermoFisher Scientific | 2215365 | level 8 |
| VWR Vacuum Aspiration System | VWR | 75870-734 | |
| ZCL 278 | Bio-Techne Tocris | 4794 | Dissolve in DMSO; store at -70 °C |
Referências
- Aoyama-Ishiwatari, S., Hirabayashi, Y. Endoplasmic reticulum-mitochondria contact sites-emerging intracellular signaling hubs. Front Cell Dev Biol. 9, 653828(2021).
- Sassano, M. L., Felipe-Abrio, B., Agostinis, P. ER-mitochondria contact sites; a multifaceted factory for Ca2+ signaling and lipid transport. Front Cell Dev Biol. 10, 988014(2022).
- Scorrano, L., et al. Coming together to define membrane contact sites. Nat Commun. 10 (1), 1287(2019).
- Voeltz, G. K., Sawyer, E. M., Hajnóczky, G., Prinz, W. A. Making the connection: How membrane contact sites have changed our view of organelle biology. Cell. 187 (2), 257-270 (2024).
- Paillusson, S., et al. There's something wrong with my MAM; the ER-mitochondria axis and neurodegenerative diseases. Trends Neurosci. 39 (3), 146-157 (2016).
- Sasi, U. S. S., Sindhu, G., Raj, P. S., Raghu, K. G. Mitochondria associated membranes (MAMs): emerging drug targets for diabetes. Curr Med Chem. 27 (20), 3362-3385 (2019).
- Joshi, A. U., Kornfeld, O. S., Mochly-Rosen, D. The entangled ER-mitochondrial axis as a potential therapeutic strategy in neurodegeneration: A tangled duo unchained. Cell Calcium. 60 (3), 218-234 (2016).
- Moltedo, O., Remondelli, P., Amodio, G. The mitochondria-endoplasmic reticulum contacts and their critical role in aging and age-associated diseases. Front Cell Dev Biol. 7, 172(2019).
- Rieusset, J. The role of endoplasmic reticulum-mitochondria contact sites in the control of glucose homeostasis: an update. Cell Death Dis. 9 (3), 388(2018).
- Bouguerra, M. D., Lalli, E. ER-mitochondria interactions: Both strength and weakness within cancer cells. Biochim Biophys Acta Mol Cell Res. 1866 (4), 650-662 (2019).
- Tepikin, A. V. Mitochondrial junctions with cellular organelles: Ca2+ signalling perspective. Pflügers Arch. 470 (8), 1181-1192 (2018).
- Masson, G. L., Przedborski, S., Abbott, L. F. A computational model of motor neuron degeneration. Neuron. 83 (4), 975-988 (2014).
- Giamogante, F., Barazzuol, L., Brini, M., Calì, T. ER-mitochondria contact sites reporters: strengths and weaknesses of the available approaches. Int J Mol Sci. 21 (21), 8157(2020).
- Rizzuto, R., et al. Close contacts with the endoplasmic reticulum as determinants of mitochondrial Ca2+ responses. Science. 280 (5370), 1763-1766 (1998).
- Valm, A. M., et al. Applying systems-level spectral imaging and analysis to reveal the organelle interactome. Nature. 546 (7656), 162-167 (2017).
- Csordás, G., et al. Imaging interorganelle contacts and local calcium dynamics at the ER-mitochondrial interface. Mol Cell. 39 (1), 121-132 (2010).
- Cieri, D., et al. SPLICS: a split green fluorescent protein-based contact site sensor for narrow and wide heterotypic organelle juxtaposition. Cell Death Differ. 25 (6), 1131-1145 (2018).
- Kakimoto, Y., et al. Visualizing multiple inter-organelle contact sites using the organelle- targeted split-GFP system. Sci Rep. 8 (1), 6175(2018).
- Wu, M. M., Covington, E. D., Lewis, R. S. Single-molecule analysis of diffusion and trapping of STIM1 and Orai1 at ER-plasma membrane junctions. Mol Biol Cell. 25 (22), Mac.E14-06-1107 (2014).
- Csordas, G., et al. Structural and functional features and significance of the physical linkage between ER and mitochondria. J Cell Biol. 174 (7), 915-921 (2006).
- de Brito, O. M., Scorrano, L. Mitofusin 2 tethers endoplasmic reticulum to mitochondria. Nature. 456 (7222), 605-610 (2008).
- Kremer, A., et al. Developing 3D SEM in a broad biological context. J Microsc. 259 (2), 80-96 (2015).
- Söderberg, O., et al. Direct observation of individual endogenous protein complexes in situ by proximity ligation. Nat Methods. 3 (12), 995-1000 (2006).
- Lim, Y., Cho, I. -T., Rennke, H. G., Cho, G. β2-adrenergic receptor regulates ER-mitochondria contacts. Sci Rep. 11 (1), 21477(2021).
- Lam, S. S., et al. Directed evolution of APEX2 for electron microscopy and proximity labeling. Nat Methods. 12 (1), 51-54 (2014).
- Cho, I. -T., et al. Ascorbate peroxidase proximity labeling coupled with biochemical fractionation identifies promoters of endoplasmic reticulum-mitochondrial contacts. J Biol Chem. 292 (39), 16382-16392 (2017).
- Lim, Y., Cho, I. -T., Schoel, L. J., Cho, G., Golden, J. A. Hereditary spastic paraplegia-linked REEP1 modulates endoplasmic reticulum/mitochondria contacts. Ann Neurol. 78 (5), 679-696 (2015).
- Arnold, T. R., Stephenson, R. E., Miller, A. L. Rho GTPases and actomyosin: Partners in regulating epithelial cell-cell junction structure and function. Exp Cell Res. 358 (1), 20-30 (2017).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados