Method Article
Essai de réassemblage de la luciférase fractionnée pour mesurer les contacts du réticulum endoplasmique et des mitochondries dans des cellules vivantes
Dans cet article
Résumé
Nous avons mis au point un test de réassemblage de la luciférase fractionnée pour surveiller les contacts réticulum-mitochondrie endoplasmique dans les cellules vivantes. À l’aide de ce test, nous décrivons un protocole permettant de mesurer quantitativement le niveau de ces couplages inter-organites dans HEK293T cellules, dans des conditions de traitement chimique.
Résumé
Les sites de contact du réticulum endoplasmique (RE)-mitochondries jouent un rôle essentiel dans la santé cellulaire et l’homéostasie, tels que la régulation du Ca2+ et de l’homéostasie lipidique, la dynamique mitochondriale, la biogenèse des autophagosomes et des mitophagosomes et l’apoptose. L’incapacité à maintenir un couplage ER-mitochondrial, normal, est impliquée dans de nombreuses maladies neurodégénératives telles que la maladie d’Alzheimer, la maladie de Parkinson, la sclérose latérale amyotrophique et la paraplégie spastique héréditaire. Il est d’une importance considérable d’explorer comment la dérégulation des contacts mitochondriaux RE pourrait conduire à la mort cellulaire et si la réparation de ces contacts à un niveau normal pourrait améliorer les conditions neurodégénératives. Ainsi, des tests améliorés qui mesurent le niveau de ces contacts pourraient aider à éclairer les mécanismes pathogènes de ces maladies. À terme, la mise en place de dosages simples et fiables facilitera le développement de nouvelles stratégies thérapeutiques. Nous décrivons ici un test de luciférase fractionnée pour mesurer quantitativement le niveau de contacts entre les mitochondries du RE et les cellules vivantes. Ce test peut être utilisé pour étudier le rôle physiopathologique de ces contacts ainsi que pour identifier leurs modulateurs dans le criblage à haut débit.
Introduction
Les interactions entre le RE et les mitochondries sont vitales pour l’homéostasie cellulaire et la survie 1,2,3,4. Des preuves antérieures indiquent que tout type de perturbation ou de dérégulation des sites de contact entre les mitochondries du RE peut contribuer à plusieurs maladies neurodégénératives, métaboliques et cardiovasculaires, ainsi qu’au cancer 5,6,7,8,9,10. Par exemple, une augmentation anormale de l’absorption de Ca2+ dans les mitochondries peut entraîner la mort cellulaire en ouvrant les pores de transition de perméabilité des mitochondries, qui sont couramment observés dans certains modèles de la maladie d’Alzheimer 5,11. De même, la réduction des contacts entre les mitochondries du RE peut entraîner une diminution de la production d’ATP et une altération de l’apport en Ca2+, comme on l’a vu dans des modèles de sclérose latérale amyotrophique 5,11,12. Au fur et à mesure que de plus en plus d’études sont menées dans le domaine des contacts RE-mitochondries, d’autres protéines et gènes liés à la maladie qui pourraient affecter ces contacts sont découverts. Malgré les connaissances actuelles et les preuves montrant le rôle des sites de contact RE-mitochondries, beaucoup de travail reste encore à faire pour élucider comment ces contacts pourraient entraîner une perte de fonction cellulaire et, en fin de compte, la mort cellulaire.
Diverses méthodes ont été développées pour évaluer la proximité des deux membranes, la morphologie structurelle et la distance entre les deux sites de contact des organites 3,4,13. Les approches de surveillance du couplage RE-mitochondries comprennent l’imagerie basée sur des marqueurs de fluorescence14,15, l’imagerie basée sur le rapporteur FRET16 et l’imagerie basée sur une sonde de fluorescence divisée17,18, qui utilisent l’épifluorescence et la microscopie confocale. La microscopie à super-résolution et la microscopie à résolution atomique sont également des outils puissants pour visualiser avec précision les contacts entre organites, bien que leur utilisation dans l’analyse des sites de contact soit encore limitée car elles nécessitent des microscopes hautement dédiés et une expertise technique19. De plus, la microscopie électronique à transmission (MET), la microscopie électronique à balayage (MEB) et d’autres techniques EM telles que la tomographie électronique (TE) et la cryomicroscopie électronique sont couramment utilisées car elles fournissent une imagerie ultrastructurale à haute résolution des sites de contact, qui sont souvent impossibles à explorer à l’aide d’autres approches expérimentales 20,21,22. Cependant, ces méthodes basées sur les EM sont une technique à très faible débit qui peut également être affectée par les procédures de fixation chimique. Plus récemment, des méthodes basées sur le marquage de proximité ont été utilisées pour détecter les sites de contact ainsi que pour identifier de nouvelles protéines de site de contact. Par exemple, le test de ligature de proximité (PLA) a été utilisé pour quantifier la proximité des organites23,24, tandis qu’une version révisée du test de l’ascorbate peroxydase (APEX) a été utilisée pour identifier de nouvelles protéines de site de contact25,26. Il est important de reconnaître que toutes ces méthodes décrites ci-dessus ont des forces et des limites intrinsèques dans la détection des contacts entre les organites. Ainsi, l’appariement de différentes techniques est nécessaire pour obtenir une interprétation approfondie des sites de contact des organites.
Auparavant, nous avons établi le test de réassemblage de la Renilla luciférase 8 (test Split-Rluc) pour surveiller le niveau de contacts membranaires des mitochondries du RE (Figure 1A)24,26,27. En bref, chaque moitié fendue de Renilla luciferase est conjuguée à une séquence ciblant le RE ou les mitochondries. Lorsqu’elle est transfectée ensemble, chaque moitié fendue de l’enzyme est exprimée soit dans le RE, soit dans la membrane mitochondriale. Lorsque le RE et les mitochondries sont positionnés à proximité l’une de l’autre, les moitiés fendues se rejoignent et reconstituent l’ensemble de l’enzyme avec l’activité de la luciférase. Pour la construction split-Rluc, nous avons utilisé Renilla luciferase 8 (Rluc8) dans pBAD/Myc-His27 pour le modèle initial. Le site de division (entre les acides aminés 91 et 92) a été déterminé sur la base des rapports précédents27. Pour la moitié N-terminale du Rluc8, les séquences d’ADN des acides aminés 1-91 du Rluc8 ont été fusionnées à l’extrémité 3' de l’étiquette FLAG et à la séquence de ciblage mitochondriale AKAP1 de la souris dans le vecteur TOPO pcDNA3.1 par PCR27. Pour la moitié C-terminale ciblée sur le RE, les séquences d’ADN qui codent pour les acides aminés 92-311 ont été fusionnées à l’extrémité 5' de l’étiquette myc et à la séquence de localisation du RE UBC6 de la levure. Ici, nous avons mis à niveau la construction du plasmide luc split-Rde sorte que les moitiés fendues de la Renilla luciferase sont exprimées dans un seul vecteur (pCAG) sous le même promoteur, puis clivées en deux fragments lorsque T2A, une séquence 2A du peptide 2A auto-clivant du virus Thosea asigna, est insérée entre les deux moitiés divisées (Figure 1B). La carte et les séquences de l’ADN plasmidique sont fournies dans le fichier supplémentaire 1 et la figure supplémentaire S1. À l’aide de ce système, nous avons mesuré les effets de trois substances chimiques (inhibitrices des GTPases impliquées dans la polymérisation de l’actine) sur les contacts RE-mitochondries. Ce test luc split-Rest un système de test simple mais robuste pour le criblage à haut débit de modulateurs de contact inter-organites24.
Protocole
1. Entretien des cellules et ensemencement (Jour 1)
- Conserver HEK293T cellules dans un milieu de culture cellulaire contenant du milieu modifié Eagle de Dulbecco (DMEM) avec 10 % de sérum fœtal bovin (FBS) (dans des boîtes de culture de 100 mm) dans un incubateur humidifié à 37 °C avec 5 % de CO2.
- Avant de commencer, vérifiez la confluence de la plaque en l’observant au microscope. Lorsque les cellules atteignent environ 80 à 90 % de confluence, préparez-vous à ensemencer les cellules dans une plaque de culture à 6 puits en retirant le milieu et en les lavant avec 10 ml de solution saline tamponnée au phosphate de Dulbecco (DPBS).
- Retirer le DPBS de la boîte de culture et traiter les cellules avec 1 mL de rouge phénolique Trypsine-EDTA à 0,05 %. Incuber pendant 3 min, puis sortir la plaque de l’incubateur et ajouter 9 mL de milieu de culture (DMEM avec 10 % de FBS) pour arrêter la réaction de la trypsine. Pipetez lentement de haut en bas pour vous assurer que toutes les cellules encore collées à la plaque sont délogées. Transférez la suspension cellulaire dans un tube de 50 ml.
- Centrifuger à 300 × g pendant 5 min à température ambiante. Retirer le milieu et remettre en suspension les granules de cellules avec 1 mL de milieu frais (DMEM + 10 % FBS).
- Comptez le nombre de cellules à l’aide d’un hémocytomètre.
- Assurez une distribution uniforme des cellules en faisant tourner doucement le tube. Retirer immédiatement 10 μL de la suspension cellulaire et l’introduire dans un tube de microcentrifugation de 1,7 mL. Ajoutez 90 μL de fluide dans le même tube.
- Après avoir mélangé doucement les cellules et les milieux, prélever la suspension cellulaire et appliquer 10 μL dans chacune des chambres de l’hémocytomètre en pipetant doucement sous la lamelle.
- À l’aide d’un objectif 10x, faites la mise au point au microscope sur les lignes de la grille de l’hémocytomètre. Une fois sélectionné, comptez le nombre de cellules dans un ensemble de 16 cases (4 x 4 cases) à l’aide d’un compteur de comptage de main. Répétez l’opération jusqu’à ce que les quatre séries de 16 carrés soient comptées.
- Calculez le nombre total de cellules/ml en prenant le nombre moyen de cellules (de chacun des 16 carrés d’angle) et en le multipliant par 105.
REMARQUE : Au lieu d’utiliser un hémocytomètre pour compter les cellules, un compteur de cellules automatisé peut être utilisé selon les instructions du fabricant.
- Une fois le comptage des cellules terminé, préparez-vous à plaquer les cellules dans une plaque à 6 puits à 7,5 × 10 à une densité de5 cellules/puits avec 2 mL de milieu/puits. Dans un tube de 50 ml, ajoutez le volume nécessaire de cellules ainsi que 13 ml de milieu. Versez 2 ml de la suspension cellulaire diluée dans chacun des 6 puits.
- Avant le placage, calculer le nombre de cellules nécessaires pour 7,5 × 105 cellules/puits en utilisant C1V1 = C2V2 où C1 est le nombre initial de cellules (nombre de cellules/mL), V1 est le volume nécessaire à partir de la suspension cellulaire initiale, C2 est la densité cellulaire cible souhaitée (nombre de cellules/mL), et V2 est le volume final nécessaire à l’ensemencement cellulaire.
- Après la distribution, tapotez doucement la plaque de tous les côtés pour étaler les cellules, puis placez-la dans un incubateur humidifié à 37 °C avec 5% de CO2 pendant la nuit.
2. Transfection cellulaire médiée par la polyéthylèneimine (PEI) et ensemencement cellulaire post-transfection (jour 2)
- Retirez la plaque à 6 puits de l’incubateur et aspirez le milieu de culture existant de chaque puits. Ajouter 2 ml de milieu frais par puits.
- Transfectez les cellules avec l’ADN plasmidique split-Rluc (pCAG-MitoRluc N-T2A-R lucCER-IRES-mCherry) dissous dans un tampon TE (pH 8,0).
- Pour chaque puits, mélangez l’ADN (750 ng) et le PEI (rapport ADN/PEI = 1:3) dans 200 μL de DMEM dans un tube de microcentrifugation de 1,7 mL. Vortex rapidement l’ADN/PEI au niveau 8 pendant environ 2 s pour s’assurer qu’ils se mélangent bien pour former un complexe. Après cela, faites-le tourner brièvement dans une mini-centrifugeuse (<3 s).
- Laissez le mélange ADN/PEI incuber à température ambiante pendant 15 min, puis ajoutez le mélange (en le laissant tomber) à la surface du milieu de culture dans la plaque à 6 puits contenant les cellules. Tapotez doucement la plaque (pour répartir uniformément le mélange) et incubez-la dans un incubateur humidifié à 37 °C avec 5% de CO2 pendant 6 h.
- Pendant l’incubation, préparer une plaque à 96 puits recouverte de poly-D-lysine (PDL) en ajoutant d’abord 70 μL de PDL (50 μg/mL dans le DPBS) dans chaque puits et en l’incubant dans un incubateur humidifié à 37 °C avec 5 % de CO2 pendant au moins 1 h. Retirer la solution de PDL, puis laver chaque puits 2 fois avec 100 μL de DPBS. À la fin du deuxième lavage, assurez-vous que tous les DPBS restants sont retirés.
- 6 h après la transfection, préparez-vous à ensemencer les cellules transfectées dans une plaque à 96 puits recouverte de PDL.
- Sortez de l’incubateur la plaque à 6 puits contenant les cellules transfectées, aspirez le milieu et lavez chaque puits avec 2 ml de DPBS. Après avoir retiré le DPBS, ajoutez 350 μL de rouge phénol Trypsine-EDTA à 0,05 % et incubez pendant 1 à 2 min. Pour arrêter la réaction de la trypsine, ajoutez 1,7 mL de milieu dans chaque puits.
- Après avoir transféré les cellules et le milieu dans un tube de 50 ml, faites tourner le tube à 300 × g pendant 5 min dans une centrifugeuse de table. Aspirez le milieu et remettez en suspension la pastille de cellule avec 1 mL de milieu frais. Comptez le nombre de cellules à l’aide d’un hémocytomètre (comme décrit à l’étape 1.5).
- Ensemencer les cellules dans une plaque de 96 puits recouverte d’un PDL à une densité de 4 × 104 cellules/puits dans 100 μL de milieu de culture. Pour cela, dans un tube de 50 ml, ajoutez le volume nécessaire de cellules (voir ci-dessous pour le calcul) ainsi que le volume approprié de média (volume total de 12 ml). À l’aide d’une pipette multicanaux, versez 100 μL de suspension cellulaire diluée dans chacun des 96 puits. Incuber les cellules pendant 18 h dans un incubateur humidifié à 37 °C avec 5% de CO2.
- Calculez le nombre de cellules nécessaires pour 4 × 104 cellules/puits à l’aide de C1V1 = C2V2 où C1 est le nombre initial de cellules (nombre de cellules/mL), V1 est le volume nécessaire à partir de la suspension cellulaire initiale, C2 est la densité cellulaire cible souhaitée (nombre de cellules/mL) et V2 est le volume final nécessaire pour l’ensemencement des cellules.
3. Traitement chimique et dosage de la luciférase dans des cellules vivantes (Jour 3)
- Dans un tube à microfuge de 1,7 mL, préparer les trois solutions fraîches de 50 μM de rhosine (50 mM de DMSO), de 25 μM d’Ehop-016 (25 mM de DMSO) et de 50 μM de ZCL278 (50 mM de DMSO) en diluant chaque solution mère (50 mM de rhosine, 25 mM d’Ehop-016 et 50 mL de ZCL278 ; toutes dans du DMSO) à 1:1 000 dans la quantité requise (50 μL/puits × nombre de puits) de milieu de culture. À chaque solution, ajouter un substrat de cellules vivantes (stock de 50 mM dans du DMSO) pour Renilla luciferase diluée à 1:2 000 jusqu’à une concentration finale de 25 μM. Pour fabriquer des solutions ZCL278 de 0,5, 5 et 50 μM, effectuez des dilutions en série en utilisant le ZCL278 initial de 50 μM dans un milieu de culture.
REMARQUE : Si le temps d’incubation du produit chimique est inférieur à 1-1,5 h, les cellules peuvent être préincubées avec un substrat de cellules vivantes (pour Renilla luciferase) pendant 1-1,5 h avant d’être traitées avec chaque produit chimique. Nous ajoutons simultanément des produits chimiques et un substrat de cellules vivantes afin d’optimiser le protocole de criblage de médicaments à haut débit. - Retirez le milieu de chaque puits de la plaque de 96 puits contenant les cellules transfectées et ajoutez 50 μL de mélange de produits chimiques et de substrat dans chaque puits.
- Après avoir incubé les cellules pendant 1 h, 2 h ou 5 h, sortez la plaque de culture de l’incubateur, chargez-la sur le lecteur de plaque de luminescence et mesurez la luminescence.
- Avant de commencer, assurez-vous que le lecteur de plaques est allumé. Accédez au logiciel de lecture de microplaques et créez un nouveau fichier en cliquant sur Nouvelle session. Réglez la température du lecteur de plaques sur 37 °C en cliquant sur Incubateur, en cochant l’option de température et en tapant 37 °C. Sous Disposition des plaques, choisissez l’option Inconnu et spécifiez les puits à mesurer en cliquant et en faisant glisser.
- Sous Protocole, cliquez sur Luminescence et conservez les paramètres par défaut. Une fois la configuration terminée, chargez la plaque avec le couvercle retiré dans le lecteur de plaques et choisissez Run Plate In. Cliquez sur Démarrer et la fenêtre d’enregistrement du fichier apparaîtra. Enregistrez le fichier sur votre bureau en le renommant et en cliquant sur Enregistrer.
REMARQUE : La luminescence continuera à être produite pendant >24 h après l’ajout du substrat de cellule vivante.
- Une fois la lecture terminée, retirez la plaque du lecteur de plaques, puis cliquez sur Exécuter la plaque pour remettre le lecteur en place. Une fois terminé, replacez la plaque dans l’incubateur humidifié (37 °C, 5% CO2) jusqu’à la lecture suivante.
- Lorsque vous avez terminé, transférez les données de luminescence dans un programme graphique pour tracer l’unité de luminescence relative (RLU) (axe y) pour chaque variable (par exemple, produits chimiques ou concentrations) (axe x) et analysez les données.
4. Validation du dosage split-Rluc avec d’autres méthodes.
- Effectuer d’autres tests tels que le test de ligature de proximité (PLA), la microscopie électronique à transmission et la surveillance de l’absorption du calcium mitochondrial pour valider les résultats obtenus du test split-Rluc comme décrit précédemment24.
Résultats
Nous avons utilisé le protocole décrit ci-dessus pour mesurer le niveau de contacts RE-mitochondries lors de l’ajout de trois composés connus pour inhiber des GTPases spécifiques. CDC42, RHO et RAC sont des GTPases qui favorisent la polymérisation de l’actine28 lorsqu’elles sont activées et sont inhibées par ZCL278, Rhosine et Ehop-016, respectivement24. HEK293T cellules transfectées avec split-Rluc ont été traitées avec du DMSO (témoin), du ZCL278 (50 μM), de la Rhosine (50 μM) ou de l’Ehop-016 (25 μM) et incubées pendant 1 h, 2 h et 5 h. À l’aide d’un lecteur de plaques, nous avons mesuré l’activité de split-Rluc à des moments définis (Figure 2).
Les cellules traitées à la rhosine (inhibiteur de la GTPase RHO) n’ont montré aucun changement significatif de l’activité de la luciférase par rapport aux cellules témoins traitées au DMSO à 1 h, 2 h et 5 h du traitement (p > 0,9999, p = 0,6956, p > 0,9999) (Figure 2). Ces résultats démontrent que la rhosine n’affecte pas les contacts ER-mitochondries, suggérant qu’il n’y a pas d’implication de l’activité de la GTPase RHO dans la régulation des contacts entre les deux organites.
En revanche, l’inhibiteur de la GTPase RAC, Ehop-016, a montré une activité luciférase significativement plus faible que celle du DMSO à 1 h, 2 h et 5 h du traitement (p = 0,0106, p = 0,0009, p = 0,0024) (Figure 2). Ces données indiquent que l’activité de la GTPase RAC est nécessaire au maintien d’une interaction normale entre le RE et les mitochondries dans la cellule.
Enfin, l’inhibiteur de CDC42, ZCL278, a montré les changements les plus drastiques de l’activité de la luciférase (Figure 2). Les cellules traitées au ZCL278 ont montré une diminution significative de l’activité de la luciférase par rapport au DMSO témoin et ont présenté les valeurs p les plus faibles aux trois points temporels, maintenant un p < 0,0001 tout au long de l’étude. Nos données démontrent une forte inhibition du couplage ER-mitochondries lorsque l’activité de la GTPase CDC42 est bloquée par ZCL278, conformément à notre rapport précédent utilisant le test original split-Rluc réalisé dans des cellules co-transfectées avec deux vecteurs codant pour chaque moitié de la split-luciferase séparément24.
Compte tenu des résultats frappants obtenus avec ZCL278, en particulier par rapport aux autres inhibiteurs de la GTPase, l’activité de ZCL278 a été testée à diverses concentrations [0,5 μM, 5 μM, 50 μM] contre un témoin DMSO. Les activités de la luciférase mesurées 1 h, 2 h et 5 h après le traitement ont montré une réponse dose-dépendante ; 0,5 μM de ZCL278 a entraîné l’effet le plus faible sur l’activité de la luciférase, tandis que 5 μM ont montré une réduction plus forte et 50 μM ont montré la plus forte réduction de l’activité enzymatique reconstituée (Figure 3). Ensemble, nos résultats montrent que ZCL278 régule à la baisse l’activité de split-Rluc de manière dose-dépendante et, au fil du temps, des concentrations plus élevées de ZCL278 (inhibiteur de CDC42) montrent toujours un changement significatif par rapport au DMSO témoin par rapport aux concentrations plus faibles.
Afin de valider notre dosage du luc split-R, il est possible d’effectuer des tests indépendants bien établis, tels que le test de ligature de proximité (PLA), la microscopie électronique à transmission (MET) et la surveillance de l’absorption du calcium mitochondrial (figure 4 et données non présentées)24. L’isoprotérénol, un agoniste des récepteurs adrénergiques β2, qui est l’un des composés identifiés dans notre criblage pour les promoteurs de contact des mitochondries du RE (Figure 4A), a été utilisé pour confirmer que l’activité de la luciférase du luc split-Rlors du traitement à l’isoprotérénol était augmentée en raison de l’augmentation des contacts entre les mitochondries du RE, comme le démontrent les changements dans l’intensité du signal PLA ainsi que dans l’absorption du calcium mitochondrial (Figure 4B-E).
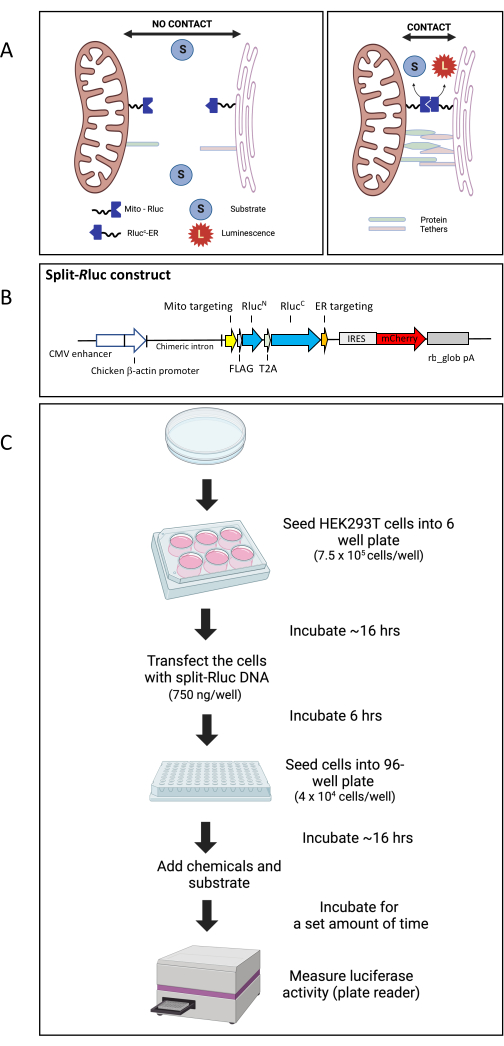
Figure 1 : Le test de réassemblage Split-R luc. (A) Représentation schématique du test Split-R Luc. Lorsqu’il n’y a pas de contacts membranaires entre les mitochondries et le réticulum endoplasmique, le Mito-R lucN et le RlucC-ER n’interagissent pas et il n’y a donc pas d’activité de Rluc8. Cependant, l’interaction entre le Mito-R lucN et le RlucC-ER, où les membranes du RE et des mitochondries entrent en contact, entraîne une activité enzymatique complète, qui est détectée par la conversion du substrat en luminescence. (B) Carte schématique de la construction de l’ADN split-Rluc (pCAG-MitoRluc N-T2A-R lucCER-IRES-mCherry). (C) Schéma du flux de travail pour le test split-Rluc dans HEK293T cellules. Abréviations : Mito = mitochondries ; RE = réticulum endoplasmique ; IRES, site d’entrée ribosomique interne ; rb_glob pA, signal de poly adénylation de la bêta-globine de lapin ; T2A, une séquence peptidique 2A auto-clivante du virus Thosea asigna. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 2 : Les activités luc split-Rcorrespondant aux niveaux de contacts ER-Mito dans les cellules traitées avec des inhibiteurs de la GTPase. (A-C) Activités reconstituées de la luciférase à (A) 1 h, (B) 2 h, ou (C) 5 h après un traitement au DMSO (contrôle), à la rhosine [50 μM], à l’Ehop-016 [25 μM] ou au ZCL278 [50 μM] (n = 12 pour chaque groupe). Toutes les données ont été analysées à l’aide d’une ANOVA à un facteur avec plusieurs comparaisons. Les valeurs p sont indiquées comme suit : p > 0,05 (ns), p ≤ 0,05 (*), p ≤ 0,01 (**), p ≤ 0,001 (***), p ≤ 0,0001 (****). Les barres d’erreur indiquent la moyenne ± l’erreur type de mesure. Abréviations : Mito = mitochondries ; RE = réticulum endoplasmique ; RLU = unité de lumière relative. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 3 : Les activités luc split-Rmesurant les niveaux de contacts ER-Mito dans les cellules traitées avec ZCL278. (A-C) Activités de la luciférase reconstituée à (A) 1 h, (B) 2 h ou (C) 5 h après l’injection de DMSO (témoin) ou de ZCL278 à une concentration de [0,5 μM], [5 μM] ou [50 μM] (n = 12 pour chaque groupe). Toutes les données ont été analysées à l’aide d’une ANOVA à un facteur avec plusieurs comparaisons. Les valeurs p sont indiquées comme suit : p > 0,05 (ns), p ≤ 0,05 (*), p ≤ 0,01 (**), p ≤ 0,001 (***), p ≤ 0,0001 (****). Les barres d’erreur indiquent la moyenne ± l’erreur type de mesure. Abréviations : Mito = mitochondries ; RE = réticulum endoplasmique ; RLU = unité de lumière relative. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 4 : Validation du dosage du Split-Rluc. (A) Un exemple de quantification de l’activité de luc split-Raprès traitement de cellules HEK293T avec 11 médicaments, y compris l’isoprotérénol, identifié dans un criblage composé pour les promoteurs de contact ER-mitochondries. (B) Un exemple de test de ligature de proximité dans des cellules HeLa traitées avec du DMSO ou de l’isoprotérénol (1 μM). Le signal PLA (rouge) indique un contact étroit entre ER et Mito. Sec61-GFP (vert) : pour étiqueter RE ; DAPI (bleu) : noyau. (C) Quantification du signal PLA dans A (n = 4 expériences avec 77-97 cellules chacune ; Test de Mann-Whitney). (D) Absorption mitochondriale de calcium en réponse à l’histamine (100 μM). Le changement d’intensité fluorescente de Mito-R-GECO1 (la moyenne a été prise sur plusieurs cellules HeLa) est tracé toutes les 2,5 s dans des cellules traitées au DMSO ou à l’isoprotérénol. La flèche indique l’ajout d’histamine. (E) À gauche : Pic maximal de l’absorption mitochondriale de calcium chez A. Test t bilatéral non apparié (n = 10, DMOS ; 14, isoprotérénol). À droite : Taux d’absorption du calcium mitochondrial (comme dans A ; 7,5 à 12,5 s). Test de Mann-Whitney (n = 12, DMSO ; 11, isoprotérénol). Cette figure est une modification de Lim et al.24. Abréviations : RLU = unité de lumière relative ; PLA = test de ligature de proximité. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Tableau 1 : Résumé des techniques actuelles pour les contacts RE-mitochondries. Liste des avantages et des limites de chaque méthode. Veuillez cliquer ici pour télécharger ce tableau.
Fichier supplémentaire 1 : Fichier zip contenant la séquence d’ADN (formats PDF et GBK) de pCAG-MitoRluc N-T2A-R luc CER-IRES-mCherry. Veuillez cliquer ici pour télécharger ce fichier.
Figure supplémentaire S1 : Carte de pCAG-MitoRluc N-T2A-R lucCER-IRES-mCherry. Veuillez cliquer ici pour télécharger ce fichier.
Discussion
Nous avons utilisé un test de réassemblage de la luciférase 8 de Renilla fendue (test R luc split-R) pour quantifier le niveau de couplages RE-mitochondries. Dans cette étude, nous avons modifié la construction originale de Split-Rluc24 en générant un seul vecteur, pCAG-MitoRluc N-T2A-R luc CER-IRES-mCherry, codant chaque composant de Split-Rluc (MitoRlucN et RlucCER) et une séquence de peptide 2A auto-clivant du virus Thosea asigna pour assurer l’expression de la même quantité de chaque moitié divisée de luciférase. Comme substrat de la Renilla luciférase, au lieu de la coelentérazine, un substrat de cellules vivantes modifié (dérivé protégé de la coelentérazine ; voir le tableau des matériaux) a été utilisé pour les raisons suivantes. Tout d’abord, contrairement à la coelenterazine qui génère une autoluminescence en milieu aqueux (particulièrement pire dans le sérum), ce dérivé protégé génère une très faible autoluminescence (souvent non détectable) dans un milieu contenant 10% de sérum, donc son rapport signal/bruit de fond est plus élevé que celui généré par la coelentérazine. Deuxièmement, contrairement à la coelentérazine, où la luminescence est la plus forte peu de temps après l’ajout du substrat mais commence à diminuer très rapidement, la luminescence produite par le substrat vivant modifié atteint son point culminant environ 1,5 h après l’ajout du substrat et reste continue pendant plus de 24 h. Ces changements dans la cinétique de réaction facilitent la mesure de la luminescence dans une plaque multipuits, ce qui est très utile pour le criblage à haut débit. De plus, la luminescence produite par le substrat protégé restant constante à long terme permet de mesurer un seul ensemble d’échantillons à plusieurs points temporels, par opposition à la mesure de plusieurs ensembles d’échantillons utilisés à différents moments (un seul point par échantillon).
À l’aide de cette version mise à jour de la construction luc split-R, nous avons testé si les GTPases RHO, RAC et CDC42 participent à la formation de contact ER-Mito. Nous avons ajouté des inhibiteurs de la GTPase, Rhosin (inhibiteur de RHO), Ehop-016 (inhibiteur de RAC) et ZCL278 (inhibiteur de CDC42), pour HEK293T cellules exprimant des composants split-Rluc. Les résultats de nos tests indiquent que ZCL278 est l’inhibiteur le plus puissant de la formation de contacts entre les mitochondries et Ehop-016 est le prochain inhibiteur significatif, tandis que la rhosine n’a pas affecté leurs contacts. Ces données suggèrent que la polymérisation de l’actine médiée par CDC42 (ainsi que celle médiée par le RAC dans une moindre mesure) est nécessaire pour maintenir les contacts entre les mitochondries du RE dans la cellule, tandis que la polymérisation de l’actine médiée par le RHO n’est probablement pas importante.
Pour garantir une efficacité maximale, de nombreuses étapes critiques doivent être prises en compte pour ce protocole. Tout d’abord, la quantité de construction transfectée doit être testée, car une trop grande quantité d’ADN peut avoir un effet toxique sur les cellules. Deuxièmement, comme le nombre de cellules peut avoir un impact significatif sur les niveaux de luciférase mesurés, il est crucial de maintenir la densité des cellules aussi proche que ce qui a été indiqué dans le protocole pour assurer la confluence idéale et constante de 80 % des cellules au moment du test. Il peut y avoir une réduction de la luminescence si les cellules atteignent la confluence pendant le temps d’exposition chimique, car les cellules confluentes produisent moins de protéines en général. Troisièmement, pour s’assurer que les cellules ne se détachent pas lors de l’ajout de produits chimiques et de substrat, nous utilisons une plaque de 96 puits pré-recouverte de Poly-D-Lysine. Quatrièmement, pour les composés (y compris le substrat) utilisés dans ce protocole, il est important de les conserver à -20 °C ou moins (-70 °C) dans des aliquotes et de les ajouter aux cellules immédiatement décongelées. Enfin, en raison des fluctuations de température possibles qui peuvent affecter l’activité de la luciférase, il peut être préférable de choisir d’utiliser les puits les plus internes de la plaque et d’ignorer les puits qui tapissent l’extérieur de la plaque.
Le test split-Rluc8 offre une méthode simple mais robuste pour déterminer les contacts des mitochondries ER, idéale pour le criblage à haut débit ; Cependant, il y a quelques limites qui doivent être notées. Pour la physiologie cellulaire, il y a la question des changements dans le comportement cellulaire dus à la surexpression constante de l’ADN construit. Actuellement, nous travaillons sur une construction avec un promoteur inductible pour lutter contre les changements potentiels de la physiologie cellulaire dus à une surexpression constante. De plus, lorsque ce test est utilisé à des fins de criblage, il convient de noter que des résultats faussement positifs peuvent découler de tout effet indirect des médicaments sur i) les niveaux d’expression, ii) l’efficacité du ciblage des organites desdits fragments de protéines, ou iii) les activités enzymatiques des luciférases reconstituées. En raison de ces limites, ce test doit être testé en conjonction avec d’autres méthodes pour valider davantage les résultats. Le tableau 1 fait référence à diverses méthodes d’essai de ces sites de contact, ainsi qu’à leurs avantages et inconvénients. Nous avons tiré parti d’autres méthodes, y compris, mais sans s’y limiter, les tests de ligature de proximité (PLA), qui détectent la proximité entre les protéines, et la microscopie électronique à transmission (MET), qui permet une visualisation claire des contacts des organites, en conjonction avec le test split-Rluc pour valider que les changements résultants dans la RLU sont effectivement dus à des changements dans les sites de contact ER-mitochondries (Figure 4 et Lim et al.24). Nous avons également montré une corrélation entre l’altération des contacts RE-mitochondries et des paramètres fonctionnels tels que le transfert de calcium et la fission mitochondriale (Figure 4 et Lim et al.24). Pour s’assurer que l’effet des médicaments sur l’activité de split-Rluc n’est pas dû à une expression accrue du gène split-Rluc, un inhibiteur de transcription ou de traduction peut être ajouté aux médicaments comme nous l’avons fait précédemment24.
Pour l’application du système split-Rluc dans des neurones différenciés en culture où le taux de transfection est faible, l’infection virale (avec des composants split-R luc exprimant des composants split-Rluc) serait une approche alternative. Son utilisation peut être étendue pour créer un système de criblage où les cellules (par exemple, les cellules souches pluripotentes induites humaines) sont modifiées pour exprimer de manière stable chaque composant du split-Rluc, MitoRlucN et RlucCER, soit par infection virale, soit par édition de gènes médiée par CRSPR. Cependant, pour un système d’expression stable, il faut être conscient qu’il pourrait y avoir une pression de sélection pour les cellules tolérant mieux le système luc split-R, ce qui pourrait ne pas être représentatif de l’état physiologique normal. Cette adaptation ou sélection pourrait introduire un biais dans les résultats expérimentaux.
En résumé, malgré certaines des limitations évoquées ci-dessus, split-Rluc est un test rapide, simple et puissant pour le criblage à haut débit de petites molécules ou de gènes régulant les contacts inter-organites dans la cellule, comme cela a été démontré dans notre précédente étude de criblage de médicaments24.
Déclarations de divulgation
Les auteurs ne déclarent aucun conflit d’intérêts.
Remerciements
Les auteurs remercient le Dr Jeffrey Golden (Cedars-Sinai Medical Center) pour l’examen critique du manuscrit. Ces travaux ont été financés en partie par le National Institute of Neurological Disease and Stroke (NINDS, R01NS113516).
matériels
| Name | Company | Catalog Number | Comments |
| 1.7 mL SafeSeal Microcentrifuge Tube | Sorenson | 16070 | |
| 6-well plate TC Treated | USA Scientific | CC7682-7506 | |
| 10 mL Pipette Tips OneTip | USA Scientific | 1110-3700 | |
| 10 μL pipette tips OneTip | USA Scientific | 1110-3700 | |
| 20-200 μL Beveled tips OneTip | USA Scientific | 1111-1210 | |
| 50 mL Polypropylene Conical Tube | Falcon | 352070 | |
| 96-Well Flipper Microtube Racks | ThermoFisher Scientific | 8770-11 | |
| 96-well plate TC Treated | USA Scientific | CC7682-7596 | |
| 100 mm x 20 mm TC Treated Dish | USA Scientific | CC7682-3394 | |
| 1250 μL Tips OneTip | USA Scientific | 1112-1720 | |
| Centrifuge 5910 Ri - Refrigerated Centrifuge | Eppendorf | 5943000131 | |
| Dimethyl sulfoxide, anhydrous, ≥99.9% | Sigma-Aldrich | 276855-100ML | |
| DMEM, high glucose | ThermoFisher Scientific | 11965092 | |
| DPBS, no calcium, no magnesium | ThermoFisher Scientific | 14190144 | |
| EHop 016 | Bio-Techne Tocris | 6248 | Dissolve in DMSO; store at -70 °C |
| EnduRen Live Cell Substrate | Promega | E6481 | Store aliquots at -70 °C |
| Eppendorf 2-20 μL pipette | Eppendorf | 3123000039 | |
| Eppendorf Research plus 100-1000 μL pipette | Eppendorf | 3123000063 | |
| Eppendorf Research Plus 1-10 µL pipette | Eppendorf | 3123000020 | |
| Eppendorf Research plus 12-channel | Eppendorf | 3125000028 | |
| Eppendorf Research plus 200 μL pipette | Eppendorf | 3123000055 | |
| Fetal Bovine Serum, qualified, USDA-approved regions | ThermoFisher Scientific | 10437028 | |
| Forma Steri-Cycle CO2 Incubator, 184 L, Polished Stainless Steel | ThermoFisher Scientific | 381 | |
| Hand tally counter | Sigma-Aldrich | HS6594 | |
| HEK 293T Cells | ATCC | CRL-3216 | |
| Hemacytometer - Neubauer Bright Line, Double-Counting Chamber | LW Scientific | CTL-HEMM-GLDR | |
| Invitrogen TE Buffer | ThermoFisher Scientific | 8019005 | |
| Microscope | Zeiss | Axiovert 25 CFL | |
| Mini centrifuge | Benchmark Scientific | C1012 | |
| Multi Tube Rack For 50ml Conical, 15ml Conical, And Microcentrifuge Tubes | Boekel Scientific | 120008 | |
| PEI MAX - Transfection Grade Linear Polyethylenimine Hydrochloride (MW 40,000) | Polysciences | 24765-100MG | |
| Pipet-Aid XP | USA Scientific | 4440-0101 | |
| Poly-D-lysine hydrobromide | Sigma-Aldrich | P6407-5MG | |
| Rhosin hydrochloride | Bio-Techne Tocris | 5003 | Dissolve in DMSO; store at -70 °C |
| Trypsin-EDTA (0.05%), phenol red | ThermoFisher Scientific | 25300054 | |
| Varioskan LUX multimode microplate reader | ThermoFisher Scientific | VL0000D0 | |
| Vortex | ThermoFisher Scientific | 2215365 | level 8 |
| VWR Vacuum Aspiration System | VWR | 75870-734 | |
| ZCL 278 | Bio-Techne Tocris | 4794 | Dissolve in DMSO; store at -70 °C |
Références
- Aoyama-Ishiwatari, S., Hirabayashi, Y. Endoplasmic reticulum-mitochondria contact sites-emerging intracellular signaling hubs. Front Cell Dev Biol. 9, 653828(2021).
- Sassano, M. L., Felipe-Abrio, B., Agostinis, P. ER-mitochondria contact sites; a multifaceted factory for Ca2+ signaling and lipid transport. Front Cell Dev Biol. 10, 988014(2022).
- Scorrano, L., et al. Coming together to define membrane contact sites. Nat Commun. 10 (1), 1287(2019).
- Voeltz, G. K., Sawyer, E. M., Hajnóczky, G., Prinz, W. A. Making the connection: How membrane contact sites have changed our view of organelle biology. Cell. 187 (2), 257-270 (2024).
- Paillusson, S., et al. There's something wrong with my MAM; the ER-mitochondria axis and neurodegenerative diseases. Trends Neurosci. 39 (3), 146-157 (2016).
- Sasi, U. S. S., Sindhu, G., Raj, P. S., Raghu, K. G. Mitochondria associated membranes (MAMs): emerging drug targets for diabetes. Curr Med Chem. 27 (20), 3362-3385 (2019).
- Joshi, A. U., Kornfeld, O. S., Mochly-Rosen, D. The entangled ER-mitochondrial axis as a potential therapeutic strategy in neurodegeneration: A tangled duo unchained. Cell Calcium. 60 (3), 218-234 (2016).
- Moltedo, O., Remondelli, P., Amodio, G. The mitochondria-endoplasmic reticulum contacts and their critical role in aging and age-associated diseases. Front Cell Dev Biol. 7, 172(2019).
- Rieusset, J. The role of endoplasmic reticulum-mitochondria contact sites in the control of glucose homeostasis: an update. Cell Death Dis. 9 (3), 388(2018).
- Bouguerra, M. D., Lalli, E. ER-mitochondria interactions: Both strength and weakness within cancer cells. Biochim Biophys Acta Mol Cell Res. 1866 (4), 650-662 (2019).
- Tepikin, A. V. Mitochondrial junctions with cellular organelles: Ca2+ signalling perspective. Pflügers Arch. 470 (8), 1181-1192 (2018).
- Masson, G. L., Przedborski, S., Abbott, L. F. A computational model of motor neuron degeneration. Neuron. 83 (4), 975-988 (2014).
- Giamogante, F., Barazzuol, L., Brini, M., Calì, T. ER-mitochondria contact sites reporters: strengths and weaknesses of the available approaches. Int J Mol Sci. 21 (21), 8157(2020).
- Rizzuto, R., et al. Close contacts with the endoplasmic reticulum as determinants of mitochondrial Ca2+ responses. Science. 280 (5370), 1763-1766 (1998).
- Valm, A. M., et al. Applying systems-level spectral imaging and analysis to reveal the organelle interactome. Nature. 546 (7656), 162-167 (2017).
- Csordás, G., et al. Imaging interorganelle contacts and local calcium dynamics at the ER-mitochondrial interface. Mol Cell. 39 (1), 121-132 (2010).
- Cieri, D., et al. SPLICS: a split green fluorescent protein-based contact site sensor for narrow and wide heterotypic organelle juxtaposition. Cell Death Differ. 25 (6), 1131-1145 (2018).
- Kakimoto, Y., et al. Visualizing multiple inter-organelle contact sites using the organelle- targeted split-GFP system. Sci Rep. 8 (1), 6175(2018).
- Wu, M. M., Covington, E. D., Lewis, R. S. Single-molecule analysis of diffusion and trapping of STIM1 and Orai1 at ER-plasma membrane junctions. Mol Biol Cell. 25 (22), Mac.E14-06-1107 (2014).
- Csordas, G., et al. Structural and functional features and significance of the physical linkage between ER and mitochondria. J Cell Biol. 174 (7), 915-921 (2006).
- de Brito, O. M., Scorrano, L. Mitofusin 2 tethers endoplasmic reticulum to mitochondria. Nature. 456 (7222), 605-610 (2008).
- Kremer, A., et al. Developing 3D SEM in a broad biological context. J Microsc. 259 (2), 80-96 (2015).
- Söderberg, O., et al. Direct observation of individual endogenous protein complexes in situ by proximity ligation. Nat Methods. 3 (12), 995-1000 (2006).
- Lim, Y., Cho, I. -T., Rennke, H. G., Cho, G. β2-adrenergic receptor regulates ER-mitochondria contacts. Sci Rep. 11 (1), 21477(2021).
- Lam, S. S., et al. Directed evolution of APEX2 for electron microscopy and proximity labeling. Nat Methods. 12 (1), 51-54 (2014).
- Cho, I. -T., et al. Ascorbate peroxidase proximity labeling coupled with biochemical fractionation identifies promoters of endoplasmic reticulum-mitochondrial contacts. J Biol Chem. 292 (39), 16382-16392 (2017).
- Lim, Y., Cho, I. -T., Schoel, L. J., Cho, G., Golden, J. A. Hereditary spastic paraplegia-linked REEP1 modulates endoplasmic reticulum/mitochondria contacts. Ann Neurol. 78 (5), 679-696 (2015).
- Arnold, T. R., Stephenson, R. E., Miller, A. L. Rho GTPases and actomyosin: Partners in regulating epithelial cell-cell junction structure and function. Exp Cell Res. 358 (1), 20-30 (2017).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon