Method Article
Saggio di riassemblaggio della split-luciferasi per misurare i contatti reticolo-mitocondri endoplasmatici in cellule vive
In questo articolo
Riepilogo
Abbiamo stabilito un saggio di riassemblaggio della luciferasi divisa per monitorare i contatti reticolo endoplasmatico-mitocondri nelle cellule vive. Utilizzando questo saggio, descriviamo un protocollo per misurare quantitativamente il livello di questi accoppiamenti inter-organelli in cellule HEK293T, in condizioni di trattamento chimico.
Abstract
I siti di contatto tra reticolo endoplasmatico (ER) e mitocondri svolgono un ruolo fondamentale nella salute cellulare e nell'omeostasi, come la regolazione del Ca2+ e dell'omeostasi lipidica, la dinamica mitocondriale, la biogenesi degli autofagosomi e dei mitofagosomi e l'apoptosi. L'incapacità di mantenere un normale accoppiamento ER-mitocondriale è implicata in molte malattie neurodegenerative come il morbo di Alzheimer, il morbo di Parkinson, la sclerosi laterale amiotrofica e la paraplegia spastica ereditaria. È di notevole importanza esplorare come la disregolazione dei contatti ER-mitocondriale possa portare alla morte cellulare e se la riparazione di questi contatti al livello normale possa migliorare le condizioni neurodegenerative. Pertanto, saggi migliorati che misurano il livello di questi contatti potrebbero aiutare a illuminare i meccanismi patogenetici di queste malattie. In definitiva, la messa a punto di saggi semplici e affidabili faciliterà lo sviluppo di nuove strategie terapeutiche. Qui descriviamo un saggio di split-luciferasi per misurare quantitativamente il livello di contatti ER-mitocondri nelle cellule vive. Questo test può essere utilizzato per studiare il ruolo fisiopatologico di questi contatti e per identificare i loro modulatori nello screening ad alto rendimento.
Introduzione
Le interazioni tra l'ER e i mitocondri sono vitali per l'omeostasi cellulare e la sopravvivenza 1,2,3,4. Prove precedenti indicano che qualsiasi tipo di interruzione o disregolazione nei siti di contatto ER-mitocondri può contribuire a diverse malattie neurodegenerative, metaboliche e cardiovascolari, nonché al cancro 5,6,7,8,9,10. Ad esempio, un aumento anomalo dell'assorbimento di Ca2+ nei mitocondri può portare alla morte cellulare aprendo i pori di transizione della permeabilità dei mitocondri, che sono comunemente osservati in alcuni modelli di malattia di Alzheimer 5,11. Allo stesso modo, la riduzione dei contatti tra ER e mitocondri può comportare una diminuzione della produzione di ATP e una compromissione dell'assunzione di Ca2+, come si è visto nei modelli di sclerosi laterale amiotrofica 5,11,12. Man mano che vengono condotti ulteriori studi nell'ambito dei contatti ER-mitocondri, vengono scoperte ulteriori proteine e geni correlati alla malattia che potrebbero influenzare questi contatti. Nonostante le attuali conoscenze e prove che mostrino il ruolo dei siti di contatto ER-mitocondri, c'è ancora molto lavoro da fare per chiarire come questi contatti possano portare alla perdita della funzione cellulare e, in ultima analisi, alla morte cellulare.
Sono stati sviluppati vari metodi per valutare la vicinanza delle due membrane, la morfologia strutturale e la distanza tra i due siti di contatto degli organelli 3,4,13. Gli approcci per monitorare l'accoppiamento ER-mitocondri includono l'imaging basato su marcatori di fluorescenza14,15, l'imaging basato su FRET-reporter16 e l'imaging basato su sonda a fluorescenza divisa17,18, che utilizzano l'epifluorescenza e la microscopia confocale. Anche la microscopia a super-risoluzione e la microscopia a risoluzione atomica sono strumenti potenti per visualizzare con precisione i contatti inter-organelli, sebbene il loro utilizzo nell'analisi del sito di contatto sia ancora limitato poiché richiedono microscopi altamente dedicati e competenze tecniche19. Inoltre, la microscopia elettronica a trasmissione (TEM), la microscopia elettronica a scansione (SEM) e altre tecniche EM come la tomografia elettronica (ET) e la microscopia crioelettronica, sono comunemente utilizzate in quanto forniscono immagini ultrastrutturali ad alta risoluzione dei siti di contatto, che sono spesso impossibili da esplorare utilizzando altri approcci sperimentali 20,21,22. Tuttavia, questi metodi basati su EM sono una tecnica a bassissima produttività che può anche essere influenzata dalle procedure di fissazione chimica. Più recentemente, sono stati utilizzati metodi basati sull'etichettatura di prossimità per rilevare i siti di contatto e per identificare nuove proteine del sito di contatto. Ad esempio, il saggio di legatura di prossimità (PLA) è stato utilizzato per quantificare le prossimità degli organelli23,24, mentre una versione rivista del test dell'ascorbato perossidasi (APEX) è stata utilizzata per identificare nuove proteine del sito di contatto25,26. È importante riconoscere che tutti questi metodi sopra descritti hanno punti di forza e limiti intrinseci nel rilevare i contatti tra gli organelli. Pertanto, è necessario l'abbinamento di diverse tecniche per ottenere un'interpretazione approfondita dei siti di contatto degli organelli.
In precedenza abbiamo stabilito il saggio di riassemblaggio split-Renilla luciferasi 8 (split-Rluc assay) per monitorare il livello dei contatti di membrana ER-mitocondri (Figura 1A)24,26,27. In breve, ogni metà divisa della Renilla luciferasi è coniugata con una sequenza di targeting ER o mitocondri. Quando vengono trasfettati insieme, ogni metà divisa dell'enzima viene espressa nell'ER o nella membrana mitocondriale. Quando l'ER e i mitocondri sono posizionati in stretta vicinanza l'uno all'altro, le metà divise si uniscono e ricostituiscono l'intero enzima con attività luciferasi. Per il costrutto split-Rluc, abbiamo usato Renilla luciferasi 8 (Rluc8) in pBAD/Myc-His27 per il modello iniziale. Il sito di divisione (tra gli amminoacidi 91 e 92) è stato determinato sulla base di precedenti rapporti27. Per la metà N-terminale dell'Rluc8, le sequenze di DNA per gli amminoacidi 1-91 dell'Rluc8 sono state fuse all'estremità 3' del tag FLAG e alla sequenza di targeting mitocondriale AKAP1 del topo di topo nel vettore TOPO pcDNA3.1 mediante PCR27. Per la metà C-terminale mirata all'ER, le sequenze di DNA che codificano gli amminoacidi 92-311 sono state fuse all'estremità 5' del tag myc e alla sequenza di localizzazione UBC6 ER del lievito. Qui, abbiamo aggiornato il costrutto plasmidico split-Rluc in modo tale che le metà divise della luciferasi Renilla siano espresse in un singolo vettore (pCAG) sotto lo stesso promotore e successivamente scisse in due frammenti quando T2A, una sequenza di peptide 2A auto-scissa dal virus Thosea asigna, viene inserita tra le due metà divise (Figura 1B). La mappa e le sequenze del DNA plasmidico sono fornite nel File Supplementare 1 e nella Figura Supplementare S1. Utilizzando questo sistema, abbiamo misurato gli effetti di tre sostanze chimiche (che inibiscono le GTPasi coinvolte nella polimerizzazione dell'actina) sui contatti ER-mitocondri. Questo saggio split-Rluc è un sistema di analisi semplice ma robusto per lo screening ad alto rendimento per modulatori di contatto inter-organelli24.
Protocollo
1. Mantenimento delle cellule e semina (Giorno 1)
- Mantenere HEK293T cellule in terreni di coltura cellulare contenenti Dulbecco's Modified Eagle Medium (DMEM) con il 10% di siero fetale bovino (FBS) (in piastre di coltura da 100 mm) in un incubatore umidificato a 37 °C con il 5% di CO2.
- Prima di iniziare, controllare la confluenza della lastra osservandola al microscopio. Quando le cellule raggiungono circa l'80-90% di confluenza, prepararsi a seminare le cellule in una piastra di coltura a 6 pozzetti rimuovendo il terreno e lavando con 10 ml di soluzione salina tamponata con fosfato di Dulbecco (DPBS).
- Rimuovere il DPBS dalla piastra di coltura e trattare le cellule con 1 mL di tripsina-EDTA fenolo rosso allo 0,05%. Incubare per 3 minuti, quindi estrarre la piastra dall'incubatore e aggiungere 9 mL di terreno di coltura (DMEM con il 10% di FBS) per fermare la reazione della tripsina. Pipettare lentamente su e giù per assicurarsi che le cellule ancora attaccate alla piastra vengano rimosse. Trasferire la sospensione cellulare in una provetta da 50 mL.
- Centrifugare a 300 × g per 5 min a temperatura ambiente. Rimuovere il terreno e risospendere i pellet cellulari con 1 mL di terreno fresco (DMEM + 10% FBS).
- Contare il numero di cellule utilizzando un emocitometro.
- Garantire una distribuzione uniforme delle cellule facendo roteare delicatamente la provetta. Estrarre immediatamente 10 μl della sospensione cellulare e metterli in una provetta da microcentrifuga da 1,7 mL. Aggiungere 90 μl di terreno nella stessa provetta.
- Dopo aver miscelato delicatamente le cellule e i terreni, prelevare la sospensione cellulare e applicare 10 μl in ciascuna delle camere dell'emocitometro pipettando delicatamente sotto il vetrino coprioggetti.
- Utilizzando un obiettivo 10x, mettere a fuoco le linee della griglia dell'emocitometro al microscopio. Una volta messo a fuoco, conta il numero di celle in una serie di 16 quadrati (4 x 4 quadrati) usando un contatore di conteggio manuale. Ripeti fino a contare tutte e quattro le serie da 16 caselle.
- Calcola il numero totale di cellule/mL prendendo il numero medio di cellule (da ciascuno dei 16 quadrati d'angolo) e moltiplicandolo per 105.
NOTA: Invece di utilizzare un emocitometro per contare le cellule, è possibile utilizzare un contatore di cellule automatizzato secondo le istruzioni del produttore.
- Una volta completato il conteggio delle cellule, preparare a piastrare, le cellule in una piastra a 6 pozzetti a 7,5 × 105 cellule/pozzetto con 2 mL di terreno/pozzetto. In una provetta da 50 mL, aggiungere il volume necessario di cellule insieme a 13 mL di terreno. Erogare 2 mL della sospensione cellulare diluita in ciascuno dei 6 pozzetti.
- Prima di procedere alla piastratura, calcolare il numero di cellule necessarie per 7,5 × 105 cellule/pozzetto utilizzando C1V1 = C2V2 dove C1 è il numero iniziale di cellule (numero di cellule/mL), V1 è il volume necessario dalla sospensione iniziale della cellula, C2 è la densità della cellula target desiderata (numero di cellule/mL), e V2 è il volume finale necessario per la semina cellulare.
- Dopo l'erogazione, picchiettare delicatamente la piastra su tutti i lati per allargare le cellule, quindi posizionarla in un incubatore umidificato a 37 °C con il 5% di CO2 per una notte.
2. Trasfezione cellulare mediata da polietilenimmina (PEI) e semina cellulare post-trasfezione (Giorno 2)
- Rimuovere la piastra a 6 pozzetti dall'incubatore e aspirare il terreno di coltura esistente da ciascun pozzetto. Aggiungere 2 ml di terreno fresco per pozzetto.
- Trasfettare le cellule con DNA plasmidico split-Rluc (pCAG-Mito,Rluc, N-T2A-R, luc,CER-IRES-mCherry) disciolto in tampone TE (pH 8.0).
- Per ogni pozzetto, mescolare il DNA (750 ng) e il PEI (rapporto DNA/PEI = 1:3) in 200 μL di DMEM in una provetta da microcentrifuga da 1,7 mL. Agitare rapidamente il DNA/PEI al livello 8 per circa 2 s per assicurarsi che si mescolino bene insieme per formare un complesso. Dopodiché, centrifugare brevemente in una mini centrifuga (<3 s).
- Lasciare incubare la miscela di DNA/PEI a temperatura ambiente per 15 minuti, quindi aggiungere la miscela (facendola cadere) sulla superficie del terreno di coltura nella piastra a 6 pozzetti contenente le cellule. Picchiettare delicatamente la piastra (per distribuire uniformemente la miscela) e incubarla in un'incubatrice umidificata a 37 °C con il 5% di CO2 per 6 ore.
- Durante l'incubazione, preparare una piastra a 96 pozzetti rivestita di poli-D-lisina (PDL) aggiungendo prima 70 μL di PDL (50 μg/mL in DPBS) a ciascun pozzetto e incubando in un incubatore umidificato a 37 °C con il 5% di CO2 per almeno 1 ora. Rimuovere la soluzione PDL e quindi lavare ogni pozzetto 2 volte con 100 μL di DPBS. Al termine del secondo lavaggio, assicurarsi che tutto il DPBS rimanente sia stato rimosso.
- A 6 ore dalla trasfezione, preparare a seminare le cellule trasfettate in una piastra a 96 pozzetti rivestita in PDL.
- Estrarre la piastra a 6 pozzetti contenente le cellule trasfettate dall'incubatore, aspirare il terreno e lavare ogni pozzetto con 2 mL di DPBS. Dopo aver rimosso il DPBS, aggiungere 350 μl di tripsina-EDTA fenolo rosso allo 0,05% e incubare per 1-2 minuti. Per fermare la reazione della tripsina, aggiungere 1,7 ml di terreno in ciascun pozzetto.
- Dopo aver trasferito le cellule insieme al terreno in una provetta da 50 ml, centrifugare la provetta a 300 × g per 5 minuti in una centrifuga da tavolo. Aspirare il terreno e risospendere il pellet di cella con 1 mL di terreno fresco. Contare il numero di cellule utilizzando un emocitometro (come descritto al punto 1.5).
- Seminare le cellule in una piastra a 96 pozzetti rivestita con PDL a una densità di 4 × 104 cellule/pozzetto in 100 μL di terreno di coltura. A tal fine, in una provetta da 50 mL, aggiungere il volume necessario di cellule (vedere di seguito per il calcolo) insieme al volume appropriato di terreno (12 mL di volume totale). Utilizzando una pipetta multicanale, erogare 100 μl della sospensione cellulare diluita in ciascuno dei 96 pozzetti. Incubare le cellule per 18 ore in un incubatore umidificato a 37 °C con CO2 al 5%.
- Calcolare il numero di cellule necessarie per 4 × 104 cellule/pozzetto utilizzando C1V1 = C2V2 dove C1 è il conteggio iniziale delle cellule (numero di cellule/mL), V1 è il volume necessario dalla sospensione iniziale delle cellule, C2 è la densità delle cellule target desiderata (numero di cellule/mL) e V2 è il volume finale necessario per la semina delle cellule.
3. Trattamento chimico e dosaggio della luciferasi in cellule vive (Giorno 3)
- In una provetta per microfuge da 1,7 mL, preparare le tre soluzioni fresche di 50 μM di Rhosin (50 mM di stock in DMSO), 25 μM di Ehop-016 (25 mM di stock in DMSO) e 50 μM di ZCL278 (50 mM di stock in DMSO) diluendo ciascuna soluzione madre (50 mM di Rhosin, 25 mM di Ehop-016 e 50 mL di ZCL278; tutte in DMSO) 1:1.000 nella quantità richiesta (50 μL/pozzetto × numero di pozzetti) di terreni di coltura. A ciascuna soluzione, aggiungere il substrato di cellule vive (50 mM stock in DMSO) per Renilla luciferasi diluita 1:2.000 a una concentrazione finale di 25 μM. Per ottenere soluzioni ZCL278 da 0,5, 5 e 50 μM, eseguire diluizioni seriali utilizzando il 50 μM iniziale di ZCL278 in terreni di coltura.
NOTA: Se si desidera un tempo di incubazione chimica inferiore a 1-1,5 ore, le cellule possono essere preincubate con substrato di cellule vive (per Renilla luciferasi) per 1-1,5 ore prima di essere trattate con ciascuna sostanza chimica. Aggiungiamo contemporaneamente sostanze chimiche e substrato di cellule vive per ottimizzare il protocollo per lo screening di farmaci ad alto rendimento. - Rimuovere il terreno da ciascun pozzetto della piastra a 96 pozzetti contenente le cellule trasfettate e aggiungere 50 μL di miscela di terreni chimici e substrati a ciascun pozzetto.
- Dopo aver incubato le cellule per 1 ora, 2 ore o 5 ore, estrarre la piastra di coltura dall'incubatore, caricarla sul lettore di piastre di luminescenza e misurare la luminescenza.
- Prima di iniziare, assicurarsi che il lettore di piastre sia acceso. Accedi al software di lettura delle micropiastre e crea un nuovo file facendo clic su Nuova sessione. Impostare la temperatura del lettore di piastre a 37 °C facendo clic su Incubatore, selezionando l'opzione di temperatura e digitando 37 °C. In Layout piastra, scegliere l'opzione Sconosciuto e specificare i pozzetti da misurare facendo clic e trascinando.
- In Protocollo, fare clic su Luminescenza e mantenere le impostazioni predefinite. Una volta terminata la configurazione, caricare la piastra con il coperchio nel lettore di piastre e scegliere Run Plate In. Fare clic su Avvia e verrà visualizzata la finestra di salvataggio del file. Salvare il file sul desktop rinominandolo e facendo clic su Salva.
NOTA: La luminescenza continuerà ad essere prodotta per >24 ore dopo l'aggiunta del substrato di cellule vive.
- Una volta completata la lettura, rimuovere la piastra dal lettore di lastre, quindi fare clic su Esegui piastra in per reinserire il lettore. Una volta completata, riposizionare la piastra nell'incubatore umidificato (37 °C, 5% CO2) fino alla lettura successiva.
- Al termine, trasferire i dati di luminescenza a un programma grafico per tracciare l'unità di luminescenza relativa (RLU) (asse y) per ogni variabile (ad esempio, sostanze chimiche o concentrazioni) (asse x) e analizzare i dati.
4. Validazione del saggio split-Rluc con altri metodi.
- Eseguire altri saggi come il saggio di legatura di prossimità (PLA), la microscopia elettronica a trasmissione e il monitoraggio dell'assorbimento di calcio mitocondriale per convalidare i risultati ottenuti dal saggio split-Rluc come descritto in precedenza24.
Risultati
Abbiamo utilizzato il protocollo sopra descritto per misurare il livello di contatti ER-mitocondri con l'aggiunta di tre composti noti per inibire specifiche GTPasi. CDC42, RHO e RAC sono GTPasi che promuovono la polimerizzazione dell'actina28 quando attivate e sono inibite rispettivamente da ZCL278, Rhosin e Ehop-01624. HEK293T cellule trasfettate con split-Rluc sono state trattate con DMSO (controllo), ZCL278 (50 μM), Rhosin (50 μM) o Ehop-016 (25 μM) e incubate per 1 h, 2 h e 5 h. Utilizzando un lettore di piastre, abbiamo misurato l'attività split-Rluc in punti temporali definiti (Figura 2).
Le cellule trattate con Rhosin (RHO GTPase inhibitor) non hanno mostrato cambiamenti significativi nell'attività della luciferasi rispetto alle cellule di controllo trattate con DMSO a 1 h, 2 h e 5 h del trattamento (p > 0,9999, p = 0,6956, p > 0,9999) (Figura 2). Questi risultati dimostrano che la Rhosin non influenza i contatti ER-mitocondri, suggerendo l'assenza di coinvolgimento dell'attività della RHO GTPasi nella regolazione dei contatti tra i due organelli.
Al contrario, l'inibitore della GTPasi RAC Ehop-016 ha mostrato un'attività della luciferasi significativamente inferiore rispetto al DMSO a 1 ora, 2 ore e 5 ore di trattamento (p = 0,0106, p = 0,0009, p = 0,0024) (Figura 2). Questi dati indicano che l'attività della GTPasi RAC è necessaria per mantenere la normale interazione ER-mitocondri nella cellula.
Infine, l'inibitore CDC42 ZCL278 ha mostrato i cambiamenti più drastici nell'attività della luciferasi (Figura 2). Le cellule trattate con ZCL278 hanno mostrato una diminuzione significativa dell'attività della luciferasi rispetto al DMSO di controllo e hanno avuto i valori p più bassi in tutti e tre i punti temporali, mantenendo un p < 0,0001 per tutto il tempo. I nostri dati dimostrano una forte inibizione dell'accoppiamento ER-mitocondri quando l'attività della GTPasi CDC42 è bloccata da ZCL278, in linea con il nostro precedente rapporto utilizzando il saggio originale split-Rluc eseguito in cellule co-trasfettate con due vettori che codificano separatamente ciascuna metà della split-luciferasi24.
Dati i risultati sorprendenti con ZCL278, soprattutto se confrontato con gli altri inibitori della GTPasi, l'attività di ZCL278 è stata ulteriormente testata a varie concentrazioni [0,5 μM, 5 μM, 50 μM] rispetto a un controllo DMSO. Le attività della luciferasi misurate a 1 ora, 2 ore e 5 ore dopo il trattamento hanno mostrato una risposta dose-dipendente; 0,5 μM di ZCL278 hanno determinato l'effetto più debole sull'attività della luciferasi, mentre 5 μM hanno mostrato una riduzione più forte e 50 μM hanno mostrato la più forte riduzione dell'attività enzimatica ricostituita (Figura 3). Insieme, i nostri risultati mostrano che ZCL278 sottoregola l'attività di split-Rluc in modo dose-dipendente e, con il passare del tempo, concentrazioni più elevate di ZCL278 (inibitore CDC42) mostrano ancora un cambiamento significativo rispetto al DMSO di controllo rispetto a concentrazioni più basse.
Per convalidare il nostro test split-Rluc, è possibile eseguire saggi indipendenti consolidati, come il saggio di legatura di prossimità (PLA), la microscopia elettronica a trasmissione (TEM) e il monitoraggio dell'assorbimento di calcio mitocondriale (Figura 4 e dati non mostrati)24. L'isoproterenolo, un agonista del recettore β2 adrenergico, che è uno dei composti identificati nel nostro screening per i promotori del contatto ER-mitocondri (Figura 4A), è stato utilizzato per confermare che l'attività luciferasica dello split-Rluc dopo il trattamento con isoproterenolo era aumentata a causa dell'aumento dei contatti ER-mitocondri, come dimostrato dai cambiamenti nell'intensità del segnale PLA e nell'assorbimento mitocondriale del calcio (Figura 4B-E).
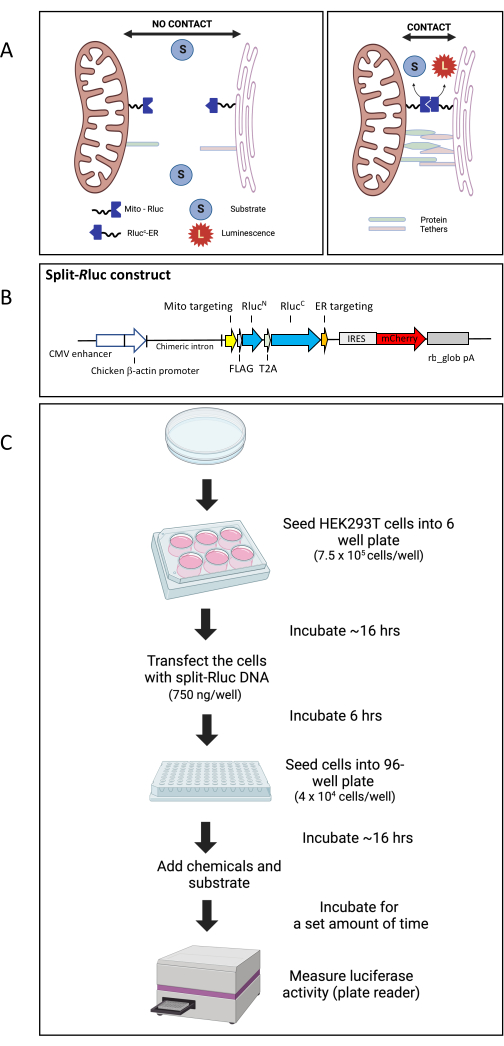
Figura 1: Il saggio di riassemblaggio split-R luc. (A) Rappresentazione schematica del saggio Split-R Luc. Dove non ci sono contatti di membrana tra i mitocondri e il reticolo endoplasmatico, il Mito-R lucN e il RlucC-ER non riescono a interagire e quindi non c'è attività di Rluc8. Tuttavia, l'interazione tra il Mito-R lucN e il RlucC-ER, dove l'ER e le membrane dei mitocondri entrano in contatto, provoca una piena attività enzimatica, che viene rilevata dalla conversione del substrato in luminescenza. (B) Mappa schematica del costrutto di DNA split-Rluc (pCAG-MitoRluc N-T2A-R lucCER-IRES-mCherry). (C) Diagramma del flusso di lavoro per il saggio split-Rluc in cellule HEK293T. Abbreviazioni: Mito = mitocondri; ER = reticolo endoplasmatico; IRES, sito di ingresso ribosomiale interno; rb_glob pA, segnale di poliadenilazione della beta-globina di coniglio; T2A, una sequenza di peptide 2A auto-scissa dal virus Thosea asigna. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 2: Le attività split-Rluc corrispondenti ai livelli di contatti ER-Mito nelle cellule trattate con inibitori della GTPasi. (A-C) Attività della luciferasi ricostituita a (A) 1 ora, (B) 2 ore o (C) 5 ore dopo il trattamento con DMSO (controllo), Rhosin [50 μM], Ehop-016 [25 μM] o ZCL278 [50 μM] (n = 12 per ciascun gruppo). Tutti i dati sono stati analizzati utilizzando l'ANOVA unidirezionale con confronti multipli. I valori di P sono riportati come p > 0,05 (ns), p ≤ 0,05 (*), p ≤ 0,01 (**), p ≤ 0,001 (***), p ≤ 0,0001 (****). Le barre di errore indicano l'errore medio ± standard di misura. Abbreviazioni: Mito = mitocondri; ER = reticolo endoplasmatico; RLU = unità di misura della luce relativa. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 3: Le attività split-Rluc che misurano i livelli di contatti ER-Mito nelle cellule trattate con ZCL278. (A-C) Attività della luciferasi ricostituita a (A) 1 ora, (B) 2 ore o (C) 5 ore dopo DMSO (controllo) o ZCL278 a [0,5 μM], [5 μM] o [50 μM] (n = 12 per ciascun gruppo). Tutti i dati sono stati analizzati utilizzando l'ANOVA unidirezionale con confronti multipli. I valori di P sono riportati come p > 0,05 (ns), p ≤ 0,05 (*), p ≤ 0,01 (**), p ≤ 0,001 (***), p ≤ 0,0001 (****). Le barre di errore indicano l'errore medio ± standard di misura. Abbreviazioni: Mito = mitocondri; ER = reticolo endoplasmatico; RLU = unità di misura della luce relativa. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 4: Convalida del saggio split-Rluc. (A) Un esempio di quantificazione dell'attività split-Rluc dopo il trattamento di cellule HEK293T con 11 farmaci, tra cui l'isoproterenolo, identificato in uno screening composto per i promotori di contatto ER-mitocondri. (B) Un esempio di saggio di legatura di prossimità in cellule HeLa trattate con DMSO o isoproterenolo (1 μM). Il segnale PLA (rosso) indica un contatto stretto tra ER e Mito. Sec61-GFP (verde): per etichettare ER; DAPI (blu): nucleo. (C) Quantificazione del segnale PLA in A (n = 4 esperimenti con 77-97 celle ciascuno; test di Mann-Whitney). (D) Captazione mitocondriale del calcio in risposta all'istamina (100 μM). La variazione di intensità fluorescente di Mito-R-GECO1 (la media è stata presa su più cellule HeLa) è stata tracciata ogni 2,5 s in cellule trattate con DMSO o isoproterenolo. La freccia indica l'aggiunta di istamina. (E) A sinistra: picco massimo di assorbimento mitocondriale di calcio in A. T-test a due code spaiato (n = 10, DMOS; 14, isoproterenolo). A destra: tasso di assorbimento del calcio mitocondriale (come in A; da 7,5 a 12,5 s). Test di Mann-Whitney (n = 12, DMSO; 11, isoproterenolo). Questa cifra è modificata da Lim et al.24. Abbreviazioni: RLU = unità di luce relativa; PLA = saggio di legatura di prossimità. Clicca qui per visualizzare una versione più grande di questa figura.
Tabella 1: Riepilogo delle tecniche attuali per i contatti ER-mitocondri. Elenco dei vantaggi e dei limiti per ogni metodo. Clicca qui per scaricare questa tabella.
File supplementare 1: File zip contenente la sequenza di DNA (formati PDF e GBK) di pCAG-MitoRluc N-T2A-R luc CER-IRES-mCherry. Clicca qui per scaricare questo file.
Figura supplementare S1: Mappa di pCAG-MitoRluc N-T2A-R lucCER-IRES-mCherry. Clicca qui per scaricare questo file.
Discussione
Abbiamo utilizzato un saggio di riassemblaggio split-Renilla luciferasi 8 (split-Rluc assay) per quantificare il livello di accoppiamenti ER-mitocondri. In questo studio, abbiamo modificato il costrutto originale split-Rluc24 generando un singolo vettore, pCAG-MitoRluc N-T2A-R lucCER-IRES-mCherry, codificante ogni componente split-Rluc (MitoRlucN e RlucCER) e una sequenza di peptide 2A auto-scissa dal virus Thosea asigna per garantire l'espressione della stessa quantità di ciascuna metà divisa della luciferasi. Come substrato per la Renilla luciferasi, al posto della celenterazina, è stato utilizzato un substrato modificato di cellule vive (derivato protetto della celenterazina; vedi la Tabella dei materiali) per i seguenti motivi. In primo luogo, a differenza della celeterazina che genera autoluminescenza in un ambiente acquoso (soprattutto peggio nel siero), questo derivato protetto genera un'autoluminescenza molto bassa (spesso non rilevabile) in un terreno contenente il 10% di siero, quindi il suo rapporto segnale/fondo è superiore a quello generato dalla celenterazina. In secondo luogo, a differenza della celenterazina, dove la luminescenza è più forte poco dopo l'aggiunta del substrato ma inizia a diminuire molto rapidamente, la luminescenza prodotta dal substrato vivo modificato raggiunge il suo punto più alto raggiungibile circa 1,5 ore dopo l'aggiunta del substrato e rimane continua per più di 24 ore. Questi cambiamenti nella cinetica di reazione facilitano la misurazione della luminescenza in una piastra a più pozzetti, il che è molto utile per lo screening ad alto rendimento. Inoltre, la luminescenza prodotta dal substrato protetto che rimane costante nel lungo periodo consente la misurazione di un singolo set di campioni in più punti temporali, invece di misurare più set di campioni utilizzati in vari punti temporali (singolo punto per campione).
Utilizzando questa versione aggiornata del costrutto luc split-R, abbiamo testato se le GTPasi RHO, RAC e CDC42 partecipano alla formazione dei contatti ER-Mito. Abbiamo aggiunto inibitori della GTPasi, Rhosin (inibitore RHO), Ehop-016 (inibitore RAC) e ZCL278 (inibitore CDC42), per HEK293T cellule che esprimono componenti split-Rluc. I risultati dei nostri test indicano che ZCL278 è l'inibitore più potente della formazione del contatto ER-mitocondri e Ehop-016 è il successivo inibitore significativo, mentre la Rhosina non ha influenzato i loro contatti. Questi dati suggeriscono che la polimerizzazione dell'actina mediata da CDC42 (così come quella mediata da RAC in misura minore) è necessaria per mantenere i contatti ER-mitocondri nella cellula, mentre la polimerizzazione dell'actina mediata da RHO probabilmente non è importante.
Per garantire la massima efficienza, ci sono molti passaggi critici che devono essere considerati per questo protocollo. Innanzitutto, la quantità di costrutto trasfettato deve essere testata poiché troppo DNA può avere un effetto tossico sulle cellule. In secondo luogo, poiché il numero di cellule può avere un impatto significativo sui livelli di luciferasi misurati, mantenere la densità delle cellule il più vicino possibile a quanto dichiarato nel protocollo è fondamentale per garantire la confluenza ideale e costante dell'80% delle cellule al momento del test. Ci può essere una riduzione della luminescenza se le cellule raggiungono la confluenza durante il tempo di esposizione chimica, poiché le cellule confluenti producono meno proteine in generale. In terzo luogo, per garantire che le cellule non si stacchino durante l'aggiunta di sostanze chimiche e substrato, utilizziamo una piastra a 96 pozzetti pre-rivestita con poli-D-lisina. In quarto luogo, per i composti (compreso il substrato) utilizzati in questo protocollo, è importante mantenerli a -20 °C o inferiori (-70 °C) in aliquote e aggiungerli alle cellule immediatamente scongelate. Infine, a causa delle possibili fluttuazioni di temperatura che possono influenzare l'attività della luciferasi, potrebbe essere meglio scegliere di utilizzare i pozzetti più interni della piastra e ignorare i pozzetti che rivestono l'esterno della piastra.
Il test split-Rluc8 offre un metodo semplice ma robusto per determinare i contatti ER-mitocondri, ideale per lo screening ad alto rendimento; Tuttavia, ci sono alcune limitazioni che devono essere notate. Per la fisiologia cellulare, c'è il problema dei cambiamenti nel comportamento cellulare dovuti alla costante sovraespressione del costrutto DNA. Attualmente, stiamo lavorando su un costrutto con un promotore inducibile per combattere i potenziali cambiamenti nella fisiologia cellulare dovuti alla costante sovraespressione. Inoltre, quando questo test viene utilizzato a scopo di screening, si noti che i risultati falsi positivi possono derivare da qualsiasi effetto indiretto dei farmaci su i) livelli di espressione, ii) efficienze di targeting degli organelli dei suddetti frammenti proteici, o iii) attività enzimatiche delle luciferasi ricostituite. A causa di queste limitazioni, questo test deve essere testato insieme ad altri metodi per convalidare ulteriormente i risultati. La tabella 1 si riferisce a vari metodi di verifica di questi siti di contatto insieme ai loro vantaggi e svantaggi. Abbiamo sfruttato altri metodi, tra cui, a titolo esemplificativo ma non esaustivo, i saggi di legatura di prossimità (PLA), che rilevano la prossimità tra le proteine, e la microscopia elettronica a trasmissione (TEM), che consente una chiara visualizzazione dei contatti degli organelli, in combinazione con il saggio split-Rluc per convalidare che i cambiamenti risultanti in RLU siano effettivamente dovuti a cambiamenti nei siti di contatto ER-mitocondri (Figura 4 e Lim et al.24). Abbiamo anche mostrato una correlazione tra l'alterazione dei contatti ER-mitocondri e parametri funzionali come il trasferimento di calcio e la fissione mitocondriale (Figura 4 e Lim et al.24). Per garantire che l'effetto dei farmaci sull'attività di split-Rluc non sia dovuto all'aumento dell'espressione del gene split-Rluc, un inibitore della trascrizione o della traduzione può essere aggiunto insieme ai farmaci come abbiamo fatto in precedenza24.
Per l'applicazione del sistema split-Rluc in neuroni differenziati in colture dove il tasso di trasfezione è basso, l'infezione virale (con lentivirus che esprimono componenti split-Rluc) sarebbe un approccio alternativo. Il suo utilizzo può essere esteso per creare un sistema di screening in cui le cellule (ad esempio, le cellule staminali pluripotenti indotte umane) sono ingegnerizzate per esprimere stabilmente ogni componente di split-Rluc, MitoRlucN e RlucCER, sia per infezione virale che per editing genico mediato da CRSPR. Tuttavia, per un sistema di espressione stabile, si deve essere consapevoli che potrebbe esserci una pressione selettiva per le cellule che tollerano meglio il sistema split-R-Luc, che potrebbe non essere rappresentativo del normale stato fisiologico. Questo adattamento o selezione potrebbe introdurre distorsioni nei risultati sperimentali.
In sintesi, nonostante alcune delle limitazioni discusse sopra, split-Rluc è un test veloce, diretto e potente per lo screening ad alto rendimento di piccole molecole o geni che regolano i contatti inter-organelli nella cellula, come è stato dimostrato nel nostro precedente studio di screening farmacologico24.
Divulgazioni
Gli autori dichiarano di non avere conflitti di interesse.
Riconoscimenti
Gli autori sono grati al Dr. Jeffrey Golden (Cedars-Sinai Medical Center) per la revisione critica del manoscritto. Questo lavoro è stato finanziato in parte dal National Institute of Neurological Disease and Stroke (NINDS, R01NS113516).
Materiali
| Name | Company | Catalog Number | Comments |
| 1.7 mL SafeSeal Microcentrifuge Tube | Sorenson | 16070 | |
| 6-well plate TC Treated | USA Scientific | CC7682-7506 | |
| 10 mL Pipette Tips OneTip | USA Scientific | 1110-3700 | |
| 10 μL pipette tips OneTip | USA Scientific | 1110-3700 | |
| 20-200 μL Beveled tips OneTip | USA Scientific | 1111-1210 | |
| 50 mL Polypropylene Conical Tube | Falcon | 352070 | |
| 96-Well Flipper Microtube Racks | ThermoFisher Scientific | 8770-11 | |
| 96-well plate TC Treated | USA Scientific | CC7682-7596 | |
| 100 mm x 20 mm TC Treated Dish | USA Scientific | CC7682-3394 | |
| 1250 μL Tips OneTip | USA Scientific | 1112-1720 | |
| Centrifuge 5910 Ri - Refrigerated Centrifuge | Eppendorf | 5943000131 | |
| Dimethyl sulfoxide, anhydrous, ≥99.9% | Sigma-Aldrich | 276855-100ML | |
| DMEM, high glucose | ThermoFisher Scientific | 11965092 | |
| DPBS, no calcium, no magnesium | ThermoFisher Scientific | 14190144 | |
| EHop 016 | Bio-Techne Tocris | 6248 | Dissolve in DMSO; store at -70 °C |
| EnduRen Live Cell Substrate | Promega | E6481 | Store aliquots at -70 °C |
| Eppendorf 2-20 μL pipette | Eppendorf | 3123000039 | |
| Eppendorf Research plus 100-1000 μL pipette | Eppendorf | 3123000063 | |
| Eppendorf Research Plus 1-10 µL pipette | Eppendorf | 3123000020 | |
| Eppendorf Research plus 12-channel | Eppendorf | 3125000028 | |
| Eppendorf Research plus 200 μL pipette | Eppendorf | 3123000055 | |
| Fetal Bovine Serum, qualified, USDA-approved regions | ThermoFisher Scientific | 10437028 | |
| Forma Steri-Cycle CO2 Incubator, 184 L, Polished Stainless Steel | ThermoFisher Scientific | 381 | |
| Hand tally counter | Sigma-Aldrich | HS6594 | |
| HEK 293T Cells | ATCC | CRL-3216 | |
| Hemacytometer - Neubauer Bright Line, Double-Counting Chamber | LW Scientific | CTL-HEMM-GLDR | |
| Invitrogen TE Buffer | ThermoFisher Scientific | 8019005 | |
| Microscope | Zeiss | Axiovert 25 CFL | |
| Mini centrifuge | Benchmark Scientific | C1012 | |
| Multi Tube Rack For 50ml Conical, 15ml Conical, And Microcentrifuge Tubes | Boekel Scientific | 120008 | |
| PEI MAX - Transfection Grade Linear Polyethylenimine Hydrochloride (MW 40,000) | Polysciences | 24765-100MG | |
| Pipet-Aid XP | USA Scientific | 4440-0101 | |
| Poly-D-lysine hydrobromide | Sigma-Aldrich | P6407-5MG | |
| Rhosin hydrochloride | Bio-Techne Tocris | 5003 | Dissolve in DMSO; store at -70 °C |
| Trypsin-EDTA (0.05%), phenol red | ThermoFisher Scientific | 25300054 | |
| Varioskan LUX multimode microplate reader | ThermoFisher Scientific | VL0000D0 | |
| Vortex | ThermoFisher Scientific | 2215365 | level 8 |
| VWR Vacuum Aspiration System | VWR | 75870-734 | |
| ZCL 278 | Bio-Techne Tocris | 4794 | Dissolve in DMSO; store at -70 °C |
Riferimenti
- Aoyama-Ishiwatari, S., Hirabayashi, Y. Endoplasmic reticulum-mitochondria contact sites-emerging intracellular signaling hubs. Front Cell Dev Biol. 9, 653828(2021).
- Sassano, M. L., Felipe-Abrio, B., Agostinis, P. ER-mitochondria contact sites; a multifaceted factory for Ca2+ signaling and lipid transport. Front Cell Dev Biol. 10, 988014(2022).
- Scorrano, L., et al. Coming together to define membrane contact sites. Nat Commun. 10 (1), 1287(2019).
- Voeltz, G. K., Sawyer, E. M., Hajnóczky, G., Prinz, W. A. Making the connection: How membrane contact sites have changed our view of organelle biology. Cell. 187 (2), 257-270 (2024).
- Paillusson, S., et al. There's something wrong with my MAM; the ER-mitochondria axis and neurodegenerative diseases. Trends Neurosci. 39 (3), 146-157 (2016).
- Sasi, U. S. S., Sindhu, G., Raj, P. S., Raghu, K. G. Mitochondria associated membranes (MAMs): emerging drug targets for diabetes. Curr Med Chem. 27 (20), 3362-3385 (2019).
- Joshi, A. U., Kornfeld, O. S., Mochly-Rosen, D. The entangled ER-mitochondrial axis as a potential therapeutic strategy in neurodegeneration: A tangled duo unchained. Cell Calcium. 60 (3), 218-234 (2016).
- Moltedo, O., Remondelli, P., Amodio, G. The mitochondria-endoplasmic reticulum contacts and their critical role in aging and age-associated diseases. Front Cell Dev Biol. 7, 172(2019).
- Rieusset, J. The role of endoplasmic reticulum-mitochondria contact sites in the control of glucose homeostasis: an update. Cell Death Dis. 9 (3), 388(2018).
- Bouguerra, M. D., Lalli, E. ER-mitochondria interactions: Both strength and weakness within cancer cells. Biochim Biophys Acta Mol Cell Res. 1866 (4), 650-662 (2019).
- Tepikin, A. V. Mitochondrial junctions with cellular organelles: Ca2+ signalling perspective. Pflügers Arch. 470 (8), 1181-1192 (2018).
- Masson, G. L., Przedborski, S., Abbott, L. F. A computational model of motor neuron degeneration. Neuron. 83 (4), 975-988 (2014).
- Giamogante, F., Barazzuol, L., Brini, M., Calì, T. ER-mitochondria contact sites reporters: strengths and weaknesses of the available approaches. Int J Mol Sci. 21 (21), 8157(2020).
- Rizzuto, R., et al. Close contacts with the endoplasmic reticulum as determinants of mitochondrial Ca2+ responses. Science. 280 (5370), 1763-1766 (1998).
- Valm, A. M., et al. Applying systems-level spectral imaging and analysis to reveal the organelle interactome. Nature. 546 (7656), 162-167 (2017).
- Csordás, G., et al. Imaging interorganelle contacts and local calcium dynamics at the ER-mitochondrial interface. Mol Cell. 39 (1), 121-132 (2010).
- Cieri, D., et al. SPLICS: a split green fluorescent protein-based contact site sensor for narrow and wide heterotypic organelle juxtaposition. Cell Death Differ. 25 (6), 1131-1145 (2018).
- Kakimoto, Y., et al. Visualizing multiple inter-organelle contact sites using the organelle- targeted split-GFP system. Sci Rep. 8 (1), 6175(2018).
- Wu, M. M., Covington, E. D., Lewis, R. S. Single-molecule analysis of diffusion and trapping of STIM1 and Orai1 at ER-plasma membrane junctions. Mol Biol Cell. 25 (22), Mac.E14-06-1107 (2014).
- Csordas, G., et al. Structural and functional features and significance of the physical linkage between ER and mitochondria. J Cell Biol. 174 (7), 915-921 (2006).
- de Brito, O. M., Scorrano, L. Mitofusin 2 tethers endoplasmic reticulum to mitochondria. Nature. 456 (7222), 605-610 (2008).
- Kremer, A., et al. Developing 3D SEM in a broad biological context. J Microsc. 259 (2), 80-96 (2015).
- Söderberg, O., et al. Direct observation of individual endogenous protein complexes in situ by proximity ligation. Nat Methods. 3 (12), 995-1000 (2006).
- Lim, Y., Cho, I. -T., Rennke, H. G., Cho, G. β2-adrenergic receptor regulates ER-mitochondria contacts. Sci Rep. 11 (1), 21477(2021).
- Lam, S. S., et al. Directed evolution of APEX2 for electron microscopy and proximity labeling. Nat Methods. 12 (1), 51-54 (2014).
- Cho, I. -T., et al. Ascorbate peroxidase proximity labeling coupled with biochemical fractionation identifies promoters of endoplasmic reticulum-mitochondrial contacts. J Biol Chem. 292 (39), 16382-16392 (2017).
- Lim, Y., Cho, I. -T., Schoel, L. J., Cho, G., Golden, J. A. Hereditary spastic paraplegia-linked REEP1 modulates endoplasmic reticulum/mitochondria contacts. Ann Neurol. 78 (5), 679-696 (2015).
- Arnold, T. R., Stephenson, R. E., Miller, A. L. Rho GTPases and actomyosin: Partners in regulating epithelial cell-cell junction structure and function. Exp Cell Res. 358 (1), 20-30 (2017).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon