Method Article
살아있는 세포에서 소포체-미토콘드리아 접촉을 측정하기 위한 Split-Luciferase 재조립 분석
요약
당사는 살아있는 세포에서 소포체-미토콘드리아 접촉을 모니터링하기 위해 split-luciferase reassembly assay를 확립했습니다. 이 분석을 사용하여 화학적 처리 조건에서 HEK293T 세포에서 이러한 세포 간 결합의 수준을 정량적으로 측정하는 프로토콜을 설명합니다.
초록
소포체(ER)-미토콘드리아 접촉 부위는 Ca2+ 및 지질 항상성 조절, 미토콘드리아 역학, 자가포식 및 미토파고좀 생물 발생, 세포 사멸과 같은 세포 건강 및 항상성에 중요한 역할을 합니다. 정상적인 ER-미토콘드리아 결합을 유지하지 못하는 것은 알츠하이머병, 파킨슨병, 근위축성 측삭 경화증 및 유전성 경련성 하반신 마비와 같은 많은 신경 퇴행성 질환과 관련이 있습니다. ER-미토콘드리아 접촉의 조절 장애가 어떻게 세포 사멸로 이어질 수 있는지, 그리고 이러한 접촉을 정상 수준으로 복구하면 신경 퇴행성 상태를 개선할 수 있는지 여부를 조사하는 것은 상당한 의미가 있습니다. 따라서 이러한 접촉의 수준을 측정하는 개선된 분석법은 이러한 질병의 병원성 메커니즘을 밝히는 데 도움이 될 수 있습니다. 궁극적으로 간단하고 신뢰할 수 있는 분석을 확립하면 새로운 치료 전략의 개발을 촉진할 수 있습니다. 여기에서는 살아있는 세포에서 ER-미토콘드리아 접촉 수준을 정량적으로 측정하기 위한 split-luciferase assay에 대해 설명합니다. 이 분석은 이러한 접촉의 병태생리학적 역할을 연구하고 고처리량 스크리닝에서 조절제를 식별하는 데 사용할 수 있습니다.
서문
ER과 미토콘드리아 사이의 상호 작용은 세포 항상성과 생존에 매우 중요합니다 1,2,3,4. 이전의 증거에 의하면 ER-미토콘드리아 접촉 부위의 모든 유형의 중단 또는 조절 장애는 암뿐만 아니라 여러 신경퇴행성, 대사성 및 심혈관 질환에 기여할 수 있다 5,6,7,8,9,10. 예를 들어, 미토콘드리아로의 Ca2+ 흡수가 비정상적으로 증가하면 알츠하이머병의 일부 모델에서 흔히 볼 수 있는 미토콘드리아 투과성 전이 기공이 열려 세포 사멸을 유발할 수 있습니다 5,11. 마찬가지로, ER-미토콘드리아 접촉이 감소하면 ATP 생성이 감소하고 Ca2+ 섭취가 손상될 수 있으며, 이는 근위축성 측삭 경화증 모델에서 볼 수 있습니다 5,11,12. ER-미토콘드리아 접촉 영역에서 더 많은 연구가 수행됨에 따라 이러한 접촉에 영향을 줄 수 있는 추가적인 질병 관련 단백질 및 유전자가 발견되고 있습니다. ER-미토콘드리아 접촉 부위의 역할을 보여주는 현재의 지식과 증거에도 불구하고, 이러한 접촉이 어떻게 세포 기능의 상실과 궁극적으로 세포 사멸로 이어질 수 있는지 설명하기 위해서는 여전히 많은 연구가 필요합니다.
두 막의 근접성, 구조적 형태 및 두 세포 기관 접촉 부위 사이의 거리를 평가하기 위해 다양한 방법이 개발되었습니다 3,4,13. ER-미토콘드리아 결합을 모니터링하는 접근 방식에는 형광 마커 기반 이미징14,15, FRET 리포터 기반 이미징16 및 분할 형광 프로브 기반 이미징17,18이 포함되며 에피형광 및 컨포칼 현미경을 사용합니다. 초고해상도 및 원자 분해능 현미경 검사는 또한 세포 기관 간 접촉을 정확하게 시각화하기 위한 강력한 도구이지만, 고도로 전용 현미경과 기술 전문 지식이 필요하기 때문에 접촉 부위 분석에서의 활용은 여전히 제한적입니다19. 또한 투과 전자 현미경(TEM), 주사 전자 현미경(SEM) 및 전자 단층 촬영(ET) 및 초저온 전자 현미경과 같은 기타 EM 기술은 다른 실험적 접근 방식을 사용하여 탐색할 수 없는 접촉 부위의 고해상도 초구조 이미징을 제공하기 때문에 일반적으로 사용됩니다 20,21,22. 그러나 이러한 EM 기반 방법은 화학적 고정 절차의 영향도 받을 수 있는 매우 낮은 처리량 기술입니다. 보다 최근에는 근접 라벨링 기반 방법을 사용하여 접촉 부위를 검출하고 새로운 접촉 부위 단백질을 식별했습니다. 예를 들어, 근접 결찰 분석(PLA)은 소기관 근접성을 정량화하는 데 사용되었으며(23,24), 아스코르브산 과산화효소(APEX) 분석의 수정된 버전은 새로운 접촉 부위 단백질(25,26)을 식별하는 데 사용되었습니다. 위에서 설명한 이러한 모든 방법은 소기관 간의 접촉을 감지하는 데 강점과 본질적인 한계가 있음을 인식하는 것이 중요합니다. 따라서, 세포 기관 접촉 부위에 대한 철저한 해석을 얻기 위해 다양한 기술의 쌍이 필요합니다.
이전에는 ER-미토콘드리아 멤브레인 접촉 수준(그림 1A)을 모니터링하기 위해 split-Renilla luciferase 8 재조립 분석(split-Rluc assay)을 확립했습니다(그림 1A)24,26,27. 간단히 말해서, Renilla luciferase의 각 분할 절반은 ER 또는 미토콘드리아 표적 서열과 접합됩니다. 함께 transfection하면 효소의 각 분할 절반이 ER 또는 미토콘드리아 막에서 발현됩니다. ER과 미토콘드리아가 서로 가까이 위치하면 분할된 반쪽이 함께 모여 루시페라아제 활성으로 전체 효소를 재구성합니다. split-Rluc 구조체의 경우 초기 템플릿에 대해 pBAD/Myc-His27의 Renilla luciferase 8(Rluc8)을 사용했습니다. 분할 부위(아미노산 91 및 92 사이)는 이전 보고(27)에 기초하여 결정되었다. Rluc8의 N-말단 절반에 대해, Rluc8의 아미노산 1-91에 대한 DNA 서열을 PCR27에 의해 pcDNA3.1 TOPO 벡터에서 FLAG 태그의 3' 말단 및 마우스 AKAP1 미토콘드리아 표적 서열에 융합시켰다. ER을 대상으로 하는 C-말단 절반의 경우, 아미노산 92-311을 암호화하는 DNA 서열을 myc 태그의 5' 말단과 효모 UBC6 ER 국소화 서열에 융합했습니다. 여기에서는 Renilla luciferase의 분할된 반쪽이 동일한 promoter 하에서 단일 벡터(pCAG)로 발현된 후 Thosea asigna 바이러스의 자가 절단 펩타이드 2A 염기서열인 T2A가 두 개의 분할 반쪽 사이에 삽입됨에 따라 두 개의 단편으로 절단되도록 split-Rluc plasmid 구조를 업그레이드했습니다(그림 1B). 플라스미드 DNA 맵 및 서열은 보충 파일 1 및 보충 그림 S1에 제공된다. 이 시스템을 사용하여 ER-미토콘드리아 접촉에 대한 세 가지 화학 물질(액틴 중합에 관여하는 GTPase 억제)의 효과를 측정했습니다. 이 split-Rluc assay는 세포기관 간 접촉 조절제(inter-organelle contact modulators24)에 대한 고처리량 스크리닝을 위한 간단하지만 강력한 분석 시스템입니다.
프로토콜
1. 세포 유지 관리 및 파종(1일차)
- 37°C의 가습 인큐베이터에서 5%CO2로 10% 소 태아 혈청(FBS)(100mm 배양 접시에 포함)이 포함된 Dulbecco의 DMEM(Modified Eagle Medium)을 포함하는 세포 배양 배지의 HEK293T 세포를 유지합니다.
- 시작하기 전에 현미경으로 관찰하여 플레이트의 밀도를 확인하십시오. 세포가 약 80-90%의 밀도에 도달하면 배지를 제거하고 10mL의 Dulbecco's phosphate-buffered saline(DPBS)으로 세척하여 6웰 배양 플레이트에서 세포를 파종할 준비를 합니다.
- 배양 접시에서 DPBS를 제거하고 세포를 0.05% 트립신-EDTA 페놀 레드 1mL로 처리합니다. 3분 동안 배양한 다음 인큐베이터에서 플레이트를 꺼내고 9mL의 배양 배지(10% FBS가 있는 DMEM)를 추가하여 트립신 반응을 중지합니다. 플레이트에 아직 붙어 있는 세포가 제거되었는지 확인하기 위해 천천히 위아래로 피펫팅합니다. 세포 현탁액을 50mL 튜브로 옮깁니다.
- 300× g 에서 실온에서 5분 동안 원심분리합니다. 배지를 제거하고 1mL의 새 배지(DMEM + 10% FBS)로 세포 펠릿을 재현탁시킵니다.
- 혈구계를 사용하여 세포 수를 계산합니다.
- 튜브를 부드럽게 소용돌이쳐 세포가 고르게 분포되도록 합니다. 즉시 세포 현탁액 10μL를 꺼내 1.7mL 미세 원심분리 튜브에 넣습니다. 동일한 튜브에 90μL의 매체를 추가합니다.
- 세포와 배지를 부드럽게 혼합한 후 세포 현탁액을 취하고 커버슬립 아래에 부드럽게 피펫팅하여 혈구계의 각 챔버에 10μL를 적용합니다.
- 10x 대물렌즈를 사용하여 현미경으로 혈구계의 격자선에 초점을 맞춥니다. 초점이 맞춰지면 손 계산 카운터를 사용하여 16개의 사각형(4 x 4 사각형)으로 구성된 한 세트의 셀 수를 계산합니다. 16개의 사각형으로 구성된 4세트가 모두 계산될 때까지 반복합니다.
- 평균 세포 수(16개의 모서리 사각형 각각에서)를 취하고 105를 곱하여 총 세포 수/mL를 계산합니다.
알림: 혈구계를 사용하여 세포를 계수하는 대신 제조업체의 지침에 따라 자동 세포 카운터를 사용할 수 있습니다.
- 세포 계수가 완료되면 7.5 × 105 세포/웰 밀도의 6-웰 플레이트에 2mL의 배지/웰로 세포를 플레이트할 준비를 합니다. 50mL 튜브에 13mL의 배지와 함께 필요한 부피의 세포를 추가합니다. 희석된 세포 현탁액 2mL를 6개의 웰 각각에 분주합니다.
- 도금하기 전에 C1V1 = C2V2를 사용하여 7.5 × 105 셀 / 웰에 필요한 셀 수를 계산하십시오. 여기서 C1은 초기 셀 수(셀 수/mL), V1은 초기 셀 현탁액에서 필요한 부피, C2는 원하는 목표 셀 밀도(셀 수/mL)입니다. V2 는 세포 파종에 필요한 최종 부피입니다.
- 분주 후 플레이트의 모든 면을 가볍게 두드려 세포를 펼친 다음 37°C의 가습 인큐베이터에 밤새 5%CO2 로 넣습니다.
2. 폴리에틸렌이민(PEI) 매개 세포 transfection 및 transfection 후 세포 seeding(2일차)
- 인큐베이터에서 6-웰 플레이트를 제거하고 각 웰에서 기존 배양 배지를 흡인합니다. 웰당 2mL의 신선한 배지를 추가합니다.
- TE 완충액(pH 8.0)에 용해된 split-Rluc plasmid DNA(pCAG-MitoRluc N-T2A-R lucCER-IRES-mCherry)로 세포를 transfection합니다.
- 각 웰에 대해 1.7mL 미세 원심분리기 튜브의 DMEM 200μL에 DNA(750ng)와 PEI(DNA/PEI 비율 = 1:3)를 혼합합니다. 레벨 8에서 DNA/PEI를 약 2초 동안 빠르게 소용돌이쳐 서로 잘 섞여 복합체를 형성하도록 합니다. 그런 다음 미니 원심분리기에서 짧게(<3초) 회전시킵니다.
- DNA/PEI 혼합물을 실온에서 15분 동안 배양한 다음 혼합물을 세포가 들어 있는 6웰 플레이트의 배양 배지 표면에 떨어뜨려 추가합니다. 플레이트를 부드럽게 두드리고(혼합물이 고르게 분포되도록) 37°C의 가습 인큐베이터에서 5% CO2 로 6시간 동안 배양합니다.
- 배양하는 동안 먼저 각 웰에 70μL의 PDL(DPBS에서 50μg/mL)을 추가하고 37°C의 가습 인큐베이터에서 5% CO2 로 최소 1시간 동안 배양하여 Poly-D-Lysine(PDL) 코팅된 96웰 플레이트를 준비합니다. PDL 용액을 제거한 다음 각 웰을 100μL의 DPBS로 2번 세척합니다. 두 번째 세척이 끝나면 나머지 DPBS가 모두 제거되었는지 확인합니다.
- transfection 후 6시간이 지나면 PDL 코팅된 96웰 플레이트에서 transfection된 세포를 seed할 준비를 합니다.
- 인큐베이터에서 transfection된 세포가 들어 있는 6웰 플레이트를 꺼내고 배지를 흡인한 다음 각 웰을 2mL의 DPBS로 세척합니다. DPBS를 제거한 후 350μL의 0.05% 트립신-EDTA 페놀 레드를 넣고 1-2분 동안 배양합니다. 트립신 반응을 중지하려면 각 웰에 1.7mL의 배지를 추가합니다.
- 세포를 배지와 함께 50mL 튜브로 옮긴 후 탁상용 원심분리기에서 300× g 으로 5분 동안 튜브를 회전시킵니다. 배지를 흡입하고 1mL의 새 배지로 세포 펠릿을 재현탁시킵니다. 혈구계를 사용하여 세포 수를 계산합니다(1.5단계에서 설명한 대로).
- 100 μL의 배양 배지에 4 × 104 cells/well의 밀도로 PDL 코팅된 96웰 플레이트에 세포를 시드합니다. 이를 위해 50mL 튜브에 적절한 양의 매체(총 부피 12mL)와 함께 필요한 세포 부피(계산은 아래 참조)를 추가합니다. 멀티채널 피펫을 사용하여 100μL의 희석된 셀 현탁액을 96웰 각각에 분주합니다. 37 ° C의 가습 인큐베이터에서 5 % CO2 로 18 시간 동안 세포를 배양합니다.
- C1V1 = C2V2를 사용하여 4 × 104 세포/웰에 필요한 세포 수를 계산합니다. 여기서 C1은 초기 세포 수(세포 수/mL), V1은 초기 세포 현탁액에서 필요한 부피, C2는 원하는 목표 세포 밀도(세포 수/mL), V2는 세포 파종에 필요한 최종 부피입니다.
3. 살아있는 세포의 화학적 처리 및 루시페라아제 분석(3일차)
- 1.7mL 미세분리 튜브에 50μM Rhosin(DMSO의 50mM 스톡), 25μM Ehop-016(DMSO의 25mM 스톡) 및 50μM ZCL278(DMSO의 50mM 스톡)의 3가지 새로운 용액을 각 스톡 용액(50mM Rhosin, 25mM Ehop-016 및 50mL ZCL278, 모두 DMSO에 포함)을 필요한 양(50μL/웰 × 웰 수)으로 1:1,000으로 희석하여 준비합니다. 각 용액에 1:2,000으로 희석된 Renilla luciferase용 생세포 기질(DMSO의 50mM 스톡)을 최종 농도 25μM로 추가합니다. 0.5, 5 및 50 μM ZCL278 용액을 만들려면 배양 배지에서 초기 50 μM ZCL278을 사용하여 연속 희석을 수행합니다.
참고: 화학적 배양 시간이 1-1.5시간 미만으로 필요한 경우 세포를 각 화학 물질로 처리하기 전에 1-1.5시간 동안 살아있는 세포 기질( Renilla luciferase의 경우)로 사전 배양할 수 있습니다. 당사는 화학 물질과 살아있는 세포 기질을 동시에 추가하여 고처리량 약물 스크리닝을 위한 프로토콜을 최적화합니다. - 형질주입된 세포가 들어 있는 96웰 플레이트의 각 웰에서 배지를 제거하고 각 웰에 50μL의 화학물질 및 기판 배지 혼합물을 추가합니다.
- 세포를 1시간, 2시간 또는 5시간 동안 배양한 후 배양기에서 배양판을 꺼내 발광 플레이트 리더에 로드하고 발광을 측정합니다.
- 시작하기 전에 플레이트 리더가 켜져 있는지 확인하십시오. 마이크로플레이트 리더 소프트웨어에 액세스하고 New Session(새 세션)을 클릭하여 새 파일을 만듭니다. Incubator를 클릭하고 온도 옵션을 선택한 다음 37 °C를 입력하여 플레이트 리더의 온도를 37 °C로 설정합니다. 플레이트 레이아웃(Plate Layout)에서 알 수 없음(Unknown) 옵션을 선택하고 클릭하고 끌어서 측정할 웰을 지정합니다.
- 프로토콜에서 발광을 클릭하고 기본 설정을 유지합니다. 설정이 완료되면 뚜껑을 닫은 플레이트를 플레이트 리더에 로드하고 Run Plate In(플레이트 인 실행)을 선택합니다. 시작을 클릭하면 파일 저장 창이 나타납니다. 파일 이름을 바꾸고 저장을 클릭하여 바탕 화면에 파일을 저장합니다.
알림: 발광은 라이브 셀 기판을 추가한 후에도 >24시간 동안 계속 생성됩니다.
- 판독이 완료되면 플레이트 리더에서 플레이트를 제거한 다음 플레이트 인 실행을 클릭하여 리더를 다시 끼웁니다. 완료되면 다음 판독 할 때까지 플레이트를 가습 인큐베이터 (37 °C, 5 % CO2)에 다시 놓습니다.
- 완료되면 발광 데이터를 그래프 프로그램으로 전송하여 각 변수(예: 화학 물질 또는 농도)(x축)에 대한 상대 발광 단위(RLU)(y축)를 플롯하고 데이터를 분석합니다.
4. 다른 방법을 사용한 split-Rluc 분석의 검증.
- 근접 결찰 분석(PLA), 투과 전자 현미경 및 미토콘드리아 칼슘 흡수 모니터링과 같은 다른 분석을 수행하여 이전에 설명한 대로 split-Rluc 분석에서 얻은 결과를 검증합니다24.
결과
우리는 위에서 설명한 프로토콜을 사용하여 특정 GTPase를 억제하는 것으로 알려진 세 가지 화합물을 추가할 때 ER-미토콘드리아 접촉 수준을 측정했습니다. CDC42, RHO 및 RAC는 활성화될 때 액틴 중합(28)을 촉진하고 각각 ZCL278, Rhosin 및 Ehop-016에 의해 억제되는 GTPase입니다(24). split-Rluc로 형질주입된 HEK293T 세포를 DMSO(대조군), ZCL278(50μM), Rhosin(50μM) 또는 Ehop-016(25μM)으로 처리하고 1시간, 2시간, 5시간 동안 배양했습니다. 플레이트 리더를 사용하여 정의된 시점에서 split-Rluc 활성을 측정했습니다(그림 2).
로신(RHO GTPase inhibitor) 처리된 세포는 처리한 1시간, 2시간, 5시간에서 대조군 DMSO 처리된 세포에 비해 루시페라아제 활성에 유의한 변화가 없었습니다(p > 0.9999, p= 0.6956, p > 0.9999)(그림 2). 이러한 결과는 로신이 ER-미토콘드리아 접촉에 영향을 미치지 않는다는 것을 보여주며, 이는 두 소기관 사이의 접촉을 조절하는 데 RHO GTPase 활성이 관여하지 않음을 시사합니다.
이에 반해 RAC GTPase 억제제인 Ehop-016은 치료 후 1시간, 2시간, 5시간에서 DMSO보다 루시페라아제 활성이 유의하게 낮았다(p=0.0106, p=0.0009, p=0.0024)(그림 2). 이러한 데이터는 세포에서 정상적인 ER-미토콘드리아 상호 작용을 유지하기 위해 RAC GTPase 활성이 필요하다는 것을 나타냅니다.
마지막으로, CDC42 억제제 ZCL278은 루시페라아제 활성에서 가장 급격한 변화를 보였습니다(그림 2). ZCL278 처리된 세포는 대조군 DMSO에 비해 루시페라아제 활성이 유의하게 감소했으며, 3개 시점 모두에서 가장 낮은 p-값을 보였으며, 전체적으로 p < 0.0001을 유지했습니다. 당사의 데이터는 CDC42 GTPase 활성이 ZCL278에 의해 차단될 때 ER-미토콘드리아 결합의 강력한 억제를 보여주며, 이는 split-luciferase의 각 절반을 별도로 인코딩하는 두 개의 벡터와 co-transfection된 세포에서 수행된 원래의 split-Rluc 분석을 사용한 이전 보고서와 일치합니다24.
ZCL278의 놀라운 결과를 감안할 때, 특히 다른 GTPase 억제제와 비교할 때 ZCL278 활성은 DMSO 대조군에 대해 다양한 농도[0.5μM, 5μM, 50μM]에서 추가로 테스트되었습니다. 처리 후 1시간, 2시간, 5시간에 측정된 루시페라아제 활성은 용량 의존적 반응을 보였다; ZCL278의 0.5μM은 루시페라아제 활성에 가장 약한 영향을 미친 반면, 5μM은 더 강했고, 50μM은 재구성된 효소 활성에서 가장 강한 감소를 보였습니다(그림 3). 이러한 결과를 종합해 볼 때, ZCL278은 용량 의존적 방식으로 split-Rluc 활성을 하향 조절하며, 시간이 지남에 따라 더 높은 농도의 ZCL278(CDC42 억제제)은 낮은 농도에 비해 대조군 DMSO에 비해 여전히 유의한 변화를 보입니다.
split-Rluc assay를 검증하는 방법으로 PLA(Proximity ligation assay), TEM(transmission electron microscopy) 및 미토콘드리아 칼슘 흡수 모니터링과 같은 잘 정립된 독립 분석을 수행할 수 있습니다(그림 4 및 데이터는 표시되지 않음)24. ER-미토콘드리아 접촉 프로모터에 대한 스크리닝에서 확인된 화합물 중 하나인 β2 아드레날린성 수용체 작용제인 이소프로테레놀(그림 4A)을 사용하여 PLA 신호 강도의 변화와 미토콘드리아 칼슘 흡수의 변화로 입증된 바와 같이 이소프로테레놀 처리 시 split-Rluc의 루시페라아제 활성이 증가했음을 확인했습니다(그림 4B-E).
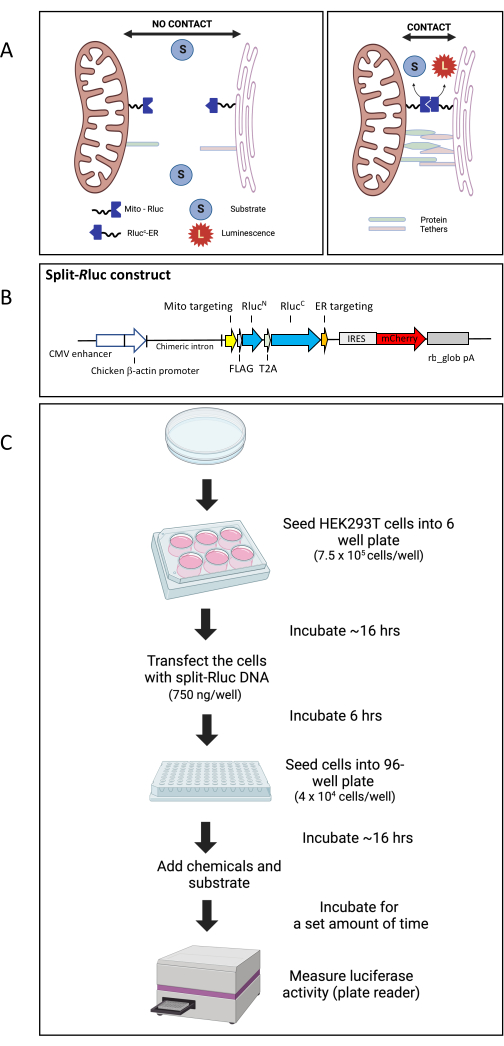
그림 1: split-Rluc 재조립 분석. (A) Split-RLuc 분석의 개략도. 미토콘드리아와 소포체 사이에 막 접촉이 없는 경우 Mito-R lucN과 RlucC-ER이 상호 작용하지 못하므로 Rluc8 활성이 없습니다. 그러나 ER과 미토콘드리아 막이 접촉하는 Mito-R lucN과 RlucC-ER 사이의 상호 작용은 기질의 발광 전환에 의해 감지되는 완전한 효소 활성을 초래합니다. (B) split-Rluc DNA 구조체(pCAG-MitoRluc N-T2A-R lucCER-IRES-mCherry)의 개략도. (C) HEK293T 세포에서 split-Rluc 분석을 위한 워크플로우 다이어그램. 약어: 미토 = 미토콘드리아; ER = 소포체; IRES, 내부 리보솜 진입 부위; rb_glob pA, 토끼 베타-글로빈 폴리 아데닐화 신호; T2A, Thosea asigna 바이러스의 자가 절단 펩타이드 2A 염기서열. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

그림 2: GTPase 억제제로 처리된 세포의 ER-Mito 접촉 수준에 해당하는 split-R luc 활성. (A-C) DMSO(대조군), Rhosin[50μM], Ehop-016[25μM] 또는 ZCL278[50μM] 처리 후 (A) 1시간, (B) 2시간 또는 (C) 5시간(각 그룹에 대해 n = 12)에서 재구성된 루시페라아제 활성. 모든 데이터는 다중 비교와 함께 일원 분산 분석을 사용하여 분석되었습니다. P 값은 p > 0.05 (ns), p ≤ 0.05 (*), p ≤ 0.01 (**), p ≤ 0.001 (***), p ≤ 0.0001 (****)로 보고됩니다. 오차 막대는 측정의 평균 ± 표준 오차를 나타냅니다. 약어: 미토 = 미토콘드리아; ER = 소포체; RLU = 상대 조명 단위. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

그림 3: ZCL278로 처리된 세포에서 ER-Mito 접촉 수준을 측정하는 split-R luc 활성.(A-C) [0.5 μM], [5 μM] 또는 [50 μM] 농도(각 그룹에 대해 n = 12)에서 DMSO(대조군) 또는 ZCL278 후 (A) 1시간, (B) 2시간 또는 (C) 5시간에서 재구성된 루시페라아제 활성. 모든 데이터는 다중 비교와 함께 일원 분산 분석을 사용하여 분석되었습니다. P 값은 p > 0.05 (ns), p ≤ 0.05 (*), p ≤ 0.01 (**), p ≤ 0.001 (***), p ≤ 0.0001 (****)로 보고됩니다. 오차 막대는 측정의 평균 ± 표준 오차를 나타냅니다. 약어: 미토 = 미토콘드리아; ER = 소포체; RLU = 상대 조명 단위. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

그림 4: split-Rluc 분석의 검증. (A) ER-미토콘드리아 접촉 프로모터에 대한 화합물 스크리닝에서 확인된 이소프로테레놀을 포함한 11가지 약물로 HEK293T 세포를 처리한 후 split-Rluc 활성의 정량화의 예. (B) DMSO 또는 이소프로테레놀(1μM)로 처리된 HeLa 세포의 근접 결찰 분석의 예. PLA 신호(빨간색)는 ER과 Mito 사이의 긴밀한 접촉을 나타냅니다. Sec61-GFP(녹색): ER 라벨링; DAPI(파란색): 핵. (C) A에서 PLA 신호의 정량화 (n = 각각 77-97 개의 세포를 사용한 4 회 실험; Mann-Whitney 테스트). (D) 히스타민(100μM)에 반응하는 미토콘드리아 칼슘 흡수. Mito-R-GECO1 형광 강도 변화(여러 HeLa 세포에 대한 평균)는 DMSO 또는 이소프로테레놀 처리된 세포에서 2.5초마다 표시되었습니다. 화살표는 히스타민 첨가를 나타냅니다. (E) 왼쪽: A에서 미토콘드리아 칼슘 흡수의 최대 피크. 쌍을 이루지 않은 양측 t-검정(n = 10, DMOS; 14, 이소프로테레놀). 오른쪽: 미토콘드리아 칼슘 흡수율( A와 같이, 7.5-12.5초). Mann-Whitney 테스트(n = 12, DMSO; 11, 이소프로테레놀). 이 그림은 Lim et al.24에서 수정되었습니다. 약어: RLU = 상대 광 단위; PLA = 근접 결찰 분석. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
표 1: ER-미토콘드리아 접촉에 대한 현재 기술 요약. 각 방법의 장점과 제한 사항 목록입니다. 이 표를 다운로드하려면 여기를 클릭하십시오.
보충 파일 1: pCAG-MitoRluc N-T2A-R lucCER-IRES-mCherry의 DNA 서열(PDF 및 GBK 형식)을 포함하는 Zip 파일. 이 파일을 다운로드하려면 여기를 클릭하십시오.
추가 그림 S1: pCAG-MitoRluc N-T2A-R lucCER-IRES-mCherry의 지도. 이 파일을 다운로드하려면 여기를 클릭하십시오.
토론
우리는 ER-미토콘드리아 커플링의 수준을 정량화하기 위해 split-Renilla luciferase 8 reassembly assay(split-Rluc assay)를 사용했습니다. 이 연구에서는 루시페라아제의 각 분할 절반이 동일한 양으로 발현되도록 하기 위해 각 split-Rluc 구성 요소(MitoRlucN 및 RlucCER)와 Thosea asigna 바이러스의 자체 절단 펩타이드 2A 서열을 인코딩하는 단일 벡터 pCAG-MitoRluc N-T2A-R lucCER-IRES-mCherry를 생성하여 원래의 split-Rluc construct24를 수정했습니다. 레닐라 루시페라제(Renilla luciferase)의 기질로서, 코엘렌테라진(coelenterazine) 대신에, 변형된 생세포 기질(코엘렌테라진의 보호된 유도체, 재료표 참조)이 다음과 같은 이유로 사용되었다. 첫째, 수성 환경(특히 혈청에서 더 나쁨)에서 자동 발광을 생성하는 코엘렌테라진과 달리, 이 보호된 유도체는 10% 혈청을 함유하는 매체에서 매우 낮은 자동 발광(종종 검출할 수 없음)을 생성하므로 신호 대 배경 비율이 코엘렌테라진에 의해 생성된 것보다 높습니다. 둘째, 기판을 추가한 직후 발광이 가장 강하지만 매우 빠르게 감소하기 시작하는 코엘렌테라진과 달리, 변형된 라이브 기판에 의해 생성된 발광은 기판 첨가 후 약 1.5시간 후에 도달할 수 있는 가장 높은 지점에 도달하고 24시간 이상 연속적으로 유지됩니다. 반응 역학의 이러한 변화는 멀티웰 플레이트에서 발광을 더 쉽게 측정할 수 있게 해주며, 이는 고처리량 스크리닝에 매우 유용합니다. 더욱이, 장기적으로 일정하게 유지되는 보호된 기판에 의해 생성된 발광은 다양한 시점(샘플당 단일 지점)에서 사용되는 여러 샘플 세트를 측정하는 것과 대조적으로 여러 시점에서 단일 샘플 세트를 측정할 수 있습니다.
이 업데이트된 버전의 split-Rluc 구조를 사용하여 RHO, RAC 및 CDC42 GTPase가 ER-Mito 접촉 형성에 참여하는지 테스트했습니다. split-Rluc 성분을 발현하는 세포HEK293T GTPase 억제제, Rhosin(RHO 억제제), Ehop-016(RAC 억제제) 및 ZCL278(CDC42 억제제)을 추가했습니다. 분석 결과에 따르면 ZCL278은 ER-미토콘드리아 접촉 형성의 가장 강력한 억제제이고 Ehop-016은 다음으로 중요한 억제제인 반면 Rhosin은 접촉에 영향을 미치지 않았습니다. 이러한 데이터는 세포 내 ER-미토콘드리아 접촉을 유지하기 위해 CDC42 매개 액틴 중합(RAC 매개 중합 포함)이 필요한 반면, RHO 매개 액틴 중합은 중요하지 않을 수 있음을 시사합니다.
최대 효율성을 보장하기 위해 이 프로토콜에 대해 고려해야 하는 많은 중요한 단계가 있습니다. 무엇보다도, 너무 많은 DNA가 세포에 독성 영향을 미칠 수 있으므로 형질주입된 구조체의 양을 테스트해야 합니다. 둘째, 세포의 수가 측정된 루시페라아제 수치에 상당한 영향을 미칠 수 있기 때문에 세포의 밀도를 프로토콜에 명시된 것과 가깝게 유지하는 것은 분석 시점까지 세포의 이상적이고 일관된 80% 밀도를 보장하는 데 중요합니다. 화학 물질 노출 시간 동안 세포가 밀도에 도달하면 발광이 감소할 수 있는데, 이는 합류 세포가 일반적으로 더 적은 단백질을 만들기 때문입니다. 셋째, 화학 물질과 기질을 추가하는 동안 세포가 벗겨지지 않도록 Poly-D-Lysine으로 사전 코팅된 96웰 플레이트를 사용합니다. 넷째, 이 프로토콜에 사용된 화합물(기질 포함)의 경우, 부분 표본에서 -20°C 이하(-70°C)로 유지하고 즉시 해동된 세포에 첨가하는 것이 중요합니다. 마지막으로, 루시페라아제 활성에 영향을 줄 수 있는 온도 변동 가능성으로 인해 플레이트의 가장 안쪽 웰을 선택하고 플레이트 외부를 감싸고 있는 웰은 무시하는 것이 가장 좋습니다.
split-Rluc8 분석은 ER-미토콘드리아 접촉을 측정하는 간편하면서도 강력한 방법을 제공하며, 고처리량 스크리닝에 이상적입니다. 그러나 주의해야 할 몇 가지 제한 사항이 있습니다. 세포 생리학의 경우, 구성 DNA의 지속적인 과발현으로 인한 세포 행동의 변화 문제가 있습니다. 현재 우리는 지속적인 과발현으로 인한 세포 생리학의 잠재적인 변화에 맞서기 위해 유도성 프로모터를 가진 구조를 연구하고 있습니다. 또한, 이 분석법이 스크리닝 목적으로 사용되는 경우, 위양성 결과가 i) 발현 수준, ii) 상기 단백질 단편의 소기관 표적화 효율, 또는 iii) 재구성된 루시페라아제의 효소 활성에 대한 약물의 간접적인 효과로 인해 발생할 수 있다는 점에 유의해야 합니다. 이러한 제한으로 인해 이 분석은 결과를 추가로 검증하기 위해 다른 방법과 함께 테스트해야 합니다. 표 1은 이러한 접촉 부위를 테스트하는 다양한 방법과 그 장점 및 단점을 나타냅니다. 단백질 간의 근접성을 감지하는 PLA(proximity ligation assay)와 세포소 접촉을 명확하게 시각화할 수 있는 TEM(transmission electron microscopy)을 포함하되 이에 국한되지 않는 다른 방법을 split-Rluc 분석과 함께 활용하여 RLU의 결과 변화가 실제로 ER-미토콘드리아 접촉 부위의 변화로 인한 것임을 검증했습니다(그림 4 및 Lim et al.24)). 또한 ER-미토콘드리아 접촉의 변화와 칼슘 전달 및 미토콘드리아 분열과 같은 기능적 매개변수 간의 상관관계를 보여주었습니다(그림 4 및 Lim et al.24). split-Rluc 활성에 대한 약물의 효과가 split-Rluc 유전자의 발현 증가로 인한 것이 아님을 보장하기 위해, 이전에 수행한 것처럼 전사 또는 번역 억제제를 약물과 함께 추가할 수 있습니다24.
transfection rate가 낮은 배양에서 분화된 뉴런에 split-Rluc 시스템을 적용하기 위해서는 바이러스 감염(split-Rluc 구성 요소를 발현하는 렌티바이러스)이 대안적인 접근법이 될 수 있습니다. 이의 사용은 세포(예: 인간 유도 만능 줄기 세포)가 바이러스 감염 또는 CRSPR 매개 유전자 편집에 의해 split-Rluc, MitoRlucN 및 RlucCER의 각 구성 요소를 안정적으로 발현하도록 엔지니어링된 스크리닝 시스템을 만들기 위해 확장될 수 있습니다. 그러나, 안정적인 발현 시스템을 위해서는 split-Rluc 시스템을 더 잘 견디는 세포에 대한 선택 압력이 있을 수 있다는 것을 알아야 하며, 이는 정상적인 생리학적 상태를 대표하지 않을 수 있습니다. 이러한 적응 또는 선택은 실험 결과에 편향을 도입할 수 있습니다.
요약하면, 위에서 논의한 몇 가지 제한 사항에도 불구하고 split-Rluc는 이전 약물 스크리닝 연구24에서 나타난 바와 같이 세포의 세포 기관 간 접촉을 조절하는 작은 분자 또는 유전자에 대한 고처리량 스크리닝을 위한 빠르고 간단하며 강력한 분석법입니다.
공개
저자는 이해 상충이 없음을 선언합니다.
감사의 말
저자들은 원고에 대한 비판적 검토를 해준 Dr. Jeffrey Golden (Cedars-Sinai Medical Center)에게 감사를 표했다. 이 연구는 미국 국립신경질환 및 뇌졸중 연구소(National Institute of Neurological Disease and Stroke, NINDS, R01NS113516)의 지원을 받았습니다.
자료
| Name | Company | Catalog Number | Comments |
| 1.7 mL SafeSeal Microcentrifuge Tube | Sorenson | 16070 | |
| 6-well plate TC Treated | USA Scientific | CC7682-7506 | |
| 10 mL Pipette Tips OneTip | USA Scientific | 1110-3700 | |
| 10 μL pipette tips OneTip | USA Scientific | 1110-3700 | |
| 20-200 μL Beveled tips OneTip | USA Scientific | 1111-1210 | |
| 50 mL Polypropylene Conical Tube | Falcon | 352070 | |
| 96-Well Flipper Microtube Racks | ThermoFisher Scientific | 8770-11 | |
| 96-well plate TC Treated | USA Scientific | CC7682-7596 | |
| 100 mm x 20 mm TC Treated Dish | USA Scientific | CC7682-3394 | |
| 1250 μL Tips OneTip | USA Scientific | 1112-1720 | |
| Centrifuge 5910 Ri - Refrigerated Centrifuge | Eppendorf | 5943000131 | |
| Dimethyl sulfoxide, anhydrous, ≥99.9% | Sigma-Aldrich | 276855-100ML | |
| DMEM, high glucose | ThermoFisher Scientific | 11965092 | |
| DPBS, no calcium, no magnesium | ThermoFisher Scientific | 14190144 | |
| EHop 016 | Bio-Techne Tocris | 6248 | Dissolve in DMSO; store at -70 °C |
| EnduRen Live Cell Substrate | Promega | E6481 | Store aliquots at -70 °C |
| Eppendorf 2-20 μL pipette | Eppendorf | 3123000039 | |
| Eppendorf Research plus 100-1000 μL pipette | Eppendorf | 3123000063 | |
| Eppendorf Research Plus 1-10 µL pipette | Eppendorf | 3123000020 | |
| Eppendorf Research plus 12-channel | Eppendorf | 3125000028 | |
| Eppendorf Research plus 200 μL pipette | Eppendorf | 3123000055 | |
| Fetal Bovine Serum, qualified, USDA-approved regions | ThermoFisher Scientific | 10437028 | |
| Forma Steri-Cycle CO2 Incubator, 184 L, Polished Stainless Steel | ThermoFisher Scientific | 381 | |
| Hand tally counter | Sigma-Aldrich | HS6594 | |
| HEK 293T Cells | ATCC | CRL-3216 | |
| Hemacytometer - Neubauer Bright Line, Double-Counting Chamber | LW Scientific | CTL-HEMM-GLDR | |
| Invitrogen TE Buffer | ThermoFisher Scientific | 8019005 | |
| Microscope | Zeiss | Axiovert 25 CFL | |
| Mini centrifuge | Benchmark Scientific | C1012 | |
| Multi Tube Rack For 50ml Conical, 15ml Conical, And Microcentrifuge Tubes | Boekel Scientific | 120008 | |
| PEI MAX - Transfection Grade Linear Polyethylenimine Hydrochloride (MW 40,000) | Polysciences | 24765-100MG | |
| Pipet-Aid XP | USA Scientific | 4440-0101 | |
| Poly-D-lysine hydrobromide | Sigma-Aldrich | P6407-5MG | |
| Rhosin hydrochloride | Bio-Techne Tocris | 5003 | Dissolve in DMSO; store at -70 °C |
| Trypsin-EDTA (0.05%), phenol red | ThermoFisher Scientific | 25300054 | |
| Varioskan LUX multimode microplate reader | ThermoFisher Scientific | VL0000D0 | |
| Vortex | ThermoFisher Scientific | 2215365 | level 8 |
| VWR Vacuum Aspiration System | VWR | 75870-734 | |
| ZCL 278 | Bio-Techne Tocris | 4794 | Dissolve in DMSO; store at -70 °C |
참고문헌
- Aoyama-Ishiwatari, S., Hirabayashi, Y. Endoplasmic reticulum-mitochondria contact sites-emerging intracellular signaling hubs. Front Cell Dev Biol. 9, 653828(2021).
- Sassano, M. L., Felipe-Abrio, B., Agostinis, P. ER-mitochondria contact sites; a multifaceted factory for Ca2+ signaling and lipid transport. Front Cell Dev Biol. 10, 988014(2022).
- Scorrano, L., et al. Coming together to define membrane contact sites. Nat Commun. 10 (1), 1287(2019).
- Voeltz, G. K., Sawyer, E. M., Hajnóczky, G., Prinz, W. A. Making the connection: How membrane contact sites have changed our view of organelle biology. Cell. 187 (2), 257-270 (2024).
- Paillusson, S., et al. There's something wrong with my MAM; the ER-mitochondria axis and neurodegenerative diseases. Trends Neurosci. 39 (3), 146-157 (2016).
- Sasi, U. S. S., Sindhu, G., Raj, P. S., Raghu, K. G. Mitochondria associated membranes (MAMs): emerging drug targets for diabetes. Curr Med Chem. 27 (20), 3362-3385 (2019).
- Joshi, A. U., Kornfeld, O. S., Mochly-Rosen, D. The entangled ER-mitochondrial axis as a potential therapeutic strategy in neurodegeneration: A tangled duo unchained. Cell Calcium. 60 (3), 218-234 (2016).
- Moltedo, O., Remondelli, P., Amodio, G. The mitochondria-endoplasmic reticulum contacts and their critical role in aging and age-associated diseases. Front Cell Dev Biol. 7, 172(2019).
- Rieusset, J. The role of endoplasmic reticulum-mitochondria contact sites in the control of glucose homeostasis: an update. Cell Death Dis. 9 (3), 388(2018).
- Bouguerra, M. D., Lalli, E. ER-mitochondria interactions: Both strength and weakness within cancer cells. Biochim Biophys Acta Mol Cell Res. 1866 (4), 650-662 (2019).
- Tepikin, A. V. Mitochondrial junctions with cellular organelles: Ca2+ signalling perspective. Pflügers Arch. 470 (8), 1181-1192 (2018).
- Masson, G. L., Przedborski, S., Abbott, L. F. A computational model of motor neuron degeneration. Neuron. 83 (4), 975-988 (2014).
- Giamogante, F., Barazzuol, L., Brini, M., Calì, T. ER-mitochondria contact sites reporters: strengths and weaknesses of the available approaches. Int J Mol Sci. 21 (21), 8157(2020).
- Rizzuto, R., et al. Close contacts with the endoplasmic reticulum as determinants of mitochondrial Ca2+ responses. Science. 280 (5370), 1763-1766 (1998).
- Valm, A. M., et al. Applying systems-level spectral imaging and analysis to reveal the organelle interactome. Nature. 546 (7656), 162-167 (2017).
- Csordás, G., et al. Imaging interorganelle contacts and local calcium dynamics at the ER-mitochondrial interface. Mol Cell. 39 (1), 121-132 (2010).
- Cieri, D., et al. SPLICS: a split green fluorescent protein-based contact site sensor for narrow and wide heterotypic organelle juxtaposition. Cell Death Differ. 25 (6), 1131-1145 (2018).
- Kakimoto, Y., et al. Visualizing multiple inter-organelle contact sites using the organelle- targeted split-GFP system. Sci Rep. 8 (1), 6175(2018).
- Wu, M. M., Covington, E. D., Lewis, R. S. Single-molecule analysis of diffusion and trapping of STIM1 and Orai1 at ER-plasma membrane junctions. Mol Biol Cell. 25 (22), Mac.E14-06-1107 (2014).
- Csordas, G., et al. Structural and functional features and significance of the physical linkage between ER and mitochondria. J Cell Biol. 174 (7), 915-921 (2006).
- de Brito, O. M., Scorrano, L. Mitofusin 2 tethers endoplasmic reticulum to mitochondria. Nature. 456 (7222), 605-610 (2008).
- Kremer, A., et al. Developing 3D SEM in a broad biological context. J Microsc. 259 (2), 80-96 (2015).
- Söderberg, O., et al. Direct observation of individual endogenous protein complexes in situ by proximity ligation. Nat Methods. 3 (12), 995-1000 (2006).
- Lim, Y., Cho, I. -T., Rennke, H. G., Cho, G. β2-adrenergic receptor regulates ER-mitochondria contacts. Sci Rep. 11 (1), 21477(2021).
- Lam, S. S., et al. Directed evolution of APEX2 for electron microscopy and proximity labeling. Nat Methods. 12 (1), 51-54 (2014).
- Cho, I. -T., et al. Ascorbate peroxidase proximity labeling coupled with biochemical fractionation identifies promoters of endoplasmic reticulum-mitochondrial contacts. J Biol Chem. 292 (39), 16382-16392 (2017).
- Lim, Y., Cho, I. -T., Schoel, L. J., Cho, G., Golden, J. A. Hereditary spastic paraplegia-linked REEP1 modulates endoplasmic reticulum/mitochondria contacts. Ann Neurol. 78 (5), 679-696 (2015).
- Arnold, T. R., Stephenson, R. E., Miller, A. L. Rho GTPases and actomyosin: Partners in regulating epithelial cell-cell junction structure and function. Exp Cell Res. 358 (1), 20-30 (2017).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유