Method Article
Produção e Otimização de LTE, um Sistema de Expressão de Proteína Livre de Células Derivadas de Leishmania tarentolae para Produção de Proteína Recombinante
Neste Artigo
Resumo
O Extrato Translacional de Leishmania (LTE) é um sistema de expressão de proteínas livres de células eucarióticas derivado do parasita unicelular, Leishmania tarentolae. Esse protocolo otimizado torna o LTE simples e econômico de fabricar. É adequado para várias aplicações focadas na expressão multiparalela e no estudo de proteínas eucarióticas complexas e suas interações.
Resumo
Este protocolo descreve a produção e otimização de um Sistema de Expressão de Proteína Livre de Células (CFPS) eucariótico derivado do flagelado unicelular Leishmania tarentolae, conhecido como Extrato Translacional de Leishmania ou LTE. Embora este organismo tenha evoluído originalmente como um parasita de lagartixas, ele pode ser cultivado de forma fácil e barata em frascos ou biorreatores. Ao contrário da Leishmania major, não é patogênica para os seres humanos e não requer precauções laboratoriais especiais. Outra vantagem de usar Leishmania para CFPS é que a adição de um único oligonucleotídeo antisense à CFPS, visando uma sequência líder de splice conservada na extremidade 5' de todos os RNAs codificadores de proteínas, pode suprimir a expressão de proteínas endógenas. Fornecemos procedimentos para interrupção celular e processamento de lisados, que foram simplificados e aprimorados em comparação com as versões anteriores. Esses procedimentos começam com culturas de frascos simples. Além disso, explicamos como introduzir informações genéticas usando vetores contendo locais de iniciação de tradução independentes da espécie (SITS) e como realizar otimização direta de lote e controle de qualidade para garantir a qualidade consistente da expressão da proteína.
Introdução
Na década de 1960, os sistemas de expressão de proteínas livres de células desempenharam um papel fundamental na descoberta do código genético1. No entanto, os sistemas de expressão de proteínas livres de células procarióticas, principalmente baseados em E. coli, atualmente dominam as aplicações laboratoriais e comerciais. Embora os sistemas baseados em E. coli ofereçam vantagens como custo-benefício, escalabilidade e altos rendimentos de expressão, eles enfrentam desafios ao produzir proteínas de múltiplos domínios em suas formas ativas e facilitar a montagem de complexos proteicos 2,3. Nos dias atuais, as formas comumente usadas de Síntese de Proteína Livre de Células Eucarióticas (CFPS) incluem extrato de gérmen de trigo (WGE), lisado de reticulócitos de coelho (RRL) e lisado de células de insetos (ICL) 4 , 5 , 6 . Este trabalho apresenta um sistema alternativo livre de células eucarióticas que é simples e escalável, baseado no parasita flagelado unicelular Leishmania tarentolae.
Leishmania tarentolae pode ser cultivada facilmente em frascos usando meios econômicos e também pode ser ampliada em biorreatores para atingir maior densidade celular. A presença de mRNAs endógenos no lisado celular, que de outra forma poderiam competir com as mensagens introduzidas, pode ser neutralizada usando oligonucleotídeos antisense direcionadosà sequência 7 do líder de splice de mRNA de Leishmania conservada. Ao contrário de seu parente próximo Leishmania major, que causa doenças humanas, L. tarentolae infecta a lagartixa-mourisca (Tarentolae mauritanica), tornando-a adequada para cultivo em ambientes de laboratório PC2 sem a necessidade de precauções especiais. Já foi usado anteriormente como um organismo transgênico para expressão de proteínas in vivo8.
Para facilitar o priming do molde em sistemas livres de células, sequências universais foram projetadas com base em estruturas poliméricas de RNA que aumentam a iniciação translacional9. Essas sequências de tradução independentes da espécie (SITS) são aplicáveis a sistemas livres de células procarióticas e eucarióticas e são adequadas para introduzir informações genéticas no LTE. Embora este protocolo não forneça uma explicação detalhada da construção do vetor para a expressão de proteínas livres de células LTE, a otimização e o controle de qualidade requerem vetores adequados contendo fusões de fluoróforos das proteínas desejadas de interesse a jusante do local STITS. Para esse fim, vetores LTE apropriados foram depositados no repositório de genes Addgene, como o vetor pCellFree_G03, que codifica uma fusão de eGFP N-terminal para a proteína de interesse desejada usando locais de clonagem Gateway.
O LTE provou seu valor em uma ampla gama de aplicações que requerem expressão de proteínas, incluindo a análise da automontagem de proteínas10,16, produção de proteínas de membrana integral humana17, pesquisa sobre candidatos a medicamentos antivirais18, desenvolvimento de enzimas biotecnologicamente úteis19, prototipagem de biossensores de proteínas20,21 e o estudo de produtos biológicos de ancilostomídeos22. O LTE também tem sido fundamental no mapeamento de redes de interação proteína-proteína nos campos da virologia e estruturas celulares21,32. O LTE foi avaliado para ter um desempenho semelhante a outros sistemas livres de células eucarióticas na expressão de proteínas de comprimento total, monodispersas e não agregadas33, ao mesmo tempo em que oferece uma produção mais econômica e escalável.
Este protocolo fornece técnicas para cultivar e interromper o organismo hospedeiro, preparar lisado e suplementar uma solução de alimentação (FS) para expressão de proteína de transcrição/tradução acoplada. Além disso, inclui um protocolo para otimizar os lotes de produção. Na versão inicial do sistema livre de células de Leishmania, foi observada variação indesejada de lote para lote nos níveis de expressão, na fração de proteínas de comprimento total e na presença de agregados proteicos, levando ao descarte de lotes34. Melhorias subsequentes no protocolo foram feitas para resolver esse problema25. O protocolo atual se baseia nessas melhorias, permitindo que lotes individuais sejam otimizados para pico de expressão e tamanho da proteína. Ele consegue isso controlando de perto a carga do disruptor celular (medida como densidade óptica a 600 nm; OD600nm) e normalizando a saída de lisado resultante usando absorbância a 280 nm (Abs280nm). Além disso, incorpora um método para suplementar parcialmente o lisado com rNTP e magnésio durante a fabricação, com subsequente otimização desses componentes da solução de alimentação durante as expressões de teste. Embora essa otimização seja apresentada como uma opção no protocolo, ela é fortemente recomendada pelos autores.
Protocolo
Este protocolo inclui receitas de mídia detalhadas e etapas que envolvem cultura, centrifugação, medição da fluorescência GFP usando um leitor de placas multimodo, medição de cultura OD600nm e avaliação de lisado Abs280nm. Também abrange a configuração e a imagem de géis de proteína SDS-PAGE. Os materiais necessários ou sugeridos para este protocolo estão listados na planilha de materiais. É importante observar que os recursos laboratoriais típicos, como componentes de mídia, centrífugas, tubos, espectrofotômetros e configurações de eletroforese em gel, provavelmente podem ser usados de forma intercambiável, a menos que especificado de outra forma. A Figura 1 fornece um resumo do processo de fabricação LTE.
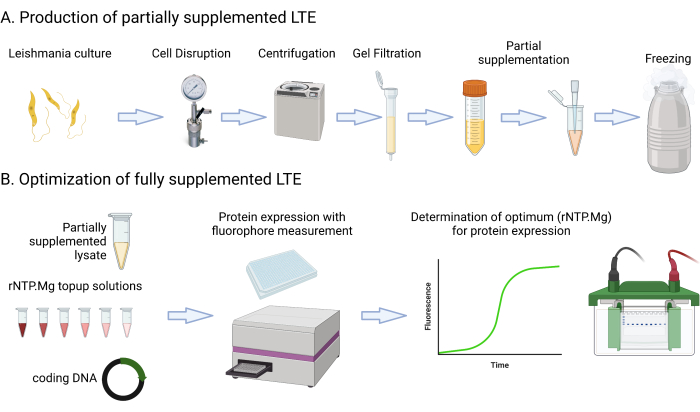
Figura 1: Visão geral do protocolo de fabricação LTE. Este desenho animado fornece um resumo conciso do protocolo de fabricação LTE. Clique aqui para ver uma versão maior desta figura.
1. Crescimento de culturas de Leishmania tarentolae
- Prepare pelo menos 3 L de meio de crescimento TBGG (bactotriptona 12 g/L, extrato de levedura 24 g/L, glicerol 8 mL/L, glicose 1 g/L, KH2PO4 2,3 g/L, K2HPO4 2,5 g/L, consulte a Tabela de Materiais). Esterilize a mídia usando um filtro de 0.22 μm sob vácuo ou uma configuração semelhante.
- Armazene o meio em temperatura ambiente (TR), com as adições finais (Hemin, antibióticos) adicionadas imediatamente antes da inoculação com L. tarentolae. Hemina (0,25% v / v em 50% de trietanolamina) é adicionada a 0,2% v / v, Penicilina (10.000 unidades / mL) mais estreptomicina (10.000 μg / mL) mistura a 0,5% v / v.
NOTA: O ponto de partida deste protocolo é uma cultura mantida de 2 x 10 mL de L. tarentolae do tipo selvagem. As culturas de manutenção são cultivadas a 27 °C em frascos padrão de 50 mL de cultura de tecidos com baixa agitação (75 rpm). Essas culturas de 10 mL podem ser mantidas indefinidamente com diluições de ~ 1/20 em estéril (TBGG + hemina, penicilina, estreptomicina) a cada 2-3 dias. Recomenda-se um gabinete de biossegurança padrão em um laboratório PC2; no entanto, as contaminações bacterianas tendem a ser evitadas pelos antibióticos adicionados, enquanto as contaminações fúngicas são geralmente superadas por L. tarentolae. - Ao longo de dois dias, expanda as culturas de manutenção de L. tarentolae para 200 mL (dia 1) e depois 2 L (dia 2) por meio de diluições de 1:10 com um volume crescente de TBGG + hemina / antibióticos a cada dia. Efectuar ambas as diluições em balões de vidro de 5 L defletores esterilizados em autoclave (cheios até um máximo de 1 L). A segunda diluição deve ocorrer no período da tarde, entre 15h e 18h, com a intenção de iniciar a produção de lisado no dia seguinte, entre 8h e 11h.
NOTA: Este protocolo usa o volume inicial mínimo para produção LTE (culturas de 2 x 1 L). Também é possível expandir a cultura até 10 L para a produção de LTE incorporando uma etapa de expansão adicional (por exemplo, Dia 1: 100 mL; Dia 2: 1 L; Dia 3: 10 L). Embora este protocolo use frascos defletores (ver Tabela de Materiais) para cultivar L. tarentolae, opcionalmente, biorreatores convencionais projetados para crescimento bacteriano com impulsores de Rushton podem ser usados, desde que a taxa de agitação seja mantida abaixo de 100 rpm. A melhor aeração e controle de pH em biorreatores geralmente estendem o crescimento da fase logarítmica das culturas de L. tarentolae , permitindo uma maior OD decolheita de 600 nm de 10 a ser usada na etapa 1.4. - Registar a DIÂMETRO DE600 nm da cultura em triplicado através de uma diluição de 1:10 em TBGG directamente na cubeta do espectrofotómetro. Uma faixa inicial adequada para fazer lisado é OD600nm = 4,0-8,0.
- Forneça tempo de incubação adicional se OD600nm < 4.0. Uma cultura com OD600nm > 8 é utilizável e resultará em um maior volume de lisado de expressão livre de células, mas com qualidade inferior devido ao início da fase de crescimento logarítmico tardio. Coloque os frascos de cultura no gelo, aguardando as etapas subsequentes.
NOTA: A medição precisa do OD decultura final de 600 nm é crítica, pois é usada para calcular o volume final das células concentradas antes da interrupção. Este cálculo substitui um método de pesagem de pellets usado em versões anteriores da fabricação de LTE para calibrar a concentração da célula antes da interrupção34, a fim de simplificar o protocolo. Certifique-se de diluir em TBGG para medição de OD600nm , caso contrário, o choque osmótico altera a forma da célula, causando erro de medição. Pipete misture a diluição 1:10 para medição OD600nm (diretamente na cubeta) segundos antes de fazer a leitura espectrofotométrica, pois as células de L. tarentolae se acomodam rapidamente com uma aparência turva distinta. Se o volume final da cultura de expressão for considerado aproximado, recomenda-se também a pesagem dos frascos na colheita (com tara de balão vazio adequada) para obter um volume melhor estimado (a 1 g = 1 ml). O OD máximo de600 nm possível do crescimento de L. tarentolae em frascos defletores é de 15-20, embora isso seja inadequado para a fabricação de lisado devido ao alcance da fase estacionária.
2. Concentração de culturas de L. tarentolae
- As células de Leishmania devem ser lavadas e concentradas aproximadamente 60 vezes antes da interrupção. Calcule o volume alvo para a concentração celular com base em OD600nm = 300 para o concentrado final. A equação é V = volume de colheita (mL) x (DO de colheita600nm/300). Por exemplo, usando uma cultura de 2 L com uma colheita OD600 = 5, o volume alvo é de 33 mL.
NOTA: O alvo OD600nm de 300 pode ser modificado; a produção anterior de LTE usou valores na faixa de 150-350. Concentrações mais altas de células que entram em ruptura tendem a produzir reações finais de expressão livre de células com maior rendimento de proteínas, mas com uma tendência aumentada de agregação de proteínas vulneráveis. OD600nm = 300 representa um alvo padrão adequado para produção LTE. - Transferir as culturas colhidas para frascos de centrifugação adequados e centrifugá-los a 2500 x g durante 10 min a 4 °C. Decantar cuidadosamente o sobrenadante para os resíduos de cultura.
NOTA: É importante minimizar a perda de células no sobrenadante descartado, pois isso afeta o cálculo da carga de interrupção. Em protocolos de produção LTE anteriores, a concentração de células de L. tarentolae para ruptura foi calibrada girando o concentrado de células em um tubo de microcentrífuga de teste e medindo o peso do pellet versus o peso total34. Em vez disso, este protocolo simplificado usa uma meta teórica de OD600nm para o concentrado, com base na OD de colheita medida de600nm, e pressupõe baixa perda de células durante a concentração e lavagem das células. - Lave o pellet celular em tampão SEB (45 mM HEPES-KOH pH 7,6, 250 mM de sacarose, 100 mM de KOAc, 3 mM de Mg(OAc)2, mantido em gelo) três vezes, cada vez centrifugando a 2500 x g por 10 min a 4 °C. Para a primeira lavagem, ressuspenda cada 1 L de cultura peletizada em 100 mL de tampão SEB e, em seguida, combine-os em um único frasco de centrífuga. Para a segunda lavagem, use também 100 mL de SEB para cada 1 L da cultura original.
NOTA: Para a ressuspensão final do pellet, adicione o tampão SEB a 50% do volume final de ressuspensão alvo (etapa 2.1). Isso permite que o concentrado agrupado seja cuidadosamente reabastecido exatamente até o volume alvo final na etapa 2.4. Cada ressuspensão deve ser o mais suave possível para evitar a lise prematura de L. tarentolae, por exemplo, girando suavemente o SEB adicionado ao redor do pellet decantado ou pipetando o SEB sobre o pellet aderido à parede do tubo da centrífuga. Pode ser mais conveniente transferir sobrenadantes para tubos de centrífuga menores para a etapa final. - Despeje o concentrado ressuspenso em um cilindro volumétrico de vidro lavado adequado, complete o volume até o volume alvo (etapa 2.1) usando SEB frio adicional e misture suavemente.
3. Lise do concentrado de L. tarentolae
- Transferir o concentrado de células para o dispositivo de cavitação de azoto (ver Tabela de Materiais) pré-arrefecido a 4 °C, pressurizá-lo a 70 bar de azoto e incubar durante 45 minutos em gelo.
NOTA: Embora os disruptores de cavitação de nitrogênio não sejam itens comuns de laboratório, eles são recomendados para a produção de LTE. Métodos alternativos, como congelamento e descongelamento celular e disruptores do tipo prensa francesa, foram tentados; no entanto, a atividade de expressão da proteína foi de <50% em comparação com o uso do método de cavitação de nitrogênio. O dispositivo de cavitação de nitrogênio deve ser cuidadosamente limpo antes do uso e entre as corridas, semelhante a todos os recipientes reutilizados que entram em contato com o lisado celular a partir desta etapa (como o frasco receptor). Um regime de limpeza adequado envolve a lavagem com detergentes de laboratório seguida de enxágue completo com água deionizada. - Abra a ventilação do dispositivo de cavitação de nitrogênio e expulse o lisado resultante para um recipiente adequadamente robusto, como um frasco receptor de vácuo no gelo. Incline o frasco receptor para garantir que todo o lisado resultante assente e possa ser pipetado em um novo tubo de centrífuga ou em um recipiente semelhante.
CUIDADO: Os disruptores de cavitação de nitrogênio dependem da transição abrupta do concentrado de células de 70 bar de nitrogênio para a pressão ambiente, obtida por meio de um forte fluxo de líquido primeiro e depois de nitrogênio através da válvula de saída do dispositivo. A ventilação deve ser feita com equipamento de proteção individual (EPI) apropriado em uma capa de segurança química. Existe o risco de quebrar o recipiente de destino e perder o lisado, e é por isso que usamos um receptor de vácuo robusto em vez de um frasco genérico. Se a válvula de saída do dispositivo for um tubo, evite colocar o tubo diretamente dentro do receptor para evitar o acúmulo excessivo de pressão no ponto de ventilação.
4. Centrifugação do lisado celular
- Transfira o lisado para tubos de centrífuga com força g adequada e centrifugue a 10.000 x g por 15 min a 4 °C. Remova o sobrenadante para tubos de centrífuga novos e semelhantes.
- Centrifugue o lisado a 30.000 x g durante 15 min a 4 °C e, em seguida, retire o sobrenadante final para um novo tubo de centrifugação ou um recipiente semelhante colocado sobre gelo. Estime o volume total.
5. Filtração em gel do lisado celular
NOTA: A filtração em gel é usada para remover a sacarose incluída no tampão SEB. Embora a sacarose auxilie na estabilização da maquinaria celular durante a ruptura celular, ela diminui o rendimento se retida nas reações de expressão de proteínas.
- Configure um número suficiente de colunas de filtragem de gel alimentadas por gravidade PD-10 (consulte a Tabela de Materiais) em um formato de rack que permita que elas gotejem em uma bandeja de coleta ou em um recipiente semelhante abaixo, garantindo que possam filtrar todo o volume de lisado a 2,5 mL por coluna. Pré-equilibre as colunas passando 10 mL de tampão EB a 4 ° C (45 mM HEPES-KOH pH 7,6, 100 mM KOAc, 3 mM Mg (OAc) 2) através delas de antemão.
NOTA: Todas as etapas a partir deste ponto se beneficiam de serem conduzidas em uma câmara fria a 4 °C. No entanto, também é adequado manter todos os lisados e reagentes em uma bandeja de gelo de bancada. Uma exceção é a etapa de filtração do gel, na qual os autores colocam um rack de colunas dentro de uma geladeira a 4 ° C durante o rebuffering. Nas versões originais deste protocolo, novas colunas de filtração de gel foram 'bloqueadas' inicialmente tamponando o lisado e descartando a primeira saída. Embora tal não seja considerado necessário, as colunas devem ser lavadas com tampão EB e armazenadas a 4 °C entre os lotes de lisados. O primeiro lisado de saída pode ter menor atividade de expressão de proteína do que as saídas subsequentes devido a alguma retenção de fundo dos componentes do lisado na nova coluna. - Adicione 2,5 mL de lisado a cada coluna e espere até que passe para a coluna. Adicione mais 0,5 mL de EB para depositar o lisado na coluna enquanto descarta o eluato.
- Eluir o lisado filtrado por gel adicionando mais 2,5 mL de EB a cada coluna, coletando a saída colocando uma bandeja nova e limpa ou outro recipiente sob as colunas.
6. Suplementação de lisado celular
- Use o espectrofotômetro nanodrop (consulte a Tabela de Materiais) para medir Abs280 nm do lisado filtrado por gel. Se exceder 60, dilua-o até atingir Abs280nm = 60 usando tampão EB adicional de 4 °C.
NOTA: Embora o controle da entrada de densidade celular na interrupção usando OD600nm determine aproximadamente a força de saída do lisado, normalizar o Abs280nm após a interrupção e o processamento do lisado melhora ainda mais a consistência do lote do desempenho do lisado. O Lysate Abs280nm pode ser ajustado para cima e para baixo, com consequências para o rendimento e agregação da expressão da proteína (consulte a seção Discussão). Se o lisado não suplementado indicar um Abs280nm < 60, pode ser necessário incluir mais biomassa de Leishmania na etapa de interrupção, ou seja, aumentar a carga do disruptor celular para OD600nm > 300 na etapa 2.1. - Adicione 5x Solução de Alimentação (5x FS, Tabela 1) ao lisado na proporção de 2:5 e misture bem o vórtice. Alicote em recipientes adequados (por exemplo, tubos de microcentrífugas de 1,5 mL) e congele-o em nitrogênio líquido. Se alguém estiver seguindo as etapas opcionais 7.1-7.3 para otimização de expressão LTE abaixo, use o rNTP.Mg reduzido 5x FS da Tabela 1 em vez do padrão 5x FS. Incluir alíquotas de 5 x 100 μL para congelação para utilização nas experiências de optimização.
NOTA: O congelamento com o padrão 5x FS em uma proporção de 2:5 cria LTE suplementado pronto para expressão, usado na expressão de 7 μL/10 μL (portanto, o 5x FS torna-se 1x FS na reação final). No entanto, os autores recomendam seguir as etapas opcionais adicionais em que 0,6x a quantidade padrão de rNTPs e magnésio é fornecida no 5x FS. Isso é seguido por uma etapa de otimização em que uma combinação equimolar dos dois (conhecida como rNTP.Mg) é adicionada para completar as reações de teste para um valor otimizado. O 5x FS parcial também contém um oligonucleotídeo que desliga a expressão endógena de mRNA (consulte a seção Introdução). A sequência do oligonucleotídeo é CAATAAAGTACAGAAACTGATACTTATATAGCGTT.
7. CQ e otimização do LTE final suplementado
NOTA: As etapas mínimas necessárias para determinar a adição apropriada de rNTP.Mg ao rNTP reduzido e lisado suplementado com magnésio envolvem a expressão de eGFP ou um fluoróforo semelhante (por exemplo, sfGFP) sem um parceiro de fusão. Concentrações crescentes de rNTP.Mg são adicionadas às reações para determinar o ponto em que o nível de expressão (medido como eGFP RFU por meio de um leitor de placas multimodo) é otimizado. As terminações prematuras do eGFP, que não são fluorescentes, tornam-se evidentes pela diminuição do eGFP RFU em concentrações de rNTP.Mg muito altas. No entanto, o mau funcionamento do LTE por produto curto ocorre com mais frequência em proteínas expressas maiores (>50 kDa). Portanto, é possível realizar essa otimização usando um modelo maior do que o eGFP, especialmente se um estiver disponível em um vetor de expressão adequado, fornecendo uma fusão de fluoróforo que se deseja produzir por LTE para uma aplicação ou estudo específico (consulte a seção Resultados representativos).
- Descongele uma alíquota de 100 μL e estabeleça seis reações de expressão de 10 μL, cada uma composta de 7 μL de lisado parcialmente suplementado da etapa 6.2, 1 μL de solução de recarga conforme a Tabela 2 e 2 μL de água ultrapura contendo molde de controle de DNA suficiente para atingir uma concentração final de 50 ng / μL na reação.
- Incubar as reações durante 2 h a 25 °C e monitorizar o aumento da fluorescência GFP utilizando um leitor de placas multimodo.
NOTA: Os valores de configuração adequados para GFP são excitação a 485 nm (largura de banda 5 nm), emissão a 516 nm (largura de banda 5 nm), com um intervalo de leitura de 1 min por 2 h. - Classifique os valores finais da expressão para determinar a concentração de rNTP.Mg correspondente à RFU eGFP mais alta. Se os dados cinéticos estiverem disponíveis, um excesso de rNTP.Mg também será indicado por um aumento bifásico na RFU eGFP durante o período de expressão de 2 h (consulte a seção Resultados representativos).
- Uma vez determinada a concentração otimizada de rNTP.Mg de complementação, adicione-a a todas as outras expressões proteicas usando o lote de LTE que foi parcialmente suplementado nas etapas anteriores.
NOTA: Se a alíquota na etapa 6.2 for feita cuidadosamente com volumes fixos, a recarga pode ser adicionada retrospectivamente a cada alíquota sem descongelar, por exemplo, com as alíquotas colocadas em gelo seco. Essas alíquotas agora são totalmente complementadas, pois a rNTP.Mg recarga correta será misturada em cada uma quando forem descongeladas e misturadas para uso.
Resultados
O objetivo da expressão de proteínas livres de células é produzir proteínas de comprimento total em uma forma ativa dobrada adequada para uma ampla gama de aplicações. O LTE (extrato de Leishmania tarentolae ) já foi comparado a outros sistemas de expressão livre de células procarióticas e eucarióticas, demonstrando uma alta capacidade de evitar truncamento e agregação quando operando de maneira ideal, particularmente em comparação com a expressão livre de células baseada em E. coli 33. No entanto, isso foi anteriormente acompanhado por uma variação significativa de lote para lote na qualidade da saída. O método atual incorpora melhorias adicionais para garantir uma qualidade de saída consistente, principalmente por meio da suplementação parcial da solução de alimentação necessária antes do congelamento inicial do LTE em alíquotas. Isso é seguido pela otimização da entrada transcricional rNTP.Mg em uma solução de recarga que pode ser adicionada a cada reação subsequente ou usada para completar as alíquotas congeladas diretamente. Vale ressaltar que as reações de otimização também representam o uso típico de LTE para expressar proteínas de forma prática, com reações realizadas a 25 °C por 2 h.
Os dados da otimização da concentração de rNTP.Mg em reações livres de células fornecem um conjunto de dados representativo. Os níveis de expressão geralmente aumentam à medida que a entrada transcricional (rNTP.Mg) aumenta, indicando expressão bem-sucedida. No entanto, um limite é atingido onde o sistema tende à expressão não produtiva de produtos truncados, particularmente no caso de proteínas maiores (>50 kDa). Essa expressão abaixo do ideal leva a uma perda de sinal de fluorescência com o aumento da rNTP.Mg, particularmente evidente com fusões de fluoróforos C-terminais, onde a tradução do polipeptídeo não atinge o próprio fluoróforo. Para fusões N-terminais, embora uma redução na RFU geral (Unidades de Fluorescência Relativa) não ocorra necessariamente com excesso de rNTP.Mg, a expressão falha é visivelmente aparente em géis SDS-PAGE como vários produtos fluorescentes de tamanhos decrescentes. Essa abordagem aproveita a capacidade da GFP (Proteína Fluorescente Verde) de manter a fluorescência mesmo quando visualizada em um gel SDS-PAGE convencional, desde que as amostras não sejam aquecidas após a mistura. Em vez disso, eles são misturados com tampão de carregamento de gel e carregados diretamente no gel. Embora os materiais e equipamentos de gel SDS-PAGE sejam geralmente intercambiáveis, o gerador de imagens de gel deve ser capaz de visualizar a fluorescência GFP. Uma configuração típica para visualização GFP é fornecida com excitação a 485 nm (largura de banda 5 nm), emissão a 516 nm (largura de banda 5 nm) e um intervalo de leitura de 1 min em 2 h.
É possível otimizar o sistema usando apenas a expressão de eGFP. A Figura 2A, B (inserção) mostra as saídas de expressão típicas de reações de otimização para dois lotes LTE, com eGFP RFU aumentando com o aumento das concentrações de rNTP.Mg, atingindo um nível ideal de +0,6x rNTP.Mg (Figura 2A) e +0,3x rNTP.Mg (Figura 2B) para RFU máxima. A solução de alimentação rNTP reduzida inclui 0,6x rNTP.Mg, resultando em níveis totais de rNTP.Mg de 1,2x e 0,9x a quantidade padrão para esses lotes LTE. A Figura 2C ilustra a cinética do aumento da RFU durante a reação para o lote LTE na Figura 2B, demonstrando uma reação bifásica com duas fases discretas ao longo da duração da reação.

Figura 2: Otimização da adição de top-up de rNTP.Mg em LTE com rNTP 5x FS reduzido. (A) Níveis de expressão de eGFP após 140 min de expressão de um plasmídeo de controle em níveis de top-up de rNTP.Mg variados na reação de expressão livre de células (n = 3, Média ± SD plotado). (B) Otimização de um lote de produção LTE diferente, mostrando expressão reduzida além de um determinado limite rNTP.Mg. (C) Cinética de acúmulo de eGFP nas mesmas reações que (B), com aumento da rNTP.Mg recarga. Esses dados também representam a cinética típica de acúmulo de proteínas na expressão do lote LTE. Clique aqui para ver uma versão maior desta figura.
No entanto, deve-se notar que a eGFP, sendo uma proteína pequena (27 kDa) que é fácil de dobrar, provavelmente será expressa, dobrada e amadurecida independentemente do sistema de expressão livre de células usado. A falha do sistema é mais provável ao expressar proteínas maiores de interesse, com produtos truncados tornando-se mais aparentes em tamanhos de proteína de entrada maiores que 70 kDa33. Portanto, a otimização do sistema com a(s) proteína(s) pretendida(s) para uso real é superior, com eGFP ainda presente para quantificação, mas como uma fusão N-terminal com a proteína de interesse.
A Figura 3 representa uma otimização típica do nível de recarga rNTP.Mg ao usar um modelo de proteína maior propenso a fornecer produtos truncados (eGFP-Sox18). Usando um formato SDS-PAGE de gel semi-nativo (ou seja, sem amostras de aquecimento), é possível visualizar a falha progressiva de expressão. A adição ideal de rNTP.Mg a +0,1x (combinada com a rNTP.Mg de 0,6x na solução de alimentação parcial, totalizando 0,7x) reduz claramente a fração da banda de proteína de comprimento total como parte do total de produtos de expressão fluorescente, significando falha do sistema com excesso de rNTP.Mg adição.
Conforme mencionado no protocolo, é possível pular a etapa de otimização rNTP.Mg e adicionar diretamente a quantidade total de rNTP.Mg na solução de alimentação "padrão" durante a suplementação imediatamente após a filtração do gel na etapa 6.2. Ao fazer isso, o protocolo essencialmente reverte para os métodos originais publicados para criar o LTE34. No entanto, os autores acreditam que adaptar o sistema para um desempenho ideal, conforme demonstrado na Figura 3 (Pista D para Pista E), supera a complexidade adicional do protocolo e aumenta o valor do LTE como uma ferramenta de expressão de proteínas.
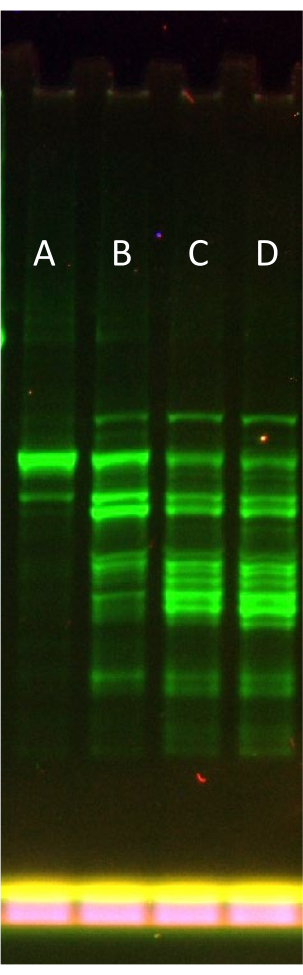
Figura 3: Efeito do aumento da recarga de rNTP.Mg na expressão de eGFP-Sox18 em LTE parcialmente suplementado. Gel SDS-PAGE semi-nativo que descreve o impacto do aumento da recarga de rNTP.Mg na expressão de eGFP-Sox18. Pista A: +0,1x (rNTP.Mg) de recarga, Pista B: +0,2x (rNTP.Mg), Pista C: +0,3x (rNTP.Mg), Pista D: +0,4x (rNTP.Mg). A fusão N-terminal eGFP é visualizada por varredura de fluorescência do gel. A banda primária na Pista A representa o eGFP-Sox18 completo. Clique aqui para ver uma versão maior desta figura.
| Componente | Concentração de estoque | 5x Solução de Alimentação | μL de estoque/mL 5x Solução de Alimentação |
| Padrão (rNTP reduzido) | Padrão (rNTP reduzido) | ||
| Espermidina | 100 mM | 1,25 mM | 12 |
| TDT | 500 mM | 10 mM | 20 |
| Fosfato de creatina | 1000 mM | 200 mM | 200 |
| HEPES-KOH pH 7,6 | 2500 mM | 100 mM | 40 |
| PEG3000 | 0,5 v/v | 0,05 v/v | 100 |
| Coquetel de inibidores de protease | 120 vezes | 5x | 43 |
| Aminoácidos | 3,6 mM (ea) | 0,68 mM (ea) | 190 |
| ATP | 100 mM | 8,5 (5,1) mM | 85 (51) |
| GTP | 100 mM | 3,2 (1,9) mM | 32 (19) |
| UTP | 100 mM | 2,5 (1,5) mM | 25 (15) |
| CTP | 100 mM | 2,5 (1,5) mM | 25 (15) |
| Mg(OAc)2 | 1 milhão | 16,7 (10) mM | 16.7 (10) |
| Oligo líder anti-emenda | 1mM | 0,05 mM | 50 |
| RNA polimerase T7 | 5 mg/ml | 0,5 mg/mL | 100 |
| Creatina Fosfoquinase | 5 unidades/μL | 0,2 unidades/μL | 42 |
| Água ultrapura | 19 (93) |
Tabela 1: Composição da solução de alimentação 5x (5x FS) para LTE. 1 mL de 5x FS é necessário para cada 2,5 mL de lisado não suplementado após a filtração do gel. A suplementação com o padrão 5x FS cria um LTE pronto para expressão para uso em reações de expressão em uma proporção de 7 μL/10 μL. A receita de rNTP.Mg reduzida (quantidades em itálico) é recomendada para otimização da expressão LTE e contém 0,6 vezes os níveis padrão de rNTPs e magnésio. Estes podem ser ajustados para níveis variáveis (0,6 a 1,1 vezes) no experimento de otimização subsequente usando as adições descritas na Tabela 2.
| Recarga de rNTP | ATP (100 mM) | GTP (100 mM) | UTP (100 mM) | CTP (100 mM) | MgOAc (1 M) | Água ultrapura |
| (1 μL/10 μL rxn) | μL/200 μL | μL/200 μL | μL/200 μL | μL/200 μL | μL/200 μL | |
| +0x | 0 | 0 | 0 | 0 | 0 | 200 |
| +0,1x | 3.4 | 1.3 | 1 | 1 | 0.7 | 193 |
| +0,2x | 6.8 | 2.5 | 2 | 2 | 1.3 | 185 |
| +0,3x | 10.2 | 3.8 | 3 | 3 | 2 | 178 |
| +0,4x | 13.6 | 5.1 | 4 | 4 | 2.7 | 171 |
| +0,5x | 17 | 6.4 | 5 | 5 | 3.3 | 163 |
Tabela 2: Composição de (rNTP.Mg) soluções de recarga para otimização LTE. Essas soluções são usadas para otimizar o LTE adicionando 1 μL de solução de recarga por 10 μL de reação de expressão de proteína. Uma vez que um nível de recarga é determinado no experimento de otimização, ele pode ser adicionado de forma consistente a todas as reações de expressão de proteína subsequentes usando as mesmas alíquotas de lote LTE. Alternativamente, pode ser adicionado diretamente às alíquotas com adição de 1 μL por 7 μL (sem descongelamento). Após o descongelamento e a mistura, esses lisados são usados a 8 μL LTE por 10 μL de expressão de proteína, mantendo o nível de topup rNTP.Mg estabelecido durante a otimização.
Discussão
Os protocolos para criação de LTE foram publicados na última década7 e passaram por atualizações periódicas25,34. No entanto, os recém-chegados à técnica geralmente encontram uma curva de aprendizado íngreme, resultando em atrasos na obtenção de expressão de proteínas de alta qualidade e alto rendimento. Desafios semelhantes foram relatados por outros grupos de pesquisa que trabalham com LTE35, particularmente em relação a variações significativas de lote para lote. O formato de protocolo baseado em vídeo pode potencialmente fornecer conhecimento de configuração adicional e menos óbvio que beneficia usuários em potencial34. Modificações foram introduzidas no protocolo, com o objetivo de aumentar a probabilidade de sucesso, simplificar o procedimento, reduzir o tempo e minimizar erros relacionados à complexidade.
Na ruptura celular, o controle preciso sobre o carregamento das células no disruptor celular de cavitação de nitrogênio é crucial34. Conseguir isso pode ser um desafio devido à alta densidade celular após a concentração e lavagem das células. Nos protocolos originais, vários métodos foram usados, como girar um pequeno volume da cultura concentrada final e quantificar o pellet de célula fracionada. No entanto, neste protocolo, é adotada uma abordagem mais simples. O volume de colheita da cultura e o OD600nm são medidos, e essas medições são usadas para calcular um volume alvo para o concentrado de células em mililitros, visando um OD final desejado de600nm de 300. Este cálculo pressupõe que não ocorra perda celular significativa durante a lavagem. Se houver suspeita de perda de células, um método alternativo envolvendo diluições seriais triplicadas de 1/10 das células concentradas após a lavagem é empregado, resultando em uma diluição de 1/1000. Isso permite medir o diâmetro externo real do concentradode 600 nm, garantindo que ele atinja o diâmetro externo alvode 600 nm = 300 antes de carregá-lo no disruptor.
Mesmo com um controle cuidadoso da carga do disruptor celular, pode ocorrer uma variabilidade significativa no conteúdo de proteína do lisado pós-interrupção, conforme indicado pelo Abs280nm do lisado não suplementado filtrado por gel34. Portanto, uma medição de Abs280nm antes da suplementação de lisado é introduzida, e o lisado é diluído para atingir um Abs280nm = 60. Como as reações de expressão de proteínas eventualmente incluem lisado de 0,5 v / v, isso resulta em um lisado de reação padronizado com Abs280nm = 30. O desempenho do lisado configurado com Abs280nm = 30 tende a produzir reações de baixa expressão e longa duração, enquanto valores maiores que Abs280nm = 30 tendem a produzir maior expressão, mas uma tendência aumentada para agregação de proteínas de saída.
A otimização do desempenho do lisado envolve o ajuste das entradas transcricionais na solução de alimentação que complementa o lisado, especificamente rNTPs e magnésio, nas etapas de reação opcionais 7.0-7.3. É importante observar que os rNTPs e o magnésio têm papéis complexos e múltiplos em um sistema de transcrição-tradução acoplado como o LTE25. No entanto, o LTE demonstrou ter uma expressão aproximada de magnésio ótima em rNTP (mM) + 1,5. Como o próprio lisado contribui com 1,5 mM de Mg para a mistura de reação final, isso fornece uma maneira direta de variar e otimizar a entrada de rNTP sem co-otimizar o Mg variando a rNTP.Mg equimolar.
O desempenho do lisado exibe variação significativa ao aumentar a rNTP.Mg, com a expressão da proteína geralmente aumentando até um limite em que a otimização se inverte, resultando em mau funcionamento da expressão na forma de produtos curtos em vez de proteínas de comprimento total25. Portanto, a otimização final do sistema para identificar esse limite é benéfica. O protocolo LTE original empregava uma receita de solução de alimentação fixa, com alguma otimização de Mg sugerida34. Essa abordagem foi posteriormente modificada com uma otimização rNTP mais extensa. No entanto, esse método exigia o congelamento instantâneo do lisado na forma não suplementada para permitir a otimização em uma alíquota, o que tendia a diminuir os níveis de expressão do lisado. Essa redução foi atribuída à perda das propriedades crioprotetoras da solução de alimentação ao congelar o lisado não suplementado imediatamente após a etapa de filtração do gel. O protocolo atual atinge um equilíbrio ao suplementar com uma solução de alimentação contendo rNTP.Mg reduzido antes do congelamento, que pode ser complementado até o nível otimizado no ponto de expressão.
Espera-se que essas melhorias de protocolo aumentem a utilidade do sistema LTE para usuários iniciantes, mitigando as principais fontes de variação e melhorando a consistência da saída da expressão da proteína.
Divulgações
Não estão presentes interesses financeiros concorrentes.
Agradecimentos
Os autores desejam reconhecer os muitos membros do laboratório Alexandrov que contribuíram para o desenvolvimento dos sistemas LTE nos últimos 10 anos, em particular Sergey Mureev, que foi pioneiro no sistema e desenvolveu o local de entrada do ribossomo SITS. A Figura 1 foi criada por Biorender.com e reproduzida sob licença.
Materiais
| Name | Company | Catalog Number | Comments |
| PD-10 SuperDex 25 Columns | Cytiva | 17085101 | Gel filtration columns |
| Nitrogen Cavitation cell disrupter | Parr Industries | 4635 or 4639 | Cell Disrupter |
| Bovine derived Hemin | Sigma-Aldrich | H5533 | Culture additive |
| Penicillin/Streptomycin 10000U/ml | Thermo-Fisher | 15140122 | Antibiotic mix |
| Optiplate 384 | Perkin-Elmer | 6007290 | Multiwell plate for 10ul expressions |
| Oligonucleotide | IDT synthesis | Oligo with sequence CAATAAAGTACAGAAACTGATAC TTATATAGCGTT | |
| Creatine Phosphokinase | Sigma-Aldrich | 9001-15-4 | Enzyme |
| Tecan Spark | Tecan | or similar Multimode Platereader | |
| Chemidoc MP Imager | Biorad | or similar SDS-PAGE gel Imager | |
| 4-12% Bis-Tris Gels | Invitrogen | NW04125 | SDS-PAGE gels |
| Biophotometer | Eppendorf | or similar Cuvette Specrophotometer | |
| Nanodrop One | Thermofisher | Nanodrop spectrophotometer | |
| Avanti JXN-26 centrifuge | Beckman Coulter | or similar centrifuge, with rotors/tubes rated 10K and 50K g | |
| 5424R microcentrifuge | Eppendorf | or similar microcentrifuge, with 1.5ml microcentrifuge tubes | |
| Flask Incubator Inova S44i | Eppendorf | or similar flask incubator shaker suitable for 5L Flasks | |
| 5L glass culture flasks | Baffled glass flasks for culture growth | ||
| Bactotryptone | BD | 211705 | Growth medium |
| Yeast Extract | Merck | VM930053 | Growth medium |
| Glycerol | Any analytical grade | ||
| Glucose | Any analytical grade | ||
| KH2PO4 | Any analytical grade | ||
| K2HPO4 | Any analytical grade | ||
| UltraPure water | Invitrogen | 10977-015 | Or output from any MilliQ-type water dispenser |
Referências
- Nirenberg, M. W., Matthaei, J. H. The dependence of cell-free protein synthesis in E.coli upon naturally occurring or synthetic polyribonucleotides. Proc Natl Acad Sci USA. 47 (10), 1588-1602 (1961).
- Caschera, F., Noireaux, V. Synthesis of 2.3 mg/ml of protein with an all Escherichia coli cell-free transcription-translation system. Biochimie. 99, 162-168 (2014).
- Kelwick, R., Webb, A. J., MacDonald, J. T., Freemont, P. S. Development of a Bacillus subtilis cell-free transcription-translation system for prototyping regulatory elements. Metab Eng. 38, 370-381 (2016).
- Ezure, T., et al. Cell-free protein synthesis system prepared from insect cells by freeze-thawing. Biotechnol Prog. 22 (6), 1570-1577 (2006).
- Harbers, M. Wheat germ systems for cell-free protein expression. FEBS Letters. 588 (17), 2762-2773 (2014).
- Kobs, G. Selecting the cell-free protein expression system that meets your experimental goals. Promega Corporation. 21, 6-9 (2008).
- Kovtun, O., et al. Leishmania cell-free protein expression system. Methods. 55 (1), 58-64 (2011).
- Basile, G., Peticca, M. Recombinant protein expression in Leishmania tarentolae. Mol Biotechnol. 43 (3), 273-278 (2009).
- Mureev, S., Kovtun, O., Nguyen, U. T., Alexandrov, K. Species-independent translational leaders facilitate cell-free expression. Nat Biotechnol. 27 (8), 747-752 (2009).
- Gambin, Y., et al. Single-molecule fluorescence reveals the oligomerization and folding steps driving the prion-like behavior of ASC. J Mol Biol. 430 (4), 491-508 (2018).
- Sierecki, E., et al. Rapid mapping of interactions between human SNX-BAR proteins measured in vitro by AlphaScreen and single-molecule spectroscopy. Mol Cell Proteomics. 13 (9), 2233-2245 (2014).
- Sierecki, E., et al. Nanomolar oligomerization and selective co-aggregation of alpha-synuclein pathogenic mutants revealed by single-molecule fluorescence. Sci Rep. 6, 37630 (2016).
- Leitao, A., Bhumkar, A., Hunter, D. J. B., Gambin, Y., Sierecki, E. Unveiling a selective mechanism for the inhibition of alpha-synuclein aggregation by beta-synuclein. Int J Mol Sci. 19 (2), 334 (2018).
- Gambin, Y., et al. Single-molecule analysis reveals self assembly and nanoscale segregation of two distinct cavin subcomplexes on caveolae. Elife. 3, e01434 (2013).
- Ve, T., et al. Structural basis of TIR-domain-assembly formation in MAL- and MyD88-dependent TLR4 signaling. Nat Struct Mol Biol. 24 (9), 743-751 (2017).
- Guo, Z., et al. Subunit organisation of in vitro reconstituted HOPS and CORVET multisubunit membrane tethering complexes. PLoS One. 8 (12), e81534 (2013).
- Ruehrer, S., Michel, H. Exploiting Leishmania tarentolae cell-free extracts for the synthesis of human solute carriers. Mol Membr Biol. 30 (4), 288-302 (2013).
- Varasteh Moradi, S., et al. Mapping Interactions among cell-free expressed Zika virus proteins. J Proteome Res. 19 (4), 1522-1532 (2020).
- Gagoski, D., et al. Cell-free pipeline for discovery of thermotolerant xylanases and endo-1,4-beta-glucanases. J Biotechnol. 259, 191-198 (2017).
- Ergun Ayva, C., et al. Exploring performance parameters of artificial allosteric protein switches. J Mol Biol. 434 (17), 167678 (2022).
- Lau, D., et al. Fluorescence biosensor for real-time interaction dynamics of host proteins with HIV-1 capsid tubes. ACS Appl Mater Interfaces. 11 (38), 34586-34594 (2019).
- Ryan, S. M., et al. Novel antiinflammatory biologics shaped by parasite-host coevolution. Proc Natl Acad Sci USA. 119 (36), e2202795119 (2022).
- McMahon, K. A., et al. Identification of intracellular cavin target proteins reveals cavin-PP1alpha interactions regulate apoptosis. Nat Commun. 10 (1), 3279 (2019).
- Sierecki, E., et al. A cell-free approach to accelerate the study of protein-protein interactions in vitro. Interface Focus. 3 (5), 20230018 (2013).
- Johnston, W. A., Moradi, S. V., Alexandrov, K. Adaption of the Leishmania cell-free expression system to high-throughput analysis of protein interactions. Methods Mol Biol. 2025, 403-421 (2019).
- Jung, W., et al. Cell-free formation and interactome analysis of caveolae. J Cell Biol. 217 (6), 2141-2165 (2018).
- Fontaine, F. R., et al. Functional domain analysis of SOX18 transcription factor using a single-chain variable fragment-based approach. MAbs. 10 (4), 596-606 (2018).
- Overman, J., et al. Pharmacological targeting of the transcription factor SOX18 delays breast cancer in mice. Elife. 6, e21221 (2017).
- Kubala, M. H., et al. Mammalian farnesyltransferase alpha subunit regulates vacuolar protein sorting-associated protein 4A (Vps4A)--dependent intracellular trafficking through recycling endosomes. Biochem Biophys Res Commun. 468 (4), 580-586 (2015).
- Han, S. P., et al. Cortactin scaffolds Arp2/3 and WAVE2 at the epithelial zonula adherens. J Biol Chem. 289 (11), 7764-7775 (2014).
- Das Gupta, K., et al. Class IIa histone deacetylases drive toll-like receptor-inducible glycolysis and macrophage inflammatory responses via pyruvate kinase M2. Cell Rep. 30 (8), 2712-2728.e8 (2020).
- Leitão, A. D. G., et al. Selectivity of protein interactions along the aggregation pathway of α-synuclein. BioRxiv. , (2021).
- Gagoski, D., et al. Performance benchmarking of four cell-free protein expression systems. Biotechnol Bioeng. 113 (2), 292-300 (2016).
- Johnston, W. A., Alexandrov, K. Production of eukaryotic cell-free lysate from Leishmania tarentolae. Methods Mol Biol. 1118, 1-15 (2014).
- Hunter, D. J. B., Bhumkar, A., Giles, N., Sierecki, E., Gambin, Y. Unexpected instabilities explain batch-to-batch variability in cell-free protein expression systems. Biotechnol Bioeng. 115 (8), 1904-1914 (2018).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados