Method Article
Produktion und Optimierung von LTE, einem von Leishmania tarentolae abgeleiteten zellfreien Proteinexpressionssystem für die rekombinante Proteinproduktion
In diesem Artikel
Zusammenfassung
Leishmania Translational Extract (LTE) ist ein eukaryotisches zellfreies Proteinexpressionssystem, das aus dem einzelligen Parasiten Leishmania tarentolae gewonnen wird. Dieses optimierte Protokoll macht LTE einfach und kostengünstig in der Herstellung. Es eignet sich für verschiedene Anwendungen, die sich auf die multiparallele Expression und Untersuchung komplexer eukaryotischer Proteine und ihrer Wechselwirkungen konzentrieren.
Zusammenfassung
Dieses Protokoll beschreibt die Produktion und Optimierung eines eukaryotischen zellfreien Proteinexpressionssystems (CFPS), das aus dem einzelligen Flagellaten Leishmania tarentolae gewonnen wird, der als Leishmania Translational Extract oder LTE bezeichnet wird. Obwohl sich dieser Organismus ursprünglich als Parasit der Geckos entwickelt hat, kann er einfach und kostengünstig in Kolben oder Bioreaktoren kultiviert werden. Im Gegensatz zu Leishmania major ist es für den Menschen nicht pathogen und erfordert keine besonderen Laborvorsichtsmaßnahmen. Ein weiterer Vorteil der Verwendung von Leishmania bei CFPS besteht darin, dass die Zugabe eines einzelnen Antisense-Oligonukleotids zum CFPS, das auf eine konservierte Spleißleader-Sequenz am 5'-Ende aller proteinkodierenden RNAs abzielt, die endogene Proteinexpression unterdrücken kann. Wir bieten Verfahren für den Zellaufschluss und die Lysatverarbeitung an, die im Vergleich zu früheren Versionen vereinfacht und verbessert wurden. Diese Verfahren beginnen mit einfachen Kolbenkulturen. Darüber hinaus erklären wir, wie genetische Informationen mithilfe von Vektoren eingeführt werden können, die speziesunabhängige Translationsinitiationsstellen (SITS) enthalten, und wie eine einfache Chargenoptimierung und Qualitätskontrolle durchgeführt werden kann, um eine gleichbleibende Proteinexpressionsqualität zu gewährleisten.
Einleitung
In den 1960er Jahren spielten zellfreie Proteinexpressionssysteme eine entscheidende Rolle bei der Aufdeckung des genetischen Codes1. Prokaryotische zellfreie Proteinexpressionssysteme, die hauptsächlich auf E. coli basieren, dominieren derzeit jedoch sowohl im Labor als auch in der kommerziellen Anwendung. Während E. coli-basierte Systeme Vorteile wie Kosteneffizienz, Skalierbarkeit und hohe Expressionsausbeuten bieten, stehen sie vor Herausforderungen bei der Herstellung von Multi-Domain-Proteinen in ihren aktiven Formen und der Erleichterung des Zusammenbaus von Proteinkomplexen 2,3. Zu den heute gebräuchlichen Formen der eukaryotischen zellfreien Proteinsynthese (CFPS) gehören Weizenkeimextrakt (WGE), Kaninchen-Retikulozytenlysat (RRL) und Insektenzelllysat (ICL)4,5,6. In dieser Arbeit wird ein alternatives eukaryotisches zellfreies System vorgestellt, das sowohl unkompliziert als auch skalierbar ist und auf dem einzelligen Flagellatenparasiten Leishmania tarentolae basiert.
Leishmania tarentolae kann leicht in Kolben mit kostengünstigen Medien kultiviert werden und kann auch in Bioreaktoren hochskaliert werden, um eine höhere Zelldichte zu erreichen. Das Vorhandensein endogener mRNAs im Zelllysat, die andernfalls mit eingeführten Nachrichten konkurrieren könnten, kann mit Antisense-Oligonukleotiden neutralisiert werden, die auf die konservierte Leishmania mRNA-Spleißleader-Sequenz7 abzielen. Im Gegensatz zu seinem nahen Verwandten Leishmania major, der den Menschen erregt, infiziert L. tarentolae den Maurischen Gecko (Tarentolae mauritanica) und eignet sich daher für die Kultivierung in PC2-Laborumgebungen, ohne dass besondere Vorsichtsmaßnahmen erforderlich sind. Es wurde zuvor als transgener Organismus für die In-vivo-Proteinexpression verwendet8.
Um das Template-Priming in zellfreien Systemen zu erleichtern, wurden universelle Sequenzen auf der Grundlage polymerer RNA-Strukturen entwickelt, die die translationale Initiation verbessern9. Diese speziesunabhängigen Translationssequenzen (SITS) sind sowohl für prokaryotische als auch für eukaryotische zellfreie Systeme anwendbar und eignen sich für die Einführung genetischer Informationen in LTE. Obwohl dieses Protokoll keine detaillierte Erklärung der Vektorkonstruktion für die zellfreie LTE-Proteinexpression liefert, erfordern Optimierung und Qualitätskontrolle geeignete Vektoren, die Fluorophorfusionen der gewünschten Proteine von Interesse stromabwärts der SITS-Stelle enthalten. Zu diesem Zweck wurden geeignete LTE-Vektoren bei dem Addgene-Genrepository hinterlegt, wie z.B. der pCellFree_G03 Vektor, der eine N-terminale eGFP-Fusion zu dem gewünschten Protein von Interesse unter Verwendung von Gateway-Klonierungsstellen kodiert.
LTE hat sich in einer Vielzahl von Anwendungen bewährt, die eine Proteinexpression erfordern, darunter die Analyse der Proteinselbstorganisation10,16, die Herstellung von humanen integralen Membranproteinen17, die Erforschung antiviraler Wirkstoffkandidaten18, die Entwicklung biotechnologisch nützlicher Enzyme19, das Prototyping von Protein-Biosensoren20,21 und die Untersuchung von Biologika aus Hakenwürmern22. LTE war auch maßgeblich an der Kartierung von Protein-Protein-Interaktionsnetzwerken in den Bereichen Virologie und zelluläre Strukturen beteiligt21,32. LTE wurde so bewertet, dass es bei der Expression von monodispersen und nicht aggregierten Proteinen in voller Länge eine ähnliche Leistung wie andere eukaryotische zellfreie Systemeaufweist 33 und gleichzeitig eine kostengünstigere und skalierbarere Produktion bietet.
Dieses Protokoll bietet Techniken zur Kultivierung und Störung des Wirtsorganismus, zur Herstellung von Lysat und zur Ergänzung einer Fütterungslösung (FS) für die gekoppelte Transkriptions-/Translationsproteinexpression. Darüber hinaus enthält es ein Protokoll zur Optimierung von Produktionschargen. In der ersten Version des zellfreien Systems von Leishmania wurde eine unerwünschte Variation der Expressionsniveaus, des Anteils von Proteinen in voller Länge und des Vorhandenseins von Proteinaggregaten von Charge zu Charge beobachtet, was zur Entsorgung der Chargenführte 34. Spätere Protokollverbesserungen wurden vorgenommen, um dieses Problem zu beheben25. Das aktuelle Protokoll baut auf diesen Verbesserungen auf und ermöglicht es, einzelne Chargen für die maximale Proteinexpression und -größe zu optimieren. Dies wird erreicht, indem die Beladung der Zelldisruptoren (gemessen als optische Dichte bei 600 nm; OD600nm) und Normalisierung des resultierenden Lysatausstoßes durch Extinktion bei 280 nm (Abs280nm). Darüber hinaus umfasst es ein Verfahren zur teilweisen Ergänzung des Lysats mit rNTP und Magnesium während der Herstellung, mit anschließender Optimierung dieser Zuführlösungskomponenten während der Testexpression. Obwohl diese Optimierung als Option im Protokoll vorgestellt wird, wird sie von den Autoren dringend empfohlen.
Protokoll
Dieses Protokoll enthält detaillierte Medienrezepte und -schritte, die das Kultivieren, Zentrifugieren, Messen der GFP-Fluoreszenz mit einem Multimode-Plattenleser, Messen des Kultur-OD600 nm und Beurteilen des Lysats Abs280 nm umfassen. Es behandelt auch den Aufbau und die Bildgebung von SDS-PAGE-Proteingelen. Die für dieses Protokoll erforderlichen oder vorgeschlagenen Materialien sind in der Materialtabelle aufgeführt. Es ist wichtig zu beachten, dass typische Laborressourcen wie Medienkomponenten, Zentrifugen, Röhrchen, Spektralphotometer und Gelelektrophorese-Setups wahrscheinlich austauschbar verwendet werden können, sofern nicht anders angegeben. Abbildung 1 zeigt eine Zusammenfassung des LTE-Herstellungsprozesses.
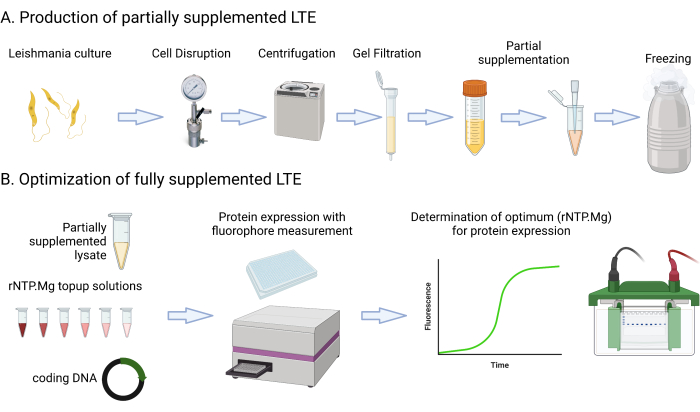
Abbildung 1: Überblick über das LTE-Herstellungsprotokoll. Dieser Cartoon bietet eine kurze Zusammenfassung des LTE-Herstellungsprotokolls. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
1. Wachstum von Leishmania tarentolae-Kulturen
- Bereiten Sie mindestens 3 l TBGG-Wachstumsmedium vor (Bactotrypton 12 g/L, Hefeextrakt 24 g/L, Glycerin 8 mL/L, Glukose 1 g/L, KH2PO4 2,3 g/L, K2HPO4 2,5 g/L, siehe Materialtabelle). Sterilisieren Sie das Medium mit einem 0,22-μm-Filter unter Vakuum oder einem ähnlichen Aufbau.
- Lagern Sie das Medium bei Raumtemperatur (RT), wobei die letzten Zusätze (Hämin, Antibiotika) kurz vor der Inokulation mit L. tarentolae hinzugefügt werden. Hemin (0,25 % v/v in 50 % Triethanolamin) wird mit 0,2 % v/v zugesetzt, Penicillin (10.000 Einheiten/ml) plus Streptomycin (10.000 μg/ml) mischen sich mit 0,5 % v/v.
HINWEIS: Der Ausgangspunkt dieses Protokolls ist eine gepflegte 2 x 10 ml-Kultur des Wildtyps L. tarentolae. Erhaltungskulturen werden bei 27 °C in Standard-50-ml-Gewebekulturflaschen mit geringem Schütteln (75 U/min) gezüchtet. Solche 10-ml-Kulturen können auf unbestimmte Zeit mit ~1/20 Verdünnungen in sterilen (TBGG + Hemin, Penicillin, Streptomycin) alle 2-3 Tage aufbewahrt werden. Eine Standard-Biosicherheitswerkbank in einem PC2-Labor wird empfohlen; bakterielle Kontaminationen werden jedoch tendenziell durch die zugesetzten Antibiotika verhindert, während Pilzkontaminationen in der Regel von L. tarentolae überwachsen werden. - Erweitern Sie die Erhaltungskulturen von L. tarentolae über einen Zeitraum von zwei Tagen auf 200 ml (Tag 1) und dann auf 2 l (Tag 2) in 1:10-Verdünnungen mit einem täglich zunehmenden Volumen an TBGG + Hämin/Antibiotika. Beide Verdünnungen werden in autoklaviersterilisierten, 5-Liter-Glaskolben mit Prallen (gefüllt bis maximal 1 l) durchgeführt. Die zweite Verdünnung muss am Nachmittag zwischen 15 und 18 Uhr erfolgen, mit der Absicht, die Lysatproduktion am nächsten Tag zwischen 8 und 11 Uhr zu starten.
HINWEIS: Dieses Protokoll verwendet das minimale Startvolumen für die LTE-Produktion (2 x 1 l Kulturen). Es ist auch möglich, die Kultur für die LTE-Produktion auf bis zu 10 L zu erweitern, indem ein zusätzlicher Expansionsschritt eingebaut wird (z. B. Tag 1: 100 mL; Tag 2: 1 Mittagessen; Tag 3: 10 L). Obwohl bei diesem Protokoll Prallkolben (siehe Materialtabelle) verwendet werden, um L. tarentolae zu züchten, können optional konventionelle Bioreaktoren verwendet werden, die für das Bakterienwachstum mit Rushton-Laufrädern ausgelegt sind, vorausgesetzt, die Rührgeschwindigkeit wird unter 100 U/min gehalten. Eine verbesserte Belüftung und pH-Kontrolle in Bioreaktoren verlängert im Allgemeinen das Wachstum der L. tarentolae-Kulturen in logarithmischer Phase, so dass ein höherer Ernte-ODvon 600 nm von 10 in Schritt 1.4 verwendet werden kann. - Erfassen Sie den OD600nm der Kultur in dreifacher Ausfertigung über eine 1:10-Verdünnung in TBGG direkt in der Spektralphotometer-Küvette. Ein geeigneter Startbereich für die Herstellung von Lysat ist OD600 nm = 4,0-8,0.
- Bieten Sie zusätzliche Inkubationszeit, wenn OD600 nm < 4.0. Eine Kultur mit OD600nm > 8 ist verwendbar und führt zu einem größeren Volumen an zellfreiem Expressionslysat, jedoch mit geringerer Qualität aufgrund des Beginns der späten logarithmischen Wachstumsphase. Legen Sie Kulturflaschen auf Eis und warten Sie auf die nachfolgenden Schritte.
HINWEIS: Eine genaue Messung des Endkultur-OD600 nm ist von entscheidender Bedeutung, da er zur Berechnung des endgültigen Volumens für konzentrierte Zellen vor dem Aufschluss verwendet wird. Diese Berechnung ersetzt eine Pellet-Wiegemethode, die in früheren Versionen der LTE-Herstellung zur Kalibrierung der Zellkonzentration vor der Unterbrechung34 verwendet wurde, um das Protokoll zu vereinfachen. Stellen Sie sicher, dass die TBGG für die OD-Messungvon 600 nm verdünnt wird, da sonst der osmotische Schock die Zellform verändert und zu Messfehlern führt. Mischen Sie die 1:10-Verdünnung für die OD-Messung von600 nm (direkt in der Küvette) Sekunden vor der spektrophotometrischen Ablesung, da sich die L . tarentolae-Zellen schnell absetzen und ein charakteristisches trübes Aussehen aufweisen. Wenn das endgültige Volumen der Expressionskultur als ungefähr angesehen wird, wird auch empfohlen, die Kolben bei der Ernte zu wiegen (mit geeignetem Tara des leeren Kolbens), um ein besseres geschätztes Volumen (bei 1 g = 1 ml) zu erhalten. Der maximal mögliche ODvon 600 nm aus dem Wachstum von L. tarentolae in Kolben mit Blende beträgt 15-20, obwohl dies für die Lysatherstellung ungeeignet ist, da die stationäre Phase erreicht wird.
2. Konzentration von L. tarentolae-Kulturen
- Die Leishmaniazellen müssen vor dem Aufschluss etwa 60x gewaschen und konzentriert werden. Berechnen Sie das Zielvolumen für die Zellkonzentration basierend auf OD600 nm = 300 für das Endkonzentrat. Die Gleichung lautet V = Erntevolumen (mL) x (Ernte OD600nm/300). Bei Verwendung einer 2-Liter-Kultur mit einem Ernte-OD600 = 5 beträgt das Zielvolumen beispielsweise 33 ml.
HINWEIS: DasOD-Ziel von 600 nm von 300 nm kann geändert werden. In der bisherigen LTE-Produktion wurden Werte im Bereich von 150-350 verwendet. Höhere Konzentrationen von Zellen, die in die Disruption geraten, führen tendenziell zu endgültigen zellfreien Expressionsreaktionen mit höheren Proteinausbeuten, aber mit einer erhöhten Tendenz für anfällige Proteine, sich zu aggregieren. OD600nm = 300 stellt ein geeignetes Standardziel für die LTE-Produktion dar. - Die geernteten Kulturen in geeignete Zentrifugenflaschen umfüllen und bei 2500 x g für 10 min bei 4 °C schleudern. Dekantieren Sie den Überstand vorsichtig in die Kulturabfälle.
HINWEIS: Es ist wichtig, den Verlust von Zellen in den verworfenen Überstand zu minimieren, da dies die Berechnung der Störungsbelastung beeinflusst. In früheren LTE-Produktionsprotokollen wurde die Konzentration von L. tarentolae-Zellen für den Aufschluss kalibriert, indem das Zellkonzentrat in einem Test-Mikrozentrifugenröhrchen heruntergeschleudert und das Pelletgewicht im Vergleich zum Gesamtgewichtgemessen wurde 34. Dieses vereinfachte Protokoll verwendet stattdessen ein theoretischesOD-Ziel von 600 nm für das Konzentrat, das auf dem gemessenen Harvest-ODvon 600 nm basiert, und geht von einem geringen Zellverlust während der Zellkonzentration und des Waschens aus. - Waschen Sie das Zellpellet dreimal in SEB-Puffer (45 mM HEPES-KOH pH 7,6, 250 mM Saccharose, 100 mM KOAc, 3 mM Mg(OAc)2, auf Eis gehalten) und zentrifugieren Sie jedes Mal bei 2500 x g für 10 min bei 4 °C. Für die erste Wäsche resuspendieren Sie jeweils 1 l der pelletierten Kultur in 100 ml SEB-Puffer und kombinieren Sie sie dann in einem einzigen Zentrifugenkolben. Verwenden Sie für die zweite Wäsche ebenfalls 100 ml SEB pro 1 l der ursprünglichen Kultur.
HINWEIS: Für die endgültige Pellet-Resuspension wird SEB-Puffer auf 50 % des endgültigen Ziel-Resuspensionsvolumens zugegeben (Schritt 2.1). Auf diese Weise kann das gepoolte Konzentrat in Schritt 2.4 vorsichtig auf genau das endgültige Zielvolumen aufgefüllt werden. Jede Resuspension muss so schonend wie möglich sein, um eine vorzeitige Lyse von L. tarentolae zu vermeiden, z. B. durch sanftes Schwenken des zugegebenen SEB um das dekantierte Pellet oder durch Pipettieren von SEB über das Pellet, das an der Wand des Zentrifugenröhrchens haftet. Für den letzten Schritt kann es bequemer sein, Überstände in kleinere Zentrifugenröhrchen zu übertragen. - Gießen Sie das resuspendierte Konzentrat in einen geeigneten Volumenzylinder aus gewaschenem Glas, füllen Sie dann das Volumen mit zusätzlichem kaltem SEB auf das Zielvolumen auf (Schritt 2.1) auf und mischen Sie vorsichtig.
3. Lyse von L. tarentolae-Konzentrat
- Das Zellkonzentrat wird in die auf 4 °C vorgekühlte Stickstoffkavitationsvorrichtung (siehe Materialtabelle) überführt, auf 70 bar Stickstoff unter Druck gesetzt und 45 Minuten lang auf Eis inkubiert.
HINWEIS: Obwohl Stickstoffkavitationsdisruptoren keine gängigen Laborartikel sind, werden sie für die LTE-Produktion empfohlen. Alternative Methoden wie zelluläre Gefrier-Auftau- und French-Press-Disruptoren wurden ausprobiert; Die Proteinexpressionsaktivität betrug jedoch <50% im Vergleich zur Verwendung der Stickstoffkavitationsmethode. Die Stickstoffkavitationsvorrichtung muss vor dem Gebrauch und zwischen den Durchläufen gründlich gereinigt werden, ähnlich wie alle wiederverwendeten Gefäße, die ab diesem Schritt mit dem Zelllysat in Berührung kommen (z. B. der Auffangkolben). Ein geeignetes Reinigungsprogramm besteht aus dem Waschen mit Laborreinigern und dem anschließenden gründlichen Spülen mit entionisiertem Wasser. - Öffnen Sie die Entlüftung an der Stickstoffkavitationsvorrichtung und stoßen Sie das resultierende Lysat in einen entsprechend robusten Behälter aus, z. B. einen Vakuum-Auffangkolben auf Eis. Kippen Sie den Auffangkolben, um sicherzustellen, dass sich das gesamte resultierende Lysat absetzt und in ein frisches Zentrifugenröhrchen oder ein ähnliches Gefäß pipettiert werden kann.
VORSICHT: Stickstoffkavitationsdisruptoren beruhen auf dem abrupten Übergang des Zellkonzentrats von 70 bar Stickstoff zu Umgebungsdruck, der durch einen starken Fluss von zuerst Flüssigkeit und dann Stickstoff durch das Auslassventil des Geräts erreicht wird. Die Entlüftung muss mit geeigneter persönlicher Schutzausrüstung (PSA) in einer Chemikalienschutzhaube erfolgen. Es besteht die Gefahr, dass das Zielgefäß zerbricht und das Lysat verloren geht, weshalb wir anstelle eines generischen Kolbens einen robusten Vakuumabscheider verwenden. Wenn es sich bei dem Auslassventil des Geräts um einen Schlauch handelt, vermeiden Sie es, den Schlauch direkt in den Empfänger zu stecken, um einen übermäßigen Druckaufbau an der Entlüftungsstelle zu vermeiden.
4. Zentrifugation von Zelllysat
- Das Lysat wird in Zentrifugenröhrchen mit geeigneter g-Kraft überführt und 15 Minuten lang bei 4 °C bei 10.000 x g zentrifugiert. Entfernen Sie den Überstand zu frischen, ähnlichen Zentrifugenröhrchen.
- Das Lysat wird 15 Minuten lang bei 4 °C bei 30.000 x g zentrifugiert und dann der endgültige Überstand in ein frisches Zentrifugenröhrchen oder einen ähnlichen Behälter auf Eis entfernt. Schätzen Sie das Gesamtvolumen.
5. Gelfiltration von Zelllysat
HINWEIS: Die Gelfiltration wird verwendet, um die im SEB-Puffer enthaltene Saccharose zu entfernen. Während Saccharose bei der Stabilisierung der zellulären Maschinerie während des Zellaufschlusses hilft, verringert sie die Ausbeute, wenn sie in Proteinexpressionsreaktionen zurückgehalten wird.
- Stellen Sie eine ausreichende Anzahl von PD-10-Schwerkraft-Gelfiltrationssäulen (siehe Materialtabelle) in einem Rackformat auf, das es ihnen ermöglicht, in eine Auffangwanne oder einen ähnlichen Behälter darunter zu tropfen, um sicherzustellen, dass sie das gesamte Lysatvolumen mit 2,5 mL pro Säule filtern können. Die Säulen werden voräquilibriert, indem zuvor 10 ml 4 ΰC EB-Puffer (45 mM HEPES-KOH PH 7,6, 100 mM KOAc, 3 mM Mg(OAc)2) durch sie geleitet werden.
HINWEIS: Alle Schritte ab diesem Punkt profitieren davon, dass sie in einem Kühlraum mit einer Temperatur von 4 °C durchgeführt werden. Es ist jedoch auch geeignet, alle Lysate und Reagenzien auf einer Tisch-Eisschale aufzubewahren. Eine Ausnahme bildet der Schritt der Gelfiltration, bei dem die Autoren ein Gestell mit Säulen in einen 4 °C heißen Kühlschrank stellen, während sie repuffern. In den ursprünglichen Versionen dieses Protokolls wurden neue Gelfiltrationssäulen "blockiert", indem das Lysat zunächst gepuffert und der erste Ausgang verworfen wurde. Obwohl dies nicht für notwendig erachtet wird, müssen die Säulen mit EB-Puffer gespült und bei 4 °C zwischen den Lysatchargen gelagert werden. Das erste Ausgangslysat kann eine geringere Proteinexpressionsaktivität aufweisen als nachfolgende Ausgaben, da eine gewisse Hintergrundretention von Lysatkomponenten auf der neuen Säule vorhanden ist. - Geben Sie 2,5 ml Lysat in jede Säule und warten Sie, bis es in die Säule übergeht. Fügen Sie weitere 0,5 ml EB hinzu, um das Lysat in der Säule abzusetzen, während das Eluat verworfen wird.
- Eluieren Sie das gelgefilterte Lysat, indem Sie jeder Säule weitere 2,5 ml EB hinzufügen und den Ausgang sammeln, indem Sie ein frisches, sauberes Tablett oder einen anderen Behälter unter die Säulen stellen.
6. Supplementierung von Zelllysat
- Verwenden Sie das Nanotropfen-Spektralphotometer (siehe Materialtabelle), um Abs280 nm des gelfiltrierten Lysats zu messen. Wenn er 60 überschreitet, verdünnen Sie ihn mit zusätzlichem EB-Puffer von 4 °C auf Abs280 nm = 60.
HINWEIS: Während die Kontrolle der Zelldichteeingabe in die Disruption mit OD600 nm die Lysatausgangsstärke ungefähr bestimmt, verbessert die Normalisierung von Abs280 nm nach der Lysataufschluss und -verarbeitung die Chargenkonsistenz der Lysatleistung weiter. Lysat Abs280nm kann nach oben und unten angepasst werden, was sich auf die Proteinexpressionsausbeute und -aggregation auswirkt (siehe Abschnitt Diskussion). Wenn das unsupplementierte Lysat einen Abs280 nm < 60 anzeigt, kann es erforderlich sein, mehr Leishmania-Biomasse in den Disruptionsschritt einzubeziehen, d. h. die Zelldisruptorbeladung in Schritt 2.1 auf OD600 nm > 300 zu erhöhen. - Geben Sie 5x Fütterungslösung (5x FS, Tabelle 1) im Verhältnis 2:5 zum Lysat und mischen Sie es gründlich durch. Aliquotieren Sie es in geeignete Behälter (z. B. 1,5-ml-Mikrofugenröhrchen) und frieren Sie es in flüssigem Stickstoff ein. Wenn Sie die optionalen Schritte 7.1 bis 7.3 für die Optimierung der LTE-Expression unten befolgen, verwenden Sie den reduzierten rNTP.Mg 5x FS aus Tabelle 1 anstelle des Standardwerts von 5x FS. Fügen Sie 5 x 100 μl Aliquots zum Einfrieren für die Verwendung in den Optimierungsexperimenten hinzu.
HINWEIS: Das Einfrieren mit dem Standardwert von 5x FS in einem Verhältnis von 2:5 erzeugt expressionsbereites, ergänztes LTE, das bei einer Expression von 7 μL/10 μL verwendet wird (daher wird das 5x FS in der Endreaktion zu 1x FS). Die Autoren empfehlen jedoch, die weiteren optionalen Schritte zu befolgen, bei denen das 0,6-fache der Standardmenge an rNTPs und Magnesium im 5x FS bereitgestellt wird. Darauf folgt ein Optimierungsschritt, bei dem eine äquimolare Mischung aus beiden (als rNTP.Mg bezeichnet) hinzugefügt wird, um die Testreaktionen auf einen optimierten Wert aufzufüllen. Das partielle 5x FS enthält auch ein Oligonukleotid, das die endogene mRNA-Expression abschaltet (siehe Abschnitt Einleitung). Die Sequenz des Oligonukleotids ist CAATAAAGTACAGAAACTGATACTTATATAGCGTT.
7. QC und Optimierung des abschließend ergänzten LTE
HINWEIS: Die minimal notwendigen Schritte zur Bestimmung der geeigneten "Auffrischungs"-Zugabe von rNTP.Mg zu dem reduzierten rNTP und dem mit Magnesium ergänzten Lysat umfassen die Expression von eGFP oder eines ähnlichen Fluorophors (z. B. sfGFP) ohne Fusionspartner. Steigende Konzentrationen von rNTP.Mg werden den Reaktionen zugesetzt, um den Punkt zu bestimmen, an dem das Expressionsniveau (gemessen als eGFP RFU über einen Multimode-Plattenleser) optimiert wird. Vorzeitige Abbrüche von eGFP, die nicht fluoreszierend sind, werden durch eine Abnahme der eGFP RFU bei zu hohen rNTP.Mg Konzentrationen deutlich. Kurzproduktstörungen von LTE treten jedoch häufiger in größeren exprimierten Proteinen (>50 kDa) auf. Daher ist es möglich, diese Optimierung mit einem größeren Template als eGFP durchzuführen, insbesondere wenn eines in einem geeigneten Expressionsvektor verfügbar ist, der eine Fluorophorfusion liefert, die von LTE für eine bestimmte Anwendung oder Studie erzeugt werden soll (siehe Abschnitt "Repräsentative Ergebnisse").
- Tauen Sie ein 100-μl-Aliquot auf und richten Sie sechs 10-μl-Expressionsreaktionen ein, die jeweils aus 7 μl teilweise supplementiertem Lysat aus Schritt 6.2, 1 μl Auffrischungslösung gemäß Tabelle 2 und 2 μl Reinstwasser bestehen, das eine ausreichende DNA-Kontrollvorlage enthält, um eine Endkonzentration von 50 ng/μl in der Reaktion zu erreichen.
- Inkubieren Sie die Reaktionen 2 Stunden lang bei 25 °C und überwachen Sie den Anstieg der GFP-Fluoreszenz mit einem Multimode-Plattenreader.
HINWEIS: Geeignete Konfigurationswerte für GFP sind Anregung bei 485 nm (Bandbreite 5 nm), Emission bei 516 nm (Bandbreite 5 nm), mit einem Leseintervall von 1 min für 2 h. - Ordnen Sie die endgültigen Expressionswerte, um die rNTP.Mg Konzentration zu bestimmen, die der höchsten eGFP-RFU entspricht. Wenn kinetische Daten verfügbar sind, wird ein Überschuss von rNTP.Mg auch durch einen biphasischen Anstieg der eGFP RFU während der 2-stündigen Expressionsperiode angezeigt (siehe Abschnitt Repräsentative Ergebnisse).
- Sobald die optimierte Auffüll- rNTP.Mg Konzentration bestimmt ist, fügen Sie sie zu allen weiteren Proteinexpressionen hinzu, indem Sie die LTE-Charge verwenden, die in den vorherigen Schritten teilweise ergänzt wurde.
HINWEIS: Wenn die Aliquotierung in Schritt 6.2 vorsichtig mit festen Volumina durchgeführt wird, kann die Auffrischung nachträglich zu jedem Aliquot hinzugefügt werden, ohne aufzutauen, z. B. indem die Aliquote auf Trockeneis gelegt werden. Diese Aliquots sind nun vollständig ergänzt, da die richtige rNTP.Mg Auffüllung durch jedes Aliquot gemischt wird, wenn sie aufgetaut und für die Verwendung gemischt werden.
Ergebnisse
Der Zweck der zellfreien Proteinexpression besteht darin, Proteine in voller Länge in einer gefalteten, aktiven Form herzustellen, die für eine Vielzahl von Anwendungen geeignet sind. LTE (Leishmania tarentolae-Extrakt ) wurde zuvor mit anderen prokaryotischen und eukaryotischen zellfreien Expressionssystemen verglichen und zeigt eine hohe Fähigkeit, Trunkierung und Aggregation bei optimalem Betrieb zu vermeiden, insbesondere im Vergleich zu zellfreier Expression auf E . coli-Basis 33. Dies ging jedoch zuvor mit erheblichen Unterschieden in der Ausgabequalität von Charge zu Charge einher. Das derzeitige Verfahren enthält weitere Verbesserungen, um eine gleichbleibende Ausgabequalität zu gewährleisten, hauptsächlich durch eine teilweise Supplementierung der notwendigen Fütterungslösung vor dem anfänglichen Einfrieren des LTE in Aliquoten. Es folgt die Optimierung des transkriptionellen Inputs rNTP.Mg in einer Auffülllösung, die zu jeder nachfolgenden Reaktion hinzugefügt oder zur direkten Vervollständigung der gefrorenen Aliquoten verwendet werden kann. Bemerkenswert ist, dass die Optimierungsreaktionen auch eine typische Verwendung von LTE zur praktischen Expression von Proteinen darstellen, wobei die Reaktionen 2 h lang bei 25 °C durchgeführt wurden.
Daten zur Optimierung der Konzentration von rNTP.Mg in zellfreien Reaktionen liefern einen repräsentativen Datensatz. Die Expressionsniveaus nehmen in der Regel mit zunehmender transkriptioneller Eingabe (rNTP.Mg) zu, was auf eine erfolgreiche Expression hinweist. Es wird jedoch eine Schwelle erreicht, bei der das System zu einer nicht-produktiven Expression von verkürzten Produkten neigt, insbesondere im Fall von größeren Proteinen (>50 kDa). Diese suboptimale Expression führt mit zunehmender rNTP.Mg zu einem Verlust des Fluoreszenzsignals, was sich besonders bei C-terminalen Fluorophorfusionen zeigt, bei denen die Translation des Polypeptids das Fluorophor selbst nicht erreicht. Bei N-terminalen Fusionen tritt zwar nicht unbedingt eine Verringerung der Gesamt-RFU (Relative Fluorescence Units) bei überschüssigem rNTP.Mg auf, während eine fehlgeschlagene Expression auf SDS-PAGE-Gelen als mehrere fluoreszierende Produkte mit abnehmender Größe sichtbar ist. Dieser Ansatz nutzt die Fähigkeit von GFP (Green Fluorescent Protein), die Fluoreszenz auch dann aufrechtzuerhalten, wenn es auf einem herkömmlichen SDS-PAGE-Gel sichtbar gemacht wird, vorausgesetzt, die Proben werden nach dem Mischen nicht erhitzt. Stattdessen werden sie mit Gel-Ladepuffer gemischt und direkt auf das Gel geladen. Während SDS-PAGE-Gelmaterialien und -geräte in der Regel austauschbar sind, muss der Gel-Imager in der Lage sein, die GFP-Fluoreszenz sichtbar zu machen. Eine typische Konfiguration für die GFP-Visualisierung wird mit Anregung bei 485 nm (Bandbreite 5 nm), Emission bei 516 nm (Bandbreite 5 nm) und einem Leseintervall von 1 Minute über 2 h bereitgestellt.
Eine Optimierung des Systems allein durch die Expression von eGFP ist möglich. Abbildung 2A,B (Einschub) zeigt typische Expressionsausgaben von Optimierungsreaktionen für zwei LTE-Chargen, wobei die eGFP RFU mit steigender rNTP.Mg Konzentrationen zunimmt und ein optimales Niveau von +0,6x rNTP.Mg (Abbildung 2A) und +0,3x rNTP.Mg (Abbildung 2B) für maximale RFU erreicht. Die reduzierte rNTP-Feed-Lösung enthält das 0,6-fache rNTP.Mg, was zu insgesamt rNTP.Mg Niveaus führt, die das 1,2-fache und das 0,9-fache des Standardbetrags für diese LTE-Chargen betragen. Abbildung 2C zeigt die Kinetik des RFU-Anstiegs während der Reaktion für die LTE-Charge in Abbildung 2B und zeigt eine biphasische Reaktion mit zwei diskreten Phasen über die Dauer der Reaktion.

Abbildung 2: Optimierung der rNTP.Mg Aufsättigung in LTE mit reduziertem rNTP 5x FS. (A) Expressionsniveaus von eGFP nach 140 min Expression aus einem Kontrollplasmid bei unterschiedlichen rNTP.Mg Auffrischungsniveaus in der zellfreien Expressionsreaktion (n = 3, Mittelwert ± SD aufgetragen). (B) Optimierung einer anderen LTE-Produktionscharge, die eine reduzierte Expression über einen bestimmten rNTP.Mg-Schwellenwert hinaus zeigt. (C) Kinetik der eGFP-Akkumulation in den gleichen Reaktionen wie (B), mit zunehmender rNTP.Mg Auffüllung. Diese Daten repräsentieren auch die typische Kinetik der Proteinakkumulation in der LTE-Batch-Expression. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Es sollte jedoch beachtet werden, dass eGFP als kleines Protein (27 kDa), das leicht zu falten ist, wahrscheinlich exprimiert, gefaltet und gereift wird, unabhängig vom verwendeten zellfreien Expressionssystem. Ein Versagen des Systems ist wahrscheinlicher, wenn größere Proteine von Interesse exprimiert werden, wobei verkürzte Produkte bei Eingangsproteingrößen von mehr als 70 kDa deutlicher werden33. Daher ist die Optimierung des Systems mit den Proteinen, die für die tatsächliche Verwendung vorgesehen sind, überlegen, wobei eGFP immer noch zur Quantifizierung vorhanden ist, aber als N-terminale Fusion mit dem interessierenden Protein.
Abbildung 3 zeigt eine typische Optimierung des rNTP.Mg Auffüllgrads bei Verwendung eines größeren Protein-Templates, das anfällig für die Abgabe von verkürzten Produkten ist (eGFP-Sox18). Unter Verwendung eines semi-nativen Gel-SDS-PAGE-Formats (d.h. ohne Erhitzen von Proben) ist es möglich, das fortschreitende Versagen der Expression zu visualisieren. Eine optimale rNTP.Mg Zugabe bei +0,1x (kombiniert mit der 0,6x rNTP.Mg in der Teilfütterungslösung, insgesamt 0,7x) reduziert deutlich den Anteil der Proteinbande in voller Länge als Teil der gesamten fluoreszierenden Expressionsprodukte, was auf einen Systemausfall mit übermäßiger rNTP.Mg Zugabe hinweist.
Wie im Protokoll erwähnt, ist es möglich, den rNTP.Mg Optimierungsschritt zu überspringen und die volle Menge an rNTP.Mg direkt in der "Standard"-Fütterungslösung während der Supplementierung unmittelbar nach der Gelfiltration in Schritt 6.2 hinzuzufügen. Auf diese Weise kehrt das Protokoll im Wesentlichen zu den ursprünglich veröffentlichten Methoden zur Erstellung von LTE34 zurück. Die Autoren glauben jedoch, dass die Anpassung des Systems an eine optimale Leistung, wie in Abbildung 3 (Lane D bis Lane E) gezeigt, die zusätzliche Komplexität des Protokolls überwiegt und den Wert von LTE als Proteinexpressionswerkzeug erhöht.
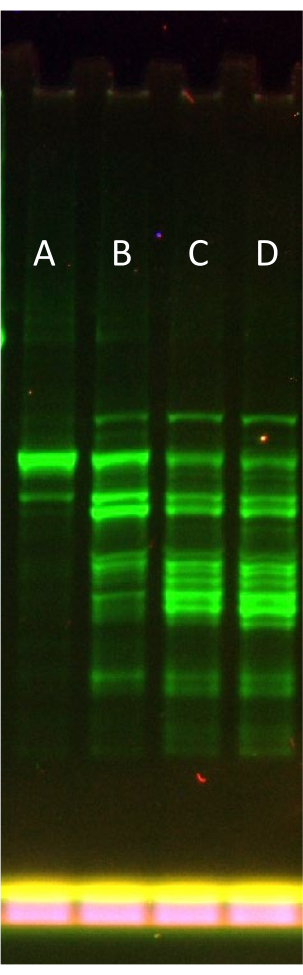
Abbildung 3: Effekt einer zunehmenden rNTP.Mg-Auffüllung auf die eGFP-Sox18-Expression in teilweise supplementiertem LTE. Semi-natives SDS-PAGE-Gel, das die Auswirkungen einer Erhöhung der rNTP.Mg-Auffüllung auf die eGFP-Sox18-Expression darstellt. Bahn A: +0,1x (rNTP.Mg) Aufladung, Bahn B: +0,2x (rNTP.Mg), Bahn C: +0,3x (rNTP.Mg), Bahn D: +0,4x (rNTP.Mg). Die N-terminale eGFP-Fusion wird durch Fluoreszenzabtastung des Gels sichtbar. Das primäre Band in Bahn A stellt eGFP-Sox18 in voller Länge dar. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
| Bestandteil | Bestandskonzentration | 5x Futterlösung | μL Stamm/ml 5x Fütterungslösung |
| Ausfall (reduzierter rNTP) | Ausfall (reduzierter rNTP) | ||
| Spermidin | ca. 100 mM | ca. 1,25 Mio. | 12 |
| DVB-T | 500 mM | ca. 10 mM | 20 |
| Kreatinphosphat | ca. 1000 mM | ca. 200 mM | 200 |
| HEPES-KOH pH7,6 | ca. 2500 mM | ca. 100 mM | 40 |
| PEG3000 | 0,5 V/V | 0,05 V/V | 100 |
| Protease-Inhibitor-Cocktail | 120-mal | 5-fach | 43 |
| Aminosäuren | 3,6 mM (Stück) | 0,68 mM (Stück) | 190 |
| ATP | ca. 100 mM | 8,5 (5,1) mM | 85 (51) |
| Zum GTP | ca. 100 mM | 3,2 (1,9) mM | 32 (19) |
| UTP | ca. 100 mM | 2,5 (1,5) mM | 25 (15) |
| CTP | ca. 100 mM | 2,5 (1,5) mM | 25 (15) |
| Mg(OAc)2 | 1 Mio. | 16,7 (10) mM | 16.7 (10) |
| Anti-Spleiß-Leader-Oligo | 1 Mio. | 0,05 Mio. | 50 |
| T7-RNA-Polymerase | 5 mg/ml | 0,5 mg/ml | 100 |
| Kreatin-Phosphokinase | 5 Einheiten/μL | 0,2 Einheiten/μl | 42 |
| Reinstwasser | 19 (93) |
Tabelle 1: Zusammensetzung der 5x Futterlösung (5x FS) für LTE. 1 ml 5x FS ist für jeweils 2,5 ml unsupplementiertes Lysat nach der Gelfiltration erforderlich. Durch die Ergänzung mit dem Standardwert von 5x FS entsteht ein expressionsfähiges LTE für den Einsatz in Expressionsreaktionen bei einem Verhältnis von 7 μL/10 μL. Das Rezept mit reduziertem rNTP.Mg (Mengen in Kursivschrift) wird für die LTE-Expressionsoptimierung empfohlen und enthält das 0,6-fache der Standardwerte von rNTPs und Magnesium. Diese können im anschließenden Optimierungsexperiment mit den in Tabelle 2 skizzierten Ergänzungen auf variable Werte (0,6 bis 1,1 mal) angepasst werden.
| rNTP-Aufladung | ATP (100 mM) | GTP (100 mM) | UTP (100 mM) | CTP (100 mM) | MgOAc (1 Mio.) | Reinstwasser |
| (1 μL/10 μL rxn) | μL/200 μL | μL/200 μL | μL/200 μL | μL/200 μL | μL/200 μL | |
| +0x | 0 | 0 | 0 | 0 | 0 | 200 |
| +0,1-mal | 3.4 | 1.3 | 1 | 1 | 0.7 | 193 |
| +0,2-mal | 6.8 | 2.5 | 2 | 2 | 1.3 | 185 |
| +0,3-mal | 10.2 | 3.8 | 3 | 3 | 2 | 178 |
| +0,4-mal | 13.6 | 5.1 | 4 | 4 | 2.7 | 171 |
| +0,5-mal | 17 | 6.4 | 5 | 5 | 3.3 | 163 |
Tabelle 2: Zusammensetzung der (rNTP.Mg) Aufladelösungen für die LTE-Optimierung. Diese Lösungen werden zur Optimierung der LTE verwendet, indem 1 μl Auffülllösung pro 10 μl Proteinexpressionsreaktion hinzugefügt werden. Sobald ein Auffüllgrad im Optimierungsexperiment bestimmt wurde, kann er konsistent zu allen nachfolgenden Proteinexpressionsreaktionen mit denselben LTE-Batch-Aliquoten hinzugefügt werden. Alternativ kann es direkt zu den Aliquoten selbst mit 1 μl Zugabe pro 7 μl (ohne Auftauen) hinzugefügt werden. Nach dem Auftauen und Mischen werden diese Lysate mit 8 μl LTE pro 10 μl Proteinexpression verwendet, wobei das während der Optimierung festgelegte rNTP.Mg Auffüllniveau beibehalten wird.
Diskussion
Protokolle zur Schaffung von LTE wurden in den letzten zehn Jahren veröffentlicht7 und regelmäßig aktualisiert25,34. Neueinsteiger in die Technik stoßen jedoch oft auf eine steile Lernkurve, was zu Verzögerungen beim Erreichen einer qualitativ hochwertigen und ertragreichen Proteinexpression führt. Ähnliche Herausforderungen wurden von anderen Forschungsgruppen, die mit LTE35 arbeiten, berichtet, insbesondere in Bezug auf signifikante Variationen von Charge zu Charge. Das videobasierte Protokollformat kann möglicherweise zusätzliches, weniger offensichtliches Einrichtungswissen liefern, das potenziellen Benutzern zugute kommt34. Es wurden Änderungen am Protokoll vorgenommen, die darauf abzielen, die Erfolgswahrscheinlichkeit zu erhöhen, das Verfahren zu vereinfachen, den Zeitaufwand zu reduzieren und Fehler im Zusammenhang mit der Komplexität zu minimieren.
Bei der Zellaufschluss ist die präzise Kontrolle über die Beladung der Zellen in den Stickstoffkavitationszelldisruptor von entscheidenderBedeutung 34. Dies zu erreichen, kann aufgrund der hohen Zelldichte nach der Zellkonzentration und dem Waschen eine Herausforderung darstellen. In den ursprünglichen Protokollen wurden verschiedene Methoden verwendet, wie z. B. das Abdrehen eines kleinen Volumens der endgültigen konzentrierten Kultur und die Quantifizierung des fraktionierten Zellpellets. In diesem Protokoll wird jedoch ein einfacherer Ansatz gewählt. Das Erntevolumen der Kultur und der OD600 nm werden gemessen, und diese Messungen werden verwendet, um ein Zielvolumen für das Zellkonzentrat in Millilitern zu berechnen, wobei ein gewünschter End-ODvon 600 nm von 300 nm angestrebt wird. Diese Berechnung geht davon aus, dass während des Waschens kein signifikanter Zellverlust auftritt. Besteht der Verdacht auf Zellverlust, wird ein alternatives Verfahren mit dreifachen seriellen 1/10-Verdünnungen der konzentrierten Zellen nach dem Waschen angewendet, was letztendlich zu einer 1/1000-Verdünnung führt. Auf diese Weise kann der tatsächliche Außendurchmesser des Konzentratsvon 600 nm gemessen werden, um sicherzustellen, dass es den Ziel-Außendurchmesservon 600 nm = 300 erreicht, bevor es in den Disruptor geladen wird.
Selbst bei sorgfältiger Kontrolle der Zelldisruptorbeladung kann eine signifikante Variabilität des Lysatproteingehalts nach der Disruption auftreten, wie durch den Abs280 nm des gelfiltrierten unsupplementierten Lysats34 angezeigt wird. Daher wird eine Messung von Abs280 nm vor der Einführung einer Lysat-Supplementierung durchgeführt und das Lysat verdünnt, um einen Abs280 nm = 60 zu erreichen. Da Proteinexpressionsreaktionen schließlich 0,5 v/v-Lysat enthalten, ergibt sich ein standardisiertes Reaktionslysat mit Abs280nm = 30. Eine Lysatleistung, die mit einem Wert von weniger als Abs280 nm = 30 konfiguriert ist, führt tendenziell zu langanhaltenden Reaktionen mit geringer Expression, während Werte über Abs280 nm = 30 tendenziell zu einer höheren Expression, aber einer erhöhten Tendenz zur Aggregation von Ausgangsproteinen führen.
Die Optimierung der Lysatleistung beinhaltet die Anpassung der transkriptionellen Inputs in der Fütterungslösung, die das Lysat, insbesondere rNTPs und Magnesium, in den optionalen Reaktionsschritten 7.0-7.3 ergänzt. Es ist wichtig zu beachten, dass rNTPs und Magnesium komplexe und vielfältige Rollen in einem gekoppelten Transkriptions-Translations-System wie LTE25 spielen. Es wurde jedoch gezeigt, dass LTE ein ungefähres Magnesiumexpressionsoptimum bei rNTP (mM) + 1,5 aufweist. Da das Lysat selbst 1,5 mM Mg zum endgültigen Reaktionsgemisch beiträgt, bietet dies eine einfache Möglichkeit, den rNTP-Input zu variieren und zu optimieren, ohne Mg durch Variation der äquimolaren rNTP.Mg zu co-optimieren.
Die Lysatleistung weist eine signifikante Variation auf, wenn die rNTP.Mg erhöht wird, wobei die Proteinexpression im Allgemeinen bis zu einem Schwellenwert ansteigt, bei dem sich die Optimierung umkehrt, was zu Expressionsstörungen in Form von kurzen Produkten anstelle von Proteinen in voller Länge führt25. Daher ist eine abschließende Optimierung des Systems zur Identifizierung dieser Schwelle von Vorteil. Das ursprüngliche LTE-Protokoll verwendete ein Rezept mit fester Fütterungslösung, wobei eine gewisse Mg-Optimierung vorgeschlagenwurde 34. Dieser Ansatz wurde später mit einer umfangreicheren rNTP-Optimierung modifiziert. Diese Methode erforderte jedoch ein Schock-Einfrieren des Lysats in unergänzter Form, um eine Optimierung auf einem Aliquot zu ermöglichen, was dazu führte, dass die eventuellen Lysatexpressionsniveaus tendenziell verringert wurden. Diese Verringerung wurde auf den Verlust der kryoprotektiven Eigenschaften der Fütterungslösung beim Schockgefrieren von unsupplementiertem Lysat unmittelbar nach dem Gelfiltrationsschritt zurückgeführt. Das derzeitige Protokoll schafft ein Gleichgewicht, indem es vor dem Einfrieren eine Fütterungslösung mit reduziertem rNTP.Mg enthält, die zum Zeitpunkt der Expression auf das optimierte Niveau aufgefüllt werden kann.
Es wird erwartet, dass diese Protokollverbesserungen den Nutzen des LTE-Systems für unerfahrene Benutzer erhöhen, indem sie die primären Variationsquellen abschwächen und die Konsistenz der Proteinexpressionsausgabe verbessern.
Offenlegungen
Es gibt keine konkurrierenden finanziellen Interessen.
Danksagungen
Die Autoren möchten sich bei den vielen Alexandrov-Labormitgliedern bedanken, die in den letzten 10 Jahren zur Entwicklung der LTE-Systeme beigetragen haben, insbesondere bei Sergey Mureev, der Pionierarbeit für das System geleistet und die SITS-Ribosomen-Eintrittsstelle entwickelt hat. Abbildung 1 wurde von Biorender.com erstellt und unter Lizenz reproduziert.
Materialien
| Name | Company | Catalog Number | Comments |
| PD-10 SuperDex 25 Columns | Cytiva | 17085101 | Gel filtration columns |
| Nitrogen Cavitation cell disrupter | Parr Industries | 4635 or 4639 | Cell Disrupter |
| Bovine derived Hemin | Sigma-Aldrich | H5533 | Culture additive |
| Penicillin/Streptomycin 10000U/ml | Thermo-Fisher | 15140122 | Antibiotic mix |
| Optiplate 384 | Perkin-Elmer | 6007290 | Multiwell plate for 10ul expressions |
| Oligonucleotide | IDT synthesis | Oligo with sequence CAATAAAGTACAGAAACTGATAC TTATATAGCGTT | |
| Creatine Phosphokinase | Sigma-Aldrich | 9001-15-4 | Enzyme |
| Tecan Spark | Tecan | or similar Multimode Platereader | |
| Chemidoc MP Imager | Biorad | or similar SDS-PAGE gel Imager | |
| 4-12% Bis-Tris Gels | Invitrogen | NW04125 | SDS-PAGE gels |
| Biophotometer | Eppendorf | or similar Cuvette Specrophotometer | |
| Nanodrop One | Thermofisher | Nanodrop spectrophotometer | |
| Avanti JXN-26 centrifuge | Beckman Coulter | or similar centrifuge, with rotors/tubes rated 10K and 50K g | |
| 5424R microcentrifuge | Eppendorf | or similar microcentrifuge, with 1.5ml microcentrifuge tubes | |
| Flask Incubator Inova S44i | Eppendorf | or similar flask incubator shaker suitable for 5L Flasks | |
| 5L glass culture flasks | Baffled glass flasks for culture growth | ||
| Bactotryptone | BD | 211705 | Growth medium |
| Yeast Extract | Merck | VM930053 | Growth medium |
| Glycerol | Any analytical grade | ||
| Glucose | Any analytical grade | ||
| KH2PO4 | Any analytical grade | ||
| K2HPO4 | Any analytical grade | ||
| UltraPure water | Invitrogen | 10977-015 | Or output from any MilliQ-type water dispenser |
Referenzen
- Nirenberg, M. W., Matthaei, J. H. The dependence of cell-free protein synthesis in E.coli upon naturally occurring or synthetic polyribonucleotides. Proc Natl Acad Sci USA. 47 (10), 1588-1602 (1961).
- Caschera, F., Noireaux, V. Synthesis of 2.3 mg/ml of protein with an all Escherichia coli cell-free transcription-translation system. Biochimie. 99, 162-168 (2014).
- Kelwick, R., Webb, A. J., MacDonald, J. T., Freemont, P. S. Development of a Bacillus subtilis cell-free transcription-translation system for prototyping regulatory elements. Metab Eng. 38, 370-381 (2016).
- Ezure, T., et al. Cell-free protein synthesis system prepared from insect cells by freeze-thawing. Biotechnol Prog. 22 (6), 1570-1577 (2006).
- Harbers, M. Wheat germ systems for cell-free protein expression. FEBS Letters. 588 (17), 2762-2773 (2014).
- Kobs, G. Selecting the cell-free protein expression system that meets your experimental goals. Promega Corporation. 21, 6-9 (2008).
- Kovtun, O., et al. Leishmania cell-free protein expression system. Methods. 55 (1), 58-64 (2011).
- Basile, G., Peticca, M. Recombinant protein expression in Leishmania tarentolae. Mol Biotechnol. 43 (3), 273-278 (2009).
- Mureev, S., Kovtun, O., Nguyen, U. T., Alexandrov, K. Species-independent translational leaders facilitate cell-free expression. Nat Biotechnol. 27 (8), 747-752 (2009).
- Gambin, Y., et al. Single-molecule fluorescence reveals the oligomerization and folding steps driving the prion-like behavior of ASC. J Mol Biol. 430 (4), 491-508 (2018).
- Sierecki, E., et al. Rapid mapping of interactions between human SNX-BAR proteins measured in vitro by AlphaScreen and single-molecule spectroscopy. Mol Cell Proteomics. 13 (9), 2233-2245 (2014).
- Sierecki, E., et al. Nanomolar oligomerization and selective co-aggregation of alpha-synuclein pathogenic mutants revealed by single-molecule fluorescence. Sci Rep. 6, 37630 (2016).
- Leitao, A., Bhumkar, A., Hunter, D. J. B., Gambin, Y., Sierecki, E. Unveiling a selective mechanism for the inhibition of alpha-synuclein aggregation by beta-synuclein. Int J Mol Sci. 19 (2), 334 (2018).
- Gambin, Y., et al. Single-molecule analysis reveals self assembly and nanoscale segregation of two distinct cavin subcomplexes on caveolae. Elife. 3, e01434 (2013).
- Ve, T., et al. Structural basis of TIR-domain-assembly formation in MAL- and MyD88-dependent TLR4 signaling. Nat Struct Mol Biol. 24 (9), 743-751 (2017).
- Guo, Z., et al. Subunit organisation of in vitro reconstituted HOPS and CORVET multisubunit membrane tethering complexes. PLoS One. 8 (12), e81534 (2013).
- Ruehrer, S., Michel, H. Exploiting Leishmania tarentolae cell-free extracts for the synthesis of human solute carriers. Mol Membr Biol. 30 (4), 288-302 (2013).
- Varasteh Moradi, S., et al. Mapping Interactions among cell-free expressed Zika virus proteins. J Proteome Res. 19 (4), 1522-1532 (2020).
- Gagoski, D., et al. Cell-free pipeline for discovery of thermotolerant xylanases and endo-1,4-beta-glucanases. J Biotechnol. 259, 191-198 (2017).
- Ergun Ayva, C., et al. Exploring performance parameters of artificial allosteric protein switches. J Mol Biol. 434 (17), 167678 (2022).
- Lau, D., et al. Fluorescence biosensor for real-time interaction dynamics of host proteins with HIV-1 capsid tubes. ACS Appl Mater Interfaces. 11 (38), 34586-34594 (2019).
- Ryan, S. M., et al. Novel antiinflammatory biologics shaped by parasite-host coevolution. Proc Natl Acad Sci USA. 119 (36), e2202795119 (2022).
- McMahon, K. A., et al. Identification of intracellular cavin target proteins reveals cavin-PP1alpha interactions regulate apoptosis. Nat Commun. 10 (1), 3279 (2019).
- Sierecki, E., et al. A cell-free approach to accelerate the study of protein-protein interactions in vitro. Interface Focus. 3 (5), 20230018 (2013).
- Johnston, W. A., Moradi, S. V., Alexandrov, K. Adaption of the Leishmania cell-free expression system to high-throughput analysis of protein interactions. Methods Mol Biol. 2025, 403-421 (2019).
- Jung, W., et al. Cell-free formation and interactome analysis of caveolae. J Cell Biol. 217 (6), 2141-2165 (2018).
- Fontaine, F. R., et al. Functional domain analysis of SOX18 transcription factor using a single-chain variable fragment-based approach. MAbs. 10 (4), 596-606 (2018).
- Overman, J., et al. Pharmacological targeting of the transcription factor SOX18 delays breast cancer in mice. Elife. 6, e21221 (2017).
- Kubala, M. H., et al. Mammalian farnesyltransferase alpha subunit regulates vacuolar protein sorting-associated protein 4A (Vps4A)--dependent intracellular trafficking through recycling endosomes. Biochem Biophys Res Commun. 468 (4), 580-586 (2015).
- Han, S. P., et al. Cortactin scaffolds Arp2/3 and WAVE2 at the epithelial zonula adherens. J Biol Chem. 289 (11), 7764-7775 (2014).
- Das Gupta, K., et al. Class IIa histone deacetylases drive toll-like receptor-inducible glycolysis and macrophage inflammatory responses via pyruvate kinase M2. Cell Rep. 30 (8), 2712-2728.e8 (2020).
- Leitão, A. D. G., et al. Selectivity of protein interactions along the aggregation pathway of α-synuclein. BioRxiv. , (2021).
- Gagoski, D., et al. Performance benchmarking of four cell-free protein expression systems. Biotechnol Bioeng. 113 (2), 292-300 (2016).
- Johnston, W. A., Alexandrov, K. Production of eukaryotic cell-free lysate from Leishmania tarentolae. Methods Mol Biol. 1118, 1-15 (2014).
- Hunter, D. J. B., Bhumkar, A., Giles, N., Sierecki, E., Gambin, Y. Unexpected instabilities explain batch-to-batch variability in cell-free protein expression systems. Biotechnol Bioeng. 115 (8), 1904-1914 (2018).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten