Method Article
Extração de cromatina de tecido hepático quimérico congelado para análise de imunoprecipitação de cromatina
Neste Artigo
Resumo
Este protocolo concentra-se na preparação de cromatina a partir de tecidos congelados instantâneos e é adequado para Imunoprecipitação de Cromatina de Reticulação (X-ChIP) seguida de análise quantitativa de PCR (X-ChIP-qPCR) ou abordagens de sequenciamento de próxima geração (X-ChIP-seq).
Resumo
A imunoprecipitação de cromatina reticulada (X-ChIP) é uma técnica amplamente utilizada para avaliar os níveis de marcas de histonas e a ocupação de fatores de transcrição na cromatina do hospedeiro e/ou patógeno. A preparação de cromatina a partir de tecidos cria desafios adicionais que precisam ser superados para a obtenção de protocolos reprodutíveis e confiáveis comparáveis aos utilizados para cultura celular. A ruptura tecidual e a fixação são etapas críticas para alcançar o cisalhamento eficiente da cromatina. A coexistência de diferentes tipos celulares e agrupamentos também pode exigir diferentes tempos de cisalhamento para atingir o tamanho ideal do fragmento e dificultar a reprodutibilidade do cisalhamento. O objetivo deste método é obter preparações de cromatina hospedeira confiáveis e reprodutíveis a partir de tecido congelado (fígado) adequadas para aplicações de ChIP-qPCR e sequenciamento de próxima geração (NGS). Observamos que a combinação de pulverização tecidual de nitrogênio líquido seguida de homogeneização leva a maior reprodutibilidade em comparação com a homogeneização apenas, uma vez que fornece uma suspensão constituída principalmente por células isoladas dissociadas que podem ser eficientemente cisalhadas. Além disso, a etapa de fixação deve ser realizada sob rotação leve para proporcionar reticulação homogênea. O material fixo é então adequado para o isolamento de núcleos baseados em tampão, para reduzir a contaminação de proteínas citoplasmáticas e DNAs e RNAs de patógenos (quando aplicável), evitando gradientes de centrifugação demorados. A sonicação subsequente completará a lise nuclear e cisalhará a cromatina, produzindo uma faixa de tamanho específica de acordo com as condições de cisalhamento escolhidas. A faixa de tamanho deve ficar entre 100 e 300 nt para aplicações NGS, enquanto pode ser maior (300-700 nt) para análise ChIP-qPCR. Tais adaptações de protocolo podem melhorar muito as análises de cromatina a partir de amostras de tecido congelado.
Introdução
Desde sua descoberta, a regulação epigenética em células de mamíferos tem ganhado crescente reconhecimento1, considerando que a compreensão de tais mecanismos forneceria insights fundamentais não apenas na biologia celular, mas também na biologia de doenças e tumores. Além disso, agentes infecciosos também podem causar alterações epigenéticas no hospedeiro2, enquanto a maquinaria da célula hospedeira também pode afetar a cromatina de patógenos, como a persistência de vírus de DNA 3,4. Essa interação patógeno-hospedeiro parece desempenhar um papel na persistência da infecção. algarismo
Através de uma associação reversível com o DNA, as proteínas histonas formam um complexo chamado nucleossomo. Os nucleossomos, por sua vez, atingem um nível mais alto de organização conhecido como cromatina. Sabe-se que o remodelamento da cromatina regula fortemente a expressão gênica, concedendo ou negando acesso a fatores de transcrição (FTs)5. Esses fatores podem desencadear ou bloquear o recrutamento da RNA polimerase II (PolII) em promotores gênicos, influenciando a síntese de RNAm a partir do molde deDNA6. As proteínas histonas contêm caudas7, flanqueando ambas as extremidades da prega das histonas, que podem estar sujeitas a modificações pós-traducionais (MPTs), permitindo uma regulação estreita da transcrição gênica por mudanças estruturais da cromatina. A maioria das PTMs de histonas está localizada no terminal N da cauda, sendo acetilação e metilação as MPTs mais bem estudadas, embora fosforilação8, ubiquitinação9 e ribosilação10 também tenham sido relatadas. Caracterizar e estudar tais proteínas é então essencial para obter uma visão profunda sobre a regulação gênica.
Atualmente, há um punhado de métodos e ferramentas bem estabelecidos disponíveis para estudar interações diretas DNA-proteína: Electrophoretic mobility shift assay (EMSA), Yeast one-hybrid assay (Y1H) e DNA footprinting11. No entanto, esses métodos se concentram per se em interações DNA-proteína única e não são aplicáveis para estudos genômicos amplos. Outra limitação dessas técnicas é a ausência de associação de histonas com os segmentos de DNA investigados. Assim, tais abordagens não pretendem refletir a complexidade da maquinaria transcricional in vivo e não levam em conta mudanças estruturais importantes12 ou outras enzimas/cofatores necessários13 que poderiam influenciar (promover ou inibir) a ligação de proteínas ao DNA.
A ideia de que a fixação de células com agentes como formaldeído (FA) poderia fornecer um instantâneo in vivo das interações proteína-DNA, criou a base para o desenvolvimento de ensaios de imunoprecipitação de cromatina (ChIP)14. Isso, juntamente com a disponibilidade da tecnologia de PCR quantitativo (qPCR) e de anticorpos altamente específicos, permitiu o desenvolvimento de ensaios de ChIP-qPCR. Posteriormente, o advento de técnicas de sequenciamento de próxima geração (NGS), cujos custos estão ficando mais acessíveis, permitiu acoplar experimentos ChIP com abordagens NGS (ChIP-seq), fornecendo assim aos pesquisadores novas ferramentas poderosas que permitem a investigação da regulação da cromatina. Nesses ensaios, células isoladas ou cultivadas são fixadas com disuccinimidil glutarato (DSG) e/ou AG, núcleos são isolados, cromatina é então fragmentada e precipitada pelo anticorpo de interesse. A partir de então, o DNA é purificado e analisado por PCR ou NGS. Em contraste com EMSA, Y1H e pegada de DNA, os ensaios ChIP têm a capacidade de fornecer um instantâneo global da interação proteína-DNA dentro da célula. Isso oferece flexibilidade e permite a análise de vários loci dentro da mesma amostra. No entanto, devido à natureza do ensaio, ChIP pode, eventualmente, detectar não apenas interações diretas, mas também indiretas, não oferecendo a precisão dos métodos acima mencionados, quando interessado em interações diretas proteína-DNA.
Os protocolos de preparação de cromatina a partir de material de cultura celular são bem estabelecidos15 e altamente reprodutíveis, permitindo ao usuário obter cromatina adequada tanto para abordagens de qPCR quanto NGS em 1-2 dias úteis. No entanto, a obtenção de cromatina de alta qualidade a partir de tecidos inteiros ainda representa um desafio devido à necessidade de dissociar as células dentro do tecido, ao mesmo tempo em que se obtém a fixação e cisalhamento ideais da cromatina. Além disso, a composição e a morfologia de diferentes tipos de tecidos variam, exigindo ajustes nos protocolos existentes16,17. O uso de tecido criopreservado oferece desafios adicionais em comparação com amostras frescas. Isso se deve à dificuldade de se obter uma suspensão unicelular sem grandes perdas de material. Isso leva ao cisalhamento indevido, dificultando as aplicações a jusante. No entanto, o acesso a espécimes de tecidos congelados em vez da contraparte fresca não só aumenta a flexibilidade do trabalho, mas também pode representar a única opção para os pesquisadores que trabalham com espécimes que se originam de estudos longitudinais ou comparativos. Um punhado de protocolos de preparação de cromatina para tecido congelado foram publicados. Estes baseiam-se, em sua maioria, no descongelamento dos espécimes seguido de etapas de picagem, dissociação manual/máquina ou pulverização de nitrogênio líquido18,19,20.
Aqui descrevemos um método otimizado de preparação de cromatina15 para espécimes de fígado congelados não fixos, que combina pulverização de tecido em nitrogênio líquido com homogeneização de pilão, para obter um cisalhamento de cromatina reprodutível adequado para abordagens X-ChIP visando analisar genomas virais e hospedeiros.
Protocolo
A amostragem de tecido de camundongos quiméricos hepáticoshumanos 21 foi realizada de acordo com a diretiva 86/609/CEE da União Europeia e aprovada pelo comitê de ética da cidade e estado de Hamburgo de acordo com os princípios da Declaração de Helsinque.
1. Preparação dos reagentes
- Preparar solução de glicina 1,25 M em água deionizada. Filtro estéril com filtro de poros de 0,22 μm. Conservar a 4 °C.
- Preparar solução de cloreto de sódio (NaCl) 5 M. Conservar à temperatura ambiente.
- Preparar solução de CaCl 2: 300 mM de CaCl2 e 10 mM de Tris-HCl pH 8 em água deionizada. Filtro estéril com filtro de poros de 0,22 μm e armazenar em RT.
- Preparar uma diluição de 10% Triton X-100 em água deionizada. Loja na RT.
- Preparar tampão Tris-EDTA: 1 mM EDTA e 10 mM Tris pH 8 em água deionizada. Conservar a 4 °C.
- Prepare os seguintes buffers de acordo com a quantidade necessária:
- Preparar tampão A: 50 mM Hepes-KOH pH 7,5, 140 mM NaCl, 1 mM ácido etilenodiaminotetracético (EDTA), 10% Glicerol, 0,5% NP-40 e 0,25% Triton X-100 em água deionizada. Filtro estéril com filtro de poros de 0,22 μm. Conservar a 4 °C.
- Preparar tampão B: 10 mM Tris-HCl pH 8, 200 mM NaCl, 1mM EDTA, 0,5 mM ácido egtazico (EGTA). Filtro estéril com filtro de poros de 0,22 μm. Conservar a 4 °C.
- Preparar tampão C: 1% SDS, 10 mM EDTA e 50 mM Tris-HCl pH 8 em água deionizada. Filtro estéril com filtro de poros de 0,22 μm. Loja na RT.
- Preparar tampão de diluição da cromatina: 0,01% SDS, 1,1% Triton X-100, 1,2 mM EDTA, 16,6 mM Tris-HCl pH 8 e 166 mM NaCl em água deionizada. Filtro estéril com filtro de poros de 0,22 μm. Conservar a 4 °C.
2. Preparação do material
- Colete gelo seco, gelo e nitrogênio líquido.
CUIDADO: Manuseie gelo seco e nitrogênio líquido com os cuidados necessários para evitar queimaduras. - Pré-resfriar a centrífuga a 4 °C.
NOTA: Esta etapa é importante para evitar a degradação de proteínas e a desreticulação durante as etapas de lavagem, pois isso reduzirá a qualidade da cromatina. - Coloque uma placa estéril no gelo seco e deixe esfriar.
NOTA: Certifique-se de que a placa é grande o suficiente para facilitar o processo de corte. Recomenda-se uma placa de Petri/placa de cultura celular de 100 mm. - Retire a alíquota necessária de glicina 1,25 M e deixe chegar ao TR.
- Retire as alíquotas necessárias dos buffers A, B e PBS. Adicionar protease e/ou inibidores de desacetilase e fosfatase para atingir 1 vez a concentração e deixá-los no gelo.
- Retire a alíquota necessária do buffer C deixando-o no RT. Não adicione a protease e/ou os inibidores da desacetilase e da fosfatase até que seja especificado.
- Retire a alíquota necessária do RT PBS.
CUIDADO: O tampão C contém dodecil sulfato de sódio (SDS). Adotar as medidas de segurança adequadas ao preparar o amortecedor.
NOTA: SDS precipita no gelo, e inibidores de protease e desacetilase não são estáveis em RT. - Pré-resfriar a argamassa despejando nitrogênio líquido em sua câmara, seguindo rigorosamente as instruções do fornecedor. Resfriar o pilão metálico em gelo seco por pelo menos 5 min.
OBS: É possível utilizar uma argamassa alternativa à proposta. Entretanto, o dispositivo utilizado neste protocolo, devido à sua construção peculiar, permite trabalhar com pequena quantidade de tecido sem perda substancial durante o processo de pulverização. - Pré-resfriar o homogeneizador Dounce com o pilão A associado no gelo.
NOTA: O pilão A tem um encaixe solto com o homogeneizador. Isso permite obter uma suspensão de célula única sem lise celular significativa.
3. Reticulação tecidual
- Corte cerca de 50 mg de tecido congelado diretamente no prato em gelo seco com a ajuda de um bisturi e pinça.
OBS: Sugere-se manter o bisturi em RT, pois isso facilitará o processo de corte. Evite aplicar muita pressão no bisturi, pois isso aumentará o risco de espalhar pedaços de tecido fora da área de corte. Para notar, 50 mg de tecido (fígado neste caso) deve render cerca de 5 milhões de células. Observe que a lâmina quente descongelará a borda de corte. Considerando o tamanho relativamente grande da peça de tecido, no entanto, isso deve ter um efeito limitado. Quando pedaços menores são cortados, pode ser benéfico usar um bisturi frio prestando atenção para evitar a dispersão do tecido. - Coloque o tecido cortado em um tubo de 1,5 mL já resfriado em gelo seco. Evite o descongelamento dos tecidos.
- Mova o tubo contendo o tecido para a argamassa, deixando-o descansar por 5 min.
NOTA: Deixar a amostra repousar na argamassa diminui sua temperatura (de -80 °C para -196 °C). Isso aumenta sua tenacidade e facilita a etapa de pulverização. - Aplique pressão na amostra com a ajuda do pilão pré-resfriado até que não sejam visíveis mais desmoronamentos sólidos.
NOTA: É importante evitar o aquecimento do pilão por forças de rotação excessivas, pois isso descongelará a amostra. Após cada pulverização da amostra, limpe o pilão com etanol 70% (EtOH) e deixe esfriar novamente em gelo seco. - Retire o tubo que contém a amostra da argamassa e adicione 950 μL de PBS gelado com os inibidores necessários. Pipetar suavemente para cima e para baixo até que a amostra seja completamente ressuspensa. Prossiga imediatamente para a etapa 3.6.
- Transfira a suspensão de tecido para o homogeneizador e aplique 20-30 tempos com pilão A para obter uma suspensão mais fina. Evite espuma.
NOTA: A quantidade de traços deve ser otimizada de acordo com a consistência do tecido. Esta etapa dissocia ainda mais os pequenos aglomerados de células obtidos após a pulverização. Uma homogeneização inadequada pode afetar a eficiência do cisalhamento. - Transfira o homogeneizado para um novo tubo de 1,5 mL, já pré-resfriado em gelo.
- Centrifugar durante 5 min a 1.300 x g a 4 °C e remover cuidadosamente o sobrenadante.
- Ressuspender completamente o pellet em 950 μL de RT PBS por pipetagem suave e adicionar 63,6 μL de FA livre de MeOH a 16% para obter uma concentração final de 1%. Prossiga imediatamente para a etapa 3.10.
CUIDADO: FA é um produto químico tóxico. Manuseie-o sob um exaustor com as medidas de segurança adequadas.
NOTA: A ressuspensão incompleta pode provocar agregação celular durante a etapa de fixação. Isso dificulta o processo de lise e cisalhamento. - Gire 10 min no RT. Prossiga imediatamente para a etapa 3.11
NOTA: A rotação é necessária para evitar agregados. O tempo necessário para fixação deve ser otimizado, de acordo com o alvo de interesse e o tipo de amostra. É importante ressaltar que tempos excessivos de fixação podem dificultar o cisalhamento adequado. - Adicionar 113 μL de glicina 1,25 M em TR para obter uma concentração final de 125 mM e girar por 5 min.
NOTA: A glicina elimina a reação fixadora evitando ligações cruzadas. - Centrifugar a 1.300 x g por 3 min a 4 °C.
- Descarte o sobrenadante e ressuspenda o pellet cuidadosamente pipetando em 950 μL de PBS gelado com os inibidores necessários.
- Centrifugar a 1.300 x g por 3 min a 4 °C.
- Repita os passos 3.13-3.14 e prossiga imediatamente para os passos de isolamento da cromatina.
4. Isolamento da cromatina
- Adicionar 950 μL de tampão A com os inibidores necessários ao pellet. Misture suavemente por pipetagem até que o pellet seja completamente ressuspenso e gire 10 min a 4 °C.
NOTA: Esta etapa lisa a suspensão fixa de célula única, sem lise nucleimétrica. Isso permite livrar a amostra de proteínas citosólicas e RNAs. Prolongar o tempo de lise pode ser benéfico para células difíceis de lisar, aumentando, no entanto, o tempo de manuseio do tecido. Neste momento é possível verificar o preparo ao microscópio após coloração com azul de tripano/DAPI para verificar o tamanho dos cachos e a presença de células únicas. No entanto, os núcleos únicos podem não ser fáceis de apreciar devido ao material tecidual fixo. - Centrifugar a 2.000 x g durante 5 min a 4 °C e remover cuidadosamente o sobrenadante.
- Adicionar 950 μL de tampão B com os inibidores necessários ao pellet. Misture suavemente por pipetagem até que o pellet seja completamente ressuspenso e gire 10 min a 4 °C.
NOTA: Esta etapa lava o tampão de lise da preparação dos núcleos para evitar mais lise indesejada. - Centrifugar a 2000 x g durante 5 min a 4 °C. Enquanto isso, adicione os inibidores necessários (o mesmo que o passo 2.5) ao tampão C.
- Retire cuidadosamente o sobrenadante.
- Adicionar 300 μL de tampão RT C ao pellet e pipetar vigorosamente.
- Vórtice a amostra por 15-30 s e gire o tubo brevemente para coletar as gotas na tampa.
NOTA: Esta etapa é importante para liberar e lisar os núcleos fixos. Para preservar a integridade da amostra e, ao mesmo tempo, evitar a precipitação de SDS, mantenha a amostra antes da sonicação em um rack de plástico mantido no gelo para manter uma temperatura de 9-11 °C.
5. Fragmentação da cromatina
- Transfira a amostra para três tubos limpos com certificação de sonicação de 0,65 mL, garantindo 100 μL de suspensão de núcleos lisados por tubo.
NOTA: É possível usar tubos certificados de sonicação de 1,5 mL com um volume máximo de 300 μL. É necessário um suporte específico para esses tubos. 0,65 mL deve oferecer cisalhamento mais homogêneo devido ao menor volume da amostra por tubo. - Sonicar a cromatina por 28 ciclos em alta intensidade com ajuste de 30 s ON e 30 s OFF. Certifique-se de que o banho do sonicador está devidamente resfriado (gelo ou dispositivo de resfriamento).
NOTA: Esta etapa precisa de otimização em quase todos os casos. O usuário deve ter em mente que o aumento do tempo de cisalhamento proporcionará fragmentos menores e mais homogêneos; no entanto, isso pode aumentar a chance de diminuir a qualidade da cromatina. Escolha o menor número de ciclos que forneça o tamanho de fragmento necessário. Durante a otimização desta etapa, é útil realizar coloração nuclear para verificar se o número de ciclos foi suficiente para lisar a maioria dos núcleos. - Transfira a cromatina sonicada para um novo tubo de 1,5 mL previamente resfriado em gelo.
- Adicionar 30 μL de solução de Triton X-100 a 10% e vórtice durante 5-10 s.
NOTA: Triton X-100 liga o SDS impedindo precipitação adicional a 4 °C. A quantidade final de Triton X-100 deve ser sempre de 1%. - Centrifugar a 16.000 x g por 15 min a 4 °C.
- Transfira o sobrenadante para um tubo limpo de 1,5 mL pré-resfriado em gelo.
- NOTA: O sobrenadante contém a cromatina cortada e deve aparecer límpido. O pellet contém restos "não cisalháveis" e deve permanecer muito pequeno (principalmente marrom no caso de tecido hepático). Procure indicação de cisalhamento malsucedido: solução de cromatina que não se tornou mais clara e dimensões de pastilha semelhantes às do passo 4.5.
6. Purificação do DNA
- Transferir 10-25 μL de cromatina cortada para um novo tubo e adicionar Buffer C para atingir um volume final de 200 μL. Conservar o resto da cromatina a -80 °C até nova utilização. Se necessário, o procedimento pode ser interrompido nesta etapa e a amostra armazenada a -20 °C.
- Adicionar 8 μL de NaCl 5 M e incubar pelo menos 6 h a 65 °C num bloco de aquecimento sob agitação a 1000 rpm.
NOTA: Esta etapa descruza a cromatina. É mais seguro estender a descrosslinking durante a noite, quando possível. A presença de NaCl torna o processo mais eficiente. - Deixar arrefecer as amostras em RT durante 5 min e adicionar 2 μL de RNase A.
- Incubar durante 1 h a 37 °C sob agitação a 1000 rpm.
- Retirar as amostras do bloco de aquecimento e adicionar 7 μL de CaCl2 300 mM e 2 μL de Proteinase K.
- Ajustar o bloco de aquecimento a 56 °C e incubar durante 30 minutos sob agitação a 1000 rpm. Enquanto isso, prepare um tubo de separação de fases para cada amostra, centrifugando-os a 16.000 x g por 1 min a 4 °C.
NOTA: Estes tubos especiais facilitam a separação de fases durante a extração de fenol-clorofórmio de ácido nucleico. - Retire os tubos do bloco de aquecimento e deixe-os equilibrar em TR por 3 min.
- Transferir 400 μL da amostra para um tubo de separação de fase previamente centrifugado.
- Adicionar 400 μL de solução de fenol-clorofórmio-álcool isoamílico (PCI) e vórtice durante 5 s.
CUIDADO: A PCI é um composto altamente volátil e tóxico. Por favor, manuseie-o com as medidas de segurança necessárias sob um exaustor. - Centrifugar a 16.000 x g por 5 min a 4 °C.
- Adicionar 400 μL de clorofórmio e vórtice durante 5 s.
CUIDADO: O clorofórmio é um composto altamente volátil e tóxico. Por favor, manuseie-o com as medidas de segurança necessárias sob um exaustor.
NOTA: Esta etapa elimina possíveis resíduos de fenol, que poderiam interferir com as aplicações de PCR a jusante. - Centrifugar a 16.000 x g por 5 min a 4 °C.
- Transferir 400 μL da fase superior para um novo tubo de 1,5 mL, onde foram adicionados 24 μL de NaCl 5 M e 0,75 μL de glicogênio. Brevemente vórtice.
- Adicionar 1.055 μL de EtOH a 100% e o vórtice completamente. Garanta a mistura adequada.
- Incubar a -80 °C durante 1 h ou a -20 °C durante a noite (ON).
NOTA: Esta etapa precipita o DNA cisalhado; para maximizar o rendimento, sugere-se a escolha da incubação ON. - Centrifugar a 16.000 x g por 30 min a 4 °C.
- Retire cuidadosamente o sobrenadante prestando atenção para não deslocar o pellet.
- Adicionar 500 μL de EtOH a frio a 70%. Incline o tubo suavemente para garantir que o pellet seja lavado.
NOTA: Esta etapa é essencial para remover resíduos de sal que poderiam ter co-precipitado com os ácidos nucleicos. Os sais podem interferir com outras aplicações a jusante. - Centrifugar a 16.000 x g por 15 min a 4 °C.
- Retire cuidadosamente todo o sobrenadante e deixe o pellet secar em RT.
NOTA: Incubar o tubo num bloco de aquecimento a 37 °C reduzirá o tempo necessário para a secagem. - Adicionar 50 μL de solução de Tris-EDTA (TE-Buffer) e colocar o tubo no bloco de aquecimento a 37 °C durante 5-10 minutos sob agitação a 300 rpm.
NOTA: Esta etapa garante a dissolução do pellet. O protocolo pode ser pausado aqui, e a amostra pode ser armazenada a 4 °C por até 1 semana ou a -20 °C para armazenamento mais longo. - Realizar análise de DNA em gel de agarose 1%.
7. Análise do tamanho do DNA
- Preparar um gel de agarose a 1% misturando 1 g de agarose por 100 ml de tampão de corrida (ou seja, Tris-acetato-EDTA (TAE) ou Tris-borato-EDTA (TBE)). Aqueça a suspensão até que a agarose esteja completamente dissolvida. Adicionar 10 μL de EtBr para cada 100 mL de solução de agarose antes de despejar a solução de gel.
CUIDADO: EtBr é um agente intercalante de DNA conhecido por ser carcinogênico. Por favor, manuseie-o com as medidas de segurança necessárias sob um exaustor.
NOTA: A coloração EtBr (diretamente em gel ou após a corrida) é fortemente sugerida. Outros corantes intercalantes de DNA não tiveram um bom desempenho em nossas mãos quando trabalhamos com esfregaços de DNA. Poços de carregamento estreitos proporcionam uma melhor resolução quando comparados aos poços mais largos. - Misturar 10 μL da amostra com 2 μL de corante de carregamento 6x. Em seguida, carregar 10 μL da amostra no gel e executá-la até que a última banda do corante de carga corra para 2/3 do gel. Certifique-se de adicionar uma escada de DNA.
- Faça uma imagem do gel e verifique se o tamanho do esfregaço está na faixa para a aplicação desejada.
Se a cromatina passar no controle de qualidade, ela pode ser usada para aplicações a jusante.
Resultados
A preparação da cromatina é um passo crucial para alcançar um ChIP bem-sucedido. A fim de preparar cromatina de boa qualidade a partir de espécimes congelados, devemos garantir uma ruptura tecidual eficiente antes da fixação para evitar a presença de aglomerados de tecido que possam dificultar o cisalhamento eficiente. A Figura 1 mostra um pipeline resumido do protocolo. A pulverização isolada não é suficiente para dissociar completamente o tecido, pois produz aglomerados celulares de tamanho variável e poucas células isoladas (Figura 1a). Associando a primeira etapa da pulverização com a homogeneização de Dounce, a quantidade de touceiras teciduais é fortemente reduzida e as demais são menores (Figura 1b). Após as etapas de fixação e lise, o número de núcleos únicos visíveis (Figura 1c) aumenta, enquanto a aparência esférica típica é perdida. Após a sonicação por 28 ciclos, a coloração nuclear (Hoechst 33258/DAPI) na maioria das vezes não é mais visível. Este é, de fato, um sinal de cisalhamento bem-sucedido (Figura 1d). Após a desreticulação de uma alíquota de cromatina e visualização do DNA em gel de agarose, o sucesso do cisalhamento pode ser reconhecido pela presença de fragmentos na faixa de 100-300 pb. (Figura 2a) A quantidade de DNA pode variar de acordo com a composição do tecido preparado. Essa cromatina pode ser usada com sucesso para ChIP-qPCR. Como mostrado na Figura 2b , a cromatina pode ser precipitada com sucesso com anticorpos H3K4me3, H3K27ac (modificações relacionadas a genes ativos) e H3K27me3 (modificação relacionada a genes silenciados). As regiões promotoras do cromossomo 1 Open Reading Frame 43 (C1orf43), do proteassoma 20S Subunit Beta 2 (PSMB2) e do Glyceraldehyde 3-phosphate desidrogenase (mGapdh) resultaram enriquecidas em H3K4me3 e H3K27ac em comparação com as regiões promotoras Homeobox C13 (HOXC13), Homeobox C12 (HOXC12) e Myelin Transcription Factor 1 (mMyt1) de camundongo (Tabela 1). Isso ocorre porque C1orf43, PSMB2 e mGapdh são transcritos constitutivamente no fígado, enquanto HOXC13, HOXC12 e mMyt1 são silenciados. H3K27me3 mostra o comportamento oposto, confirmando o sucesso do ensaio ChIP. O fato de o fígado desses camundongos ser uma quimera, nos permitiu analisar tanto a cromatina murina quanto a humana. Além disso, a mesma cromatina pode ser usada com sucesso em experimentos de ChIP-seq. Após a etapa de sequenciamento, as leituras foram alinhadas a um índice composto por genomas murinos e humanos para reduzir a quantidade de fragmentos desalinhados. Posteriormente, as leituras foram separadas de acordo com a espécie e posteriormente analisadas com EaSeq22 . A intensidade de sinal foi então medida no local de início da transcrição (TSS) de cada gene e o resultado classificado para a intensidade do sinal H3K4me3. A Figura 3a e a Figura 3c mostram uma presença marcante de H3K4me3 e H3K27ac no TSS para uma porção considerável dos genes dentro da cromatina humana e de camundongo. Além disso, H3K27me3 se correlaciona com H3K4me3/H3K27ac. A H3K27me3 está presente em toda a extensão do gene e não apenas na SST, como esperado desta PTM. A Figura 3b e a Figura3d mostram o cluster HOXC/HoxC conhecido por ser enriquecido para H3K27me3 e transcricionalmente inativo em fígados de camundongos e humanos. O perfil de H3K4me3 e H3K27ac mostra picos para essas duas MPTs, enquanto a intensidade de sinal de H3K27me3 tende a ser menor e mais distribuída.
Devido à complexidade da preparação da cromatina, pode ocorrer sobrefixação, o tempo de lise ou sonicação pode ser subótimo, aglomerados de grandes células podem persistir ou o manuseio inadequado da amostra pode ser inadequado. Todos estes são acontecimentos que afectam a qualidade da preparação. Em alguns casos, o enriquecimento de fragmentos de cromatina dentro do tamanho correto ainda estará presente ou será deslocado para um tamanho maior. Em outros casos, pode haver perda de material devido à lise prematura ou cisalhamento malsucedido. A Figura 4 mostra alguns exemplos desses resultados negativos e subótimos. As faixas 3 e 4 mostram um enriquecimento do tamanho do fragmento entre 200 pb e 800 pb. No entanto, é claro que o tamanho do fragmento varia de 100 pb a >10.000 pb. Nas faixas 5 e 6 está presente um enriquecimento na faixa de 100-250 pb com uma clara perda de material durante a preparação. Isso poderia explicar por que a sonicação produziu fragmentos menores. A faixa 7 mostra uma preparação ligeiramente abaixo do ideal com a faixa de fragmentos aumentada, enquanto a faixa 8 mostra uma perda quase completa de material. Isso pode ser causado por lise nuclear prematura ou dissociação tecidual insuficiente com consequente perda após o passo 5.5.

Figura 1: Visão geral do protocolo de preparação da cromatina. As fotos foram obtidas após pulverização do tecido (a), homogeneização manual adicional (b), após lise nuclear (c) e após sonicação (antes da centrifugação) (d). A coloração nuclear foi realizada com Hoechst 33258/DAPI. Barra de escala = 200 μm. Clique aqui para ver uma versão maior desta figura.

Figura 2: Cisalhamento representativo da cromatina e sua qualidade avaliada por ChIP-qPCR . Gel de agarose a 1% com amostras de cromatina fragmentadas de acordo com protocolos de diferentes preparações de cromatina. É adicionado um controlo da cromatina não cisalhada para garantir que não haja degradação prévia da cromatina/ADN (a). Cromatina tesourada foi testada quanto à qualidade realizando um ensaio de ChIP-qPCR. Anticorpos H3K4me3, H3K27ac e H3K27me3 foram usados para precipitar a cromatina recém-preparada. (b ) a análise de qPCR foi realizada em promotores ativos humanos (C1orf43 e PSMB2), murinos (Gapdh) e humanos (HOXC13, HOXC12), murinos (Myt1). Clique aqui para ver uma versão maior desta figura.
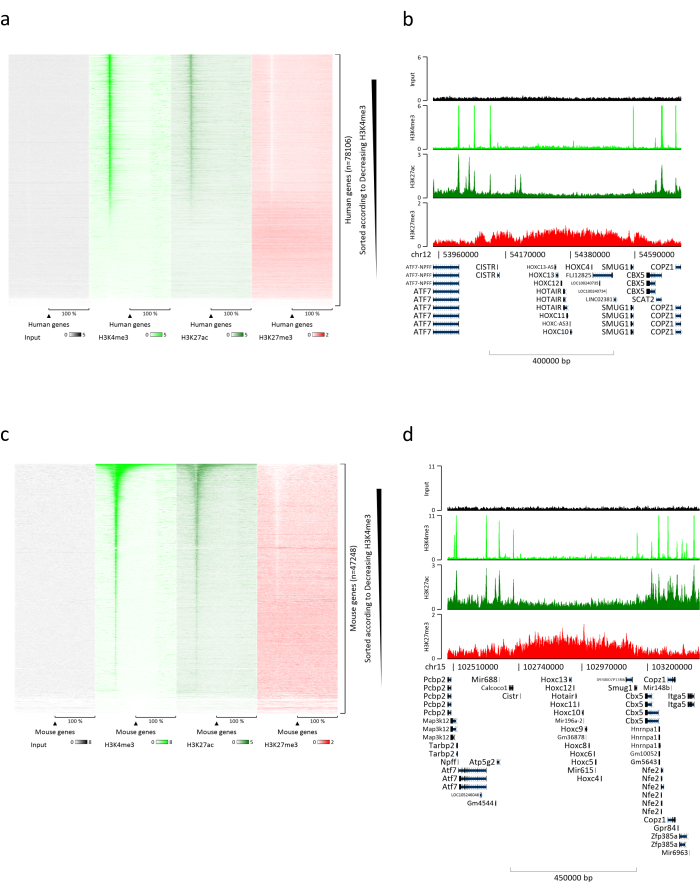
Figura 3: Análise representativa de ChIP-seq. As leituras foram alinhadas a um índice criado com genomas humanos e de camundongos (hg19 e mm10). Após o alinhamento, as leituras humana e murina foram separadas e posteriormente analisadas. Mapa de calor de genes humanos onde o sinal foi quantificado no TSS e mostrado em ordem decrescente para intensidade de H3K4me3 (a). Exemplo de cluster genético humano de genes suprimidos (cluster HOX) cercado por genes ativos (b). Mapa de calor de genes murinos onde o sinal foi quantificado no TSS e mostrado em ordem decrescente para intensidade de H3K4me3 (c). Exemplo de um grupo de genes murinos de genes suprimidos (cluster Hox) cercado por genes ativos (d). Todos os dados mostrados foram normalizados pelo EaSeq por milhão de leituras. Clique aqui para ver uma versão maior desta figura.

Figura 4: Preparações de cromatina subótimas e com falha. Gel de agarose a 1% com amostras de cromatina fragmentadas de acordo com protocolo. A figura contém cromatina não cisalhada usada como controle (faixa 2), cisalhamento não ideal (faixa 3-4), cisalhamento ótimo com perda clara de material (faixa 5-6), cisalhamento subótimo (faixa 7) e perda material extensa (faixa 8). Clique aqui para ver uma versão maior desta figura.
| Nome da cartilha | Seqüenciar | |
| Promotor C1orf43 | Encaminhar | AGTGGGTGGAGAATGCAGAC |
| Inverter | GAGATTACCCCACCCCATTC | |
| Promotor PSMB2 | Encaminhar | CTTATTCAACCCCCGACAAA |
| Inverter | GATGAAGGACGGTGAGAGGA | |
| Promotor distal HOXC13 | Encaminhar | GAGCCCGAGATTCACTCAAC |
| Inverter | TTATGCCCAGTTTTGGGGTA | |
| Promotor distal HOXC12 | Encaminhar | AAAGCTTCCCACTGCAAAGA |
| Inverter | AAATCTGGGGGCGAACTACT | |
| Promotor mGAPDH | Encaminhar | GGTCCAAAGAGAGGGAGGAG |
| Inverter | GCCCTGCTTATCCAGTCCTA | |
| Promotor mMYT | Encaminhar | CAGCCCAATTCTAGCCACAT |
| Inverter | CCAAAGCAGGGGAGTAGGAG |
Tabela 1: Lista de primers qPCR para genes ativos e inativos usados para ensaios de ChIP-qPCR.
Discussão
A preparação de cromatina a partir de tecido congelado instantâneo permanece um desafio devido ao número de etapas que precisam ser otimizadas para alcançar resultados reprodutíveis e confiáveis. A maioria dos protocolos já publicados 16,23 requer a picagem tecidual antes da dissociação manual (douncing). Procurou-se evitar ao máximo etapas que pudessem provocar degradação proteica antes da fixação da amostra. A etapa de pulverização já é utilizada em preparações hepáticas congeladas24 e torna a dissociação manual mais fácil e reprodutível (ver Figura 2a). Com o uso de uma argamassa projetada especificamente para tubos de 1,5 mL (ver Protocolos), a perda de espécime durante o processo de pulverização é reduzida, permitindo processar pequenas quantidades de tecido, como espécimes de biópsia hepática. Em princípio, é possível utilizar a homogeneização direta dos tecidos sem etapas de trituração; no entanto, a homogeneização tecidual sem pulverização prévia tem uma reprodutibilidade pior em nossa experiência e o aparecimento de problemas para aplicações a jusante foi maior (dados não mostrados).
A maioria dos problemas encontrados pela preparação da cromatina a partir de tecidos deriva da natureza dessas amostras e da incapacidade de verificar adequadamente se os aglomerados celulares são pequenos o suficiente para a fixação sem perder a qualidade. Além disso, a verificação de cada alíquota em cada etapa seria demorada, aumentando a chance de degradação proteica.
A fixação (passo 3.9) é uma parte fundamental e crucial da preparação da cromatina. Devido à natureza do tecido, a etapa de fixação foi adiada até que o tecido fosse homogeneizado. Esta etapa de fixação postergada tem a vantagem de produzir uma suspensão celular mais homogênea. No entanto, reconhecemos que, no caso de alvos particularmente sensíveis à manipulação, pode ser necessário realizar a fixação imediatamente antes do passo 3.6. Isso ajudaria a proteger proteínas extremamente sensíveis ou PTMs, embora possa aumentar o tamanho dos aglomerados celulares, que quando fixados podem resultar em cisalhamento não homogêneo. A concentração da solução de AF utilizada no protocolo é padrão, porém pode ser modificada para tentar melhorar a fixação global. O tempo de fixação aqui escolhido também reflete as condições padrão comumente usadas no campo. Em caso de maior concentração da solução fixadora, o tempo de fixação pode ser reduzido, enquanto em caso de menor quantidade deve ser aumentado. O operador deve considerar que uma alteração do tempo de fixação pode conduzir a uma sobrefixação da amostra ou dar espaço à degradação das proteínas. No caso de precipitar grandes complexos (ou parte deles) e FTs, seria vantajoso realizar uma fixação em duplo passo com uma solução de DSG seguida de uma solução de AG25,26. O DSG, nesse caso, estabilizaria as interações proteína-proteína, enquanto o formaldeído atua principalmente nas interações diretas DNA-proteína27.
O operador deve ter em conta a possibilidade de implementar um kit baseado em colunas para purificação do ADN a partir do passo 6.7, que é mais rápido e não utiliza compostos tóxicos. No entanto, sempre haverá uma certa quantidade de DNA não ligado que será perdida. Por esta razão, sugerimos o uso da extração clássica fenol-clorofórmio seguida de precipitação de EtOH. Além disso, antes de executar o gel de agarose (passo 7.2) pode ser benéfico medir a concentração de DNA e carregar a mesma quantidade para cada poço para ter uma imagem mais clara.
Uma limitação deste protocolo decorre do fato de que exploramos e utilizamos este protocolo apenas utilizando espécimes hepáticos derivados de camundongos quiméricos humano-hepáticos28. Per se, o fígado é constituído por tecido epitelial e conjuntivo29. Em caso de doença, tecido fibrótico e tecido adiposo podem estar presentes30,31, criando desafios adicionais durante a ruptura tecidual. No entanto, reconhecemos que nosso protocolo não pode ser utilizado em tecido ósseo, muscular e adiposo sem otimização das etapas de dissociação e sonicação. Ressalta-se que todo tecido necessita de algum tipo de otimização devido à ausência de um protocolo adequado para todos eles, como para amostras de cultura celular15. Acreditamos, no entanto, que com pouca ou nenhuma otimização, esse protocolo poderia ser aplicado com sucesso a outros tecidos que compartilham semelhanças com o fígado em composição, como pulmão, intestino, estômago, pâncreas ou tecidos renais.
Nosso protocolo também tem sido usado com sucesso para analisar FTs e modificações de histonas no ecrossoma de DNA covalentemente fechado do HBV (cccDNA)32. Isso abre a chance de aplicar tal abordagem para outros genomas virais que afetam o fígado, como o citomegalovírus33 humano (hCMV) e o adenovírus humano34 (HAdV). Não está excluído que seria possível analisar outros vírus de DNA que estabelecem uma infecção persistente em outros tecidos como Kaposi Sarcoma Herpes Virus35 (KHSV), Herpes Simplex Virus 36 (HSV1/2) Polyoma viruses, Epstein-Barr Virus37 (EBV).
Divulgações
Os autores não têm nada a revelar.
Agradecimentos
O estudo foi apoiado pela Fundação Alemã de Pesquisa (DFG) por uma bolsa para Maura Dandri (SFB 841 A5) e pelo Estado de Hamburgo com o Programa de Pesquisa (LFF-FV44: EPILOG).
Agradecemos ao Dr. Tassilo Volz, Yvonne Ladiges e Annika Volmari pela ajuda técnica e pela leitura crítica do manuscrito. Dr. Thomas Günther e Prof. Adam Grundhoff por fornecerem sugestões muito úteis e os conjuntos de primers para a análise de ChIP-qPCR.
Materiais
| Name | Company | Catalog Number | Comments |
| 0.22µm sterile syringe filter | Labsolute | 7699822 | |
| 1.5 mL Safeseal tubes | Sarstedt | 7,27,06,400 | |
| 6x orange loading dye | Thermofisher | R0631 | |
| Benchtop refrigerated centrifuge | |||
| Bioruptor NGS | Diagenode | ||
| Blade or Scalpel | |||
| Calcium chloride dihydrate | Carl Roth | HN04 | |
| Chloroform | Sigma Aldrich (Merck) | C2432 | |
| cOmplete Protease Inhibitor Cocktail | Roche | 11697498001 | |
| Deacetylase Inhibitor | Active Motif | 37494 | |
| Dounce tissue grinder set | Sigma Aldrich (Merck) | DWK885300-0001-1EA | |
| EDTA 500 mM solution | PanReac AppliChem | A4892 | |
| EGTA | Sigma Aldrich (Merck) | E4378 | |
| EtBr | Carl Roth | 2218 | Concentration 10mg/mL |
| Ethanol absolute | CHEMSOLUTE | 2273 | |
| Glycerol | Sigma Aldrich (Merck) | G9012 | |
| Glycin | Carl Roth | 0079 | |
| Glycogen | Roche | 10901393001 | Concentration: 20mg/mL |
| Heating block | |||
| HEPES | Sigma Aldrich (Merck) | H4034 | |
| LE Agarose | Biozym | 840000 | |
| Liquid nitrogen cooled mini mortar | Bel-Art | H37260-0100 | |
| MeOH free Formaldehyde 16% | Thermofisher | 28908 | |
| NP-40 | Roche | 11332473001 | |
| PBS 1x | Thermofisher | 10010015 | |
| Pefabloc SC-Protease-Inhibitor | Sigma Aldrich (Merck) | 11429868001 | |
| Phase Lock Gel - Heavy | QuantaBio | 2302830 | |
| Phenol:Chloroform:Isoamyl alcohol 25:24:1 | Sigma Aldrich (Merck) | P3803 | |
| Potassium chloride | Carl Roth | 6781 | |
| Potassium hydroxyde | Merck | 105033 | |
| Proteinase K | Lucigen | MPRK092 | Concentration: 50 µg/µL |
| RNAse A | Lucigen | MRNA092 | Concentration: 5 mg/mL |
| SDS 10% solution | PanReac AppliChem | A3950 | |
| Sodium carbonate anhydrous | Carl Roth | A135 | |
| Sodium chloride | Sigma Aldrich (Merck) | S7653 | |
| Sterile Petri dishes | Sarstedt | 83,39,02,500 | |
| Tris-HCl solution | Sigma Aldrich (Merck) | T2694 | |
| Triton-X100 | Sigma Aldrich (Merck) | X100 |
Referências
- Waddington, C. H., Pantelouris, E. M. Transplantation of nuclei in newt's eggs. Nature. 172 (4388), 1050-1051 (1953).
- Silmon de Monerri, N. C., Kim, K. Pathogens hijack the epigenome: A new twist on host-pathogen interactions. American Journal of Pathology. 184 (4), 897-911 (2014).
- Knipe, D. M., et al. Snapshots: chromatin control of viral infection. Virology. 435 (1), 141-156 (2013).
- Tropberger, P., et al. Mapping of histone modifications in episomal HBV cccDNA uncovers an unusual chromatin organization amenable to epigenetic manipulation. Proceedings of the National Academy of Sciences of the United States of America. 112 (42), 5715-5724 (2015).
- Sproul, D., Gilbert, N., Bickmore, W. A. The role of chromatin structure in regulating the expression of clustered genes. Nature Reviews Genetics. 6 (10), 775-781 (2005).
- Fischle, W., Wang, Y., Allis, C. D. Histone and chromatin cross-talk. Current Opinion in Cell Biology. 15 (2), 172-183 (2003).
- Ling, X., Harkness, T. A., Schultz, M. C., Fisher-Adams, G., Grunstein, M. Yeast histone H3 and H4 amino termini are important for nucleosome assembly in vivo and in vitro: redundant and position-independent functions in assembly but not in gene regulation. Genes & Development. 10 (6), 686-699 (1996).
- Zhang, L., Eugeni, E. E., Parthun, M. R., Freitas, M. A. Identification of novel histone post-translational modifications by peptide mass fingerprinting. Chromosoma. 112 (2), 77-86 (2003).
- Wang, H., et al. Role of histone H2A ubiquitination in Polycomb silencing. Nature. 431 (7010), 873-878 (2004).
- Hassa, P. O., Haenni, S. S., Elser, M., Hottiger, M. O. Nuclear ADP-ribosylation reactions in mammalian cells: where are we today and where are we going. Microbiology and Molecular Biology Reviews. 70 (3), 789-829 (2006).
- Dey, B., et al. DNA-protein interactions: methods for detection and analysis. Molecular and Cellular Biochemistry. 365 (1-2), 279-299 (2012).
- Hager, G. L., McNally, J. G., Misteli, T. Transcription dynamics. Molecular Cell. 35 (6), 741-753 (2009).
- Nagy, Z., Tora, L. Distinct GCN5/PCAF-containing complexes function as co-activators and are involved in transcription factor and global histone acetylation. Oncogene. 26 (37), 5341-5357 (2007).
- Solomon, M. J., Larsen, P. L., Varshavsky, A. Mapping protein-DNA interactions in vivo with formaldehyde: evidence that histone H4 is retained on a highly transcribed gene. Cell. 53 (6), 937-947 (1988).
- Gunther, T., Theiss, J. M., Fischer, N., Grundhoff, A. Investigation of viral and host chromatin by ChIP-PCR or ChIP-Seq analysis. Current Protocols in Microbiology. 40, 11-21 (2016).
- Cotney, J. L., Noonan, J. P. Chromatin immunoprecipitation with fixed animal tissues and preparation for high-throughput sequencing. Cold Spring Harbor Protocols. 2015 (4), 419 (2015).
- Haim, Y., Tarnovscki, T., Bashari, D., Rudich, A. A chromatin immunoprecipitation (ChIP) protocol for use in whole human adipose tissue. American Journal of Physiology-Endocrinology and Metabolism. 305 (9), 1172-1177 (2013).
- Castellano-Castillo, D., et al. Chromatin immunoprecipitation improvements for the processing of small frozen pieces of adipose tissue. PLoS One. 13 (2), 0192314 (2018).
- Savic, D., Gertz, J., Jain, P., Cooper, G. M., Myers, R. M. Mapping genome-wide transcription factor binding sites in frozen tissues. Epigenetics Chromatin. 6 (1), 30 (2013).
- Perna, A., Alberi, L. A. TF-ChIP method for tissue-specific gene targets. Frontiers Cell Neuroscience. 13, 95 (2019).
- Allweiss, L., et al. Proliferation of primary human hepatocytes and prevention of hepatitis B virus reinfection efficiently deplete nuclear cccDNA in vivo. Gut. 67 (3), 542-552 (2018).
- Lerdrup, M., Johansen, J. V., Agrawal-Singh, S., Hansen, K. An interactive environment for agile analysis and visualization of ChIP-sequencing data. Nature Structural & Molecular Biology. 23 (4), 349-357 (2016).
- Perna, A., Alberi, L. A. TF-ChIP Method for Tissue-Specific Gene Targets. Frontiers in Cellular Neuroscience. 13, 95 (2019).
- Liang, N., Fan, R., Goni, S., Treuter, E. Preparation of Frozen Liver Tissues for Integrated Omics Analysis. Methods in Molecular Biology. 1951, 167-178 (2019).
- Liu, Z., et al. Proteomic and network analysis of human serum albuminome by integrated use of quick crosslinking and two-step precipitation. Scientific Reports. 7 (1), 9856 (2017).
- Singh, A. A., et al. Optimized ChIP-seq method facilitates transcription factor profiling in human tumors. Life Science Alliance. 2 (1), 201800115 (2019).
- Aoki, T., et al. Bi-functional cross-linking reagents efficiently capture protein-DNA complexes in Drosophila embryos. Fly. 8 (1), 43-51 (2014).
- Allweiss, L., Dandri, M. Experimental in vitro and in vivo models for the study of human hepatitis B virus infection. Journal of Hepatology. 64, 17-31 (2016).
- Krishna, M. Microscopic anatomy of the liver. Clinics in Liver Disease. 2, 4-7 (2013).
- Tannapfel, A., et al. Histopathological diagnosis of non-alcoholic and alcoholic fatty liver disease. Virchows Archiv. 458 (5), 511-523 (2011).
- Schuppan, D., Afdhal, N. H. Liver cirrhosis. Lancet. 371 (9615), 838-851 (2008).
- Allweiss, L., et al. Therapeutic shutdown of HBV transcripts promotes reappearance of the SMC5/6 complex and silencing of the viral genome in vivo. Gut. , 322571 (2021).
- Gerna, G., Kabanova, A., Lilleri, D. Human cytomegalovirus cell tropism and host cell receptors. Vaccines. 7 (3), (2019).
- Echavarria, M. Adenoviruses in immunocompromised hosts. Clinical Microbiology Reviews. 21 (4), 704-715 (2008).
- Frohlich, J., Grundhoff, A. Epigenetic control in Kaposi sarcoma-associated herpesvirus infection and associated disease. Seminars in Immunopathology. 42 (2), 143-157 (2020).
- Nicoll, M. P., Proenca, J. T., Efstathiou, S. The molecular basis of herpes simplex virus latency. FEMS Microbiology Reviews. 36 (3), 684-705 (2012).
- Thorley-Lawson, D. A., Hawkins, J. B., Tracy, S. I., Shapiro, M. The pathogenesis of Epstein-Barr virus persistent infection. Current Opinion in Virology. 3 (3), 227-232 (2013).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados