Method Article
Chromatinextraktion aus gefrorenem chimärem Lebergewebe für die Chromatin-Immunpräzipitationsanalyse
In diesem Artikel
Zusammenfassung
Dieses Protokoll konzentriert sich auf die Chromatinpräparation aus tiefgefrorenem Gewebe und eignet sich für die Vernetzung der Chromatin-Immunpräzipitation (X-ChIP), gefolgt von einer quantitativen PCR-Analyse (X-ChIP-qPCR) oder einem Next-Generation-Sequencing-Ansatz (X-ChIP-seq).
Zusammenfassung
Die Crosslinking Chromatin Immunpräzipitation (X-ChIP) ist eine weit verbreitete Technik zur Beurteilung des Niveaus von Histonmarkierungen und der Belegung von Transkriptionsfaktoren auf dem Wirts- und/oder Pathogenchromatin. Die Chromatinpräparation aus Geweben stellt zusätzliche Herausforderungen dar, die überwunden werden müssen, um reproduzierbare und zuverlässige Protokolle zu erhalten, die mit denen für die Zellkultur vergleichbar sind. Gewebeauflösung und Fixierung sind entscheidende Schritte, um eine effiziente Scherung des Chromatins zu erreichen. Die Koexistenz verschiedener Zelltypen und Cluster kann auch unterschiedliche Scherzeiten erfordern, um eine optimale Fragmentgröße zu erreichen, und behindert die Reproduzierbarkeit der Scherung. Ziel dieser Methode ist es, zuverlässige und reproduzierbare Wirtschromatinpräparationen aus gefrorenem Gewebe (Leber) zu erhalten, die sowohl für ChIP-qPCR- als auch für Next Generation Sequencing (NGS)-Anwendungen geeignet sind. Wir beobachteten, dass die Kombination von Flüssigstickstoff-Gewebepulverisierung mit anschließender Homogenisierung zu einer erhöhten Reproduzierbarkeit im Vergleich zur reinen Homogenisierung führt, da sie eine Suspension liefert, die hauptsächlich aus dissoziierten Einzelzellen besteht, die effizient geschert werden können. Darüber hinaus sollte der Fixierungsschritt unter leichter Rotation durchgeführt werden, um eine homogene Vernetzung zu gewährleisten. Das fixierte Material eignet sich dann für die Isolierung von Kernen auf Pufferbasis, um die Kontamination von zytoplasmatischen Proteinen und pathogenen DNAs und RNAs (falls zutreffend) zu reduzieren und zeitaufwändige Zentrifugationsgradienten zu vermeiden. Die anschließende Beschallung schließt die Kernlyse ab und schert das Chromatin, wodurch ein spezifischer Größenbereich entsprechend den gewählten Scherbedingungen entsteht. Der Größenbereich sollte für NGS-Anwendungen zwischen 100 und 300 nt liegen, während er für die ChIP-qPCR-Analyse höher sein könnte (300-700 nt). Solche Protokollanpassungen können die Chromatinanalyse von gefrorenen Gewebeproben erheblich verbessern.
Einleitung
Seit ihrer Entdeckung hat die epigenetische Regulation in Säugetierzellenzunehmend an Bedeutung gewonnen1, da das Verständnis solcher Mechanismen nicht nur wichtige Erkenntnisse für die Zellbiologie, sondern auch für die Krankheits- und Tumorbiologie liefern würde. Darüber hinaus können Infektionserreger auch epigenetische Veränderungen des Wirts verursachen2, während die Wirtszellmaschinerie auch das Chromatin von Krankheitserregern, wie z. B. persistierenden DNA-Viren, beeinflussen kann 3,4. Dieses Wirt-Pathogen-Wechselspiel scheint eine Rolle bei der Persistenz von Infektionen zu spielen. arabische Ziffer
Durch eine reversible Assoziation mit der DNA bilden Histonproteine einen Komplex, der als Nukleosom bezeichnet wird. Nukleosomen erreichen ihrerseits eine höhere Organisationsebene, die als Chromatin bekannt ist. Es ist bekannt, dass der Chromatin-Umbau die Genexpression stark reguliert und den Zugang zu Transkriptionsfaktoren (TFs) gewährt oder verweigert5. Diese Faktoren können die Rekrutierung der RNA-Polymerase II (PolII) auf Genpromotoren entweder auslösen oder blockieren und so die mRNA-Synthese aus dem DNA-Templatebeeinflussen 6. Histonproteine enthalten Schwänze7, die beide Enden der Histonfaltung flankieren, die posttranslationalen Modifikationen (PTMs) unterliegen können, was eine enge Regulierung der Gentranskription durch strukturelle Chromatinänderungen ermöglicht. Die meisten Histon-PTMs befinden sich am Schwanz-N-Terminus, wobei Acetylierung und Methylierung die am besten untersuchten PTMs sind, obwohl auch Phosphorylierung8, Ubiquitinierung9 und Ribosylierung10 beschrieben wurden. Die Charakterisierung und Untersuchung solcher Proteine ist dann unerlässlich, um einen tiefen Einblick in die Genregulation zu erhalten.
Derzeit gibt es eine Handvoll etablierter Methoden und Werkzeuge, um direkte DNA-Protein-Interaktionen zu untersuchen: Electrophoretic Mobility Shift Assay (EMSA), Yeast One-Hybrid Assay (Y1H) und DNA Footprinting11. Diese Methoden konzentrieren sich jedoch per se auf einzelne DNA-Protein-Interaktionen und sind nicht für genomweite Studien anwendbar. Eine weitere Einschränkung dieser Techniken ist die fehlende Histon-Assoziation mit den untersuchten DNA-Abschnitten. Daher sind solche Ansätze nicht dazu gedacht, die Komplexität der Transkriptionsmaschinerie in vivo widerzuspiegeln, und sie berücksichtigen keine wichtigen strukturellen Veränderungen12 oder andere erforderliche Enzyme/Cofaktoren13 , die die Proteinbindung an die DNA beeinflussen (entweder fördern oder hemmen) könnten.
Die Idee, dass die Fixierung von Zellen mit Wirkstoffen wie Formaldehyd (FA) eine In-vivo-Momentaufnahme von Protein-DNA-Interaktionen liefern könnte, schuf die Grundlage für die Entwicklung von Chromatin-Immunpräzipitations-Assays (ChIP)14. Zusammen mit der Verfügbarkeit der quantitativen PCR-Technologie (qPCR) und hochspezifischer Antikörper ermöglichte dies die Entwicklung von ChIP-qPCR-Assays. In der Folge ermöglichte das Aufkommen von Next-Generation-Sequencing-Techniken (NGS), deren Kosten erschwinglicher werden, die Kopplung von ChIP-Experimenten mit NGS-Ansätzen (ChIP-seq), wodurch den Forschern neue leistungsfähige Werkzeuge zur Verfügung gestellt wurden, mit denen die Chromatinregulation untersucht werden kann. In diesen Assays werden isolierte oder kultivierte Zellen mit Disuccinimidylglutarat (DSG) und/oder FA fixiert, Zellkerne werden isoliert, das Chromatin wird dann fragmentiert und durch den zu untersuchenden Antikörper ausgefällt. Anschließend wird die DNA gereinigt und mittels PCR- oder NGS-Ansätzen analysiert. Im Gegensatz zu EMSA, Y1H und DNA-Footprinting sind ChIP-Assays in der Lage, eine globale Momentaufnahme der Protein-DNA-Interaktion innerhalb der Zelle zu liefern. Dies bietet Flexibilität und ermöglicht die Analyse mehrerer Loci innerhalb derselben Probe. Aufgrund der Art des Assays kann ChIP jedoch letztendlich nicht nur direkte, sondern auch indirekte Wechselwirkungen erkennen, die nicht die Präzision der oben genannten Methoden bieten, wenn es um direkte Protein-DNA-Interaktionen geht.
Chromatinpräparationsprotokolle aus Zellkulturmaterial sind gut etabliert15 und hochgradig reproduzierbar, so dass der Benutzer in 1-2 Arbeitstagen Chromatin erhalten kann, das sowohl für qPCR- als auch für NGS-Ansätze geeignet ist. Die Gewinnung von qualitativ hochwertigem Chromatin aus ganzen Geweben stellt jedoch immer noch eine Herausforderung dar, da die Zellen innerhalb des Gewebes dissoziiert werden müssen und gleichzeitig eine optimale Fixierung und Scherung des Chromatins erreicht werden muss. Darüber hinaus variieren die Zusammensetzung und Morphologie der verschiedenen Gewebetypen, so dass eine Anpassung bestehender Protokolle erforderlich ist16,17. Die Verwendung von kryokonserviertem Gewebe bietet im Vergleich zu frischen Proben zusätzliche Herausforderungen. Dies liegt an der Schwierigkeit, eine Einzelzellensuspension ohne großen Materialverlust zu erhalten. Dies führt zu unsachgemäßem Scheren und behindert nachgelagerte Anwendungen. Nichtsdestotrotz erhöht der Zugriff auf gefrorene Gewebeproben anstelle des frischen Gegenstücks nicht nur die Arbeitsflexibilität, sondern kann auch die einzige Option für Forscher darstellen, die mit Proben arbeiten, die aus Längsschnitt- oder Vergleichsstudien stammen. Eine Handvoll Protokolle zur Chromatinpräparation für gefrorenes Gewebe wurden veröffentlicht. Diese basieren meist auf dem Auftauen der Probe mit anschließendem Zerkleinern, manueller/maschineller Dissoziation oder Pulverisierung mit flüssigem Stickstoff18,19,20.
In dieser Arbeit beschreiben wir eine optimierte Chromatinpräparationsmethode15 für gefrorene, nicht fixierte Leberproben, die die Gewebepulverisierung in flüssigem Stickstoff mit der Homogenisierung des Stößels kombiniert, um eine reproduzierbare Chromatinscherung zu erreichen, die für X-ChIP-Ansätze geeignet ist, die darauf abzielen, sowohl virale als auch Wirtsgenome zu analysieren.
Protokoll
Die Gewebeentnahme von chimären Mäusen der menschlichen Leber21 wurde in Übereinstimmung mit der Richtlinie 86/609/EWG der Europäischen Union durchgeführt und von der Ethikkommission der Stadt und des Landes Hamburg gemäß den Grundsätzen der Deklaration von Helsinki genehmigt.
1. Vorbereitung der Reagenzien
- 1,25 M Glycinlösung in deionisiertem Wasser herstellen. Sterilfilter mit 0,22 μm porengroßem Filter. Bei 4 °C lagern.
- Bereiten Sie 5 M Natriumchlorid (NaCl)-Lösung vor. Bei Raumtemperatur lagern.
- CaCl2-Lösung vorbereiten: 300 mM CaCl2 und 10 mM Tris-HCl pH 8 in deionisiertem Wasser. Sterilfilter mit 0,22 μm porengroßem Filter und bei RT lagern.
- Bereiten Sie eine 10%ige Triton X-100-Verdünnung in deionisiertem Wasser vor. Bei RT speichern.
- Tris-EDTA-Puffer vorbereiten: 1 mM EDTA und 10 mM Tris pH 8 in deionisiertem Wasser. Bei 4 °C lagern.
- Bereiten Sie je nach benötigter Menge folgende Puffer vor:
- Puffer A: 50 mM Hepes-KOH pH 7,5, 140 mM NaCl, 1 mM Ethylendiamintetraessigsäure (EDTA), 10 % Glycerin, 0,5 % NP-40 und 0,25 % Triton X-100 in deionisiertem Wasser. Sterilfilter mit 0,22 μm porengroßem Filter. Bei 4 °C lagern.
- Bereiten Sie Puffer B vor: 10 mM Tris-HCl pH 8, 200 mM NaCl, 1 mM EDTA, 0,5 mM Egtazinsäure (EGTA). Sterilfilter mit 0,22 μm porengroßem Filter. Bei 4 °C lagern.
- Bereiten Sie Puffer C: 1 % SDS, 10 mM EDTA und 50 mM Tris-HCl pH 8 in deionisiertem Wasser vor. Sterilfilter mit 0,22 μm porengroßem Filter. Bei RT speichern.
- Chromatin-Verdünnungspuffer vorbereiten: 0,01 % SDS, 1,1 % Triton X-100, 1,2 mM EDTA, 16,6 mM Tris-HCl pH 8 und 166 mM NaCl in deionisiertem Wasser. Sterilfilter mit 0,22 μm porengroßem Filter. Bei 4 °C lagern.
2. Materialvorbereitung
- Sammle Trockeneis, Eis und flüssigen Stickstoff.
VORSICHT: Behandeln Sie Trockeneis und flüssigen Stickstoff mit der notwendigen Sorgfalt, um Verbrennungen zu vermeiden. - Die Zentrifuge auf 4 °C vorkühlen.
HINWEIS: Dieser Schritt ist wichtig, um einen Proteinabbau und eine De-Vernetzung während der Waschschritte zu vermeiden, da dies die Qualität des Chromatins verringert. - Stellen Sie einen sterilen Teller auf Trockeneis und lassen Sie ihn abkühlen.
Anmerkungen: Stellen Sie sicher, dass die Platte groß genug ist, um den Schneidevorgang zu erleichtern. Eine 100 mm Petriplatte/Zellkulturschale wird empfohlen. - Nehmen Sie das erforderliche Aliquot von Glycin 1,25 M heraus und lassen Sie es RT erreichen.
- Nehmen Sie die notwendigen Aliquots von Puffer A, B und PBS heraus. Fügen Sie Protease und/oder Deacetylase- und Phosphatase-Hemmer hinzu, um eine 1-fache Konzentration zu erreichen, und lassen Sie sie auf Eis.
- Nehmen Sie das notwendige Aliquot von Puffer C heraus und belassen Sie es bei RT. Fügen Sie die Protease- und/oder Deacetylase- und Phosphatase-Hemmer erst nach Angabe hinzu.
- Nehmen Sie das erforderliche Aliquot von RT PBS heraus.
VORSICHT: Puffer C enthält Natriumdodecylsulfat (SDS). Ergreifen Sie bei der Vorbereitung des Puffers die entsprechenden Sicherheitsmaßnahmen.
HINWEIS: SDS fällt auf Eis aus, und Protease- und Deacetylase-Inhibitoren sind bei RT nicht stabil. - Kühlen Sie den Mörtel vor, indem Sie flüssigen Stickstoff in seine Kammer gießen, und befolgen Sie dabei strikt die Anweisungen des Lieferanten. Den Metallstößel in Trockeneis mindestens 5 Minuten abkühlen lassen.
HINWEIS: Es ist möglich, einen alternativen Mörtel als den vorgeschlagenen zu verwenden. Das in diesem Protokoll verwendete Gerät ermöglicht jedoch aufgrund seiner besonderen Konstruktion die Arbeit mit kleinen Gewebemengen ohne erheblichen Verlust während des Pulverisierungsprozesses. - Kühlen Sie den Dounce-Homogenisator mit dem zugehörigen Stößel A auf Eis vor.
Anmerkungen: Stößel A hat eine lockere Passform mit dem Homogenisator. Dies ermöglicht es, eine Einzelzellsuspension ohne nennenswerte Zelllyse zu erhalten.
3. Vernetzung des Gewebes
- Schneiden Sie mit Hilfe eines Skalpells und einer Pinzette etwa 50 mg gefrorenes Gewebe direkt auf die Schale auf Trockeneis.
Anmerkungen: Es wird empfohlen, das Skalpell auf RT zu halten, da dies den Schneidevorgang erleichtert. Vermeiden Sie es, zu viel Druck auf das Skalpell auszuüben, da dies die Gefahr erhöht, dass Gewebestücke außerhalb des Schneidebereichs verstreut werden. Zu beachten ist, dass 50 mg Gewebe (in diesem Fall Leber) etwa 5 Millionen Zellen ergeben sollten. Beachten Sie, dass die warme Klinge die Schneide auftaut. In Anbetracht der relativ großen Größe des Gewebestücks sollte dies jedoch nur eine begrenzte Wirkung haben. Wenn kleinere Stücke geschnitten werden, kann es von Vorteil sein, ein kaltes Skalpell zu verwenden, um ein Streuen des Gewebes zu vermeiden. - Legen Sie das geschnittene Gewebe in ein 1,5-ml-Röhrchen, das bereits auf Trockeneis gekühlt ist. Vermeiden Sie das Auftauen von Gewebe.
- Bewegen Sie das Röhrchen mit dem Gewebe in den Mörser und lassen Sie es dort 5 Minuten einwirken.
Anmerkungen: Wenn Sie die Probe im Mörtel ruhen lassen, sinkt ihre Temperatur (von -80 °C auf -196 °C). Dies erhöht die Zähigkeit und erleichtert den Pulverisierungsschritt. - Üben Sie mit Hilfe des vorgekühlten Stößels Druck auf die Probe aus, bis keine festen Krümel mehr sichtbar sind.
Anmerkungen: Es ist wichtig, eine Erwärmung des Stößels durch übermäßige Rotationskräfte zu vermeiden, da dies die Probe auftaut. Reinigen Sie den Stößel nach jeder Probenzerkleinerung mit 70%igem Ethanol (EtOH) und lassen Sie ihn erneut auf Trockeneis abkühlen. - Nehmen Sie das Röhrchen mit der Probe aus dem Mörser und fügen Sie 950 μl eiskaltes PBS mit den erforderlichen Inhibitoren hinzu. Pipettieren Sie vorsichtig auf und ab, bis die Probe vollständig resuspendiert ist. Fahren Sie sofort mit Schritt 3.6 fort.
- Übertragen Sie die Gewebesuspension in den Homogenisator und wenden Sie 20-30 Striche mit Stößel A an, um eine feinere Suspension zu erhalten. Vermeiden Sie Schaumbildung.
HINWEIS: Die Anzahl der Schläge sollte entsprechend der Gewebekonsistenz optimiert werden. Dieser Schritt dissoziiert weiter die kleinen Zellcluster, die nach der Pulverisierung erhalten werden. Eine unsachgemäße Homogenisierung kann die Schereffizienz beeinträchtigen. - Übertragen Sie das Homogenat in ein neues 1,5-ml-Röhrchen, das bereits auf Eis vorgekühlt ist.
- 5 min bei 1.300 x g bei 4 °C zentrifugieren und den Überstand vorsichtig entfernen.
- Resuspendieren Sie das Pellet vollständig in 950 μl RT PBS durch vorsichtiges Pipettieren und fügen Sie 63,6 μl 16 % MeOH-freies FA hinzu, um eine Endkonzentration von 1 % zu erreichen. Fahren Sie sofort mit Schritt 3.10 fort.
ACHTUNG: FA ist eine giftige Chemikalie. Behandeln Sie es unter einem Abzug mit geeigneten Sicherheitsmaßnahmen.
HINWEIS: Eine unvollständige Resuspension kann während des Fixierungsschritts eine Zellaggregation hervorrufen. Dies behindert die Lyse und den Scherprozess. - Drehen Sie 10 Minuten bei RT. Fahren Sie dann sofort mit Schritt 3.11 fort
Anmerkungen: Eine Rotation ist notwendig, um Aggregate zu vermeiden. Die für die Fixierung erforderliche Zeit sollte je nach Zielobjekt und Probentyp optimiert werden. Es ist wichtig zu beachten, dass übermäßige Fixierzeiten das ordnungsgemäße Scheren behindern können. - Fügen Sie 113 μl 1,25 M Glycin bei RT hinzu, um eine Endkonzentration von 125 mM zu erhalten, und drehen Sie sie 5 Minuten lang.
Anmerkungen: Glycin löscht die Fixierreaktion ab und vermeidet eine Übervernetzung. - Zentrifugieren bei 1.300 x g für 3 min bei 4 °C.
- Verwerfen Sie den Überstand und resuspendieren Sie das Pellet vorsichtig, indem Sie 950 μl eiskaltes PBS mit den erforderlichen Inhibitoren pipettieren.
- Zentrifugieren bei 1.300 x g für 3 min bei 4 °C.
- Wiederholen Sie die Schritte 3.13-3.14 und fahren Sie sofort mit den Schritten zur Chromatinisolierung fort.
4. Chromatin-Isolierung
- Fügen Sie dem Pellet 950 μl Puffer A mit den erforderlichen Inhibitoren hinzu. Vorsichtig durch Pipettieren mischen, bis das Pellet vollständig resuspendiert ist, und 10 min bei 4 °C drehen.
HINWEIS: In diesem Schritt wird die fixierte Einzelzellsuspension ohne Zelllyse lysiert. Dies ermöglicht es, die Probe von zytosolischen Proteinen und RNAs zu befreien. Eine Verlängerung der Lysezeit könnte für schwer zu lysierende Zellen von Vorteil sein und die Handhabungszeit des Gewebes erhöhen. An dieser Stelle ist es möglich, die Präparation nach der Trypanblau/DAPI-Färbung unter dem Mikroskop zu überprüfen, um die Größe der Cluster und das Vorhandensein einzelner Zellen zu überprüfen. Die einzelnen Zellkerne sind jedoch aufgrund des fixierten Gewebematerials möglicherweise nicht leicht zu erkennen. - Bei 2.000 x g 5 min bei 4 °C zentrifugieren und den Überstand vorsichtig entfernen.
- Fügen Sie dem Pellet 950 μl Puffer B mit den erforderlichen Inhibitoren hinzu. Vorsichtig durch Pipettieren mischen, bis das Pellet vollständig resuspendiert ist, und 10 min bei 4 °C drehen.
HINWEIS: Bei diesem Schritt wird der Lysepuffer aus der Kernpräparation ausgewaschen, um eine weitere unerwünschte Lyse zu vermeiden. - Zentrifugieren bei 2000 x g für 5 min bei 4 °C. In der Zwischenzeit fügen Sie die erforderlichen Inhibitoren (wie in Schritt 2.5) hinzu, um C zu puffern.
- Entfernen Sie vorsichtig den Überstand.
- Geben Sie 300 μl RT-Puffer C in das Pellet und pipettieren Sie kräftig.
- Wirbeln Sie die Probe für 15-30 s und drehen Sie das Röhrchen kurz, um die Tropfen auf dem Deckel aufzufangen.
HINWEIS: Dieser Schritt ist wichtig, um die fixierten Kerne zu befreien und zu lysieren. Um die Integrität der Probe zu erhalten und gleichzeitig SDS-Fällungen zu vermeiden, bewahren Sie die Probe vor der Beschallung in einem Kunststoffgestell auf, das auf Eis gehalten wird, um eine Temperatur von 9-11 °C aufrechtzuerhalten.
5. Chromatin-Fragmentierung
- Übertragen Sie die Probe in drei saubere, 0,65-ml-Röhrchen, die für die Beschallung zertifiziert sind, und stellen Sie sicher, dass 100 μl lysierte Zellkernsuspension pro Röhrchen vorhanden sind.
HINWEIS: Es ist möglich, 1,5-ml-Ultraschallröhrchen mit einem maximalen Volumen von 300 μl zu verwenden. Für diese Röhren wird eine spezielle Halterung benötigt. 0,65 ml sollten aufgrund des geringeren Volumens der Probe pro Röhrchen eine homogenere Scherung ermöglichen. - Beschallen Sie das Chromatin 28 Zyklen lang bei hoher Intensität mit den Einstellungen 30 s EIN und 30 s AUS. Stellen Sie sicher, dass das Ultraschallbad ordnungsgemäß gekühlt ist (Eis oder Kühlgerät).
HINWEIS: Dieser Schritt muss in fast jedem Fall optimiert werden. Der Benutzer sollte bedenken, dass mit zunehmender Scherzeit kleinere und homogenere Fragmente entstehen. Dies kann jedoch die Wahrscheinlichkeit erhöhen, dass die Chromatinqualität sinkt. Wählen Sie die niedrigste Anzahl von Zyklen, die die erforderliche Fragmentgröße bereitstellt. Bei der Optimierung dieses Schritts ist es sinnvoll, eine Kernfärbung durchzuführen, um zu überprüfen, ob die Anzahl der Zyklen ausreicht, um die Mehrheit der Kerne zu lysieren. - Übertragen Sie das beschallte Chromatin in ein neues 1,5-ml-Röhrchen, das zuvor auf Eis gekühlt wurde.
- Fügen Sie 30 μl Triton X-100 10%ige Lösung hinzu und wirbeln Sie 5-10 s lang.
HINWEIS: Triton X-100 bindet das SDS und verhindert weitere Niederschläge bei 4 °C. Die Endmenge von Triton X-100 sollte immer 1% betragen. - Zentrifugieren bei 16.000 x g für 15 min bei 4 °C.
- Übertragen Sie den Überstand in ein sauberes 1,5-ml-Röhrchen, das auf Eis vorgekühlt ist.
- HINWEIS: Der Überstand enthält das geschorene Chromatin und sollte klar erscheinen. Das Pellet enthält "nicht scherbare" Reste und sollte recht klein bleiben (bei Lebergewebe meist braun). Achten Sie auf Anzeichen für eine erfolglose Scherung: Chromatinlösung, die nicht klarer geworden ist, und ähnliche Pelletabmessungen wie in Schritt 4.5.
6. DNA-Reinigung
- Übertragen Sie 10-25 μl geschertes Chromatin in ein neues Röhrchen und fügen Sie Puffer C hinzu, um ein Endvolumen von 200 μl zu erreichen. Lagern Sie den Rest des Chromatins bis zur weiteren Verwendung bei -80 °C. Bei Bedarf kann der Vorgang in diesem Schritt unterbrochen und die Probe bei -20 °C gelagert werden.
- 8 μL 5 M NaCl zugeben und mindestens 6 h bei 65 °C in einem Heizblock unter Schütteln bei 1000 U/min inkubieren.
HINWEIS: In diesem Schritt wird das Chromatin entvernetzt. Es ist sicherer, die Entkopplung nach Möglichkeit über Nacht zu verlängern. Das Vorhandensein von NaCl macht den Prozess effizienter. - Lassen Sie die Proben 5 min bei RT abkühlen und fügen Sie 2 μl RNase A hinzu.
- 1 h bei 37 °C unter Schütteln bei 1000 U/min inkubieren.
- Nehmen Sie die Proben aus dem Heizblock und fügen Sie 7 μl 300 mM CaCl 2 und2 μl Proteinase K hinzu.
- Stellen Sie den Heizblock auf 56 °C ein und inkubieren Sie ihn 30 Minuten lang unter Schütteln bei 1000 U/min. Bereiten Sie in der Zwischenzeit für jede Probe ein Phasentrennröhrchen vor, indem Sie sie bei 16.000 x g für 1 min bei 4 °C zentrifugieren.
HINWEIS: Diese speziellen Röhrchen erleichtern die Phasentrennung bei der Nukleinsäure-Phenol-Chloroform-Extraktion. - Entfernen Sie die Schläuche vom Heizblock und lassen Sie sie 3 Minuten lang bei RT ausgleichen.
- 400 μl der Probe werden in ein zuvor zentrifugiertes Phasentrennröhrchen überführt.
- 400 μl Phenol-Chloroform-Isoamylalkohollösung (PCI) zugeben und 5 s lang wirbeln.
VORSICHT: PCI ist eine leicht flüchtige und toxische Verbindung. Bitte behandeln Sie es mit den erforderlichen Sicherheitsmaßnahmen unter einem Abzug. - Zentrifugieren bei 16.000 x g für 5 min bei 4 °C.
- Fügen Sie 400 μl Chloroform hinzu und wirbeln Sie 5 s lang.
ACHTUNG: Chloroform ist eine leicht flüchtige und giftige Verbindung. Bitte behandeln Sie es mit den erforderlichen Sicherheitsmaßnahmen unter einem Abzug.
HINWEIS: In diesem Schritt werden mögliche Phenolrückstände ausgewaschen, die nachgeschaltete PCR-Anwendungen beeinträchtigen könnten. - Zentrifugieren bei 16.000 x g für 5 min bei 4 °C.
- 400 μl der oberen Phase werden in ein neues 1,5-ml-Röhrchen überführt, in dem 24 μl 5 M NaCl und 0,75 μl Glykogen hinzugefügt wurden. Kurz wirbeln.
- Fügen Sie 1.055 μl 100 % EtOH hinzu und wirbeln Sie gründlich vor. Achten Sie auf das richtige Mischen.
- Bei -80 °C für 1 h oder bei -20 °C über Nacht (ON) inkubieren.
HINWEIS: Dieser Schritt fällt die geschorene DNA aus; Um die Ausbeute zu maximieren, wird empfohlen, die ON-Inkubation zu wählen. - Zentrifugieren bei 16.000 x g für 30 min bei 4 °C.
- Entfernen Sie vorsichtig den Überstand und achten Sie darauf, dass sich das Pellet nicht verschiebt.
- Fügen Sie 500 μl kaltes 70% EtOH hinzu. Kippen Sie das Rohr vorsichtig, um sicherzustellen, dass das Pellet gewaschen wird.
HINWEIS: Dieser Schritt ist wichtig, um Salzrückstände zu entfernen, die mit den Nukleinsäuren ausgefällt haben könnten. Salze können andere nachgelagerte Anwendungen beeinträchtigen. - Zentrifugieren bei 16.000 x g für 15 min bei 4 °C.
- Entfernen Sie vorsichtig den gesamten Überstand und lassen Sie das Pellet bei RT trocknen.
Anmerkungen: Die Inkubation des Röhrchens auf einem Heizblock bei 37 °C verkürzt die Trocknungszeit. - Fügen Sie 50 μl Tris-EDTA-Lösung (TE-Puffer) hinzu und legen Sie den Schlauch bei 37 °C für 5-10 min unter Schütteln bei 300 U/min auf den Heizblock.
Anmerkungen: Dieser Schritt stellt die Auflösung des Pellets sicher. Hier kann das Protokoll pausiert werden, und die Probe kann bei 4 °C für bis zu 1 Woche oder bei -20 °C für eine längere Lagerung gelagert werden. - Führen Sie eine DNA-Analyse mit 1%igem Agarosegel durch.
7. Analyse der DNA-Größe
- Bereiten Sie ein 1%iges Agarose-Gel vor, indem Sie 1 g Agarose pro 100 ml Laufpuffer mischen (d. h. Tris-Acetat-EDTA (TAE) oder Tris-Borat-EDTA (TBE)). Erhitzen Sie die Suspension, bis sich die Agarose vollständig aufgelöst hat. Fügen Sie 10 μl EtBr pro 100 ml Agaroselösung hinzu, bevor Sie die Gellösung einfüllen.
VORSICHT: EtBr ist ein DNA-Interkalationsmittel, von dem bekannt ist, dass es krebserregend ist. Bitte behandeln Sie es mit den erforderlichen Sicherheitsmaßnahmen unter einem Abzug.
HINWEIS: Eine EtBr-Färbung (direkt im Gel oder nach dem Lauf) wird dringend empfohlen. Andere DNA-interkalierende Farbstoffe schnitten in unseren Händen bei der Arbeit mit DNA-Abstrichen nicht gut ab. Schmale Ladeschächte bieten im Vergleich zu breiteren Vertiefungen eine bessere Auflösung. - Mischen Sie 10 μl der Probe mit 2 μl 6x Loading Dye. Laden Sie als Nächstes 10 μl der Probe in das Gel und lassen Sie es laufen, bis die letzte Bande des geladenen Farbstoffs für 2/3 des Gels verläuft. Stellen Sie sicher, dass Sie eine DNA-Leiter hinzufügen.
- Bilde das Gel ab und überprüfe, ob die Abstrichgröße in den Bereich für die gewünschte Anwendung fällt.
Wenn das Chromatin die Qualitätskontrolle besteht, kann es für nachgelagerte Anwendungen verwendet werden.
Ergebnisse
Die Herstellung von Chromatin ist ein entscheidender Schritt auf dem Weg zu einem erfolgreichen ChIP. Um aus gefrorenen Proben qualitativ hochwertiges Chromatin herzustellen, sollten wir vor der Fixierung eine effiziente Gewebezerstörung sicherstellen, um das Vorhandensein von Gewebeklumpen zu vermeiden, die eine effiziente Scherung behindern könnten. Abbildung 1 zeigt eine zusammengefasste Pipeline des Protokolls. Die Pulverisierung allein reicht nicht aus, um das Gewebe vollständig zu dissoziieren, da Zellhaufen unterschiedlicher Größe und wenige Einzelzellen entstehen (Abbildung 1a). Verbindet man den ersten Pulverisierungsschritt mit der Dounce-Homogenisierung, so wird die Menge der Gewebeklumpen stark reduziert und die verbleibenden kleiner (Abbildung 1b). Nach den Fixierungs- und Lyseschritten nimmt die Anzahl der sichtbaren Einzelkerne zu (Abbildung 1c), während das typische sphärische Erscheinungsbild verloren geht. Nach 28 Beschallungszyklen ist die Kernfärbung (Hoechst 33258/DAPI) meist nicht mehr sichtbar. Dies ist in der Tat ein Zeichen für erfolgreiches Scheren (Abbildung 1d). Nach der De-Vernetzung eines Chromatin-Aliquots und der Visualisierung der DNA auf Agarosegel kann eine erfolgreiche Scherung durch das Vorhandensein von Fragmenten im Bereich von 100-300 bp erkannt werden. (Abbildung 2a) Die DNA-Menge kann je nach Zusammensetzung des präparierten Gewebestücks variieren. Dieses Chromatin kann erfolgreich für die ChIP-qPCR eingesetzt werden. Wie in Abbildung 2b dargestellt, konnte das Chromatin erfolgreich mit den Antikörpern H3K4me3, H3K27ac (aktive genbezogene Modifikationen) und H3K27me3 (stillgelegte genesverwandte Modifikationen) ausgefällt werden. Chromosom 1 Open Reading Frame 43 (C1orf43), Proteasom 20S Untereinheit Beta 2 (PSMB2) und Glyceraldehyd-3-phosphat-Dehydrogenase (mGapdh) Promotorregionen waren im Vergleich zu den Promotorregionen der Homeobox C13 (HOXC13), der Homeobox C12 (HOXC12) und der Maus Myelin Transkription Faktor 1 (mMyt1) angereichert in H3K4me3 und H3K27ac (Tabelle 1). Dies liegt daran, dass C1orf43, PSMB2 und mGapdh konstitutiv in der Leber transkribiert werden, während HOXC13, HOXC12 und mMyt1 stillgelegt werden. H3K27me3 zeigt das gegenteilige Verhalten, was den Erfolg des ChIP-Assays bestätigt. Die Tatsache, dass die Leber dieser Mäuse eine Chimäre ist, ermöglichte es uns, sowohl das murine als auch das menschliche Chromatin zu analysieren. Darüber hinaus konnte das gleiche Chromatin erfolgreich für ChIP-seq-Experimente verwendet werden. Nach dem Sequenzierungsschritt wurden die Reads mit einem Index abgeglichen, der sowohl aus murinen als auch aus menschlichen Genomen bestand, um die Anzahl der nicht ausgerichteten Fragmente zu reduzieren. Anschließend wurden die Reads nach Spezies getrennt und mit EaSeq22 weiter analysiert. Die Signalintensität wurde dann an der Transkriptionsstartstelle (TSS) jedes Gens gemessen und das Ergebnis nach der H3K4me3-Signalintensität sortiert. Abbildung 3a und Abbildung 3c zeigen eine deutliche Präsenz von H3K4me3 und H3K27ac an der TSS für einen beträchtlichen Teil der Gene sowohl im Maus- als auch im humanen Chromatin. Darüber hinaus antikorreliert H3K27me3 mit H3K4me3/H3K27ac. H3K27me3 ist auf der gesamten Länge des Gens vorhanden und nicht nur am TSS, wie von diesem PTM erwartet. Abbildung 3b und Abbildung3d zeigen den HOXC/HoxC-Cluster, von dem bekannt ist, dass er mit H3K27me3 angereichert und sowohl in der Leber von Mäusen als auch von Menschen transkriptionell inaktiv ist. Die Profilierung von H3K4me3 und H3K27ac zeigt Peaks für diese beiden PTMs, während die Signalintensität von H3K27me3 tendenziell niedriger und stärker verteilt ist.
Aufgrund der Komplexität der Chromatinpräparation kann es zu einer Überfixierung kommen, die Lyse- oder Beschallungszeit kann suboptimal sein, große Zellklumpen können bestehen bleiben oder eine unzureichende Handhabung der Probe kann unzureichend sein. Dies sind alles Ereignisse, die sich auf die Qualität der Zubereitung auswirken. In einigen Fällen ist die Anreicherung von Chromatinfragmenten in der richtigen Größe noch vorhanden oder wird auf eine höhere Größe verschoben. In anderen Fällen kann es zu Materialverlusten durch vorzeitige Lyse oder erfolgloses Scheren kommen. Abbildung 4 zeigt einige Beispiele für solche negativen und suboptimalen Ergebnisse. Bahn 3 und 4 zeigen eine Anreicherung der Fragmentgröße zwischen 200 bp und 800 bp. Es ist jedoch klar, dass die Fragmentgröße von 100 bp bis >10.000 bp reicht. In Bahn 5 und 6 liegt eine Anreicherung im Bereich von 100-250 bp mit deutlichem Materialverlust während der Präparation vor. Dies könnte erklären, warum bei der Beschallung kleinere Fragmente entstanden. Bahn 7 zeigt eine leicht suboptimale Präparation mit erhöhter Fragmentreichweite, während Bahn 8 einen fast vollständigen Materialverlust zeigt. Dies kann durch eine vorzeitige Kernlyse oder eine unzureichende Gewebedissoziation mit anschließendem Verlust nach Schritt 5.5 verursacht werden.

Abbildung 1: Übersicht über das Protokoll zur Chromatinpräparation. Die Bilder wurden nach Gewebepulverisierung (a), zusätzlicher manueller Homogenisierung (b), nach Kernlyse (c) und nach Beschallung (vor der Zentrifugation) (d) aufgenommen. Die Kernfärbung wurde mit Hoechst 33258/DAPI durchgeführt. Maßstabsbalken = 200 μm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.

Abbildung 2: Repräsentative Chromatinscherung und deren Qualität mittels ChIP-qPCR bewertet . 1% Agarosegel mit fragmentierten Chromatinproben nach Protokollen verschiedener Chromatinpräparationen. Eine Kontrolle des nicht gescherten Chromatins wird hinzugefügt, um sicherzustellen, dass kein Chromatin/DNA-Abbau im Voraus stattfindet (a). Geschertes Chromatin wurde in einem ChIP-qPCR-Assay auf seine Qualität getestet. Die Antikörper H3K4me3, H3K27ac und H3K27me3 wurden verwendet, um das frisch hergestellte Chromatin auszufällen. (b ) Die qPCR-Analyse wurde an humanen (C1orf43 und PSMB2), murinen (Gapdh) aktiven Promotoren und humanen (HOXC13, HOXC12), murinen (Myt1) inaktiven Promotoren durchgeführt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
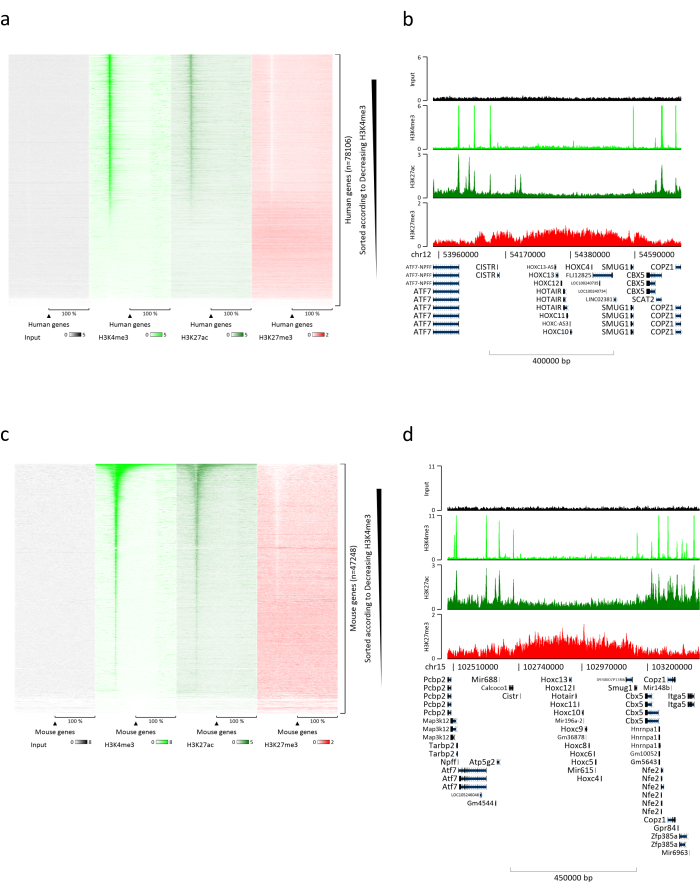
Abbildung 3: Repräsentative ChIP-seq-Analyse. Die Lesevorgänge wurden an einen Index angepasst, der sowohl mit menschlichen als auch mit Mausgenomen (hg19 und mm10) erstellt wurde. Nach dem Alignment wurden menschliche und murine Reads getrennt und weiter analysiert. Heatmap der menschlichen Gene, bei der das Signal am TSS quantifiziert und in absteigender Reihenfolge für die H3K4me3-Intensität angezeigt wurde (a). Beispiel eines menschlichen Genclusters unterdrückter Gene (HOX-Cluster), umgeben von aktiven Genen (b). Heatmap von murinen Genen, bei denen das Signal am TSS quantifiziert und in absteigender Reihenfolge für die H3K4me3-Intensität dargestellt wurde (c). Beispiel eines murinen Genclusters von unterdrückten Genen (Hox-Cluster), umgeben von aktiven Genen (d). Alle angezeigten Daten wurden von EaSeq pro Million Lesevorgänge normalisiert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.

Abbildung 4: Suboptimale und fehlgeschlagene Chromatinpräparationen. 1% Agarosegel mit fragmentierten Chromatinproben gemäß Protokoll. Die Abbildung enthält nicht abgeschertes Chromatin, das als Kontrolle verwendet wurde (Spur 2), nicht optimales Scheren (Spur 3-4), optimales Scheren mit deutlichem Materialverlust (Spur 5-6), suboptimales Scheren (Spur 7) und ausgedehntes Materialverlust (Spur 8). Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
| Name des Primers | Reihenfolge | |
| C1orf43 Promotor | Vorwärts | AGTGGGTGGAGAATGCAGAC |
| Rückwärts | GAGATTACCCCACCCCATTC | |
| PSMB2-Promotor | Vorwärts | CTTATTCAACCCCCGACAAA |
| Rückwärts | GATGAAGGACGGTGAGAGGA | |
| Hoxc13 distaler Promotor | Vorwärts | GAGCCCGAGATTCACTCAAC |
| Rückwärts | TTATGCCCAGTTTTGGGGTA | |
| Hoxc12 distaler Promotor | Vorwärts | AAAGCTTCCCACTGCAAAGA |
| Rückwärts | AAATCTGGGGGCGAACTACT | |
| mGAPDH-Promotor | Vorwärts | GGTCCAAAGAGAGGGAGGAG |
| Rückwärts | GCCCTGCTTATCCAGTCCTA | |
| mMYT-Promotor | Vorwärts | CAGCCCAATTCTAGCCACAT |
| Rückwärts | CCAAAGCAGGGGAGTAGGAG |
Tabelle 1: Liste der qPCR-Primer für aktive und inaktive Gene, die für ChIP-qPCR-Assays verwendet werden.
Diskussion
Die Chromatinpräparation aus schnappgefrorenem Gewebe bleibt eine Herausforderung, da die Anzahl der Schritte optimiert werden muss, um reproduzierbare und zuverlässige Ergebnisse zu erzielen. Die meisten der bereits veröffentlichten Protokolle 16,23 erfordern eine Gewebezerkleinerung vor der manuellen Dissoziation (Douncing). Wir haben versucht, Schritte, die einen Proteinabbau provozieren könnten, vor der Fixierung der Probe so weit wie möglich zu vermeiden. Der Pulverisierungsschritt wird bereits in gefrorenen Leberpräparaten24 verwendet und macht die manuelle Dissoziation einfacher und reproduzierbarer (siehe Figur 2a). Durch die Verwendung eines Mörtels, der speziell für 1,5-ml-Röhrchen entwickelt wurde (siehe Protokolle), wird der Probenverlust während des Pulverisierungsprozesses reduziert, so dass kleine Gewebemengen wie Leberbiopsieproben verarbeitet werden können. Prinzipiell ist eine direkte Gewebehomogenisierung ohne Mahlschritte möglich; Allerdings hat die Gewebehomogenisierung ohne vorherige Pulverisierung unserer Erfahrung nach eine schlechtere Reproduzierbarkeit und das Auftreten von Problemen für nachgelagerte Anwendungen war höher (Daten nicht gezeigt).
Die meisten Probleme, die bei der Herstellung von Chromatin aus Geweben auftreten, ergeben sich aus der Beschaffenheit dieser Proben und der Unfähigkeit, richtig zu überprüfen, ob die Zellhaufen klein genug sind, um ohne Qualitätsverlust fixiert zu werden. Darüber hinaus wäre die Überprüfung jedes Aliquots bei jedem Schritt zeitaufwändig und würde die Wahrscheinlichkeit eines Proteinabbaus erhöhen.
Die Fixierung (Schritt 3.9) ist ein grundlegender und entscheidender Teil der Chromatinpräparation. Aufgrund der Beschaffenheit des Gewebes wurde der Fixierungsschritt verzögert, bis das Gewebe homogenisiert war. Ein solcher aufgeschobener Fixierungsschritt hat den Vorteil, eine homogenere Zellsuspension zu erzeugen. Wir sind uns jedoch bewusst, dass es bei besonders manipulationsempfindlichen Zielen notwendig sein kann, die Fixierung erst kurz vor Schritt 3.6 durchzuführen. Dies würde dazu beitragen, extrem empfindliche Proteine oder PTMs zu schützen, obwohl es die Größe der Zellhaufen vergrößern kann, die, wenn sie fixiert werden, zu inhomogener Scherung führen können. Die Konzentration der im Protokoll verwendeten FA-Lösung ist Standard, kann jedoch modifiziert werden, um die Gesamtfixierung zu verbessern. Die hier gewählte Fixierzeit entspricht auch den im Feld üblichen Standardbedingungen. Bei einer höheren Konzentration der Fixierlösung kann die Fixierzeit verkürzt werden, während sie bei einer geringeren Menge erhöht werden sollte. Der Bediener sollte bedenken, dass eine Änderung der Fixierungszeit entweder zu einer Überfixierung der Probe führen oder Raum für den Proteinabbau schaffen kann. Im Falle der Ausfällung großer Komplexe (oder eines Teils davon) und TFs wäre es vorteilhaft, eine zweistufige Fixierung mit einer DSG-Lösung gefolgt von einer FA-Lösung durchzuführen25,26. DSG würde in diesem Fall Protein-Protein-Interaktionen stabilisieren, während Formaldehyd hauptsächlich auf direkte DNA-Protein-Interaktionen wirkt27.
Der Betreiber sollte die Möglichkeit in Betracht ziehen, ab Schritt 6.7 ein säulenbasiertes Kit für die DNA-Aufreinigung zu implementieren, das schneller ist und keine toxischen Verbindungen verwendet. Es wird jedoch immer eine gewisse Menge an ungebundener DNA geben, die verloren geht. Aus diesem Grund empfehlen wir die klassische Phenol-Chloroform-Extraktion mit anschließender EtOH-Fällung. Darüber hinaus kann es vor dem Ausführen des Agarosegels (Schritt 7.2) von Vorteil sein, die DNA-Konzentration zu messen und für jede Vertiefung die gleiche Menge zu laden, um ein klareres Bild zu erhalten.
Eine Einschränkung dieses Protokolls ergibt sich aus der Tatsache, dass wir dieses Protokoll nur mit Leberproben von chimären Mäusen der menschlichen Leber erforscht und angewendet haben28. Die Leber besteht per se aus Epithel- und Bindegewebe29. Im Falle einer Erkrankung kann fibrotisches Gewebe und Fettgewebe vorhanden sein30,31, was zu zusätzlichen Herausforderungen bei der Gewebezerstörung führt. Wir sind uns jedoch bewusst, dass unser Protokoll nicht auf Knochen, Muskeln und Fettgewebe angewendet werden darf, ohne die Dissoziations- und Beschallungsschritte zu optimieren. Zu beachten ist, dass jedes Gewebe in irgendeiner Weise optimiert werden muss, da es kein Protokoll gibt, das für alle geeignet ist, wie z. B. für Zellkulturproben15. Wir glauben jedoch, dass dieses Protokoll mit wenig oder gar keiner Optimierung erfolgreich auf andere Gewebe angewendet werden könnte, die in ihrer Zusammensetzung Ähnlichkeiten mit der Leber aufweisen, wie z. B. Lungen-, Darm-, Magen-, Bauchspeicheldrüsen- oder Nierengewebe.
Unser Protokoll wurde auch erfolgreich zur Analyse von TFs und Histonmodifikationen auf dem HBV-Episom mit kovalent geschlossener DNA (cccDNA) eingesetzt32. Dies eröffnet die Chance, einen solchen Ansatz auf andere virale Genome anzuwenden, die die Leber betreffen, wie z. B. das humane Cytomegalievirus33 (hCMV) und das humane Adenoviren34 (HAdV). Es ist nicht ausgeschlossen, dass es möglich wäre, andere DNA-Viren zu analysieren, die eine persistierende Infektion in anderen Geweben etablieren, wie z.B. Kaposi-Sarkom-Herpesvirus35 (KHSV), Herpes-simplex-Virus 36 (HSV1/2), Polyomaviren, Epstein-Barr-Virus37 (EBV).
Offenlegungen
Die Autoren haben nichts zu verraten.
Danksagungen
Die Studie wurde von der Deutschen Forschungsgemeinschaft (DFG) durch eine Förderung von Maura Dandri (SFB 841 A5) und vom Land Hamburg mit dem Forschungsprogramm (LFF-FV44: EPILOG) unterstützt.
Wir danken Dr. Tassilo Volz, Yvonne Ladiges und Annika Volmari für die technische Hilfe und die kritische Lektüre des Manuskripts. Dr. Thomas Günther und Prof. Adam Grundhoff für die sehr hilfreichen Anregungen und die Primer-Sets für die ChIP-qPCR-Analyse.
Materialien
| Name | Company | Catalog Number | Comments |
| 0.22µm sterile syringe filter | Labsolute | 7699822 | |
| 1.5 mL Safeseal tubes | Sarstedt | 7,27,06,400 | |
| 6x orange loading dye | Thermofisher | R0631 | |
| Benchtop refrigerated centrifuge | |||
| Bioruptor NGS | Diagenode | ||
| Blade or Scalpel | |||
| Calcium chloride dihydrate | Carl Roth | HN04 | |
| Chloroform | Sigma Aldrich (Merck) | C2432 | |
| cOmplete Protease Inhibitor Cocktail | Roche | 11697498001 | |
| Deacetylase Inhibitor | Active Motif | 37494 | |
| Dounce tissue grinder set | Sigma Aldrich (Merck) | DWK885300-0001-1EA | |
| EDTA 500 mM solution | PanReac AppliChem | A4892 | |
| EGTA | Sigma Aldrich (Merck) | E4378 | |
| EtBr | Carl Roth | 2218 | Concentration 10mg/mL |
| Ethanol absolute | CHEMSOLUTE | 2273 | |
| Glycerol | Sigma Aldrich (Merck) | G9012 | |
| Glycin | Carl Roth | 0079 | |
| Glycogen | Roche | 10901393001 | Concentration: 20mg/mL |
| Heating block | |||
| HEPES | Sigma Aldrich (Merck) | H4034 | |
| LE Agarose | Biozym | 840000 | |
| Liquid nitrogen cooled mini mortar | Bel-Art | H37260-0100 | |
| MeOH free Formaldehyde 16% | Thermofisher | 28908 | |
| NP-40 | Roche | 11332473001 | |
| PBS 1x | Thermofisher | 10010015 | |
| Pefabloc SC-Protease-Inhibitor | Sigma Aldrich (Merck) | 11429868001 | |
| Phase Lock Gel - Heavy | QuantaBio | 2302830 | |
| Phenol:Chloroform:Isoamyl alcohol 25:24:1 | Sigma Aldrich (Merck) | P3803 | |
| Potassium chloride | Carl Roth | 6781 | |
| Potassium hydroxyde | Merck | 105033 | |
| Proteinase K | Lucigen | MPRK092 | Concentration: 50 µg/µL |
| RNAse A | Lucigen | MRNA092 | Concentration: 5 mg/mL |
| SDS 10% solution | PanReac AppliChem | A3950 | |
| Sodium carbonate anhydrous | Carl Roth | A135 | |
| Sodium chloride | Sigma Aldrich (Merck) | S7653 | |
| Sterile Petri dishes | Sarstedt | 83,39,02,500 | |
| Tris-HCl solution | Sigma Aldrich (Merck) | T2694 | |
| Triton-X100 | Sigma Aldrich (Merck) | X100 |
Referenzen
- Waddington, C. H., Pantelouris, E. M. Transplantation of nuclei in newt's eggs. Nature. 172 (4388), 1050-1051 (1953).
- Silmon de Monerri, N. C., Kim, K. Pathogens hijack the epigenome: A new twist on host-pathogen interactions. American Journal of Pathology. 184 (4), 897-911 (2014).
- Knipe, D. M., et al. Snapshots: chromatin control of viral infection. Virology. 435 (1), 141-156 (2013).
- Tropberger, P., et al. Mapping of histone modifications in episomal HBV cccDNA uncovers an unusual chromatin organization amenable to epigenetic manipulation. Proceedings of the National Academy of Sciences of the United States of America. 112 (42), 5715-5724 (2015).
- Sproul, D., Gilbert, N., Bickmore, W. A. The role of chromatin structure in regulating the expression of clustered genes. Nature Reviews Genetics. 6 (10), 775-781 (2005).
- Fischle, W., Wang, Y., Allis, C. D. Histone and chromatin cross-talk. Current Opinion in Cell Biology. 15 (2), 172-183 (2003).
- Ling, X., Harkness, T. A., Schultz, M. C., Fisher-Adams, G., Grunstein, M. Yeast histone H3 and H4 amino termini are important for nucleosome assembly in vivo and in vitro: redundant and position-independent functions in assembly but not in gene regulation. Genes & Development. 10 (6), 686-699 (1996).
- Zhang, L., Eugeni, E. E., Parthun, M. R., Freitas, M. A. Identification of novel histone post-translational modifications by peptide mass fingerprinting. Chromosoma. 112 (2), 77-86 (2003).
- Wang, H., et al. Role of histone H2A ubiquitination in Polycomb silencing. Nature. 431 (7010), 873-878 (2004).
- Hassa, P. O., Haenni, S. S., Elser, M., Hottiger, M. O. Nuclear ADP-ribosylation reactions in mammalian cells: where are we today and where are we going. Microbiology and Molecular Biology Reviews. 70 (3), 789-829 (2006).
- Dey, B., et al. DNA-protein interactions: methods for detection and analysis. Molecular and Cellular Biochemistry. 365 (1-2), 279-299 (2012).
- Hager, G. L., McNally, J. G., Misteli, T. Transcription dynamics. Molecular Cell. 35 (6), 741-753 (2009).
- Nagy, Z., Tora, L. Distinct GCN5/PCAF-containing complexes function as co-activators and are involved in transcription factor and global histone acetylation. Oncogene. 26 (37), 5341-5357 (2007).
- Solomon, M. J., Larsen, P. L., Varshavsky, A. Mapping protein-DNA interactions in vivo with formaldehyde: evidence that histone H4 is retained on a highly transcribed gene. Cell. 53 (6), 937-947 (1988).
- Gunther, T., Theiss, J. M., Fischer, N., Grundhoff, A. Investigation of viral and host chromatin by ChIP-PCR or ChIP-Seq analysis. Current Protocols in Microbiology. 40, 11-21 (2016).
- Cotney, J. L., Noonan, J. P. Chromatin immunoprecipitation with fixed animal tissues and preparation for high-throughput sequencing. Cold Spring Harbor Protocols. 2015 (4), 419 (2015).
- Haim, Y., Tarnovscki, T., Bashari, D., Rudich, A. A chromatin immunoprecipitation (ChIP) protocol for use in whole human adipose tissue. American Journal of Physiology-Endocrinology and Metabolism. 305 (9), 1172-1177 (2013).
- Castellano-Castillo, D., et al. Chromatin immunoprecipitation improvements for the processing of small frozen pieces of adipose tissue. PLoS One. 13 (2), 0192314 (2018).
- Savic, D., Gertz, J., Jain, P., Cooper, G. M., Myers, R. M. Mapping genome-wide transcription factor binding sites in frozen tissues. Epigenetics Chromatin. 6 (1), 30 (2013).
- Perna, A., Alberi, L. A. TF-ChIP method for tissue-specific gene targets. Frontiers Cell Neuroscience. 13, 95 (2019).
- Allweiss, L., et al. Proliferation of primary human hepatocytes and prevention of hepatitis B virus reinfection efficiently deplete nuclear cccDNA in vivo. Gut. 67 (3), 542-552 (2018).
- Lerdrup, M., Johansen, J. V., Agrawal-Singh, S., Hansen, K. An interactive environment for agile analysis and visualization of ChIP-sequencing data. Nature Structural & Molecular Biology. 23 (4), 349-357 (2016).
- Perna, A., Alberi, L. A. TF-ChIP Method for Tissue-Specific Gene Targets. Frontiers in Cellular Neuroscience. 13, 95 (2019).
- Liang, N., Fan, R., Goni, S., Treuter, E. Preparation of Frozen Liver Tissues for Integrated Omics Analysis. Methods in Molecular Biology. 1951, 167-178 (2019).
- Liu, Z., et al. Proteomic and network analysis of human serum albuminome by integrated use of quick crosslinking and two-step precipitation. Scientific Reports. 7 (1), 9856 (2017).
- Singh, A. A., et al. Optimized ChIP-seq method facilitates transcription factor profiling in human tumors. Life Science Alliance. 2 (1), 201800115 (2019).
- Aoki, T., et al. Bi-functional cross-linking reagents efficiently capture protein-DNA complexes in Drosophila embryos. Fly. 8 (1), 43-51 (2014).
- Allweiss, L., Dandri, M. Experimental in vitro and in vivo models for the study of human hepatitis B virus infection. Journal of Hepatology. 64, 17-31 (2016).
- Krishna, M. Microscopic anatomy of the liver. Clinics in Liver Disease. 2, 4-7 (2013).
- Tannapfel, A., et al. Histopathological diagnosis of non-alcoholic and alcoholic fatty liver disease. Virchows Archiv. 458 (5), 511-523 (2011).
- Schuppan, D., Afdhal, N. H. Liver cirrhosis. Lancet. 371 (9615), 838-851 (2008).
- Allweiss, L., et al. Therapeutic shutdown of HBV transcripts promotes reappearance of the SMC5/6 complex and silencing of the viral genome in vivo. Gut. , 322571 (2021).
- Gerna, G., Kabanova, A., Lilleri, D. Human cytomegalovirus cell tropism and host cell receptors. Vaccines. 7 (3), (2019).
- Echavarria, M. Adenoviruses in immunocompromised hosts. Clinical Microbiology Reviews. 21 (4), 704-715 (2008).
- Frohlich, J., Grundhoff, A. Epigenetic control in Kaposi sarcoma-associated herpesvirus infection and associated disease. Seminars in Immunopathology. 42 (2), 143-157 (2020).
- Nicoll, M. P., Proenca, J. T., Efstathiou, S. The molecular basis of herpes simplex virus latency. FEMS Microbiology Reviews. 36 (3), 684-705 (2012).
- Thorley-Lawson, D. A., Hawkins, J. B., Tracy, S. I., Shapiro, M. The pathogenesis of Epstein-Barr virus persistent infection. Current Opinion in Virology. 3 (3), 227-232 (2013).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten