Method Article
Extracción de cromatina del tejido hepático quimérico congelado para el análisis de inmunoprecipitación de la cromatina
En este artículo
Resumen
Este protocolo se centra en la preparación de cromatina a partir de tejidos congelados a presión y es adecuado para la inmunoprecipitación de cromatina reticulada (X-ChIP) seguida de un análisis cuantitativo de PCR (X-ChIP-qPCR) o enfoques de secuenciación de próxima generación (X-ChIP-seq).
Resumen
La inmunoprecipitación de cromatina reticulada (X-ChIP) es una técnica ampliamente utilizada para evaluar los niveles de marcas de histonas y la ocupación de factores de transcripción en la cromatina del huésped y / o patógeno. La preparación de la cromatina a partir de tejidos crea desafíos adicionales que deben superarse para obtener protocolos reproducibles y confiables comparables a los utilizados para el cultivo celular. La disrupción y fijación del tejido son pasos críticos para lograr un cizallamiento eficiente de la cromatina. La coexistencia de diferentes tipos de células y grupos también puede requerir diferentes tiempos de cizallamiento para alcanzar el tamaño óptimo del fragmento y dificulta la reproducibilidad del cizallamiento. El propósito de este método es lograr preparaciones de cromatina huésped confiables y reproducibles a partir de tejido congelado (hígado) adecuadas tanto para aplicaciones de ChIP-qPCR como de secuenciación de próxima generación (NGS). Observamos que la combinación de pulverización tisular de nitrógeno líquido seguida de homogeneización conduce a una mayor reproducibilidad en comparación con la homogeneización solamente, ya que proporciona una suspensión que consiste principalmente en células individuales disociadas que pueden cortarse de manera eficiente. Además, la etapa de fijación debe realizarse bajo rotación suave para proporcionar una reticulación homogénea. El material fijo es entonces adecuado para el aislamiento de núcleos basados en tampón, para reducir la contaminación de proteínas citoplasmáticas y ADN y ARN patógenos (cuando corresponda), evitando gradientes de centrifugación que consumen mucho tiempo. La sonicación posterior completará la lisis nuclear y cortará la cromatina, produciendo un rango de tamaño específico de acuerdo con las condiciones de cizallamiento elegidas. El rango de tamaño debe estar entre 100 y 300 nt para aplicaciones NGS, mientras que podría ser mayor (300-700 nt) para el análisis ChIP-qPCR. Tales adaptaciones al protocolo pueden mejorar en gran medida los análisis de cromatina de muestras de tejido congelado.
Introducción
Desde su descubrimiento, la regulación epigenética en células de mamíferos ha ganado un reconocimiento creciente1, considerando que la comprensión de tales mecanismos proporcionaría información clave no solo en biología celular, sino también en biología de enfermedades y tumores. Además, los agentes infecciosos también pueden causar cambios epigenéticos en el huésped2, mientras que la maquinaria de la célula huésped también puede afectar la cromatina de los patógenos, como la persistencia de virus de ADN 3,4. Esta interacción huésped-patógeno parece desempeñar un papel en la persistencia de la infección. número arábigo
A través de una asociación reversible con el ADN, las proteínas histonas forman un complejo llamado nucleosoma. Los nucleosomas alcanzan a su vez un nivel más alto de organización conocido como cromatina. Se sabe que la remodelación de la cromatina regula estrictamente la expresión génica, otorgando o negando el acceso a los factores de transcripción (TF)5. Estos factores pueden desencadenar o bloquear el reclutamiento de la ARN polimerasa II (PolII) en los promotores de genes, influyendo en la síntesis de ARNm a partir de la plantilla de ADN6. Las proteínas histonas contienen colas7, flanqueando ambos extremos del pliegue de histonas, que pueden estar sujetas a modificaciones postraduccionales (PTM), lo que permite una regulación estricta de la transcripción génica mediante cambios estructurales de la cromatina. La mayoría de los PTM de histonas se localizan en el extremo N de la cola, siendo la acetilación y la metilación los PTM mejor estudiados, aunque también se han descrito fosforilación8, ubiquitinación9 y ribosilación10 . Caracterizar y estudiar tales proteínas es esencial para obtener una visión profunda de la regulación génica.
Actualmente, hay un puñado de métodos y herramientas bien establecidos disponibles para estudiar las interacciones directas ADN-proteína: ensayo de cambio de movilidad electroforética (EMSA), ensayo de levadura de un híbrido (Y1H) y huella de ADN11. Sin embargo, estos métodos se centran per se en interacciones únicas ADN-proteína y no son aplicables para estudios de todo el genoma. Otra limitación de esas técnicas es la falta de asociación de histonas con los segmentos de ADN investigados. Por lo tanto, tales enfoques no pretenden reflejar la complejidad de la maquinaria transcripcional in vivo y no tienen en cuenta cambios estructurales importantes12 u otras enzimas/cofactores requeridos13 que podrían influir (ya sea promoviendo o inhibiendo) la unión de proteínas al ADN.
La idea de que la fijación de células con agentes como el formaldehído (FA) podría proporcionar una instantánea in vivo de las interacciones proteína-ADN, creó la base para el desarrollo de ensayos de inmunoprecipitación de cromatina (ChIP)14. Esto, junto con la disponibilidad de la tecnología de PCR cuantitativa (qPCR) y de anticuerpos altamente específicos, permitió el desarrollo de ensayos ChIP-qPCR. Posteriormente, el advenimiento de las técnicas de secuenciación de próxima generación (NGS), cuyos costos son cada vez más asequibles, concedió acoplar los experimentos ChIP con los enfoques NGS (ChIP-seq), proporcionando así a los investigadores nuevas herramientas poderosas que permiten la investigación de la regulación de la cromatina. En estos ensayos, las células aisladas o cultivadas se fijan con glutarato de disuccinimidil (DSG) y / o FA, los núcleos se aíslan, la cromatina se fragmenta y precipita por el anticuerpo de interés. A partir de entonces, el ADN se purifica y se analiza mediante enfoques de PCR o NGS. A diferencia de EMSA, Y1H y la huella de ADN, los ensayos ChIP tienen la capacidad de proporcionar una instantánea global de la interacción proteína-ADN dentro de la célula. Esto ofrece flexibilidad y permite el análisis de múltiples loci dentro de la misma muestra. Sin embargo, debido a la naturaleza del ensayo, ChIP puede, eventualmente, detectar no solo interacciones directas, sino también indirectas, no ofreciendo la precisión de los métodos mencionados anteriormente, cuando esté interesado en interacciones directas proteína-ADN.
Los protocolos de preparación de la cromatina a partir de material de cultivo celular están bien establecidos15 y son altamente reproducibles, lo que permite al usuario obtener cromatina adecuada tanto para los enfoques de qPCR como NGS en 1-2 días hábiles. Sin embargo, la obtención de cromatina de alta calidad a partir de tejidos enteros todavía representa un desafío debido a la necesidad de disociar las células dentro del tejido mientras se logra una fijación y cizallamiento óptimos de la cromatina. Además, la composición y la morfología de distintos tipos de tejidos varían, lo que requiere el ajuste de los protocolos existentes16,17. El uso de tejido criopreservado ofrece desafíos adicionales en comparación con las muestras frescas. Esto se debe a la dificultad de obtener una suspensión de una sola célula sin una gran pérdida de material. Esto conduce a un cizallamiento inadecuado, lo que dificulta las aplicaciones posteriores. No obstante, acceder a muestras de tejido congelado en lugar de la contraparte fresca no solo aumenta la flexibilidad del trabajo, sino que también puede representar la única opción para los investigadores que trabajan con muestras que se originan en estudios longitudinales o comparativos. Se han publicado un puñado de protocolos de preparación de cromatina para tejido congelado. Estos se basan principalmente en la descongelación de muestras seguida de la picación, la disociación manual / basada en máquinas o los pasos de pulverización de nitrógeno líquido18,19,20.
Aquí describimos un método optimizado de preparación de cromatina15 para muestras hepáticas congeladas no fijadas, que combina la pulverización tisular en nitrógeno líquido con la homogeneización del mortero, para lograr un cizallamiento de cromatina reproducible adecuado para los enfoques X-ChIP destinados a analizar los genomas virales y del huésped.
Protocolo
El muestreo de tejido de ratones quiméricos hepáticos humanos21 se realizó de acuerdo con la directiva de la Unión Europea 86/609 / CEE y aprobado por el comité ético de la ciudad y el estado de Hamburgo de acuerdo con los principios de la Declaración de Helsinki.
1. Preparación de reactivos
- Preparar solución de glicina 1,25 M en agua desionizada. Filtro estéril con filtro de 0,22 μm del tamaño de un poro. Conservar a 4 °C.
- Prepare una solución de cloruro de sodio (NaCl) 5 M. Conservar a temperatura ambiente.
- Preparar la solución de CaCl 2: 300 mM CaCl2 y 10 mM Tris-HCl pH 8 en agua desionizada. Filtro estéril con filtro de 0,22 μm de tamaño de poro y almacenar en RT.
- Prepare una dilución Triton X-100 al 10% en agua desionizada. Tienda en RT.
- Preparar tampón Tris-EDTA: 1 mM EDTA y 10 mM Tris pH 8 en agua desionizada. Conservar a 4 °C.
- Prepare los siguientes buffers de acuerdo con la cantidad requerida:
- Preparar tampón A: 50 mM Hepes-KOH pH 7.5, 140 mM NaCl, 1 mM ácido etilendiaminotetraacético (EDTA), 10% glicerol, 0.5% NP-40 y 0.25% Triton X-100 en agua desionizada. Filtro estéril con filtro de 0,22 μm del tamaño de un poro. Conservar a 4 °C.
- Preparar tampón B: 10 mM Tris-HCl pH 8, 200 mM NaCl, 1mM EDTA, 0,5 mM de ácido egtázico (EGTA). Filtro estéril con filtro de 0,22 μm del tamaño de un poro. Conservar a 4 °C.
- Preparar tampón C: 1% SDS, 10 mM EDTA y 50 mM Tris-HCl pH 8 en agua desionizada. Filtro estéril con filtro de 0,22 μm del tamaño de un poro. Tienda en RT.
- Preparar tampón de dilución de cromatina: 0,01% SDS, 1,1% Triton X-100, 1,2 mM EDTA, 16,6 mM Tris-HCl pH 8 y 166 mM NaCl en agua desionizada. Filtro estéril con filtro de 0,22 μm del tamaño de un poro. Conservar a 4 °C.
2. Preparación del material
- Recoja hielo seco, hielo y nitrógeno líquido.
PRECAUCIÓN: Manipule el hielo seco y el nitrógeno líquido con el cuidado necesario para evitar quemaduras. - Preenfriar la centrífuga a 4 °C.
NOTA: Este paso es importante para evitar la degradación de proteínas y la desreticulación durante los pasos de lavado, ya que esto reducirá la calidad de la cromatina. - Ponga un plato estéril en hielo seco y déjelo enfriar.
NOTA: Asegúrese de que la placa sea lo suficientemente grande como para facilitar el proceso de corte. Se recomienda una placa de Petri de 100 mm/placa de cultivo celular. - Saque la alícuota necesaria de glicina 1.25 M y deje que llegue a RT.
- Sacar las alícuotas necesarias de los buffer A, B y PBS. Agregue inhibidores de la proteasa y / o desacetilasa y fosfatasa para alcanzar la concentración de 1 vez y déjelos en hielo.
- Saque la alícuota necesaria del tampón C dejándola en RT. No agregue los inhibidores de la proteasa y/o la desacetilasa y la fosfatasa hasta que se especifique.
- Sacar la alícuota necesaria de RT PBS.
PRECAUCIÓN: El tampón C contiene dodecil sulfato de sodio (SDS). Adoptar las medidas de seguridad adecuadas a la hora de preparar el tampón.
NOTA: SDS precipita en hielo, y los inhibidores de la proteasa y la desacetilasa no son estables en RT. - Pre-enfríe el mortero vertiendo nitrógeno líquido en su cámara, siguiendo estrictamente las instrucciones del proveedor. Enfríe el mortero metálico en hielo seco durante al menos 5 minutos.
NOTA: Es posible utilizar un mortero alternativo al propuesto. Sin embargo, el dispositivo utilizado en este protocolo, debido a su peculiar construcción, permite trabajar con pequeña cantidad de tejido sin pérdida sustancial durante el proceso de pulverización. - Pre-enfríe el homogeneizador Dounce con el mortero A asociado sobre hielo.
NOTA: El mortero A tiene un ajuste suelto con el homogeneizador. Esto permite obtener una suspensión de una sola célula sin lisis celular significativa.
3. Reticulación tisular
- Corte unos 50 mg de tejido congelado directamente sobre el plato sobre hielo seco con la ayuda de un bisturí y unas pinzas.
NOTA: Se sugiere mantener el bisturí en RT, ya que esto facilitará el proceso de corte. Evite aplicar demasiada presión sobre el bisturí, ya que esto aumentará el riesgo de dispersar trozos de tejido fuera del área de corte. Para tener en cuenta, 50 mg de tejido (hígado en este caso) deben producir alrededor de 5 millones de células. Tenga en cuenta que la cuchilla caliente descongelará el filo de corte. Sin embargo, teniendo en cuenta el tamaño relativamente grande de la pieza de tejido, esto debería tener un efecto limitado. Cuando se cortan piezas más pequeñas, podría ser beneficioso usar un bisturí frío prestando atención para evitar la dispersión del tejido. - Coloque el tejido cortado en un tubo de 1,5 ml ya enfriado en hielo seco. Evite la descongelación de tejidos.
- Mueva el tubo que contiene el tejido al mortero, dejándolo reposar allí durante 5 minutos.
NOTA: Dejar reposar la muestra en el mortero disminuye su temperatura (de -80 °C a -196 °C). Esto aumenta su dureza y facilita el paso de pulverización. - Aplique presión a la muestra con la ayuda del mortero preenfriado hasta que no se vean más desmoronamientos sólidos.
NOTA: Es importante evitar el calentamiento del mortero por fuerzas de rotación excesivas, ya que esto descongelará la muestra. Después de cada pulverización de la muestra, limpie el mortero con etanol al 70% (EtOH) y déjelo enfriar nuevamente en hielo seco. - Retirar del mortero el tubo que contiene la muestra y añadir 950 μL de PBS helado con los inhibidores necesarios. Pipetear suavemente hacia arriba y hacia abajo hasta que la muestra esté completamente resuspendida. Continúe inmediatamente con el paso 3.6.
- Transfiera la suspensión de tejido al homogeneizador y aplique 20-30 golpes con mortero A para obtener una suspensión más fina. Evite la formación de espuma.
NOTA: La cantidad de trazos debe optimizarse de acuerdo con la consistencia del tejido. Este paso disocia aún más los pequeños grupos de células obtenidas después de la pulverización. Una homogeneización inadecuada puede afectar la eficiencia del cizallamiento. - Transfiera el homogeneizado a un nuevo tubo de 1,5 ml, ya preenfriado en hielo.
- Centrifugar durante 5 min a 1.300 x g a 4 °C y retirar con cuidado el sobrenadante.
- Resuspender el pellet completamente en 950 μL de RT PBS mediante un pipeteo suave y añadir 63,6 μL de FA libre de MeOH al 16% para tener una concentración final del 1%. Continúe inmediatamente con el paso 3.10.
PRECAUCIÓN: FA es un químico tóxico. Manéjelo bajo una campana extractora con las medidas de seguridad adecuadas.
NOTA: La resuspensión incompleta puede provocar la agregación celular durante el paso de fijación. Esto dificulta la lisis y el proceso de cizallamiento. - Gire 10 minutos en RT. Continúe inmediatamente con el paso 3.11
NOTA: La rotación es necesaria para evitar agregados. La cantidad de tiempo requerido para la fijación debe optimizarse, de acuerdo con el objetivo de interés y el tipo de muestra. Es importante tener en cuenta que los tiempos de fijación excesivos podrían dificultar el cizallamiento adecuado. - Añadir 113 μL de glicina a 1,25 M a RT para obtener una concentración final de 125 mM y rotar durante 5 min.
NOTA: La glicina apaga la reacción fijadora evitando la reticulación excesiva. - Centrifugar a 1.300 x g durante 3 min a 4 °C.
- Desechar el sobrenadante y resuspender el pellet cuidadosamente pipeteando en 950 μL de PBS helado con los inhibidores necesarios.
- Centrifugar a 1.300 x g durante 3 min a 4 °C.
- Repita los pasos 3.13-3.14 y proceda inmediatamente a los pasos de aislamiento de la cromatina.
4. Aislamiento de cromatina
- Añadir 950 μL de tampón A con los inhibidores necesarios al pellet. Mezclar suavemente pipeteando hasta que el pellet esté completamente resuspendido y girar 10 min a 4 °C.
NOTA: Este paso lisa la suspensión fija de una sola célula, sin lisis de núcleos. Esto permite eliminar la muestra de proteínas citosólicas y ARN. Prolongar el tiempo de lisis podría ser beneficioso para las células difíciles de lisar, aumentando, sin embargo, el tiempo de manipulación del tejido. En este punto, es posible verificar la preparación bajo el microscopio después de la tinción de azul de tripano / DAPI para verificar el tamaño de los grupos y la presencia de células individuales. Sin embargo, los núcleos individuales pueden no ser fáciles de apreciar debido al material tisular fijo. - Centrifugar a 2.000 x g durante 5 min a 4 °C y retirar con cuidado el sobrenadante.
- Añadir 950 μL de tampón B con los inhibidores necesarios al pellet. Mezclar suavemente pipeteando hasta que el pellet esté completamente resuspendido y girar 10 min a 4 °C.
NOTA: Este paso elimina el tampón de lisis de la preparación de núcleos para evitar más lisis no deseada. - Centrifugar a 2000 x g durante 5 min a 4 °C. Mientras tanto, agregue los inhibidores necesarios (igual que el paso 2.5) para amortiguar C.
- Retire con cuidado el sobrenadante.
- Añadir 300 μL de tampón RT C al pellet y pipetear vigorosamente.
- Vortex la muestra durante 15-30 s y girar el tubo brevemente para recoger las gotas en la tapa.
NOTA: Este paso es importante para liberar y lisar los núcleos fijos. Para preservar la integridad de la muestra y al mismo tiempo evitar la precipitación SDS, mantenga la muestra antes de la sonicación en un recipiente de plástico mantenido en hielo para mantener una temperatura de 9-11 ° C.
5. Fragmentación de la cromatina
- Transfiera la muestra a tres tubos limpios con certificación de sonicación de 0,65 ml que garanticen 100 μL de suspensión de núcleos lisados por tubo.
NOTA: Es posible utilizar tubos certificados por sonicación de 1,5 ml con un volumen máximo de 300 μL. Se necesita un soporte específico para esos tubos. 0,65 ml debe ofrecer un cizallamiento más homogéneo debido al menor volumen de la muestra por tubo. - Sonicar la cromatina durante 28 ciclos a alta intensidad con el ajuste 30 s ON y 30 s OFF. Asegúrese de que el baño sonicador esté bien enfriado (hielo o dispositivo de enfriamiento).
NOTA: Este paso necesita optimización en casi todos los casos. El usuario debe tener en cuenta que aumentar el tiempo de cizallamiento proporcionará fragmentos más pequeños y homogéneos; sin embargo, esto puede aumentar la posibilidad de disminuir la calidad de la cromatina. Elija el número más bajo de ciclos que proporcione el tamaño de fragmento requerido. Durante la optimización de este paso, es útil realizar tinción nuclear para comprobar si el número de ciclos fue suficiente para lisar la mayoría de los núcleos. - Transfiera la cromatina sonicada a un nuevo tubo de 1,5 ml previamente enfriado en hielo.
- Añadir 30 μL de solución Triton X-100 al 10% y vórtice durante 5-10 s.
NOTA: Triton X-100 se une a la SDS evitando más precipitaciones a 4 °C. La cantidad final de Triton X-100 siempre debe ser del 1%. - Centrifugadora a 16.000 g durante 15 min a 4 °C.
- Transfiera el sobrenadante a un tubo limpio de 1,5 ml preenfriado en hielo.
- NOTA: El sobrenadante contiene la cromatina cortada y debe aparecer claro. El pellet contiene restos "no cortables" y debe permanecer bastante pequeño (principalmente marrón en el caso del tejido hepático). Busque indicios de cizallamiento fallido: solución de cromatina que no se haya vuelto más clara y dimensiones de pellet similares a las del paso 4.5.
6. Purificación del ADN
- Transfiera 10-25 μL de cromatina cortada a un tubo nuevo y agregue tampón C para alcanzar un volumen final de 200 μL. Almacene el resto de la cromatina a -80 °C hasta su uso posterior. Si es necesario, el procedimiento puede interrumpirse en este paso y la muestra puede almacenarse a -20 °C.
- Añadir 8 μL de NaCl 5 M e incubar al menos 6 h a 65 °C en un bloque calefactor agitado a 1000 rpm.
NOTA: Este paso desenlaza la cromatina. Es más seguro extender la reticulación durante la noche cuando sea posible. La presencia de NaCl hace que el proceso sea más eficiente. - Deje que las muestras se enfríen a RT durante 5 min y agregue 2 μL de RNasa A.
- Incubar durante 1 h a 37 °C agitando a 1000 rpm.
- Retirar las muestras del bloque calefactor y añadir 7 μL de CaCl 2 a 300 mM y2 μL de proteinasa K.
- Ajuste el bloque calefactor a 56 °C e incubar durante 30 minutos agitando a 1000 rpm. Mientras tanto, prepare un tubo de separación de fases para cada muestra centrifugando a 16.000 x g durante 1 minuto a 4 °C.
NOTA: Estos tubos especiales facilitan la separación de fases durante la extracción de fenol-cloroformo de ácidos nucleicos. - Retire los tubos del bloque calefactor y déjelos equilibrarse a RT durante 3 minutos.
- Transfiera 400 μL de la muestra a un tubo de separación de fases previamente centrifugado.
- Añadir 400 μL de solución de fenol-cloroformo-alcohol isoamílico (PCI) y vórtice durante 5 s.
PRECAUCIÓN: PCI es un compuesto altamente volátil y tóxico. Por favor, manéjelo con las medidas de seguridad necesarias bajo una campana extractora. - Centrifugar a 16.000 x g durante 5 min a 4 °C.
- Añadir 400 μL de cloroformo y vórtice durante 5 s.
PRECAUCIÓN: El cloroformo es un compuesto altamente volátil y tóxico. Por favor, manéjelo con las medidas de seguridad necesarias bajo una campana extractora.
NOTA: Este paso elimina los posibles residuos de fenol, que podrían interferir con las aplicaciones posteriores de PCR. - Centrifugar a 16.000 x g durante 5 min a 4 °C.
- Transfiera 400 μL de la fase superior a un nuevo tubo de 1,5 ml donde se agregaron 24 μL de 5 M NaCl y 0,75 μL de glucógeno. Vórtice breve.
- Agregue 1,055 μL de 100% EtOH y vórtice a fondo. Asegúrese de mezclar correctamente.
- Incubar a -80 °C durante 1 h o a -20 °C durante la noche (ON).
NOTA: Este paso precipita el ADN cortado; para maximizar el rendimiento se sugiere elegir la incubación ON. - Centrifugadora a 16.000 g durante 30 min a 4 °C.
- Retire con cuidado el sobrenadante prestando atención para no dislocar el pellet.
- Añadir 500 μL de EtOH frío al 70%. Incline el tubo suavemente para asegurarse de que el pellet esté lavado.
NOTA: Este paso es esencial para eliminar los residuos de sal que podrían haber coprecipitado con los ácidos nucleicos. Las sales pueden interferir con otras aplicaciones posteriores. - Centrifugadora a 16.000 g durante 15 min a 4 °C.
- Retire con cuidado todo el sobrenadante y deje que el pellet se seque a RT.
NOTA: La incubación del tubo en un bloque de calentamiento a 37 °C reducirá el tiempo necesario para el secado. - Añadir 50 μL de solución de Tris-EDTA (TE-Buffer) y colocar el tubo en el bloque de calentamiento a 37 °C durante 5-10 min bajo agitación a 300 rpm.
NOTA: Este paso asegura la disolución del pellet. El protocolo se puede pausar aquí, y la muestra se puede almacenar a 4 °C durante un máximo de 1 semana o a -20 °C para un almacenamiento más prolongado. - Realice análisis de ADN en gel de agarosa al 1%.
7. Análisis del tamaño del ADN
- Prepare un gel de agarosa al 1% mezclando 1 g de agarosa por tampón de 100 ml (es decir, Tris-acetato-EDTA (TAE) o Tris-borato-EDTA (TBE)). Caliente la suspensión hasta que la agarosa se disuelva por completo. Añadir 10 μL de EtBr por cada 100 ml de solución de agarosa antes de verter la solución de gel.
PRECAUCIÓN: EtBr es un agente intercalante de ADN conocido por ser cancerígeno. Por favor, manéjelo con las medidas de seguridad necesarias bajo una campana extractora.
NOTA: Se recomienda encarecidamente la tinción de EtBr (directamente en gel o después de la ejecución). Otros tintes intercalantes de ADN no funcionaron bien en nuestras manos cuando trabajamos con frotis de ADN. Los pozos de carga estrechos proporcionan una mejor resolución en comparación con los más anchos. - Mezclar 10 μL de la muestra con 2 μL de 6x tinte de carga. A continuación, cargue 10 μL de la muestra en el gel y pásela hasta que la última banda del tinte de carga corra durante 2/3 del gel. Asegúrese de agregar una escalera de ADN.
- Imagen del gel y verifique si el tamaño del frotis cae en el rango para la aplicación deseada.
Si la cromatina pasa el control de calidad, se puede utilizar para aplicaciones posteriores.
Resultados
La preparación de la cromatina es un paso crucial para lograr un ChIP exitoso. Para preparar cromatina de buena calidad a partir de muestras congeladas, debemos garantizar una alteración tisular eficiente antes de la fijación para evitar la presencia de grumos de tejido que podrían dificultar el cizallamiento eficiente. La figura 1 muestra una canalización resumida del protocolo. La pulverización por sí sola no es suficiente para disociar completamente el tejido, ya que produce grupos de células de tamaño variable y pocas células individuales (Figura 1a). Asociando el primer paso de pulverización con la homogeneización Dounce, la cantidad de grupos de tejido se reduce fuertemente y los restantes son más pequeños (Figura 1b). Después de los pasos de fijación y lisis, el número de núcleos únicos visibles (Figura 1c) aumenta, mientras que la apariencia esférica típica se pierde. Después de la sonicación durante 28 ciclos, la tinción nuclear (Hoechst 33258 / DAPI) ya no es visible. Esto es de hecho un signo de cizallamiento exitoso (Figura 1d). Después de la reticulación de una alícuota de cromatina y la visualización del ADN en gel de agarosa, se puede reconocer un cizallamiento exitoso por la presencia de fragmentos en el rango de 100-300 pb. (Figura 2a) La cantidad de ADN puede variar según la composición de la pieza de tejido preparada. Dicha cromatina se puede utilizar con éxito para ChIP-qPCR. Como se muestra en la Figura 2b , la cromatina podría precipitarse con éxito con anticuerpos H3K4me3, H3K27ac (modificaciones relacionadas con genes activos) y H3K27me3 (modificación relacionada con genes silenciados). Las regiones promotoras del cromosoma 1 Open Reading Frame 43 (C1orf43), Proteasome 20S Subunit Beta 2 (PSMB2) y Glyceraldehyde 3-phosphate dehydrogenase (mGapdh) resultaron enriquecidas en H3K4me3 y H3K27ac en comparación con Homeobox C13 (HOXC13), Homeobox C12 (HOXC12) y las regiones promotoras del factor de transcripción de mielina 1 (mMyt1) de ratón (Tabla 1). Esto se debe a que C1orf43, PSMB2 y mGapdh se transcriben constitutivamente en el hígado, mientras que HOXC13, HOXC12 y mMyt1 se silencian. H3K27me3 muestra el comportamiento opuesto que confirma el éxito del ensayo ChIP. El hecho de que el hígado de estos ratones sea una quimera, nos permitió analizar tanto la cromatina murina como la humana. Además, la misma cromatina podría usarse con éxito para experimentos ChIP-seq. Después del paso de secuenciación, las lecturas se alinearon a un índice compuesto por genomas murinos y humanos para reducir la cantidad de fragmentos no alineados. Posteriormente, las lecturas se separaron según la especie y se analizaron más a fondo con EaSeq22 . La intensidad de la señal se midió en el sitio de inicio de la transcripción (TSS) de cada gen y el resultado se clasificó para la intensidad de la señal H3K4me3. La Figura 3a y la Figura 3c muestran una marcada presencia de H3K4me3 y H3K27ac en el TSS para una parte considerable de los genes dentro de la cromatina humana y de ratón. Además de eso, H3K27me3 se correlaciona con H3K4me3 / H3K27ac. H3K27me3 está presente en toda la longitud del gen y no solo en el TSS, como se esperaba de este PTM. La Figura 3b y la Figura3d muestran el grupo HOXC/HoxC conocido por estar enriquecido para H3K27me3 y transcripcionalmente inactivo tanto en hígados de ratón como humanos. El perfil de H3K4me3 y H3K27ac muestra picos para estos dos PTM, mientras que la intensidad de la señal de H3K27me3 tiende a ser menor y más distribuida.
Debido a la complejidad de la preparación de la cromatina, puede ocurrir una fijación excesiva, el tiempo de lisis o sonicación puede ser subóptimo, los grupos de células grandes pueden persistir o el manejo inadecuado de la muestra puede ser inadecuado. Todos estos son eventos que afectan la calidad de la preparación. En algunos casos, el enriquecimiento de fragmentos de cromatina dentro del tamaño correcto seguirá presente o se desplazará a un tamaño superior. En otros casos, puede haber una pérdida de material debido a la lisis prematura o al cizallamiento fallido. La Figura 4 muestra algunos ejemplos de tales resultados negativos y subóptimos. Los carriles 3 y 4 muestran un enriquecimiento del tamaño del fragmento entre 200 pb y 800 pb. Sin embargo, está claro que el tamaño del fragmento abarca de 100 pb a >10.000 pb. En los carriles 5 y 6 está presente un enriquecimiento en el rango de 100-250 pb con una clara pérdida de material durante la preparación. Esto podría explicar por qué la sonicación produjo fragmentos más pequeños. El carril 7 muestra una preparación ligeramente subóptima con el rango de fragmentos aumentado, mientras que el carril 8 muestra una pérdida casi completa de material. Esto puede ser causado por una lisis nuclear prematura o una disociación tisular insuficiente con la consiguiente pérdida después del paso 5.5.

Figura 1: Descripción general del protocolo de preparación de la cromatina. Las imágenes se tomaron después de la pulverización del tejido (a), homogeneización manual adicional (b), después de la lisis nuclear (c) y después de la sonicación (antes de la centrifugación) (d). La tinción nuclear se realizó con Hoechst 33258/DAPI. Barra de escala = 200 μm. Haga clic aquí para ver una versión más grande de esta figura.

Figura 2: Cizalla representativa de la cromatina, y su calidad evaluada por ChIP-qPCR . Gel de agarosa al 1% con muestras de cromatina fragmentadas según protocolos de diferentes preparaciones de cromatina. Se añade un control de la cromatina sin cizallamiento para garantizar que no se produzca degradación de la cromatina/ADN de antemano (a). La cromatina cortada ha sido probada para determinar su calidad realizando un ensayo ChIP-qPCR. Se utilizaron anticuerpos H3K4me3, H3K27ac y H3K27me3 para precipitar la cromatina recién preparada. (b ) el análisis de qPCR se realizó en promotores activos humanos (C1orf43 y PSMB2), murinos (Gapdh) y humanos (HOXC13, HOXC12), inactivos murinos (Myt1). Haga clic aquí para ver una versión más grande de esta figura.
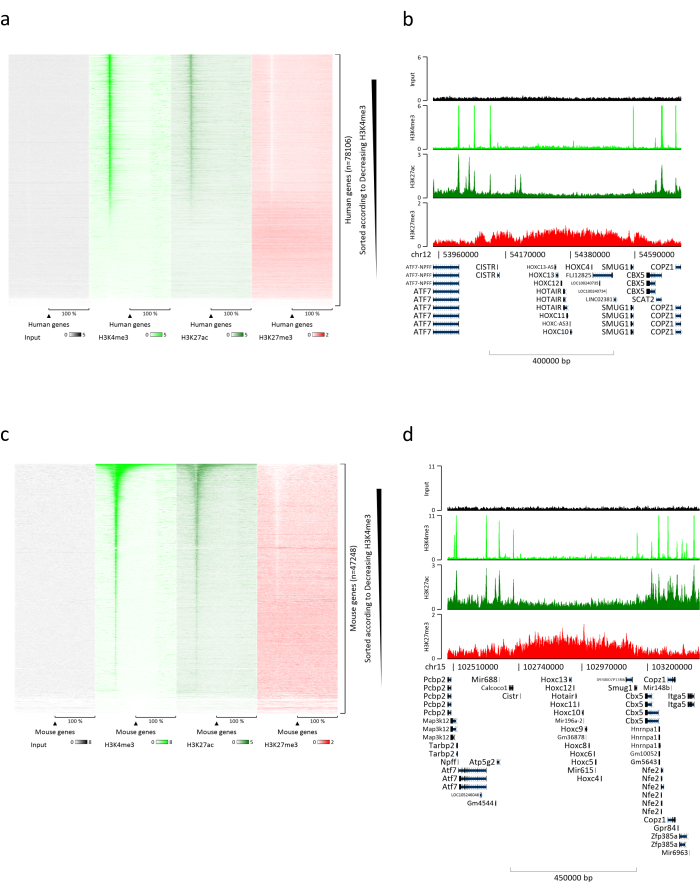
Figura 3: Análisis representativo de ChIP-seq. Las lecturas se han alineado con un índice creado con genomas humanos y de ratón (hg19 y mm10). Después de la alineación, las lecturas humanas y murinas se separaron y analizaron más a fondo. Mapa de calor de genes humanos donde la señal se cuantificó en el TSS y se mostró en orden descendente para la intensidad de H3K4me3 (a). Ejemplo de grupo de genes humanos de genes suprimidos (grupo HOX) rodeado de genes activos (b). Mapa de calor de genes murinos donde la señal se cuantificó en el TSS y se mostró en orden descendente para la intensidad de H3K4me3 (c). Ejemplo de un grupo de genes murinos de genes suprimidos (grupo Hox) rodeado de genes activos (d). Todos los datos mostrados han sido normalizados por EaSeq por millón de lecturas. Haga clic aquí para ver una versión más grande de esta figura.

Figura 4: Preparaciones de cromatina subóptimas y fallidas. Gel de agarosa al 1% con muestras de cromatina fragmentadas según protocolo. La figura contiene cromatina sin cortar utilizada como control (carril 2), cizallamiento no óptimo (carril 3-4), cizallamiento óptimo con pérdida de material claro (carril 5-6), cizallamiento subóptimo (carril 7) y pérdida de material extensiva (carril 8). Haga clic aquí para ver una versión más grande de esta figura.
| Nombre de la cartilla | Secuencia | |
| C1orf43 promotor | Adelante | AGTGGGTGGAGAATGCAGAC |
| Marcha atrás | GAGATTACCCACCCCATTC | |
| Promotor PSMB2 | Adelante | CTTATTCAACCCCCGACAAA |
| Marcha atrás | GATGAAGGACGGTGAGAGGA | |
| Promotor distal HOXC13 | Adelante | GAGCCCGAGATTCACTCAAC |
| Marcha atrás | TTATGCCCAGTTTTGGGGTA | |
| Promotor distal HOXC12 | Adelante | AAAGCTTCCCACTGCAAAGA |
| Marcha atrás | AAATCTGGGGGCGAACTACT | |
| Promotor mGAPDH | Adelante | GGTCCAAAGAGAGGGAGGAGAG |
| Marcha atrás | GCCCTGCTTATCCAGTCCTA | |
| Promotor mMYT | Adelante | CAGCCCAATTCTAGCCACAT |
| Marcha atrás | CCAAAGCAGGGGAGTAGGAG |
Tabla 1: Lista de cebadores de qPCR para genes activos e inactivos utilizados para ensayos ChIP-qPCR.
Discusión
La preparación de la cromatina a partir de tejido congelado a presión sigue siendo un desafío debido a la cantidad de pasos que deben optimizarse para lograr resultados reproducibles y confiables. La mayoría de los protocolos ya publicados16,23 requieren picado de tejidos antes de la disociación manual (douncing). Tratamos de evitar los pasos que podrían provocar la degradación de la proteína antes de la fijación de la muestra tanto como sea posible. La etapa de pulverización ya se utiliza en preparaciones hepáticas congeladas24 y hace que la disociación manual sea más fácil y reproducible (ver Figura 2a). Con el uso de un mortero diseñado específicamente para tubos de 1,5 ml (ver Protocolos), se reduce la pérdida de muestras durante el proceso de pulverización, lo que permite procesar pequeñas cantidades de tejido, como muestras de biopsia hepática. En principio, es posible utilizar la homogeneización directa del tejido sin ningún paso de molienda; Sin embargo, la homogeneización tisular sin pulverización previa tiene una peor reproducibilidad en nuestra experiencia y la aparición de problemas para aplicaciones posteriores fue mayor (datos no mostrados).
La mayoría de los problemas encontrados por la preparación de la cromatina a partir de tejidos se derivan de la naturaleza de estas muestras y de la incapacidad de comprobar adecuadamente si los grupos de células son lo suficientemente pequeños para la fijación sin perder calidad. Además, verificar cada alícuota en cada paso llevaría mucho tiempo, lo que aumentaría la posibilidad de degradación de proteínas.
La fijación (paso 3.9) es una parte fundamental y crucial de la preparación de la cromatina. Debido a la naturaleza del tejido, la etapa de fijación se ha retrasado hasta que el tejido se homogeneizó. Tal paso de fijación pospuesto tiene la ventaja de producir una suspensión celular más homogénea. Sin embargo, reconocemos que, en caso de objetivos particularmente sensibles a la manipulación, puede ser necesario realizar la fijación justo antes del paso 3.6. Esto ayudaría a proteger proteínas extremadamente sensibles o PTM, aunque puede aumentar el tamaño de los grupos de células, que cuando se fijan pueden resultar en un cizallamiento no homogéneo. La concentración de la solución de FA utilizada en el protocolo es estándar, sin embargo, se puede modificar para tratar de mejorar la fijación general. El tiempo de fijación elegido aquí también refleja las condiciones estándar comúnmente utilizadas en el campo. En caso de mayor concentración de la solución de fijación, el tiempo de fijación puede reducirse, mientras que en caso de una cantidad menor debe aumentarse. El operador debe considerar que un cambio en el tiempo de fijación puede dar lugar a una fijación excesiva de la muestra o dar lugar a la degradación de proteínas. En caso de apuntar a precipitar grandes complejos (o parte de ellos) y TFs, sería ventajoso realizar una fijación de doble paso utilizando una solución DSG seguida de una FA25,26. DSG en este caso estabilizaría las interacciones proteína-proteína, mientras que el formaldehído actúa principalmente sobre las interacciones directas ADN-proteína27.
El operador debe tener en cuenta la posibilidad de implementar un kit basado en columnas para la purificación del ADN a partir del paso 6.7, que es más rápido y no utiliza compuestos tóxicos. Sin embargo, siempre habrá una cierta cantidad de ADN no unido que se perderá. Por esta razón, sugerimos utilizar la extracción clásica de fenol-cloroformo seguida de precipitación de EtOH. Además, antes de ejecutar el gel de agarosa (paso 7.2) podría ser beneficioso medir la concentración de ADN y cargar la misma cantidad para que cada pocillo tenga una imagen más clara.
Una limitación de este protocolo se deriva del hecho de que exploramos y utilizamos este protocolo solo utilizando muestras de hígado derivadas de ratones quiméricos de hígado humano28. Per se el hígado está constituido por tejido epitelial y conectivo29. En caso de enfermedad, el tejido fibrótico y el tejido graso pueden estar presentes30,31 creando desafíos adicionales durante la alteración del tejido. Sin embargo, reconocemos que nuestro protocolo no se puede utilizar en huesos, músculos y tejido adiposo sin la optimización de los pasos de disociación y sonicación. Cabe destacar que cada tejido requiere algún tipo de optimización debido a la ausencia de un protocolo adecuado para todos ellos como para las muestras de cultivo celular15. Creemos, sin embargo, que con poca o ninguna optimización en absoluto, este protocolo podría aplicarse con éxito a otros tejidos que comparten similitudes con el hígado en su composición, como los tejidos pulmonares, intestinales, estomacales, pancreáticos o renales.
Nuestro protocolo también se ha utilizado con éxito para analizar TFs y modificaciones de histonas en el episoma de ADN cerrado covalentemente del VHB (cccDNA)32. Esto abre la oportunidad de aplicar este enfoque para otros genomas virales que afectan al hígado, como el citomegalovirus humano33 (hCMV) y los adenovirus humanos34 (HAdV). No se excluye que sea posible analizar otros virus de ADN que establecen una infección persistente en otros tejidos como el virus del herpes del sarcoma de Kaposi35 (KHSV), el virus del polioma del virus del herpes simple36 (HSV1/2), el virus de Epstein-Barr37 (EBV).
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
El estudio fue apoyado por la Fundación Alemana de Investigación (DFG) por una subvención a Maura Dandri (SFB 841 A5) y por el Estado de Hamburgo con el Programa de Investigación (LFF-FV44: EPILOG).
Nos gustaría agradecer al Dr. Tassilo Volz, Yvonne Ladiges y Annika Volmari por la ayuda técnica y por leer críticamente el manuscrito. Dr. Thomas Günther y Prof. Adam Grundhoff por proporcionar sugerencias muy útiles y los conjuntos de cebadores para el análisis ChIP-qPCR.
Materiales
| Name | Company | Catalog Number | Comments |
| 0.22µm sterile syringe filter | Labsolute | 7699822 | |
| 1.5 mL Safeseal tubes | Sarstedt | 7,27,06,400 | |
| 6x orange loading dye | Thermofisher | R0631 | |
| Benchtop refrigerated centrifuge | |||
| Bioruptor NGS | Diagenode | ||
| Blade or Scalpel | |||
| Calcium chloride dihydrate | Carl Roth | HN04 | |
| Chloroform | Sigma Aldrich (Merck) | C2432 | |
| cOmplete Protease Inhibitor Cocktail | Roche | 11697498001 | |
| Deacetylase Inhibitor | Active Motif | 37494 | |
| Dounce tissue grinder set | Sigma Aldrich (Merck) | DWK885300-0001-1EA | |
| EDTA 500 mM solution | PanReac AppliChem | A4892 | |
| EGTA | Sigma Aldrich (Merck) | E4378 | |
| EtBr | Carl Roth | 2218 | Concentration 10mg/mL |
| Ethanol absolute | CHEMSOLUTE | 2273 | |
| Glycerol | Sigma Aldrich (Merck) | G9012 | |
| Glycin | Carl Roth | 0079 | |
| Glycogen | Roche | 10901393001 | Concentration: 20mg/mL |
| Heating block | |||
| HEPES | Sigma Aldrich (Merck) | H4034 | |
| LE Agarose | Biozym | 840000 | |
| Liquid nitrogen cooled mini mortar | Bel-Art | H37260-0100 | |
| MeOH free Formaldehyde 16% | Thermofisher | 28908 | |
| NP-40 | Roche | 11332473001 | |
| PBS 1x | Thermofisher | 10010015 | |
| Pefabloc SC-Protease-Inhibitor | Sigma Aldrich (Merck) | 11429868001 | |
| Phase Lock Gel - Heavy | QuantaBio | 2302830 | |
| Phenol:Chloroform:Isoamyl alcohol 25:24:1 | Sigma Aldrich (Merck) | P3803 | |
| Potassium chloride | Carl Roth | 6781 | |
| Potassium hydroxyde | Merck | 105033 | |
| Proteinase K | Lucigen | MPRK092 | Concentration: 50 µg/µL |
| RNAse A | Lucigen | MRNA092 | Concentration: 5 mg/mL |
| SDS 10% solution | PanReac AppliChem | A3950 | |
| Sodium carbonate anhydrous | Carl Roth | A135 | |
| Sodium chloride | Sigma Aldrich (Merck) | S7653 | |
| Sterile Petri dishes | Sarstedt | 83,39,02,500 | |
| Tris-HCl solution | Sigma Aldrich (Merck) | T2694 | |
| Triton-X100 | Sigma Aldrich (Merck) | X100 |
Referencias
- Waddington, C. H., Pantelouris, E. M. Transplantation of nuclei in newt's eggs. Nature. 172 (4388), 1050-1051 (1953).
- Silmon de Monerri, N. C., Kim, K. Pathogens hijack the epigenome: A new twist on host-pathogen interactions. American Journal of Pathology. 184 (4), 897-911 (2014).
- Knipe, D. M., et al. Snapshots: chromatin control of viral infection. Virology. 435 (1), 141-156 (2013).
- Tropberger, P., et al. Mapping of histone modifications in episomal HBV cccDNA uncovers an unusual chromatin organization amenable to epigenetic manipulation. Proceedings of the National Academy of Sciences of the United States of America. 112 (42), 5715-5724 (2015).
- Sproul, D., Gilbert, N., Bickmore, W. A. The role of chromatin structure in regulating the expression of clustered genes. Nature Reviews Genetics. 6 (10), 775-781 (2005).
- Fischle, W., Wang, Y., Allis, C. D. Histone and chromatin cross-talk. Current Opinion in Cell Biology. 15 (2), 172-183 (2003).
- Ling, X., Harkness, T. A., Schultz, M. C., Fisher-Adams, G., Grunstein, M. Yeast histone H3 and H4 amino termini are important for nucleosome assembly in vivo and in vitro: redundant and position-independent functions in assembly but not in gene regulation. Genes & Development. 10 (6), 686-699 (1996).
- Zhang, L., Eugeni, E. E., Parthun, M. R., Freitas, M. A. Identification of novel histone post-translational modifications by peptide mass fingerprinting. Chromosoma. 112 (2), 77-86 (2003).
- Wang, H., et al. Role of histone H2A ubiquitination in Polycomb silencing. Nature. 431 (7010), 873-878 (2004).
- Hassa, P. O., Haenni, S. S., Elser, M., Hottiger, M. O. Nuclear ADP-ribosylation reactions in mammalian cells: where are we today and where are we going. Microbiology and Molecular Biology Reviews. 70 (3), 789-829 (2006).
- Dey, B., et al. DNA-protein interactions: methods for detection and analysis. Molecular and Cellular Biochemistry. 365 (1-2), 279-299 (2012).
- Hager, G. L., McNally, J. G., Misteli, T. Transcription dynamics. Molecular Cell. 35 (6), 741-753 (2009).
- Nagy, Z., Tora, L. Distinct GCN5/PCAF-containing complexes function as co-activators and are involved in transcription factor and global histone acetylation. Oncogene. 26 (37), 5341-5357 (2007).
- Solomon, M. J., Larsen, P. L., Varshavsky, A. Mapping protein-DNA interactions in vivo with formaldehyde: evidence that histone H4 is retained on a highly transcribed gene. Cell. 53 (6), 937-947 (1988).
- Gunther, T., Theiss, J. M., Fischer, N., Grundhoff, A. Investigation of viral and host chromatin by ChIP-PCR or ChIP-Seq analysis. Current Protocols in Microbiology. 40, 11-21 (2016).
- Cotney, J. L., Noonan, J. P. Chromatin immunoprecipitation with fixed animal tissues and preparation for high-throughput sequencing. Cold Spring Harbor Protocols. 2015 (4), 419 (2015).
- Haim, Y., Tarnovscki, T., Bashari, D., Rudich, A. A chromatin immunoprecipitation (ChIP) protocol for use in whole human adipose tissue. American Journal of Physiology-Endocrinology and Metabolism. 305 (9), 1172-1177 (2013).
- Castellano-Castillo, D., et al. Chromatin immunoprecipitation improvements for the processing of small frozen pieces of adipose tissue. PLoS One. 13 (2), 0192314 (2018).
- Savic, D., Gertz, J., Jain, P., Cooper, G. M., Myers, R. M. Mapping genome-wide transcription factor binding sites in frozen tissues. Epigenetics Chromatin. 6 (1), 30 (2013).
- Perna, A., Alberi, L. A. TF-ChIP method for tissue-specific gene targets. Frontiers Cell Neuroscience. 13, 95 (2019).
- Allweiss, L., et al. Proliferation of primary human hepatocytes and prevention of hepatitis B virus reinfection efficiently deplete nuclear cccDNA in vivo. Gut. 67 (3), 542-552 (2018).
- Lerdrup, M., Johansen, J. V., Agrawal-Singh, S., Hansen, K. An interactive environment for agile analysis and visualization of ChIP-sequencing data. Nature Structural & Molecular Biology. 23 (4), 349-357 (2016).
- Perna, A., Alberi, L. A. TF-ChIP Method for Tissue-Specific Gene Targets. Frontiers in Cellular Neuroscience. 13, 95 (2019).
- Liang, N., Fan, R., Goni, S., Treuter, E. Preparation of Frozen Liver Tissues for Integrated Omics Analysis. Methods in Molecular Biology. 1951, 167-178 (2019).
- Liu, Z., et al. Proteomic and network analysis of human serum albuminome by integrated use of quick crosslinking and two-step precipitation. Scientific Reports. 7 (1), 9856 (2017).
- Singh, A. A., et al. Optimized ChIP-seq method facilitates transcription factor profiling in human tumors. Life Science Alliance. 2 (1), 201800115 (2019).
- Aoki, T., et al. Bi-functional cross-linking reagents efficiently capture protein-DNA complexes in Drosophila embryos. Fly. 8 (1), 43-51 (2014).
- Allweiss, L., Dandri, M. Experimental in vitro and in vivo models for the study of human hepatitis B virus infection. Journal of Hepatology. 64, 17-31 (2016).
- Krishna, M. Microscopic anatomy of the liver. Clinics in Liver Disease. 2, 4-7 (2013).
- Tannapfel, A., et al. Histopathological diagnosis of non-alcoholic and alcoholic fatty liver disease. Virchows Archiv. 458 (5), 511-523 (2011).
- Schuppan, D., Afdhal, N. H. Liver cirrhosis. Lancet. 371 (9615), 838-851 (2008).
- Allweiss, L., et al. Therapeutic shutdown of HBV transcripts promotes reappearance of the SMC5/6 complex and silencing of the viral genome in vivo. Gut. , 322571 (2021).
- Gerna, G., Kabanova, A., Lilleri, D. Human cytomegalovirus cell tropism and host cell receptors. Vaccines. 7 (3), (2019).
- Echavarria, M. Adenoviruses in immunocompromised hosts. Clinical Microbiology Reviews. 21 (4), 704-715 (2008).
- Frohlich, J., Grundhoff, A. Epigenetic control in Kaposi sarcoma-associated herpesvirus infection and associated disease. Seminars in Immunopathology. 42 (2), 143-157 (2020).
- Nicoll, M. P., Proenca, J. T., Efstathiou, S. The molecular basis of herpes simplex virus latency. FEMS Microbiology Reviews. 36 (3), 684-705 (2012).
- Thorley-Lawson, D. A., Hawkins, J. B., Tracy, S. I., Shapiro, M. The pathogenesis of Epstein-Barr virus persistent infection. Current Opinion in Virology. 3 (3), 227-232 (2013).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados