Method Article
Extraction de chromatine à partir de tissu hépatique chimérique congelé pour l’analyse de l’immunoprécipitation de la chromatine
Dans cet article
Résumé
Ce protocole se concentre sur la préparation de la chromatine à partir de tissus congelés et convient à l’immunoprécipitation de la chromatine par réticulation (X-ChIP) suivie d’une analyse PCR quantitative (X-ChIP-qPCR) ou d’approches de séquençage de nouvelle génération (X-ChIP-seq).
Résumé
L’immunoprécipitation de la chromatine réticulée (X-ChIP) est une technique largement utilisée pour évaluer les niveaux de marques d’histones et l’occupation des facteurs de transcription sur la chromatine de l’hôte et/ou de l’agent pathogène. La préparation de la chromatine à partir de tissus crée des défis supplémentaires qui doivent être surmontés pour obtenir des protocoles reproductibles et fiables comparables à ceux utilisés pour la culture cellulaire. La perturbation et la fixation des tissus sont des étapes essentielles pour obtenir un cisaillement efficace de la chromatine. La coexistence de différents types de cellules et de grappes peut également nécessiter des temps de cisaillement différents pour atteindre une taille optimale des fragments et entrave la reproductibilité du cisaillement. Le but de cette méthode est d’obtenir des préparations de chromatine hôte fiables et reproductibles à partir de tissus congelés (foie) adaptées à la fois aux applications ChIP-qPCR et de séquençage de nouvelle génération (NGS). Nous avons observé que la combinaison de pulvérisation tissulaire à l’azote liquide suivie d’homogénéisation conduit à une reproductibilité accrue par rapport à l’homogénéisation seule, car elle fournit une suspension constituée principalement de cellules individuelles dissociées qui peuvent être efficacement cisaillées. De plus, l’étape de fixation doit être effectuée sous rotation légère pour fournir une réticulation homogène. Le matériel fixé convient alors à l’isolement des noyaux à base de tampon, afin de réduire la contamination des ADN et des ARN des protéines cytoplasmiques et des agents pathogènes (le cas échéant), en évitant les gradients de centrifugation fastidieux. La sonication ultérieure complétera la lyse nucléaire et cisaillera la chromatine, produisant une gamme de tailles spécifique en fonction des conditions de cisaillement choisies. La plage de taille devrait se situer entre 100 et 300 nt pour les applications NGS, alors qu’elle pourrait être plus élevée (300-700 nt) pour l’analyse ChIP-qPCR. De telles adaptations de protocole peuvent grandement améliorer les analyses de chromatine à partir d’échantillons de tissus congelés.
Introduction
Depuis sa découverte, la régulation épigénétique dans les cellules de mammifères a gagné en reconnaissance1, considérant que la compréhension de tels mécanismes fournirait des informations clés non seulement en biologie cellulaire, mais aussi en biologie des maladies et des tumeurs. De plus, les agents infectieux peuvent également provoquer des modifications épigénétiques de l’hôte2 tandis que la machinerie des cellules hôtes peut également affecter la chromatine des agents pathogènes, tels que les virus à ADN persistants 3,4. Cette interaction hôte-pathogène semble jouer un rôle dans la persistance de l’infection. deux
Grâce à une association réversible avec l’ADN, les protéines histones forment un complexe appelé nucléosome. Les nucléosomes atteignent à leur tour un niveau d’organisation plus élevé connu sous le nom de chromatine. Le remodelage de la chromatine est connu pour réguler étroitement l’expression des gènes, accordant ou refusant l’accès aux facteurs de transcription (TF)5. Ces facteurs peuvent déclencher ou bloquer le recrutement de l’ARN polymérase II (PolII) sur les promoteurs de gènes, influençant la synthèse de l’ARNm à partir du modèle d’ADN6. Les protéines histones contiennent des queues7, flanquant les deux extrémités du pli des histones, qui peuvent être soumises à des modifications post-traductionnelles (PTM), permettant une régulation étroite de la transcription du gène par des changements structurels de la chromatine. La plupart des PTM d’histones sont situés à l’extrémité N de la queue, l’acétylation et la méthylation étant les PTM les mieux étudiés, bien que la phosphorylation8, l’ubiquitination9 et la ribosylation10 aient également été rapportées. La caractérisation et l’étude de ces protéines sont alors essentielles pour obtenir un aperçu approfondi de la régulation des gènes.
Actuellement, il existe une poignée de méthodes et d’outils bien établis pour étudier les interactions directes ADN-protéine : test de déplacement de mobilité électrophorétique (EMSA), test mono-hybride de levure (Y1H) et empreinte ADN11. Cependant, ces méthodes se concentrent en soi sur les interactions ADN et protéines uniques et ne sont pas applicables aux études à l’échelle du génome. Une autre limite de ces techniques est l’absence d’association d’histones avec les segments d’ADN étudiés. Ainsi, de telles approches ne sont pas censées refléter la complexité de la machinerie transcriptionnelle in vivo et elles ne tiennent pas compte des changements structurels importants12 ou d’autres enzymes/cofacteursrequis 13 qui pourraient influencer (favoriser ou inhiber) la liaison des protéines à l’ADN.
L’idée que la fixation des cellules avec des agents comme le formaldéhyde (FA) pourrait fournir un instantané in vivo des interactions protéine-ADN a jeté les bases du développement de tests d’immunoprécipitation de la chromatine (ChIP)14. Ceci, associé à la disponibilité de la technologie de PCR quantitative (qPCR) et d’anticorps hautement spécifiques, a permis le développement de tests ChIP-qPCR. Par la suite, l’avènement des techniques de séquençage de nouvelle génération (NGS), dont les coûts deviennent de plus en plus abordables, a concédé de coupler les expériences ChIP avec les approches NGS (ChIP-seq), fournissant ainsi aux chercheurs de nouveaux outils puissants permettant d’étudier la régulation de la chromatine. Dans ces essais, des cellules isolées ou cultivées sont fixées avec du glutarate de disuccinimidyl (DSG) et/ou de l’AF, les noyaux sont isolés, la chromatine est ensuite fragmentée et précipitée par l’anticorps d’intérêt. Par la suite, l’ADN est purifié et analysé par PCR ou NGS. Contrairement à l’EMSA, à l’Y1H et à l’empreinte ADN, les tests ChIP ont la capacité de fournir un instantané global de l’interaction protéine-ADN dans la cellule. Cela offre une flexibilité et permet l’analyse de plusieurs loci au sein d’un même échantillon. Cependant, en raison de la nature du test, ChIP peut, à terme, détecter non seulement des interactions directes, mais aussi des interactions indirectes, n’offrant pas la précision des méthodes mentionnées ci-dessus, lorsqu’il s’agit d’interactions directes protéine-ADN.
Les protocoles de préparation de la chromatine à partir de matériel de culture cellulaire sont bien établis15 et hautement reproductibles, permettant à l’utilisateur d’obtenir une chromatine adaptée à la fois aux approches qPCR et NGS en 1-2 jours ouvrables. Cependant, l’obtention de chromatine de haute qualité à partir de tissus entiers représente toujours un défi en raison de la nécessité de dissocier les cellules dans le tissu tout en obtenant une fixation et un cisaillement optimaux de la chromatine. En outre, la composition et la morphologie des différents types de tissus varient, ce qui nécessite un ajustement des protocoles existants16,17. L’utilisation de tissus cryoconservés présente des défis supplémentaires par rapport aux échantillons frais. Cela est dû à la difficulté d’obtenir une suspension à une seule cellule sans perte matérielle importante. Cela conduit à un cisaillement inapproprié, entravant les applications en aval. Néanmoins, l’accès à des échantillons de tissus congelés plutôt qu’à la contrepartie fraîche augmente non seulement la flexibilité du travail, mais peut également représenter la seule option pour les chercheurs travaillant avec des échantillons provenant d’études longitudinales ou comparatives. Une poignée de protocoles de préparation de la chromatine pour les tissus congelés ont été publiés. Celles-ci sont principalement basées sur la décongélation des échantillons suivie des étapes de hachage, de dissociation manuelle / mécanique ou de pulvérisation à l’azote liquide18,19,20.
Nous décrivons ici une méthode optimisée de préparation de la chromatine15 pour des échantillons de foie congelés non fixés, qui combine la pulvérisation des tissus dans l’azote liquide avec l’homogénéisation du pilon, pour obtenir un cisaillement de la chromatine reproductible adapté aux approches X-ChIP visant à analyser à la fois les génomes viraux et hôtes.
Protocole
L’échantillonnage de tissus sur des souris chimériques hépatiques humaines21 a été effectué conformément à la directive 86/609/CEE de l’Union européenne et approuvé par le comité d’éthique de la ville et de l’État de Hambourg conformément aux principes de la Déclaration d’Helsinki.
1. Préparation des réactifs
- Préparer la solution de glycine 1,25 M dans de l’eau désionisée. Filtre stérile avec filtre de la taille d’un pore de 0,22 μm. Conserver à 4 °C.
- Préparer une solution de chlorure de sodium (NaCl) 5 M. Conserver à température ambiante.
- Préparerla solution de CaCl2 : 300 mM de CaCl2et 10 mM de Tris-HCl pH 8 dans de l’eau désionisée. Filtre stérile avec filtre de 0,22 μm de la taille d’un pore et conserver à TA.
- Préparer une dilution Triton X-100 à 10% dans de l’eau désionisée. Magasin chez RT.
- Préparer le tampon Tris-EDTA : 1 mM EDTA et 10 mM Tris pH 8 dans de l’eau désionisée. Conserver à 4 °C.
- Préparez les tampons suivants en fonction de la quantité requise :
- Préparer le tampon A : 50 mM Hepes-KOH pH 7,5, 140 mM NaCl, 1 mM acide éthylènediaminetétraacétique (EDTA), 10 % de glycérol, 0,5 % de NP-40 et 0,25 % de Triton X-100 dans de l’eau désionisée. Filtre stérile avec filtre de la taille d’un pore de 0,22 μm. Conserver à 4 °C.
- Préparer le tampon B : 10 mM Tris-HCl pH 8, 200 mM NaCl, 1mM EDTA, 0,5 mM d’acide égtazique (EGTA). Filtre stérile avec filtre de la taille d’un pore de 0,22 μm. Conserver à 4 °C.
- Préparer le tampon C : 1% SDS, 10 mM EDTA et 50 mM Tris-HCl pH 8 dans de l’eau désionisée. Filtre stérile avec filtre de la taille d’un pore de 0,22 μm. Magasin chez RT.
- Préparer le tampon de dilution de la chromatine : 0,01 % SDS, 1,1 % Triton X-100, 1,2 mM EDTA, 16,6 mM Tris-HCl pH 8 et 166 mM NaCl dans de l’eau désionisée. Filtre stérile avec filtre de la taille d’un pore de 0,22 μm. Conserver à 4 °C.
2. Préparation du matériel
- Recueillez de la glace sèche, de la glace et de l’azote liquide.
ATTENTION : Manipulez la glace sèche et l’azote liquide avec le soin nécessaire pour éviter les brûlures. - Prérefroidir la centrifugeuse à 4 °C.
REMARQUE: Cette étape est importante pour éviter la dégradation des protéines et la réticulation pendant les étapes de lavage, car cela réduira la qualité de la chromatine. - Mettez une assiette stérile sur de la glace sèche et laissez-la refroidir.
REMARQUE: Assurez-vous que la plaque est assez grande pour faciliter le processus de coupe. Une plaque de Petri / boîte de culture cellulaire de 100 mm est recommandée. - Sortez l’aliquote nécessaire de glycine 1,25 M et laissez-la atteindre RT.
- Retirer les aliquotes nécessaires des tampons A, B et PBS. Ajouter de la protéase et/ou de la désacétylase et des inhibiteurs de phosphatase pour atteindre une concentration de 1 fois et laissez-les sur la glace.
- Retirez l’aliquote nécessaire du tampon C en la laissant à RT. N’ajoutez pas les inhibiteurs de la protéase et/ou de la désacétylase et de la phosphatase avant d’être spécifiés.
- Sortez l’aliquote nécessaire de RT PBS.
ATTENTION : Le tampon C contient du dodécylsulfate de sodium (SDS). Adopter les mesures de sécurité appropriées lors de la préparation du tampon.
REMARQUE : Le SDS précipite sur la glace, et les inhibiteurs de la protéase et de la désacétylase ne sont pas stables à la RT. - Prérefroidissez le mortier en versant de l’azote liquide dans sa chambre, en suivant strictement les instructions du fournisseur. Refroidir le pilon métallique dans de la glace sèche pendant au moins 5 min.
NOTE: Il est possible d’utiliser un mortier alternatif à celui proposé. Cependant, le dispositif utilisé dans ce protocole, en raison de sa construction particulière, permet de travailler avec une petite quantité de tissu sans perte substantielle pendant le processus de pulvérisation. - Pré-refroidir l’homogénéisateur Dounce avec le pilon A associé sur la glace.
REMARQUE: Le pilon A a un ajustement lâche avec l’homogénéisateur. Cela permet d’obtenir une suspension unicellulaire sans lyse cellulaire significative.
3. Réticulation tissulaire
- Couper environ 50 mg de tissu congelé directement sur le plat sur de la glace sèche à l’aide d’un scalpel et d’une pince à épiler.
REMARQUE: Il est suggéré de garder le scalpel à RT, car cela facilitera le processus de coupe. Évitez d’appliquer trop de pression sur le scalpel, car cela augmenterait le risque de dispersion des morceaux de tissu à l’extérieur de la zone de coupe. A noter, 50 mg de tissu (foie dans ce cas) devraient donner environ 5 millions de cellules. Notez que la lame chaude dégèlera le tranchant. Compte tenu de la taille relativement grande du morceau de tissu, cependant, cela devrait avoir un effet limité. Lorsque de plus petits morceaux sont coupés, il pourrait être avantageux d’utiliser un scalpel froid en faisant attention pour éviter la dispersion du tissu. - Mettez le tissu coupé dans un tube de 1,5 ml déjà refroidi sur de la glace sèche. Évitez la décongélation des tissus.
- Déplacez le tube contenant le tissu vers le mortier, en le laissant reposer là pendant 5 minutes.
NOTE: Laisser l’échantillon reposer dans le mortier diminue sa température (de -80 ° C à -196 ° C). Cela augmente sa ténacité et facilite l’étape de pulvérisation. - Appliquez une pression sur l’échantillon à l’aide du pilon pré-refroidi jusqu’à ce qu’il n’y ait plus de miettes solides.
REMARQUE: Il est important d’éviter l’échauffement du pilon par des forces de rotation excessives, car cela décongèlerait l’échantillon. Après chaque pulvérisation de l’échantillon, nettoyez le pilon avec de l’éthanol à 70% (EtOH) et laissez-le refroidir à nouveau sur de la glace sèche. - Retirer le tube contenant l’échantillon du mortier et ajouter 950 μL de PBS glacé avec les inhibiteurs requis. Pipeter doucement de haut en bas jusqu’à ce que l’échantillon soit complètement remis en suspension. Passez immédiatement à l’étape 3.6.
- Transférer la suspension tissulaire dans l’homogénéisateur et appliquer 20-30 coups avec le pilon A pour obtenir une suspension plus fine. Évitez la formation de mousse.
REMARQUE: La quantité de traits doit être optimisée en fonction de la consistance tissulaire. Cette étape dissocie davantage les petits amas de cellules obtenus après pulvérisation. Une homogénéisation incorrecte peut affecter l’efficacité du cisaillement. - Transférer l’homogénat dans un nouveau tube de 1,5 mL, déjà prérefroidi sur glace.
- Centrifuger pendant 5 min à 1 300 x g à 4 °C et retirer délicatement le surnageant.
- Remettez complètement en suspension la pastille dans 950 μL de PBS RT par pipetage doux et ajoutez 63,6 μL de FA sans MeOH à 16 % pour obtenir une concentration finale de 1 %. Passez immédiatement à l’étape 3.10.
ATTENTION : L’AF est un produit chimique toxique. Manipulez-le sous une hotte avec les mesures de sécurité appropriées.
REMARQUE: Une remise en suspension incomplète peut provoquer une agrégation cellulaire pendant l’étape de fixation. Cela entrave le processus de lyse et de cisaillement. - Rotation de 10 min à RT. Passez immédiatement à l’étape 3.11
NOTE: La rotation est nécessaire pour éviter les agrégats. Le temps nécessaire à la fixation doit être optimisé en fonction de la cible d’intérêt et du type d’échantillon. Il est important de noter que des temps de fixation excessifs pourraient nuire à un cisaillement approprié. - Ajouter 113 μL de glycine 1,25 M à TA pour obtenir une concentration finale de 125 mM et faire tourner pendant 5 min.
REMARQUE: La glycine éteint la réaction fixatrice en évitant la réticulation excessive. - Centrifuger à 1 300 x g pendant 3 min à 4 °C.
- Jeter le surnageant et remettre la pastille en suspension avec précaution en l’incorporant 950 μL de PBS glacé avec les inhibiteurs requis.
- Centrifuger à 1 300 x g pendant 3 min à 4 °C.
- Répétez les étapes 3.13-3.14 et passez immédiatement aux étapes d’isolement de la chromatine.
4. Isolement de la chromatine
- Ajouter 950 μL de tampon A avec les inhibiteurs requis à la pastille. Mélanger doucement par pipetage jusqu’à ce que la pastille soit complètement remise en suspension et tourner 10 min à 4 °C.
NOTE: Cette étape lyse la suspension unicellulaire fixe, sans lyse des noyaux. Cela permet de débarrasser l’échantillon des protéines cytosoliques et des ARN. Prolonger le temps de lyse pourrait être bénéfique pour les cellules difficiles à lyser, en augmentant, cependant, le temps de manipulation du tissu. À ce stade, il est possible de vérifier la préparation au microscope après coloration au bleu de trypan / DAPI pour vérifier la taille des grappes et la présence de cellules individuelles. Cependant, les noyaux uniques peuvent ne pas être faciles à apprécier en raison du matériau tissulaire fixe. - Centrifuger à 2 000 x g pendant 5 min à 4 °C et retirer délicatement le surnageant.
- Ajouter 950 μL de tampon B avec les inhibiteurs requis à la pastille. Mélanger doucement par pipetage jusqu’à ce que la pastille soit complètement remise en suspension et tourner 10 min à 4 °C.
REMARQUE: Cette étape élimine le tampon de lyse de la préparation des noyaux pour éviter toute autre lyse indésirable. - Centrifuger à 2000 x g pendant 5 min à 4 °C. Pendant ce temps, ajoutez les inhibiteurs requis (identiques à l’étape 2.5) au tampon C.
- Retirez délicatement le surnageant.
- Ajouter vigoureusement 300 μL de tampon RT C à la pastille et à la pipette.
- Tourbillonner l’échantillon pendant 15-30 s et faire tourner brièvement le tube pour recueillir les gouttes sur le couvercle.
REMARQUE: Cette étape est importante pour libérer et lyser les noyaux fixes. Pour préserver l’intégrité de l’échantillon et éviter en même temps les précipitations SDS, conservez l’échantillon avant la sonication dans une grille en plastique maintenue sur glace pour maintenir une température de 9-11 ° C.
5. Fragmentation de la chromatine
- Transférer l’échantillon dans trois tubes propres certifiés sonication de 0,65 mL assurant 100 μL de suspension de noyaux lysés par tube.
REMARQUE: Il est possible d’utiliser des tubes certifiés par sonication de 1,5 mL avec un volume maximal de 300 μL. Un support spécifique pour ces tubes est nécessaire. 0,65 mL devrait offrir un cisaillement plus homogène en raison du plus petit volume de l’échantillon par tube. - Sonicez la chromatine pendant 28 cycles à haute intensité avec le réglage ON et 30 s OFF. Assurez-vous que le bain du sonicateur est correctement refroidi (glace ou dispositif de refroidissement).
REMARQUE: Cette étape nécessite une optimisation dans presque tous les cas. L’utilisateur doit garder à l’esprit que l’augmentation du temps de cisaillement fournira des fragments plus petits et plus homogènes; Cependant, cela peut augmenter les chances de réduire la qualité de la chromatine. Choisissez le plus petit nombre de cycles qui fournit la taille de fragment requise. Lors de l’optimisation de cette étape, il est utile d’effectuer une coloration nucléaire pour vérifier si le nombre de cycles était suffisant pour lyser la majorité des noyaux. - Transférer la chromatine soniquée dans un nouveau tube de 1,5 mL préalablement refroidi sur de la glace.
- Ajouter 30 μL de solution Triton X-100 à 10% et vortex pendant 5-10 s.
NOTE: Triton X-100 lie la FDS empêchant de nouvelles précipitations à 4 ° C. La quantité finale de Triton X-100 devrait toujours être de 1%. - Centrifuger à 16 000 x g pendant 15 min à 4 °C.
- Transférer le surnageant dans un tube propre de 1,5 mL prérefroidi sur de la glace.
- NOTE: Le surnageant contient la chromatine cisaillée et doit apparaître clair. La pastille contient des restes « non tondables » et elle doit rester assez petite (principalement brune dans le cas du tissu hépatique). Recherchez une indication de cisaillement infructueux: solution de chromatine qui n’est pas devenue plus claire et des dimensions de granulés similaires à celles de l’étape 4.5.
6. Purification de l’ADN
- Transférer 10-25 μL de chromatine cisaillée dans un nouveau tube et ajouter le tampon C pour atteindre un volume final de 200 μL. Conserver le reste de la chromatine à -80 °C jusqu’à nouvelle utilisation. Si nécessaire, la procédure peut être interrompue à cette étape et l’échantillon stocké à -20 °C.
- Ajouter 8 μL de NaCl 5 M et incuber au moins 6 h à 65 °C dans un bloc chauffant sous agitation à 1000 tr/min.
REMARQUE: Cette étape dé-réticulation de la chromatine. Il est plus sûr de prolonger la réticulation du jour au lendemain lorsque cela est possible. La présence de NaCl rend le processus plus efficace. - Laisser refroidir les échantillons à TA pendant 5 min et ajouter 2 μL de RNase A.
- Incuber pendant 1 h à 37 °C sous agitation à 1000 tr/min.
- Retirer les échantillons du bloc chauffant et ajouter 7 μL de CaCl 2 à 300 mM et2 μL de protéinase K.
- Régler le bloc chauffant à 56 °C et incuber pendant 30 min sous agitation à 1000 tr/min. Pendant ce temps, préparez un tube de séparation de phase pour chaque échantillon en les centrifugeant à 16 000 x g pendant 1 min à 4 °C.
REMARQUE: Ces tubes spéciaux facilitent la séparation des phases lors de l’extraction phénol-chloroforme des acides nucléiques. - Retirez les tubes du bloc chauffant et laissez-les s’équilibrer à TA pendant 3 min.
- Transférer 400 μL de l’échantillon dans un tube de séparation de phase préalablement centrifugé.
- Ajouter 400 μL de solution d’alcool phénol-chloroforme-isoamylique (PCI) et vortex pendant 5 s.
ATTENTION : Le PCI est un composé hautement volatil et toxique. Veuillez le manipuler avec les mesures de sécurité nécessaires sous une hotte. - Centrifuger à 16 000 x g pendant 5 min à 4 °C.
- Ajouter 400 μL de chloroforme et vortex pendant 5 s.
ATTENTION : Le chloroforme est un composé hautement volatil et toxique. Veuillez le manipuler avec les mesures de sécurité nécessaires sous une hotte.
REMARQUE: Cette étape élimine les résidus possibles de phénol, qui pourraient interférer avec les applications de PCR en aval. - Centrifuger à 16 000 x g pendant 5 min à 4 °C.
- Transférer 400 μL de la phase supérieure dans un nouveau tube de 1,5 mL où 24 μL de NaCl 5 M et 0,75 μL de glycogène ont été ajoutés. Brièvement vortex.
- Ajouter 1 055 μL d’EtOH à 100 % et bien vortex. Assurez-vous d’un bon mélange.
- Incuber à -80 °C pendant 1 h ou à -20 °C pendant une nuit (ON).
NOTE: Cette étape précipite l’ADN cisaillé; pour maximiser le rendement, il est suggéré de choisir l’incubation ON. - Centrifuger à 16 000 x g pendant 30 min à 4 °C.
- Retirez délicatement le surnageant en faisant attention à ne pas disloquer le granulé.
- Ajouter 500 μL d’EtOH froid à 70%. Inclinez doucement le tube pour vous assurer que le granulé est lavé.
NOTE: Cette étape est essentielle pour éliminer les résidus de sel qui auraient pu co-précipiter avec les acides nucléiques. Les sels peuvent interférer avec d’autres applications en aval. - Centrifuger à 16 000 x g pendant 15 min à 4 °C.
- Retirez soigneusement le surnageant entier et laissez sécher la pastille à TA.
NOTE: L’incubation du tube sur un bloc chauffant à 37 °C réduira le temps nécessaire au séchage. - Ajouter 50 μL de solution de Tris-EDTA (TE-Buffer) et placer le tube sur le bloc chauffant à 37 °C pendant 5-10 min sous agitation à 300 tr/min.
REMARQUE: Cette étape assure la dissolution des granulés. Le protocole peut être mis en pause ici, et l’échantillon peut être conservé à 4 °C jusqu’à 1 semaine ou à -20 °C pour un stockage plus long. - Effectuer une analyse ADN sur du gel d’agarose à 1%.
7. Analyse de la taille de l’ADN
- Préparer un gel d’agarose à 1 % en mélangeant 1 g d’agarose par tampon courant de 100 ml (c.-à-d. tris-acétate-EDTA (TAE) ou tris-borate-EDTA (TBE)). Chauffer la suspension jusqu’à ce que l’agarose soit complètement dissoute. Ajouter 10 μL d’EtBr pour chaque 100 mL de solution d’agarose avant de verser la solution de gel.
ATTENTION : L’EtBr est un agent intercalant de l’ADN connu pour être cancérigène. Veuillez le manipuler avec les mesures de sécurité nécessaires sous une hotte.
REMARQUE: La coloration EtBr (directement dans le gel ou après la course) est fortement suggérée. D’autres colorants intercalants d’ADN n’ont pas bien fonctionné entre nos mains lorsque nous travaillions avec des frottis d’ADN. Les puits de chargement étroits offrent une meilleure résolution que les puits plus larges. - Mélanger 10 μL de l’échantillon avec 2 μL de colorant de chargement 6x. Ensuite, chargez 10 μL de l’échantillon dans le gel et faites-le fonctionner jusqu’à ce que la dernière bande du colorant de chargement fonctionne sur les 2/3 du gel. Assurez-vous d’ajouter une échelle d’ADN.
- Imagez le gel et vérifiez si la taille du frottis se situe dans la plage pour l’application souhaitée.
Si la chromatine passe le contrôle qualité, elle peut être utilisée pour des applications en aval.
Résultats
La préparation de la chromatine est une étape cruciale dans l’obtention d’un ChIP réussi. Afin de préparer une chromatine de bonne qualité à partir d’échantillons congelés, nous devons assurer une perturbation tissulaire efficace avant la fixation afin d’éviter la présence d’amas tissulaires qui pourraient entraver un cisaillement efficace. La figure 1 montre un pipeline résumé du protocole. La pulvérisation seule n’est pas suffisante pour dissocier complètement le tissu puisqu’elle produit des amas cellulaires de taille variable et peu de cellules individuelles (Figure 1a). En associant la première étape de pulvérisation à l’homogénéisation de Dounce, la quantité d’amas tissulaires est fortement réduite et les autres sont plus petits (Figure 1b). Après les étapes de fixation et de lyse, le nombre de noyaux simples visibles (Figure 1c) augmente, tandis que l’aspect sphérique typique est perdu. Après la sonication pendant 28 cycles, la coloration nucléaire (Hoechst 33258 / DAPI) n’est presque plus visible. C’est en effet un signe de cisaillement réussi (Figure 1d). Après la réticulation d’une chromatine aliquote et la visualisation de l’ADN sur gel d’agarose, un cisaillement réussi peut être reconnu par la présence de fragments dans la plage de 100-300 bp. (Figure 2a) La quantité d’ADN peut varier en fonction de la composition du morceau de tissu préparé. Une telle chromatine peut être utilisée avec succès pour ChIP-qPCR. Comme le montre la figure 2b , la chromatine a pu être précipitée avec succès avec les anticorps H3K4me3, H3K27ac (modifications liées aux gènes actifs) et H3K27me3 (modification liée aux gènes silencieux). Le chromosome 1 Open Reading Frame 43 (C1orf43), le protéasome 20S Subunit Beta 2 (PSMB2) et les régions promotrices de la glycéraldéhyde 3-phosphate déshydrogénase (mGapdh) se sont enrichis en H3K4me3 et H3K27ac par rapport aux régions promotrices Homeobox C13 (HOXC13), Homeobox C12 (HOXC12) et du facteur de transcription 1 de la myéline de souris (mMyt1) (tableau 1). En effet, C1orf43, PSMB2 et mGapdh sont transcrits de manière constitutive dans le foie, tandis que HOXC13, HOXC12 et mMyt1 sont réduits au silence. H3K27me3 montre le comportement inverse confirmant le succès du test ChIP. Le fait que le foie de ces souris soit une chimère nous a permis d’analyser à la fois la chromatine murine et la chromatine humaine. En outre, la même chromatine pourrait être utilisée avec succès pour les expériences ChIP-seq. Après l’étape de séquençage, les lectures ont été alignées sur un index composé à la fois de génomes murin et humain afin de réduire la quantité de fragments non alignés. Par la suite, les lectures ont été séparées selon les espèces et analysées plus en détail avec EaSeq22 . L’intensité du signal a ensuite été mesurée au site de début de la transcription (TSS) de chaque gène et le résultat a été trié pour l’intensité du signal H3K4me3. Les figures 3a et 3c montrent une présence marquée de H3K4me3 et H3K27ac au SCT pour une partie considérable des gènes dans la chromatine de souris et humaine. En plus de cela, les anticorrélats H3K27me3 avec H3K4me3/H3K27ac. H3K27me3 est présent sur toute la longueur du gène et pas seulement au niveau du SCT, comme on peut s’y attendre de ce PTM. La figure 3b et la figure3d montrent le cluster HOXC/HoxC connu pour être enrichi en H3K27me3 et inactif transcriptionnellement dans les foies de souris et d’humains. Le profilage de H3K4me3 et H3K27ac montre des pics pour ces deux PTM tandis que l’intensité du signal de H3K27me3 tend à être plus faible et plus distribuée.
En raison de la complexité de la préparation de la chromatine, une surfixation peut se produire, le temps de lyse ou de sonication peut être sous-optimal, les amas de grosses cellules peuvent persister ou une manipulation inadéquate de l’échantillon peut être inadéquate. Ce sont tous des événements affectant la qualité de la préparation. Dans certains cas, l’enrichissement des fragments de chromatine dans la taille correcte sera toujours présent ou sera déplacé vers une taille supérieure. Dans d’autres cas, il peut y avoir une perte de matériau due à une lyse prématurée ou à un cisaillement infructueux. La figure 4 montre quelques exemples de ces résultats négatifs et sous-optimaux. Les voies 3 et 4 montrent un enrichissement de la taille du fragment entre 200 bp et 800 bp. Cependant, il est clair que la taille du fragment s’étend de 100 pb à 10 000 pb >. Dans les voies 5 et 6, un enrichissement de l’ordre de 100 à 250 pb est présent avec une perte nette de matière pendant la préparation. Cela pourrait expliquer pourquoi la sonication a produit des fragments plus petits. La voie 7 montre une préparation légèrement sous-optimale avec l’augmentation de la portée des fragments, tandis que la voie 8 montre une perte presque complète de matériau. Cela peut être causé par une lyse nucléaire prématurée ou une dissociation tissulaire insuffisante avec une perte conséquente après l’étape 5.5.

Figure 1 : Vue d’ensemble du protocole de préparation de la chromatine. Des photos ont été prises après pulvérisation tissulaire (a), homogénéisation manuelle supplémentaire (b), après lyse nucléaire (c) et après sonication (avant centrifugation) (d). La coloration nucléaire a été réalisée avec Hoechst 33258/DAPI. Barre d’échelle = 200 μm. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 2 : Cisaillement représentatif de la chromatine et évaluation de sa qualité par ChIP-qPCR. Gel d’agarose à 1% avec des échantillons de chromatine fragmentés selon des protocoles de différentes préparations de chromatine. Un contrôle de la chromatine non cisaillée est ajouté pour s’assurer qu’il n’y a pas de dégradation de la chromatine/ADN au préalable (a). La chromatine cisaillée a été testée pour la qualité en effectuant un test ChIP-qPCR. Les anticorps H3K4me3, H3K27ac et H3K27me3 ont été utilisés pour précipiter la chromatine fraîchement préparée. b) l’analyse qPCR a été réalisée sur des promoteurs actifs humains (C1orf43 et PSMB2), murins (Gapdh) et inactifs humains (HOXC13, HOXC12) et murins (Myt1). Veuillez cliquer ici pour voir une version agrandie de cette figure.
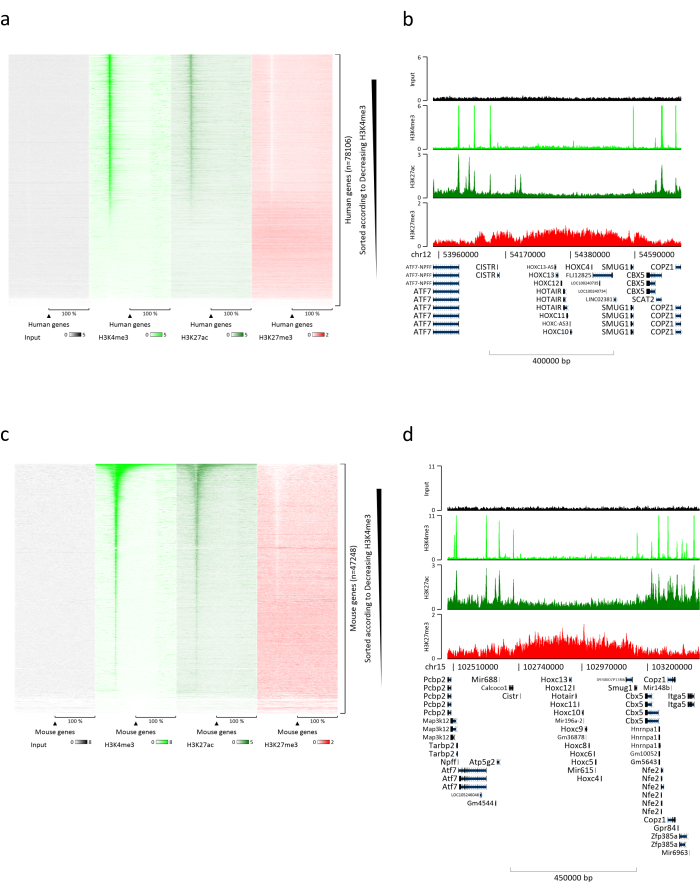
Figure 3 : Analyse ChIP-seq représentative. Les lectures ont été alignées sur un index créé avec les génomes humain et murin (hg19 et mm10). Après alignement, les lectures humaines et murines ont été séparées et analysées plus avant. Carte thermique des gènes humains où le signal a été quantifié au TSS et montré par ordre décroissant pour l’intensité H3K4me3 (a). Exemple de groupe de gènes humains de gènes supprimés (groupe HOX) entouré de gènes actifs (b). Carte thermique des gènes murins où le signal a été quantifié au TSS et montré par ordre décroissant pour l’intensité H3K4me3 (c). Exemple d’un groupe de gènes murins de gènes supprimés (amas Hox) entouré de gènes actifs (d). Toutes les données affichées ont été normalisées par EaSeq par million de lectures. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 4 : Préparations de chromatine sous-optimales et échouées. Gel d’agarose à 1% avec échantillons de chromatine fragmentés selon le protocole. La figure contient la chromatine non cisaillée utilisée comme témoin (voie 2), le cisaillement non optimal (voie 3-4), le cisaillement optimal avec perte de matière nette (voie 5-6), le cisaillement sous-optimal (voie 7) et la perte importante de matière (voie 8). Veuillez cliquer ici pour voir une version agrandie de cette figure.
| Nom de l’amorce | Séquence | |
| Promoteur C1orf43 | En avant | AGTGGGTGGAGAATGCAGAC |
| Inverse | GAGATTACCCCACCCCATTC | |
| Promoteur PSMB2 | En avant | CTTATTCAACCCCCGACAAA |
| Inverse | GATGAAGGACGGTGAGAGGA | |
| Promoteur distal HOXC13 | En avant | GAGCCCGAGATTCACTCAAC |
| Inverse | TTATGCCCAGTTTTGGGGTA | |
| Promoteur distal HOXC12 | En avant | AAAGCTTCCCACTGCAAAGA |
| Inverse | AAATCTGGGGGCGAACTACT | |
| Promoteur mGAPDH | En avant | GGTCCAAAGAGAGGGAGGAG |
| Inverse | GCCCTGCTTATCCAGTCCTA | |
| Promoteur mMYT | En avant | CAGCCCAATTCTAGCCACAT |
| Inverse | CCAAAGCAGGGGAGTAGGAG |
Tableau 1 : liste des amorces qPCR pour les gènes actifs et inactifs utilisés pour les tests ChIP-qPCR.
Discussion
La préparation de la chromatine à partir de tissus congelés reste un défi en raison du nombre d’étapes qui doivent être optimisées afin d’obtenir des résultats reproductibles et fiables. La plupart des protocoles déjà publiés 16,23 nécessitent un hachage tissulaire avant la dissociation manuelle (décollage). Nous avons essayé d’éviter autant que possible les étapes qui pourraient provoquer une dégradation des protéines avant la fixation de l’échantillon. L’étape de pulvérisation est déjà utilisée dans les préparations hépatiques congelées24 et rend la dissociation manuelle plus facile et reproductible (voir figure 2a). Avec l’utilisation d’un mortier spécialement conçu pour les tubes de 1,5 mL (voir Protocoles), la perte d’échantillons pendant le processus de pulvérisation est réduite, ce qui permet de traiter de petites quantités de tissu telles que des échantillons de biopsie du foie. En principe, il est possible d’utiliser l’homogénéisation directe des tissus sans aucune étape de broyage; Cependant, l’homogénéisation tissulaire sans pulvérisation préalable a une moins bonne reproductibilité selon notre expérience et l’apparition de problèmes pour les applications en aval était plus élevée (données non présentées).
La plupart des problèmes rencontrés lors de la préparation de la chromatine à partir de tissus proviennent de la nature de ces échantillons et de l’incapacité de vérifier correctement si les amas cellulaires sont suffisamment petits pour être fixés sans perte de qualité. De plus, la vérification de chaque aliquote à chaque étape prendrait beaucoup de temps, ce qui augmenterait les chances de dégradation des protéines.
La fixation (étape 3.9) est une partie fondamentale et cruciale de la préparation de la chromatine. En raison de la nature du tissu, l’étape de fixation a été retardée jusqu’à ce que le tissu soit homogénéisé. Une telle étape de fixation différée a l’avantage de produire une suspension cellulaire plus homogène. Cependant, nous reconnaissons que dans le cas de cibles particulièrement sensibles à la manipulation, il peut être nécessaire d’effectuer la fixation juste avant l’étape 3.6. Cela aiderait à protéger les protéines extrêmement sensibles ou PTM, bien que cela puisse augmenter la taille des amas cellulaires, ce qui, lorsqu’il est fixé, peut entraîner un cisaillement non homogène. La concentration de la solution FA utilisée dans le protocole est standard, cependant, elle peut être modifiée pour essayer d’améliorer la fixation globale. Le temps de fixation choisi ici reflète également les conditions standard couramment utilisées sur le terrain. En cas de concentration plus élevée de la solution fixatrice, le temps de fixation peut être réduit, tandis qu’en cas de quantité inférieure, il doit être augmenté. L’opérateur doit considérer qu’une modification du temps de fixation peut soit entraîner une fixation excessive de l’échantillon, soit laisser place à la dégradation des protéines. Dans le cas de viser à précipiter de gros complexes (ou une partie de ceux-ci) et des TF, il serait avantageux d’effectuer une fixation en double étape en utilisant une solution DSG suivie d’une solution FA25,26. Dans ce cas, le DSG stabiliserait les interactions protéine-protéine, tandis que le formaldéhyde agit principalement sur les interactions directes ADN-protéine27.
L’opérateur doit tenir compte de la possibilité de mettre en œuvre un kit à base de colonne pour la purification de l’ADN à partir de l’étape 6.7, qui est plus rapide et n’utilise pas de composés toxiques. Cependant, il y aura toujours une certaine quantité d’ADN non lié qui sera perdue. Pour cette raison, nous suggérons d’utiliser l’extraction classique phénol-chloroforme suivie d’une précipitation EtOH. De plus, avant d’exécuter le gel d’agarose (étape 7.2), il pourrait être avantageux de mesurer la concentration d’ADN et de charger la même quantité pour chaque puits afin d’avoir une image plus claire.
Une limite de ce protocole découle du fait que nous avons exploré et utilisé ce protocole uniquement en utilisant des échantillons de foie dérivés de souris chimériques humaines28. En soi, le foie est constitué de tissu épithélial et conjonctif29. En cas de maladie, du tissu fibrotique et du tissu adipeux peuvent être présents30,31, créant des défis supplémentaires lors de la perturbation des tissus. Cependant, nous reconnaissons que notre protocole ne peut pas être utilisé sur les os, les muscles et le tissu adipeux sans optimisation des étapes de dissociation et de sonication. Il est à noter que chaque tissu nécessite une sorte d’optimisation en raison de l’absence d’un protocole adapté à tous comme pour les échantillons de culture cellulaire15. Nous pensons, cependant, qu’avec peu ou pas d’optimisation du tout, ce protocole pourrait être appliqué avec succès à d’autres tissus qui partagent des similitudes avec le foie dans la composition, comme les tissus pulmonaires, intestinaux, estomac, pancréas ou rénaux.
Notre protocole a également été utilisé avec succès pour analyser les TF et les modifications des histones sur l’épisome d’ADN fermé par covalence du VHB (ADNcc)32. Cela ouvre la possibilité d’appliquer une telle approche à d’autres génomes viraux affectant le foie tels que le cytomégalovirushumain 33 (hCMV) et les adénovirushumains 34 (HAdV). Il n’est pas exclu qu’il soit possible d’analyser d’autres virus à ADN qui établissent une infection persistante dans d’autres tissus comme le virus de l’herpès du sarcome de Kaposi35 (KHSV), le virus de l’herpès simplex36 (HSV1/2), le virus d’Epstein-Barr37 (EBV).
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
L’étude a été soutenue par la Fondation allemande pour la recherche (DFG) par une subvention à Maura Dandri (SFB 841 A5) et par l’État de Hambourg avec le programme de recherche (LFF-FV44: EPILOG).
Nous tenons à remercier le Dr Tassilo Volz, Yvonne Ladiges et Annika Volmari pour leur aide technique et pour la lecture critique du manuscrit. Le Dr Thomas Günther et le professeur Adam Grundhoff pour avoir fourni des suggestions très utiles et les ensembles d’amorces pour l’analyse ChIP-qPCR.
matériels
| Name | Company | Catalog Number | Comments |
| 0.22µm sterile syringe filter | Labsolute | 7699822 | |
| 1.5 mL Safeseal tubes | Sarstedt | 7,27,06,400 | |
| 6x orange loading dye | Thermofisher | R0631 | |
| Benchtop refrigerated centrifuge | |||
| Bioruptor NGS | Diagenode | ||
| Blade or Scalpel | |||
| Calcium chloride dihydrate | Carl Roth | HN04 | |
| Chloroform | Sigma Aldrich (Merck) | C2432 | |
| cOmplete Protease Inhibitor Cocktail | Roche | 11697498001 | |
| Deacetylase Inhibitor | Active Motif | 37494 | |
| Dounce tissue grinder set | Sigma Aldrich (Merck) | DWK885300-0001-1EA | |
| EDTA 500 mM solution | PanReac AppliChem | A4892 | |
| EGTA | Sigma Aldrich (Merck) | E4378 | |
| EtBr | Carl Roth | 2218 | Concentration 10mg/mL |
| Ethanol absolute | CHEMSOLUTE | 2273 | |
| Glycerol | Sigma Aldrich (Merck) | G9012 | |
| Glycin | Carl Roth | 0079 | |
| Glycogen | Roche | 10901393001 | Concentration: 20mg/mL |
| Heating block | |||
| HEPES | Sigma Aldrich (Merck) | H4034 | |
| LE Agarose | Biozym | 840000 | |
| Liquid nitrogen cooled mini mortar | Bel-Art | H37260-0100 | |
| MeOH free Formaldehyde 16% | Thermofisher | 28908 | |
| NP-40 | Roche | 11332473001 | |
| PBS 1x | Thermofisher | 10010015 | |
| Pefabloc SC-Protease-Inhibitor | Sigma Aldrich (Merck) | 11429868001 | |
| Phase Lock Gel - Heavy | QuantaBio | 2302830 | |
| Phenol:Chloroform:Isoamyl alcohol 25:24:1 | Sigma Aldrich (Merck) | P3803 | |
| Potassium chloride | Carl Roth | 6781 | |
| Potassium hydroxyde | Merck | 105033 | |
| Proteinase K | Lucigen | MPRK092 | Concentration: 50 µg/µL |
| RNAse A | Lucigen | MRNA092 | Concentration: 5 mg/mL |
| SDS 10% solution | PanReac AppliChem | A3950 | |
| Sodium carbonate anhydrous | Carl Roth | A135 | |
| Sodium chloride | Sigma Aldrich (Merck) | S7653 | |
| Sterile Petri dishes | Sarstedt | 83,39,02,500 | |
| Tris-HCl solution | Sigma Aldrich (Merck) | T2694 | |
| Triton-X100 | Sigma Aldrich (Merck) | X100 |
Références
- Waddington, C. H., Pantelouris, E. M. Transplantation of nuclei in newt's eggs. Nature. 172 (4388), 1050-1051 (1953).
- Silmon de Monerri, N. C., Kim, K. Pathogens hijack the epigenome: A new twist on host-pathogen interactions. American Journal of Pathology. 184 (4), 897-911 (2014).
- Knipe, D. M., et al. Snapshots: chromatin control of viral infection. Virology. 435 (1), 141-156 (2013).
- Tropberger, P., et al. Mapping of histone modifications in episomal HBV cccDNA uncovers an unusual chromatin organization amenable to epigenetic manipulation. Proceedings of the National Academy of Sciences of the United States of America. 112 (42), 5715-5724 (2015).
- Sproul, D., Gilbert, N., Bickmore, W. A. The role of chromatin structure in regulating the expression of clustered genes. Nature Reviews Genetics. 6 (10), 775-781 (2005).
- Fischle, W., Wang, Y., Allis, C. D. Histone and chromatin cross-talk. Current Opinion in Cell Biology. 15 (2), 172-183 (2003).
- Ling, X., Harkness, T. A., Schultz, M. C., Fisher-Adams, G., Grunstein, M. Yeast histone H3 and H4 amino termini are important for nucleosome assembly in vivo and in vitro: redundant and position-independent functions in assembly but not in gene regulation. Genes & Development. 10 (6), 686-699 (1996).
- Zhang, L., Eugeni, E. E., Parthun, M. R., Freitas, M. A. Identification of novel histone post-translational modifications by peptide mass fingerprinting. Chromosoma. 112 (2), 77-86 (2003).
- Wang, H., et al. Role of histone H2A ubiquitination in Polycomb silencing. Nature. 431 (7010), 873-878 (2004).
- Hassa, P. O., Haenni, S. S., Elser, M., Hottiger, M. O. Nuclear ADP-ribosylation reactions in mammalian cells: where are we today and where are we going. Microbiology and Molecular Biology Reviews. 70 (3), 789-829 (2006).
- Dey, B., et al. DNA-protein interactions: methods for detection and analysis. Molecular and Cellular Biochemistry. 365 (1-2), 279-299 (2012).
- Hager, G. L., McNally, J. G., Misteli, T. Transcription dynamics. Molecular Cell. 35 (6), 741-753 (2009).
- Nagy, Z., Tora, L. Distinct GCN5/PCAF-containing complexes function as co-activators and are involved in transcription factor and global histone acetylation. Oncogene. 26 (37), 5341-5357 (2007).
- Solomon, M. J., Larsen, P. L., Varshavsky, A. Mapping protein-DNA interactions in vivo with formaldehyde: evidence that histone H4 is retained on a highly transcribed gene. Cell. 53 (6), 937-947 (1988).
- Gunther, T., Theiss, J. M., Fischer, N., Grundhoff, A. Investigation of viral and host chromatin by ChIP-PCR or ChIP-Seq analysis. Current Protocols in Microbiology. 40, 11-21 (2016).
- Cotney, J. L., Noonan, J. P. Chromatin immunoprecipitation with fixed animal tissues and preparation for high-throughput sequencing. Cold Spring Harbor Protocols. 2015 (4), 419 (2015).
- Haim, Y., Tarnovscki, T., Bashari, D., Rudich, A. A chromatin immunoprecipitation (ChIP) protocol for use in whole human adipose tissue. American Journal of Physiology-Endocrinology and Metabolism. 305 (9), 1172-1177 (2013).
- Castellano-Castillo, D., et al. Chromatin immunoprecipitation improvements for the processing of small frozen pieces of adipose tissue. PLoS One. 13 (2), 0192314 (2018).
- Savic, D., Gertz, J., Jain, P., Cooper, G. M., Myers, R. M. Mapping genome-wide transcription factor binding sites in frozen tissues. Epigenetics Chromatin. 6 (1), 30 (2013).
- Perna, A., Alberi, L. A. TF-ChIP method for tissue-specific gene targets. Frontiers Cell Neuroscience. 13, 95 (2019).
- Allweiss, L., et al. Proliferation of primary human hepatocytes and prevention of hepatitis B virus reinfection efficiently deplete nuclear cccDNA in vivo. Gut. 67 (3), 542-552 (2018).
- Lerdrup, M., Johansen, J. V., Agrawal-Singh, S., Hansen, K. An interactive environment for agile analysis and visualization of ChIP-sequencing data. Nature Structural & Molecular Biology. 23 (4), 349-357 (2016).
- Perna, A., Alberi, L. A. TF-ChIP Method for Tissue-Specific Gene Targets. Frontiers in Cellular Neuroscience. 13, 95 (2019).
- Liang, N., Fan, R., Goni, S., Treuter, E. Preparation of Frozen Liver Tissues for Integrated Omics Analysis. Methods in Molecular Biology. 1951, 167-178 (2019).
- Liu, Z., et al. Proteomic and network analysis of human serum albuminome by integrated use of quick crosslinking and two-step precipitation. Scientific Reports. 7 (1), 9856 (2017).
- Singh, A. A., et al. Optimized ChIP-seq method facilitates transcription factor profiling in human tumors. Life Science Alliance. 2 (1), 201800115 (2019).
- Aoki, T., et al. Bi-functional cross-linking reagents efficiently capture protein-DNA complexes in Drosophila embryos. Fly. 8 (1), 43-51 (2014).
- Allweiss, L., Dandri, M. Experimental in vitro and in vivo models for the study of human hepatitis B virus infection. Journal of Hepatology. 64, 17-31 (2016).
- Krishna, M. Microscopic anatomy of the liver. Clinics in Liver Disease. 2, 4-7 (2013).
- Tannapfel, A., et al. Histopathological diagnosis of non-alcoholic and alcoholic fatty liver disease. Virchows Archiv. 458 (5), 511-523 (2011).
- Schuppan, D., Afdhal, N. H. Liver cirrhosis. Lancet. 371 (9615), 838-851 (2008).
- Allweiss, L., et al. Therapeutic shutdown of HBV transcripts promotes reappearance of the SMC5/6 complex and silencing of the viral genome in vivo. Gut. , 322571 (2021).
- Gerna, G., Kabanova, A., Lilleri, D. Human cytomegalovirus cell tropism and host cell receptors. Vaccines. 7 (3), (2019).
- Echavarria, M. Adenoviruses in immunocompromised hosts. Clinical Microbiology Reviews. 21 (4), 704-715 (2008).
- Frohlich, J., Grundhoff, A. Epigenetic control in Kaposi sarcoma-associated herpesvirus infection and associated disease. Seminars in Immunopathology. 42 (2), 143-157 (2020).
- Nicoll, M. P., Proenca, J. T., Efstathiou, S. The molecular basis of herpes simplex virus latency. FEMS Microbiology Reviews. 36 (3), 684-705 (2012).
- Thorley-Lawson, D. A., Hawkins, J. B., Tracy, S. I., Shapiro, M. The pathogenesis of Epstein-Barr virus persistent infection. Current Opinion in Virology. 3 (3), 227-232 (2013).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon