Method Article
Combinando Hibridização In Situ por Fluorescência Multiplex com Imunohistoquímica Fluorescente em Seções Cerebrais de Camundongos Frescos Congelados ou Fixos
Neste Artigo
Resumo
Este protocolo descreve um método para combinar hibridização in situ fluorescente (FISH) e imunohistoquímica de fluorescência (IHQ) em cortes frescos congelados e fixos de camundongos em cérebros, com o objetivo de obter sinais de FISH multimarcados e IHC de fluorescência. IHQ teve como alvo proteínas citoplasmáticas e ligadas à membrana.
Resumo
Hibridização in situ fluorescente (FISH) é uma técnica molecular que identifica a presença e distribuição espacial de transcritos de RNA específicos dentro das células. A fenotipagem neuroquímica de neurônios funcionalmente identificados geralmente requer marcação simultânea com múltiplos anticorpos (proteína-alvo) usando imunohistoquímica (IHQ) e otimização da hibridização in situ (RNA-alvo), em conjunto. Uma "assinatura neuroquímica" para caracterizar neurônios particulares pode ser alcançada, no entanto, fatores complicadores incluem a necessidade de verificar alvos de FISH e IHQ antes de combinar os métodos, e o número limitado de RNAs e proteínas que podem ser visados simultaneamente dentro da mesma seção de tecido.
Aqui descrevemos um protocolo, usando preparações cerebrais de camundongos frescos congelados e fixos, que detecta múltiplos mRNAs e proteínas na mesma seção cerebral usando RNAscope FISH seguido de imunomarcação por fluorescência, respectivamente. Usamos o método combinado para descrever o padrão de expressão de mRNAs de baixa abundância (por exemplo, receptor de galanina 1) e mRNAs de alta abundância (por exemplo, transportador de glicina 2), em núcleos do tronco cerebral imunohistoquimicamente identificados.
As principais considerações para a rotulagem de proteínas a jusante do ensaio FISH vão além da preparação de tecidos e da otimização da rotulagem de sondas de FISH. Por exemplo, descobrimos que a especificidade de ligação e rotulagem de anticorpos pode ser afetada negativamente pela etapa de protease dentro do ensaio de sonda FISH. As proteases catalisam a clivagem hidrolítica de ligações peptídicas, facilitando a entrada da sonda FISH nas células, mas também podem digerir a proteína alvo do ensaio IHQ subsequente, produzindo ligação fora do alvo. A localização subcelular da proteína-alvo é outro fator que contribui para o sucesso da IHQ após o ensaio com sonda FISH. Observamos que a especificidade da IHQ deve ser retida quando a proteína-alvo está ligada à membrana, enquanto a IHQ direcionada à proteína citoplasmática requer solução de problemas extensa. Finalmente, achamos que o manuseio do tecido congelado fixo montado em lâmina é mais desafiador do que o tecido fresco congelado, no entanto, a qualidade da IHQ foi globalmente melhor com o tecido congelado fixo, quando combinado com o RNAscope.
Introdução
Proteínas e RNAm que definem neuroquimicamente subpopulações de neurônios são comumente identificados com uma combinação de imunohistoquímica (IHQ) e/ou hibridização in situ (ISH), respectivamente. A combinação de ISH com técnicas de IHQ facilita a caracterização de padrões de colocalização exclusivos de neurônios funcionais (codificação neuroquímica), maximizando a capacidade de marcação multiplex.
Métodos de ISH fluorescente (FISH), incluindo RNAscope, têm maior sensibilidade e especificidade em comparação com métodos anteriores de detecção de RNA, como ISH radioativa e ISH cromogênica não radioativa. O FISH permite a visualização de transcritos de RNAm único como pontos corados puntiformes1. Além disso, o ensaio RNAscope permite que um número maior de alvos de RNA seja marcado de cada vez, usando diferentes tags de fluoróforo. Apesar dessas vantagens, limitações técnicas podem afetar o número de fluoróforos/cromógenos que podem ser utilizados em um único experimento. Estes incluem a disponibilidade de conjuntos de filtros para microscópios; tais considerações são agravadas quando a identificação neuroquímica utiliza FISH e IHQ combinadas, em comparação com o uso de cada técnica isoladamente, uma vez que etapas inerentes ótimas para um método podem ser prejudiciais para o outro.
A aplicação prévia de FISH combinada com IHQ demonstrou a expressão de alvos celulares específicos em linfomas de células B humanas2, embriões de pintinhos3, embriões de peixe-zebra4, retina de camundongo 5 e células da orelha internade camundongos6. Nesses estudos, o preparo tecidual foi fixado em formalina e incluído em parafina (FFPE)2,3,5 ou montaria inteira a fresco 4,6. Outros estudos aplicaram o RNAscópio cromogênico em preparações cerebrais fixas de camundongos e ratos 7,8,9. Em particular, Baleriola et al.8 descreveram duas preparações teciduais diferentes para a combinação de IHQ-HIS; seções fixas do cérebro de camundongos e seções do cérebro humano FFPE. Em uma publicação recente, combinamos FISH e IHQ fluorescente em cortes frescos congelados, para visualizar simultaneamente mRNA de baixa abundância (receptor de galanina 1, GalR1), mRNA de alta abundância (transportador de glicina 2, GlyT2) e proteína10 do transportador vesicular de acetilcolina (vAChT) na formação reticular do tronco encefálico.
O núcleo do trato solitário (NTS) é uma importante região cerebral envolvida na função autonômica. Localizada no rombencéfalo, essa população heterogênea de neurônios recebe e integra um vasto número de sinais autonômicos, incluindo aqueles que regulam a respiração. O NTS abriga várias populações neuronais, que podem ser fenotipicamente caracterizadas pelo padrão de expressão de alvos de RNAm, incluindo GalR1 e GlyT2 e marcadores proteicos para a enzima tirosina hidroxilase (TH) e o fator de transcrição Paired-like homeobox 2b (Phox2b).
O proprietário do RNAscope recomenda preparações de tecidos frescos congelados, mas o tecido preparado por fixação transcárdica de perfusão animal inteiro, juntamente com crioproteção a longo prazo (armazenamento a -20 °C) de cortes fixos de tecido congelado, é comum em muitos laboratórios. Assim, buscou-se estabelecer protocolos para FISH em combinação com IHQ utilizando preparações de tecidos frescos congelados e congelados fixos. Aqui, nós fornecemos para seções de cérebro congelado fresco e congelado fixo: (1) um protocolo para FISH combinado e IHC fluorescente (2) uma descrição da qualidade do mRNA e rotulagem de proteínas produzidas, ao utilizar cada preparação (3) uma descrição da expressão de GalR1 e GlyT2 no NTS.
Nosso estudo revelou que, quando combinado com a metodologia RNAscope, o sucesso da IHQ variou em preparações congeladas frescas e fixas, dependendo da localização das proteínas-alvo dentro da célula. Em nossas mãos, a rotulagem de proteínas ligadas à membrana sempre foi bem-sucedida. Em contraste, a IHQ para proteína citoplasmática exigiu solução de problemas mesmo nos casos em que a proteína citoplasmática foi superexpressa em um animal transgênico (Phox2b-GFP)11. Finalmente, enquanto GalR1 é expressa em neurônios não-catecolaminérgicos no NTS, a expressão de GlyT2 está ausente no NTS.
Protocolo
Um resumo das etapas de pré-processamento tecidual pode ser encontrado na Figura 1. Todos os procedimentos foram realizados de acordo com o Comitê de Ética e Cuidados com Animais da Universidade de Nova Gales do Sul, de acordo com as diretrizes para o uso e cuidados de animais para fins científicos (Australian National Health and Medical Research Council).
1. Preparação da amostra de tecido cerebral fresco congelado
- Perfusão Transcárdica
- Preparar tampão fosfato (PB) 0,1 M heparinizado (2500 U/L), pH 7,5. Faça pasta de etanol de gelo seco misturando gelo seco com etanol. Este terá uma temperatura de aproximadamente -72 °C e será usado para congelamento imediato do tecido colhido.
- Eutanásia de camundongos adultos C57BL/6 e Phox2b-GFP11 (Mouse Genome Informatics database ID MGI:5776545) anestesiando com pentobarbital sódico (70 mg/kg, i.p.), usando um calibre de agulha de 27,5 polegadas.
CUIDADO: O pentobarbital é um barbitúrico. É agudamente tóxico em altas doses e pode causar a morte por parada respiratória. Consulte as diretrizes locais de saúde, legais e de segurança de materiais antes de usar. - Expor o coração e canular o ventrículo esquerdo com uma agulha de desenho (calibre de 23 polegadas). Realizar perfusão transcárdica com PB 0,1 M heparinizado até que o sangue clareie (2-3 minutos) a uma taxa de fluxo de 11-13 mL/min. Determinar a depuração sanguínea monitorando a coloração do fígado e o efusato do átrio direito12.
- Isole o cérebro da cavidade craniana, incorpore-o imediatamente no Composto de Temperatura de Corte Ótima (OCT) em um criomold ou folha de alumínio e coloque-o no banho de etanol de gelo seco. Armazenar o tecido embutido congelado em um recipiente hermético a - 80 °C por até 3 meses.
- Secção de tecido fresco congelado
- Ajuste a temperatura do criostato para -20 °C. Deixe o tecido embutido em OCT e um mandril de criostato no criostato por ~30 minutos para permitir o equilíbrio à nova temperatura.
OBS: Manter o tecido congelado o tempo todo; transportar o tecido do congelador de -80 °C para o criostato em gelo seco. - Fixe o tecido no mandril de criostato pré-resfriado usando o composto OCT. Nesse protocolo, blocos de tecido foram montados sobre o mandril no plano coronal.
NOTA: Aparar o excesso de OCT do tecido, usando uma lâmina de barbear, para minimizar a quantidade de OCT que está sendo cortada pelo criostato e posteriormente transferida para a lâmina de vidro. - Corte seções coronais de 14 μm de espessura e monte-as em lâminas de microscopia de vidro carregadas.
- Aqueça as lâminas à temperatura ambiente antes de montar as seções. Depois que a seção tiver sido montada, mantenha as lâminas em uma caixa de slides no criostato.
- Se mais de uma seção precisar ser montada em uma lâmina, aqueça a área para a segunda seção colocando um dedo no lado oposto da lâmina por 5-10 segundos para ajudar a aderência da seção à lâmina. Uma seção de tecido frio não se prenderá a uma lâmina fria. As seções devem aderir às lâminas planas; A dobra fará com que eles caiam das lâminas durante as etapas de lavagem.
- Se forem notadas rachaduras nas seções, aumente a temperatura do criostato em 1-5 °C para evitar isso. É particularmente importante colocar cortes de tecido próximos uns dos outros na mesma lâmina. Isso evitará o desperdício de sondas e reagentes FISH durante o ensaio.
- Armazene seções de tecido montadas em lâminas de vidro em um recipiente hermético a -80 °C por até 6 meses.
NOTA: Mantenha as seções congeladas o tempo todo e evite ciclos de congelamento de descongelamento, para evitar a degradação do RNA. Transporte a caixa deslizante do interior do criostato para o congelador de -80 °C em gelo seco.
- Ajuste a temperatura do criostato para -20 °C. Deixe o tecido embutido em OCT e um mandril de criostato no criostato por ~30 minutos para permitir o equilíbrio à nova temperatura.
- Fixação de tecido fresco congelado
- No dia da realização do ensaio com sonda de FISH, preparar paraformaldeído (PFA) a 4% em PB 0,1 M, pH 7,5 (solução de PFA a 4%). Filtrar passando por papel de filtro (Grau 1: 11 μm, Tabela de Materiais) em um funil de Buchner ou filtro de cadinho.
CUIDADO: O PFA é nocivo e tóxico por contato com a pele ou inalação. Todos os procedimentos com solução de PFA devem ser realizados em um gabinete de exaustor. Os resíduos de soluções de PFA devem ser descartados cuidadosamente, seguindo os protocolos de segurança institucionais. - Arrefecer a solução de PFA a 4% a 4 °C. Transporte o tecido montado na lâmina do congelador de -80 °C em gelo seco e mergulhe-o imediatamente no fixador pré-resfriado por 15 minutos.
NOTA: É importante que esta etapa de fixação não exceda 15 minutos, pois a fixação excessiva resultará em rotulagem de fundo não específica.
- No dia da realização do ensaio com sonda de FISH, preparar paraformaldeído (PFA) a 4% em PB 0,1 M, pH 7,5 (solução de PFA a 4%). Filtrar passando por papel de filtro (Grau 1: 11 μm, Tabela de Materiais) em um funil de Buchner ou filtro de cadinho.
- Desidratação de tecido fresco congelado
- Desidratar cortes de tecido submergindo as lâminas em concentrações graduadas de etanol. Em um frasco de Coplin, primeiro submergir em 50%, depois 70% e finalmente etanol absoluto, por 5 minutos cada em temperatura ambiente. Repetir a incubação absoluta final do etanol uma segunda vez.
- Lâminas secas ao ar e, delinear o grupo de seções usando uma caneta de barreira hidrofóbica, garantindo que a área interna seja mantida ao mínimo.
NOTA: Certifique-se de que a lâmina de vidro está completamente seca antes de desenhar a barreira hidrofóbica. A barreira hidrofóbica deve envolver os cortes de tecido completamente sem lacunas e deve ser seca antes do processamento posterior.
2. Preparação da amostra de tecido cerebral congelado fixo
- Fixação transcárdica da perfusão
- Eutanásia de camundongos anestesiando com pentobarbital sódico (70 mg/kg, i.p.) seguido de perfusão transcárdica, primeiro com solução de PFA 0,1 M PB e depois 4%. Fixar com 10 minutos de perfusão a 11-13 mL/min.
- Isolar o cérebro da cavidade craniana após a fixação da perfusão e submergir durante a noite em solução de PFA a 4%, a 4 °C.
- Secção tecidual de tecido fixado
- Enxaguar o cérebro em solução salina tamponada com fosfato 0,1 M (PBS) estéril antes de remover as camadas meníngeas, com o auxílio de um microscópio dissecante, usando pinça fina.
- Corte o cérebro precisamente em blocos (separe o tronco cerebral do prosencéfalo antes da secção do vibratom) usando uma matriz cerebral (Tabela de Materiais). Especificamente, cortar o tronco cerebral caudalmente na decussação piramidal e dissecar o cerebelo. Da mesma forma, corte o prosencéfalo imediatamente rostral ao quiasma óptico.
- Fixar o tecido em um mandril de micrótomo vibratório usando cianoacrilato e incorporar em solução de ágar 2%.
- Cortar cortes de tecido de 30 μm de espessura usando micrótomo vibratório e armazenar cortes em solução crioprotetora (sacarose livre de RNase 30%, etilenoglicol 30%, polivinilpirrolidona 1% (PVP-40), em PB 0,1 M, pH 7,4). Os cortes de tecido podem ser armazenados em crioprotetor a -20 °C por até 6 meses.
- Preparação de secções fixas antes do FISH
- No dia do FISH, lave as seções flutuantes livres três vezes, por 10 minutos por lavagem, para remover a solução crioprotetora. Para lavar, colocar as seções em PBS 0,1 M em uma placa de cultura de células de 12 poços e agitar em um agitador de plataforma giratória (90 - 100 rpm).
- Após as lavagens, use um pincel para montar seções em lâminas de microscopia de vidro e seque ao ar por pelo menos 2 horas.
NOTA: As seções devem aderir planas nas lâminas, pois quaisquer dobras pronunciadas farão com que elas se desprendam durante as lavagens. - Usando uma caneta de barreira hidrofóbica, desenhe uma barreira ao redor das seções para restringir os reagentes FISH às seções. Mais uma vez, é importante minimizar a área interna do contorno desenhado com a caneta barreira.
POSSÍVEL PONTO DE RUPTURA: As seções poderiam ser armazenadas à temperatura ambiente, durante a noite, para continuar o ensaio no dia seguinte.
3. Ensaio FISH
NOTA: O resto do protocolo aplica-se tanto ao tecido fresco congelado como ao tecido congelado fixo.
- Preparar os reagentes e instrumentos para as etapas de hibridização e amplificação.
- Coloque uma incubadora de bancada e banho-maria a 40 °C.
- Prepare uma câmara umidificada e protegida contra a luz para incubar lâminas. A umidificação evita a secagem dos tecidos - as lâminas estão localizadas de forma segura acima de um reservatório úmido. Idealmente, a câmara é feita de poliestireno para serviço pesado, é à prova de luz e hermética para manter uma atmosfera saturada de vapor de água. O fechamento da câmara depende do mínimo de atrito para evitar o movimento. Utilizou-se uma caixa de slides forrada com lenços umedecidos de laboratório (Tabela de Materiais) na parte inferior. Coloque a caixa deslizante dentro da incubadora para pré-aquecê-la a 40 °C.
- Aqueça o tampão de lavagem de 50x (Tabela de Materiais) e as sondas a 40 °C por 10 minutos, usando o banho-maria, e depois resfrie até a temperatura ambiente.
- Prepare 1 L de 1x Wash Buffer a partir da concentração de estoque de 50x.
- Preparar mistura de sonda (Tabela de Materiais): a sonda C1 está pronta para uso na concentração de estoque, enquanto as sondas C2 e C3 são enviadas como concentração de 50x e requerem diluição com o diluente fornecido no kit.
NOTA: As misturas de sonda podem ser armazenadas a 4 °C por até 6 meses.
- Tratamento de protease
- Incubar os cortes com Protease III (Tabela de Materiais) à temperatura ambiente por 30 minutos.
NOTA: Certifique-se de que a Protease III e os reagentes de incubação em processos a jusante (mistura de sonda, soluções de amplificação, tampão de bloqueio e soros de anticorpos) cubram totalmente as secções. Uma ponta de pipeta pode ser usada para espalhar o reagente na seção para cobrir toda a área dentro da barreira hidrofóbica. - Lave as lâminas duas vezes com PBS 0,1 M, por 2 min de cada vez, em uma grande placa de Petri quadrada de plástico. Utilizou-se uma placa de bioensaio quadrada de 245 mm x 245 mm (Tabela de Materiais). Segure de um lado do prato e incline suavemente 3-5 vezes. Após as lavagens, passe o excesso de 0,1 M PBS da lâmina e adicione imediatamente o próximo reagente. Não deixe secar as secções de tecido.
NOTA: Durante cada lavagem, as lâminas são imersas em solução à temperatura ambiente. Este é o fluxo de trabalho para todas as etapas de lavagem subsequentes. As seções fixas de 30 μm de espessura deslocam das lâminas mais facilmente do que as seções de 14 μm de espessura, seja suave durante as lavagens.
- Incubar os cortes com Protease III (Tabela de Materiais) à temperatura ambiente por 30 minutos.
- Hibridização e amplificação
- Depois de lavar a solução de protease, coloque as lâminas na câmara umidificada e pré-aquecida. Incubar seções com mistura de sonda (Tabela de Materiais) por 2 horas a 40 °C dentro de uma incubadora de bancada.
NOTA: Certifique-se de que há pelo menos 2 seções reservadas para sondas de controle positivo e negativo para avaliar a qualidade do RNA da amostra e a permeabilização ideal. Sondas de controle positivo têm como alvo genes de manutenção doméstica; aqui, estes foram um coquetel de RNAs visando ubiquitina C (UBC; alta abundância), peptidilpropilisomerase B (PPIB; abundância moderada) e RNA polimerase 2a (POLR2A; baixa abundância). As sondas de controle negativo têm como alvo o gene bacteriano 4-hidroxi-tetraidrodipicolinato redutase (DapB), que normalmente está ausente em amostras de cérebro de camundongos. O sinal DapB positivo indica sinal inespecífico e/ou contaminação bacteriana da amostra. - Após a hibridização com a mistura de sonda, as etapas de amplificação do sinal consistem na incubação com Amp 1-FL (30 minutos), depois com Amp 2-FL (15 minutos), seguido por Amp 3-FL (30 minutos) e, finalmente, Amp 4-FL (15 minutos) - cada um a 40 °C. Usando os frascos conta-gotas fornecidos, cubra os cortes de tecido com solução de amplificação. Prossiga para o ensaio IHC após a última etapa de amplificação.
- Enxaguar as lâminas com Wash Buffer duas vezes por 2 minutos entre a hibridização da sonda e cada etapa de amplificação.
- Depois de lavar a solução de protease, coloque as lâminas na câmara umidificada e pré-aquecida. Incubar seções com mistura de sonda (Tabela de Materiais) por 2 horas a 40 °C dentro de uma incubadora de bancada.
4. Ensaio IHC
- Etapa de bloqueio IHC
- Para evitar a ligação inespecífica de anticorpos, incubar os cortes por 1 h à temperatura ambiente com solução de bloqueio contendo 10% de soro normal de cavalo, 0,3% de Tween20 em 1x TBSm (50 mM Tris-Cl, pH 7,5, 150 mM NaCl, 0,05% de mertiolato) após o ensaio de FISH. Preparar anticorpos primários em tampão de diluição contendo 1x TBSm, 5% de soro normal de cavalo e 0,1% de Tween20. Os principais fornecedores de anticorpos estão listados na Tabela de Materiais.
- Imuno-histoquímica
- Remova o excesso de tampão de bloqueio agitando a lâmina e incube as seções com anticorpos primários durante a noite a 4 °C.
- Lavar as lâminas 3 vezes (5 minutos cada) com 1x TBSm e incubar com anticorpo secundário em diluente contendo 1x TBSm, 1% de soro normal de cavalo e 0,1% de Tween20 por 2 horas à temperatura ambiente. Os anticorpos secundários utilizados neste protocolo estão listados na Tabela de Materiais.
- Lave as lâminas 3 vezes com 1x TBSm (5 minutos cada) antes de cobrir com meio de montagem com ou sem DAPI (Tabela de Materiais).
5. Exames por imagem
- Examine a imunomarcação sob um microscópio de epifluorescência equipado com uma câmera (consulte a Tabela de Material para obter detalhes). Adquira imagens representativas com ampliação de 20x e salve como arquivos TIFF.
- Exporte imagens representativas para um software de processamento de imagens (Tabela de Materiais) para ajuste de brilho/contraste para aumentar a clareza e refletir a renderização verdadeira.
6. OPCIONAL: Análise quantitativa das transcrições alvo
Observação : este é um artigo de métodos e resultados quantitativos não são fornecidos. O método de quantificação aqui apresentado é proveniente de Dereli et al.10º.
- Adquirir imagens das regiões de interesse, conforme explicado no item 5.1, e aplicar as mesmas configurações do microscópio e da câmera (como tempo de exposição e intensidade da luz) a todas as imagens do mesmo fluoróforo.
- Plotar os perfis neuronais utilizando um software de análise de imagens (Tabela de Materiais).
- Alinhar as seções com referência ao nível de Bregma de acordo com um atlas cerebral estereotáxico13.
- Aplique o mesmo brilho e contraste a todas as imagens do mesmo fluoróforo. Considere apenas os neurônios com núcleos corados com DAPI.
- Contar manualmente o número de células de mRNA, expressão de proteínas, mRNA/mRNA, proteína/proteína e mRNA/proteína coexpressando células dentro da região de interesse.
- Para diminuir o viés nos resultados experimentais, peça à pessoa que quantifica os resultados experimentais cega para os grupos experimentais.
- Aplique a correção14 do Abercrombie às contagens totais de células usando a seguinte equação de Abercombie:
Contagem de células corrigida = contagem manual de células x espessura da seção / (espessura da seção + tamanho nuclear)
Por exemplo, para seções de 14 μm de espessura, a largura nuclear média é calculada em 7,7 ± 0,3 μm e a espessura média da seção é de 14 ± 1 μm com base em 30 células e 10 cortes, respectivamente, em 5 animais10. De acordo com a equação de Abercrombie, a contagem de células corrigida seria a contagem manual de células x 14/(14+7,7).

Figura 1: Fluxo de trabalho paralelo das etapas de pré-processamento do tecido para o tecido fresco congelado e paraformaldeído fixo. As etapas de processamento para o tecido fresco congelado são exibidas nas caixas delineadas em vermelho, enquanto as etapas para o tecido fixo com paraformaldeído (PFA) são exibidas nas caixas delineadas em azul. Clique aqui para ver uma versão maior desta figura.

Figura 2: Resumo da combinação de sonda de FISH e procedimento de imunohistoquímica. Após o pré-processamento do tecido, o tecido montado na lâmina é circundado com uma caneta de barreira hidrofóbica, como visto no primeiro quadro, e incubado em uma solução de protease à temperatura ambiente. Após as lavagens, o tecido é transferido para uma incubadora de bancada para hibridização por 2 horas antes das etapas de amplificação sequencial. O sistema de hibridização in situ utiliza um design proprietário de 'sonda Z', pré-amplificadores e amplificadores como visto nos quadros 3-66. Uma vez que o tecido tenha sido submetido ao processamento da sonda de FISH, ele é lavado antes do bloqueio com soro normal de cavalo. A incubação primária do anticorpo é realizada durante a noite a 4 °C para maximizar a ligação anticorpo-antígeno. A incubação do anticorpo secundário (2 horas) foi realizada à temperatura ambiente. Clique aqui para ver uma versão maior desta figura.
Resultados
Aqui, nós delineamos um método para combinar FISH multiplex com IHQ fluorescente para localizar a expressão de RNAm para GalR1 e GlyT2 usando tecidos frescos congelados e fixados em paraformaldeído, respectivamente, no NTS de camundongos. Um pipeline dos procedimentos de processamento de tecidos, FISH e IHQ descritos nos métodos é mostrado na Figura 1 e na Figura 2. A Tabela 1 fornece um resumo das combinações de sonda FISH e anticorpos usadas em cada figura.
As sondas de controle são rotineiramente ensaiadas simultaneamente com a sonda de destino, para garantir a integridade do fluxo de trabalho e confirmar a qualidade da amostra. A ausência de marcação DapB confirma a boa qualidade e integridade dos tecidos e a ausência de contaminação bacteriana (Figura 3A). A marcação de sondas de controle positivo visando ubiquitina C (UBC, alta abundância), peptidilpropilisomerase B (PPIB, abundância moderada) e RNA polimerase 2a (POLR2A, baixa abundância) confirma a integridade do RNA e o sinal observado entre os ensaios pode ser usado para calibrar a variabilidade interensaio (Figura 3B). Para validar a expressão da sonda FISH, usamos tecidos controles previamente descritos para expressar o transcrito do RNAm. Por exemplo, a expressão do RNAm de GalR1 foi confirmada como positiva no tálamo, como descrito anteriormente10,15. A distribuição do mRNA Phox2b foi adicionalmente verificada pela comarcação com o anticorpo Phox2b; confirmamos que a marcação de FISH estava presente apenas em neurônios que também foram corados positivamente pelo anticorpo Phox2b (Figura 5).
Para distinguir os neurônios GalR1+ no NTS dos núcleos vizinhos, usamos marcadores neuroquímicos adicionais. A imunorreatividade de TH, Phox2b ou Phox2b-GFP (Figura 4-6) e a FISH Phox2b (Figura 5 e Figura 6) diferenciaram o NTS de outros núcleos no tronco encefálico dorsal, uma vez que neurônios NTS já foram relatados para expressar Phox2b e TH 16,17. Como o NTS está aninhado por núcleos colinérgicos - situa-se dorsal ao núcleo do hipoglosso e ao núcleo motor dorsal do vago (DMNX) e ventral ao núcleo vestibular - co-marcamos com o marcador colinérgico vAChT18 (Figura 4). Portanto, a expressão de GalR1 dentro do NTS foi avaliada em relação a HT e Phox2b, enquanto a marcação vAChT auxiliou a orientação espacial em relação às coordenadas rostrocaudal, dorsoventral e médio-lateral. Descobrimos que todos os neurônios imunorreativos de HT e positivos para RNAm de GalR1 no NTS eram imunorreativos a Phox2b-GFP, mas nem todos os neurônios imunorreativos de Phox2b-GFP no NTS eram imunorreativos de HT ou positivos para mRNA de GalR1 (Figura 4). Além disso, demonstramos que o RNAm para o receptor de baixa abundância GalR1 estava ausente nos neurônios imunorreativos de HT e vAChT.
Em preparações frescas congeladas, quando combinadas com o ensaio de sonda FISH, o sucesso da IHQ foi dependente da localização subcelular da proteína-alvo. Por exemplo, a vAChT (uma proteína ligada à membrana da vesícula sináptica) foi claramente imunomarcada, enquanto TH e GFP (proteínas citoplasmáticas) foram imunomarcadas indefinidamente e apenas fracamente observadas (Figura 4). Descrevemos esta rotulagem indefinida como "floculante" porque as células não tinham um contorno claro e se revelaram difíceis de distinguir do fundo. No mesmo corte de tecido fresco congelado, a marcação com sonda GalR1 FISH do mRNA citoplasmático de GalR1 foi puntiforme e claramente observada (Figura 4).
Além disso, como os anticorpos TH e vAChT estão elevados no mesmo hospedeiro, ambas as proteínas foram marcadas usando o mesmo anticorpo secundário e, portanto, o mesmo fluoróforo de cor (luz de excitação: 594). Eles são facilmente distinguidos por duas razões: eles nunca co-marcam nos mesmos neurônios, e a localização subcelular é diferente para essas proteínas; vAChT em vesículas exibindo aparência puntiforme e HT no citoplasma e processos neuronais.
Para apoiar nossa hipótese de que a qualidade da IHQ (em preparações frescas congeladas) é dependente da localização subcelular da proteína, comparamos a marcação para o mRNA Phox2b (localizado no citoplasma), GFP (superexpresso no citoplasma) e proteína Phox2b (encontrada principalmente no núcleo) em neurônios. Como esperado, nossos resultados mostram sobreposição de marcação de anticorpos Phox2b mRNA, GFP e Phox2b em neurônios individuais do NTS (Figura 5). Células com marcação de RNAm citoplasmático corresponderam com células exibindo marcação nuclear da proteína Phox2b, fornecendo validação do método combinado FISH-IHC. Embora a Phox2b-GFP citoplasmática tivesse uma aparência floculenta, o sinal da proteína Phox2b nuclear foi claro e específico. Em conclusão, quando combinadas com FISH em preparações frescas congeladas, as proteínas ligadas à membrana, incluindo vAChT e Phox2B, exibem marcação imunológica de maior qualidade do que as proteínas citoplasmáticas.
Em contraste, a IHQ foi confiável, independentemente da localização subcelular, quando realizada em cortes fixos congelados em combinação com FISH. FISH multiplex para mRNA de GlyT2 e mRNA de Phox2b foi bem sucedido, como mostrado na Figura 6. Os neurônios positivos para RNAm de GlyT2 estavam localizados ventralmente ao NTS e não dentro do NTS. Os neurônios GlyT2+ e Phox2b+ não colocalizaram. Uma subpopulação de neurônios Phox2b+ NTS era imunorreativa ao HT e nenhum continha mRNA de GlyT2. Os neurônios imunorreativos do HT são aparentes no mesmo corte tecidual, exibindo soma e processos neuronais marcados positivamente (Figura 6). Isso contrasta com a aparência "floculenta" dos neurônios imunorreativos de HT em cortes de tecido fresco congelado. Assim, a preparação congelada fixa descrita aqui é um método alternativo de preparação de tecidos que permite o direcionamento confiável de proteínas citoplasmáticas imunohistoquimicamente, em combinação com o RNAscope.

Figura 3: Imagens microscópicas representativas de cortes coronais do prosencéfalo de camundongos ao nível do septo lateral (Bregma 1.1 a -0.1) mostrando marcação de sondas de controle positivo e negativo . (A) A falta de sinal após ISH com 4-hidroxi-tetraidrodipicolinato redutase bacteriana (DapB) confirma a ausência de sinais de fundo. (B) A marcação com sondas de controle positivo visando ubiquitina C (UBC), peptidilpropilisomerase B (PPIB) e RNA polimerase 2a (POLR2A) ilustra o sinal a ser esperado de alvos de alta, moderada e baixa abundância, respectivamente. As barras de escala são de 50 μm. Todas as imagens foram adquiridas com objetiva de 20x. Clique aqui para ver uma versão maior desta figura.
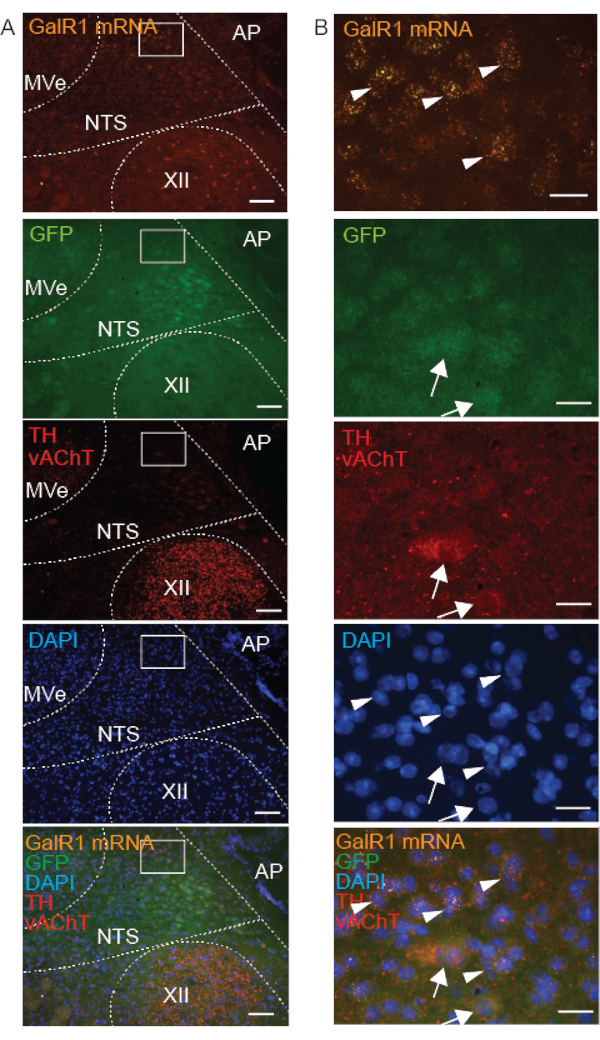
Figura 4: Imagens microscópicas representativas de uma seção coronal fresca congelada do tronco cerebral de um camundongo Phox2b-GFP mostrando marcação combinada de RNAm de GalR1 (FISH) e 3 proteínas (IHQ) no núcleo da região do trato solitário (NTS). As inserções em A estão aumentadas em B. O mRNA GalR1 é indicado pela marcação puntiforme da sonda FISH (pontas de setas). Anticorpos direcionados às proteínas citoplasmáticas GFP e tirosina hidroxilase (TH) exibiram marcação "floculenta" (setas). A imunorreatividade vesicular do transportador vesicular de acetilcolina (vAChT) é demonstrada (marcação puntiforme vermelha) no núcleo do hipoglosso (XII). As barras de escala são 100 μm em A e 25 μm em B. Todas as imagens foram adquiridas com objetiva de 20x. Outras abreviações: área postrema (AP), núcleo vestibular medial (MVe). Clique aqui para ver uma versão maior desta figura.
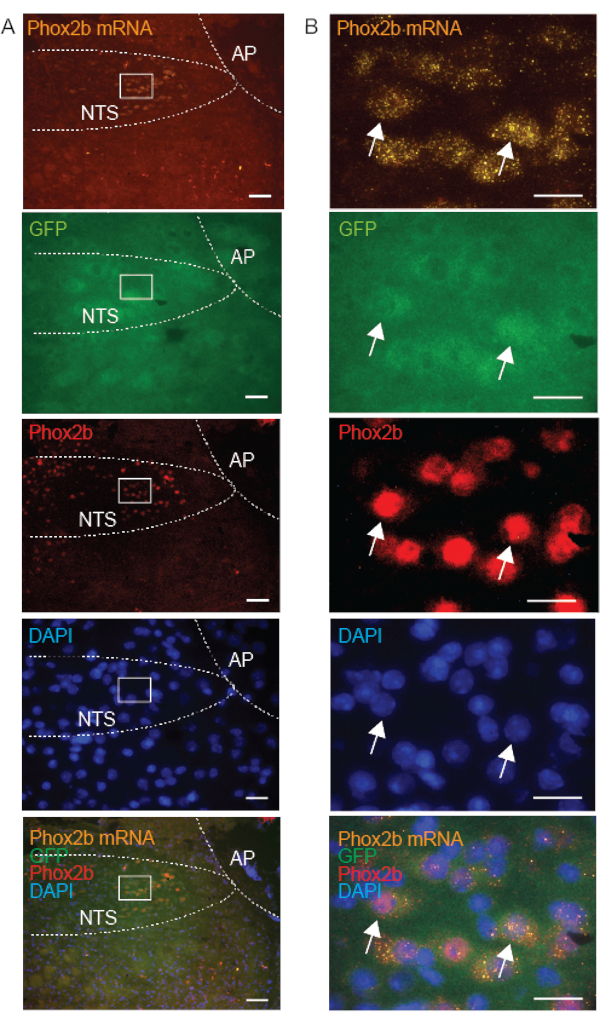
Figura 5: Imagens microscópicas representativas de uma seção coronal congelada fresca do tronco cerebral de um camundongo Phox2b-GFP, ilustrando o alvo de Phox2b no núcleo do trato solitário (NTS) com três abordagens diferentes: Phox2b mRNA (FISH), GFP (IHC) e Phox2b protein (IHQ). A proteína Phox2b está localizada no núcleo. As inserções em A estão aumentadas em B. As setas indicam neurônios que são triplamente marcados com sonda Phox2b (laranja-550), anticorpo GFP (verde-488) e anticorpo Phox2b (vermelho-647). As barras de escala são 100 μm em A e 25 μm em B. Todas as imagens são adquiridas com objetiva de 20x. Outras abreviaturas: area postrema (AP). Clique aqui para ver uma versão maior desta figura.

Figura 6: Imagens representativas de cortes fixos do tronco cerebral coronal congelado demonstrando FISH bem-sucedida combinada com imunomarcação confiável de proteínas citoplasmáticas (tirosina hidroxilase [TH]). FISH duplo mostrando marcação de mRNA do transportador de glicina 2 (GlyT2-vermelho-647, cabeças de setas preenchidas) e Phox2b (amarelo-550, setas) no núcleo da região do trato solitário (NTS). A FISH foi combinada com IHQ para proteína TH (azul-346, pontas de seta vazias). As inserções em A são ampliadas em B. As barras de escala são de 25 μm. Todas as imagens foram adquiridas com objetiva de 20x. Clique aqui para ver uma versão maior desta figura.
| Sonda primária de anticorpos ou RNAscope | Anticorpo secundário ou Módulo de exibição Amp 4-FL-Alt | Excitação (nm) | Preparo de tecidos | ||
| Figura 3 | sondar | POLR2A (C1) | Amp 4-FL-Alt B Módulo de exibição | 647 | fresco congelado |
| sondar | PPIB (C2) | Amp 4-FL-Alt B Módulo de exibição | 488 | ||
| sondar | UBC (C3) | Amp 4-FL-Alt B Módulo de exibição | 550 | ||
| sondar | DapB (C1, C2, C3) | Amp 4-FL-Alt B Módulo de exibição | 647, 488, 550 | ||
| DAPI | 346 | ||||
| Figura 4 | anticorpo | coelho-anti-GFP | burro-anti-coelho | 488 | fresco congelado |
| anticorpo | ovelha-anti-TH | burro-anti-ovelhas | 647 | ||
| anticorpo | cabra-anti-vAChT | burro-anti-cabra | 647 | ||
| sondar | GalR1 (C1) | Amp 4-FL-Alt B Módulo de exibição | 550 | ||
| DAPI | 346 | ||||
| Figura 5 | anticorpo | coelho-anti-GFP | burro-anti-coelho | 488 | fresco congelado |
| anticorpo | rato-anti-Phox2b | burro-anti-rato | 647 | ||
| sondar | Phox2b (C2) | Amp 4-FL-Alt A Módulo de exibição | 550 | ||
| DAPI | 346 | ||||
| Figura 6 | anticorpo | mouse-anti-TH | burro-anti-rato | 346 | fixo |
| sondar | GlyT2 | Amp 4-FL-Alt A Módulo de exibição | 647 | ||
| sondar | Phox2b | Amp 4-FL-Alt A Módulo de exibição | 550 |
Tabela 1: Sonda de FISH, anticorpo e combinações correspondentes de fluróforo utilizadas nas Figuras 3 a 6.
Discussão
Nas neurociências, FISH e IHQ são rotineiramente usadas para investigar a organização espacial e o significado funcional de RNAm ou proteínas dentro de subpopulações neuronais. O protocolo descrito neste estudo aumenta a capacidade de detecção simultânea de mRNAs e proteínas em cortes cerebrais. Nosso ensaio combinado multiplex FISH-IHC permitiu a identificação fenotípica de subpopulações neuronais distintas no NTS em preparações cerebrais frescas congeladas e fixas. A IHQ de FISH em preparações de tecidos congelados fixos produziu resultados confiáveis de IHQ. Por exemplo, FISH multiplex para mRNAs de baixa e alta abundância (GalR1 e GlyT2 respectivamente) e IHC (visando tirosina hidroxilase) revelaram que GalR1 e GlyT2 são expressos em neurônios NTS não catecolaminérgicos. A IHQ para HT não foi bem sucedida em tecidos frescos congelados, destacando a capacidade limitada de IHQ de FISH em preparações frescas congeladas.
A ISH pode ser mais apropriada do que a IHC em uma variedade de cenários. Primeiro, a IHQ pode não ter um bom desempenho ao detectar proteínas de baixa abundância, como receptores. O uso de ISH para atingir mRNAs de abundância relativamente maior para essas proteínas melhora a detectabilidade1. Em segundo lugar, proteínas como neuropeptídeos são frequentemente trafegadas para os terminais axonais após a tradução no soma celular19. Quando os neuropeptídeos são visados com IHQ, os processos axonais e terminais das células marcam-se com o anticorpo, mas não com o soma, reduzindo a capacidade de identificar a célula de origem ou realizar análise quantitativa do número de células. No entanto, como os RNAm que codificam todas as proteínas são encontrados localizados no soma, a técnica de ISH é vantajosa. Finalmente, os anticorpos não estão prontamente disponíveis para algumas espécies de proteínas, ou os anticorpos disponíveis são apropriados para outras técnicas proteômicas (por exemplo, western blot), mas não para IHQ. Nestas circunstâncias, os métodos de marcação de mRNA revelam-se úteis. Uma ressalva é que os mRNAs nem sempre podem ser traduzidos em proteína e, portanto, eles fornecem apenas um proxy para a identificação de proteínas. Uma vez que os kits comerciais de FISH podem ser caros e as sondas ISH têm menor probabilidade de estarem comercialmente disponíveis em comparação com os anticorpos, a combinação de FISH com IHQ apresenta uma estratégia eficaz em termos de custo e tempo para aumentar o número de alvos que podem ser rotulados simultaneamente.
A preparação fresca de tecidos congelados versus fixos foi um fator que conferiu sucesso à IHQ após o ensaio com sonda de FISH. Testamos a IHQ usando anticorpos direcionados a proteínas nucleares, vesiculares e citoplasmáticas e encontramos marcação confiável de proteínas ligadas à membrana (vAChT e Phox2b) em amostras frescas congeladas, mas não em proteínas citoplasmáticas (HT e GFP). A coexpressão da proteína Phox2b e do RNAm com a marcação "floculenta" de Phox2b-GFP validou que os neurônios que expressam o transcrito também expressaram a proteína relacionada, confirmando a identidade neuroquímica dos neurônios (Figura 5). Em contraste, preparações de tecidos congelados fixos produziram marcação confiável de IHQ, independentemente da localização subcelular do antígeno. Estudos anteriores demonstraram que o pré-tratamento com protease (por exemplo, pronase 8,20) pode ter um efeito prejudicial sobre a imunoistoquímica. O conteúdo da solução de protease utilizada no protocolo RNAscope é proprietário, e a permeabilização por protease é recomendada para o acesso da sonda RNAscope às células. A marcação de proteínas citoplasmáticas utilizando os anticorpos aqui descritos foi previamente verificada em cortes de cérebro de camundongo congelados fixos de 30 μm flutuantes 10,21,22. Amostras fixas de 30 μm de espessura montadas em lâmina e realizamos o protocolo FISH-IHQ, em oposição aos cortes frescos congelados de 14 μm de espessura recomendados pelo fabricante. Na ausência de modificações no ensaio ou alteração de outras variáveis (recuperação de antígenos, maior concentração de anticorpos, mudança de protease), foi obtida uma imunoimunodeficiência confiável em amostras espessas e fixas, com marcação demonstrada do citoplasma e dos processos axonais juntamente com a marcação da sonda FISH (Figura 6). Enquanto abordagens semelhantes foram empregadas por outros grupos de pesquisa 7,8,9, o presente estudo obteve combinação de IHQ-ISH em neurônios e em configuração fluorescente.
Havia uma série de etapas críticas nos métodos a serem observados. Para a preparação fresca congelada, o tempo de fixação não deve exceder 15 minutos; tempos de fixação mais longos provocaram maior rotulagem de fundo. A etapa de protease foi otimizada, uma vez que tecidos de diferentes espessuras e de vários órgãos necessitam de diferentes tipos de protease para atingir a permeabilização. As seções congeladas fixas aderem menos às lâminas de vidro e desalojam mais facilmente durante as etapas de lavagem. Assim, cuidados redobrados devem ser tomados no manuseio manual de cortes fixos congelados, para evitar perda ou dano tecidual.
Embora tenhamos encontrado a combinação de FISH e IHQ como uma estratégia eficaz, as desvantagens incluem custo e ensaio tecnicamente exigente ao combinar os dois métodos. Uma limitação do estudo é que não foi realizada uma comparação lado a lado dos dois protocolos de preparo tecidual. Além disso, nossa avaliação dos resultados foi limitada pelo número de canais que o microscópio epifluorescente poderia acomodar; A configuração permitia um máximo de 4 canais em um determinado momento: 346, 488, 550 e 647 nm (luz de excitação). Conseguimos marcar multiplex de 5 alvos marcando duas proteínas com localizações subcelulares diferentes usando o mesmo fluróforo (Figura 4, Tabela 1). Usando um microscópio confocal, a excitação discreta de muitos fluoróforos adicionais pode ser usada para marcação de proteínas individuais via IHQ, ou para imagens de moléculas fluorescentes expressas por transgenes.
A combinação de FISH e IHQ fluorescente pode reduzir a confiabilidade de cada técnica isoladamente. No futuro, pretendemos melhorar a marcação de proteínas citoplasmáticas em tecido fresco congelado com um tratamento de recuperação de antígeno23. Estudos prévios mostram que a recuperação antigênica induzida pelo calor aumenta a acessibilidade do epítopo proteico24,25,26. O tratamento térmico cliva as ligações cruzadas e os grupos metilol da proteína e desdobra os antígenos nos tecidos, expondo epítopos que, de outra forma, estariam ocultos na estrutura terciária da proteína em condições biológicas. Essa acessibilidade pode melhorar o sucesso da marcação de proteínas26,27. Adicionalmente, visaremos diferentes epítopos da mesma proteína citoplasmática para determinar se o sucesso da marcação proteína-anticorpo depende dos clones de anticorpos específicos utilizados.
Em conclusão, a combinação de FISH e IHQ é útil para a identificação neuroquímica de populações heterogêneas de células no cérebro, como as do NTS. Este estudo apresenta dois protocolos de ensaio de diferentes preparações de tecido do tronco cerebral de camundongos - fresco, congelado ou fixo - para marcação fluorescente multiplex simultânea de RNAm e proteínas in situ. Ambos os protocolos podem ser amplamente aplicados para detectar o padrão de expressão de mRNAs de baixa abundância, como GalR1. Preparações congeladas fixas espessas (30 μm) permeabilizadas com protease conferiram detecção mais confiável de proteínas citoplasmáticas e mais desafios de manuseio tecidual, quando comparadas a preparações congeladas frescas finas (14 μm).
Agradecimentos
Este trabalho foi financiado pelo Australian Research Council Discovery Project grant DP180101890 e Rebecca L Cooper Medical Research Foundation grant PG2018110
Materiais
| Name | Company | Catalog Number | Comments |
| ANIMALS | |||
| C57BL/6 mouse | Australian BioResources, Moss Vale | MGI: 2159769 | |
| Phox2b-eGFP mouse | Australian BioResources, Moss Vale | MGI: 5776545 | |
| REAGENTS | |||
| Cyanoacrylate | Loctite | ||
| Ethylene Glycol | Sigma-Aldrich | 324558 | |
| Heparin-Sodium | Clifford Hallam Healthcare | 1070760 | Consult local veterinary supplier or pharmacy. |
| Lethabarb (Sodium Pentabarbitol) Euthanasia Injection | Virbac (Australia) Pty Ltd | N/A | Consult a veterinarian for local pharmaceutical regulations regarding Sodium Pentabarbitol |
| Molecular grade agarose powder | Sigma Aldrich | 5077 | |
| OCT Compound, 118mL | Scigen Ltd | 4586 | |
| Paraformaldehyde, prilled, 95% | Sigma-Aldrich | 441244-1KG | |
| Polyvinylpyrrolidone, average mol wt 40,000 (PVP-40) | Sigma-Aldrich | PVP40 | |
| ProLong Gold Antifade Mountant | Invitrogen | P36930 | With or without DAPI |
| RNAscope Multiplex Fluorescent Reagent Kit (up to 3-plex capability) | Advanced Cell Diagnostics, Inc. (ACD Bio) | ADV320850 | Includes 50x Wash buffer and Protease III |
| RNase Away | Thermo-Fisher Scientific | 7003 | |
| Tris(hydroxymethyl)aminomethane | Sigma-Aldrich | 252859 | |
| Tween-20, for molecular biology | Sigma-Aldrich | P9416 | |
| EQUIPMENT | |||
| Benchtop incubator | Thermoline scientific micro incubator | Model: TEI-13G | |
| Brain Matrix, Mouse, 30g Adult, Coronal, 1mm | Ted Pella | 15050 | |
| Cryostat | Leica | CM1950 | |
| Drawing-up needle (23 inch gauge) | BD | 0288U07 | |
| Hydrophobic Barrier Pen | Vector labs | H-4000 | |
| Kimtech Science Kimwipes Delicate Task Wipes | Kimberley Clark Professional | 34120 | |
| Olympus BX51 | Olympus | BX-51 | |
| Peristaltic pump | Coleparmer Masterflex | L/S Series | |
| Retiga 2000R Digital Camera | QImaging | RET-2000R-F-CLR | colour camera |
| SuperFrost Plus Glass Slides (White) | Thermo-Fisher Scientific | 4951PLUS4 | |
| Vibrating Microtome (Vibratome) | Leica | VT1200S | |
| Whatman qualitative filter paper, Grade 1, 110 mm diameter | Merck | WHA1001110 | |
| SOFTWARES | |||
| CorelDRAW | Corel Corporation | Version 7 | |
| FIJI (ImageJ Distribution) | Open Source/GNU General Public Licence (GPL) | N/A | ImageJ 2.x: Rueden, C. T.; Schindelin, J. & Hiner, M. C. et al. (2017), "ImageJ2: ImageJ for the next generation of scientific image data", BMC Bioinformatics 18:529, PMID 29187165, doi:10.1186/s12859-017-1934-z and Fiji: Schindelin, J.; Arganda-Carreras, I. & Frise, E. et al. (2012), "Fiji: an open-source platform for biological-image analysis", Nature methods 9(7): 676-682, PMID 22743772, doi:10.1038/nmeth.2019 |
| PRIMARY ANTIBODIES | |||
| Anti-Tyrosine Hydroxylase Antibody | Millipore Sigma | AB1542 | Sheep polyclonal (1:1000 dilution), RRID: AB_90755 |
| Anti-Tyrosine Hydroxylase Antibody, clone LNC1 | Millipore Sigma | MAB318 | Mouse monoclonal (1:1000 dilution), RRID: AB_2201528 |
| Anti-Vesicular Acetylcholine Transporter (VAchT) Antibody | Sigma-Aldrich | ABN100 | Goat polyclonal (1:1000 dilution), RRID: AB_2630394 |
| GFP Antibody | Novus Biologicals | NB600-308 | Rabbit polyclonal (1:1000 dilution), RRID: AB_10003058 |
| Phox2b Antibody (B-11) | Santa Cruz Biotechnology | sc-376997 | Mouse monoclonal (1:1000 dilution), RRID: AB_2813765 |
| SECONDARY ANTIBODIES | |||
| Alexa Fluor 488 AffiniPure Donkey Anti-Rabbit IgG (H+L) (min X Bov, Ck, Gt, GP, Sy Hms, Hrs, Hu, Ms, Rat, Shp Sr Prot) | Jackson ImmunoResearch | 711-545-152 | Donkey anti-Rabbit (1:400 dilution), RRID: AB_2313584 |
| AMCA AffiniPure Donkey Anti-Sheep IgG (H+L) (min X Ck, GP, Sy Hms, Hrs, Hu, Ms, Rb, Rat Sr Prot) | Jackson ImmunoResearch | 713-155-147 | Donkey anti-Sheep (1:400 dilution), RRID: AB_AB_2340725 |
| Cy5 AffiniPure Donkey Anti-Goat IgG (H+L) (min X Ck, GP, Sy Hms, Hrs, Hu, Ms, Rb, Rat Sr Prot) | Jackson ImmunoResearch | 705-175-147 | Donkey anti-Goat (1:400 dilution), RRID: AB_2340415 |
| Cy5 AffiniPure Donkey Anti-Mouse IgG (H+L) (min X Bov, Ck, Gt, GP, Sy Hms, Hrs, Hu, Rb, Rat, Shp Sr Prot) | Jackson ImmunoResearch | 715-175-151 | Donkey anti-Mouse (1:400 dilution), RRID: AB_2619678 |
| Cy5 AffiniPure Donkey Anti-Sheep IgG (H+L) (min X Ck, GP, Sy Hms, Hrs, Hu, Ms, Rb, Rat Sr Prot) | Jackson ImmunoResearch | 713-175-147 | Donkey anti-Sheep (1:400 dilution), RRID: AB_2340730 |
| RNASCOPE PROBES | |||
| Galanin Receptor 1 oligonucleotide probe | ACDBio | 448821-C1 | targets bp 482 - 1669 (Genebank ref: NM_008082.2) |
| Glycine transporter 2 oligonucleotide probe | ACDBio | 409741-C3 | targets bp 925 - 2153 (Genebank ref: NM_148931.3) |
| Phox2b oligonucleotide probe | ACDBio | 407861-C2 | targets bp 1617 - 2790 (Genebank ref: NM_008888.3) |
Referências
- Wang, F., et al. RNAscope: a novel in situ RNA analysis platform for formalin-fixed, paraffin-embedded tissues. Journal of Molecular Diagnostics. 14 (1), 22-29 (2012).
- Annese, T., et al. RNAscope dual ISH-IHC technology to study angiogenesis in diffuse large B-cell lymphomas. Histochemistry and Cell Biology. 153 (3), 185-192 (2020).
- Morrison, J. A., McKinney, M. C., Kulesa, P. M. Resolving in vivo gene expression during collective cell migration using an integrated RNAscope, immunohistochemistry and tissue clearing method. Mechanisms of Development. 148, 100-106 (2017).
- Gross-Thebing, T., Paksa, A., Raz, E. Simultaneous high-resolution detection of multiple transcripts combined with localization of proteins in whole-mount embryos. BMC Biology. 12, 55(2014).
- Stempel, A. J., Morgans, C. W., Stout, J. T., Appukuttan, B. Simultaneous visualization and cell-specific confirmation of RNA and protein in the mouse retina. Molecular Vision. 20, 1366-1373 (2014).
- Kersigo, J., et al. A RNAscope whole mount approach that can be combined with immunofluorescence to quantify differential distribution of mRNA. Cell and Tissue Research. 374 (2), 251-262 (2018).
- Grabinski, T. M., Kneynsberg, A., Manfredsson, F. P., Kanaan, N. M. A method for combining RNAscope in situ hybridization with immunohistochemistry in thick free-floating brain sections and primary neuronal cultures. PLoS One. 10 (3), 0120120(2015).
- Baleriola, J., Jean, Y., Troy, C., Hengst, U. Detection of axonally localized mRNAs in brain sections using high-resolution in situ hybridization. Journal of Visualized Experiments. (100), e52799(2015).
- Fe Lanfranco, M., Loane, D. J., Mocchetti, I., Burns, M. P., Villapol, S. Combination of fluorescent in situ hybridization (FISH) and immunofluorescence imaging for detection of cytokine expression in microglia/macrophage cells. Bio-Protocol. 7 (22), (2017).
- Dereli, A. S., Yaseen, Z., Carrive, P., Kumar, N. N. Adaptation of respiratory-related brain regions to long-term hypercapnia: focus on neuropeptides in the RTN. Frontiers in Neuroscience. 13, 1343(2019).
- Lazarenko, R. M., et al. Acid sensitivity and ultrastructure of the retrotrapezoid nucleus in Phox2b-EGFP transgenic mice. Journal of Comparative Neurology. 517 (1), 69-86 (2009).
- Gage, G. J., Kipke, D. R., Shain, W. Whole animal perfusion fixation for rodents. Journal of Visualized Experiments. (65), e3564(2012).
- Paxinos, G., Franklin, K. B. The mouse brain in stereotaxic coordinates. , Academic Press. (2004).
- Abercrombie, M. Estimation of nuclear population from microtome sections. Anatomical Records. 94, 239-247 (1946).
- Kerr, N., et al. The generation of knock-in mice expressing fluorescently tagged galanin receptors 1 and 2. Molecular and Cellular Neurosciences. 68, 258-271 (2015).
- Kachidian, P., Pickel, V. M. Localization of tyrosine hydroxylase in neuronal targets and efferents of the area postrema in the nucleus tractus solitarii of the rat. Journal of Comparative Neurology. 329 (3), 337-353 (1993).
- Stornetta, R. L., et al. Expression of Phox2b by brainstem neurons involved in chemosensory integration in the adult rat. Journal of Neuroscience. 26 (40), 10305-10314 (2006).
- Gilmor, M. L., et al. Expression of the putative vesicular acetylcholine transporter in rat brain and localization in cholinergic synaptic vesicles. Journal of Neuroscience. 16 (7), 2179-2190 (1996).
- Fisher, J. M., Sossin, W., Newcomb, R., Scheller, R. H. Multiple neuropeptides derived from a common precursor are differentially packaged and transported. Cell. 54 (6), 813-822 (1988).
- Towle, A. C., Lauder, J. M., Joh, T. H. Optimization of tyrosine-hydroxylase immunocytochemistry in paraffin sections using pretreatment with proteolytic-enzymes. Journal of Histochemistry and Cytochemistry. 32 (7), 766-770 (1984).
- Biancardi, V., et al. Mapping of the excitatory, inhibitory, and modulatory afferent projections to the anatomically defined active expiratory oscillator in adult male rats. Journal of Comparative Neurology. 529 (4), 853-884 (2021).
- Matthews, D. W., et al. Feedback in the brainstem: an excitatory disynaptic pathway for control of whisking. Journal of Comparative Neurology. 523 (6), 921-942 (2015).
- Ramos-Vara, J. A. Principles and methods of immunohistochemistry. Methods in Molecular Biology. 1641, 115-128 (2017).
- Shi, S. R., Key, M. E., Kalra, K. L. Antigen retrieval in formalin-fixed, paraffin-embedded tissues: an enhancement method for immunohistochemical staining based on microwave oven heating of tissue sections. Journal of Histochemistry and Cytochemistry. 39 (6), 741-748 (1991).
- Yamashita, S., Katsumata, O. Heat-induced antigen retrieval in immunohistochemistry: mechanisms and applications. Methods in Molecular Biology. 1560, 147-161 (2017).
- Yamashita, S., Okada, Y. Mechanisms of heat-induced antigen retrieval: analyses in vitro employing SDS-PAGE and immunohistochemistry. Journal of Histochemistry and Cytochemistry. 53 (1), 13-21 (2005).
- Yamashita, S. Heat-induced antigen retrieval: mechanisms and application to histochemistry. Progress in Histochemistry and Cytochemistry. 41 (3), 141-200 (2007).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados