Method Article
Kombination von Multiplex-Fluoreszenz-In-situ-Hybridisierung mit fluoreszierender Immunhistochemie an frischen, gefrorenen oder fixierten Mäusehirnschnitten
In diesem Artikel
Zusammenfassung
Dieses Protokoll beschreibt eine Methode zur Kombination von Fluoreszenz-in-situ-Hybridisierung (FISH) und Fluoreszenz-Immunhistochemie (IHC) sowohl in frisch gefrorenen als auch in fixierten Maus-Hirnschnitten, mit dem Ziel, Multilabel-FISH und Fluoreszenz-IHC-Signal zu erreichen. IHC zielte auf zytoplasmatische und membrangebundene Proteine ab.
Zusammenfassung
Die Fluoreszenz-in-situ-Hybridisierung (FISH) ist eine molekulare Technik, die das Vorhandensein und die räumliche Verteilung spezifischer RNA-Transkripte in Zellen identifiziert. Die neurochemische Phänotypisierung von funktionell identifizierten Neuronen erfordert in der Regel die gleichzeitige Markierung mit mehreren Antikörpern (Targeting-Protein) mittels Immunhistochemie (IHC) und die Optimierung der In-situ-Hybridisierung (Targeting-RNA ). Eine "neurochemische Signatur" zur Charakterisierung bestimmter Neuronen kann erreicht werden, aber zu den erschwerenden Faktoren gehören die Notwendigkeit, FISH- und IHC-Ziele vor der Kombination der Methoden zu verifizieren, und die begrenzte Anzahl von RNAs und Proteinen, die gleichzeitig innerhalb desselben Gewebeschnitts anvisiert werden können.
Hier beschreiben wir ein Protokoll, das sowohl frisch gefrorene als auch fixierte Mäusegehirnpräparate verwendet, das mehrere mRNAs und Proteine im selben Gehirnschnitt mit RNAscope FISH und anschließender Fluoreszenz-Immunfärbung nachweist. Wir verwenden die kombinierte Methode, um das Expressionsmuster von mRNAs mit geringer Häufigkeit (z. B. Galaninrezeptor 1) und mRNAs mit hoher Häufigkeit (z. B. Glycintransporter 2) in immunhistochemisch identifizierten Hirnstammkernen zu beschreiben.
Die wichtigsten Überlegungen für die Proteinmarkierung nach dem FISH-Assay gehen über die Gewebevorbereitung und die Optimierung der FISH-Sondenmarkierung hinaus. Zum Beispiel haben wir herausgefunden, dass die Antikörperbindung und die Markierungsspezifität durch den Proteaseschritt innerhalb des FISH-Sonden-Assays nachteilig beeinflusst werden können. Proteasen katalysieren die hydrolytische Spaltung von Peptidbindungen und erleichtern so den Eintritt der FISH-Sonde in die Zellen, aber sie können auch das Protein verdauen, auf das der nachfolgende IHC-Assay abzielt, wodurch eine Off-Target-Bindung entsteht. Die subzelluläre Lokalisation des Zielproteins ist ein weiterer Faktor, der zum Erfolg der IHC nach dem FISH-Sondentest beiträgt. Wir beobachteten, dass die IHC-Spezifität erhalten blieb, wenn das Zielprotein membrangebunden ist, während IHC, das auf das zytoplasmatische Protein abzielte, eine umfangreiche Fehlerbehebung erforderte. Schließlich stellten wir fest, dass die Handhabung von fixiertem gefrorenem Gewebe auf Objektträgern schwieriger war als die von frischem gefrorenem Gewebe, jedoch war die IHC-Qualität mit fixiertem gefrorenem Gewebe in Kombination mit RNAscope insgesamt besser.
Einleitung
Proteine und mRNAs, die neurochemisch Subpopulationen von Neuronen definieren, werden üblicherweise mit einer Kombination aus Immunhistochemie (IHC) und/oder In-situ-Hybridisierung (ISH) identifiziert. Die Kombination von ISH mit IHC-Techniken erleichtert die Charakterisierung von Kolokalisationsmustern, die für funktionelle Neuronen einzigartig sind (neurochemische Kodierung), indem die Multiplex-Markierungskapazität maximiert wird.
Fluoreszierende ISH (FISH)-Methoden, einschließlich RNAscope, haben eine höhere Sensitivität und Spezifität im Vergleich zu früheren RNA-Nachweismethoden wie radioaktivem ISH und nicht-radioaktivem chromogenem ISH. FISH ermöglicht die Visualisierung einzelner mRNA-Transkripte als punktgefärbte Spots1. Darüber hinaus ermöglicht der RNAscope-Assay die gleichzeitige Markierung einer größeren Anzahl von RNA-Zielen unter Verwendung verschiedener Fluorophor-Tags. Trotz dieser Vorteile können technische Einschränkungen die Anzahl der Fluorophore/Chromogene beeinflussen, die in einem einzigen Experiment verwendet werden können. Dazu gehören die Verfügbarkeit von Mikroskop-Filtersets; Solche Überlegungen werden noch verstärkt, wenn die neurochemische Identifizierung kombinierte FISH und IHC verwendet, verglichen mit der Verwendung beider Techniken allein, da inhärente Schritte, die für eine Methode optimal sind, für die andere nachteilig sein können.
Frühere Anwendungen von FISH in Kombination mit IHC haben die Expression spezifischer zellulärer Ziele in humanen B-Zell-Lymphomen2, Kükenembryonen3, Zebrafischembryonen4, Maus-Retina5 und Maus-Innenohrzellen6 gezeigt. In diesen Studien wurde das Gewebepräparat entweder in formalinfixiertes Paraffin eingebettet (FFPE)2,3,5 oder in frisches ganzesMount-4,6 eingelassen. In anderen Studien wurde chromogenes RNAscope auf fixierte Gehirnpräparate von Mäusen und Ratten angewendet 7,8,9. Insbesondere Baleriola et al.8 beschrieb zwei verschiedene Gewebepräparate für die kombinierte ISH-IHC; Korrigierte Maus-Gehirn-Schnitte und FFPE-Sektionen des menschlichen Gehirns. In einer kürzlich erschienenen Veröffentlichung haben wir FISH und fluoreszierende IHC auf frischen Gefrierschnitten kombiniert, um gleichzeitig mRNA mit geringer Häufigkeit (Galaninrezeptor 1, GalR1), mRNA mit hoher Häufigkeit (Glycintransporter 2, GlyT2) und vesikuläres Acetylcholintransporter (vAChT)-Protein10 in der retikulären Formation des Hirnstamms sichtbar zu machen.
Der Kern des Solitärtrakts (NTS) ist eine wichtige Hirnregion, die an der autonomen Funktion beteiligt ist. Diese heterogene Population von Neuronen befindet sich im Hinterhirn und empfängt und integriert eine große Anzahl autonomer Signale, einschließlich solcher, die die Atmung regulieren. Das NTS beherbergt mehrere neuronale Populationen, die phänotypisch durch das Expressionsmuster von mRNA-Zielen wie GalR1 und GlyT2 sowie Proteinmarker für das Enzym Tyrosinhydroxylase (TH) und den Transkriptionsfaktor Paired-like homeobox 2b (Phox2b) charakterisiert werden können.
Der RNAscope-Inhaber empfiehlt frische, gefrorene Gewebepräparate, aber Gewebe, das durch transkardiale Perfusionsfixierung bei Ganztieren hergestellt wurde, zusammen mit langfristiger Kryoprotektion (Lagerung bei -20 °C) von fixierten gefrorenen Gewebeschnitten, ist in vielen Labors üblich. Daher haben wir versucht, Protokolle für FISH in Kombination mit IHC unter Verwendung von frisch gefrorenen und fixierten gefrorenen Gewebepräparaten zu erstellen. Hier stellen wir für frisch gefrorene und fixierte gefrorene Hirnschnitte zur Verfügung: (1) ein Protokoll für kombinierte FISH und fluoreszierende IHC, (2) eine Beschreibung der Qualität der mRNA- und Proteinmarkierung, die bei Verwendung jedes Präparats erzeugt wird, (3) eine Beschreibung der Expression von GalR1 und GlyT2 im NTS.
Unsere Studie zeigte, dass in Kombination mit der RNAscope-Methodik der IHC-Erfolg in frisch gefrorenen und fixierten gefrorenen Präparaten variierte und von der Lokalisierung der Zielproteine in der Zelle abhing. In unseren Händen war die Markierung membrangebundener Proteine immer erfolgreich. Im Gegensatz dazu erforderte die IHC für zytoplasmatische Proteine eine Fehlerbehebung auch in Fällen, in denen das zytoplasmatische Protein in einem transgenen Tier überexprimiert wurde (Phox2b-GFP)11. Während GalR1 im NTS in nicht-katecholaminergen Neuronen exprimiert wird, fehlt die GlyT2-Expression im NTS.
Protokoll
Eine Zusammenfassung der Schritte zur Gewebevorverarbeitung ist in Abbildung 1 zu finden. Alle Verfahren wurden in Übereinstimmung mit dem Animal Care and Ethics Committee der University of New South Wales in Übereinstimmung mit den Richtlinien für die Verwendung und Pflege von Tieren für wissenschaftliche Zwecke (Australian National Health and Medical Research Council) durchgeführt.
1. Probenvorbereitung von frischem, gefrorenem Hirngewebe
- Transkardiale Perfusion
- Bereiten Sie heparinisierten (2500 U/L) 0,1 M Phosphatpuffer (PB) mit einem pH-Wert von 7,5 vor. Stellen Sie Trockeneis-Ethanol-Aufschlämmung her, indem Sie Trockeneis mit Ethanol mischen. Dieser hat eine Temperatur von ca. −72 °C und wird zum sofortigen Einfrieren des entnommenen Gewebes verwendet.
- Euthanasieren adulte Mäuse mit C57BL/6 und Phox2b-GFP11 (Mouse Genome Informatics database ID MGI:5776545) durch Anästhesie mit Natrium-Pentobarbital (70 mg/kg, i.p.) unter Verwendung einer 27,5-Zoll-Nadelstärke.
ACHTUNG: Pentobarbital ist ein Barbiturat. Es ist in hohen Dosen akut toxisch und kann zum Tod durch Atemstillstand führen. Konsultieren Sie vor der Verwendung die örtlichen Gesundheits-, Rechts- und Materialsicherheitsrichtlinien. - Legen Sie das Herz frei und kanülieren Sie die linke Herzkammer mit einer Aufziehnadel (23 Zoll Gauge). Führen Sie eine transkardiale Perfusion mit heparinisierten 0,1 M PB durch, bis das Blut mit einer Flussrate von 11-13 ml/min klar ist (2-3 Minuten). Bestimmung der Blutreinigung durch Überwachung der Färbung der Leber und des Ausflusses aus dem rechten Vorhof12.
- Isolieren Sie das Gehirn von der Schädelhöhle, betten Sie es sofort in Optimal Cutting Temperature Compound (OCT) in einer Kryomol- oder Aluminiumfolie ein und legen Sie es auf das Trockeneis-Ethanolbad. Lagern Sie das gefrorene eingebettete Gewebe in einem luftdichten Behälter bei - 80 °C bis zu 3 Monate.
- Schneiden von frischem, gefrorenem Gewebe
- Stellen Sie die Kryostattemperatur auf -20 °C ein. Lassen Sie das OCT-eingebettete Gewebe und ein Kryostat-Chuck ~30 Minuten lang im Kryostaten, um ein Gleichgewicht mit der neuen Temperatur zu ermöglichen.
HINWEIS: Halten Sie das Taschentuch immer gefroren; Transportieren Sie das Gewebe aus dem -80 °C-Gefrierschrank auf Trockeneis zum Kryostaten. - Befestigen Sie das Gewebe mit OCT-Compound auf dem vorgekühlten Kryostatfutter. In diesem Protokoll wurden Gewebeblöcke auf das Futter in der koronalen Ebene montiert.
HINWEIS: Schneiden Sie überschüssige OCT mit einer Rasierklinge aus dem Gewebe, um die Menge an OCT zu minimieren, die vom Kryostaten geschnitten und anschließend auf den Objektträger übertragen wird. - Schneiden Sie 14 μm dicke koronale Schnitte und montieren Sie sie auf geladene Glasobjektträger.
- Erwärmen Sie die Objektträger auf Raumtemperatur, bevor Sie die Profile montieren. Sobald der Abschnitt montiert ist, bewahren Sie die Objektträger in einer Objektträgerbox im Kryostaten auf.
- Wenn mehr als ein Abschnitt auf einem Objektträger montiert werden muss, erwärmen Sie den Bereich für den zweiten Teil, indem Sie einen Finger 5-10 Sekunden lang auf die gegenüberliegende Seite des Objektträgers legen, um das Anhaften des Abschnitts am Objektträger zu erleichtern. Ein kalter Gewebeschnitt lässt sich nicht an einem kalten Objektträger befestigen. Die Abschnitte sollten flach auf den Schienen haften; Das Zusammenklappen führt dazu, dass sie während der Waschschritte von den Objektträgern fallen.
- Wenn Risse in den Abschnitten festgestellt werden, erhöhen Sie die Kryostattemperatur um 1-5 °C, um dies zu vermeiden. Besonders wichtig ist es, Gewebeschnitte in unmittelbarer Nähe zueinander auf demselben Objektträger zu platzieren. Dadurch wird die Verschwendung von FISH-Sonden und Reagenzien während des Assays vermieden.
- Auf Objektträgern montierte Gewebeschnitte in einem luftdichten Behälter bei -80 °C bis zu 6 Monate lagern.
HINWEIS: Halten Sie die Abschnitte immer gefroren und vermeiden Sie Einfrier-Auftauzyklen, um einen RNA-Abbau zu verhindern. Transportieren Sie die Objektträgerbox aus dem Inneren des Kryostaten in den -80 °C Gefrierschrank auf Trockeneis.
- Stellen Sie die Kryostattemperatur auf -20 °C ein. Lassen Sie das OCT-eingebettete Gewebe und ein Kryostat-Chuck ~30 Minuten lang im Kryostaten, um ein Gleichgewicht mit der neuen Temperatur zu ermöglichen.
- Fixierung von frischem, gefrorenem Gewebe
- An dem Tag, an dem der FISH-Sondentest durchgeführt werden soll, werden 4 % Paraformaldehyd (PFA) in 0,1 M PB, pH 7,5 (4 % PFA-Lösung) hergestellt. Filtern Sie, indem Sie Filterpapier (Güteklasse 1: 11 μm, Materialtabelle) in einem Buchner-Trichter oder Tiegelfilter durchlaufen.
VORSICHT : PFA ist bei Hautkontakt oder Einatmen schädlich und giftig. Alle Verfahren mit PFA-Lösung sollten in einem Abzug durchgeführt werden. PFA-Lösungsabfälle sollten sorgfältig gemäß den institutionellen Sicherheitsprotokollen entsorgt werden. - Die 4%ige PFA-Lösung auf 4 °C abkühlen lassen. Transportieren Sie das Objektträgergewebe aus dem -80 °C Gefrierschrank in Trockeneis und tauchen Sie es sofort für 15 Minuten in das vorgekühlte Fixiermittel.
HINWEIS: Es ist wichtig, dass dieser Fixierungsschritt 15 Minuten nicht überschreitet, da eine Überfixierung zu einer unspezifischen Hintergrundbeschriftung führt.
- An dem Tag, an dem der FISH-Sondentest durchgeführt werden soll, werden 4 % Paraformaldehyd (PFA) in 0,1 M PB, pH 7,5 (4 % PFA-Lösung) hergestellt. Filtern Sie, indem Sie Filterpapier (Güteklasse 1: 11 μm, Materialtabelle) in einem Buchner-Trichter oder Tiegelfilter durchlaufen.
- Dehydrierung von frischem, gefrorenem Gewebe
- Dehydrieren Sie Gewebeschnitte, indem Sie die Objektträger in abgestufte Ethanolkonzentrationen eintauchen. In einem Coplin-Glas zuerst 50 %, dann 70 % und schließlich absolutes Ethanol für jeweils 5 Minuten bei Raumtemperatur eintauchen. Wiederholen Sie die abschließende absolute Ethanol-Inkubation ein zweites Mal.
- Trocknen Sie die Objektträger an der Luft und skizzieren Sie die Schnittgruppe mit einem hydrophoben Barrierestift, wobei darauf zu achten ist, dass der Innenbereich auf ein Minimum reduziert wird.
HINWEIS: Stellen Sie sicher, dass der Objektträger vollständig trocken ist, bevor Sie die hydrophobe Barriere ziehen. Die hydrophobe Barriere sollte die Gewebeschnitte völlig lückenlos umschließen und muss vor der Weiterverarbeitung trocken sein.
2. Probenvorbereitung von fixiertem gefrorenem Hirngewebe
- Transkardiale Perfusionsfixierung
- Euthanasie der Mäuse durch Anästhesie mit Natrium-Pentobarbital (70 mg/kg, i.p.), gefolgt von transkardialer Perfusion, zuerst mit 0,1 M PB, dann mit 4%iger PFA-Lösung. Fixieren Sie mit 10 Minuten Perfusion bei 11-13 ml/min.
- Das Gehirn wird nach der Perfusionsfixierung von der Schädelhöhle isoliert und über Nacht in eine 4%ige PFA-Lösung bei 4 °C getaucht.
- Gewebeschnitt von fixiertem Gewebe
- Spülen Sie das Gehirn mit steriler 0,1 M phosphatgepufferter Kochsalzlösung (PBS) ab, bevor Sie die Hirnhautschichten mit Hilfe eines Präpariermikroskops mit einer feinen Pinzette entfernen.
- Schneiden Sie das Gehirn präzise in Blöcke (trennen Sie den Hirnstamm vom Vorderhirn vor dem Vibratomschnitt) mit Hilfe einer Gehirnmatrix (Materialtabelle). Konkret wird der Hirnstamm kaudal an der pyramidalen Dekussion durchtrennt und das Kleinhirn wegseziert. In ähnlicher Weise schneidest du das Vorderhirn unmittelbar rostral zum Chiasma opticus.
- Befestigen Sie das Gewebe mit Cyanacrylat auf einem vibrierenden Mikrotomfutter und betten Sie es in eine 2%ige Agarlösung ein.
- Schneiden Sie 30 μm dicke Gewebeschnitte mit einem vibrierenden Mikrotom und lagern Sie die geschnittenen Abschnitte in kryoprotektiver Lösung (30 % RNase-freie Saccharose, 30 % Ethylenglykol, 1 % Polyvinylpyrrolidon (PVP-40), in 0,1 M PB, pH 7,4). Gewebeschnitte können bis zu 6 Monate in Kryoprotektivum bei -20 °C gelagert werden.
- Vorbereitung von fixierten Schnitten vor FISH
- Waschen Sie am Tag des FISH die frei schwimmenden Abschnitte dreimal für 10 Minuten pro Wäsche, um die Kryoprotektivlösung zu entfernen. Zum Waschen werden die Abschnitte in 0,1 M PBS in eine 12-Well-Zellkulturplatte gegeben und auf einem rotierenden Plattformschüttler (90 - 100 U/min) gerührt.
- Verwenden Sie nach dem Waschen einen Pinsel, um Abschnitte auf Glasobjektträgern zu befestigen, und lassen Sie sie mindestens 2 Stunden lang an der Luft trocknen.
HINWEIS: Die Abschnitte sollten flach auf den Objektträgern haften, da sich ausgeprägte Falten beim Waschen lösen. - Ziehen Sie mit einem hydrophoben Barrierestift eine Barriere um die Abschnitte, um die FISH-Reagenzien auf die Abschnitte zu beschränken. Auch hier ist es wichtig, den Innenbereich des mit dem Barrierestift gezeichneten Umrisses zu minimieren.
MÖGLICHER BRUCHPUNKT: Die Abschnitte könnten über Nacht bei Raumtemperatur gelagert werden, um den Assay am nächsten Tag fortzusetzen.
3. FISH-Analyse
HINWEIS: Der Rest des Protokolls gilt sowohl für frisch gefrorenes als auch für fixiertes gefrorenes Gewebe.
- Bereiten Sie die Reagenzien und Instrumente für die Hybridisierungs- und Amplifikationsschritte vor.
- Stellen Sie einen Tischinkubator und ein Wasserbad auf 40 °C ein.
- Bereiten Sie eine befeuchtete, lichtgeschützte Kammer für die Inkubation von Objektträgern vor. Die Befeuchtung verhindert das Austrocknen des Gewebes - die Objektträger befinden sich sicher über einem feuchten Reservoir. Im Idealfall besteht die Kammer aus strapazierfähigem Polystyrol, sie ist licht- und luftdicht, um eine gesättigte Wasserdampfatmosphäre aufrechtzuerhalten. Der Verschluss der Kammer beruht auf minimaler Reibung, um Bewegungen zu vermeiden. Wir haben unten eine Objektträgerbox verwendet, die mit feuchten Labortüchern (Materialtabelle) ausgekleidet war. Stellen Sie die Objektträgerbox in den Inkubator, um sie auf 40 °C vorzuwärmen.
- Erwärmen Sie den 50x Waschpuffer (Materialtabelle) und die Sonden 10 Minuten lang im Wasserbad auf 40 °C und kühlen Sie sie dann auf Raumtemperatur ab.
- Bereiten Sie 1 l 1x Waschpuffer aus der 50-fachen Vorratskonzentration vor.
- Sondenmischung vorbereiten (Materialtabelle): Die C1-Sonde ist bei Lagerkonzentration gebrauchsfertig, während C2- und C3-Sonden als 50-fache Konzentration geliefert werden und mit dem im Kit enthaltenen Verdünnungsmittel verdünnt werden müssen.
HINWEIS: Sondenmischungen können bei 4 °C bis zu 6 Monate gelagert werden.
- Protease-Behandlung
- Schnitte mit Protease III (Materialtabelle) 30 Minuten lang bei Raumtemperatur inkubieren.
HINWEIS: Stellen Sie sicher, dass Protease III und Inkubationsreagenzien in nachgeschalteten Prozessen (Sondenmischung, Amplifikationslösungen, Blockierungspuffer und Antikörperseren) die Abschnitte vollständig abdecken. Eine Pipettenspitze kann verwendet werden, um das Reagenz auf dem Abschnitt zu verteilen, um den gesamten Bereich innerhalb der hydrophoben Barriere abzudecken. - Waschen Sie die Objektträger zweimal mit 0,1 M PBS jeweils 2 Minuten lang in einer großen quadratischen Petrischale aus Kunststoff. Hier wurde eine 245 mm x 245 mm große quadratische Bioassay-Schale verwendet (Materialtabelle). Halten Sie die Schale von einer Seite und kippen Sie sie vorsichtig 3-5 Mal. Nach dem Waschen überschüssige 0,1 M PBS vom Objektträger entfernen und sofort das nächste Reagenz hinzufügen. Gewebeschnitte nicht austrocknen lassen.
HINWEIS: Bei jedem Waschen werden die Objektträger bei Raumtemperatur in Lösung getaucht. Dies ist der Arbeitsablauf für alle nachfolgenden Waschschritte. Die festen 30 μm dicken Abschnitte lösen sich leichter von den Objektträgern als 14 μm dicke Abschnitte, seien Sie während der Wäsche schonend.
- Schnitte mit Protease III (Materialtabelle) 30 Minuten lang bei Raumtemperatur inkubieren.
- Hybridisierung und Amplifikation
- Nach dem Abwaschen der Proteaselösung legen Sie die Objektträger in die befeuchtete, vorgewärmte Kammer. Inkubieren Sie Schnitte mit Sondenmischung (Materialtabelle) für 2 Stunden bei 40 °C in einem Tischinkubator.
HINWEIS: Stellen Sie sicher, dass mindestens 2 Abschnitte für Positiv- und Negativkontrollsonden reserviert sind, um die RNA-Qualität der Probe und die optimale Permeabilisierung zu beurteilen. Positive Kontrollsonden zielen auf Haushaltsgene ab; hier handelte es sich um einen Cocktail von RNAs, die auf Ubiquitin C (UBC; hohe Abundanz), Peptidylpropylisomerase B (PPIB; moderate Abundanz) und RNA-Polymerase 2a (POLR2A; geringe Abundanz) abzielen. Negative Kontrollsonden zielen auf das bakterielle 4-Hydroxy-Tetrahydrodipicolinat-Reduktase-Gen (DapB) ab, das normalerweise in Gehirnproben von Mäusen fehlt. Ein positives DapB-Signal weist auf ein unspezifisches Signal und/oder eine bakterielle Kontamination der Probe hin. - Nach der Hybridisierung mit dem Sondengemisch bestehen die Signalverstärkungsschritte aus der Inkubation mit Amp 1-FL (30 Minuten), dann mit Amp 2-FL (15 Minuten), gefolgt von Amp 3-FL (30 Minuten) und schließlich Amp 4-FL (15 Minuten) - jeweils bei 40 °C. Decken Sie die Gewebeabschnitte mit den mitgelieferten Tropfflaschen mit Amplifikationslösung ab. Fahren Sie nach dem letzten Amplifikationsschritt mit dem IHC-Assay fort.
- Spülen Sie die Objektträger zwischen der Sondenhybridisierung und jedem Amplifikationsschritt zweimal 2 Minuten lang mit Waschpuffer.
- Nach dem Abwaschen der Proteaselösung legen Sie die Objektträger in die befeuchtete, vorgewärmte Kammer. Inkubieren Sie Schnitte mit Sondenmischung (Materialtabelle) für 2 Stunden bei 40 °C in einem Tischinkubator.
4. IHC-Analyse
- IHC-Sperrschritt
- Um eine unspezifische Bindung von Antikörpern zu verhindern, werden die Abschnitte nach dem FISH-Assay 1 h lang bei Raumtemperatur mit einer Blockierungslösung inkubiert, die 10 % normales Pferdeserum und 0,3 % Tween20 in 1x TBSm (50 mM Tris-Cl, pH 7,5, 150 mM NaCl, 0,05 % Merthiolat) enthält. Präparation der Primärantikörper in einem Verdünnungspuffer, der 1x TBSm, 5 % normales Pferdeserum und 0,1 % Tween20 enthält. Die Lieferanten von Primärantikörpern sind in der Materialtabelle aufgeführt.
- Immunhistochemie
- Überschüssigen Blockierungspuffer durch Schnippen des Objektträgers entfernen und Abschnitte mit Primärantikörpern über Nacht bei 4 °C inkubieren.
- Objektträger 3 Mal (je 5 Minuten) mit 1x TBSm waschen und mit sekundärem Antikörper in einem Verdünnungsmittel mit 1x TBSm, 1 % normalem Pferdeserum und 0,1 % Tween20 für 2 Stunden bei Raumtemperatur inkubieren. Sekundäre Antikörper, die in diesem Protokoll verwendet werden, sind in der Materialtabelle aufgeführt.
- Objektträger 3 mal mit 1x TBSm (je 5 Minuten) waschen, bevor sie mit Eindeckmedium mit oder ohne DAPI (Table of Materials) abgedeckt werden.
5. Bildgebung
- Untersuchen Sie die Immunfärbung unter einem Epifluoreszenzmikroskop, das mit einer Kamera ausgestattet ist (siehe Materialtabelle für Details). Erfassen Sie repräsentative Bilder mit 20-facher Vergrößerung und speichern Sie sie als TIFF-Dateien.
- Exportieren Sie repräsentative Bilder in eine Bildverarbeitungssoftware (Materialtabelle), um Helligkeit und Kontrast anzupassen, um die Klarheit zu erhöhen und die tatsächliche Wiedergabe widerzuspiegeln.
6. OPTIONAL: Quantitative Analyse der Zieltranskripte
HINWEIS: Dies ist ein Methodenartikel und quantitative Ergebnisse werden nicht bereitgestellt. Die hier vorgestellte Quantifizierungsmethode stammt von Dereli et al.10. Anmelden
- Nehmen Sie Bilder aus den Regionen von Interesse auf, wie in Abschnitt 5.1 beschrieben, und wenden Sie die gleichen Mikroskop- und Kameraeinstellungen (z. B. Belichtungszeit und Lichtintensität) auf alle Bilder desselben Fluorophors an.
- Zeichnen Sie die neuronalen Profile mit einer Bildanalysesoftware (Table of Materials) auf.
- Richten Sie die Abschnitte in Bezug auf das Bregma-Niveau gemäß einem stereotaktischen Hirnatlas13 aus.
- Wenden Sie die gleiche Helligkeit und den gleichen Kontrast auf alle Bilder desselben Fluorophors an. Betrachten Sie nur die Neuronen mit DAPI-gefärbten Kernen.
- Zählen Sie manuell die Anzahl der mRNA-, Protein-exprimierenden, mRNA/mRNA-, Protein-/Protein- und mRNA/Protein-koexprimierenden Zellen innerhalb der interessierenden Region.
- Um die Verzerrung in den experimentellen Ergebnissen zu verringern, lassen Sie die Person, die die experimentellen Ergebnisse quantifiziert, für die experimentellen Gruppen verblinden.
- Wenden Sie die Abercrombie-Korrektur14 auf die Gesamtzahl der Zellen an, indem Sie die folgende Abercombie-Gleichung verwenden:
Korrigierte Zellzahl = manuelle Zellzahl x Schnittdicke / (Schnittdicke + Kerngröße)
Zum Beispiel wird für 14 μm dicke Abschnitte die durchschnittliche Kernbreite mit 7,7 ± 0,3 μm berechnet und die durchschnittliche Querschnittsdicke beträgt 14 ± 1 μm, basierend auf 30 Zellen bzw. 10 Abschnitten bei 5 Tieren10. Nach der Abercrombie-Gleichung wäre die korrigierte Zellzahl die manuelle Zellzahl x 14/(14+7,7).

Abbildung 1: Paralleler Arbeitsablauf von Gewebevorverarbeitungsschritten sowohl für frisch gefrorenes als auch für paraformaldehydfixiertes Gewebe. Die Verarbeitungsschritte für frisch gefrorenes Gewebe werden in den rot umrandeten Kästchen angezeigt, während die für paraformaldehyd (PFA) fixiertes Gewebe in den blau umrandeten Kästchen angezeigt werden. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 2: Zusammenfassung des kombinierten FISH-Sonden- und immunhistochemischen Verfahrens. Nach der Vorverarbeitung des Gewebes wird das auf dem Objektträger montierte Gewebe mit einem hydrophoben Barrierestift eingekreist, wie im ersten Bild zu sehen, und in einer Proteaselösung bei Raumtemperatur inkubiert. Nach dem Waschen wird das Gewebe zur Hybridisierung für 2 Stunden in einen Tischinkubator überführt, bevor die sequentiellen Amplifikationsschritte fortgesetzt werden. Das In-situ-Hybridisierungssystem verwendet ein proprietäres "Z-Sonden"-Design, Vorverstärker und Verstärker, wie in den Frames 3-66 zu sehen. Sobald das Gewebe einer FISH-Sondenverarbeitung unterzogen wurde, wird es vor der Blockierung mit normalem Pferdeserum gewaschen. Die Primärantikörper-Inkubation wird über Nacht bei 4 °C durchgeführt, um die Antikörper-Antigen-Bindung zu maximieren. Die Sekundärantikörper-Inkubation (2 Stunden) wurde bei Raumtemperatur durchgeführt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Ergebnisse
In dieser Arbeit skizzieren wir eine Methode zur Kombination von Multiplex-FISH mit fluoreszierender IHC, um die mRNA-Expression für GalR1 und GlyT2 unter Verwendung von frisch gefrorenem bzw. paraformaldehydfixiertem Gewebe im NTS der Maus zu lokalisieren. Eine Pipeline der in den Methoden beschriebenen Gewebeverarbeitungs-, FISH- und IHC-Verfahren ist in Abbildung 1 und Abbildung 2 dargestellt. Tabelle 1 enthält eine Zusammenfassung der in jeder Abbildung verwendeten FISH-Sonden- und Antikörperkombinationen.
Kontrollsonden werden routinemäßig gleichzeitig mit der Zielsonde analysiert, um die Integrität des Arbeitsablaufs zu gewährleisten und die Probenqualität zu bestätigen. Das Fehlen einer DapB-Markierung bestätigt die einwandfreie Qualität und Integrität des Gewebes sowie die Abwesenheit einer bakteriellen Kontamination (Abbildung 3A). Die Markierung von Positivkontrollsonden, die auf Ubiquitin C (UBC, hohe Abundanz), Peptidylpropylisomerase B (PPIB, moderate Abundanz) und RNA-Polymerase 2a (POLR2A, geringe Abundanz) mRNA abzielen, bestätigt die RNA-Integrität und das zwischen den Assays beobachtete Signal kann zur Kalibrierung der Inter-Assay-Variabilität verwendet werden (Abbildung 3B). Um die Expression der FISH-Sonde zu validieren, verwendeten wir Kontrollgewebe, die zuvor beschrieben wurden, um das mRNA-Transkript zu exprimieren. Zum Beispiel wurde die GalR1-mRNA-Expression im Thalamus als positiv bestätigt, wie zuvor beschrieben10,15. Die Verteilung der Phox2b-mRNA wurde zusätzlich durch Co-Markierung mit dem Phox2b-Antikörper verifiziert; Wir bestätigten, dass die FISH-Markierung nur in Neuronen vorhanden war, die auch mit dem Phox2b-Antikörper positiv gefärbt wurden (Abbildung 5).
Um GalR1+-Neuronen im NTS von benachbarten Zellkernen zu unterscheiden, verwendeten wir zusätzliche neurochemische Marker. TH, Phox2b oder Phox2b-GFP-Immunreaktivität (Abbildung 4-6) und Phox2b FISH (Abbildung 5 und Abbildung 6) unterschieden das NTS von anderen Kernen im dorsalen Hirnstamm, da zuvor berichtet wurde, dass NTS-Neuronen Phox2b und TH 16 exprimieren,17. Da das NTS von cholinergen Kernen umgeben ist - es liegt dorsal des Nucleus hypoglossus und des dorsalen motorischen Kerns des Vagus (DMNX) und ventral des Nucleus vestibularis - haben wir mit dem cholinergen Marker vAChT18 co-markiert (Abbildung 4). Daher wurde die Expression von GalR1 innerhalb des NTS in Bezug auf TH und Phox2b untersucht, während die vAChT-Markierung die räumliche Orientierung in Bezug auf rostrocaudale, dorsoventrale und mediolaterale Koordinaten unterstützte. Wir fanden heraus, dass alle TH-immunreaktiven und GalR1-mRNA-positiven Neuronen im NTS Phox2b-GFP-immunreaktiv waren, aber nicht alle Phox2b-GFP-immunreaktiven Neuronen im NTS waren TH-immunreaktiv oder GalR1-mRNA-positiv (Abbildung 4). Außerdem konnten wir zeigen, dass mRNA für den Low-Venanz-Rezeptor GalR1 in TH- und vAChT-immunreaktiven Neuronen fehlte.
In frisch gefrorenen Präparaten hing der IHC-Erfolg in Kombination mit dem FISH-Sondenassay von der subzellulären Lokalisation des Zielproteins ab. Zum Beispiel war vAChT (ein synaptisches Vesikelmembran-gebundenes Protein) eindeutig immunmarkiert, während TH und GFP (zytoplasmatische Proteine) unbegrenzt immunmarkiert waren und nur schwach beobachtet wurden (Abbildung 4). Wir bezeichnen diese unbestimmte Markierung als "flockig", da die Zellen keinen klaren Umriss hatten und sich als schwer vom Hintergrund zu unterscheiden erwiesen. Auf demselben frisch gefrorenen Gewebeschnitt war die GalR1 FISH-Sondenmarkierung der zytoplasmatischen GalR1-mRNA punktgenau und deutlich zu beobachten (Abbildung 4).
Da die TH- und vAChT-Antikörper im selben Wirt gezüchtet werden, wurden beide Proteine mit dem gleichen Sekundärantikörper und damit mit dem gleichen Farbfluorophor markiert (Anregungslicht: 594). Sie sind aus zwei Gründen leicht zu unterscheiden: Sie markieren nie in denselben Neuronen, und die subzelluläre Lokalisation ist für diese Proteine unterschiedlich; vAChT in Vesikeln, die ein punktförmiges Aussehen aufweisen, und TH im Zytoplasma und in neuronalen Prozessen.
Um unsere Hypothese zu untermauern, dass die IHC-Qualität (in frisch gefrorenen Präparaten) von der subzellulären Lokalisation des Proteins abhängt, verglichen wir die Markierung von Phox2b-mRNA (im Zytoplasma), GFP (überexprimiert im Zytoplasma) und Phox2b-Protein (hauptsächlich im Zellkern zu finden) in Neuronen. Wie erwartet, zeigen unsere Ergebnisse eine Überlappung der Markierung von Phox2b-mRNA-, GFP- und Phox2b-Antikörpern in einzelnen Neuronen des NTS (Abbildung 5). Zellen mit zytoplasmatischer mRNA-Markierung korrespondierten mit Zellen, die eine nukleäre Markierung des Phox2b-Proteins aufwiesen, was eine Validierung der kombinierten FISH-IHC-Methode darstellt. Obwohl zytoplasmatisches Phox2b-GFP ein flockiges Aussehen hatte, war das Signal des nukleären Phox2b-Proteins klar und spezifisch. Zusammenfassend lässt sich sagen, dass membrangebundene Proteine, einschließlich vAChT und Phox2B, in Kombination mit FISH auf frisch gefrorenen Präparaten eine höhere Qualität der Immunmarkierung aufweisen als zytoplasmatische Proteine.
Im Gegensatz dazu war die IHC unabhängig von der subzellulären Lokalisation zuverlässig, wenn sie an fixierten Gefrierschnitten in Kombination mit FISH durchgeführt wurde. Multiplex FISH für GlyT2 mRNA und Phox2b mRNA war erfolgreich, wie in Abbildung 6 dargestellt. GlyT2-mRNA-positive Neuronen befanden sich ventral des NTS und nicht innerhalb des NTS. Die Neuronen von GlyT2+ und Phox2b+ kolokalisierten nicht. Eine Subpopulation von Phox2b+ NTS-Neuronen war TH-immunreaktiv und keines enthielt GlyT2-mRNA. TH-immunreaktive Neuronen sind auf demselben Gewebeschnitt nachweisbar und weisen positiv markierte Soma- und neuronale Fortsätze auf (Abbildung 6). Dies steht im Gegensatz zum "flockigen" Auftreten von immunreaktiven TH-Neuronen in frisch gefrorenen Gewebeschnitten. Somit ist das hier beschriebene fixierte gefrorene Präparat eine alternative Methode der Gewebepräparation, die ein zuverlässiges Targeting von zytoplasmatischen Proteinen immunhistochemisch in Kombination mit RNAscope ermöglicht.

Abbildung 3: Repräsentative mikroskopische Aufnahmen von koronalen Vorderhirnschnitten der Maus auf Höhe des lateralen Septums (Bregma 1.1 bis -0.1) zeigen die Markierung von Positiv- und Negativkontrollsonden . (A) Ein fehlendes Signal nach ISH mit bakterieller 4-Hydroxy-Tetrahydrodipicolinat-Reduktase (DapB) bestätigt das Fehlen von Hintergrundsignalen. (B) Die Markierung mit Positivkontrollsonden, die auf Ubiquitin C (UBC), Peptidylpropylisomerase B (PPIB) und RNA-Polymerase 2a (POLR2A) abzielen, veranschaulicht das Signal, das von Zielen mit hoher, mittlerer bzw. niedriger Häufigkeit zu erwarten ist. Die Maßstäbe betragen 50 μm. Alle Bilder wurden mit einem 20-fach-Objektiv aufgenommen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
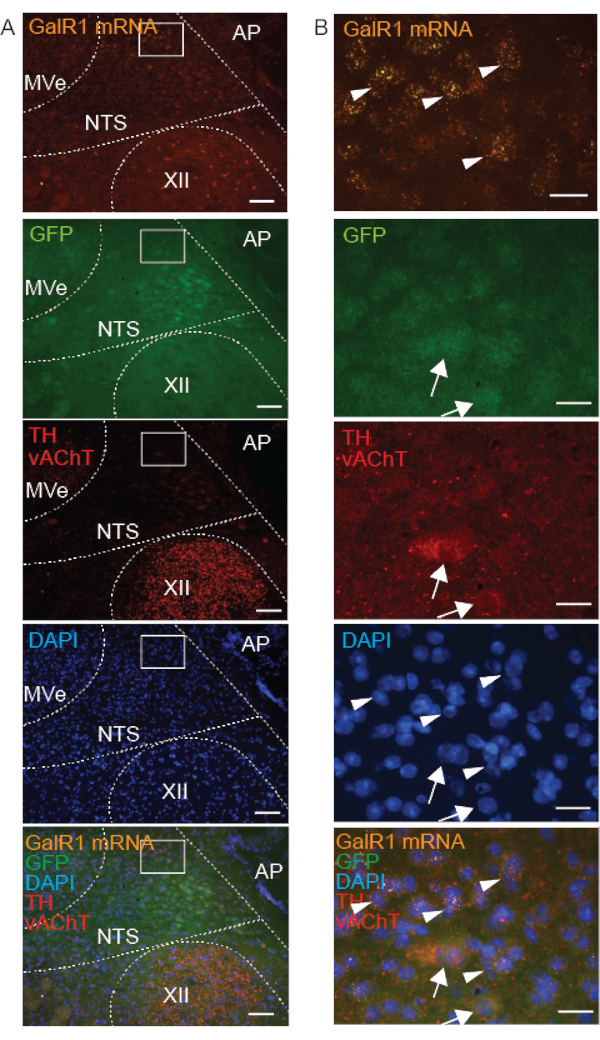
Abbildung 4: Repräsentative mikroskopische Aufnahmen eines frisch gefrorenen koronalen Hirnstammschnitts einer Phox2b-GFP-Maus, die eine kombinierte Markierung von GalR1-mRNA (FISH) und 3 Proteinen (IHC) im Zellkern der Region des Solitärtrakts (NTS) zeigt. Einschübe in A sind in B vergrößert. GalR1-mRNA wird durch punktförmige FISH-Sondenmarkierung (Pfeilspitzen) angezeigt. Antikörper, die auf die zytoplasmatischen Proteine GFP und Tyrosinhydroxylase (TH) abzielen, wiesen eine "flockenlose" Markierung auf (Pfeile). Die Immunreaktivität des vesikulären Acetylcholintransporters (vAChT) wird im Nucleus hypoglossus nachgewiesen (Red Punctate Markierung) (XII). Die Maßstäbe betragen 100 μm in A und 25 μm in B. Alle Bilder wurden mit einem 20-fach-Objektiv aufgenommen. Andere Abkürzungen: Area postrema (AP), Nucleus vestibularis medialis (MVe). Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
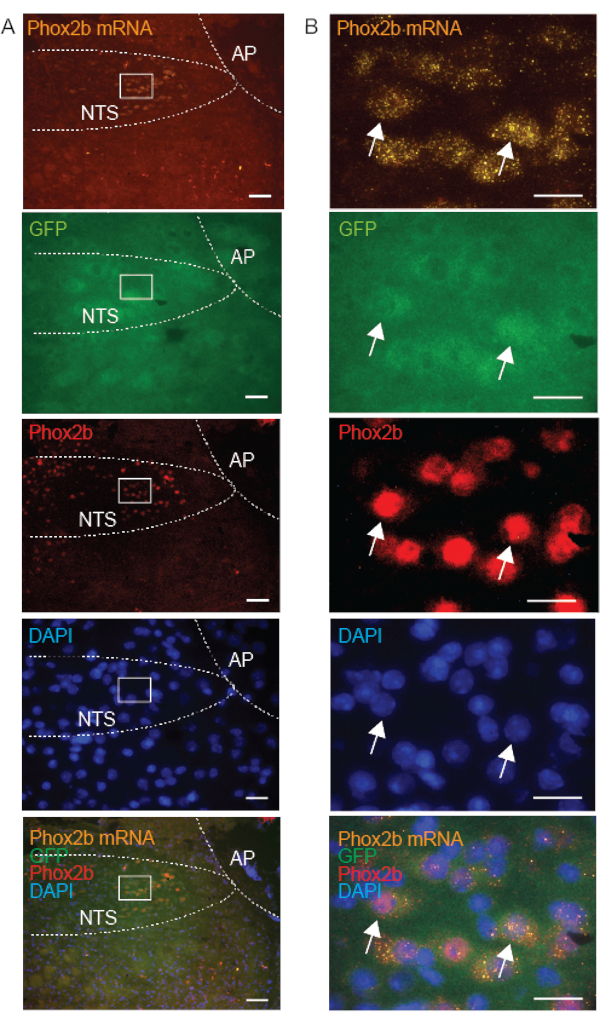
Abbildung 5: Repräsentative mikroskopische Aufnahmen eines frisch eingefrorenen koronalen Hirnstammschnitts einer Phox2b-GFP-Maus, die das Targeting von Phox2b im Zellkern des Solitärtrakts (NTS) mit drei verschiedenen Ansätzen veranschaulichen: Phox2b mRNA (FISH), GFP (IHC) und Phox2b Protein (IHC). Das Phox2b-Protein ist im Zellkern lokalisiert. Die Einsätze in A sind in B vergrößert. Die Pfeile zeigen Neuronen an, die dreifach mit der Phox2b-Sonde (orange-550), dem GFP-Antikörper (grün-488) und dem Phox2b-Antikörper (rot-647) markiert sind. Die Maßstäbe betragen 100 μm in A und 25 μm in B. Alle Bilder werden mit einem 20-fach-Objektiv aufgenommen. Andere Abkürzungen: area postrema (AP). Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 6: Repräsentative Bilder von fixierten gefrorenen koronalen Hirnstammschnitten, die eine erfolgreiche FISH in Kombination mit einer zuverlässigen Immunmarkierung zytoplasmatischer Proteine (Tyrosinhydroxylase [TH]) zeigen. Doppel-FISH zeigt die mRNA-Markierung von Glycin-Transporter 2 (GlyT2-red-647, gefüllte Pfeilspitzen) und Phox2b (gelb-550, Pfeile) im Zellkern der Region des Solitärtrakts (NTS). FISH wurde mit IHC für das TH-Protein (blau-346, leere Pfeilspitzen) kombiniert. Die Einschübe in A sind in B vergrößert. Die Maßstabsbalken betragen 25 μm. Alle Bilder wurden mit einem 20-fach-Objektiv aufgenommen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
| Primärer Antikörper oder RNAscope-Sonde | Sekundärer Antikörper oder Amp 4-FL-Alt Display-Modul | Anregung (nm) | Präparation des Gewebes | ||
| Abbildung 3 | Sonde | POLR2A (C1) | Amp 4-FL-Alt B Display-Modul | 647 | frisch gefroren |
| Sonde | PPIB (C2) | Amp 4-FL-Alt B Display-Modul | 488 | ||
| Sonde | UBC (C3) | Amp 4-FL-Alt B Display-Modul | 550 | ||
| Sonde | DapB (C1, C2, C3) | Amp 4-FL-Alt B Display-Modul | 647, 488, 550 | ||
| DAPI | 346 | ||||
| Abbildung 4 | Antikörper | Kaninchen-Anti-GFP | Esel-Anti-Kaninchen | 488 | frisch gefroren |
| Antikörper | Schaf-Anti-TH | Esel-Anti-Schaf | 647 | ||
| Antikörper | ziege-anti-vAChT | Esel-Anti-Ziege | 647 | ||
| Sonde | GalR1 (C1) | Amp 4-FL-Alt B Display-Modul | 550 | ||
| DAPI | 346 | ||||
| Abbildung 5 | Antikörper | Kaninchen-Anti-GFP | Esel-Anti-Kaninchen | 488 | frisch gefroren |
| Antikörper | Maus-Anti-Phox2b | Esel-Anti-Maus | 647 | ||
| Sonde | Phox2b (C2) | Amp 4-FL-Alt A Display-Modul | 550 | ||
| DAPI | 346 | ||||
| Abbildung 6 | Antikörper | Maus-Anti-TH | Esel-Anti-Maus | 346 | Fest |
| Sonde | GlyT2-KARTON | Amp 4-FL-Alt A Display-Modul | 647 | ||
| Sonde | Phox2b | Amp 4-FL-Alt A Display-Modul | 550 |
Tabelle 1: FISH-Sonde, Antikörper und entsprechende Flurophor-Kombinationen, die in den Abbildungen 3-6 verwendet werden.
Diskussion
In den Neurowissenschaften werden FISH und IHC routinemäßig eingesetzt, um die räumliche Organisation und funktionelle Bedeutung von mRNA oder Proteinen innerhalb neuronaler Subpopulationen zu untersuchen. Das in dieser Studie beschriebene Protokoll verbessert die Fähigkeit zum gleichzeitigen Nachweis von mRNAs und Proteinen in Hirnschnitten. Unser kombinierter Multiplex-FISH-IHC-Assay ermöglichte die phänotypische Identifizierung verschiedener neuronaler Subpopulationen im NTS sowohl in frisch gefrorenen als auch in fixierten Gehirnpräparaten. FISH-IHC in fixierten gefrorenen Gewebepräparaten führte zu zuverlässigen IHC-Ergebnissen. Zum Beispiel zeigten Multiplex-FISH für mRNAs mit niedriger und hoher Häufigkeit (GalR1 bzw. GlyT2) und IHC (Targeting Tyrosinhydroxylase), dass GalR1 und GlyT2 in nicht-katecholaminergen NTS-Neuronen exprimiert werden. Die IHC für TH war in frischem gefrorenem Gewebe nicht erfolgreich, was die begrenzte Kapazität von FISH-IHC in frisch gefrorenen Zubereitungen unterstreicht.
ISH kann in einer Reihe von Szenarien besser geeignet sein als IHC. Erstens kann IHC beim Nachweis von Proteinen mit geringer Häufigkeit, wie z. B. Rezeptoren, nicht gut abschneiden. Die Verwendung von ISH als Ziel für mRNAs mit relativ höherer Häufigkeit für diese Proteine verbessert die Nachweisbarkeit1. Zweitens werden Proteine wie Neuropeptide nach der Translation im Zellsoma oft zu den axonalen Endigungen transportiert19. Wenn Neuropeptide mit IHC angegriffen werden, markieren die axonalen Fortsätze und Endigungen der Zellen mit dem Antikörper, aber nicht mit dem Soma, wodurch die Fähigkeit verringert wird, die Ursprungszelle zu identifizieren oder eine quantitative Analyse der Anzahl der Zellen durchzuführen. Da jedoch mRNAs, die für alle Proteine kodieren, im Soma lokalisiert sind, ist die ISH-Technik von Vorteil. Schließlich sind Antikörper für einige Proteinspezies nicht ohne weiteres verfügbar, oder die verfügbaren Antikörper sind für andere Proteomik-Techniken (z. B. Western Blot), aber nicht für IHC geeignet. Unter diesen Umständen erweisen sich mRNA-Markierungsmethoden als nützlich. Ein Vorbehalt ist, dass mRNAs möglicherweise nicht immer in Proteine übersetzt werden und daher nur einen Proxy für die Proteinidentifikation darstellen. Da kommerzielle FISH-Kits kostspielig sein können und die ISH-Sonden im Vergleich zu Antikörpern mit geringerer Wahrscheinlichkeit kommerziell erhältlich sind, stellt die Kombination von FISH mit IHC eine kosten- und zeiteffiziente Strategie dar, um die Anzahl der gleichzeitig markierten Ziele zu erhöhen.
Die Präparation von frischem, gefrorenem und fixiertem Gewebe war ein Faktor, der eine erfolgreiche IHC nach dem FISH-Sondentest ermöglichte. Wir testeten IHC mit Antikörpern, die gegen nukleäre, vesikuläre und zytoplasmatische Proteine gerichtet sind, und fanden eine zuverlässige Markierung von membrangebundenen Proteinen (vAChT und Phox2b) auf frisch gefrorenen Proben, aber nicht von zytoplasmatischen Proteinen (TH und GFP). Die Koexpression des Phox2b-Proteins und der mRNA mit der "flockigen" Phox2b-GFP-Markierung bestätigte, dass Neuronen, die das Transkript exprimierten, auch das verwandte Protein exprimierten, was die neurochemische Identität der Neuronen bestätigte (Abbildung 5). Im Gegensatz dazu lieferten fixierte gefrorene Gewebepräparate eine zuverlässige IHC-Markierung unabhängig von der subzellulären Lokalisation des Antigens. Frühere Studien haben gezeigt, dass eine Protease-Vorbehandlung (z. B. Pronase 8,20) eine nachteilige Wirkung auf die IHC haben kann. Der Inhalt der Proteaselösung, die im RNAscope-Protokoll verwendet wird, ist proprietär, und die Permeabilisierung durch Protease wird für den Zugang der RNAscope-Sonde in die Zellen empfohlen. Die Markierung von zytoplasmatischen Proteinen mit den hier beschriebenen Antikörpern wurde bereits an frei schwebenden 30 μm fixierten gefrorenen Maushirnschnitten nachgewiesen 10,21,22. Wir haben 30 μm dicke fixierte Proben auf Objektträger montiert und das FISH-IHC-Protokoll durchgeführt, im Gegensatz zu den vom Hersteller empfohlenen 14 μm dicken frischen Gefrierschnitten. In Abwesenheit von Assay-Modifikationen oder Veränderungen anderer Variablen (Antigen-Retrieval, höhere Antikörperkonzentration, Änderung der Protease) wurde eine zuverlässige IHC an dicken, fixierten Proben mit nachgewiesener Markierung des Zytoplasmas und der axonalen Prozesse zusammen mit der Markierung der FISH-Sonde erreicht (Abbildung 6). Während ähnliche Ansätze von anderen Forschungsgruppen verwendet wurden 7,8,9, erzielte die aktuelle Studie eine kombinierte ISH-IHC an Neuronen und in einem fluoreszierenden Aufbau.
Es gab eine Reihe kritischer Schritte in den Methoden, die es zu beachten galt. Für die frisch gefrorene Zubereitung sollte die Fixierzeit 15 Minuten nicht überschreiten; Längere Fixierungszeiten führten zu einer höheren Hintergrundbeschriftung. Der Proteaseschritt wurde optimiert, da Gewebe unterschiedlicher Dicke und aus verschiedenen Organen unterschiedliche Arten von Protease benötigen, um eine Permeabilisierung zu erreichen. Fixierte gefrorene Abschnitte haften weniger an Glasobjektträgern und lösen sich bei Waschschritten leichter. Daher ist bei der manuellen Handhabung von festsitzenden Gefrierschnitten besondere Vorsicht geboten, um Gewebeverlust oder -beschädigung zu vermeiden.
Obwohl wir festgestellt haben, dass die Kombination von FISH und IHC eine effektive Strategie ist, gehören zu den Nachteilen die Kosten und der technisch anspruchsvolle Assay bei der Kombination der beiden Methoden. Eine Einschränkung der Studie besteht darin, dass kein direkter Vergleich der beiden Gewebepräparationsprotokolle durchgeführt wurde. Außerdem war unsere Auswertung der Ergebnisse durch die Anzahl der Kanäle begrenzt, die das Epifluoreszenzmikroskop aufnehmen konnte. Der Aufbau erlaubte maximal 4 Kanäle gleichzeitig: 346, 488, 550 und 647 nm (Anregungslicht). Wir waren in der Lage, eine Multiplex-Markierung von 5 Zielen zu erreichen, indem wir zwei Proteine mit unterschiedlichen subzellulären Lokalisationen mit demselben Flurophor markierten (Abbildung 4, Tabelle 1). Durch die Verwendung eines konfokalen Mikroskops kann die diskrete Anregung vieler zusätzlicher Fluorophore für die Markierung einzelner Proteine mittels IHC oder für die Abbildung von fluoreszierenden Molekülen, die von Transgenen exprimiert werden, verwendet werden.
Die Kombination von FISH und fluoreszierender IHC kann die Zuverlässigkeit jeder einzelnen Technik für sich genommen verringern. In Zukunft wollen wir die zytoplasmatische Proteinmarkierung auf frischem, gefrorenem Gewebe mit einer Antigen-Retrieval-Behandlung verbessern23. Frühere Studien zeigen, dass die hitzeinduzierte Antigengewinnung die Zugänglichkeit des Proteinepitops24,25,26 erhöht. Durch die Wärmebehandlung werden die Vernetzungen und Methylolgruppen des Proteins gespalten und die Antigene im Gewebe entfaltet, wodurch Epitope freigelegt werden, die sonst unter biologischen Bedingungen in der tertiären Proteinstruktur verborgen wären. Diese Zugänglichkeit könnte den Erfolg der Proteinmarkierung verbessern26,27. Darüber hinaus werden wir verschiedene Epitope desselben zytoplasmatischen Proteins anvisieren, um festzustellen, ob der Erfolg der Protein-Antikörper-Markierung von den verwendeten spezifischen Antikörperklonen abhängt.
Zusammenfassend lässt sich sagen, dass die Kombination von FISH und IHC für die neurochemische Identifizierung heterogener Zellpopulationen im Gehirn, wie z.B. im NTS, nützlich ist. In dieser Studie werden zwei Protokolle vorgestellt, in denen verschiedene Hirnstammgewebepräparate der Maus - frisch gefroren oder fixiert - für die gleichzeitige Multiplex-Fluoreszenzmarkierung von mRNA und Proteinen in situ getestet werden. Beide Protokolle können häufig angewendet werden, um das Expressionsmuster von mRNAs mit geringer Häufigkeit, wie z. B. GalR1, zu detektieren. Dicke (30 μm) fixierte, gefrorene Präparate, die mit Protease permeabilisiert waren, boten im Vergleich zu dünnen (14 μm) frischen gefrorenen Präparaten einen zuverlässigeren Nachweis zytoplasmatischer Proteine und mehr Herausforderungen bei der Handhabung des Gewebes.
Danksagungen
Diese Arbeit wurde durch das Australian Research Council Discovery Project Grant DP180101890 und das Rebecca L Cooper Medical Research Foundation Project Grant PG2018110 finanziert
Materialien
| Name | Company | Catalog Number | Comments |
| ANIMALS | |||
| C57BL/6 mouse | Australian BioResources, Moss Vale | MGI: 2159769 | |
| Phox2b-eGFP mouse | Australian BioResources, Moss Vale | MGI: 5776545 | |
| REAGENTS | |||
| Cyanoacrylate | Loctite | ||
| Ethylene Glycol | Sigma-Aldrich | 324558 | |
| Heparin-Sodium | Clifford Hallam Healthcare | 1070760 | Consult local veterinary supplier or pharmacy. |
| Lethabarb (Sodium Pentabarbitol) Euthanasia Injection | Virbac (Australia) Pty Ltd | N/A | Consult a veterinarian for local pharmaceutical regulations regarding Sodium Pentabarbitol |
| Molecular grade agarose powder | Sigma Aldrich | 5077 | |
| OCT Compound, 118mL | Scigen Ltd | 4586 | |
| Paraformaldehyde, prilled, 95% | Sigma-Aldrich | 441244-1KG | |
| Polyvinylpyrrolidone, average mol wt 40,000 (PVP-40) | Sigma-Aldrich | PVP40 | |
| ProLong Gold Antifade Mountant | Invitrogen | P36930 | With or without DAPI |
| RNAscope Multiplex Fluorescent Reagent Kit (up to 3-plex capability) | Advanced Cell Diagnostics, Inc. (ACD Bio) | ADV320850 | Includes 50x Wash buffer and Protease III |
| RNase Away | Thermo-Fisher Scientific | 7003 | |
| Tris(hydroxymethyl)aminomethane | Sigma-Aldrich | 252859 | |
| Tween-20, for molecular biology | Sigma-Aldrich | P9416 | |
| EQUIPMENT | |||
| Benchtop incubator | Thermoline scientific micro incubator | Model: TEI-13G | |
| Brain Matrix, Mouse, 30g Adult, Coronal, 1mm | Ted Pella | 15050 | |
| Cryostat | Leica | CM1950 | |
| Drawing-up needle (23 inch gauge) | BD | 0288U07 | |
| Hydrophobic Barrier Pen | Vector labs | H-4000 | |
| Kimtech Science Kimwipes Delicate Task Wipes | Kimberley Clark Professional | 34120 | |
| Olympus BX51 | Olympus | BX-51 | |
| Peristaltic pump | Coleparmer Masterflex | L/S Series | |
| Retiga 2000R Digital Camera | QImaging | RET-2000R-F-CLR | colour camera |
| SuperFrost Plus Glass Slides (White) | Thermo-Fisher Scientific | 4951PLUS4 | |
| Vibrating Microtome (Vibratome) | Leica | VT1200S | |
| Whatman qualitative filter paper, Grade 1, 110 mm diameter | Merck | WHA1001110 | |
| SOFTWARES | |||
| CorelDRAW | Corel Corporation | Version 7 | |
| FIJI (ImageJ Distribution) | Open Source/GNU General Public Licence (GPL) | N/A | ImageJ 2.x: Rueden, C. T.; Schindelin, J. & Hiner, M. C. et al. (2017), "ImageJ2: ImageJ for the next generation of scientific image data", BMC Bioinformatics 18:529, PMID 29187165, doi:10.1186/s12859-017-1934-z and Fiji: Schindelin, J.; Arganda-Carreras, I. & Frise, E. et al. (2012), "Fiji: an open-source platform for biological-image analysis", Nature methods 9(7): 676-682, PMID 22743772, doi:10.1038/nmeth.2019 |
| PRIMARY ANTIBODIES | |||
| Anti-Tyrosine Hydroxylase Antibody | Millipore Sigma | AB1542 | Sheep polyclonal (1:1000 dilution), RRID: AB_90755 |
| Anti-Tyrosine Hydroxylase Antibody, clone LNC1 | Millipore Sigma | MAB318 | Mouse monoclonal (1:1000 dilution), RRID: AB_2201528 |
| Anti-Vesicular Acetylcholine Transporter (VAchT) Antibody | Sigma-Aldrich | ABN100 | Goat polyclonal (1:1000 dilution), RRID: AB_2630394 |
| GFP Antibody | Novus Biologicals | NB600-308 | Rabbit polyclonal (1:1000 dilution), RRID: AB_10003058 |
| Phox2b Antibody (B-11) | Santa Cruz Biotechnology | sc-376997 | Mouse monoclonal (1:1000 dilution), RRID: AB_2813765 |
| SECONDARY ANTIBODIES | |||
| Alexa Fluor 488 AffiniPure Donkey Anti-Rabbit IgG (H+L) (min X Bov, Ck, Gt, GP, Sy Hms, Hrs, Hu, Ms, Rat, Shp Sr Prot) | Jackson ImmunoResearch | 711-545-152 | Donkey anti-Rabbit (1:400 dilution), RRID: AB_2313584 |
| AMCA AffiniPure Donkey Anti-Sheep IgG (H+L) (min X Ck, GP, Sy Hms, Hrs, Hu, Ms, Rb, Rat Sr Prot) | Jackson ImmunoResearch | 713-155-147 | Donkey anti-Sheep (1:400 dilution), RRID: AB_AB_2340725 |
| Cy5 AffiniPure Donkey Anti-Goat IgG (H+L) (min X Ck, GP, Sy Hms, Hrs, Hu, Ms, Rb, Rat Sr Prot) | Jackson ImmunoResearch | 705-175-147 | Donkey anti-Goat (1:400 dilution), RRID: AB_2340415 |
| Cy5 AffiniPure Donkey Anti-Mouse IgG (H+L) (min X Bov, Ck, Gt, GP, Sy Hms, Hrs, Hu, Rb, Rat, Shp Sr Prot) | Jackson ImmunoResearch | 715-175-151 | Donkey anti-Mouse (1:400 dilution), RRID: AB_2619678 |
| Cy5 AffiniPure Donkey Anti-Sheep IgG (H+L) (min X Ck, GP, Sy Hms, Hrs, Hu, Ms, Rb, Rat Sr Prot) | Jackson ImmunoResearch | 713-175-147 | Donkey anti-Sheep (1:400 dilution), RRID: AB_2340730 |
| RNASCOPE PROBES | |||
| Galanin Receptor 1 oligonucleotide probe | ACDBio | 448821-C1 | targets bp 482 - 1669 (Genebank ref: NM_008082.2) |
| Glycine transporter 2 oligonucleotide probe | ACDBio | 409741-C3 | targets bp 925 - 2153 (Genebank ref: NM_148931.3) |
| Phox2b oligonucleotide probe | ACDBio | 407861-C2 | targets bp 1617 - 2790 (Genebank ref: NM_008888.3) |
Referenzen
- Wang, F., et al. RNAscope: a novel in situ RNA analysis platform for formalin-fixed, paraffin-embedded tissues. Journal of Molecular Diagnostics. 14 (1), 22-29 (2012).
- Annese, T., et al. RNAscope dual ISH-IHC technology to study angiogenesis in diffuse large B-cell lymphomas. Histochemistry and Cell Biology. 153 (3), 185-192 (2020).
- Morrison, J. A., McKinney, M. C., Kulesa, P. M. Resolving in vivo gene expression during collective cell migration using an integrated RNAscope, immunohistochemistry and tissue clearing method. Mechanisms of Development. 148, 100-106 (2017).
- Gross-Thebing, T., Paksa, A., Raz, E. Simultaneous high-resolution detection of multiple transcripts combined with localization of proteins in whole-mount embryos. BMC Biology. 12, 55(2014).
- Stempel, A. J., Morgans, C. W., Stout, J. T., Appukuttan, B. Simultaneous visualization and cell-specific confirmation of RNA and protein in the mouse retina. Molecular Vision. 20, 1366-1373 (2014).
- Kersigo, J., et al. A RNAscope whole mount approach that can be combined with immunofluorescence to quantify differential distribution of mRNA. Cell and Tissue Research. 374 (2), 251-262 (2018).
- Grabinski, T. M., Kneynsberg, A., Manfredsson, F. P., Kanaan, N. M. A method for combining RNAscope in situ hybridization with immunohistochemistry in thick free-floating brain sections and primary neuronal cultures. PLoS One. 10 (3), 0120120(2015).
- Baleriola, J., Jean, Y., Troy, C., Hengst, U. Detection of axonally localized mRNAs in brain sections using high-resolution in situ hybridization. Journal of Visualized Experiments. (100), e52799(2015).
- Fe Lanfranco, M., Loane, D. J., Mocchetti, I., Burns, M. P., Villapol, S. Combination of fluorescent in situ hybridization (FISH) and immunofluorescence imaging for detection of cytokine expression in microglia/macrophage cells. Bio-Protocol. 7 (22), (2017).
- Dereli, A. S., Yaseen, Z., Carrive, P., Kumar, N. N. Adaptation of respiratory-related brain regions to long-term hypercapnia: focus on neuropeptides in the RTN. Frontiers in Neuroscience. 13, 1343(2019).
- Lazarenko, R. M., et al. Acid sensitivity and ultrastructure of the retrotrapezoid nucleus in Phox2b-EGFP transgenic mice. Journal of Comparative Neurology. 517 (1), 69-86 (2009).
- Gage, G. J., Kipke, D. R., Shain, W. Whole animal perfusion fixation for rodents. Journal of Visualized Experiments. (65), e3564(2012).
- Paxinos, G., Franklin, K. B. The mouse brain in stereotaxic coordinates. , Academic Press. (2004).
- Abercrombie, M. Estimation of nuclear population from microtome sections. Anatomical Records. 94, 239-247 (1946).
- Kerr, N., et al. The generation of knock-in mice expressing fluorescently tagged galanin receptors 1 and 2. Molecular and Cellular Neurosciences. 68, 258-271 (2015).
- Kachidian, P., Pickel, V. M. Localization of tyrosine hydroxylase in neuronal targets and efferents of the area postrema in the nucleus tractus solitarii of the rat. Journal of Comparative Neurology. 329 (3), 337-353 (1993).
- Stornetta, R. L., et al. Expression of Phox2b by brainstem neurons involved in chemosensory integration in the adult rat. Journal of Neuroscience. 26 (40), 10305-10314 (2006).
- Gilmor, M. L., et al. Expression of the putative vesicular acetylcholine transporter in rat brain and localization in cholinergic synaptic vesicles. Journal of Neuroscience. 16 (7), 2179-2190 (1996).
- Fisher, J. M., Sossin, W., Newcomb, R., Scheller, R. H. Multiple neuropeptides derived from a common precursor are differentially packaged and transported. Cell. 54 (6), 813-822 (1988).
- Towle, A. C., Lauder, J. M., Joh, T. H. Optimization of tyrosine-hydroxylase immunocytochemistry in paraffin sections using pretreatment with proteolytic-enzymes. Journal of Histochemistry and Cytochemistry. 32 (7), 766-770 (1984).
- Biancardi, V., et al. Mapping of the excitatory, inhibitory, and modulatory afferent projections to the anatomically defined active expiratory oscillator in adult male rats. Journal of Comparative Neurology. 529 (4), 853-884 (2021).
- Matthews, D. W., et al. Feedback in the brainstem: an excitatory disynaptic pathway for control of whisking. Journal of Comparative Neurology. 523 (6), 921-942 (2015).
- Ramos-Vara, J. A. Principles and methods of immunohistochemistry. Methods in Molecular Biology. 1641, 115-128 (2017).
- Shi, S. R., Key, M. E., Kalra, K. L. Antigen retrieval in formalin-fixed, paraffin-embedded tissues: an enhancement method for immunohistochemical staining based on microwave oven heating of tissue sections. Journal of Histochemistry and Cytochemistry. 39 (6), 741-748 (1991).
- Yamashita, S., Katsumata, O. Heat-induced antigen retrieval in immunohistochemistry: mechanisms and applications. Methods in Molecular Biology. 1560, 147-161 (2017).
- Yamashita, S., Okada, Y. Mechanisms of heat-induced antigen retrieval: analyses in vitro employing SDS-PAGE and immunohistochemistry. Journal of Histochemistry and Cytochemistry. 53 (1), 13-21 (2005).
- Yamashita, S. Heat-induced antigen retrieval: mechanisms and application to histochemistry. Progress in Histochemistry and Cytochemistry. 41 (3), 141-200 (2007).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten