Method Article
Investigando a Motilidade Impulsionada por Flagella em Escherichia coli aplicando três técnicas estabelecidas em uma série
Neste Artigo
Resumo
Muitas bactérias usam motilidade impulsionada por flagela para navegar em seu ambiente e colonizar ambientes favoráveis tanto individualmente quanto como coletivos. Demonstrado aqui é o uso de três métodos estabelecidos que exploram a motilidade como ferramenta de seleção para identificar componentes/caminhos que contribuem para a natação e a motilidade.
Resumo
A motilidade é crucial para a sobrevivência e o sucesso de muitas espécies bacterianas. Existem muitas metodologias para explorar a motilidade para entender caminhos de sinalização, para elucidar a função e montagem de partes flageladoras, e para examinar e entender padrões de movimento. Aqui demonstramos uma combinação de três dessas metodologias. A motilidade no ágar macio é a mais antiga, oferecendo uma forte seleção para isolar mutações supressoras de ganho de função em cepas prejudicadas pela motilidade, onde a motilidade é restaurada através de uma segunda mutação. A técnica de amarração celular, primeiramente empregada para demonstrar a natureza rotativa do motor flagelador, pode ser usada para avaliar o impacto dos efeitos de sinalização na velocidade do motor e sua capacidade de alternar a direção rotacional. O ensaio de "travessia de fronteiras" é mais recente, onde bactérias nadando podem ser preparadas para a transição para se mover coletivamente como um enxame. Em combinação, esses protocolos representam uma abordagem sistemática e poderosa para identificar componentes da maquinaria de motilidade, e para caracterizar seu papel em diferentes facetas da natação e do enxame. Eles podem ser facilmente adaptados para estudar motilidade em outras espécies bacterianas.
Introdução
As bactérias empregam muitos apêndices para movimentação e dispersão em seus nichos ecológicos1. A motilidade conduzida por Flagella é a mais rápida delas, promovendo a colonização de locais favoráveis em resposta aos sinais ambientais, contribuindo significativamente para a capacidade patogênica de algumas espécies2,3. Bactérias flageladas podem nadar individualmente em líquido a granel, ou enxame como um coletivo sobre uma superfície semissólida4. A flagela extracelular se prende e é conduzida por motores rotativos embutidos na membrana, que aproveitam a potência dos gradientes de íons para gerar torque que causa rotação1,2,4,5,6,7,8. Em E. coli, cujos motores funcionam em um torque constante9, a saída do motor pode ser categorizada em termos de velocidade rotacional e comutação do rotor entre as direções no sentido anti-horário (CCW) e sentido horário (CW). A rotação CCW promove a formação de um feixe de flagelar coerente que impulsiona a célula para a frente (corrida), enquanto um interruptor transitório na direção rotacional (CW) faz com que o feixe se desmonte parcial ou totalmente10, e a célula reoriente sua direção de natação (tombo). E. coli normalmente corre por um segundo e cai por um décimo de segundo. A frequência de comutação do rotor ou do "viés de queda" é controlada pelo sistema de sinalização de quimiotaxis, onde os quimoreceptores transmembrano detectam sinais químicos externos e transmitem-nos via fosfora ao motor flagelador para estender as corridas em resposta aos atrativos, ou suprimi-los em resposta a produtos químicos tóxicos11,12. A motilidade da natação é avaliada em 0,3% de ágar macio.
Durante o enxame, as bactérias navegam em uma superfície semissólida como um coletivo denso, onde pacotes de bactérias fluem em um movimento contínuo de redemoinho2,13,14,15. Os enxames de E. coli apresentam fisiologia quimioensory alterada (viés de queda mais baixo), maiores velocidades e maior tolerância a antimicrobianos sobre células que nadam em líquido a granel16,17. Os enxames variam em sua implantação de uma infinidade de estratégias que auxiliam o movimento, incluindo produção surfactante, hiperflagellação e alongamento celular2. O swarming oferece às bactérias uma vantagem competitiva nos ambientes ecológicos e clínicos18,19,20. Existem duas categorias de bactérias em enxame: enxames temperados, que só podem enxamear na mídia solidificada com 0,5-0,8% de ágar, e enxames robustos, que podem navegar através de concentrações de ágar mais altas21.
Existem uma variedade de ensaios para interrogar a motilidade da natação e sua regulamentação. Quando prejudicada por mutações ou condições ambientais, a própria motilidade oferece uma forte seleção para identificar mutações supressoras de ganho de função. Esses supressores podem ser revertentes genuínos da mutação original, ou pseudo-revertentes, onde uma segunda mutação restaura a funcionalidade. Tais mutantes podem ser identificados por sequenciamento de genomas inteiros (WGS). Uma alternativa à seleção de supressores imparcial é uma estratégia de mutagênese direcionada tendenciosa (por exemplo, mutagênese PCR). Essas metodologias muitas vezes lançam luz sobre a função ou regulação ambiental do aparelho de motilidade. Se o objetivo é estudar a função motora, então a restauração da motilidade do tipo selvagem medida em ágar macio pode não necessariamente indicar a restauração da saída do motor do tipo selvagem. O ensaio de amarração celular, no qual as células são anexadas a uma superfície de vidro por um único flagellum e a rotação do corpo celular é posteriormente monitorada, pode ser o ensaio inicial de escolha para avaliar o comportamento motor. Embora metodologias mais sofisticadas estejam agora disponíveis para monitorar as propriedades do motor, a configuração necessária da câmera de alta velocidade e a aplicação de pacotes de software para análise de movimento limitam seu uso generalizado22,23,24,25. O ensaio de amarração da célula requer apenas que a flagela seja amarrada, permitindo a fixação dos filamentos curtos a um slide de vidro, seguido de filmagem da rotação do corpo celular. Embora as velocidades motoras registradas sejam baixas neste ensaio devido à alta carga que o corpo celular exerce sobre o flagelo, este ensaio contribuiu, no entanto, para insights valiosos sobre as respostas chemotactic26,27,28,29, e continua sendo uma ferramenta investigativa válida como discutido abaixo.
A motilidade repleta representa um conjunto diferente de desafios para os pesquisadores. A seleção de supressores de ganho de função só funciona em enxames que produzem surfactantes abundantes e enxames prontamente13. Não-produtores surfactantes como o E. coli são exigentes com relação à escolha do ágar, composição da mídia e umidade do ambiente2,13,14,21. Uma vez estabelecidas as condições de enxame, o ensaio de travessia de fronteiras17 é uma metodologia útil para interrogar a capacidade de um enxame de navegar em novas/condições duras. Embora os protocolos apresentados abaixo se relacionem com E. coli,eles podem ser prontamente adaptados para aplicação em outras espécies.
Protocolo
1. Isolamento de mutantes supressores em cepas deficientes de motilidade
NOTA: Use este método como um amplo "catch-all" para identificar a natureza geral do defeito de motilidade.
- Preparação de placas de ágar macio
NOTA: O ágar macio, também chamado de ágar de motilidade ou natação, é um ágar de baixa porcentagem (~0,2-0,35% w/v), muito usado para avaliarquimiotaxis 31,32.- Adicione 3 g de bacto-agar (0,3% w/v) e 20 g de LB a um frasco fundo redondo de 2 L. Adicione 1 L de ddH2O (água duplamente destilada) ao frasco e misture uniformemente a suspensão usando uma haste de mexida e placa de agitação magnética.
- Autoclave por 20 min a 121 °C.
- Deixe esfriar com agitação suave usando a haste/placa como acima. Quando a temperatura atingir aproximadamente 50 °C, despeje 25 mL em placas estéreis de Petri (100 mm x 15 mm), e permita que o ágar derretido coloque a tampa no lugar por pelo menos 1h, para uso dentro de 16 h.
- Preparação cultural, inoculação e isolamento de mutantes supressores
NOTA: E. coli inoculado no centro de mídia rica em nutrientes solidificada com ágar macio consome nutrientes localmente, criando um gradiente de nutrientes que eles seguem. À medida que se movem para fora, aparecem 'anéis' definidos(Figura 1A), que estão relacionados a quimioattractants específicos a que as bactérias respondem. Defeitos no sistema de quimiotaxis ou componentes estruturais do motor flagela podem comprometer o desempenho neste ensaio. Muitas vezes, mutantes com uma vantagem de motilidade surgem durante a exibição, e podem ser vistos emergindo de um único ou de vários pontos ao longo da periferia do anel, de onde eles 'flare' para fora(Figura 1C). Nota-se que a borda mais externa da frente de natação contrasta prontamente contra o ágar macio virgem não colonizado.- Cresça culturas noturnas da variedade deficiente de motilidade desejada em 5 mL de Caldo de Lennox (LB; triptona de 10 g/L, extrato de levedura de 5 g/L e 5 g/L NaCl, Tabela de Materiais) a 30 °C com agitação horizontal (220 r.p.m.). No dia seguinte, subcultura (diluição de 1:100) em LB fresco, crescendo sob as mesmas condições para fase exponencial (OD600 de 0,6).
- Inocular 6 μL da cultura no centro de uma placa de ágar macio (1.1) usando uma pipeta, empurrando a ponta estéril carregada para dentro do ágar para expulsar suavemente o conteúdo. Transfira para 30 °C e incubar(Figura 1B),até que as "erupções" de motilidade sejam evidentes, emanando do ponto de inoculação ou da periferia dos anéis de motilidade, tipicamente em 24-36 h(Figura 1C).
NOTA: Nos ensaios de motilidade, inocular uma cepa do tipo selvagem ao lado dos isolados mutantes para comparação. Esta cepa de tipo selvagem mostrará os anéis quimotacticos concêntricos característicos(Figura 1A) e encherá a placa dentro de 8-10 h. - Use um laço de arame estéril para levantar as células da região 'flare' e listras para purificar colônias únicas em uma placa de ágar duro LB (LB preparado como acima, solidificado com 15 g/L bacto-ágar).
- Escolha colônias únicas da placa de raia usando um laço de arame estéril e re purifique, redimensionando-se para colônias únicas para garantir o isolamento de uma colônia "pura".
- Confirmação e caracterização de mutantes supressores
- Confirme que os mutantes isolados do supressor restauraram a motilidade. Prepare placas de ágar macio (1.1) e culturas para cepas de interesse (como em 1.2.1), incluindo o tipo selvagem e a tensão inicial "deficiente de motilidade" para comparação.
- Inocular as placas (como em 1.2.2) e incubar a 30 °C por 8-10 h.
- Regissuir o diâmetro do anel mais externo (borda do círculo) e compare para estabelecer qual dos isolados restaurou substancialmente a motilidade.
NOTA: Recomenda-se que as placas sejam fotografadas durante todo o período de tempo do experimento. Para obter melhores resultados, use um dispositivo "balde de luz"30,onde uma câmera digital é montada acima de uma fonte de luz para melhor iluminação para medir o diâmetro da colônia de natação e distingui-lo do ágar não colonizado. - O sujeito verificou isolados para WGS conforme necessário, permitindo uma "cobertura de sequência" suficiente para identificar positivamente as mutações que restauraram a função do tipo selvagem.
2. Quantificar o comportamento motor de flagela através da amarração celular
NOTA: Use este método quando o comportamento normal de queda de corrida (quimiotaxis) parece estar comprometido.
-
Preparação cultural e flagela tesoura
- Prepare uma cultura de fase exponencial da tensão de interesse, conforme descrito na etapa 1.2.1.
- Pelota 10 mL de células por centrifugação a 2.000 x g por 3 minutos antes de reutilizar em 10 mL de tampão de motilidade esterilizado por filtro (MB; Tampão fosfato de potássio de 10 mM [0,0935 M K2HPO4, 0,0065 M KH2PO4, pH 7.0], 0,1 mM EDTA [pH 7.0], 10 mM NaCl, 75 mM KCl).
NOTA: MB suporta motilidade, mas não suporta crescimento bacteriano - Repita o passo 2.1.2 mais duas vezes antes de reutilizar a pelota final em 1 mL de MB.
- Transfira a suspensão da célula para uma seringa de 1 mL e conecte uma agulha 23G até o final. Monte um aparelho de seringa/agulha idêntico e conecte os dois juntos através de 6 polegadas de tubos de polietileno (diâmetro interno de 0,58 mm) firmemente em baia sobre cada ponta de agulha.
- Tesoura a flagela (eles são frágeis e quebram facilmente) passando suavemente a suspensão da célula para frente e para trás de uma seringa para a outra 50x, com 1 min pausas entre cada 10 passes.
- Centrifugar as células desarmadas a 2.000 x g por 3 min e resuspend em um volume final de 500 μL de MB.
-
Preparação de slides e amarração celular
- Prepare uma câmara de fixação celular empilhando uma tampa de 18 mm x 18 mm sobre um slide de microscópio de vidro de 3 polegadas x 1 polegada x 1 mm, separado por fita dupla face(Figura S1).
- Lave a câmara com solução de poli-lise de poli-lise de 0,01% (w/v) aplicando-se ao topo da câmara. Incline a borda inferior (a mancha de cobertura alinhada com o slide do microscópio) em um limpador de tarefas (papel de tecido) para ajudar a desenhar a solução através da câmara(Figura S1). Em seguida, incubar em temperatura ambiente por 10 minutos.
- Lave a câmara três vezes com 40 μL de MB usando como descrito na etapa 2.2.2
- Adicione 40 μL da suspensão da célula de corte (preparada acima) ao topo da câmara e incubar à temperatura ambiente por 10 minutos para permitir que as células se conectem ao deslizamento de tampas.
- Lave suavemente a câmara com 40 μL de MB como em 2.2.3 para remover células nãoectadas.
-
Gravação e quantificação de rotação celular
- Transfira o slide do microscópio carregado com células amarradas para o estágio do microscópio.
- Utilizando microscopia de contraste de fase e um objetivo de 100x, escaneie a população em busca de células fixadas no lugar e gire em um único eixo, ou seja, rotações suaves em um ponto fixo em vez de apresentar em um ângulo onde a célula se move dentro e fora de foco(Vídeo 1).
- Use um microscópio comercial e uma câmera associada. Abra o software associado, garanta que as células de interesse estejam em foco e clique na aquisição de vídeo para gravar a rotação do celular por um minuto (a 10 quadros por segundo ou superior).
- A partir da reprodução de vídeo, quantifique o número de rotações completas por minuto e o número de vezes que a célula muda de direção (frequência de comutação).
NOTA: As velocidades de rotação e a frequência de comutação podem ser muito rápidas para medir olho por olho, por isso é recomendado usar software de vídeo que ofereça reprodução lenta/fina, ou adote um sistema de software automatizado para quantificar padrões de rotação33. Uma alternativa seria aumentar a viscosidade do MB usando celulose de metila (ou agente similar) para ajudar a retardar e resolver a rotação de bactérias mais rápidas ou compensar quando câmeras com esquadragens baixas estão em uso. - Repita o passo 2.3.2 com réplicas biológicas para compilar uma representação da população de interesse.
3. Preparação de enxames em um ensaio de travessia de fronteiras
NOTA: Use este método para avaliar o impacto de uma mutação ou condição na motilidade do grupo. Swarm-agar refere-se ao ágar onde a porcentagem é tipicamente maior do que a do ágar macio. Em ágar macio (0,3%), as células nadam individualmente dentro do ágar. No ágar enxame (0,5% ou mais), as células se movem como um grupo na superfície. Enquanto as placas de enxame devem ser usadas como detalhadas aqui, as placas de natação têm uma vida útil mais longa, e podem ser usadas por vários dias. Nossa preferência pessoal é usar em 1-2 dias.
-
Preparação do enxame ágar
- Adicione 5 g de agar Eiken (0,5% c/v) e 20 g de LB a um frasco fundo redondo de 2 L. Adicione 1 L de ddH2O ao frasco e misture uniformemente a suspensão usando uma haste de mexida e placa de agitação magnética.
- Autoclave por 20 min a 121 °C.
- Deixe esfriar com agitação suave para evitar quaisquer bolhas de ar usando a haste/placa como acima. Quando aproximadamente 50 °C, adicione glicose esterilizada por filtro para uma concentração final de 0,5 %.
- Despeje 25 mL em placas de Petri estéreis (100 mm x 15 mm) e deixe fixar em temperatura ambiente por pelo menos 14 h e não mais do que 20 h. Não guarde para uso futuro.
-
Inoculação e incubação de placas de enxame
- Inocular 6 μL de uma cultura exponencial média (preparada como em 1,2) ao detectar em cima do ágar.
- Deixe a tampa desligada por 5-10 min e substitua quando o inóculo secar na superfície do ágar.
- Incubar a 30 °C por 8 h. Evite a tentação de inspecionar o progresso do enxame removendo a tampa, pois isso contribuirá para a secagem do ágar e prejudicará o enxame.
NOTA: O tempo de incubação pode variar dependendo do fenótipo da cepa. Algumas mutações isoladas podem dificultar a capacidade de enxame e exigirão uma porcentagem reduzida de ágar ou um período mais prolongado de incubação.
-
Elaboração de placas de ensaio de travessia de fronteira
NOTA: Este ensaio utiliza uma placa de Petri modificada, onde uma divisão de plástico (borda) cria duas câmaras, em vez de uma(Figura 2A). Cada câmara pode ser preparada independentemente da outra, oferecendo condições diferentes para enxame, antes de "conectar" as duas. Dependendo do design experimental, a primeira câmara (designada à esquerda) pode ser preparada com ágar nadador (0,3% c/v) ou ágar enxame (0,5% c/v) de onde as bactérias podem migrar através da fronteira para a câmara direita contendo ágar enxame +/- qualquer suplemento ou desafio necessário (por exemplo, antibióticos). A migração em ambos os ágars é tipicamente medida/comparada registrando o diâmetro mais amplo da colonização bacteriana (borda a borda) do ponto original da inoculação.- Prepare o ágar de natação, conforme descrito em 1.1, se necessário.
- Prepare o ágar do enxame, como descrito em 3.1.
- Despeje ~30 mL de ágar enxame (com suplementação desejada, se necessário) na câmara direita de uma placa de Petri de compartimento duplo (100 mm x 15 mm), ao ponto em que está nivelado com o divisor de plástico entre as câmaras, mas não transbordando para a esquerda(Figura 2B).
- Depois que o ágar endurecer, encha a câmara esquerda com ~30 mL de natação ou ágar enxame, novamente ao ponto de contato com a divisão de plástico(Figura 2C). Antes de definir, use uma ponta de pipeta estéril para arrastar suavemente o ágar sobre a borda para conectar os dois lados com uma ponte de ágar de ~1 mm de altura que abrange toda a extensão da divisão(Figura 2D).
- Deixe a placa secar à temperatura ambiente (3.1).
NOTA: Um método alternativo de criação de ponte é permitir que o ágar da câmara esquerda seque e, em seguida, lentamente pipeta ~100 μL de ágar enxame derretido ao longo do divisor de plástico para fazer a ponte das duas câmaras (3.4). Inocular as placas na câmara esquerda como detalhada acima para natação (1.2.2) ou enxame (3.2) ágar, antes de incubar a 30 °C por 12-16 h, ou até que enxames tenham feito progresso suficiente sobre a câmara direita para permitir comparações entre cepas de interesse.
Resultados
O isolamento de pseudo-revertentes em uma cepa de E. coli cuja motilidade é prejudicada por altos níveis da molécula de sinalização c-di-GMP, foi detalhado em trabalhos recentes do nosso laboratório34. Esta cepa (JP1442) abrigava duas mutações: ΔyhjH e ΔycgR. YhjH é o fosfodiesterase mais ativo que degrada c-di-GMP em E. coli. A ausência de YhjH leva a níveis elevados de c-di-GMP e inibição da motilidade. YcgR é um efeito c-di-GMP. Em complexo com c-di-GMP, YcgR liga-se ao rotor flagelar para induzir pela primeira vez a rotação do motor CCW e, posteriormente, diminuir a velocidade do motor. Os ensaios de tethering celular e contas mostraram que o comportamento motor retornou ao normal no mutante duplo, mas a motilidade no ágar macio não34. Então, implantamos o passo 1 do protocolo para isolar sinalizadores pseudo-reversantes no mutante duplo(Figura 1C). A maioria das mutações mapeadas pelo WGS (plataforma HiSeq 4000, configuração PE 2 x 15034) para rssB, que codifica para um regulador de resposta/proteína adaptadora que normalmente direciona o ClpXP protease para atingir σS para degradação34. Um desses revertentes, que exibia motilidade próxima ao tipo selvagem (AW405, compare a Figura 1A,D),foi usado para gerar resultados representativos para as etapas 2 e 3 da seção de protocolo, utilizando como controles tanto seu pai mutante duplo (Figura 1B) quanto a cepa isogênica do tipo selvagem(Figura 1A).
Para a etapa 2 da seção de protocolo, foram analisadas capturas de vídeo para calcular rotações por minuto (cada rotação completa de 360°) e CWBias (a fração de motores de tempo giram em direção CW, ou viés de queda). O ΔyhjH mostrou menos rotações por minuto e um viés CW mais baixo em comparação com o tipo selvagem, como esperado(Figura 3). Tanto o ΔyhjH Δ ycgR duplo mutante quanto seu supressor mostraram comportamento motor semelhante ao tipo selvagem, observações apoiadas por uma análise prévia usando o ensaio 'contas' de maior resolução detalhado na introdução acima no trabalho anterior34.
Para o passo 3 da seção de protocolo, o ensaio de travessia de fronteira (Figura 2) foi usado para comparar as habilidades do tipo selvagem e do isolador supressor, primeiro para enxame, e depois para atravessar a fronteira e enxame em ágar complementado com kanamicina. Os resultados mostram que ambas as cepas chegaram à fronteira em um momento semelhante (dados não mostrados) indicando taxas semelhantes de enxame de um ponto de inoculação idêntico. No entanto, a cruz do enxame para a câmara direita (antibiótico) foi marginalmente, mas consistentemente maior para o tipo selvagem do que o supressor a 20 μg/mL kanamycin(Figura 4). A diferença entre as duas cepas foi mais acentuada em 40 μg/mL kanamycin. Juntos, esses dados sugerem que as mutações no rssB que restauraram a motilidade em placas de ágar macio(Figura 1D),impactam negativamente a resistência a antibióticos da cepa supressora durante o enxame(Figura 4).
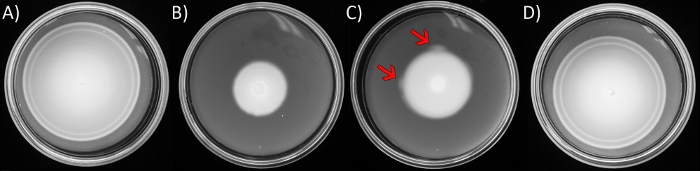
Figura 1: Ensaios de motilidade de ágar macio e surgimento de sinalizadores supressores.
As placas contêm LB solidificado com 0,3 % de ágar. As cepas de E. coli foram inoculadas no centro de cada placa e incubadas a 30°C por 8h, com exceção de C, que foi incubada por 16 h.(A) E. coli (AW405). (B) Variante deficiente de motilidade ∆yhjH ∆ycgR (JP1442). (C) Como em B, exceto tempos mais longos de incubação. As setas apontam para "sinalizadores" em movimento mais rápido emergindo no anel periférico da colônia de natação em expansão. (D) Um supressor isolado de um sinalizador em C. Clique aqui para ver uma versão maior desta figura.
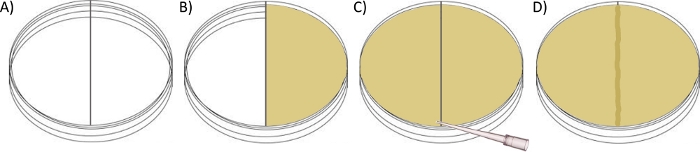
Figura 2: Esquema para a criação de um ensaio de placa de travessia de fronteira.
(A) Despeje ~30 mL de ágar enxame (com antibiótico desejado) na câmara direita de uma placa de petri dividida até nivelar com o divisor plástico e deixe definir com tampa fechada. (B) Encha a câmara esquerda com ~30 mL de natação ou ágar enxame até o ponto de contato com a parte superior do divisor plástico. (C) Use uma ponta de pipeta estéril para arrastar suavemente o ágar enxame derretido sobre a borda, conectando assim os dois lados com uma ponte de ágar de ~1 mm de altura e deixe definir com tampa fechada. (D) Deixe a placa secar ainda mais à temperatura ambiente durante a noite antes de vacinar a câmara esquerda com a cepa desejada e incubar a 30 °C. Clique aqui para ver uma versão maior desta figura.
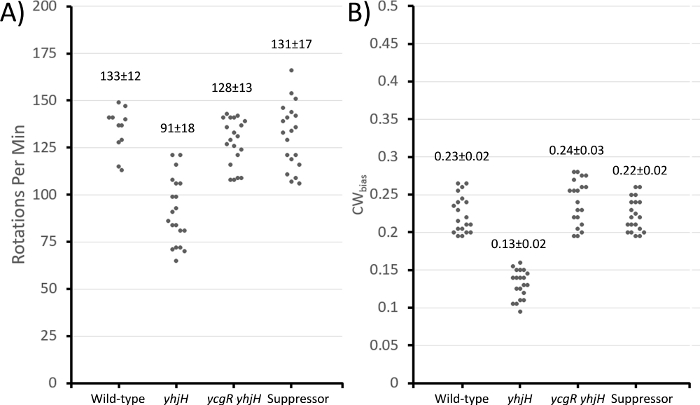
Figura 3: Propriedades motoras de várias cepas medidas pela técnica de amarração celular.
Tipo selvagem (AW405), ∆yhjH (VN133), ∆ycgR ∆yhjH (JP1442), e seu supressor (JP1836) foram cultivados em LB a 30 °C a fase exponencial média antes do amarração. (A) Rotações por minuto (completado 360° voltas) e (B)viés CW (fração de motores de tempo giram em uma direção CW). Desvio padrão da média (±). Foram observadas 20 células amarradas por 60 segundos em cada cepa. Clique aqui para ver uma versão maior desta figura.
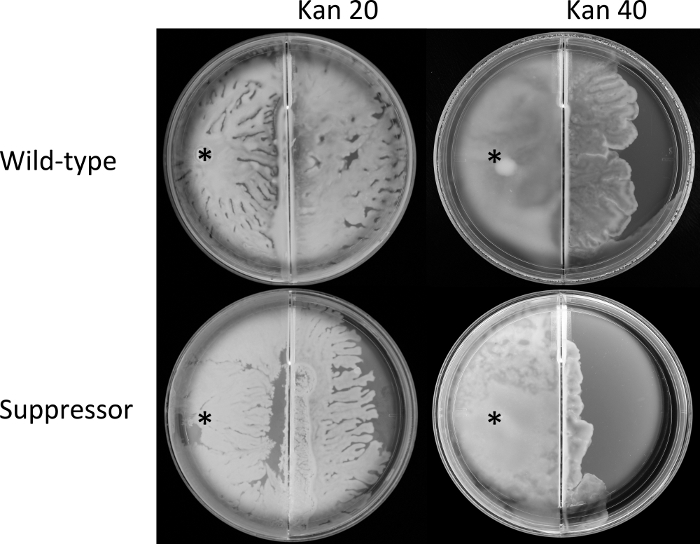
Figura 4: Ensaios de travessia de fronteiras.
Culturas de fase mid-exponencial do tipo selvagem E. coli (AW405) e do mutante supressor (JP1836) foram inoculadas na posição indicada (*) no compartimento esquerdo da placa dividida contendo mídia de enxame, e incubadas a 30 °C. Eles chegaram à fronteira em momentos comparáveis. As placas foram incubadas por mais 6h, durante as quais o enxame atravessou para a câmara direita, na qual a mídia foi complementada com kanamicina (Kan; números indicam μg/mL). As placas são representativas de três réplicas biológicas cada uma realizadas em triplicado. Clique aqui para ver uma versão maior desta figura.
Figura suplementar 1: Preparação de um deslizamento de câmara para amarração celular. (A) Deite duas peças de fita dupla face antes(B) usando uma lâmina de barbear para cortar o excesso. (C) Retire a camada superior para expor o adesivo antes (D) afixando um deslizamento de tampa e pressionando-o suavemente na posição (indicado por [ ), garantindo que todo o ar seja empurrado para fora da interface entre o deslizamento de tampa e a fita abaixo. (E) Amostra de carga (mostrada aqui com corante de carregamento de DNA [30% v/v glicerol, 0,25% w/v azul bromofenol, e 0,25% w/v xileno cyanol] adicionado para ajudar a visualização) na parte superior do canal criado (seta) enquanto(F) angling em limpo, limpeza de tarefas teciduais para ajudar a desenhar a solução através da câmara como o tecido absorve o líquido (seta) no canal e puxa-lo através. Clique aqui para baixar este número.
Vídeo 1: Rotação de células E. coli amarradas. Clique aqui para baixar este vídeo.
Vídeo 2: Um enxame ativo de E. coli filmado sob ampliação de 60x, demonstrando seu movimento característico girando atrás da borda da frente em movimento. Clique aqui para baixar este vídeo.
Discussão
O isolamento e a caracterização das mutações supressoras contribuíram com sucesso para identificar componentes-chave do sistema de quimioterapia35,36,37, bem como da própria máquina motora38,39,40. Ao usar o Protocolo 1, é importante incluir múltiplas réplicas independentes para garantir o isolamento de um grande espectro de possíveis mutações que poderiam compensar a perda de motilidade. Aumentar o número de bactérias, espalhando a cultura em uma linha em vez de um ponto, pode melhorar as chances de gerar supressores41. O isolamento da mesma mutação (determinada pelo sequenciamento do DNA) aumenta várias vezes a confiança em sua autenticidade. O WGS invariavelmente revelará a presença de outras mutações no genoma. Por isso, é importante verificar os resultados transduzindo (sempre que possível) a mutação identificada de volta ao fundo original deficiente de motilidade. A abordagem mutante supressora está enraizada na restauração da função através de uma mutação secundária, de modo que uma limitação deste método é que se um gene estrutural crítico for excluído, ou seja, um que sustenta toda a via ou estrutura, pode não haver margem para compensação. Apesar de ser um método antigo, nosso recente trabalho34 demonstra sua utilidade contínua na elucidação de novos caminhos que contribuem para a motilidade bacteriana.
Para a quantificação da saída do motor, a abordagem de amarração celular permanece uma ferramenta universalmente acessível que requer apenas um microscópio com um acessório de câmera. O tethering celular já foi usado em um número diversificado de espécies bacterianas, incluindo Salmonella42, Pseudomonas43, Streptococcus44e Rhodobacter33. O sucesso do protocolo depende em grande parte da adequada tesoura e apego das células. A tesoura muito agressiva ou omitindo a pausa entre tesouras (2.1.5) tende a promover o enlatado inconsistente ou incompleto de filamentos, resultando em células não motil ou células amarradas em um eixo distorcido. A relevância duradoura deste protocolo permanece, apesar da adoção do ensaio de contas de maior resolução por muitos grupos de pesquisa (incluindo o nosso). A limitação primária do ensaio de contas vem da necessidade de a conta aderir ao filamento das bactérias de interesse. Essa técnica tem se beneficiado muito de estudos em E. coli que identificaram um alelo flagellin 'pegajoso', o que facilita a adesão45. A variante pegajosa também é superior no ensaio de amarração celular. Tal variante ainda não está disponível para a maioria das bactérias flageladas. A situação é ainda mais complicada com alguns organismos possuindo múltiplas proteínas flagellin46, e no caso de Vibrio sp., também possuindo uma baia membranous47. A amarração celular também pode ser realizada usando um anticorpo antiflagelante específico da espécie ou um anticorpo para uma etiqueta de epítope projetada.
Embora as bactérias possam nadar imediatamente após a introdução em um meio líquido, isso não é verdade para o enxame, onde as células devem ser primeiro preparadas para um estado de enxame. O contato superficial desencadeia uma mudança fisiológica necessária para que as células iniciem o enxame48,49,50,51, resultando em uma fase de defasagem e um acúmulo de alta densidade celular. As alterações fisiológicas incluem a remodelação do sistema de quimotáxi em E. coli16, e adaptações como alongamento celular e/ou hiperflagellação para outras bactérias13,14,21. Dadas as mudanças fisiológicas necessárias para iniciar o enxame, fazemos uma nota de advertência sobre estudos que tentaram imitar facetas selecionadas de enxames - como alta densidade ou aumento do comprimento celular - simplesmente concentrando culturas de células planctônicas para aumentar a densidade e/ou induzir o alongamento celular pelo uso de antibióticos que inibem a divisão celular52,53. Se usar o alongamento celular como marcador, alertamos também que, em comparação com as células planctônicas, há apenas um aumento marginal no comprimento médio das células swarmer em Salmonella ou E. coli54. Ensaios de enxame são mais difíceis de estabelecer do que ensaios de natação. As variáveis incluem a fonte comercial do ágar usado para solidificar a mídia (o ágar eiken especial é essencial para o enxame em E. coli, enquanto o ágar Difco mais padrão suporta enxame para todas as outras bactérias), uso de mídia rica versus mínima(E. coli e Salmonella requerem suplementação de glicose), e mais criticamente umidade ambiente2,13,14,21. Destes, manter a umidade ideal pode ser o mais frustrante. [Uma excelente, metódica, otimização para enxame em um organismo de escolha (Pseudomonas aeruginosa) foi demonstrada por Morales-Soto e colegas de trabalho55.] A mídia enxame deve ser suficientemente úmida para promover o enxame, mas não tão úmido a fim de permitir a propagação passiva/deslizamento, o que pode ser facilmente confundido com o enxame ativo56. Portanto, é fundamental que enxames sejam verificados sob um microscópio para confirmar os padrões distintos de movimento associados a essa motilidade coletiva(Vídeo 2). A temperatura também é uma consideração importante para otimizar o ensaio de enxame. Temperaturas mais altas, por exemplo, 37 °C, secarão as placas mais cedo do que a 30 °C. O uso de uma incubadora com controle de umidade (~70-80%) pode ajudar a mitigar esses problemas, incluindo mudanças sazonais que podem afetar as temperaturas internas de construção e a umidade. Uma vez estabelecido com sucesso, o protocolo 3 fornece uma maneira poderosa de investigar um dos aspectos mais interessantes das bactérias em enxame, resistência elevada aos antibióticos17. Todos os protocolos descritos aqui podem ser aplicados a novos organismos para identificar caminhos que especificam e controlam a motilidade mediada pela flagela.
Divulgações
Os autores não têm nada a revelar.
Agradecimentos
Este trabalho foi apoiado pelo Instituto Nacional de Saúde de subvenção GM118085 e em parte pela Fundação Robert Welch (conceder F-1811 a R.M.H.).
Materiais
| Name | Company | Catalog Number | Comments |
| Reagents | |||
| Bacto Dehydrated Agar | Fisher Scientific | DF0140-15-4 | |
| EDTA Disodium Salt, Dihydrate | Fisher Scientific | 02-002-786 | |
| Eiken agar | Eiken Chemical Co. Japan | E-MJ00 | Essential for E. coli swarming |
| Glucose D (+) | Fisher Scientific | 410955000 | |
| LB (Lennox) Broth | Fisher Scientific | BP1427-500 | |
| Poly-L-lysine Solution (0.1%) | Sigma-Aldrich | P8920 | |
| Potassium chloride (KCl) | Fisher Scientific | 18-605-496 | |
| Potassium Phosphate monobasic (KH2PO4) | Fisher Scientific | BP362-500 | |
| Potassium Phosphate dibasic (K2HPO4) | Fisher Scientific | BP363-500 | |
| Sodium chloride (NaCl) | Fisher Scientific | S271-500 | |
| Materials and Equipment | |||
| CellSense microscope imaging software (V. 1.6) | Olympus | Or equivalent software for microscope used | |
| Electron Microscopy Sciences Scotch 666 Doube Sided Tape | Fisher | 50-285-28 | |
| Frosted microscope slides 3x1x1mm | Fisher | 12-550-343 | |
| Olympus BX53 microscope | Olympus | BX53 | Any upright or inverted phase microscope can be used |
| Petri dishes (100 mm diameter) | Fisher Scientific | FB0875712 | For soft-agar assays |
| Polyethylene Nebulizer Capillary Tubing (0.58mm x 99mm 3.0m) | Perkin Elmer | 9908265 | |
| Round Petri Dish with 2 Compartments | VWR | 89200-944 | For border-crossing assays |
| Safety Hypodermic Needles (23G) | Fisher Scientific | 14-826A | |
| Sterile Syringe - 1 mL | Fisher scientific | 14-955-450 | |
| Task/Tissue wipes | Fisher scientific | 06-666 | Or equivalent single use tissue wipes |
| VWR micro cover-glass 18x18mm | VWR | 48366205 | |
| XM10 camera | Olympus | XM10 | Or equivalent microscope camera |
Referências
- Jarrell, K. F., McBride, M. J. The surprisingly diverse ways that prokaryotes move. Nature Reviews in Microbiology. 6 (6), 466-476 (2008).
- Harshey, R. M. Bacterial motility on a surface: many ways to a common goal. Annual Reviews Microbiology. 57, 249-273 (2003).
- Duan, Q., Zhou, M., Zhu, L., Zhu, G. Flagella and bacterial pathogenicity. Journal of Basic Microbiology. 53 (1), 1-8 (2013).
- Nakamura, S., Minamino, T. Flagella-Driven Motility of Bacteria. Biomolecules. 9 (7), (2019).
- Haiko, J., Westerlund-Wikstrom, B. The role of the bacterial flagellum in adhesion and virulence. Biology (Basel). 2 (4), 1242-1267 (2013).
- Berg, H. C. . E. coli in Motion. 1 edn. , (2004).
- Berg, H. C. The rotary motor of bacterial flagella. Annual Review of Biochemistry. 72, 19-54 (2003).
- Xing, J., Bai, F., Berry, R., Oster, G. Torque-speed relationship of the bacterial flagellar motor. Proceedings of the National Academy of Sciences U. S. A. 103 (5), 1260-1265 (2006).
- Chen, X., Berg, H. C. Torque-speed relationship of the flagellar rotary motor of Escherichia coli. Biophysics Journal. 78 (2), 1036-1041 (2000).
- Turner, L., Ryu, W. S., Berg, H. C. Real-time imaging of fluorescent flagellar filaments. Journal of Bacteriology. 182 (10), 2793-2801 (2000).
- Brown, M. T., Delalez, N. J., Armitage, J. P. Protein dynamics and mechanisms controlling the rotational behaviour of the bacterial flagellar motor. Current Opinion in Microbiology. 14 (6), 734-740 (2011).
- Parkinson, J. S., Hazelbauer, G. L., Falke, J. J. Signaling and sensory adaptation in Escherichia coli chemoreceptors: 2015 update. Trends in Microbiology. 23 (5), 257-266 (2015).
- Kearns, D. B. A field guide to bacterial swarming motility. Nature Reviews Microbiology. 8 (9), 634-644 (2010).
- Harshey, R. M., Partridge, J. D. Shelter in a Swarm. Journal of Molecular Biology. 427 (23), 3683-3694 (2015).
- Ariel, G., et al. Swarming bacteria migrate by Levy Walk. Nature Communications. 6, 8396 (2015).
- Partridge, J. D., Nhu, N. T. Q., Dufour, Y. S., Harshey, R. M. Escherichia coli Remodels the Chemotaxis Pathway for Swarming. mBio. 10 (2), (2019).
- Butler, M. T., Wang, Q., Harshey, R. M. Cell density and mobility protect swarming bacteria against antibiotics. Proceedings of the National Academy of Science U. S. A. 107 (8), 3776-3781 (2010).
- Mobley, H. L., Belas, R. Swarming and pathogenicity of Proteus mirabilis in the urinary tract. Trends in Microbiology. 3 (7), 280-284 (1995).
- Burall, L. S., et al. Proteus mirabilis genes that contribute to pathogenesis of urinary tract infection: identification of 25 signature-tagged mutants attenuated at least 100-fold. Infections and Immunity. 72 (5), 2922-2938 (2004).
- Mazzantini, D., et al. FlhF Is Required for Swarming Motility and Full Pathogenicity of Bacillus cereus. Frontiers in Microbiology. 7, 1644 (2016).
- Partridge, J. D., Harshey, R. M. Swarming: flexible roaming plans. Journal of Bacteriology. 195 (5), 909-918 (2013).
- Yuan, J., Berg, H. C. Resurrection of the flagellar rotary motor near zero load. Proceedings of the National Academy of Science U. S. A. 105 (4), 1182-1185 (2008).
- Yuan, J., Fahrner, K. A., Berg, H. C. Switching of the bacterial flagellar motor near zero load. Journal of Molecular Biology. 390 (3), 394-400 (2009).
- Terasawa, S., et al. Coordinated reversal of flagellar motors on a single Escherichia coli cell. Biophysics Journal. 100 (9), 2193-2200 (2011).
- Nord, A. L., Sowa, Y., Steel, B. C., Lo, C. J., Berry, R. M. Speed of the bacterial flagellar motor near zero load depends on the number of stator units. Proceedings of the National Academy of Science. 114 (44), 11603-11608 (2017).
- Block, S. M., Segall, J. E., Berg, H. C. Adaptation kinetics in bacterial chemotaxis. J Bacteriol. 154 (1), 312-323 (1983).
- Segall, J. E., Block, S. M., Berg, H. C. Temporal comparisons in bacterial chemotaxis. Proceedings of the National Academy of Science U. S. A. 83 (23), 8987-8991 (1986).
- Wolfe, A. J., Conley, M. P., Kramer, T. J., Berg, H. C. Reconstitution of signaling in bacterial chemotaxis. Journal of Bacteriology. 169 (5), 1878-1885 (1987).
- Blair, D. F., Berg, H. C. Restoration of torque in defective flagellar motors. Science. 242 (4886), 1678-1681 (1988).
- Parkinson, J. S. A "bucket of light" for viewing bacterial colonies in soft agar. Methods Enzymol. 423, 432-435 (2007).
- Adler, J. Chemotaxis in bacteria. Science. 153 (3737), 708-716 (1966).
- Wolfe, A. J., Berg, H. C. Migration of bacteria in semisolid agar. Proceedings of the National Academy of Science U. S. A. 86 (18), 6973-6977 (1989).
- Kojadinovic, M., Sirinelli, A., Wadhams, G. H., Armitage, J. P. New motion analysis system for characterization of the chemosensory response kinetics of Rhodobacter sphaeroides under different growth conditions. Applied and Environmental Microbiology. 77 (12), 4082-4088 (2011).
- Nieto, V., et al. Under Elevated c-di-GMP in Escherichia coli, YcgR Alters Flagellar Motor Bias and Speed Sequentially, with Additional Negative Control of the Flagellar Regulon via the Adaptor Protein RssB. Journal of Bacteriology. 202 (1), (2019).
- Parkinson, J. S., Parker, S. R., Talbert, P. B., Houts, S. E. Interactions between chemotaxis genes and flagellar genes in Escherichia coli. Journal of Bacteriology. 155 (1), 265-274 (1983).
- Roman, S. J., Meyers, M., Volz, K., Matsumura, P. A chemotactic signaling surface on CheY defined by suppressors of flagellar switch mutations. Journal of Bacteriology. 174 (19), 6247-6255 (1992).
- Sanna, M. G., Simon, M. I. Isolation and in vitro characterization of CheZ suppressors for the Escherichia coli chemotactic response regulator mutant CheYN23D. Journal of Biological Chemistry. 271 (13), 7357-7361 (1996).
- Sockett, H., Yamaguchi, S., Kihara, M., Irikura, V. M., Macnab, R. M. Molecular analysis of the flagellar switch protein FliM of Salmonella typhimurium. Journal of Bacteriology. 174 (3), 793-806 (1992).
- Irikura, V. M., Kihara, M., Yamaguchi, S., Sockett, H., Macnab, R. M. Salmonella typhimurium fliG and fliN mutations causing defects in assembly, rotation, and switching of the flagellar motor. Journal of Bacteriology. 175 (3), 802-810 (1993).
- Ishida, T., et al. Sodium-powered stators of the bacterial flagellar motor can generate torque in the presence of phenamil with mutations near the peptidoglycan-binding region. Molecular Microbiology. 111 (6), 1689-1699 (2019).
- Barker, C. S., Meshcheryakova, I. V., Kostyukova, A. S., Samatey, F. A. FliO regulation of FliP in the formation of the Salmonella enterica flagellum. PLoS Genetics. 6 (9), 1001143 (2010).
- Paul, K., Nieto, V., Carlquist, W. C., Blair, D. F., Harshey, R. M. The c-di-GMP binding protein YcgR controls flagellar motor direction and speed to affect chemotaxis by a "backstop brake" mechanism. Molecular Cell. 38 (1), 128-139 (2010).
- Qian, C., Wong, C. C., Swarup, S., Chiam, K. H. Bacterial tethering analysis reveals a "run-reverse-turn" mechanism for Pseudomonas species motility. Applied and Environmental Microbiology. 79 (15), 4734-4743 (2013).
- Manson, M. D., Tedesco, P. M., Berg, H. C. Energetics of flagellar rotation in bacteria. Journal of Molecular Biology. 138 (3), 541-561 (1980).
- Kuwajima, G. Construction of a minimum-size functional flagellin of Escherichia coli. Journal of Bacteriology. 170 (7), 3305-3309 (1988).
- Kuhn, M. J., et al. Spatial arrangement of several flagellins within bacterial flagella improves motility in different environments. Nature Communication. 9 (1), 5369 (2018).
- Hranitzky, K. W., Mulholland, A., Larson, A. D., Eubanks, E. R., Hart, L. T. Characterization of a flagellar sheath protein of Vibrio cholerae. Infections and Immun. 27 (2), 597-603 (1980).
- Wang, Q., Frye, J. G., McClelland, M., Harshey, R. M. Gene expression patterns during swarming in Salmonella typhimurium: genes specific to surface growth and putative new motility and pathogenicity genes. Molecular Microbiology. 52 (1), 169-187 (2004).
- Gode-Potratz, C. J., Kustusch, R. J., Breheny, P. J., Weiss, D. S., McCarter, L. L. Surface sensing in Vibrio parahaemolyticus triggers a programme of gene expression that promotes colonization and virulence. Molecular Microbiology. 79 (1), 240-263 (2011).
- McCarter, L., Silverman, M. Surface-induced swarmer cell differentiation of Vibrio parahaemolyticus. Molecular Microbiology. 4 (7), 1057-1062 (1990).
- Pearson, M. M., Rasko, D. A., Smith, S. N., Mobley, H. L. Transcriptome of swarming Proteus mirabilis. Infections and Immunity. 78 (6), 2834-2845 (2010).
- Swiecicki, J. M., Sliusarenko, O., Weibel, D. B. From swimming to swarming: Escherichia coli cell motility in two-dimensions. Integrative Biology (Cambridge). 5 (12), 1490-1494 (2013).
- Colin, R., Drescher, K., Sourjik, V. Chemotactic behaviour of Escherichia coli at high cell density. Nature Communication. 10 (1), 5329 (2019).
- Partridge, J. D., Harshey, R. M. More than motility: Salmonella flagella contribute to overriding friction and facilitating colony hydration during swarming. Journal Bacteriology. 195 (5), 919-929 (2013).
- Morales-Soto, N., et al. Preparation, imaging, and quantification of bacterial surface motility assays. Journal Visualized Experiment. (98), e52338 (2015).
- Chawla, R., Ford, K. M., Lele, P. P. Torque, but not FliL, regulates mechanosensitive flagellar motor-function. Science Reports. 7 (1), 5565 (2017).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados