Method Article
Método de genética reversa simplificado para recuperar rotavírus recombinantes expressando proteínas de repórter
Neste Artigo
Resumo
A geração de rotavírus recombinantes a partir do DNA plasmático fornece uma ferramenta essencial para o estudo da replicação de rotavírus e patogênese, e o desenvolvimento de vetores e vacinas de expressão rotavírus. Aqui, descrevemos uma abordagem genética reversa simplificada para a geração de rotavírus recombinantes, incluindo cepas expressando proteínas fluorescentes de repórteres.
Resumo
Os rotavírus são uma população grande e em evolução de vírus de RNA segmentados de dupla cadeia que causam gastroenterite severa em jovens de muitas espécies de mamíferos e hospedeiros aviários, incluindo os humanos. Com o recente advento dos sistemas de genética reversa do rotavírus, tornou-se possível usar mutagênese direcionada para explorar a biologia do rotavírus, modificar e otimizar as vacinas rotavírus existentes e desenvolver vetores de vacinas multialvo rotavírus. Neste relatório, descrevemos um sistema de genética reversa simplificado que permite a recuperação eficiente e confiável de rotavírus recombinantes. O sistema baseia-se na cofecção de vetores de transcrição T7 expressando rotavírus de comprimento total (+)RNAs e um vetor CMV codificando uma enzima de capping de RNA em células BHK que produzem comstitutivamente a polimerase De T7 RNA (BHK-T7). Os rotavírus recombinantes são amplificados pela supervisão das células BHK-T7 transfeccionadas com células MA104, uma linha de células renais de macaco que é altamente permissiva para o crescimento do vírus. Neste relatório, também descrevemos uma abordagem para a geração de rotavírus recombinantes que expressam uma proteína repórter fluorescente separada através da introdução de um elemento de parada translacional 2A no segmento genoma 7 (NSP3). Esta abordagem evita excluir ou modificar qualquer um dos quadros de leitura abertos virais, permitindo assim a produção de rotavírus recombinantes que retêm proteínas virais totalmente funcionais enquanto expressam uma proteína fluorescente.
Introdução
Os rotavírus são as principais causas de gastroenterite grave em bebês e crianças pequenas, bem como os filhotes de muitas outras espécies de mamíferos e aviários1. Como membros da família Reoviridae, os rotavírus têm um genoma de RNA de dupla cadeia segmentada (dsRNA). Os segmentos do genoma estão contidos dentro de uma virion icosaedro não envolta formada a partir de três camadas concêntricas de proteína2. Com base no sequenciamento e análise filogenética dos segmentos do genoma, foram definidas nove espécies de rotavírus (A−D, F−J)3. Essas cepas que compreendem a espécie rotavírus A são responsáveis pela grande maioria da doença humana4. A introdução de vacinas rotavírus em programas de imunização infantil a partir da última década está correlacionada com reduções significativas na mortalidade e morbidade de rotavírus. Mais notavelmente, o número de mortes infantis associadas ao rotavírus diminuiu de aproximadamente 528.000 em 2000 para 128.500 em 20164,5. As vacinas rotavírus são formuladas a partir de cepas atenuantes vivas do vírus, com 2 a 3 doses administradas em crianças até 6 meses de idade. O grande número de cepas de rotavírus geneticamente diversas que circulam em humanos e outras espécies de mamíferos, combinadas com sua capacidade de evoluir rapidamente através de mutagênese e reclassificação, pode levar a alterações antigênicas nos tipos de rotavírus que infectam crianças6,7,8. Tais alterações podem minar a eficácia das vacinas existentes, exigindo sua substituição ou modificação.
O desenvolvimento de sistemas de genética reversa totalmente baseados em plasmídeos que permitem a manipulação de qualquer um dos 11 segmentos do genoma rotavírus foi alcançado recentemente9. Com a disponibilidade desses sistemas, tornou-se possível desvendar detalhes moleculares de replicação de rotavírus e patogênese, desenvolver métodos aprimorados de triagem de alto desempenho para compostos anti-rotavírus e criar novas classes potencialmente mais eficazes de vacinas rotavírus. Durante a replicação do rotavírus, as RNAs virais (+)não só orientam a síntese de proteínas virais, mas também servem como modelos para a síntese dos segmentos do genoma dsRNA da progêia10,11. Todos os sistemas genéticos reversos de rotavírus descritos até hoje dependem da transfecção de vetores de transcrição T7 em linhas celulares de mamíferos como fonte de cDNA derivado (+)RNAs usados na recuperação de vírus recombinantes9,,12,13. Dentro dos vetores de transcrição, os cDNAs virais de comprimento completo são posicionados entre um promotor T7 a montante e o ribozyme de hepatite delta (HDV) de tal forma que as rnas virais (+)RNAs são sintetizadas por polimerase T7 RNA que contêm autênticos 5' e 3'-termini(Figura 1A). No sistema de genética reversa de primeira geração, os vírus recombinantes foram feitos por células renais de hamster de bebê transfeccionaexpressas T7 RNA polimerase (BHK-T7) com 11 vetores de transcrição T7 (pT7), cada uma síntese direcional de um Único (+)RNA da cepa do vírus SA11 símio, e três plasmídeos de expressão de unidade de promotor cmv, um codificando a proteína de fusão aviária reovirus p10FAST e duas subunidades codificadoras do vírus da vaccinia D1R-D12L capping enzima complexo9. Os vírus SA11 recombinantes gerados em células BHK-T7 transfeccionadas foram amplificados pela supervisão com células MA104, uma linha celular permissiva para o crescimento de rotavírus. Uma versão modificada do sistema de genética reversa de primeira geração foi descrita que não usa mais plasmídeos de suporte12. Em vez disso, o sistema modificado gera com sucesso rotavírus recombinantes simplesmente transfecting células BHK-T7 com os vetores de transcrição 11 SA11 T7, com a ressalva de que os vetores para os blocos de construção da fábrica viral (viroplasma) (proteínas não estruturais NSP2 e NSP5) são adicionados a níveis 3 vezes maiores que os outros vetores14,15. Também foram desenvolvidas versões modificadas do sistema genético reverso que suportam a recuperação das cepas humanas de KU e Odelia do rotavírus16,17. O genoma rotavírus é notavelmente passível de manipulação por genética reversa, com vírus recombinantes gerados até hoje com mutações introduzidas em VP418, NSP19, NSP219, NSP320,21, e NSP522,23. Entre os vírus mais úteis gerados até agora estão aqueles que foram projetados para expressar proteínas fluorescentes de repórteres (FPs)9,12,21,24,25.
Nesta publicação, fornecemos o protocolo para o sistema de genética reversa que usamos em nosso laboratório para gerar cepas recombinantes de rotavírus SA11. A principal característica do nosso protocolo é a cofecção de células BHK-T7 com os vetores de transcrição 11 pT7 (modificados para incluir níveis de 3x da enzima de capping pT7/NSP2SA11 e pT7/NSP5SA11) e um vetor de expressão CMV codificando o vírus da peste suína africana (ASFV) NP868R enzique21 (Figura 2). Em nossas mãos, a presença do plasmídeo NP868R leva à produção de títulos mais altos de vírus recombinantes por células BHK-T7 transfeccionadas. Nesta publicação, também fornecemos um protocolo para modificar o plasmídeo pT7/NSP3SA11 para que vírus recombinantes possam ser gerados que expressem não apenas o produto de proteína nSP3 do segmento 7, mas também um FP separado. Isso é feito pela reengenharia do quadro de leitura aberta NSP3 (ORF) no plasmídeo pT7/NSP3SA11 para conter um elemento de reinicialização translacional 2A a jusante seguido de um FP ORF(Figura 1B)24,26. Através desta abordagem, geramos rotavírus recombinantes expressando vários FPs: UnaG (verde), mKate (vermelho distante), mRuby (vermelho), TagBFP (azul), CFP (ciano) e YFP (amarelo)24,27,28. Esses rotavírus que expressam FP são feitos sem excluir o NSP3 ORF, produzindo assim vírus que devem codificar um complemento completo de proteínas virais em funcionamento.
Protocolo
1. Preparação da mídia e manutenção da cultura celular
- Obter células renais de hamster bebê expressando com stitutivamente t7 rna polimerase (BHK-T7) e células de rim de macaco verde africano MA104.
NOTA: As células BHK-T7 (ou BSR-T7) não estão disponíveis comercialmente, mas são uma linha celular comum de laboratórios que usam genética reversa para estudar a biologia do vírus RNA. A linha celular BHK-T7 utilizada neste protocolo foi obtida da Dra. Ursula J. Buchholz (National Institutes of Health, Bethesda, MD, USA), uma co-desenvolvedora da linha de células BHK original expressando T7 RNA polimerase29. As células MA104 estão disponíveis na European Collection of Authenticated Cell Cultures (ECACC) e na American Type Culture Collection (ATCC). - Prepare os seguintes meios de cultura em um ambiente estéril, de preferência um gabinete de segurança biológica classe II. Armazene a mídia no escuro a 4 °C e aqueça a 37 °C imediatamente antes de usar.
NOTA: As fontes de componentes de mídia são fornecidas na Tabela de Materiais.- Para preparar o meio incompleto do DMEM, combine 500 mL do meio mínimo essencial (MEM) da Águia modificada de Dulbecco contendo 4,5 g/L de glicose e 1% de glutamina com 5 mL de 100x pen-strep.
- Para preparar o meio completo do DMEM, combine 500 mL de Meio Incompleto de DMEM com 25 mL de soro bovino fetal (FBS).
- Para preparar o meio incompleto gmem, combine 500 mL de glasgow médio essencial mínimo, 5 mL de glutamina 100x, 50 mL de caldo de triptose-fosfato (TPB), 5 mL de aminoácidos não essenciais de 100x (NEAA) e 5 mL de 100x pen-strep.
- Para preparar o meio completo GMEM, combine 500 mL de meio incompleto gmem com 25 mL de FBS inativado por calor.
- Para preparar o meio completo GMEM+G, adicione 10 mL de 50 mg/mL de genética (G418) a 500 mL de meio completo GMEM.
- Para preparar o meio incompleto do SMEM, combine 500 mL do MEM da Águia modificada de Joklik, 5 mL de glutamina de 100x, 50 mL de TPB, 5 mL de 100x NEAA e 5 mL de 100x pen-strep.
- Células Cultura MA104 em frascos T75 ou T175 contendo 12 ou 25 mL de meio completo DMEM, respectivamente. Para passar células MA104 que atingiram 100% de confluência, enxaguar a monocamada celular 2x com solução salina tamponada por fosfato (PBS), dissociar com tripsina (0,05%)-EDTA (0,1%) solução e resuspender em 5 (frasco T75) ou 10 mL de (frasco T175) de meio incompleto DMEM.
- Coloque 0,5-1,0 mL de células ressuspensas em frascos frescos e traga para o volume final apropriado adicionando o meio completo DMEM. Coloque frascos em uma incubadora de 37 °C, 5% CO2.
- Propagar células BHK-T7 em frascos T75, alternando entre o uso de GMEM e GMEM+G meio completo a cada rodada de passagem. Para subculturar células BHK-T7 que atingiram 100% de confluência, enxaguar a monocamada celular 2x com PBS, dissociar com solução trypsin-EDTA e resuspender em 5 mL de meio.
- Depois de colocar 15 mL de GEM ou GMEM+G completo em um frasco T75 fresco, adicione quatro gotas de células BHK-T7 ressuspensas. Coloque o frasco em uma incubadora de 37 °C, 5% CO2.
2. Preparação plasmídea
- Obter os seguintes plasmídeos expressando rotavírus SA11 (+)RNAs de Addgene: pT7/VP1SA11, pT7/VP2SA11, pT7/VP3SA11, pT7/VP4SA11, pT7/VP6SA11, pT7/VP7SA11, pT7/NSP1SA11, pT7/NSP2SA11, pT7/NSP3SA11, pT7/NSP4SA11 e pT7/NSP5SA11. Obter o plasmídeo que expressa a enzima de capping ASFV (pCMV/NP868R) dos autores21.
NOTA: Os plasmídeos pT7/NSP3SA11 modificados projetados para expressar um FP através da atividade de um elemento translacional 2A (pT7/NSP3-2A-3xFL-FP) também podem ser obtidos dos autores24,26. os plasmídeos pT7/NSP3-2A-3xFL-FP que expressam os seguintes FPs estão disponíveis: UnaG (verde), mKate (vermelho), mRuby (vermelho), TagBFP (azul), CFP (ciano) e YFP (amarelo)24. - Transforme plasmídeos em Ecompetentes . coli DH5α e espalhar bactérias em placas de ágar de caldo luria contendo o antibiótico apropriado. Cultivar culturas bacterianas colhidas em colônias individuais e preparar quantidades menores de plasmídeo (20 μg) usando um kit de purificação de miniprep de spin(Tabela de Materiais), deacordo com as instruções do fabricante. Preparar quantidades maiores de plasmídeo de culturas bacterianas usando kits de purificação midi e maxi (Tabela de Materiais).
NOTA: Plasmídeos adequados para uso no sistema genético reverso também foram preparados com outros kits de purificação plasmídeos. Não é necessário preparar plasmídeos usando kits especificamente projetados para gerar material livre de endotoxinas. - Ajuste as concentrações de plasmídeos purificados para 1 mg/mL em água de grau de biologia molecular livre de nuclease. Verifique a pureza plasmídea confirmando uma relação de absorvância de 260/280 de ~1,8 usando um espectrofotômetro. Além disso, use eletroforese em géis de agarose de 0,8% para verificar se os plasmídeos são predominantemente supercoiled.
- Aquotpuria plasmídeos em tubos de microcentrífuga estéril de 0,5 mL, cada um contendo 10 μL. Armazene plasmídeos a -80 °C.
3. Geração de vírus recombinante
NOTA: A pesquisa de rotavírus humanos e animais, incluindo a geração e caracterização de cepas de rotavírus recombinantes, deve ser tratada sob condições de Biossegurança Nível 2 (BSL-2) e exigirá aprovação prévia do Comitê Institucional de Biossegurança (IBC). As condições laboratoriais apropriadas da BSL-2 são descritas em Biossegurança em Laboratórios Microbiológicos e Biomédicos (BMBL) produzidos pelos Centros de Controle e Prevenção de Doenças (CDC)30.
- Dia 1: semeamento de células BHK-T7 em placas de 12 poços
- Enxágüe uma monocamada recém-confluente de células BHK-T7 contidas em um frasco T75 2x com PBS. Interrompa a monocamada celular com a solução trypsin-EDTA e resuspenda as células em 5 mL de meio completo GMEM.
- Use um contador de células automatizado e uma solução trypan-blue para determinar a concentração de células BHK-T7 viáveis no meio. Células de sementes em placas de cultura celular de 12 poços, com cada poço contendo 2 x 105 células em um volume total de 1 mL de GMEM (livre de G418) de meio completo. Incubar em uma incubadora de 37 °C, 5% CO2. As células devem atingir 80-90% de confluência até o dia 2.
- Dia 2: transfecção de células BHK-T7 com misturas plasmáticas
- Para preparar a mistura plasmídea, combine o seguinte em um tubo de microcentrífuga de 0,5 mL usando estoques plasmáticos ajustados a 1 mg/mL: 0,8 μL cada um dos plasmídeos SA11 pT7 VP1, VP2, VP3, VP4, VP6, VP7, NSP1, NSP3 e NSP4, 2,4 μL cada um dos plasmídeos SA11 pT7 NSP2 e NSP5, e 0,8 μL de pCMV/NP868R. Misture suavemente os plasmídeos tocando no tubo e colete o conteúdo por centrifugação de pulso. Para preparar vírus recombinantes que expressem FPs, substitua o pT7/NSP3SA11 pelo plasmídeo pT7/NSP3-2A-3xFL-FP apropriado.
NOTA: As misturas plasmáticas devem ser armazenadas no gelo até serem utilizadas. Um tubo separado deve ser preparado para cada transfecção. - Para preparar a mistura de reagente de reagente médio/transfecção do soro plasmídeo/reduzido: Adicione 110 μL de meio soro pré-aquecido (37 °C) reduzido(Tabela de Materiais)a cada mistura plasmídea e misture suavemente pipetando para cima e para baixo. Em seguida, adicione 32 μL de reagente de transfecção(Tabela de Materiais)a cada mistura média plasmídea/reduzida do soro; isso produz uma concentração de 2,5 μL de reagente de transfecção por μg de plasmídeo em misturas. As misturas de vórtice suavemente e incubam em temperatura ambiente por 20 min.
- Durante o período de incubação de 20 min, enxágue as células BHK-T7 em placas de 12 poços (preparadas no dia 1) uma vez com 2 mL de mídia incompleta GMEM. Em seguida, adicione 1 mL de SMEM de meio incompleto a cada poço e retorne as placas à incubadora.
- Após o período de incubação de 20 min, adicione a mistura média/transfecção do soro plasmídeo/reduzido que da daca a cada poço das placas de 12 poços usando um pipettor de 200 μL. Acerte suavemente as placas e retorne a uma incubadora de 37 °C, 5% CO2.
- Para preparar a mistura plasmídea, combine o seguinte em um tubo de microcentrífuga de 0,5 mL usando estoques plasmáticos ajustados a 1 mg/mL: 0,8 μL cada um dos plasmídeos SA11 pT7 VP1, VP2, VP3, VP4, VP6, VP7, NSP1, NSP3 e NSP4, 2,4 μL cada um dos plasmídeos SA11 pT7 NSP2 e NSP5, e 0,8 μL de pCMV/NP868R. Misture suavemente os plasmídeos tocando no tubo e colete o conteúdo por centrifugação de pulso. Para preparar vírus recombinantes que expressem FPs, substitua o pT7/NSP3SA11 pelo plasmídeo pT7/NSP3-2A-3xFL-FP apropriado.
- Dia 4: supervisionando células BHK-T7 transfeccionadas com células MA104
- Enxágüe uma monocamada recém-confluente de células MA104 contidas em um frasco T75 2x com PBS. Interrompa a monocamada usando a solução trypsin-EDTA e resuspenda as células em 5 mL de meio completo DMEM.
- Utilizando um contador de células automatizado e uma solução trypan-blue, determine a concentração de células MA104 viáveis no meio. Ajuste a concentração para 8 x 105 células MA104/mL no meio incompleto do DMEM e adicione 0,25 mL de células suspensas (2 x 105 células) dropwise aos poços que contêm células BHK-T7 transfeccionadas.
- Ajuste a concentração de tripsina (pancreático suíno tipo IX) no meio a ~0,5 μg/mL adicionando 0,8 μL de 1 mg/mL de estoque de tripspsina a cada poço.
NOTA: Os estoques de tripsina devem ser preparados em PBS, alicitados e armazenados a -20 °C. - Use as células MA104 restantes para preparar placas de 6 poços que serão necessárias no dia 7 para amplificar vírus recombinantes. Para semear placas de 6 poços, diluir as células MA104 ressuspensas para uma concentração de 1,5 x 105 células/mL no meio completo DMEM e colocar 2 mL em cada poço. Coloque as placas em uma incubadora de 37 °C, 5% CO2.
- Dia 7: recuperação e amplificação do vírus recombinante de células transinfectadas
- Sujeito células BHK-T7/MA104 em placas de 12 poços a três ciclos de congelamento-degelo em condições estéreis, movendo placas entre -20 °C freezer e uma superfície de temperatura ambiente. Depois de transferir lysates para tubos de 1,5 mL, tubos de centrífuga por 10 min a 500 x g (4 °C) para pellet grandes detritos celulares. Coletar sobrenadante e armazenar a 4 °C (curto prazo) ou -20 °C (longo prazo).
- Lave as monocamadas MA104 em placas de 6 poços (preparadas no dia 4) 2x com PBS. Coloque 2 mL de Meio Incompleto de DMEM para cada poço que contenha 0,5 μg/mL de tripsina. Adicione 300 μL de sobrenadante recuperado de talatos de células BHK-T7/MA104 em poços e coloque placas em uma incubadora de 37 °C, 5% CO2.
- Incubar placas por 7 dias ou até que sejam observados efeitos citopáticos completos (CPE). Células Lyse MA104 em placas de 6 poços por três ciclos de congelamento-degelo, em seguida, transferir lisatos para tubos de microcentrífuga de 1,5 mL. Pelotas grandes detritos celulares por centrifugação por 10 min a 500 x g (4 °C). Transfira as células clarificadas para tubos de microcentrífuge de 1,5 mL e armazene a -20 °C.
4. Isolamento da placa de vírus recombinantes
- Ativar vírus em 100 μL de lysates de células esclarecidas adicionando tripsina a uma concentração final de 10 μg/mL e incubando a 37 °C por 1 h. Prepare uma série de diluição serial de 10 vezes variando de 10-1 a 10-7 (1 mL cada) em meio incompleto.
- Enxágüe as monocamadas MA104 em placas de 6 poços 2x com 2 mL de PBS e uma vez com meio incompleto de DMEM. Adicione 400 mL de diluições lisato supér8as nas placas. Incubar as placas por 1 h em uma incubadora de 37 °C, 5% CO2, balançando a cada 10-15 min para redistribuir as diluições através da monocamada.
- Prepare uma solução de sobreposição agarose-MEM combinando volumes iguais do meio essencial mínimo de 2x Eagle (EMEM; pré-aquecido a 37 °C) com 1,5% de agarose que foi derretida em água usando um forno microondas e pré-resfriada a 45 °C. Mantenha a solução de sobreposição a 42 °C usando um banho de água e ajuste-se para uma concentração final de 0,5 μg/mL de tripsina imediatamente antes de colocar nas células.
- Retire as diluições de lysate de placas de 6 poços e enxágue as células uma vez com 2 mL de DMEM incompleto. Sobreponha suavemente 3 mL da solução de sobreposição agarose-MEM sobre a monocamada celular contida em cada poço. Deixe a sobreposição de agarose endurecer à temperatura ambiente e, em seguida, retorne as placas para a incubadora.
- Três dias depois, prepare uma solução de sobreposição agarose-MEM como na etapa 4.3 e leve a 42 °C. Imediatamente antes de usar, ajuste a solução de sobreposição para uma concentração final de vermelho neutro de 50 μg/mL(Tabela de Materiais).
- Adicione 2 mL da solução de sobreposição em cima da camada de agarose existente nas placas de 6 poços. Depois de permitir que a nova camada de agarose endureça, retorne as placas para a incubadora. Proteger soluções e placas contendo vermelho neutro da exposição à luz.
- Durante as próximas 6h, identifique placas rotavírus nas placas de 6 poços com o auxílio de uma caixa de luz. Escolha placas claramente definidas usando pipetas de transferência descartáveis, recuperando plugues de agarose que se estendem totalmente à camada celular.
- Expelir o plugue em um tubo de 1,5 mL contendo 0,5 mL de Meio Incompleto e vórtice de DMEM para 30 s. Amplificar o vírus isolado da placa eluido no meio pela propagação em monocamadas MA104 em placas de 6 poços ou frascos T25 contendo meio incompleto E 0,5 μg/mL trypsin.
NOTA: Informações adicionais sobre isolamento e telerotação de rotavírus por ensaio de placa estão disponíveis em Arnold et al.31.
5. Eletroforese em gel de dsRNA viral
- Coloque 600 μL de lysates celulares infectados esclarecidos e 400 μL de tiocianato de guanidinium em tubos de microcentrífuge de 1,5 mL, vórtice para 30 s e incubar à temperatura ambiente por 5 minutos. Adicione 200 μL de clorofórmio, vórtice para 30 s e incubar por 3 minutos. Depois de centrifugar por 5 min a 13.000 x g (4 °C), transfira ~550 μL da fase aquosa superior para tubos frescos.
- Recuperar dsRNA viral da fase aquosa adicionando 2 volumes (~900 μL) de álcool isopropílico frio e tubos invertidos 4-6x. Depois de incubar em temperatura ambiente por 10 min, tubos de centrífuga por 10 min a 13.000 x g (4 °C). Descarte os sobrenatantes e retenha as pelotas de RNA.
- Lave as pelotas adicionando 1 mL de 75% de etanol ao tubo, invertendo uma vez e centrifugando por 5 min a 7.500 x g (4 °C). Depois de remover cuidadosamente a lavagem de etanol com um pipettor, permita que as pelotas de RNA sequem a ar no banco de laboratório por 5-10 min. Dissolva as pelotas de RNA em 15 μL de água de grau de biologia molecular sem nuclease e armazene a -20 °C.
- Combine 10 μL de amostras de RNA dissolvidas com 2 μL de tampão de carregamento de DNA de 6x, carregue em mini géis de poliacrilamida pré-moldados (ou equivalentes fundidos à mão) e resolva RNAs por eletroforese em tampão de execução tris-glicina por 2 h sob uma corrente constante (16 mA). Mergulhe géis por 5-10 min em água contendo ~1 μg/mL brometo de etídio e detecte segmentos do genoma do rotavírus com um transiluminador UV.
6. Recuperação e sequenciamento de dsRNA viral
- Localize bandas de dsRNA virais em géis de poliacrilamida preferencialmente usando luz UV de comprimento de onda longa de baixa intensidade, para evitar gerar ligações nucleicas-ácidas. Usando uma lâmina de barbear fresca, corte fragmentos de gel contendo bandas de dsRNA e transfira para tubos de microcentrífuga de 1,5 mL.
- Depois de adicionar 10-20 μL de água livre de RNase, esmague cada fragmento com um pilão de pelota descartável sem RNase projetado para caber um tubo de microcentrífuga de 1,5 mL ou desenhando para cima e para baixo através de uma agulha de 18 G. Incubar fragmentos esmagados durante a noite a 4 °C.
- Recuperar 3 μL de líquido de tubos contendo fragmentos de gel esmagados. Gerar cDNAs dos dsRNAs virais usando um kit de reação em cadeia de polimerase de transcrição reversa de um passo (RT-PCR) e primers oligonucleotídeos apropriados.Table of Materials
NOTA: Os produtos PCR são purificados em gel com um kit de limpeza PCR (Tabela de Materiais) e enviados para sequenciamento noturno, juntamente com primers, por serviços de seqüência de DNA comercial.
7. Análise imunoblotde proteínas virais
- Sementes placas de 6 poços com 3 x 105 células MA104 por poço em um volume total de 2 mL de meio completo DMEM. Coloque as placas em uma incubadora de 37 °C, 5% CO2 e deixe até que as células atinjam a confluência (3-5 dias).
NOTA: Poços com monocamadas confluentes conterão ~1,2 x 106 células. - Trate os supernatantes esclarecidos incubando com tripsina de 10 μg/mL a 37 °C por 1h para ativar partículas de rotavírus recombinantes contidas dentro delas.
- Enxágüe as monocamadas MA104 em placas de 6 poços 2x com 2 mL de PBS. Infectar células adicionando, em cada poço, 200 μL de inóculo contendo 3-5 unidades formadoras de placas (PFU) por célula de rotavírus ativado por tripsina em meio incompleto DMEM. Devolva as placas para a incubadora.
- A cada 10 min, remova as placas e balance suavemente para redistribuir o inóculo através da monocamada celular. Após 1h, substitua o inóculo por 2 mL de meio incompleto de DMEM.
- Às 8h após a infecção, enxágue as células em placas de 6 poços 2x com PBS. Raspar células em 750 μL de PBS e transferir volume para um tubo de microcentrífuga de 1,5 mL. Enxágüe a placa com outros 750 μL de PBS e combine com a amostra coletada anterior de 750 μL.
- Células de pelotas em amostra por centrifugação a 5.000 x g por 10 min a 4 °C. Depois de descartar o sobrenadante, armazene as pelotas de células a -80 °C até que seja processada posteriormente.
- Prepare o tampão de lyse celular contendo 300 mM NaCl, 100 mM Tris-HCl, pH 7.4, 2% Triton X-100 e 1x inibidor de protease completa sem EDTA. Depois de adicionar 300 μL de tampão de lyse a pelotas de células congeladas, brevemente amostras de vórtice e incubar no gelo por 10 min.
- Repita o processo de vórtice e incubação de amostras no gelo 3x. Depois, amostras de centrífugas a 15.000 x g por 10 min a 4 °C, depois coletam sobrenatantes e armazenam a -80 °C.
- Resolver proteínas contidas em volumes de 20 μL de amostras sobrenatantes por eletroforese em mini géis lineares pré-fundidos de 8 a 16% de poliacrilamida e transferência para membranas de nitrocelulose. Bloquear membranas por incubação com PBS-Tween 20 (0,02%) solução contendo 5% de leite seco sem gordura.
- Sonda as membranas incubando com um ou mais anticorpos primários (por exemplo, cobaia anti-NSP3 ou anti-VP6 anti-soro, anticorpo anti-Bandeira M2 monoclonal do rato ou anticorpo anti-PCNA monoclonal). Detecte anticorpos primários incubando membranas com o cavalo anti-rato IgG, anti-cobaia IgG, ou anti-coelho igG de cabra anti-coelho anti-rabanete peroxidase-conjugado anticorpos secundários, seguido pelo substrato de quimiolum (ECL) aprimorado. Visualize sinais de luminescência usando um sistema de imagem em gel(Table of Materials)ou filme de raios-X.
8. Imagens em células vivas de células infectadas com vírus que expressam FP
- Sementes placas de 6 poços com 3 x 105 células MA104 por poço em um total de 2 mL de meio completo DMEM. Coloque as placas em uma incubadora de 37 °C, 5% CO2 e deixe até que as células atinjam a confluência (3-5 dias).
NOTA: Poços com monocamadas confluentes conterão ~1,2 x 106 células. - Ativar amostras de rotavírus, de título conhecido, por incubação com tripsina de 10 μg/mL a 37 °C por 1 h.
- Enxágüe placas de 6 poços com monocamadas MA104 2x com 2 mL de PBS. Infectar células adicionando a cada poço 200 μL de inóculo contendo 3-5 PFU por célula de rotavírus ativado por tripsina em meio incompleto DMEM. Retorne as placas para a incubadora e suavemente as placas de rocha para redistribuir o inóculo através da monocamada celular a cada 10 min.
NOTA: Poços que são simulados infectados com inóculo DMEM livre de vírus devem ser incluídos como controles. - Após 1h de adsorção de vírus, enxágue a monocamada 2x com PBS e substitua o meio por 0,5 mL por poço de meio incompleto do DMEM. A 7,5 h pós-infecção, substitua o meio de cultura pelo DMEM por fluorescência de fundo baixa (Tabela de Materiais). Às 8h após a infecção, use um imager celular vivo para examinar as células em ampliação de 20x para sinal de fluorescência verde, vermelha ou azul.
Resultados
O protocolo de genética reversa descrito neste artigo prossegue através de múltiplas etapas distintas: (1) co-transfecção de células BHK-T7 com vetores de transcrição de rotavírus pT7 e um plasmídeo de expressão pCMV/NP868R, (2) supervisão de células BHK-T7 transfeccionadas com células MA104, (3) amplificação de vírus recombinantes presentes em células BHK-T7/MA104 usando células MA104 e (4) isolamento de placa de vírus recombinante usando células MA104(Figura 2). Em nossas mãos, o protocolo é eficiente, produzindo títulos de vírus wildtype sa11 recombinante (rSA11/wt) em lisatos celulares BHK-T7/MA104 de ~104 PFU/mL e em lisatos celulares MA104 amplificados de >1 x 107 PFU/mL. Os vírus recombinantes SA11 gerados pela genética reversa usando plasmídeos pT7/NSP3SA11 modificados expressando FPs (por exemplo, rSA11/NSP3-2A-3xFL-UnaG) crescem até títulos que são ~4 vezes menos que rSA11/wt.
Seguindo o protocolo de genética reversa, geramos vírus SA11 recombinantes que foram facilmente identificados por ensaio de placa em células MA104, permitindo assim o isolamento da placa(Figura 3D). Os vírus em placas foram colhidos com uma tubulação de transferência descartável de ponta longa e amplificados em células MA104. Os genomas dsRNA dos vírus rSA11/wt purificados por placa e rSA11/NSP3-2A-3xFL-UnaG foram extraídos com guanidinium thiocyanato, resolvidos por eletroforese em um gel de poliacrilamida de 10% e detectados por coloração com brometo de ethidium(Figura 3A). Como esperado, o segmento 7 (NSP3) dsRNA de rSA11/NSP3-2A-3xFL-UnaG migrou muito mais lento do que o de rSA11/wt(Figura 3A)devido à presença de seqüências 2A-3xFL-UnaG. O segmento 7 (NSP3) dsRNA de rSA11/NSP3-2A-3xFL-UnaG foi purificado em gel, convertido em forma de cDNA por RT-PCR, e sequenciado para confirmar a precisão.
Para verificar a expressão da proteína fluorescente UnaG, as células MA104 em placas de 6 poços foram infectadas com 3 PFU por célula de rSA11/wt e rSA11/NSP3-2A-3xFL-UnaG. Às 8h pós-infecção, o meio de cultura em placas foi substituído por 0,5 mL de DMEM com fluorescência de fundo baixo por poço e as placas incubadas por um adicional de 30 min a 37 °C em uma incubadora de CO2. Posteriormente, as placas foram examinadas para expressão UnaG usando um imager de célula viva. A análise mostrou que o rSA11/NSP3-2A-3xFL-UnaG produziu fluorescência verde, verificando a funcionalidade do gene UnaG no vírus recombinante(Figura 3B). Em contrapartida, a fluorescência verde não foi detectada em células infectadas com rSA11/wt. Para abordar se o elemento 2A no segmento modificado 7 de rSA11/NSP3-2AxFL-UnaG promoveu a expressão de duas proteínas separadas (NSP3-2A e 3xFL-UnaG), as células MA104 foram infectadas com rSA11/NSP3-2A-3xFL-UnaG e rSA11/wt. Os lysates celulares foram preparados a partir de células colhidas a 8h após a infecção, resolvidas por eletroforese em gel, e borrifadas em filtros de nitrocelulose. As manchas foram sondadas com anticorpos específicos para rotavírus VP6 e NSP3, e tag FLAG. A análise mostrou que nsp3-2A e 3xFL-UnaG foram expressos como proteínas separadas em células infectadas com rSA11/NSP3-2A-3xFL-UnaG, indicando que o elemento 2A era funcional(Figura 3C). As células infectadas com rSA11/wt não expressaram proteína reconhecida pelo anticorpo anti-FLAG. A proteína NSP3 presente em células infectadas rSA11/NSP3-2A-3xFL-UnaG por anticorpo anti-NSP3 migrou ligeiramente mais lentamente do que nsp3 presente em células infectadas por rSA11/wt devido à presença de resíduos remanescentes de 2A no Terminal C de NSP3.

Figura 1: Plasmídeos de genética reversa do rotavírus. (A) Os cDNAs de comprimento total dos 11 segmentos do genoma rotavírus SA11 estão posicionados dentro de plasmídeos pT7, ligados a montante com um promotor para polimerase T7 RNA e rio abaixo com um ribozyme HDV. Na presença de T7 RNA polymerase, os plasmídeos rotavírus pT7 produzem SA11 (+)RNAs de comprimento completo com autênticos 5' e 3' termini. (B) Esquemas dos produtos (+)RNA e proteínas fabricados pelo plasmídeo pT7/NSP3-2A-3xFL-UnaG. O esquema inclui a seqüência de cDNA NSP3, e seqüências para o elemento porcino teschovirus 2A-like (2A), a tag 3x FLAG (FL) e a proteína verde fluorescente UnaG. A posição do local de reinicialização translacional 2A é indicada com uma seta vermelha. Devido à atividade do elemento 2A, a tradução do RNA produz duas proteínas. A porção NSP3 contém restos do elemento 2A e a porção UnaG é fundida a uma tag FLAG de 3x. Clique aqui para ver uma versão maior desta figura.

Figura 2: Sistema genético reverso rotavírus. As monocamadas BHK-T7 são transfeccionadas com plasmídeos de 11 pT7, cada uma expressando um SA11 (+)RNA diferente, e um vetor pCMV expressando a enzima de capping NP868R do vírus da peste suína africana (ASFV). A 3 dias após a infecção (d.p.i.), as células BHK-T7 são supervisionadas com células MA104. Aos 7 dias após a infecção, os rotavírus recombinantes em lisatos celulares BHK-T7/MA104 são amplificados pela passagem em células MA104, depois isolados pela purificação da placa. Clique aqui para ver uma versão maior desta figura.
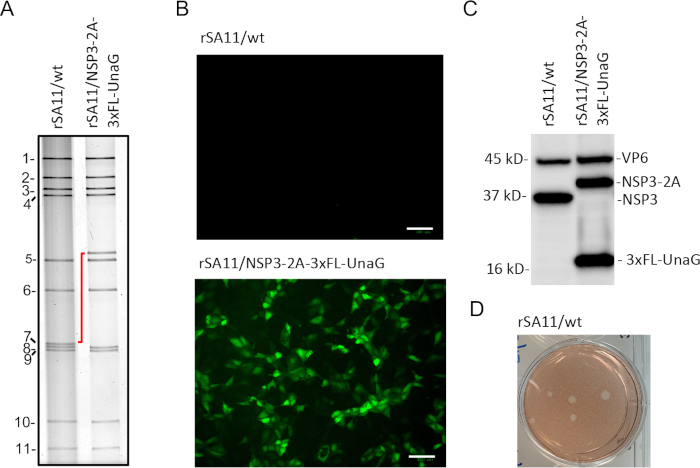
Figura 3: Características das cepas recombinantes rSA11/wt e rSA11/NSP3-2A-3xFL-UnaG. ( A) Perfis eletroforéticos dos segmentos do genoma dsRNA de cepas rSA11 isoladas por placas. Os 11 segmentos do genoma estão numerados e a mudança na posição do segmento 7 (NSP3) é indicada com uma linha vermelha. (B) Fluorescência detectada em células MA104 infectadas por rSA11 a 8 h após a infecção usando um imager celular vivo (ampliação de 20x) definido no canal de detecção verde. Barra de escala = 100 μm. (C) Análise de imunoblot de proteínas presentes a 8 h pós-infecção em células MA104 infectadas com cepas rSA11 utilizando cobaia anti-VP6 e anti-anti-nsp3 anti-nsp3 e anticorpo monoclonal anti-FLAG do rato. (D) Placas produzidas por rSA11/wt em células MA104 a 3 dias após a infecção e detectadas por coloração vermelha neutra. Clique aqui para ver uma versão maior desta figura.
Discussão
Em nosso laboratório, contamos rotineiramente com o protocolo genético reverso descrito aqui para produzir rotavírus SA11 recombinantes. Com essa abordagem, indivíduos com pouca experiência em técnicas de biologia molecular ou trabalhando com rotavírus recuperam vírus recombinantes mesmo em sua primeira tentativa. Geramos cerca de 100 vírus recombinantes seguindo este protocolo, incluindo aqueles com genomas que foram reprojetados para expressar proteínas estrangeiras (por exemplo, FPs) e que contêm adições de seqüência, exclusões e mutações pontuais.
As condições e os tempos de incubação dados neste protocolo aplicam-se à recuperação de cepas de bem crescimento de vírus recombinantes. Os ajustes devem ser considerados se a tentativa de recuperar os vírus SA11 que, devido à modificação genética, pode crescer mal. Em particular, como o título desses vírus em lysates celulares transfeccionados BHK-T7/MA014 pode ser baixo, normalmente dobramos a quantidade de lysate usada como inóculo em etapas de amplificação subseqüentes. Além disso, na etapa de amplificação, vírus de baixo crescimento podem exigir tempos mais longos de incubação antes de atingir níveis de CPE suficientes para a colheita celular. De fato, com esses vírus, podemos permitir que as infecções prossigam por 10-14 dias, ou até mais, antes de colher as células. Finalmente, vírus de baixo crescimento provavelmente gerarão placas pequenas e de crescimento lento em células MA104. Assim, para aplacar esses vírus, pode ser necessário permitir que as placas se desenvolvam até 6-10 dias após a infecção antes de manchar células com vermelho neutro e placas de colheita.
Em nossa experiência, o fator mais importante na recuperação confiável do rotavírus recombinante é o uso de células BHK-T7 saudáveis e bem conservadas. Em nosso laboratório, rotineiramente passamos células BHK-T7 2x por semana na mesma diluição usando meio suplementado não só com 10% de FBS, mas também com NEAA, TPB e altos níveis de glicose (meio completo GMEM). Os suplementos adicionais ajudam as células BHK-T7 a manter a viabilidade estendida após a transfecção plasmídea, um fator provavelmente crucial para a recuperação de vírus mutantes de crescimento ruim. Complementamos o meio todas as outras passagens com G418, um antibiótico que seleciona para manutenção da expressão de polimerase T7 plasmid. As condições de passagem devem ser tais que as células BHK-T7 nunca são permitidas a crescer além da confluência, condições que rapidamente levam à diminuição da viabilidade celular. Para nós, as células BHK-T7 que foram autorizadas a crescer demais têm um desempenho ruim no protocolo genético reverso, mesmo que as células sejam posteriormente perversas apropriadamente várias vezes. Em vez de tentar reabilitar células BHK-T7 crescidas, reiniciamos a linhagem com células previamente armazenadas em nitrogênio líquido.
A contaminação por mycoplasma das células BHK-T7 e MA104 pode ser um fator importante na falha do sistema genético reverso do rotavírus para gerar vírus recombinante. Em nosso laboratório, usamos um kit de detecção de mycoplasma baseado em PCR(Table of Materials)para verificar a contaminação das linhas celulares e, quando detectado, é mais frequentemente associado às nossas células BHK-T7. Não tentamos curar linhas celulares de micoplasma contaminante, em vez de restabelecer as linhas com passagens anteriores sem mycoplasma armazenadas em nitrogênio líquido. Antes de iniciar novas linhas celulares, descartamos todos os suplementos médios e médios usados anteriormente e descontaminamos completamente incubadoras, armários de segurança biológica, banhos de água, bancos de laboratório e pipettors. Também usamos kits de detecção de mycoplasma para verificar estoques de vírus recombinantes para contaminação. Devido à resistência das partículas rotavírus à desnaturação por solventes orgânicos, como o Vertrel VF32,é possível livrar os estoques de vírus da contaminação do mycoplasma, negando a necessidade de regenerar vírus recombinantes por genética reversa. É importante ressaltar que as linhas celulares e as preparações de vírus recebidas em laboratório devem ser verificadas para contaminação por mycoplasma antes do uso rotineiro.
Apesar de dia após dia, usamos o mesmo protocolo para gerar vírus recombinantes, sabemos que certas modificações podem ser feitas que não impedirão a recuperação do vírus. Por exemplo, (i) a cofecção da enzima de capping pCMV/NSP868R com vetores SA11 pT7 não é necessária para a recuperação de vírus recombinantes. Embora a adição do plasmídeo de capping produza títulos de vírus mais elevados em lysates celulares BHK-T7/MA104 transfeccionados, temos sido capazes de recuperar inúmeros vírus, incluindo aqueles que expressam FPs, sem ele. No entanto, concluímos que a expressão da enzima de capping pelo pCMV/NP868R pode contribuir significativamente para a recuperação de vírus menos aptos. (ii) Descobrimos que os vírus recombinantes podem ser gerados mesmo que a quantidade do reagente de transfecção (Tabela de Materiais) usada no protocolo de genética reversa seja reduzida pela metade, uma modificação que pode reduzir significativamente os gastos. (iii) Da mesma forma, determinamos que o meio completo M199 pode ser usado no lugar do meio completo Do MMEM. (iv) Finalmente, não há um requisito definido sobre o tipo de espinha dorsal vetorial que deve ser usado na produção de vetores de transcrição SA11 T7. Enquanto o CDNA viral no plasmídeo é cercado por um promotor T7 a montante e um exterminador hdv ribozyme e T7 a jusante, o plasmídeo pode ser esperado para suportar a recuperação do vírus recombinante. Notavelmente, vetores baseados em pGEM, pBluescript e pUC têm sido usados com sucesso no sistema genético reverso.
Divulgações
Os autores não têm nada para revelar.
Agradecimentos
Este trabalho foi apoiado pelas bolsas NIH R03 AI131072 e R21 AI144881, Indiana University Start-Up Funding, e lawrence M. Blatt Endowment. Agradecemos aos membros do laboratório Rotahoosier da UI, Ulrich Desselberger e Guido Papa por suas muitas contribuições e sugestões no desenvolvimento do protocolo genético reverso.
Materiais
| Name | Company | Catalog Number | Comments |
| Baby Hamster Kidney - T7 RdRP (BHK-T7) Cells | Contact: ubuchholz@niaid.nih.gov | ||
| Bio-Rad 8-16% Tris-Glycine Polyacrylamide Mini-Gel | Bio-Rad | 45608105 | |
| Cellometer AutoT4 viable cell counter | Nexcelom | ||
| ChemiDoc MP Gel Imaging System | Bio-Rad | ||
| Chloroform | MP | 194002 | |
| Clarity Western Enhanced Chemiluminescence (ECL) Substrate | Bio-Rad | 170-5060 | |
| Competent E.coli DH5alpha Bacteria | Lucigen | 60602-2 | |
| Complete Protease Inhibitor | Pierce | A32965 | |
| Disposable Transfer Pipettes, Ultrafine Extended Tips | MTC Bio | P4113-11 | |
| Dulbecco's Modified Eagle Medium (DMEM) | Lonza | 12-604F | |
| Eagle's Minimal Essential Medium, 2x (2xEMEM) | Quality Biological | 115-073-101 | |
| Ethanol, Absolute (200 proof) | Fisher Bioreagents | BP2818-500 | |
| Ethidium Bromide Solution (10 mg/ml) | Invitrogen | 15585-011 | |
| Fetal Bovine Serum (FBS) | Corning | 35-010-CV | |
| Fetal Bovine Serum (FBS), Heat Inactivated | Corning | 35-011-CV | |
| Flag M2 Antibody, Mouse Monoclonal | Sigma-Aldrich | F1804 | |
| GenEluate HP Plasmid Midiprep Kit | Sigma | NA0200-1KT | |
| Geneticin (G-418) | Invitrogen | 10131-027 | |
| Gibco FluroBrite DMEM | ThermoFisher | A1896701 | DMEM with low background fluorescence |
| Glasgow Minimal Essential Medium (GMEM) | Gibco | 11710-035 | |
| Goat Anti-Rabbit IgG, Horseradish Peroxidase (HRP) Conjugated | Cell-Signaling Technology | 7074S | |
| Guinea Pig Anti-NSP3 Antiserum | Patton lab | lot 55068 | |
| Guinea Pig Anti-VP6 Antierum | Patton lab | lot 53963 | |
| Horse Anti-Guinea Pig IgG, Horseradish Peroxidase (HRP) Conjugated | KPL | 5220-0366 | |
| Horse Anti-Mouse IgG, Horseradish eroxidase (HRP) Conjugated | Cell-Signaling Technology | 7076S | |
| iNtRON Biotechnology e-Myco Mycoplasma PCR Detection Kit | JH Science | 25235 | |
| Isopropyl alcohol | Macron | 3032-02 | |
| L-glutamine Solution (100x) | Gibco | 25030-081 | |
| Luria Agar Powder (Miller's LB Agar) | RPI research products | L24020-2000.0 | |
| Medium 199 (M199) Culture Medium | Hyclone | Sh30253.01 | |
| Minimal Essential Medium -Eagle Joklik's Forumation (SMEM) | Lonza | 04-719Q | |
| Monkey Kidney (MA104) Cells | ATCC | ATCC CRL-2378.1 | |
| NanoDrop One Spectrophotometer | ThermoScientific | ||
| Neutral Red Solution (0.33%) | Sigma-Aldrich | N2889-100ml | |
| Non-Essential Amino Acid Solution (100x) | Gibco | 11140-050 | |
| Novex 10% Tris-Glycine Polyacrylamide Mini-Gel | Invitrogen | XP00102BOX | |
| Nuclease-Free Molecular Biology Grade Water | Invitrogen | 10977-015 | |
| NucleoSpin Gel and PCR Clean-Up Kit | Takara | 740609.25 | |
| Opti-MEM Reduced Serum Medium | Gibco | 31985-070 | |
| Pellet pestle (RNase-free, disposable) | Fisher | 12-141-368 | |
| Penicillin-Streptomycin Solution, (100x penn-strep) | Corning | 30-002-Cl | |
| Phosphate Buffered Saline (PBS), 10x | Fisher Bioreagents | BP399-20 | |
| Porcine Trypsin, Type IX-S | Sigma-Aldrich | T0303 | |
| PureYield Plasmid Miniprep System | Promega | A1223 | |
| Qiagen Plasmid Maxi Kit | Qiagen | 12162 | |
| Qiagen Plasmid Midi Kit | Qiagen | 12143 | |
| QIAprep Spin Miniprep Kit | Qiagen | 27104 | |
| SA11 pT7 Transcription Vectors | Addgene | 89162-89172 | |
| SA11 pT7/NSP3 Transcription Vectors Expressing Fluorescent Proteins | Contact: jtpatton@iu.edu | ||
| SeaKem LE Agarose | Lonza | 50000 | For gel electrophoresis |
| SeaPlaque agarose | Lonza | 50100 | For plaque assay |
| Superscript III One-Step RT-PCR kit | Invitrogen | 12574-035 | |
| Trans-Blot Turbo Nitrocellulose Transfer Kit | Bio-Rad | 170-4270 | |
| Trans-Llot Turbo Transfer System | Bio-Rad | ||
| TransIT-LTI Transfection Reagent | Mirus | MIR2306 | |
| Tris-Glycine-SDS Gel Running Buffer (10x) | Bio-Rad | 161-0772 | |
| Triton X 100 | Fisher Bioreagents | BP151-500 | |
| Trizol RNA Extraction Reagent | Ambion | 15596026 | |
| Trypan blue | Corning | 25-900-CI | |
| Trypsin (0.05%)-EDTA (0.1%) Cell Dissociation Solution | Quality Biological | 118-087-721 | |
| Tryptose Phosphate Broth | Gibco | 18050-039 | |
| Tween-20 | VWR | 0777-1L | |
| Vertrel VF solvent | Zoro | G0707178 | |
| Zoe Fluorescent Live Cell Imager | Bio-Rad |
Referências
- Crawford, S. E., et al. Rotavirus infection. Nature Reviews Disease Primers. 3 (17083), 1-16 (2017).
- Settembre, E. C., Chen, J. Z., Dormitzer, P. R., Grigorieff, N., Harrison, S. C. Atomic model of an infectious rotavirus particle. The EMBO Journal. 30 (2), 408-416 (2011).
- International Committee on Taxonomy of Viruses. Taxonomic information. Virus taxonomy: 2018b release. , Available from: https://talk.ictvonline.org/taxonomy (2019).
- Tate, J. E., Burton, A. H., Boschi-Pinto, C., Parashar, U. D. World Health Organization-Coordinated Global Rotavirus Surveillance Network. Global, regional, and national estimates of rotavirus mortality in children <5 years of age. Clinical Infectious Diseases. 62, Suppl 2 96-105 (2016).
- Troeger, C., et al. Rotavirus vaccination and the global burden of rotavirus diarrhea among children younger than 5 years. JAMA Pediatrics. 172 (10), 958-965 (2018).
- Matthijnssens, J., Van Ranst, M. Genotype constellation and evolution of group A rotaviruses infecting humans. Current Opinion in Virology. 2 (4), 426-433 (2012).
- McDonald, S. M., Nelson, M. I., Turner, P. E., Patton, J. T. Reassortment in segmented RNA viruses: mechanisms and outcomes. Nature Reviews Microbiology. 14 (7), 448-460 (2016).
- Patton, J. T. Rotavirus diversity and evolution in the post-vaccine world. Discovery Medicine. 13 (68), 85-97 (2012).
- Kanai, Y., et al. Entirely plasmid-based reverse genetics system for rotaviruses. Proceedings of the National Academy of Sciences of the United States of America. 114 (9), 2349-2354 (2017).
- Trask, S. D., McDonald, S. M., Patton, J. T. Structural insights into the coupling of virion assembly and rotavirus replication. Nature Reviews Microbiology. 10 (3), 165-177 (2012).
- Guglielmi, K. M., McDonald, S. M., Patton, J. T. Mechanism of intraparticle synthesis of the rotavirus double-stranded RNA genome. Journal of Biological Chemistry. 285 (24), 18123-18128 (2010).
- Komoto, S., et al. Generation of recombinant rotaviruses expressing fluorescent proteins by using an optimized reverse genetics system. Journal of Virology. 92 (13), 00588-00618 (2018).
- Trask, S. D., Taraporewala, Z. F., Boehme, K. W., Dermody, T. S., Patton, J. T. Dual selection mechanisms drive efficient single-gene reverse genetics for rotavirus. Proceedings of the National Academy of Sciences of the United States of America. 107, 18652-18657 (2010).
- Fabbretti, E., Afrikanova, I., Vascotto, F., Burrone, O. R. Two non-structural rotavirus proteins, NSP2 and NSP5, form viroplasm-like structures in vivo. Journal of General Virology. 80, 333-339 (1999).
- Eichwald, C., Rodriguez, J. F., Burrone, O. R. Characterization of rotavirus NSP2/NSP5 interactions and the dynamics of viroplasm formation. Journal of General Virology. 85, 625-634 (2004).
- Kawagishi, T., et al. Reverse genetics system for a human group A rotavirus. Journal of Virology. , (2019).
- Komoto, S., et al. Generation of infectious recombinant human rotaviruses from just 11 cloned cDNAs encoding the rotavirus genome. Journal of Virology. 93 (8), 02207-02218 (2019).
- Mohanty, S. K., et al. A point mutation in the rhesus rotavirus VP4 protein generated through a rotavirus reverse genetics system attenuates biliary atresia in the murine model. Journal of Virology. 91 (15), 00510-00517 (2017).
- Navarro, A., Trask, S. D., Patton, J. T. Generation of genetically stable recombinant rotaviruses containing novel genome rearrangements and heterologous sequences by reverse genetics. Journal of Virology. 87, 6211-6220 (2013).
- Duarte, M., et al. Rotavirus infection alters splicing of the stress-related transcription factor XBP1. Journal of Virology. 93 (5), 01739-01818 (2019).
- Philip, A. A., et al. Generation of recombinant rotavirus expressing NSP3-UnaG fusion protein by a simplified reverse genetics system. Journal of Virology. , 1616-1619 (2019).
- Papa, G., et al. Recombinant rotaviruses rescued by reverse genetics reveal the role of NSP5 hyperphosphorylation in the assembly of viral factories. Journal of Virology. , (2019).
- Komoto, S., et al. Reverse genetics system demonstrates that rotavirus nonstructural protein NSP6 is not essential for viral replication in cell culture. Journal of Virology. 91 (21), 00695-00717 (2017).
- Philip, A. A., et al. Collection of recombinant rotaviruses expressing fluorescent reporter proteins. Microbiology Resource Announcements. 8 (27), 00523-00619 (2019).
- Kanai, Y., et al. Development of stable rotavirus reporter expression systems. Journal of Virology. , 01774-01818 (2019).
- Donnelly, M. L., et al. The 'cleavage' activities of foot-and-mouth disease virus 2A site-directed mutants and naturally occurring '2A-like' sequences. Journal of General Virology. 82, 1027-1041 (2001).
- Kumagai, A., et al. A bilirubin-inducible fluorescent protein from eel muscle. Cell. 153, 1602-1611 (2013).
- Rodriguez, E. A., et al. The growing and glowing toolbox of fluorescent and photoactive proteins. Trends in Biochemical Sciences. 42, 111-129 (2017).
- Buchholz, U. J., Finke, S., Conzelmann, K. K. Generation of bovine respiratory syncytial virus (BRSV) from cDNA: BRSV NS2 is not essential for virus replication in tissue culture, and the human RSV leader region acts as a functional BRSV genome promoter. Journal of Virology. 73, 251-259 (1999).
- Centers for Disease Control and Prevention (CDC). Biosafety in microbiological and biomedical laboratories (BMBL), 5th edition. , HHS publication no. CDC 21-1112 (2009).
- Arnold, M., Patton, J. T., McDonald, S. M. Culturing, storage, and quantification of rotaviruses. Current Protocols in Microbiology. 15 (1), (2009).
- Benureau, Y., Huet, J. C., Charpilienne, A., Poncet, D., Cohen, J. Trypsin is associated with the rotavirus capsid and is activated by solubilization of outer capsid proteins. Journal of General Virology. 86 (11), 3143-3151 (2005).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados