Method Article
Metodo di genetica inversa semplificato per recuperare rotavirus ricombinanti che esprimono proteine Reporter
In questo articolo
Riepilogo
La generazione di rotavirus ricombinanti dal DNA plasmide fornisce uno strumento essenziale per lo studio della replicazione e della patogenesi del rotavirus e per lo sviluppo di vettori e vaccini di espressione del rotavirus. Qui, descriviamo un approccio semplificato di genetica inversa per la generazione di rotavirus ricombinanti, compresi ceppi che esprimono proteine reporter fluorescenti.
Abstract
I rotavirus sono una grande e in continua evoluzione di virus segmentati dell'RNA a doppio filamento che causano una grave gastroenterite nei giovani di molte specie di mammiferi e aviari, compresi gli esseri umani. Con il recente avvento dei sistemi di genetica inversa del rotavirus, è diventato possibile utilizzare la mutagenesi diretta per esplorare la biologia del rotavirus, modificare e ottimizzare i vaccini esistenti sul rotavirus e sviluppare vettori di vaccino multitarget del rotavirus. In questa relazione, descriviamo un sistema di genetica inversa semplificato che consente il recupero efficiente e affidabile dei rotavirus ricombinanti. Il sistema si basa sulla co-trascrizione di vettori di trascrizione T7 che esprimono rotavirus a lunghezza intera e su un vettore CMV che codifica un enzima di aggancio dell'RNA nelle cellule BHK producendo constitutively produrre polimerasi T7 RNA (BHK-T7). I rotavirus ricombinanti sono amplificati sovrasseminando le cellule BHK-T7 trafette con cellule MA104, una linea cellulare renale delle scimmie che è altamente permissiva per la crescita del virus. In questo rapporto, descriviamo anche un approccio per generare rotavirus ricombinanti che esprimono una proteina reporter fluorescente separata attraverso l'introduzione di un elemento di stop-restart traslazionale 2A nel segmento 7 del genoma (NSP3). Questo approccio evita di cancellare o modificare uno qualsiasi dei fotogrammi di lettura aperti virali, consentendo così la produzione di rotavirus ricombinanti che mantengono proteine virali completamente funzionali e allo stesso tempo di esprimere una proteina fluorescente.
Introduzione
I rotavirus sono le principali cause di gastroenterite grave nei neonati e nei bambini piccoli, così come i giovani di molti altri mammiferi e specie aviarie1. Come membri della famiglia Reoviridae, i rotavirus hanno un genoma di RNA a doppio filamento (dsRNA) segmentato. I segmenti del genoma sono contenuti all'interno di un virione icosaedro non avvolto formato da tre strati concentrici di proteine2. Sulla base del sequenziamento e dell'analisi filogenetica dei segmenti del genoma, sono state definite nove specie di rotavirus (A, D, F,J)3. I ceppi che comprendono la specie a rotavirus A sono responsabili della stragrande maggioranza delle malattie umane4. L'introduzione di vaccini rotavirus nei programmi di immunizzazione infantile a partire dall'ultimo decennio è correlata a riduzioni significative della mortalità e morbilità del rotavirus. In particolare, il numero di decessi infantili associati al rotavirus è diminuito da circa 528.000 nel 2000 a 128.500 nel 20164,5. I vaccini Rotavirus sono formulati da ceppi vivi attenuati del virus, con 2 a 3 dosi somministrate ai bambini entro i 6 mesi di età. Il gran numero di ceppi di rotavirus geneticamente diversi che circolano negli esseri umani e in altre specie di mammiferi, combinato con la loro capacità di evolversi rapidamente attraverso la mutagenesi e il riassortimento, può portare a cambiamenti antigenici nei tipi di rotavirus che infettano i bambini6,7,8. Tali cambiamenti possono compromettere l'efficacia dei vaccini esistenti, richiedendone la sostituzione o la modifica.
Lo sviluppo di sistemi di genetica inversa completamente basati su plasmide che consentono la manipolazione di uno qualsiasi degli 11 segmenti del genoma del rotavirus è stato raggiunto solo di recente9. Con la disponibilità di questi sistemi, è diventato possibile svelare i dettagli molecolari della replicazione del rotavirus e della patogenesi, sviluppare migliori metodi di screening ad alto throughput per i composti anti-rotavirus e creare nuove classi potenzialmente più efficaci di vaccini rotavirus. Durante la replicazione del rotavirus, gli RNA con tappo non solo guidano la sintesi delle proteine virali, ma fungono anche da modelli per la sintesi dei segmenti del genoma della progenie dsRNA10,11. Tutti i sistemi di genetica inversa del rotavirus descritti fino ad oggi si basano sulla trasfezione dei vettori di trascrizione T7 nelle linee cellulari dei mammiferi come fonte di RNA derivati da cDNA , utilizzati per recuperare virus ricombinanti9,12,13. All'interno dei vettori di trascrizione, i cDNA virali a tutta lunghezza sono posizionati tra un promotore T7 a monte e il virus dell'epatite delta a valle (HDV) ribozyme in modo tale che gli RNA virali sono sintetizzati da polimerasi T7 RNA che contengono autentici 5' e 3 termini(Figura 1A). Nel sistema di genetica inversa di prima generazione, i virus ricombinanti sono stati fatti traducendo cellule renali del criceto per bambini che esprimono la polimerasi t7 RNA (BHK-T7) con 11 vettori di trascrizione T7 (pT7), ogni sintesi diretta di un unico (-)RNA del ceppo virus simian SA11, e tre plasmidi di espressione promotore-drive CMV, uno che codifica la proteina di fusione aviaria reovirus p10FAST e due sottounità di codifica del virus vaccini D1R-D12L filare enzima complesso9. I virus SA11 ricombinanti generati nelle cellule BHK-T7 trasfette sono stati amplificati dal semina con cellule MA104, una linea cellulare permissiva per la crescita del rotavirus. È stata descritta una versione modificata del sistema di genetica inversa di prima generazione che non utilizza più plasmidi di supporto12. Invece, il sistema modificato genera con successo rotavirus ricombinanti semplicemente traducendo le cellule BHK-T7 con i 11 vettori di trascrizione SA11 T7, con l'avvertenza che i vettori per la fabbrica virale (viroplasmo) blocchi costitutivi (proteine non strutturali NSP2 e NSP5) vengono aggiunti a livelli 3 volte superiori rispetto agli altri vettori14,15. Sono state sviluppate anche versioni modificate del sistema di genetica inversa che supportano il recupero dei ceppi umani KU e Odelia del rotavirus16,17. Il genoma del rotavirus è notevolmente suscettibile alla manipolazione da parte della genetica inversa, con virus ricombinanti generati fino ad oggi con mutazioni introdotte in VP418, NSP19, NSP219, NSP320,21e NSP522,23. Tra i virus più utili generati finora ci sono quelli che sono stati progettati per esprimere proteine reporter fluorescenti (FP)9,12,21,24,25.
In questa pubblicazione, forniamo il protocollo per il sistema di genetica inversa che usiamo nel nostro laboratorio per generare ceppi ricombinanti del rotavirus SA11. La caratteristica chiave del nostro protocollo è la co-transfezione delle cellule BHK-T7 con i vettori di trascrizione 11 pT7 (modificati per includere livelli 3x dei vettori pT7/NSP2SA11 e pT7/NSP5SA11) e unFigure 2vettore di espressione CMV che codifica il virus della peste suina africana (ASFV) NP868R).21 Nelle nostre mani, la presenza del plasmide NP868R porta alla produzione di più alti titer di virus ricombinanti da parte di cellule BHK-T7 trafette. In questa pubblicazione, forniamo anche un protocollo per modificare il pLasmide pT7/NSP3SA11 in modo che possano essere generati virus ricombinanti che esprimono non solo il prodotto proteico segmento 7 NSP3 ma anche un FP separato. Questa operazione viene eseguita ri-ingegnerizzando il frame di lettura aperto NSP3 (ORF) nel plasmid pT7/NSP3SA11 per contenere un elemento downstream 2A di arresto traslazionale 2A seguito da un FP ORF (Figura 1B)24,26. Attraverso questo approccio, abbiamo generato rotavirus ricombinanti che esprimono vari FP: UnaG (verde), mKate (molto rosso), mRuby (rosso), TagBFP (blu), CFP (ciano) e YFP (giallo)24,27,28. Questi rotavirus che esprimono FP sono fatti senza eliminare l'OrF NSP3, producendo così virus che dovrebbero codificare un completo complemento di proteine virali funzionanti.
Protocollo
1. Preparazione dei supporti e manutenzione della coltura cellulare
- Ottenere cellule renali di criceto bambino che esprimono le cellule del rene T7 RNA (BHK-T7) e il rene della scimmia verde africana MA104.
NOTA: Le cellule BHK-T7 (o BSR-T7) non sono disponibili in commercio, ma sono una linea cellulare comune di laboratori che utilizzano la genetica inversa per studiare la biologia del virus dell'RNA. La linea cellulare BHK-T7 utilizzata in questo protocollo è stata ottenuta dalla Dott.ssa Ursula J. Buchholz (National Institutes of Health, Bethesda, MD, USA), co-sviluppatore della linea cellulare BHK originale che esprime la polimerasi T7 RNA29. Le celle MA104 sono disponibili presso la European Collection of Authenticated Cell Cultures (ECACC) e dalla American Type Culture Collection (ATCC). - Preparare i seguenti supporti di coltura in un ambiente sterile, preferibilmente un mobile di sicurezza biologica di classe II. Conservare i supporti al buio a 4 gradi centigradi e riscaldare a 37 gradi centigradi immediatamente prima dell'uso.
NOTA: le fonti dei componenti multimediali sono fornite nella Tabella dei materiali.- Per preparare il mezzo incompleto DMEM, unire 500 mL del mezzo minimo essenziale dell'Aquila modificato di Dulbecco (MEM) contenente 4,5 g di glucosio e 1% glutammina con 5 mL di 100x pen-strep.
- Per preparare il supporto completo DMEM, combinare 500 mL di DMEM con un supporto incompleto di 25 mL di siero bovino fetale (FBS).
- Per preparare il supporto incompleto GMEM, combinare 500 mL di Glasgow mezzo minimo essenziale, 5 mL di 100x glutammina, 50 mL di brodo triptosi-fosfato (TPB), 5 mL di 100x aminoacidi non essenziali (NEAA), e 5 mL di 100x pen-strep.
- Per preparare il supporto completo GMEM, combinare 500 mL di supporto GMEM incompleto con 25 mL di FBS inattivati dal calore.
- Per preparare il supporto completo GMEM-G, aggiungere 10 mL di 50 mg/mL di genetica (G418) a 500 mL di GMEM mezzo completo.
- Per preparare il mezzo incompleto SMEM, combina 500 mL del MEM MODIFICATO di Eagle di Joklik, 5 mL di 100x glutammina, 50 mL di TPB, 5 mL di 100x NEAA e 5 mL di 100x pen-strep.
- Colture ma104 cellule in flaconi T75 o T175 contenenti 12 o 25 mL di DMEM mezzo completo, rispettivamente. Per passare le cellule MA104 che hanno raggiunto il 100% di confluenza, sciacquare il monomeoro cellulare 2 volte con salina tamponata di fosfati (PBS), dissociate con la trypsin (0,05%) -EDTA (0,1%) soluzione e risospendere in 5 (T75 fiaschetta) o 10 mL di (T175 fiaschetta) di supporto incompleto DMEM.
- Mettete in flisci da 0,5,0 ml di celle risospese e portate al volume finale appropriato aggiungendo il supporto completo DMEM. Collocare i flaconi in un'incubatrice di CO2 del 5% di 37 gradi centigradi.
- Propagare le cellule BHK-T7 in flaconi T75, alternando l'uso di GMEM e GMEM-G mezzo completo con ogni giro di passaggio. Per sottocultura le cellule BHK-T7 che hanno raggiunto il 100% di confluenza, risciacquare il monostrato cellulare 2x con PBS, dissociate con soluzione trypsin-EDTA e risospendere in 5 mL di media.
- Dopo aver posizionato 15 mL di GEM o GMEM-G mezzo completo in un pallone T75 fresco, aggiungere quattro gocce di cellule BHK-T7 risospese. Mettere il flacone in un'incubatrice di CO2 a 37 gradi centigradi.
2. Preparazione plasmide
- Ottenere i seguenti plasmidi che esprimono rlaavirus SA11 (z)RNA da Addgene: pT7/VP1SA11, pT7/VP2SA11, pT7/VP3SA11, pT7/VP4SA11, pT7/VP6SA11, pT7/VP7SA11, pT7/NSP1SA11, pT7/NSP2SA11, pT7/NSP3SA11, pT7/NSP4SA11 e pT7/N5SA111. Ottenere il plasmide esprimendo l'enzima di tappatura ASFV (pCMV/NP868R) dagli autori21.
NOTA: i plasmidi pT7/NSP3SA11 modificati progettati per esprimere un FP attraverso l'attività di un elemento traslazionale 2A (pT7/NSP3-2A-3xFL-FP) possono essere ottenuti anche dagli autori24,26. sono disponibili pT7/NSP3-2A-3xFL-FP che esprimono i seguenti FpP: UnaG (verde), mKate (di colore rosso), mRuby (rosso), TagBFP (blu), CFP (ciano) e YFP (giallo)24. - Trasforma i plasmidi in competenti E. coli e diffondere i batteri su piastre di agar brodo di Luria contenenti l'antibiotico appropriato. Coltivare colture batteriche raccolte da singole colonie e preparare piccole quantità di plasmide (20 g) utilizzando un kit di purificazione miniprep spin (Tabella dei materiali), secondo le istruzioni del produttore. Preparare grandi quantità di plasmide da colture batteriche utilizzando midi e maxi kit di purificazione (Tabella dei materiali).
NOTA: Plasmidi adatti all'uso nel sistema di genetica inversa sono stati preparati anche con altri kit di purificazione plasmide. Non è necessario preparare plasmidi utilizzando kit appositamente progettati per generare materiale privo di endotossina. - Regolare le concentrazioni di plasmidi purificati a 1 mg/mL in acqua di grado di biologia molecolare priva di nucleasi. Controllare la purezza del plasmide confermando un rapporto di assorbimento 260/280 di 1,8 dollari utilizzando uno spettrofotometro. Inoltre, utilizzare l'elettroforesi su 0.8% gel agarose per verificare che i plasmidi sono prevalentemente supercoiled.
- I plasmidi annidati in tubi di microcentrifuga sterili da 0,5 ml, ciascuno contenente 10 plasmidi di immagazzinamento a -80 gradi centigradi.
3. Generazione di virus ricombinante
NOTA: la ricerca sul rotavirus umano e animale, compresa la generazione e la caratterizzazione di ceppi di rotavirus ricombinante, deve essere gestita in condizioni di biosicurezza di livello 2 (BSL-2) e richiederà un'approvazione preventiva da parte del Comitato di biosicurezza istituzionale (IBC). Le condizioni di laboratorio BSL-2 appropriate sono descritte in Biosicurezza nei laboratori microbiologici e biomedici (BMBL) prodotti dai Centers for Disease Control and Prevention (CDC)30.
- Giorno 1: semina delle cellule BHK-T7 in piastre 12-well
- Sciacquare un monostrato appena confluente di cellule BHK-T7 contenute in un pallone T75 2x con PBS. Interrompere il monostrato cellulare con la soluzione trypsin-EDTA e risospendere le cellule in 5 mL di GMEM mezzo completo.
- Utilizzare un contatore cellulare automatizzato e una soluzione trypan-blu per determinare la concentrazione di cellule BHK-T7 vitali nel mezzo. Le cellule di semi in piastre di coltura cellulare 12-well, con ogni pozzo contenente 2 x 105 celle in un volume totale di 1 mL di GMEM (G418-free) mezzo completo. Incubare in un incubatore di 37 gradi centigradi, 5% di CO2. Le cellule devono raggiungere l'80-90% di confluenza entro il giorno 2.
- Giorno 2: trasfezione di cellule BHK-T7 con miscele di plasmide
- Per preparare la miscela di plasmide, unire quanto segue in un tubo di microcentrismo da 0,5 ml utilizzando supporti plasmici regolati a 1 mg/mL: 0,8 litri ciascuno di SA11 pT7 plasmidi VP1, VP2, VP3, VP4, VP6, VP7, NSP1, NSP3 e NSP4, 2,4 litri ciascuno di plasmids SA11 pT7 NSP2 e NSP5 e 0,8 L di pCMV/NP868R. Mescolare delicatamente i plasmidi toccando il tubo e raccogliere i contenuti mediante centrifugazione a impulsi. Per preparare virus ricombinanti che esprimono FP, sostituire pT7/NSP3SA11 con il pT7/NSP3-2A-3xFL-FP plasmide appropriato.
NOTA: Le miscele di plasmide devono essere conservate sul ghiaccio fino a quando non vengono utilizzate. Un tubo separato deve essere preparato per ogni trasfezione. - Per preparare la miscela reagente di plasmide/riduzione del siero medio/trasfezione: aggiungere 110 ll di preriscaldato (37 gradi centigradi) di mezzo siero ridotto (Tabella dei materiali) ad ogni miscela di plasmide e mescolare con delicatamente pipettando su e giù. Successivamente, aggiungere 32 L di reagente di trasfezione (Tabella dei materiali) ad ogni miscela media di plasmide/ridotto; questo produce una concentrazione di 2,5 litri di reagente di trasfezione per g di plasmide nelle miscele. Il vortice mescola delicatamente e incubare a temperatura ambiente per 20 min.
- Durante il periodo di incubazione di 20 minuti, sciacquare le cellule BHK-T7 in piastre 12-well (preparate il giorno 1) una volta con 2 mL di GMEM incompleto supporto. Successivamente, aggiungere 1 mL di SMEM mezzo incompleto ad ogni pozzo e restituire piastre all'incubatrice.
- Dopo il periodo di incubazione di 20 min, aggiungere la miscela di plasmide/ridotto medio/trasversale goccia per goccia a ciascun pozzetto delle piastre da 12 pozze utilizzando un pipettorale da 200 luna. Piastre rocciose delicatamente e ritorno a un incubatore di CO2 del 57% e del 5%.
- Per preparare la miscela di plasmide, unire quanto segue in un tubo di microcentrismo da 0,5 ml utilizzando supporti plasmici regolati a 1 mg/mL: 0,8 litri ciascuno di SA11 pT7 plasmidi VP1, VP2, VP3, VP4, VP6, VP7, NSP1, NSP3 e NSP4, 2,4 litri ciascuno di plasmids SA11 pT7 NSP2 e NSP5 e 0,8 L di pCMV/NP868R. Mescolare delicatamente i plasmidi toccando il tubo e raccogliere i contenuti mediante centrifugazione a impulsi. Per preparare virus ricombinanti che esprimono FP, sostituire pT7/NSP3SA11 con il pT7/NSP3-2A-3xFL-FP plasmide appropriato.
- Giorno 4: sovrasedendo cellule BHK-T7 trasfette con cellule MA104
- Sciacquare un monoteman appena confluente di cellule MA104 contenute in un pallone T75 2x con PBS. Interrompere il monostrato utilizzando la soluzione trypsin-EDTA e rimettere in sospensione le cellule in 5 mL di DMEM mezzo completo.
- Utilizzando un contatore cellulare automatizzato e una soluzione trypan-blu, determinare la concentrazione di cellule MA104 vitali nel mezzo. Regolare la concentrazione a 8 x 105 cellule MA104 / mL nel mezzo incompleto DMEM e aggiungere 0,25 mL di cellule sospese (2 x 105 cellule) dropwise ai pozzi contenenti cellule BHK-T7 trasfette.
- Regolare la concentrazione di orpina (tipo pancreatico porcino IX) nel mezzo a 0,5 g/mL aggiungendo a ogni pozzo 0,8 .l di 1 00/mL di brodo di 1 mg/mL.
NOTA: le scorte di Trypsin devono essere preparate in PBS, aliquote e conservate a -20 gradi centigradi. - Utilizzare le cellule MA104 rimanenti per preparare piastre a 6 piani che saranno necessarie il giorno 7 per amplificare i virus ricombinanti. Per seme 6-popla, diluire rielutta cellule MA104 ad una concentrazione di 1,5 x 105 cellule / mL in DMEM completano il mezzo e posizionare 2 mL in ogni pozzo. Collocare le piastre in un incubatore di CO2 del 5% di C.
- Giorno 7: recupero e amplificazione del virus ricombinante da cellule trasfette
- Soggetto BHK-T7/MA104 cellule in piastre 12-well a tre cicli di congelamento-scongelamento in condizioni sterili, piastre mobili tra -20 gradi congelatore e una superficie di temperatura ambiente. Dopo aver trasferito i lismi ai tubi da 1,5 mL, centrifugare i tubi per 10 min a 500 x g (4 gradi centigradi) per pellet recidiferi cellulari di grandi dimensioni. Raccogliere supernatali e conservare a 4 gradi centigradi (a breve termine) o -20 gradi centigradi (a lungo termine).
- Lavare i monostrati MA104 in piastre a 6 pozze (preparate il giorno 4) 2x con PBS. Posizionare 2 mL di DMEM incompleto medio per ogni pozzo che contiene 0,5 g/mL trypsin. Aggiungere 300 l di supernatante recuperato da BHK-T7/MA104 lisa riesci a essere cella in pozzetti e posizionare le piastre in un incubatore di CO2 a 37 gradi centigradi.
- Incubare piastre per 7 giorni o fino a quando si osservano effetti citopatici completi (CPE). Lyse MA104 cellule in piastre 6-well da tre cicli di congelare-scongelamento, quindi trasferire lisati a tubi di microcentrifuga 1,5 mL. Pellet grandi detriti cellulari per centrifugazione per 10 min a 500 x g (4 gradi centigradi). Trasferire le lingue chiarificate in tubi di microcentrifuga da 1,5 ml e conservarli a -20 gradi centigradi.
4. Isolamento della placca di virus ricombinanti
- Attivare i virus in 100 - l di liselli cellulari chiarificati aggiungendo trippsina ad una concentrazione finale di 10 g/mL e incubando a 37-1 gradi centigradi per 1 h. Preparare una serie di diluizione seriale da 10-1 a 10-7 (1 mL ciascuno) nel mezzo incompleto DMEM.
- Sciacquare i monostrati MA104 in piastre a 6 pozze 2x con 2 mL di PBS e una volta con un supporto incompleto DMEM. Aggiungere 400 mL di diluizioni lisate in duplicati alle piastre. Incubare le piastre per 1 h in un incubatore di 37 gradi, 5% DI CO2, dondolando ogni 10-15 min per ridistribuire le diluizioni sul monostrato.
- Preparare una soluzione di sovrapposizione agarose-MEM combinando volumi uguali del mezzo essenziale minimo 2x Eagle (EMEM; preriscaldato a 37 gradi centigradi) con l'1,5% di agarose che è stato fuso in acqua utilizzando un forno a microonde e preraffreddato a 45 gradi centigradi. Mantenere la soluzione di sovrapposizione a 42 gradi centigradi utilizzando un bagno d'acqua e regolare ad una concentrazione finale di 0,5 g/mL trypsin immediatamente prima di posizionare sulle cellule.
- Estrarre le diluizioni lisate da piastre a 6 pozze, quindi risciacquare le cellule una volta con 2 mL di DMEM incompleto. Sovrappone delicatamente 3 mL della soluzione di sovrapposizione agarose-MEM sul monostrato cellulare contenuto in ogni pozzo. Lasciare che la sovrapposizione di agarose si indurisca a temperatura ambiente, quindi riportare le piastre all'incubatrice.
- Tre giorni dopo, preparare una soluzione di overlay agarose-MEM come nel passaggio 4.3 e portare a 42 gradi centigradi. Immediatamente prima dell'uso, regolare la soluzione di sovrapposizione a una concentrazione finale di 50 g/mL di rosso neutro (Tabella dei materiali).
- Aggiungete 2 mL della soluzione di sovrapposizione sopra lo strato di agarose esistente nelle piastre a 6 pozze. Dopo aver permesso al nuovo strato di agarose di indurirsi, riportare le piastre all'incubatrice. Proteggere le soluzioni e le piastre che contengono il rosso neutro dall'esposizione alla luce.
- Nei successivi 6 h, identificare le placche del rotavirus nelle piastre 6-pozzo con l'aiuto di una scatola luminosa. Scegli placche chiaramente definite utilizzando pipetta di trasferimento usa e getta, recuperando tappi di agarose che si estendono completamente allo strato cellulare.
- Espellere il tappo in un tubo da 1,5 mL contenente 0,5 mL di DMEM incompleto medio e vortice il campione per 30 s. Amplificare il virus isolato dalla placca esoluto nel mezzo per propagazione su monostrati MA104 in piastre a 6 pozzetti o flaconi T25 contenenti mezzo incompleto DMEM e 0,5 g/mL trypsin.
NOTA: Ulteriori informazioni sull'isolamento e il titering del rotavirus per saggio placca sono disponibili in Arnold et al.31.
5. Elettroforesi gel di dsRNA virale
- Mettete 600 lunati con cellule infette chiarie e 400 luna di tiocianato di guanidio in tubi di microcentrifuga da 1,5 mL, vortice per 30 s, e incubate a temperatura ambiente per 5 min. Dopo la centrifuga per 5 min a 13.000 x g (4 gradi centigradi), trasferire 550 dollari di fase acquosa superiore in tubi freschi.
- Recupera dsRNA virale dalla fase acquosa aggiungendo 2 volumi (900 dollari ll) di alcool isopropile freddo e tubi invertiti 4/6x. Dopo aver incubato a temperatura ambiente per 10 min, centrifugare i tubi per 10 min a 13.000 x g (4 gradi centigradi). Eliminare i supernatanti e trattenere i pellet di RNA.
- Lavare i pellet aggiungendo 1 mL del 75% di etanolo al tubo, invertendo una volta e centrifugeno per 5 min a 7.500 x g (4 gradi centigradi). Dopo aver rimosso con attenzione il lavaggio dell'etanolo con un pipettor, lasciare che i pellet di RNA si asciughino sul banco di laboratorio per un valore di 5-10 min.
- Unire 10 l di campioni di RNA disciolto con 2 luna di 6 x buffer di carico del DNA, caricare su prefabbricati 10% di gel poliacrilamide (o equivalenti lanciati a mano) e risolvere gli RNA mediante elettroforesi nel buffer di corsa Tris-glicina per 2 h sotto una corrente costante (16 mA). I gel per un'acqua di immersione per 5/10 min di acqua che contengono 1 bromuro di etidio di g/mL e rilevano i segmenti del genoma del rotavirus con un transilluminatore UV.
6. Recupero e sequenziamento del dsRNA virale
- Individuare bande di dsRNA virale su gel di poliacrilammide preferibilmente utilizzando la luce UV a lunga lunghezza d'onda a bassa intensità, per evitare di generare collegamenti a acido nucleico. Utilizzando una lama di rasoio fresca, ritagliare frammenti di gel contenenti bande di dsRNA e trasferirli in tubi di microcentrifuga da 1,5 mL.
- Dopo aver aggiunto 10-20 l di acqua senza RNase, schiacciare ogni frammento con un pestello pellet usa e getta senza RNase progettato per adattarsi a un tubo di microcentrifuga da 1,5 ml o disegnando su e giù attraverso un ago da 18 G. Incubare frammenti schiacciati durante la notte a 4 gradi centigradi.
- Recuperare 3 -L di liquido da tubi contenenti frammenti di gel schiacciati. Generare cDNA dei dsRNA virali utilizzando un kit di reazione a catena di trascrizione inversa in un passaggio (RT-PCR) (Tabella dei materiali) e i primer oligonucleotidi appropriati.
NOTA: i prodotti PCR vengono purificati in gel con un kit di pulizia PCR (Table of Materials) e inviati per il sequenziamento notturno, insieme ai primer, dai servizi commerciali di sequenza del DNA.
7. Analisi immunoblot delle proteine virali
- Piastre di semi 6 pozzetti con 3 x 105 celle MA104 per pozzo in un volume totale di 2 mL di DMEM mezzo completo. Collocare le piastre in un incubatore di 37 gradi centigradi, il 5% di CO2 e lasciare fino a quando le cellule non raggiungono la confluenza (3-5 giorni).
NOTA: I pozzi con monostrati confluenti conterranno 1,2 x 106 celle. - Trattare i supernatanti chiarificati incubando con 10 g/mL di metapsina a 37 gradi centigradi per 1 h per attivare particelle di rotavirus ricombinante contenute al loro interno.
- Sciacquare i monostrati MA104 in piastre a 6 pozze 2x con 2 mL di PBS. Infettare le cellule aggiungendo, in ogni pozzo, 200 L di inoculo contenente unità di formazione di placca 3-5 (PFU) per cellula del rotavirus incompleto della triessipina nel mezzo incompleto del DMEM. Riportare le piastre all'incubatrice.
- Ogni 10 min, rimuovere piastre e delicatamente roccia per ridistribuire l'inoculum attraverso il monostrato cellulare. Dopo 1 h, sostituire inoculum con 2 mL di DMEM incompleto medio.
- A 8 h dopo l'infezione, risciacquare le cellule in piastre 6-well 2x con PBS. Le cellule di raschiare in 750 L di PBS e il volume di trasferimento in un tubo di microcentrifuga da 1,5 mL. Sciacquare la piastra con altri 750 l di PBS e combinarla con il precedente campione raccolto 750 L.
- Cellule di pellet nel campione centrifugando a 5.000 x g per 10 min a 4 gradi centigradi. Dopo aver rigettato i supernatali, conservare i pellet cellulari a -80 gradi centigradi fino a quando non vengono elaborati ulteriormente.
- Preparare il buffer di lisi cellulare contenente 300 mM NaCl, 100 mM Tris-HCl, pH 7,4, 2% Triton X-100 e 1x inibitore della proteasi completa senza EDTA. Dopo aver aggiunto 300 l di lis tampone a pellet di cellule congelate, brevemente campioni di vortice e incubare sul ghiaccio per 10 min.
- Ripetere il processo di vortice e incubazione dei campioni sul ghiaccio 3x. Successivamente, centrifugare campioni a 15.000 x g per 10 min a 4 gradi centigradi, quindi raccogliere i supernatanti e conservare a -80 gradi centigradi.
- Risolvere le proteine contenute in 20 volumi di campioni di supernatante mediante elettroforesi su mini gel poliacrilamina timore l'8-16% di poliacrilamina e il trasferimento nelle membrane di nitrocellulosa. Membrane di blocco mediante incubazione con PBS-Tween 20 (0,02%) soluzione contenente il 5% di latte secco non grasso.
- Membrane sonda mediante incubazione di uno o più anticorpi primari (ad esempio, anti-NSP3 o antisera anti-VP6, anticorpo m2 monoclonale del topo o anticorpo anti-PCNA monoclonale del coniglio). Rilevare gli anticorpi primari incubando membrane con IgG anti-topo di cavallo, IgG di maiale anti-guinea o gora anti-coniglio IgG rafano IgG perossidase-coniugato anticorpi secondari, seguita dalla maggiore chemiluminescenza (ECL) horseradish chemiluminescenza substrato. Visualizzare i segnali di luminescenza utilizzando un sistema di imaging gel (Tabella dei materiali) o pellicola a raggi X.
8. Immagini a cellule vive di cellule infettate da virus che esprimono FP
- Seme 6-pozze con 3 x 105 ma104 cellule per pozzo in un totale di 2 mL di DMEM mezzo completo. Collocare le piastre in un incubatore di 37 gradi centigradi, il 5% di CO2 e lasciare fino a quando le cellule non raggiungono la confluenza (3-5 giorni).
NOTA: I pozzi con monostrati confluenti conterranno 1,2 x 106 celle. - Attivare i campioni di rotavirus, di tibo noto, per incubazione con 10 g/mL di metapsina a 37 s per 1 h.
- Risciacquare 6 lastre con monostrati MA104 2x con 2 mL di PBS. Infettare le cellule aggiungendo ad ogni pozzo 200 -L di inoculo contenente 3/5 PFU per cellula di rotavirus incompleto con la trypsina nel mezzo incompleto del DMEM. Riportare le piastre all'incubatrice e le piastre di roccia delicatamente per ridistribuire l'inoculo sul monostrato cellulare ogni 10 min.
NOTA: I pozzi che sono simulati infettati con inoculum DMEM privo di virus devono essere inclusi come controlli. - Dopo 1 h di adsorbimenti del virus, risciacquare il monostrato 2x con PBS e sostituire il supporto con 0,5 mL per pozzo di DMEM incompleto medio. A 7,5 h dopo l'infezione, sostituire il mezzo di coltura con DMEM con bassa fluorescenza di fondo (Tabella dei materiali). A 8 h dopo l'infezione, utilizzare un imager di cellule vive per esaminare le cellule con ingrandimento 20x per il segnale di fluorescenza verde, rosso o blu.
Risultati
Il protocollo di genetica inversa descritto in questo articolo procede attraverso più passaggi distinti: (1) co-trasfezione di cellule BHK-T7 con vettori di trascrizione del rotavirus pT7 e pCMV/NP868R espressione plasmid, (2) semina di cellule BHK-T7 trafette con cellule MA104, (3) amplificazione dei virus ricombinanti presenti nelle cellule BHK-T7/MA104 che utilizzano cellule MA104 e (4) isolamento della placca del virus ricombinante utilizzando cellule MA104(Figura 2). Nelle nostre mani, il protocollo è efficiente, producendo titers del virus ricombinante wildtype SA11 (rSA11/wt) in BHK-T7/MA104 lisate cellulari di 104 PFU /mL e in lisati cellulari MA104 amplati di >1 x 107 PFU/mL. I virus ricombinanti SA11 generati dalla genetica inversa utilizzando plasmidi modificati di pT7/NSP3SA11 che esprimono FP (ad esempio, rSA11/NSP3-2A-3xFL-UnaG) crescono in titer che sono di 4 volte inferiori rispetto a rSA11/wt.
Seguendo il protocollo di genetica inversa, abbiamo generato virus SA11 ricombinanti che sono stati facilmente identificabili dal test della placca sulle cellule MA104, consentendo così l'isolamento della placca (Figura 3D). I virus nelle placche sono stati raccolti con un pipet di trasferimento usa e getta a punta lunga e amplificati sulle cellule MA104. I genomi dsRNA dei virus rSA11/wt e rSA11/NSP3-2A-3XFL-UnaG sono stati estratti con tiocianato di guanidio, risolto dall'elettroforesi su un gel poliacrilamina al 10% e rilevato dalla colorazione con broe etidiomico (3A). Come previsto, il dsRNA del segmento 7 (NSP3) di rSA11/NSP3-2A-3xFL-UnaG è migrato molto più lentamente di quello di rSA11/wt (Figura 3A) a causa della presenza di sequenze 2A-3xFL-UnaG. Il dsRNA del segmento 7 (NSP3) di rSA11/NSP3-2A-3xFL-UnaG è stato purificato in gel, convertito in cDNA da RT-PCR e sequenziato per confermare la precisione.
Per verificare l'espressione della proteina fluorescente UnaG, le cellule MA104 in piastre a 6 pozzetti sono state infettate da 3 PFU per cellula di rSA11/wt e rSA11/NSP3-2A-3xFL-UnaG. A 8 h dopo l'infezione, il mezzo di coltura nelle piastre è stato sostituito da 0,5 mL di DMEM con bassa fluorescenza di fondo per pozzo e le piastre incubate per ulteriori 30 min a 37 gradi centigradi in un'incubatrice di CO2. Successivamente, sono state esaminate le piastre per l'espressione UnaG utilizzando un imager di cellule vive. L'analisi ha mostrato che rSA11/NSP3-2A-3xFL-UnaG produceva fluorescenza verde, verificando la funzionalità del gene UnaG nel virus ricombinante (Figura 3B). Al contrario, la fluorescenza verde non è stata rilevata nelle cellule infettate da rSA11/wt. Per capire se l'elemento 2A nel segmento modificato 7 di rSA11/NSP3-2AxFL-UnaG ha promosso l'espressione di due proteine separate (NSP3-2A e 3xFL-UnaG), le cellule MA104 sono state infettate da rSA11/NSP3-2A-3xFL-UnaG e rSA11/wt. Le lisati cellulari sono state preparate da cellule raccolte a 8 h dopo l'infezione, risolte dall'elettroforesi gel e cancellate sui filtri di nitrocellulosa. Le macchie sono state sondate con anticorpi specifici per rotavirus VP6 e NSP3 e flag tag. L'analisi ha mostrato che NSP3-2A e 3xFL-UnaG erano espressi come proteine separate nelle cellule infettate da rSA11/NSP3-2A-3xFL-UnaG, indicando che l'elemento 2A era funzionale (Figura 3C). Le cellule infettate da rSA11/wt non hanno espresso proteine riconosciute dall'anticorpo anti-FLAG. La proteina NSP3 presente nelle cellule infettate da rSA11/NSP3-2A-3xFL-UnaG da anticorpi anti-NSP3 migrati leggermente più lentamente di NSP3 presenti nelle cellule rSA11/wt-infected a causa della presenza di residui residui 2A al Capolino C di NSP3.

Figura 1: Plasmidi genetica inversa del Rotavirus. (A) I cDNA a lunghezza intera degli 11 segmenti del genoma del rotavirus SA11 sono posizionati all'interno di plasmidi di pT7, si legano a monte con un promotore per la polimerasi t7 RNA e a valle con un ribozyme HDV. In presenza della polimerasi t7 RNA, i plasmidi dell'RNA tavirus pT7 producono SA11 (O) RNA a tutti gli effetti con autentici termini da 5' e 3'. (B) Schemi dei prodotti di RNA e proteine realizzati dal pT7/NSP3-2A-3xFL-UnaG plasmide. Lo schema include la sequenza cDNA NSP3 e sequenze per l'elemento di tipo porcine teschovirus 2A (2A), il tag 3x FLAG (FL) e la proteina fluorescente verde UnaG. La posizione del sito di arresto della traduzione 2A è indicata con una freccia rossa. A causa dell'attività dell'elemento 2A, la traduzione dell'RNA produce due proteine. La parte NSP3 contiene i resti dell'elemento 2A e la porzione UnaG viene fusa in un tag 3x FLAG. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 2: Sistema di genetica inversa Rotavirus. I monostrati BHK-T7 sono trasincati da plasmidi 11 pT7, ognuno dei quali esprime un diverso RNA SA11 e un vettore pCMV che esprime il virus della peste suina africano (ASFV) NP868R (pCMV/NP868R). A 3 giorni dopo l'infezione (d.p.i.), le cellule BHK-T7 sono sottoposte a seminato con cellule MA104. A 7 giorni dopo l'infezione, rotavirus ricombinanti in BHK-T7/MA104 lismi cellulari sono amplificati dal passaggio sulle cellule MA104, poi isolati per purificazione della placca. Fare clic qui per visualizzare una versione più grande di questa figura.
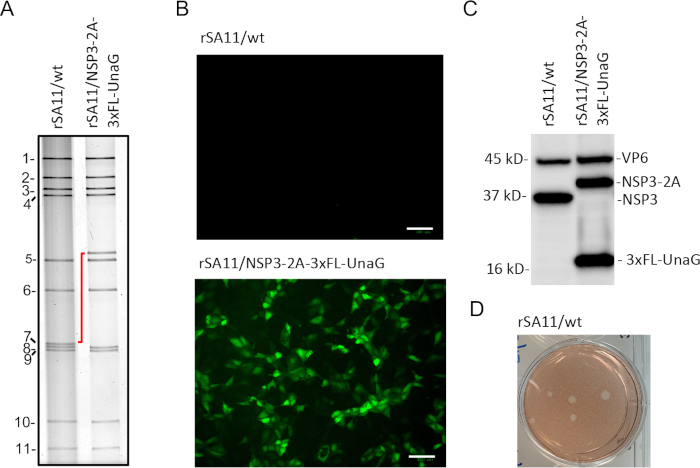
Figura 3: Caratteristiche dei ceppi ricombinanti rSA11/wt e rSA11/NSP3-2A-3xFL-UnaG. (A) Profili elettroforici dei segmenti del genoma del dsRNA dei ceppi di rSA11 isolati in placca. Gli 11 segmenti del genoma sono numerati e lo spostamento della posizione del segmento 7 (NSP3) è indicato con una linea rossa. (B) Fluorescenza rilevata nelle cellule MA104 infettate da rSA11 a 8 h dopo l'infezione utilizzando un imager a cellule vive (20x ingrandimento) impostato sul canale di rilevamento verde. Barra di scala (C) Analisi immunoblot delle proteine presenti a 8 h dopo l'infezione nelle cellule MA104 infettate da ceppi di rSA11 che utilizzano antiseraantino anti-VICEPRESIDENTe della cavia e anticorpo monoclonale anti-FLAG anti-FLAG anti-FLAG anti-FLAG. (D) Placche prodotte da rSA11/wt sulle cellule MA104 a 3 giorni dopo l'infezione e rilevate dalla colorazione rossa neutra. Fare clic qui per visualizzare una versione più grande di questa figura.
Discussione
Nel nostro laboratorio, ci affidiamo regolarmente al protocollo di genetica inversa descritto qui per produrre rotavirus SA11 ricombinanti. Con questo approccio, gli individui con poca esperienza nelle tecniche di biologia molecolare o che lavorano con i rotavirus recuperano i virus ricombinanti anche al loro primo tentativo. Abbiamo generato quasi 100 virus ricombinanti seguendo questo protocollo, compresi quelli con genomi che sono stati riprogettati per esprimere proteine estranee (ad esempio, SP) e che contengono aggiunte di sequenze, delezioni e mutazioni puntiformi.
Le condizioni e i tempi di incubazione indicati in questo protocollo si applicano al recupero di ceppi ben crescenti di virus ricombinanti. Gli adeguamenti dovrebbero essere presi in considerazione se si tenta di recuperare i virus SA11 che, a causa di modifiche genetiche, potrebbero crescere male. In particolare, poiché il tiborendo di tali virus nei lismi cellulari BHK-T7/MA014 trasfettati può essere basso, in genere raddoppiamo la quantità di lisato utilizzata come inoculo nelle successive fasi di amplificazione. Inoltre, nella fase di amplificazione, virus in mal crescita possono richiedere tempi più lunghi di incubazione prima di raggiungere i livelli di CPE sufficienti per la raccolta delle cellule. Infatti, con tali virus, possiamo permettere alle infezioni di procedere per 10/14 giorni, o anche più a lungo, prima di raccogliere le cellule. Infine, è probabile che virus in mal crescita generino placche piccole e a crescita lenta sulle cellule MA104. Quindi, per isolare questi virus in placca, potrebbe essere necessario consentire alle placche di svilupparsi fino a 6-10 giorni dopo l'infezione prima di colorare le cellule con il rosso neutro e raccogliere placche.
Nella nostra esperienza, il fattore più importante nel recupero affidabile del rotavirus ricombinante è l'uso di cellule BHK-T7 sane e ben tenute. Nel nostro laboratorio, di routine passaggiamo le cellule BHK-T7 2 volte a settimana alla stessa diluizione utilizzando un mezzo integrato non solo con il 10% di FBS, ma anche con NEAA, TPB e alti livelli di glucosio (GMEM completo medio). I supplementi aggiuntivi aiutano le cellule BHK-T7 mantenere la vitalità estesa dopo la trasfezione plasmide, un fattore probabilmente cruciale per il recupero di virus mutanti ricombinanti in mal crescita. Completiamo il mezzo ad ogni altro passaggio con G418, un antibiotico che seleziona per la manutenzione del plasmide di espressione polimerasi T7. Le condizioni di passaggio dovrebbero essere tali che le cellule BHK-T7 non sono mai autorizzate a crescere oltre la confluenza, condizioni che portano rapidamente alla diminuzione della vitalità cellulare. Per noi, le cellule BHK-T7 che sono state autorizzate a crescere troppo scarsamente nel protocollo di genetica inversa, anche se le cellule vengono successivamente passate in modo appropriato più volte. Invece di tentare di riabilitare le cellule BHK-T7 incolte inmodo, riavviiamo il lignaggio con cellule precedentemente conservate in azoto liquido.
La contaminazione da micoplasma delle cellule BHK-T7 e MA104 può essere un fattore importante nel fallimento del sistema genetico inverso del rotavirus per generare virus ricombinante. Nel nostro laboratorio, utilizziamo un kit di rilevamento del micoplasma basato su PCR (Tabella dei materiali) per verificare la presenza di contaminazione delle linee cellulari e, quando rilevato, è più spesso associato alle nostre cellule BHK-T7. Non abbiamo tentato di curare le linee cellulari del micoplasma contaminato, ristabilendo invece le linee con precedenti passaggi privi di micoplasma conservati nell'azoto liquido. Prima di iniziare nuove linee cellulari, scartiamo tutti gli integratori medi e medi utilizzati in precedenza e decontaminano accuratamente incubatori, armadi di sicurezza biologici, bagni d'acqua, panchine da laboratorio e pipettori. Utilizziamo anche kit di rilevamento del micoplasma per controllare le scorte di virus ricombinanti per la contaminazione. A causa della resistenza delle particelle di rotavirus alla denaturazione da solventi organici, come Vertrel VF32, è possibile liberare gli stock di virus dal mioplasma contaminante, negando la necessità di rigenerare i virus ricombinanti con la genetica inversa. È importante sottolineare che le linee cellulari e i preparati per i virus ricevuti in laboratorio devono essere controllati per verificarne la contaminazione da micoplasma prima dell'uso di routine.
Anche se giorno dopo giorno, usiamo lo stesso protocollo per generare virus ricombinanti, sappiamo che alcune modifiche possono essere apportate che non precluderà il recupero dei virus. Ad esempio, (i) la co-trasfezione del plasmide ipalizzatore/NSP868R con vettori SA11 pT7 non è necessaria per il recupero di virus ricombinanti. Mentre l'aggiunta del plasmide capping produce titoli di virus più elevati in lismi di cellule BHK-T7/MA104 trasfette, siamo stati in grado di recuperare numerosi virus, compresi quelli che esprimono PSp, senza di essa. Tuttavia, abbiamo concluso che l'espressione dell'enzima di tappatura da parte di pCMV/NP868R può contribuire in modo significativo al recupero di virus meno adatti. (ii) Abbiamo scoperto che i virus ricombinanti possono essere generati anche se la quantità del reagente di trasfezione (Tabella dei materiali) utilizzato nel protocollo di genetica inversa è ridotta della metà, una modifica che può ridurre significativamente le spese. (iii) Allo stesso modo, abbiamo stabilito che il supporto completo M199 può essere utilizzato al posto del supporto completo DMEM. (iv) Infine, non vi è alcun requisito prestabilito per quanto riguarda il tipo di spina dorsale vettoriale che deve essere utilizzato nella produzione di vettori di trascrizione SA11 T7. Finché il cDNA virale nel plasmide è circondato da un promotore T7 a monte e da un ribozyme HDV a valle e un terminatore T7, il plasmide può supportare il recupero del virus ricombinante. In particolare, i vettori basati su pGEM, pBluescript e pUC sono stati utilizzati con successo nel sistema di genetica inversa.
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Questo lavoro è stato supportato da NIH sovvenzioni R03 AI131072 e R21 AI144881, Indiana University Start-Up Funding, e il Lawrence M. Blatt Endowment. Ringraziamo i membri del laboratorio IU Rotahoosier, Ulrich Desselberger, e Guido Papa per i loro numerosi contributi e suggerimenti nello sviluppo del protocollo di genetica inversa.
Materiali
| Name | Company | Catalog Number | Comments |
| Baby Hamster Kidney - T7 RdRP (BHK-T7) Cells | Contact: ubuchholz@niaid.nih.gov | ||
| Bio-Rad 8-16% Tris-Glycine Polyacrylamide Mini-Gel | Bio-Rad | 45608105 | |
| Cellometer AutoT4 viable cell counter | Nexcelom | ||
| ChemiDoc MP Gel Imaging System | Bio-Rad | ||
| Chloroform | MP | 194002 | |
| Clarity Western Enhanced Chemiluminescence (ECL) Substrate | Bio-Rad | 170-5060 | |
| Competent E.coli DH5alpha Bacteria | Lucigen | 60602-2 | |
| Complete Protease Inhibitor | Pierce | A32965 | |
| Disposable Transfer Pipettes, Ultrafine Extended Tips | MTC Bio | P4113-11 | |
| Dulbecco's Modified Eagle Medium (DMEM) | Lonza | 12-604F | |
| Eagle's Minimal Essential Medium, 2x (2xEMEM) | Quality Biological | 115-073-101 | |
| Ethanol, Absolute (200 proof) | Fisher Bioreagents | BP2818-500 | |
| Ethidium Bromide Solution (10 mg/ml) | Invitrogen | 15585-011 | |
| Fetal Bovine Serum (FBS) | Corning | 35-010-CV | |
| Fetal Bovine Serum (FBS), Heat Inactivated | Corning | 35-011-CV | |
| Flag M2 Antibody, Mouse Monoclonal | Sigma-Aldrich | F1804 | |
| GenEluate HP Plasmid Midiprep Kit | Sigma | NA0200-1KT | |
| Geneticin (G-418) | Invitrogen | 10131-027 | |
| Gibco FluroBrite DMEM | ThermoFisher | A1896701 | DMEM with low background fluorescence |
| Glasgow Minimal Essential Medium (GMEM) | Gibco | 11710-035 | |
| Goat Anti-Rabbit IgG, Horseradish Peroxidase (HRP) Conjugated | Cell-Signaling Technology | 7074S | |
| Guinea Pig Anti-NSP3 Antiserum | Patton lab | lot 55068 | |
| Guinea Pig Anti-VP6 Antierum | Patton lab | lot 53963 | |
| Horse Anti-Guinea Pig IgG, Horseradish Peroxidase (HRP) Conjugated | KPL | 5220-0366 | |
| Horse Anti-Mouse IgG, Horseradish eroxidase (HRP) Conjugated | Cell-Signaling Technology | 7076S | |
| iNtRON Biotechnology e-Myco Mycoplasma PCR Detection Kit | JH Science | 25235 | |
| Isopropyl alcohol | Macron | 3032-02 | |
| L-glutamine Solution (100x) | Gibco | 25030-081 | |
| Luria Agar Powder (Miller's LB Agar) | RPI research products | L24020-2000.0 | |
| Medium 199 (M199) Culture Medium | Hyclone | Sh30253.01 | |
| Minimal Essential Medium -Eagle Joklik's Forumation (SMEM) | Lonza | 04-719Q | |
| Monkey Kidney (MA104) Cells | ATCC | ATCC CRL-2378.1 | |
| NanoDrop One Spectrophotometer | ThermoScientific | ||
| Neutral Red Solution (0.33%) | Sigma-Aldrich | N2889-100ml | |
| Non-Essential Amino Acid Solution (100x) | Gibco | 11140-050 | |
| Novex 10% Tris-Glycine Polyacrylamide Mini-Gel | Invitrogen | XP00102BOX | |
| Nuclease-Free Molecular Biology Grade Water | Invitrogen | 10977-015 | |
| NucleoSpin Gel and PCR Clean-Up Kit | Takara | 740609.25 | |
| Opti-MEM Reduced Serum Medium | Gibco | 31985-070 | |
| Pellet pestle (RNase-free, disposable) | Fisher | 12-141-368 | |
| Penicillin-Streptomycin Solution, (100x penn-strep) | Corning | 30-002-Cl | |
| Phosphate Buffered Saline (PBS), 10x | Fisher Bioreagents | BP399-20 | |
| Porcine Trypsin, Type IX-S | Sigma-Aldrich | T0303 | |
| PureYield Plasmid Miniprep System | Promega | A1223 | |
| Qiagen Plasmid Maxi Kit | Qiagen | 12162 | |
| Qiagen Plasmid Midi Kit | Qiagen | 12143 | |
| QIAprep Spin Miniprep Kit | Qiagen | 27104 | |
| SA11 pT7 Transcription Vectors | Addgene | 89162-89172 | |
| SA11 pT7/NSP3 Transcription Vectors Expressing Fluorescent Proteins | Contact: jtpatton@iu.edu | ||
| SeaKem LE Agarose | Lonza | 50000 | For gel electrophoresis |
| SeaPlaque agarose | Lonza | 50100 | For plaque assay |
| Superscript III One-Step RT-PCR kit | Invitrogen | 12574-035 | |
| Trans-Blot Turbo Nitrocellulose Transfer Kit | Bio-Rad | 170-4270 | |
| Trans-Llot Turbo Transfer System | Bio-Rad | ||
| TransIT-LTI Transfection Reagent | Mirus | MIR2306 | |
| Tris-Glycine-SDS Gel Running Buffer (10x) | Bio-Rad | 161-0772 | |
| Triton X 100 | Fisher Bioreagents | BP151-500 | |
| Trizol RNA Extraction Reagent | Ambion | 15596026 | |
| Trypan blue | Corning | 25-900-CI | |
| Trypsin (0.05%)-EDTA (0.1%) Cell Dissociation Solution | Quality Biological | 118-087-721 | |
| Tryptose Phosphate Broth | Gibco | 18050-039 | |
| Tween-20 | VWR | 0777-1L | |
| Vertrel VF solvent | Zoro | G0707178 | |
| Zoe Fluorescent Live Cell Imager | Bio-Rad |
Riferimenti
- Crawford, S. E., et al. Rotavirus infection. Nature Reviews Disease Primers. 3 (17083), 1-16 (2017).
- Settembre, E. C., Chen, J. Z., Dormitzer, P. R., Grigorieff, N., Harrison, S. C. Atomic model of an infectious rotavirus particle. The EMBO Journal. 30 (2), 408-416 (2011).
- . Taxonomic information. Virus taxonomy: 2018b release Available from: https://talk.ictvonline.org/taxonomy (2019)
- Tate, J. E., Burton, A. H., Boschi-Pinto, C., Parashar, U. D. World Health Organization-Coordinated Global Rotavirus Surveillance Network. Global, regional, and national estimates of rotavirus mortality in children <5 years of age. Clinical Infectious Diseases. 62, 96-105 (2016).
- Troeger, C., et al. Rotavirus vaccination and the global burden of rotavirus diarrhea among children younger than 5 years. JAMA Pediatrics. 172 (10), 958-965 (2018).
- Matthijnssens, J., Van Ranst, M. Genotype constellation and evolution of group A rotaviruses infecting humans. Current Opinion in Virology. 2 (4), 426-433 (2012).
- McDonald, S. M., Nelson, M. I., Turner, P. E., Patton, J. T. Reassortment in segmented RNA viruses: mechanisms and outcomes. Nature Reviews Microbiology. 14 (7), 448-460 (2016).
- Patton, J. T. Rotavirus diversity and evolution in the post-vaccine world. Discovery Medicine. 13 (68), 85-97 (2012).
- Kanai, Y., et al. Entirely plasmid-based reverse genetics system for rotaviruses. Proceedings of the National Academy of Sciences of the United States of America. 114 (9), 2349-2354 (2017).
- Trask, S. D., McDonald, S. M., Patton, J. T. Structural insights into the coupling of virion assembly and rotavirus replication. Nature Reviews Microbiology. 10 (3), 165-177 (2012).
- Guglielmi, K. M., McDonald, S. M., Patton, J. T. Mechanism of intraparticle synthesis of the rotavirus double-stranded RNA genome. Journal of Biological Chemistry. 285 (24), 18123-18128 (2010).
- Komoto, S., et al. Generation of recombinant rotaviruses expressing fluorescent proteins by using an optimized reverse genetics system. Journal of Virology. 92 (13), 00588-00618 (2018).
- Trask, S. D., Taraporewala, Z. F., Boehme, K. W., Dermody, T. S., Patton, J. T. Dual selection mechanisms drive efficient single-gene reverse genetics for rotavirus. Proceedings of the National Academy of Sciences of the United States of America. 107, 18652-18657 (2010).
- Fabbretti, E., Afrikanova, I., Vascotto, F., Burrone, O. R. Two non-structural rotavirus proteins, NSP2 and NSP5, form viroplasm-like structures in vivo. Journal of General Virology. 80, 333-339 (1999).
- Eichwald, C., Rodriguez, J. F., Burrone, O. R. Characterization of rotavirus NSP2/NSP5 interactions and the dynamics of viroplasm formation. Journal of General Virology. 85, 625-634 (2004).
- Kawagishi, T., et al. Reverse genetics system for a human group A rotavirus. Journal of Virology. , (2019).
- Komoto, S., et al. Generation of infectious recombinant human rotaviruses from just 11 cloned cDNAs encoding the rotavirus genome. Journal of Virology. 93 (8), 02207-02218 (2019).
- Mohanty, S. K., et al. A point mutation in the rhesus rotavirus VP4 protein generated through a rotavirus reverse genetics system attenuates biliary atresia in the murine model. Journal of Virology. 91 (15), 00510-00517 (2017).
- Navarro, A., Trask, S. D., Patton, J. T. Generation of genetically stable recombinant rotaviruses containing novel genome rearrangements and heterologous sequences by reverse genetics. Journal of Virology. 87, 6211-6220 (2013).
- Duarte, M., et al. Rotavirus infection alters splicing of the stress-related transcription factor XBP1. Journal of Virology. 93 (5), 01739-01818 (2019).
- Philip, A. A., et al. Generation of recombinant rotavirus expressing NSP3-UnaG fusion protein by a simplified reverse genetics system. Journal of Virology. , 1616-1619 (2019).
- Papa, G., et al. Recombinant rotaviruses rescued by reverse genetics reveal the role of NSP5 hyperphosphorylation in the assembly of viral factories. Journal of Virology. , (2019).
- Komoto, S., et al. Reverse genetics system demonstrates that rotavirus nonstructural protein NSP6 is not essential for viral replication in cell culture. Journal of Virology. 91 (21), 00695-00717 (2017).
- Philip, A. A., et al. Collection of recombinant rotaviruses expressing fluorescent reporter proteins. Microbiology Resource Announcements. 8 (27), 00523-00619 (2019).
- Kanai, Y., et al. Development of stable rotavirus reporter expression systems. Journal of Virology. , 01774-01818 (2019).
- Donnelly, M. L., et al. The 'cleavage' activities of foot-and-mouth disease virus 2A site-directed mutants and naturally occurring '2A-like' sequences. Journal of General Virology. 82, 1027-1041 (2001).
- Kumagai, A., et al. A bilirubin-inducible fluorescent protein from eel muscle. Cell. 153, 1602-1611 (2013).
- Rodriguez, E. A., et al. The growing and glowing toolbox of fluorescent and photoactive proteins. Trends in Biochemical Sciences. 42, 111-129 (2017).
- Buchholz, U. J., Finke, S., Conzelmann, K. K. Generation of bovine respiratory syncytial virus (BRSV) from cDNA: BRSV NS2 is not essential for virus replication in tissue culture, and the human RSV leader region acts as a functional BRSV genome promoter. Journal of Virology. 73, 251-259 (1999).
- Centers for Disease Control and Prevention (CDC). . Biosafety in microbiological and biomedical laboratories (BMBL), 5th edition. , (2009).
- Arnold, M., Patton, J. T., McDonald, S. M. Culturing, storage, and quantification of rotaviruses. Current Protocols in Microbiology. 15 (1), (2009).
- Benureau, Y., Huet, J. C., Charpilienne, A., Poncet, D., Cohen, J. Trypsin is associated with the rotavirus capsid and is activated by solubilization of outer capsid proteins. Journal of General Virology. 86 (11), 3143-3151 (2005).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon