Method Article
Ferramenta de peixe de DNA Multicolor 3D para estudar arquitetura nuclear em células primárias humanas
Neste Artigo
Resumo
O DNA 3D multicolor FISH representa uma ferramenta para visualizar loci múltiplogenômico dentro de núcleos preservados em 3D, definindo inequivocamente suas interações recíprocas e localização dentro do espaço nuclear em um único nível celular. Aqui, um protocolo passo a passo é descrito para um amplo espectro de células primárias humanas.
Resumo
Uma questão importante na biologia celular é a organização genômica dentro do espaço nuclear e como a arquitetura da cromatina pode influenciar processos como expressão genética, identidade celular e diferenciação. Muitas abordagens desenvolvidas para estudar a arquitetura 3D do genoma podem ser divididas em duas categorias complementares: tecnologias baseadas em captura de conformação cromossa (C-technologies) e imagem. Enquanto o primeiro é baseado na captura da conformação cromossomo e interações de DNA proximal em uma população de células fixas, este último, baseado na fluorescência de DNA na hibridização situ (FISH) em núcleos preservados em 3D, permite a visualização contemporânea de loci múltiplo em um único nível celular (multicolor), examinando suas interações e distribuição dentro do núcleo (3D multicolor DNA FISH). A técnica do DNA 3D multicolor FISH tem uma limitação de visualizar apenas alguns loci pré-determinados, não permitindo uma análise abrangente da arquitetura nuclear. No entanto, dada a robustez de seus resultados, o DNA FISH multicolorido 3D em combinação com microscopia 3D e reconstrução de imagens é um método possível para validar resultados baseados em tecnologia C e estudar inequivocamente a posição e organização de loci específico em um único nível celular. Aqui, propomos um método passo a passo de Peixe sna multicolorido 3D adequado para uma ampla gama de células primárias humanas e discutimos todas as ações práticas, passos cruciais, noções de imagem 3D e análise de dados necessárias para obter uma multicolor 3D bem sucedida e informativa Peixe de DNA em diferentes contextos biológicos.
Introdução
Os eucalitos mais altos precisam condensar sistematicamente e compactar uma enorme quantidade de informações genéticas no espaço 3D do núcleo1,2,3,4. Hoje, sabemos que o genoma é espacialmente encomendado em compartimentos e domínios topologicamente associados5 e que os múltiplos níveis de dobramento de DNA geram contatos entre diferentes regiões genômicas que podem envolver a formação de loop de cromromatina6,7. O looping dinâmico 3D da cromromatina pode influenciar muitos processos biológicos diferentes, como transcrição8,9, diferenciação e desenvolvimento10,11, reparação de DNA12,13, enquanto suas perturbações estão envolvidas em várias doenças14,15,16 e defeitos de desenvolvimento15,17,18.
Muitas abordagens foram desenvolvidas para estudar a organização do genoma 3D. Tecnologias baseadas em conformação cromossa (c-technologies, 3C, 4C, 5C, Hi-C e derivados) foram desenvolvidas para estudar a organização do genoma em células fixas3,4,19,20. Tais abordagens baseiam-se na capacidade de capturar as frequências de contato entre loci genômico na proximidade física. C-tecnologias, dependendo de sua complexidade, capturam a organização global do genoma 3D e a topologia nuclear de uma população celular3,4,19,20. No entanto, as interações 3D são dinâmicas no tempo e no espaço, altamente variáveis entre células individuais que consistem em interações multiplex, e são extensivamente heterogênitas21,22.
Fluorescência de DNA multicolor 3D na hibridização situ (FISH) é uma técnica que permite a visualização de loci genômico específico em um único nível celular, permitindo a investigação direta da arquitetura nuclear 3D de forma complementar às tecnologias C. Representa uma tecnologia atualmente usada para validar inequivocamente os resultados C. O DNA FISH 3D multicolor usa sondas com rótulo fluorescente complementares aos loci genômicos de interesses. O uso de diferentes fluoroforóres e equipamentos adequados de microscopia permitem a visualização contemporânea de múltiplos alvos dentro do espaço nuclear23,24. Nos últimos anos, o FISH tem sido combinado com os avanços tecnológicos na microscopia para obter a visualização de estruturas em escala fina em alta resolução25,26 ou com abordagens CRISPR-Cas para a visualização dos ácidos nucleicos em imagens vivas27,28. Apesar da ampla adoção, a abordagem 3D multicolor DNA FISH ainda é considerada difícil em muitos laboratórios porque o material biológico utilizado deve ser adaptado.
Aqui, fornecemos um protocolo abrangente para peixes de DNA multicoloridos 3D (da preparação celular/sonda à análise de dados) aplicável a uma ampla gama de células primárias humanas, permitindo a visualização de múltiplos loci genômicos e preservando a estrutura 3D de núcleos. Para estudar a arquitetura nuclear, a estrutura 3D dos núcleos deve ser preservada. Por essa razão, contrastando com outros protocolos existentes29,30,31, evitamos o uso de um gradiente de álcool e o armazenamento dos comprovantes de cobertura no álcool que podem afetar a estrutura de cromatina32. O método é adaptado a partir de protocolos de Peixe sna 3D preservados24,33 a serem aplicados a uma ampla gama de células primárias humanas, tanto ex vivo isoladas quanto cultizadas in vitro. Existem parâmetros de permeabilização e desproteína para diferentes características citológicas e citológicas nucleares (por exemplo, diferentes graus de compactação nuclear, abundância de citoesqueleto)34. Esses parâmetros são geralmente descritos em outros protocolos24,33, sem fornecer uma clara discriminação do procedimento dentro de diferentes tipos de células. Além disso, desenvolvemos uma ferramenta específica chamada NuCLεD (localizador de contatos nucleares em 3D)16, fornecendo princípios para análise de dados que melhorarão a proximidade 3D entre diferentes loci e sua distribuição topológica nuclear dentro do espaço nuclear de forma automatizada.
Protocolo
1. Procedimentos de preparação e rotulagem de sondas de DNA com tradução de nick
- Cromossomos artificiais bacterianos purificados e limpos (BACs), plasmídeos ou produtos PCR com kits específicos (Tabela de Materiais),resuspendem no ddH2O, verifiquem por eletroforese em gel de agarose e quantificar.
- Realize a tradução de nick em 1,5-2 μg de DNA a partir da etapa 1.1 em um volume final de 50 μL misturando todos os reagentes em um tubo de DNA de baixa ligação de 0,5 mL de acordo com a Tabela 1.
NOTA: Sondas diretamente rotuladas podem melhorar a relação sinal/ruído. Kits para produzir sondas indiretas e diretamente rotuladas com vários fluorocromáticos Alexa por tradução de nick estão disponíveis comercialmente. - Incubar a mistura de tradução nick em uma batedeira térmica a 16 °C por um tempo, dependendo do comprimento do material de DNA inicial: 45 min para produtos PCR (um pool de produtos PCR de 2.000 bp cada) e até 4 h para DNA BAC e plasmídeos.
- Verifique o tamanho das sondas produzidas na etapa 1.3 por eletroforrose em um gel de agarose de 2,2%.
NOTA: O tamanho ideal da sonda é <200 bp(Figura 1A).- Se o DNA não for digerido o suficiente, adicione 5 U de DNA polymerase I e 0,05 U de DNase I à reação e incubar por 1-2 h a 16 °C e posteriormente re-verificar. Pare a reação com 0,5 mM EDTA (concentração final). Armazene sondas a -20 °C.
- Para cada experimento de DNA FISH, precipitar as seguintes quantidades de sondas dependendo do material de DNA inicial do qual as sondas são produzidas: 200 ng de produtos PCR traduzidos por Nick, 100 ng de BACs traduzidos por Nick, ou 300 ng de nick traduzido plasmid. Adicione ddH2O até 150 μL, 20 μg de DNA de esperma torça de salmão não rotulado, 3,5 μg de DNA Cot-1 específico de espécies, 3 volumes de 100% EtOH e 1/10 volume de 3 M de acetato de sódio pH 5.2. Precipitar a -80 °C por 1h.
- Centrífuga em velocidade máxima por 1 h a 4 °C e descarte o supernatante. Lave a pelota duas vezes com 70% de EtOH.
- Resuspender a pelota em 2 μL de 100% de pH 7.0, tremer a 40 °C por 30 min (pode levar até algumas horas) e, em seguida, adicionar um volume igual de citrato salina-sódio 4x (SSC)/20% dextran sulfato.
- Prepare 20x SSC misturando 175,3 g de NaCl e 88,2 g de citrato de sódio em um volume final de 1 L de H2O. Autoclave, filtrar e preparar alíquotas.
NOTA: Para peixes de DNA multicoloridos, as diferentes sondas podem ser precipitadas juntas, exceto para sondas que podem anrneá-los uns com os outros. Nestes casos específicos, trate-os separadamente, precipitar e resuspender as sondas que dividem os reagentes mencionados nas etapas 1.5-1,7 em relação ao número de sondas. Pool as sondas somente após a resuspensão em formamide. Se forem utilizadas tampas de vidro superiores a 10 mm ou menos de 10 mm, dimensione para cima/para baixo o volume dos reagentes utilizados nos passos 1,5-1,7 proporcionalmente ao tamanho do slide. As sondas de hibridização podem ser armazenadas a -20 °C por um longo período de tempo (até dois meses).
- Prepare 20x SSC misturando 175,3 g de NaCl e 88,2 g de citrato de sódio em um volume final de 1 L de H2O. Autoclave, filtrar e preparar alíquotas.
2. Fixação celular, pré-tratamento e permeabilização
NOTA: As passagens de permeabilização e desproteína são passos cruciais. O tempo de reação e concentração dos reagentes dependem fortemente do tipo celular, da abundância de citoplasma e da morfologia nuclear.
- Fixação celular, pré-tratamento e permeabilização para linfócitos t primários humanos
NOTA: Este protocolo é adequado para pequenas células, com pequenos núcleos e uma baixa quantidade de citoplasma.- Use tampas de vidro (10 mm, espessura nº 1,5H).
- Lave o vidro com ddH2O, depois com 70% de EtOH e deixe-os secar. Use um copo para cada poço de uma placa de 24 poços.
- Adicione 200 μL de 0,1% poli-L-lysine (w/v) (Tabela de Materiais) diretamente no vidro para formar uma gota. Preste atenção, pois a gota deve permanecer na superfície do vidro sem tocar no poço por 2-5 min. Deixe de fora a queda cuidadosamente, e deixe o vidro secar por 30 min.
- Faça a etapa 2.1.3 duas vezes mais.
- Coloque 200 μL de células de suspensão (2 x 106/mL, suspensão PBS de linfócitos ex vivo primárioT) diretamente no vidro, permita que as células semarem à temperatura ambiente (RT) por 30 min.
- Remova rapidamente a queda. Adicione recém-feito 4% PFA (preparado na PBS/0,1% TWEEN 20, pH 7.0, filtrado) por 10 min e fixar as células semeadas na RT.
- Lave três vezes por 5 min cada com 0,05% Triton X-100/PBS (TPBS) na RT. Permeabilize com 0,5% TPBS por 10 min na RT.
- Realizar o tratamento RNase adicionando 2,5 μL de coquetel RNase (Tabela de Materiais) em 250 μL de PBS/bem em uma placa de 24 poços por 1 h a 37 °C.
- Enxágüe na PBS, adicione 20% de glicerol/PBS e incuba durante a noite (ON) a 4°C.
NOTA: Este passo pode variar de 1h a ON. Os slides podem ser mantidos em 20% glicerol/PBS a 4 °C até 7 dias. - Congele no gelo seco (15-30 s), descongele gradualmente na RT e mergulhe em 20% glicerol/PBS. Repita o passo 2.1.9 mais três vezes.
- Lave em 0,5% TPBS por 5 min na RT. Wash em 0,05% TPBS, duas vezes por 5 min na RT. Incubar em 0,1 N HCl por 12 min na RT. Rinse em 2x SSC.
- Incubar em 50% de pH 7.0/2x SSC ON na RT.
NOTA: O tempo de incubação pode ser otimizado e eventualmente reduzido. Os slides podem ser mantidos em 50% de formamide/2x SSC por vários dias.
- Fixação celular, pré-tratamento e permeabilização para mioblastos primários humanos
NOTA: Este protocolo é adequado para células grandes, com uma alta quantidade de citoplasma.- Cultivar mioblastos primários humanos diretamente nos óculos de deslizamento de cobertura em placas de 24 poços em meio de crescimento (o meio eagle modificado de Dulbecco (DMEM), 20% de soro bovino fetal (FBS), fator de crescimento do fibroblasto de 25 ng/mL (FGF), fator de crescimento epidérmico de 10 ng/mL (EGF), 10 μg/mL insulina humana, 1x glutamina, 1x penicilina/estreptomia) por pelo menos 24 h (atingindo 50-70% de confluência).
NOTA: Para facilitar a adesão, gelatina ou colágeno ou poli-l-lysina pode ser usado para cobrir o deslizamento de cobertura antes da semente. - As células de enxágüe em 2-3 alterações de PBS. Adicione recém-feito 4% pfa e fixe as células por 10 min na RT.
- Lave três vezes por 3 min cada em 0,01% TPBS na RT. Permeabilize em 0,5% TPBS por 10 min na RT.
- Realizar o tratamento RNase adicionando 2,5 μL de coquetel RNase (Tabela de Materiais) em 250 μL de PBS/bem em uma placa de 24 poços por 1 h a 37 °C.
- Enxágüe na PBS, adicione 20% de glicerol/PBS e incuba na RT.
NOTA: Este passo pode variar de 1h a ON. Os slides podem ser mantidos em 20% glicerol/PBS a 4 °C até 7 dias. - Congele no gelo seco (15-30 s), descongele gradualmente na RT e mergulhe em 20% glicerol/PBS. Repita esta etapa três vezes.
- Lave três vezes por 10 min cada em PBS. Incubar em 0,1 N HCl por 5 min na RT. Rinse em 2x SSC.
- Incubar em 50% de pH 7.0/2x SSC ON na RT.
NOTA: O tempo de incubação pode ser otimizado e eventualmente reduzido. Os slides podem ser mantidos em 50% de formamide/2x SSC por vários dias. - Deslizamentos de equilíbrio (mantidos em 50% de formamide/2x SSC) em 2x SSC por 2 min. Em seguida, equilibre na PBS por 3 min.
- Trate com 0,01 N HCl/0,0025% pepsin a partir de alguns segundos até 5 minutos, dependendo do tipo celular. Durante esta etapa, observe as células um microscópio óptico e pare a reação (passo 2.2.11) assim que os núcleos estiverem livres do citoplasma, mantendo sua estrutura intacta (por exemplo, o nucleoli ainda está visível e intacto).
- Inative a pepsina lavando duas vezes por 5min cada em 50 mM MgCl 2/PBS.
- Pós-fixação em 1% PFA/PBS por 1 min. Lave por 5 min na PBS. Lave duas vezes por 5 min cada em 2x SSC, e depois adicione 50% de formamide/2x SSC por pelo menos 30 min.
- Cultivar mioblastos primários humanos diretamente nos óculos de deslizamento de cobertura em placas de 24 poços em meio de crescimento (o meio eagle modificado de Dulbecco (DMEM), 20% de soro bovino fetal (FBS), fator de crescimento do fibroblasto de 25 ng/mL (FGF), fator de crescimento epidérmico de 10 ng/mL (EGF), 10 μg/mL insulina humana, 1x glutamina, 1x penicilina/estreptomia) por pelo menos 24 h (atingindo 50-70% de confluência).
3. Hibrid de DNA 3D
- Desnatura as sondas a 80 °C por 5 min, e depois coloque rapidamente no gelo.
- Carregue as sondas de hibridização em um slide de microscópio limpo. Gire o deslizamento de cobertura com células de cabeça para baixo na queda de sondas de hibridização.
- Selar o deslizamento de cobertura com cimento de borracha. Deixe o cimento de borracha secar completamente. Em seguida, coloque slides em um bloco de aquecimento e desnatureza a 75 °C por 4 min.
NOTA: O tempo de desnaturação e temperatura de desnaturação podem ser aumentados, até 80 °C. - Hibridize a 37 °C ON em uma caixa metálica flutuando em um banho de água.
NOTA: Para melhorar a relação sinal/ruído, a temperatura da hibridização pode subir para 42 °C. - Retire o cimento de borracha, mergulhe os slides em 2x SCC, retire o deslizamento de cobertura de vidro e transfira-o para 2x SSC em placa de 6 poços.
- Lave em 2x SSC três vezes por 5 min cada a 37 °C, tremendo a 90 rpm em um agitador de incubadora. Lave em 0,1x SSC três vezes por 5 min cada a 60 °C, tremendo a 90 rpm em um agitador de incubadora. Enxágüe brevemente em 4x SSC/0,2% TWEEN 20.
4. Detecção de PEIXE sna de DNA multicolorido 3D
NOTA: Para sondas diretamente rotuladas, pule as etapas 4.1 e 4.2.
- Bloqueie em 4x SSC/0,2% TWEEN 20/4% de albumina de soro bovino (BSA) por 20 min a 37 °C em uma placa de 24 poços, tremendo a 20 rpm em um shaker incubadora.
- Incubar com a concentração adequada de anti-digoxygenin (1:150), e/ou streptavidin (1:1.000) (Tabela de Materiais) diluída em 4x SSC/0,2% TWEEN 20/4% BSA por 35 min em uma câmara escura e úmida a 37 °C.
- Lave em 4x SSC/0,2% TWEEN 20 três vezes por 5 min cada a 37 °C, tremendo a 90 rpm em uma placa de 6 poços em um shaker incubadora.
- Equilibre-se na PBS e pós-fixação em 2% formaldeído/PBS por 2 min na RT em uma placa de 24 poços.
NOTA: Sondas diretamente rotuladas não precisam de pós-fixação. - Lave 5x brevemente na PBS. Mancha com 1 ng/mL DAPI (4,6-diamidino-2-phenylindole)/PBS por 5 min na RT.
- Lave 5x brevemente na PBS. Montar com solução antifumada (Tabela de Materiais).
5. Microscopia e análise de Peixe sna multicolorido 3D
- Adquirir imagens 3D com um sistema de microscópio.
NOTA: Aqui, um microscópio widefield com uma distância axial de 0,2-0,25 μm entre seções consecutivas(Figura 1B) é usado. - Analise pilhas de imagens 3D usando diferentes softwares e ferramentas (aqui, NuCLεD ou Localizador de Contatos Nucleares em 3D).
NOTA: A ferramenta NuCLεD foi desenvolvida para analisar automaticamente o DNA de DNA 3D em pilhas de imagem celular de fluorescência. O NuCLεD reconstrói os núcleos das pilhas de imagem celular em 3D, bem como detecta e localiza manchas 3D fluorescentes. Mede o posicionamento relativo das manchas no núcleo (por exemplo, distância do centroide dos núcleos e/ou periferia dos núcleos) e o raio máximo para cada núcleo e distâncias intermanchas. A ferramenta e o algoritmo utilizado são totalmente descritos em Cortesi et al.16. - Analise os dados (por exemplo, distâncias 3D entre loci genômico especificado e as frequências nucleares e de contato) recuperadas pelo NuCLεD.
NOTA: Para distâncias 3D entre loci genômico especificado e o centroide nuclear, normalize distâncias no raio máximo para cada núcleo e represente esses dados como distribuições de frequência de distâncias normalizadas do centroide nuclear. Para estudos de interações de longo alcance, as frequências de contato devem estar na faixa de 10-20%21 com um limiar inter-distância que pode variar e pode ser colocada em torno de 2 μm35.- Para realizar análiseestatística, analise aproximadamente 100 núcleos por replicação biológica. Represente distâncias 3D entre loci genômico especificado como distribuições de frequência cumulativa de distâncias que estão abaixo do limiar inter-distância selecionada e usam um teste tpara avaliar a significância das diferenças nas distribuições. Além disso, calcule o percentual de núcleos positivos para as interações que estão abaixo do limiar inter-distância selecionado, e use o teste exato de Fisher para avaliar a significância das diferenças nos percentuais.
Resultados
O método de DNA 3D multicolorido FISH descrito neste artigo permite a visualização contemporânea de diferentes loci genômicos dentro de núcleos 3D preservados (Figura 1B). Este protocolo permite a medição de distâncias entre alelos, e diferentes loci genômicos para avaliar sua proximidade espacial, e avaliar sua localização dentro do espaço nuclear (por exemplo, a distância loci do centroide ou da periferia dos núcleos)16. No entanto, existem muitas etapas cruciais que devem ser configuradas com precisão e especificamente para cada tipo de célula utilizada; é altamente recomendado prestar atenção especial aos seguintes passos para o sucesso do DNA 3D multicolor FISH.
Para a preparação da sonda de DNA, verifique se o tamanho da sonda é <200 bp(Figura 1A). Este tamanho garante um procedimento bem sucedido de 3D multicolor DNA FISH (Figura 1B). Sondas subótimas de DNA FISH produzidas por tradução de nick podem ser parcialmente digeridas(Figura 2A) ou sobredigeridas(Figura 2B). Com sondas parcialmente digeridas, o procedimento não terá sinal nas células, devido à incapacidade da sonda de entrar nos núcleos e hibridizar adequadamente para os loci genômicos complementares. Sobre as sondas digeridas resultará em um sinal inespecífico, devido à perda de especificidade na hibridização e a um consequente aumento do fundo. Um exemplo representativo de sondas sobredigeridas é mostrado na Figura 3A em comparação com uma sonda digerida ideal na Figura 3B.
Para desiproteção e pepsinização, siga esses passos de acordo com o tipo de célula. Em particular, leve em consideração o tamanho nuclear e a abundância de citoplasma. Para linfócitos e células T isoladas do ex vivo humano com núcleos pequenos e altamente compactados e citoplasma abundante baixo, a desproteína do HCl é crucial. O tratamento com 0,1 N HCl para 5 min não é suficiente para visualização de Peixes de DNA. 0.1 N HCl tratamento por 12 min é recomendado para promover a acessibilidade de núcleos a sondas de DNA e preservar a integridade nuclear (Figura 4A). A digestão de pepsina do citoplasma não é necessária para obter um bom sinal de DNA FISH (Figura 4B).
Para mioblastos primários humanos e células que possuem grandes núcleos e citoplasma abundante, o passo de pepsinização é fundamental. Uma pepsinização curta e subótima do citoesqueleto dificultará a entrada da sonda nos núcleos (Figura 4C), terminando na ausência de um sinal de DNA FISH. No entanto, se as células forem sobre pepsinizadas, os núcleos não permanecerão intactos (Figura 4D), perdendo sua estrutura 3D. Um exemplo de peixe de DNA multicolorido 3D bem sucedido é fornecido na Figura 4E.
Durante a hibridização, veda o deslizamento de cobertura com precisão; caso contrário, a sonda se dispersará e secará. As etapas de desnaturação e hibridização devem ser realizadas rapidamente de modo que a sonda e o DNA genômico não reanneal. A duração da desnaturação pode ser aumentada.

Figura 1: Sondas representativas de DNA e PEIXE de DNA multicolorido 3D. (A) Nick traduziu sondas de DNA de tamanho ideal executados em um gel de agarose de 2,2% (faixa 1, 2), marcador de 50 bp (M). (B) Núcleo de PEIXE sna multicolorido 3D representativo utilizando sondas mapeando 3q11,2 região (verde), 10q26,3 região (vermelha) e 8q24,13 região (magenta) em mioblastos primários humanos. Os núcleos são neutralizados com daPI (azul). Ampliação de 63x. Barra de escala = 5 μm. Clique aqui para ver uma versão maior deste valor.

Figura 2: Exemplos de sondas de Peixe de DNA não digeridas de forma ideal. (A) Não digeridos (pista 1, 2) ou parcialmente digeridos (pista 3) sondas de DNA traduzidas em um gel de agarose de 2,2%, marcador de 2log (M). (B) Sobre os testes de DNA traduzidos de Nick foram executados em um gel de agarose de 2,2%, marcador de 2log (M). Clique aqui para ver uma versão maior deste valor.
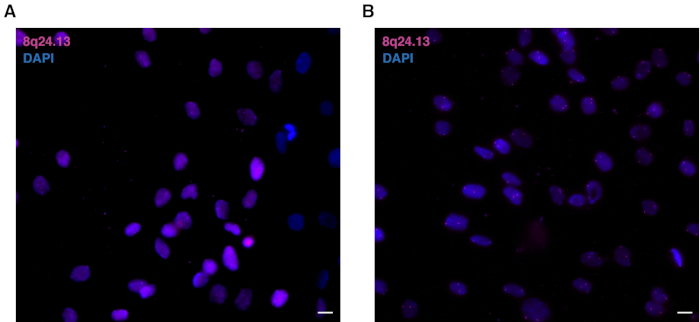
Figura 3: Comparação de PEIXE sNA multicolorido 3D usando sondas subótimas ou ideais de DNA FISH. (A)Núcleos de Peixes de DNA multicoloridos 3D representativos usando mapeamento de sondas digeridas para 8q24,13 região (magenta) em mioblastos primários humanos. Os núcleos são neutralizados com daPI (azul). Ampliação de 63x. Barra de escala = 10 μm.(B) Núcleos de Peixe saque de DNA multicolor 3D representativos usando mapeamento de sondas digeridos idealmente para 8q24,13 região (magenta) em mioblastos primários humanos. Os núcleos são neutralizados com daPI (azul). Ampliação de 63x. Barra de escala = 10 μm. Clique aqui para ver uma versão maior deste valor.
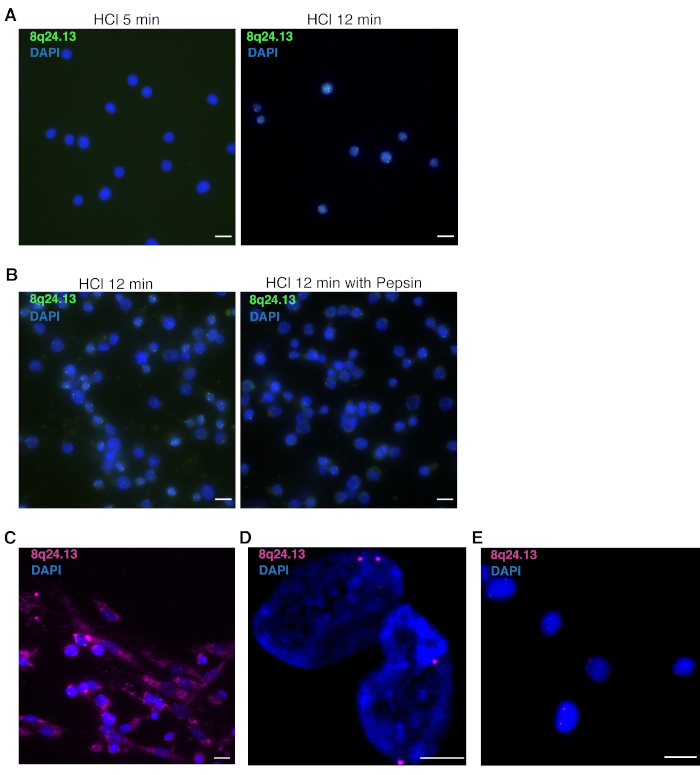
Figura 4: Possíveis resultados de desproteína subótima e etapas de pepsinização nos resultados do DNA de DNA multicolorido 3D. (A) Núcleos de Peixe sem tons 3D representativos de linfócitos T primários humanos tratados por 5 min (esquerda) ou 12 min (direita) com 0,1 N HCl, utilizando mapeamento de sondas para 8q24,13 região (verde). Os núcleos são neutralizados com daPI (azul). Ampliação de 100x. Barra de escala = 10 μm.(B) Representante 3D núcleos de peixe multicolor de linfócitos t primários humanos tratados para 12 min com 0,1 N HCl (esquerda) ou acoplado com 0,01 N HCl/0,0025% pepsin para 2 min (direita), utilizando mapeamento de sondas para 8q24,13 região (verde). Os núcleos são neutralizados com daPI (azul). Ampliação de 100x. Barra de escala = 10 μm.(C) Representante 3D núcleos de peixe multicolor de mioblastos primários humanos tratados com pepsinização curta e subótima, utilizando mapeamento de sondas para 8q24,13 região (magenta). Os núcleos são neutralizados com daPI (azul). Ampliação de 63x. Barra de escala = 10 μm.(D) Representante 3D núcleos de peixe multicolor de mioblastos primários humanos tratados com passo de pepsinização prolongado, utilizando mapeamento de sondas para 8q24,13 região (magenta). Os núcleos são neutralizados com daPI (azul). Ampliação de 63x. Barra de escala = 5 μm.(E) Representante 3D núcleos de peixe multicolorido de mioblastos primários humanos tratados com condições ideais de HCl/pepsina usando mapeamento de sondas para 8q24,13 região (magenta). Os núcleos são neutralizados com daPI (azul). Ampliação de 63x. Barra de escala = 25 μm. Clique aqui para ver uma versão maior deste valor.
| Reagentes de tradução de Nick | Concentração inicial | Concentração final |
| dNTPs (C-G-A) | 0,5 mM | 0,05 mM |
| dTTP | 0,1 mM | 0,01 mM |
| Biotina/Dig/Cy3 dUTP | 1 mM | 0,02 mM |
| Tris HCl pH 7.8 | 1 M | 50 mM |
| MgCl2 | 100 mM | 5 mM |
| β-mercaptoetanol | 100 mM | 10 mM |
| Bsa | 100 ng/μL | 10 ng/μL |
| DNA Pol I | 10 U/μL | 0,1 U/μL |
| DNase I | 1 U/μL | 0,002 U/μL |
| DNA 2 μg | X | X |
| ddH2O | Até 50 μL |
Tabela 1: Tradução de Nick. Tabela descrevendo todos os reagentes, sua concentração e tempo sugerido para reação de tradução de Nick.
Discussão
O método atual descreve um protocolo passo a passo para executar peixes de DNA multicoloridos 3D em uma ampla gama de células primárias humanas. Embora o DNA FISH seja uma tecnologia em grande uso, o Peixe de DNA multicolorido 3D em núcleos interfásicos 3D preservados ainda é difícil de executar em muitos laboratórios, principalmente devido às características das amostras utilizadas23,24.
A tradução de nick de sonda é um passo fundamental para o sucesso do DNA multicolor 3D FISH; muitos substratos diferentes (BAC, fosmid, plasmid, produtos PCR) podem ser usados para essa reação, e o tempo da reação e concentração enzimática pode ser ajustado em conformidade com relação ao comprimento do substrato. Uma digestão de sonda adequada é fundamental (Figura 1), pois sondas não ideais(Figura 2) resultarão em nenhum sinal ou um sinal não específico(Figura 3A). As etapas de permeabilização, desproteína e pepsinização são passagens cruciais que dependem fortemente do tipo celular utilizado. Células com pequenos núcleos e baixa abundância de citoesqueleto, como linfócitos T isolados ex vivo, requerem desproteinização com um tratamento prolongado de 0,1 N HCl. Além disso, lava-jatos na PBS com percentuais mais altos de Triton X-100 pode ajudar a entrada da sonda nos núcleos dessas células. Pelo contrário, mioblastos primários humanos cultivados in vitro que apresentam núcleos maiores, com alto teor de citoesqueleto, precisam de digestão das estruturas citosólicas com pepsina. Essas funções gerais podem ser aplicadas a uma ampla gama de células, eventualmente combinando os diferentes passos dependendo das características celulares específicas.
O uso de material biológico recém-preparado, soluções frescas (em particular soluções com detergente) e reagentes fluorescentes são fortemente sugeridos: PFA filtrado no pH 7.0; autocelava e filtrada 20x SSC em pH 7.0; formamida filtrada em pH 7.0; nuclease água livre; e alíquotas descartáveis de UTP modificado. A incubação prolongada com glicerol/PBS de 20%, ou 50% de formamide/2x SSC pode facilitar a hibridização. O tratamento de HCl e/ou pepsina pode ser ainda mais aumentado. O tempo de hibridização, a quantidade de sondas, a concentração e o tempo de incubação de antidigoxigenina e streptavidina podem ser ajustados ainda mais para melhorar a relação sinal-ruído.
O DNA FISH 3D multicolor representa uma ferramenta complementar às tecnologias C, o método padrão para validar resultados baseados em C. Se combinado com microscopia e análise 3D, o DNA 3D multicolor FISH pode monitorar a proximidade entre loci genômico e sua distribuição topológica dentro do espaço nuclear a um único nível celular. O Peixe de DNA multicolor 3D pode ser integrado com outras metodologias, como RNA FISH e imunofluorescência para uma visão geral abrangente da dinâmica e interações entre loci genômico, RNAs (RNA mensageiro ou RNA de não codificação regulatória) e uma ampla gama de proteínas, proporcionando uma oportunidade única para visualizar a estrutura nuclear e investigar os mecanismos epigenéticos que subtendem a identidade celular.
Apesar da enorme melhoria das tecnologias FISH com super resolução25,26, imagens de células vivas27,28,36, detecção de molécula única37, e visualização contemporânea de múltiplos alvos com matrizes oligonucleotídeas como Oligopaint37,38 com abordagens39 39, uma limitação da tecnologia continua sendo o número discreto de loci genômica predeterminado que pode ser visualizado. Isso impede uma ampla análise da arquitetura nuclear. Vários estudos descreveram recentemente métodos sequenciais de hibridização para abordar a organização do genoma em células únicas, como o DNA FISH40,41,42,43. Mais esforços serão necessários para acoplar a natureza celular única de Peixes de DNA multicoloridos 3D a características wide do genoma para visualizar amplamente a heterogeneidade da arquitetura nuclear com tecnologias de imagem, já que o número de loci que podem ser testados por vez aumentará.
Divulgações
Os autores não têm nada para divulgar.
Agradecimentos
Os autores reconhecem a assistência técnica do INM Imaging Facility (Istituto Nazionale di Genetica Molecolare "Romeo ed Enrica Invernizzi" (INGM), Milão, Itália), em particular C. Cordiglieri, para assistência durante imagens 3D multicolor DNA FISH Aquisição. Este trabalho tem sido apoiado pelas seguintes subvenções ao B.B.: Programa principal italiano epigen, Associação Française contre les Myopathies (AFM-Telethon, grant nr 18754) e Giovani Ricercatori, Ministério da Saúde italiano (GR-2011-02349383). Este trabalho tem sido apoiado pela seguinte concessão ao F.M.: Fondazione Cariplo (Bando Giovani, bolsa nR 2018-0321).
Materiais
| Name | Company | Catalog Number | Comments |
| 24-well plates | Thermo Fisher Scientific | 142475 | |
| 6-well plates | Thermo Fisher Scientific | 140675 | |
| Anti-Digoxigenin 488 | DBA | DI7488 | |
| b-Mercaptoethanol | Sigma | M3148 | |
| bFGF | PeproTech | 100-18B | |
| Biotin 11 d-UTP | Thermo Fisher Scientific | R0081 | |
| BSA (bovine serum albumine) | Sigma | A7030 | |
| Coverlsips | Marienfeld | 117500 | |
| CY3 d-UTP | GE Healthcare | PA53022 | |
| DAPI (4,6-diamidino-2-phenylindole) | Thermo Fisher Scientific | D21490 | |
| Deoxyribonucleic acids single strand from salmon testes | Sigma | D7656 | |
| Dextran sulfate (powder) | Santa Cruz | sc-203917A | |
| Digoxigenin 11 d-UTP | Roche | 11093088910 | |
| DMEM | Thermo Fisher Scientific | 21969-035 500mL | |
| DNA polymerase I | Thermo Fisher Scientific | 18010-017 | |
| DNase I | Sigma | AMPD1 | |
| dNTPs (C-G-A-T) | Euroclone | BL0423A/C/G | |
| EGF | Sigma | E9644.2MG | |
| Ethanol | Sigma | 02860-1L | |
| FBS Hyclone | Thermo Fisher Scientific | SH30109 | |
| Formaldehyde solution | Sigma | F8775-25mL | |
| Formamide | Sigma | F9037 | |
| Glutammine | Thermo Fisher Scientific | 25030-024 100mL | |
| Glycerol | Sigma | G5516-100mL | |
| Glycogen | Thermo Fisher Scientific | AM9510 | |
| HCl | Sigma | 30721 | |
| Human Cot-1 DNA | Thermo Fisher Scientific | 15279-001 | |
| Insulin Human | Sigma | I9278-5 mL | |
| MgCl2 | Sigma | 63069 | |
| NaAc (Sodium Acetate, pH 5.2, 3 M) | Sigma | S2889 | |
| NaCl | Sigma | S9888 | |
| Paraformaldehyde | Sigma | 158127-25G | |
| PBS (phosphate-buffered saline) | Sigma | P4417 | |
| Pennycillin/Streptavidin | Thermo Fisher Scientific | 15070-063 100mL | |
| Pepsin | Biorad | P6887 | |
| PhasePrep BAC DNA Kit | Sigma | NA0100-1KT | |
| Poly-L-lysine solution | Sigma | P8920 | |
| ProLong Diamond Antifade Mountant | Thermo Fisher Scientific | P36970 | |
| PureLink Quick Gel Extraction & PCR Purification Combo Kit | Thermo Fisher Scientific | K220001 | |
| PureLink Quick Plasmid Miniprep Kit | Thermo Fisher Scientific | K210010 | |
| RNAse cocktail | Thermo Fisher Scientific | AM2288 | |
| Rubbercement | Bostik | ||
| Slides | VWR | 631-0114 | |
| Streptavidina Alexa fluor 647 | Thermo Fisher Scientific | S21374 | |
| Tri-Sodium Citrate | Sigma | 1110379026 | |
| Tris-HCl | Sigma | T3253-500g | |
| Triton X-100 | Sigma | T8787-250mL | |
| TWEEN 20 | Sigma | P9416-100mL |
Referências
- Cremer, T., Cremer, C. Chromosome territories, nuclear architecture and gene regulation in mammalian cells. Nature Reviews: Genetics. 2 (4), 292-301 (2001).
- Lieberman-Aiden, E., et al. Comprehensive mapping of long-range interactions reveals folding principles of the human genome. Science. 326 (5950), 289-293 (2009).
- Schmitt, A. D., Hu, M., Ren, B. Genome-wide mapping and analysis of chromosome architecture. Nature Reviews: Molecular Cell Biology. 17 (12), 743-755 (2016).
- Bonev, B., Cavalli, G. Organization and function of the 3D genome. Nature Reviews: Genetics. 17 (12), 772(2016).
- van Steensel, B., Furlong, E. E. M. The role of transcription in shaping the spatial organization of the genome. Nature Reviews: Molecular Cell Biology. 20 (6), 327-337 (2019).
- Rajarajan, P., Gil, S. E., Brennand, K. J., Akbarian, S. Spatial genome organization and cognition. Nature Reviews: Neuroscience. 17 (11), 681-691 (2016).
- Pombo, A., Dillon, N. Three-dimensional genome architecture: players and mechanisms. Nature Reviews: Molecular Cell Biology. 16 (4), 245-257 (2015).
- Fanucchi, S., Shibayama, Y., Burd, S., Weinberg, M. S., Mhlanga, M. M. Chromosomal contact permits transcription between coregulated genes. Cell. 155 (3), 606-620 (2013).
- Therizols, P., et al. Chromatin decondensation is sufficient to alter nuclear organization in embryonic stem cells. Science. 346 (6214), 1238-1242 (2014).
- Gonzalez-Sandoval, A., et al. Perinuclear Anchoring of H3K9-Methylated Chromatin Stabilizes Induced Cell Fate in C. elegans Embryos. Cell. 163 (6), 1333-1347 (2015).
- Hubner, B., et al. Remodeling of nuclear landscapes during human myelopoietic cell differentiation maintains co-aligned active and inactive nuclear compartments. Epigenetics Chromatin. 8, 47(2015).
- Aymard, F., et al. Genome-wide mapping of long-range contacts unveils clustering of DNA double-strand breaks at damaged active genes. Nature Structural & Molecular Biology. 24 (4), 353-361 (2017).
- Sellou, H., et al. The poly(ADP-ribose)-dependent chromatin remodeler Alc1 induces local chromatin relaxation upon DNA damage. Molecular Biology of the Cell. 27 (24), 3791-3799 (2016).
- Krijger, P. H., de Laat, W. Regulation of disease-associated gene expression in the 3D genome. Nature Reviews: Molecular Cell Biology. 17 (12), 771-782 (2016).
- Lupianez, D. G., Spielmann, M., Mundlos, S. Breaking TADs: How Alterations of Chromatin Domains Result in Disease. Trends in Genetics. 32 (4), 225-237 (2016).
- Cortesi, A., et al. 4q-D4Z4 chromatin architecture regulates the transcription of muscle atrophic genes in facioscapulohumeral muscular dystrophy. Genome Research. 29 (6), 883-895 (2019).
- Woltering, J. M., Noordermeer, D., Leleu, M., Duboule, D. Conservation and divergence of regulatory strategies at Hox Loci and the origin of tetrapod digits. PLoS Biology. 12 (1), 1001773(2014).
- Woltering, J. M., Duboule, D. Tetrapod axial evolution and developmental constraints; Empirical underpinning by a mouse model. Mechanisms of Development. 138, Pt 2 64-72 (2015).
- de Laat, W., Dekker, J. 3C-based technologies to study the shape of the genome. Methods. 58 (3), 189-191 (2012).
- Denker, A., de Laat, W. The second decade of 3C technologies: detailed insights into nuclear organization. Genes & Development. 30 (12), 1357-1382 (2016).
- Finn, E. H., et al. Extensive Heterogeneity and Intrinsic Variation in Spatial Genome Organization. Cell. 176 (6), 1502-1515 (2019).
- Zheng, M., et al. Multiplex chromatin interactions with single-molecule precision. Nature. 566 (7745), 558-562 (2019).
- Solovei, I. Fluorescence in situ hybridization (FISH) on tissue cryosections. Methods in Molecular Biology. 659, 71-82 (2010).
- Cremer, M., et al. Multicolor 3D fluorescence in situ hybridization for imaging interphase chromosomes. Methods in Molecular Biology. 463, 205-239 (2008).
- Legant, W. R., et al. High-density three-dimensional localization microscopy across large volumes. Nature Methods. 13 (4), 359-365 (2016).
- Jungmann, R., et al. Quantitative super-resolution imaging with qPAINT. Nature Methods. 13 (5), 439-442 (2016).
- Chen, B., et al. Dynamic imaging of genomic loci in living human cells by an optimized CRISPR/Cas system. Cell. 155 (7), 1479-1491 (2013).
- Nelles, D. A., et al. Programmable RNA Tracking in Live Cells with CRISPR/Cas9. Cell. 165 (2), 488-496 (2016).
- Byron, M., Hall, L. L., Lawrence, J. B. A multifaceted FISH approach to study endogenous RNAs and DNAs in native nuclear and cell structures. Current Protocol of Human Genetics. , Chapter 4, Unit 4 15(2013).
- Chaumeil, J., Augui, S., Chow, J. C., Heard, E. Combined immunofluorescence, RNA fluorescent in situ hybridization, and DNA fluorescent in situ hybridization to study chromatin changes, transcriptional activity, nuclear organization, and X-chromosome inactivation. Methods in Molecular Biology. 463, 297-308 (2008).
- Takizawa, T., Gudla, P. R., Guo, L., Lockett, S., Misteli, T. Allele-specific nuclear positioning of the monoallelically expressed astrocyte marker GFAP. Genes & Development. 22 (4), 489-498 (2008).
- Kraus, F., et al. Quantitative 3D structured illumination microscopy of nuclear structures. Nature Protocols. 12 (5), 1011-1028 (2017).
- Solovei, I., Cremer, M. 3D-FISH on cultured cells combined with immunostaining. Methods in Molecular Biology. 659, 117-126 (2010).
- Skinner, B. M., Johnson, E. E. Nuclear morphologies: their diversity and functional relevance. Chromosoma. 126 (2), 195-212 (2017).
- Schoenfelder, S., et al. The pluripotent regulatory circuitry connecting promoters to their long-range interacting elements. Genome Research. 25 (4), 582-597 (2015).
- Lubeck, E., Cai, L. Single-cell systems biology by super-resolution imaging and combinatorial labeling. Nature Methods. 9 (7), 743-748 (2012).
- Beliveau, B. J., et al. Single-molecule super-resolution imaging of chromosomes and in situ haplotype visualization using Oligopaint FISH probes. Nature Communication. 6, 7147(2015).
- Beliveau, B. J., et al. Versatile design and synthesis platform for visualizing genomes with Oligopaint FISH probes. Proceedings of the National Academy of Sciences. 109 (52), 21301-21306 (2012).
- Samacoits, A., et al. A computational framework to study sub-cellular RNA localization. Nature Communication. 9 (1), 4584(2018).
- Cardozo Gizzi, A. M., et al. Microscopy-Based Chromosome Conformation Capture Enables Simultaneous Visualization of Genome Organization and Transcription in Intact Organisms. Molecular Cell. 74 (1), 212-222 (2019).
- Mateo, L. J., et al. Visualizing DNA folding and RNA in embryos at single-cell resolution. Nature. 568 (7750), 49-54 (2019).
- Nir, G., et al. Walking along chromosomes with super-resolution imaging, contact maps, and integrative modeling. PLoS Genetics. 14 (12), 1007872(2018).
- Bintu, B., et al. Super-resolution chromatin tracing reveals domains and cooperative interactions in single cells. Science. 362 (6413), (2018).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados