Method Article
3D Multicolor DNA FISH Outil pour étudier l'architecture nucléaire dans les cellules primaires humaines
Dans cet article
Résumé
3D multicolor DNA FISH représente un outil pour visualiser les loci génomiques multiples dans les noyaux préservés 3D, définissant sans ambiguïté leurs interactions réciproques et leur localisation dans l'espace nucléaire à un seul niveau cellulaire. Ici, un protocole étape par étape est décrit pour un large éventail de cellules primaires humaines.
Résumé
Une question majeure en biologie cellulaire est l'organisation génomique dans l'espace nucléaire et comment l'architecture de chromatine peut influencer des processus tels que l'expression des gènes, l'identité cellulaire et la différenciation. De nombreuses approches développées pour étudier l'architecture 3D du génome peuvent être divisées en deux catégories complémentaires : les technologies basées sur la conformation chromosomique (technologies C) et l'imagerie. Alors que le premier est basé sur la capture de la conformation chromosomique et des interactions d'ADN proximal dans une population de cellules fixes, le second, basé sur la fluorescence de l'ADN in situ hybridation (FISH) sur des noyaux conservés en 3D, permet la visualisation contemporaine de loci multiples à un seul niveau cellulaire (multicolor), examinant leurs interactions et leur distribution dans le noyau (3D MULTIcolor DNA FISH). La technique de l'ADN multicolore 3D FISH a une limitation de visualiser seulement quelques loci prédéterminés, ne permettant pas une analyse complète de l'architecture nucléaire. Cependant, compte tenu de la robustesse de ses résultats, 3D multicolor DNA FISH en combinaison avec la microscopie 3D et la reconstruction d'image est une méthode possible pour valider les résultats basés sur la technologie C et d'étudier sans ambiguïté la position et l'organisation de loci spécifiques au niveau d'une seule cellule. Ici, nous proposons une méthode étape par étape de 3D MULTIcolor DNA FISH adapté à un large éventail de cellules primaires humaines et discuter de toutes les actions pratiques, étapes cruciales, notions d'imagerie 3D et l'analyse des données nécessaires pour obtenir un succès et instructif 3D multicolore DNA FISH dans différents contextes biologiques.
Introduction
Les eucaryotes plus élevés doivent condenser et compacter systématiquement une énorme quantité d'informations génétiques dans l'espace 3D minute du noyau1,2,3,4. Aujourd'hui, nous savons que le génome est classé spatialement dans des compartiments et des domaines topologiquement associés5 et que les niveaux multiples de pliage de l'ADN génèrent des contacts entre différentes régions génomiques qui peuvent impliquer la formation de boucle de chromatine6,7. La boucle dynamique 3D de la chromatine peut influencer de nombreux processus biologiques différents tels que la transcription8,9, la différenciation et le développement10,11, réparation de l'ADN12,13, tandis que ses perturbations sont impliqués dans diverses maladies14,15,16 et les défauts de développement15,17,18.
De nombreuses approches ont été développées pour étudier l'organisation du génome 3D. Des technologies basées sur le capture de conformation de chromosome (C-technologies, 3C, 4C, 5C, Hi-C et dérivés) ont été développées pour étudier l'organisation de génome dans les cellules fixes3,4,19,20. Ces approches sont basées sur la capacité de capturer les fréquences de contact entre les loci génomiques à proximité physique. C-technologies, en fonction de leur complexité, attraper l'organisation mondiale du génome 3D et la topologie nucléaire d'une population cellulaire3,4,19,20. Néanmoins, les interactions 3D sont dynamiques dans le temps et l'espace, très variables entre les cellules individuelles composées d'interactions multiplexes, et sont largement hétérogènes21,22.
L'hybridation 3D de fluorescence in situ de l'ADN multicolore (FISH) est une technique qui permet la visualisation de loci génomiques spécifiques à un seul niveau de cellule, permettant une étude directe de l'architecture nucléaire 3D d'une manière complémentaire aux technologies C. Il représente une technologie actuellement utilisée pour valider sans ambiguïté les résultats C. 3D multicolor DNA FISH utilise des sondes étiquetées fluorescentes complémentaires aux loci génomiques d'intérêts. L'utilisation de différents fluorophores et d'équipements de microscopie appropriés permet la visualisation contemporaine de cibles multiples dans l'espace nucléaire23,24. Ces dernières années, FISH a été combiné avec les progrès technologiques dans la microscopie pour obtenir la visualisation des structures à échelle fine à haute résolution25,26 ou avec des approches CRISPR-Cas pour la visualisation des acides nucléiques en imagerie en direct27,28. Malgré une large adoption, l'approche 3D multicolor DNA FISH est encore considérée comme difficile dans de nombreux laboratoires car le matériel biologique utilisé doit être adapté.
Ici, nous fournissons un protocole complet pour 3D multicolor DNA FISH (de la préparation cellulaire / sonde à l'analyse des données) applicable à un large éventail de cellules primaires humaines, permettant la visualisation de loci génomiques multiples et la préservation de la structure 3D des noyaux. Afin d'étudier l'architecture nucléaire, la structure 3D des noyaux doit être préservée. Pour cette raison, contrairement à d'autres protocoles existants29,30,31, nous évitons l'utilisation d'un gradient d'alcool et le stockage des couvertures dans l'alcool qui peut affecter la structure de chromatine32. La méthode est adaptée à partir de protocoles FISH d'ADN 3D conservés24,33 à appliquer à un large éventail de cellules primaires humaines, à la fois isolées ex vivo ou cultivées in vitro. Il existe des paramètres de perméabilisation et de déprotéinisation pour différentes morphologies nucléaires et caractéristiques cytologiques (p. ex., différents degrés de compactage nucléaire, abondance de cytosquelettes)34. Ces paramètres sont souvent généralement décrits dans d'autres protocoles24,33, sans fournir une discrimination claire de la procédure au sein de différents types de cellules. En outre, nous avons développé un outil spécifique nommé NuCLD (localisateur de contacts nucléaires en 3D)16, fournissant des principes pour l'analyse des données qui permettra d'améliorer la proximité 3D entre les différents loci et leur distribution topologique nucléaire dans l'espace nucléaire d'une manière automatisée.
Protocole
1. Procédures de préparation et d'étiquetage de la sonde d'ADN avec traduction de pseudo
- Purifiez et nettifiez les chromosomes artificiels bactériens (BAC), les plasmides ou les produits PCR avec des kits spécifiques (Table of Materials), resuspend in ddH2O, vérifier par électrophorèse sur un gel agarose et quantifier.
- Effectuer une traduction de pseudo sur 1,5 à 2 g d'ADN à partir de l'étape 1.1 dans un volume final de 50 L en mélangeant tous les réactifs dans un tube d'ADN liant de 0,5 mL bas selon le tableau 1.
REMARQUE : Les sondes directement étiquetées peuvent améliorer le rapport signal/bruit. Des kits pour produire des sondes indirectement et directement étiquetées avec divers fluorochromes Alexa par traduction de pseudo sont disponibles dans le commerce. - Incuber le mélange de traduction de pseudo dans un mélangeur thermique à 16 oC pendant un certain temps en fonction de la longueur du matériel d'ADN de départ : 45 min pour les produits PCR (un pool de produits PCR de 2 000 bp chacun) et jusqu'à 4 h pour l'ADN BAC et les plasmides.
- Vérifiez la taille des sondes produites à l'étape 1.3 par électrophoresis sur un gel agarose de 2,2%.
REMARQUE : La taille optimale de la sonde est de 200 pb(figure 1A).- Si l'ADN n'est pas assez digéré, ajoutez 5 U de polymérase d'ADN I et 0,05 U de DNase I à la réaction et incubez pendant 1 à 2 h à 16 oC et vérifiez par la suite. Arrêtez la réaction avec 0.5 mM EDTA (concentration finale). Stocker les sondes à -20 oC.
- Pour chaque expérience DNA FISH, précipitez les quantités suivantes de sondes en fonction du matériel d'ADN de départ à partir duquel les sondes sont produites : 200 ng de produits PCR traduits en pseudo, 100 ng de BAC traduits en pseudo, ou 300 ng de pseudo traduit plasmide. Ajouter ddH2O jusqu'à 150 O, 20 g d'ADN de sperme de saumon non étiqueté, 3,5 g d'ADN Cot-1 spécifique à l'espèce, 3 volumes de 100 % EtOH et 1/10 volume de 3 M d'acétate de sodium pH 5,2. Précipitation à -80 oC pendant 1 h.
- Centrifugeàeur à vitesse maximale pendant 1 h à 4 oC et jetez le supernatant. Laver la pastille deux fois avec 70% EtOH.
- Resuspendre la pastille en 2 l de 100% de pH 7.0 formamide, secouer à 40 oC pendant 30 minutes (peut prendre jusqu'à quelques heures) puis ajouter un volume égal de 4x de citrate saline-sodium (SSC)/20% de sulfate de dextran.
- Préparer 20x SSC en mélangeant 175,3 g de NaCl et 88,2 g de citrate de sodium dans un volume final de 1 L de H2O. Autoclave, filtrer et préparer les aliquots.
REMARQUE: Pour l'ADN multicolore FISH, les différentes sondes peuvent être précipitées ensemble, sauf pour les sondes qui peuvent annexer les uns avec les autres. Dans ces cas spécifiques, traitez-les séparément, précipitez et suspendez de nouveau les sondes divisant les réactifs mentionnés dans les étapes 1.5-1.7 en ce qui concerne le nombre de sondes. Pool les sondes seulement après la résuspension dans formamide. Si des couvercles en verre de plus de 10 mm ou moins de 10 mm sont utilisés, augmenter/descendre le volume des réactifs utilisés dans les étapes 1,5 à 1,7 proportionnellement à la taille de la glissière. Les sondes d'hybridation peuvent être stockées à -20 oC pendant une longue période (jusqu'à deux mois).
- Préparer 20x SSC en mélangeant 175,3 g de NaCl et 88,2 g de citrate de sodium dans un volume final de 1 L de H2O. Autoclave, filtrer et préparer les aliquots.
2. Fixation cellulaire, prétraitement et perméabilisation
REMARQUE : Les passages de perméabilisation et de déprotéinisation sont des étapes cruciales. Le temps de réaction et de concentration des réactifs dépend fortement du type de cellule, de l'abondance du cytoplasme et de la morphologie nucléaire.
- Fixation cellulaire, prétraitement et perméabilisation pour les lymphocytes T primaires humains
REMARQUE: Ce protocole est adapté pour les petites cellules, avec de petits noyaux et une faible quantité de cytoplasme.- Utilisez des couvercles en verre (10 mm, épaisseur No. 1.5H).
- Laver le verre avec ddH2O, puis avec 70% EtOH et les laisser sécher. Utilisez un verre pour chaque puits d'une assiette de 24 puits.
- Ajouter 200 l de 0,1 % de polylysine (w/v)(tableau des matériaux)directement sur le verre pour former une goutte. Faites attention car la goutte doit rester sur la surface de verre sans toucher le puits pendant 2 à 5 min. Laissez la goutte soigneusement, et laissez le verre sécher pendant 30 min.
- Effectuez l'étape 2.1.3 deux fois plus.
- Mettre 200 l de cellules de suspension (2 x 106/mL, suspension PBS des lymphocytes T primaires ex vivo) directement sur le verre, permettent aux cellules de semer à température ambiante (RT) pendant 30 min.
- Retirez rapidement la goutte. Ajouter le PFA fraîchement préparé à 4 % (préparé en PBS/0,1 % TWEEN 20, pH 7,0, filtré) pendant 10 min et fixer les cellules ensemoit à RT.
- Laver trois fois pendant 5 min chacun avec 0,05% Triton X-100/PBS (TPBS) à RT. Permeabilize avec 0,5% TPBS pour 10 min à RT.
- Effectuer le traitement de RNase en ajoutant 2,5 l de cocktail RNase(Table of Materials) dans 250 'L de PBS/puits dans une assiette de 24 puits pendant 1 h à 37 oC.
- Rincer dans le PBS, ajouter 20 % de glycérol/PBS et couver toute la nuit (ON) à 4 oC.
REMARQUE: Cette étape peut varier de 1 h à ON. Les glissières peuvent être conservées dans 20 % de glycérol/PBS à 4 oC jusqu'à 7 jours. - Congeler sur de la glace sèche (15 à 30 s), décongeler graduellement à rt et tremper dans 20 % de glycérol/PBS. Répétez l'étape 2.1.9 encore trois fois.
- Laver en TPBS de 0,5 % pendant 5 min à RT. Wash en 0,05 % TPBS, deux fois pendant 5 min à RT. Incubate en 0.1 N HCl pendant 12 min à RT. Rinse en 2x SSC.
- Incubate dans 50% formamide pH 7.0/2x SSC ON à RT.
REMARQUE : Le temps d'incubation peut être optimisé et éventuellement réduit. Les glissières peuvent être conservées dans 50% formamide/2x SSC pendant plusieurs jours.
- Fixation cellulaire, prétraitement et perméabilisation des myoblastes primaires humains
REMARQUE: Ce protocole est adapté pour les grandes cellules, avec une grande quantité de cytoplasme.- Cultivez les myoblastes primaires humains directement sur les verres de couverture dans des plaques de 24 puits dans le milieu de croissance (le milieu d'aigle modifié de Dulbecco (DMEM), 20% de sérum bovin foetal (FBS), le facteur de croissance de fibroblaste de 25 ng/mL (FGF), le facteur épidermique de 10 ng/mL (EGF), l'insuline humaine de 10 g/mL, 1x glutamine, 1x pénicilline/streptomycine) pendant au moins 24 h (atteignant 50 à 70% de la confluence).
REMARQUE : Pour faciliter l'adhérence, la gélatine ou le collagène ou la poly-l-lysine peuvent être utilisés pour enrober le bordereau avant l'ensemencement. - Rincer les cellules dans les changements de 2 à 3 de PBS. Ajouter fraîchement fait 4% PFA et fixer les cellules pendant 10 min à RT.
- Laver trois fois pendant 3 min chacun en 0,01% TPBS à RT. Permeabilize en 0,5% TPBS pour 10 min à RT.
- Effectuer le traitement de RNase en ajoutant 2,5 l de cocktail RNase(Table of Materials) dans 250 'L de PBS/puits dans une assiette de 24 puits pendant 1 h à 37 oC.
- Rincer dans PBS, ajouter 20% de glycérol/PBS, et incuber ON à RT.
REMARQUE: Cette étape peut varier de 1 h à ON. Les glissières peuvent être conservées dans 20 % de glycérol/PBS à 4 oC jusqu'à 7 jours. - Congeler sur de la glace sèche (15 à 30 s), décongeler graduellement à rt et tremper dans 20 % de glycérol/PBS. Répétez cette étape trois fois.
- Laver trois fois pendant 10 min chacun en PBS. Incuber en 0.1 N HCl pendant 5 min à RT. Rinse en 2x SSC.
- Incubate dans 50% formamide pH 7.0/2x SSC ON à RT.
REMARQUE : Le temps d'incubation peut être optimisé et éventuellement réduit. Les glissières peuvent être conservées dans 50% formamide/2x SSC pendant plusieurs jours. - Diapositives d'équilibre (maintenues dans 50% formamide/2x SSC) en 2x SSC pendant 2 min. Ensuite, équilibrez en PBS pendant 3 min.
- Traiter avec 0,01 N HCl/0.0025% pepsine de quelques secondes jusqu'à 5 min, selon le type de cellule. Au cours de cette étape, observez les cellules sous un microscope optique et arrêtez la réaction (étape 2.2.11) dès que les noyaux sont exempts du cytoplasme, tout en maintenant leur structure intacte (par exemple, les nucléoles sont encore visibles et intacts).
- Inactiver la pepsine en lavant deux fois pendant 5 min chacun e en 50 mM MgCl2/PBS.
- Post-fixer dans 1% PFA/PBS pendant 1 min. Laver pendant 5 min en PBS. Laver deux fois pendant 5 min chacun en 2x SSC, puis ajouter 50% formamide/2x SSC pendant au moins 30 min.
- Cultivez les myoblastes primaires humains directement sur les verres de couverture dans des plaques de 24 puits dans le milieu de croissance (le milieu d'aigle modifié de Dulbecco (DMEM), 20% de sérum bovin foetal (FBS), le facteur de croissance de fibroblaste de 25 ng/mL (FGF), le facteur épidermique de 10 ng/mL (EGF), l'insuline humaine de 10 g/mL, 1x glutamine, 1x pénicilline/streptomycine) pendant au moins 24 h (atteignant 50 à 70% de la confluence).
3. Hybridation 3D multicolorE D'ADN FISH
- Dénaturer les sondes à 80 oC pendant 5 min, puis les mettre rapidement sur la glace.
- Chargez les sondes d'hybridation sur une lame de microscope propre. Tournez le bordereau avec des cellules à l'envers sur la goutte de sondes d'hybridation.
- Scellez la glissière de couverture avec du ciment en caoutchouc. Laisser sécher complètement le ciment en caoutchouc. Ensuite, placez les glissières sur un bloc de chauffage et dénaturez à 75 oC pendant 4 min.
REMARQUE : Le moment de la dénaturation et la température de la dénaturation peuvent être augmentés, jusqu'à 80 oC. - Hybrider à 37 oC ON dans une boîte métallique flottant dans un bain d'eau.
REMARQUE : Pour améliorer le rapport signal/bruit, la température d'hybridation peut aller jusqu'à 42 oC. - Épluchez le ciment en caoutchouc, immerger les lames dans 2x SCC, dépouiller le bordereau en verre et le transférer à 2x SSC dans une assiette de 6 puits.
- Laver en 2x SSC trois fois pendant 5 min chacun à 37 oC, en secouant à 90 tr/min dans un shaker d'incubateur. Laver en 0,1x SSC trois fois pendant 5 min chacun à 60 oC, en secouant à 90 tr/min dans un shaker d'incubateur. Rincer brièvement dans 4x SSC/0.2% TWEEN 20.
4. Détection 3D multicolore d'ADN FISH
REMARQUE : Pour les sondes directement étiquetées, sautez les étapes 4.1 et 4.2.
- Bloquer dans 4x SSC/0.2% TWEEN 20/4% d'albumine de sérum bovin (BSA) pendant 20 min à 37 oC dans une assiette de 24 puits, en secouant à 20 tr/min dans un shaker d'incubateur.
- Incuber avec la concentration appropriée d'anti-digoxygénine (1:150), et/ ou de streptavidin (1:1,000) (Tableau des matériaux) dilué dans 4x SSC/0.2% TWEEN 20/4% BSA pendant 35 min dans une chambre sombre et humide à 37 oC.
- Laver en 4x SSC/0,2% TWEEN 20 trois fois pendant 5 min chacun à 37 oC, en secouant à 90 tr/min dans une assiette de 6 puits dans un shaker.
- Équilibre dans PBS et post-fixe dans 2% formaldéhyde/PBS pendant 2 min à RT dans une plaque de 24 puits.
REMARQUE : Les sondes directement étiquetées n'ont pas besoin de post-fixation. - Laver 5x brièvement dans PBS. Tache avec 1 ng/mL DAPI (4,6-diamidino-2-phenylindole)/PBS pendant 5 min à RT.
- Laver 5x brièvement dans PBS. Mont avec solution antifade (Tableau des Matériaux).
5. Microscopie et analyse 3D multicolorDNA FISH
- Acquérir des images 3D avec un système de microscope.
REMARQUE : Ici, un microscope à champ large avec une distance axiale de 0,2 à 0,25 m entre les sections consécutives (figure 1B) est utilisé. - Analysez les piles d'images 3D à l'aide de différents logiciels et outils (ici, NuCLMD ou Nuclear Contacts Locator en 3D).
REMARQUE : L'outil NuCLMD a été développé afin d'analyser automatiquement l'ADN multicolor edeur3D FISH dans les piles z d'image cellulaire de fluorescence. NuCL-D reconstitue les noyaux à partir de piles d'images cellulaires en 3D et détecte et localise les taches 3D fluorescentes. Il mesure le positionnement relatif des taches dans le noyau (p. ex., la distance par rapport au centroïde des noyaux et/ou de la périphérie des noyaux) et le rayon maximal pour chaque noyau et chaque distance entre les taches. L'outil et l'algorithme utilisé sont entièrement décrits dans Cortesi et al.16. - Analyser les données (p. ex., distances 3D entre les loci génomiques spécifiés et les fréquences de contact et de centroïdes nucléaires) récupérées par NuCL-D.
REMARQUE : Pour les distances 3D entre les loci génomiques spécifiés et le centroïde nucléaire, normalisez les distances sur le rayon maximum de chaque noyau et représentent ces données comme des distributions de fréquences des distances normalisées du centroïde nucléaire. Pour les études d'interactions à longue portée, les fréquences de contact sont censées être de l'ordre de 10 à 20 %21 avec un seuil inter-distance qui peut varier et peut être mis autour de 2 m35.- Pour effectuer une analyse statistique, analysez environ 100 noyaux par réplique biologique. Représenter les distances 3D entre les loci génomiques spécifiés comme des distributions cumulatives de fréquences inférieures au seuil inter-distance sélectionné et utiliser un test tpour évaluer l'importance des différences dans les distributions. En outre, calculer le pourcentage de noyaux positifs pour les interactions qui sont en dessous du seuil inter-distance sélectionné, et utiliser le test exact de Fisher pour évaluer l'importance des différences dans les pourcentages.
Résultats
La méthode de l'ADN multicolore 3D FISH décrit dans cet article permet la visualisation contemporaine de différents loci génomiques dans les noyaux 3D conservés (Figure 1B). Ce protocole permet de mesurer les distances entre les allèles et les différents loci génomiques afin d'évaluer leur proximité spatiale et d'évaluer leur emplacement dans l'espace nucléaire (p. ex., distance loci du centroïde ou de la périphérie des noyaux)16. Cependant, il existe de nombreuses étapes cruciales qui doivent être correctement et spécifiquement mis en place pour chaque type de cellule utilisée; il est fortement recommandé de prêter une attention particulière aux étapes suivantes pour le succès de 3D multicolor DNA FISH.
Pour la préparation de la sonde d'ADN, vérifiez que la taille de la sonde est de 200 pb (Figure 1A). Cette taille assure une procédure réussie de 3D multicolor DNA FISH (Figure 1B). Les sondes FISH d'ADN sous-optimaleproduites produites par la traduction de pseudopeuvent peuvent être partiellement digérées (Figure 2A) ou surdisées (Figure 2B). Avec des sondes partiellement digérées, la procédure n'aura aucun signal dans les cellules, en raison de l'incapacité de la sonde à entrer dans les noyaux et à s'hybrider correctement aux loci génomiques complémentaires. Les sondes surdisées entraîneront un signal non spécifique, en raison d'une perte de spécificité dans l'hybridation et d'une augmentation conséquente de l'arrière-plan. Un exemple représentatif de sondes surdigérées est montré dans la figure 3A par rapport à une sonde digérée optimale dans la figure 3B.
Pour la déprotéinisation et la pepsinisation, suivez ces étapes en fonction du type de cellule. En particulier, prendre en considération la taille nucléaire et l'abondance du cytoplasme. Pour les lymphocytes T et les cellules T isolés ex vivo humains avec de petits noyaux très compactés et un cytoplasme faiblement abondant, la déprotéinisation de HCl est cruciale. Le traitement avec 0.1 N HCl pendant 5 min n'est pas suffisant pour la visualisation d'ADN FISH. 0.1 N Le traitement HCl pendant 12 min est recommandé pour promouvoir l'accessibilité des noyaux aux sondes d'ADN et préserver l'intégrité nucléaire (Figure 4A). La digestion pepsine du cytoplasme n'est pas nécessaire pour obtenir un bon signal d'ADN FISH (Figure 4B).
Pour les myoblastes primaires humains et les cellules qui ont de grands noyaux et cytoplasme abondant, l'étape de pepsinisation est fondamentale. Une pepsinisation courte et sous-optimale du cytosquelette entravera l'entrée de la sonde dans les noyaux (Figure 4C), se terminant par l'absence d'un signal FISH D'ADN. Cependant, si les cellules sont surpepsiées, les noyaux ne resteront pas intacts (figure 4D), perdant leur structure 3D. Un exemple de FISH d'ADN multicolore 3D réussi est fourni dans la figure 4E.
Pendant l'hybridation, sceller la glissière de couverture avec précision; sinon, la sonde se dispersera et séchera. Les étapes de dénaturation et d'hybridation doivent être effectuées rapidement de telle sorte que la sonde et l'ADN génomique ne soient pas rénannés. La durée de la dénaturation peut être augmentée.

Figure 1 : Sondes d'ADN représentatives et FISH d'ADN multicolore 3D. (A) Nick a traduit des sondes d'ADN de taille optimale exécuté sur un gel agarose de 2,2% (voie 1, 2), marqueur de 50 bp (M). (B) Representative 3D multicolor DNA FISH nucleus using probes mapping to 3q11.2 region (green), 10q26.3 region (red) and 8q24.13 region (magenta) in human primary myoblasts. Les noyaux sont contrecarrés avec DAPI (bleu). 63x grossissement. Barre d'échelle de 5 m. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.

Figure 2 : Exemples de sondes FISH d'ADN non digérées de façon optimale. (A) Non digéré (voie 1, 2) ou partiellement digéré (voie 3) pseudo traduit sondes d'ADN exécuté sur un gel agarose 2,2%, marqueur 2log (M). (B) Sur les sondes d'ADN traduites par pseudo digérée s'exécutent sur un gel agarose de 2,2 %, marqueur 2log (M). Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
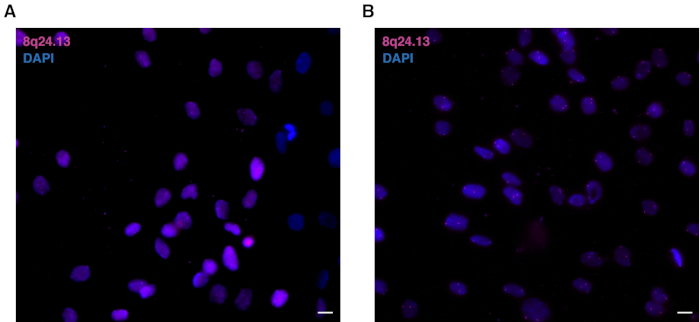
Figure 3 : Comparaison de FISH d'ADN multicolore 3D à l'aide de sondes FISH sous-optimales ou optimales. (A) Représentant 3D multicolor DNA FISH noyaux utilisant la cartographie de sonde surdigérée à 8q24.13 région (magenta) dans les myoblastes primaires humains. Les noyaux sont contrecarrés avec DAPI (bleu). 63x grossissement. Barre d'échelle de 10 m. (B) Les noyaux d'ADN multicolore 3D représentant FISH utilisant la cartographie de sonde digérée de manière optimale à la région 8q24.13 (magenta) dans les myoblastes primaires humains. Les noyaux sont contrecarrés avec DAPI (bleu). 63x grossissement. Barre d'échelle de 10 m. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
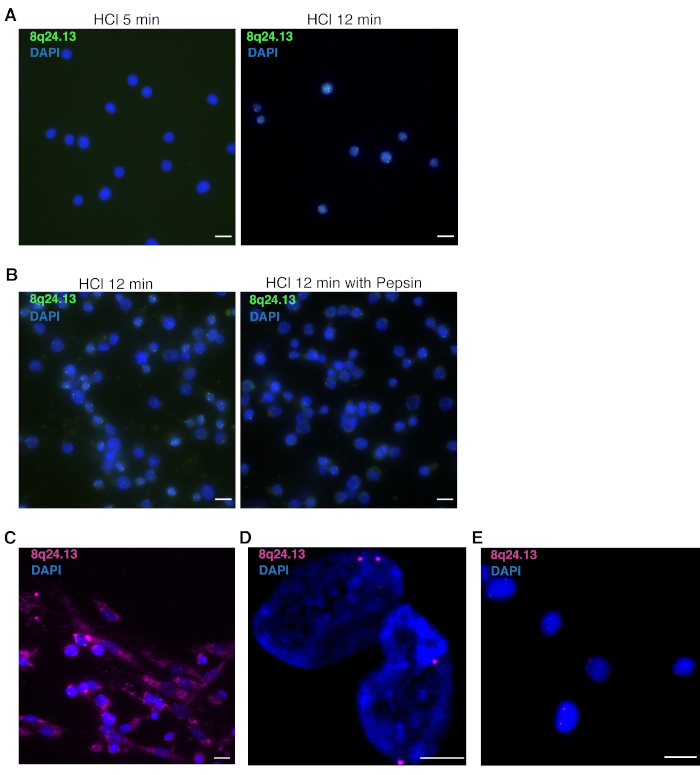
Figure 4 : Résultats possibles de la déprotéinisation sous-optimale et des étapes de pepsinisation sur les résultats 3D multicolor DNA FISH. (A) Représentant 3D multicolor DNA FISH noyaux de lymphocytes T primaires humains traités pendant 5 min (gauche) ou 12 min (à droite) avec 0,1 N HCl, en utilisant la cartographie de la sonde à 8q24.13 région (vert). Les noyaux sont contrecarrés avec DAPI (bleu). 100x grossissement. Barre d'échelle de 10 m. (B) Les noyaux 3D multicolors d'ADN FISH de lymphocytes T primaires humains traités pendant 12 min avec 0,1 N HCl (à gauche) ou couplés à 0,01 N HCl/0,0025% de pepsine pendant 2 min (droite), à l'aide de la cartographie de la sonde à 8q24.13 région (vert). Les noyaux sont contrecarrés avec DAPI (bleu). 100x grossissement. Barre d'échelle de 10 m. (C) Les noyaux 3D multicolors DE FISH d'ADN 3D des myoblastes primaires humains traités avec la pepsinisation courte et sous-optimale, utilisant la cartographie de sonde à la région 8q24.13 (magenta). Les noyaux sont contrecarrés avec DAPI (bleu). 63x grossissement. Barre d'échelle de 10 m. (D) Les noyaux 3D multicolors d'ADN FISH de myoblastes primaires humains traités avec l'étape prolongée de pepsinisation, utilisant la cartographie de sonde à la région 8q24.13 (magenta). Les noyaux sont contrecarrés avec DAPI (bleu). 63x grossissement. Barre d'échelle de 5 m. (E) Les noyaux 3D multicolors DE FISH de l'ADN humain des myoblastes primaires traités avec des conditions optimales de HCl/pepsin utilisant la cartographie de sonde à la région 8q24.13 (magenta). Les noyaux sont contrecarrés avec DAPI (bleu). 63x grossissement. Barre d'échelle de 25 m. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
| Nick réactifs de traduction | Concentration initiale | Concentration finale |
| dNTPs (C-G-A) | 0,5 mm | 0,05 mm |
| dTTP (en) | 0,1 mm | 0,01 mM |
| Biotine/Dig/Cy3 dUTP | 1 mM | 0,02 mm |
| Tris HCl pH 7,8 | 1 M | 50 mM |
| MgCl2 | 100 mm | 5 mM |
| -mercaptoéthanol | 100 mm | 10 mM |
| Bsa | 100 ng/L | 10 ng/L |
| ADN Pol I | 10 U/L | 0,1 U/L |
| DNase I | 1 U/L | 0,002 U/L |
| ADN 2 g | X | X |
| ddH2O | Jusqu'à 50 l |
Tableau 1 : Traduction de Nick. Tableau décrivant tous les réactifs, leur concentration et le moment suggéré pour la réaction de traduction de pseudo.
Discussion
La méthode actuelle décrit un protocole étape par étape pour effectuer 3D MULTIcolor DNA FISH sur un large éventail de cellules primaires humaines. Bien que l'ADN FISH soit une technologie largement utilisée, l'ADN 3D multicolore FISH sur les noyaux interphases 3D conservés est encore difficile à réaliser dans de nombreux laboratoires, principalement en raison des caractéristiques des échantillons utilisés23,24.
La traduction de pseudo de sonde est une étape fondamentale pour le FISH d'ADN multicolore 3D réussi ; de nombreux substrats différents (BAC, fosmid, plasmide, PCR) peuvent être utilisés pour cette réaction, et le moment de la réaction et la concentration enzymatique peuvent être ajustés en conséquence en fonction de la longueur du substrat. Une bonne digestion des sondes est fondamentale (figure 1), car les sondes non optimales (figure 2) n'entraîneront aucun signal ou un signal non spécifique (figure 3A). Perméabilisation, déprotéinisation, et les étapes de pepsinisation sont des passages cruciaux qui dépendent fortement du type de cellule utilisé. Les cellules avec de petits noyaux et une faible abondance de cytosquelette, telles que les lymphocytes T isolés ex vivo, nécessitent une déprotéinisation avec un traitement prolongé de 0,1 N HCl. En outre, les lavages dans PBS avec des pourcentages plus élevés de Triton X-100 peuvent aider l'entrée de la sonde dans les noyaux de ces cellules. Au contraire, les myoblastes primaires humains cultivés in vitro qui présentent de plus grands noyaux, avec une teneur élevée en cytosquelette, ont besoin de digestion des structures cytosoliques avec de la pepsine. Ces rôles généraux peuvent être appliqués à un large éventail de cellules, éventuellement en combinant les différentes étapes en fonction des caractéristiques cellulaires spécifiques.
L'utilisation de matériel biologique fraîchement préparé, de solutions fraîches (en particulier de solutions avec détergent) et de réactifs fluorescents sont fortement suggérées : PFA filtrée au pH 7.0 ; autoclaved et filtré 20x SSC au pH 7.0; formamide filtré au pH 7.0; nucléase sans eau; aliquots jetables d'UTP modifié. Une incubation prolongée avec 20 % de glycérol/PBS, ou 50 % de formamide/2x SSC peut faciliter l'hybridation. Le traitement au HCl et/ou à la pepsine peut être encore augmenté. Le moment de l'hybridation, la quantité de sondes, la concentration et le moment de l'incubation de l'anti-digoxinine et de la streptavidine peuvent tous être ajustés pour améliorer le rapport signal/bruit.
3D multicolor DNA FISH représente un outil complémentaire aux technologies C, la méthode standard pour valider les résultats basés sur C. S'il est combiné avec la microscopie et l'analyse 3D, l'ADN multicolor 3D FISH peut surveiller la proximité entre les loci génomiques et leur distribution topologique dans l'espace nucléaire au niveau d'une seule cellule. 3D multicolor DNA FISH peut être intégré à d'autres méthodologies telles que l'ARN FISH et l'immunofluorescence pour un aperçu complet de la dynamique et des interactions entre les loci génomiques, les ARN (ARN messager ou ARN régulateur non codant) et un large éventail des protéines, offrant une occasion unique de visualiser la structure nucléaire et d'étudier les mécanismes épigénétiques qui sous-tendent l'identité cellulaire.
Malgré l'énorme amélioration des technologies FISH avec super résolution25,26, imagerie cellulaire en direct27,28,36, détection de molécules uniques37, et la visualisation contemporaine de cibles multiples avec des tableaux oligonucléotides tels que Oligopaint37,38 avec 3D approches à haut débit39, une limitation de la technologie reste le nombre discret de loci génomique prédéterminé qui peut être visualisé. Cela empêche une analyse de grande envergure de l'architecture nucléaire. Plusieurs études ont récemment décrit des méthodes séquentielles d'hybridation pour aborder l'organisation du génome dans des cellules uniques telles que le code-barres DNA FISH40,41,42,43. D'autres efforts seront nécessaires pour coupler la nature cellulaire unique de l'ADN multicolor e 3D FISH à des caractéristiques génomiques larges pour visualiser largement l'hétérogénéité de l'architecture nucléaire avec les technologies d'imagerie, car le nombre de loci qui peuvent être testés à la fois augmentera.
Déclarations de divulgation
Les auteurs n'ont rien à révéler.
Remerciements
Les auteurs reconnaissent l'assistance technique de l'INSTALLATION d'imagerie INGM (Istituto Nazionale di Genetica Molecolare "Romeo ed Enrica Invernizzi" (INGM), Milan, Italie), en particulier C. Cordiglieri, pour l'assistance lors des images 3D multicolor DNA FISH Achat. Ce travail a été soutenu par les subventions suivantes à B.B.: EPIGEN programme phare italien, Association Française contre les Myopathies (AFM-Téléthon, subvention nr 18754) et Giovani Ricercatori, ministère italien de la Santé (GR-2011-02349383). Ce travail a été soutenu par la subvention suivante à F.M.: Fondazione Cariplo (Bando Giovani, subvention nr 2018-0321).
matériels
| Name | Company | Catalog Number | Comments |
| 24-well plates | Thermo Fisher Scientific | 142475 | |
| 6-well plates | Thermo Fisher Scientific | 140675 | |
| Anti-Digoxigenin 488 | DBA | DI7488 | |
| b-Mercaptoethanol | Sigma | M3148 | |
| bFGF | PeproTech | 100-18B | |
| Biotin 11 d-UTP | Thermo Fisher Scientific | R0081 | |
| BSA (bovine serum albumine) | Sigma | A7030 | |
| Coverlsips | Marienfeld | 117500 | |
| CY3 d-UTP | GE Healthcare | PA53022 | |
| DAPI (4,6-diamidino-2-phenylindole) | Thermo Fisher Scientific | D21490 | |
| Deoxyribonucleic acids single strand from salmon testes | Sigma | D7656 | |
| Dextran sulfate (powder) | Santa Cruz | sc-203917A | |
| Digoxigenin 11 d-UTP | Roche | 11093088910 | |
| DMEM | Thermo Fisher Scientific | 21969-035 500mL | |
| DNA polymerase I | Thermo Fisher Scientific | 18010-017 | |
| DNase I | Sigma | AMPD1 | |
| dNTPs (C-G-A-T) | Euroclone | BL0423A/C/G | |
| EGF | Sigma | E9644.2MG | |
| Ethanol | Sigma | 02860-1L | |
| FBS Hyclone | Thermo Fisher Scientific | SH30109 | |
| Formaldehyde solution | Sigma | F8775-25mL | |
| Formamide | Sigma | F9037 | |
| Glutammine | Thermo Fisher Scientific | 25030-024 100mL | |
| Glycerol | Sigma | G5516-100mL | |
| Glycogen | Thermo Fisher Scientific | AM9510 | |
| HCl | Sigma | 30721 | |
| Human Cot-1 DNA | Thermo Fisher Scientific | 15279-001 | |
| Insulin Human | Sigma | I9278-5 mL | |
| MgCl2 | Sigma | 63069 | |
| NaAc (Sodium Acetate, pH 5.2, 3 M) | Sigma | S2889 | |
| NaCl | Sigma | S9888 | |
| Paraformaldehyde | Sigma | 158127-25G | |
| PBS (phosphate-buffered saline) | Sigma | P4417 | |
| Pennycillin/Streptavidin | Thermo Fisher Scientific | 15070-063 100mL | |
| Pepsin | Biorad | P6887 | |
| PhasePrep BAC DNA Kit | Sigma | NA0100-1KT | |
| Poly-L-lysine solution | Sigma | P8920 | |
| ProLong Diamond Antifade Mountant | Thermo Fisher Scientific | P36970 | |
| PureLink Quick Gel Extraction & PCR Purification Combo Kit | Thermo Fisher Scientific | K220001 | |
| PureLink Quick Plasmid Miniprep Kit | Thermo Fisher Scientific | K210010 | |
| RNAse cocktail | Thermo Fisher Scientific | AM2288 | |
| Rubbercement | Bostik | ||
| Slides | VWR | 631-0114 | |
| Streptavidina Alexa fluor 647 | Thermo Fisher Scientific | S21374 | |
| Tri-Sodium Citrate | Sigma | 1110379026 | |
| Tris-HCl | Sigma | T3253-500g | |
| Triton X-100 | Sigma | T8787-250mL | |
| TWEEN 20 | Sigma | P9416-100mL |
Références
- Cremer, T., Cremer, C. Chromosome territories, nuclear architecture and gene regulation in mammalian cells. Nature Reviews: Genetics. 2 (4), 292-301 (2001).
- Lieberman-Aiden, E., et al. Comprehensive mapping of long-range interactions reveals folding principles of the human genome. Science. 326 (5950), 289-293 (2009).
- Schmitt, A. D., Hu, M., Ren, B. Genome-wide mapping and analysis of chromosome architecture. Nature Reviews: Molecular Cell Biology. 17 (12), 743-755 (2016).
- Bonev, B., Cavalli, G. Organization and function of the 3D genome. Nature Reviews: Genetics. 17 (12), 772 (2016).
- van Steensel, B., Furlong, E. E. M. The role of transcription in shaping the spatial organization of the genome. Nature Reviews: Molecular Cell Biology. 20 (6), 327-337 (2019).
- Rajarajan, P., Gil, S. E., Brennand, K. J., Akbarian, S. Spatial genome organization and cognition. Nature Reviews: Neuroscience. 17 (11), 681-691 (2016).
- Pombo, A., Dillon, N. Three-dimensional genome architecture: players and mechanisms. Nature Reviews: Molecular Cell Biology. 16 (4), 245-257 (2015).
- Fanucchi, S., Shibayama, Y., Burd, S., Weinberg, M. S., Mhlanga, M. M. Chromosomal contact permits transcription between coregulated genes. Cell. 155 (3), 606-620 (2013).
- Therizols, P., et al. Chromatin decondensation is sufficient to alter nuclear organization in embryonic stem cells. Science. 346 (6214), 1238-1242 (2014).
- Gonzalez-Sandoval, A., et al. Perinuclear Anchoring of H3K9-Methylated Chromatin Stabilizes Induced Cell Fate in C. elegans Embryos. Cell. 163 (6), 1333-1347 (2015).
- Hubner, B., et al. Remodeling of nuclear landscapes during human myelopoietic cell differentiation maintains co-aligned active and inactive nuclear compartments. Epigenetics Chromatin. 8, 47 (2015).
- Aymard, F., et al. Genome-wide mapping of long-range contacts unveils clustering of DNA double-strand breaks at damaged active genes. Nature Structural & Molecular Biology. 24 (4), 353-361 (2017).
- Sellou, H., et al. The poly(ADP-ribose)-dependent chromatin remodeler Alc1 induces local chromatin relaxation upon DNA damage. Molecular Biology of the Cell. 27 (24), 3791-3799 (2016).
- Krijger, P. H., de Laat, W. Regulation of disease-associated gene expression in the 3D genome. Nature Reviews: Molecular Cell Biology. 17 (12), 771-782 (2016).
- Lupianez, D. G., Spielmann, M., Mundlos, S. Breaking TADs: How Alterations of Chromatin Domains Result in Disease. Trends in Genetics. 32 (4), 225-237 (2016).
- Cortesi, A., et al. 4q-D4Z4 chromatin architecture regulates the transcription of muscle atrophic genes in facioscapulohumeral muscular dystrophy. Genome Research. 29 (6), 883-895 (2019).
- Woltering, J. M., Noordermeer, D., Leleu, M., Duboule, D. Conservation and divergence of regulatory strategies at Hox Loci and the origin of tetrapod digits. PLoS Biology. 12 (1), 1001773 (2014).
- Woltering, J. M., Duboule, D. Tetrapod axial evolution and developmental constraints; Empirical underpinning by a mouse model. Mechanisms of Development. 138, 64-72 (2015).
- de Laat, W., Dekker, J. 3C-based technologies to study the shape of the genome. Methods. 58 (3), 189-191 (2012).
- Denker, A., de Laat, W. The second decade of 3C technologies: detailed insights into nuclear organization. Genes & Development. 30 (12), 1357-1382 (2016).
- Finn, E. H., et al. Extensive Heterogeneity and Intrinsic Variation in Spatial Genome Organization. Cell. 176 (6), 1502-1515 (2019).
- Zheng, M., et al. Multiplex chromatin interactions with single-molecule precision. Nature. 566 (7745), 558-562 (2019).
- Solovei, I. Fluorescence in situ hybridization (FISH) on tissue cryosections. Methods in Molecular Biology. 659, 71-82 (2010).
- Cremer, M., et al. Multicolor 3D fluorescence in situ hybridization for imaging interphase chromosomes. Methods in Molecular Biology. 463, 205-239 (2008).
- Legant, W. R., et al. High-density three-dimensional localization microscopy across large volumes. Nature Methods. 13 (4), 359-365 (2016).
- Jungmann, R., et al. Quantitative super-resolution imaging with qPAINT. Nature Methods. 13 (5), 439-442 (2016).
- Chen, B., et al. Dynamic imaging of genomic loci in living human cells by an optimized CRISPR/Cas system. Cell. 155 (7), 1479-1491 (2013).
- Nelles, D. A., et al. Programmable RNA Tracking in Live Cells with CRISPR/Cas9. Cell. 165 (2), 488-496 (2016).
- Byron, M., Hall, L. L., Lawrence, J. B. A multifaceted FISH approach to study endogenous RNAs and DNAs in native nuclear and cell structures. Current Protocol of Human Genetics. , 15 (2013).
- Chaumeil, J., Augui, S., Chow, J. C., Heard, E. Combined immunofluorescence, RNA fluorescent in situ hybridization, and DNA fluorescent in situ hybridization to study chromatin changes, transcriptional activity, nuclear organization, and X-chromosome inactivation. Methods in Molecular Biology. 463, 297-308 (2008).
- Takizawa, T., Gudla, P. R., Guo, L., Lockett, S., Misteli, T. Allele-specific nuclear positioning of the monoallelically expressed astrocyte marker GFAP. Genes & Development. 22 (4), 489-498 (2008).
- Kraus, F., et al. Quantitative 3D structured illumination microscopy of nuclear structures. Nature Protocols. 12 (5), 1011-1028 (2017).
- Solovei, I., Cremer, M. 3D-FISH on cultured cells combined with immunostaining. Methods in Molecular Biology. 659, 117-126 (2010).
- Skinner, B. M., Johnson, E. E. Nuclear morphologies: their diversity and functional relevance. Chromosoma. 126 (2), 195-212 (2017).
- Schoenfelder, S., et al. The pluripotent regulatory circuitry connecting promoters to their long-range interacting elements. Genome Research. 25 (4), 582-597 (2015).
- Lubeck, E., Cai, L. Single-cell systems biology by super-resolution imaging and combinatorial labeling. Nature Methods. 9 (7), 743-748 (2012).
- Beliveau, B. J., et al. Single-molecule super-resolution imaging of chromosomes and in situ haplotype visualization using Oligopaint FISH probes. Nature Communication. 6, 7147 (2015).
- Beliveau, B. J., et al. Versatile design and synthesis platform for visualizing genomes with Oligopaint FISH probes. Proceedings of the National Academy of Sciences. 109 (52), 21301-21306 (2012).
- Samacoits, A., et al. A computational framework to study sub-cellular RNA localization. Nature Communication. 9 (1), 4584 (2018).
- Cardozo Gizzi, A. M., et al. Microscopy-Based Chromosome Conformation Capture Enables Simultaneous Visualization of Genome Organization and Transcription in Intact Organisms. Molecular Cell. 74 (1), 212-222 (2019).
- Mateo, L. J., et al. Visualizing DNA folding and RNA in embryos at single-cell resolution. Nature. 568 (7750), 49-54 (2019).
- Nir, G., et al. Walking along chromosomes with super-resolution imaging, contact maps, and integrative modeling. PLoS Genetics. 14 (12), 1007872 (2018).
- Bintu, B., et al. Super-resolution chromatin tracing reveals domains and cooperative interactions in single cells. Science. 362 (6413), (2018).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon